DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2015
VALIDASI
Single Nucleotide Polymorphisms
(SNP) BERBASIS
GENOM DAN PEMANFAATANNYA UNTUK UJI
KEKERABATAN 50 AKSESI KEDELAI
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini Saya menyatakan bahwa skripsi berjudul Validasi Single Nucleotide Polymorphisms (SNP) Berbasis Genom dan Pemanfaatannya untuk Uji Kekerabatan 50 Aksesi Kedelai adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor dan Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian.
ABSTRAK
ANDHIKA FAISAL MUBAROK. Validasi Single Nucleotide Polymorphisms (SNP) Berbasis Genom dan Pemanfaatannya untuk Uji Kekerabatan 50 Aksesi Kedelai. Dibimbing oleh I MADE ARTIKA dan I MADE TASMA.
Single Nucleotide Polymorphisms (SNP) sudah terdeteksi dalam genom kedelai varietas Anjasmoro, Tanggamus dan Wilis melalui sekuensing menggunakan Next Generation Sequencing. Sekuen genom tersebut dijajarkan dengan sekuen genom rujukan kedelai varietas William 82. SNP tersebut masih berpeluang sebagai false SNP. Tujuan dalam penelitian ini memvalidasi SNP hasil penjajaran sekuen genom kedelai varietas Anjasmoro, Tanggamus, dan Wilis dengan genom rujukan William 82 menggunakan Single Nucleotide Amplified Polymorphisms (SNAP) dan memanfaatkannya untuk menguji kekerabatan 50 aksesi kedelai. Sebanyak 19 pasang primer SNAP dirancang menggunakan program SNAPER untuk membuktikan keberadaan SNP pada 19 posisi pilihan dalam genom. Hasil dari penelitian ini bahwa sembilan SNP valid keberadaannya dalam genom dari 19 SNP yang diuji. SNP tervalidasi tersebut belum mampu untuk membedakan secara genetik beberapa aksesi dari 50 aksesi kedelai sehingga kekerabatan antara 50 aksesi kedelai sangat tinggi dengan koefisien jarak genetik 0 sampai 0.3.
Kata kunci: Aksesi, genom, kedelai, SNP, SNAP
ABSTRACT
ANDHIKA FAISAL MUBAROK. Single Nucleotide Polymorphisms (SNP) Validation Based on Genomic and Its Utilization for Genetic Relationship Test of 50 Soybean Accessions. Supervised by I MADE ARTIKA dan I MADE TASMA. Single Nucleotide Polymorphisms (SNPs) have been detected in Anjasmoro, Tanggamus and Wilis soybean variety genomes through sequencing using Next Generation Sequencing. The genomic sequences obtained were aligned with the soybean reference genomic sequence, William 82. Identified SNPs from the NGS still have possibility to be false SNPs. The purposes of this research was to validate SNPs from the alignment of Anjasmoro, Tanggamus, and Wilis sequences with the reference genomic sequence, William 82 using Single Nucleotide Amplified Polymorphisms (SNAP) method and apply it to analyze the genetic relationship of 50 soybean accessions. There were 19 pairs of SNAP primers were designed using SNAPER program to prove SNP existence in 19 selected positions in genome. Result of this study were 9 valid SNPs out of 19 SNPs in genomic sequences analyzed. Those validated SNPs were not capable yet to differentiate genetically some accessions from 50 soybean accessions, so the linkage values between 50 soybean accessions were relatively high with genetic distance coefficient 0 to 0.3.
Skripsi
sebagai salah satu syarat memperoleh gelar Sarjana Sains pada
Departemen Biokimia
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
BOGOR 2015
VALIDASI
Single Nucleotide Polymorphisms
(SNP) BERBASIS
GENOM DAN PEMANFAATANNYA UNTUK UJI
KEKERABATAN 50 AKSESI KEDELAI
Judul : Validasi Single Nucleotide Polymorphisms (SNP) Berbasis Genom dan Pemanfaatannya untuk Uji Kekerabatan 50 Aksesi Kedelai
Nama : Andhika Faisal Mubarok NIM : G84110012
Disetujui oleh
Dr Ir I Made Artika, MAppSc Dr Ir I Made Tasma, MSc
Pembimbing I Pembimbing II
Diketahui oleh
Dr Ir I Made Artika, MAppSc Ketua Departemen
PRAKATA
Puji syukur kepada Allah SWT atas rahmat dan karunia-Nya sehingga penulis dapat menyelesaikan penelitian yang berjudul Validasi Single Nucleotide Polymorphisms (SNP) Berbasis Genom dan Pemanfaatannya untuk Uji Kekerabatan 50 Aksesi Kedelai. Penelitian ini dilaksanakan pada bulan Desember hingga April 2015 di Laboratorium Biologi Molekuler, Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian (BB Biogen).
Penulis menyampaikan terima kasih kepada Bapak Dr Ir I Made Artika MAppSc selaku pembimbing utama dan Bapak Dr Ir I Made Tasma MSc selaku pembimbing kedua atas bimbingan dan pengarahan yang diberikan berikut kritik dan sarannya dalam penyusunan skripsi ini. Penulis juga mengucapkan terima kasih kepada Ibu Dr Puji Lestari SP MSi, para peneliti, staf, serta rekan sesama mahasiswa penelitian di Laboratorium Biologi Molekuler, BB Biogen. Ucapan terima kasih juga tak lupa penulis sampaikan kepada kedua orang tua atas dukungan yang selalu diberikan. Penulis menyadari masih banyak kekurangan dalam penyusunan usulan penelitian ini, maka diharapkan kritik dan saran yang membangun untuk perbaikan dalam penulisan selanjutnya.
DAFTAR ISI
DAFTAR TABEL viii
DAFTAR GAMBAR viii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
METODE 2
Bahan dan Alat 2
Prosedur Penelitian 2
HASIL 5
PEMBAHASAN 16
SIMPULAN DAN SARAN 22
Simpulan 22
Saran 23
DAFTAR PUSTAKA 23
LAMPIRAN 27
DAFTAR TABEL
1 Kuantitas dan kemurnian DNA genom kedelai 7
2 Posisi dan SNP target 19 pasang primer SNAP 8
3 Perbandingan hasil PCR observasi berdasarkan 19 pasang primer SNAP
dengan hasil PCR yang diharapkan 9
4 SNP pada 50 aksesi kedelai berdasarkan kinerja 12 pasang primer SNAP dengan rujukan SNP pada varietas William 82, Wilis, Tanggamus dan
Anjasmoro dari database di IAARD GC 12
5 Tingkat heterozigositas dan polimorfisme sembilan marka SNAP melalui
50 aksesi kedelai 14
6 Matriks rerata jarak genetik antar daerah asal 50 aksesi kedelai 16
DAFTAR GAMBAR
1 Elektroforegram DNA genom tiga varietas kedelai 6
2 Elektroforegram DNA genom 50 aksesi kedelai 6
3 Elektroforegram hasil PCR menggunakan primer SOY-SNPR1 sampai
SOY-SNPR19 dengan 28 siklus PCR 10
4 Elektroforegram hasil PCR menggunakan primer SNPR4, SOY-SNPR8, SOY-SNPR10, SOY-SNPR17, dan SOY-SNPR19 dengan 38
siklus PCR 11
5 Elektroforegram hasil PCR menggunakan primer SOY-SNPR16 11 6 Dendogram 50 aksesi kedelai berdasarkan sembilan marka SNAP 15
DAFTAR LAMPIRAN
1 Diagram alir penelitian 27
2 Tanaman kedelai yang digunakan dalam penelitian 28 3 Sekuen dan Melting Temperature (Tm) 19 pasang primer SNAP 30
4 Ukuran primer SNAP dan amplikon 31
5 Tingkat validitas SNP 32
6 Tampilan program SNAPER 33
PENDAHULUAN
Kedelai (Glycine max (L.) Merr.) merupakan komoditas pangan terpenting di Indonesia setelah komoditas padi dan jagung (Supadi 2009). Budidaya kedelai di Indonesia masih mengalami berbagai permasalahan baik dari segi kualitas dan kuantitas. Salah satu permasalahannya adalah produktivitas yang masih rendah sehingga terjadi impor kedelai. Tahun 2010 sampai 2014, kebutuhan kedelai setiap tahun sekitar 2300000 ton biji kering, sedangkan produksi nasional hanya sekitar 807.57 ton (Eka et al. 2015). Peningkatan kuantitas dan kualitas kedelai lokal masih terus diusahakan, salah satu caranya adalah pemuliaan tanaman.
Marka molekuler sangat membantu para pemulia salah satunya untuk kegiatan seleksi. Seleksi menggunakan marka molekuler jauh lebih akurat dibandingkan melalui pengamatan fenotipe karena tidak dipengaruhi oleh lingkungan (Nuraida 2012). Single Nucleotide Polymorphisms (SNP) merupakan marka molekuler yang sudah banyak dikembangkan saat ini karena mempunyai kelebihan dibandingkan marka molukuler lain. Marka tersebut keberadaannya sangat berlimpah, mudah untuk diskoring dan laju mutasinya rendah (Murani et al. 2005). SNP merupakan nukleotida tunggal di lokasi spesifik dalam genom yang mengalami variasi diantara individu dalam populasi yang sama (Jehan dan Lakhanpaul 2006). Menurut Kumar et al. (2012), SNP dapat dengan cepat dan relatif murah untuk ditemukan setelah adanya Next Generation Sequencing (NGS). Hasil sekuensing dijajarkan dengan sekuen rujukan sehingga dapat dideteksi adanya SNP.
Menurut You et al. (2011), SNP yang ditemukan dari sekuensing menggunakan NGS harus divalidasi. Faktor yang mendukung bahwa validasi sangat dibutuhkan adalah adanya potensi kesalahan pada sekuensing atau kesalahan pada penjajaran sekuen genom. Validasi dapat menggunakan beberapa cara seperti Illumina Goldengate dan KBiosciences Competitive Allele Specific-PCR SNP genotyping system (KASPar) (Kumar et al. 2012). Salah satu cara validasi SNP yang saat ini sedang dikembangkan pada berbagai organisme karena mudah digunakan dan biayanya relatif murah adalah Single Nucleotide Amplified Polymorphisms (SNAP). SNAP berupa primer yang dirancang pada ujung 3 OH spesifik terhadap salah satu dari dua nukleotida yang merupakan SNP. Primer tersebut juga ditambahkan modifikasi pada salah satu dari tiga nukleotida dekat ujung 3 OH sehingga tidak komplemen terhadap DNA cetakan (Lestari dan Koh 2013).
validasi SNP menggunakan marka SNAP yang murah dan mudah serta memanfaatkanya melalui aksesi kedelai. Pemanfaatan SNP salah satunya dapat digunakan untuk membedakan secara genetik antar individu (Montes et al. 2014).
Tujuan penelitian ini adalah memvalidasi SNP hasil sekuensing genom menggunakan SNAP dan memanfaatkannya untuk menguji kekerabatan 50 aksesi kedelai. Hipotesis penelitian ini adalah marka SNAP berhasil membuktikan adanya SNP dengan tingkat validitas tinggi dan SNP tersebut dapat membedakan 50 aksesi kedelai dengan jarak genetik tinggi. Manfaat penelitian ini adalah memberikan alternatif pemilihan marka molekuler, yaitu marka SNAP yang lebih efisien dan efektif untuk kegiatan pemuliaan tanaman agar lebih cepat menghasilkan jenis kedelai unggul Indonesia. Penelitian dilakukan di Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian.
METODE
Bahan dan Alat
Bahan-bahan yang digunakan adalah 50 benih aksesi kedelai dan 3 benih varietas kedelai yang merupakan koleksi BB Biogen (Lampiran 2), nitrogen cair, larutan cetyltrimethylammonium bromide (CTAB) 2%, polivinilpirolidon (PVP)
2%, β-merkaptoetanol 0.2%, natrium bisulfit 0.38 %, natrium asetat (NaOAc) 3M, kloroform:isoamilalkohol (chisam) 24:1, isopropanol dingin, etanol 70% dingin, bufer Tris-EDTA (TE) 1× pH 8.0, RNAase, KAPA2G Fast ReadyMix PCR Kit yang terdiri atas KAPA2G Fast DNA Polymerase (0.5 U per 25 µL reaksi) dalam bufer reaksi (dNTPs 0.2 mM untuk 1 × reaksi, MgCl2 1.5 mM untuk 1 × reaksi,
stabilizer dan tracking dye 2x), primer SNAP forward dan reverse masing-masing berjumlah 19, Double Distilled Water (ddH2O), gel agarosa, bufer Tris Asetat
EDTA (TAE) 1× pH 8.0, Blue JuiceTM Gel Loading Buffer (4×), SYBR® Gold Nucleic Acid Gel Stain (100 × in TE) dan TrackltTM 1 Kb Plus DNA Ladder Invitrogen.
Alat-alat yang digunakan adalah media semai, penangas air, inkubator thermostat, mikropipet, spin down, tip pipet, autoklaf, parafilm, blue sterile pastle, tabung eppendorf 1.5 mL, tabung 2 mL, labu Erlenmeyer, gelas ukur, 96-well PCR plate, sentrifus 5810R eppendorf, batang magnet, mesin pendingin, Vortex, sarung tangan karet, neraca analitik, sudip, thermal cycler biometra T1, spektrofotometer nanodrop thermoscientific 2000c, ice maker, microwave, perangkat elektroforesis dan UV transilluminator High Performance 2UV.
Prosedur Penelitian
Penanaman dan Pengambilan Sampel (Santoso et al. 2006)
mL dengan jumlah maksimal 10 helai daun. Daun kedelai dengan karakteristik yang sesuai dan masih terdapat di tanaman diambil dan dimasukkan ke dalam plastik untuk cadangan. Tabung 2 mL dan plastik yang berisi daun kedelai disimpan di dalam mesin pendingin -50 oC.
Isolasi DNA Menggunakan Modifikasi Metode Doyle & Doyle (1987)
Daun yang berada di tabung 2 mL digerus sampai halus menggunakan blue sterile pastle yang sebelumnya tabung dan pastle telah dibasuh dengan nitrogen cair. Bufer ekstraksi (CTAB 2%, 0.2 % β-merkaptoetanol, 2% PVP dan 0.38% natrium bisulfit) yang telah dipanaskan sebelumnya pada suhu 60 oC ditambahkan sebanyak 750 µL pada daun yang telah halus. Sampel diinkubasi pada suhu 65 oC selama 60 menit dan secara berkala dilakukan pencampuran. Sebanyak 750 µL kloroform isoamilalkohol ditambahkan ke dalam sampel dan dicampurkan dengan cara dibolak-balik secara perlahan selama 5 menit. Campuran tersebut kemudian disentrifus selama 15 menit pada 12000 rpm (20 oC) kemudian lapisan atas hasil sentrifus dipindahkan ke dalam tabung 1.5 mL baru. Sebanyak (1/10×volume supernatan) Na asetat 3M ditambahkan ke dalam supernatan dan dicampurkan dengan cara dibolak-balik secara perlahan, kamudian sebanyak (1×volume supernatan) isopropanol dingin ditambahkan ke dalam supernatan. Selanjutnya, sampel diinkubasi pada suhu -20 oC selama 1 jam, kemudian dibolak-balik sebentar dan diinkubasi kembali pada -20 oC selama 15 menit.
Selanjutnya, sampel disentrifus selama 20 menit pada 12000 rpm (10 oC). Supernatan yang dihasilkan dibuang dan pelet yang dihasilkan dibilas dengan 200 µL etanol 70 % dingin. Sampel disentrifus kembali pada 12000 rpm selama 5 menit dengan suhu 4 oC. Supernatan hasil sentrifus dibuang dan pelet dikeringkan pada udara terbuka selama satu malam. Endapan hasil pengeringan kemudian dilarutkan dengan 100 µL bufer TE pH 8.0 dan dipanaskan pada 65 oC hingga larut. Enzim RNAse kemudian ditambahkan sebanyak 2 µL dan kemudian diinkubasi kembali dengan suhu 37 oC selama 30 menit. Selanjutnya, DNA hasil isolasi diuji kualitas dan kuantitasnya.
Analisis Kualitas dan Kuantitas DNA
Analisis kualitatif DNA menggunakan modifikasi Sambrook dan Russel (2001). Uji kualitatif menggunakan elektroforesis gel agarosa sebesar 1.5 %. Gel agarosa dibuat dengan menambahkan agarosa sebanyak 0.9 g ke dalam 60 mL larutan TAE 1×. Campuran tersebut dipanaskan hingga larut, kemudian didinginkan hingga hangat dan dituang ke dalam cetakan gel. Gel yang sudah padat dipindahkan ke dalam bak elektroforesis yang berisi TAE 1×. Selanjutnya dibuat loading bufer dengan campuran blue juice 4× sebanyak 2 μL, SYBR Gold 100× sebanyak 0.5 µL dan ddH2O sebanyak 2.5 µL. Loading bufer tersebut
Uji kuantitatif DNA. Uji kuantitatif DNA dilakukan dengan menggunakan spektrofotometer nanodrop thermoscientific dengan blanko yang digunakan adalah larutan TE pH 8.0. Sebelum digunakan lubang optik dibersihkan terlebih dahulu dengan menggunakan nuclease water. Sebanyak 2 ul Larutan TE dimasukkan ke dalam lubang optik nanodrop thermoscientific dan kemudian dipilih menu measure blank pada komputer. Kemudian, lubang optik dibersihkan kembali dan sampel DNA sebanyak 2 μL dimasukkan ke dalam
lubang optik. Setelah itu, hasil pengukuran akan muncul dalam konsentrasi ng/μL
dan kemurnian DNA dapat dilihat langsung dalam A260/A280 dan A260/A230.
Perancangan Primer (Drenkard et al. 2000)
Database SNP kedelai untuk varietas Wilis, Tanggamus dan Anjasmoro digunakan untuk perancangan primer yang didapatkan di situs iaardgc.or.id. Database tersebut berupa sekuen DNA yang mengandung SNP. Sekuen tersebut dijadikan bahan untuk desain primer menggunakan progam SNAPER yang dapat diakses di ausubellab.mgh.harvard.edu. Pilihan sekuen primer yang dapat digunakan akan diberikan oleh SNAPER melalui surat elektronik yang sebelumnya sudah didaftarkan. Salah satu pasangan primer dari sekian pilihan dipilih dan dikirim ke perusahaan pembuat primer, yaitu Genetica Science untuk disintesis sesuai sekuen primer yang mengapit SNP.
Validasi SNP (Lestari dan Koh 2013)
Validasi SNP pada genom varietas Tanggamus, Anjasmoro dan Wilis. DNA genom kedelai varietas Tanggamus, Wilis dan Anjasmoro diamplifikasi menggunakan primer SNAP yang berjumlah 19 pasang. PCR dilakukan dengan volume 10 µL dalam tabung PCR dengan komponen 40 ng/µ L DNA genom sebanyak 1 µL, 10 µM primer forward dan reverse masing-masing sebanyak 0.5 µL, 5 µL KAPA2G dan sebanyak 3 µL ddH2O. Setiap tabung PCR berisi
komponen reaksi PCR dimasukkan dalam mesin PCR dan reaksi PCR dilakukan dengan progam sebagai berikut: denaturasi awal pada suhu 94 oC selama 5 menit, amplifikasi DNA sebanyak 28 siklus atau 38 siklus (denaturasi pada suhu 94 oC selama 30 detik, annealing pada suhu 57 oC selama 1 menit) dan tahap terakhir adalah ekstensi pada suhu 72 oC selama 10 menit.
Hasil amplifikasi dipisahkan menggunakan elektroforesis gel agarosa 1.5 % dengan tegangan listrik 100 volt selama 1 jam. Hasil elektroforesis berupa ada dan tidaknya adanya pita DNA dibandingkan dengan hasil elektroforesis yang diharapkan berdasarkan rancangan primer. Hasil elektroforesis yang menunjukkan hasil yang sama dengan hasil yang diharapkan dinyatakan valid, sedangkan adanya perbedaan dengan hasil yang diharapkan dinyatakan tidak valid.
Validasi SNP pada genom 50 aksesi kedelai. DNA genom 50 aksesi kedelai diamplifikasi menggunakan primer yang sudah valid berdasarkan hasil amplifikasi DNA genomik Tanggamus, Anjasmoro dan Wilis. PCR dilakukan dengan volume 10 µL dalam tabung PCR dengan komponen 40 ng/µL DNA genom sebanyak 1 µL, 10 µM primer forward dan reverse masing-masing sebanyak 0.5 µL, 5 µL KAPA2G dan sebanyak 3 µL ddH2O. Setiap tabung PCR
suhu 94 oC selama 30 detik, annealing pada suhu 57 oC selama 1 menit) dan tahap terakhir adalah ekstensi pada suhu 72 oC selama 10 menit.
Hasil amplifikasi dipisahkan menggunakan elektroforesis gel agarosa 1.5 % dengan tegangan listrik 100 volt selama 1 jam. Selanjutnya hasil elektroforesis diamati dengan bantuan lampu UV. Setiap pita amplikon yang terbentuk atau tidak terbentuk amplikon dikonversi menjadi data alel SNP. Konversi tersebut disesuaikan dengan rancangan primer dan database SNP pada IARRD GC. Primer yang menunjukkan pola monomorfik alel SNP untuk 50 aksesi kedelai tidak dapat digunakan untuk uji kekerabatan. Tingkat validitas SNP dapat dihitung menggunakan rumus yang dikembangkan oleh Chagne et al. (2012), sebagai berikut:
Tingkat validitas = Jumlah SN Valid
Total SN yang Diuji x 100%
Uji Kekerabatan 50 Aksesi Kedelai Menggunakan SNP Tervalidasi
Analisis tingkat polimorfisme dan heterozigositas (Liu dan Muse 2005). Hasil elektroforesis amplikon 50 aksesi kedelai menggunakan primer yang sudah valid diskoring berdasarkan ada dan tidak adanya pita DNA. Nilai 1/1 untuk keberadaan pita dan 2/2 untuk ketidakberadaan pita. Hasil skoring disimpan dalam format txt. Format tersebut dimasukkan dalam program PowerMarker V3.25 melalui menu File – Import – Dataset. Analisis statistika berupa heterozigositas, diversitas gen (heterozigositas harapan) dan Polymorphisms Information Content (PIC) didapatkan melalui menu Analysis – Summary – Summary Statistics.
Jarak genetik antar daerah aksesi kedelai (Liu dan Muse 2005). PowerMarker juga digunakan untuk mendapatkan jarak genetik antar daerah asal aksesi kedelai. Menu yang digunakan adalah Analysis – Phylogeny – Frequency Based Distance.
Konstruksi filogenetik (Tamura et al. 2011). Hasil elektroforesis amplikon 50 aksesi kedelai menggunakan primer yang sudah valid dikonversi menjadi data alel SNP yang disimpan dalam format Fasta. Selanjutnya, data tersebut diolah dalam program MEGA5 yang sebelumnya diubah terlebih dahulu menjadi format MEGA. Pohon filogenetik didapatkan melalui menu Analysis – Phylogeny – Construct/Test Unweighted Pair Group Method with Arithmetic Mean (UPGMA) Tree.
HASIL
DNA Genom Kedelai
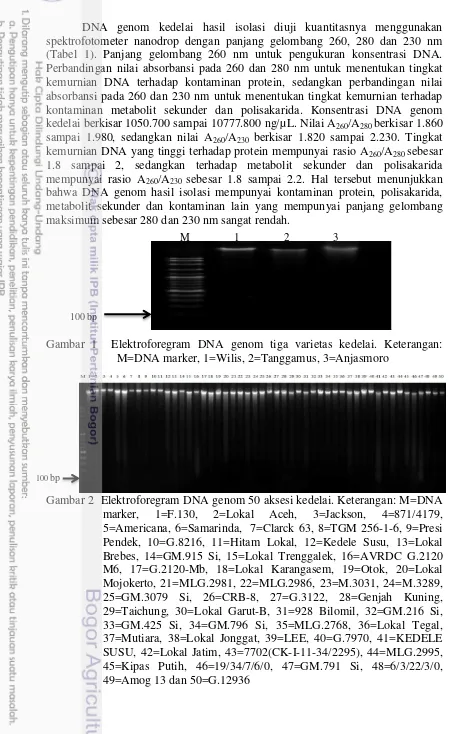
M 1 2 3
DNA genom kedelai hasil isolasi diuji kuantitasnya menggunakan spektrofotometer nanodrop dengan panjang gelombang 260, 280 dan 230 nm (Tabel 1). Panjang gelombang 260 nm untuk pengukuran konsentrasi DNA. Perbandingan nilai absorbansi pada 260 dan 280 nm untuk menentukan tingkat kemurnian DNA terhadap kontaminan protein, sedangkan perbandingan nilai absorbansi pada 260 dan 230 nm untuk menentukan tingkat kemurnian terhadap kontaminan metabolit sekunder dan polisakarida. Konsentrasi DNA genom kedelai berkisar 1050.700 sampai 10777.800 ng/µL. Nilai A260/A280 berkisar 1.860
sampai 1.980, sedangkan nilai A260/A230 berkisar 1.820 sampai 2.230. Tingkat
kemurnian DNA yang tinggi terhadap protein mempunyai rasio A260/A280 sebesar
1.8 sampai 2, sedangkan terhadap metabolit sekunder dan polisakarida mempunyai rasio A260/A230 sebesar 1.8 sampai 2.2. Hal tersebut menunjukkan
bahwa DNA genom hasil isolasi mempunyai kontaminan protein, polisakarida, metabolit sekunder dan kontaminan lain yang mempunyai panjang gelombang maksimum sebesar 280 dan 230 nm sangat rendah.
Gambar 1 Elektroforegram DNA genom tiga varietas kedelai. Keterangan: M=DNA marker, 1=Wilis, 2=Tanggamus, 3=Anjasmoro
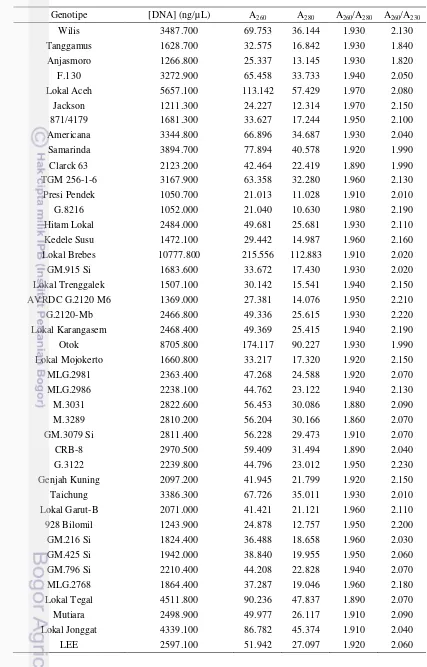
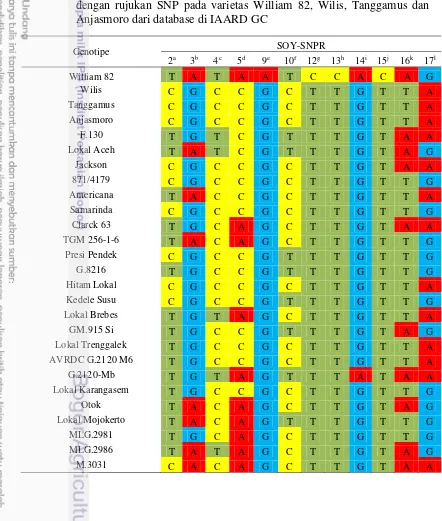
Gambar 2 Elektroforegram DNA genom 50 aksesi kedelai. Keterangan: M=DNA marker, 1=F.130, 2=Lokal Aceh, 3=Jackson, 4=871/4179, 5=Americana, 6=Samarinda, 7=Clarck 63, 8=TGM 256-1-6, 9=Presi Pendek, 10=G.8216, 11=Hitam Lokal, 12=Kedele Susu, 13=Lokal Brebes, 14=GM.915 Si, 15=Lokal Trenggalek, 16=AVRDC G.2120 M6, 17=G.2120-Mb, 18=Lokal Karangasem, 19=Otok, 20=Lokal Mojokerto, 21=MLG.2981, 22=MLG.2986, 23=M.3031, 24=M.3289, 25=GM.3079 Si, 26=CRB-8, 27=G.3122, 28=Genjah Kuning, 29=Taichung, 30=Lokal Garut-B, 31=928 Bilomil, 32=GM.216 Si, 33=GM.425 Si, 34=GM.796 Si, 35=MLG.2768, 36=Lokal Tegal, 37=Mutiara, 38=Lokal Jonggat, 39=LEE, 40=G.7970, 41=KEDELE SUSU, 42=Lokal Jatim, 43=7702(CK-I-11-34/2295), 44=MLG.2995, 45=Kipas Putih, 46=19/34/7/6/0, 47=GM.791 Si, 48=6/3/22/3/0, 49=Amog 13 dan 50=G.12936
100 bp
Tabel 1 Kuantitas dan kemurnian DNA genom kedelai (lanjutan)
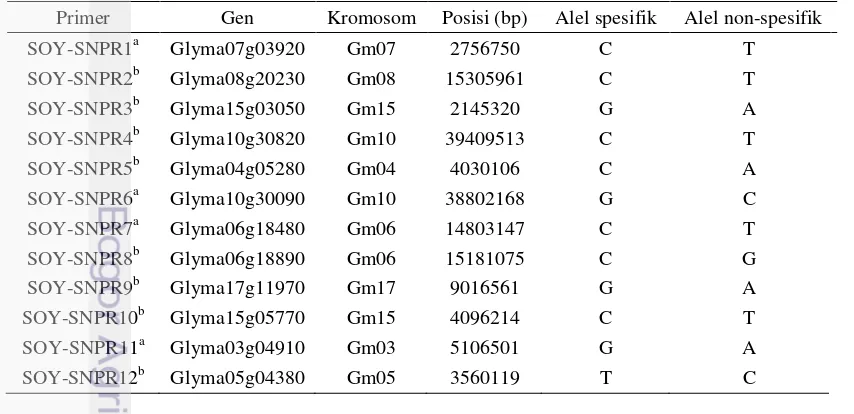
Genotipe [DNA] (ng/µL) A260 A280 A260/A280 A260/A230
G.7970 1802.700 36.054 18.291 1.970 2.060 KEDELE SUSU 4240.700 84.813 44.279 1.920 2.130 Lokal Jatim 4741.200 94.824 50.000 1.900 2.040 7702(CK-I-11-34/2295) 2100.700 42.014 21.791 1.930 2.000 MLG.2995 1821.300 36.426 18.627 1.960 2.170 Kipas Putih 1902.000 38.041 19.678 1.930 2.190 19/34/7/6/0 3588.500 71.770 37.782 1.900 2.020 GM.791 Si 2528.100 50.561 26.628 1.900 2.050 6/3/22/3/0 1953.700 39.074 20.040 1.950 2.110 Amog 13 3935.200 78.704 40.744 1.930 2.000 G.12936 2623.600 52.472 27.856 1.880 2.070
Primer SNAP
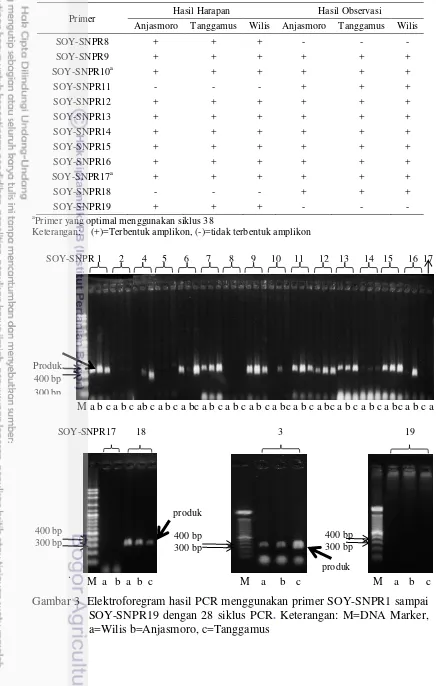
Sebanyak 19 pasang primer SNAP dirancang untuk mendeteksi keberadaan SNP pada 19 posisi yang berbeda dalam genom. Sebagai contoh, SOY-SNPR1 untuk mendeteksi SNP (C/T) pada gen Glyma07g03920, posisi 2756750 bp dalam kromosom nomor 7 (Tabel 2). SOY-SNPR1, SOY-SNPR6, SOY-SNPR7, SOY-SNPR11 dan SOY-SNPR18 dirancang spesifik terhadap alel yang dimiliki William 82 dan tidak spesifik terhadap alel yang dimiliki Anjasmoro, Tanggamus dan Wilis. Sebanyak 14 pasang primer lainnya dirancang spesifik terhadap alel yang dimiliki Anjasmoro, Tanggamus dan Wilis dan tidak spesifik terhadap alel yang dimiliki William 82. Primer SNAP yang digunakan mempunyai Melting Temperature (Tm) sebesar 56.992 sampai 63.305 oC (Lampiran 3). Panjang sekuen primer dan amplikon sekitar 22 sampai 36 bp dan 325 sampai 375 bp (Lampiran 4).
Tabel 2 Posisi dan SNP target 19 pasang primer SNAP
Primer Gen Kromosom Posisi (bp) Alel spesifik Alel non-spesifik
Tabel 2 Posisi dan SNP target 19 Primer SNAP (lanjutan)
Primer Gen Kromosom Posisi (bp) Alel spesifik Alel non-spesifik SOY-SNPR13b Glyma01g32160 Gm01 43606825 T C
Primer dirancang spesifik terhadap alel yang dimiliki varietas William 82 dan tidak spesifik terhadap alel yang dimiliki varietas Anjasmoro, Wilis dan Tanggamus. bPrimer dirancang spesifik terhadap alel yang dimiliki varietas Anjasmoro, Wilis dan Tanggamus dan tidak spesifik terhadap alel yang dimiliki varietas William 82.
Validasi SNP
Validasi SNP Pada Genom Varietas Tanggamus, Anjasmoro dan Wilis
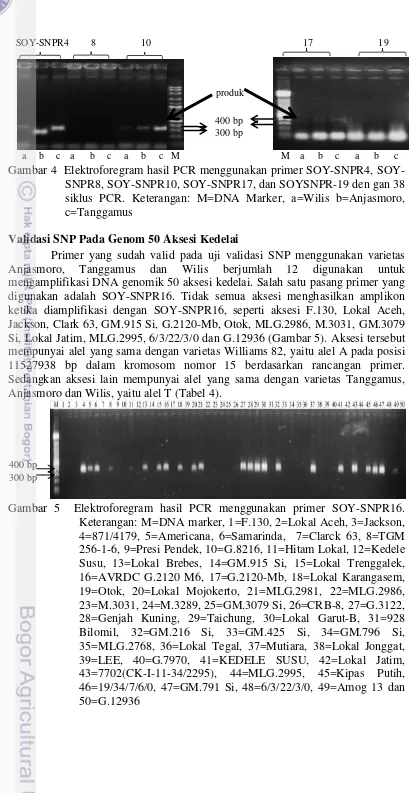
Primer SNAP yang berjumlah 19 pasang mayoritas berhasil mengamplifikasi DNA varietas Wilis, Tanggamus dan Anjasmoro dengan 28 siklus PCR (Gambar 3). Namun, SOY-SNPR4, SOY-SNPR10 dan SOY-SNPR17 tidak berhasil membentuk amplikon menggunakan siklus 28 dan berhasil menghasilkan amplikon jika menggunakan siklus 38 (Gambar 4). SOY-SNPR1, SOY-SNPR6, SOY-SNPR7, SOY-SNPR11 dan SOY-SNPR18 dirancang sebagai primer yang spesifik terhadap alel yang dimiliki varietas William 82 sehingga seharusnya tidak berhasil mengamplifikasi DNA varietas Wilis, Tanggamus dan Anjasmoro. Namun, elektroforegram menunjukkan adanya amplikon pada ketiga varietas, sehingga SNP pada lokasi yang sesuai target primer tersebut tidak valid (Tabel 3). SNP pada lokasi yang sesuai target primer SNPR8 dan SOY-SNPR19 juga belum valid keberadaanya karena amplikon tidak terbentuk pada penggunaan 28 dan 38 siklus PCR. SOY-SNPR8 dan SOY-SNPR19 dirancang spesifik terhadap alel Wilis, Tanggamus dan Anjasmoro sehingga seharusnya terbentuk amplikon pada varietas tersebut. SNP yang valid berdasarkan primer SNAP hanya 12 dari 19 SNP dengan tingkat validitas sebesar 63% (Lampiran 5). Tabel 3 Perbandingan hasil PCR observasi berdasarkan 19 pasang primer SNAP
dengan hasil PCR yang diharapkan
Primer Hasil Harapan Hasil Observasi
Anjasmoro Tanggamus Wilis Anjasmoro Tanggamus Wilis
Produk 400 bp 300 bp
SOY-SNPR 1 2 4 5 6 7 8 9 10 11 12 13 14 15 16 17
M a b c a b c ab c a b c a bc a b c a b c a b c a bc a b c a bc a b c a b c a bc a b c a
produk 400 bp 300 bp
400 bp 300 bp
SOY-SNPR17 18 3 19
M a b a b c M a b c M a b c produk
Tabel 3 Perbandingan hasil PCR sebenarnya berdasarkan 19 primer SNAP dengan hasil PCR yang diharapkan (lanjutan)
Primer Hasil Harapan Hasil Observasi
Anjasmoro Tanggamus Wilis Anjasmoro Tanggamus Wilis
SOY-SNPR8 + + + - - -
SOY-SNPR9 + + + + + +
SOY-SNPR10a + + + + + +
SOY-SNPR11 - - - + + +
SOY-SNPR12 + + + + + +
SOY-SNPR13 + + + + + +
SOY-SNPR14 + + + + + +
SOY-SNPR15 + + + + + +
SOY-SNPR16 + + + + + +
SOY-SNPR17a + + + + + +
SOY-SNPR18 - - - + + +
SOY-SNPR19 + + + - - -
a
Primer yang optimal menggunakan siklus 38
Keterangan: (+)=Terbentuk amplikon, (-)=tidak terbentuk amplikon
.
Gambar 3 Elektroforegram hasil PCR menggunakan primer SOY-SNPR1 sampai SOY-SNPR19 dengan 28 siklus PCR. Keterangan: M=DNA Marker, a=Wilis b=Anjasmoro, c=Tanggamus
a b c a b c a b c M M a b c a b c 400 bp
300 bp
SOY-SNPR4 8 10 17 1 9
400 bp 300 bp
produk
Gambar 4 Elektroforegram hasil PCR menggunakan primer SNPR4, SOY-SNPR8, SOY-SNPR10, SOY-SNPR17, dan SOYSNPR-19 den gan 38 siklus PCR. Keterangan: M=DNA Marker, a=Wilis b=Anjasmoro, c=Tanggamus
Validasi SNP Pada Genom 50 Aksesi Kedelai
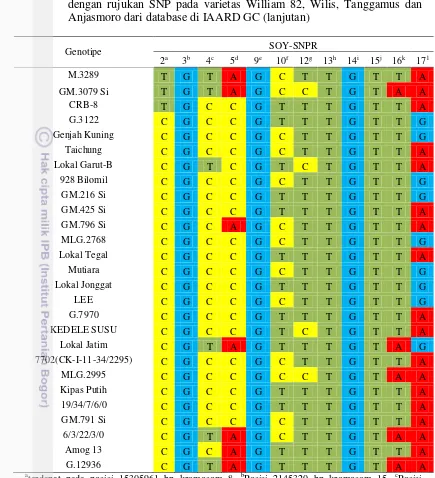
Primer yang sudah valid pada uji validasi SNP menggunakan varietas Anjasmoro, Tanggamus dan Wilis berjumlah 12 digunakan untuk mengamplifikasi DNA genomik 50 aksesi kedelai. Salah satu pasang primer yang digunakan adalah SOY-SNPR16. Tidak semua aksesi menghasilkan amplikon ketika diamplifikasi dengan SOY-SNPR16, seperti aksesi F.130, Lokal Aceh, Jackson, Clark 63, GM.915 Si, G.2120-Mb, Otok, MLG.2986, M.3031, GM.3079 Si, Lokal Jatim, MLG.2995, 6/3/22/3/0 dan G.12936 (Gambar 5). Aksesi tersebut mempunyai alel yang sama dengan varietas Williams 82, yaitu alel A pada posisi 11527938 bp dalam kromosom nomor 15 berdasarkan rancangan primer. Sedangkan aksesi lain mempunyai alel yang sama dengan varietas Tanggamus, Anjasmoro dan Wilis, yaitu alel T (Tabel 4).
Primer SNAP lain juga dapat membuktikan tersebarnya SNP pada 50 aksesi kedelai (Tabel 4). Namun primer SNPR9, SNPR13 dan SOY-SNPR15 tidak dapat membuktikan bahwa SNP terjadi pada posisi 9016561 bp kromosom 17, 43606825 bp kromosom satu dan 45105095 bp kromosom empat. Aksesi tersebut mempunyai alel yang sama dengan Tanggamus, Wilis dan Anjasmoro. Tidak ada yang sama dengan William 82. Pola yang ditunjukkan monomorfik sehingga SNP pada posisi tersebut tidak valid. Jumlah SNP yang valid menurun dari 12 menjadi 9 SNP dengan tingkat validitas sebesar 47% (Lampiran 5). Sembilan SNP yang valid berada pada posisi 15305961 bp kromosom 8, 2145320 bp kromosom 15, 39409513 bp kromosom 10, 4030106 bp kromosom 4, 4096214 bp kromosom 15, 3560119 bp kromosom 5, 8455691 bp kromosom 18, 11527938 kromosom 15 dan 49289270 bp kromosom 14.
Tabel 4 SNP pada 50 aksesi kedelai berdasarkan kinerja 12 pasang primer SNAP dengan rujukan SNP pada varietas William 82, Wilis, Tanggamus dan Anjasmoro dari database di IAARD GC (lanjutan)
Genotipe SOY-SNPR 39409513 bp kromosom 10. dPosisi 4030106 bp kromosom 4. ePosisi 9016561 bp kromosom 17.
f
Posisi 4096214 bp kromosom 15. gPosisi 3560119 bp kromosom 5. hPosisi 43606825 bp kromosom 1. iPosisi 8455691 bp kromosom 18. jPosisi 45105095 bp kromosom 4. kPosisi 11527938 bp kromosom 15. lPosisi 49289270 bp kromosom 14.
Kekerabatan 50 Aksesi Kedelai Menggunakan SNP Tervalidasi Tingkat Polimorfisme dan Heterozigositas
SNPR10 dan SNPR17 mempunyai nilai PIC tertinggi, yaitu 0.371. SOY-SNPR14 mempunyai nilai PIC terendah, yaitu 0.038. Nilai heterozigositas untuk semua marka sebesar nol, berarti tidak ada individu yang bersifat heterozigot. Rerata nilai diversitas gen atau heterozigositas harapan untuk semua marka, yaitu 0.341. SOY-SNPR10 dan SOY-SNPR17 mempunyai nilai diversitas gen tertinggi, yaitu 0.493. SOY-SNPR14 mempunyai nilai diversitas gen yang rendah, yaitu 0.039. Alel yang terlibat dalam marka sebanyak dua karena SNP hanya melibatkan dua alel. Rerata frekuensi alel utama, yaitu alel yang dimiliki varietas Wilis, Tanggamus dan Anjasmoro sebesar 0.742.
Tabel 5 Tingkat polimorfisme dan heterozigositas sembilan marka SNAP melalui 50 aksesi kedelai
Penyebaran SNP pada 50 aksesi kedelai berdasarkan sembilan pasang primer SNAP yang sudah valid dapat dikembangkan untuk mencari nilai kekerabatan antar aksesi. Besarnya koefisien jarak genetik antar aksesi kedelai berkisar 0 sampai 0.3 yang tergambar dalam filogenetik (Gambar 6). Aksesi kedelai berjumlah 50 dapat dibedakan menjadi dua kelompok besar berdasarkan nilai koefisien jarak genetik sebesar 0.3. Kelompok pertama terdiri atas 38 aksesi, sedangkan kelompok kedua hanya terdiri atas 12 aksesi. Dendogram tersebut mempunyai enam kelompok yang mempunyai koefisien kesamaan jarak genetik sebesar nol atau sangat identik. Salah satu kelompok yang mempunyai jarak genetik sebesar nol, yaitu kelompok yang terdiri atas 871/4179, 928 Bilomil, Genjah Kuning, Lee, MLG.2768, Mutiara dan Samarinda.
koefisien jarak genetik
Tabel 6 Matriks rerata jarak genetik antar daerah asal 50 aksesi kedelai
Aksesi kedelai yang daerah asalnya belum diketahui
PEMBAHASAN
DNA Genom Kedelai
Daun kedelai seperti tanaman lain mengandung polisakarida dan metabolit sekunder yang dapat menurunkan kemurnian hasil isolasi DNA. Kontaminan seperti polisakarida dapat mengganggu studi genetik karena menghambat aktivitas enzim, seperti DNA polimerase dalam proses PCR (Fang et al. 1992). Polifenol merupakan salah satu metabolit sekunder yang juga dapat menghambat studi genetik dengan merusak DNA. Senyawa tersebut akan teroksidasi dan berikatan secara kovalen dengan DNA dan protein. DNA akan tampak berwarna coklat ketika berikatan dengan polifenol yang teroksidasi dan DNA tidak dapat digunakan untuk proses selanjutnya (Hussain et al. 2015).
Isolasi DNA menggunakan bufer CTAB pada metode Doyle & Doyle merupakan pilihan yang tepat untuk isolasi DNA tanaman. Menurut Varma et al. (2007), CTAB akan mengendapkan protein dan karbohidrat dengan membentuk kompleks, namun tidak mengendapkan DNA pada kondisi kekuatan ionik yang tinggi. Senyawa metabolit sekunder, seperti polifenol dapat diatasi dengan PVP,
β-merkaptoetanol dan natrium bisulfit. PVP membentuk kompleks dengan polifenol melalui ikatan hidrogen sehingga polifenol akan terpisah dengan DNA. Natrium bisulfit dan β-merkaptoetanol berperan sebagai agen pereduksi untuk mencegah oksidasi polifenol (Choundhary et al. 2008). Menurut Varma et al. (2007), kontaminan polisakarida dan metabolit sekunder dapat dikurangi dengan menggunakan daun yang segar dan muda. Semakin tua umur daun maka kadar polisakarida dan metabolit sekunder semakin tinggi.
DNA semakin lambat kecepatan migrasinya (Gusmiaty et al. 2012). DNA genom 50 aksesi dan tiga varietas kedelai bermigrasi sangat lambat (Gambar 1 dan 2). Berdasarkan perbandingan dengan DNA marker, ukuran DNA genom sangat besar terletak dekat sumur gel. Hal tersebut disebabkan bahwa ukuran genom kedelai sebesar 1.1-1.15 gb (Shultz et al. 2006). Elektroforegram menunjukkan pita DNA yang tebal dengan sedikit smear. DNA diduga mengalami degradasi sehingga terbentuk smear yang diakibatkan aktivitas nuklease. Enzim nuklease dapat bersumber dari dalam sel yang gagal dihilangkan dan lingkungan (Karimnasab et al. 2013). Namun, degradasi pada level rendah masih dapat ditolerir dalam tahap selanjutnya.
Uji kuantitatif hasil isolasi DNA dilakukan menggunakan spektrofotometer nanodrop. DNA genom untuk semua genotipe mengandung kontaminan protein, polisakarida dan polifenol yang rendah. Hal tersebut ditunjukkan oleh A260/280 dan A260/230 berada pada nilai optimum (Tabel 1).
Menurut Al-Ashwal dan Hamdan (2013), tingkat kemurnian DNA sangat tinggi jika A260/280 dan A260/230 sebesar 1.8 sampai 2 dan 1.7 sampai 2.2. Nilai absorbansi
pada panjang gelombang 280 dan 230 nm lebih kecil dibandingkan 260 nm yang berbanding lurus dengan konsentrasi kontaminan yang sangat sedikit dibandingkan DNA. Nilai A260/230 juga dapat menunjukkan rendahnya kontaminan
pereaksi-pereaksi isolasi DNA yang tertinggal, seperti EDTA dan senyawa organik (Bhandari et al. 2014).
Rendahnya kontaminan yang berada pada hasil isolasi DNA genom kedelai dipengaruhi oleh ketepatan metode isolasi DNA. Selain karbohidrat dan polifenol yang berhasil dipisahkan dari DNA, kontaminan protein juga dapat dengan mudah untuk dipisahkan karena penggunaan metode yang tepat. Penggunaan kloroform isoamilalkohol sangat berperan penting untuk memisahkan protein dari DNA. Protein didenaturasi oleh kloroforom dan larut dalam fase organik atau interfase, sedangkan DNA larut dalam fase air sehingga protein mudah untuk dipisahkan dari DNA. Sedangkan, isoamilalkohol meningkatkan pemisahan antara fase organik dan fase air (Susanti dan Ariani 2004). Kualitas dan kuantitas DNA hasil isolasi menunjukkan bahwa DNA genom dapat digunakan untuk tahap selanjutnya, yaitu PCR untuk validasi SNP.
Primer SNAP
Program SNAPER memberikan sekitar 32 alternatif sekuen primer SNAP untuk membuktikan SNP pada posisi yang sama. Hanya satu pasang primer yang digunakan dalam penelitian dari 32 pasang primer. Sebanyak 19 primer SNAP digunakan untuk membuktikan SNP terjadi pada 19 posisi pilihan (Tabel 2). Pemilihan dilakukan secara acak dengan memperhatikan keterangan “high end self complementary”. rimer dengan keterangan tersebut tidak boleh digunakan karena dapat mengganggu proses PCR. Menurut Singh et al. (2000), sekuen primer yang komplemen terhadap sekuennya sendiri mengakibatkan terbentuknya
Setiap sekuen primer SNAP pada ujung 3 OH akan komplemen terhadap nukleotida spesifik dalam DNA cetakan. Jika pada ujung 3 OH SOY-SNPR2 mempunyai basa C maka primer hanya menempel pada DNA cetakan yang mempunyai basa C pada lokasi sesuai target primer tersebut. DNA cetakan yang mempunyai basa lain selain C akan menyebabkan primer tidak akan menempel dan gagal mengamplifikasi DNA cetakan. Hal tersebut sesuai dengan Lestari dan Koh (2013), bahwa primer SNAP dapat membedakan individu yang mempunyai perbedaan basa tunggal di lokasi spesifik dalam genom.
Modifikasi primer juga dilakukan pada salah satu dari tiga nukleotida dekat ujung 3 OH. Nukleotida pada posisi pertama dari ujung 3 OH pada SOY-SNPR2 dibuat tidak komplemen (mismatch) dengan cetakan DNA. Hal tersebut meningkatkan kegagalan penempelan primer pada DNA yang membawa nukleotida tidak spesifik karena mengandung dua mismatch, yaitu pada ujung 3 OH primer dan posisi satu dari ujung 3 OH primer. Sedangkan cetakan DNA yang membawa nukleotida spesifik hanya mengalami mismatch pada satu posisi antara posisi dekat dari ujung 3 OH primer dengan cetakan DNA sehingga primer tetap berhasil mengamplifikasi DNA. Hal tersebut disebabkan karena primer masih mempunyai kemampuan untuk menempel pada cetakan DNA dengan satu posisi mismatch yang berada di dalam (Kwok et al. 1990). Modifikasi tersebut bertujuan meningkatkan spesifisitas primer dalam membedakan individu yang mempunyai basa tunggal berbeda karena jika hanya mengandalkan satu posisi mismatch menyebabkan spesifisitas sangat rendah (Drenkard et al. 2000).
Primer SNAP yang digunakan dalam penelitian mempunyai panjang sekuen sebesar 22 sampai 36 bp dan Tm sebesar 56.992 sampai 63.305 oC. Menurut Borah (2011), secara umum panjang sekuen dan Tm untuk primer sebesar 18 sampai 22 bp dan 52 sampai 58 oC. Primer yang terlalu panjang mengakibatkan sulitnya penempelan primer terhadap DNA. Tm yang terlalu tinggi berpotensi terjadi secondary annealing atau penempelan primer secara tidak spesifik. Berdasarkan beberapa penelitian, penggunaan primer SNAP dengan karakteristik panjang sekuen dan Tm lebih dari optimum tetap menghasilkan amplikon yang spesifik. Hal tersebut dibuktikan melalui penggunaan primer SNAP sebagai marka fenotipe kualitas beras (Lestari dan Koh 2013). Menurut Drenkard et al. (2000), pemilihan siklus PCR yang lebih berpengaruh terhadap hasil PCR.
Validasi SNP
Validasi SNP Pada Genom Varietas Tanggamus, Anjasmoro dan Wilis
terjadi karena sekuensing dan penjajaran sekuen masih berpotensi mengalami kesalahan (You et al. 2011). Menurut Schirmer et al. (2015), tahap pembacaan sekuen dalam sekuensing melalui penerimaan spektra warna yang dikeluarkan oleh nukleotida bermuatan fluoresen merupakan tahap yang rentan mengalami kesalahan. Menurut Nielsen et al. (2011), keakuratan penjajaran sekuen mempunyai peran penting agar tidak terjadi kesalahan pendektesian SNP.
Ujung 3 OH primer SNPR1, SNPR6, SNPR7, SOY-SNPR11 dan SOY-SNPR18 dirancang spesifik atau match dengan nukleotida yang dimiliki William 82 dan tidak spesifik atau mismatch dengan Anjasmoro, Tanggamus dan Wilis. Posisi mismatch juga ditambahkan pada nukleotida dekat ujung 3 OH yang mengakibatkan peningkatan kegagalan amplifikasi DNA genomik ketiga varietas. Menurut Sutanto et al. (2013), primer SNAP dirancang seperti itu agar dapat membedakan antara beberapa individu yang mempunyai perbedaan nukleotida tunggal dalam posisi tertentu. Individu yang mempunyai nukleotida T dengan individu yang mempunyai nukleotida G dapat dibedakan menggunakan SNAP. Namun, DNA Wilis, Tanggamus dan Anjasmoro berhasil diamplifikasi dengan membentuk amplikon (Tabel 3). Hal tersebut menunjukkan bahwa posisi 2756750 bp kromosom 7, 38802168 bp kromosom 10, 14803147 bp kromosom 6, 5106501 bp kromosom 3 dan 4853774 bp kromosom 3 pada genom Wilis, Tanggamus dan Anjasmoro mempunyai nukleotida yang sama dengan William 82 atau false SNP.
Pemilihan banyaknya siklus PCR yang digunakan pada SNAP sangat berdampak terhadap hasil PCR. Siklus PCR untuk primer SNAP yang umum digunakan sebanyak 28 dan 38 siklus. SOY-SNPR2, SOY-SNPR3, SOY-SNPR5, SOY-SNPR9, SOY-SNPR12, SOY-SNPR13, SOY-SNPR14, SOY-SNPR15 dan SNPR16 optimal menggunakan siklus 28 (Gambar 3). Sedangkan, SOY-SNP4, SOY-SNP10 dan SOY-SNP17 pada siklus 28 tidak menghasilkan amplikon. Menurut Drenkard et al. (2000), primer yang tidak membentuk amplikon menggunakan siklus 28 disebabkan primer membutuhkan konsentrasi DNA cetakan yang lebih tinggi. Penggunaan siklus 38 sangat tepat untuk mengatasi hal tersebut. Ketiga primer tersebut tidak boleh dipaksakan menggunakan siklus 28 karena akan menyebabkan false negative. Suatu posisi yang dinyatakan tidak terdapat SNP, namun sebenarnya terdapat SNP merupakan false negative.
mismatch maka semakin tinggi kegagalan menghasilkan produk PCR (Sutanto et al. 2013). Sehingga kedua primer tersebut belum mampu mendeteksi keberadaan SNP.
Validasi SNP Pada Genom 50 Aksesi Kedelai
SOY-SNPR2, SOY-SNPR3, SOY-SNPR4, SOY-SNPR5, SOY-SNPR10 SOY-SNPR12, SOY-SNPR14, SOY-SNPR16 dan SOY-SNPR17 berhasil membuktikan keberadaan SNP pada posisi spesifik dalam genom 50 aksesi kedelai (Tabel 4). Primer tersebut dirancang spesifik terhadap varietas Wilis, Tanggamus dan Anjasmoro. Jika terbentuk amplikon maka aksesi tersebut mempunyai alel yang sama dengan ketiga varietas tersebut, sedangkan aksesi yang tidak terbentuk amplikon mempunyai alel yang sama dengan William 82. Beberapa aksesi ada yang mempunyai alel yang sama dengan William 82 dan ada yang mempunyai alel yang sama dengan Wilis, Tanggamus dan Anjasmoro. SNP tersebut berpeluang dijadikan marka molekuler yang berkorelasi dengan karakter tanaman kedelai. Menurut Thomas et al. (2011), perubahan satu basa nitrogen berpotensi mengubah asam amino yang akan disintesis melalui translasi dan transkripsi. Perubahan asam amino tersebut akan mengubah fungsi protein yang mengakibatkan perubahan ekspresi gen. Hal tersebut dibuktikan bahwa perubahan basa A menjadi T dalam gen GmNARK mengubah karakter supernodulasi pada kedelai (Kim et al. 2005). SNP terbagi menjadi dua jenis, yaitu SNP synonymous dan SNP non-synonymous. SNP yang dapat mengubah asam amino disebut SNP non-synonymous, sedangkan SNP yang tidak dapat mengubah asam amino disebut SNP synonymous (Bromberg dan Rost 2007).
SOY-SNPR9, SOY-SNPR13 dan SOY-SNPR15 tidak dapat membuktikan adanya SNP pada 50 aksesi kedelai (Tabel 4). Semua aksesi kedelai mempunyai alel yang sama dengan varietas Wilis, Tanggamus dan Anjasmoro. Jika 50 aksesi kedelai pada posisi 9016561 kromosom 17, 43606825 kromosom 1 dan 45105095 krosomom 4 mempunyai nukleotida yang sama dengan pola monomorfik berarti tidak terjadi SNP pada posisi tersebut. Menurut Vignal et al. (2002), SNP seharusnya berupa nukleotida tunggal yang mengalami variasi, umumnya melibatkan dua nukleotida, seperti A/G. Beberapa individu mempunyai nukleotida A dan lainnya mempunyai nukleotida G. Sehingga hanya sembilan SNP yang valid keberadaanya dalam genom 50 aksesi kedelai yang menunjukkan pola polimorfik.
Kekerabatan 50 Aksesi Kedelai Menggunakan SNP Tervalidasi Tingkat Polimorfisme dan Heterozigositas
alel SNP mempunyai frekuensi yang sama menunjukkan bahwa SNP tersebut tersebar merata dalam populasi. Sehingga SNP tersebut dapat membedakan secara genetik 50 aksesi kedelai lebih baik dibandingkan SNP pada posisi lainnya. Sedangkan SOY-SNPR14 mempunyai tingkat polimorfisme terendah dengan nilai PIC 0.038 sehingga kemampuan untuk membedakan secara genetik 50 aksesi kedelai sangat rendah terbukti bahwa hanya satu aksesi yang dapat dibedakan dari 50 aksesi kedelai.
Suatu marka molekuler harus mempunyai nilai PIC yang tinggi untuk meningkatkan keakuratan dalam pengujian kekerabatan genetik. Sembilan marka SNAP mempunyai tingkat informatif yang kurang sampai sedang berdasarkan nilai PIC. Menurut Tasliah et al. (2013), marka yang mempunyai nilai PIC sebesar 0.25-0.5 tingkat informatifnya sedang dan jika dibawah 0.25 kurang informatif. Kisaran nilai PIC tertinggi untuk marka bialel, seperti SNAP sebesar 0 sampai 0.5. Jika dibandingkan dengan marka SSR yang multialel mempunyai nilai PIC 0 sampai 1 (Singh et al. 2013).
Menurut Bassil et al. (2005), diversitas gen atau heterozigositas harapan merupakan frekuensi dua alel yang berbeda terpilih secara acak pada suatu populasi. SOY-SNPR10 dan SOY-SNPR17 mempunyai diversitas gen tertinggi, yaitu 0.493 (Tabel 5). Hal tersebut menyatakan bahwa peluang individu mempunyai alel A dan G dalam posisi target SOY-SNPR10 serta alel C dan T dalam posisi target SOY-SNPR17 sebesar 49.30 %. Individu yang mempunyai dua alel yang berbeda pada posisi yang sama dalam genom merupakan individu heterozigot (Keating dan MD 1992). Sehingga nilai diversitas gen juga dapat memprediksi seberapa besar individu dalam populasi yang bersifat heterozigot. Frekuensi alel utama dan alel alternatif yang tidak terlalu berbeda pada marka tersebut menyebabkan diversitas gen tinggi. Hal tersebut sesuai White et al. (2007), bahwa diversitas gen dipengaruhi oleh fekuensi alel utama dan alel alternatifnya, semakin kecil selisihnya maka diversitas gen semakin tinggi. SOY-SNPR14 mempunyai nilai diversitas gen yang rendah, yaitu 0.039. Peluang individu yang mempunyai dua alel (A dan G) dalam posisi target SOY-SNPR14 sangat kecil. Hal tersebut disebabkan frekuensi masing-masing alel mempunyai selisih yang besar.
Filogenetik dan Koefisien Jarak Genetik
Keberadaan SNP pada 50 aksesi kedelai berdasarkan kinerja sembilan pasang primer SNAP menunjukkan perbedaan genetik pada aksesi tersebut. Perbedaan genetik antar aksesi kedelai dapat ditunjukkan melalui koefisien jarak genetik atau koefisien kesamaan genetik. Filogenetik dapat dikonstruksi berdasarkan jarak genetik antar aksesi kedelai. Filogenetik menunjukkan bahwa antar 50 aksesi kedelai mempunyai koefisien jarak genetik yang rendah bahkan beberapa aksesi kedelai mempunyai kekerabatan yang sangat dekat dengan koefisien jarak genetik sebesar nol (Gambar 6). Menurut Chaerani et al. (2011), aksesi kedelai tidak ada yang mempunyai koefisien jarak genetik sebesar nol berdasarkan marka SSR. Hal tersebut disebabkan bahwa sembilan marka SNAP yang digunakan hanya melibatkan SNP yang terletak pada tujuh pasang kromosom 50 aksesi kedelai. Marka tersebut masih belum cukup dan mampu untuk membedakan beberapa aksesi kedelai secara genetik. Jumlah kromosom untuk kedelai sebanyak 20 pasang kromosom (Walling et al. 2006). Kemungkinan SNP dalam kromosom lain selain 7 pasang kromosom tersebut mengandung SNP dengan tingkat polimorfisme yang lebih tinggi dan dapat membedakan seluruh 50 aksesi kedelai. Hal tersebut didukung bahwa SNP merupakan marka yang keberadaannya melimpah di dalam genom dan polimorfisme tinggi (Blair et al. 2012). Sembilan SNP tersebut juga tidak tersebar secara merata pada genom 50 aksesi kedelai sehingga beberapa aksesi kedelai tidak dapat dibedakan.
Lima puluh aksesi kedelai yang terkelompok dalam daerah asalnya dapat dibedakan melalui rerata koefisien jarak genetik antar daerah, yaitu sebesar 0.101 sampai 0.500 (Tabel 6). Antara aksesi yang berasal dari Jawa Barat dan aksesi yang berstatus admixture sangat dekat jarak genetiknya dengan koefisien jarak genetik sebesar 0.101. Hal tersebut mengindikasikan bahwa sebagian besar askesi yang berstatus admixture merupakan aksesi yang berasal dari Jawa Barat. Aksesi kedelai yang belum diketahui daerah asalnya diberi status Admixture. Antara aksesi yang berasal dari Filipina dan Aceh mempunyai jarak genetik tertinggi, yaitu sebesar 0.500. Antara aksesi yang berasal dari Filipina dan Aceh berpotensi dijadikan sebagai tetua yang dapat disilangkan karena jarak genetik jauh lebih tinggi dibandingkan daerah lainnya. Menurut Chaerani et al. (2011), aksesi dengan jarak genetik yang tinggi berpotensi dijadikan tetua untuk persilangan yang menghasilkan varietas unggul. Namun, karakteristik yang dimiliki tetua juga harus diperhatikan karena sangat menentukan keberhasilan pemuliaan tanaman untuk mendapatkan varietas unggul.
SIMPULAN DAN SARAN
Simpulan
5, 8455691 bp kromosom 18, 11527938 kromosom 15 dan 49289270 bp kromosom 14. SNP tervalidasi tersebut belum mampu membedakan secara genetik beberapa aksesi dari 50 aksesi kedelai sehingga kekerabatan 50 aksesi kedelai sangat tinggi dengan koefisien jarak genetik sebesar 0 sampai 0.3. Aksesi kedelai yang berasal Filipina dan Aceh kekerabatannya paling jauh, sedangkan aksesi yang berasal dari Jawa Barat dan berstatus admixture kekerabatannya paling dekat.
Saran
Perlu dilakukan optimasi suhu annealing primer SNAP agar menghasilkan hasil yang lebih akurat dan diperlukan penggunaan primer SNAP yang lebih banyak dengan SNP target yang mempunyai posisi disetiap kromosom agar dapat membedakan 50 aksesi kedelai secara genetik. Penggunaan dua pasang primer SNAP yang masing-masing spesifik terhadap alel utama dan alel alternatif sangat diperlukan agar 50 aksesi kedelai dapat dibedakan secara genetik dan dapat mengidentifikasi individu heterozigot.
DAFTAR PUSTAKA
Al-Ashwal R, Hamdan S. 2013. Optimization for expression of coxacki adenovirus receptor on chinese hamster ovary cell line using different transfection methods. Life Sci J. 10(4):1119-1128.
Bassil NV, Botta R, Mehlenbacher SA. 2005. Microsatellites markers in hazelnuts: isolation, characterization, cross-species amplification. J Amer Soc Hort Sci. 130(4):543-549.
Bhandari A, Lamichhane N, Lamichhane S, Shakya P. 2014. An efficient method for isolation of genomic DNA from silica dried leaves of Taxus sps. Int J Biosci. 5(5):147-152.
Blair MW, Cortes AJ, Penmetsa RV, Farmer A, Carrasquilla-Garcia N, Cook DR. 2012. A high-throughput SNP marker system for parental polymorphism screening, and diversity analysis in common bean (Phaseolus vulgaris L.). Theor Appl Genet. 126(2):535-548.doi:10.1007/s00122-012-1999-z.
Bolibok-Brągoszewska H, Targonska M, Bolibok L, Kilian A, Rakoczy-Trojanowska M. 2014. Genome-wide characterization of genetic diversity and population structure in Secale. BMC Plant Biol. 14(184):1-15.
Borah P. 2011. Primer design for PCR. Sci Vis. 11(3):134-136.
Bromberg Y, Rost B. 2007. SNAP: predict effect of non-synonymous polymorphisms on function. Nucleic Acids Res. 35(11):3823-3835. doi:10.1093/nar/gkm238.
Chaerani, Hidayatun N, Utami DW. 2011. Keragaman genetik 50 aksesi plasma nutfah kedelai berdasarkan sepuluh penanda mikrosatelit. J AgroBiogen. 7(2):96-105.
SNP detection, validation, and development of an 8K SNP array for apple. Plos One. 7(2):1-12.
Choundhary K, Mathur N, Choundhary OP, Pillai U. 2008. Protocol for isolation of genomic dna from dry and fresh leaves of Vigna species suitable for RAPD restriction digestion. Adv Biol Res. 2(5):83-89.
Doyle JJ and Doyle JL. 1987. A rapid DNA isolation procedure from small quantities of fresh leaf tissue. Phytochem Bull. 19(1):11-15.
Drenkard E, Richter BG, Rozen S, Stutius LM, Angell NA, Mindrinos M, Cho RJ, Oefner PJ, Davis RW, Ausubel FM. 2000. A simple procedure for the analysis of single nucleotide polymorphisms facilitates map-based cloning in arabidopsis. Plant Physiol. 124:1483-1492.
Eka A, Hanafiah DS, Nuraidi I. 2015. Respon morfologis dan fisiologis beberapa varietas kedelai (Glycine max L. Merrill) di tanah masam. J Onl Agroekotek. 3(2):507-514.
Fang G, Hammar S, Grumet R. 1992. A quick and inexpensive method for removing polyscaccharides from plant genomic DNA. BioTechniq. 13(1):52-54.
Gusmiaty, Restu M, Pongtuluran I. 2012. Seleksi primer untuk analisis keragaman genetik jenis bitti (Vitex coffassus). J Perennial. 8(1):25-29.
Handini AS. 2014. Analisis keragaman morfologi dan biokimia pada anggrek Phalaenopsis serta analisis keragaman genetik dengan marka SNAP [Skripsi]. Bogor (ID): Institut Pertanian Bogor.
Hussain H, Shujah S, Haider SZ, Rehman S, Iqbal Z, Naz S, Yasmin N, Khattak NS, Khan SA, Naeem R. 2015. Inhibition of poly phenol oxidation during genomic DNA isolation and morphological variations in Berberis lycium royle genotypes. JBMS. 3(1):25-34.
Jehan T, Lakhanpaul S. 2006. Single nucleotide polymorphism (SNP)-methods and applications in plant genetics: a review. Indian J Biotechnol. 5:453-459. Johari S, Sutopo, Kurnianto E, Hasviara E. 2008. Polimorfisme Protein Darah
Ayam KEDU. J Indon Trop Anim Agric. 33(4):313-318.
Karimnasab, Tadayon N, Khaki P, Bidhendi PM, Ghaderi S, Sekhavati R, Asadi M. 2013. An optimized affordable DNA-extraction method from Salmonella enterica enteritidis for pcr experiments. Archives Razi Institute. 68(2):105-109.
Keating M, MD. 1992. Linkage analysis and long QT syndrome using genetics to study cardiovascular disease. Circulat. 85(6):1973-1986.
Kim MY, Van K, Lestari P, Moon JK, Lee SH. 2005. SNP identification and SNAP marker development for a GmNARK gene controlling supernodulation in soybean. Theor Appl Genet. 10(110):1003-1010.
Kumar S, Banks TW, Cloutier S. 2012. SNP discovery through next-generation sequencing and its applications. Int J Plant Genom. 2012(831460):1-15.doi:10.1155/2012/831460.
Kwok S, Kellogg DE, McKinney N, Spasic D, Goda L, Levenson C, Sninsky JJ. 1990. Effects of primer-template mismatches on the polymerase chain reaction: human immunodeficiency virus type 1 model studies. Nucleic Acids Res. 18:999-1005.
[Internet]. [diunduh 2015 Juni 6]. Tersedia pada: http://iaardgc.or.id/detail-publikasi/12.
Lestari P dan Koh HJ. 2013. Development of new CAPS/dCAPS and SNAP markers for rice eating quality. Hayati J Biosci. 20(1):15-23.
Liu K, Muse SV. 2005. PowerMarker: integrated analysis environment for genetic marker data. Bioinform. 21(9):2128-2129.
Millah M, Habibah NA, Suwarni E. 2010. Analisis keanekaragaman genetik dan diferensiasi jati jawa dan madura berdasarkan marka mikrosatelit untuk mendukung fingerprinting jati. Biosain. 2(2):101-109.
Montes JM, Technow F, Martin M, Becker K. 2014. Genetic diversity in Jatropha curcas L. assessed with SSR and SNP markers. Diversity. 6:551-566.
Sambrook J, Russel DW. 2001. Molecular Cloning: A Laboratory Manual. Edisi ke-3. New York (US): Cold-Spring Harbor Laboratory Pr.
Santoso TJ, Utami DW, Septiningsih EM. 2006. Analisis sidik jari DNA plasma nutfah kedelai menggunakan markah SSR. J AgroBiogen. 2(1):1-7.
Schirmer M, Ijaz UZ, D’Amore R, Hall N, Sloan WT, Quince C. 2015. Insight into biases and sequencing errors for amplicon sequencing with the Illumina MiSeq platform. Nucleic Acid Res. 43(6):1-16.doi: 10.1093/nar/gku134. Shultz JL, Kurunam D, Shopinski K, Iqbal MJ, Kazi S, Zobrist K, Bashir R,
Yaegashi S, Lavu N, Afzal AJ et al. 2006. The soybean genome database (SoyGD): a browser for display of duplicated, polyploid, regions and sequence tagged sites on the integrated physical and genetic maps of
Glycine max. Nucleic Acids Res. 34:758-765.doi:10.1093/nar/gkj050.
Singh N, Choudhury DR, Singh AK, Kumar S, Srinivasan K, Tyagi RK, Singh NK, Singh R. 2013. Comparison of SSR and SNP markers in estimation of genetic diversity and population structure of Indian rice varieties. Plos One. 8(12).1-14.
Singh VK, Govindarajan R, Naik S, Kumar A. 2000. The effect of hairpin structure on PCR amplification efficiency. Mol Biol Today. 1(3):67-69. Siqueira MVBM, Dequigiovanni G, Corazon-Guivin MA, Feltran JC, Veasey EA.
2012. DNA fingerprinting of water yam (Dioscorea alata) cultivars in Brazilbased on microsatellite markers. Hortic Bras. 30(4):653-659.
Supadi. 2009. Dampak impor kedelai berkelanjutan terhadap ketahanan pangan. Analis Kebijak Pertan.7(1):87-102.
Susanti EVH, Ariani SRD. 2004. Kloning gen penisilin V asilase dari Bacillus sp BAC4 melalui pembuatan pustaka genom. Biodiversitas. 5(1):1-6.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol Biol Evol. 28(10):2731-2739.doi: 10.1093/molbev/msr121.
Tasliah, Rijzaani H, Hariyadi TZP, Yuriyah S, Rebin, Ma’sumah, Silitonga TS.
2013. Analisis keragaman genetik 161 aksesi mangga indonesia
menggunakan marka mikrosatelit. J AgroBiogen. 9(3):125-134.
Terwilliger JD, Ott J. 1994. Handbook of Human Genetic Linkage. Baltimore (US): The Jons Hopkins University Press.
Thomas PE, Klinger R, Furlong LI, Hofmann-Apitius M, Friedrich CM. 2011. Challenges in the association of human single nucleotide polymorphism mentions with unique database identifiers. BMC Bioinform. 12(4):1-18. Varma A, Padh H, Shrivastava N. 2007. Plant genomic DNA isolation: an art or a
science. Biotechnol J. 2:386-392.doi:10.1002/biot.200600195.
Vignal A, Milan D, SanCristobal M, Eggen A. 2002. A review on SNP and other types of molecular markers and their use in animal genetics. Genet Sel Evol. 34(2002):275-305.doi:10.1051/gse:2002009.
Walling JG, Shoemaker R, Young N, Mudge J, Jackson S. 2006. Chromosome-level homeology in paleopolyploid soybean (Glycine max) revealed through integration of genetic and chromosome maps. Genet. 172:1893-1900.doi: 10.1534/genetics.105.051466.
Wan-Ping F, Meinhardt LW, Hua-Wei T, Zhou L, Mischke S, Zhang D. 2014. Varietal identification of tea (Camellia sinensis) using nanofluidic array of single nucleotide polymorphism (SNP) markers. Hortic Res. 1(14035):1-8.doi:10.1038/hortres.2014.35.
White TL, Adams WT, Nealse DB. 2007. Forest Genetics. Cambridge (US): CABI Publishing.
LAMPIRAN
Lampiran 1 Diagram alur penelitian
Penanaman Tanaman Kedelai
Desain Primer SNAP
Validasi Primer SNAP Pada Genom Varietas Wilis, Tanggamus, dan
Anjasmoro
Validasi Primer SNAP Pada Genom 50 Aksesi Kedelai
Uji Kekerabatan 50 Aksesi Kedelai Menggunakan Marka SNAP yang
Valid
Lampiran 2 Tanaman kedelai yang digunakan dalam penelitian
No. Aksesi Genotipe Asal Daerah
05003-03460 Wilis Admixture
05003-01409 Americana Bogor (Kota), Jawa Barat 05003-01459 Samarinda Jember, Jawa Timur
05003-01654 Clarck 63 Filipina
05003-01730 TGM 256-1-6 Admixture
05003-02323 Presi Pendek Banyuwangi, Jawa Timur
05003-02758 G.8216 Admixture
05003-03184 Hitam Lokal Subang, Jawa Barat 05003-03207 Kedele Susu Subang, Jawa Barat 05003-03246 Lokal Brebes Brebes, Jawa Tengah 05003-03562 GM.915 Si Subang, Jawa Barat 05003-03605 Lokal Trenggalek Ponorogo, Jawa Timur 05003-03628 AVRDC G.2120 M6 Taiwan
05003-03639 G.2120-Mb Taiwan
05003-03699 Lokal Karangasem Karangasem, Bali 05003-03740 Otok Bojonegoro, Jawa Timur 05003-03744 Lokal Mojokerto Mojokerto, Jawa Timur
05003-03763 MLG.2981 Jawa Tengah
05003-03765 MLG.2986 Jawa Timur
05003-03803 M.3031 Malang, Jawa Timur 05003-03806 M.3289 Malang, Jawa Timur
05003-04000 GM.3079 Si Admixture
05003-04133 CRB-8 Cirebon(kodya), Jawa Barat
05003-04149 G.3122 Jawa Barat
05003-04153 Genjah Kuning Jawa Barat
05003-04232 Taichung Taiwan
05003-04239 Lokal Garut-B Garut, Jawa Barat
05003-04301 928 Bilomil Taiwan
05003-04340 GM.216 Si Admixture
05003-04393 GM.425 Si Admixture
05003-04395 GM.796 Si Subang, Jawa Barat 05003-04405 MLG.2768 Malang, Jawa Timur 05003-04425 Lokal Tegal Tegal, Jawa Tengah
05003-04453 Mutiara Admixture
05003-04440 Lokal Jonggat Jonggat, Nusa Tenggara Barat
Lampiran 2 Tanaman kedelai yang digunakan dalam penelitian (lanjutan)
No. Aksesi Genotipe Asal Daerah
05003-02623 G.7970 Taiwan
05003-03200 KEDELE SUSU Subang, Jawa Barat 05003-03478 Lokal Jatim Jawa Timur 05003-03521 7702(CK-I-11-34/2295) Admixture
05003-03770 MLG.2995 Jawa Timur
05003-03846 Kipas Putih Aceh
05003-04307 19/34/7/6/0 USA
05003-04394 GM.791 Si Subang, Jawa Barat
05003-03911 6/3/22/3/0 USA
05003-04464 Amog 13 Admixture
Lampiran 3 Sekuen dan Melting Temperature (Tm) 19 pasang primer SNAP Primer Sekuen Primer (5’ 3’) Tm (oC)
SOY-SNPR1 F-GGTGGGTTGCCTTGGAAGGGTTAC 60.800
R-TACTGGTCAGTCCTCCATAGATGCAGTGG 62.920
SOY-SNPR2 F-TGTTGCTATTAAATATCTTAAACAGATCCTAAAACC 57.611 R-ACCTTGAATTACTTTAGGTGTGGGAAACTTAAGGAC 62.166 SOY-SNPR3 F-TCATACCTGTATAATAACAAAAGAGCATTTGGCG 59.594
R-GCTTACTTAACTCAATGGCCAATGGCG 59.737
SOY-SNPR4 F- CAAACACTCAACTCTCTGTTCCAACAACTCAAC 61.918
R-GATATAATCCACAACCGGTTCCCCACA 59.737
SOY-SNPR5 F-GAAGCATTGTCACTTTCATTTAAGTTTCGCC 59.080 R-ACAAGTAAGTACAAGTAAGGAGCAGGCATCAATGT 62.088
SOY-SNPR6 F-CTCACTACTCATCCTGGGGGCCTG 62.508
R-GTCTACCTCTACCCATCTGCTTTGGAGAAAACT 63.160
SOY-SNPR7 F-GCGGCTTCCAATGATTTCAGTGAGC 59.324
R-CAAATTTGATACATTACAGTGACAAGCCTGGAA 59.433
SOY-SNPR8 F-CTGGAGCCACAGACACGTGTCGTC 62.508
R-CACCCATCGACGCCAAGAGTAGTAAGAA 61.385
SOY-SNPR9 F-CAGGGGTGAAGGAAATGGAAGCG 58.839
R-CGATGCACCCATTGTCAGGGAAGT 59.091
SOY-SNPR10 F-GTCTCTAATGTCATGTCCCTACTCTGAAAGTGAATC 63.305
R-TTCATCAATCACTTGTTCTTTTCCCACACA 57.520
SOY-SNPR11 F-GGAAATGTGGGGTCTACATACCCTACACG 62.920
R-CCCAACGACGTGGATTAATAGAGATAAATAATAAAT 58.750
SOY-SNPR12 F-TGCGCACACACCCAGCTTCAGT 58.563
R-CCTAATTCTTGAATCTTGGACAAAAAGTACACCAA 59.743 SOY-SNPR13 F-GATGTTGACTCTACATTTTCTTGGGAGCGT 60.253
R-CAAAATTTGCCTAAACCGAACATCTCGTTATT 57.981
SOY-SNPR14 F-ACAGCATTTTTCATTGGGTTTTGGAGTG 56.992
R-TAATCGTATCTCCACAGAGTGGATTCACATGA 60.543
SOY-SNPR15 F-GGCAACCATTTGCTACTAGTGTATGATTTTTCAT 59.594
R-CCGTAACAATGTCATCACCTAACTGAGCAAG 61.725
SOY-SNPR16 F-AGCAATTGATCATGTACCTTTAGACTACCTTACCCT 62.166
R-AAGACACCAAAGTCATTAACACATGTACAACCG 60.675
SOY-SNPR17 F-TCAAAAGCTGATGGAGCATTCGCTATTTTA 57.520
R-TGAAAATCAATAACATAGCATCTGTGAACACTTGTC 59.888 SOY-SNPR18 F-GGCATGAATAAACGTTGCAAGAGCTCC 59.737
R-AGATGAAATTCTCTTTTTCATGGGACCAGTCTAA 59.594
SOY-SNPR19 F-GTAGCCCTGATCCCACGCTGAAAA 59.091
Lampiran 5 Tingkat validitas SNP
Tingkat validitas SNP pada genom varietas Anjasmoro, Tanggamus, dan Wilis Tingkat validitas =
x 100%
Tingkat validitas =
x 100% = 63%
Tingkat validitas SNP pada genom 50 aksesi kedelai
Tingkat validitas =
x 100%
Tingkat validitas =
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta pada tanggal 18 Desember 1993 dari bapak Didi Sulaiman dan ibu Eka Tisah. Penulis merupakan anak pertama dari dua bersaudara. Penulis sekolah di SMA Negeri 1 Cikarang Utara dan lulus dari SMA tersebut pada tahun 2011 dan pada tahun yang sama lulus seleksi masuk IPB melalui jalur SMPTN Undangan. Penulis memilih mayor Biokimia, Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam dan minor Manajemen Fungsional, Departemen Manajemen, Fakultas Ekonomi dan Manajemen.
Selama mengikuti perkuliahan, penulis pernah melakukan Praktek Lapangan (PL) di Balai Besar Penelitian dan Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian, Bogor selama periode Juni 2014 hingga Agustus 2014 dengan judul Optimasi Primer untuk Analisis Marka Single Nucleotide Polymorphisms (SNP) Kakao (Theobroma Cacao L.). Penulis juga aktif dalam kegiatan kemahasiswaan di IPB, seperti Himpunan Profesi Community of Research and Education in Biochemistry (CREBs) sebagai staf divisi keilmuan Metabolisme pada tahun 2012/2013.