ABSTRAK
NUR HASANAH. Analisis Taksonometri pada Karakter Morfologi Daun Dikotiledon Kelas Magnoliopsida menggunakan SOM Kohonen. Dibimbing oleh AZIZ KUSTIYO dan ARIEF RAMADHAN.
Taksonometri atau taksonomi numerik merupakan cabang dari ilmu taksonomi yang menggunakan metode kuantitatif dengan bantuan teknologi komputasi untuk melakukan identifikasi dan klasifikasi organisme. Karakter morfologi daun sebagai organ vegetatif tumbuhan diketahui memiliki pola tertentu yang memperlihatkan keteraturan, sehingga dapat dikelompokkan secara sistematis. Penelitian ini menggunakan 126 data spesimen daun kelas Magnoliopsida yang terdiri atas 21 family, 17 ordo, dan 4 subclass untuk membentuk tiga model taksonometri dengan metode SOM Kohonen. Identifikasi dilakukan terhadap 16 karakter primer morfologi daun.
Model taksonometri family, ordo, dan subclass dibentuk dengan neuron output masing-masing berjumlah 21, 17, dan 4 neuron, sesuai dengan banyaknya setiap tingkatan taksa. Data latih dan data uji dibentuk dengan metode 3-fold cross validation. Fungsi jarak yang digunakan adalah Mahalanobis, dengan jumlah iterasi maksimum, laju pembelajaran awal (αi) dan lebar tetangga awal (δi) yang telah ditentukan. Dari evaluasi terhadap Indeks Davies-Bouldin (IDB) diperoleh model taksonometri family terbaik dengan δi bernilai 10 dan αi bernilai 0.9, taksonometri ordo dengan δi bernilai 5 dan αi bernilai 0.9, dan taksonometri subclass dengan δi bernilai 3 dan αi bernilai 0.5.
Evaluasi cluster precision (CP) dan recall (CR) menghasilkan rata-rata CP dan CR dari ketiga model taksonometri sebesar 0.629 dan 0.644. Nilai ini menggambarkan adanya variasi morfologi daun yang cukup tinggi pada kelas Magnoliopsida. Representasi pengetahuan menunjukkan adanya konsistensi pada hasil taksonometri ketiga model tersebut, sehingga SOM Kohonen terbukti dapat digunakan untuk analisis taksonometri terhadap karakter morfologi daun. Dapat disimpulkan pula bahwa karakter morfologi daun memberikan hasil yang prospektif untuk digunakan sebagai salah satu cara dalam identifikasi dan pengelompokan tumbuhan.
ANALISIS TAKSONOMETRI PADA KARAKTER MORFOLOGI
DAUN DIKOTILEDON KELAS MAGNOLIOPSIDA
MENGGUNAKAN SOM KOHONEN
NUR HASANAH
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ANALISIS TAKSONOMETRI PADA KARAKTER MORFOLOGI
DAUN DIKOTILEDON KELAS MAGNOLIOPSIDA
MENGGUNAKAN SOM KOHONEN
NUR HASANAH
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ANALISIS TAKSONOMETRI PADA KARAKTER MORFOLOGI
DAUN DIKOTILEDON KELAS MAGNOLIOPSIDA
MENGGUNAKAN SOM KOHONEN
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer
pada Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
NUR HASANAH
G64104022
DEPARTEMEN ILMU KOMPUTER
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
NUR HASANAH. Analisis Taksonometri pada Karakter Morfologi Daun Dikotiledon Kelas Magnoliopsida menggunakan SOM Kohonen. Dibimbing oleh AZIZ KUSTIYO dan ARIEF RAMADHAN.
Taksonometri atau taksonomi numerik merupakan cabang dari ilmu taksonomi yang menggunakan metode kuantitatif dengan bantuan teknologi komputasi untuk melakukan identifikasi dan klasifikasi organisme. Karakter morfologi daun sebagai organ vegetatif tumbuhan diketahui memiliki pola tertentu yang memperlihatkan keteraturan, sehingga dapat dikelompokkan secara sistematis. Penelitian ini menggunakan 126 data spesimen daun kelas Magnoliopsida yang terdiri atas 21 family, 17 ordo, dan 4 subclass untuk membentuk tiga model taksonometri dengan metode SOM Kohonen. Identifikasi dilakukan terhadap 16 karakter primer morfologi daun.
Model taksonometri family, ordo, dan subclass dibentuk dengan neuron output masing-masing berjumlah 21, 17, dan 4 neuron, sesuai dengan banyaknya setiap tingkatan taksa. Data latih dan data uji dibentuk dengan metode 3-fold cross validation. Fungsi jarak yang digunakan adalah Mahalanobis, dengan jumlah iterasi maksimum, laju pembelajaran awal (αi) dan lebar tetangga awal (δi) yang telah ditentukan. Dari evaluasi terhadap Indeks Davies-Bouldin (IDB) diperoleh model taksonometri family terbaik dengan δi bernilai 10 dan αi bernilai 0.9, taksonometri ordo dengan δi bernilai 5 dan αi bernilai 0.9, dan taksonometri subclass dengan δi bernilai 3 dan αi bernilai 0.5.
Evaluasi cluster precision (CP) dan recall (CR) menghasilkan rata-rata CP dan CR dari ketiga model taksonometri sebesar 0.629 dan 0.644. Nilai ini menggambarkan adanya variasi morfologi daun yang cukup tinggi pada kelas Magnoliopsida. Representasi pengetahuan menunjukkan adanya konsistensi pada hasil taksonometri ketiga model tersebut, sehingga SOM Kohonen terbukti dapat digunakan untuk analisis taksonometri terhadap karakter morfologi daun. Dapat disimpulkan pula bahwa karakter morfologi daun memberikan hasil yang prospektif untuk digunakan sebagai salah satu cara dalam identifikasi dan pengelompokan tumbuhan.
Judul : Analisis Taksonometri pada Karakter Morfologi Daun Dikotiledon
Kelas Magnoliopsida menggunakan SOM Kohonen
Nama : Nur Hasanah
NIM : G64104022
Menyetujui:
Pembimbing I,
Aziz Kustiyo, S.Si, M.Kom
NIP 132 206 241
Pembimbing II,
Arief Ramadhan, S.Kom
Mengetahui:
Dekan Fakultas Matematika dan Ilmu Pengetahuan Alam
Institut Pertanian Bogor
Dr. drh. Hasim, DEA
NIP 131 578 806
RIWAYAT HIDUP
Penulis dilahirkan sebagai putri pertama dari tujuh bersaudara di Batang, 16 Mei 1987, dari pasangan Achmad dan Yudiwanti Wahyu Endro Kusumo. Pendidikan formal semenjak TK hingga SMA ditempuhnya di kota hujan Bogor, hingga pada tahun 2004 penulis lulus dari SMA Negeri 1 Bogor. Pada tahun yang sama, penulis melanjutkan ke jenjang pendidikan S1 di Departemen Ilmu Komputer, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor, melalui jalur Undangan Seleksi Masuk IPB (USMI).
Selama mengikuti kegiatan perkuliahan, penulis aktif pada beberapa kegiatan kelembagaan mahasiswa, seperti Himpunan Mahasiswa Ilmu Komputer (HIMALKOM) periode 2004/2005 dan periode 2005/2006, serta Badan Eksekutif Mahasiswa (BEM) FMIPA IPB periode 2006/2007. Selain itu, penulis juga mengikuti organisasi pembinaan pada SMA asal penulis, yaitu Forum Alumni Muslim SMAN 1 Bogor (FORKOM ALIMS) pada periode 2004/2005 hingga periode 2008/2009. Pada lingkup Departemen Ilmu Komputer, penulis turut aktif sebagai asisten praktikum untuk beberapa mata kuliah pada tahun akademik 2006/2007 hingga 2007/2008.
PRAKATA
Alhamdulillahi rabbil ‘alamin, puji dan syukur penulis panjatkan kepada Allah SWT atas segala rahmat dan karunia-Nya sehingga penulis dapat menyelesaikan tugas akhir sebagai salah satu syarat untuk memperoleh gelar Sarjana Komputer di IPB. Penelitian yang berjudul “Analisis Taksonometri pada Karakter Morfologi Daun Dikotiledon Kelas Magnoliopsida menggunakan SOM Kohonen” merupakan usaha penulis dalam implementasi ilmu komputer yang telah didapatkan selama perkuliahan untuk digunakan pada disiplin ilmu lain, terutama berkaitan dengan ranah dasar Institut Pertanian Bogor sebagai perguruan tinggi bernafaskan pertanian.
Penghargaan dan rasa terima kasih penulis sampaikan kepada Bapak Aziz Kustiyo, S.Si, M.Kom dan Bapak Arief Ramadhan, S.Kom selaku pembimbing, atas dukungan, bimbingan, dan arahan yang telah dicurahkan selama pengerjaan tugas akhir; serta Bapak Ir. Agus Buono, M.Si, M.Kom atas kesediaannya menjadi penguji dan atas bimbingan dan masukannya untuk perbaikan tugas akhir ini.
Kepada Ibu Dr. Ir. Sri Nurdiati, M.Sc selaku Ketua Departemen Ilmu Komputer, penulis ingin menyampaikan rasa terima kasih atas bimbingan dan dukungannya terhadap segala aktivitas yang penulis jalani pada masa perkuliahan. Demikian pula untuk seluruh dosen pengajar yang telah mendidik dan membangun wawasan serta kepribadian penulis selama menuntut ilmu di Departemen Ilmu Komputer.
Untuk Ibu yang senantiasa berperan sebagai pembimbing pribadi bagi penulis, untuk Bapak atas dukungan dan dorongan semangat kepada penulis untuk selalu mencapai yang terbaik, juga untuk nenek tercinta dan adik-adik tersayang yang selalu menyediakan keceriaan dan pelepas penat di rumah, terima kasih atas bantuan dan doanya.
Terima kasih atas kebersamaan, pengertian, dan nasihat yang telah diberikan oleh keluarga kecil penulis: Aghiez, Ratih, Weni, Inna, dan Lia. Kepada Listya, terima kasih telah mengajarkan arti persahabatan. Untuk Danang, Bowo, dan Dika, terima kasih untuk inspirasinya tentang visi, mimpi, dan kerja keras. Atas penerimaan, kegembiraan, dan masa-masa indah selama perkuliahan, terima kasih kepada teman-teman Ilkomerz 41 dan keluarga besar Ilkomerz IPB.
Kepada rekan-rekan aktivis kampus dan sekolah, terima kasih untuk mengajarkan begitu banyak hal kepada penulis. Untuk seluruh staf Departemen Ilmu Komputer, terima kasih atas kemudahan dan kenyamanan dalam kegiatan perkuliahan. Terima kasih juga penulis sampaikan untuk pihak-pihak lain yang tidak dapat disebutkan satu persatu.
Penulis menyadari bahwa penelitian ini masih jauh dari sempurna, baik dalam pelaksanaannya maupun pada hasilnya. Meskipun demikian, penulis berharap agar penelitian ini dapat memberikan manfaat bagi seluruh pihak.
Bogor, Januari 2009
DAFTAR ISI
Halaman
DAFTAR TABEL ... vi
DAFTAR GAMBAR ... vi
DAFTAR LAMPIRAN ... vii
PENDAHULUAN Latar Belakang ... 1
Tujuan ... 1
Ruang Lingkup ... 1
Manfaat ... 1
TINJAUAN PUSTAKA Taksonomi ... 2
Taksonometri ... 2
Analisis Korelasi ... 2
Koefisien Korelasi Spearman ... 3
Analisis Cluster ... 3
Self-Organizing Maps (SOM) Kohonen ... 3
Topologi Jaringan ... 4
Algoritme ... 4
Fungsi Jarak ... 4
Fungsi Tetangga ... 4
Laju Pembelajaran ... 4
K-Fold Cross Validation ... 4
Indeks Davies-Bouldin ... 5
ClusterRecall dan Precision ... 5
METODE PENELITIAN Praproses ... 5
Pembentukan Model Taksonometri ... 6
Analisis Taksonometri ... 7
Spesifikasi Pengembangan ... 7
HASIL DAN PEMBAHASAN Praproses ... 7
Pembentukan Data Latih dan Data Uji ... 9
Pembentukan Model Taksonometri Family ... 9
Pembentukan Model Taksonometri Ordo... 10
Pembentukan Model Taksonometri Subclass ... 10
Analisis Taksonometri ... 10
Evaluasi ClusterRecall dan Precision ... 10
Representasi Pengetahuan ... 11
Taksonometri Family ... 12
Taksonometri Ordo ... 13
Taksonometri Subclass ... 14
Representasi Umum ... 15
KESIMPULAN DAN SARAN Kesimpulan ... 16
Saran ... 16
DAFTAR PUSTAKA ... 16
DAFTAR TABEL
Halaman
1 Parameter pelatihan dan pengujian model taksonometri ... 7
2 Daftar anggota tingkatan taksa subclass, ordo, dan family ... 7
3 Jumlah data spesimen daun pada setiap family ... 8
4 Jumlah data spesimen daun pada setiap ordo ... 8
5 Jumlah data spesimen daun pada setiap subclass ... 8
6 Karakter dan state spesimen daun dari speciesAglaia multinervis... 9
7 Persebaran jumlah taksa pada setiap subset data ... 9
8 Nilai CP dan CR untuk ketiga model taksonometri ... 11
9 Deskripsi cluster hasil taksonometri family ... 12
10 Deskripsi taksa hasil taksonometri family ... 12
11 Deskripsi cluster hasil taksonometri ordo ... 13
12 Deskripsi taksa hasil taksonometri ordo ... 13
13 Deskripsi cluster hasil taksonometri subclass ... 15
14 Deskripsi taksa hasil taksonometri subclass ... 15
DAFTAR GAMBAR
Halaman 1 Susunan taksonomi tumbuhan ... 22 Struktur jaringan SOM Kohonen satu dimensi ... 3
3 Topologi SOM Kohonen satu dimensi ... 4
4 Diagram alir metode penelitian ... 5
5 Diagram alir praproses... 5
6 Diagram alir pembentukan model taksonometri ... 6
7 Diagram alir analisis taksonometri ... 7
8 IDB pada percobaan model taksonometri family ... 9
9 IDB pada percobaan model taksonometri ordo ... 10
10 IDB pada percobaan model taksonometri subclass ... 10
11 Cluster precision taksonometri family ... 12
12 Cluster recall taksonometri family ... 12
13 Cluster precision taksonometri ordo ... 13
14 Cluster recall taksonometri ordo ... 13
15 Cluster precision taksonometri subclass ... 15
DAFTAR LAMPIRAN
Halaman
1 Contoh data spesimen daun sebelum kodefikasi ... 19
2 Contoh daftar karakter morfologi daun sebelum kodefikasi ... 20
3 Rincian kodefikasi karakter morfologi daun ... 21
4 Ilustrasi karakter morfologi daun ... 23
5 Contoh data spesimen daun setelah kodefikasi ... 27
6 Hasil uji taraf nyata dua arah pada koefisien korelasi Spearman... 28
7 Rincian pembagian taksa ke dalam setiap subset data ... 29
8 IDB pada percobaan model taksonometri family ... 30
9 Ilustrasi model taksonometri family untuk bobot akhir pada neuron output pertama ... 30
10 Daftar bobot akhir model taksonometri family ... 31
11 IDB pada percobaan model taksonometri ordo ... 32
12 Ilustrasi model taksonometri ordo untuk bobot akhir pada neuron output pertama ... 32
13 Daftar bobot akhir model taksonometri ordo ... 33
14 IDB pada percobaan model taksonometri subclass ... 34
15 Ilustrasi model taksonometri subclass untuk bobot akhir pada neuron output pertama ... 34
16 Daftar bobot akhir model taksonometri subclass... 34
17 Deskripsi hasil taksonometri family ... 35
18 Deskripsi hasil taksonometri ordo ... 36
PENDAHULUAN
Latar Belakang
Dunia biologi mengenal adanya konsep identifikasi untuk setiap organisme di dunia yang telah dilakukan penelitian atasnya. Cara yang umum digunakan untuk mengidentifikasi suatu organisme adalah melalui taksonomi. Sejak bermulanya pada abad ke-4 sebelum Masehi, ilmu taksonomi telah mengalami perkembangan yang pesat. Penggunaan teknologi komputer sebagai alat bantu pada taksonomi modern melahirkan bidang baru yang dinamakan taksonomi numerik atau taksonometri (Tjitrosoepomo 2005).
Stace (1980) memberikan lima cara yang
sering digunakan untuk melakukan
identifikasi terhadap tumbuhan. Kelima cara tersebut yaitu melalui karakteristik morfologi dan anatomi tumbuhan, unsur kimiawi penyusun tumbuhan, struktur kromosom, breeding system, dan lokasi geografis serta ekologi dari tumbuhan.
Jika ditinjau dari segi kemudahan dan kecepatan dalam mendapatkan data, maka karakteristik morfologi dan anatomi menjadi acuan pertama dalam proses identifikasi tumbuhan. Karakteristik ini dapat diamati pada organ vegetatif tumbuhan, seperti daun, batang, dan cabang, serta pada organ generatif tumbuhan, seperti bunga dan buah pada tumbuhan dikotiledon. Kedua organ tumbuhan ini memiliki perbedaan waktu observasi. Organ generatif tumbuhan hanya dapat diamati pada waktu tertentu, sedangkan organ vegetatif tumbuhan cenderung tersedia sebagai sumber pengamatan sepanjang waktu. Sebagai salah satu organ vegetatif tumbuhan, karakter morfologi daun khususnya pada tumbuhan dikotiledon diketahui mempunyai pola tertentu yang memper-lihatkan keteraturan, sehingga dapat dikelompokkan secara sistematis. Di lain pihak, karakter ini memiliki beberapa kelemahan, antara lain sifatnya yang kurang stabil akibat pengaruh lingkungan, adanya morfologi yang serupa pada anggota taksa yang tidak saling berhubungan, dan sifat polimorfisme yang terdapat pada tahap tertentu pertumbuhan daun (Hickey 1973 dalam Rasnovi 2001).
Pengkajian terhadap pemakaian karakter morfologi daun dalam identifikasi jenis telah dilakukan dalam beberapa penelitian. Rasnovi (2001) dalam tesisnya menyimpulkan bahwa
karakter morfologi daun dapat memisahkan species contoh dengan indikasi nilai separation coefficient gabungan bernilai satu. Klasifikasi citra daun menggunakan PNN (Probabilistic Neural Network) dengan data berupa 12 fitur citra daun yang dilakukan oleh Wu et al. (2007) menghasilkan akurasi hingga melebihi 90%. Morfologi daun juga telah digunakan bersama dengan karakter bunga dan buah dalam identifikasi species Endos-permum duodenum (Salwana et al. 2007).
Data spesimen daun yang berupa data kualitatif dapat diolah dalam proses identifikasi kesamaan ciri menggunakan self-organizing maps (SOM) Kohonen (Madarum 2006). Penelitian tersebut menghasilkan nilai cluster recall sebesar 0.429 dan cluster precision sebesar 0.530. Hasil yang belum optimal ini membuka peluang bagi penelitian lebih lanjut untuk membangun model taksonometri tumbuhan berdasarkan karakter morfologi daun dengan hasil yang lebih baik. Tujuan
Penelitian ini bertujuan :
1 Mengimplementasikan algoritme SOM Kohonen dalam clustering data spesimen daun dikotiledon kelas Magnoliopsida untuk membentuk model taksonometri tumbuhan.
2 Mendapatkan karakteristik data hasil taksonometri dan membandingkannya dengan taksonomi tumbuhan yang telah ditentukan oleh pakar taksonomi.
Ruang Lingkup
Penelitian ini meliputi pembuatan model taksonometri tumbuhan menggunakan ciri organ vegetatif tumbuhan berupa morfologi daun dikotiledon kelas Magnoliopsida. Metode yang digunakan untuk membangun model taksonometri adalah jaringan SOM Kohonen satu dimensi, dengan jumlah karakter morfologi daun sebanyak 16 karakter. Tingkatan taksa yang menjadi target penelitian adalah tingkat family, ordo, dan subclass. Jumlah neuron output pada setiap model taksonometri ditentukan berdasarkan jumlah taksa yang bersesuaian. Sebanyak 126 data spesimen daun, yang terdiri atas 21 family, 17 ordo, dan 4 subclass, kesemuanya merupakan anggota dari kelas Magnoliopsida.
Manfaat
spesimen daun dikotiledon berdasarkan kesamaan cirinya. Dari hasil analisis, akan diketahui kemampuan dan efektivitas SOM Kohonen dalam melakukan pengelompokan terhadap spesimen tumbuhan berdasarkan karakter morfologi daun. Selain itu, dari hasil clustering dapat diketahui pula hubungan antara karakter morfologi daun dengan taksonomi tumbuhan.
TINJAUAN PUSTAKA
Taksonomi
Taksonomi dapat dideskripsikan sebagai studi dan deskripsi mengenai variasi dalam organisme, investigasi terhadap sebab dan akibat variasi tersebut, dan penggunaan data yang diperoleh untuk menciptakan sistem klasifikasi (Stace 1980). Kelas-kelas yang dihasilkan oleh proses taksonomi disebut taksa, misalnya phylum, family, atau species. Penggunaan istilah ini mengindikasikan tingkatan suatu kelas dan organisme yang berada di dalam kelas tersebut.
Dalam ilmu taksonomi, tumbuhan diklasi-fikasikan ke dalam tujuh grup taksa, yaitu Kingdom, Divisio/Phylum, Class, Ordo, Family, Genus, dan Species. Di antara grup tersebut terdapat beberapa subgrup seperti superordo dan subclass. Ilustrasi taksonomi tumbuhan dapat diamati pada Gambar 1.
Gambar 1 Susunan taksonomi tumbuhan. Taksonometri
Seiring perkembangan dunia ilmu pengetahuan, penggunaan komputer dalam mengembangkan metode kuantitatif untuk melakukan klasifikasi tumbuhan semakin meningkat, sehingga menghasilkan bidang baru dalam taksonomi tumbuhan yang
dinamakan taksonomi numerik atau
taksonometri (Tjitrosoepomo 2005).
Sokal & Sneath (1963) mendefinisikan taksonometri sebagai metode evaluasi kuantitatif mengenai kesamaan atau kemiripan sifat antar golongan organisme, dan penataan golongan-golongan tersebut melalui analisis cluster ke dalam kategori takson yang lebih tinggi atas dasar kesamaan-kesamaan tersebut. Taksonometri didasarkan atas bukti-bukti fenetik, yaitu kemiripan yang diperlihatkan objek studi yang diamati dan dicatat, dan bukan berdasarkan kemungkinan perkem-bangan filogenetiknya.
Terdapat lima kegiatan dalam analisis taksonometri, yang diawali dengan pemilihan objek studi yang mewakili golongan organisme tertentu, yang disebut dengan OTU (Operational Taxonomic Unit). Kegiatan berikutnya adalah pemilihan karakter, pengukuran kemiripan, analisis cluster, dan penarikan kesimpulan (Tjitrosoepomo 2005).
Pengukuran kemiripan antar OTU didasarkan pada karakter yang dimilikinya. Menurut Sokal & Sneath (1963), karakter yang digunakan sebagai identifikasi OTU merupakan deskripsi terhadap bentuk, struktur, atau sifat yang membedakan sebuah unit taksonomi dengan unit lainnya.
Setiap karakter memiliki nilai yang dapat bersifat kualitatif ataupun kuantitatif. Karakter yang berkaitan dengan bentuk dan struktur merupakan karakter kualitatif, sedangkan karakter yang mendeskripsikan ukuran, panjang, dan jumlah merupakan karakter kuantitatif. Secara umum, karakter kualitatif lebih berguna dalam membedakan taksa pada tingkat taksonomi yang lebih tinggi, sementara karakter kuantitatif banyak digunakan untuk membedakan kategori taksonomi pada tingkatan yang lebih rendah (Naik 1985).
Analisis Korelasi
Koefisien Korelasi Spearman
Pada statistika nonparametrik, nilai koefisien korelasi antara dua peubah yang terdapat pada skala -1 hingga 1 sulit untuk diinterpretasikan secara tepat, sehingga digunakan pengambilan keputusan dengan pengujian pada taraf nyata tertentu α. Pengukuran yang digunakan adalah koefisien korelasi Spearman (Paulson 2003).
Data yang diolah untuk mendapatkan koefisien korelasi Spearman merupakan data berskala ordinal atau interval yang dapat diurutkan dari yang terkecil hingga terbesar. Uji nyata satu arah maupun dua arah dapat diaplikasikan pada koefisien korelasi yang diperoleh.
Koefisien korelasi Spearman didapatkan dengan rumus: ) 1 ( 6 1 2 1 2 − − =
∑
= n n d r n i i c ,dengan nilai di2 adalah:
( )
( )
[
]
. 1 2 1 2∑
∑
= = − = n i i i n ii R x R x
d
Analisis Cluster
Analisis cluster adalah mekanisme eksplorasi data yang umum digunakan dalam permasalahan klasifikasi. Analisis ini bertujuan mengelompokkan data ke dalam grup atau cluster sedemikian rupa sehingga derajat asosiasi di antara anggota dari satu cluster bersifat kuat dan derajat asosiasi antara anggota dari satu cluster dengan cluster lain bersifat lemah (Astel et al. 2007).
Clustering berbeda dengan klasifikasi dalam hal variabel target yang ditentukan. Dalam proses clustering, tidak ada proses klasifikasi, peramalan, atau prediksi terhadap nilai dari variabel target (Larose 2004).
Penggunaan analisis cluster dalam taksonometri bertujuan membentuk kelompok taksa dengan pengukuran kemiripan karakter (Tjitrosoepomo 2005). Hasil dari analisis cluster telah terbukti dapat dibandingkan dengan sistem taksonomi yang dibangun oleh pakar (Naik 1985; Sokal & Sneath 1963). Self-Organizing Maps (SOM) Kohonen
Metode Self-Organizing Maps (SOM) atau dikenal sebagai SOM Kohonen pertama kali diperkenalkan oleh Malsburg pada tahun 1973, kemudian diperbaiki dan dikembangkan
oleh Teuvo Kohonen pada tahun 1982. Kohonen (2001) mendeskripsikan SOM sebagai metode pemetaan yang bersifat nonlinear dan terurut dari data input dengan dimensi tinggi ke dalam array tujuan dengan dimensi yang lebih rendah. Metode pembelajaran yang digunakan bersifat unsupervised, artinya pembelajaran yang dilakukan terhadap data input tidak disertai dengan target ekspektasi terhadap hasil yang diinginkan (Freeman & Skapura 1991).
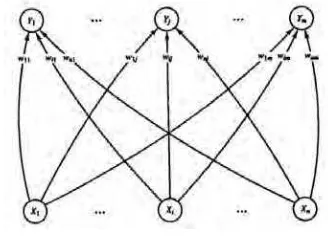
Gambar 2 Struktur jaringan SOM Kohonen satu dimensi.
Pada Gambar 2 dapat diamati struktur SOM Kohonen satu dimensi (Fausett 1994) yang terdiri atas dua lapisan, yaitu lapisan input (Xn) dan lapisan output (Ym). Setiap
neuron pada lapisan input terhubung dengan setiap neuron pada lapisan output melalui vektor bobot (wnm). Cluster yang terbentuk direpresentasikan oleh setiap neuron pada lapisan output.
Dalam proses pembelajaran jaringan SOM Kohonen, setiap neuron output saling
berkompetisi untuk menjadi neuron
pemenang, yang diperoleh dari perhitungan jarak yang paling dekat dengan neuroninput. Oleh karena itu, setiap neuron output akan bereaksi terhadap pola input tertentu, sehingga hasil dari SOM Kohonen akan menunjukkan adanya kesamaan ciri antar anggota clusternya (Larose 2004).
Topologi Jaringan
Beberapa topologi SOM Kohonen yang umum digunakan pada SOM Kohonen dua dimensi antara lain topologi grid, heksagonal, dan random (Kohonen 2001). SOM Kohonen satu dimensi hanya memiliki satu jenis topologi, seperti yang diilustrasikan pada Gambar 3. Pada gambar tersebut terdapat sembilan neuron dalam topologi satu dimensi, dengan neuron kelima sebagai neuron pemenang ditunjukkan dengan simbol #, dan neuron tetangganya ditunjukkan dengan simbol *, disertai dengan keterangan lebar tetangga 0, 1, dan 2 (Fausett 1994).
Gambar 3 Topologi SOM Kohonen satu dimensi.
Algoritme
Diketahui n adalah dimensi vektor input x = [x1, x2,..., xn]T. Vektor bobot pada neuron output j memiliki dimensi yang sama dengan vektor input, sehingga dapat dilambangkan dengan wj = [wj1, wj2,..., wjn]T.
Algoritme SOM dalam Kohonen (2001) dijelaskan sebagai berikut. Untuk setiap vektor input x, lakukan:
• Kompetisi. Untuk setiap simpul output j, hitung nilai D(wj,xn) yang diperoleh dari fungsi jarak. Tentukan simpul pemenang J (Best Matching Unit (BMU)) yang meminimumkan jarak antara vektor input x dengan semua simpul output.
• Kooperasi. Identifikasikan semua simpul output j dalam lingkungan simpul pemenang J menggunakan fungsi node tetangga (neighborhood function) h(t). Untuk setiap simpul dalam lingkungan tersebut, lakukan :
o Adaptasi. Perbaharui nilai bobot: wj(t+1) = wj(t) + h(t) * [xni – wj(t)]. • Perbaharui learning rate (α) dan lebar
tetangga δ.
• Hentikan perlakuan ketika kriteria pemberhentian telah dicapai.
Fungsi Jarak
Fungsi jarak digunakan untuk melakukan komputasi terhadap similaritas vektor input dengan vektor bobot pada setiap neuron output. Jarak Mahalanobis digunakan untuk mengukur jarak antara atribut yang
berkorelasi satu sama lain (Tan et al. 2004). Fungsi ini didefinisikan sebagai:
D(wj,xn) = (wj-xi) Σ-1 (wj-xi)T,
dengan Σ merupakan matriks kovarian dari vektor input (xn):
Fungsi Tetangga
Fungsi tetangga adalah derajat pengubahan terhadap bobot neuron pemenang dan tetangganya relatif terhadap lebar tetangga, yang akan berkurang seiring dengan langkah pembelajaran. Fungsi tetangga yang digunakan adalah fungsi Gauss (Kohonen 2001) dengan rumus :
dengan :
||ri-rc||2 = jarak neuron ke-i dengan neuron pemenang dalam grid
δ(t) = lebar tetangga, berkurang seiring dengan t langkah pembelajaran ri = neuron ke-i
rc = neuron pemenang
Perubahan lebar tetangga didapatkan dari perhitungan berikut (Tirozzi et al. 2007) :
dengan :
δi = nilai awal lebar tetangga δf = nilai akhir lebar tetangga tmax = iterasi maksimum
Laju Pembelajaran
Laju pembelajaran adalah fungsi penurunan tingkat pembelajaran seiring perubahan waktu (Fausett 1994). Nilai laju pembelajaran diperoleh dari rumus berikut (Kohonen 2001) :
dengan αi adalah nilai awal laju pembelajaran dan tmax adalah iterasi maksimum.
K-Fold Cross Validation
K-Fold Cross Validation adalah salah satu metode estimasi error. Dalam metode ini, akan dilakukan proses pengulangan sebanyak k-kali untuk himpunan contoh secara acak yang akan dibagi menjadi k-subset yang saling bebas. Pada setiap tahap pengulangan
, ) ( 2 || || exp * ) ( ) ( 2 2 − − = t rc ri t t h
δ
α
).
)(
(
1
1
1, ij j ik k
n
i k
j
x
x
x
x
n
−
∑
−
−
=
∑
= , ) ( max t t i f i t =δ
δ
δ
δ
, 1 ) ( max − = t tt
α
iakan diambil satu subset untuk data pengujian dan sisanya untuk data pelatihan (Fu 1994). Indeks Davies-Bouldin
Indeks Davies-Bouldin (IDB) merupakan salah satu metode validasi cluster untuk evaluasi kuantitatif dari hasil clustering. Pengukuran ini bertujuan memaksimalkan jarak inter-cluster antara satu cluster dengan cluster yang lain (separation value) sekaligus meminimalkan jarak intra-cluster antara titik dalam sebuah cluster (compactness value) (Bolshakova & Azuaje 2002; Gunter & Bunke 2002).
Jarak inter-cluster dkl didefinisikan sebagai berikut:
dkl = ||Ck – Cl|| ,
dengan Ck dan Cl adalah centroid cluster k dan cluster l.
Jarak intra-cluster sc(Qk) dalam cluster Qk dihitung dengan rumus:
dengan Nk adalah banyak titik yang termasuk dalam cluster Qk, dan Ck adalah centroid dari cluster Qk.
Dengan demikian, Indeks Davies-Bouldin didefinisikan sebagai berikut:
dengan nc adalah banyaknya cluster. Dari beberapa percobaan, akan dicari skema cluster yang optimal, yaitu skema yang memiliki nilai IDB paling rendah (Salazar et al. 2002). Cluster Recall dan Precision
Evaluasi kualitatif terhadap hasil clustering dapat diperoleh dari nilai cluster recall (CR) dan cluster precision (CP). Cluster recall menunjukkan besarnya proporsi jumlah data yang tercluster dengan benar dibandingkan dengan jumlah data dalam kelas yang sebenarnya. Adapun cluster precision menunjukkan proporsi jumlah data yang tercluster dengan benar dibandingkan dengan jumlah data dalam kelas hasil clustering. Nilai dari CR dan CP akan semakin baik jika mendekati satu.
Pencarian CR dan CP dilakukan dengan rumus berikut, dengan nij adalah jumlah anggota kelas i dalam cluster j, ni adalah jumlah anggota kelas i, dan nj adalah jumlah anggota cluster j (Madarum 2006):
METODE PENELITIAN
Penelitian ini dilakukan melalui tiga tahap utama seperti yang diilustrasikan pada Gambar 4, yaitu:
1 Praproses.
2 Pembentukan model taksonometri. 3 Analisis taksonometri.
Gambar 4 Diagram alir metode penelitian.
Praproses
Diagram alir langkah praproses yang dilakukan dalam penelitian ini dapat diamati pada Gambar 5. Kegiatan yang dilakukan dalam praproses data adalah kodefikasi, seleksi karakter, dan analisis korelasi.
Data yang digunakan pada penelitian ini diperoleh dari basis data Divora yang dikelola oleh World Agroforestry Centre (ICRAF) yang berlokasi di Bogor. Di dalam basis data ini terdapat informasi spesimen daun dari hutan karet yang terletak di wilayah Jambi dan Lampung. Sebanyak 126 spesimen daun akan digunakan dalam penelitian ini. Lampiran 1 memuat contoh data spesimen daun yang didapatkan dari basis data Divora.
Gambar 5 Diagram alir praproses. , || || ) ( k k i i k c N C X Q
s =∑ −
, ) , ( ) ( ) ( max 1 ) ( 1
∑
= + = nck kl k l
l c k c Q Q d Q s Q s n nc DB , ) , ( i ij n n j i
CR = (, ) .
Setiap spesimen daun dikotiledon pada basis data tersebut memiliki karakter-karakter tertentu yang melambangkan morfologinya. Secara keseluruhan terdapat 282 state yang terbagi ke dalam 10 level karakter. Pada level pertama, yaitu level primer, terdapat 21 karakter yang setiap karakternya memiliki state atau subkarakter pada level berikutnya. Level kedua memiliki 60 state dan satu state N.A. (Not Applicable). Level berikutnya memiliki lebih banyak jumlah state, demikian seterusnya hingga level terakhir. Contoh daftar karakter morfologi daun yang didapatkan dari basis data dapat diamati pada Lampiran 2.
Dalam penelitian ini hanya digunakan karakter-karakter daun yang terletak pada level pertama dan kedua, karena telah dapat mewakili karakter umum yang dimiliki oleh spesimen daun. Selain itu, tidak semua spesimen daun memiliki karakter-karakter pada level yang lebih dalam.
Untuk memudahkan pemrosesan data, perlu dilakukan pengubahan kode (kodefikasi) karakter yang terdapat pada basis data menjadi nilai yang dapat dikenali dan diproses oleh SOM Kohonen. Oleh karena itu, dibangun sistem kodefikasi yang khusus digunakan dalam penelitian ini.
Langkah selanjutnya adalah melakukan seleksi terhadap 21 karakter primer yang akan digunakan dalam proses clustering. Seleksi penting untuk dilakukan, karena ada kemungkinan tidak semua karakter dapat mewakili keunikan ciri dari masing-masing spesimen daun.
Hasil dari kodefikasi dan seleksi karakter adalah data spesimen daun yang siap digunakan untuk proses selanjutnya. Langkah berikutnya adalah melakukan analisis korelasi dari perhitungan terhadap koefisien korelasi Spearman, untuk mengetahui ada atau tidaknya korelasi antar karakter. Hasil dari analisis korelasi berguna untuk menentukan fungsi jarak yang akan digunakan pada jaringan SOM Kohonen.
Pembentukan Model Taksonometri
Proses pembentukan model taksonometri dapat diamati pada Gambar 6, meliputi: 1 Pembentukan data latih dan data uji. 2 Pelatihan dan pengujian.
3 Evaluasi IDB (Indeks Davies-Bouldin). 4 Penetapan model taksonometri.
Gambar 6 Diagram alir pembentukan model taksonometri.
Pembentukan data latih dan data uji didasarkan kepada metode k-fold cross validation. Dalam penelitian ini digunakan 3-fold cross validation, sehingga data spesimen daun akan dibagi ke dalam tiga subset dengan proporsi data setiap subset yang diusahakan mendekati proporsi data secara keseluruhan.
Proses pelatihan dan pengujian, evaluasi IDB, dan penetapan model taksonometri akan dilakukan sebanyak tiga kali, sesuai dengan jumlah model taksonometri yang ingin dibentuk. Model taksonometri yang pertama
akan melambangkan clustering untuk
tingkatan taksa family, sedangkan model taksonometri kedua dan ketiga masing-masing akan menggambarkan clustering untuk tingkat ordo dan subclass.
Pelatihan dan pengujian pada setiap model taksonometri menggunakan parameter yang tercantum pada Tabel 1, dengan n adalah jumlah neuron output, αi adalah laju pembelajaran awal, δi adalah lebar tetangga awal, dan tmax adalah iterasi maksimum. Tabel 1 Parameter pelatihan dan pengujian
model taksonometri.
Para-meter
Model Taksonometri
Family Ordo Subclass
n 21 17 4
αi 0.9, 0.5, 0.1 0.9, 0.5, 0.1 0.9, 0.5, 0.1
δi 20, 15, 10, 5 15, 10, 5 3, 2, 1
tmax 50 50 50
Metode inisialisasi nilai vektor bobot menggunakan nilai acak, dengan nilai maksimum setiap bobot diperoleh dari nilai maksimum karakter yang bersesuaian. Fungsi jarak yang digunakan adalah jarak Mahalanobis, karena adanya indikasi korelasi antar karakter. Kombinasi berbagai parameter tersebut akan diterapkan pada jaringan SOM Kohonen. Penetapan model taksonometri untuk setiap kategori didasarkan kepada jaringan dengan kinerja terbaik, yaitu jaringan yang menghasilkan nilai IDB minimum. Analisis Taksonometri
Model taksonometri yang telah
ditetapkan, bersama dengan data spesimen daun secara keseluruhan, selanjutnya akan diproses sebagai bahan untuk analisis taksonometri. Langkah-langkah dalam analisis ini dapat diamati pada Gambar 7.
Gambar 7 Diagram alir analisis taksono-metri.
Data spesimen daun yang telah diclusterkan menggunakan model taksono-metri family, ordo, dan subclass akan dievaluasi untuk mendapatkan nilai cluster precision (CP) dan cluster recall (CR). Selanjutnya, hasil taksonometri untuk setiap
tingkatan taksa akan diamati dan
dibandingkan dengan sistem taksonomi umum untuk mendapatkan representasi pengetahuan.
Spesifikasi Pengembangan
Dalam penelitian ini digunakan perangkat keras berupa laptop dengan prosesor Intel Centrino Duo 1.7 GHz dan memori 1 GB.
Model taksonometri dibangun pada
lingkungan pemrograman Java dengan Netbeans 6.0.1 sebagai IDE. SPSS 13.0 digunakan untuk analisis korelasi. Seluruh perangkat lunak berjalan pada sistem operasi Ubuntu ME 8.04.
HASIL DAN PEMBAHASAN
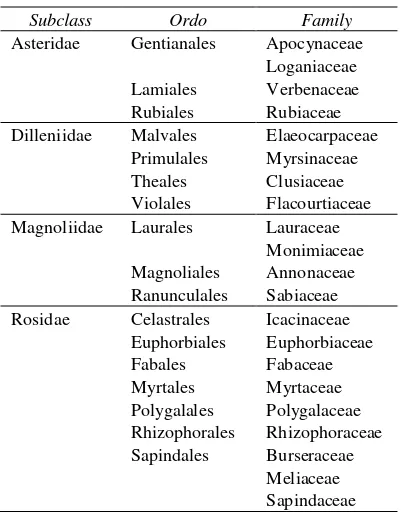
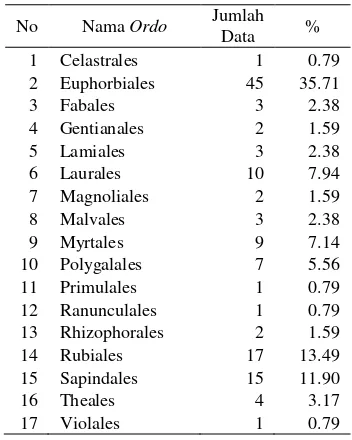
PraprosesDari basis data Divora didapatkan 126 spesimen daun kelas Magnoliopsida yang akan digunakan dalam penelitian ini. Jumlah tersebut terdiri atas 86 species, 44 genus, 21 family, 17 ordo, dan 4 subclass. Penelitian ini akan berfokus kepada tiga tingkatan taksa, yaitu family, ordo, dan subclass. Daftar anggota untuk ketiga tingkatan taksa ini dapat diamati pada Tabel 2. Rincian jumlah data spesimen daun untuk setiap tingkatan taksa disajikan pada Tabel 3, Tabel 4, dan Tabel 5.
Dari ketiga tabel tersebut dapat diamati bahwa terdapat persebaran data yang tidak merata di setiap kelasnya. Jumlah data terbesar terdapat pada family Euphorbiaceae (35.71%), ordo Euphorbiales (35.71%) dan subclass Rosidae (65.08%).
Tabel 2 Daftar anggota tingkatan taksa subclass, ordo, dan family.
Subclass Ordo Family
Asteridae Gentianales Apocynaceae
Loganiaceae
Lamiales Verbenaceae
Rubiales Rubiaceae
Dilleniidae Malvales Elaeocarpaceae
Primulales Myrsinaceae
Theales Clusiaceae
Violales Flacourtiaceae
Magnoliidae Laurales Lauraceae
Monimiaceae
Magnoliales Annonaceae
Ranunculales Sabiaceae
Rosidae Celastrales Icacinaceae
Euphorbiales Euphorbiaceae
Fabales Fabaceae
Myrtales Myrtaceae
Polygalales Polygalaceae
Rhizophorales Rhizophoraceae
Sapindales Burseraceae
Meliaceae
Tabel 3 Jumlah data spesimen daun pada setiap family.
No Nama Family Jumlah
Data %
1 Annonaceae 2 1.59
2 Apocynaceae 1 0.79
3 Burseraceae 8 6.35
4 Clusiaceae 4 3.17
5 Elaeocarpaceae 3 2.38
6 Euphorbiaceae 45 35.71
7 Fabaceae 3 2.38
8 Flacourtiaceae 1 0.79
9 Icacinaceae 1 0.79
10 Lauraceae 8 6.35
11 Loganiaceae 1 0.79
12 Meliaceae 2 1.59
13 Monimiaceae 2 1.59
14 Myrsinaceae 1 0.79
15 Myrtaceae 9 7.14
16 Polygalaceae 7 5.56
17 Rhizophoraceae 2 1.59
18 Rubiaceae 17 13.49
19 Sabiaceae 1 0.79
20 Sapindaceae 5 3.97
21 Verbenaceae 3 2.38
Tabel 4 Jumlah data spesimen daun pada setiap ordo.
No Nama Ordo Jumlah
Data %
1 Celastrales 1 0.79
2 Euphorbiales 45 35.71
3 Fabales 3 2.38
4 Gentianales 2 1.59
5 Lamiales 3 2.38
6 Laurales 10 7.94
7 Magnoliales 2 1.59
8 Malvales 3 2.38
9 Myrtales 9 7.14
10 Polygalales 7 5.56
11 Primulales 1 0.79
12 Ranunculales 1 0.79
13 Rhizophorales 2 1.59
14 Rubiales 17 13.49 15 Sapindales 15 11.90 16 Theales 4 3.17 17 Violales 1 0.79
Tabel 5 Jumlah data spesimen daun pada setiap subclass.
No Nama Subclass Jumlah
Data %
1 Asteridae 22 17.46
2 Dilleniidae 9 7.14
3 Magnoliidae 13 10.32
4 Rosidae 82 65.08
Kodefikasi dilakukan dengan menetapkan kisaran nilai untuk setiap karakter primer sesuai dengan jumlah state yang dimilikinya. Setiap karakter primer memiliki sebuah state N.A. (Not Applicable) yang dikodekan dengan angka nol (0). Selanjutnya, apabila karakter tersebut memiliki dua buah state atau lebih, setiap state dikodekan dengan angka 1, 2, dan seterusnya.
Rincian kodefikasi karakter morfologi daun yang dilakukan dimuat pada Lampiran 3. Beberapa karakter morfologi daun tersebut diilustrasikan pada Lampiran 4. Ilustrasi tersebut didapatkan dari Manual of Leaf Architecture (Ash et.al. 1999). Dari data awal, akan dilakukan kodefikasi terhadap setiap karakter sehingga menghasilkan satu nilai state untuk karakter tersebut. Apabila spesimen daun memiliki lebih dari satu state pada salah satu karakternya, maka state yang akan diperhatikan hanyalah salah satunya saja. Dari proses kodefikasi akan diperoleh hasil berupa sekuen angka yang menunjukkan karakteristik sebuah spesimen daun. Pada Tabel 6 disajikan contoh hasil kodefikasi dari species Aglaia multinervis. Karakteristik spesimen daun dari species ini adalah 211131911212111222112. Contoh data spesi-men daun hasil kodefikasi lainnya dapat diamati pada Lampiran 5.
Pengamatan terhadap data spesimen daun hasil kodefikasi menunjukkan bahwa terdapat lima karakter dengan variasi sangat kecil. Tiga karakter, yaitu karakter ke-4, 15, dan 20, memiliki state yang sama untuk seluruh data spesimen. Dua karakter lainnya, yaitu karakter ke-10 dan 21, hanya memiliki satu atau dua data spesimen yang berbeda state dengan spesimen lainnya. Oleh karena itu, kelima karakter ini tidak diikutsertakan dalam proses clustering, sehingga jumlah karakter yang akan digunakan dalam proses clustering adalah 16 karakter. Pada species Aglaia multinervis di atas, karakteristiknya menjadi 2113191112112221.
Tabel 6 Karakter dan state spesimen daun dari speciesAglaia multinervis.
No Karakter State Kode
1 Jenis daun
berdasarkan ada tidaknya anak daun
Daun majemuk, anak daun > 1
2
2 Susunan daun pada
batang
Tersebar 1
3 Ada tidaknya stipula Tidak ada 1
4 Ada tidaknya stipel Tidak ada 1
5 Helaian daun Bulat telur 3
6 Ujung daun Runcing 1
7 Pangkal daun Asimetri 9
8 Tepi daun Rata, halus 1
9 Ada tidaknya kelenjar daun
Tidak ada 1
10 Tangkai daun atau ibu tangkai daun
Ada 2
11 Tipe pertulangan daun
Menyirip 1
12 Pola urat daun tersier Menjala 2
13 Arah urat daun primer Lurus tak bercabang
1
14 Ada tidaknya vena
intramarginal
Tidak ada 1
15 Ada tidaknya duri Tidak ada 1
16 Ada tidaknya gabus Ada 2
17 Ada tidaknya getah Ada 2
18 Ada tidaknya rambut Ada 2
19 Ada tidaknya bintik Tidak ada 1
20 Ada tidaknya domatia Tidak ada 1
21 Paralel tidaknya urat daun sekunder
Tidak ada 2
Pembentukan Data Latih dan Data Uji
Pembagian 126 data spesimen daun ke dalam tiga subset, masing-masing sejumlah 42 data, dilakukan untuk mendapatkan data latih dan data uji. Rincian pembagian taksa ke dalam setiap subset data disajikan pada Lampiran 6, sedangkan banyaknya masing-masing tingkatan taksa pada setiap subset data dapat diamati pada Tabel 7.
Tabel 7 Persebaran jumlah taksa pada setiap subset data.
Taksa Subset Data
1 2 3
Family 15 17 14
(%) 71.4 81.0 66.7
Ordo 12 14 13
(%) 70.6 82.4 76.5
Subclass 4 4 4
(%) 100 100 100
Jumlah data yang tidak merata di setiap family dan ordo menyebabkan setiap subset
data tidak dapat sepenuhnya menyamai proporsi data secara keseluruhan. Taksa family yang memiliki 21 kelompok hanya dapat tersebar dengan jumlah maksimum 17 family di dalam satu subset data. Demikian pula taksa ordo yang tersebar dengan jumlah maksimum 14 dari total 17 ordo yang terdapat dalam data.
Pada Lampiran 6 dapat diamati bahwa beberapa family maupun ordo hanya memiliki satu data spesimen daun, sehingga tidak seluruh subset dapat memperoleh data family atau ordo tersebut. Contohnya adalah family Apocynaceae yang hanya dimiliki oleh subset data kedua, dan genus Celastrales yang hanya terdapat pada subset data pertama.
Ketiga subset data ini akan digunakan secara bergantian dalam proses pelatihan dan pengujian jaringan SOM Kohonen. Pada setiap iterasi, digunakan dua subset sebagai data latih dan satu subset sebagai data uji. Pembentukan Model Taksonometri Family
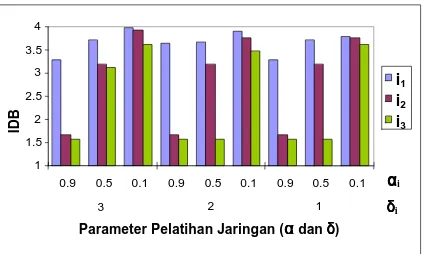
Kombinasi parameter-parameter yang telah ditetapkan akan digunakan untuk membangun jaringan SOM Kohonen sebagai model taksonometri family. Dari 36 percobaan, didapatkan grafik IDB yang disajikan pada Gambar 8, adapun data lengkapnya dapat diamati pada Lampiran 8.
Gambar 8 IDB pada percobaan model takso-nometri family.
IDB minimum dengan nilai 2.0058 diperoleh pada percobaan ketujuh di iterasi ketiga (i3) dengan parameter ukuran lebar tetangga awal (δi) sebesar 10 dan nilai laju pembelajaran awal (αi) sebesar 0.9. Oleh karena itu, bobot yang dihasilkan oleh jaringan pada percobaan ini akan digunakan sebagai model taksonometri family. Ilustrasi model SOM yang dihasilkan terdapat pada Lampiran 9, sedangkan daftar bobot akhirnya secara lengkap terdapat pada Lampiran 10.
1 1.5 2 2.5 3
0.9 0.5 0.1 0.9 0.5 0.1 0.9 0.5 0.1 0.9 0.5 0.1
i1 i2 i3 α αα αi δδδδi
15 10 5
20
Parameter Pelatihan Jaringan (αααα dan δδδδ) )
ID
Pembentukan Model Taksonometri Ordo
Model taksonometri ordo dibentuk dari ukuran neuron output sebanyak 17 dengan ukuran lebar tetangga awal, laju pembelajaran, dan iterasi maksimum seperti yang dijelaskan pada metode penelitian. Grafik IDB yang diperoleh dari 27 percobaan disajikan pada Gambar 9, sedangkan data lengkapnya dapat diamati pada Lampiran 11.
Gambar 9 IDB pada percobaan model takso-nometri ordo.
Untuk model taksonometri ordo, Indeks Davies-Bouldin minimum yang bernilai 2.0514 didapatkan pada percobaan ketujuh di iterasi ketiga (i3). Parameter yang digunakan adalah ukuran lebar tetangga awal (δi) sebesar 5 dan nilai laju pembelajaran awal (αi) sebesar 0.9. Dengan demikian, analisis taksonometri untuk tingkatan taksa ordo akan menggunakan bobot yang dihasilkan oleh jaringan SOM Kohonen pada percobaan ini sebagai model taksonometri ordo. Ilustrasi model ini terdapat pada Lampiran 12, dan daftar bobot akhir dapat diamati pada Lampiran 13.
Pembentukan Model Taksonometri Subclass
Sebanyak 27 percobaan dilakukan untuk mengombinasikan berbagai parameter yang telah ditetapkan dalam pembentukan model taksonomi subclass. Hasil dari percobaan ini adalah grafik IDB pada Gambar 10, dan data lengkapnya dapat diamati pada Lampiran 14.
Pada Gambar 10 dapat diamati adanya beberapa IDB yang bernilai jauh lebih kecil dibandingkan yang lainnya, yaitu berkisar antara 1.5 – 1.7, sementara nilai IDB yang lainnya berkisar antara 3 – 4. Pemeriksaan terhadap hasil clustering data pada jaringan SOM Kohonen yang memiliki IDB sangat kecil menunjukkan adanya underfitting, yaitu kegagalan jaringan untuk melakukan clustering terhadap data dengan baik akibat
Gambar 10 IDB pada percobaan model takso-nometri subclass.
menurunnya kemampuan jaringan dalam melakukan generalisasi terhadap data (Palit & Popovic 2005). Pada kasus ini, jaringan tersebut gagal melakukan pengelompokan data ke dalam empat cluster yang tersedia secara merata, karena didapatkan adanya jumlah data pada satu cluster yang mencapai 80% dari data keseluruhan, yang berakibat pada buruknya hasil clustering secara keseluruhan.
Dengan demikian, nilai IDB terbaik didapatkan dari seleksi pada jaringan yang tidak mengalami underfitting. Diperoleh nilai IDB minimum sebesar 3.1228 pada percobaan kedua di iterasi ketiga (i3), dengan parameter ukuran lebar tetangga awal (δi) sebesar 3 dan nilai laju pembelajaran awal (αi) sebesar 0.5. Bobot yang dihasilkan oleh jaringan ini akan digunakan sebagai model pada analisis taksonometri untuk tingkatan taksa subclass. Ilustrasi model ini dapat diamati pada Lampiran 15, sedangkan daftar bobot lengkapnya terdapat pada Lampiran 16. Analisis Taksonometri
Ketiga model taksonometri family, ordo, dan subclass selanjutnya digunakan untuk melakukan clustering terhadap seluruh data spesimen daun. Setiap model taksonometri diharapkan akan menghasilkan cluster yang dapat merepresentasikan kesamaan ciri yang menyerupai tingkatan taksa model tersebut seperti yang ditetapkan oleh para pakar. • Evaluasi Cluster Recall dan Precision
Perhitungan nilai CP dan CR dilakukan untuk hasil clustering setiap tingkatan taksa untuk mengetahui efektivitas pengelompokan yang telah dilakukan. Pada ketiga model taksonometri, nilai CP diperoleh dari setiap cluster, sedangkan nilai CR didapatkan dari setiap tingkatan taksa. Rata-rata nilai CP dan CR disajikan pada Tabel 11.
1.5 2 2.5 3
0.9 0.5 0.1 0.9 0.5 0.1 0.9 0.5 0.1
i1
i2
i3
15 10 5
α αα αi δδδδi
1
Parameter Pelatihan Jaringan (αααα dan δδδδ)
ID B 1 1.5 2 2.5 3 3.5 4
0.9 0.5 0.1 0.9 0.5 0.1 0.9 0.5 0.1
i1
i2
i3
3 2 1
α αα αi δδδδi
Parameter Pelatihan Jaringan (αααα dan δδδδ)
ID
Tabel 11 Nilai CP dan CR untuk ketiga model taksonometri.
Model
Taksonometri CP CR
Family 0.601 0.636
Ordo 0.596 0.626
Subclass 0.691 0.669
Rata-rata 0.629 0.644
Nilai ideal dari CP dan CR adalah 1, yang berarti setiap cluster tepat terisi oleh satu taksa, dan setiap taksa tepat mengisi satu cluster. Pada kondisi ideal, CP dan CR yang bernilai 1 menunjukkan bahwa kesesuaian model taksonometri dengan hasil taksonomi dari para pakar bernilai 100%.
Dari Tabel 11 didapatkan rata-rata nilai CP untuk seluruh model taksonometri adalah 0.629, sedangkan rata-rata nilai CR sebesar 0.644. Dengan demikian, dapat ditarik kesimpulan bahwa model taksonometri yang dibangun pada penelitian ini belum mencapai tingkat kesesuaian 100% dengan hasil taksonomi dari para pakar.
Salah satu penyebab kurang optimalnya tingkat kesesuaian ini terdapat pada metode yang digunakan, antara lain representasi data input dan fungsi jarak. Penggunaan fungsi jarak Mahalanobis diperkirakan belum dapat menggambarkan secara tepat jarak antar vektor dengan representasi data yang digunakan dalam penelitian ini. Jarak Mahalanobis pada umumnya dapat digunakan secara optimal pada data kontinu. Untuk tipe data yang digunakan pada penelitian ini, pengukuran jaraknya dapat diperbaiki dengan cara menggunakan pengukuran similaritas, yang lebih memperhatikan tingkat kesamaan antara dua vektor dibandingkan dengan perbedaannya (Pedrycz 2005).
Penentuan target clustering yang disesuaikan dengan jumlah taksa pada model taksonometri juga berperan dalam nilai CP dan CR yang diperoleh. Dari target awal yang ditetapkan pada ruang lingkup penelitian ini, diperoleh kesimpulan bahwa setiap taksa tidak dapat tercluster dengan tepat ke cluster yang berbeda. Di lain pihak, apabila target clustering dibebaskan, maka terdapat kemungkinan bahwa akan ditemukan model taksonometri pada setiap tingkatan taksa yang menghasilkan nilai CP dan CR yang lebih baik, dengan jumlah cluster yang berbeda dengan jumlah taksa pada awalnya.
Apabila ditinjau dari sisi ilmu taksonomi, kurang optimalnya hasil clustering juga disebabkan oleh penggunaan karakter tumbuhan pada penelitian ini yang terbatas pada morfologi daun, sementara taksonomi yang dilakukan oleh para pakar menggunakan seluruh karakter yang terdapat pada tumbuhan. Meskipun demikian, tingkat kesesuaian ini menunjukkan bahwa karakter morfologi daun memiliki potensi untuk digunakan dalam identifikasi dan klasifikasi tumbuhan.
• Representasi Pengetahuan
Representasi pengetahuan didapatkan dari analisis terhadap hasil clustering yang dilakukan pada taksonometri family, ordo, dan subclass. Pembahasan terhadap hasil taksono-metri akan dilakukan untuk setiap model taksonometri. Representasi pengetahuan secara umum akan diperoleh dari kesimpulan terhadap pembahasan tersebut.
Hasil clustering untuk setiap model disajikan dalam dua tabel, yaitu tabel deskripsi cluster dan tabel deskripsi taksa. Tabel deskripsi cluster mencakup jumlah anggota setiap cluster, persentasenya terhadap jumlah data secara keseluruhan, dan nilai CP untuk cluster tersebut. Nilai CP didapatkan dari banyaknya anggota taksa yang secara dominan mengisi cluster tersebut dibagi dengan banyaknya anggota cluster. Cluster dengan nilai CP sebesar 1 berarti cluster tersebut tepat terisi oleh satu taksa.
Tabel deskripsi taksa menjelaskan mengenai jumlah anggota taksa, jumlah cluster yang terisi oleh taksa tersebut (sebaran cluster), dan nilai CR untuk taksa tersebut. Nilai CR diperoleh dari banyaknya anggota taksa tersebut yang secara dominan mengisi salah satu cluster dibagi dengan banyaknya anggota taksa tersebut. Nilai CR sebesar 1 pada suatu taksa memiliki arti bahwa taksa tersebut tepat mengisi satu cluster.
Taksonometri Family
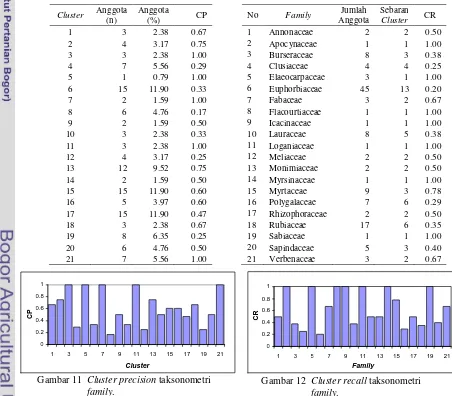
Gambar 11 dan 12 memperlihatkan diagram batang untuk nilai CP dan CR setiap cluster dan family pada hasil taksonometri ini. Dari kedua gambar tersebut, dapat diamati bahwa terdapat beberapa cluster dan family yang memiliki nilai CP dan CR yang tinggi, sementara yang lainnya memiliki nilai yang cukup rendah.
Pada Gambar 11 dapat diamati bahwa sebanyak 5 dari 21 cluster memiliki nilai CP sebesar 1, yang artinya cluster tersebut terisi oleh tepat satu family. Meskipun demikian, pengamatan pada Lampiran 17 menunjukkan bahwa diantara kelima cluster tersebut, terdapat cluster 3, 5, dan 11 yang diisi oleh family yang sama, yaitu Euphorbiaceae. Seperti yang ditunjukkan pada Tabel 13 dan Gambar 12, family ini memiliki jumlah
anggota terbesar, namun ia juga tersebar ke banyak cluster sehingga ia memiliki nilai CR yang terendah diantara family yang lain. Hal ini menunjukkan adanya variasi morfologi daun yang cukup tinggi pada family ini, sehingga tidak seluruhnya dapat terkelompok menjadi satu cluster.
Family Euphorbiaceae juga dominan pada cluster dengan anggota terbanyak, yaitu cluster 6, 15, dan 17. Dari Tabel 12 dapat diamati bahwa ketiga cluster ini memiliki anggota terbanyak, tetapi nilai CP ketiganya hanya berkisar antara 0.3-0.6. Family lainnya yang juga dominan di ketiga cluster ini adalah family Rubiaceae, seperti yang ditunjukkan pada Lampiran 17. Terkelompoknya kedua family ini secara dominan pada beberapa cluster yang sama memperlihatkan adanya kesamaan dalam morfologi daun keduanya.
Tabel 12 Deskripsi cluster hasil taksonometri family.
Cluster Anggota
(n)
Anggota
(%) CP
1 3 2.38 0.67
2 4 3.17 0.75
3 3 2.38 1.00
4 7 5.56 0.29
5 1 0.79 1.00
6 15 11.90 0.33
7 2 1.59 1.00
8 6 4.76 0.17
9 2 1.59 0.50
10 3 2.38 0.33
11 3 2.38 1.00
12 4 3.17 0.25
13 12 9.52 0.75
14 2 1.59 0.50
15 15 11.90 0.60
16 5 3.97 0.60
17 15 11.90 0.47
18 3 2.38 0.67
19 8 6.35 0.25
20 6 4.76 0.50
21 7 5.56 1.00
Tabel 13 Deskripsi taksa hasil taksonometri family.
No Family Jumlah
Anggota
Sebaran
Cluster CR
1 Annonaceae 2 2 0.50
2 Apocynaceae 1 1 1.00
3 Burseraceae 8 3 0.38
4 Clusiaceae 4 4 0.25
5 Elaeocarpaceae 3 1 1.00
6 Euphorbiaceae 45 13 0.20
7 Fabaceae 3 2 0.67
8 Flacourtiaceae 1 1 1.00
9 Icacinaceae 1 1 1.00
10 Lauraceae 8 5 0.38
11 Loganiaceae 1 1 1.00
12 Meliaceae 2 2 0.50
13 Monimiaceae 2 2 0.50
14 Myrsinaceae 1 1 1.00
15 Myrtaceae 9 3 0.78
16 Polygalaceae 7 6 0.29
17 Rhizophoraceae 2 2 0.50
18 Rubiaceae 17 6 0.35
19 Sabiaceae 1 1 1.00
20 Sapindaceae 5 3 0.40
21 Verbenaceae 3 2 0.67
Gambar 11 Cluster precision taksonometri family.
Gambar 12 Cluster recall taksonometri family. 0 0.2 0.4 0.6 0.8 1
1 3 5 7 9 11 13 15 17 19 21
Family C R C P 0 0.2 0.4 0.6 0.8 1
1 3 5 7 9 11 13 15 17 19 21
Diantara cluster dengan CP tertinggi, hanya satu cluster yang memiliki jumlah anggota lebih dari lima, yaitu cluster 21. Lampiran 17 menunjukkan bahwa tujuh dari anggota family Myrtaceae merupakan anggota cluster tersebut, sementara dua sisanya tersebar ke cluster lain. Dari Tabel 13 diperoleh bahwa family ini memiliki nilai CR sebesar 0.78, yang merupakan nilai CR terbesar pada family dengan jumlah anggota lebih dari lima. Dapat diamati bahwa family ini memiliki variasi morfologi daun yang relatif rendah hingga sebagian besarnya dapat terkelompok ke dalam satu cluster.
Pada umumnya, cluster lainnya terisi oleh family yang beragam, baik pada cluster dengan jumlah anggota kurang dari lima ataupun lebih. Misalnya, cluster 14 memiliki dua anggota yang berasal dari family Lauraceae dan Myrtaceae. Cluster dengan nilai CP terendah, yaitu cluster 8, terisi oleh 6 anggota dari 6 family yang berbeda, seperti terlihat pada Gambar 11 dan Lampiran 17.
Gambar 12 menunjukkan bahwa terdapat 7 dari 21 family dengan nilai CR sebesar 1, berarti family tersebut tepat mengisi satu cluster. Akan tetapi hal ini tidak cukup menunjukkan kinerja model taksonometri ini, karena dapat dilihat pada Tabel 13 bahwa 6 dari 7 family tersebut hanya memiliki satu anggota, dan Lampiran 17 menunjukkan bahwa cluster-cluster yang diisi oleh keenam family ini juga terisi oleh family lainnya, yang membuat nilai CP mereka tidak maksimal. Taksonometri Ordo
Diagram nilai CP dan CR dari hasil taksonometri ordo disajikan pada Gambar 13 dan 14. Dari 17 cluster hasil taksonometri ini, terdapat enam cluster dengan CP bernilai 1, sementara satu cluster tidak memiliki anggota, yaitu cluster 15. Dari keenam cluster dengan nilai CP tertinggi, tiga diantaranya yaitu cluster 6, 9, dan 12 terisi oleh ordo yang sama, yaitu ordo Euphorbiales. Tabel 15
menunjukkan bahwa meskipun jumlah
anggotanya terbanyak, ordo ini memiliki nilai
Tabel 14 Deskripsi cluster hasil taksonometri ordo.
Cluster Anggota
(n)
Anggota
(%) CP
1 2 1.59 0.50
2 4 3.17 0.25
3 23 18.25 0.35
4 5 3.97 0.60
5 7 5.56 0.43
6 3 2.38 1.00
7 4 3.17 0.50
8 3 2.38 1.00
9 6 4.76 1.00
10 8 6.35 0.25
11 23 18.25 0.17
12 26 20.63 0.58
13 3 2.38 1.00
14 2 1.59 0.50
15 0 0.00 0.00
16 5 3.97 1.00
17 2 1.59 1.00
Tabel 15 Deskripsi taksa hasil taksonometri ordo.
No Ordo Jumlah
Anggota
Sebaran
Cluster CR
1 Celastrales 1 1 1.00
2 Euphorbiales 45 12 0.33
3 Fabales 3 1 1.00
4 Gentianales 2 2 0.50
5 Lamiales 3 2 0.67
6 Laurales 10 5 0.30
7 Magnoliales 2 2 0.50
8 Malvales 3 1 1.00
9 Myrtales 9 4 0.56
10 Polygalales 7 4 0.43
11 Primulales 1 1 1.00
12 Ranunculales 1 1 1.00
13 Rhizophorales 2 2 0.50
14 Rubiales 17 4 0.41
15 Sapindales 15 7 0.20
16 Theales 4 4 0.25
17 Violales 1 1 1.00
Gambar 13 Cluster precision taksonometri ordo.
Gambar 14 Cluster recall taksonometri ordo. 0 0.2 0.4 0.6 0.8 1
1 3 5 7 9 11 13 15 17
Cluster C P 0 0.2 0.4 0.6 0.8 1
1 3 5 7 9 11 13 15 17
Ordo
C
CR yang rendah karena tersebar ke 12 cluster. Nilai CR sebesar 0.33 yang dimilikinya cukup rendah dibandingkan dengan ordo yang lain, seperti terlihat pada
Gambar 14. Seperti halnya family
Euphorbiaceae, dapat disimpulkan bahwa ordo ini juga memiliki variasi morfologi daun yang cukup tinggi.
Cluster 16 dan 17 yang juga memiliki nilai CP sebesar 1 terisi oleh ordo Myrtales, yang pada taksonometri family juga menempati satu cluster yang sama. Pengamatan pada Tabel 15 menunjukkan bahwa ordo ini memiliki nilai CR sebesar 0.58. Meskipun pada Gambar 14 dapat diamati bahwa nilai ini cukup rendah dibandingkan dengan nilai keseluruhan, tetapi nilai ini adalah yang terbesar diantara ordo lainnya dengan jumlah anggota lebih dari lima. Dengan demikian, dapat disimpulkan bahwa variasi morfologi daun pada ordo Myrtales cukup rendah, sehingga dapat terkelompok ke dalam cluster yang berbeda.
Nilai CP terendah adalah 0.17 yang terdapat pada cluster 11, yang juga merupakan salah satu cluster dengan jumlah anggota terbanyak, seperti terlihat pada Tabel 14. Selain cluster 11, jumlah anggota terbanyak juga terdapat pada cluster 3 dan 12. Ketiga cluster ini diisi oleh beragam ordo, tetapi seperti yang terdapat pada taksonometri family, secara umum ketiga cluster tersebut masih didominasi oleh ordo Euphorbiales dan Rubiales. Seperti halnya pada taksonometri family, terkelompoknya kedua ordo ini ke dalam cluster yang sama menguatkan kesimpulan bahwa terdapat kesamaan morfologi daun yang cukup tinggi pada kedua ordo tersebut.
Tabel 14 juga menunjukkan bahwa jumlah cluster yang memiliki anggota kurang dari lima masih cukup besar. Terdapat 9 dari 17 cluster yang termasuk ke dalam kategori ini. Salah satu cluster yang mempertahankan konsistensi anggotanya adalah cluster 14 yang diisi oleh ordo Laurales dan Myrtales, sama halnya dengan hasil dari taksonometri family.
Pada Gambar 14 dapat diamati bahwa sebanyak 6 dari 17 ordo memiliki nilai CR sebesar 1. Akan tetapi, keenamnya memiliki jumlah anggota kurang dari lima. Di sisi lain, nilai CR terendah terdapat pada ordo Sapindales, yaitu sebesar 0.20. Hal ini dikarenakan ordo Sapindales yang anggotanya berjumlah 15 tersebar ke 7 cluster, yang menunjukkan tingginya variasi morfologi
daun pada ordo ini, seperti halnya ordo Euphorbiales dan Laurales pada Tabel 15. Taksonometri Subclass
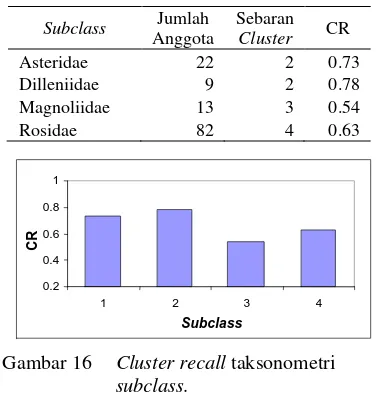
Taksonometri subclass menghasilkan empat buah cluster dengan nilai CP dan CR yang beragam, seperti ditunjukkan pada Gambar 15 dan 16. Keempat cluster ini memiliki karakteristik yang unik dan dapat memberikan informasi mengenai hubungan dari morfologi daun antara beberapa subclass, ordo, dan family pada class Magnoliopsida.
Gambar 15 menunjukkan bahwa nilai CP tertinggi terdapat pada cluster 1. Dari Tabel 16 dapat diamati bahwa cluster ini diisi oleh tujuh anggota, dan dari Lampiran 12 diperoleh bahwa ketujuhnya berasal dari subclass Rosidae. Penelusuran pada data menunjukkan bahwa ketujuh anggota tersebut berasal dari ordo Myrtales dan family Myrtaceae. Lebih jauh lagi, enam dari tujuh anggota cluster 1 berasal dari satu genus yang sama, yaitu genus Syzygium. Genus ini memiliki jumlah species terbesar dari seluruh data spesimen daun, yaitu 7 dari 86 species, atau sama dengan 8.14% dari seluruh data.
Cluster 2 yang memiliki CP terendah hanya diisi oleh dua anggota, yaitu dari subclass Magnoliidae dan Rosidae, seperti ditunjukkan pada Lampiran 12. Kedua anggota ini berasal dari ordo Laurales dan Myrtales, yang juga menempati satu cluster pada taksonometri ordo, yaitu cluster 14. Pada tingkatan taksa family, kedua anggota ini berasal dari family Lauraceae dan Myrtaceae, yang juga menempati satu cluster pada taksonometri family, yaitu cluster 14. Model taksonomi menempatkan keduanya secara konsisten ke dalam cluster yang sama. Dengan demikian, SOM Kohonen mengung-kapkan adanya kedekatan karakter morfologi daun antara kedua jenis tanaman ini, meskipun secara taksonomi keduanya berbeda subclass, ordo, dan family.
Tabel 16 Deskripsi cluster hasil taksonometri subclass.
Cluster Anggota
(n)
Anggota
(%) CP
1 7 5.56 1.00
2 2 1.59 0.50
3 35 27.78 0.63
4 82 65.08 0.63
Tabel 17 Deskripsi taksa hasil taksonometri subclass.
Subclass Jumlah
Anggota
Sebaran
Cluster CR
Asteridae 22 2 0.73
Dilleniidae 9 2 0.78
Magnoliidae 13 3 0.54
Rosidae 82 4 0.63
Gambar 15 Cluster precision taksonometri subclass.
Gambar 16 Cluster recall taksonometri subclass.
Sebagian kecil dari anggota cluster 3 dan 4 juga berasal dari kedua subclass lainnya, yaitu Dilleniidae dan Magnoliidae. Meskipun kedua subclass ini memiliki jumlah anggota yang relatif sedikit, dikelompokkan-nya mereka ke dalam cluster yang sama dengan subclass Asteridae dan Rosiidae menunjukkan adanya kesamaan morfologi daun yang cukup tinggi diantara keempat subclass ini, yang menyebabkan sulitnya memisahkan mereka secara sepenuhnya ke dalam empat cluster sesuai dengan subclass masing-masing.
Tabel 17 bersama dengan Gambar 16 mengungkapkan tinggi atau rendahnya variasi morfologi daun pada setiap subclass. Subclass Magnoliidae memiliki variasi morfologi daun yang relatif tinggi, ditunjukkan dengan nilai CR yang terendah diantara subclass lainnya. Nilai CR yang cukup tinggi yang dimiliki subclass Dilleniidae dan Asteridae dapat diartikan bahwa kedua subclass ini memiliki variasi morfologi daun yang cukup rendah. Subclass Rosidae dengan jumlah anggota terbesar ternyata memiliki variasi morfologi daun yang cukup tinggi diantara anggotanya, hal ini ditunjukkan dengan tersebarnya anggota subclass Rosidae ke dalam keempat cluster hasil taksonometri subclass.
Representasi Umum
Ketiga model taksonometri yang dibangun tidak dapat memisahkan sepenuhnya setiap tingkatan taksa ke dalam cluster-<