KARAKTERISASI
ISOLAT BAKTERI PENGHASIL FITASE
ASAL KUTU JAGUNG (Sitophilus zeamays)
ROSWITA AMELIA
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa Tesis yang berjudul Karakterisasi Isolat Bakteri Penghasil Fitase Asal Kutu Jagung (Sitophilus zeamays) adalah karya saya dengan arahan dari komisi pembimbing. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir Tesis ini.
Bogor, Agustus 2010
ABSTRACT
ROSWITA AMELIA. Characterization of Phytase Producing Bacteria from Corn Weefil (Sitophilus zeamays). Under direction of ANTONIUS SUWANTO and ARIS TRIWAHYUDI
Phytic acid, the main storage form of phosphorus in seed, could not be digested by monogastric animal. The negatively charge phosphate of this compound strongly bind to metallic cations, proteins and polysaccharides making them insoluble and unavailable as nutritional factors. This compound can be hydrolyzed by phytases to released phosphate, inositol and nutrients. In the study about community of corn weevil bacteria some phytase producing bacterial isolates have been isolated. In this study, four of these isolates have been characterized. Morphological and physiological identification using microbact showed that all four isolates have 99% similarity to Serratia rubidaea. Partial amplification of their 16S rRNA gene also showed highest identity to S. rubidaea
(84%, 93%, 98%, and 80% respectively for KJ07, KJ13, KJ14, and KJ16). Employing from these result we proposed that all isolates were closely related to
S. rubidaea. Using primers for phytase putative gene designed from Escherichia coli phytase, a 1500 bp DNA fragment was amplified. One of these fragments originated from KJ07 was chosen for further analysis. This fragment was cloned in E. coli and sequenced. Sequence analysis indicated that it showed similarity to phosphoenol piruvate protein phospho transferase (89%) and prolipoprotein diacylglycerol transferase (93%). Therefore, this strategy failed to isolate phytase gene from S. rubidaea.
RINGKASAN
ROSWITA AMELIA. Karakterisasi Isolat Bakteri Penghasil Fitase Asal Kutu Jagung (Sitophilus zeamays). Dibimbing oleh ANTONIUS SUWANTO dan ARIS TRIWAHYUDI
Asam fitat merupakan komponen esensial pada semua biji sebagai bentuk penyimpanan fosfor yang utama. Akan tetapi, senyawa ini tidak dapat dicerna oleh manusia dan hewan-hewan monogastrik. Senyawa ini juga dapat menurunkan kualitas bahan makanan karena bisa membentuk kompleks tidak larut dengan kation logam protein, vitamin, dan polisakarida. Kompleks yang tidak dapat dicerna tersebut akan dikeluarkan melalui feces dan bila mengalami penumpukan akan menyebabkan pencemaran lingkungan.
Asam fitat dapat diuraikan menjadi mioinositol dan gugus fosfat melalui serangkaian reaksi enzimatis oleh enzim fitase. Fitase (mioinositol heksakisfosfat fosfohidrolase) adalah enzim fosfatase yang bekerja pada ikatan ester (phosphoric monoester hydrolase) yang memotong gugus fosfat dari asam fitat. Pentingnya peranan enzim fitase dalam mengatasi masalah yang berkaitan dengan asam fitat memacu para ahli untuk melakukan berbagai penelitian yang berkaitan dengan hal ini. Berbagai organisme dari taksonomi yang berbeda-beda dieksplorasi untuk diuji kemampuannya dalam memproduksi fitase termasuk bakteri. Hal ini disebabkan dunia bakteri menempati habitat yang sangat luas bahkan termasuk daerah ekstrim sehingga sangat memungkinkan ditemukannya anggota organisme kelompok ini di daerah yang banyak mengandung fitat dan memanfaatkan senyawa tersebut sebagai sumber fosfor satu-satunya.
Yogiara et al. (2008) telah berhasil menapis sejumlah isolat bakteri penghasil fitase yang berasal dari kutu jagung (Sitophilus zeamays). Pada penelitian ini empat dari isolat-isolat tersebut diidentifikasi. Berdasarkan hasil pengamatan diketahui bahwa keempat isolat bakteri tersebut yaitu KJ07, KJ13, KJ14, dan KJ16 mempunyai koloni yang berwarna krem, berbentuk bundar, dengan elevasi cembung, dan tepian licin. Setelah dilakukan pewarnaan gram dan pengamatan di bawah mikroskop, diketahui bahwa sel-selnya berbentuk coccus, motil, dengan tipe dinding gram negatif. Kemudian dari hasil uji katalase dan oksidase diketahui bahwa isolat-isolat tersebut termasuk ke dalam kelompok bakteri anaerob fakultatif. Berdasarkan data-data ini, selanjutnya dilakukan serangkaian uji fisiologis menggunakan microbact™ tipe 12A+12B. Setelah dilakukan analisis menggunakan microbact™ computer aided identification package didapatkan probabilitas isolat-isolat tersebut sebesar 99% terhadap
Serratia rubidaea. Sementara itu, hasil amplifikasi parsial dari sekuen gen penyandi 16S rRNAnya, menunjukkan % identitas sebesar 84%, 93%, 98%, 80% secara berturut-turut dari isolat KJ07, KJ13, KJ14, KJ16 terhadap S. rubidaea. Oleh sebab itu, diusulkan bahwa keempat isolat tersebut berkerabat dekat dengan
S. rubidaea.
filogenetik ini dapat terlihat bahwa isolat KJ07 memiliki hubungan kekerabatan yang lebih dekat dengan KJ16 daripada KJ13 dan KJ14. Sementara itu empat galur S. rubidaea berada pada posisi yang berbeda-beda tapi tetap dekat dengan keempat isolat tersebut. Kemudian apabila dilihat dari skala jaraknya terlihat bahwa bakteri-bakteri endosimbion kutu jagung ini setelah S. rubidaea lebih dekat terhadap bakteri-bakteri endosimbion serangga lainnya yang ada di database seperti endosimbion dari S. zeamays, Curculio sikkimensis, Glossina palpalis, dan Sodalis glossinidius dari pada bakteri-bakteri enterik dan gram negatif lainnya dan sangat jauh dengan bakteri gram positif yang diwakili oleh Bacillus subtilis.
Salah satu cara untuk mengetahui kekhasan enzim fitase yang dihasilkan oleh suatu organisme adalah dengan mengisolasi gen yang menyandikannya. Pada penelitian ini deteksi gen putatif penyandi fitase dilakukan menggunakan PCR. Setelah dilakukan amplifikasi parsial terhadap sekuen gen yang diduga penyandi fitase dari keempat isolat bakteri asal kutu jagung didapatkan pita DNA berukuran 1500 bp. Kemudian salah satu dari keempat fragmen DNA ini yaitu yang berasal dari isolat KJ07 dipilih untuk analisis lebih lanjut. Fragmen DNA ini disisipkan pada pCR®2.1TOPO® kemudian diintroduksikan pada E. coli TOP 10. Koloni transforman dianalisis menggunakan PCR dengan primer M13f dan M13r. Hasil amplifikasi menunjukkan pita berukuran 1700 bp yang merupakan gabungan dari fragmen sisipan yang berukuran 1500 bp dan DNA pengapitnya pada plasmid rekombinan. Verifikasi plasmid rekombinan juga dilakukan dengan cara mengisolasi plasmid dari bakteri transforman lalu memotongnya dengan enzim restriksi EcoRI. Hasil pemotongan menunjukan dua pita yaitu yang berukuran 3.9 kb (vektor) dan 1.5 kb (DNA sisipan). Plasmid rekombinan ini kemudian digunakan dalam proses sekuensing untuk menentukan urutan sekuen DNA dari fragmen 1500bp. Setelah dianalisis dengan blastx didapatkan % kemiripan tertinggi terhadap phosphoenolpiruvate phosphotransferase (89%) dan prolipoprotein diacylglycerol transferase (93%). Oleh sebab itu, dapat disimpulkan bahwa strategi ini tidak memungkinkan untuk mendapatkan gen penyandi fitase dari S. rubidaea.
@Hak Cipta milik IPB tahun 2010
Hak Cipta dilindungi Undang-undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah
b. Pengutipan tidak merugikan kepentingan yang wajar IPB
KARAKTERISASI
ISOLAT BAKTERI PENGHASIL FITASE
ASAL KUTU JAGUNG (Sitophilus zeamays)
ROSWITA AMELIA
Tesis
Sebagai salah satu syarat untuk memperoleh gelar
Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Karakterisasi isolat bakteri penghasil fitase asal kutu jagung (Sitophilus zeamays)
Nama : Roswita Amelia
NIM : P052060081
Disetujui, Komisi Pembimbing
Prof. Dr. Antonius Suwanto Dr. Aris Triwahyudi
Ketua Anggota
Diketahui,
Ketua Program Studi Bioteknologi Dekan Sekolah Pascasarjana IPB
Dr. Suharsono Prof. Dr. Ir. Khairil Anwar Notodiputro, MS
PRAKATA
Alhamdulillaahirabbil’alamin. Segala puji bagi Allah SWT yang telah memberikan kesempatan kepada penulis untuk meneyelesaiakn Tesis yang berjudul Karakterisasi Isolat Bakteri Penghasil Fitase Asal Kutu Jagung (Sitophilus zeamays). Tulisan ini merupakan laporan dari rangkaian penelitian yang dilaksanakan sejak bulan Juni 2009 sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada Program Studi Bioteknologi, SPs, IPB.
Terima kasih penulis ucapkan kepada Bapak Prof. Dr. Antonius Suwanto sebagai pembimbing I dan Bapak Dr. Aris Triwahyudi sebagai pembimbing II, serta Bapak Dr. Iman Rusmana atas saran-sarannya sebagai penguji luar komisi. Selain itu, penulis juga mengucapkan terima kasih kepada Kak Yogiara Msi atas bantuan dan saran-sarannya selama proses penelitian dan juga kepada Ibu Prof. Dr. Maggy T. Suhartono yang telah memberikan kesempatan kepada penulis untuk melaksanakan sebagian percobaan penelitian di Laboratorium Mikrobiologi dan Biokimia, PPSHB, IPB.
Ucapan terima kasih juga penulis sampaikan pada teman-teman di Laboratorium Mikrobiologi dan Biokimia diantaranya Ibu Ika, Ibu Eni, Pak Ace, Agus, dan teman-teman lainnya di lingkungan PPSHB, IPB, begitu juga kepada teman-teman di Laboratorium Riset dan Teknologi DNA, Fakultas Teknobiologi, Unika Atmajaya diantaranya Arhat, Alvin, Mesa, Pak Bambang, Nurdin, Ridwan, Yuni, Mbak Nouri dan lainnya yang terlalu panjang untuk disebutkan satu per satu atas bantuan dan dukungannya, serta teman-teman PS Bioteknologi angkatan 2006 dan 2007 yang telah memberikan semangat dan dorongan untuk menyelesaikan studi. Kemudian penghargaan penulis berikan kepada Papa, Mama, dan kakak-kakak atas segala do’a dan dukungannya sehingga penulis berhasil menyelesaikan Tesis ini. Akhir kata, semoga karya ini bermanfaat bagi dunia ilmu pengetahuan.
Bogor, Agustus 2010
RIWAYAT HIDUP
Penulis dilahirkan di Bukittinggi pada tanggal 7 Desember 1978 dari pasangan Rusli dan Djusmaniar sebagai anak ke 7 dari 7 bersaudara. Pada tahun 1997 penulis diterima sebagai mahasiswi di Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor melaui jalur Undangan Seleksi Masuk IPB (USMI) dan lulus pada bulan April 2002.
Pada bulan Juli 2002 penulis diterima sebagai pegawai honorer di Laboratorium Biologi Molekuler, SEAMEO BIOTROP. Kemudian pada bulan Januari 2003 dpenulis dikontrak sebagai Asisten Laboratorium di tempat yang sama. Pada tahun 2004 penulis menjadi staf di Laboratorium Bioteknologi Tanaman dan Laboratorium Servis di instansi yang sama. Selama bekerja penulis terlibat dalam berbagai proyek penelitian di bidang mikrobiologi dan bioteknologi, serta aktif mengikuti seminar dan pelatihan di bidang yang sama.
Pada bulan September 2006 penulis diterima sebagai mahasiswi di Program Studi Bioteknologi, Sekolah Pascasarjana IPB. Oleh sebab itu, pada tahun 2007 penulis tidak memperpanjang kontrak sebagai pegawai di SEAMEO BIOTROP dan fokus pada studi. Penulis pernah mengalami masa non aktif sebagai mahasiswi SPs selama 2 semester yaitu pada semester genap tahun 2007/2008 dan semester ganjil tahun 2008/2009 karena sakit.
DAFTAR ISI
Asam fitat, bioavailabilitas dan pencemaran lingkungan ... 5
Fitase dan klasifikasinya ... 7
Bakteri penghasil fitase ... 8
BAHAN DAN METODE ... 11
Isolat dan media pertumbuhan bakteri... ... 11
Pengamatan morfologi bakteri... ... 11
Pengamatan sifat-sifat fisiologis ... 12
Isolasi DNA genom ... 13
Amplifikasi gen penyandi 16S rRNA ... 13
Amplifikasi gen penyandi fitase... 14
Kloning gen penyandi fitase ... 14
Verifikasi transforman ... 15
Verifikasi plasmid rekombinan ... 15
Penentuan urutan sekuen DNA ... 17
Analisis sekuen DNA ... 17
HASIL DAN PEMBAHASAN ... 19
Ciri-ciri morfologi dan fisiologi bakteri ... 19
Identifikasi menggunakan sekuen gen penyandi 16S rRNA... 21
Deteksi gen putatif penyandi fitase ... 25
KESIMPULAN DAN SARAN ... 29
DAFTAR PUSTAKA ... 31
DAFTAR TABEL
Halaman
1. Kandungan fitat pada beberapa jenis biji ... 6
2. Pengelompokan fitase berdasarkan strukturnya ... 7
3. Uji fisiologis yang tersedia pada kit microbact™ tipe 12A dan 12B ... 12
4. Ciri-ciri morfologi bakteri ... 19
5. Ciri-ciri fisiologi bakteri ... 20
6. Hasil identifikasi dengan microbact™ ... 21
7. Hasil identifikasi berdasarkan sekuen gen penyandi 16S rRNA ... 22
DAFTAR GAMBAR
Halaman
1. Struktur asam fitat ... 5
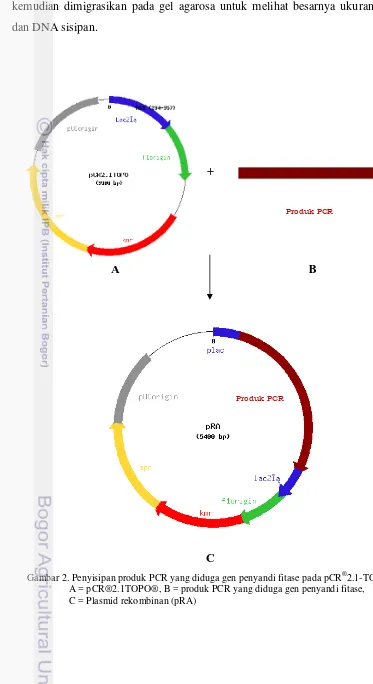
2. Penyisipan gen penyandi fiase pada pCR®2.1-TOPO ... 16
3. Pohon filogenetik hubungan kekerabatan keempat isolat bakteri asal kutu jagung dan bakteri-bakteri lainnya ... 24
4. Hasil amplifikasi sekuen gen putatif penyandi fitase ... 25
5. Verifikasi transforman ... 26
DAFTAR LAMPIRAN
Halaman
PENDAHULUAN
Latar Belakang
Asam fitat (mio-inositol heksakisfosfat) merupakan bentuk penyimpanan fosfor yang utama pada serealia, polong-polongan, dan oil seed. Akan tetapi, senyawa ini tidak dapat dicerna oleh manusia dan hewan-hewan monogastrik seperti babi, unggas dan ikan walaupun berbagai spesies dari tanaman tersebut telah digunakan secara luas sebagai bahan makanan. Akibatnya fosfor anorganik seringkali ditambahkan pada pakan ternak untuk melengkapi kebutuhan gizi mereka. Asam fitat juga dapat menurunkan kualitas bahan makanan karena bisa membentuk kompleks tidak larut dengan kation logam (Ca, Mg, Fe, K, Zn), protein, vitamin, dan polisakarida (menurunkan bioavailibilitas bahan-bahan nutrisi tersebut) (Mullaney et al. 2000). Kompleks yang tidak dapat dicerna tersebut akan dikeluarkan melalui feces dan bila mengalami penumpukan akan menyebabkan pencemaran lingkungan (Glick & Pasternak 2003).
Asam fitat dapat diuraikan menjadi mioinositol dan gugus fosfat melalui serangkaian reaksi enzimatis oleh enzim fitase (mioinositol heksafosfat fosfohidrolase). Penguraian ini sekaligus melepaskan mineral-mineral dan senyawa-senyawa lain yang diikatnya (Bohn et al. 2008). Oleh sebab itu, enzim ini dapat digunakan sebagai suplemen pada pakan ternak dan ikan. Dengan demikian unsur P dan bahan nutrisi lainnya bisa dimanfaatkan secara optimal oleh hewan-hewan monogastrik dan tidak perlu adanya tambahan P anorganik ke dalam pakan yang pada akhirnya mengurangi polusi fosfat dari buangan hewan.
2
di bagian dalam. Telur-telur tersebut kemudian menetas lalu larva yang keluar dari telur akan memakan bagian dalam biji (PestWeb 2010). Dengan habitat yang khusus ini maka serangga tersebut kemungkinan mempunyai jalur metabolisme tertentu untuk dapat memanfaatkan asam fitat dari jagung sebagai sumber P. Kemungkinan lain kemampuan untuk memanfaatkan fosfat dari asam fitat tersebut diperoleh atas bantuan mikroorganisme penghasil fitase yang berasosiasi dengannya.
Yogiara et al. (2008) telah berhasil menapis sejumlah isolat bakteri penghasil fitase yang berasal dari kutu jagung. Untuk mengetahui kelompok taksonominya, isolat-isolat bakteri tersebut perlu diidentifikasi. Ada berbagai cara untuk melakukan identifikasi terhadap organisme yang tidak dikenal mulai dari pengamatan ciri-ciri morfologi, fisiologis maupun molekuler. Analisis secara molekuler dianggap lebih teliti dibandingkan dua analisis sebelumnya, namun untuk hasil yang lebih baik digunakan gabungan dari ketiga teknik ini. Analisis yang paling banyak digunakan saat ini (khususnya bagi prokariot) adalah identifikasi menggunakan sekuen gen penyandi rRNA 16S. Hal ini berdasarkan pemikiran bahwa molekul rRNA 16S adalah salah satu perangkat biosintesis protein dan terdapat dalam sel prokariot dan juga mitokondria dan kloroplas. Disamping itu, pada molekul ini terdapat daerah dengan sekuen konservatif dan hipervariabel. Perbandingan sekuen konservatif berguna untuk mengkonstruksi pohon filogenetik universal sedangkan sekuen hipervariabel digunakan untuk mencirikan organisme dalam takson yang lebih sempit (Madigan et al. 1997).
Mengingat beragamnya tipe fitase yang telah diketahui maka kekhasan enzim tersebut tidak cukup hanya dilihat dari aktivitasnya saja tetapi juga perlu dilakukan deteksi terhadap gen yang menyandikannya. Berbagai rangkaian metode dapat ditempuh untuk mendeteksi suatu gen. Teknik deteksi gen berdasarkan PCR seringkali menjadi alternatif yang dipilih karena preparasinya yang lebih sederhana dan lebih cepat. Sejumlah gen penyandi fitase telah berhasil diisolasi menggunakan teknik ini diantaranya adalah gen penyandi fitase dari
3
Tujuan Penelitian
TINJAUAN PUSTAKA
Asam fitat, bioavailabilitas dan pencemaran lingkungan
Asam fitat (mio inositol (1,2,3,4,5,6) heksakisfosfat) merupakan turunan gula heksosa siklik (mioinositol) dengan keenam posisi hidroksilnya (OH) diganti dengan gugus fosfat (Gambar 1). Masing-masing gugus fosfat membentuk ikatan ester pada cincin inositol dan antara gugus P yang satu dengan lainnya tidak terdapat ikatan internal. Senyawa ini mempunyai efek pengkelat karena dapat berikatan dengan kation logam membetuk garam (fitat) (Bohn et al. 2008). Selain itu pada pH asam gugus P dari asam fitat dapat berikatan dengan gugus amino dari asam amino sedangkan pada pH netral gugus karboksil dari asam amino akan berikatan dengan asam fitat melalui kation divalen. Pengikatan senyawa ini pada polisakarida juga dapat terjadi baik secara langsung maupun melalui perantara protein (Kornegay 1996).
Gambar 1 Struktur asam fitat (Wikipedia 2009).
6
Tabel 1 Kandungan fitat pada beberapa jenis biji (Kornegay, 1996)
Jenis biji P fitat (g/kg) % fitat P terhadap P total
Bunga matahari 3,2-5,1 35-47
7
hipoksia, dan kematian organisme akuatik serta produksi nitrous oksida yang menyebabkan efek rumah kaca (Madigan et al. 1997).
Fitase dan Klasifikasinya
Fitase (mioinositol heksakisfosfat fosfohidrolase) adalah enzim fosfatase yang bekerja pada ikatan ester (phosphoric monoester hydrolase) yang memotong gugus fosfat dari asam fitat. Menurut Scoglund et al. (1997), lintasan hidrosis asam fitat dimulai dengan terbentuknya mioinositol pentafosfat. Kemudian produk hidrolisis pertama ini akan berikatan kembali dengan enzim sehingga terjadi reaksi yang menghasilkan mioinositol tetrafosfat dan seterusnya sampai akhirnya menghasilkan mioinositol monofosfat.
Tabel 2 Pengelompokan fitase berdasarkan strukturnya (Lei et al 2007)
Famili enzim Struktur unik Mekanisme katalitik/adaptasi
untuk menghidrolisis fitat
fosfat/paket situs aktif yang lebih
dalam mengakomodasi fitat
dekat dengan PAP tanaman/tidak
diketahui
Glycine max,
8
Berdasarkan tempat awal pemotongan gugus fosfatnya terdapat tiga kelompok fitase yaitu 3 fitase yang memotong fosfat ke-3 dari asam fitat, 6 fitase/4 fitase yang memotong fosfat di sebelah fosfat ke-5 dari asam fitat dan 5 fitase yang memotong fosfat ke-5 dari asam fitat. Berdasarkan pH optimum aktivitasnya ada 3 kelompok fitase yaitu yang bekerja pada pH asam, netral, dan alkalin (Bohn et al. 2008). Kemudian berdasarkan strukturnya (Tabel 2) enzim fitase terdapat pada empat kelas enzim yaitu Histidin Acid Phosphatase (HAP), β
Propeller Phytase (BPP), Purple Acid Phosphatase (PAP), dan cysteine phytase
(Cphy) (Mullaney & Ullah 2003, Chu et al. 2004, Lei et al. 2007).
Bakteri Penghasil Fitase
Selain terdapat pada tumbuhan, fitat juga mencapai saluran pencernaan akibat dikonsumsi oleh hewan dan manusia, kemudian tersebar ke tanah dan daerah perairan melalui feses maupun jatuhnya bagian tumbuhan yang mengandung fitat itu sendiri. Sementara itu, dunia bakteri menempati habitat yang sangat luas bahkan termasuk daerah ekstrim. Oleh sebab itu, sangat memungkinkan ditemukannya anggota organisme kelompok ini di daerah yang banyak mengandung fitat dan memanfaatkan senyawa tersebut sebagai sumber fosfor satu-satunya. Untuk melepaskan unsur P dari senyawa ini maka bakteri-bakteri tersebut menghasilkan enzim fitase.
Dari sejumlah penelitian yang telah dilakukan akhir-akhir ini diketahui bahwa bakteri-bakteri penghasil fitase berasal dari hampir semua filum dari domain bakteria dan terutama merupakan bakteri gram negatif, walaupun sebagian yang ditemukan tergolong pada bakteri gram positif. Dan bila ditelusuri secara taksonomi bakteri-bakteri gram negatif penghasil fitase terutama termasuk ke dalam filum Proteobacteria, dan sebagian besar termasuk kelas Gammaproteobacteria. Dibandingkan kelompok taksonomi lainnya, famili Enterobacteriaceae paling banyak dipelajari dalam kaitannya menghasilkan enzim fitase. Contoh bakteri penghasil fitase pada kelompok ini adalah Escherichia coli
(Dassa et al. 1990), Citrobacter amalonaticus (Luo et al 2007), Yersinia intermedia (Huang et al. 2006), Pectobacterium wasanabe (Na et al. 2008),
9
2004). Sementara itu, bakteri penghasil fitase gram positif yang paling banyak dipelajari adalah famili Bacillaceae yang salah satu anggotanya adalah Bacillus subtilis (Kerovuo et al. 1998).
Sebagian besar bakteri penghasil fitase yang telah dipublikasi sudah dilengkapi dengan data sekuen gen penyandi enzim tersebut. Data-data tersebut dapat diakses melalui database dan dijadikan bahan perbandingan bagi suatu fitase yang baru dipelajari. Data sekuen yang tersedia dapat berupa sekuen gen yang utuh maupun parsial. Akan tetapi, sejauh ini kajian mendalam mengenai struktur gen dan mekanisme ekspresi gen fitase masih sangat sedikit. Data terbaru menunjukkan bahwa suatu gen fitase dapat berupa monosistronik seperti pada
Bacillus subtilis 168 maupun polisistronik seperti pada Bacillus amyloliquefaciens
BAHAN DAN METODE
Isolat dan Media Pertumbuhan Bakteri
Isolat-isolat bakteri penghasil fiase yang digunakan pada penelitian ini merupakan koleksi Laboratorium Riset, Fakultas Teknobiologi, Universitas Katolik Atmajaya (Yogiara et al. 2008). Untuk menumbuhkan bakteri-bakteri tersebut digunakan media Luria Berthani dengan komposisi perliternya adalah 5 g yeast extract, 10 g tripton, 10 g NaCl, dan dH2O sampai volume mencapai 1 L dan jika diperlukan media padat maka ditambahkan 20 g agar.
Pengamatan morfologi bakteri
Identifikasi awal yang dilakukan berupa pengamatan terhadap koloni bakteri yang meliputi pengamatan terhadap warna, elevasi, tepian, bentuk, dan ukurannya. Selain itu, juga dilakukan pengamatan bentuk dan penataan/pengelompokan sel serta motilitasnya menggunakan mikroskop. Untuk mengetahui tipe dinding sel masing-masing isolat bakteri dilakukan uji pewarnaan gram sesuai dengan prosedur dalam Hadioetomo (1993). Biakan bakteri yang akan diuji dioleskan pada kaca objek lalu dikeringanginkan. Untuk mempercepat proses pengeringan dibantu dengan fiksasi panas. Kemudian kaca objek tersebut diletakkan pada rak kawat. Setelah itu digenangi dengan pewarna primer ungu kristal selama 1 menit. Lalu kaca objek dimiringkan dengan pinset untuk membuang kelebihan zat warna dan dibilas dengan akuades. Sisa air yang masih menempel dihisap dengan kertas serap. Selanjutnya olesan tersebut di beri iodin selama 2 menit dengan cara yang sama. Setelah dibilas dengan akuades, olesan dicuci dengan etanol 95% setetes demi setetes selama 30 detik lalu dibilas dengan akuades. Kemudian diwarnai dengan safranin selama 30 detik dengan cara yang sama dengan pewarna pertama. Preparat yang sudah jadi lalu diamati di bawah mikroskop dan dibandingkan dengan kontrol. Sebagai kontrol bakteri gram positif digunakan Bacillus sp. Sedangkan kontrol bakteri gram negatif adalah
12
Pengamatan sifat-sifat fisiologis
Uji fisiologis awal yang dilakukan meliputi uji katalase dan uji oksidase. Uji katalase dilakukan dengan cara meletakkan satu tetes H2O2 3% pada kaca objek lalu sebanyak satu lup biakan bakteri yang diuji dicampurkan pada tetesan tersebut dengan cara mengaduknya. Hasil positif ditunjukkan dengan terbentuknya gelembung gas setelah beberapa saat. Uji oksidase dilakukan dengan cara mengoleskan satu lup koloni biakan yang diuji pada kertas strip oksidase. Hasil positif ditandai dengan terbentuknya warna biru atau hitam.
Sifat-sifat fisiologis lainnya diuji menggunakan tipe 12A + 12B dari kit microbact™ (Oxoid, UK). Sebanyak 2-3 koloni masing-masing isolat bakteri yang berumur 18-24 jam disuspensikan dalam 5 ml garam fisiologis. Kemudian masing-masing sebanyak 4 tetes suspensi dimasukkan kedalam setiap sumur kit mucrobact yang telah berisi substrat yang berbeda-beda untuk menguji reaksi fisiologis yang berbeda pula (Tabel 3). Setelah itu pada sumur nomor 1, 2, dan 3 untuk tipe 12A dan sumur nomor 8 dan 12 pada tipe 12B diberi satu tetes mineral oil. Semua sumur ditutup dan diinkubasi pada suhu ruang selama 18-24 jam. Reaksi positif atau negatif dicatat dengan membandingkannya dengan tabel warna. Pada sumur nomor 8 pada tipe 12A ditambahkan 2 tetes reagen indol lalu dilakukan pembacaan setelah 2 menit. Pada sumur nomor 10 tipe 12A ditambahkan 1 tetes reagen VP I dan 1 tetes reagen VP II lalu dilakukan pembacaan setelah 15-30 menit. Pada sumur nomor 12 dari tipe 12A ditambahkan 1 tetes reagen TDA lalu langsung dilakukan pembacaan. Setelah pembacaan pada semua sumur selesai kemudian dilakukan uji tambahan pada sumur nomor 7 dari tipe 12A yaitu uji nitrat. Uji ini dilakukan dengan penambahan 1 tetes reagen nitrat A dan 1 tetes reagen nitrat B.
Tabel 3 Uji fisiologis yang tersedia pada kit microbact™ tipe 12A dan 12B
13
Isolasi DNA Genom
DNA genom bakteri diisolasi menggunakan metode CTAB (Murray & Thomson 1980) dengan sedikit modifikasi. Bakteri ditumbuhkan pada media LB selama ± 24 jam lalu dipanen. Masing-masing biakan disentrifusi pada 6000 rpm selama 2 menit dan supernatannya dibuang. Endapan sel direuspensi dala buffer TE lalu disentrifusi kembali pada 6000 rpm selama 2 menit dan supernatannya dibuang. Endapan diresuspensi dalam 500 µl TE, selanjutnya ditambahkan 40 µl 10% SDS dan 8 µl proteinase-K (10 mg/ml) lalu dicampur dengan cara membolak-balikan tabung. Kemudian diinkubasi pada 370C selama 1 jam. Setelah itu ditambahkan 100 µl 5 M NaCl dan 100 µl 10% CTAB/NaCl yang sudah dipanaskan pada 650C. Setelah dicampur dengan baik, diinkubasi pada 650C selama 20 menit.
Kemudian ditambahkan 500 µl PCI (25:24:1) dan dicampur dengan membolak-balikan tabung secara kuat. Lalu tabung disentrifugasi pada 10 000 rpm selama 10 menit. Lapisan atas yang terbentuk dipindahkan pada tabung baru. Ekstrasi kedua menggunakan 500 µl CI (24:1) dengan cara yang sama. Selanjutnya ditambahkan 500 µl isopropanol dingin dan dicampur perlahan lalu diinkubasi pada -200C selama 20 menit. Setelah itu dilakukan sentrifusi pada kecepatan maksimum selama 5 menit dan supernatan dibuang. Endapan DNA ditambah dengan 1 ml etanol 70% lalu dicampur perlahan. Kemudian dilakukan sentrifusi pada kecepatan maksimum selama 2 menit dan supernatannya dibuang. Tabung yang berisi DNA ini disimpan pada temperatur ruang secara terbuka sampai semua etanol menguap. Selanjutnya DNA dilarutkan dalam ± 50 µl buffer elusi atau ddH2O. DNA dideteksi menggunakan elektroforesis gel agarosa dengan pewarna 0.5 µg/ml ethidium bromida.
Amplifikasi Gen Penyandi 16S rRNA
Gen penyandi 16S rRNA diamplifikasi menggunakan primer 63f
(5’-CAGGCCTAACACATGCAAGTC) dan primer 1387r
14
rantai pada 750C selama 1 menit dan post PCR pada 750C selama 20 menit. PCR dilakukan sebanyak 30 siklus. Komposisi PCR yang digunakan adalah 100 ng DNA, 5 pmol primer 63f, 5 pmol primer 1387r, 0.5 µl enzim polymerase termostabil (5U/µl), 2.5 µl 10 X buffer, 2 µl dNTPmix, dan ddH2O sampai volume mencapai 25 µl. Setelah itu, fragmen DNA hasil PCR dipurifikasi menggunakan QiaexII Gel Extraction Kit (Qiagen, USA)
Amplifikasi gen penyandi fitase
Mengingat belum diketahuinya termasuk ke dalam kelas apa enzim fitase yang dihasilkan oleh keempat isolat pada penelitian ini maka dipakai sepasang primer yang diambil dari sekuen gen penyandi fitase dari bakteri yang sekerabat
yaitu Escherichia coli dengan sekuen primer Ecf
5’-GCTAATCCCCTATCTCGGAC-3’ dan Ecr 5’-TAATAACGGGGTGGCGCGG-3’.
Komposisi PCR yang digunakan adalah 100 ng DNA, 5 pmol primer forward, 5 pmol primer riverse, 22.5 µl Platinum Blue PCR supermix (Invitrogen, USA), dengan volume akhir mencapai 25 µl. Kondisi PCR terdiri dari prePCR pada 94ºC selama 4 menit, denaturasi pada 92ºC selama 2 menit, penempelan primer pada 50ºC selama 30 detik, perpanjangan primer pada 72ºC selama 1 menit, dan post PCR pada 72ºC selama 10 menit dengan jumlah siklus sebanyak 30. Hasil amplifikasi kemudian dideteksi menggunakan elektroforesis gel mini lalu dipurifikasi menggunakan QiaexII Gel Extraction Kit (Qiagen, USA).
Kloning gen penyandi fitase
Kloning gen fitase dilakukan menggunakan TOPO TA Cloning® kit (Invitrogen, USA). Fragmen DNA hasil amplifikasi gen penyandi enzim fitase disisipkan pada pCR®2.1-TOPO (Gambar 2). Ligasi dilakukan dalam suatu reaksi menggunakan 1 µl dilute salt solution, 1µl (10 ng/µl) vektor, 2 µl DNA hasil PCR dan ddH2O sampai volume mencapai 6 µl. Kemudian diinkubasi pada suhu ruang selama 20 menit lalu dipindahkan ke es.
15
Instructions and Applications Guide (Biorad, USA). Kemudian sebanyak 2,5 µl plasmid rekombinan dicampur dengan 40 µl sel E. coli kompeten. Campuran ini diinkubasi di atas es selama 1 menit lalu dipindahkan pada kuvet berukuran 0,2 cm. Elektroporasi dilakukan menggunakan micropulser™ electroporator apparatus (Biorad, USA). Setelah itu ditambahkan 800 µl Luria Berthani Broth dan diresuspensi lalu dibagi dalam 2 tabung. Inkubasi dilakukan selama 1 jam pada suhu 370C pada shaker bath lalu disebar pada media LA yang mengandung 40 µg/ml X-gal dan 50 µg/ml kanamisin dan diinkubasi selama semalam. Hasil transformasi diamati keesokan harinya. Koloni bakteri transforman yaitu yang berwarna putih diambil dan dibiakkan. Sebagai kontrol negatif digunakan E. coli
kompeten yang tidak ditambahkan plasmid, sedangkan control positif terdiri dari kontrol ligasi yaitu E. coli kompeten yang diintroduksi dengan plasmid vector yang diligasikan dengan kontrol insert, dan kontrol transformasi yaitu E. coli
kompeten yang diintroduksi dengan pUC19.
Verifikasi transforman
Koloni transforman yang sudah dibiakkan masing-masing diambil menggunakan tusuk gigi steril lalu diresuspensi dalam pereaksi PCR yang terdiri dari 5 pmol primer M13f, 5 pmol primer M13r, 22.5 µl Platinum Blue PCR supermix, dengan volume akhir mencapai 25 µl. Kemudian diinkubasi pada 940C selama 10 menit untuk melisis sel dan menonaktifkan nuklease. Amplifikasi dilakukan sebanyak 25 siklus yang terdiri dari denaturasi pada 940C selama 1 menit, penempelan primer pada 550C selama 1 menit, perpanjangan primer pada 720C selama 1 menit dan post PCR pada 72oC selama 10 menit. Hasil amplifikasi dideteksi dengan elektroforesis gel agarosa lalu dipurifikasi menggunakan
QiaQuick PCR Purification Kit (Qiagen, USA).
Verifikasi Plasmid Rekombinan
16
kemudian dimigrasikan pada gel agarosa untuk melihat besarnya ukuran vector dan DNA sisipan.
+
A B
CCCCCCC
C
17
Penentuan Urutan Sekuen DNA
Fragmen DNA ditentukan urutan sekuennya dengan cara mengamplifikasinya dalam suatu reaksi cycle sequencing. Pada proses ini digunakan salah satu dari pasangan primer (F/R), DNA cetakan, Bigdye terminator ready reaction mix (ddNTP berlabel, dNTP, DNA polimerase, MgCl2, buffer Tris-HCl pH 9.0). Setelah dilakukan purifikasi, hasil amplifikasi ini dideteksi dengan elektroforesis menggunakan ABI Prism 310 Genetic analyzer®.
Analisis Sekuen DNA
Data sekuen DNA disimpan pada notepad dengan format fasta. Selanjutnya data sekuen DNA ini dibandingkan dengan data base pada genebank (http://www.ebi.ac.uk) menggunakan program Blast (Basic local aligment search tool). Dari hasil ini dipilih sejumlah data sekuen DNA yang diperlukan sesuai dengan kata kunci yang diinginkan lalu dipindahkan pada notepad dengan format fasta. Kemudian dilakukan multiple sequence alignment melalui program Clustal W. Data yang keluar dari hasil analisis ini dipindahkan ke notepad (dalam format
HASIL DAN PEMBAHASAN
Ciri-ciri morfologi dan fisiologi bakteri
Dalam upaya mengelompokkan organisme, para ahli menggunakan perbedaan karakter yang ditemukan untuk menggambarkan suatu bentuk kehidupan atau mengidentifikasinya. Karakter yang digunakan dapat berupa ciri-ciri morfologi, fisiologi maupun molekuler.
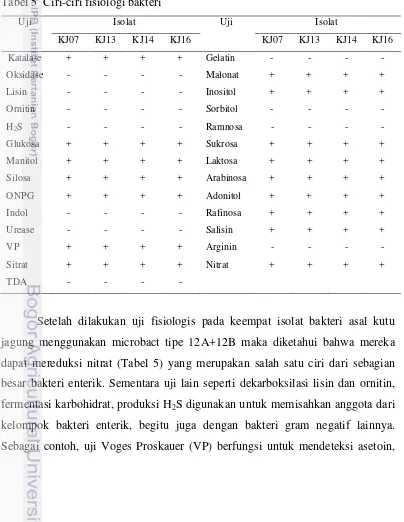
Untuk mengidentifikasi suatu bakteri ciri-ciri morfologi yang diamati diantaranya adalah warna, elevasi, tepian, bentuk, dan ukuran koloni; serta bentuk, motilitas, dan tipe dinding sel (gram negatif atau positif). Berdasarkan hasil pengamatan diketahui bahwa keempat isolat bakteri asal kutu jagung yaitu KJ07, KJ13, KJ14, dan KJ16 mempunyai koloni yang berwarna krem, berbentuk bundar, dengan elevasi cembung, dan tepian licin. Setelah dilakukan pewarnaan gram dan pengamatan di bawah mikroskop, maka keempat isolat tersebut diketahui mempunyai sel yang berbentuk coccus, motil, dengan tipe dinding sel gram negatif (Tabel 4).
Tabel 4 Ciri-ciri morfologi bakteri
Ciri-ciri Isolat
KJ07 KJ13 KJ14 KJ16
Ciri-ciri koloni
Warna krem krem krem krem
Elevasi cembung cembung cembung cembung
Tepian licin licin licin licin
Bentuk bundar bundar bundar bundar
Ukuran 1.2 mm 1.25 mm 1 mm 1 mm
Ciri-ciri sel
Bentuk coccus coccus coccus coccus
Motilitas motil motil motil motil
Spora - - - -
Gram negatif negatif negatif negatif
20
Setelah dilakukan uji katalase dan oksidase maka diketahui bahwa keempat isolat bakteri tersebut bersifat katalase positif dan oksidase negatif. Hal ini menunjukkan bahwa isolat-isolat tersebut adalah bakteri anaerobik fakultatif. Berdasarkan informasi ini, ditambah hasil pengujian gram yang negatif maka diputuskan untuk menggunakan microbact tipe 12A+12B untuk uji fisiologis lebih lanjut. Microbact™ gram negative identification system dirancang untuk mengidentifikasi bakteri gram negatif aerob dan anaerob fakultatif . Secara umum kit ini digunakan untuk mengidentifikasi famili Enterobacteriaceae dan bakteri gram negatif batang lainnya.
Tabel 5 Ciri-ciri fisiologi bakteri
Uji Isolat Uji Isolat
21
prekursor 2,3 butanadiol yang diproduksi dari fermentasi glukosa oleh bakteri gram negatif penghuni usus khususnya Enterobacter dan Serratia, serta beberapa spesies dari Erwinia, Bacillus dan Aeromonas. Hasil positif dari uji ini merupakan salah satu tanda bahwa isolat yang diuji termasuk ke dalam kelompok tersebut.
Setelah dilakukan analisis terhadap hasil uji fisiologis ini menggunakan
microbact™ computer aided identification package didapatkan % probabilitas isolat-isolat tersebut sebesar 99.92% terhadap Serratia rubidaea (Tabel 6). Setelah dibandingkan dengan Bergey’s Manual of Determinative Bacteriology (Holt et al. 1994) maka hasil pengamatan ini secara umum sesuai. Warna koloni yang krem merupakan keragaman yang ditemukan dalam genus Serratia
meskipun secara umum anggota kelompok ini berwarna merah. Begitu juga dengan kemampuan menghidrolisis gelatin yang dinyatakan secara umum dimiliki oleh anggota kelompok ini sehingga ada kemungkinan ada yang tidak memilikinya. Akan tetapi bentuk sel yang coccus, merupakan fenomena yang perlu diamati lebih lanjut apakah benar-benar coccus ataukah batang pendek atau oval karena dengan mikroskop cahaya sampai perbesaran 1000x sel-sel bakteri tersebut masih terlihat sangat kecil dibandingkan bakter-bakteri kontrol. Pengamatan terbaik dapat dilakukan menggunakan mikroskop elektron yang tidak dilakukan pada penelitian ini.
Tabel 6 Hasil identifikasi dengan micobact™ tipe 12A+12B
Isolat Morfologi-Fisiologi
Spesies homolog Probabilitas (%)
KJ07 Serratia rubidaea 99,92
KJ13 Serratia rubidaea 99,92
KJ14 Serratia rubidaea 99,92
KJ16 Serratia rubidaea 99,92
Identifikasi menggunakan sekuen gen penyandi 16S rRNA
22
prokariot dan juga mitokondria dan kloroplas. Ukurannya yang tidak terlalu panjang (bila dibandingkan dengan rRNA 23S) dan tidak terlalu pendek (bila dibandingkan dengan rRNA 5S) membuat molekul ini menjadi pilihan dalam melacak filogeni prokariot. Disamping itu, pada molekul ini terdapat daerah dengan sekuen konservatif dan hipervariabel. Perbandingan sekuen konservatif berguna untuk mengkonstruksi pohon filogenetik universal sedangkan sekuen hipervariabel digunakan untuk mencirikan organisme dalam takson yang lebih sempit (Madigan et al. 1997).
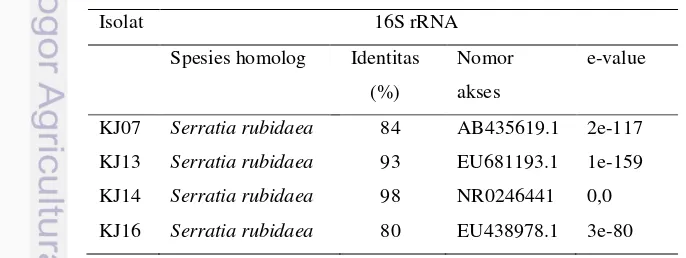
Setelah dilakukan analisis BLAST terhadap hasil amplifikasi parsial sekuen gen penyandi 16S rRNA dari keempat isolat bakteri kutu jagung, maka didapatkan % identitas dari isolat KJ07, KJ13, KJ14, KJ16 sebesar 84%, 93%, 98%, 80% terhadap Serratia rubidaea (Tabel 7). Menurut Madigan et al. (1997), jika sekuen gen penyandi 16S rRNA suatu isolat yang diuji mempunyai identitas kurang dari 97% terhadap sekuen yang ada di database maka dipertimbangkan sebagai spesies baru. Keadaan ini berdasarkan pengamatan DNA dari dua bakteri kesamaan sekuen 16S rRNAnya kurang dari 97% berhibridisasi kurang dari 70%, nilai minimal yang menunjukkan organisme berada dalam satu spesies. Oleh sebab itu, jika hanya dilihat hasil uji 16S rRNAnya saja maka hanya isolat KJ14 yang bisa diterima sebagai Serratia rubidaea. Namun, karena hasil fisiologis menunjukkan % probabilitas yang sangat tinggi terhadap spesies ini, maka diusulkan keempat isolat tersebut sangat dekat dengan spesies Serratia rubidaea. Hal ini juga mengingat bahwa uji terhadap sekuen 16S rRNA hanya dilakukan secara parsial.
Tabel 7 Hasil identifikasi berdasarakan sekuen gen penyandi 16S rRNA
Isolat 16S rRNA
KJ07 Serratia rubidaea 84 AB435619.1 2e-117
KJ13 Serratia rubidaea 93 EU681193.1 1e-159
KJ14 Serratia rubidaea 98 NR0246441 0,0
23
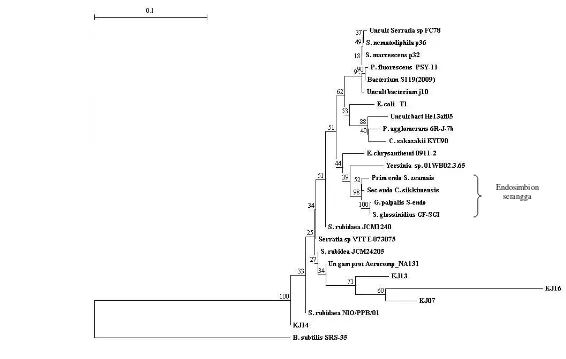
Kemudian berdasarkan sekuen gen penyandi 16S rRNA ini didapatkan pohon filogenetik yang memperlihatkan hubungan kekerabatan antara keempat isolat bakteri asal kutu jagung dengan sebagian bakteri-bakteri lainnya dari database. Pada pohon filogenetik ini terlihat bahwa isolat KJ07 memiliki hubungan kekerabatan yang lebih dekat dengan KJ16 daripada KJ13 dan KJ14. Sementara itu empat galur Serratia rubidaea berada pada posisi yang berbeda-beda tapi tetap dekat dengan keempat isolat tersebut. Kemudian apabila dilihat dari skala jaraknya terlihat bahwa bakteri-bakteri endosimbion kutu jagung ini setelah S. rubidaea lebih dekat terhadap bakteri-bakteri endosimbion serangga lainnya yang ada di database seperti endosimbion dari S. zeamays, Curculio sikkimensis, Glossina palpalis, dan Sodalis glossinidius dari pada bakteri-bakteri enterik dan gram negatif lainnya dan sangat jauh dari bakteri gram positif yang diwakili oleh Bacillus subtilis (Gambar 2).
Gambar 3 Pohon filogenetik hubungan kekerabatan keempat isolat bakteri asal kutu jagung dan bakteri-bakteri lainnya. Angka 0.1 menunjukkan skala persentase
perbedaan (distance scale). Angka pada nodus menunjukkan nilai bootstrap dengan 100 kali replikasi.
25
Deteksi gen putatif penyandi fitase
Kemampuan hidrolisis fitat dari bakteri melibatkan enzim fitase. Enzim ini dikodekan oleh suatu gen yang dinamakan appa atau phy. Untuk mendeteksi gen penyandi fitase dirancang sepasang primer berdasarkan sekuen gen fitase dari
E. coli (Dassa et al. 1990). Setelah dilakukan amplifikasi parsial terhadap sekuen gen putatif penyandi fitase dari keempat isolat bakteri asal kutu jagung didapatkan pita DNA berukuran 1500 bp (Gambar 3). Ukuran ini termasuk ke dalam rentangan ukuran gen penyandi fitase bakteri pada umumnya diantaranya telah diketahui bahwa gen fitase pada E. coli berukuran 1500 bp (Dassa et al. 1990), dan gen fitase pada B. subtilis berukuran 1300 bp (Kerovuo et al. 1998).
Gambar 4 Hasil amplifikasi sekuen gen putatif penyandi fitase. 1=1 kb DNA ladder, 2=KJ07, 3=KJ13, 4=KJ14, 5=KJ16
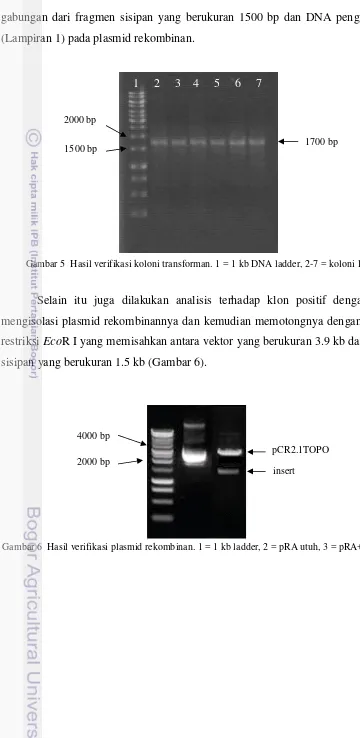
Karena keterbatasan waktu, maka untuk selanjutnya salah satu isolat saja yang dipilih untuk proses kloning yaitu isolat KJ07. Fragmen DNA berukuran 1500 bp yang telah diamplifikasi dari genom isolat ini disisipkan pada pCR®2.1TOPO®, kemudian diintroduksikan pada E. coli TOP 10. Koloni transforman dianalisis menggunakan PCR dengan primer M13f dan M13r. Hasil amplifikasi menunjukkan pita berukuran 1700 bp (Gambar 5) yang merupakan
1 2 3 4 5
1500 bp
26
gabungan dari fragmen sisipan yang berukuran 1500 bp dan DNA pengapitnya (Lampiran 1) pada plasmid rekombinan.
Gambar 5 Hasil verifikasi koloni transforman. 1 = 1 kb DNA ladder, 2-7 = koloni 1-7
Selain itu juga dilakukan analisis terhadap klon positif dengan cara mengisolasi plasmid rekombinannya dan kemudian memotongnya dengan enzim restriksi EcoR I yang memisahkan antara vektor yang berukuran 3.9 kb dan DNA sisipan yang berukuran 1.5 kb (Gambar 6).
Gambar 6 Hasil verifikasi plasmid rekombinan. 1 = 1 kb ladder, 2 = pRA utuh, 3 = pRA+ EcoR I 1 2 3 4 5 6 7
1500 bp 2000 bp
1700 bp
4000 bp
2000 bp
insert
27
Penentuan sekuen fragmen hasil PCR dilakukan menggunakan templat plasmid rekombinan dengan primer M13f dan M13r. Sekuen yang didapat kemudian digabungkan dan dianalisis menggunakan BLAST yang tersedia di http://www.ncbi.nlm.nih.gov. Analisis dilakukan menggunakan sekuen nukleotida (blastn) dan sekuen asam amino yang dideduksi dari nukleotida tersebut (blastx). Hasil blastn menunjukkan identitas tertinggi sebesar 80% terhadap Serratia proteamaculans 568, complete genome dengan subjek pada bagian sekuen tersebut adalah prolipoprotein diacylglyceryl transferase dan PTSINtr with GAF domain, PtsP. Sementara itu hasil blastx menunjukkan identitas tertinggi sebesar 89% terhadap phosphoenolpyruvate-protein phosphotransferase [Serratia odorifera DSM 4582] dan 93% terhadap prolipoprotein diacylglyceryl transferase [Serratia odorifera DSM 4582] (Tabel 8).
Tabel 8 Hasil analisis BLAST dari fragmen DNA 1,5 kb
Analisis Sekuen homolog Identitas
(%)
E-value No. akses
blastn Serratia proteamaculans 568, complete genome dengan subjek transferase [Serratia odorifera DSM 4582]
Berdasarkan hasil ini maka dapat disimpulkan bahwa strategi yang digunakan tidak memungkinkan untuk mendeteksi gen fitase pada Serratia rubidaea. Diduga pasangan primer menempel secara tidak spesifik pada genom isolat yang diuji. Hal ini kemungkinan terjadi akibat gen penyandi fitase pada E. coli tidak berkerabat dekat dengan gen penyandi fitase pada Serratia meskipun kedua genus ini berada dalam famili yang sama. Pada waktu perancangan primer tidak ditemukan pada database sekuen DNA penyandi fitase yang berasal dari
28
putatif penyandi fitase dari Serratia proteamaculan 568 (Lim et al 2007). Namun penelitian tersebut hanya pada skala bioinformatika dimana dilacak sekuen DNA yang mirip gen penyandi fitase pada kumpulan genom yang ada di database dan tidak diuji pada skala laboratorium. Setelah ditelusuri menggunakan blastx dan blastp kemiripan gen putatif penyandi fitase pada Serratia proteamaculan 568 tersebut hanya sebesar 33% terhadap gen penyandi fitase E. coli. Selain itu motif tipe fitase HAP yang dimiliki oleh Escherichia coli (RHGXRXP dan HDTN) tidak dimiliki secara utuh oleh sekuen ini dimana G diganti dengan N dan T diganti dengan S.
Setelah dilakukan alignment langsung antara sekuen produk PCR dari KJ07 ini dengan sekuen DNA penyandi fitase dari E. coli, identitas yang diperoleh hanya sebesar 45,5% sedangkan hasil alignmen langsung terhadap sekuen gen putatif penyandi fitase dari S. proteamaculan 568 hanya menunjukkan identitas sebesar 43,5%. Selain itu, juga tidak ditemukan motif tipe fitase HAP dari hasil terjemahan sekuen tersebut ke dalam bentuk asam amino dari keenam frame yang mungkin baik forward maupun reverse. Berdasarkan data-data tersebut maka dapat dipastikan bahwa sekuen yang diperoleh bukanlah gen penyandi fitase, melainkan bagian dari genom S. rubidaea yang terdiri dari sepertiga akhir dari gen penyandi phosphoenolpyruvate-protein phosphotransferase dan duapertiga awal dari gen penyandi prolipoprotein diacylglyceryl transferase (Lampiran 2).
KESIMPULAN DAN SARAN
Kesimpulan
Keempat isolat bakteri yang berasal dari kutu jagung yaitu KJ07, KJ13, KJ14, KJ16 berkerabat dekat dengan Serratia rubidaea. Penggunaan PCR dengan primer Ecf dan Ecr tidak memungkinkan kami untuk mendapatkan gen penyandi fitase dengan teknik ini.
Saran
DAFTAR PUSTAKA
Bohn L, Meyer A S, Rasmussen SK. 2008. Phytate: impact on environment and human nutrition. A challenge for molecular breeding. J Zhejiang Univ Sci B 9:165-191.
Chu HM, Guo RT, Lin TW, Chou CC, Shr HL, Lai HL, Tang TY, Cheng KJ, Selinger BL, Wang AH. 2004. Structures of Selenomonas ruminantium
phytase in complex with persulfated phytate: DSP phytase fold and mechanism for sequential substrate hydrolysis Structure, 12:2015-2024. Dassa J, Christian M, Boquet D L. 1990. The complete nucleotide sequence of the
Escherichia coli gene appA reveals significant homology between pH 2,5 acid phosphatase and glucose 1-phosphatase. J Bacteriol 172:5497-5500. Glick B R, Pasternak J J. 2003. Molecular Biotechnology: Principles and
Applications of Recombinant DNA. New York: American society for microbiology.
Hadiutomo R S. 1993. Microbiologi Dasar dalam Praktek: Teknik dan Prosedur Dasar Laboratorium. Jakarta: Penerbit PT Gramedia
Holt J G, Krieg N R, Sneath P H A, Staley J Y, Williams S T. 1994. Bergey’s Manual of Determinative Bacteriology Edisi ke-9. New York:Lippincott Williams &Wilkins.
Huang H, Luo H, Yang P, Meng K, Wang Y, Yun T, Bai Y, Yau B. 2006. A novel phytase with preferable characteristic from Yersinia intermedia.
Biochem Biophys Res Commun 350:884-889.
Kerovuo J, Lauraeus M, Nurminen P, Kalkkinen N, Apajalahti J. 1998. Isolation, characterization, molecular gene cloning, and sequencing of a novel phytase from Bacillus subtilis. Appl Envir Microbiol 64:2079-2085.
Kornegay E T. 1996. Nutritional, environmental, and economic considerations for using phytase in pig and poultry diets. Di dalam: Kornegay E T, editor.
32
Lei X G, Porres J M, Mullaney E J, Brinch-Pedersen H. 2007 Phytase: source, structure, and application. Di dalam: Polaina, MacCabe, editor. Industrial Enzymes. Hlm 505-529. US Government
Lim B L, Yeung P, Cheng C, Hill J E. 2007. Distribution and diversity of phytate-mineralizing bacteria. ISME J 1:321-330
Luo H, Huang H, Yang P, Wang y, Yuan T, W N, Yao B, Fan Y. 2007. A novel phytase appA from Citrobacter amalonaticus C6MCC 1696: Gene cloning and overexpression in Pichia pastoris. Curr Microbiol 55:185-192. Madigan M T, Martinko J M, Parker J. 1997. Biology of microorganism Ed-8.
New York: Prentice Hall International Inc.
Makarewicz O, Dubrac S, Msadek T, Borriss R.2006. Dual role of the PhoP~P response regulator: Bacillus amyloliquefaciens FZB45 phytase gene transcription is directed by positive and negative interactions with the
phyC promoter. J Bacteriol 188: 6953–6965.
Marchesi JR, Sato T, Weightman AJ, Martin TA, Fry JC, Hiom SJ, Wade WG. 1998. Design and evaluation of useful bacterium specific primers that amplify gene coding for bacterial 16S rRNA. App Environ Microbiol. 64:795-799.
Mullaney E J, Daly C B, Ullah A H. 2000. Advance in phytase research. Adv Appl Microbiol 47:157-199.
Murray M G, Thomson W F. 1980. Rapid isolation of high molecular weigh plant DNA. Nucleic Acid Res 19:823-881
Na S, Huang H, Meng K, Luo H, Wang Y, Yang P, Yao B. 2008. Cloning, expression and characterization of a new phytase from the phytophatogenic bacteria Pectobacterium wasanabe DSMS 18074. J Microbiol Biotechnol 18:1221-1226.
Pestweb. 2010. Greater rice weevil. http//:www.Agspsrv34.agric.wa.gov.au. [Juni 2009]
33
Skoglund E, Carlsson N G, Sanberg A S. 1997. Analysis of mono – and diphosphate isomers using high-performance ion chromatography and pulsed amperometric detection. J Agric Food Chem 45:4668-4673.
Van de Peer Y, De Wachter R. 1994. Treecon for windows: a software package for construction and drawing of evolutionary trees for Microsoft window environment. Comput Appl Biosci 10:569-570.
Wikipedia. 2009. Phytase. http//:www. wikipedia.org. [Juni 2009]
Zinin N, Serkina A V, Gelfand M S. 2004. Gene cloning, expression and characterization of novel phytase from Obesumbacterium proteus. FEMS Microbiol Lett 236:283-290.
37
38
Lampiran 2. Hasil blastx sekuen DNA dari fragmen 1,5 kb.
> ref|ZP_06638960.1| phosphoenolpyruvate-protein phosphotransferase [Serratia odorifera DSM 4582]
gb|EFE96031.1| phosphoenolpyruvate-protein phosphotransferase [Serratia odorifera DSM 4582] Length=748
Score = 400 bits (1028), Expect = 3e-109
Identities = 203/228 (89%), Positives = 217/228 (95%), Gaps = 0/228 (0%) Frame = +3
Query 45 GWRGIRVTLDQPEIFLIQVRAMLRANAGTGNLGILLPMVTSLDEIDEAKSLIERAGHEVE 224 GWRGIRVTLDQPEIFLIQVRAMLRANAGTGNLGILLPM+TSLDEIDEAK LIERAG+EVE Sbjct 521 GWRGIRVTLDQPEIFLIQVRAMLRANAGTGNLGILLPMITSLDEIDEAKRLIERAGNEVE 580
Query 225 EMLGYAIPKPRIGVMLEVPSMIFLIPQLAERVDFISVGTNDLTQYLLAVDRNNTRVASLY 404 E+LGYAIPKP+IGVMLEVPSMIFLIP LA RVDFISVGTNDLTQYLLAVDRNNTRVASLY Sbjct 581 EVLGYAIPKPKIGVMLEVPSMIFLIPFLARRVDFISVGTNDLTQYLLAVDRNNTRVASLY 640
Query 405 DSLHPSMLRVLQTIVEQGEAAGLQVSLCGELAGDPIGALLLVGMGYRHLSMNGRSVARIK 584 DSLHPSMLRVL+ IVEQGEAAGLQV LCGELAGDP+GALLLVGMGYRHLSMNGRSVARIK Sbjct 641 DSLHPSMLRVLKMIVEQGEAAGLQVGLCGELAGDPMGALLLVGMGYRHLSMNGRSVARIK 700
Query 585 YLLRHIELADAQRLTQQVLRASLTNEVRHLAATFMEQQNMGGLVRGGR 728 YLLRHIELA+AQ L+Q+VL A +T EVRHL A FME+++MGGL+RGGR Sbjct 701 YLLRHIELAEAQALSQRVLNAQMTTEVRHLVAAFMERRSMGGLIRGGR 748
> ref|ZP_06638961.1| prolipoprotein diacylglyceryl transferase [Serratia odorifera
DSM 4582]
gb|EFE96032.1| prolipoprotein diacylglyceryl transferase [Serratia odorifera DSM 4582]
Length=290
Score = 348 bits (893), Expect = 1e-93
Identities = 185/198 (93%), Positives = 192/198 (96%), Gaps = 1/198 (0%) Frame = +3
Query 939 MSNSYLRFPEFDPVLFSIGPVSLHWYGLMYLVGFVFAMWLAIRRANKPGSGWTKDEVENL 1118 MSNSYL FP+FDPV+FSIGPVSLHWYGLMYLVGFVFAMWLAIRRANKPGSGWTKDEVENL Sbjct 1 MSNSYLAFPKFDPVIFSIGPVSLHWYGLMYLVGFVFAMWLAIRRANKPGSGWTKDEVENL 60
Query 1119 LYAgflgvfvggrvgyvlfyNLPLFLENPLYLFKVWDGGMSFHGGLMGVILVMFWFARRT 1298 LYAGFLGVFVGGRVGYVLFYNLPLFL+NPLYLFKVWDGGMSFHGGL+GVILVMFWFARRT Sbjct 61 LYAGFLGVFVGGRVGYVLFYNLPLFLDNPLYLFKVWDGGMSFHGGLLGVILVMFWFARRT 120
Query 1299 KRTFFQVSDFIAPLIPFGLGAGRLGNFINGELWGRVTTDTPWAMLFPGSRSEDIAIAAAD 1478 KRTFFQVSDFIAPLIPFGLGAGRLGNFINGELWGRVT+D PWAMLFPGSR+EDIAIAAAD Sbjct 121 KRTFFQVSDFIAPLIPFGLGAGRLGNFINGELWGRVTSDVPWAMLFPGSRNEDIAIAAAD 180