IDENTIFIKASI KERAGAMAN GEN LAKTOFERIN (LTF EcoRI)
PADA SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI DAN BET CIPELANG
SKRIPSI
GABBY ELFANDA MUMPUNIE
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
GABBY ELFANDA MUMPUNIE. D14070166. 2011. Idetifikasi Keragaman Gen Laktoferin (LTF EcoRI) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari dan BET Cipelang. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D.
Laktoferin merupakan komponen dari protein susu yang bersifat antimikroba. Laktoferin yang terkandung di dalam susu berfungsi untuk mencegah diare, sedangkan bagi sapi laktoferin berfungsi untuk mencegah mastitis pada ambing. Dilihat dari peranan laktoferin tersebut, maka perlu adanya suatu usaha untuk meningkatkan kadar laktoferin di dalam susu. Salah satu cara untuk meningkatkan kemampuan sapi yang menghasilkan susu berkadar laktoferin tinggi adalah dengan melakukan seleksi pada tingkat DNA. Hal ini dapat dilakukan dengan cara mencari keragaman susunan nukleotida dalam gen yang berpengaruh terhadap kadar laktoferin susu. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen LTF EcoRI pada sapi Friesian Holstein (FH) di Balai Besar Inseminasi Buatan (BBIB) Singosari, Balai Inseminasi Buatan (BIB) Lembang dan Balai Embrio Ternak (BET) Cipelang; serta sapi pedaging di BET Cipelang sebagai pembanding.
Materi penelitian berupa sampel darah sapi FH sebanyak 89 ekor yang terdiri dari sapi jantan berasal dari BBIB Singosari (32 ekor) dan BIB Lembang (17 ekor), sapi betina dari BET Cipelang (40 ekor); serta sapi pedaging betina sebagai pembanding meliputi Limosin (14 ekor), Angus (5 ekor), Simental (13 ekor) dan Brahman (5 ekor) dari BET Cipelang. Amplifikasi gen LTF EcoRI dilakukan dengan
Polymerase Chain Reaction (PCR) pada gel agarose 1,5 %. Pendeteksian keragaman gen LTF EcoRI menggunakan metode Polymerase Chain Reaction-Restricsion Fragment Length Polymorphism (PCR-RFLP) dengan enzim restriksi EcoRI, sehingga didapatkan tiga macam fragmen yang panjangnya 301 pasang basa (pb) untuk alel A, 201 pb dan 100 pb untuk alel B. Keragaman genetik gen LTF EcoRI dianalisis dengan menghitung nilai frekuensi alel, frekuensi genotipe, keseimbangan Hardy-Weinberg, dan heterozigositas.
Hasil perhitungan frekuensi alel gen LTF EcoRI menunjukkan bahwa sapi FH pada ketiga lokasi dan sapi pedaging yang digunakan sebagai pembanding mempunyai frekuensi alel A yang lebih tinggi dibandingkan dengan alel B. Frekuensi total alel A dan B dari semua sapi FH pengamatan yaitu 0,77 dan 0,23. Hal tersebut juga tidak berbeda jauh dengan sapi pedaging yang diteliti. Genotipe yang ditemukan untuk sapi FH adalah AA dan AB, sebaliknya tidak ditemukan genotipe BB. Hasil perhitungan frekuensi genotipe sapi FH memiliki genotipe AA sebesar 0,55 dan genotipe AB sebesar 0,45, sedangkan genotipe BB bernilai 0. Nilai heterozigositas gen LTF EcoRI pengamatan (Ho=0,5) lebih tinggi dibandingkan dengan nilai heterozigositas harapan (He=0,38) untuk keseluruhan sapi pengamatan, baik sapi FH maupun sapi pedaging.
ABSTRACT
Identification of the Lactoferrin (LTF EcoRI) Gene Polymorphism in HolsteinFriesian Cattle in BIB Lembang, BBIB Singosari and BET Cipelang
Mumpunie, G. E. , C. Sumantri, and A. Anggraeni
Lactoferrin (LTF) is a gene controlling milk protein component in which having the characteristic as antimicrobial. LTF contained in the milk has function to prevent diarhea, whereas the function of LTF for the cows is to prevent mastitis in the udder. By looking at the role of the LTF gene, an attempt of improving LTF component in the milk is necessary through the selection process conducted at the DNA level. This study was aimed to identify the polymorphism of the LTF EcoRI gene in Holstein-Friesian (HF) cattle. The LTF EcoRI gene polymorphism was identified with Polymerase Chain Reaction-Restricsion Fragment Length Polymorphism (PCR-RFLP) method using EcoRI enzyme. Genotyping was perfomed in 89 HF cattle from Balai Inseminasi Buatan (BIB) Lembang (17 heads), Balai Besar Inseminasi Buatan (BBIB) Singosari (32 heads) and Balai Embrio Ternak (BET) Cipelang (40 heads). Genotyping was also conducted in four breeds of beef cattle as a comparison for Limosin (14 heads), Angus (5 heads), Simental (13 heads) and Brahman (5 heads) from BET Cipelang.
Results from the analysis of the allele frequencies of the LTF EcoRI gene in HF cattle in three locations and beef cattle showed that the A allele had a higher frequency than the B allele. The total frequencies of the A and B allels in HF cattle were 0,77 and 0,23 respectively. Genotypes for HF cattle observed were found for AA and AB, whereas the BB genotype was not found. The genotype frequencies of all cattle (HF and beef cattle) had the same proportion, genotype AA and AB had the same value (0,5), whereas the genotype BB was 0. Heterozygosity (Ho) value of LTF EcoRI gene were higher than those of heterozygosity value for all cattle. The conclusion was that LTF EcoRI gene in HF and beef cattle observed was polymorphic.
IDENTIFIKASI KERAGAMAN GEN LAKTOFERIN (LTF EcoRI)
PADA SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI DAN BET CIPELANG
GABBY ELFANDA MUMPUNIE D14070166
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen laktoferin (LTF EcoRI) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari dan BET Cipelang
Nama : Gabby Elfanda Mumpunie NIM : D14070166
Menyetujui,
Mengetahui: Ketua Departemen,
Ilmu Produksi dan Teknologi Peternakan
Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. NIP. 19591212 198603 1 004
Tanggal Ujian : 3 Mei 2011 Tanggal Lulus:
Pembimbing Utama, Pembimbing Anggota,
Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. NIP. 19591212 198603 1 004
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 3 September 1989 di Cilacap, Jawa Tengah. Penulis adalah putri tunggal dari pasangan Bapak Yuswadi dan Ibu Nuning Listyarini.
Penulis mengawali pendidikan dasar pada tahun 1995 di SD Pius, Cilacap dan diselesaikan pada tahun 2001. Pendidikan lanjutan menengah pertama dimulai pada tahun 2001 dan diselesaikan pada tahun 2004 di SMP Bunda Hati Kudus, Jakarta Barat. Penulis melanjutkan pendidikan di Sekolah Menengah Atas pada tahun 2004 diselesaikan pada tahun 2007 di SMA N 65, Jakarta Barat.
KATA PENGANTAR
Puji syukur kepada Tuhan Yang Maha Esa atas segala karunia dan penyertaanNya sehingga penulis dapat menyelesaikan skripsi yang berjudul “Identifikasi Keragaman Gen Laktoferin (LTF EcoRI) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari dan BET Cipelang”yang merupakan salah satu syarat untuk memperoleh gelar sarjana peternakan di Fakultas Peternakan, Institut Pertanian Bogor. Penulis juga mengucapkan terima kasih kepada seluruh pihak yang telah memberi dukungan moril maupun material.
Laktoferin merupakan protein yang bersifat antimikroba, sehingga dapat digunakan untuk mencegah dan mengontrol mastitis pada sapi perah. Salah satu upaya yang dapat dilakukan untuk meningkatkan produksi susu segar dengan kadar laktoferin tinggi adalah melalui seleksi bibit sapi perah yang berkemampuan memproduksi susu yang susu berkadar laktoferin tinggi. Kemajuan dalam bidang bioteknologi molekuler memungkinkan upaya seleksi dapat dilakukan pada tingkat DNA, yaitu dengan cara mencari keragaman susunan nukleotida dalam gen yang berpengaruh terhadap produksi dan kualitas susu. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen LTF EcoRI pada sapi Friesian Holstein (FH) pada tiga Balai Bibit Nasional (BIB Lembang, BBIB Singosari dan BET Cipelang).
Penulis menyadari bahwa dalam penulisan skripsi ini masih banyak terdapat kekurangan, oleh karena itu kritik dan saran yang membangun sangat diharapkan demi kesempurnaan tulisan ini. Semoga tulisan ini dapat bermanfaat bagi pembaca dan memberikan kontribusi bagi kemajuan ilmu pengetahuan.
Bogor, Mei 2011
Penulis
Keseimbangan Hardy Weinberg (HW) ... 13
Heterozigositas ... 14
HASIL DAN PEMBAHASAN ... 15
Amplifikasi Gen LTF EcoRI ... 15
Pendeteksian Keragaman Gen LTF EcoRI ... 16
Frekuensi Genotip dan Alel Gen LTF EcoRI ... 18
Keseimbangan Hardy-Weinberg Gen LTF EcoRI ... 20
Pendugaan Nilai Heterozigositas Gen LTF EcoRI ... 21
KESIMPULAN DAN SARAN ... 23
Kesimpulan ... 23
Saran ... 23
UCAPAN TERIMAKASIH ... 24
DAFTAR PUSTAKA ... 25
DAFTAR TABEL
Nomor Halaman
1. Nilai Frekuensi Genotipe dan Alel Gen LTF EcoRI Sapi dari
BIB Lembang, BBIB Singosari dan BET Cipelang…………. 18
2. Uji Hasil Uji Signifikasi χ2 Keseimbangan Hardy-Weinberg
Gen LTF EcoRI ... 20
3. Pendugaan Nilai Heterozigositas Pengamatan (Ho) dan Nilai
DAFTAR GAMBAR
Nomor Halaman
1. Sapi Friesian Holstein ... 3 2. Struktur Gen Laktoferin ... 5 3. Hasil Amplifikasi Gen LTF EcoRI Sapi FH dengan Metode PCR 15 4. Posisi Penempelan Primer (Cetak Tebal) pada Sekuen Gen
LTF EcoRI (L19986)... 16 5. Pola Pita Gen LTF EcoRI Sapi FH pada Gel Agarose 2%
dengan Enzim Retriksi EcoRI ... .. 17 6. Jumlah Sapi FH dan Sapi Pedaging dengan Genotipe tertentu
DAFTAR LAMPIRAN
Nomor Halaman
PENDAHULUAN
Latar Belakang
Sapi Friesian Holstein merupakan salah satu bangsa sapi perah yang paling banyak menghasilkan air susu. Sapi ini merupakan sapi yang umum dibudidayakan di Indonesia karena dapat beradaptasi di daerah tropis. Susu yang dihasilkan sapi perah menyediakan kebutuhan protein hewani yang bermanfaat bagi manusia. Konsumsi susu saat ini mulai meningkat seiring dengan kesadaran masyarakat akan pentingnya minum susu. Susu kaya akan nutrisi yang berguna bagi tubuh yang terdiri atas kasein dan whey protein yang mengandung laktoferin, laktoperoksidase, lisozim, dan immunoglobulin yang sering disebut sebagai protein anti mikroba. Kandungan protein yang tinggi dalam susu akan meningkatkan kualitas susu sebagai pangan fungsional.
Laktoferin yang disebut sebagai protein anti mikroba, merupakan salah satu protein minor yang secara alami ditemukan dalam susu sapi. Laktoferin adalah salah satu faktor penting untuk mencegah dan mengontrol mastitis pada sapi perah. Protein disintesis granulosit dan sel-sel epitel susu sebagai respons terhadap infeksi seperti mastitis. Laktoferin mampu mengikat ion besi (Fe) dari mikroba, sehingga dapat menghambat pertumbuhan mikroba. Laktoferin ditemukan dalam jumlah besar dalam sekresi mamalia seperti air susu, air mata, saliva, dan cairan seminal, sebagaimana pada beberapa sel darah putih. Laktoferin pada mamalia mempunyai fungsi utama yang berhubungan langsung dengan sistem kekebalan tubuh.
Tujuan
TINJAUAN PUSTAKA
Sapi Friesian Holstein
Bangsa sapi Friesian Holstein (FH) berasal dari negara Belanda tepatnya di Provinsi North Holland dan West Friesland, kedua daerah tersebut memiliki padang rumput yang bagus. Sapi FH memiliki kemampuan berkembang biak yang baik, rata-rata bobot badan sapi FH adalah 750 kg dengan tinggi bahu 139,65 cm. Kemampuan produksi susu sapi FH lebih tinggi dibandingkan bangsa sapi perah lain. Suhu lingkungan yang optimum untuk sapi perah dewasa berkisar antara suhu 5-21 ºC, sedangkan kelembaban udara yang baik untuk pemeliharaan sapi perah adalah sebesar 60% dengan kisaran 50% -75% (Enseminger dan Howard, 2006).
Syarief dan Sumopastowo (1984) menyatakan bahwa tanda-tanda yang dimiliki bangsa ini antara lain memiliki warna putih dengan belang hitam, dapat juga hitam dengan belang putih sampai warna hitam. Ekor harus putih, warna hitam tidak diperkenankan, juga tidak diperbolehkan warna hitam di daerah bawah persendian siku dan lutut, tetapi warna hitam diperbolehkan pada kaki mulai dari bahu atau paha sampai ke kuku. Gambar sapi FH dapat dilihat pada Gambar 1.
Sumber: http://dompi.co.id
Gambar 1. Sapi Friesian Holstein
(Dwiyanto et al., 2001). Talib et al. (2000) melaporkan bahwa rataan produksi susu sapi FH pada perusahaan lebih banyak dibandingkan dengan peternakan rakyat di pulau Jawa, yaitu sebesar 3565,8 ± 159,7 liter pada perusahaan dan 3266,4 ± 210,4 liter pada peternakan rakyat.
Gen Laktoferin
Gen laktoferin mengandung 17 ekson dalam suatu unit 34,5 kb transkripsional (Seyfert et al., 1994). Lebih dari enam dekade yang lalu, laktoferin ditemukan dalam susu sapi (Sorensen dan Sorensen, 1993). Laktoferin merupakan ikatan glikoprotein pengikat besi (Fe3+) yang pertama sekali diisolasi dari susu sapi dan selanjutnya dari ASI. Laktoferin ditemukan dalam jumlah besar dalam sekresi mamalia seperti susu, air mata, saliva, dan cairan seminal, begitu juga pada beberapa sel darah putih (Naim, 2003).
Laktoferin aktif dalam modulasi dan peraturan makrofag, fungsi limfosit dan neutrofil (Smith dan Oliver, 1981; Sordillo et al., 1997). Laktoferin adalah salah satu faktor penting untuk mencegah dan mengontrol mastitis pada sapi perah karena sifat-sifatnya (Teng, 2002). Penelitian sebelumnya menemukan bahwa laktoferin dapat digunakan sebagai konsentrasi sel somatik penanda genetik dalam susu, yaitu sebagai penanda kerentanan atau ketahanan terhadap mastitis pada sapi perah (Wojdak et al., 2006). Laktoferin terdiri atas rantai polipeptida tunggal dengan dua globular lobes (N-lobe dan C-lobe) dan relatif resisten terhadap proteolisis. Laktoferin merupakan protein yang disekresikan oleh jaringan ektodermal dan mempunyai struktur mirip transferin.
Keterangan :
= Exon 1 (81 pb), Exon 2 (163 pb), Exon 3 (108 pb), Exon 4 (182 pb), Exon 5 (147 pb), Exon 6 (55 pb), Exon 7 (178 pb), Exon 8 (174 pb), Exon 9 (154 pb), Exon 10 (90 pb), Exon 11 (47 pb), Exon 12 (155 pb), Exon 13 (141 pb), Exon 14
(68 pb), Exon 15 (183 pb), Exon 16 (189 pb), Exon 17 (224 pb)
= Intron 1 (32 pb), Intron 2 (49 pb), Intron 3 (292 pb), Intron 4 (478 pb), Intron 5 (941 pb), Intron 6 (1486 pb), Intron 7 (447 pb), Intron 8 (526 pb),Intron 9 (527 pb), Intron 10 (1092 pb), Intron 11 (630 pb), Intron 12(12 pb), Intron 13 (918 pb), Intron 14 (495 pb), Intron 15 (79 pb), Intron 16 (60 pb)
Gambar 2. Struktur gen Laktoferin (Seyfert et al., 1994)
Kandungan tertinggi laktoferin ditemukan dalam kolostrum, kemudian kandungan laktoferin ditemukan ada pada susu (Adam et al., 2008). Konsentrasi laktoferin dalam susu sapi rata-rata 0,2 gram/liter. Dalam kolostrum, kandungan laktoferin dapat setinggi 0,5-1,0 gram/liter (Naim, 2003). Susu sapi yang terinfeksi
E.coli memiliki konsentrasi laktoferin yang lebih tinggi 30 kali dibandingkan dengan susu normal (Hyvonen, 2006). Tingginya kadar laktoferin pada sapi yang terinfeksi
E.coli disebabkan untuk pertahanan tubuh dari serangan E.coli, dimana laktoferin mengikat Fe dari bakteri tersebut. Laktoferin menurut Robble et al. (2003) dapat mereduksi keberadaan E.coli di dalam usus anak sapi dan mengurangi serangan diare.
Peranan Laktoferin
Laktoferin mampu menghambat aktivitas bakteri dan menyebabkan bakteri kehilangan kemampuan untuk membentuk koloni dengan cepat (Rainard, 1992). Adam et al. (2008) menyatakan bahwa laktoferin dapat mencegah tumor pada usus kecil atau lokasi lain. Laktoferin yang berasal dari sapi mempunyai antibakteri terhadap E.coli dan S. Aureus lebih tinggi bila dibandingkan dengan laktoferin yang berasal dari manusia, tikus dan kambing (Vorland et al., 1998). Secara alami protein yang bersifat antimikrobial mempunyai kemampuan aktivitas sebagai antiviral. Laktoferin mempunyai kemampuan menghambat replikasi Herpes Simplex Virus (HVS) dengan cara memblokir virus tersebut masuk ke dalam sitoplasma sel inang (Jenssen, 2005). Selain bersifat sebagai antimikroba dan antivirus, laktoferin
mempunyai kemampuan sebagai antistres seperti dilaporkan oleh Zimecki et al. (2005) bahwa pemberian laktoferin melalui air minum pada mencit
dapat meningkatkan respon imunitas baik secara seluler maupun humoral. Menurut Sumantri (2006), sapi perah FH yang menghasilkan susu berlaktoferin tinggi mempunyai beberapa manfaat, yaitu menurunkan kemungkinan mastitis, menurunkan total plate count (TPC) susu, menurunkan cemaran antibiotik pada produk susu akibat langsung dari pengobatan mastitis.
Keragaman Genetik
Dalam suatu populasi, keragaman genetik digunakan untuk mengetahui dan melestarikan bangsa-bangsa dalam populasi terkait dengan penciri atau suatu sifat khusus. Pengetahuan keragaman genetik suatu bangsa sangat bermanfaaat bagi keamanan dan ketersediaan bahan pangan yang berkesinambungan (Blott et al., 1998). Semakin beragam sumber daya genetik, akan semakin tahan populasi tersebut untuk hidup dalam jangka yang lama, serta semakin tinggi daya adaptasi populasi terhadap perubahan lingkungan (Frankham et al., 2002).
Keragaman genetik dihasilkan mutasi, sedangkan perubahan frekuensi alel disebabkan migrasi, seleksi, dan genetic drift (Frankham et al., 2002). Menurut Noor (2000) frekuensi genotipe dalam suatu populasi yang cukup besar akan selalu dalam keadaan seimbang bila tidak ditemukan seleksi, migrasi, mutasi dan genetik drift. Selain itu, silang luar dan silang dalam juga mempengaruhi frekuensi genetik. Keragaman genetik antara subpopulasi dapat diketahui dengan melihat persamaan dan perbedaan frekuensi alel diantara subpopulasi (Li et al., 2000).
Semakin tinggi derajat heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan semakin tinggi (Avise, 1994). Menurut Nei (1987), derajat heterozigositas adalah rataan presentase lokus heterozigositas tiap individu atau rataan presentase individu heterozigot dalam populasi. Avise (1994) menambahkan bahwa seiring dengan derajat heterozigositas yang menurun akibat dari silang dalam dan fregmentasi populasi, sebagian besar alel resesif yang bersifat lethal akan semakin meningkat frekuensinya.
yaitu AA (37,90%), BB (2,42%), dan AB (59,68%). Chang-hong et al. (2008) yang menggunakan enzim restriksi HinfI untuk menganalisis gen laktoferin pada peternakan sapi perah di Xinjiang Shihezi; menemukan dua alel A dan B dengan frekuensi masing-masing 0,78 dan 0,22 pada kelompok sapi kontrol, serta 0,17 dan 0,73 pada kelompok sapi pengamatan . Seyfert dan Kuhn (1994) menemukan mutasi pada intron 6 dengan menggunakan enzim restriksi EcoRI dan terdapat alel A dan B dengan masing-masing frekuensi sebesar 0,755 dan 0,245.
Kadar laktoferin sangat nyata dipengaruhi genetik dan gen laktoferin dikontrol dua alel yaitu A dan B, individu bergenotipe AA mempunyai kadar laktoferin lebih tinggi dari genotipe AB dan BB (Lee et al., 1997). Dengan demikian
laktoferin diwariskan secara kodominan dan mengikuti hukum Mendel (Lee et al., 1997).
Analisis Keragaman DNA
Polimerase Chain Reaction (PCR) adalah suatu reaksi in vitro untuk menggandakan molekul DNA pada target tertentu dengan cara mensintesis molekul DNA baru yang berkomplemen dengan molekul DNA tersebut dengan enzim polimerase dan oligonukleotida pendek sebagai primer dalam mesin thermocycler. Primer merupakan oligonukleotida spesifik yang menempel pada bagian sampel DNA yang akan diperbanyak. Hasil dari proses PCR dapat divisualisasikan melalui elektroforesis (Williams, 2005).
Proses yang terjadi dalam mesin PCR meliputi tiga tahap utama yaitu denaturasi (pemisahan untai ganda DNA), annealing (penempelan primer), dan ekstensi (pemanjangan primer) (Muladno, 2002). Keragaman DNA amplikon atau produk PCR dapat dianalisis dengan berbagai cara, antara lain Restriction Fragment Length Polymorphism (RFLP), Single Strand Conformation Polymorphism (SSCP),
Denaturing Gradient Gel Electrophoresis (DGGE), dan sekuensing .
Kelebihan dari RFLP adalah dapat mendeteksi sifat kodominan, artinya dapat membedakan antara yang homozigot dan heterozigot (Gupta et al., 2002).
Selain PCR-RFLP, PCR-SSCP juga dapat digunakan untuk analisis keragaman DNA. Asumsi yang mendasari metode analisis SSCP adalah bahwa perubahan yang terjadi pada nukleotida meskipun hanya terjadi pada satu basa, akan mempengaruhi bentuk (conformation) dari fragmen DNA pada kondisi untai tunggal (Bastos et al., 2001). Metode SCCP dapat mendeteksi mutasi pada fragmen DNA, akan tetapi tidak dapat memberikan informasi tentang posisi dimana terjadinya
mutasi pada fragmen DNA dan memiliki keterbatasan dalam menentukan jumlah alel (Baroso et al., 1999).
Temperature Gradient Gel Electrophoresis (TGGE) merupakan salah satu metode analisis keragaman yang mendeteksi mutasi dengan menggunakan gel yang memiliki perbedaan suhu (Jasik dan Reichert, 2006). DGGE merupakan salah satu metode yang dapat membedakan mutasi berdasarkan berat molekul fragmen DNA pada gel yang memiliki perbedaan konsentrasi bahan untuk menyamakan berat molekul (Liu et al., 2008).
MATERI DAN METODE
Lokasi dan Waktu
Penelitian ini dilaksanakan pada bulan Juni hingga Nopember 2010. Penelitian dilakukan di Laboratorium Pemuliaan dan Genetik Molekuler, Bagian Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Materi Sampel
Sampel yang digunakan berupa darah sapi Friesian Holstein (FH) sebanyak 89 sampel yang terdiri atas sapi jantan yang berasal dari BBIB Singosari sebanyak 32 ekor dan BIB Lembang 17 ekor; sapi betina dari BET Cipelang sebanyak 40 ekor. Darah dikoleksi juga dari sapi pedaging sebagai pembanding sebanyak 37 ekor sapi betina yang meliputi sapi Limosin sebanyak 14 ekor, Angus lima ekor, Simental 13 ekor dan Brahman lima ekor yang berasal dari BET Cipelang.
Pengambilan Sampel
Bahan yang digunakan adalah alkohol 70%, es, dan kapas. Alat yang digunakan antara lain jarum venoject, tabung vacutainer 10 ml dan termos. Sampel-sampel darah yang digunakan merupakan data sekunder hasil koleksi laboratorium Pemuliaan dan Genetika Molekuler, Bagian Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan.
Ekstraksi DNA
Bahan yang digunakan adalah Destilation Water (DW), NaCl, Proteinase-K, 1x STE (5M NaCl, 2M tris HCl, 0,2M EDTA), SDS 10% (sodium dodesil sulfat), CIAA (klorofom iso amil alkohol), fenol, etanol absolut, etanol 70% dan buffer TE 80% (tris EDTA). Alat yang digunakan adalah autoclave, satu set pipet mikro,
vortexmixer, alat pemutar, alat sentrifugasi, refrigerator dan freezer.
Primer
forward: 5´-GCC TCA TGA CAA CTC CCA CAC-3´; reverse: 3´-CAG GTT GAC ACA TCG GTT GAC-5´. Amplifikasi DNA dengan Polymerase Chain Reaction (PCR)
Bahan yang digunakan adalah DW, sampel DNA, buffer, MgCl2, pasangan primer, enzim taq dan dNTP, enzim retriksi EcoRI dan buffer EcoRI. Alat yang digunakan adalah satu set pipet mikro, alat sentrifugasi, tabung PCR, mesin PCR,
vortex, lemari es. Elektroforesis
Bahan yang digunakan adalah air destilasi, agarose, 0,5xTBE, Etidium Bromide, loading dye (0,01% Xylene Cyanol, 0,01% Bromtimol blue, 50% gliserol), dan marker 100 pb. Alat yang digunakan antara lain microwave, stearer, magnet stearer, gelas ukur, tabung erlenmeyer, gel tray, pencetak untuk sumur (comb),
power supply 100 volt, gelas ukur, pipet makro dan mikro.
Genotyping
Bahan-bahan yang digunakan yaitu loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%) dan untuk membuat 1 lembar gel agarose 2% adalah sebagai berikut: agarose 0,6 g; 0,5 TBE 30 ml, dan 2,5 µl Etidium Bromide. Alat-alat yang digunakan antara lain adalah microwave, stearer, magnet stearer, gelas ukur, tabung erlenmeyer, gel tray, pencetak untuk sumur (comb),
power supply 100 volt, gelas ukur, dan pipet mikro.
Prosedur Pengambilan Sampel
Ekstraksi DNA
Sampel darah yang disimpan dalam alkohol 70% diambil sebanyak 200 l, kemudian ditambahkan 1000µl Destilation Water (DW). Sampel digetarkan lalu didiamkan selama lima menit. Setelah itu disentrifuse pada kecepatan 8000 rpm selama lima menit, lalu supernatan dibuang, dan diulangi seperti proses sebelumnya, kemudian ditambahkan 10 l proteinase-K yang berfungsi untuk menghancurkan protein, 350 l 1xSTE (sodium tris-EDTA) dan 40 l 10% SDS (sodium dodesil sulfat) yang berfungsi untuk melisiskan membran sel. Campuran tersebut kemudian diinkubasi pada suhu 55oC selama dua jam sambil dikocok pelan dengan menggunakan alat pemutar (tilting).
Molekul DNA kemudian dimurnikan dengan metode fenol-chloroform, yaitu dengan menambahkan 40 l 5M NaCl, 400 l larutan fenol dan CIAA (chloroform iso amil alcohol), lalu dikocok pelan (tilting) pada suhu ruang selama 1 jam. Molekul DNA yang larut dalam fase air dipisahkan dari fase fenol dengan alat sentrifugasi pada kecepatan 12.000 rpm selama lima menit. Setelah terbentuk fase DNA, diambil sebanyak 40 l pada fase DNA untuk dipindahkan ke tabung baru 1,5 ml. Kemudian ditambahkan 5M NaCl sebanyak 40 l dan etanol absolut sebanyak 800 l. Molekul DNA kemudian dimalamkan (over night) pada suhu -20oC.
Molekul DNA kemudian dipisahkan dari etanol absolut dengan cara sentrifugasi pada kecepatan 12.000 rpm selama lima menit, kemudian supernatan yang diperoleh dibuang. Endapan yang terbentuk kemudian dicuci dengan menambahkan 70% etanol sebanyak 800 l dan disentrifugasi pada kecepatan 12.000 rpm selama lima menit. Supernatan yang diperoleh kemudian dibuang sehingga didapatkan endapan molekul DNA. Endapan tersebut didiamkan sampai kering. Lalu endapan DNA disuspensikan dalam 100 l 80% buffer TE (tris EDTA).
Amplifikasi DNA
detik, anneling 60 oC selama 45 detik dan elongasi 72 oC selama satu menit) dan
elongasi akhir 72 oC selama lima menit. Elektroforesis
Elektroforesis produk PCR dilakukan menggunakan 5 μl produk PCR pada gel agarose 1.5% pada tegangan 100 volt selama 30 menit. Gel dibuat dengan cara memanaskan agarose 0,45 g yang dilarutkan dalam larutan 0,5xTBE 30 ml serta menambahkan 2,5 µl EtBr pada saat distearer sampai didapatkan larutan jernih. Larutan yang masih cair dituangkan ke dalam pencetak gel serta menempatkan sisir di dekat tepian gel dan gel dibiarkan mengeras. Apabila gel sudah mengeras, sisir dicabut sehingga akan terbentuk sumur-sumur yang digunakan untuk menempatkan sebanyak 5µl produk PCR dicampur dengan loading dye (bromthymol blue 0,01%, Xylene cyanol 0,01% dan gliserol 50%). Gel ditempatkan ke dalam gel tray
elektroforesis yang sudah berisi larutan buffer dan dialiri listrik, molekul DNA yang bermuatan negatif pada pH netral akan bergerak (migrasi) ke arah positif (anode). Setelah elektroforesis selesai gel agarose diambil untuk dilihat panjang pita DNA dengan menggunakan sinar Ultraviolet yaitu dengan menarik garis lurus antara posisi pita dari masing-masing sampel DNA yang ingin diukur dengan posisi pita DNA marker, dapat mengestimasi ukuran sampel DNA karena ukuran DNA pengukur telah diketahui.
Genotyping
PCR-RFLP dilakukan dengan menggunakan lima μl produk PCR dipindahkan ke dalam tabung 0,5 ml yang ditambahkan 1 µl DW, 0,3 µl enzim restriksi EcoRI dan 0,7 µl buffer EcoRI. Campuran tersebut diinkubasikan dalam inkubator pada suhu 37 °C selama 16 jam. Sampel DNA yang telah dipotong dengan enzim restriksi dielektroforesis pada gel agarose 2% pada tegangan 100 volt selama 30 menit. Setelah elektroforesis selesai gel agarose diambil untuk melihat panjang pita DNA dengan menggunakan sinar ultraviolet dan dibandingkan dengan
sepanjang 301 pb, 201 pb dan 100 pb. Alel A pada fragmen 301 pb, sedangkan alel B pada fragmen 201 pb dan 100 pb. Genotipe AA ditunjukkan dengan hanya ditemukan satu fragmen 301 pb, genotip BB ditunjukkan dengan ditemukan tiga fragmen yaitu 301 pb, 201 pb, dan 100 pb, sedangkan untuk genotipe AB terdapat dua fragmen yaitu 201 pb, dan 100 pb.
Rancangan Frekuensi Alel dan Genotipe
Frekuensi genotipe merupakan rasio dari jumlah suatu genotipe terhadap jumlah populasi. Keragaman genotipe pada masing-masing individu ternak dapat ditentukan melalui pita-pita DNA yang ditemukan. Frekuensi genotipe dapat diketahui dengan menghitung perbandingan jumlah genotipe tertentu pada setiap populasi, dengan rumus Nei dan Kumar (2000) sebagai berikut :
Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada suatu lokus dalam populasi. Frekuensi alel ( ) gen laktoferin dapat dihitung berdasarkan rumus Nei dan Kumar (2000), sebagai berikut :
Keterangan :
= frekuensi genotipe ke-ii = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii
nij = jumlah individu bergenotipe ij
N = jumlah individu sampel
Keseimbangan Hardy-Weinberg (HW)
Keterangan: χ2
= chi-kuadrat
O = jumlah pengamatan genotipe ke-i E = jumlah harapan genotipe ke-i
Derajat bebas (db) dihitung berdasarkan Allendrof dan Luikart (2007) dengan menggunakan rumus:
Db = (Genotipe-1) – (Alel -1)
Heterozigositas
Keragaman genetik dapat diketahui melalui estimasi frekuensi heterozigositas pengamatan yang diperoleh dari masing-masing populasi, dengan menggunakan rumus Weir (1996) sebagai berikut :
Keterangan :
Ho = heterozigositas pengamatan (populasi) nij = jumlah individu heterozigot
N = jumlah individu yang diamati
Heterozigositas harapan (He) berdasarkan frekuensi alel dihitung menggunakan rumus Nei dan Kumar (2000) sebagai berikut :
Keterangan :
HASIL DAN PEMBAHASAN
Amplifikasi Gen LTF EcoRI
Proses amplifikasi gen LTF EcoRI pada sapi Friesian Holstein (FH) di Balai Inseminasi Buatan (BIB) Lembang, Balai Besar Inseminasi Buatan (BBIB) Singosari dan Balai Embrio Ternak (BET) Cipelang dilakukan dengan metode Polymerase
Chain Reaction (PCR). Pasangan primer yang digunakan berdasarkan Wojdak et al. (2006) yaitu dengan forward 5´-GCC TCA TGA CAA CTC CCA
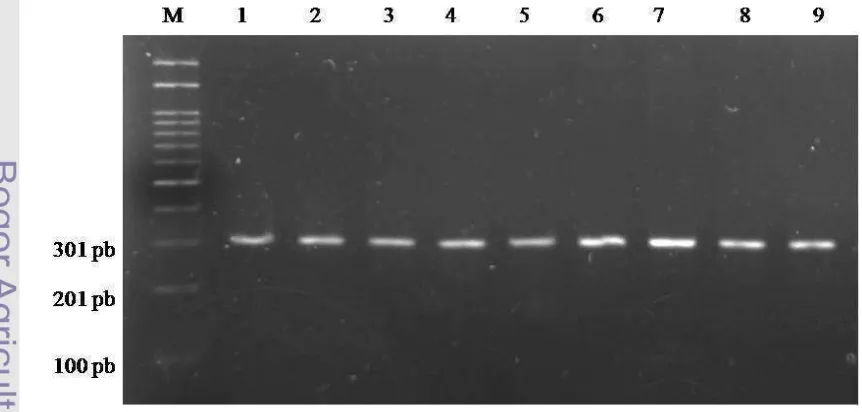
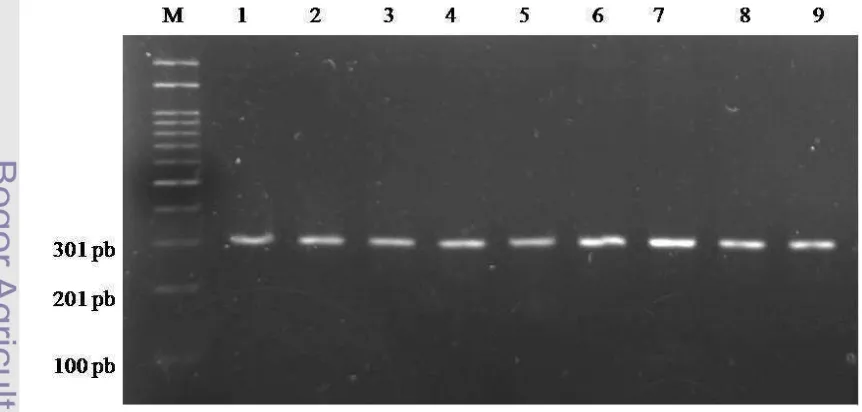
CAC-3´ dan reverse 3´-CAG GTT GAC ACA TCG GTT GAC-5´. Amplifikasi gen LTF EcoRI berhasil 100% (126/126) untuk semua sampel yang diteliti.
Amplifikasi gen LTF EcoRI menggunakan mesin Thermal Cycler pada kondisi annealing 60 oC selama 45 detik sehingga diperoleh produk PCR dengan panjang 301 pasang basa (pb). Suhu annealing menentukan keberhasilan amplifikasi karena proses pemanjangan DNA dimulai dari primer. Suhu annealing yang digunakan pada penelitian ini berdasarkan penelitian Chang-hong et al. (2008) yaitu 60oCselama 45 detik. Namun hal ini berbeda dengan penelitian yang dilakukan oleh Wojdak et al. (2006) yang menggunakan suhu 65 oC. Namun perbedaan suhu tersebut tidak terlalu berpengaruh terhadap hasil amplifikasi. Hasil amplifikasi fragmen gen LTF EcoRI sepanjang 301 pb pada gel agarose 1,5% disajikan pada Gambar 3.
Faktor-faktor yang menyebabkan berhasilnya amplifikasi gen LTF EcoRI diantaranya adalah kondisi penempelan primer DNA genom (gen target), bahan pereaksi PCR dan kondisi mesin PCR (thermal cycler). Viljoen et al. (2005) menyatakan bahwa beberapa hal umum dilakukan untuk optimasi hasil PCR diantaranya adalah suhu annealing (penempelan primer), konsentgrasi Mg2+, konsentrasi primer, dan konsentrasi DNA target. Posisi penempelan primer pada sekuen gen LTF EcoRI (Gen Bank Nomor Akses L19986.1) disajikan pada Gambar 4.
Forward
3241 aaaaaaactc aatatcccac ataaggtcac ttaacatagc ctcatgacaa ctcccacacc 3301 aaaacagtac tttattttgt aaattttgac cattattact cccatgttat ggtcttttca 3061 attggccact ttggccctgc tggcatatgt ttgggggtct tcatttattt attttttaat 3121 ccaatagcat tgacttttat tcattcatgt taagtcacct gaaatggtat ctgttgcatt 3181 taaatgctca attaatgtaa atggctgaac aaatccattt taatggatat tcaatccatt 3241 aaaaaaactc aatatcccac ataaggtcac ttaacatagc ctcatgacaa ctcccacacc 3301 aaaacagtac tttattttgt aaattttgac cattattact cccatgttat ggtcttttca 3361 gctgtcaagc aaacaaggtg aagaaaaaat ttagttagat gggggttgca cctggaaaat 3421 aaatttctta aactccatat acatgtttca aatctgctgg gtcccaagtc catctatgaa 3481 ttcccaggct gccagtatca tatgcagcat actaaagcta cgctatctga atagcttatt 3541 aattctgcat atatcaggtc aaccgatgtg tcaacctgta acagactgcg acacttttaa
Reverse Keterangan:
Terjadi Mutasi pada Situs Pemotongan Enzin Restriksi EcoRI (gaattc) pada Posisi 3419 (Intron 6) Alel A : 5‟…….CATCTATGATTTCCCAGGCT…..3‟
Alel B : 5‟…….CATCTATGAATTCCCAGGCT…..3‟
Gambar 4. Posisi Penempelan Primer (Cetak Tebal) pada Sekuen Gen LTF EcoRI (Gen Bank Nomor Akses L19986.1)
Pendeteksian Keragaman Gen LTF EcoRI
Pendeteksian keragaman gen LTF EcoRI pada sapi FH menggunakan metode
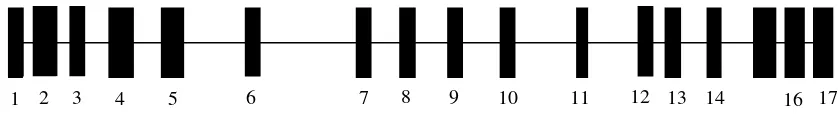
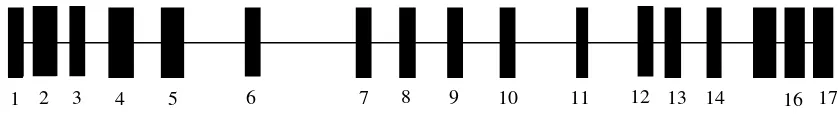
Gambar 5. Pola Pita Gen LTF EcoRI Sapi FH pada Gel Agarose 2% dengan Enzim Restriksi EcoRI. AA(1, 2, 5, dan 6), AB (3, 4, 7 dan 8), M (marker) Keragaman gen pada sapi FH di ketiga lokasi tersebut terjadi akibat mutasi. Mutasi adalah suatu perubahan struktur kimia gen yang berakibat perubahan fungsi gen. Mutasi yang terjadi pada penelitian ini adalah mutasi subtitusi yaitu perubahan basa antar basa-purin (A-G) atau basa pirimidin (T-C) (Gambar 4).
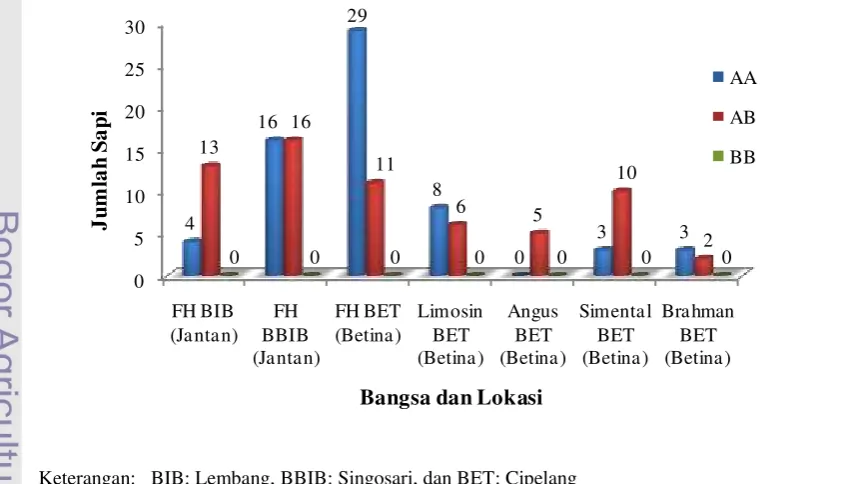
Jumlah sapi FH dan sapi pedaging dengan genotipe tertentu gen LTF EcoRI untuk setiap lokasi pengamatan diilustrasikan pada Gambar 6.
0
Keterangan: BIB: Lembang, BBIB: Singosari, dan BET: Cipelang
Alel yang muncul pada sapi FH di BIB Lembang, BBIB Singosari dan BET Cipelang adalah alel A dan alel B. Genotipe yang ditemukan untuk ketiga lokasi tersebut adalah AA dan AB, sebaliknya tidak ditemukan genotipe BB. Sapi yang bergenotipe AA akan lebih tinggi kandungan laktoferinnya dibandingkan dengan sapi yang bergenotipe AB atau BB, sehingga sapi yang bergenotipe AA lebih dipertahankan untuk dikembangbiakan. Menurut Lee et al. (1997) kadar laktoferin sangat nyata dipengaruhi oleh faktor genetik dan gen laktoferin dikontrol oleh dua alel A dan B, individu bergenotipe AA mempunyai kadar laktoferin lebih tinggi dari AB dan BB.
Frekuensi Genotip dan Alel Gen LTF EcoRI
Nilai frekuensi genotipe dan frekuensi alel gen LTF EcoRI populasi sapi di BIB Lembang, BBIB Singosari dan BET Cipelang dapat dilihat secara lengkap pada Tabel 1.
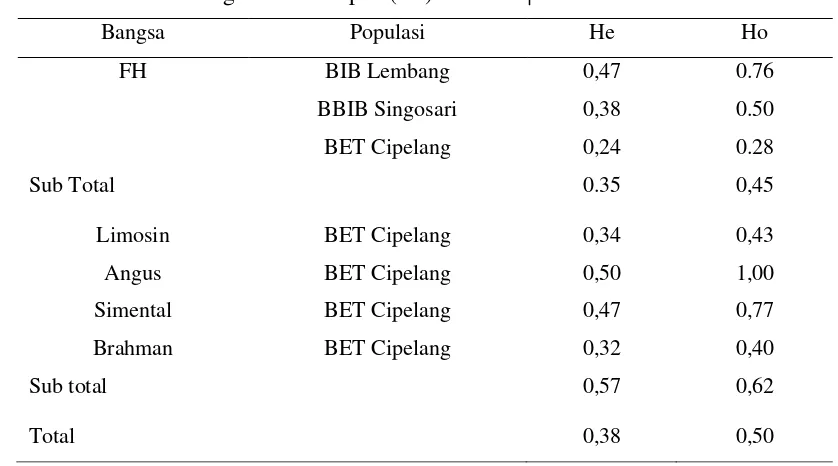
Tabel 1. Nilai Frekuensi Genotipe dan Alel Gen LTF EcoRI Sapi dari BIB Lembang, BBIB Singosari dan BET Cipelang
Bangsa/Populasi Genotipe Alel
AA AB BB A B
FH BIB Lembang (17) (4) 0,24 (13) 0,76 (0) 0,00 0,62 0,38
FH BBIB Singosari (32) (16) 0,50 (16) 0,50 (0) 0,00 0,75 0,25
FH BET Cipelang (40) (29) 0,72 (11) 0,28 (0) 0,00 0,86 0,14
Sub total (49) 0,55 (40) 0,45 (0) 0,00 0,77 0,23
Limosin (14) BET Cipelang (8) 0,57 (6) 0,43 (0) 0,00 0,79 0,21
Angus (5) BET Cipelang (0) 0,00 (5) 1,00 (0) 0,00 0,50 0,50
Simental (13) BET Cipelang (3) 0,23 (10) 0,77 (0) 0,00 0,62 0,38
Brahman (5) BET Cipelang (3) 0,60 (2) 0,40 (0) 0,00 0,80 0,20
Sub total (14) 0,38 (23) 0,62 (0) 0,00 0,69 0,31
Total (126) (63) 0,50 (63) 0,50 (0) 0,00 0,75 0,25
Keterangan : (..) = jumlah sapi
ditemukannya genotipe BB diduga karena jantan atau betina yang dipakai lebih banyak membawa genotipe AA dan AB dibandingkan dengan genotipe BB.
Nilai frekuensi genotipe AA tertinggi terdapat pada sapi FH di BET Cipelang sebesar 0,72, sedangkan frekuensi genotipe AB tertinggi terdapat pada sapi FH di BIB Lembang sebesar 0,76. Nilai frekuensi genotipe pada sapi pedaging untuk genotipe AA, paling tinggi nilainya terdapat pada sapi Brahman di BET Cipelang sebesar 0,60; dan sapi bergenotipe AB tertinggi terdapat pada sapi Angus sebesar 1,00. Frekuensi genotipe total untuk sapi FH dengan sapi pedaging berbanding terbalik, dimana pada sapi FH nilai frekuensi genotipe AA lebih besar dibandingkan dengan frekuensi genotipe AB, sedangkan pada sapi pedaging nilai frekuensi genotipe AB lebih besar dibandingkan dengan frekuensi genotipe AA. Total frekuensi genotipe AA dan AB untuk ketiga lokasi mempunyai proporsi yang sama, yaitu masing-masing 0,5.
Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi atau jumlah suatu alel terhadap jumlah total alel yang terdapat dalam suatu populasi (Nei dan Kumar, 2000). Sapi FH pada ketiga lokasi mempunyai frekuensi alel A yang lebih tinggi dibandingkan dengan alel B. Hal ini juga terlihat pada sapi pedaging bangsa Limosin, Angus, Simental dan Brahman yang digunakan sebagai pembanding. Frekuensi alel A dan B sapi FH pengamatan yaitu 0,77 dan 0,23. Hasil penelitian ini tidak berbeda jauh dengan penelitian sebelumnya oleh Wojdak et al. (2006), yang memperoleh bahwa sapi FH Polandia ditemukan alel A dan alel B dengan frekuensi alelnya masing-masing sebesar 67,74% dan 32,58%. Hal tersebut tidak berbeda dengan sapi pedaging, namun pada sapi Angus nilai frekuensi alel A dan B memiliki proporsi yang sama yaitu 0,5 dan 0,5.
Keseimbangan Hardy-Weinberg Gen LTF EcoRI
Hukum Hardy-Weinberg (HW) menggambarkan keseimbangan suatu lokus dalam populasi diploid yang mengalami perkawinan secara acak yang bebas dari faktor yang berpengaruh terhadap terjadinya proses evolusi seperti mutasi, migrasi dan pergeseran genetik (Gillespie, 1998). Noor (2000) juga menyatakan bahwa hukum Hardy-Weinberg menyatakan frekuensi genotipe suatu populasi yang cukup besar akan selalu dalam keadaan seimbang bila tidak ada seleksi, migrasi, mutasi dan
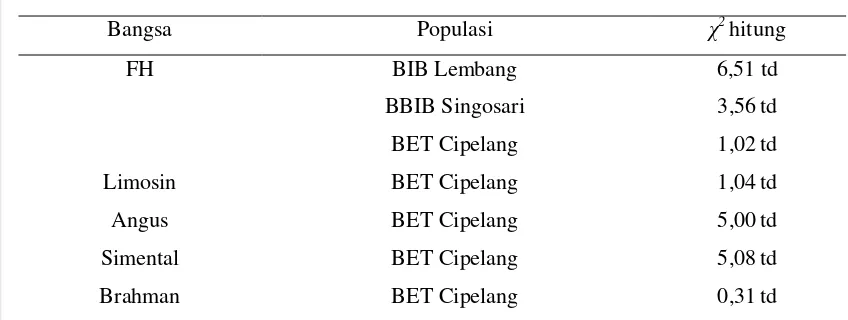
genetic drift. Hasil pengujian keseimbangan HW gen LTF EcoRI pada sapi di lokasi BIB Lembang, BBIB Singosari dan BET Cipelang disajikan pada Tabel 2.
Tabel 2. Uji Hasil Uji Signifikasi χ2 Keseimbangan Hardy-Weinberg Gen LTF EcoRI
Bangsa Populasi χ2 hitung
FH BIB Lembang 6,51 td
BBIB Singosari 3,56td
BET Cipelang 1,02td
Limosin BET Cipelang 1,04td
Angus BET Cipelang 5,00td
Simental BET Cipelang 5,08td
Brahman BET Cipelang 0,31td
Keterangan: td = tidak dapat dianalisis
Tabel 2 memperlihatkan bahwa pada sapi FH di ketiga lokasi,
keseimbangan HW tidak dapat dihitung atau dianalisis. Begitu juga pada sapi pedaging di BET Cipelang. Hal ini dikarenakan derajat bebas χ2 bernilai nol yang disebabkan pada hasil analisis genotipe hanya terdapat 2 macam genotipe dan 2 macam alel. Menurut Allendrof dan Luikart (2007), derajat bebas χ2
merupakan hasil pengurangan antara jumlah genotipe dengan jumlah alel.
Pendugaan Nilai Heterozigositas Gen LTF EcoRI
Keragaman genetik dapat diukur secara akurat dengan nilai heterozigositas (ĥ) (Nei, 1987). Pendugaan nilai heterozigositas pengamatan (Ho) dan nilai heterozigositas harapan (He) gen LTF EcoRI pada ketiga lokasi dapat dilihat secara lengkap pada Tabel 3.
Tabel 3. Pendugaan Nilai Heterozigositas Pengamatan (Ho) dan Nilai Heterozigositas Harapan (He) Gen LTF EcoRI
Bangsa Populasi He Ho
FH BIB Lembang 0,47 0.76
BBIB Singosari 0,38 0.50
BET Cipelang 0,24 0.28
Sub Total 0.35 0,45
Limosin BET Cipelang 0,34 0,43
Angus BET Cipelang 0,50 1,00
Simental BET Cipelang 0,47 0,77
Brahman BET Cipelang 0,32 0,40
Sub total 0,57 0,62
Total 0,38 0,50
Tabel 3 menunjukkan bahwa nilai heterozigositas gen LTF EcoRI pengamatan (Ho) lebih tinggi dibandingkan dengan nilai heterozigositas harapan
(He) untuk keseluruhan sapi pengamatan, baik sapi FH maupun sapi pedaging. Tambasco et al. (2003) menyatakan bahwa jika nilai heterozigositas pengamatan
(Ho) lebih rendah dari heterozigositas harapan (He) dapat mengindikasikan adanya derajat endogami (perkawinan dalam kelompok) sebagai akibat dari adanya proses seleksi yang intensif. Nilai Ho yang lebih tinggi dari He pada penelitian ini mengindikasikan bahwa tidak adanya proses seleksi yang intensif terhadap gen laktoferin.
KESIMPULAN DAN SARAN
Kesimpulan
Gen LTF EcoRI pada sapi FH di BIB Lembang, BBIB Singosari dan BET Cipelang serta sapi pedaging di BET Cipelang bersifat polimorfik. Tipe alel teridentifikasi yaitu A dan B dengan genotipe yang ditemukan hanya AA dan AB. Keseimbangan HW sapi FH dan pedaging pengamatan tidak dapat dihitung atau dianalisis. Nilai heterozigositas gen LTF EcoRI pengamatan (Ho) lebih tinggi bila dibandingkan dengan nilai heterozigositas harapan (He) untuk keseluruhan sapi pengamatan. Nilai heterozigositas tertinggi terdapat pada sapi FH di BIB Lembang sebesar 0,76 dan sapi angus di BET Cipelang sebesar 1,00.
Saran
UCAPAN TERIMAKASIH
Puji dan syukur Penulis panjatkan kehadirat Tuhan YME atas segala berkat dan karunia serta penyertaan-Nya sehingga Penulis dapat menyelesaikan penelitian dan penulisan skripsi ini. Penulis menyampaikan terimakasih sebesar-besarnya kepada Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. sebagai Pembimbing Utama dan Ir. Anneke Anggraeni, M.Si, Ph.D. sebagai Pembimbing Anggota yang banyak memberikan masukan, saran, dan pengarahan sehingga penulisan skripsi ini dapat diselesaikan. Terima kasih kepada Ir. Rini H. Mulyono, M.Si., Dr. Ir. Ahmad Darobin Lubis, M.Sc., M. Baihaqi, S.Pt., M.Sc. sebagai dosen penguji ujian lisan yang memberikan banyak masukan dan koreksi terhadap skripsi ini. Terima kasih kepada Dr. Ir. Rukmiasih, M.Si. selaku Pembimbing Akademik yang telah memberi pengarahan mulai tingkat awal hingga tingkat akhir. Terimakasih pula kepada seluruh staf pengajar Fakultas Peternakan yang telah memberikan ilmu dan pengalaman selama menyelesaikan pendidikan di Fakultas Peternakan IPB.
Tidak lupa penulis mengucapkan terimakasih kepada orang tua untuk dukungan, doa, kasih sayang, bantuan moril dan materil yang selalu diberikan hingga menyelesaikan tugas akhir. Terima kasih kepada Andika Widhi atas semua doa, dukungan, pengorbanan, kesabaran dan perhatian yang diberikan kepada penulis. Terimakasih pada teman-teman seperjuangan di Laboratorium Genetika Molekuler (Tifanny, Wieke, Icha, Diny, Paulina, Iren, Revi, Priskila, Rahmah, Leni, Ferdy, dan Erwin) serta Kak Erik, Kak Restu, Kak Surya, Pak Ihsan atas bantuannya selama penelitian. Terimakasih kepada teman-teman „Pondok Bidadari‟ (Sherli, Indah P, Winda, Tika, Listika, Nurul, Fatma, Selvi, Retno) atas dukungan, persahabatan, kebersamaan dan pengalaman selama ini. Terimakasih penulis ucapkan juga kepada Nur‟adhadinia dan seluruh teman-teman IPTP 44 yang tidak dapat disebutkan satu per satu atas semangat, persahabatan dan dukungannya selama kuliah hingga menyelesaikan tugas akhir. Semoga skripsi ini dapat bermanfaat.
DAFTAR PUSTAKA
Adam, V., O. Zitka, P. Dolezal, L. Zeman, A. Horna, J. Hubalek, J. Sileny, S. Kriskova, L. Trnkova, & R. Kizek. 2008. Lactoferrin isolation using monolithic column coupled with spectrometric or micro-amperometric detector. Sensors. 8: 464-487.
Allendrof, F. W. & G. Luikart. 2007. Conversation and the Genetics of Populations. Blackwell Publishing, USA.
Arnould, V. M. R., Soyeurt, H., Gengler, N., Colinet. F. G., Georges, M. V., Bertozzi, C., Portetelle, D., & Renaville, R. 2009. Genetic analysis of lactoferrin contentin bovine milk. J. Dairy. Sci. 92: 2151-2158.
Avise, J. C. 1994. Molecular Markers. Natural History Evolution. Chapman and Hall, Inc., Washington.
Barroso, A., S. Dunner, & J. Canon. 1999. Technical note: use of PCR-single-strand-conformation polymorphism analysis for detection of Bovine –casein variants A1, A2, A3, and B. J. Anim. Sci. 77: 2629-2632.
Bastos, E., A. Cradavor, J. Azevedo, & H. G. Pinto. 2001. Single strand conformation polymorphism (SSCP) detection in six genes in Portuguese indigenous sheep bred “Churra da Terra Quente”. Biothecnol. Agron. Soc. Environ. 5: 7-15.
Blott, S. C., J. L. Williams, & C. S. Haley. 1998. Genetic relationship among European cattle breeds. Anim. Genet. 29: 273-282.
Chang-hong, Z., Gao-ming, H. E., Yan-liang, W., & Zhao-xia, Z. 2008. Polymorphism analysis of the promoter of cow lactoferrin gene with PCR-RFLP and its correlation with subclinical mastitis. Acta. Agric. Slov. 92: 185-187.
Connely, O. M. 2001. Review: Antiinflamatory activities of lactoferrin. J. Am. Coll. Nutr. 20: 389-395.
Dwiyanto, K., A. Anggraeni, T. Sugiarti, Nurhasanah, H. Setyanto, & L. Praharani. 2001. Pengkajian system budidaya sapi perah untuk meningkatkan produktivitas. Prosiding Hasil Penelitian PUSLITBANGNAK, Bogor. Enseminger, M. E & D. T. Howard. 2006. Dairy Cattle Science. 4th Ed. The
Interstate Printers and Publisher, Inc., Danville.
Falconer, D. S. & Mackay, T. F. C. 1996. Introduction to Quantitative Genetics. Ed ke-4, New York.
Frankham, R., J. D. Ballou & D. A. Briscoe. 2002. Introduction to Conservation Genetics, Cambrigde University Press.
Green, E. K. 1998. Restriction fragment Length Polymorphism. In: Rapley, R and J. M. Walker. (Eds). Molecular Biology Methods Handbook. Human Press.Totowa, New Jersey.
Gupta, P. K., R. K. Vashney, & M. Prasad. 2002. Molecular Markers: Pronciples and Methodology. Dalam: Jain, S. M., D. S. Brar, and B. S. Ahloowalia (Eds.). Molecular Techniques in Crop Improvement. 9-54.
Hyvonen, P. 2006. Transgenic cows that produce recombinant human lactoferrin in milk are not protected from experimental Escherichia coli intramammary infection. Infect. Immun. 74: 6206-6212.
Jasik, A. & M. Reichert. 2006. Application of temperature-gradient gel electrophoresis for detection of prion protein gene polymorphism in Pollish Siniarka sheep. J. Vet. Diadn. Invest. 18: 170-274.
Javanmard, A., N. Asadazadeh., M. H. Banabazi, & J. Tavakolian. 2005. The allele and genotype frequencies of bovine pituitaryspecific transcription factor and leptin genes in Iranian cattle and buffalo populations using PCR-RFLP. Irianian. J. of Biotechnol. 3: 104-108.
Jenssen, H. 2005. Anti herpes simplex activity of lactoferrin/lactoferricin an example of antiviral activity of antimicrobial protein/peptide. Cell. Mol. Life. Sci. 62: 3002-3013.
Kanyshkova, T. G., S. E. Rabina, D. V. Semenov, N. Isaeva, A. V. Vlassov, K. N. Neustroev, A. A. Kulminskaya, V. N. Buneva, & G. A. Wevinsky. 2003. Multiple enzymatic of human milk lactoferrin. Eur. J. Biochem. 270: 3353-3361.
Lee, T. H., K. Shimizaki, S. L., Yu, M. S. Nam, S. J. Kim, K. K. Lee, & D. Y. Yu. 1997. Polymorphic sequence of Korean native goat lactoferrin exhibiting greater antibacterial activity. Anim. Genet. 28: 367-369.
Li, X., K. Li, B. Fan, y. Gong, S. Zhao, Z. Peng, & B. Liu. 2000. The genetic diversity of seven pigs breeds in China, estimated by means of microsatellites. J. Anim. Sci. 9: 1193-1195.
Liu, G., T. Amemiya, & K. Itoh. 2008. Two-dimensional DNA gel electrophoresis mapping: a novel approach to diversity analysis of bacterial communities in environmental soil. J. of Biosci. and Bioengine. 105: 127-133.
Muladno. 2002. Seputar Teknologi Rekayasa Genetik. Pustaka Wira Usaha Muda, Bogor.
Naim, R. 2003. Fungsi Biologik Lactoferrin. http://www.kompas.com/kompas-cetak/0308/11/ilpeng/4580666.htm. [22 Februari 2011].
Nei, M. 1987. Molecular Evolutionary Genetics. Columbia University Press, New York.
Noor, R. R. 2000. Genetica Ternak. Penebar Swadaya, Jakarta.
Orita, M., H. Iwahana, H. Kanazawa, K. Hayashi, & T. Sekiya. 1989. Detection of polymorphism of human DNA by gel electrophoresis in pigs. Anim. Sci. Pap. and Rep. 22: 57-64.
Putranto, E. H. 2006. Analisis keuntungan usaha peternakan sapi perah rakyat di Jawa Tengah. Tesis. Sekolah Pasca Sarjana, Universitas Diponegoro, Semarang.
Rainard, P. 1992. Binding of bovine lactoferrin to Streptococcus agalactiae. FEMS Microbiol. Lett. 177: 235-239.
Robble, E. D., P. S. Erickson, N. L. Whitehouse, A. M. McLaughlin, C. G. Schwab, J. J. Rejman, & R. E. Rompala. 2003. Supplemental lactoferrin improves health and growth of Holstein calves during the preweaning phase. J. Dairy Sci. 86: 1458-1464.
Sachaerczuk, M., T. Zagulski, B. Sadowski, M. Barchikowska, & R. Pluta. 2005. Lactoferrin in the central nervous system. Neurol. Neurochir. Pol. 39: 482-489.
Seyfert, H. M. & Kuhn, C. 1994. Characterization of a first bovine lactoferrin gene variant, based on an EcoRI polymorphism. Anim. Genec. 24-25.
Seyfert, H. M., Tuckoricz, A., Interthal, H., Koczan, D., & Hobom, G. 1994. Structure of the bovine lactoferrin-encoding gene and its promoter. Gene 143: 265-269.
Smith, K. L. & Oliver, S. P. 1981. Lactoferrin: A Component of Nonspecific Defense of The Involuting Bovine Mammary Gland. In: Butler J.E. (ed.): The Ruminant Immune System. Plenum Press, New York.
Sordillo, L. M., Shafer-Weaver, & K., DeRosa. 1997. Immunobiology of the
Suryanto, D. 2008. Melihat keanekaragaman organism melalui beberapa teknik genetika molekuler. Progam Studi Biologi. Fakultas Matematika dan Ilmu Pengetahuan Alam. Universitas Sumatera Utara. http://library. Usu. ac. Id. [4 April 2011].
Syarief, M. Z. & Sumoprastowo, R. M. 1984. Ternak Perah. Yasaguna, Jakarta. Talib, C., A. Anggraeni, & K. Dwiyanto. 2000. Evaluasi potensi genetiK sapi perah
Tambasco, D. D., C. C. P. Paz, M. Tambasco-Studart, A. P. Pereira, M. M. Alencor, A. R. Freitas, L L. Coutinho, I. U. Packer, & L. C. A. Regitano. 2003. Candidate genes for growth traits in beef cattle crosses Bos Taurus x Bos imdicus. J. Anim. Breed. Genet. 120: 51-60.
Teng, C. T. 2002. Lactoferrin gene expression and regulation: an overview. Biochem. and Cell Biol. 80: 7–16.
Viljoen, G. J., L. H. Nel, & J. R. Crowther. 2005. Molecular Diagnostic PCR Handbook. Springer, Dordrecht, Netherland.
Vorland, L. H., H. Ulvatne, J. Andersen, H. Haukland, O. Rekdal, J. S. Svendsen, & T. J. Gutterberg. 1998. Lactoferricin of bovine origin is more active than lactoferricins of human, murine and caprine origin, Scand. J. Infect. Dis. 30: 513-517.
Williams, J. L. 2005. The use of marker-asissted selection in animal breeding and biothecnology. Rev. Sci. Tech. Off. Int. Epiz. 24: 379-391.
Wojdak, M. K., M. Kmiec, & J. Ziemak. 2006. Associations between bovine lactoferrin gene polymorphism and somatic cell count in milk. Vet. Med. 51: 14–20.
Lampiran 1. Sekuen Gen Laktoferin pada Bos Taurus (Nomor Akses L19986)
Bos taurus lactoferrin gene, exons 3-6 </nuccore/L19986.1>
GenBank: L19986.1
FASTA </nuccore/L19986.1?report=fasta> Graphics </nuccore/L19986.1?report=graph> Related Sequences
</sites/entrez?Db=nuccore&DbFrom=nuccore&Cmd=Link&LinkName=nuccore_nuccore&I dsFromResult=408916>
* Features <#feature_408916> * Sequence <#sequence_408916>
LOCUS AH000852S03 3751 bp DNA linear MAM 11-OCT-2005
DEFINITION Bos taurus lactoferrin gene, exons 3-6. ACCESSION L19986 L19987
VERSION L19986.1 GI:408916 KEYWORDS lactoferrin.
SEGMENT 3 of 11 </nuccore/AH000852.1> SOURCE Bos taurus (cattle)
ORGANISM Bos taurus </Taxonomy/Browser/wwwtax.cgi?id=9913> Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;
Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 3751)
AUTHORS Seyfert,H.M., Tuckoricz,A., Interthal,H., Koczan,D. and Hobom,G. TITLE Structure of the bovine lactoferrin-encoding gene and its
promoter
JOURNAL Gene 143 (2), 265-269 (1994) PUBMED 8206385 </pubmed/8206385>
repeat_region </nuccore/408916?from=819&to=829&report=gbwithparts> 819..829
/note="putative" /rpt_type=direct
/rpt_unit_range=968..978
misc_feature </nuccore/408916?from=842&to=967&report=gbwithparts> 842..967
/note="retroposon; putative"
exon </nuccore/408916?from=1119&to=1266&report=gbwithparts> 1119..1266
/number=5
exon </nuccore/408916?from=2209&to=2264&report=gbwithparts> 2209..2264
/number=6 ORIGIN
1741 gcctcgcact gaagctgatg tttctccttt taccccatct gtctctcgga ctcaggcatt 1801 ttgtgtgtgg gggggaggcc tccccgattc tctgctgctc agggcacgtt taccacttag 1861 ccctgggagg ggaaaaaacc acgtggccgc atgtactgct tttaccctct ggtgcagagg 1921 cttggaaact cttttacaga gaaagacaaa gttttgccct ctcaatcaac ccttaaaggg 1981 gaaagtgggg ggtctctagc cagcatcctc tggttctttc ttcctcagaa tccatctgca 2041 ggtggatcta ttccctccat gggggcccca gagcctcaga gacctgtcct ctgcaggtca 2101 ggagcccaca gaagacaagc cccagggccc ctttccacag gcaggctcga ggaacaagcc 2161 gggctggctg ctctgtgctg acccttctgg tgctttctca tcccacaggt gtctgcagga 2221 cggggctgga gacgtggctt ttgttaaaga gacgacagtg tttggtaagt ggaggagaga 2281 agccacgggt actgcctcct ttgctttgct ttcctttcat ttcattgtaa atccaagtag 2341 tgagctaatt tgactcctca cttcctggca cacgattatc aaagctctct gtagagcaca 2401 gctcttattt ttatgtgact ttatatttta agcaatcagt tttctttgaa actcccgtgc 2461 ccgctatctt tctcttctgc atcaaagacg cccatgtcaa ggtatctgac acatgtcctc 2521 cgagacagat gtagtcttgc aaagtgtatg gggctgatct gtgtttctgg attctaagct 2581 cacagaaagt tttattgatc cgttattgat gatgagtgga tcaagtttcc cgctcagcat 2641 cctgttttag agatttagtg ttgttgtgct gtacatgtct ggttcagtgg ttctaacact 2701 gagacgtgct ctgtggagag cacacaacac atttattatc cacggtcgta aagcggtaga 2761 tacgtcagtg tggtttatct tgcatttctc tggcatctgg tgagtttgat catctcagta 2821 tatttgctgg tagtttgggc ttccctttct tcctttttca cctttgacta tctttctatt 2881 gattttcatg cctgtttctt catgattttg attcaagctg aggcattcct cctctctagg 2941 taacccgcca tggttggttc tagatagact ttaggaaaat ctcttctcag taaaaaaaaa 3001 aaaaaaaatc tgtcagtaac aggtttcttg aacagaagtc cttgatattg ataaagtcaa 3061 attggccact ttggccctgc tggcatatgt ttgggggtct tcatttattt attttttaat 3121 ccaatagcat tgacttttat tcattcatgt taagtcacct gaaatggtat ctgttgcatt 3181 taaatgctca attaatgtaa atggctgaac aaatccattt taatggatat tcaatccatt 3241 aaaaaaactc aatatcccac ataaggtcac ttaacatagc ctcatgacaa ctcccacacc 3301 aaaacagtac tttattttgt aaattttgac cattattact cccatgttat ggtcttttca 3361 gctgtcaagc aaacaaggtg aagaaaaaat ttagttagat gggggttgca cctggaaaat 3421 aaatttctta aactccatat acatgtttca aatctgctgg gtcccaagtc catctatgaa 3481 ttcccaggct gccagtatca tatgcagcat actaaagcta cgctatctga atagcttatt 3541 aattctgcat atatcaggtc aaccgatgtg tcaacctgta acagactgcg acacttttaa 3601 ctgaacatgg caaaatattc actctcttga actttacatc atctttgata tcaaacaatg 3661 aggcaaatcc taacagtata tcaaaaaaca acttttgagt tttacaaaag attgttccca 3721 tactgattaa tcaggcaagc taactcttag g
//
Lampiran 2. Perhitungan Derajat Bebas χ2 Gen LTF EcoRI
IDENTIFIKASI KERAGAMAN GEN LAKTOFERIN (LTF EcoRI)
PADA SAPI
FRIESIAN HOLSTEIN
DI BIB LEMBANG,
BBIB SINGOSARI DAN BET CIPELANG
SKRIPSI
GABBY ELFANDA MUMPUNIE
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
GABBY ELFANDA MUMPUNIE. D14070166. 2011. Idetifikasi Keragaman Gen Laktoferin (LTF EcoRI) pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari dan BET Cipelang. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D.
Laktoferin merupakan komponen dari protein susu yang bersifat antimikroba. Laktoferin yang terkandung di dalam susu berfungsi untuk mencegah diare, sedangkan bagi sapi laktoferin berfungsi untuk mencegah mastitis pada ambing. Dilihat dari peranan laktoferin tersebut, maka perlu adanya suatu usaha untuk meningkatkan kadar laktoferin di dalam susu. Salah satu cara untuk meningkatkan kemampuan sapi yang menghasilkan susu berkadar laktoferin tinggi adalah dengan melakukan seleksi pada tingkat DNA. Hal ini dapat dilakukan dengan cara mencari keragaman susunan nukleotida dalam gen yang berpengaruh terhadap kadar laktoferin susu. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen LTF EcoRI pada sapi Friesian Holstein (FH) di Balai Besar Inseminasi Buatan (BBIB) Singosari, Balai Inseminasi Buatan (BIB) Lembang dan Balai Embrio Ternak (BET) Cipelang; serta sapi pedaging di BET Cipelang sebagai pembanding.
Materi penelitian berupa sampel darah sapi FH sebanyak 89 ekor yang terdiri dari sapi jantan berasal dari BBIB Singosari (32 ekor) dan BIB Lembang (17 ekor), sapi betina dari BET Cipelang (40 ekor); serta sapi pedaging betina sebagai pembanding meliputi Limosin (14 ekor), Angus (5 ekor), Simental (13 ekor) dan Brahman (5 ekor) dari BET Cipelang. Amplifikasi gen LTF EcoRI dilakukan dengan
Polymerase Chain Reaction (PCR) pada gel agarose 1,5 %. Pendeteksian keragaman gen LTF EcoRI menggunakan metode Polymerase Chain Reaction-Restricsion Fragment Length Polymorphism (PCR-RFLP) dengan enzim restriksi EcoRI, sehingga didapatkan tiga macam fragmen yang panjangnya 301 pasang basa (pb) untuk alel A, 201 pb dan 100 pb untuk alel B. Keragaman genetik gen LTF EcoRI dianalisis dengan menghitung nilai frekuensi alel, frekuensi genotipe, keseimbangan Hardy-Weinberg, dan heterozigositas.
Hasil perhitungan frekuensi alel gen LTF EcoRI menunjukkan bahwa sapi FH pada ketiga lokasi dan sapi pedaging yang digunakan sebagai pembanding mempunyai frekuensi alel A yang lebih tinggi dibandingkan dengan alel B. Frekuensi total alel A dan B dari semua sapi FH pengamatan yaitu 0,77 dan 0,23. Hal tersebut juga tidak berbeda jauh dengan sapi pedaging yang diteliti. Genotipe yang ditemukan untuk sapi FH adalah AA dan AB, sebaliknya tidak ditemukan genotipe BB. Hasil perhitungan frekuensi genotipe sapi FH memiliki genotipe AA sebesar 0,55 dan genotipe AB sebesar 0,45, sedangkan genotipe BB bernilai 0. Nilai heterozigositas gen LTF EcoRI pengamatan (Ho=0,5) lebih tinggi dibandingkan dengan nilai heterozigositas harapan (He=0,38) untuk keseluruhan sapi pengamatan, baik sapi FH maupun sapi pedaging.
ABSTRACT
Identification of the Lactoferrin (LTF EcoRI) Gene Polymorphism in HolsteinFriesian Cattle in BIB Lembang, BBIB Singosari and BET Cipelang
Mumpunie, G. E. , C. Sumantri, and A. Anggraeni
Lactoferrin (LTF) is a gene controlling milk protein component in which having the characteristic as antimicrobial. LTF contained in the milk has function to prevent diarhea, whereas the function of LTF for the cows is to prevent mastitis in the udder. By looking at the role of the LTF gene, an attempt of improving LTF component in the milk is necessary through the selection process conducted at the DNA level. This study was aimed to identify the polymorphism of the LTF EcoRI gene in Holstein-Friesian (HF) cattle. The LTF EcoRI gene polymorphism was identified with Polymerase Chain Reaction-Restricsion Fragment Length Polymorphism (PCR-RFLP) method using EcoRI enzyme. Genotyping was perfomed in 89 HF cattle from Balai Inseminasi Buatan (BIB) Lembang (17 heads), Balai Besar Inseminasi Buatan (BBIB) Singosari (32 heads) and Balai Embrio Ternak (BET) Cipelang (40 heads). Genotyping was also conducted in four breeds of beef cattle as a comparison for Limosin (14 heads), Angus (5 heads), Simental (13 heads) and Brahman (5 heads) from BET Cipelang.
Results from the analysis of the allele frequencies of the LTF EcoRI gene in HF cattle in three locations and beef cattle showed that the A allele had a higher frequency than the B allele. The total frequencies of the A and B allels in HF cattle were 0,77 and 0,23 respectively. Genotypes for HF cattle observed were found for AA and AB, whereas the BB genotype was not found. The genotype frequencies of all cattle (HF and beef cattle) had the same proportion, genotype AA and AB had the same value (0,5), whereas the genotype BB was 0. Heterozygosity (Ho) value of LTF EcoRI gene were higher than those of heterozygosity value for all cattle. The conclusion was that LTF EcoRI gene in HF and beef cattle observed was polymorphic.
PENDAHULUAN
Latar Belakang
Sapi Friesian Holstein merupakan salah satu bangsa sapi perah yang paling banyak menghasilkan air susu. Sapi ini merupakan sapi yang umum dibudidayakan di Indonesia karena dapat beradaptasi di daerah tropis. Susu yang dihasilkan sapi perah menyediakan kebutuhan protein hewani yang bermanfaat bagi manusia. Konsumsi susu saat ini mulai meningkat seiring dengan kesadaran masyarakat akan pentingnya minum susu. Susu kaya akan nutrisi yang berguna bagi tubuh yang terdiri atas kasein dan whey protein yang mengandung laktoferin, laktoperoksidase, lisozim, dan immunoglobulin yang sering disebut sebagai protein anti mikroba. Kandungan protein yang tinggi dalam susu akan meningkatkan kualitas susu sebagai pangan fungsional.
Laktoferin yang disebut sebagai protein anti mikroba, merupakan salah satu protein minor yang secara alami ditemukan dalam susu sapi. Laktoferin adalah salah satu faktor penting untuk mencegah dan mengontrol mastitis pada sapi perah. Protein disintesis granulosit dan sel-sel epitel susu sebagai respons terhadap infeksi seperti mastitis. Laktoferin mampu mengikat ion besi (Fe) dari mikroba, sehingga dapat menghambat pertumbuhan mikroba. Laktoferin ditemukan dalam jumlah besar dalam sekresi mamalia seperti air susu, air mata, saliva, dan cairan seminal, sebagaimana pada beberapa sel darah putih. Laktoferin pada mamalia mempunyai fungsi utama yang berhubungan langsung dengan sistem kekebalan tubuh.
Tujuan
TINJAUAN PUSTAKA
Sapi Friesian Holstein
Bangsa sapi Friesian Holstein (FH) berasal dari negara Belanda tepatnya di Provinsi North Holland dan West Friesland, kedua daerah tersebut memiliki padang rumput yang bagus. Sapi FH memiliki kemampuan berkembang biak yang baik, rata-rata bobot badan sapi FH adalah 750 kg dengan tinggi bahu 139,65 cm. Kemampuan produksi susu sapi FH lebih tinggi dibandingkan bangsa sapi perah lain. Suhu lingkungan yang optimum untuk sapi perah dewasa berkisar antara suhu 5-21 ºC, sedangkan kelembaban udara yang baik untuk pemeliharaan sapi perah adalah sebesar 60% dengan kisaran 50% -75% (Enseminger dan Howard, 2006).
Syarief dan Sumopastowo (1984) menyatakan bahwa tanda-tanda yang dimiliki bangsa ini antara lain memiliki warna putih dengan belang hitam, dapat juga hitam dengan belang putih sampai warna hitam. Ekor harus putih, warna hitam tidak diperkenankan, juga tidak diperbolehkan warna hitam di daerah bawah persendian siku dan lutut, tetapi warna hitam diperbolehkan pada kaki mulai dari bahu atau paha sampai ke kuku. Gambar sapi FH dapat dilihat pada Gambar 1.
Sumber: http://dompi.co.id
Gambar 1. Sapi Friesian Holstein
(Dwiyanto et al., 2001). Talib et al. (2000) melaporkan bahwa rataan produksi susu sapi FH pada perusahaan lebih banyak dibandingkan dengan peternakan rakyat di pulau Jawa, yaitu sebesar 3565,8 ± 159,7 liter pada perusahaan dan 3266,4 ± 210,4 liter pada peternakan rakyat.
Gen Laktoferin
Gen laktoferin mengandung 17 ekson dalam suatu unit 34,5 kb transkripsional (Seyfert et al., 1994). Lebih dari enam dekade yang lalu, laktoferin ditemukan dalam susu sapi (Sorensen dan Sorensen, 1993). Laktoferin merupakan ikatan glikoprotein pengikat besi (Fe3+) yang pertama sekali diisolasi dari susu sapi dan selanjutnya dari ASI. Laktoferin ditemukan dalam jumlah besar dalam sekresi mamalia seperti susu, air mata, saliva, dan cairan seminal, begitu juga pada beberapa sel darah putih (Naim, 2003).
Laktoferin aktif dalam modulasi dan peraturan makrofag, fungsi limfosit dan neutrofil (Smith dan Oliver, 1981; Sordillo et al., 1997). Laktoferin adalah salah satu faktor penting untuk mencegah dan mengontrol mastitis pada sapi perah karena sifat-sifatnya (Teng, 2002). Penelitian sebelumnya menemukan bahwa laktoferin dapat digunakan sebagai konsentrasi sel somatik penanda genetik dalam susu, yaitu sebagai penanda kerentanan atau ketahanan terhadap mastitis pada sapi perah (Wojdak et al., 2006). Laktoferin terdiri atas rantai polipeptida tunggal dengan dua globular lobes (N-lobe dan C-lobe) dan relatif resisten terhadap proteolisis. Laktoferin merupakan protein yang disekresikan oleh jaringan ektodermal dan mempunyai struktur mirip transferin.