KONSTRUKSI VEKTOR RNAi DARI FRAGMEN 3’UTR GEN
PENYANDI COPPER/ZINC SUPEROXIDE DISMUTASE DARI
Melastoma malabathricum, L.
Abstrak
Tehnik RNA silencing adalah sebuah cara yang efektif untuk menguji fungsi biologi mRNA target pada tanaman. Perkembangan terkini mengenai teknologi pengklonan GATEWAYTM memudahkan untuk mengkonstruksi vector RNAi yang mempunyai sekuen pemicu dan untuk menganalisis fungsi gen target. Tujuan penelitian ini adalah mengkonstruksi RNAi dari fragmen 3’UTR gen penyandi CuZn-SOD dari Melastoma malabathricum L. (MmCuZn-SOD). Vektor RNAi telah berhasil dikonstruksi menggunakan teknologi pengklonan GATEWAYTM dimana fragmen 3’UTRMmCuZn-SOD digunakan sebagai sekuen pemicu RNA utas ganda (dsRNA), pENTRTM/D-TOPO® sebagai entry vector dan vektor pANDA sebagai destination vector. RNAi telah diintroduksikan ke tanaman M. malabathricum L. melalui Agrobacterium tumefaciens LBA4404 untuk mempelajari peranan gen MmCuZn-SOD dalam detoksifikasi cekaman Al. Uji toleransi tanaman transgenik terhadap cekaman Al menunjukkan bahwa pada media MS yang mengandung 1 mM Al (AlCl3.6H2O), tanaman transgenik mengalami hambatan pertumbuhan sampai mati, sementara non-transgenik tidak mengalami hambatan. Hal ini menunjukkan bahwa penghambatan ekspresi gen MmCuZn-SOD dengan RNAi pada tanaman M. malabathricum L. menyebabkan tanaman menjadi sensitif terhadap Al.
Abstract
The RNA silencing technique is an effective tool to examine the biological function of the target mRNAs in plants. The recent development of regarding GATEWAYTM cloning technology makes it easy to construct the RNAi vectors with trigger sequences and to analyze the function of a target gene. The objective of this study is to construct RNAi including the 3'UTR fragment of the gene coding CuZn-SOD from Melastoma malabathricum L. (MmCuZn-SOD). RNAi vector had been successfully constructed using Gateway cloning technology with the 3'UTR MmCuZn-SOD was used as double stranded RNA (dsRNA) trigger sequence, pENTRTM/D-TOPO® as entry vector, and pANDA as destination vector. RNAi had been successfully introduced into M. malabathricum L. mediated by Agrobacterium tumefaciens LBA 4404 to analyze the function of MmCuZn-SOD gene in the detoxifying Al stress. Transgenic plants tolerance analyzes to Al stress showed that in the MS medium containing 1 mM Al (AlCl3.6H2O), the transgenic plants underwent growth suppression, whereas non-transgenic plants underwent growth normally. These results suggested that suppression of MmCuZn-SOD gene expression by RNAi to M. malabathricum L. caused the plant became sensitive to Al.
Pendahuluan
Perkembangan teknologi rekayasa genetika yang sangat pesat memberikan harapan baru dalam memanipulasi tanaman agar dihasilkan varietas-varietas baru dengan sifat-sifat unggul termasuk tanaman yang tahan terhadap cekaman lingkungan. Langkah awal dalam rekayasa genetik adalah mempelajari peranan gen tertentu. Peranan suatu gen dalam tanaman dapat dipelajari minimal dengan pendekatan dua arah, yaitu meningkatkan ekspresi gen dengan mengkonstruksi vektor over expression serta menghentikan dan/atau menurunkan ekspresi gen antara lain dengan mengkonstruksi RNAi (RNA interference).
RNAi merupakan potongan kecil RNA yang dapat menginduksi penghancuran mRNA tertentu sebelum proses transkripsi di dalam sitoplasma. Prinsip dasarnya adalah bahwa masuknya RNA utas ganda (dsRNA) ke dalam sitoplasma akan membungkam ekspresi suatu gen di tingkat post-transkripsi (Fire et al. 1998; Kalantidis et al. 2008). Adanya RNAi menyebabkan RNA yang ada di dalam sel dapat diikat oleh RNAi untuk membentuk RNA utas ganda (dsRNA). RNA utas ganda dipotong oleh enzim Dicer di sitoplasma menjadi fragmen dsRNA pendek berukuran 21-26 nukleotida dengan ujung 3’ overhangs dua nukleotida (Fire et al. 1998; Waterhouse et al. 2001; Hannon 2002; Pickford & Cogoni 2003). Salah satu dari kedua utas dari masing-masing fragmen, yang diketahui sebagai utas pemandu (guide strand), bergabung dengan RISC (RNA-induced silencing complex) dan berasosiasi dengan mRNA target (Hammond et al. 2000), dan kemudian mengaktivasi fungsi RISC untuk mendegradasi mRNA target dan menekan ekspresi gen pada berbagai level (Fire et al. 1998; Meister & Tuschl 2004).
Berdasarkan mekanisme selulernya, pembentukan dsRNA dapat terjadi melalui dua cara, yaitu secara eksogenous dan endogenous (Bagasra & Priliman 2004). Secara eksogenous, dsRNA yang berasal dari infeksi virus dengan sebuah genom RNA atau dari manipulasi laboratorium yang secara langsung masuk dalam sitoplasma dan dipotong menjadi fragmen kecil oleh enzim Dicer. Secara endogenous, dsRNA berasal dari dalam sel sebagai pre-microRNA yang diekspresikan dari gen penyandi RNA dalam genom. Transkrip dari gen tersebut membentukstruktur jepit rambut (stem-loop) yang kemudian dikeluarkan ke sitoplasma untuk dipotong oleh Dicer.
Teknik RNAi ini merupakan cara yang efektif untuk menguji fungsi biologi mRNA target pada tanaman. Perkembangan terkini mengenai vektor RNAi, yang menggunakan promotor kostitutif dan teknologi teknik pengklonan Gateway, memudahkan untuk mengkonstruksi vektor RNAi yang mempunyai sekuen pemicu dsRNA dan memudahkan pula untuk menganalisis fungsi gen target. Salah satu vektor RNAi yang dapat digunakan untuk mempelajari fungsi gen target adalah vektor pANDA yang dikembangkan oleh Miki & Shimamoto (2004). Vektor ini menggunakan promoter ubiquitin1 dari jagung yang mengontrol ekspresi mRNA berulang terbalik dari sekuen gen target. Orientasi mRNA berulang terbalik merupakan pemicu pembentukan siRNA. Aplikasi vektor ini dengan gen penyandi green fluorescent protein (GFP) dan phytoene desaturase (PDS) dapat menekan ekspresi kedua gen tersebut pada tanaman padi (Miki et al. 2005).
Miki et al. (2005) juga menggabungkan sekuen unik berulang terbalik dari 3’UTR (untranslated region) yang diperoleh dari family gen OsRac padi dengan promotor kuat dan stabil lalu diintroduksikan ke dalam padi. Masing-masing dari sembilan anggota family gen OsRac eksperesinya ditekan oleh kontruksi berulang terbalik tersebut. Ekspresi mutan berulang terbalik menggunakan gabungan dari beberapa 3’UTR membungkam ekspresi tiga anggota family gen OsRac. Selanjutnya, dengan menggunakan daerah yang terkonservasi tinggi dari family gen OsRac, Miki et al. (2005) juga berhasil menekan ekspresi semua anggota family gen OsRac dengan efesiansi yang berbeda-beda. Hasil tersebut menunjukkan bahwa RNAi merupakan metoda yang bermanfaat untuk menganalisis fungsi famili gen pada padi dan tanaman lain.
Teknologi RNAi ini telah digunakan untuk mempelajari fungsi suatu gen pada tanaman (Wesley et al. 2001; DeBoer et al. 2011; Muzuni 2011), menapis gen fungsional untuk mengidentifikasi target terapi pada hewan (Soutchek et al. 2004; Shen et al. 2005; Raoul et al; 2005), pengobatan infeksi virus berbahaya pada manusia (Leonard & Schaffer 2006; Ma et al. 2007), dan pengobatan terapi kanker (Yin et al. 2003; Abdelrahim et al. 2006; Pai et al. 2006).
Salah satu gen yang diduga terlibat dalam toleransi tanaman terhadap cekaman lingkungan termasuk cekaman aluminium adalah gen penyandi superoxide dismutase (SOD) (Darko et al. 2004; Du et al. 2010). Superoxide dismutase (SOD) termasuk kelompok metalloenzim yang mampu menetralkan radikal bebas dengan mengkatalisis dismutase radikal superoksida menjadi
molekul O2 dan H2O2 (Tseng et al. 2008). Salah satu tipenya adalah copper/zinc superoksida dismutase (CuZn-SOD) yang memiliki kofactor logam copper/zinc. Pada tanaman CuZn-SOD pada umumnya ditemukan di sitosol dan kloroplast.
cDNA utuh dari gen CuZn-SOD dari Melastoma malabathricum L. telah berhasil diisolasi dari penelitian sebelumnya. Pada percobaan ini, kami menguji peranan gen penyandi MmCuZn-SOD dalam toleransinya terhadap cekaman Al. Pada pengujian ini, kostruksi RNAi menggunakan vektor pANDA dan dua sekuen 3’UTR dari gen MmCuZn-SOD yang ekspresinya berlawanan sebagai sekuen berulang terbalik. Vektor hasil konstruksi diintroduksikan ke dalam tanaman M. malabathricum L. melalui perantara A. tumefaciens LBA 4404. Tanaman transgenik selanjutnya diuji pada media yang mengandung Al. Tanaman M. malabathricum adalah tanaman yang sangat toleran Al, bilamana MmCuZn-SOD berperan dalam toleransi M.malabathricum L. terhadap cekaman Al, maka gangguan ekspresi gen ini melalui RNAi menyebabkan tanaman transgenik yang diperoleh adalah sensitif terhadap cekaman Al.
Bahan dan Metode Penelitian Bahan Penelitian
Plasmid pGEM-MmCuZn-SOD digunakan sebagai sumber fragmen 3’UTR MmCuZn-SOD. Plasmid biner pANDA (koleksi Prof. Ko Shimamoto, NAIST, Japan) digunakan sebagai vektor RNAi, plasmid pENTR (Invitrogen, USA) sebagai entry clone ke plasmid biner pANDA, A. tumefaciens LBA 4404 digunakan untuk mentransfer gen ke genom tanaman, E. coli HB 101 yang membawa helper pRK 2013 sebagai plasmid konyugasi untuk memindahkan plasmid biner rekombinan yang mengandung fragmen 3’UTR MmCuZn-SOD dari bakteri E.coli ke A. tumefaciens. E.coli DH5α digunakan untuk memperbanyak plasmid RNAi rekombinan yang telah mengandung fragmen 3’UTR MmCuZn-SOD, dan kultur invitro M.malabathricum digunakan sebagai bahan tanaman.
Metode Penelitian
Perbanyakan Fragmen 3’UTR MmCuZn-SOD. Fragmen 3’UTR MmCuZn-SOD diamplifikasi menggunakan enzim DNA polimerase proofreading untuk menghasilkan produk PCR ujung tumpul (blunt end) dengan primer spesifik (SOD3UTR-F1 : 5’-CAC CAG GGT TAA GCT ACC TCT G -3’ &
SOD3UTR-R1 : CCC AAG ATC AGT AAC ACC CGG TT) dengan penambahan nukleotida CACC pada primer forward agar dapat disisipkan pada situs pengklonan plasmid pENTR-D/TOPO (Gambar 2) dan pGEM-MmCuZn-SOD (Gambar 13) sebagai cetakan. Komposisi PCR yang digunakan adalah 10 ng pGEM-MmCuZn-SOD, 0.5 µM primer SOD3UTR-F, 0.5 µM primer SOD3UTR-R, 1x buffer Tag, 0.2 mM dNTP mix, 1.25 U Platinum Taq (invitrogen) DNA polymerase dan ddH2O hingga volume reaksi 20 µM. PCR dilakukan sebanyak 30 siklus dengan kondisi pra-PCR pada 95oC selama 5 menit, denaturasi pada 94oC selama 1 menit, penempelan primer pada 58oC selama 30 detik, pemanjangan 72oC selama 1 menit, dan pasca-PCR pada 72oC selama 5 menit. Produk PCR selanjutnya dielektroforesis menggunakan gel agarose 1 % untuk mengetahui integritas dan kuantitasnya.
Penyisipan Fragmen 3’UTRMmCuZn-SOD ke dalam Entry Vector. Metode penyisipan fragmen 3’UTR MmCuZn-SOD ke dalam entry vector pENTRTM/D-TOPO® sesuai protokol Invitrogen.
Pengklonan ke dalam Vektor pANDA. Vektor pENTR-D/TOPO rekombinan (entry clone) yang telah mengandung 3’UTR MmCuZn-SOD direkombinasikan dengan vektor pANDA menggunakan enzim LR clonase. Campuran reaksinya adalah sebagai berikut: 2 µl buffer reaksi LR (5x), 300 ng entry clone, 300 ng vektor pANDA, dan buffer TE hingga volume 8 µl. Selanjutnya 2 µl enzim LR clonase ditambahkan ke dalam campuran dan diinkubasi pada suhu 25oC selama semalam. Reaksi pada campuran dihentikan dengan menambahkan 1 µl proteinase K (2 µg/ µl) dan diinkubasi pada suhu 37oC selama 10 menit. Hasil rekombinasi diintroduksikan ke dalam E.coli DH5α dengan mencampur 10 µl larutan rekombinasi dengan 100 µl E.coli DH5α kompeten. Tahapan transformasi dilakukan sama dengan sebelumnya. Hasil proses transformasi disebar pada media LB padat yang mengandung 50 mg/L kanamisin dan 50 mg/L higromisin. Bakteri yang tumbuh diverifikasi dengan PCR koloni menggunakan primer spesifik SOD3UTR-F dan SOD3UTR-R.
Transformasi A. tumefaciens LBA 4404 dengan Vektor RNAi. Vektor pANDA yang telah mengandung fragmen 3’UTRMmCuZn-SOD (pAND-MmCuZn-SOD) diintroduksikan ke dalam bakteri A.tumefaciens melalui metode triparental mating (TPM) seperti pada BAB V. Bakteri A. tumefaciens yang tumbuh pada media seleksi yang mengandung 50 mg/L kanamisin, 50 mg/L higromisin, dan
streptomisin 50 mg/L diverifikasi dengan PCR koloni menggunakan primer spesifik SOD3UTR-F dan SOD3UTR-R.
Transformasi M.malabathricum dengan Vektor RNAi. Transformasi dilakukan dengan menggunakan A.tumefaciens dan eksplan daun menurut prosedur Akashi et al. (2005) yang telah dimodifikasi oleh Darojat (2010).
Eksplan daun dari tanaman M. malabathricum hasil perbanyakan in vitro berumur empat minggu dipotong dengan ukuran 1-2 cm. Ko-kultivasi dilakukan dengan merendam eksplan dalam suspensi A. tumefaciens OD600 0,5-0,8 selama 60 menit, digoyang 100 rpm pada suhu ruang. Selanjutnya eksplan dikeringkan dengan kertas tissue steril dan diletakkan dalam media MS0 dengan penambahan 0,1 mg/L NAA, 1 mg/L BAP dan 40 mg/L asetosiringon selama 3 hari di ruang gelap. Setelah ko-kultivasi, eksplan dicuci dengan air steril sebanyak tiga kali dan dilanjutkan dengan larutan 500 mg/L carbenicillin dan 200 mg/L cefotaxime. Eksplan dikeringkan dan dipindahkan ke dalam media MS0 yang mengandung 1 mg/L NAA, 1 mg/L BAP, 100 mg/L kanamisin, 10 mg/L higromisin, 200 mg/L carbenicillin dan 100 mg/L cefotaxime, dan kultur tanaman disimpan dalam ruang dengan suhu 26º-27ºC. Setelah 4 minggu, eksplan yang berkalus dipindahkan ke dalam media MS0 yang mengandung 1 mg/L NAA, 1 mg/L BAP, 100 mg/L kanamisin dan 20 mg/L higromisin. Setiap 4 minggu sekali kalus yang masih hidup disubkultur (regenerasi) ke media yang sama sampai bertunas. Tunas-tunas yang muncul disubkultur ke media seleksi MS0 yang mengandung 100 mg/L kanamisin dan 30 mg/L higromisin sampai tunas menjadi besar.
Identifikasi Tanaman Transgenik. Tanaman transgenik diidentifikasi menggunakan PCR dengan primer higromisin-F 5’-AAG GAA TCG GTC AAT ACA CTA C-3’ dan higromisin-R 5’-ACT ATC GGC GAG TAC TTC TAC A-3’. Isolasi DNA mengikuti metode CTAB. Daun sebanyak 0.1 g digerus dengan bantuan nitrogen cair di dalam mortar sampai menjadi tepung. Hasil gerusan dimasukkan ke ependorf yang telah berisi 500 µl buffer ekstraksi (2 % CTAB, 2% PVP 40000, 100 mM Tris-HCl pH 8, 20 mM EDTA, 1.4 M NaCl dan 1% β-mercapto ethanol), kemudian divortex dan diinkubasikan pada suhu 65oC selama 10 menit. Sebelum ditambahkan 1 x volume kloroform:isoamil alkohol (24:1), suspensi didinginkan terlebih dahulu. Campuran kemudian divortex dan disentrifus pada kecepatan 18000 x g (TOMY MX-205) pada suhu 4oC selama 10 menit. Cairan bagian atas yang mengandung DNA total diambil, dimasukkan ke
dalam tabung 1.5 ml dan diekstraksi kembali dengan 1 x volume phenol:kloroform:isoamilalkohol (25:24:1), divortek dan disentrifus pada kecepatan 18000 x g suhu 4oC selama 10 menit. Cairan bagian atas dipindahkan ke ependof baru, ditambahkan 2 x volume etanol absolut dan diinkubasi pada suhu -30oC selama 2 jam. Campuran disentrifus pada 18000 x g pada suhu 4oC selama 15 menit. Cairan dibuang, dan endapan DNA disuspensikan dalam 500 µl TE (10 mM Tris pH 7.4, 1 mM EDTA) dan ditambahkan 1 x volume phenol pH 8, divortek dan disentrifus pada 18000 x g pada suhu 20oC selama 10 menit. Cairan bagian atas yang mengandung DNA diambil, dimasukkan ke dalam tabung baru 1.5 ml dan ditambahkan 2 x volume etanol absolut dan diinkubasi pada -30oC selama 3 jam. Cairan disentrifus pada kecepatan 18000 x g, suhu 4oC selama 15 menit. Endapan DNA dibilas dengan 500 µl alkohol 70% dan disentrifus pada 18000 x g suhu 4oC selama 5 menit. Endapan DNA dikeringkan dengan vaccum dryer, dan disuspensikan di dalam dH2O. Untuk menghilangkan RNA ditambahkan RNAse . Kualitas dan kuantitas
DNA ditentukan dengan spektrometer UV pada panjang gelombang 260 nm dan 280 nm. Keutuhan DNA dianalisis dengan elektroforesis pada gel agarose 1% di dalam larutan penyangga TAE 1x. Visualisasi DNA dilakukan di atas UV transiluminator GelDoc (Labquip) setelah diwarnai dengan EtBr (0.5 µg/ml) selama 15 menit dan dibilas dengan air.
Komposisi PCR untuk mendeteksi tanaman transgenik adalah 10 ng DNA total, 0.5 µM primer higromisin-F, 0.5 µM primer higromisin-R, 1x buffer Tag, 0.2 mM dNTP mix, 1.25 U DNA Polymerase (Fermentas) dan ddH2O
hingga volume reaksi 20 µM. PCR dilakukan sebanyak 30 siklus dengan kondisi pra-PCR 95oC, 5 menit; denaturasi 94oC, 1 menit; penempelan primer 58oC, 1 detik; pemanjangan 72oC, 1 menit; dan pasca-PCR 72oC, 5 menit.
Uji Toleransi Tanaman Transgenik Terhadap Cekaman Al. Tanaman transgenik yang telah berumur 6 bulan setelah transformasi dipotong bagian atasnya hingga 3-4 buku dari pucuk lalu ditumbuhkan di dalam media MS0 yang mengandung Al 1 mM AlCl3.6H2O pH 4.5. Kemudian diamati penghambatan
Hasil dan Pembahasan Konstruksi Vektor RNAi
Fragmen 3’UTR MmCuZn-SOD yang berukuran 200 pb telah berhasil diisolasi dari plasmid pGEM-MmCuZn-SOD (Gambar 31). PCR dilakukan dengan menggunakan enzim Taq polymerase yang menghasilkan ujung yang tumpul (blunt end) yang tidak mengandung nukleotida A. Hal ini dimaksudkan agar ujung 5’ fragmen hasil PCR adalah CACC supaya dapat tersisipi ke vektor pENTRTM/D-TOPO®.
Gambar 31. Hasil isolasi 3’UTR MmCuZn-SOD melalui PCR pGEM-MmCuZn-SOD sebagai cetakan dan primer pGEM-MmCuZn-SOD3UTR-F dan pGEM-MmCuZn-SOD3UTR-R.
Produk PCR yang berukuran 200 pb ini telah berhasil diligasikan dengan pENTR-D/TOPO. Keberhasilan ini ditunjukkan oleh koloni bakteri E.coli yang tumbuh dalam media seleksi LB padat yang mengandung antibiotik kanamisin 50 mg/L setelah ditransformasikan dengan hasil ligasi tersebut. Hasil PCR koloni dengan primer SOD3UTR-F dan SOD3UTR-R menunjukkan bahwa koloni yang tumbuh pada media seleksi tersebut membawa fragmen 3’UTR gen MmCuZn-SOD (Gambar 32). Hal ini menunjukkan bahwa fragmen 3’UTR gen MmCu/Zn-SOD telah berhasil tersisipi ke vektor pENTR-D/TOPO yang selanjutnya disebut plasmid pENTR-3UTRMmCuZn-SOD. cDNA yang tersisipi ke dalam pENTR dapat dipastikan memiliki arah yang tepat karena kesepesifikan pada ujung 5’nya.
Gambar 32. Hasil PCR dengan primer spesifik SOD3UTR-F dan SOD3UTR-R terhadap lima koloni bakteri E.coli hasil transformasi membawa plasmid pENTR-D/TOPO rekombinan (pENTR-3UTRMmCuZn-SOD). (M=Marka, 1-4 = koloni E.coli transforman: 5 = pGEM-MmCuZn-SOD sebagai kontrol positif).
Fragmen 3’UTR MmCuZn-SOD yang dibawa vektor pENTRTM
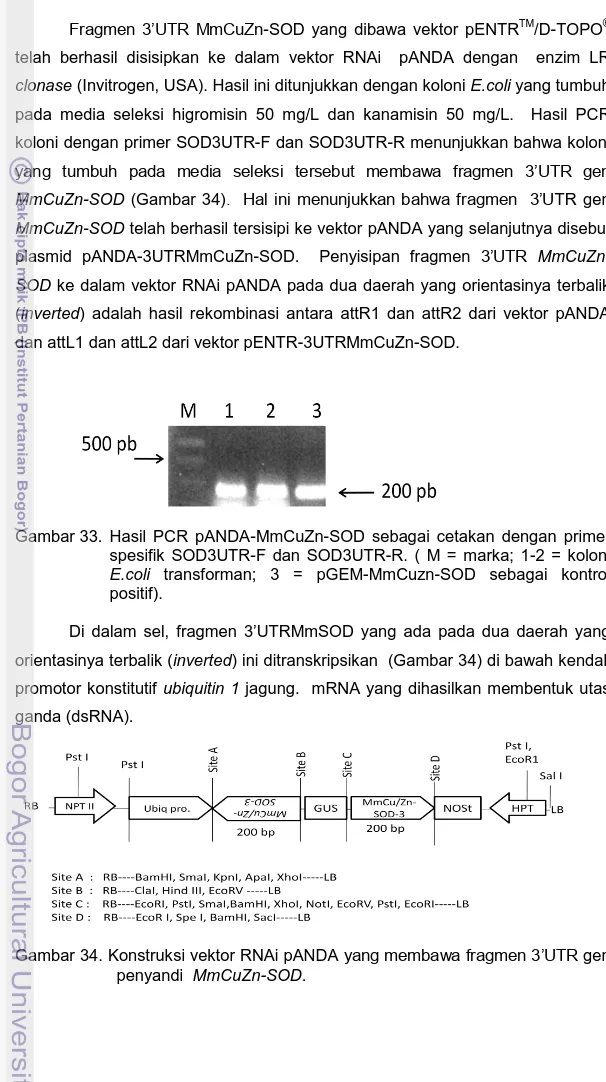
/D-TOPO® telah berhasil disisipkan ke dalam vektor RNAi pANDA dengan enzim LR clonase (Invitrogen, USA). Hasil ini ditunjukkan dengan koloni E.coli yang tumbuh pada media seleksi higromisin 50 mg/L dan kanamisin 50 mg/L. Hasil PCR koloni dengan primer SOD3UTR-F dan SOD3UTR-R menunjukkan bahwa koloni yang tumbuh pada media seleksi tersebut membawa fragmen 3’UTR gen MmCuZn-SOD (Gambar 34). Hal ini menunjukkan bahwa fragmen 3’UTR gen MmCuZn-SOD telah berhasil tersisipi ke vektor pANDA yang selanjutnya disebut plasmid pANDA-3UTRMmCuZn-SOD. Penyisipan fragmen 3’UTR MmCuZn-SOD ke dalam vektor RNAi pANDA pada dua daerah yang orientasinya terbalik (inverted) adalah hasil rekombinasi antara attR1 dan attR2 dari vektor pANDA dan attL1 dan attL2 dari vektor pENTR-3UTRMmCuZn-SOD.
Gambar 33. Hasil PCR pANDA-MmCuZn-SOD sebagai cetakan dengan primer spesifik SOD3UTR-F dan SOD3UTR-R. ( M = marka; 1-2 = koloni E.coli transforman; 3 = pGEM-MmCuzn-SOD sebagai kontrol positif).
Di dalam sel, fragmen 3’UTRMmSOD yang ada pada dua daerah yang orientasinya terbalik (inverted) ini ditranskripsikan (Gambar 34) di bawah kendali promotor konstitutif ubiquitin 1 jagung. mRNA yang dihasilkan membentuk utas ganda (dsRNA).
Gambar 34. Konstruksi vektor RNAi pANDA yang membawa fragmen 3’UTR gen penyandi MmCuZn-SOD.
Double strand RNA yang dihasilkan oleh suatu organisme akan dikenali oleh enzim dicer dan dipotong menjadi dsRNA yang berukuran kecil dan selanjutnya salah satu utasnya (antisense) berasosiasi dengan RISC (RNA-induced silencing complex) untuk mendegradasi mRNA target sehingga ekspresi gen dapat ditekan dalam berbagai level (Hannon 2002; Meister & Tuschl 2004).
Transformasi A. tumefaciens dengan Plasmid pAND-3’UTRMmCuZn-SOD Plasmid pAND-3’UTRMmCuZn-SOD telah berhasil diintroduksikan ke dalam A.tumefaciens LBA 4044 dengan menggunakan metode triparental mating (TPM). Plasmid pAND-3’UTRMmCuZn-SOD yang terdapat pada E.coli DH5α hasil transformasi (sebagai donor) dipindahkan ke dalam A.tumefaciens LBA 4044 (sebagai resipien) melalui proses konyugasi dengan bantuan E.coli HB 101 (pRK 2013) yang resisten terhadap antibiotik kanamisin. Bakteri yang mampu hidup pada media seleksi yang mengandung 50 mg/L kanamisin +50 mg/L higromisin + 50 mg/L streptomisin hanya A. tumefaciens LBA 4404 yang mengandung plasmid pAND-3’UTRMmCuZn-SOD.
Koloni hasil TPM yang tumbuh pada media seleksi selanjutnya telah dianalisis dengan PCR untuk membuktikan adanya plasmid pAND-3’UTRMmCuZn-SOD di dalam A.tumefaciens. PCR dengan primer spesifik SOD3UTR-F dan SOD3UTR-R terhadap koloni A. tumefaciens menghasilkan produk PCR berukuran 200 pb (Gambar 35). Hasil ini menunjukkan bahwa proses TPM telah berhasil dan menghasilkan A.tumefaciens yang mengandung plasmid RNAi pAND-3’UTRMmCuZn-SOD. A.tumefaciens tersebut siap diintroduksikan ke dalam tanaman target M. malabathricum.
Gambar 35. Hasil PCR dengan primer spesifik SOD3UTR-F dan SOD3UTR-R terhadap 2 koloni bakteri A. tumefaciens LBA 4404 hasil triparental mating. (M=Marka, 1-2 = koloni A. tumefaciens transforman 3UTRMmCuZn-SOD; 3 = plasmid pAND-3UTRMmCuZn-SOD).
Transformasi Tanaman M. malabathricum dengan A. tumefaciens
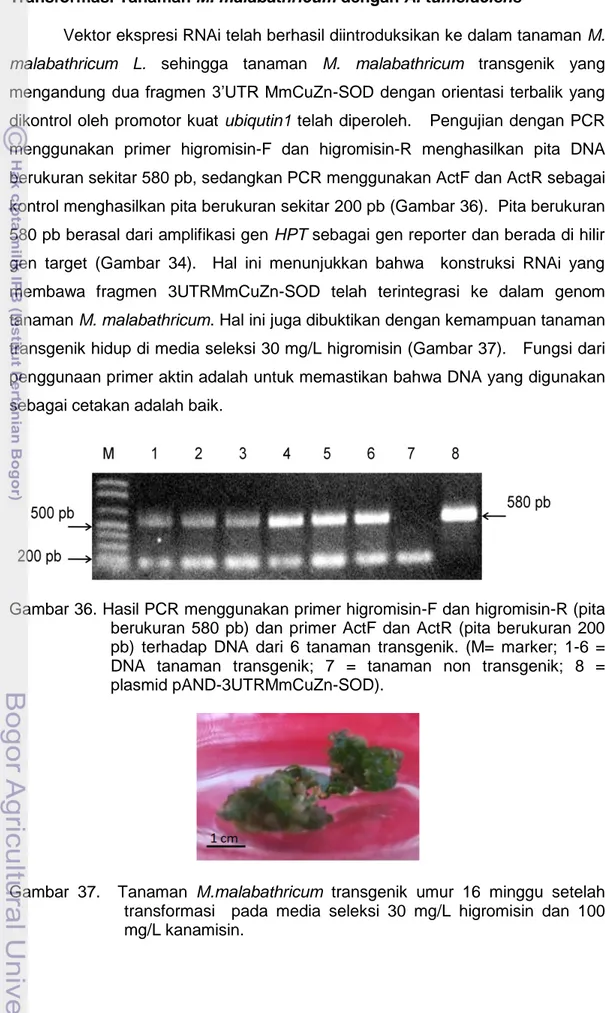
Vektor ekspresi RNAi telah berhasil diintroduksikan ke dalam tanaman M. malabathricum L. sehingga tanaman M. malabathricum transgenik yang mengandung dua fragmen 3’UTR MmCuZn-SOD dengan orientasi terbalik yang dikontrol oleh promotor kuat ubiqutin1 telah diperoleh. Pengujian dengan PCR menggunakan primer higromisin-F dan higromisin-R menghasilkan pita DNA berukuran sekitar 580 pb, sedangkan PCR menggunakan ActF dan ActR sebagai kontrol menghasilkan pita berukuran sekitar 200 pb (Gambar 36). Pita berukuran 580 pb berasal dari amplifikasi gen HPT sebagai gen reporter dan berada di hilir gen target (Gambar 34). Hal ini menunjukkan bahwa konstruksi RNAi yang membawa fragmen 3UTRMmCuZn-SOD telah terintegrasi ke dalam genom tanaman M. malabathricum. Hal ini juga dibuktikan dengan kemampuan tanaman transgenik hidup di media seleksi 30 mg/L higromisin (Gambar 37). Fungsi dari penggunaan primer aktin adalah untuk memastikan bahwa DNA yang digunakan sebagai cetakan adalah baik.
Gambar 36. Hasil PCR menggunakan primer higromisin-F dan higromisin-R (pita berukuran 580 pb) dan primer ActF dan ActR (pita berukuran 200 pb) terhadap DNA dari 6 tanaman transgenik. (M= marker; 1-6 = DNA tanaman transgenik; 7 = tanaman non transgenik; 8 = plasmid pAND-3UTRMmCuZn-SOD).
Gambar 37. Tanaman M.malabathricum transgenik umur 16 minggu setelah transformasi pada media seleksi 30 mg/L higromisin dan 100 mg/L kanamisin.
Uji Toleransi Tanaman Transgenik Terhadap Cekaman Al
Tanaman M.malabathricum L. transgenik yang telah ditumbuhkan pada MS yang mengandung 1 mM AlCl3.6H2O mulai minggu ke 3 perlakuan Al menampakkan gejala daun menguning dan terus mengalami perubahan ke coklat dan mengalami kematian 7 minggu kemudian. Sementara kontrol tetap menunjukkan pertumbuhan yang baik yang diindikasikan dengan warna daun yang tetap hijau. Pada penelitian ini pertumbuhan tanaman transgenik terhambat oleh perlakuan 1 mM Al (Gambar 38). Hal tersebut menunjukkan bahwa konstruksi RNAi dari 3’UTRMmCuZn-SOD diduga dapat menghentikan dan/atau menurunkan ekspresi gen penyandi MmCuZn-SOD pada M. malabathricum L. Kondisi ini juga dapat memberikan dugaan bahwa gen penyandi MmCuZn-SOD pada M. malabathricum merupakan salah satu gen yang terlibat dalam toleransi tanaman dari cekaman Al. Hal ini sejalan dengan pernyataan bahwa konstruksi RNAi dapat digunakan untuk mempelajari peranan suatu gen pada tanaman (Miki et al. 2005; Moritoh et al. 2005; DeBoer et al. 2010; Muzuni 2011).
Gambar 38. Tanaman M.malabathricum transgenik dan non transgenik yang diperlakukan dengan 1mM AlCl3.6H2O. dan pH 4.5 pada media MS selama 7 minggu. A. Tanaman non trangenik, B. Tanaman transgenik, C. Tanaman transgenik pada media MS tanpa cekaman pH dan Al.
Simpulan
Vektor RNAi yang membawa fragmen 3’UTRMmCuZn-SOD berhasil dikonstruksi dan disisipkan ke genom tanaman Melastoma malabathricum L. melalui Agrobacterium tumefaciens LBA 4404. Pada media MS yang mengandung cekaman Al (1 mM AlCl3.6H2O), tanaman transgenik mengalami hambatan pertumbuhan, sedangkan non trasngenik tidak mengalami hambatan. Hal ini menunjukkan bahwa CuZn-SOD berperan dalam toleransi Al pada tanaman M. malabathricum.