1
ANALISIS Escherichia coli O157:H7
PADA SUSU DAN SALAD MENGGUNAKAN REAL TIME PCR
(ANALYSIS ofEscherichia coli O157:H7
IN MILK AND SALAD USING REAL TIME PCR)
Eva Nikastri, Suci Yuliangsih, Tanti Lanovia dan Tepy Usia Pusat Riset Obat dan Makanan, Badan POM
Jl. Percetakan Negara No. 23, Jakarta, Indonesia [email protected]
ABSTRACT
Escherichia coli O157: H7 is a foodborne pathogen, when entry in the human digestive tract can cause illness and even death. Some cases have occurred are contamination of this pathogen to frozen meat products, milk, juice and salad. This study focuses on the isolation and identification of E. coli O157: H7 on milk and salad. Contamination of E. coli O157: H7 on salad can be sourced from fertilizers derived from animal waste.
Identification and quantification was using a Real Time PCR. Isolates DNA was amplified using reagents KAPA Fast Probe Master Mix, primers and specific probes labeled with FAM and BHQ. Development of the analytical method for E. coli O157: H7 based on artificial contamination. It was using Qiagen diagnostic kit which has been modified, with the forward primer Stx1-418-F (SynthID 1,222,546), reverse primer Stx1-617-R (SynthID 1,222,547) and probe STX1- P.
Validation parameters obtained for accuracy in the form of recovery from 92.07 to 119.70% (R2 = 0.9671), and a precision
of 7.31% RSD. Limit of Detection (LOD) obtained was 7,0 CFU/mL with R2 = 0.9771. Based on the specificity of primer,
showed that this method can detect other than E. coli O157: H7, such as L. monocytogenes, S. Typhimurium, B. subtilis, C. sakazakii and S. sonnei but with a different Ct values obtained for E. coli O157: H7 at 16.94 while for other bacterial Ct above 34.0.
Keywords : E. coli O157:H7, Real Time PCR, validation
ABSTRAK
Bakteri Escherichia coli O157:H7 merupakan foodborne pathogen, bila masuk dalam saluran pencernaan manusia dapat menyebabkan sakit bahkan kematian. Beberapa kasus yang terjadi adalah tercemarnya produk daging beku, susu, jus dan salad sayur. Penelitian ini menitik beratkan pada isolasi dan identifikasi E. coli O157:H7 pada produk susu dan salad sayur. Kontaminasi E. coli O157:H7 pada salad dapat berasal dari sayur yang tercemar pupuk yang berasal dari kotoran hewan.
Identifikasi dan kuantifikasi menggunakan alat Real Time PCR. Kontrol negatif untuk uji spesifisitas adalah Escherichia coli ATCC 25922, S. Typhimurium (ATCC 14028), E. aerogenes (ATCC 13048), B. subtilis, Listeria monocytogenes, S. sonnei, dan C. muytjensii. Isolat DNA diamplifikasi menggunakan pereaksi KAPA Probe Fast Master Mix, primer dan probe spesifik yang dilabel dengan FAM dan BHQ. Pengembangan metode analisis E.coli O157:H7 pada produk susu dan salad sayur menggunakan metode kit diagnostik Qiagen yang telah dimodifikasi, dengan primer forward Stx1-418-F (SynthID 1222546) dan primer reverse Stx1-617-R (SynthID 1222547) dan probe STX1-P, berdasarkan kontaminasi buatan (spiked sample).
Parameter validasi yang diperoleh untuk akurasi berupa recovery 92,07-119,70% (R2 = 0,9671), dan RSD presisi sebesar
7,31%. Limit of Detection (LOD) yang diperoleh adalah 10 CFU/mL dengan R2 = 0,9771. Parameter validasi yang
2
lainnya seperti L. monocytogenes, S. Typhimurium, B. subtilis, C. sakazakii dan Shigella sonnei karena nilai Ct yang diperoleh untuk E. coli O157:H7 pada 16,94 sedangkan Ct untuk bakteri lainnya di atas 34,0.
Keywords : E. coli O157:H7, Real Time PCR, validation
PENDAHULUAN
Keracunan pangan adalah penyakit yang disebabkan infeksi atau intoksikasi akibat mengkonsumsi makanan, minuman atau air yang telah terkontaminasi (Sharp dan Reilly, 2000). Kejadian Luar Biasa (KLB) keracunan pangan di Indonesia dapat terjadi akibat kontaminasi mikroba patogen atau bahan kimia berbahaya seperti toksin alami, pestisida, logam berat dan lain-lain. Salah satu permasalahan KLB keracunan pangan adalah tidak diketahuinya penyebabnya karena data epidemiologi di lapangan tidak lengkap, sampel tidak representatif, hasil pengujian sampel negatif atau salah menetapkan hipotesa. Berdasarkan Laporan Tahunan Badan POM (tahun 2010-2013), KLB keracunan pangan akibat cemaran mikroba lebih banyak dibanding kimia. Dari Laporan tersebut juga dapat dinyatakan bahwa sumber terbesar pangan penyebab KLB berasal dari masakan rumah tangga, jasa boga dan pangan jajanan. Salah satu produk pangan hasil masakan rumah tangga, jasa boga dan pangan jajanan yang banyak dikonsumsi masyarakat adalah produk susu dan salad sayur.
Bakteri patogen yang sering mengkontaminasi produk susu dan salad sayur antara lain E. coli, S. aureus dan Salmonella. Dalam kurun tahun 1982-2007, E. coli O157:H7 merupakan bakteri yang selalu menyebabkan penyakit melalui STEC (shigalike toxin E. coli). E. coli O157:H7 bersifat tidak tahan panas tetapi tahan pembekuan dan pH rendah. Dalam dua tahun terakhir, STEC juga telah mengakibatkan penyakit di berbagai negara bagian di Amerika Serikat karena konsumsi bayam siap santap. E. coli O157:H7 mengakibatkan diare berdarah (hemorrhagic colitis) dan gejala gagal ginjal (hemolytic uremic syndrome) terutama pada anak-anak dan lansia (Hariyadi, 2004). Pada tahun 2011, telah terjadi KLB di Jerman yang berasal dari kecambah. Korban mengalami diare berdarah dan hemolytic uremic syndrome (HUS). Bahan pangan yang sering terkontaminasi E. coli antara lain salami ayam, salami sapi, salami babi selama penyembelihan, ikan dan pangan hasil laut lainnya, telur dan produk olahannya, sayuran, buah-buahan, sari buah, serta bahan minuman seperti susu dan lainnya.
Untuk mengetahui penyebab KLB keracunan pangan, pangan yang diduga sebagai penyebabnya perlu diuji di laboratorium dan selanjutnya dapat dilakukan langkah-langkah pencegahan dan penanggulangan. Salah satu metode yang akurat dilakukan saat ini adalah identifikasi secara molekular dengan cara isolasi dan amplifikasi DNA bakteri patogen menggunakan PCR. PCR adalah salah satu metode in vitro untuk memperbanyak sekuens DNA tertentu. Metode ini jauh lebih cepat dan spesifik dibanding metode konvensional.
3 Perkembangan PCR terbaru adalah Real-time PCR, yaitu amplifikasi PCR dan verifikasi dilakukan sekali jalan dengan probe khusus (oligonukleotida berlabel fluoresen yang spesiifik terhadap gen target). Real-time PCR melakukan amplifikasi dan deteksi dalam satu tahapan sebab akumulasi produk spesifik dicatat secara kontinyu selama siklus. Kuantitas produk Real-time PCR dihitung berdasarkan threshold cycle (Ct) yaitu waktu dimana intensitas fluoresen lebih besar daripada fluoresen yang ditimbulkan oleh noise (background fluorescence).
Badan POM sebagai institusi pemerintah yang salah satu wewenangnya adalah mengawasi keamanan produk pangan yang beredar di masyarakat, melalui unit Pusat Riset Obat dan Makanan telah melakukan riset pengembangan metode analisis untuk mendeteksi beberapa bakteri patogen pada sampel pangan sejak tahun 2008. Penggunaan sampel pangan ini adalah sebagai tahap lanjutan dari penggunaan model pangan pada tahun 2007. Hasil penelitian sebelumnya menunjukkan bahwa pita DNA dari beberapa bakteri patogen yang diperoleh masih tipis, karena DNA yang diperoleh sedikit, pengaruh pengotor yang berasal dari matriks pangan dan primer yang digunakan belum spesifik.
Metode analisis yang dikembangkan divalidasi dengan melakukan amplifikasi menggunakan kontrol positif (standar), kontrol negatif, dan sampel. Kontrol negatif adalah bakteri yang sudah diketahui secara pasti tidak mengandung amplificate target yang dimaksud, sedangkan kontrol positif sebaliknya. Validasi metode kuantitatif menurut Trullols et al. (2004) mempunyai 7 (tujuh) parameter yang harus dipenuhi, yaitu (1) akurasi (ketepatan dan presisi, accuracy), (2) ketidakpastian (uncertainty), (3) sensitivitas dan spesifisitas, (4) selektifitas : interferensi, (5) daerah linier pengukuran dan daerah kerja (linearity and range), (6) limit deteksi dan (7) ketangguhan dan kekuatan (ruggedness and robustness). Parameter yang dipilih berdasarkan metode analisis yang akan divalidasi. Tahapan preparasi sampel untuk mengisolasi DNA yang berkualitas tinggi dari bakteri dalam susu, juga merupakan hal yang sering menjadi masalah dan membutuhkan waktu semalaman untuk prosedur pengkayaan yang selektif (Phuektes et al., 2001; Meiri-Bendek et al, 2002; Ramesh et al, 2002). Kesulitan ini diakibatkan oleh sedikitnya konsentrasi DNA patogen yang ada dalam sampel, termasuk berbagai faktor yang mempengaruhi DNA recovery, tingkat lisis sel, ikatan DNA dengan material, dan pemutusan atau degradasi DNA.
Tujuan penelitian ini adalah melakukan pengembangan metoda analisis untuk isolasi Deoxyribonucleic Acid (DNA) bakteri patogen Escherichia coli O157:H7 dalam produk susu dan salad sayur yang disimpan dingin. Isolasi DNA dari matriks pangan menggunakan metode kit komersial Qiagen kemudian diidentifikasi dan dikuantifikasi menggunakan alat Real Time Polymerase Chain Reaction. Prinsipnya dilakukan kontaminasi buatan (artificial contamination) Escherichia coli O157:H7 ke dalam sampel susu dan salad sayur dingin dan kemudian DNA target diisolasi. Isolat DNA diamplifikasi dengan menggunakan
4 pereaksi KAPA Probe Fast Master Mix, primer dan probe spesifik. Probe dilabel dengan FAM dan BHQ dan dideteksi menggunakan Real Time PCR. Pembacaan produk hasil Real-Time PCR ditampilkan dalam bentuk grafik amplifikasi dan kurva standar. Tahapan validasi metoda analisis meliputi penentuan spesifisitas, limit deteksi, keseksamaan atau presisi, dan kecermatan atau akurasi.
BAHAN DAN METODE
Bahan
Sampel yang digunakan adalah susu dan salad sayur yang dijual di supermarket dan disimpan dingin. Sebagai baku pembanding (kontrol positif) digunakan E. coli 0157:H7 (2126, Kyoto). Untuk kontrol negatif digunakan Escherichia coli ATCC 25922, S. Typhimurium (ATCC 14028), E. aerogenes (ATCC 13048), B. subtilis, Listeria monocytogenes, S. sonnei, dan C. muytjensii. Media untuk pertumbuhan dan isolasi bakteri adalah Sorbitol MacKonkey Agar (media selektif E.coli O157:H7), cemitex tellurite, Eosin Methylene Blue Agar (EMBA, Oxoid), Nutrient Agar, Nutrient Broth dan Trypticase Soy Broth (TSB, Oxoid) dan XLDA.
Bahan isolasi DNA meliputi kit diagnostik (QIAamp DNA Mini and Blood Mini 250 rx) terdiri dari bufer AL, bufer AW 1, bufer AW 2, dan bufer AE. Proteinase K (Nacalai Tesque), CTAB (Merck), PBS (Merck), EDTA Solution (Merck), Tris HCl solution (USB), Etanol absolut (Merck), RNase A (USB), dan RNase free water (USB). Bahan Real Time PCR antara lain KAPA Probe Fast Mastermix (2x) Bio Rad iCycler (KAPA), primer forward Stx1-418-F (SynthID 1222546) dan primer reverse Stx1-617-R (SynthID 1222547), dan probe Stx1-P. Instrumen utama yang digunakan adalah Real Time PCR-IQ5 (BioRAD), laminar air flow, spektrofotometer UV-1800 (Shimadzu), refrigerator microsentrifuge (Hettich) dan peralatan gelas lainnya.
Metode
1. Persiapan Kultur Bakteri dan Sampel
Bakteri uji ditumbuhkan di media spesifiknya masing-masing. Untuk pengembangan metode analisis disiapkan sampel negatif yaitu susu dan salad sayur yang telah disterilkan. Sampel positif yaitu sampel yang dispike dengan E.coli O157:H7 pada konsentrasi 10o, 101, 102, 103, 104 dan 105 CFU/mL. Sampel yang telah dispike, diaduk/dikocok agar homogen dan diinkubasi pada 5-10 ºC selama 1 jam. Selanjutnya untuk sampel salad sayur dihancurkan menggunakan stomaker selama 15 detik untuk persiapan tahapan selanjutnya yaitu isolasi DNA
5 2. Ekstraksi DNA
Isolasi DNA dilakukan menggunakan metode ekstraksi kit untuk bakteri Gram Negatif (Metode QIAamp® DNA Blood Mini Kit Handbook, 2007, dengan modifikasi Bioteknologi PROM, 2012). Kultur murni (kontrol positif dan kontrol negatif), sampel negatif dan sampel yang telah dispike (sampel positif) dihomogenisasi dan diambil masing-masing 1,5 mL ke dalam tabung eppendorf, lalu disentrifus pada kecepatan 10.000 rpm selama 5 menit pada suhu 4 oC. Supernatan dibuang dengan menyisakan ± 200 µL larutan dalam tabung, ditambahkan 180 µL bufer CTAB, dan divortex selama 10 detik. Ditambahkan 20 µL proteinase K untuk kultur murni atau 100 µL proteinase K untuk sampel pangan. Diinkubasi pada suhu 65°C selama 1 jam dalam penangas air dan selama inkubasi sampel divortex sebanyak 2-3 kali. Disentrifus 6000 rpm, 20 detik, suhu 4 oC. Dilakukan penambahan RNase A sebanyak 4 µL, divortex selama 15 detik, diinkubasi pada suhu ruang (30 0C) selama 2 menit. Disentrifus 6000 rpm, 20 detik pada suhu 4 oC. Ditambahkan bufer AL, divortex selama 15 detik, diinkubasi pada 70°C selama 10 menit dan disentrifus 5 detik. Ditambahkan 200 µL etanol (96-100%), divortex selama 15 detik, disentrifus 5 detik, kemudian pindahkan cairan ke dalam kolom mini yang berada di dalam collection tube.
Penutup kolom mini kemudian ditutup, disentrifus pada 8000 rpm selama 1 menit pada suhu 4 oC, lalu cairan dalam collection tube dibuang. Dibuka dengan hati-hati kolom mini tersebut dan ditambahkan 500 µL bufer AW1, tabung eppendorf ditutup dan disentrifus pada 8000 rpm selama 1 menit pada suhu 4 oC. Cairan dalam collection tube dibuang lalu tempatkan kembali kolom mini pada collection tube yang baru. Tutup eppendorf dibuka perlahan lalu ditambahkan 500 µL bufer AW2, kolom mini ditutup kembali, kemudian disentrifus dengan kecepatan 14000 rpm selama 3 menit pada suhu 4 oC lalu disentrifus kembali selama 1 menit pada suhu 4 oC. Collection tube dibuang, kolom mini diletakkan di dalam tabung eppendorf baru. Kolom mini dibuka perlahan dan ditambahkan 200 µL bufer AE, selanjutnya diinkubasi pada suhu ruang selama 1 menit dan disentrifus pada 10,000 rpm selama 1 menit pada suhu 4 oC. Kemudian tahap selanjutnya ditambahkan kembali 200 µL bufer AE, diulang disentrifus pada 10,000 rpm selama 1 menit pada suhu 4 oC. Kolom mini dibuang, dan kemudian isolat DNA yang diperoleh disimpan dalam freezer (-20°C) sampai akan digunakan.
Kualitas DNA hasil isolasi ditentukan dengan cara diukur absorbansinya menggunakan spektrofotometer UV pada panjang gelombang 260 nm dan 280 nm. Hasil pembacaan dari spektrofotometer meliputi absorbansi pada λ 260 nm, absorbansi λ 280 nm, perbandingan antara absorbansi λ 260 nm dan absorbansi λ 280 nm serta konsentrasi DNA (ng/µL). Konsentrasi DNA hasil isolasi ditentukan dengan mengukur serapannya pada panjang gelombang λ 260 nm (1 unit absorban sebanding dengan 50 µg/mL dari DNA untai ganda). Kriteria keberterimaan untuk kemurnian DNA hasil isolasi adalah 1,8 – 2,0.
6 3. Optimasi Suhu Penempelan Primer/Probe (Annealing)
Suhu annealing dioptimasi dengan mengaktifkan program gradient pada instrumen real time PCR, kemudian di tentukan rentang suhu yang akan diuji. Protokol Real Time PCR E.coli O157:H7 (Bellin, et al., 2001) meliputi pra-denaturasi 94°C, 120 detik, untuk 40 siklus denaturasi 94°C, 20 detik, dan annealing 60°C, 30 detik.
4. Pembuatan Baku Kerja dan Optimasi Konsentrasi Primer
Pembuatan baku kerja primer dilakukan berdasarkan protokol pengenceran primer dari 1st base (Anonim, 2012). Baku kerja primer dibuat dengan konsentrasi 20 dan 10 µM, selanjutnya dilakukan pengenceran beberapa konsentrasi primer yaitu 4, 8, 12, 16 dan 20 µM yang akan digunakan untuk optimasi konsentrasi primer dengan range konsentrasi akhir 0,1 – 0,5 µM. Optimasi dilakukan dengan menggunakan kultur murni E.coli O157:H7. Kemudian dipilih konsentrasi primer yang sesuai/tepat. Konsentrasi primer yang sesuai/tepat adalah konsentrasi primer yang menghasilkan nilai Ct yang paling rendah dan tanpa menghasilkan atau seminimal mungkin menghasilkan primer-dimer pada kurva puncak pelelehan (Pestana et al. 2010).
5. Pembuatan Baku Kerja dan Optimasi Konsentrasi Pelacak (Probe)
Penentuan konsentrasi probe dilakukan setelah dilakukan penentuan konsentrasi primer. Pelacak (probe) dibuat dengan konsentrasi 20 dan 10 µM, selanjutnya dilakukan pengenceran beberapa konsentrasi primer yaitu 4, 8, 12, 16 dan 20 µM yang akan digunakan untuk optimasi konsentrasi probe dengan range konsentrasi akhir 0,1 – 0,5 µM. Optimasi dilakukan dengan menggunakan kultur murni E.coli O157:H7. Kemudian dipilih konsentrasi probe yang sesuai/tepat. Konsentrasi probe yang sesuai/tepat adalah konsentrasi primer yang menghasilkan nilai Ct yang paling rendah dan tanpa menghasilkan atau seminimal mungkin menghasilkan primer-dimer pada kurva puncak pelelehan (Pestana et al. 2010).
6. Pembuatan Master Mix
Master mix PCR (probe) pada penelitian ini terdiri dari 5,5 µL buffer TE, 10 µL KAPA Probe Fast qPCR. Ditambahkan masing-masing 1 µL primer reverse dan forward pada konsentrasi 6 µM. Ditambahkan 0,5 µL probe konsentrasi 4 µM dan dihomogenisasi. Volume master mix keseluruhan untuk pengujian satu jenis template DNA adalah 18 µL. Bahan-bahan tersebut dicampur dalam satu tabung eppendorf 2 ml berwarna gelap dan divortex. Untuk melakukan pengujian beberapa jenis template DNA, maka volume masing-masing reagensia dikali dengan banyaknya jenis template DNA yang akan diuji pada Real-Time PCR dan dicampur di dalam tabung eppendorf. Sejumlah 18 µL campuran master mix dipipet ke dalam setiap well yang kemudian ditambahkan dengan 2 µL template DNA, ditutup dengan penutup well, selanjutnya divortex pada MixMate PCR 96 dan dimasukkan pada alat Real-Time PCR. Semua pembuatan master mix ini
7 sebaiknya dalam kondisi gelap. Amplifikasi DNA E. coli 0157:H7 menggunakan Real Time PCR (PROM, 2012) pada kondisi protokol Real Time PCR pra-denaturasi (94°C, 2 menit), untuk 40 siklus yaitu denaturasi (94°C, 20 detik) dan annealing (60°C, 30 detik). 7. Validasi Metode Analisis
Untuk validasi metode analisis, baku kerja primer dibuat dengan konsentrasi 6 µM dan pelacak (probe) dengan konsentrasi 4 µM (hasil optimasi metode). Parameter yang diukur adalah akurasi, presisi, spesifisitas dan LOD (limit of detection). Akurasi dinyatakan sebagai persen perolehan kembali (recovery) analit yang ditambahkan. Akurasi ditentukan dengan cara penambahan kultur murni yang telah diketahui jumlahnya (5 tingkat konsentrasi) dan dilakukan sebanyak 3 (tiga) kali ulangan. Persen perolehan kembali (recovery) dinyatakan sebagai rasio antara hasil yang diperoleh dengan hasil yang sebenarnya dan syarat keberterimaannya adalah ≥ 70% (USP, 2008).
Presisi dilakukan dengan cara mengisolasi dan identifikasi kultur murni E. coli O157:H7 pada satu konsentrasi dengan menggunakan metode analisis yang telah dikembangkan. Dilakukan sebanyak 10 replikasi (USP, 2008). Kriteria keberterimaannya adalah RSD dibawah 15% (USP, 2008).
Uji spesifisitas dilakukan dengan menguji spesifisitas primer bukan hanya terhadap bakteri uji (kontrol positif) saja, namun terhadap bakteri kontrol negatif. Spesifisitas suatu metode adalah kemampuan untuk hanya mengukur zat tertentu secara spesifik, mampu membedakan dengan adanya bakteri atau komponen lain yang mungkin ada dalam matriks sampel.
Konsentrasi yang diuji untuk menentukan LOD adalah konsentrasi di atas dan di bawah dari kemungkinan nilai LOD (Burd, 2010). Kurva standar dapat digunakan untuk menghitung nilai LOD yaitu dengan memasukkan nilai Ct dari konsentrasi terendah yang masih dapat terdeteksi ke dalam persamaan kurva standar. Semakin rendah konsentrasi analit atau mikroorganisme yang dapat dideteksi, maka semakin tinggi sensitivitas pengujiannya (Burd, 2010).
HASIL DAN PEMBAHASAN
Optimasi Primer dan Probe
Pada penelitian ini dilakukan optimasi konsentrasi primer akhir yaitu dengan kisaran konsentrasi optimum 100-500 nM. Hasil optimasi konsentrasi primer menunjukkan bahwa primer stx, memiliki nilai Ct terkecil pada konsentrasi berturut-turut adalah 300 nM dan 500 nM. Konsentrasi primer tersebut menghasilkan nilai Ct yang tidak jauh berbeda, sehingga untuk efisiensi penggunaan primer, untuk uji selanjutnya digunakan konsentrasi primer terendah yaitu 300 nM.
8 Tabel 1. Hasil optimasi primer stx pada kultur murni E.coli O157:H7
Baku kerja primer (probe [400 nM])
Konsentrasi
primer akhir Nilai Ct
Nilai Ct Rata-rata 2 µM 100 nM 15.29 15.51 15.02 14.76 4 µM 200 nM 16.90 14.50 13.92 15.70 6 µM 300 nM 15.03 14.31 14.28 13.63 8 µM 400 nM 14.21 14.23 13.86 14.62 10 µM 500 nM 14.32 14.10 13.87
Penentuan konsentrasi primer merupakan tahap kritis dalam amplifikasi dengan real time PCR serta menjadi hal yang pertama yang perlu dilakukan dalam pengujian real time PCR, yaitu dengan mengevaluasi dan mengkaji penggunaan konsentrasi primer yang tepat (Pestana et al., 2010). Hal ini menjadi penting karena konsentrasi primer akan mempengaruhi reaksi dan hasil dari proses amplifikasi real time PCR. Konsentrasi primer yang tidak tepat yaitu terlalu rendah, akan menyebabkan primer tidak menempel pada target DNA (DNA template), sedangkan konsentrasi primer yang terlalu tinggi akan menyebabkan terjadinya misprimming, yaitu primer akan menempel pada non target DNA. Hasil optimasi primer ini juga berada pada rentang konsentrasi yang direkomendasi dari protokol Kapa Probe Fast qPCR Kit master mix, yaitu untuk memberikan hasil amplifikasi yang optimal, maka konsentrasi primer akhir yang digunakan adalah antara 100 nM – 400 nM.
Hasil optimasi konsentrasi probe akhir dengan kisaran konsentrasi 100-500 nM, dipilih konsentrasi 200 nM karena nilai Ct yang diperoleh kecil dengan konsentrasi probe yang tidak terlalu banyak. Hasil optimasi konsentrasi probe menunjukkan bahwa probe stx memiliki nilai CT terkecil pada konsentrasi 500 nM. Nilai CT hasil optimasi dapat dilihat pada Tabel 2.
9 Tabel 2. Hasil optimasi probe stx pada kultur murni E.coli O157:H7
Baku kerja probe (primer [250 nM])
Konsentrasi
probe akhir Nilai Ct
Nilai Ct Rata-rata 2 µM 100 nM 15.07 14.55 14.30 14.28 4 µM 200 nM 13.87 14.14 13.88 14.67 6 µM 300 nM 14.55 15.17 15.40 15.55 8 µM 400 nM 14.81 14.88 14.94 10 µM 500 nM 13.77 14.00 13.53 14.69
Seperti halnya primer, konsentrasi probe yang semakin tinggi atau semakin rendah, tidak menunjukkan semakin optimumnya kerja probe, karena dalam penentuan hasil optimasi konsentrasi probe, konsentrasi probe yang optimum ditunjukkan dengan nilai CT terkecil (Edwards, 2004). Dimana pada konsentrasi optimum tersebut, probe dapat melacak DNA target dengan optimum pula.
Akurasi
Berdasarkan Gambar 1 dan Tabel 3, nilai akurasi yang ditunjukkan oleh kurva standar adalah recovery sebesar 83,54-108,61% dengan nilai r2 0,9671. Nilai recovery ini memenuhi kriteria keberterimaan dari USP, yaitu ≥ 70%. Nilai Ct yang diperoleh cukup tinggi, hal ini disebabkan konsentrasi DNA E. coli O157:H7 yang diperoleh rendah. Selain itu semakin rendah konsentrasi E. coli 0157:H7 yang dispike dalam sampel maka akan semakin tinggi nilai Ct nya.
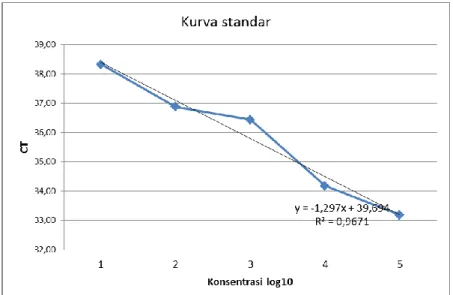
10 Gambar 1. Kurva standar sampel yang dispike dengan E.coli O157:H7
Tabel 3. Hasil pengukuran Ct isolasi DNA E.coli O157:H7 yang dispike ke dalam salad sayur dengan konsentrasi 1-5 log10 CFU/ml E. coli 015:H7 yang ditambahkan (log10) Ulangan CT Rata-rata CT Konsentrasi Log10 % Recovery 1,00 1 38,58 38,33 1,05 105,17 2 37,88 3 38,52 2,00 1 36,97 36,88 2,17 108,61 2 36,97 3 36,69 3,00 1 36,27 36,44 2,51 83,54 2 36,60 3 36,46 4,00 1 33,64 34,18 4,25 106,28 2 33,80 3 35,10 5,00 1 32,79 33,19 5,01 100,29 2 32,89 3 33,89 Rata2 35,80 % Recovery 100,78 SD 1,98 RSD 5,52
11 Presisi
Pada penentuan presisi dengan kultur murni E. coli O157:H7 konsentrasi 104 CFU/mL diperoleh RSD metode analisis ini sebesar 7,31% sehingga masuk dalam kriteria keberterimaan yaitu dibawah 15% (USP, 2008).
Spesifikasi Primer
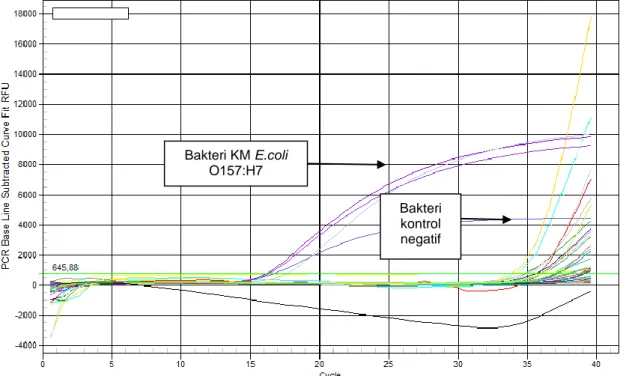
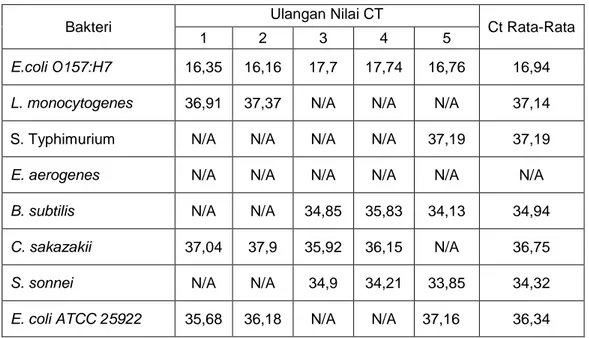
Uji spesifisitas primer dilakukan terhadap 7 bakteri kontrol negatif dan dilihat pada Gambar 2 dan Tabel 4. Hasil spesifisitas menunjukkan bahwa primer Stx spesifik untuk bakteri E.coli O157:H7 sampai pada nilai CT 16-17. Rata-rata nilai CT bakteri kontrol negatif yang diuji berkisar antara 34-39. Pada Gambar 2 terlihat hasil amplifikasi spesifisitas primer, bahwa bakteri kontrol negatif tidak mengganggu hasil Ct E.coli O157:H7, yaitu dapat dibedakan dengan bakteri kontrol negatif. Tetap terjadi peningkatan nilai Ct untuk kontrol negatif L. monocytogenes, S. Typhimurium, B. subtilis, C. sakazakii dan S. sonnei karena primer Stx yang digunakan sifatnya kurang spesifik, juga dapat mendeteksi bakteri yang mempunyai shigalike toksin.
Gambar 2. Kurva amplifikasi spesifisitas primer Stx
Bakteri KM E.coli O157:H7
Bakteri kontrol negatif
12 Tabel. 4. Nilai CT hasil amplifikasi uji spesifisitas primer stx
Limit of Detection (LOD)
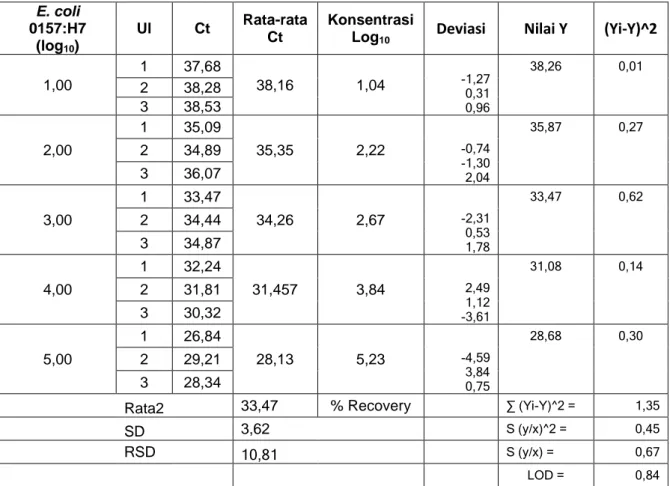
Pada Gambar 3, kurva standar untuk kultur murni E.coli O157:H7 yang diisolasi dengan metode kit Qiagen yang dimodifikasi terhubung secara linear yang ditunjukkan dengan nilai r2 = 0,9771. Linieritas sudah memenuhi syarat keberterimaan yaitu r2>0.95. Pada Tabel 5, dapat ditentukan bahwa LOD untuk E.coli O157:H7 sebesar 7 cfu/mL dengan nilai Ct rata-rata 38,16 dan recovery 89,04 – 110,81%
Gambar 3. Kurva standar kultur murni E.coli O157:H7
Bakteri Ulangan Nilai CT Ct Rata-Rata
1 2 3 4 5
E.coli O157:H7 16,35 16,16 17,7 17,74 16,76 16,94
L. monocytogenes 36,91 37,37 N/A N/A N/A 37,14
S. Typhimurium N/A N/A N/A N/A 37,19 37,19
E. aerogenes N/A N/A N/A N/A N/A N/A
B. subtilis N/A N/A 34,85 35,83 34,13 34,94
C. sakazakii 37,04 37,9 35,92 36,15 N/A 36,75
S. sonnei N/A N/A 34,9 34,21 33,85 34,32
13 Berdasarkan kurva standar dari kultur murni dan sampel yang dispike, dapat diperoleh persamaan regresi, nilai RSD dan LOD (Tabel 5). Nilai r2 untuk kultur murni lebih tinggi (0,9771) dibanding dari sampel (0,9671), namun tetap masuk dalam kriteria keberterimaan (>0,95). LOD untuk kultur murni 7 cfu/mL sedangkan untuk sampel sedikit lebih tinggi yaitu 10,23 cfu/mL. Menurut Chen et al., (1998), limit deteksi E. coli dengan PCR sekitar 1-5 cfu per campuran PCR jika kultur murni yang digunakan, namun jika digunakan sampel pangan limit deteksi 3 cfu/25 g pangan setelah dilakukan tahap preenrinchment semalam. Dengan kondisi optimal real-time multiplex PCR, Wang et al. (2007) menemukan limit deteksi kultur murni E. coli O157:H7 adalah 102 sampai 109 CFU/mL, sedangkan limit deteksi E. coli O157:H7 dengan sampel salami sapi yang dikontaminasi secara artifisial sekitar 105 CFU/g.
Tabel 5. Hasil pengukuran Ct isolasi DNA kultur murni E.coli O157:H7 dengan berbagai tingkat konsentrasi
E. coli 0157:H7 (log10) Ul Ct Rata-rata Ct Konsentrasi
Log10 Deviasi Nilai Y (Yi-Y)^2 1,00 1 37,68 38,16 1,04 -1,27 0,31 0,96 38,26 0,01 2 38,28 3 38,53 2,00 1 35,09 35,35 2,22 -0,74 -1,30 2,04 35,87 0,27 2 34,89 3 36,07 3,00 1 33,47 34,26 2,67 -2,31 0,53 1,78 33,47 0,62 2 34,44 3 34,87 4,00 1 32,24 31,457 3,84 2,49 1,12 -3,61 31,08 0,14 2 31,81 3 30,32 5,00 1 26,84 28,13 5,23 -4,59 3,84 0,75 28,68 0,30 2 29,21 3 28,34
Rata2 33,47 % Recovery ∑ (Yi-Y)^2 = 1,35
SD 3,62 S (y/x)^2 = 0,45
RSD 10,81 S (y/x) = 0,67
14
KESIMPULAN
Metode analisis ini dapat digunakan untuk identifikasi bakteri patogen E. coli 0157:H7 dari sampel susu dan salad sayuran. DNA target berhasil diekstraksi menggunakan metode kit komersial QIAamp® DNA Blood Mini Kit (Qiagen). Berdasarkan optimasi, diperoleh konsentrasi primer stx dan probe sebesar 500 nM untuk menghasilkan nilai Ct terendah. Hasil spesifisitas menunjukkan bahwa primer stx spesifik untuk E.coli O157:H7 dengan nilai Ct < 20 sedangkan bakteri kontrol negatif mempunyai nilai Ct > 35. Terhadap parameter validasi, diperoleh nilai akurasi dan presisi yang memenuhi kriteria keberterimaan yaitu recovery sebesar 83,54-108,61% dengan nilai r2 0,9671 dan RSD sebesar 7,31%.
LOD untuk E.coli O157:H7 sebesar 7,0 CFU/mL dengan nilai Ct rata-rata 38,16 dengan recovery 89,04 – 110,81%. Untuk r2 E.coli O157:H7 diperoleh 0,96 dan r2 untuk E.coli O157:H7 di dalam sampel salad sayuran diperoleh 0,992. Nilai ini telah memenuhi kriteria keberterimaan validasi.
TERIMA KASIH
Terimakasih disampaikan kepada Pusat Riset Obat dan Makanan sebagai penyandang dana yang bersumber dari DIPA Badan POM tahun 2012 dan 2013.
PUSTAKA
Anonim. 2007. QIAmp® DNA Mini and Blood Mini Handbook. Qiagen. Second Edition Badan POM RI. Laporan Tahunan 2010-2013. Badan POM RI. Jakarta
Bellin, T., M. Pulz, A. Matussek, H.G. Hempen, F. Gunzer. 2001. Rapid Detection of Enterohemorrhagic Escherichia coli by Real Time PCR with Fluoresencent Hybridization Probes. J. Clinical Microbiology. 370-374
Burd, E. M. 2010. Validation of Laboratory-Developed Molecular Assays for Infectious Diseases. J. Clinical Microbiology Reviews. 550-555
Cui, S., C. M. Schroeder, D. Y. Zhang, J. Meng. 2003. Rapid sample preparation method for PCR-based detection of Eschericia coli O157: H7 in ground beef. J. Applied Microbiology 95. 129-134
Edward, KJ. 2004. Performing Real Time PCR. Dalam Edwards, K., J. Logan, N. Saunders (eds.). Real Time PCR An Essential Guide. UK: Horizon Bioscience. Elizaquivel, P., J.A. Gabaldon, R. Aznar. 2011. Quantification of Salmonella spp., Listeria
monocytogenes and Escherichia coli O157:H7 in non-spiked food products and evaluation of real-time PCR as a diagnostic tool in routine food analysis. J. Food Control. 22 : 158-164. Elsevier
Fu, Z., S. Rogelj, T.L. Kieft. 2005. Rapid detection of Escherichia coli O157:H7 by immunomagnetic separation and real time PCR. Int. J. of Food Microbiology. 99 : 45-57. Elsevier
Hariyadi, R.D. 2004. Keracunan Pangan Tak Hanya Sebabkan Diare. Di dalam
15 Harmita, 2004. Petunjuk pelaksanaan validasi metode dan cara perhitungannya. Di dalam : Majalah Ilmu Kefarmasian. Desember, Vol. 1, No. 3, 117-135. Departemen Farmasi, FMIPA-UI.
Pestana, E.A., S. Belak, A. Diallo, JR. Crowther, G.J. Viljoen. 2010. Early, Rapid and Sensitive Veterinary Molecular Diagnostic Real Time PCR Application. Dordrecht : Springer.
Sharp, J. C. M, W. (Bill) J. Reilly. 2000. Surveillance of Foodborne Disease. Di dalam Lund, Barbara, M., T. C. Baird-Parker, G. W. Gould (eds.). 2000. The Microbiological Safety and Quality of Food Vol. II Aspen Publisher, Inc. Gaithersburg, Maryland.
USP NF. 2008. Volume 1. The United States Pharmacopeia Convention. Port City Press. Baltimore. The United States
Wang, R.F., W.W. Chao, C.E. Cemiglia. 1997. A Universal Protocol for PCR Detection of 13 Species of Foodborne Pathogens in Food. J. Applied Microbiology. 83 : 727-736 Yuliangsih, S., E. Nikastri, N. Pusparini, K. Khotimah. 2012. Validasi Metode Analisa L.
monocytogenes, S. aureus dan E. coli dalam Pangan Dengan Menggunakan Real Time PCR. Laporan Penelitian. Badan POM