TUGAS AKHIR – SS141501
OPTIMASI PARAMETER
SUPPORT VECTOR
MACHINE
MENGGUNAKAN
GENETIC ALGORITHM
UNTUK KLASIFIKASI
MICROARRAY DATA
AGENG PRAMESTHI KUSUMANINGRUM NRP 1313 100 022
Dosen Pembimbing
Santi Wulan Purnami, M.Si, Ph.D Irhamah, M.Si, Ph.D
PROGRAM STUDI SARJANA DEPARTEMEN STATISTIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT TEKNOLOGI SEPULUH NOPEMBER
TUGAS AKHIR – SS141501
OPTIMASI PARAMETER
SUPPORT VECTOR
MACHINE
MENGGUNAKAN
GENETIC ALGORITHM
UNTUK KLASIFIKASI
MICROARRAY DATA
AGENG PRAMESTHI KUSUMANINGRUM NRP 1313 100 022
Dosen Pembimbing
Santi Wulan Purnami, M.Si, Ph.D Irhamah, M.Si, Ph.D
PROGRAM STUDI SARJANA DEPARTEMEN STATISTIKA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT TEKNOLOGI SEPULUH NOPEMBER
FINAL PROJECT – SS141501
OPTIMIZATION OF SUPPORT VECTOR MACHINE
PARAMETERS USING GENETIC ALGORITHM
FOR MICROARRAY DATA CLASSIFICATION
AGENG PRAMESTHI KUSUMANINGRUM NRP 1313 100 022
Supervisor
Santi Wulan Purnami, M.Si, Ph.D Irhamah, M.Si, Ph.D
UNDERGRADUATE PROGRAMME DEPARTMENT OF STATISTICS
FACULTY OF MATHEMATICS AND NATURAL SCIENCES INSTITUT TEKNOLOGI SEPULUH NOPEMBER
vi
OPTIMASI PARAMETER
SUPPORT VECTOR
MACHINE
MENGGUNAKAN
GENETIC ALGORITHM
UNTUK KLASIFIKASI
MICROARRAY DATA
Nama Mahasiswa : Ageng Pramesthi Kusumaningrum
NRP : 1313 100 022
Departemen : Statistika
Dosen Pembimbing 1 : Santi Wulan Purnami, M.Si, Ph.D Dosen Pembimbing 2 : Irhamah, M.Si, Ph.D
Abstrak
Support Vector Machine (SVM) merupakan metode machine learning untuk mengklasifikasikan data yang telah berhasil digunakan utuk menyelesaikan permasalahan dalam berbagai bidang. Prinsip risk minimization yang digunakan dapat menghasilkan model SVM dengan kemampuan generalisasi yang baik. Permasalahan yang terdapat dalam metode SVM adalah kesulitan dalam menentukan hyperparameter SVM yang optimal, padahal pengaturan nilai parameter secara tepat akan meningkatkan akurasi klasifikasi SVM. Penelitian ini menggunakan Genetic Algorithm (GA) untuk mengoptimasi hyperparameter SVM. Optimasi GA pada SVM dibandingkan dengan optimasi Grid Search untuk membentuk model SVM yang digunakan untuk mengklasifikasikan data pada data microarray, yatu Data Colon Cancer dan Data Leukemia. Dari hasil analisis, metode GA-SVM dapat menghasilkan performa klasifikasi yang lebih baik dibandingkan metode Grid Search SVM untuk data Colon. Pada data Leukemia, metode GA-SVM menghasilkan performa klasifikasi yang sama dengan metode Grid Search SVM, yaitu 100% untuk masing masing ukuran performa klasifikasi.
vii
viii
OPTIMIZATION OF SUPPORT VECTOR MACHINE
PARAMETERS USING GENETIC ALGORITHM
FOR MICROARRAY DATA CLASSIFICATION
Name : Ageng Pramesthi Kusumaningrum
Student’s Number : 1313 100 022 Department : Statistics
Supervisor 1 : Santi Wulan Purnami, M.Si, Ph.D Supervisor 2 : Irhamah, M.Si, Ph.D
Abstract
Support Vector Machine (SVM) is a machine learning method to classify data that has been successfully used to solve problems in various fields. The principle of risk minimization that can be used to produce SVM model have good generalization capability. The problem in the SVM method is the difficulty in determining the optimal SVM hyperparameter, whereas setting the parameter values appropriately will improve the accuracy of SVM classification. This study uses Genetic Algorithm (GA) to optimize SVM hyperparameter. GA optimization in SVM compared with Grid Search optimization to form the SVM model use to classify data on microarray data, Colon Cancer dataset and Leukemia dataset. From the analysis result, GA-SVM method can yield better classification performance than Grid Search SVM for Colon data. In the Leukemia data, GA-SVM method resulted in the same classification performance with the Grid Search SVM method, which is 100% for each classification performance measure.
ix
x
KATA PENGANTAR
Puji syukur penulis panjatkan ke hadirat Allah SWT atas limpahan rezeki, rahmat, dan hidayah-Nya, sehingga penulis dapat menyelesaikan Tugas Akhir dengan judul Optimasi Parameter Support Vector Machine menggunakan Genetic Algorithm untuk Klasifikasi Microarray Data. Penulisan Tugas Akhir dapat berjalan dengan lancar atas bantuan yang diberikan oleh banyak pihak. Oleh karena itu, penulis mengucapkan terima kasih kepada:
1. Dr. Suhartono selaku Ketua Departemen Statistika ITS dan Dr. Sutikno, M.Si selaku Ketua Program Studi S1 Departemen Statistika ITS.
2. Santi Wulan Purnami, M.Si, Ph.D dan Irhamah, M.Si, Ph.D selaku dosen pembimbing yang telah memberikan arahan dan bimbingan dalam menyelesaikan Tugas Akhir ini. 3. Dr.rer.pol. Dedy Dwi Prastyo dan Shofi Andari, S.Stat,
M.Si selaku dosen penguji yang telah memberikan kritik dan saran dalam penyempurnaan Tugas Akhir ini.
4. Dr. Muhammad Mashuri, MT selaku dosen wali penulis atas nasehat yang disampaikan, serta dosen dan karyawan Departemen Statistika.
5. Kedua orang tua, kakak, dan keluarga penulis atas do’a dan dukungan yang telah diberikan.
6. Sahabat penulis atas dukungan yang diberikan.
7. Serta semua pihak yang telah memberikan bantuan kepada penulis
Penulis menyadari bahwa Tugas Akhir ini masih jauh dari kesempurnaan, sehingga besar harapan penulis untuk menerima kritik dan saran untuk perbaikan ke depan. Penulis berharap semoga Tugas Akhir ini dapat bermanfaat.
xi
xii
DAFTAR LAMPIRAN ... xviii
BAB I PENDAHULUAN ... 1
2.1.1 Klasifikasi SVM Linier ... 7
2.1.2 Klasifikasi SVM Nonlinier ... 13
2.2 Optimasi Genetic Algorithm ... 17
2.3 Pre-Processing Data ... 20
2.4 k-fold Cross-validation ... 24
2.5 Ukuran Performa Klasifikasi ... 25
2.6 Microarray Data ... 27
BAB IIIMETODOLOGI PENELITIAN ... 29
3.1 Deskripsi Data ... 29
3.2 Struktur Data ... 30
3.3 Langkah Penelitian ... 31
3.4 Diagram Penelitian ... 33
BAB IVANALISIS DAN PEMBAHASAN ... 37
xiii
4.1.1 Karakteristik Data Colon Cancer ... 37
4.1.2 Karakteristik Data Leukemia ... 39
4.2 Pre-Processing Data ... 41
4.2.1 Transformasi Data ... 41
4.2.2 Seleksi Fitur ... 42
4.3 Klasifikasi dengan Grid Search SVM ... 43
4.3.1 Klasifikasi dengan Grid Search SVM pada Data Colon Cancer ... 44
4.3.2 Klasifikasi dengan Grid Search SVM pada Data Leukemia ... 47
4.4 Prosedur Optimasi Parameter SVM dengan Genetic Algorithm ... 50
4.5 Klasifikasi dengan GA-SVM ... 56
4.5.1 Klasifikasi dengan GA-SVM pada Data Colon Cancer ... 56
4.5.2 Klasifikasi dengan GA-SVM pada Data Leukemia ... 58
4.6 Perbandingan Hasil Klasifikasi menggunakan Metode Grid Search SVM dengan GA-SVM .. 60
BAB VKESIMPULAN DAN SARAN ... 63
5.1 Kesimpulan ... 63
5.2 Saran ... 63
DAFTAR PUSTAKA ... 65
LAMPIRAN ... 71
xiv
DAFTAR GAMBAR
Halaman
Gambar 2.1 Konsep Hyperplane pada SVM Linier ... 8
Gambar 2.2 Hyperplane dan Margin SVM pada Data yang Dapat Dipisahkan secara Linier (Linearly Separable) ... 9
Gambar 2.3 Hyperplane dan Margin SVM pada Data yang Tidak Dapat Dipisahkan secara Linier (Linearly Nonseparable SVM) ... 12
Gambar 2.4 Ilustrasi Pengaruh Parameter C ... 13
Gambar 2.5 Hyperplane pada SVM Nonlinier ... 14
Gambar 2.6 Ilustrasi Pengaruh Parameter γ ... 16
Gambar 2.7 Algoritma FCBF ... 23
Gambar 2.8 Ilustrasi k-fold Cross-validation ... 24
Gambar 2.9 Ilustrasi ROC Curve dan AUC ... 26
Gambar 3.1 Diagram Alir Penelitian ... 33
Gambar 3.2 Diagram Alir Analisis Klasifikasi menggunakan Grid Search SVM ... 34
Gambar 3.3 Diagram Alir Analisis Klasifikasi menggunakan GA-SVM ... 35
Gambar 4.1 Pengamatan pada Data Colon Cancer ... 38
Gambar 4.2 Persebaran Data dari Beberapa Fitur pada Data Colon Cancer ... 39
Gambar 4.3 Pengamatan pada Data Leukemia ... 40
Gambar 4.4 Persebaran Data dari Beberapa Fitur pada Data Leukemia ... 40
Gambar 4.5 Ilustrasi Satu Buah Kromosom dengan Dua Gen ... 50
Gambar 4.6 Proporsi Kromosom Terpilih ... 51
Gambar 4.7 Ilustrasi Proses Pindah Silang ... 54
Gambar 4.8 Ilustrasi Proses Mutasi ... 54
Gambar 4.9 Ilustrasi Elitisme pada Generasi ke-1 ... 55
xv
xvi
DAFTAR TABEL
Halaman Tabel 2.1 Confusion Matrix ... 25 Tabel 3.1 Struktur Data pada Data Colon Cancer ... 30 Tabel 3.2 Struktur Data pada Data Leukemia ... 30 Tabel 4.1 Rata-rata dan Standar Deviasi Sebelum dan
Sesudah Transformasi pada D Akurasi (%)ata
Colon ... 41 Tabel 4.2 Rata-rata dan Standar Deviasi Sebelum dan
Sesudah Transformasi pada Data Leukemia ... 42 Tabel 4.3 Jumlah Fitur Sebelum dan Sesudah FCBF ... 43 Tabel 4.4 Hasil Kombinasi Range Parameter pada Data
Colon Cancer (Training) ... 44 Tabel 4.5 Hasil Percobaan Grid Search SVM pada Data
Colon Cancer (Training) dengan range C=[23,27]dan γ=[2-9, 2-3] ... 45 Tabel 4.6 Performa Klasifikasi mengggunakan Parameter
Terbaik dari Grid Search SVM pada Data Colon Cancer (Testing) ... 46 Tabel 4.7 Hasil Kombinasi Range Parameter Data
Leukemia (Training) ... 47 Tabel 4.8 Hasil Percobaan Grid-Search SVM pada Data
Leukemia (Training) dengan range C=[2-1, 23]
dan γ=[2-3
, 23] ... 48 Tabel 4.9 Performa Klasifikasi menggunakan Parameter
Terbaik dari Grid Search SVM pada Data Leukemia (Testing) ... 49 Tabel 4.10 Ilustrasi Nilai Fitness tiap Kromosom ... 51 Tabel 4.11 Ilustrasi Nilai Fitness, Fitness Relatif, Fitness
Kumulatif dan Bilangan Acak... 52 Tabel 4.12 Hasil GA-SVM pada Data Colon Cancer ... 57 Tabel 4.13 Performa Klasifikasi Parameter Terbaik dari
xvii
xviii
DAFTAR LAMPIRAN
Halaman Lampiran 1 Fitur pada Data Colon Hasil Seleksi Fitur ... 71 Lampiran 2 Fitur pada Data Leukemia Hasil Seleksi Fitur .. 71 Lampiran 3 Program Grid Search SVM untuk Data Colon
Cancer pada R ... 72 Lampiran 4 Program Grid Search SVM untuk Data
Leukemia pada R ... 73 Lampiran 5 Program Genetic Algorithm SVM untuk Data
Colon Cancer pada R ... 74 Lampiran 6 Program Genetic Algorithm SVM untuk Data
Leukemia pada R ... 75 Lampiran 7 Program Menghitung Performa Klasifikasi
SVM untuk Data Colon Cancer pada R ... 76 Lampiran 8 Program Menghitung Performa Klasifikasi
xix
1
BAB I
PENDAHULUAN
1.1 Latar Belakang
Support Vector Machine (SVM) merupakan metode pattern recognition yang akhir-akhir ini banyak mendapatkan perhatian (Byun & Lee, 2002). Pattern recognition bertujuan untuk mengklasifikasikan data berdasarkan pengetahuan apriori atau informasi statistik yang terkandung dalam data mentah yang merupakan suatu alat yang berguna dalam pemisahan data. Sebagai bagian dari supervised learning, SVM membentuk suatu model klasifikasi menggunakan data pelatihan yang tersedia untuk memprediksi keanggotaan dari data pengamatan baru serta menggunakan data pengujian untuk melakukan validasi model.
Pembentukan model klasifikasi pada SVM didasarkan pada risk minimization yang menghasilkan kemampuan untuk menggeneralisasi permasalahan dengan baik dan mengatasi adanya overfitting (Gunn, 1998). Dengan adanya kemampuan generalisasi, SVM mampu menghasilkan akurasi yang tinggi dan tingkat kesalahan yang relatif kecil. Pada perkembangannya, SVM telah berhasil digunakan untuk menyelesaikan permasalahan dalam berbagai bidang, di antaranya adalah klasifikasi pada data microarray (Furey, Cristianini, Duffy, Bednarski, Schummer, & Haussler, 2000), diagnosis penyakit (Novianti & Purnami, 2012), digital images and audio identification (Guo, Li, & Chan, 2000), dan plant disease recognition (Tian, Hu, Ma, & Han, 2012).
Andres (2006) menunjukkan bahwa metode SVM memberikan tingkat kesalahan yang lebih kecil dibandingkan dengan metode Diagonal Linear Discriminant Analysis (DLDA). Statnikov, Wang, dan Aliferis (2008) juga melakukan klasifikasi pada 10 jenis data microarray menggunakan SVM dan dibandingkan dengan Random Forest dengan hasil yaitu SVM memberikan rata-rata kinerja hasil klasifikasi lebih baik dibandingkan dengan metode Random Forest.
Metode SVM memiliki kelemahan yaitu SVM mengalami kesulitan dalam menentukan nilai parameter yang optimal. Yenaeng, Saelee dan Samai (2014) menyatakan bahwa permasalahan terbesar dalam mengatur model SVM adalah menentukan nilai hyperparameter dari SVM. Padahal, pengaturan nilai parameter secara tepat akan meningkatkan akurasi klasifikasi dari model SVM (Huang & Wang, 2006). Untuk mendapatkan parameter yang akan menghasilkan model SVM yang paling baik, maka dilakukan optimasi parameter pada model SVM. Optimasi parameter tersebut berarti menetukan hyperparameter model SVM yang paling optimal dan menghasilkan model SVM dengan hasil klasifikasi yang paling baik. Metode Grid Search merupakan metode yang paling banyak digunakan untuk optimasi parameter (Chen, Ling, Tang & Xia, 2016). Beberapa metode optimasi parameter lainnya yang dapat dilakukan pada SVM di antaranya adalah Genetic algorithm (GA), Clonal section algorithm (CSA), Ant colony optimization (ACO), Particle swarm optimization (PSO), dan Simulated annealing (SA) (Huang & Wang, 2006; Rossi & de Cavarlho, 2008; Syarif, Bennett, & Wills, 2013; Härdle, Prastyo, & Hafner, 2014). Pada penelitian ini, metode GA akan digunakan untuk mengoptimasi nilai parameter pada model SVM, sehingga dengan parameter yang optimal tersebut diharapkan dapat meningkatkan akurasi hasil klasifikasi.
3
Keuntungan menentukan parameter menggunakan GA yaitu, GA sangat bermanfaat untuk diimplementasikan pada saat range terbaik dari parameter SVM sama sekali tidak diketahui (Syarif dkk., 2013). Selain itu, GA mampu menghasilkan parameter SVM yang optimal secara bersamaan (Yenaeng dkk., 2014). Penelitian tentang GA-SVM sebelumnya telah dilakukan oleh Irawati (2010), yaitu tentang optimasi parameter SVM menggunakan GA untuk menyelesaikan permasalahan klasifikasi pada 3 data dari UCI Machine Learning Repository (Image Letter Recognition, Pima Indians Diabetes, dan Protein Localization Site), dengan hasil yaitu metode SVM dengan GA menghasilkan nilai akurasi yang lebih baik dibandingkan dengan metode SVM tanpa GA. Berdasarkan penelitian yang dilakukan oleh Syarif dkk. (2013), waktu yang diperlukan GA untuk mengoptimasi parameter SVM 15,9 kali lebih cepat dibandingkan dengan metode Grid Search.
SVM, yaitu Yu dan Liu (2003) menyatakan bahwa data yang berdimensi tinggi dapat berisi informasi yang tidak relevan serta berlebihan yang dapat menurunkan kinerja dari SVM. Untuk mengatasi permasalahan tersebut perlu dilakukan seleksi fitur untuk memilih fitur yang terbaik. Menurut Wang, Song, Wei, dan Li (2011), seleksi fitur merupakan bagian penting untuk mengoptimalkan kinerja dari classifier. Metode seleksi fitur yang akan digunakan pada penelitian ini adalah metode Fast Correlation Based Filter (FCBF) (Yu & Liu, 2003). Metode seleksi fitur dengan FCBF dapat memberikan hasil yang lebih baik dibandingkan dengan metode seleksi fitur lainnya dalam hal menangani seleksi fitur pada microarray. Hal ini telah dibuktikan sebelumnya oleh Rusydina dan Purnami (2016), yaitu metode FCBF menghasilkan fitur terseleksi yang menghasilkan akurasi klasifikasi yang lebih baik dan waktu komputasi yang lebih cepat dibandingkan dengan menggunakan seluruh fitur dan metode Correlation Based Filter (CFS). Selain itu, metode FCBF dapat mereduksi lebih banyak fitur daripada metode CFS (Rusydina & Purnami, 2016).
5
Untuk menunjukkan efektivitas metode GA-SVM dalam melakukan klasifikasi, maka GA-SVM dibandingkan dengan metode Grid Search SVM. Masing-masing metode akan digunakan untuk menyelesaikan permasalahan klasifikasi yang terdapat pada Data Colon Cancer dan Data Leukemia. Hasil klasifikasi dinilai berdasarkan ukuran performa klasifikasi meliputi akurasi, sensitivitas, spesifisitas, G-mean, dan AUC (Sokolova & Lapalme, 2009; Bekkar, Djemaa, & Alitouche, 2013).
1.2 Rumusan Masalah
1.3 Tujuan Penelitian
Berdasarkan permasalahan, tujuan dari penelitian ini adalah sebagai berikut.
1. Mengklasifikasikan data pada microarray data menggunakan Grid Search Support Vector Machine. 2. Mendapatkan prosedur optimasi parameter pada Support
Vector Machine menggunakan Genetic Algorithm pada microarray data.
3. Menerapkan optimasi Genetic Algorithm pada Support Vector Machine untuk klasifikasi pada microarray data.
1.4 Manfaat Penelitian
Dengan melakukan penelitian ini, manfaat yang diperoleh adalah mampu menyelesaikan permasalahan optimasi parameter SVM menggunakan GA pada high dimensional data jenis microarray.
1.5 Batasan Masalah
Batasan masalah yang digunakan pada penelitian ini adalah:
1. Data yang digunakan merupakan dua data microarray, yaitu Data Colon Cancer (Alon dkk., 1999). dan Data Leukemia (Golub dkk., 1999) .
2. Fungsi kernel yang digunakan adalah Gaussian RBF Kernel.
3. Nilai probabilitas pindah silang Pc yang digunakan sebesar 0,6; 0,7; dan 0,8.
4. Nilai probabilitas mutasi Pm yang digunakan sebesar 0,01; 0,02; dan 0,03.
7
BAB II
TINJAUAN PUSTAKA
2.1 Support Vector Machine
Support Vector Machine (SVM) dikembangkan oleh Vapnik pada tahun 1992 bersama dengan Bernhard Boser dan Isabelle Guyon (Han, Kamber, & Pei, 2012). SVM merupakan metode machine learning yang melakukan suatu teknik untuk menemukan fungsi pemisah (classifier) yang dapat memisahkan data menjadi dua kelas berbeda (Vapnik, 2002). Strategi yang digunakan adalah meminimalkan kesalahan pada data training dan dimensi Vapnik-Chervokinensis (VC) yang disebut dengan Structural Risk Minimization (SRM). Tujuan dari SVM adalah mendapatkan hyperplane terbaik yang memisahkan dua buah kelas (Han dkk, 2012). Mendapatkan hyperplane terbaik adalah sama dengan memaksimalkan jarak antara hyperplane dengan pattern terdekat dari masing-masing kelas (margin). Kelebihan dari metode SVM adalah kemampuan generalisasi, yaitu kemampuan untuk mengklasifikasikan data lain yang tidak termasuk dalam data yang dipakai pada machine learning (Gun, 1998). Tingkat generalisasi yang dihasilkan oleh SVM tidak dipengaruhi oleh dimensi dari vektor input, sehingga SVM mampu mengatasi permasalahan curse of dimensionality. Kelebihan lainnya menurut Gunn (1998) adalah konsep SRM yang dimiliki SVM mampu mengatasi permasalahan overfitting. Prinsip dasar SVM adalah linear classifier yang kemudian dikembangkan agar dapat bekerja pada permasalahan yang non linier (Nugroho, Witarto, & Handoko, 2013).
2.1.1 Klasifikasi SVM Linier
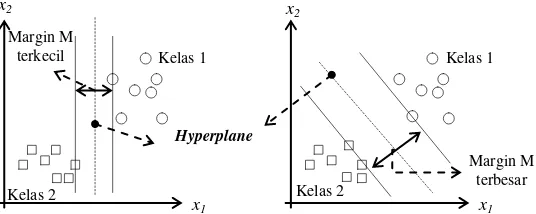
Gambar 2.1 Konsep Hyperplane pada SVM Linier
Gambar 2.1 menunjukkan beberapa pengamatan yang merupakan anggota kelas 1 dan kelas 2. Hyperplane ditunjukkan oleh garis putus-putus pada gambar, sedangkan margin adalah jarak antara hyperplane dengan data yang paling dekat dengan hyperplane pada tiap kelas. Kemampuan generalisasi tergantung pada lokasi hyperplane, dan hyperplane dengan margin terbesar disebut dengan hyperplane yang optimal (Abe, 2010). Usaha untuk mendapatkan lokasi hyperplane ini adalah inti dari proses pembelajaran pada SVM (Nugroho dkk., 2013).
Diberikan data training input berdimensi m, yaitu xi
dengan (i 1, ,M)yang termasuk dalam kelas 1 atau kelas 2, sehingga yi1 untuk kelas 1 dan yi 1 untuk kelas 2. Apabila
data terpisah secara linier, maka fungsi pemisah/hyperplane didefinisikan oleh (Abe, 2010)
( ) T
D x w x b (2.1)
dimana wadalah vektor berdimensi m, b adalah bias, dan untuk 1, 2, , .
9
pemisahan kelas dilakukan dengan mempertimbangkan pertidaksamaan berikut.
( T ) 1, 1, 2, , .
i i
y w x b i M (2.4)
Ilustrasi hyperplane pemisah dan margin SVM pada data yang dapat dipisahkan secara linier (linearly separable) terdapat pada gambar berikut.
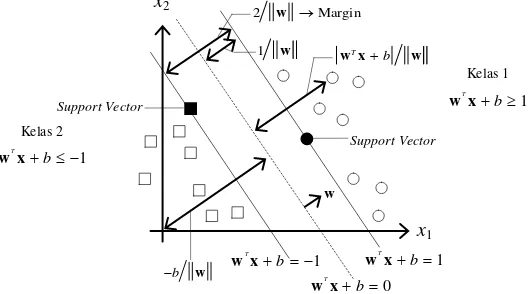
Gambar 2.2 Hyperplane dan Margin SVM pada Data yang Dapat Dipisahkan
secara Linier (Linearly Separable)
Nilai margin diketahui dengan menghitung jarak terdekat hyperplane dengan data yang paling dekat dengan hyperplane pada tiap kelas. Jarak antara data x pada tiap dengan hyperplane pada tersebut adalah
Jarak terdekat antara antara data x dengan hyperplane pada kelas 1 dan 2 masing-masing adalah 1
w , maka nilai margin diperoleh
Hyperplane yang optimal diperoleh dengan memaksimumkan nilai margin 2
w . Nilai 2
w akan maksimum
jika nilai w minimum. Meminimumkan nilai w dapat diperoleh dengan meminimumkan nilai 1 2,
2 w sehingga
formulasi permasalahan optimasi pada SVM untuk klasifikasi linier dalam bentuk primal adalah
2
1 min
2 w (2.6)
yang memenuhi batasan pada persamaan (2.4). Solusi dari permasalahan persamaan kuadratik dengan fungsi batasan berupa pertidaksamaan tersebut dapat diperoleh dengan fungsi Lagrange Multipliers (Lagrangian) berikut.
dari persamaan 2.7 dapat dihitung dengan meminimalkan LP
terhadap w dan b serta memaksimalkan LP terhadap i.
Persamaan (2.7) merupakan permasalahan primal, sehingga perlu ditransformasi menjadi bentuk permasalahan dual dengan menggunakan kondisi Karush-Kuhn-Tucker (KKT), yaitu
11
Persamaan dual diperoleh dengan mensubstitusikan pers. (2.8) dan (2.9) ke dalam pers. (2.7), maka permasalahan secara dual
terhadap i dengan fungsi batasan
1
Memaksimumkan persamaan (2.10) dengan batasan pada persamaan (2.11) akan menentukan nilai pengganda Lagrange,
.
i
Data yang berasosiasi positif dengan i adalah support
vectors untuk kelas 1 dan 2. Kemudian hyperplane pemisah yang optimal adalah
support vector, kemudian b diberikan oleh 1
Selanjutnya, data testing x akan diklasifikasikan menjadi
Kelas 1, jika ( ) 0,
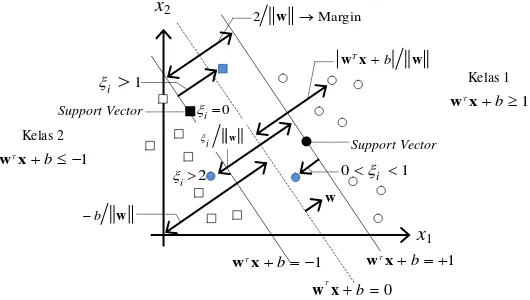
Gambar 2.3 Hyperplane dan Margin SVM pada Data yang Tidak Dapat Dipisahkan secara Linier (Linearly Nonseparable SVM)
Teknik soft margin dilakukan dengan memodifikasi persamaan 2.4 dengan memasukkan variabel slack (ξi ≥ 0) pada persamaan tersebut (Ben-Hur & Weston, 2010), sehingga diperoleh
( T ) 1 , 1, 2, ,
i i i
y w x b i M (2.15)
dimana ξi merupakan variabel slack yang memungkinkan suatu data berada pada margin (0≤ ξi ≤1, disebut margin error) atau dipilih untuk mengontrol trade off antara margin dengan kesalahan klasifikasi ξ. Nilai C yang besar berarti akan memberikan penalti yang lebih besar terhadap kesalahan klasifikasi tersebut (Nugroho dkk., 2013). Nilai C akan memberikan pengaruh terhadap bentuk hyperplane serta hasil klasifikasi seperti pada ilustrasi berikut.
13
Sumber: Ben-Hur dan Weston, 2010
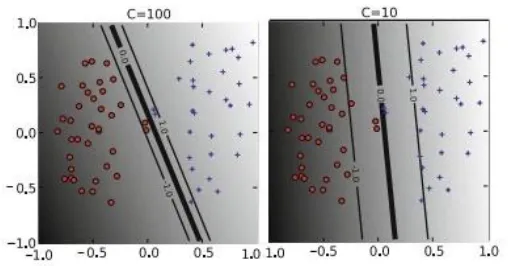
Gambar 2.4 Ilustrasi Pengaruh Parameter C (Ben-Hur dan Weston, 2010)
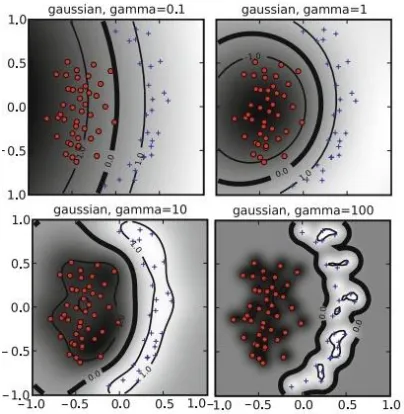
Gambar 2.4 menunjukkan pengaruh parameter C terhadap bentuk hyperplane dan hasil klasifikasi yang dilakukan pada dua kelas, yaitu Kelas 1 (biru) dan Kelas 2 (merah). Semakin besar nilai parameter C, maka semakin besar pula penalti yang diberikan kepada kesalahan klasifikasi. Nilai parameter C yang semakin kecil akan mengabaikan pengamatan yang dekat dengan hyperplane dan memperbesar margin. Pada saat nilai C besar, penalti yang besar diberikan kepada margin error. Seperti pada Gambar 2.4, saat C=100, dua pengamatan dari Kelas 1 yang berada paling dekat dengan hyperplane adalah support vectors dan mempengaruhi orientasi hyperplane. Saat nilai C semakin kecil (C=10), dua pengamatan dari Kelas 1 yang disebutkan sebelumnya berubah menjadi margin error, orientasi hyperplane berubah, dan memberikan margin yang lebih besar.
2.1.2 Klasifikasi SVM Nonlinier
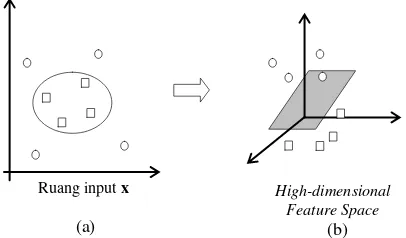
permasalahan yang tidak dapat dipisahkan secara linier (Nugroho dkk., 2013). Penyelesaian permasalahan yang bersifat nonlinear separable dilakukan dengan memetakan ruang input ke ruang berdimensi lebih tinggi yang disebut feature space.
Gambar 2.2 memberikan ilustrasi mengenai konsep hyperplane pada SVM nonlinier. Ruang input (Gambar 2.2 (a)) dengan dua dimensi tidak dapat memisahkan data ke dalam dua kelas secara linier. Maka dari itu dilakukan pemetaan vektor input oleh fungsi ( )x ke ruang vektor baru yang berdimensi lebih tinggi (3 dimensi) (Gambar 2.5 (b)). Gambar 2.5 (b) tersebut menunjukkan bahwa dengan 3 dimensi data dapat dipisahkan dalam dua kelas secara linier oleh sebuah hyperplane.
Sumber:
Gambar 2.5 Hyperplane pada SVM Nonlinier (Nugroho dkk., 2003)
Pemetaan pada vektor input x berdimensi m ke feature space berdimensi l dilakukan menggunakan fungsi pemetaan x, yaitu ( ) ( ( ), , ( ))1
T l
x
x
x berupa fungsi kernel yang ditentukan oleh pengguna. Fungsi kernel dirumuskan sebagai berikut.( , ) ( )T ( )
i j i j
K x x x x (2.17)
15
menyelesaikan permasalahan SVM nonlinier adalah sebagai berikut (Abe, 2010).
c. Gaussian/Radial Basis Function (RBF)
2
( , ) expi j i j
K x x
x x (2.20)Misalkan terdapat tiga pengamatan dengan tiga fitur yaitu
1 2 3
T
a , b
5 4 6
T, dan c
4 2 5
T. Data tersebut akan ditransformasi menggunakan fungsi kernel linier seperti pada pers. (2.20), untuk i, j =1, 2, 3. Dari data pengamatan tersebut diperoleh 1
1 5 4 ,
2
2 4 2 , dan
dalam sebuah matriks kernel K sebagai berikut.dapat mengatasi permasalahan nonlinieritas pada data. Hsu, Chang, dan Lin (2003) merekomendasikan fungsi kernel RBF untuk digunakan karena kemampuannya dalam mengatasi nonlinieritas dan RBF memiliki kesulitan numerik yang lebih sedikit dibandingkan fungsi kernel lainnya.
Pada fungsi kernel RBF, terdapat parameter γ yang nilainya perlu diatur untuk mendapatkan hasil klasifikasi yang baik. Ilustrasi pengaruh nilai parameter γ (C tetap) terhadap pembentukan hyperplane ditunjukkan oleh Gambar 2.4. Parameter γ menentukan bagaimana data training dipetakan ke feature space. Pada saat γ bernilai kecil, hyperplane yang terbentuk mendekati linier.
Gambar 2.6 Ilustrasi Pengaruh Parameter γ(Ben-Hur dan Weston, 2010)
Fungsi keputusan pada SVM nonlinier diperoleh melalui persamaan
( ) i i ( , )i ,
i S
D y K b
17
dimana nilai b diperoleh dari
1
dimana U adalah himpunan indeks unbounded support vector. Selanjutnya, data testing diklasifikasikan menggunakan fungsi keputusan berikut.
2.2 Optimasi Genetic Algorithm
Genetic algorithm (GA) pertama kali ditemukan oleh John Holand pada tahun 1975. Konsep GA didasarkan pada teori evolusi dengan prinsip seleksi alam yang dikembangkan oleh Darwin. GA merupakan teknik identifikasi pendekatan solusi untuk permasalahan optimasi. Optimasi dengan GA menggunakan kriteria kinerja (fitness) untuk mendapatkan solusi optimum. Dalam GA, solusi optimum diperoleh melalui proses seleksi, mutasi dan persilangan yang dilakukan secara berulang. GA memanipulasi populasi struktur simbolis, yang mewakili solusi, agar mendapatkan adaptasi yang terbaik yang menghasilkan solusi yang terbaik untuk suatu permasalahan. Sebuah solusi yang dibangkitkan dalam algoritma genetika disebut sebagai kromosom, sedangkan kumpulan kromosom-kromosom tersebut disebut sebagai populasi (Petrus, Soewono, Agung, & Sihana, 2009). Kromosom dari satu populasi diambil dan digunkan untuk membentuk populasi baru. Tujuan utama dari GA adalah mendapatkan populasi baru yang lebih baik dibandingkan populasi sebelumnya.
Parameter tersebut di antaranya adalah parameter penalti C dan parameter fungsi kernel. Pengaturan parameter yang tepat akan meningkatkan akurasi klasifikasi pada SVM (Yenaeng dkk., 2014).
Langkah-langkah dilakukan pada metode GA menurut Ismail dan Irhamah (2008) adalah sebagai berikut.
Langkah 1 : Define, yaitu mendefinisikan operator pada GA yang sesuai dengan permasalahan.
Langkah 2 : Initialize, yaitu membentuk populasi awal yang terdiri atas N buah kromosom.
Langkah 3 : Fitness, yaitu mengevaluasi fitness dari setiap kromosom pada populasi.
Langkah 4 : Selection, yaitu menerapkan metode seleksi roulette wheel yang memberikan suatu set populasi perkawinan M dengan ukuran N.
Langkah 5 : Crossover, yaitu proses persilangan. Proses ini memasangkan semua kromosom pada M secara acak sehingga membentuk N/2 pasang. Persilangan diterapkan peluang Pc pada setiap pasang dan bentuk N keturunan kromosom (offspring), apabila
nilai bilangan acak ≥ Pc maka keturunan
merupakan salinan dari orang tua yang tepat. Langkah 6 : Mutation, yaitu menggunakan peluang mutasi (Pm)
untuk melakukan proses mutasi keturunan.
Langkah 7 : Replace, yaitu mengganti populasi yang lama dengan populasi baru. Populasi baru diperoleh dengan memilih N kromosom terbaik yang diperoleh dengan cara mengevaluasi nilai fitness dari orang tua dan keturunan baru
Langkah 8 : Test, yaitu apabila kriteria telah terpenuhi, maka proses berhenti dan kembali ke solusi terbaik dari populasi saat ini. Apabila kriteria belum terpenuhi, maka kembali ke langkah 2.
19
ukuran yang menunjukkan baik tidaknya sebuah kromosom dan menentukan apakah baik tidaknya kromosom tersebut dalam sebuah populasi. Nilai fitness menunjukkan kemampuan solusi untuk bertahan, yaitu peluang untuk menjadi anggota dari populasi selanjutnya dan menghasilkan keturunan dengan karakteristik yang sama dengan menurunkan informasi genetik melalui mekanisme evolusioner (Lessmann, Stahbolck, & Crone, 2005).
Selection (seleksi) menentukan kromosom yang akan digunakan pada tahap selanjutnya (pindah silang) dari populasi yang ada saat ini (Härdle, Prastyo, & Hafner, 2014). Kromosom diseleksi dengan mengevaluasi nilai fitnessnya berdasarkan konsep aturan evolusi Darwin. Kromosom yang memiliki nilai fitness yang tinggi akan memiliki peluang yang lebih besar untuk terpilih pada tahap selanjutnya (Weise, 2007 diacu dalam Petrus dkk., 2009). Kromosom terpilih selanjutnya digunakan pada operasi pindah silang dan mutasi. Metode seleksi yang paling banyak digunakan adalah roulette wheel.
Crossover (pindah silang) merupakan operator pada GA yang utama. Langkah crossover yaitu melakukan pertukaran antar kromosom pada satu generasi sehingga membentuk kromosom baru yang memiliki harapan yang lebih baik daripada induknya. Kromosom-kromosom baru tersebut disebut dengan keturunan (offspring). Kemungkinan suatu kromosom mengalami crossover ditentukan berdasarkan nilai probabilitas crossover (Pc) yang sebelumnya telah ditentukan. Lessman dkk. (2005) merekomendasikan nilai Pc yang besar. Schaffer, Caruana, Eshelman, dan Das (1989) menyebutkan nilai Pc optimal terletak pada range 0,75-0,95.
adanya mutasi, individu baru dapat diciptakan dengan melakukan pengubahan terhadap satu atau lebih nilai gen pada individu yang sama. Peluang dari jumlah total gen pada populasi yang mengalami mutasi ditentukan oleh peluang mutasi (Pm). Lessman dkk. (2005) merekomendasikan nilai Pm yang kecil. Nilai Pm yang sering digunakan pada implementasi GA adalah pada range 0,001 dan 0,05 (Davis, 1991 dalam Ismail & Irhamah, 2008).
Berdasarkan pada teori Darwin, yatu “Survival of Fittest”, individu yang lebih baik memiliki peluang yang lebih besar untuk dibawa pada generasi yang berikutnya. Proses pembentukan generasi berikutnya dilakukan dengan mengganti beberapa offspring maupun induk dari individu yang dilakukan oleh operator pengganti berdasarkan pada nilai fitnessnya. Elitisme merupakan salah satu teknik yang dilakukan untuk mempertahankan suatu individu terbaik yang memiliki nilai fitness tertinggi untuk dapat bertahan hidup untuk generasi yang selanjutnya (Irawati, 2010). Pada penelitian ini, banyaknya individu yang bertahan untuk generasi yang selanjutnya adalah sebanyak 5 individu untuk setiap generasi.
2.3 Pre-Processing Data
Sebelum data diproses menggunakan teknik data mining, data mentah perlu dipersiapkan terlebih dahulu. Pre-processing data merupakan proses yang dilakukan untuk meningkatkan kualitas data mentah, sehingga dapat meningkatkan akurasi dan efisiensi untuk proses data mining selanjutnya. Apabila input data berkualitas, maka akan menghasilkan analisis data yang berkualitas (Han dkk., 2012).
a. Transformasi
21
besar mendominasi fitur dengan range nilai yang lebih kecil. Selain itu, scaling dapat menghindari kesulitan numerik selama perhitungan (Hsu dkk., 2010). Setiap fitur secara linier ditransformasi menjadi range [0, 1] menggunakan persamaan berikut.
Seleksi fitur merupakan proses dalam pre-processing data yang digunakan untuk menghapus fitur yang tidak relevan dan redundant (berlebihan) (Gorunescu, 2011). Proses ini menyeleksi fitur yang berguna untuk membangun prediksi yang baik dan mengurangi jumlah fitur yang akan dibawa pada analisis. Yu dan Liu (2003) menyatakan bahwa seleksi fitur secara efektif mampu mereduksi dimensi data, menghapus fitur yang tidak relevan dan tidak diperlukan untuk analisis, meningkatkan efisiensi machine learning, memperbaiki kinerja machine learning, dan membuat hasil dari machine learning lebih dapat dimengerti. Selain itu, semakin kecil jumlah fitur akan mempercepat proses komputasi. Dalam klasifikasi, seleksi fitur merupakan bagian penting untuk mengoptimalkan kinerja dari classifier (Wang dkk., 2011) serta mempengaruhi akurasi dari klasifikasi (Huang & Wang, 2006). Pada dasarnya, algoritma seleksi fitur dapat dibedakan menjadi tiga jenis, yaitu filter, wrapper, dan embedded (Guyon & Elisseeff, 2003).
diartikan pula bahwa fitur yang baik adalah fitur yang berkorelasi tinggi terhadap kelas tetapi tidak berkorelasi terhadap fitur yang lainnya. Maka dari itu, Yu dan Liu (2003) melakukan dua pendekatan untuk mengukur korelasi, yaitu dengan linear correlation coefficient dan teori informasi.
Pendekatan linear correlation coefficient untuk setiap fitur (X, Y) dengan n pengamatan dirumuskan sebagai
menggunakan pendekatan ini yaitu fitur yang tidak relevan mudah untuk dihilangkan dengan memilih fitur yang nilai korelasinya 0 dan membantu mengurangi redundant pada fitur-fitur yang sudah dipilih. Namun, keterbatasan dari pendekatan ini yaitu hanya dapat digunakan pada fitur dengan nilai numerik.23
dimana P x( )i adalah prior probabilities untuk semua nilai X dan
i j
P x y adalah posterior probabilities dari X jika diketahui nilai
Y. Dari entropy tersebut dapat diperoleh Information Gain (IG) sebagai berikut
H
IG X Y H X X Y (2.28)
Korelasi antar fitur diukur melalui nilai symmetrical uncertainty. Nilai symmetrical uncertainty terdapat pada range [0,1]. Symmetrical uncertainty (SU) dirumuskan sebagai berikut
,
2
Gambar 2.7 Algoritma FCBF ( Yu & Liu, 2003)
input: S(F1,F2,...,FN,C) //Data Training
//Nilai threshold yang ditentukan
output: Sbest //Fitur Optimal
19 Fp= getNextElement(S’list, Fp);
20 end until (Fp ==NULL)
21 Sbest=S’list;
Pemilihan fitur yang baik untuk klasifikasi berdasarkan analisis korelasi pada fitur (serta kelas) melibatakan dua aspek, yaitu (1) bagaimana menentukan apakah suatu fitur relevan dengan kelas atau tidak, dan (2) bagaimana memutuskan apakah suatu fitur yang relevan tersebut redundant (berlebihan) atau tidak saat mempertimbangkannya dengan fitur yang relevan lainnya. Algoritma yang digunakan pada pemilihan fitur berdasarkan FCBF terdapat pada gambar 2.7.
2.4 k-fold Cross-validation
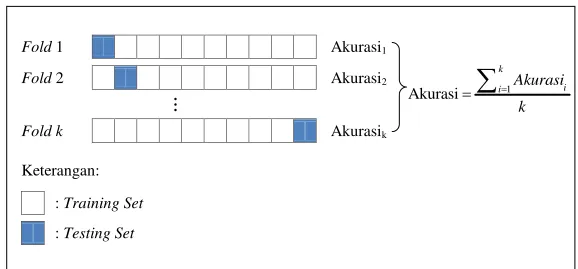
Salah satu teknik untuk mengevaluasi kinerja sebuah model adalah k-fold cross-validation. Dalam k-fold cross-validation, sebuah data (D) secara acak dibagi menjadi k subsets data (folds), yaitu D1, D2,..., Dk dengan ukuran yang sama (Han dkk., 2012). Model dibentuk menggunakan k-1 subsets sebagai data training dan diuji menggunakan 1 subset yang tersisa sebagai data testing. Proses cross-validation ini diulang sebanyak k kali dengan masing-masing k subset tersebut digunakan tepat satu kali sebagai data testing. Kinerja klasifikasi diperoleh dengan menghitung rata-rata dari nilai kinerja klasifikasi yang diperoleh pada setiap fold. Pada umumnya, banyaknya fold (k) yang digunakan untuk mengestimasi kinerja klasifikasi adalah 10 (Han dkk., 2012). Ilustrasi k-fold cross-validation dengan kinerja klasifikasi berupa akurasi terdapat pada gambar berikut.
Gambar 2.8 Ilustrasi k-fold Cross-validation
25
2.5 Ukuran Performa Klasifikasi
Hasil dari klasifikasi dapat dievaluasi dengan menghitung banyaknya prediksi benar pada kelas positif (TP), banyaknya prediksi benar pada kelas negatif (TN), dan banyaknya prediksi salah pada kelas positif (FP) serta banyaknya prediksi salah pada kelas negatif (FN). Keempat nilai tersebut dapat disusun dalam confusion matrix berikut.
Tabel 2.1 Confusion Matrix
Kelas
Ketepatan klasifikasi dapat diukur menggunakan akurasi, sensitivitas, spesifisitas (Sokolova dan Lapalme, 2009). Akurasi klasifikasi menunjukkan efektivitas classifier secara keseluruhan. Semakin tinggi nilai akurasi, maka semakin baik pula kinerja classifier dalam mengklasifikasikan data. Sensitivitas mengukur efektivitas sebuah classifier untuk mengidentifikasi kelas positif, sedangkan spesifisitas mengukur efektivitas classifier untuk mengidentifikasi kelas negatif.
TN+TP
dihasilkan. G-mean diperoleh dari rata-rata ukur dari sensitivitas dan spesifisitas, yaitu
G-mean= Sensitivitas×Spesifisitas (2.33)
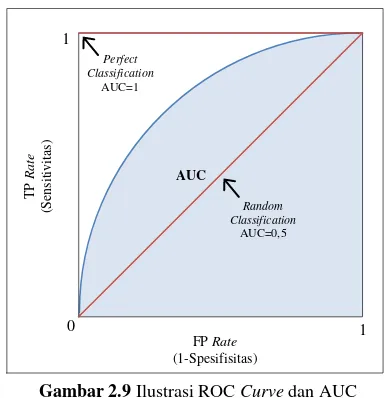
Receiver Operating Characteristic (ROC) Curve menunjukkan hubungan antara true positive rate (TP rate) dan false positive rate (FP rate). TP rate disebut juga dengan sensitivitas, sedangkan nilai FP rate diperoleh dari 1-spesifisitas. Secara teknis, ROC Curve digambarkan oleh nilai FP rate pada sumbu X dan sensitivitas pada sumbu Y.
Gambar 2.9 Ilustrasi ROC Curve dan AUC
Titik (0,0) menunjukkan bahwa TP dan FP bernilai 0 dan kondisi sebaliknya ditunjukkan oleh titik (1,1), yaitu TN dan FN bernilai 0. Titik (0,1) menunjukkan klasifikasi yang sempurna, yaitu tidak terdapat FP dan FN. Pada ROC Curve terdapat diagonal yang membagi grafik menjadi dua bagian. Titik yang berada di atas diagonal menunjukkan hasil klasifikasi yang baik, sedangkan titik yang berada di bawah diagonal menunjukkan hasil klasifikasi yang buruk. AUC merangkum performa klasifikasi pada ROC Curve menjadi suatu nilai ukuran tunggal.
27
AUC dapat diestimasi dengan menggunakan metode trapesium untuk menghitung luasan di bawah ROC Curve, sehingga AUC dapat dihitung menggunakan persamaan berikut.
1
AUC= Sensitivitas+Spesifisitas
2 (2.34)
Nilai AUC bernilai 0,5 sampai dengan 1. Nilai AUC yang semakin besar menunjukkan bahwa hasil klasifikasi semakin baik.
2.6 Microarray Data
Microarray merupakan salah satu teknologi yang memungkinkan peneliti untuk mengukur tingkat ekspresi dari ribuan gen secara bersamaan dalam satu pengamatan dan muncul sebagai perangkat penting dalam penelitian biomedis. Hasil pengukuran dari microarray tersebut biasanya dirangkum dalam daftar gen yang dinyatakan dalam dua kondisi atau diklasifikasikan berdasarkan fenotipnya. Microarray data merupakan jenis dari high dimensional data karena memiliki jumlah gen (fitur) ratusan bahkan ribuan, sedangkan jumlah pengamatan yang biasanya tidak mencapai 100 atau jauh lebih kecil dari jumlah fitur (Yu dan Liu, 2011). Dua metode umum yang dilakukan untuk menganalisis microarray data adalah clustering dan klasifikasi (Selvaraj dan Natarajan, 2011). Berdasarkan infomasi yang dimiliki, microarray memiliki peranan penting dalam penelitian biomedis sebagai alat untuk identifikasi dan klasifikasi penyakit, khususnya kanker.
29
BAB III
METODOLOGI PENELITIAN
3.1 Deskripsi Data
Deskripsi masing-masing data yang akan digunakan dalam penelitian, yaitu Data Colon Cancer dan Data Leukemia adalah sebagai berikut.
a. Data Colon Cancer
Data Colon Cancer merupakan data microarray yang berasal dari penelitian yang dilakukan oleh Alon U, Barkai N, Notterman DA, Gish K, Ybarra S, Mack D, Levine AJ pada tahun 1999. Data diperoleh dari website http://datam.i2r.a-star.du.sg/datasets/krbd. Data Colon ini menyimpan informasi berupa ekspresi gen yang diperoleh dari pengamatan pada jaringan usus yang terkena tumor (tumor colon tissues) dan jaringan usus normal atau tanpa tumor (normal colon tissues). Data Colon terdiri dari 62 pengamatan, 40 diantaranya merupakan pengamatan kelas Tumor dan 22 lainnya merupakan pengamatan kelas Normal. Jumlah fitur yang terdapat pada data Colon adalah sebanyak 2000 fitur. Pada penelitian ini, data dibagi menjadi data training dan data testing. Data training terdiri dari 15 pengamatan kelas Normal dan 27 pengamatan kelas Tumor. Data testing terdiri dari 7 pengamatan kelas Normal dan 13 pengamatan kelas Tumor.
b. Data Leukemia
Tiap pengamatan terdiri dari 7129 fitur yang berasal dari ekspresi gen pasien. Pada penelitian ini, data training terdiri dari 32 pengamatan kelas ALL dan 17 pengamatan kelas AML, sedangkan data testing terdiri dari 15 pengamatan kelas ALL dan 8 pengamatan kelas AML.
3.2 Struktur Data
Berikut ini adalah struktur data untuk masing-masing data yang digunakan dalam penelitian.
a. Struktur Data Colon Cancer
Tabel 3.1 Struktur Data pada Data Colon Cancer
Pengamatan Fitur
b. Struktur Data Leukemia
Tabel 3.2 Struktur Data pada Data Leukemia
ke-31
3.3 Langkah Penelitian
Langkah analisis yang akan digunakan dalam penelitian ini adalah sebagai berikut.
1. Mendeskripsikan data, yaitu data Colon Cancer dan data Leukemia.
2. Melakukan pre-processing data pada masing-masing data. a. Melakukan transformasi pada tiap fitur menggunakan
persamaan (2.22)
b. Melakukan seleksi fitur menggunakan metode FCBF 3. Menentukan fungsi kernel yang digunakan. Pada penelitian
ini menggunakan fungsi kernel Gaussian (RBF).
4. Analisis klasifikasi menggunakan metode Grid search SVM pada masing-masing data.
a. Menentukan range nilai parameter C dan
.
b. Melakukan klasifikasi SVM dengan kombinasi nilai parameter C dan
.c. Menghitung akurasi klasifikasi.
d. Apabila terdapat kombinasi nilai parameter C dan
yang belum dilakukan, maka kembali ke langkah 4b, dan apabila semua kombinasi sudah dilakukan, maka dilanjutkan pada langkah 4e.e. Menentukan nilai parameter C dan
yang paling optimal dari seluruh kombinasi parameter yang sudah dilakukan.f. Menghitung performa klasifikasi
5. Analisis klasifikasi menggunakan metode GA-SVM pada masing-masing data.
a. Menentukan fitness, nilai Pc, Pm, dan stopping criteria. Fitness yang digunakan pada penelitian ini adalah nilai akurasi klasifikasi. Nilai Pc dan Pm yang digunakan merupakan kombinasi dari Pc = 0,6; 0,7; dan 0,8 dengan Pm = 0,01; 0,02; dan 0,03. Stopping criteria yang digunakan antara lain adalah:
- Total generasi yang terbentuk adalah 1000
b. Menyusun kromosom dengan membangkitkan 100 kromosom. Kromosom yang dibangkitkan terdiri dari 2 gen yang menunjukkan hyperparameter SVM, yaitu C dan
.
Nilai inisial kromosom diperoleh dari nilai parameter C dan
yang paling optimal dari langkah 4 (Grid Search SVM).c. Mengevaluasi kromosom berdasarkan nilai fitness. d. Melakukan proses seleksi sebanyak 100 kromosom dari
100 induk yang berasal dari populasi menggunakan seleksi roulette wheel.
e. Melakukan proses pindah silang apabila nilai bilangan acak yang dibangkitkan kurang dari Pc.
f. Melakukan proses mutasi apabila nilai bilangan acak yang dibangkitkan kurang dari probabilitas mutasi Pm. g. Melakukan proses elitisme.
h. Melakukan pergantian populasi lama dengan generasi baru dengan cara memilih sejumlah kromosom dengan nilai fitness terbaik yang telah melalui proses seleksi, pindah silang dan elitisme.
i. Melakukan pengecekan setiap solusi yang telah didapatkan. Apabila salah satu stopping criteria belum terpenuhi, maka kembali ke langkah 5c, dan apabila salah satu stopping criteria terpenuhi, maka dilanjutkan ke langkah 5j.
j. Apabila terdapat kombinasi nilai Pc dan Pm yang belum dilakukan, maka kembali ke langkah 5c, dan apabila semua kombinasi sudah dilakukan, maka dilanjutkan pada langkah 5k.
k. Menentukan nilai parameter C dan
yang paling optimal.l. Menghitung performa klasifikasi
6. Melakukan perbandingan hasil klasifikasi metode Grid Search SVM dengan GA-SVM.
33
3.4 Diagram Penelitian
Gambar 3.1Diagram Alir Penelitian
Mulai
Microarray datasets
Mendeskripsikan data microarray
Melakukan transformasi data
Melakukan seleksi fitur dengan FCBF
Menentukan fungsi kernel
Melakukan klasifikasi menggunakan metode
Grid Search SVM
Melakukan klasifikasi menggunakan metode GA-SVM
Membandingkan metode Grid Search SVM dengan GA-SVM berdasarkan performa klasifikasi
Kesimpulan
Gambar 3.2 Diagram Alir Analisis Klasifikasi menggunakan Grid Search SVM Tidak
Mulai
Data hasil pre-processing
Menentukan range nilai parameter C dan γ
Menghitung akurasi klasifikasi Melakukan SVM dengan kombinasi nilai
parameter C dan γ
Menentukan nilai parameter C dan γ yang paling optimal
Menghitung performa klasifikasi
Selesai Melakukan semua kombinasi parameter?
35
Gambar 3.3 Diagram Alir Analisis Klasifikasi menggunakan GA-SVM
Mulai
Data hasil pre-processing
Menentukan nilai fitness,Pc, Pm, dan stopping criteria
Menentukan nilai parameter C dan γ yang paling optimal
Menghitung performa klasifikasi
Selesai Solusi memenuhi
stopping criteria?
Menyusun kromosom dan inisialisasi
Mengevaluasi kromosom berdasarkan nilai fitness
Melakukan proses seleksi
Melakukan pindah silang
Melakukan mutasi
Elitisme
Menghasilkan populasi baru
Melakukan semua kombinasi Pc dan Pm?
Tidak
Tidak
Ya
37
BAB IV
ANALISIS DAN PEMBAHASAN
Pada bab ini akan diuraikan mengenai karakteristik microarray data yang digunakan pada penelitian dan tahapan pre-processing yang dilakukan pada data tersebut. Selanjutnya akan dibahas mengenai klasifikasi microarray data menggunakan Support Vector Machine (SVM) dimana parameternya diperoleh dari metode grid search. Kemudian akan dijelaskan prosedur mendapatkan parameter SVM yang optimal menggunakan Genetic Algorithm (GA) dan menerapkan metode tersebut untuk klasifikasi pada microarray data. Setelah mendapatkan hasil klasifikasi dari metode Grid Search SVM dan GA-SVM, maka performa klasifikasi dari kedua metode tersebut dibandingkan untuk mengetahui metode yang dapat mengklasifikasikan data dengan lebih baik.
4.1 Karakteristik Data
Data yang digunakan dalam penelitian ini merupakan data high dimensional berjenis microarray. Terdapat dua data yang digunakan, yaitu Data Colon Cancer (Alon dkk., 1999) dan Data Leukemia (Golub dkk., 1999). Karakteristik data dilihat dari banyaknya pengamatan tiap kelas dan pola persebaran data dari tiap fitur dan kelas. Karakteristik data dari masing-masing data tersebut adalah sebagai berikut.
4.1.1 Karakteristik Data Colon Cancer
dalam kelas Tumor, sedangkan pengamatan yang dilakukan pada normal colon tissue dimasukkan ke dalam kelas Normal.
Gambar 4.1 Pengamatan pada Data Colon Cancer
Data Colon Cancer terdiri dari 62 pengamatan yang diperoleh dari jaringan usus manusia. Dari 62 pengamatan yang, 22 pengamatan diantaranya merupakan pengamatan yang dilakukan pada normal colon tissue yaitu kelas Normal dan 40 pengamatan lainnya merupakan kelas Tumor yang diperoleh dari tumor colon tissue. Pada penelitian ini, pengamatan yang dilakukan pada normal colon tissue merupakan kelas positif (+), sedangkan pengamatan pada tumor colon tissue merupakan kelas negatif (-).
Data Colon Cancer memiliki 2000 fitur. Fitur tersebut menggambarkan gen yang diamati pada tiap pengamatan, sehingga terdapat 2.000 ekspresi gen yang diamati. Ekspresi gen pada tiap pengamatan memiliki nilai yang berbeda-beda. Pola persebaran nilai ekspresi gen pada beberapa fitur, terdapat pada Gambar 4.2. Nilai ekspresi gen untuk pengamatan kelas Normal ditunjukkan oleh lingkaran berwarna hitam dan pengamatan Tumor ditunjukkan oleh lingkaran berwarna merah.
Gambar 4.2 menunjukkan bahwa nilai ekspresi gen pada kedua kelas yang diamati dari beberapa fitur yang terdapat pada Data Colon Cancer menyebar secara merata. Nilai ekspresi gen dari pengamatan kelas Normal dan pengamatan kelas Tumor pada tiap fitur adalah hampir sama, ditunjukkan oleh titik merah dan
22 pengamatan
40 pengamatan
Jumlah Pengamatan Normal
(Positif)
39
hitam yang terlihat hampir menyatu. Hal tersebut menunjukkan bahwa kedua kelas tidak dapat dipisahkan secara linier.
15000
Gambar 4.2 Persebaran Data dari Beberapa Fitur pada Data Colon Cancer
Dalam penelitian ini, nilai dari ekspresi gen tersebut akan digunakan untuk membentuk model SVM yang dapat mengklasifikasikan pengamatan ke dalam dua kelas, yaitu apakah pengamatan tersebut termasuk dalam kelas Tumor atau kelas Normal.
4.1.2 Karakteristik Data Leukemia
Gambar 4.3 Pengamatan pada Data Leukemia
Data Leukemia memiliki 7.129 fitur yang berisi nilai ekspresi gen. Pola nilai ekspresi gen dari beberapa fitur yang terdapat pada data Leukemia ditunjukkan melalui persebaran data pada Gambar 4.4 berikut.
0
Gambar 4.4 Persebaran Data dari Beberapa Fitur pada Data Leukemia
41
akan digunakan untuk membuat model SVM yang dapat memisahkakan data ke dalam dua kelas, yaitu ALL dan AML. 4.2 Pre-Processing Data
Sebelum melakukan klasifikasi menggunakan SVM, tahap pre-processing dilakukan pada masing-masing data. Pre-processing ini dilakukan untuk meningkatkan kualitas data yang akan dianalisis, sehingga dapat meningkatkan akurasi dan efisiensi analisis klasifikasi mengggunakan SVM. Pada penelitian ini, tahap pre-processing yang dilakukan pada masing-masing data adalah transformasi dan seleksi fitur.
4.2.1 Transformasi Data
Pada penelitian ini, transformasi yang dilakukan adalah scaling. Transformasi dilakukan secara linier pada setiap fitur, sehingga nilai pengamatan pada setiap fitur setelah ditransformasi berada pada range [0, 1]. Berikut merupakan hasil transformasi yang dilakukan pada data Colon.
Tabel 4.1 Rata-rata dan Standar Deviasi Sebelum dan Sesudah Transformasi
pada Data Colon
Rata-rata Standar Deviasi Rata-rata Standar Deviasi
1 H55933 6566 2650,840 0,374 0,213
mejadi pada range [0,1]. Setelah dilakukan transformasi, diperoleh rata-rata setiap fitur pada data Colon yang nilainya lebih kecil dibandingkan dengan rata-rata sebelum transformasi. Karena setiap nilai pengamatan berubah menjadi lebih kecil, maka standar deviasi setiap fitur pada data Colon juga menjadi lebih kecil karena data sudah ditransformasi menjadi range [0,1].
Transformasi serupa dilakukan pada data Leukemia. Hasil transformasi pada data Leukemia terdapat pada Tabel 4.2.
Tabel 4.2 Rata-rata dan Standar Deviasi Sebelum dan Sesudah Transformasi
pada Data Leukemia memiliki rata-rata dan standar deviasi yang besar. Setelah dilakukan transformasi menjadi range [0, 1], setiap fitur pada data Leukemia memiliki nilai sebaran yang lebih kecil yaitu diantara 0 sampai dengan 1.
4.2.2 Seleksi Fitur
43
dipilih tidak digunakan untuk analisis lebih lanjut. Berikut merupakan hasil seleksi fitur pada masing-masing data.
Tabel 4.3 Jumlah Fitur Sebelum dan Sesudah FCBF
Data Jumlah Fitur
Sebelum FCBF Sesudah FCBF
Colon Cancer 2.000 17
Leukemia 7.129 44
Sebelum dilakukan seleksi fitur, Data Colon ancer memiliki 2.000 fitur. Seleksi fitur dengan FCBF memilih 17 dari 2.000 fitur tersebut untuk digunakan pada analisis SVM. Pada Data Leukemia, seleksi fitur dengan FCBF mendapatkan 44 dari 7.129 fitur untuk digunakan pada analisis selanjutnya. Fitur yang digunakan untuk analisis SVM terdapat pada Lampiran 1-2. 4.3 Klasifikasi dengan Grid Search SVM
Metode pertama yang akan digunakan untuk mengklasifikasikan data adalah metode Grid Search SVM. Pada metode ini, pengaturan parameter yang membentuk model optimal diperoleh menggunakan prinsip grid search. Pada penelitian ini, metode SVM menggunakan fungsi kernel Radial Basis Function (RBF), sehingga terdapat dua parameter dalam model yang perlu ditentukan, yaitu parameter C dan γ.
4.3.1 Klasifikasi dengan Grid Search SVM pada Data Colon akan menghasilkan nilai parameter optimal dan nilai akurasi yang berbeda-beda, maka pada penelitian ini percobaan dilakukan pada data training sebanyak 10 kali untuk setiap kombinasi range parameter. Kemudian, rata-rata akurasi dihitung dari akurasi pada 10 percobaan tersebut. Berikut merupakan hasil rata-rata akurasi dari setiap kombinasi range parameter.
Tabel 4.4 Hasil Kombinasi Range Parameter pada Data Colon Cancer
(Training)
Range Parameter Rata-rata Akurasi
(%)
45
akurasi yang diperoleh dari kombinasi range parameter C dan γ lainnya. Rata-rata akurasi yang diperoleh dari 10 kali percobaan dengan menggunakan parameter C pada range 23–27 dan parameter γ pada range 2-9–2-3 adalah 92,40%.
Setelah mendapatkan range nilai parameter yang optimal dari beberapa kombinasi, selanjutnya adalah mendapatkan nilai parameter yang optimal yang berada pada range tersebut. Parameter optimal dari range tersebut diperoleh dengan membandingkan nilai akurasi yang diperoleh dari 10 percobaan yang telah dilakukan pada kombinasi parameter C pada range 23 – 27 dengan parameter γ pada range 2-9 – 2-3 sebelumnya. Akurasi diperoleh melalui 10-fold Cross-validation yang dilakukan pada data training. Nilai parameter optimal dan akurasi yang diperoleh dari 10 percobaan yang dilakukan terdapat pada tabel berikut.
Tabel 4.5 Hasil Percobaan Grid Search SVM pada Data Colon Cancer (Training) dengan range C=[23 , 27]dan γ=[2-9, 2-3]
Percobaan ke- Parameter Optimal Akurasi
( ) i i ( , )i
dimana fungsi kernel yang digunakan adalah Radial Basis Function (RBF) dengan parameter γ diperoleh sebesar 2-4, yaitu
Sehingga fungsi hyperplane yang diperoleh menjadi
2 4 2
Dengan menerapkan model SVM optimal pada data testing, maka diperoleh performa klasifikasi. Performa klasifikasi yang dihitung meliputi akurasi, sensitivitas, spesifisitas, G-mean, dan AUC. Hasil performa klasifikasi diperoleh menggunakan parameter C=27 dan parameter γ=2-4 terdapat pada tabel berikut.
Tabel 4.6 Performa Klasifikasi mengggunakan Parameter Terbaik dari Grid
47
menunjukkan nilai 73,48% dan AUC bernilai 75%. Berdasarkan nilai AUC tersebut, model SVM dapat mengklasifikasikan data dengan cukup baik. kombinasi range parameter. Kemudian dihitung rata-rata akurasi dari 10 percobaan tersebut. Hasil rata-rata akurasi dari setiap kombinasi range parameter yang dilakukan pada data training adalah sebagai berikut.
Tabel 4.7 Hasil Kombinasi Range Parameter Data Leukemia (Training)
Range Parameter Rata-rata Akurasi
(%)
menunjukkan bahwa pada kombinasi range tersebut, setiap percobaan yang dilakukan akan menghasilkan akurasi yang sama yaitu sebesar 100%, yang berarti bahwa semua pengamatan pada data training dapat diklasifikasikan dengan benar. Karena seluruh percobaan pada 12 kombinasi range paramater menghasilkan nilai akurasi yang sama, maka pada penelitian ini, ditentukan parameter optimal yang diperoleh yaitu pada range C=[2-1, 23] dan range γ=[2-3, 23]. Setelah mendapatkan range parameter yang terbaik dari beberapa kombinasi, selanjutnya adalah mendapatkan nilai parameter yang optimal diantara range tersebut. Parameter optimal dari range tersebut diperoleh dengan membandingkan nilai akurasi yang diperoleh dari 10 percobaan yang telah dilakukan pada kombinasi range parameter terbaik.
Tabel 4.8 Hasil Percobaan Grid-Search SVM pada Data Leukemia (Training)
dengan range C=[2-1, 23] dan γ=[2-3, 23]
Percobaan ke- Parameter Optimal Akurasi
(%)
49
Berdasarkan nilai parameter C dan γ yang telah diperoleh sebelumnya, fungsi hyperplane yang terbentuk untuk klasifikasi pada data Leukemia menggunakan Grid Search SVM adalah
( ) i i ( , )i
dimana fungsi kernel yang digunakan adalah Radial Basis Function (RBF) dengan parameter γ diperoleh sebesar 2-3, yaitu Sehingga fungsi hyperplane yang diperoleh menjadi
2
Performa klasifikasi meliputi akurasi, sensitivitas, spesifisitas, G-mean, dan AUC yang diperoleh menggunakan model SVM dengan parameter optimal serta dilakukan pada data testing. Hasil performa klasifikasi terdapat pada tabel berikut.
Tabel 4.9 Performa Klasifikasi menggunakan Parameter Terbaik dari Grid
Search SVM pada Data Leukemia (Testing) Ukuran Performa
4.4 Prosedur Optimasi Parameter SVM dengan Genetic Algorithm
Setelah menentukan parameter SVM optimal menggunakan Grid Search, selanjutnya adalah menentukan parameter SVM yang optimal menggunakan Genetic Algorithm (GA). Penggunaan GA dimaksudkan untuk mendapatkan parameter SVM yang akan menghasilkan akurasi lebih tinggi. GA-SVM ini akan menggunakan range nilai parameter terbaik yang diperoleh dari hasil Grid Search SVM untuk mendapatkan nilai awal parameter. Langkah awal yang dilakukan adalah melakukan inisialisasi kromosom sebanyak 100. Kromosom yang dibangkitkan memiliki dua gen yang menunjukkan dua parameter SVM, yaitu C dan γ. Nilai dari parameter C dan γ berada pada range nilai parameter terbaik yang diperoleh dari hasil Grid Search SVM.
Misalkan nilai parameter C berada pada range 1,5-2,5 dan
nilai parameter γ pada range 0,1-0,5, maka ilustrasi kromosom
dengan dua gen adalah sebagai berikut.
Parameter C γ
Kromosom 2 0,12500
Gambar 4.5 Ilustrasi Satu Buah Kromosom dengan Dua Gen
Gambar 4.5 menunjukkan ilustrasi satu buah kromosom dengan dua gen, yaitu parameter C dan γ. Kromosom yang terbentuk tersebut akan menjalani proses GA, meliputi seleksi, pindah silang, mutasi, dan elitisme sehingga diperoleh parameter yang akan menghasilkan nilai akurasi tinggi.
51
Tabel 4.10 Ilustrasi Nilai Fitness tiap Kromosom
Kromosom
Proses seleksi yang dilakukan pada penelitian ini menggunakan metode seleksi roulette wheel. Seleksi roulette wheel merupakan salah satu metode untuk menentukan kromosom orang tua yang dapat bertahan untuk generasi selanjutnya atau menentukan suatu populasi dari populasi yang ada saat ini untuk digunakan pada pindah silang. Kromosom yang bertahan untuk generasi selanjutnya dipilih dengan melibatkan nilai fitness pada kromosom tersebut. Apabila *
i
f merupakan nilai
fitness pada kromosom ke-i, maka peluang kromosom terpilih yang disebut dengan fitness relatif dihitung dengan
*
dimana N adalah banyaknya kromosom dalam 1 populasi (ukuran populasi). Metode seleksi roulette wheel memilih kromosom dengan peluang kromosom terpilih sebanding dengan nilai fitnessnya. Semakin besar fitness suatu kromosom, maka semakin besar pula peluang kromosom tersebut terpilih (Gambar 4.6).