IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH|
Hae
III) PADA SAPI
FRIESIAN HOLSTEIN DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
SKRIPSI
ANNISA OKTAVIA RINI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
ANNISA OKTAVIA RINI. D14070118. 2011. Identifikasi Keragaman Gen
Growth Hormone Releasing Hormone (GHRH|HaeIII) Pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang. Skripsi. Mayor Ilmu Produksi dan Teknologi Peternakan. Institut Pertanian Bogor
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D.
Salah satu usaha yang dapat dilakukan untuk mendapatkan ternak yang memiliki produktivitas tinggi adalah dengan perbaikan genetik melalui seleksi. Seleksi perlu mengetahui karakteristik genetik ternak yang berpengaruh terhadap sifat kuantitatif, meliputi sifat pertumbuhan. Sifat pertumbuhan dikontrol oleh banyak gen dan sebagian besar aksinya bersifat aditif. Gen GHRH merupakan hormon yang menstimulasi sintesis dan sekresi Growth Hormone. Gen GHRH dapat mempengaruhi produktivitas dan meningkatkan pertumbuhan ternak. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen GHRH pada sapi perah Friesian Holstein (FH) di BIB Lembang, BBIB Singosari, dan BET Cipelang; serta sapi pedaging di BET Cipelang sebagai pembanding.
Sampel darah sapi yang digunakan berjumlah 126 sampel, yakni 49 ekor sapi FH jantan berasal dari BIB Lembang (17 ekor), BBIB Singosari (32 ekor), dan sapi FH betina berasal dari BET Cipelang (40 ekor); serta sejumlah 37 ekor sapi pedaging betina terdiri dari Simmental (13 ekor), Limousin (14 ekor), Brahman (5 ekor), dan Angus (5 ekor) dari BET Cipelang. Penelitian ini menggunakan metode PCR-RFLP dengan menggunakan enzim restriksi HaeIII. Analisis keragaman menggunakan frekuensi genotipe dan alel, keseimbangan Hardy-Weinberg, dan heterozigositas. Enzim HaeIII memotong sekuen gen GHRH pada posisi GG | CC di posisi 118, 312, dan 406 pb dan menghasilkan empat fragmen masing-masing dengan panjang 118, 194, 94, dan 45 pb.
Genotyping gen GHRH menghasilkan dua jenis alel yaitu alel A dan B. Kedua alel tersebut menghasilkan tiga jenis genotipe, yaitu genotipe AA (312, 94, dan 45 pb), AB (312, 194, 118, 94, dan 45 pb), dan BB (194, 118, 94, dan 45 pb). Frekuensi alel B adalah lebih tinggi dibanding alel A. Frekuensi alel A dan B di BIB Lembang adalah 0,235 dan 0,765, BBIB Singosari 0,234 dan 0,766, dan BET Cipelang 0,225 dan 0,775. Analisis Chi-Kuadrat menunjukkan bahwa sapi perah FH dan sapi pedaging yang diamati dalam keseimbangan Hardy-Weinberg (χ2 < χ2(0,05)), kecuali pada sapi FH di BIB Lembang, sapi Brahman dan Angus di BET Cipelang. Nilai heterozigositas tertinggi sapi FH ditemukan di BIB Lembang sebesar 0,471, sebaliknya terendah pada FH di BBIB Singosari yaitu 0,344. Nilai heterozigositas pengamatan pada sapi FH tidak jauh berbeda dengan nilai heterozigositas harapan. Nilai heterozigositas sapi FH lebih rendah daripada sapi Simmental dan sapi Limousin. Adanya keragaman gen GHRH|HaeIII dapat dijadikan pertimbangan untuk informasi dasar kegiatan seleksi pada sifat pertumbuhan dari sapi FH dan sapi pedaging.
ABSTRACT
Identification of The Polymorphismof The Growth Hormone Releasing Hormone (GHRH|HaeIII) Gene in Holstein-Friesian Cattle in
BIB Lembang, BBIB Singosari, and BET Cipelang Rini, A.O., C. Sumantri, and A. Anggraeni
This study was aimed to identify polymorphism of the GHRH gene in Holstein Friesian (FH) cattle from BIB Lembang, BBIB Singosari, and BET Cipelang; and beef cattle from BET Cipelang for a comparison. Blood samples used were 126 samples, consisting of 49 HF bulls from BIB Lembang (17 hds), BBIB Singosari (32 hds), and HF cows from BET Cipelang (40 hds); and a number of 37 hds of beef cattle consisting of Simmental (13 hds), Limousin (14 hds), Brahman (5 hds), and Angus (5 hds) from BET Cipelang. This study used PCR-RFLP method using restriction enzyme HaeIII. This enzyme restricted the GHRH gene sequences at the bases GG | CC at the positions of 118, 312, and 406 bp resulting four fragments with the lengths of 118, 194, 94, and 45 bp respectively. Genotyping GHRH gene produced two alleles, namely A and B alleles. These alleles produced three genotypes, namely the genotypes of AA (312, 94, and 45 bp), AB (312, 194, 118, 94, and 45 bp), and BB (194, 118, 94, and 45 bp). Frequency of the B allele was higher than the A allele. Frequencies of the A and B alleles in HF were in BIB Lembang 0,235 and 0,765, BBIB Singosari 0,234 and 0,766, and BET Cipelang 0,225 and 0,775. Chi-square analysis showed that all of HF and beef cattles were in Hardy-Weinberg equilibrium (χ2 < χ2(0.05)), the exception in HF cattles in BIB Lembang, Brahman and Angus cattles in BET Cipelang. The highest heterozigosity values in HF was found in BIB Lembang (0,471), while the lowest was in BBIB Singosari (0,344). Heterozygosity values in the HF were lower than those of Simmental and Limousin beef cattle. GHRH gene in HF and beef cattle were polimorfic, the exception in Brahman cattle as possesing only the B allele.
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH|
Hae
III) PADA SAPI
FRIESIAN HOLSTEIN DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
ANNISA OKTAVIA RINI D14070118
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
Judul : Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH|HaeIII) Pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang
Nama : Annisa Oktavia Rini NIM : D14070118
Menyetujui,
Pembimbing Utama, Pembimbing Anggota,
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) (Ir. Anneke Anggraeni, M.Si, Ph.D.) NIP. 19591212 198603 1 004 NIP. 19630924 199803 2 001
Mengetahui, Ketua Departemen
Ilmu Produksi dan Teknologi Peternakan
(Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc.) NIP. 19591212 198603 1 004
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 14 Oktober 1989 di Jakarta. Penulis adalah anak kedua dari dua bersaudara dari pasangan bapak H. Adi Suwardi (alm.) dan Hj. Sulfanah Ma’ani. Kakak kandung penulis bernama Andhika Fandrian Apriyandi, S.T.
Pendidikan Taman Kanak-kanak diselesaikan pada tahun 1994 di TK Gembira, Bekasi. Pendidikan dasar diselesaikan pada tahun 2001 di SDN Cipinang Melayu 03 Pagi Jakarta Timur. Pendidikan lanjutan menengah pertama diselesaikan pada tahun 2004 di SLTP N 109 Jakarta Timur, dan pendidikan lanjutan menengah atas diselesaikan pada tahun 2007 di SMA N 71 Jakarta Timur.
Penulis diterima sebagai mahasiswa pada Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan Institut Pertanian Bogor melalui jalur
Undangan Seleksi Masuk IPB (USMI) pada tahun 2007. Selama mengikuti pendidikan, penulis aktif di organisasi Himpunan Mahasiswa Ilmu Produksi Peternakan (HIMAPROTER), pada periode 2008-2009 menjabat sebagai staf club Ruminansia dan pada periode 2009-2010 menjabat sebagai ketua divisi Keprofesian. Selain itu penulis juga pernah menjadi anggota Animal Breeding and Genetic Student Community (ABGSCi) periode 2010-2011
KATA PENGANTAR
Puji dan sukur senantiasa saya panjatkan kepada Allah SWT, karena atas segala rahmat dan karunia-Nya, penelitian dan penulisan skripsi ini dapat terselesaikan. Skripsi yang berjudul Identifikasi Keragaman Gen Growth Hormone Releasing Hormone (GHRH|HaeIII) Pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang merupakan salah satu syarat untuk memperoleh gelar sarjana pada Fakultas Peternakan Institut Pertanian Bogor.
Beberapa upaya dapat dilakukan untuk meningkatkan produktivitas sapi diantaranya dengan perbaikan pakan, manajemen pemeliharaan, dan perbaikan genetik. Perbaikan genetik dapat dilakukan dengan cara seleksi. Seleksi secara konvensional dilakukan berdasarkan performa ternak. Dengan adanya kemajuan dibidang teknologi molekuler, memungkinkan seleksi dilakukan pada level DNA.
Seleksi pada level DNA dilakukan utamanya pada gen-gen yang berkaitan dengan sifat ekonomis. Gen GHRH adalah gen yang berhubungan dengan ekspresi hormon pertumbuhan yang akan mempengaruhi sifat pertumbuhan dan bobot karkas pada ternak pedaging. Gen GHRH juga berhubungan dengan produksi susu dan lemak susu pada ternak perah.
Tujuan dari penelitian ini adalah untuk mengidentifikasi keragaman gen GHRH menggunakan enzim restriksi HaeIII (GHRH|HaeIII) dengan metode PCR-RFLP pada sapi Friesian Holstein (FH) di beberapa UPT Ditjen Peternakan dan sapi pedaging sebagai pembanding. Penulis berharap semoga skripsi ini memiliki manfaat dan memberikan sumbangan bagi kemajuan peternakan Indonesia. Amin.
Bogor, 11 April 2011
DAFTAR ISI
RINGKASAN ... i
ABSTRAK ... ii
LEMBAR PERNYATAAN ... iii
LEMBAR PENGESAHAN ... iv
RIWAYAT HIDUP ... v
KATA PENGANTAR ... vi
DAFTAR ISI ... vii
DAFTAR TABEL ... viii
DAFTAR GAMBAR ... ix
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 2
TINJAUAN PUSTAKA ... 3
Sapi Friesian Holstein ... 3
Sapi Pedaging ... 4
Hormon Pertumbuhan ... 4
Metode PCR-RFLP ... 5
Keragaman Genetik ... 6
Keragaman Gen Growth Hormone Releasing Hormone (GHRH) .... 7
MATERI DAN METODE ... 10
Lokasi dan Waktu ... 10
Materi ... 10
Penanganan dan Pengambilan Sampel ... 10
Ekstraksi DNA ... 11
Polymerase Chain Reaction (PCR) ... 11
PCR-RFLP ... 11
Elektroforesis ... 11
Prosedur ... 12
Pengambilan Sampel Darah ... 12
Ekstraksi DNA ... 12
Amplifikasi Ruas Gen GHRH ... 13
Analisis PCR-RFLP ... 13
Elektroforesis DNA ... 13
Pendeteksian Keragaman DNA ... 13
Analisis Data ... 14
Frekuensi Genotipe dan Alel ... 14
Keseimbangan Hardy-Weinberg (HW) ... 14
Heterozigositas ... 15
9
HASIL DAN PEMBAHASAN ... 16
Amplifikasi Gen GHRH ... 16
Pendeteksian Keragaman Gen GHRH dengan Metode PCR-RFLP .. 17
Frekuensi Genotipe dan Alel Gen GHRH ... 19
Keseimbangan Hardy-Weinberg ... 22
Heterozigositas ... 24
KESIMPULAN DAN SARAN ... 26
Kesimpulan ... 26
Saran ... 26
UCAPAN TERIMA KASIH ... 27
DAFTAR PUSTAKA ... 28
LAMPIRAN ... 32
DAFTAR TABEL
Nomor Halaman
1. Keragaman Gen GHRH Menurut Beberapa Penelitian pada
Beberapa Jenis Ternak ... 9
2. Sampel Darah Sapi Perah dan Sapi Pedaging yang Digunakan ... 10
3. Frekuensi Genotipe dan Alel dari Gen GHRH ... 20
4. Keseimbangan Hardy-Weinberg Gen GHRH ... 23
2
DAFTAR GAMBAR
Nomor Halaman
1. Sapi Friesian Holstein, Jenis Sapi Perah yang Banyak
Dibudidayakan Peternak di Indonesia ... 4 2. Rekonstruksi Sruktur Gen GHRH Berdasarkan Sekuen Gen GHRH di
GenBank, Nomor Akses AF242855 ... 8
3. Visualisasi Hasil Amplifikasi Ruas Gen GHRH pada Gel
Agarosa 1,5% ... 16 4. Posisi Primer, Produk PCR dan Perbedaan Sekuen Gen GHRH
(Nomor Akses GenBank AF242855) dan Pemotongan Enzim Restriksi HaeIII ... 17
5. Hasil Pemotongan Fragmen Gen GHRH Menggunakan Metode
PCR-RFLP dengan Enzim Pemotong HaeIII pada Gel Agarosa 2% .. 18 6. Keragaman Gen GHRH|HaeIII pada Sapi Friesian Holstein
PENDAHULUAN Latar Belakang
Usaha peternakan sapi perah di Indonesia umumnya berupa peternakan rakyat dengan skala usaha kecil atau sebagai usaha sampingan. Masalah yang dihadapi berupa rendahnya produksi susu dibandingkan terhadap jumlah permintaan. Konsumsi susu tahun 2008 sebesar 6,92 kg per kapita per tahun (Badan Pusat Statistik, 2009). Produksi susu dalam negeri hanya mampu memenuhi 30% kebutuhan (Pusat Penelitian dan Pengembangan Peternakan, 2009). Usaha yang dapat dilakukan untuk meningkatkan produksi susu dalam negeri antara lain melalui peningkatan populasi dan produktivitas ternak.
Upaya meningkatkan produktivitas sapi perah diantaranya ditempuh dengan perbaikan pakan, manajemen pemeliharaan, perbaikan genetik. Seleksi secara konvensional dilakukan berdasarkan performa ternak. Kemajuan dibidang teknologi molekuler memungkinkan seleksi dilakukan pada level DNA. Seleksi pada level DNA dilakukan pada gen-gen yang berkaitan dengan sifat ekonomis, seperti produksi susu dan lemak susu, maupun pertumbuhan dan bobot karkas.
Dalam menerapkan program seleksi, berbagai penciri DNA telah banyak tersedia untuk digunakan. Penggunaan penciri DNA dalam program seleksi sering
disebut sebagai Marker Assisted Selection (MAS) yang diharapkan mampu meningkatkan respons seleksi yang jauh lebih baik daripada tanpa menggunakan penciri DNA. Penggunakan penciri DNA memudahkan identifikasi keragaman genetik dalam populasi ternak sehingga akan memberikan hasil seleksi yang efektif.
Growth Hormone Releasing Hormone (GHRH) merupakan hormon yang menstimulasi sintesis dan sekresi Growth Hormone. Hormon GHRH mempengaruhi pertumbuhan, meningkatkan produktivitas pada ternak perah, seperti meningkatkan produksi susu dan lemak susu; serta pada ternak pedaging, seperti meningkatkan pertumbuhan dan bobot karkas. Gen GHRH merupakan penciri genetik sekaligus gen major yang dapat dipertimbangkan untuk dipakai sebagai gen potensial (marker) dalam melakukan seleksi sifat pertumbuhan dan produksi susu ternak sapi perah.
2
Balai BIB Lembang dan BBIB Singosari sebagai salah satu unit yang melaksanakan replacement pejantan unggul dan produsen bibit unggul secara berkesinambungan melalui penyediaan semen beku, BET Cipelang sebagai unit yang mampu melakukan produksi, penyimpanan dan pendistribusian embrio ternak serta aplikasi transfer embrio ternak.
Tujuan
TINJAUAN PUSTAKA Sapi Friesian Holstein
Sapi memiliki peran utama dalam evolusi kebudayaan manusia dan penting dalam segi ekonomi. Semua ternak sapi saat ini diperkirakan telah di domestikasi dari Bos primigenius yang sekarang sudah punah. Domestikasi sapi pertama kali ditemukan di Turki (Perkins, 1969) dengan dua spesies utama ternak sapi di dunia, yaitu tipe berpunuk (Zebu) dan tidak berpunuk (Taurin). Tipe berpunuk termasuk dalam spesies Bos indicus yang tersebar di wilayah Asia dan Afrika Selatan, sedangkan tipe tidak berpunuk (Taurine) termasuk dalam spesies Bos taurus yang tersebar di wilayah Eropa dan Afrika Barat. Kedua spesies ini kemungkinan besar didomestikasi secara bebas (Loftus et al., 1994) sekitar 10.000 tahun sebelum sekarang.
Pada umumnya sapi perah yang dipelihara di Indonesia adalah jenis sapi Friesian Holstein (FH) yang termasuk keturunan dari sapi B. taurus. Bangsa sapi Friesian Holstein murni memiliki warna bulu hitam dan putih (black Holstein) atau merah dan putih (red Holstein) dengan batas-batas warna yang jelas, seperti pada dahi umumnya terdapat warna putih berbentuk segitiga dan bulu kipas ekor, bagian perut serta kaki dari teracak sampai lutut (knee) atau (hock) berwarna putih, memiliki
4
Gambar 1. Sapi Friesian Holstein, Jenis Sapi Perah yang Banyak Dibudidayakan Peternak di Indonesia (Sumber: Pusat Penelitian dan Pengembangan Peternakan, 2009)
Sapi Pedaging
Tipe sapi pedaging atau sapi potong memiliki keunggulan dalam menghasilkan karkas berkualitas dan tingkat pertumbuhan yang tinggi. Beberapa bangsa sapi dari spesies B. taurus yaitu sapi Limousin dan Simmental. Sapi Limousin memiliki perdagingan yang bagus dengan laju pertumbuhan yang tinggi (Phillips, 2001), dengan bobot badan sapi betina normalnya adalah 600 kg dan bobot sapi jantan mencapai 1000 kg. Bangsa sapi Simmental memiliki karakter berat sapih dan pertambahan berat badan pasca sapih yang tinggi (Williamson dan Payne, 1993). Sapi yang termasuk dalam spesies B. indicus, seperti sapi Brahman, memiliki ciri khas yaitu berpunuk di bagian punggungnya, berambut pendek dan halus, serta sebagian besar berwarna putih. Spesies B. indicus memiliki kemampuan beradaptasi dengan lingkungan panas dan tahan terhadap penyakit caplak (Phillips, 2001). Penghambat perkembangan industri sapi potong antara lain terbatasnya sapi lokal sehingga belum siap mengisi kebutuhan bakalan industri peternakan (feedlotter) dan beroperasinya rumah pemotongan hewan (RPH) tradisional dan ilegal di hampir seluruh wilayah Indonesia (Williamson dan Payne, 1993).
Hormon Pertumbuhan
Lawrence dan Fowler (2002) menyatakan bahwa pertumbuhan merupakan suatu proses deposisi, pemindahan substansi sel-sel, serta peningkatan ukuran dan jumlah sel pada tingkat dan titik berbeda dalam suatu waktu tertentu. Pertumbuhan
dengan hormon Insulin-Like Growth Factor-1 (IGF-1) berperan sangat penting dalam mengatur pertumbuhan kelenjar susu dan produksi susu, metabolisme, laktasi, dan komposisi tubuh.
Proses sintesis dan pelepasan hormon pertumbuhan atau somatotropin dikontrol oleh dua macam hormon yang terdapat di hipotalamus, yaitu Growth Hormone Releasing Hormone (GHRH) yang berfungsi sebagai penggertak (stimulator) dan Somatotropin Releasing-Inhibitory Factor (SRIF) atau somatostatin sebagai penghambat (inhibitor) (Anderson et al., 2004). Kedua hormon tersebut disekresikan oleh neuron sekretoris dalam hipotalamus dan masuk ke dalam pembuluh darah pituitari (Hartman, 2000). Neurotransmiter dan neuropeptida mengontrol sekresi somatotropin secara langsung pada bagian somatotrop atau secara tidak langsung melalui jalur hipotalamus (Franklin dan Ferry, 2006).
Metode PCR-RFLP (Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism)
PCR merupakan suatu teknik untuk menggandakan jumlah molekul DNA pada ruas-ruas tertentu dan monomer-monomer nukleotida yang dilakukan secara in vitro. Proses ini berjalan dengan bantuan primer dan enzim polymerase. Primer merupakan oligonukleotida spesifik yang menempel pada bagian sampel DNA yang akan diperbanyak. Enzim polymerase merupakan enzim yang dapat mencetak urutan DNA baru. Hasil dari proses PCR dapat divisualisasikan dengan elektroforesis (Williams, 2005). Muladno (2002) menyatakan bahwa Polymerase Chain Reaction (PCR) merupakan suatu reaksi in vitro untuk menggandakan jumlah molekul DNA pada target tertentu dengan cara mensintesis molekul DNA baru yang berkomplemen dengan molekul DNA target tersebut dengan bantuan enzim polimerase dan oligonukleotida pendek sebagai primer dalam suatu mesin thermocycler. Secara umum, reaksi yang terjadi dalam mesin PCR dapat dibagi menjadi tiga tahap, yaitu tahap denaturasi (pemisahan untai ganda DNA), tahap annealing (penempelan primer), dan tahap extension (pemanjangan primer).
6
menyatakan bahwa enzim pemotong yang dapat mengenal sekuens DNA spesifik disebut recognition sequences dan biasanya memiliki panjang empat sekuens basa atau lebih dan bersifat palindrome.
PCR-RFLP merupakan suatu metode yang sederhana dan biasa digunakan untuk mencari keragaman genotipe (Yahyaoi et al., 2001). Penyisipan (insersi), penghilangan (delesi), maupun subtitusi nukleotida yang terjadi pada daerah rekognisi suatu enzim restriksi menyebabkannya tidak lagi dikenalinya situs pemotongan enzim restriksi dan terjadinya menyebabkannya perbedaan pola pemotongan DNA (Lewin, 1994). Teknik RFLP yang dikombinasikan dengan teknik PCR telah secara luas digunakan untuk mendapatkan variasi pada setiap daerah atau lokasi DNA, baik pada daerah yang bersifat penyandi (coding region) pada genom maupun pada daerah yang tidak penyandi atau daerah non-coding (Vasconcellos et al., 2003).
Meghen et al. (1995) menyatakan bahwa jumlah dan ukuran fragmen DNA yang dihasilkan oleh enzim pemotong memiliki pola pita ada atau tidaknya tempat restriksi. Apabila tidak terpotong ada indikasi terjadi mutasi pada situs tersebut sehingga tidak ada variasi hasil pemotongan dan ekspresinya bersifat kodominan. Nei dan Kumar (2000) menyatakan bahwa atas dasar terpotong atau tidaknya fragmen DNA dengan enzim pemotong, hasil fragmen potongan DNA tersebut dapat
divisualisasi melalui teknik elektroforesis yang hasilnya menunjukkan ada tidaknya polimorfisme pada suatu individu dalam populasi.
Keragaman Genetik
(Noor, 2008). Selain itu silang dalam dan silang luar juga dapat mempengaruhi frekuensi genotipe. Keragaman genetik dapat digunakan sebagai parameter dalam mempelajari genetika populasi dan genetika evolusi. Tingkat keragaman dalam populasi dapat digambarkan dari frekuensi alel. Frekuensi alel merupakan rasio relatif suatu alel terhadap keseluruhan alel yang ditemukan dalam satu populasi (Nei dan Kumar, 2000).
Derajat heterozigositas, menurut Nei (1987), merupakan rataan persentase lokus heterozigositas tiap individu atau rataan persentase individu heterozigot dalam populasi. Suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0,99. Hartl dan Clark (1997) menyatakan bahwa polimorfisme genetik dalam suatu populasi dapat digunakan dalam menentukan hubungan antar subpopulasi yang terfragmentasi dalam suatu spesies. Perhitungan keragaman genetik dalam populasi secara kuantitatif dapat diperoleh melalui dua ukuran keragaman variasi populasi yaitu proporsi lokus polimorfisme dalam populasi dan rata-rata proporsi individu heterozigot dalam setiap lokus (Nei dan Kumar, 2000). Keragaman genetik antara subpopulasi dapat diketahui dengan melihat persamaan dan perbedaan frekuensi alel di antara subpopulasi (Li et al., 2000).
Javanmard et al. (2005) menyatakan bahwa nilai heterozigositas di bawah 0,5 (50%) mengindikasikan rendahnya variasi suatu gen dalam populasi dan jika nilai Ho
(heterozigositas pengamatan) lebih rendah dari He (heterozigositas harapan) maka dapat mengindikasikan adanya proses seleksi yang intensif (Tambasco et al., 2003). Avise (1994) juga menyatakan bahwa semakin tinggi derajat heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan semakin tinggi. Seiring dengan menurunnya derajat heterozigositas akibat dari silang dalam dan fragmentasi populasi, sebagian besar alel resesif yang bersifat lethal semakin meningkat frekuensinya.
Keragaman Gen Growth Hormone Releasing Hormone (GHRH)
8
hipotalamus. Berdasarkan fungsi protein, somatoliberin sama seperti glukagon, sekretin, dan VIP (Vasoactive Intestinal Peptide).
Beberapa penelitian telah dilakukan pada ternak sapi dan diketahui bahwa somatotropin, somatoliberin dan sejenis sintesisnya meningkatkan produksi baik pada ternak perah yaitu meningkatkan produksi dan lemak susu (Bonneau dan Laarveld, 1999) maupun ternak pedaging (Achtung et al., 2001) yang dapat meningkatkan laju pertumbuhan, sehingga mempersingkat waktu yang dibutuhkan untuk mencapai bobot potong. Cheong et al. (2006) menambahkan berdasarkan hasil penelitiannya bahwa gen GHRH merupakan salah satu penanda genetik untuk produksi daging.
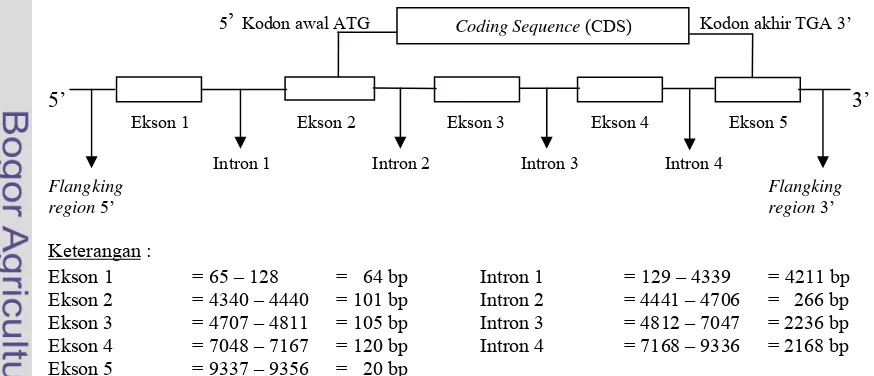
Gen adalah bagian segmen DNA termasuk semua nukleotida yang ditranskripsi ke dalam mRNA yang akan ditranslasi menjadi protein (Nicholas, 1996; Brown, 1999; Muladno, 2002). Gen GHRH pada sapi terletak pada kromosom nomor 13 (Barendse et al., 1994) terdiri dari lima ekson dan empat intron (Gambar 2). Bagian gen yang mengkode asam amino dan menghasilkan protein disebut daerah penyandi (coding sequence) (CDS). Selain itu terdapat pula bagian segmen depan (leader segment) dan segmen belakang (trailer segment) yang mengapit daerah CDS. Beberapa gen pada eukaryot bersifat tidak kontinyu karena adanya ekson (pengkode protein) dan intron (space internal antara pengkode protein). Pada saat transkripsi,
bagian intron hilang (splicing), sehingga proses translasi berjalan baik (Brown, 1999).
5’ Kodon awal ATG Kodon akhir TGA 3’
5’ 3’
Ekson 1 Ekson 2 Ekson 3 Ekson 4 Ekson 5
Intron 1 Intron 2 Intron 3 Intron 4
Flangking Flangking
region 5’ region 3’
Keterangan :
Ekson 1 = 65 – 128 = 64 bp Intron 1 = 129 – 4339 = 4211 bp
Ekson 2 = 4340 – 4440 = 101 bp Intron 2 = 4441 – 4706 = 266 bp
Ekson 3 = 4707 – 4811 = 105 bp Intron 3 = 4812 – 7047 = 2236 bp
Ekson 4 = 7048 – 7167 = 120 bp Intron 4 = 7168 – 9336 = 2168 bp
Ekson 5 = 9337 – 9356 = 20 bp
Gambar 2. Rekonstruksi Struktur Gen GHRH Berdasarkan Sekuens Gen GHRH di GenBank, Nomor Akses AF242855
Panjang gen GHRH pada sapi adalah 9356 pb (pasang basa). Moody et al. (1995) melaporkan adanya keragaman gen GHRH pada sapi pedaging dengan metode PCR-RFLP menggunakan primer GHRH forward
5’-GTAAGGATGC(C/T)(A/G)CTCTGGGT-3’ dan GHRH reverse
5’TGCCTGCTCATGATGTCCTGGA-3’; serta enzim restriksi HaeIII yang menghasilkan dua buah alel yaitu 317, 83, 55 pb (alel A) dan 196, 121, 83, 55 pb (alel B). Keragaman ditemukan pada exon ketiga gen GHRH pada sapi (no. Akses GenBank U29611). Ruas fragmen untuk gen GHRH|HaeIII pada sapi Polish Black and White menurut Dybus dan Grzesiak (2006) terletak menutupi bagian ekson 2, seluruh intron 2, dan sebagian dari ekson 3; analisis keragaman terletak di intron 2 (no. Akses GenBank AF242855), menghasilkan tiga genotipe, yaitu AA (242 dan 55 pb), genotipe AB (242, 194, 55, dan 48 pb), dan genotipe BB (194, 55, dan 48 pb). Kmiéc et al. (2007) melakukan penelitian terhadap keragaman gen GHRH|HaeIII dan hubungannya dengan sifat produksi susu pada sapi Polish Red-and-White (salah satu varietas bangsa Friesian Holstein). Produk PCR teramplifikasi sepanjang 297 pb, menghasilkan tiga genotipe, yaitu BB (194, 55, dan 48 pb), AB (242, 194, 55, dan 48 pb) dan AA (242, 55, dan 48 pb). Menghasilkan frekuensi genotipe AA sebesar 9,6%, genotipe AB sebesar 37% dan genotipe BB sebesar 53,4%. Frekuensi alel A sebesar 28,1% dan frekuensi alel B sebesar 71,9%.
Tabel 1. Keragaman Gen GHRH Menurut Beberapa Penelitian pada Beberapa Jenis Ternak
Ternak Jumlah Alel Posisi Marker Metode Sumber
Sapi Perah dan Pedaging Sapi Pedaging Sapi Perah Sapi Pedaging Sapi Perah Babi Babi 2 2 2 12 tipe 2 2 2 Intron 1 Intron 2 Intron 2 5’UTR dan intron Intron 2 Ekson 3 Ekson 3 RFLP RFLP RFLP Analisis sekuen RFLP RFLP RFLP
Moody et al. (1995)
Dybus et al. (2003) Dybus dan Grzesiak
(2006) Cheong et al. (2006)
Kmiec et al. (2007) Pierzchala et al. (2003)
MATERI DAN METODE Lokasi dan Waktu
Penelitian ini dilaksanakan di Laboratorium Genetika Molekuler Ternak (LGMT), Bagian Pemuliaan dan Genetika, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian dilaksanakan pada bulan Agustus hingga Nopember 2010.
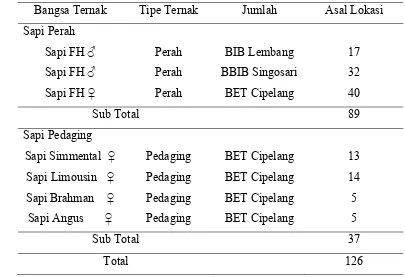
Materi
Sampel darah yang digunakan sebanyak 126 ekor sapi yang terdiri atas 89 ekor sapi FH dan 37 ekor sapi pedaging dengan bangsa yang berbeda sebagai pembanding (Tabel 2). Sampel darah yang digunakan merupakan koleksi sampel Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika Ternak, Fakultas Peternakan, Institut Pertanian Bogor.
Tabel 2. Sampel Darah Sapi Perah dan Sapi Pedaging yang Digunakan
Bangsa Ternak Tipe Ternak Jumlah Asal Lokasi
Sapi Perah
Sapi FH ♂ Perah BIB Lembang 17
Sapi FH ♂ Perah BBIB Singosari 32
Sapi FH ♀ Perah BET Cipelang 40
Sub Total 89
Sapi Pedaging
Sapi Simmental ♀ Pedaging BET Cipelang 13 Sapi Limousin ♀ Pedaging BET Cipelang 14 Sapi Brahman ♀ Pedaging BET Cipelang 5
Sapi Angus ♀ Pedaging BET Cipelang 5
Sub Total 37
Total 126
Keterangan : ♂= Jantan dan ♀ = Betina
Penanganan dan Pengambilan Sampel
Ekstraksi DNA
Bahan yang digunakan yaitu sampel darah 200 µl, Destilata Water 1000 µl, 40 µl SDS 10% (Sodium Docecyl Sulfat), 10 µl Proteinase K 5 mg/ml, 350 µl 1 x STE (Sodium Tris EDTA), 400 µl larutan phenol, 400 µl CIAA (Chloroform Isoamil Alkohol), 40 µl NaCl 5M, 800 µl etanol absolut dan etanol 70%, dan 100 µl TE 80% atau Elution buffer. Alat yang digunakan antara lain kotak penyimpan sampel, rak tabung ependorf, tabung ependorf 1,5 ml, mikro pipet (200-1000 µl, 20-100 µl, 2-20 µl) beserta tip (warna biru dan kuning), alat mikrosentrifugasi berpendingin, vortex mixer, sarung tangan, autoclave, inkubator, freezer, rotary mixer, gunting dan pinset steril, dan alat tulis.
Polymerase Chain Reaction (PCR)
Reaksi PCR dilakukan pada mesin thermocycler menggunakan enzim taq polymerase dan buffernya. Primer gen GHRH yang digunakan dalam penelitian ini adalah mengikuti Moody et al. (1995) yang telah dimodifikasi dengan runutan primer GHRH forward 5’-TGA AGG ATG CTG CTC TGG GT-3’ dan GHRH reverse 5’TGC CTG TTC ATG ATA TCC TGG A-3’ (Nomor akses GenBank AF242855). Panjang daerah target yang akan diamplifikasi adalah 451 pb dan berada di daerah sebagian ekson 2, intron 2, dan sebagian ekson 3.
Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism
Bahan-bahan yang digunakan untuk Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism (PCR-RFLP) adalah produk PCR fragmen gen GHRH, destilated water, enzim pemotong HaeIII serta buffer R. Peralatan yang digunakan dalam analisis PCR-RFLP antara lain microtube PCR, mikropipet 10 P, 20 P, dan 200 P Gilson beserta tip nya, dan inkubator 37 ˚C.
Elektroforesis
12
Prosedur Pengambilan Sampel Darah
Sampel darah diambil melalui vena jugularis di leher sapi menggunakan jarum vennoject dan tabung vaccutainer tanpa heparin. Sampel darah ditambahkan etanol absolute (1:2) dan disimpan pada suhu ruang. Sampel yang digunakan merupakan koleksi Laboratorium Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika, Fakultas Peternakan, Institut Pertanian Bogor.
Ekstraksi DNA
Ekstraksi DNA dilakukan dari darah sapi. Prosedur ekstraksi mengikuti metode phenol-chloroform (Sambrook et al., 1989):
Preparasi Sampel. Sampel dari darah dalam alkohol sebanyak 200 µl dimasukkan ke dalam tabung 1,5 ml. Alkohol dihilangkan dari sampel dengan menambahkan air destilasi sampai 1000 µl, dan dibiarkan selama 20 menit. Sampel kemudian diendapkan dengan sentrifugasi pada kecepatan 8000 rpm selama lima menit, supernatan dibuang.
Degradasi Protein. Sampel yang telah bersih dari alkohol ditambahkan 1xSTE (sodium tris EDTA) sampai volume 350 µl, 40 µl SDS 10% dan 10 µl proteinase K 5 mg/ml. Campuran diinkubasi pada suhu 55 ˚C selama dua jam digoyang pelan.
Degradasi Bahan Organik. Ditambahkan 400 µl larutan phenol, 400 µl chloroform:isoamyl alcohol (24:1) dan 40 µl NaCl 5M. Campuran digoyang pada suhu ruang selama satu jam.
Amplifikasi Ruas Gen GHRH
Amplifikasi ruas gen GHRH dilakukan dengan metode PCR (Polymerase Chain Reaction). Pereaksi yang digunakan untuk amplifikasi gen GHRH merupakan campuran yang terdiri atas sampel DNA 1 µl, destilated water 9,7 µl, primer (forward 5’-TGA AGG ATG CTG CTC TGG GT-3’ dan GHRH reverse 5’TGC CTG TTC ATG ATA TCC TGG A-3’) 0,1 μl, Taq polymerase 0,05 µl, buffer 1,25 µl, dNTP 0,1 µl, dan MgCl2 0,25 µl. Campuran tersebut kemudian diinkubasi dalam mesin thermocycler dengan kodisi suhu pradenaturasi 94 °C selama lima menit, denaturasi 94 °C selama 45 detik, anneling 62 °C selama 45 detik, ekstensi 72 °C selama satu menit, dan ekstensi akhir 72 °C selama lima menit. Siklus PCR yang digunakan sebanyak 35 siklus.
Analisis PCR-RFLP
Enzim pemotong yang digunakan untuk ruas gen target adalah HaeIII. Produk PCR lima µl dipindahkan ke tabung 0,5 ml, ditambahkan destilated water 1 µl, enzim HaeIII 0,3 µl serta buffer R 0,7 µl, dan diinkubasi pada suhu 37 °C selama 16 jam.
Elektroforesis DNA
Penentuan genotipe masing-masing individu dilakukan dengan pendekatan Restriction Fragment Lenght Polymorphism (RFLP) yang divisualisasikan pada gel
agarosa 2% dengan TBE 0,5 x (Tris Borat EDTA) pada 100 v selama 40 menit. Gel diwarnai dengan 2,5 µl bromida yang divisualisasikan pada UV transiluminator.
Pendeteksian Keragaman DNA
14
terdapatnya empat fragmen yaitu 194, 118, 94, dan 45 pb. Genotipe AB ditunjukkan dengan lima fragmen dengan panjang 312, 194, 118, 94, dan 45 pb. Genotipe AA dan BB merupakan homozigot, sedangkan genotipe AB heterozigot (kombinasi).
Analisis Data Frekuensi Genotipe dan Alel
Frekuensi genotipe merupakan rasio dari jumlah suatu genotipe terhadap jumlah populasi. Keragaman genotipe pada masing-masing individu ternak dapat ditentukan melalui pita-pita DNA yang ditemukan. Frekuensi genotipe dapat diketahui dengan menghitung perbandingan jumlah genotipe tertentu pada setiap populasi, dengan rumus Nei dan Kumar (2000) sebagai berikut :
Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada suatu lokus dalam populasi. Frekuensi alel ( ) gen GHRH|HaeIII dapat dihitung
berdasarkan rumus Nei dan Kumar (2000), sebagai berikut :
∑
Keterangan :
= frekuensi genotipe ke-ii = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii nij = jumlah individu bergenotipe ij N = jumlah individu sampel
Keseimbangan Hardy-Weinberg (HW)
Keseimbangan Hardy-Weinberg diuji dengan menggunakan perhitungan Chi-Kuadrat (Hartl dan Clark, 1997) :
Keterangan:
χ2 = chi-kuadrat
Heterozigositas
Keragaman genetik dapat diketahui melalui estimasi frekuensi heterozigositas pengamatan yang diperoleh dari masing-masing lokasi, dengan menggunakan rumus Weir (1996) sebagai berikut :
Keterangan :
Ho = heterozigositas pengamatan nij = jumlah individu heterozigot N = jumlah individu yang diamati
Heterozigositas harapan (He) berdasarkan frekuensi alel dihitung menggunakan rumus Nei dan Kumar (2000) sebagai berikut :
Keterangan :
He = nilai heterozigositas harapan = frekuensi alel
q = jumlah alel
400 pb
100 pb
451 pb
HASIL DAN PEMBAHASANAmplifikasi Gen GHRH
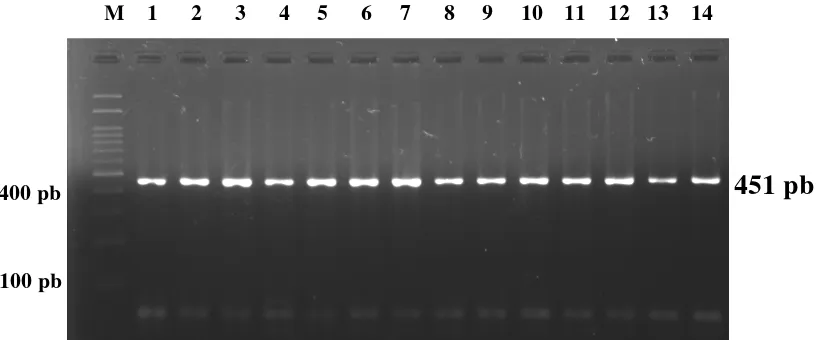
Ruas gen GHRH pada sapi FH dari lokasi BIB Lembang, BBIB Singosari, dan BET Cipelang diamplifikasi menggunakan metode PCR (Polymerase Chain Reaction). Pasangan primer yang digunakan mengikuti Moody et al. (1995) yang telah dimodifikasi. Amplifikasi ruas gen GHRH dilakukan pada mesin thermocycler dengan suhu annealing 62 ˚C. Suhu annealing adalah suhu yang memungkinkan terjadinya penempelan primer pada DNA cetakan selama proses PCR. Primer merupakan bahan yang sangat menentukan pada proses spesifikasi PCR. Pada usaha untuk mengamplifikasi DNA sekuen spesifik, sekuen dari situs penempelan primer pada DNA target harus diketahui (Verkuil et al., 2008). Suhu annealing sangat menentukan keberhasilan amplifikasi karena proses perpanjangan DNA dimulai dari primer. Persentase keberhasilan amplifikasi gen GHRH dalam penelitian ini adalah 100%. Hasil amplifikasi ruas gen yang divisualisasikan pada gel agarosa 1,5% ditampilkan pada Gambar 3.
Panjang produk hasil PCR dari ruas gen GHRH sapi berukuran 451 pb (pasang basa), yang terletak di sebagian ekson 2, intron 2, dan sebagian ekson 3. Moody et al. (1995) yang melakukan penelitian menggunakan sapi Hereford dan
Angus, menemukan adanya keragaman gen GHRH pada intron 2. Selanjutnya Dybus M 1 2 3 4 5 6 7 8 9 10 11 12 13 14
Keterangan: M= Marker; Nomor 1-14= Ruas gen GHRH
dan Grzesiak (2006) menyatakan adanya pengaruh nyata dari keragaman tersebut terhadap protein susu dan kadar lemak pada sapi perah Polandia. Posisi penempelan pasangan primer pada sekuen gen GHRH ditampilkan pada Gambar 4.
Keterangan: Garis bawah menunjukkan primer; Cetak tebal menunjukkan situs pemotongan; Cetak tebal bergaris bawah menunjukkan situs mutasi; Alel A Mempunyai Basa A pada Posisi Basa ke- 4474; Alel B Mempunyai Basa C pada Posisi Basa ke- 4474
Gambar 4. Posisi Primer, Produk PCR dan Perbedaan Sekuen Gen GHRH (Nomor Akses GenBank AF242855) dan Pemotongan Enzim Restriksi HaeIII
Pendeteksian Keragaman Gen GHRH dengan Metode PCR-RFLP
Pendeteksian keragaman pada ruas gen GHRH dilakukan melalui metode RFLP menggunakan enzim pemotong HaeIII (GHRH|HaeIII). Enzim HaeIII mengenali situs pemotongan GG|CC. Berdasarkan sekuen DNA ruas gen GHRH yang diamplifikasi ditemukan tiga titik potong enzim restriksi HaeIII, yaitu pada nukleotida ke-4472, 4666, dan 4760 atau pada posisi basa ke 118, 312, dan 406 dari produk PCR. Pemotongan tersebut menghasilkan empat fragmen yang panjangnya adalah 118, 194, 94, dan 45 pb. Keempat pita tersebut menunjukkan alel B. Apabila terdapat perubahan basa pada posisi ke-4474, yaitu dari basa C (Citosin) menjadi A (Adenin) menyebabkan perubahan fragmen (pita) hasil potongan HaeIII yang dikenal dengan alel A (Gambar 4).
Keragaman ditemukan pada daerah intron 2. Intron merupakan space internal
antara pengkode protein dan saat proses transkripasi akan hilang (splicing) (Brown,
Alel A
5’
-GCAAGAGA
GGCA
CTTTGA-3’
Alel B
5’
-GCAAGAGA
GGCC
CTTTGA-3’
Forward
tgaagg atgctgctct gggtgttctt
4381 cctcgtgacc ctcaccctca gcagcggctc ccacggttcc ctgccttccc agcctctcag
4441 gtaagcagtt ctgagaagag aagcaagaga gg|ccctttga ggatgcgact cgagctggtc
4501 cccagctggg tcctcaggca gcctcccttg ctcatctctg ggagggtggc agactgagcc
4561 ccagagaggt caccacccag ccctggttcc agccctctct ggggacgagc agggcaagag
4621 gcgacagaaa gacctcacag agaccaagtg agcacagtcc cctggg|cctc ccaccccacc
4681 ctttgacctc tgactccttc tactaggatt ccacggtacg cagatgccat cttcactaac
4741 agctaccgga aggttctggg|ccagctgtct gcccgcaagc tactccagga tatcatgaac
18
100 pb 200 pb 300 pb 400 pb
45 pb
94 pb
118 pb
194 pb
312 pb
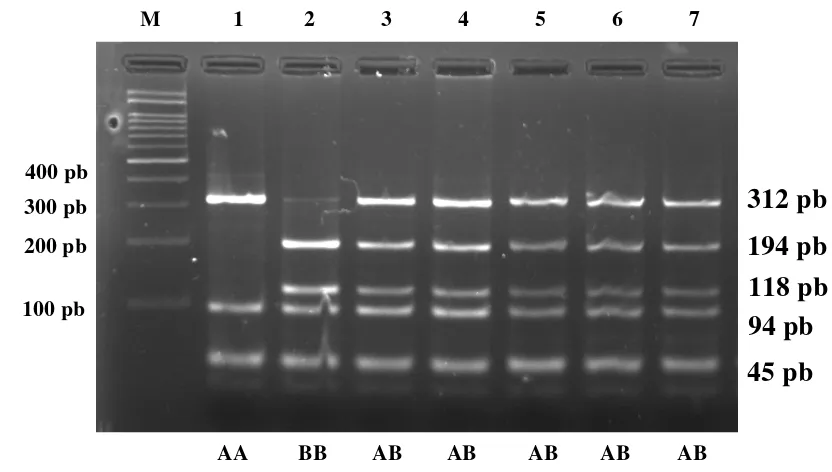
1999). Berdasarkan tipe mutasi yang terdapat pada penciri, mutasi tersebut termasuk dalam tipe mutasi subtitusi, yaitu tipe mutasi transversi pada penciri PCR-RFLP fragmen gen GHRH|HaeIII. Sebagaimana dinyatakan Li dan Graur (1991), mutasi tipe transversi yaitu pertukaran atau subtitusi antara basa purin (A = Adenin, G = Guanin) dengan basa pirimidin (C = Citosin, T = Timin).Berdasarkan hasil analisis menggunakan PCR-RFLP terhadap ruas gen GHRH|HaeIII bersifat polimorfik. Terdapat tiga genotipe, yaitu genotipe AA, AB, dan BB yang berasal dari dua alel, yaitu alel A dan alel B. Nei (1987) menyatakan bahwa suatu alel dikatakan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0,99.
Keterangan: M= Marker; 1-7= Sampel; AA, AB, BB= Genotipe
Gambar 5. Hasil Pemotongan Fragmen Gen GHRH Menggunakan Metode PCR-RFLP dengan Enzim Pemotong HaeIII pada Gel Agarosa 2%
Tiga varian genotipe ditemukan pada sapi FH jantan dan betina serta sapi pedaging betina dari penelitian ini yaitu AA, BB, dan AB (Gambar 5). Ternak sapi dikatakan memiliki genotipe AA apabila mempunyai tiga pita DNA dengan panjang 312, 94, dan 45 pb. Genotipe BB memiliki empat pita dengan panjang 194, 118, 94,
dan 45 pb. Oleh karena itu, genotipe AB memiliki lima pita dengan panjang 312, 194, 118, 94, dan 45 pb. Ternak sapi dengan genotipe homozigot yaitu AA dan BB menunjukkan bahwa kedua tetua menyumbangkan gen (alel) yang sama, sedangkan
[image:30.595.85.492.264.581.2]
sapi dengan genotipe heterozigot AB menunjukkan ternak tersebut memiliki kombinasi gen yang berbeda dari kedua tetuanya. Identifikasi keragaman gen GHRH pada sapi telah dilakukan dengan metode PCR-RFLP menggunakan enzim restriksi HaeIII oleh Moody et al. (1995) yang melaporkan adanya dua jenis alel, yaitu alel A dan alel B dengan tiga jenis genotipe, yaitu AA, AB dan BB. Keragaman gen GHRH|HaeIII pada sapi FH serta sapi pedaging Simmental, Limousin, Brahman, dan Angus ditunjukkan dengan jumlah genotipe yang muncul dari masing-masing bangsa (Gambar 6).
Keterangan : Sapi FH = BIB Lembang (♂), BBIB Singosari (♂), dan BET Cipelang (♀); Sapi Pedaging BET Cipelang (♀) = Simental, Limousin, Angus, dan Brahman
Gambar 6. Keragaman Gen GHRH|HaeIII pada Sapi Friesian Holstein dan Sapi Pedaging.
Frekuensi Genotipe dan Alel Gen GHRH
Frekuensi genotipe merupakan rasio dari jumlah suatu genotipe terhadap jumlah populasi. Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada suatu lokus dalam populasi dan merupakan parameter dasar dalam mempelajari proses terjadinya evolusi, karena perubahan genetik pada sebuah populasi biasanya
0 5 10 15 20 25
FH BIB FH BBIB FH BET Simmental Limousin Brahman Angus
0
2 2
1 1
0
3 8
11
14
7
9
0
2 9
19
24
5
4
5
0
Jumlah Sapi
(ekor)
Bangsa Sapi
AA
AB
20
[image:31.595.74.523.182.491.2]
digambarkan dengan adanya perubahan pada frekuensi alel (Nei and Kumar, 2000). Frekuensi genotipe dan alel gen GHRH sapi FH dan pedaging ditampilkan pada Tabel 3.
Tabel 3. Frekuensi Genotipe dan Alel dari Gen GHRH
Bangsa
(ekor)* Lokasi
Genotipe Alel
AA AB BB A B
Sapi Perah FH ♂
(17)
BIB Lembang 0,000 (0)
0,471 (8)
0,529
(9) 0,235 0,765 FH ♂
(32)
BBIB Singosari 0,063 (2)
0,344 (11)
0,594
(19) 0,234 0,766 FH ♀
(40)
BET Cipelang 0,050 (2)
0,350 (14)
0,600
(24) 0,225 0,775
Sub Total (89) 0,045 (4)
0,371 (33)
0,584
(52) 0,230 0,770 Sapi Pedaging
Simmental ♀ (13)
BET Cipelang 0,077 (1)
0,538 (7)
0,385
(5) 0,346 0,654 Limousin ♀
(14)
BET Cipelang 0,071 (1)
0,643 (9)
0,286
(4) 0,393 0,607 Brahman ♀
(5)
BET Cipelang 0,000 (0)
0,000 (0)
1,000
(5) 0,000 1,000 Angus ♀
(5)
BET Cipelang 0,600 (3)
0,4000 (2)
0,000
(0) 0,800 0,200
Sub Total (37) 0,135 (5)
0,486 (18)
0,378
(14) 0,378 0,622
Keterangan: (...)* = Jumlah sampel; ♂= Jantan dan ♀ = Betina
Berdasarkan hasil analisis keragaman menggunakan frekuensi genotipe, ditemukan bahwa frekuensi genotipe BB (0,584) pada sapi perah di BIB Lembang, BBIB Singosari, dan BET Cipelang lebih tinggi dibanding dengan frekuensi genotipe AB (0,371) dan frekuensi genotipe AA (0,045). Pada sapi pedaging betina di BET Cipelang ditemukan frekuensi genotipe AB (0,486) yang lebih tinggi dibandingkan frekuensi genotipe AA (0,135) dan BB (0,378). Sapi pedaging Brahman memiliki frekuensi genotipe BB sangat tinggi (1,000). Frekuensi genotipe AB pada sapi pedaging Simmental (0,538) dan sapi Limousin (0,643) lebih tinggi dibanding genotipe AA dan BB. Pada sapi Angus, genotipe AA (0,600) lebih tinggi dibandingkan genotipe lainnya.
(0,230). Frekuensi alel A dan B di BIB Lembang, BBIB Singosari, dan BET Cipelang berturut-turut adalah 0,235 dan 0,765; 0,234 dan 0,766; serta 0,225 dan 0,775. Pada sapi pedaging di BET Cipelang, sapi Simmental memiliki frekuensi alel A (0,346) dan alel B (0,654), sapi Limousin memiliki frekuensi alel A (0,393) dan alel B (0,607), dan sapi Brahman memiliki frekuensi alel A (0,000) dan alel B (1,000). Sapi Brahman bersifat monomorfik karena ditemukan hanya satu tipe alel, yaitu alel B, hal tersebut kemungkinan disebabkan oleh manajemen perkawinan yang tidak acak, seleksi terhadap sifat tertentu dan tingkat silang dalam yang tinggi (Bourdon, 2000). Namun, perbedaan ditemukan pada sapi Angus karena memiliki frekuensi alel A (0,800) yang lebih tinggi dibanding alel B (0,200). Moody et al. (1995) menemukan bahwa frekuensi alel A sebesar 0,700 pada sapi Angus.
Dybus dan Grzesiak (2006) dalam penelitiannya tentang evaluasi keragaman gen GHRH dengan produksi susu pada sapi perah Polandia, ditemukan tiga genotipe dan dua alel, dengan genotipe AA (0,0545), AB (0,3133) dan BB (0,6322), serta alel A (0,2111) dan alel B (0,7889). Sapi yang memiliki alel A (genotipe AA dan AB) dapat menghasilkan lebih banyak lemak dalam susunya. Szatkowska et al. (2009) pada penelitiannya menggunakan sapi perah Polish Holstein dan Jersey menemukan frekuensi genotipe dan alel pada sapi Polish Holstein berturut-turut adalah genotipe AA (0,078), AB (0,339), BB (0,583); serta alel A (0,248) dan alel B (0,752). Sapi
22
Frekuensi genotipe AA pada sapi pedaging jarang ditemukan dibanding genotipe AB dan BB. Moody et al. (1995) melaporkan bahwa genotipe AA jarang ditemukan pada gen GHRH. Dybus et al. (2003) melaporkan bahwa sapi Limousin yang dilahirkan dengan genotipe homozigot AA lebih rendah (p≤0,01) daripada yang dilahirkan dengan genotipe AB dan genotipe BB dengan frekuensi genotipe dan alel masing-masing adalah AA (0,0154), AB (0,1692), BB (0,8154), serta alel A (0,1) dan alel B (0,9). Secara statistik terdapat perbedaan yang signifikan (P ≤ 0,01) antara individu-individu dari genotipe gen GHRH yang berbeda. Pada umur 210 hari, sapi Limousin dengan genotipe AA memiliki ketinggian sakrum (cm) dan tinggi pundak (cm) lebih pendek (-8,14 dan -8,33 cm) daripada individu bergenotipe AB dan BB. Curi et al. (2005) dalam penelitiannya tentang keragaman gen GHRH|HaeIII pada sapi dengan komposisi 50% darah Simmental, dilaporkan memiliki genotipe AA (0,000), AB (0,633), dan BB (0,367).
Menurut Cheong et al. (2006) Gen GHRH merupakan salah satu penanda genetik untuk produksi daging. Hasil yang diperoleh dalam penelitiannya menunjukkan terdapat hubungan yang nyata antara keragaman genotipe dari gen GHRH dengan sifat karkas yang meliputi persentase bobot karkas dan perototan bagian longisimus pada ternak pedaging asli Korea (Hanwoo). Hasil tersebut menunjukkan bahwa keragaman gen GHRH dapat menjadi faktor penting yang
berpengaruh terhadap produksi karkas pada ternak pedaging.
Pierzchala et al. (2003) melaporkan keragaman gen GHRH dengan enzim restriksi AluI dan hubungannya dengan laju pertumbuhan dan kualitas karkas pada ternak babi. Keragaman gen GHRH|AluI menghasilkan frekuensi genotipe AA (8,4%), AB (29,8%), dan BB (61,8%). Ketebalan lemak pundak berhubungan dengan genotipe AA. Ternak babi dengan genotipe AB menunjukkan berat lemak babi yang lebih rendah tetapi lebih tinggi kandungan dagingnya dibandingkan genotipe AA.
Keseimbangan Hardy-Weinberg
[image:34.595.88.505.158.409.2]
derajat bebas tertentu (Nei, 1987). Keseimbangan Hardy-Weinberg gen GHRH yang diuji dengan Chi-kuadrat (χ2) ditampilkan pada Tabel 4.
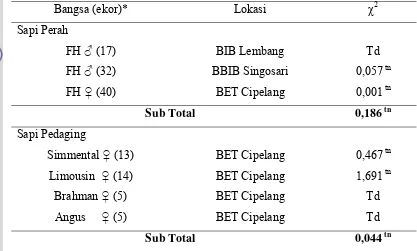
Tabel 4. Keseimbangan Hardy-Weinberg Gen GHRH
Bangsa (ekor)* Lokasi χ2
Sapi Perah
FH ♂ (17) BIB Lembang Td
FH ♂ (32) BBIB Singosari 0,057 tn
FH ♀ (40) BET Cipelang 0,001 tn
Sub Total 0,186 tn
Sapi Pedaging
Simmental ♀ (13) BET Cipelang 0,467 tn Limousin ♀ (14) BET Cipelang 1,691 tn
Brahman ♀ (5) BET Cipelang Td
Angus ♀ (5) BET Cipelang Td
Sub Total 0,044 tn
Keterangan: (...)* = Jumlah sampel; tn = tidak nyata pada taraf α = 0,05; Td = tidak dapat dihitung; χ2
(0,05) = 3,84; ♂= Jantan dan ♀ = Betina
Analisis Chi-Kuadrat pada Tabel 4 menunjukkan bahwa sapi FH jantan di BBIB Singosari dan sapi FH betina di BET Cipelang bersifat tidak nyata atau berada pada keseimbangan Hardy-Weinberg. Sapi pedaging betina bangsa Simmental dan Limousin berada pada keseimbangan Hardy-Weinberg karena nilai Chi-kuadrat χ2
< χ2(0,05). Suatu populasi dinyatakan dalam keadaan keseimbangan Hardy-Weinberg, jika frekuensi genotipe dan frekuensi alel konstan dari generasi ke generasi, sebagai akibat penggabungan gamet yang terjadi secara acak ke dalam populasi yang besar (Nei, 1987). Hukum Hardy-Weinberg menyatakan frekuensi genotipe suatu populasi yang cukup besar akan selalu dalam keadaan seimbang bila tidak ada seleksi, migrasi, mutasi dan genetic drift (Noor, 2008), namun keseimbangan Hardy-Weinberg pada bangsa sapi FH jantan di BIB Lembang, sapi Brahman dan sapi Angus betina di BET Cipelang tidak dapat dihitung karena tidak memenuhi syarat pengujian Hardy-Weinberg.
24
GHRH|HaeIII. Dybus dan Grzesiak (2006) menemukan tidak adanya hubungan yang signifikan antara keragaman GHRH|HaeIII dengan sifat produksi susu dari sapi perah Polandia. Penelitian pada sapi perah Polandia tidak menunjukkan efek dari genotipe terhadap sifat produksi susu, masih tetap tidak jelas bahwa analisis keragaman dapat mempengaruhi fisiologi produksi lemak susu sapi perah (Szatkowska et al., 2009).
Heterozigositas
[image:35.595.81.504.415.663.2]Pendugaan nilai heterozigositas memiliki arti penting untuk diketahui, yaitu untuk mendapatkan gambaran variabilitas genetik (Marson et al., 2005). Heterozigositas disebut juga sebagai keragaman genetik. Nilai heterozigositas merupakan cara yang paling akurat untuk mengukur keragaman genetik suatu populasi (Nei, 1987). Nilai heterozigositas dipengaruhi oleh jumlah sampel, jumlah alel dan frekuensi alel. Hasil analisis heterozigositas sapi FH pada lokasi yang berbeda dan sapi pedaging ditampilkan pada Tabel 5.
Tabel 5. Nilai Heterozigositas Pengamatan (Ho) dan Harapan (He) Gen GHRH
Bangsa (ekor)* Lokasi Ho He
Sapi Perah
FH ♂ (17) BIB Lembang 0,471 0,360
FH ♂ (32) BBIB Singosari 0,344 0,359
FH ♀ (40) BET Cipelang 0,350 0,349
Sub Total 0,371 0,355
Sapi Pedaging
Simmental ♀ (13) BET Cipelang 0,538 0,453 Limousin ♀ (14) BET Cipelang 0,643 0,477 Brahman ♀ (5) BET Cipelang 0,000 0,000 Angus ♀ (5) BET Cipelang 0,400 0,320
Sub Total 0,486 0,470
Keterangan: (...)* = Jumlah sampel; ♂= Jantan dan ♀ = Betina
Nilai heterozigositas yang diobservasi lebih besar daripada nilai heterozigositas harapan kecuali pada sapi FH jantan di BBIB Singosari. Nilai heterozigositas pengamatan yang lebih rendah dari harapan kemungkinan dikarenakan adanya seleksi yang intensif terhadap sifat pertumbuhan di BBIB Singosari. Nilai heterozigositas tertinggi sebesar 0,471 terdapat pada bangsa sapi FH jantan di BIB Lembang. Nilai heterozigositas pengamatan pada sapi pedaging betina berkisar antara 0,000-0,643. Heterozigositas terendah pada bangsa sapi pedaging betina terdapat pada bangsa Brahman karena tidak menunjukkan adanya variasi genotipe (0,000), sebaliknya heterozigositas tertinggi terdapat pada bangsa sapi Limousin sebesar 0,643. Tingginya nilai heterozigositas ini menunjukan tingkat keragaman gen GHRH|HaeIII dalam populasi tersebut adalah tinggi. Nilai heterozigositas dipengaruhi oleh jumlah sampel, jumlah alel dan frekuensi alel (Nei, 1987). Hartl dan Clark (1997) menambahkan bahwa nilai heterozigositas harapan juga dapat digunakan sebagai salah satu cara untuk menduga nilai koefisien biak dalam (inbreeding) pada suatu kelompok ternak.
Berdasarkan hasil analisis, nilai Ho dan He pada sapi FH jantan dan betina tidak berada pada perbedaan nilai yang terlalu besar (rataan Ho= 0,371 dan He=0,355). Nilai Ho dan He pada sapi pedaging juga tidak menunjukkan perbedaan nilai yang besar (rataan Ho=0,486 dan He= 0,470), namun nilai Ho pada sapi
KESIMPULAN DAN SARAN Kesimpulan
Gen GHRH|HaeIII pada sapi FH bersifat polimorfik dengan dua alel, yaitu alel A dan B dan tiga genotipe, yaitu AA, AB, dan BB. Frekuensi alel B ditemukan lebih tinggi dari alel A. Frekuensi tertinggi genotipe AA ditemukan pada sapi FH di BBIB Singosari, genotipe AB pada sapi FH di BIB Lembang, dan genotipe BB pada sapi FH di BET Cipelang. Analisis Chi-Kuadrat menunjukkan bahwa sapi FH berada pada keseimbangan Hardy-Weinberg, dengan tingkat keragaman gen GHRH|HaeIII rendah (0,344-0,471). Gen GHRH|HaeIII pada sapi pedaging di BET Cipelang bersifat polimorfik kecuali pada sapi Brahman bersifat monomorfik dengan alel B (1,00). Frekuensi alel A tertinggi ditemukan pada sapi Angus, sedangkan frekuensi alel B tertinggi ditemukan pada sapi Brahman. Analisis Chi-Kuadrat menunjukkan
bahwa seluruh sapi pedaging berada dalam keseimbangan Hardy-Weinberg dengan tingkat keragaman gen GHRH|HaeIII tinggi pada sapi Simmental dan Limousin (0,538 dan 0,643), sebaliknya sapi Brahman dan Angus memiliki tingkat keragaman gen GHRH|HaeIII rendah (0,000 dan 0,400).
Saran
UCAPAN TERIMA KASIH
Puji dan syukur penulis panjatkan kepada Allah SWT, atas berkat rahmat dan karunianya sehingga penulis bisa menyelesaikan penulisan skripsi ini. Kepada Papa tersayang H. Adi Suwardi (alm.), Mama tercinta Hj. Sulfanah Ma’ani yang senantiasa memberikan kasih sayang, dukungan, dan doa untuk kesuksesan penulis, dan Kakakku tersayang Andhika Fandrian Apriyandi atas perhatian yang diberikan.
Penulis mengucapkan banyak terima kasih kepada Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc sebagai pembimbing utama dan Ir. Anneke Anggraeni, M.Si, Ph.D sebagai pembimbing anggota atas segala bimbingan, arahan dan motivasi bagi penulis dalam penelitian dan penyusunan skripsi ini. Semoga Allah SWT memberikan balasan terbaik atas segala pengorbanan, waktu dan tenaga serta ilmu pengetahuan yang diberikan. Kepada dosen pembahas seminar Ir. Rini Herlina Mulyono, M.Si serta dosen penguji sidang Dr. Jakaria, S.Pt, M.Si, Dr. Despal, S.Pt, M.Si, dan Muhammad Baihaqi, S.Pt, M.Sc atas masukan yang diberikan. Kepada Dr. Ir. Moh. Yamin, M.Agr.Sc sebagai pembimbing akademik yang selalu memberikan kekuatan dan motivasi yang membangun.
Penulis mengucapkan terima kasih kepada Ka’Eryk, Mba’Restu, Mba’Mira, Ka’Surya, Bpk. Ihsan, Bpk. Andi, dan Ibu Suriana yang senantiasa memberikan
informasi terkait penulisan skripsi ini. Kepada tim penelitian (Diny, Wike, Tiffany, dan Gabby) dan anggota laboratorium GMT (Ulin, Irine, Priskilla, Ferdy, Revy, Lenny, Rahmah, Erwin, Desy, dan Gina). Kepada Diny W, Gilang A, Anis, Maya R, dan Bariah atas keakraban yang terjalin, warga IPTP 44, dan Basriansyah atas doa dan keceriaan yang diberikan. Terima kasih kepada rekan-rekan asisten TPTDU (Mba’Devi, Mba’Dewi, Walfitri, Wulan, Rahmah, Rani, dan Desti), asisten PKTT, rekan-rekan PKM (Tantia, Leli, Dodi, dan Bayu), rekan-rekan Istiqomah Farm (Gilang A, Edys, Kuswanto, dan Hassan), Ka Dudi, Ka Tito, Arzia R.C, Dyhart, Resty, Kartika, Putri, dan warga Kos MEGA I atas dukungan yang diberikan. Penulis juga tidak lupa menyampaikan terima kasih kepada Muhamad Wiriawan atas perhatian dan semangat yang diberikan. Semoga karya ilmiah ini bermanfaat bagi dunia peternakan Indonesia. Amin.
DAFTAR PUSTAKA
Achtung, T. L., D. S. Buchanan, C. A. Lents, S. M. Barao, & G. E. Dahl. 2001. Growth hormone response to growth hormone-releasing hormone in beef cows divergently selected for milk production. J. Anim. Sci. 79: 1295-1300.
Anderson, L. L., S. Jeftinija & C. G. Scanes. 2004. Growth hormone secretion: molecular and cellular mechanism and in vivo approaches. Exp. Biol. Med. 229: 291–302.
Avise, J. C. 1994. Molecular Markers, Natural History and Evolution. Chapman and Hall, Inc., New York.
Badan Pusat Statistik. 2009. Statistik Indonesia. Badan Pusat Statistik Indonesia, Jakarta.
Beker, A. R. C. L., F. Izadyar, B. Colenbrander, & M. M. Bevers. 2000. Effect of growth hormone releasing hormone (GHRH) and Vasoactive Intestinal Peptide (VIP) on in vitro bovine oocyte maturation. Theriogenology. 53: 1771-1782.
Barendse, W., S. M. Armitage, L. M. Kosarek, A. Shalom, B. W. Kirkpatrick, A. M. Ryan, D. Clayton, L. Li, H. L. Neibergs, N. Zhang, W. M. Grosse, J. Weiss, P. Creighton, F. McCarthy, M. Ron, A. J. Teale, R. Fries, R. A. McGraw, S. S. Moore, M. Georges, M. Soller, J. E. Womack, & D. J. S. Hetzel. 1994. A genetic linkage map of the bovine genome. Nat. Genet. 6: 227-235.
Blakely, J. & D. H. Bade. 1998. Ilmu Peternakan. 4th ed. Terjemahan: Bambang Srigandono. Gadjah Mada University Press, Yogyakarta.
Blott, S. C., J. L. Williams, & C. S. Haley. 1998. Genetic relationship among European cattle breeds. Anim. Genet. 29: 273-282.
Bonneau, M. & B. Laarveld. 1999. Biotechnology in animal nutrition, physiology and health. Lives. Prod. Sci. 59: 223-241.
Bourdon, R. M. 2000. Understanding Animal Breeding. 2nd ed. Prentice-Hall, Inc. Upper Saddle River, New Jersey.
Brown, T. A. 1999. Genomes. Garland Science Publishing, New York.
Cheong, H. S., D. H. Yoon, L. H. Kim, B. L. Park, Y. H. Choi, E. R. Chung, Y. M Cho, E. W. Park, I. C. Cheong, S. J. Oh, S. G. Yi, T. Park, & H. D. Shin. 2006. Growth hormone-releasing hormone (GHRH) polymorphisms associated with carcass traits of meat in Korean cattle. J. BioMed. Cent. 7: 1471-2156.
Connor, E. E., M. S. Ashwell, & G. E. Dahl. 2002. Characterization and expression of the bovine growth hormone-releasing hormone (GHRH) receptor. Dom. Anim. Endoc. 22: 189-200.
Curi, R. A,. H. N. de Oliveira, A. C. Silveira, & C. R. Lopes. 2005. Association between IGF-1, IGF-IR, and GHRH gene polymorphisms and growth and carcass traits in beef cattle. Lives. Prod. Sci. 94: 159-167.
Dwiyanto, K., Anggraeni, A., Sugiarti, Nurhasanah, Setyanto, H., & Praharani, L. 2000. Pengkajian sistem budidaya sapi perah untuk meningkatkan produktivitas. Prosiding Hasil Penelitian Bagian Proyek Rekayasa Teknologi Peternakan/ARMP-II. Pusat Penelitian dan Pengembangan Peternakan. Badan Penelitian dan Pengembangan Pertanian. Departemen Pertanian, Bogor.
Dybus, A., M. Kmiéc, Z. Sobek, W. Pietrzyk, & B. Wiśniewski. 2003. Associations between polymorphisms growth hormone releasing hormone (GHRH) and pituitary transcription factor 1 (PIT1) genes and production traits of Limousine cattle. Arch. Tierz., Dummerstorf 46: 527-534.
Dybus, A. & W. Grzesiak. 2006. GHRH/HaeIII gene polymorphism and its associations with milk production traits in Polish Black-and-White cattle. Arch. Tierz., Dummerstorf 49: 434-438.
Franco, M. M., R. C. Antunes, H. D. Silva, & L. R. Goulart. 2005. Association of PIT1, GH and GHRH polymorphisms with performance and carcass traits in Landrance pigs. J. Appl. Genet. 46: 195-200.
Franklin, S. & R. J. Ferry. 2006. Hyposomatotropism. Departement of Pediatrics. Division of Endocrinology. Pediatric Endocrinology of San Diego Medical Group.
Gillespie, J. H. 1998. Population Genetics, A Concies Guide. The Johns Hopkins University Press, London.
Hartl, D. L & A. G. Clark. 1997. Principle of Population Genetic. Sinauer Associates, Sunderland.
Hartman, M. L. 2000. Physiological Regulations of Growth Hormone Secretion. 2nd ed. Cambridge University Press, UK.
Javanmard, A., N. Asadazadeh., M. H. Banabazi., & J. Tavakolian. 2005. The allele and genotype frequencies of bovine pituitary specific transcription factor and leptin genes in Iranian cattle and buffalo populations using PCR-RFLP. Irianian J. of Biotechnol. 3: 104-108.
30
Lawrence, T. L. J. & Fowler, V. R. 2002. Growth of Farm Animals. 2nd ed. CABI Publishing, New York.
Lewin, B. 1994. Genes V. Oxford University Press, New York.
Li, W. H & D. Graur. 1991. Fundamentals of Molecular Evolution. Sinauer Associates Inc. Publishers, Sunderland.
Li, X., F. B. Li, Y. Gong, S. Zhao, Z. Peng & B. Liu. 2000. The genetic diversity of seven pig breeds in China, evaluated by means of microsatellites. Asian-Aus. J. Anim. Sci. 13: 100-102.
Loftus, R. T., D. E. MacHugh, D. G. Bradley, P. M. Sharp, & P. Cunningham. 1994. Evidence for two independent domestications of cattle. Proc. Natl. Acad. Sci. USA 91: 2757-2761.
Marson, E. P., J. B. S. Ferraz, F. V. Meirelles, J. C. D. C. Balieiro, J. P. Eler, L. G. G. Figueiredo, & G. B. Mourau. 2005. Genetic characterization of European Zebu composite bovine using RFLP markers. Genet. Mol. Res. 4: 496-505.
Meghen, C., D. E. MacHugh, & D. G. Bradley. 1995. Genetic characterization and west African cattle. Departement of Genetic. Trinity College. Dublin, Ireland.
Montaldo, H. H. & C. A. M. Herrera. 1998. Use of molecular markers and major genes in the genetic improvement of livestock. J. of Biotech. 1: 2.
Moioli B., F. Napolitano, & G. Catillo. 2004. Genetic diversity between Piedmontese, Maremmana, and Podolica cattle breeds. J. Hered. 95: 250-256.
Moody, D. E., D. Pomp, & W. Barendse. 1995. Restriction fragment lenght polymorphism in amplification products of the bovine growth hormone releasing hormone gene. J. Anim. Sci. 73: 3789.
Muladno. 2002. Seputar Teknologi Rekayasa Genetika. Pustaka Wirausaha Muda dan USESE Foundation, Bogor.
Nei, M. 1987. Molecular Evolutionary Genetics. Colombia University Press, New York.
Nei, M. & S. Kumar. 2000. Molecular Evolution and Phylogenetics. Oxford University Press, New York.
Nicholas, F. W. 1996. Introduction to Veterinary Genetics. 2nd ed. Oxford University Press, Inc., New York.
Noor, R. R. 2008. Genetika Ternak. Penebar Swadaya, Jakarta.
Perkins, D. Jr. 1969. Fauna of Catal Huyuk: Evidence for early cattle domestication in Anatolia. Sci. 164: 177-179.
Pierzchala, M., T. Blicharski, & J. Kuryl. 2003. Growth rate and carcass quality in pigs as related to genotype at loci POU1F1/RsaI (Pit1/RsaI) and GHRH/AluI*. J. Anim. Sci. 21: 159-166.
Pusat Penelitian Pengembangan dan Peternakan. 2009. Strategi Menjaring Calon Bibit Sapi Perah. Warta Penelitian dan Pengembangan Pertanian. 31: 4.
Sambrook, J., E. F. Fritsch, & T. Maniatis. 1989. Molecular Cloning, A Laboratory Manual. 2nd ed. Cold Spring Harbour Lab. Press, New York.
Szatkowska, I., A. Dybus, W. Grzesiak, M. Jdrzejczak, & M. Muszyska. 2009. Association between the growth hormone releasing hormone (GHRH) gene polymorphism and milk production traits of dairy cattle. J. of App. Anim. Res. 36: 1.
Tambasco, D. D., C. C. P. Paz, M. Tambasco-Studart, A. P. Pereira, M. M. Alencar, A. R. Freitas, L. L. Coutinho, I. U. Packer, & L. C. A. Regitano. 2003. Candidate genes for growth traits in beef cattle crosses Bos Taurus x Bos Indicus. J. Anim. Breed. Genet. 120 : 51-56.
Vasconcellos, L. P. M. K, D. T. Talhari, A. P. Pereira, L. L. Coutinho, & L. C. A. Regitano. 2003. Genetic characterization of Aberdeen Angus cattle using molecular markers. Genet. and Mol. Biol. 26: 133-137.
Verkuil, E. V. P., A. V. Belkum, & J. P. Hays. 2008. Principles and Technical Aspects of PCR Amplification. Springer, Rotterdam.
Weir, B. S. 1996. Genetic Data Analysis II : Method for Discrete Population Genetic Data. 2nd ed. Sinauer Associates. Sunderland.
Williams, J. L. 2005. The use of marker-assisted selection in animal breeding and biotechnology. Rev. Sci. Tech. Off. Int. Epiz. 24: 379-391.
Williamson, G. & W. J. A. Payne. 1993. Pengantar Peternakan di Daerah Tropis. Terjemahan: Prof. Dr. SGN Djiwa Darmadja. Gadjah Mada University Press, Yogyakarta.
Lampiran 1. Sekuen Gen GHRH pada Bos taurus (Nomor Akses AF242855) (Moody et al., 1995)
LOCUS AF242855 9356 bp DNA linear MAM 30-JUL-2000 DEFINITION Bos taurus growth hormone releasing hormone gene, complete cds. ACCESSION AF242855
VERSION AF242855.1 GI:9587127 KEYWORDS .
SOURCE Bos taurus (cattle)
ORGANISM Bos taurus </Taxonomy/Browser/wwwtax.cgi?id=9913> Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;
Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 9356)
AUTHORS Zhou,P., Kazmer,G.W. and Yang,X. TITLE Direct Submission
JOURNAL Submitted (08-MAR-2000) Animal Science, University of Connecticut,
3636 Horsebarn Hill Road, Storrs, CT 06269, USA FEATURES Location/Qualifiers
source 1..9356
/organism="Bos taurus" /mol_type="genomic DNA" /db_xref="taxon:9913 </Taxonomy/Browser/wwwtax.cgi?id=9913>" mRNA </nuccore/9587127?itemid=7&report=gbwithparts> join(65..128,4340..4440,4707..4811,7048..7167, 9337..>9356)
/product="growth hormone releasing hormone"
exon </nuccore/9587127?from=65&to=128&report=gbwithparts> 65..128 /number=1 exon </nuccore/9587127?from=4340&to=4440&report=gbwithparts> 4340..4440 /number=2 CDS </nuccore/9587127?itemid=1&report=gbwithparts> join(4361..4440,4707..4811,7048..7167,9337..9352) /codon_start=1
34
3901 tccagtcttc tcgcctgaag aatccccacg gacagaggag cctggtgggt cgcaaagtgt 3961 cggacgcaac tgaatcgatt tagcacacat tattatatta ttccttgctg aggcctgagt 4021 cccacgtaga atgccctgta ggaagcggta gtttcaccat tgtctttcat tgtatgttca 4081 accctgtgag gtcagcggca tagccttatc atgaccattt tacagatgga gaaaatgagg 4141 ccagagtttg tgactggcag aggcagaact tgaacccagg tgtgtcagac tttgagcctg 4201 ctctgtcttc cctaccccac agcttccaaa agacaaggat catagctctc acggaccagg 4261 ccgagggcca gagctagcag agggcatctc agtgtctatg gctatgcttg accttttctt
4321 cctgtctgtc atttcccagg taccagcaca ggggtgaagg atgctgctct gggtgttctt
4381 cctcgtgacc ctcaccctca gcagcggctc ccacggttcc ctgccttccc agcctctcag 4441 gtaagcagtt ctgagaagag aagcaagaga ggccctttga ggatgcgact cgagctggtc 4501 cccagctggg tcctcaggca gcctcccttg ctcatctctg ggagggtggc agactgagcc 4561 ccagagaggt caccacccag ccctggttcc agccctctct ggggacgagc agggcaagag 4621 gcgacagaaa gacctcacag agaccaagtg agcacagtcc cctgggcctc ccaccccacc 4681 ctttgacctc tgactccttc tactaggatt ccacggtacg cagatgccat cttcactaac
4741 agctaccgga aggttctggg ccagctgtct gcccgcaagc tactccagga tatcatgaac
4801 aggcagcagg ggtgagccgg cgttctcgtg acttctccct gcaccctcgg ttcatcatga
36
7801 gaactccatt tcttcaaaac gacacgactg tttgcagaaa ctgtgctgta ttttagagcc 7861 cgatttttga actgtgagca caaccatgcc ccagcgtgac tctgccagac acctccagcc 7921 caaactatta agcaaatcca gttcccagca gagttctccg tgtgccccct cgccttcttc 7981 ccagcctgat tagacaccga gagctcagag atctggccaa gtattgtcct ggggagatgc 8041 aagccgcagc aggcggacag gcagagcgta gcagaggtga gaaccaaccc cggaggcagg 8101 atggacacct gagccccagg tctgcttgtg cagccaagtc tttgcacaca gccagtcatc 8161 ttccccgttc ccacctctgt gaagggagca gatttgctcc aatggccacc aaggtcctgg 8221 aggatagaat tcttttttta aataaatgtc cctagagaat aatgcctccc ccctccacac 8281 cgtcccagcc ccgtgcctct catgctggga ttaggtcact attcacctgc ttcagggctc 8341 ccaccaggcc ccagtggggt agagtggccc caaggagggc cacgtcctaa gaatgcaggc 8401 ttggttcctg caggttaaaa gctagaggct tcacagagct gaaagtccta gagcggcagg 8461 aaggccccag aggttgcccc agccattgtt ccccctccag ctcaactgtt gcaaatccca 8521 ggacatgagg cgggggcggg aggggggggg ggcggtgctg agaaaacaga actgctgaat 8581 gaattcttac attaatacgt taagagtaga atgatttggc agttagcagc ctttcatacc 8641 caccatgctt tatgtccata taacagtcct ctaagttaat aagttcaggt ttctccatct 8701 tcccagctct taaaagtcat ccagggactt ccctggtggt ccagtggtta agacttcacc 8761 ttccaatgca gggggcatgg gttcagttcc tggtctggga aataagattc catatgcctt 8821 gtggccaaaa catgaaaaca taaaacagaa gcaatattgt aactaattca gtaaagtctt 8881 ctaaaatggt ccatataaaa aaaaatctta aaaaacaaaa agtcatccaa agatggccag 8941 gctctgggct gaggggatgg ccatggatgg gccagtcttg gtttctgctc tcatggaatt 9001 cataacacac tgaagtagac agaaattaaa caagtacgtg cacagtggat tatttaattt 9061 gtgataaatg gtaagggatg tggtgaacgt gtgtgctgga aagaatgacc tcatctaccc 9121 ctgtttggga gttagaagtg gcttcttcaa gggccagtga ttgtccaggc tctcaatagg 9181 agtgggttgg gagagctggg gctaagactg aggtcatctt gggagtcggt cagtctcaac 9241 attggagtca cggtcaccct agttccaagt gtcctgtcct accttgcctt ggtttcggtg
9301 cccccagact aagctttatc tttctttctg caataggaac tcccaaggat gaagca
IDENTIFIKASI KERAGAMAN GEN
GROWTH HORMONE
RELEASING HORMONE
(GHRH|
Hae
III) PADA SAPI
FRIESIAN HOLSTEIN DI BIB LEMBANG,
BBIB SINGOSARI, DAN BET CIPELANG
SKRIPSI
ANNISA OKTAVIA RINI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
ANNISA OKTAVIA RINI. D14070118. 2011. Identifikasi Keragaman Gen
Growth Hormone Releasing Hormone (GHRH|HaeIII) Pada Sapi Friesian Holstein di BIB Lembang, BBIB Singosari, dan BET Cipelang. Skripsi. Mayor Ilmu Produksi dan Teknologi Peternakan. Institut Pertanian Bogor
Pembimbing Utama : Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Ir. Anneke Anggraeni, M.Si, Ph.D.
Salah satu usaha yang dapat dilakukan untuk mendapatkan ternak yang memiliki produktivitas tinggi adalah dengan perbaikan genetik melalui seleksi. Seleksi perlu mengetahui karakteristik genetik ternak yang berpengaruh terhadap sifat kuantitatif, meliputi sifat pertumbuhan. Sifat pertumbuhan dikontrol oleh banyak gen dan sebagian besar aksinya bersifat aditif. Gen GHRH merupakan hormon yang menstimulasi sintesis dan sekresi Growth Hormone. Gen GHRH dapat mempengaruhi produktivitas dan meningkatkan pertumbuhan ternak. Penelitian ini bertujuan untuk mengidentifikasi keragaman gen GHRH pada sapi perah Friesian Holstein (FH) di BIB Lembang, BBIB Singosari, dan BET Cipelang; serta sapi pedaging di BET Cipelang sebagai pembanding.
Sampel darah sapi yang digunakan berjumlah 126 sampel, yakni 49 ekor sapi FH jantan berasal dari BIB Lembang (17 ekor), BBIB Singosari (32 ekor), dan sapi FH betina berasal dari BET Cipelang (40 ekor); serta sejumlah 37 ekor sapi pedaging betina terdiri dari Simmental (13 ekor), Limousin (14 ekor), Brahman (5 ekor), dan Angus (5 ekor) dari BET Cipelang. Penelitian ini menggunakan metode PCR-RFLP