KONSTRUKSI DAN ANALISIS PUSTAKA METAGENOMIK
YANG MENGEKSPRESIKAN SENYAWA BIOAKTIF
UNTUK MENGENDALIKAN Xanthomonas oryzae pv. oryzae
PADA TANAMAN PADI
TATIT SASTRINI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Konstruksi dan Analisis Pustaka Metagenomik yang Mengekspresikan Senyawa Bioaktif untuk Mengendalikan Xanthomonas oryzae pv. oryzae pada Tanaman Padi adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
TATIT SASTRINI. Konstruksi dan analisis pustaka metagenomik yang mengekspresikan senyawa bioaktif untuk mengendalikan Xanthomonas oryzae pv. oryzae pada tanaman padi. Dibimbing oleh GIYANTO dan KIKIN HAMZAH MUTAQIN.
Xanthomonas oryzae pv. oryzae penyebab penyakit hawar daun bakteri merupakan bakteri patogen penting pada pertanaman padi di dunia, termasuk Indonesia. Berbagai upaya telah dilakukan dalam pengembangan teknologi pengendalian patogen tersebut seperti eksplorasi agens hayati atau senyawa bioaktifnya untuk menekan pertumbuhan X. oryzae pv. oryzae, namun metode yang digunakan terbatas untuk agens hayati dan senyawa bioaktif dari mikrob yang dapat dikulturkan pada media buatan. Disisi lain, biosfer didominasi oleh berbagai jenis mikrob yang tidak dapatdikulturkan pada media buatan. Teknologi baru yang disebut metagenomika dikembangkan untuk mengatasi keterbatasan dari metode konvensional. Tujuan penelitian ini adalah mengonstruksi pustaka metagenomik dari mikrob pada rizosfer tanaman padi, melakukan analisis fungsional dari klon pustaka metagenomik terhadap X. oryzae pv. oryzae untuk menghasilkan klon yang mempunyai aktivitas antibiosis terhadap X. oryzae pv.
oryzae, memperoleh informasi tentang DNA sisipan yang mengodekan senyawa bioaktif anti-X. oryzae pv. oryzae, dan menganalisis potensi senyawa bioaktif dari pustaka metagenomik dalam menekan pertumbuhan X. oryzae pv. oryzae.
Pengambilan sampel rizosfer padi dilakukan dengan metode random purposive sampling dari beberapa daerah, yaitu Sleman, Daerah Istimewa Yogyakarta; Subang, Jawa Barat; Barito Kuala, Kalimantan Selatan; dan Indragiri Hilir, Riau. Konstruksi pustaka metagenomik dilakukan melalui tahapan ekstraksi DNA total secara langsung dari rizosfer tanaman padi yang dilanjutkan dengan fragmentasi, ligasi DNA sisipan dengan vektor pUC19, transformasi plasmid rekombinan mengandung DNA sisipan ke dalam sel inang kompeten (E. coli DH5α), dan seleksi transforman dengan seleksi biru putih. Klon pustaka metagenomik berupa koloni bakteri berwarna putih dipilih untuk analisis lebih lanjut.
Analisis fungsional klon pustaka metagenomik terhadap X. oryzae pv. oryzae dilakukan dengan metode paper disc diffusion assays untuk melihat aktivitas antibiosis dalam penekanan X. oryzae pv. oryzae secara in-vitro. Konfirmasi keberadaan DNA sisipan pada klon pustaka metagenomik potensial dilakukan dengan teknik PCR koloni menggunakan pasangan primer M13F (-20) (5´- GTA AAA CGA CGG CCA GT-3´) dan primer M13R (-24) (5´-GGA AAC AGC TAT GAC CAT G-3´).
bioaktif dari klon pustaka metagenomik menggunakan metode paper disc diffusion. Ekstrak kasar senyawa bioaktif diperoleh dari hasil ekstraksi menggunakan pelarut n-heksan, kloroform, dan etil asetat.
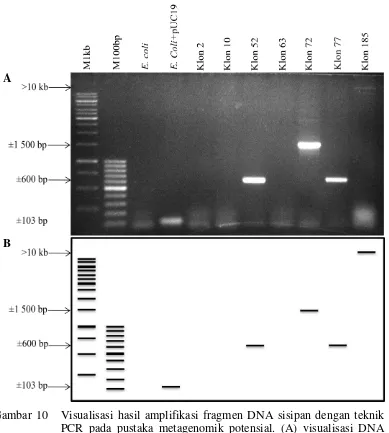
Berdasarkan analisis fungsional dari ratusan klon pustaka metagenomik, diperoleh tujuh klon (2, 10, 52, 63, 72, 77, dan 185) memproduksi senyawa bioaktif anti-X. oryzae pv. oryzae. Konfirmasi lebih lanjut dengan teknik PCR menggunakan pasangan primer M13/pUC menunjukkan empat klon pustaka metagenomik, yaitu klon 52, 72, 77, dan 185 masing-masing mengandung DNA sisipan berukuran ±600 bp, ±1 500 bp, ±600 bp, dan lebih dari >10 kb. Hal ini menunjukkan bahwa seluruh atau sebagian dari DNA sisipan yang terdapat di dalamnya merupakan gen fungsional yang terlibat dalam pengendalian X. oryzae pv. oryzae.
Analisis urutan nukleotida difokuskan pada dua fragmen DNA sisipan dengan panjang ±600 bp yang terdapat pada klon 52 dan ±1 500 bp pada klon 72. Kedua amplikon ini dipilih karena menunjukkan hasil yang konsisten dalam mempertahankan keberadaan DNA sisipan di dalam selnya. Hasil analisis homologi menunjukkan bahwa urutan nukleotida dari fragmen DNA sisipan pada klon pustaka metagenomik 52 dan 72 tidak memiliki kesamaan dengan urutan DNA gen lain pada GenBank database. Hasil penjajaran hypothetical protein dari kedua klon pada analisis lanjutan menunjukkan bahwa amplikon dari klon pustaka metagenomik 52 (±600 bp) memiliki tingkat kesamaan 62%-64% dengan histidinol dehidrogenase dan amplikon kedua dari klon pustaka metagenomik 72 (±1 500 bp) memiliki tingkat kesamaan 57% dengan enzim 3'-5' eksonuklease. Hal ini menunjukkan kebaruan dari gen pengkode senyawa bioaktif anti-X. oryzae pv. oryzae yang diperoleh dengan teknik metagenomik.
Pembacaan nilai OD600 dilakukan untuk melihat pengaruh senyawa bioaktif anti patogen pada substrat cair dari pustaka metagenomik 52, 72, dan 185 terhadap pertumbuhan X. oryzae pv. oryzae. Hasil analisis menunjukkan bahwa setiap perlakuan yang diuji memiliki pengaruh yang signifikan terhadap pertumbuhan X. oryzae pv. oryzae. Pertumbuhan X. oryzae pv. oryzae paling rendah diamati pada perlakuan senyawa bioaktif dari klon 72 dengan hasil yang konsisten dari awal hingga akhir pengamatan. Ekstraksi dilakukan pada senyawa bioaktif dari klon 52 dan 72. Kedua klon pustaka metagenomik tersebut dipilih karena menunjukkan hasil yang paling konsisten berdasarkan uji aktivitas antibiosis dan konfirmasi keberadaan DNA sisipan. Hasil yang diperoleh menunjukkan bahwa senyawa bioaktif dari klon 52 dapat diekstraksi menggunakan pelarut etil asetat, senyawa bioaktif dari klon 72 dapat diekstraksi menggunakan pelarut n-heksan dan etil asetat dengan tingkat penekanan yang berbeda. Senyawa bioaktif dari klon 72 yang diekstraksi menggunakan pelarut n-heksan menunjukkan penekanan pertumbuhan X. oryzae pv. oryzae paling baik. Dengan demikian konstruksi pustaka metagenomik yang mengekspresikan senyawa bioaktif anti-X. oryzae pv. oryzae telah berhasil dilakukan dan diperoleh 2 klon potensial, yaitu klon 52 dan 72 berdasarkan berbagai tahapan analisis yang dilakukan.
SUMMARY
TATIT SASTRINI. Construction and analysis of metagenomic libraries expressing bioactive compound for controlling Xanthomonas oryzae pv. oryzae on paddy. Supervised by GIYANTO and KIKIN HAMZAH MUTAQIN.
Xanthomonas oryzae pv. oryzae causing bacterial leaf blight of rice is an important plant pathogenic bacterium in the world including Indonesia. Several attempts has been reported to develop control technologies for bacterial leaf blight disease, including exploration of biological control agents (BAC) or development of bioactive compound expressed by BAC to suppress Xanthomonas oryzae pv. oryzae. However, those methods are limited to the substances conventionally product by culturable microbes in artificial medium. Whereas, biosphere is believed to be predominated by unculturable microbes which remain unexplored. A new technology called metagenomics is developed to overcome the disadvantage of conventional methods. The objectives of this research are to construct and analysis of metagenomic libraries from rice rhizosphere that expressing anti-X. oryzae pv. oryzae bioactive compound. The aim of this research is to construct of metagenomic libraries from rice rhizosphere microbial community, conducting a functional analysis of metagenomic library against X. oryzae pv. oryzae to obtain clones which show antibiosis activity in suppressing the growth of X. oryzae pv oryzae, this study was also conducted to obtain information about the DNA insert that expressing anti-X. oryzae pv. oryzae bioactive compound and analyze the potential of bioactive compound from metagenomic libraries in suppressing the growth of X. oryzae pv oryzae.
Rice rhizosphere samples were collected with random purposive sampling method from Sleman, Special Region of Yogyakarta; Subang, West Java; Barito Kuala, South Kalimantan; and Indragiri Hilir, Riau. Metagenomic library construction methods consist of direct extraction of total DNA from the paddy rhizosphere followed by DNA fragmentation, ligation on pUC19 vector, then introducing them into a host (E. coli DH5α) cell, and selecting transformants by blue white selection. White colonies (metagenomic library) were selected for further analysis. Functional analysis of metagenomic library clones of X.oryzae pv. oryzae was carried out using a paper disc diffusion assays to observe antibiosis activity of clones inhibit in-vitro growth of X.oryzae pv. oryzae. Subsequently, the inserted DNA fragment from the metagenomic library which is expressing the bioactive compound was confirmed by colony PCR using primer pairs: primer M13F (-20) (5´- GTA AAA CGA CGG CCA GT-3´) and primer M13R (-24) (5´-GGA AAC AGC TAT GAC CAT G-3´).
compounds were extracted with organic solvents i.e. n-hexane, chloroform, and ethyl acetate.
Based on functional analysis of hundreds selected of metagenomic library clones, seven metagenomic library have been found such as clones (2, 10, 52, 63, 72, 77, and 185) producing anti-X. oryzae pv. oryzae bioactive compounds. Further confirmation by PCR using M13/pUC primer pair showed that four metagenomic clones i.e. 52, 72, 77, and 185 contained ±600, ±1 500, ±600 bp, and > 10 kb fragment size, respectively. This indicate that the whole or part of insert DNA in recombinant plamids is functional gene which is responsible for antibiosis mechanism in controlling of X. oryzae pv oryzae.
Analysis of inserts DNA fragment was focused on the two amplicon i.e. ±600 bp (clone 52) and ±1 500 bp (clone 72). Both of the amplicons were selected because they still maintaining the presence of insert DNA after cultured in artificial media for several times. Nucleotide sequence analysis of DNA insert on clones 52 and 72 showed no similarity to known gene(s) in GenBank database. Then, we further performed hypothetical protein alignment and indicated that amplicon obtained from metagenomic libraries clone 52 (±600 bp) has similarity 62%-64% to protein histidinol dehydrogenase and amplicon obtained from metagenomic libraries clone 72 (± 1 500 bp) has similarity 57% with genes encoding 3'-5' exonuclease. Indicated that novel gene(s) encoding anti-X. oryzae pv. oryzae bioactive compound has been obtained with this metagenomics technique.
There are no any similarities of both DNA sequences to other sequences available in GenBank database. This data indicated that both DNA insert may be a novel gene encoding bioactive compound anti-X. oryzae pv. oryzae that never been characterized yet. Then, we further performed hypothetical protein alignment and indicated that the first amplicon obtained from metagenomic libraries clone 52 (±600 bp) has a level of similarity 62%-64% to protein histidinol dehydrogenase which function as a catalyst in the last two steps in the L-histidine biosynthesis pathway (Marchler-Bauer et al., 2015) as shown on Table 1.
Optical density value have been read to observe the effect of anti-pathogenic bioactive compounds contained in liquid substrate of 52 and 72 against growth of X. oryzae pv. oryzae. The results showed that each treatment which was tested had significant influence against growth of X. oryzae pv. oryzae with the lowest growth came from bioactive compound of clone 72. Extraction performed on bioactive compounds from clones 52 and 72. Both clones are chosen because they showed the most consistent results based on antibiosis activity test and confirm the presence of DNA insert. The results showed that bioactive compound of clone 52 can be extracted using a solvent ethyl acetate, bioactive compounds of clone 72 can be extracted using a solvent n-hexane and ethyl acetate with different suppression rate. The highest growth suppression of X. oryzae pv. oryzae came from crude bioactive compound of clone 72 was extracted with n-hexane. Thus, the construction of metagenomic libraries expressing anti-X. oryzae pv. oryzae bioactive compounds has been successfully and obtained two potential clones (clones 52 and 72) based on various stages of analysis performed.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Fitopatologi
KONSTRUKSI DAN ANALISIS PUSTAKA METAGENOMIK
YANG MENGEKSPRESIKAN SENYAWA BIOAKTIF
UNTUK MENGENDALIKAN Xanthomonas oryzae pv. oryzae
PADA TANAMAN PADI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2016
Judul Tesis : Konstruksi dan analisis pustaka metagenomik yang mengekspresikan senyawa bioaktif untuk mengendalikan Xanthomonas oryzae pv. oryzae pada tanaman padi Nama : Tatit Sastrini
NIM : A352120101
Disetujui oleh Komisi Pembimbing
Dr Ir Giyanto, MSi Ketua
Dr Ir Kikin Hamzah Mutaqin, MSi Anggota
Diketahui oleh
Ketua Program Studi Fitopatologi
Prof Dr Ir Sri Hendrastuti H, MSc
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT yang telah memberikan rahmat dan hidayah-Nya sehingga penulis dapat menyelesaikan karya ilmiah yang berjudul “Konstruksi dan analisis pustaka metagenomik yang mengekspresikan senyawa bioaktif untuk mengendalikan Xanthomonas oryzae pv. oryzae pada tanaman padi”.
Penulis menyampaikan ucapan terima kasih dan penghargaan yang setinggi-tingginya kepada Dr Ir Giyanto, MSi dan Dr Ir Kikin Hamzah Mutaqin, MSi selaku dosen pembimbing yang telah memberikan pengarahan dan motivasi tiada henti selama pelaksanaan kegiatan penelitian hingga penulisan karya ilmiah ini. Penulis menyampaikan ucapan terima kasih kepada Prof Dr Ir Sri Hendrastuti Hidayat, MSc selaku Ketua Program Studi Fitopatologi yang telah memberikan pengarahan kepada penulis selama pelaksanaan studi. Penulis juga mengucapkan terima kasih kepada Direktorat Jenderal Pendidikan Tinggi (DIKTI) yang telah memberikan beasiswa pendidikan melalui program Beasiswa Unggulan Dalam Negeri 2012. Ucapan terima kasih penulis ucapkan juga kepada Kementrian Penelitian dan Pengembangan Pertanian Indonesia atas dukungan biaya penelitian melalui Kemitraan Kolaborasi pada Penelitian dan Pengembangan Pertanian Nasional Nomor Hibah: 50/PL.220/I.1/3/2014.K tanggal 10 Maret 2014.
Rasa bangga dan ucapan terima kasih yang begitu besar penulis sampaikan kepada teman-teman Fitopatologi 2012, Mikrobiologi 2012, teman-teman di Laboratorium Bakteriologi Tumbuhan, dan sahabat-sahabat terbaik atas kebersamaan yang luar biasa dan sumbangan pemikiran selama menyelesaikan studi ini.
Ucapan terima kasih yang sebesar-besarnya penulis sampaikan kepada orang tua terbaik yang tiada henti memberikan do'a dan dukungan yang begitu besar agar penulis memperoleh pendidikan terbaik sebagai bekal masa depan. Semoga karya ilmiah ini dapat bermanfaat bagi kemajuan ilmu pengetahuan khususnya dalam bidang fitopatologi.
DAFTAR ISI
DAFTAR TABEL xiii
DAFTAR GAMBAR xiii
DAFTAR LAMPIRAN xiii
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 4
Xanthomonas oryzae pv. oryzae Penyebab Penyakit Hawar Daun
Bakteri 4
Pengendalian Hayati Patogen Tumbuhan 4
Eksplorasi Potensi Agens Hayati dengan Teknik Konvensional 5 Eksplorasi Potensi Agens Hayati dengan Teknik Metagenomika 5
3 METODE 11
Tempat dan Waktu 11
Bahan dan Alat 11
Pengambilan Sampel Rizosfer Tanaman Padi 11
Konstruksi Pustaka Metagenomik 12
Isolasi DNA Total dari Sampel Rizosfer Padi dan Fragmentasi
DNA 12
Penyiapan Sel Inang Kompeten (E. coli DH5α) 13
Penyiapan Vektor (plasmid pUC19) 13
Ligasi DNA Sisipan pada Plasmid pUC19 15
Transformasi Plasmid Rekombinan ke dalam Sel Inang Kompeten
(E. coli DH5α) 15
Seleksi Transforman dengan Metode Seleksi Biru Putih 16 Analisis Fungsional Klon Pustaka Metagenomik terhadap
X. oryzae pv. oryzae 16
Penyiapan Patogen Uji 16
Uji Antibiosis Klon Pustaka Metagenomik terhadap
X. oryzae pv. oryzae 17
Konfirmasi Klon Pustaka Metagenomik 17
Analisis Fragmen DNA Sisipan yang Terlibat dalam Penekanan
Pertumbuhan X. oryzae pv. oryzae 18
Pertumbuhan Klon Pustaka Metagenomik Potensial 18 Isolasi Plasmid Rekombinan Pada Klon Pustaka Metagenomik
Potensial 18
Perunutan Nukleotida dan Analisis Homologi Fragmen DNA
Sisipan 19
Analisis Potensi Senyawa Bioaktif dari Klon Pustaka Metagenomik
dalam Menekan Pertumbuhan X. oryzae pv. oryzae 19 Analisis Potensi Senyawa Bioaktif yang Terdapat pada Supernatan dari Klon Pustaka Metagenomik dalam Menekan Pertumbuhan
X. oryzae pv. oryzae 20
Analisis Potensi Ekstrak Kasar Senyawa Bioaktif dari Klon Pustaka Metagenomik dalam Menekan Pertumbuhan X. oryzae pv. oryzae 20
4 HASIL DAN PEMBAHASAN 22
Konstruksi Pustaka Metagenomik 22
Isolasi dan Fragmentasi DNA Sisipan 22
Sel Inang Kompeten dan Vektor (plasmid pUC19) 24 Transformasi dan Seleksi Bakteri Transforman 25 Analisis Fungsional Klon Pustaka Metagenomik terhadap X. oryzae
pv. oryzae 27
Patogen Uji 27
Aktivitas Antibiosis Klon Pustaka Metagenomik dalam Menekan
Pertumbuhan X. oryzae pv. oryzae 28
Analisis Fragmen DNA Sisipan yang Terlibat dalam Penekanan
Pertumbuhan X. oryzae pv. oryzae 33
Pengaruh Senyawa Bioaktif dari Pustaka Metagenomik terhadap
Pertumbuhan X. oryzae pv. oryzae 36
Pengaruh Senyawa Bioaktif yang Terdapat pada Supernatan dari Klon Pustaka Metagenomik terhadap Pertumbuhan X. oryzae
pv. oryzae 36
Pengaruh Ekstrak Kasar Senyawa Bioaktif dari Klon Pustaka
Metagenomik terhadap Pertumbuhan X. oryzae pv. oryzae 36
5 PEMBAHASAN UMUM 40
6 SIMPULAN DAN SARAN 43
Simpulan 43
Saran 43
DAFTAR PUSTAKA 44
LAMPIRAN 50
DAFTAR TABEL
1 Konsentrasi dan kemurnian DNA total mikrob hasil isolasi dari sampel rizosfer pertanaman padi pada beberapa agroekosistem
23 2 Tingkat kemiripan urutan asam amino DNA sisipan pada klon 52
dengan database protein di pusat data GenBank
34 3 Tingkat kemiripan urutan asam amino DNA sisipan pada klon 72
dengan database protein di pusat data GenBank
35
DAFTAR GAMBAR
1 Diagram alir penelitian konstruksi dan analisis pustaka metagenomik yang mengekspresikan senyawa bioaktif untuk mengendalikan X. oryzae
pv. oryzae pada tanaman padi 3
2 Peta struktur dan genetik plasmid pUC19 7
3 Visualisasi sampel DNA total asal rizosfer diikuti hasil pemotongan secara parsial dengan enzim restriksi HindIII 22 4 Perbanyakan plasmid pada sel kompeten E. coli DH5α 24 5 Konfirmasi DNA plasmid dengan pemotongan menggunakan enzim
restriksi HindIII dan DNA plasmid hasil defosforilasi 25 6 Hasil seleksi pustaka metagenomik dengan metode seleksi biru-putih 26
7 Konfirmasi X. oryzae pv. oryzae 27
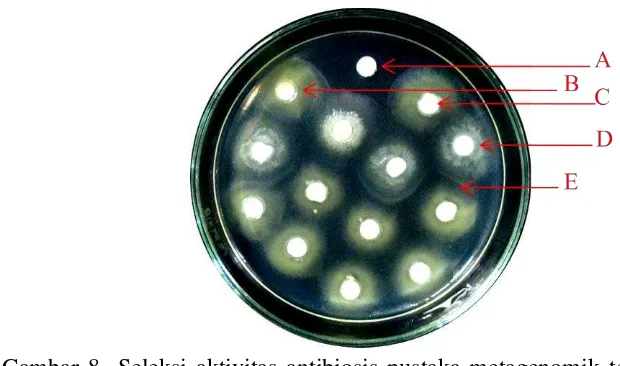
8 Seleksi aktivitas antibiosis pustaka metagenomik terhadap
penekanan X. oryzae pv. oryzae 28
9 Penghambatan pertumbuhan X. oryzae pv. oryzae oleh beberapa
klon pustaka metagenomik 30
10 Visualisasi hasil amplifikasi fragmen DNA sisipan dengan teknik
PCR pada pustaka metagenomik potensial 31
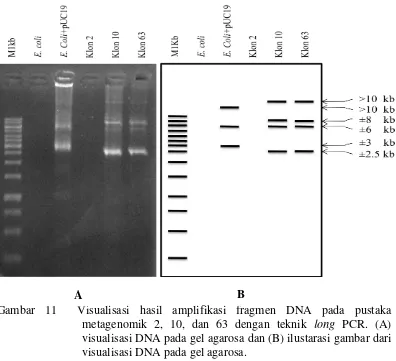
11 Visualisasi hasil amplifikasi fragmen DNA pada pustaka
metagenomik 2, 10, dan 63 dengan teknik long PCR 32 12 Pengaruh senyawa bioaktif klon pustaka metagenomik 52, 72, dan
185 terhadap pertumbuhan X. oryzae pv. oryzae 36 13 Hasil bioassay ekstrak kasar senyawa bioaktif dari klon pustaka
metagenomik dengan pelarut n-heksan, kloroform, dan etil asetat
terhadap X. oryzae pv. oryzae 37
14 Pengaruh ekstrak kasar senyawa bioaktif dari klon pustaka
metagenomik 52 dan 72 terhadap X. oryzae pv. oryzae 38
DAFTAR LAMPIRAN
1 Urutan nukleotida fragmen DNA sisipan pada klon pustaka
metagenomik 52 50
2 Urutan nukleotida fragmen DNA sisipan pada klon pustaka
metagenomik 72 51
3
Perbandingan urutan asam amino DNA sisipan dari klon pustaka metagenomik 52 dengan histidinol dehydrogenase [Candidatus
Koribacter versetilis] (WP 011524483.1) 52 4 Perbandingan urutan asam amino DNA sisipan dari klon pustaka
metagenomik 52 dengan histidinol dehydrogenase [Acidobacteria
bacterium KBS146] (WP 026387161.1) 53
5 Perbandingan urutan asam amino DNA sisipan dari klon pustaka metagenomik 72 dengan hypotetical protein [Pedoshaera parvula]
(WP 040550142.1) 54
6 Perbandingan urutan asam amino DNA sisipan dari klon pustaka metagenom 72 dengan 3'-5' exonuclease [bacterium Ellin514]
[Pedosphere parvula Ellin514] (EEF58269.1) 55 7 Pengaruh senyawa bioaktif klon pustaka metagenomik 52, 72, dan
185 terhadap pertumbuhan X. oryzae pv. oryzae 56 8 Analisis ragam pengaruh senyawa bioaktif klon pustaka
metagenomik 52, 72, dan 185 terhadap pertumbuhan
X. oryzae pv. oryzae 57
9 Ekstrak kasar masing-masing senyawa bioaktif dari klon pustaka
metagenomik 52 dan 72 58
10 Analisis ragam pengaruh ekstrak kasar senyawa bioaktif dari klon
pustaka metagenomik 52 dan 72 terhadap X. oryzae pv. oryzae 58 11 Pengaruh ekstrak kasar senyawa bioaktif dari klon pustaka
1 PENDAHULUAN
Latar Belakang
Penyakit hawar daun bakteri (HDB) yang disebabkan oleh X. oryzae pv. oryzae merupakan salah satu penyakit penting pada pertanaman padi di dunia, termasuk Indonesia. Luas pertanaman padi di Indonesia sampai tahun 2012 adalah 13 445 524 ha dan sebesar 43 719 ha terserang penyakit HDB (Ditjen Tanaman Pangan 2013). Di Indonesia, dilaporkan terdapat 12 patotipe X. oryzae pv. oryzae yang menyerang pertanaman padi. Tingginya kemampuan patogen untuk membentuk patotipe baru menjadi salah satu tantangan dalam upaya pengendalian (Suparyono et al. 2004). Menurut Suparyono dan Sudir (1992) serta BB Padi (2013), penurunan hasil produksi padi di Indonesia akibat serangan patogen tersebut sebesar 35.8%-60%. Teknik pengendalian yang dapat digunakan untuk penyakit ini adalah dengan penerapan praktik budi daya yang baik termasuk penggunaan benih sehat, penggunaan varietas tahan, pengendalian kimia, dan pengendalian hayati (Nino-Liu et al. 2006). Pengendalian kimia yang dilakukan dengan penggunaan bakterisida sintetik menjadi salah satu teknik pengendalian yang banyak digunakan, namun kenyataannya berbahaya bagi keseimbangan ekosistem. Dengan demikian pengendalian hayati mulai banyak dikaji karena dinilai lebih ramah terhadap lingkungan dan kesehatan manusia dibandingkan dengan pengendalian kimia, serta tidak menyebabkan resistensi patogen sasaran. Pengendalian hayati dilakukan dengan memanfaatkan agens hayati atau senyawa bioaktif yang dihasilkannya untuk mengendalikan patogen. Potensi agens hayati dapat dieksplorasi dari mikrob pada sampel lingkungan. Berbagai penelitian tentang komunitas mikrob pada sampel lingkungan menunjukkan bahwa 1% dari komunitas mikrob tersebut merupakan kelompok mikrob yang dapat dikulturkan (culturable microbes), sedangkan sebagian besarnya yaitu 99% merupakan kelompok mikrob yang tidak dapat dikulturkan (unculturable microbes) (Borneman et al. 1996; Torsvik dan Ovreas 2002). Berdasarkan studi Torsvik dan Ovreas (2002) dalam 1 gram tanah terdapat kurang lebih 4 000 spesies mikrob dan hanya 1% yang dapat dikulturkan pada media buatan.
2
transformasi ini disebut transforman. Kumpulan transforman yang mengandung vektor dengan fragmen DNA sisipan (gen sisipan) yang berbeda disebut pustaka metagenomik (Rondon et al. 2000; Daniel 2004; Liles et al. 2003; Amorim et al. 2008; Donato et al. 2010). Terdapat dua pendekatan dalam analisis pustaka metagenomik, yaitu analisis fungsional (function-driven analysis) dan analisis berdasarkan urutan nukleotida (sequence-driven analysis).
Teknik metagenomika dapat digunakan untuk mengeksplorasi potensi genetik yang belum diketahui. Teknik ini sangat potensial untuk mempelajari gen-gen fungsional yang berasal dari sampel lingkungan (Shen et al. 2007). Pendekatan teknik metagenomika juga dapat digunakan untuk studi pengendalian patogen tumbuhan. Van Elsas et al. (2011) menggunakan pendekatan teknik metagenomika untuk mempelajari komunitas mikrob tanah yang dapat mengendalikan penyakit tumbuhan. Sessitsch et al. (2012) juga menggunakan pendekatan metagenomika untuk analisis peranan bakteri endofit yang berasosiasi dengan akar tanaman padi termasuk salah satunya sebagai agens biokontrol terhadap patogen.
Tujuan Penelitian
Tujuan penelitian adalah mengonstruksi pustaka metagenomik dari mikrob pada rizosfer tanaman padi, melakukan analisis fungsional dari klon pustaka metagenomik untuk menghasilkan klon yang mempunyai aktivitas antibiosis terhadap X. oryzae pv. oryzae, memperoleh informasi tentang DNA sisipan yang mengodekan senyawa bioaktif anti-X. oryzae pv. oryzae, dan menganalisis potensi
senyawa bioaktif dari pustaka metagenomik dalam menekan pertumbuhan X. oryzae pv. oryzae.
Ruang Lingkup Penelitian
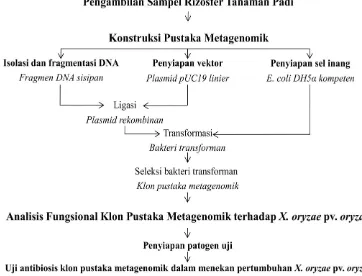
3
Gambar 1 Diagram alir penelitian Konstruksi dan Analisis Pustaka Metagenomik
4
2 TINJAUAN PUSTAKA
Xanthomonas oryzae pv. oryzae Penyebab Penyakit Hawar Daun Bakteri
Xanthomonas oryzae pv. oryzae merupakan bakteri patogen tanaman yang menyebabkan penyakit hawar daun bakteri (HDB) pada padi. Koloni bakteri X. oryzae pv. oryzae yang tumbuh pada media Yeast Extract-Dextrose-Calcium Carbonate Agar (YDCA) biasanya berwarna kuning, cembung, dan mukoid. Warna kuning pada koloni bakteri terbentuk karena bakteri tersebut memproduksi pigmen xanthomonadin (Swings et al. 1990; Schaad et al. 2000). X. oryzae pv. oryzae bersifat aerobik, berbentuk batang, dan tergolong Gram negatif. Bakteri tersebut masuk ke daun melalui hidatoda pada ujung dan tepi daun (Schaad et al. 2000; Nino-Liu et al. 2006; Wahyudi et al. 2011). Gejala penyakit yang disebabkan oleh bakteri patogen ini diawali dengan bintik-bintik berwarna kuning yang berkembang dan bergabung menjadi klorotik kemudian menjadi nekrotik, daun berwarna putih keabuan pada ujung dan tepi daun. Patogen bersifat tular benih sehingga benih merupakan sumber inokulum primer. Hal tersebut menunjukkan bahwa desinfeksi benih penting dilakukan sebagai salah satu teknik pengendalian X. oryzae pv. oryzae. Di lapangan, eksudat patogen menyebar karena angin atau melalui gesekan antara daun dari tanaman sakit ke tanaman sehat dan melaui percikan air hujan (Suparyono dan Sudir 1992; Nino-Liu et al. 2006).
X. oryzae pv. oryzae menyebabkan kerugian yang tinggi pada usaha produksi padi di Indonesia. Tingginya kemampuan patogen dalam membentuk patotipe baru merupakan salah satu tantangan dalam upaya pengendaliannya. Di Indonesia, dilaporkan terdapat 12 patotipe X. oryzae pv. oryzae berdasarkan virulensinya terhadap tanaman padi diferensial. Di Pulau Jawa terutama Jawa Barat, Jawa Tengah, dan Yogyakarta pada umumnya didominasi oleh patotipe VIII dan IV, sedangkan patotipe III rata-rata kurang dari 10%. X. oryzae pv. oryzae patotipe III dan IV hanya berkembang di daerah dataran rendah. Penurunan hasil produksi padi di Indonesia akibat serangan X. oryzae pv. oryzae cukup besar. Hal tersebut terjadi karena sebagian besar varietas padi komersial yang ada rentan terhadap patotipe VIII dan VI yang mendominasi daerah sentra produksi padi di Indonesia terutama di Jawa Barat, Jawa Tengah, dan Yogyakarta (Suparyono et al. 2004; BB Padi 2013).
Pengendalian Hayati Patogen Tumbuhan
5 lebih mengarah kepada menjaga keseimbangan lingkungan, sehingga dapat menciptakan pertanian yang berkelanjutan (Munif et al. 2012).
Pengendalian hayati mengacu pada pemanfaatan secara sengaja organisme lokal atau yang diintroduksi untuk pengendalian patogen tumbuhan. Salah satu hal penting dalam praktik pengendalian hayati adalah manipulasi lingkungan untuk meningkatkan aktivitas agens hayati sehingga secara tidak langsung dapat menekan patogen. Pengendalian hayati penyakit tanaman dengan menggunakan mikrob antagonis bertumpu pada mekanisme antibiosis, kompetisi, hiperparasitisme, dan induksi resistensi pada tanaman inang (Sigee 1993; Cook dan Baker 1996; Pal dan Gardener 2006; Suryanto et al. 2010). Eksplorasi agens hayati merupakan salah satu komponen utama pada studi pengendalian hayati. Dengan demikian, pemahaman tentang prinsip dan metode dalam isolasi dan seleksi agens hayati menjadi salah satu hal yang penting dalam penerapan teknik ini. Baker dan Cook (1974) menjelaskan bahwa agens hayati harus dicari pada area dimana penyakit yang disebabkan oleh patogen tidak ada, menurun, atau tidak dapat berkembang walaupun tanamannya rentan.
Eksplorasi Potensi Agens Hayati dengan Teknik Konvensional
Eksplorasi potensi agens hayati dengan teknik konvensional telah banyak dilakukan dan dikembangkan. Teknik konvensional dilakukan dengan mengisolasi mikrob calon agens hayati dari suatu sampel pada media biakan untuk selanjutnya dilakukan seleksi, karakterisasi, dan uji potensi agens hayati dengan metode standar yang lebih sederhana (Wahyudi et al. 2011; Munif et al. 2012). Berbagai penelitian mengenai eksplorasi dan karakterisasi agens hayati telah dilakukan. Hastuti et al. (2012) menemukan 10 isolat Streptomyces spp. yang juga efektif menekan patogen tersebut berdasarkan pengujian in planta dan in-vitro. Berdasarkan hasil uji in-vitro, dua isolat diantaranya (LBR02 dan AB131-1) memiliki kemampuan memproduksi kitinase, fosfatase, dan siderofor yang merupakan karakteristik dari agens biokontrol. Menurut Alabouvette et al. (2006), agens hayati yang melakukan pengendalian patogen melalui mekanisme antibiosis memiliki kemampuan untuk menghasilkan suatu senyawa bioaktif antipatogen. Cao et al. (2004) dan Verma et al. (2009) menjelaskan bahwa penelitian tentang eksplorasi agens hayati juga dapat digunakan untuk mengeksplorasi berbagai senyawa bioaktif alami baru yang dapat mengendalikan patogen.
Penelitian-penelitian yang dilakukan telah berhasil mengeksplorasi berbagai agens hayati dan senyawa bioaktif yang dihasilkannya untuk berbagai patogen tumbuhan menggunakan teknik konvensional. Teknik konvensional hanya dapat digunakan untuk mengeksplorasi potensi agens hayati dari mikroorganisme yang dapat dikulturkan pada media buatan. Berdasarkan hasil penelitian tentang keragaman mikrob, diketahui bahwa hanya 0.1-10% dari seluruh mikrob di alam yang dapat dikulturkan dibawah kondisi laboratorium. Dengan demikian, sebagian besar dari potensi mikrob lingkungan tidak dapat dipelajari dan dieksplorasi menggunakan teknik konvensional (Borneman et al. 1996; Torsvik dan Ovreas 2002; Zeyaullah et al. 2009).
Eksplorasi Potensi Agens Hayati dengan Teknik Metagenomika
6
dikembangkannya suatu teknik penelitian baru yang disebut teknik metagenomika. Metagenomika adalah analisis keseluruhan genom mikrob dari suatu sampel lingkungan (Rondon et al. 2000; Schlos dan Handelsman 2003; Handelsman 2004). Teknik ini banyak digunakan untuk mempelajari fisiologi dan ekologi mikrob pada sampel lingkungan dan mulai diperkenalkan pada akhir tahun 1990 (Rondon et al. 2000; Schlos dan Handelsman 2003; Handelsman 2004; Ghazanfar et al. 2010).
Teknik metagenomika dapat digunakan untuk mengeksplorasi potensi genetik yang belum diketahui dan dimanfaatkan. Proses kultivasi bakteri tidak diperlukan dalam teknik metagenomika karena sasaran analisis bukan sel hidup melainkan molekul DNA. Hal ini memungkinkan diperolehnya gen-gen fungsional baru yang terlibat dalam pengendalian patogen tumbuhan seperti gen pengkode antibiotik, enzim, atau senyawa volatil antimikrob dari kelompok mikroorganisme yang tidak dapat dibiakkan pada media buatan (Schloss dan Handelsman 2003; Van Elsas et al. 2011; Sessitsch et al. 2012). Teknik ini terdiri atas tahapan konstruksi pustaka metagenomik dan analisis pustaka metagenomik.
Konstruksi pustaka metagenomik. Konstruksi pustaka metagenomik dilakukan dengan mengekstraksi DNA total mikrob secara langsung dari suatu sampel lingkungan, penyiapan vektor, ligasi DNA sisipan pada vektor, kloning DNA dan transformasi pada sel kompeten, serta seleksi transforman (Sabree et al. 2009). Daniel (2004) menjelaskan bahwa keberhasilan dari teknik metagenomika sangat berhubungan dengan kualitas DNA yang dipengaruhi pula oleh metode ekstraksi DNA. Oleh sebab itu, penelitian tentang metode ekstraksi atau isolasi DNA secara langsung dari sampel lingkungan mulai banyak dikembangkan. Miller et al. (1999) melakukan evaluasi sembilan prosedur metode ekstraksi dan jenis sampel yang berbeda. Pada teknik metagenomik, DNA total mikrob dari suatu sampel lingkungan diekstraksi secara langsung sehingga memungkinkan didapatkannya sumber daya genetik baik dari mikroba yang dapat dikulturkan maupun yang tidak dapat dikulturkan sebagai bahan analisis (Rondon et al. 2000; Sabree et al. 2009).
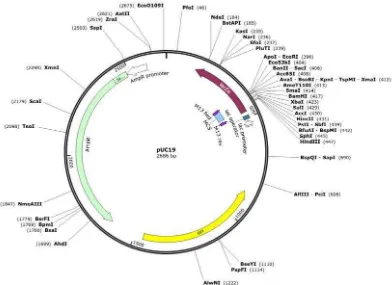
7
Gambar 2 Peta struktur dan genetic plasmid pUC19 (http://www.snapgene.com /resources/ plasmid_files/basic_cloning_vectors/pUC19)
Proses ligasi merupakan penggabungan fragmen DNA yang telah dipotong menggunakan enzim restriksi untuk menghasilkan suatu molekul DNA rekombinan. Pada teknik metagenomika, DNA total dari sampel lingkungan dipotong menggunakan enzim restriksi endonuklease tipe II. Menurut Old dan Primrose (2003), enzim ini memotong ikatan fosfodiester pada situs pengenalan yang bersifat spesifik dari suatu molekul DNA sehingga digunakan dalam teknik rekayasa genetika. Fragmen DNA tersebut kemudian digabungkan dengan vektor yang juga telah dipotong dengan enzim restriksi yang sesuai. Proses penggabungan ini melibatkan enzim ligase. Sambrook dan Russell (2001) menjelaskan bahwa enzim ligase dapat mengkatalisis pembentukan ikatan fosfodiester diantara ujung 3'-hidroksil dan 5'-fosforil DNA pada saat proses replikasi DNA di dalam sel inang.
8
Cohen et al. 1972). Bakteri biasanya mengandung sistem restriksi yang berperan dalam pengenalan dan pendegradasian DNA asing. Hal tersebut dapat mempengaruhi efisiensi proses transformasi. Oleh karena itu biasanya digunakan sel kompeten (E. coli) hasil mutasi yang tidak mengandung restriksi sebagai sel inang (Old dan Primrose 2003). Berbagai penelitian telah dilakukan untuk mengetahui faktor-faktor yang mempengaruhi efisiensi transformasi. Hasil yang diperoleh menunjukkan bahwa E.coli dan DNA plasmid melakukan interaksi secara produktif pada kondisi dimana ion kalsium tersedia, temperatur rendah (0-5 o
C), dan adanya kejutan panas pada suhu 37-45 oC. Pengujian keberhasilan transformasi dan stabilitas plasmid di dalam sel inang (sel kompeten) penting dilakukan karena dalam transformasi biasanya jumlah sel yang berhasil ditransformasi lebih sedikit dari pada jumlah sel yang tidak berhasil ditransformasi (Old dan Primrose (2003).
Seleksi bakteri transforman yang mengandung vektor rekombinan atau disebut pustaka metagenomik dalam teknik metagenomika merupakan salah satu tahap penting. Plasmid yang digunakan sebagai vektor biasanya mengandung gen resisten terhadap antibiotik tertentu seperti ampisilin, tetrasiklin, atau kloramfenikol sebagai penanda seleksi sehingga sel-sel yang tidak mengandung plasmid akan mati ketika ditumbuhkan pada media yang mengandung ampisilin (Old dan Primrose 2003). Seleksi tranforman juga dapat dilakukan dengan pengamatan ekspresi dari gen pelapor yang dimiliki oleh vektor seperti gel LacZ untuk seleksi biru-putih (Dale dan Park 2010).
9 Analisis klon pustaka metagenomik. Analisis pustaka metagenomik dilakukan dengan dua pendekatan, yaitu analisis berdasarkan urutan nukleotida (sequence-driven analysis) dan analisis fungsional (function-driven analysis). Sequence-driven analysis biasanya digunakan dalam penelitian yang bertujuan untuk mengetahui keragaman organisme dan filogeni dari suatu sampel lingkungan. Function-driven analysis digunakan dalam penelitian yang bertujuan untuk mengetahui peranan berbagai mikrob pada sampel lingkungan dan untuk memperoleh klon yang mengekspresikan sifat yang diinginkan. Analisis dilakukan berdasarkan sifat fenotipik yang diekspresikan oleh klon pustaka metagenomik, diikuti analisis urutan DNA sisipan, dan alisis biokimia (Schloss dan Handelsman 2003). Pendekatan ini baik digunakan untuk menghasilkan klon atau produk alami seperti protein yang bermanfaat dalam pengendalian patogen tumbuhan (Van Elsas et al. 2011; Sessitsch et al. 2012).
Ekspresi gen merupakan salah satu hal yang perlu difahami dalam teknik metagenomika terutama untuk analisis pustaka metagenomik menggunakan pendekatan function-driven analysis (Rondon et al. 2000; MacNeil et al. 2001; Schloss dan Handelsman 2003). Ekspresi gen membahas tentang bagaimana informasi genetik dikendalikan dan digunakan oleh sel (Paolella 1998). Ekspresi sifat genetik pada organisme termasuk prokariot diawali suatu proses yang disebut transkripsi. Transkripsi adalah proses penyalinan kode genetik yang ada pada urutan DNA menjadi RNA (mRNA, tRNA, dan rRNA). Berbeda dengan replikasi yang terjadi pada keseluruha DNA, transkripsi hanya terjadi pada segmen DNA yang mengandung kelompok gen tertentu saja (unit transkripsi). Gen yang lengkap terdiri atas tiga komponen utama, yaitu promoter sebagai pengendali proses transkripsi, bagian struktural yang mengandung informasi genetik, dan terminator yang berperan dalam pengakhiran proses transkripsi. Pada proses selanjutnya yaitu translasi, mRNA yang membawa informasi genetik kemudian diterjemahkan menjadi asam amino penyusun suatu polipeptida atau protein tertentu (Paolella 1998; Yuwono 2005).
E. coli yang biasa digunakan sebagai sel inang dalam rekayasa genetik memiliki operon lac yang mengendalikan metabolisme laktosa. Operon lac ini terdiri atas tiga gen structural, yaitu gen lacZ, lacY, dan lacA. Pada vektor ekspresi seperti plasmid pUC19, daerah penyisipan DNA atau gen asing (MCS) berada pada gen lacZ. Dengan demikian proses ekspresi gen asing pada sel inang E. coli terjadi dibawah kendali Promotor lac (Plac) yang terdapat pada vektor tersebut. Promotor ini merupakan situs yang dikenali oleh RNA polimerase sel inang untuk memulai proses transkripsi (Liaw dan Srinivasan et al. 1989; Paolella 1998; Yuwono 2005; Dale dan Park 2010).
10
11
3 METODE
Tempat dan Waktu
Penelitian dilaksanakan di Laboratorium Bakteriologi Tumbuhan serta di Laboratorium Fisiologi dan Toksikologi Serangga, Departemen Proteksi Tanaman, Institut Pertanian Bogor pada bulan Mei 2013 sampai Juli 2015.
Bahan dan Alat
Bahan yang digunakan dalam pelaksanaan penelitian ini adalah sampel rizosfer pertanaman padi, PowerSoil® DNA Isolation Kit (MO-BIO), GeneJET Plasmid Miniprep Kit (Thermo Scientific), bakteri kompeten Eschericia coli DH5α, enzim restriksi HindIII (Thermo Scientific), vektor transformasi berupa plasmid pUC19 (Thermo Scientific), T4 DNA Ligase (Thermo Scientific), 5-bromo-4-cloro-3-indolylbeta-D-galactopyranocide atau X-gal (Thermo Scientific), isopropyl-β-D-thiogalactopyranoside atau IPTG (Thermo Scientific), Dream Taq Green PCR Master Mix 2X (Thermo Scientific), Dream Taq KAPA HiFi HotStart PCR Kit (Kapabiosystems), primer M13F (-20) (5'- GTA AAA CGA CGG CCA GT-3') dan primer M13R (-24) (5'-GGA AAC AGC TAT GAC CAT G-3'), GeneRuler 1 kb DNA Ladder (Thermo Scientific), GeneRuler 100 bp DNA Ladder (Thermo Scientific), bakteri patogen tumbuhan Xanthomonas oryzae pv. oryzae patotipe IV, n-heksan, etil asetat, kloroform, aseton, dan sebagainya. Alat yang diperlukan dalam penelitian adalah sentrifuge (Hettich Mikro 200R Centrifuge), shaker waterbath (Advantec TBK202AA), Nanodrop 2000 (Thermo Scientific), alat elektroforesis, transilluminator UV, mesin PCR (GeneAmp PCR System 9700), syringe-filter 0.20 µm (Sartorius Stendim Biotech), corong pisah, rotary evaporator, dan sebagainya.
Pengambilan Sampel Rizosfer Tanaman Padi
12
Konstruksi Pustaka Metagenomik
Metode konstruksi pustaka metagenomik meliputi isolasi DNA total dari sampel rizosfer padi dan fragmentasi DNA, penyiapan sel inang kompeten (E. coli DH5α), penyiapan vektor (plasmid pUC19), ligasi DNA sisipan pada vektor pUC19, transformasi plasmid ke dalam sel inang kompeten (E. coli DH5α), dan seleksi tranforman dengan metode seleksi biru putih.
Isolasi DNA Total dari Sampel Rizosfer Padi dan Fragmentasi DNA
Isolasi DNA total dari sampel rizosfer tanaman padi dilakukan secara langsung dengan menggunakan PowerSoil® DNA Isolation Kit. Proses pelisisan sel mikrob dalam metode isolasi ini terjadi secara mekanik dan kimia. Masing-masing sebanyak 0.25 g sampel rizosfer dimasukkan ke dalam power bead tube kemudian dihomogenkan menggunakan vortex secara perlahan. Sebanyak 60 µL solution C1 (60 oC) ditambahkan kemudian dihomogenkan menggunakan vortex pada kecepatan 12 000 x g selama 10 menit. Sentrifugasi dilakukan pada 10 000 x g selama 30 detik pada suhu ruang. Supernatan yang diperoleh kemudian dipindahkan ke dalam 2 mL collection tube baru. Sebanyak 250 µL solution C2 ditambahkan kemudian dihomogenkan menggunakan vortex selama 5 detik. Inkubasi dilakukan selama 5 menit pada 4 oC kemudian suspensi tersebut disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Sebanyak 600 µL solution C3 dipindahkan ke dalam 2 mL collection 2 mL baru kemudian ditambahkan Solution C3 sebanyak 200 µL dan dihomogenkan menggunakan vortex beberapa saat. Inkubasi dilakukan pada suhu 4 oC selama 5 menit. Suspensi tersebut kemudian disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Sebanyak 750 µL supernatan dipindahkan ke dalam collection tube 2 mL baru dan ditambah 1.2 mL solution C4 kemudian dihomogenkan menggunakan vortex selama 5 detik. Sebanyak 675 µL supernatan dimasukkan ke dalam spin filter kemudian disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Cairan yang tertampung pada tabung bagian bawah dibuang. Sebanyak 675 µL supernatan dimasukkan ke dalam spin filter kemudian disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Cairan yang tertampung pada tabung bagian bawah dibuang. Supernatan yang masih tersisa dimasukkan kembali ke dalam spin filter dan disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Sebanyak 500 µL solution C5 ditambahkan ke dalam spin filter kemudian disentrifugasi pada 10 000 x g selama 30 detik pada suhu ruang. Cairan yang tertampung pada tabung bagian bawah dibuang. Kemudian tabung dengan spin filter disentrifugasi pada 10 000 x g selama 1 menit pada suhu ruang. Spin filter dipindahkan pada 2 mL collection tube baru. Sebanyak 100 µL solution C6 ditambahkan ke bagian tengah membran penyaring kemudian disentrifugasi pada 10 000 x g selama 30 detik pada suhu ruang. Spin filter dibuang, suspensi DNA yang diperoleh disimpan pada suhu -20 oC sebelum digunakan lebih lanjut.
13 Visualisasi dilakukan dengan menggunakan transilluminator UV. Pengukuran konsentrasi dan kemurnian DNA dilakukan menggunakan nanodrop.
Fragmentasi DNA tersebut dilakukan dengan pemotongan parsial menggunakan enzim restriksi HindIII selama 60 detik untuk menyiapkan fragmen DNA sisipan yang selanjutnya dianalisis lebih lanjut. DNA hasil isolasi (0.5-1 µg) masing-masing diambil dan dimasukkan ke dalam tabung mikro yang berisi 2 µL enzim restriksi HindIII (10 U µL-1), 2 µL 10x bufer R, dan nuclease-free water sampai dengan volume 21 µL. Campuran tersebut kemudian diinkubasi pada suhu 37 oC selama 60 detik. Inaktivasi enzim restriksi dilakukan dengan cara pemanasan pada suhu 80 oC selama 20 menit.
Penyiapan Sel Inang Kompeten (E. coli DH5α)
Strain E. coli DH5α digunakan sebagai sel inang pada penelitian ini. Penyiapan sel E. coli DH5α kompeten dilakukan dengan metode Inoue (Sambrook dan Russell 2001). Tahapan ini dilakukan untuk meningkatkan keefektifan pengambilan DNA asing oleh sel inang dalam proses transformasi. Biakan E. coli DH5α berumur 16-20 jam yang ditumbuhkan pada media NA (3 g L-1 beef extract, 5 g L-1 pepton, dan 15 g L-1 agar) diambil sebanyak 1 koloni kemudian Pengamatan dilakukan setiap 45 menit sampai diperoleh kultur bakteri dengan nilai OD600=0.55. Masing-masing sebanyak 1 mL kultur bakteri dipindah ke dalam tabung mikro kemudian sel dipanen dengan sentrifugasi 2 500 x g selama 10 menit pada suhu 4 oC. Supernatan dibuang, pelet diresuspensikan dengan 320 µL bufer Inoue transformasi (55 mM MnCl2∙4H2O, 15 mM CaCl2∙2H2O, 250 mM KCl, 10 mM PIPES pH 6.7, dan aquades). Sel bakteri dipanen dengan sentrifugasi 2 500 x g selama 10 menit pada suhu 4 oC.
Pelet bakteri yang diperoleh masing-masing diresuspensikan dengan 80 µL bufer Inoue transformasi dingin (-20 oC) dan 6 µL DMSO kemudian diinkubasi pada es selama 10 menit. Ujung tabung mikro berisi suspensi bakteri dicelupkan pada nitrogen cair kemudian disimpan pada suhu -70 oC.
Penyiapan Vektor (plasmid pUC19)
Plasmid pUC19 digunakan untuk konstruksi pustaka metagenomik pada penelitian ini. Penyiapan plasmid dilakukan dengan beberapa tahapan, yaitu perbanyakan plasmid, isolasi plasmid, pemotongan plasmid, dan defosforilasi plasmid.
14
dalam tabung reaksi steril dan diinkubasi pada suhu 37 oC selama 45 menit pada shaker waterbath. Sel bakteri dipanen dengan cara disentrifugasi pada kecepatan 10 000 x g selama 10 menit. Supernatan disisakan sebanyak 200 µL dan diresuspensikan kembali dengan pelet bakteri transforman. Suspensi bakteri disebar pada media SOB agar (2% pepton, 0.5% yeast extract, 10 mM NaCl, 2.5 mM KCl, 20 mM MgSO4, 10 mM MgCl, dan agar 15 g L-1) mengandung antibiotik ampisilin 50 µg mL-1. Inkubasi dilakukan selama 12-48 jam. Kontrol positif dalam percobaan ini berupa sel bakteri kompeten yang tidak mengandung plasmid dan ditumbuhkan pada media tanpa antibiotik. Kontrol negatif dalam percobaan ini berupa sel bakteri kompeten yang tidak mengandung plasmid dan ditumbuhkan pada media dengan antibiotik.
Isolasi plasmid pUC19 dilakukan dengan menggunakan GeneJET Plasmid Miniprep Kit #K0502. Proses sentrifugasi pada setiap tahapan dilakukan dengan kecepatan 12 000 x g pada suhu ruang. E. coli DH5α yang mengandung plasmid pUC19 hasil transformasi dikulturkan pada 5 mL media LB selama satu malam pada inkubator bergoyang. Sebanyak 1 mL suspensi bakteri dipanen dengan sentrifugasi selama 10 menit. Pelet yang terbentuk diresuspensikan dengan 250 µL resuspension solution yang mengandung 1% (v/v) RNase A (10 mg mL-1) menggunakan pipet atau vortex sampai tidak ada lagi sel yang menggumpal. Sebanyak 250 µL lysis solution ditambahkan ke dalam suspensi kemudian tabung berisi suspensi dibolak-balik 4-6 kali sampai larutan menjadi kental dan agak jernih. Sebanyak 350 µL neutralization solution ditambahkan kemudian dibolak-balik sebanyak 4-6 kali. Selanjutnya dilakukan sentrifugasi selama 5 menit untuk mengendapkan sisa-sisa pelisisan sel. Supernatan dipindahkan ke dalam GeneJET spin column pada collection tube kemudian disentrifugasi selama 1 menit. Larutan hasil penyaringan dibuang, column ditempatkan kembali pada collection tube. Tahapan pencucian dilakukan dengan menambahkan 500 µL wash solution (yang telah diencerkan dengan etanol) ke dalam GeneJET spin column kemudian disentrifus selama 30-60 detik. Larutan hasil penyaringan kembali dibuang, column kembali ditempatkan pada collection tube. Prosedur pencucian diulangi dengan menggunakan 500 µL wash solution. Larutan hasil penyaringan kembali dibuang, GeneJET spin column pada collection tube disentrifus selama 1 menit untuk menghilangkan sisa wash solution. GeneJET spin column dipindahkan ke dalam 1.5 mL tabung mikro baru. Sebanyak 50 µL elution buffer ditambahkan pada bagian tengah membran GeneJET spin column dan diinkubasi selama 2 menit agar DNA plasmid terelusi secara sempurna. GeneJET spin column disentrifugasi selama 2 menit. Column dibuang dan DNA plasmid disimpan pada suhu -20oC. Pengukuran konsentrasi dan kemurnian DNA plasmid dilakukan menggunakan nanodrop.
15 pada 75 volt. Penanda yang digunakan adalah GeneRuler 1 kb DNA Ladder. Visualisasi dilakukan dengan menggunakan transilluminator UV.
Defosforilasi plasmid dilakukan dengan metode Sambrook dan Russell (2001). Proses ini diawali dengan pemotongan DNA plasmid menggunakan enzim restriksi HindIII. Komponen reaksi pemotongan plasmid terdiri atas 0.5-1 µg DNA plasmid, 2 µL enzim HindIII (10 U µL-1), 2 µL 10X bufer R, dan nuclease free water sampai dengan 21 µL. Proses restriksi dilakukan selama 1 jam pada suhu 37 oC. Inaktivasi enzim restriksi dilakukan dengan inkubasi pada suhu 80 oC selama 20 menit. Sebanyak 0.1 µg plasmid hasil pemotongan dianalisis dengan elektroforesis pada 0.7% gel agarosa selama 30 menit pada 75 volt. Visualisasi dilakukan dengan menggunakan transilluminator UV. DNA plasmid kemudian diekstraksi menggunakan fenol:kloroform kemudian DNA plasmid diendapkan dengan proses presipitasi standar menggunakan 10 l 3M NaOAc pH 5.2 dan 250 l etanol absolut (-20 oC). Larutan tersebut disimpan dalam es selama 15 menit. phosphatase dilakukan dengan penambahan SDS (10% w/v) dengan konsentrasi akhir 5% dan EDTA (pH 0.8) dengan konsentrasi akhir 5 mM. Komponen tersebut dicampur secara sempurna dan ditambahkan proteinase K dengan konsentrasi akhir 100 µg mL-1. Inkubasi dilakukan selama 30 menit pada suhu 55 o
C. Campuran reaksi didinginkan pada suhu ruang dan DNA plasmid diekstrak kembali secara berturut-turut menggunakan fenol dan fenol:kloroform. DNA plasmid dipresipitasi kembali menggunakan prosedur presipitasi standar dengan menambahkan 10 l 3M NaOAc pH 5.2 dan 250 l etanol absolut (-20 oC). Inkubasi dilakukan selama selama 15 menit di dalam es. DNA plasmid diendapkan dengan sentrifugasi pada kecepatan 12 000 x g selama 10 menit pada suhu 4 oC. Pelet dicuci dengan 70% etanol dingin (4 oC) kemudian sentrifugasi kembali. Supernatan dibuang, DNA plasmid dikeringanginkan kemudian dilarutkan dengan TE (pH 0.8) sampai konsentrasi 100 ug/mL. Penyimpanan DNA dilakukan pada suhu -20 oC.
Ligasi DNA Sisipan pada Plasmid pUC19
Fragmen DNA sisipan yang telah disiapkan sebelumnya diligasikan pada vektor pUC19 dengan menggunakan T4 DNA Ligase (Thermo scientific). Reaksi ligasi terdiri atas 20-100 ng DNA plasmid linier, DNA sisipan dengan perbandingan 1:1 sampai 5:1 dengan DNA plasmid, 2 µL 10X T4 DNA ligase buffer, 1 unit T4 DNA ligase, dan nuclease free water ditambahkan sampai total volume mencapai 20 µL. Reaksi ligasi diinkubasi selama 10 menit pada 22 oC. Transformasi Plasmid Rekombinan ke dalam Sel Inang Kompeten (E. coli DH5α)
16
Seleksi Transforman dengan Metode Seleksi Biru Putih
Seleksi sel bakteri tranforman dilakukan dengan menumbuhkan bakteri transforman pada media SOB agar mengandung ampisilin 50 µg mL-1 media, 10 µL IPTG 0.1 M dan 40 µL X-gal 20 mg mL-1 per 90 mm agar cawan. Bakteri yang membawa vektor rekombinan (bakteri transforman) akan resisten terhadap antibiotik yang digunakan sehingga tumbuh pada media, sedangkan bakteri non-transforman tidak akan tumbuh pada media tersebut. Keberhasilan penyisipan ditandai dengan terbentuknya koloni bakteri berwarna putih. Apabila terjadi insersi gen atau fragmen DNA pada multiple cloning site (MCS) maka gen lacZ tidak dapat berfungsi sebagaimana mestinya sehingga tidak terjadi penguraian X-gal (substrat). Hal ini menyebabkan koloni bakteri tetap berwarna putih. Sedangkan apabila tidak terjadi insersi maka koloni bakteri akan berwarna biru yang disebabkan oleh aktivitas gen LacZ (Dale dan Park 2010). Koloni berwarna putih (klon pustaka metagenomik) dipilih untuk analisis lebih lanjut. Klon Pustaka metagenomik disimpan dalam stok gliserol 40%.
Analisis Fungsional Klon Pustaka Metagenomik terhadap X. oryzae pv. oryzae
Penyiapan Patogen Uji
Isolat bakteri X. oryzae pv. oryzae patotipe IV yang digunakan sebagai patogen uji diperoleh dari Balai Besar Penelitian Tanaman Padi (BB Padi), Sukamandi, Jawa Barat. Tahapan penyiapan patogen uji diawali dengan konfirmasi patogen uji melalui analisis secara fenotipik dan genotipik. Analisis fenotipik dilakukan dengan pengamatan karakter morfologi isolat bakteri pada media Wakimoto agar (300 g L-1 kentang, 7 g L-1 pepton, 17 g L-1 sukrosa, 0.5 g L-1 Ca(NO3)2∙4H2O, 1 g L-1 Na2HPO4∙12H2O, dan 17 g L-1 agar). Selain itu, dilakukan uji patogenisitas pada daun tanaman padi dengan metode pengguntingan daun. Pengujian dilakukan dengan cara memotong daun tanaman padi dengan gunting yang telah direndam oleh kultur cair isolat X. oryzae pv. oryzae berumur 48 jam. Pengamatan dilakukan terhadap gejala kerusakan yang muncul pada daun yang diinokulasi isolat bakteri patogen tersebut.
17 elongasi pada suhu 72 oC selama 1 menit yang dilakukan sebanyak 29 siklus, serta tahapan ekstensi akhir pada suhu 72 oC selama 7 menit (Keshavarz et al. 2011). Sebanyak 5 µL produk PCR dianalisis menggunakan gel agarosa 1.5% dalam 1X bufer TAE. Penanda yang digunakan adalah GeneRuler 100 bp DNA Ladder. Elektroforesis dilakukan dengan menggunakan gel agarosa 1.5 % selama 30 menit pada 75 volt. Gel diwarnai dengan etidium bromide dan difoto dibawah sinar UV. Uji Antibiosis Klon Pustaka Metagenomik terhadap X. oryzae pv. oryzae
Pengujian dilakukan dengan menggunakan teknik paper disc diffusion assays berdasarkan metode Ng dan Amsaveni (2012) yang dimodifikasi untuk melihat mekanisme antibiosis dalam penekanan X. oryzae pv. oryzae oleh isolat pustaka metagenomik. Pustaka metagenomik dari stok gliserol ditumbuhkan pada media SOB agar mengandung ampisilin 50 µg mL-1 media. Sebelum pustaka metagenomik ditumbuhkan pada media agar tersebut, sebanyak 10 µL IPTG 0.1 M dan 40 µL X-gal 20 mg/mL disebar pada permukaan agar cawan berdiameter 90 mm. Koloni tunggal pustaka metagenomik ditumbuhkan pada media SOB mengandung 50 µg mL-1 ampisilin dan diinkubasi pada shaker waterbath (100 rpm pada 37 oC). IPTG ditambahkan hingga konsentrasi akhir mencapai 0.1 mM ketika kultur bakteri mencapai nilai OD600=0.55 (fase logaritmik) kemudian diinkubasi kembali pada kondisi yang sama selama 12 jam. Sebanyak 3 µL suspensi pustaka metagenomik diteteskan pada kertas saring steril berdiameter 0.6 mm yang diletakkan diatas permukaan media PSA (10 g L-1 pepton, 10 g L-1 sukrosa, 1 g L-1 sodium glutamate, agar 15 g L-1 agar) mengandung 50 µg mL-1 ampisilin, 10 µL IPTG 0.1 M dan X. oryzae pv. oryzae 108 cfu per 90 mm agar cawan. Inkubasi dilakukan selama 4 hari pada suhu ruang. Perlakuan kontrol negatif dilakukan menggunakan isolat E. coli DH5α mengandung plasmid pUC1λ tanpa DNA sisipan. Pengamatan dilakukan terhadap zona bening yang terbentuk sebagai hasil penghambatan pertumbuhan X. oryzae pv. oryzae oleh senyawa bioaktif yang diproduksi oleh klon pustaka metagenomik. Penekanan dengan mekanisme antibiosis ditandai dengan pembentukan zona bening.
Konfirmasi Klon Pustaka Metagenomik
Tahapan konfirmasi keberadaan DNA sisipan pada klon pustaka metagenomik potensial dilakukan dengan PCR koloni menggunakan pasangan primer M13F (-20) (5´- GTA AAA CGA CGG CCA GT-3´) dan primer M13R (-24) (5´-GGA AAC AGC TAT GAC CAT G-3´) yang dapat mengamplifikasi fragmen DNA vektor (plasmid pUC19) pada situs kloning yang dimilikinya (Yanisch-Perron et al. 1985; Dechyeva 2006). Komposisi reaksi PCR terdiri atas 12.5 µL Dream Taq Green PCR Master Mix 2X, 10 pmol primer M13/pUC forward, 10 pmol primer M13/pUC reverse, dan PCR grade water sampai dengan volume 25 µL. Sel bakteri dari koloni tunggal pustak metagenomik diambil menggunakan ujung white tips kemudian diresuspensikan dengan bahan PCR yang sebelumnya telah disiapkan. Program PCR yang digunakan, yaitu 93 oC selama 5 menit; 35 siklus yang terdiri atas denaturation pada suhu 94 oC selama 30 detik, annealing pada suhu 56 oC selama 30 detik, dan extension pada suhu 72 o
18
100 bp DNA Ladder dan GeneRuler 1 kb DNA Ladder. Visualisasi dilakukan dengan menggunakan transilluminator UV.
Teknik long PCR digunakan untuk mengamplifikasi fragmen DNA sisipan yang berukuran lebih dari 3 Kb. Teknik ini diawali dengan penyiapan campuran bahan untuk reaksi PCR yang terdiri atas 12.5 µL 2X KAPA HiFi Hot Star ReadyMix mengandung 2.5 mM MgCl2 pada konsentrasi 1X, 10 pmol primer M13F (-20), 10 pmol primer M13R (-24), 1 µL DNA cetakan, dan PCR grade water sampai dengan 25 µL. Setiap sampel dikerjakan sebanyak dua reaksi PCR. Program PCR yang digunakan, yaitu pre-denaturation pada suhu 95 oC selama 5 menit; 25 siklus yang terdiri atas denaturation pada suhu 98oC selama 20 detik, annealing sekaligus extension pada suhu 68oC selama 7 menit; final extension pada suhu 72 oC selama 5 menit; dan cooling pada suhu 4 oC. Analisis ukuran DNA atau gen sisipan dilakukan dengan elektroforesis pada gel agarosa 1%. Visualisasi dilakukan dengan menggunakan transilluminator UV.
Analisis Fragmen DNA Sisipan yang Terlibat dalam Penekanan Pertumbuhan X. oryzae pv. oryzae
Penumbuhan Klon Pustaka Metagenomik Potensial
Stok pustaka metagenomik ditumbuhkan kembali pada media SOB agar mengandung antibiotik ampisilin 50 µg mL-1 media, 10 µL IPTG 0.1 M dan 40 µL X-gal 20 mg mL-1 per 90 mm agar cawan. Penambahan antibiotik, IPTG, dan X-gal dilakukan untuk menjaga plasmid yang mengandung DNA sisipan tetap utuh di dalam sel dan memastikan bahwa bakteri yang tumbuh pada media adalah pustaka metagenomik.
19 x g selama 2 menit pada suhu 4 oC. Supernatan (lapisan atas) dipindah ke dalam tabung mikro baru. Presipitasi DNA (plasmid rekombinan) yang terdapat pada supernatan dilakukan dengan menambahkan etanol (suhu ruang) sebanyak 2 kali volume supernatan yang diperoleh. Supernatan dicampur menggunakan vortex kemudian diinkubasi selama 2 menit pada suhu ruang. Supernatan tersebut kemudian disentrifugasi pada kecepatan 12 000 x g selama 5 menit pada suhu 4 o
C. Supernatan dibuang dan endapan DNA dicuci dua kali dengan menambahkan 1 mL etanol 70 % dingin (-20 oC) kemudian disentrifugasi pada kecepatan 12 000 x g selama 2 menit. Etanol dibuang, pelet asam nukleat yang diperoleh dikeringanginkan kemudian diresuspensikan dengan bufer TE (pH 8.0) mengandung 20 µg RNase A. Larutan DNA disimpan pada suhu -20 oC.
Amplifikasi Fragmen DNA Sisipan pada Plasmid Rekombina
Amplifikasi gen sisipan dilakukan dengan PCR standar menggunakan primer M13F (-20) dan primer M13R (-24). Reaksi PCR menggunakan Dream Taq Green PCR Master Mix 2X dengan DNA cetakan berupa DNA plasmid pUC19 mengandung DNA sisipan yang diisolasi dari pustaka metagenomik. Komposisi reaksi PCR terdiri atas 12.5 µL Dream Taq Green PCR Master Mix 2X, 10 pmol primer M13/pUC forward, 10 pmol primer M13/pUC reverse, 1 µL DNA cetakan, dan PCR grade water sampai dengan volume 25 µL. Program PCR yang digunakan, yaitu 93 oC selama 5 menit; 35 siklus yang terdiri atas denaturation pada suhu 94 oC selama 30 detik, annealing pada suhu 56 oC selama 30 detik, dan extension pada suhu 72 oC selama 90 detik; dan final extension pada suhu 72 oC selama 5 menit (Yanisch-Perron et al. 1985; Dechyeva 2006). Analisis ukuran DNA atau gen sisipan dilakukan dengan elektroforesis pada gel agarosa 1%. Visualisasi dilakukan dengan menggunakan transilluminator UV.
Perunutan Nukleotida dan Analisis Homologi Fragmen DNA Sisipan
Analisis urutan nukleotida dilakukan terhadap fragmen DNA sisipan yang memiliki ukuran berbeda. Perunutan nukleotida dari fragmen DNA sisipan yang terlibat dalam mekanisme pengendalian X. oryzae pv. oryzae dilakukan di Laboratorium First Base Asia, Malaysia. Hasil perunutan nukleotida diolah menggunakan software Sequence Scanner v1.0 dan CLC sequence viewer 6.7.1. Analisis lebih lanjut dilakukan untuk menganalisis tingkat homologi atau kemiripan dari fragmen DNA sisipan tersebut dengan gen lain yang tersedia di GenBank database menggunakan program BLASTX (Basic Local Alignment Search Tool).
Analisis Potensi Senyawa Bioaktif dari Klon Pustaka Metagenomik dalam Menekan Pertumbuhan X. oryzae pv. oryzae
Analisis lebih lanjut dilakukan untuk melihat potensi senyawa bioaktif dari klon pustaka metagenomik melalui uji potensi senyawa bioaktif yang terdapat
pada supernatan dari pustaka metagenomik dalam menekan pertumbuhan X. oryzae pv. oryzae menggunakan teknik peracunan media dan uji potensi
20
Analisis Potensi Senyawa Bioaktif yang Terdapat pada Supernatan dari Klon Pustaka Metagenomik dalam Menekan Pertumbuhan X. oryzae pv. oryzae
Supernatan dari pustaka metagenomik yang menunjukkan aktivitas penekanan terhadap X. oryzae pv. oryzae diuji menggunakan teknik peracunan media (Dhingra dan Sinclair 1995). Pengujian dilakukan untuk melihat pengaruh senyawa bioaktif anti patogen yang terkandung di dalam supernatan terhadap laju pertumbuhan X. oryzae pv. oryzae menggunakan teknik peracunan media. Supernatan diperoleh dengan menumbuhkan koloni tunggal pustaka metagenomik pada media LB mengandung 15 µg mL-1 ampisilin yang diinkubasi pada shaker waterbath (100 rpm pada suhu 37 oC). IPTG ditambahkan hingga konsentrasi akhir mencapai 0.1 mM ketika kultur bakteri mencapai nilai OD600=0.55 (fase logaritmik) kemudian diinkubasi kembali pada kondisi yang sama selama 4 hari. Suspensi bakteri disentrifugasi pada kecepatan 10 000 x g selama 20 menit. Supernatan yang diperoleh disaring menggunakan syringe-filter 0.22 µm. Bakteri X. oryzae pv. oryzae 107 cfu ditumbuhkan di dalam media PSB yang mengandung 10% supernatan yang diuji kemudian diinkubasi pada inkubator bergoyang (100 rpm pada suhu ruang). Percobaan ini terdiri atas 4 perlakuan supernatan yang berasal dari biakan klon 52, 72, 185, dan perlakuan kontrol berupa supernatan dari biakan E. coli DH5α mengandung pUC1λ. Masing-masing perlakuan diulang sebanyak 3 kali dan pengukuran nilai OD600 dilakukan sebanyak 3 kali pada setiap ulangan. Rancangan percobaan yang dipergunakan pada pengujian ini adalah Rancangan Acak Lengkap (RAL). Data diolah menggunakan analisis ragam (ANOVA) dengan program Statistical Analysis System (SAS) versi 9.1.3 untuk Windows. Perlakuan yang berpengaruh nyata diuji lanjut dengan uji Beda Nyata Terkecil (BNT) pada taraf nyata α=0.01.
Analisis Potensi Ekstrak Kasar Senyawa Bioaktif dari Klon Pustaka Metagenomik dalam Menekan Pertumbuhan X. oryzae pv. oryzae
Pengujian dilakukan untuk melihat pengaruh ekstrak kasar senyawa bioaktif dari klon pustaka metagenomik dalam menekan pertumbuhan X. oryzae pv. oryzae dan mengetahui jenis pelarut yang tepat untuk mengekstraksi senyawa bioaktif yang diekspresikan oleh DNA sisipan pada klon pustaka metagenomik. Ekstrak kasar senyawa bioaktif diperoleh dari hasil ekstraksi menggunakan pelarut n-heksan, etil asetat, dan kloroform.
21 Filtrat dievaporasi menggunakan rotary evaporator, proses ekstraksi diulang sebanyak 2 kali. Ekstrak kasar senyawa bioaktif yang diperoleh kemudian disimpan pada ruang asam selama 7 hari.
Bioassay dari ekstrak kasar yang diperoleh dilakukan terhadap X. oryzae pv. oryzae menggunakan metode paper disc diffusion assays. Ekstrak kasar senyawa bioaktif disuspensikan dengan aseton hingga konsentrasi 10%. Sebanyak 10 µL suspensi, diteteskan pada paper disc berdiameter 6 mm kemudian dikeringkan selama 2 menit. Kontrol negatif yang digunakan adalah aseton dan kontrol positif berupa media LB yang mengandung ampisilin 50 µg mL-1. Kertas saring dari setiap perlakuan diletakkan diatas media PSA yang mengandung X. oryzae pv. oryzae 108 cfu per 90 mm agar cawan. Inkubasi dilakukan selama 2 hari pada suhu ruang. Pengamatan dilakukan terhadap jari-jari zona bening yang terbentuk sebagai hasil penghambatan pertumbuhan X. oryzae pv. oryzae oleh ekstrak kasar senyawa bioaktif yang diproduksi oleh klon pustaka metagenomik.
22
4 HASIL DAN PEMBAHASAN
Konstruksi Pustaka Metagenomik
Isolasi dan Fragmentasi DNA Sisipan
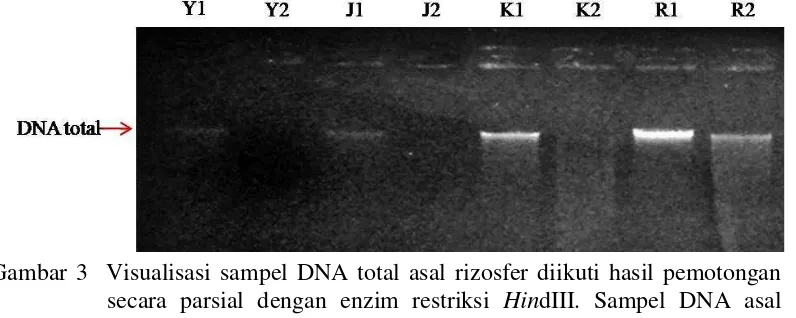
DNA mikrob yang digunakan sebagai sumber genetik diisolasi secara langsung dari sampel rizosfer. Menurut Rondon et al. (2000) dan Sabree et al. (2009), isolasi DNA total dari suatu sampel lingkungan secara langsung memungkinkan didapatkannya sumber genetik baik dari mikrob yang bersifat culturable maupun unculturable. Oleh karena itu, metode isolasi ini digunakan dalam teknik metagenomika baik untuk analisis keragaman mikrob maupun eksplorasi gen fungsional yang bersumber dari keseluruhan DNA genom mikrob pada sampel lingkungan. Gambar 3 menunjukkan hasil visualisasi DNA total dari mikrob asal rizosfer pertanaman padi pada beberapa agroekosistem yang berbeda. Keberhasilan proses isolasi DNA total dari mikrob pada sampel rizosfer ditandai dengan tervisualisasinya pita DNA pada gel agarosa. Menurut Berendsen et al. (2012) dan Spence et al. (2014) interaksi alami antara akar tanaman dan mikrob pada rizosfer sangat penting dalam mendukung kebugaran tanaman, meningkatkan pertumbuhan, dan pengendalian penyakit tanaman. Hal tersebut menunjukkan bahwa rizosfer merupakan salah satu sumber yang baik dalam eksplorasi gen-gen potensial yang terlibat dalam pengendalian hayati patogen tumbuhan.
Fragmentasi DNA hasil isolasi menggunakan enzim restriksi HindIII sebagai DNA sisipan yang dianalisis lebih lanjut juga telah berhascil dilakukan. Pada gambar 3, visualisasi DNA yang telah dipotong secara parsial menunjukkan hasil smear berbeda dengan hasil isolasi awal yang tervisualisasi sebagai pita DNA tunggal. Hal ini menunjukkan bahwa DNA telah terpotong menjadi ukuran yang lebih pendek dengan berbagai ukuran yang berbeda. Menurut Menurut Sambrook dan Russel (2001), fragmen DNA dengan ukuran lebih panjang akan Gambar 3 Visualisasi sampel DNA total asal rizosfer diikuti hasil pemotongan