1254
PROFIL SEBARAN HASIL idock UNTUK KELOMPOK SENYAWA AKTIF,
DECOYS, DAN ZINC-INDUSER TERHADAP PROTEIN PPAR-Gamma
Broto Santoso
Fakultas Farmasi Universitas Muhammadiyah Surakarta Jl. A Yani Pabelan Kartasura Sukoharjo Jawa Tengah 57102
email: Broto.Santoso@ums.ac.id
Abstract
Ligands that can interact strongly with the peroxisome proliferator-activated receptor gamma (PPAR-), proven to provide anti-diabetic effect. Design of new drugs for anti-diabetes can be accelerated using in silico techniques. Selection of the protein is important step to minimize the refraction that can occur and affect the result. This study aimed to determine the distribution profile of docking of PPAR- using idock for Windows. Three groups of compounds used as active ligands sourced from Drug-Bank database; decoys and PPAR- inducer, the last two obtained from ZINC database. Results gained as RF-score (pKd) and idock-score (kcal/mol) converted into value of minus logs and prediction Ki (µM). There were three proteins (4R06, 3FUR and 2Q5P) which can be used as the main target of the search of new anti-diabetic drugs. They had a good profile when their binding affinity was higher to inducers (outlier less than 1.7% of 3567 compounds) and lower against the decoys (outliers more than 4.8% of 747 compounds). In addition, idock-score of their natives were less than -10 kcal/mol indicated that ligands interacted strongly with protein. Two other remaining proteins, 4EMA and 2PRG, had idock-score ratio better than decoys and inducer were 2:23 and 2:15, respectively.
Keywords: PPAR-gamma, idock, decoys, native 1. PENDAHULUAN
Kajian penemuan obat baru merupakan hal yang tidak akan pernah usang. Hal ini dikarenakan beberapa faktor berikut: belum adanya obat yang mumpuni mengobati suatu penyakit terutama yang bersifat degeneratif dan seumur hidup, adanya resistensi baik dari host (pasien) atau vektor atau sumber penyakit, adanya efek samping yang merugikan, kompatibilitas sediaan yang selalu akan mempengaruhi efektivitas dari pengobatan dan masih banyak hal lainnya. Program ini tetap harus dijalankan sebagai langkah pertahanan diri bagi manusia.
Besarnya pembiayaan dalam program rancang obat tersebut menjadi alasan utama mengapa jumlah obat baru yang ditemukan tidak menunjukkan peningkatan bahkan selalu berkurang. Lamanya tahapan suatu obat baru bisa diloloskan menjadi obat yang boleh dipasarkan juga memberikan kontribusi yang signifikan dalam memperlambat penemuan senyawa baru. Adanya teknik in silico, salah
satu langkahnya dapat dipercepat dan dipermudah.
Pemodelan molekular dengan bantuan komputasi masih dalam tataran prediksi. Keakuratan hasilnya masih perlu diuji secara laboratorium baik in vitro atau in vivo. Senyawa decoys (pengumpan) diperlukan dalam docking molekular selain kelompok senyawa aktif. Kelompok senyawa ini, salah satunya dapat dikoleksi melalui data decoys ZINC untuk membuktikan bahwa nilai afinitas ikatan yang diperoleh spesifik dan selektif terhadap protein.
Reseptor peroxisome
proliferator-activated isoform gamma (PPAR-)
merupakan salah satu protein yang bertanggung jawab terhadap peningkatan sensitivitas insulin pada diabetes tipe 2 dan pengobatan hipertrigliseridemia. Isoform gamma PPAR berperan penting dalam adipogenesis dan menjadi protein target dari obat anti-diabetes kelas thiazolidinedione (TZD) (van Marrewijk et al., 2016; Liberato et al., 2012; Motani et al., 2009; Bruning et
1255
al., 2007; Nolte et al., 1998). Interaksi kuat suatu ligan terhadap PPAR- melalui studi docking molekular mengindikasikan bahwa ligan tersebut mempunyai aktivitas sebagai anti-diabetes.
Beberapa program docking yang gratis diantaranya adalah DOCK6, AutoDock, dan Vina. Program terakhir lebih banyak digunakan dalam berbagai modifikasinya, baik untuk kemudahan tampilan antar muka seperti PyRx atau penambahan perhitungan hasil akhir, contohnya adalah idock. Kedua program ini dapat digunakan dalam semua sistem operasi. Program idock dapat digunakan secara online menggunakan ligan yang dikoleksi dari database ZINC. Hasil idock standalone mendekati hasil dari uji laboratorium seperti yang telah disajikan oleh Santoso and Yuliani (2016). Penelitian ini ditujukan untuk mengetahui profil sebaran hasil docking molekular menggunakan idock versi Windows.
2. KAJIAN LITERATUR DAN PENGEMBANGAN HIPOTESIS
Reseptor PPAR- terekspresi sangat kuat pada jaringan adiposa dan makrofag yang menginduksi adipogenesis. Ligan endogen PPAR- terbukti sebagai regulator penting dalam adipogenesis. Kelompok senyawa TZD dapat mengaktivasi reseptor dan sensitisasi jaringan insulin dapat terjadi (Nolte et al., 1998).
Bruning et al. (2007) menyatakan bahwa stabilisasi helix 12 (H12) dalam proses aktivasi PPAR- diperlukan ligan yang bersifat agonis penuh bukan sebagian atau intermediat. Stabilisasi terkait dengan efek samping dari analog TZD yang berbeda dibandingkan dengan TZD. Mekanisme aktivasi melalui H12 memberikan perubahan interaksi ligan-reseptor berakibat perbedaan efek farmakologisnya.
Ligan selektif poten, INT131, terbukti memberikan stimulasi rendah pada diferensiasi jaringan adiposa dan dapat mengaktivasi parsial PPAR-. Secara bersamaan, senyawa ini menghasilkan aktivitas agonis yang berpengaruh secara langsung terhadap sensitisasi insulin (Motani et al., 2009).Asam lemak rantai medium telah
diteliti dapat menjadi aktivator bahkan agonis sebagian bagi PPAR- (Liberato et al., 2012). Senyawa sintetik SR2067, suatu agonis parsial berhasil menginformasikan bahwa ligan PPAR- mempunyai sidik jari struktur dan kinetika unik yang menjanjikan penurunan kejadian efek samping obat dibadnigkan kelompok agonis penuh (van Marrewijk et al., 2016).
3. METODE PENELITIAN
Penelitian telah dilakukan menggunakan komputer dengan sistem operasi Windows versi 64bit. Protein target, 4EMA, 4R06, 3FUR, 2Q5P dan 2PRG, merupakan PPAR- yang dikoleksi dengan cara mengunduhnya dari www.rcsb.org. Terdapat tiga kelompok senyawa yang digunakan sebagai ligan uji, yaitu kelompok aktif yang bersumber dari
database drugbank (www.drugbank.ca),
kelompok senyawa decoys (747 senyawa) dan kelompok induser PPAR- (3567 senyawa), keduanya berasal dari database ZINC pada laman dude.docking.org dan zinc.docking.org. Tahap preparasi dan konversi protein dan ligan dilakukan menggunakan program Chimera, JChem for Excel dan OpenBabel.
Docking molekular telah dilakukan
menggunakan idock versi 2.2.1 untuk Windows yang dapat diunduh secara gratis pada
https://github.com/HongjianLi/idock/releases. Beberapa contoh visualisasi dari interaksi ligan-protein dihasilkan menggunakan PLIP versi online (http://projects.biotec.tu-dresden.de/plip-web), PyMOL dan poseview
server versi online
(http://proteinsplus.zbh.uni-hamburg.de/). Data yang diperoleh berupa idock-score (kkal/mol) yang dikonversi menjadi nilai minus log dan prediksi Ki (pKi; µM) serta nilai RF-score (pKd; prediksi tetapan disosiasi). Analisis data dikerjakan dengan menggunakan bantuan program Microsoft Excel.
4. HASIL DAN PEMBAHASAN
Nilai idock-score tertinggi ligan native diperoleh pada ID 3FUR. Dua nilai yang terendah terdapat pada native dari 4EMA dan 2PRG sehingga ditemukan ada senyawa aktif
1256 yang mampu melampaui nilai ini (Tabel 1). Termasuk 3FUR, 4R06 dan 2Q5P juga menunjukkan bahwa tidak ada senyawa aktif yang diuji memiliki nilai idock-score melebihi
native. Fenomena ini mendorong peningkatan selektifitas ligan terhadap protein target dalam penemuan senyawa baru.
Tabel 1. Hasil docking ligan native dan kelompok senyawa aktif terhadap kelima protein target Kode PDB Resolusi X-Ray (Å) native idS* (kkal/mol) RMSD
active idS* (kkal/mol)
DOI Number lowest highest 4EMA 2,54 -8,46 1,126 -6,87 -10,77 10.2210/pdb4ema/pdb 4R06 2,22 -11,40 0,155 -7,29 -11,40 10.2210/pdb4r06/pdb 3FUR 2,30 -11,67 0,333 -6,40 -11,67 10.2210/pdb3fur/pdb 2Q5P 2,30 -10,38 0,281 -6,96 -10,38 10.2210/pdb2q5p/pdb 2PRG 2,30 -8,62 1,606 -6,64 -10,97 10.2210/pdb2prg/pdb *idS = idock-Score
Terdapat senyawa aktif yang memiliki nilai idock-score untuk semua protein target berada di daerah abu-abu untuk algoritma turunan vina, yaitu pada rentang [-7,3]-[-6,4] kkal/mol. Arti abu-abu adalah masih belum pasti karena rasio afinitas ikatan ligan-protein kuat terjadi atau tidak adalah 1:1. Validasi hasil docking native terhadap ligan kristalnya masih berada di nilai root-mean-square deviation (RMSD) yang diperkenankan.
Gambar 1. Hasil pencarian binding site pocket (BSP) untuk protein 2PRG. Letak BSP ditunjukkan pada area dengan warna yang berbeda dan native berada pada BSP dengan volume terbesar, 1889,92 Å (jingga).
Proses ini harus dilakukan untuk menguji besaran gridbox perhitungan untuk docking dan pusat massa terpilih yang umumnya berdasarkan pusat massa native. Besaran nilai
gridbox ditetapkan dengan memperhatikan volume dari binding site pocket (BSP) protein, contoh dapat dilihat pada Gambar 1. Umumnya native berada pada volume BSP yang terbesar dalam protein target.
Gambar 2. Konformasi native 2PRG hasil docking (hijau) disandingkan dengan hasil pengukuran kristalografi (ungu) dengan RMSD sebesar 1,606.
Contoh hasil validasi proses docking 2PRG seperti yang diperlihatkan pada Gambar 2. Kesejajaran konformasi native hasil docking terhadap konformasi native hasil pengukuran kristalografi. Letak 3-Dimensi dari ligan native menggambarkan interaksi yang terjadi dengan residu protein. Kedekatan konformasi kedua konformasi mengarahkan bahwa keduanya memiliki interaksi ligan-protein yang mirip dan menunjukkan bahwa sistem docking terpilih sudah tepat.
Penelitian sebelumnya dengan program DOCK6 berhasil memetakan batas maksimal RMSD yang masih diperbolehkan, yaitu kurang dari 5 (lima). Ada beberapa yang membatasinya pada angka kurang dari 2,0. Kelima native setiap protein berada di bawah kedua batasan tersebut. Sistem docking
1257
terpilih dapat digunakan untuk docking ligan lainnya pada kelima protein target.
A. B.
Gambar 3. Sebaran nilai idS (ordinat kiri, hasil terletak di bawah) dan pKd (ordinat kanan, atas) untuk kelompok senyawa aktif (grafik atas), decoys (A) dan ZINC-induser untuk PPAR- (B).
Distribusi nilai idS dan pKd pada Gambar 3 menyatakan bahwa tidak ada perbedaan berarti dari sebaran ketiga kelompok. Terdapat 2 protein target, 4EMA dan 2PRG yang memiliki jumlah senyawa dengan idS melampaui native. Hal ini baik untuk kelompok aktif dan ZINC-induser tapi tidak bagus jika dikaitkan dengan decoys.
Secara berurutan, ditemukan sebesar 24,5 dan 43,1% dari 747 senyawa decoys dengan idS lebih baik dari native 4EMA dan 2PRG. Sebaliknya, tidak semua senyawa ZINC-induser berhasil mempunyai idS mengungguli native, yaitu secara berurut 59,0 dan 67,7% dari 3567 senyawa. Dalam penemuan senyawa baru, penggunaan sistem pembanding seharusnya memiliki komposisi senyawa aktif (ZINC-induser) berbanding senyawa decoys pada bilangan 1:10. Idealnya, jika digunakan 100 senyawa aktif berarti
harus ada 1000 senyawa decoys yang representatif.
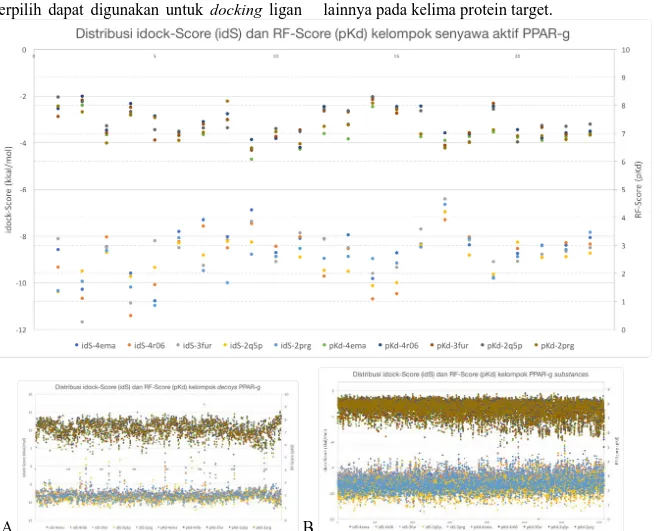
Berbeda dengan ketiga protein target lainnya, tidak diperoleh idS melebihi dari 7% (decoys) dan 8,7% (ZINC-induser). Bahkan untuk protein 3FUR, nilai idS tertinggi hanya dimiliki oleh native. Perhitungan lanjutan dari nilai idS pun, log minus idS dan pKi, tidak memberikan profil sebaran yang lebih baik seperti yang terlihat pada Gambar 4.
Hasil ini menunjukkan area untuk memilih senyawa aktif pembanding yang terletak di bagian antara kedua sebaran nilai. Senyawa decoys dapat dipilih pada bagian terbawah atau teratas dari profil. Terkait dengan penemuan senyawa obat baru yang diarahkan untuk mengobati diabetes melalui sensititasi insulin dengan jalur aktivasi PPAR- direkomendasikan menggunakan 4R06, 3FUR dan 2Q5P sebagai protein target.
1258 Ketiganya memiliki senyawa outlier (di luar populasi) berdasarkan hasil docking berurutan untuk kelompok decoys dan ZINC-induser adalah lebih dari 4,8% dan kurang dari 1,7%.
Rasio idS antara decoys dan induser untuk protein 4EMA dan 2PRG adalah 0,087 dan 0,133. Nilai ini dikatakan lebih baik dikarenakan terdapat banyak senyawa pilihan baik dari kelompok decoys dan induser yang
bisa dianalisis untuk dijadikan sebagai senyawa penuntun. Alasan lainnya adalah bahwa residu yang berperan aktif dalam perhitungan docking atau interaksi ligan-protein dapat diketahui. Hal ini diperoleh dengan memanfaatkan program PLIP atau program pencarian interaksi ligan-protein lainnya.
A. B.
Gambar 4. Sebaran nilai log[(-)idS] (ordinat kiri, hasil terletak di bawah) dan pKi (ordinat kanan, atas) untuk kelompok senyawa aktif (grafik atas), decoys (A) dan ZINC-induser untuk PPAR- (B).
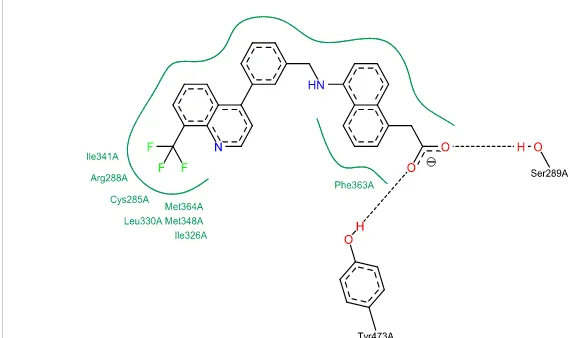
Analisis lanjutan terhadap kelompok senyawa induser diperoleh 50 senyawa yang ternyata memiliki idS melebihi -9,5 kkal/mol terhadap kelima protein target dengan profil interaksi ligan-protein dua contoh senyawa terpilih seperti pada Gambar 5-6. Keduanya menunjukkan bahwa interaksi didominasi oleh banyaknya interaksi hidrofobik. Terdapat 8-10 residu yang terlibat. Hal yang menjadi perhatian adalah senyawa calon obat baru sebaiknya dirancang dengan jumlah dan jarak antara penyumbang interaksi hidrofobik yang mirip. Faktor lain yang harus dipertimbangkan
adalah jumlahnya, sehingga menjadikan senyawa baru tersebut tidak memenuhi prinsip ADME.
1259 Gambar 5. Interaksi 2D ligan-protein untuk ZINC-induser ke-1012 dengan protein 4EMA.
Gambar 6. Interaksi 2D ligan-protein untuk ZINC-induser ke-2864 dengan protein 4R06.
Senyawa induser ke-2864 merupakan satu-satunya senyawa yang memiliki afinitas ikatan pada semua protein target. Nilai idS senyawa ini adalah -11,41 kkal/mol, melebihi native 4R06. Sepuluh residu protein 4R06 yang berperan dalam interaksi hidrofobik dengan ligan adalah leusin (330; 453; 465; 469), isoleusin (326; 341), glutamin (286), tirosin (327), arginin (288), dan serin (342). 5. KESIMPULAN
Terdapat tiga protein dengan kode 4R06, 3FUR dan 2Q5P yang dapat dijadikan target utama pencarian obat baru anti-diabetes. Profil ketiganya memiliki afinitas ikatan outlier tinggi untuk kelompok induser kurang dari 1,7% dan rendah terhadap kelompok decoys (lebih dari 4,8%). Selain itu, idock-score untuk native yang dimiliki ketiganya kurang dari -10 kkal/mol yang berarti afinitas native terjadi sangat kuat. Dua protein lainnya, 4EMA dan 2PRG, memiliki rasio nilai idock-score lebih baik dari native untuk decoys-induser berurutan adalah 2:23 dan 2:15.
Berman, H.M., Westbrook, J., Feng, Z., Gilliland, G., Bhat, T.N., Weissig, H., Shindyalov, I.N., and Bourne, P.E. (2000). The Protein Data Bank. Nucleic Acids Res., 28(1):235-242. DOI: 10.1093/nar/28.1.235.
Bruning, J.B., Chalmers, M.J., Prasad, S., Bushby, S.A., Kamenecka, T.A., He, Y., Nettles, K.W., and Griffin, P.R. (2007). Partial agonists activate PPAR gamma using a helix12 independent mechanism.
Structure, 15:1258. DOI:
10.1016/j.str.2007.07.014.
JChem for Excel 16.5.1600, 2016, ChemAxon (http://www.chemaxon.com).
Li, H., Leung, K.S. and Wong, M.H. (2012). idock: A Multithreaded Virtual Screening Tool for Flexible Ligand Docking. IEEE Symposium on Computational Intelligence in Bioinformatics and Computational Biology (CIBCB), pp.77-84, San Diego, United States, 9-12 May 2012. DOI: 10.1109/CIBCB.2012.6217214.
Liberato, M.V., Nascimento, A.S., Ayers, S.D., Lin, J.Z., Cvoro, A., Silveira, R.L., Martínez, L., Souza, P.C.T., Saidemberg, D., Deng, T., Amato, A.A., Togashi, M., Hsueh, W.A., Phillips, K. Palma, M.S., Neves, F.A.R., Skaf, M.S., Webb, P., and Polikarpov, I. (2012). Medium Chain Fatty Acids Are Selective Peroxisome Proliferator Activated Receptor (PPAR) Activators and Pan-PPAR Partial Agonists. PLoS ONE 7(5):e36297. DOI: 10.1371/journal.pone.0036297.
Motani, A., Wang, Z., Weiszmann, J., McGee L.R., Lee, G., Liu, Q., Staunton, J., Fang, Z., Fuentes, H., Lindstrom, M., Liu, J., Biermann, D.H., Jaen, J., Walker, N.P., Learned, R.M., Chen, J.L., and Li, Y. (2009). INT131: a selective modulator of PPAR gamma. J Mol Biol. 386(5) 1301-1311. DOI: 10.1016/j.jmb.2009.01.025. Mysinger, M.M., Carchia, M., Irwin, J.J., and
Brian K. Shoichet, B.K. (2012). Directory of Useful Decoys, Enhanced (DUD-E): Better Ligands and Decoys for Better
1260 Benchmarking. J. Medicinal Chemistry, 55 (14):6582-6594. DOI: 10.1021/jm300687e. Nolte, R.T., Wisely, G.B., Westin, S., Cobb, J.E., Lambert, M.H., Kurokawa, R., Rosenfeld, M.G., Willson, T.M., Glass, C.K., and Milburn, M.V. (1998). Ligand binding and co-activator assembly of the peroxisome proliferator-activated receptor-. Nature, 395(6698):137-143. DOI: 10.1038/25931.
O'Boyle, N.M., Banck, M., James, C.A., Morley, C., Vandermeersch, T., and Hutchison, G.R. (2011). Open Babel: An open chemical toolbox. J.
Cheminformatics, 3:33. DOI:
10.1186/1758-2946-3-33.
Pettersen, E.F., Goddard, T.D., Huang, C.C., Couch, G.S., Greenblatt, D.M., Meng, E.C., and Ferrin, T.E. (2004). UCSF Chimera--a visualization system for exploratory research and analysis. J. Comput. Chem., 25(13):1605-12. DOI: 10.1002/jcc.20084.
Salentin, S., Schreiber, S. Haupt, V.J., Adasme, M.F., and Schroeder, M. (2015). PLIP: Fully Automated Protein-Ligand Interaction Profiler. Nucl. Acids Res., 43(W1): W443-W447. DOI: 10.1093/nar/gkv315.
Santoso, B. and Yuliani, R. (2016). Program idock dalam Skrining Virtual Berbasis Struktur Senyawa Alam sebagai Inhibitor Enzim Ligase Ala dan D-Ala-D-Lac. Disampaikan pada Seminar Nasional Perkembangan Anti-Infeksi: dari Bahan
Alam hingga Layanan Kefarmasian.
Fakultas Farmasi UGM, 10 Desember 2016, Gedung Grha Sabha Pramana Lantai I, Bulaksumur, Yogyakarta.
Sterling, T. and Irwin, J.J. (2015). ZINC 15 – Ligand Discovery for Everyone. J. Chemical Information and Modeling, 55 (11), 2324-2337. DOI: 10.1021/acs.jcim.5b00559.
Stierand, K. and Rarey, M. (2010). Drawing the PDB-Protein-Ligand Complexes in two Dimensions. Medicinal Chemistry Letters, 1(9):540-545. DOI: 10.1021/ml100164p. The PyMOL Molecular Graphics System,
Version 1.8.4 Schrödinger, LLC. Open Source version.
van Marrewijk, L.M., Polyak, S.W., Hijnen, M., Kuruvilla, D., Chang, M.R., Shin, Y., Kamenecka, T.M., Griffin, P.R., and John B. Bruning, J.B. (2016). ACS Chemical
Biology 11(1):273-283. DOI:
10.1021/acschembio. 5b00580.
A. Volkamer, D. Kuhn, T. Grombacher, F. Rippmann, M. Rarey. (2012). Combining global and local measures for structure-based druggability predictions. J. Chem.
Inf. Model, 52:360-372. DOI:
10.1021/ci200454v.
Wishart, D.S., Knox, C., Guo, A.C., Shrivastava, S., Hassanali, M., Stothard, P., Chang, Z., and Woolsey, J. (2006). DrugBank: a comprehensive resource for in silico drug discovery and exploration.
Nucleic Acids Res., 34 (Database

![Gambar 4. Sebaran nilai log[(-)idS] (ordinat kiri, hasil terletak di bawah) dan pKi (ordinat kanan, atas) untuk kelompok senyawa aktif (grafik atas), decoys (A) dan ZINC-induser untuk PPAR- (B).](https://thumb-ap.123doks.com/thumbv2/123dok/2414429.2207751/5.893.134.785.298.801/gambar-sebaran-ordinat-terletak-ordinat-kelompok-senyawa-induser.webp)