DAFTAR PUSTAKA
1. Ballschmiter, M., Futterer, O., Liebl, W. (2006), Identification and characterization of a novel intracellular alkaline α-amylase from the hyperthermophilic bacterium Thermotoga maritima MSB8, Appl. Environ. Microbiol., 72, 2206-2211
2. Bentley, I.S. and Williams, E.C. (1996), Industrial applications, dalam Industrial Enzymology, Godfrey, T., West, S., Editor, Stockton Press, New York, 2, 341, 345.
3. Boyer, R. (2000), Modern Experimental Biochemistry, Addison Wesley Longman, San Fransisco, 3, 119.
4. Brock, T. D., Madigan, M. T., Martinko, J. M. (1994), Biology of Microorganisms, Prentice-Hall International Inc., London, 7.
5. Chandrasekaran, M. (1997), Industrial enzymes from marine microorganisms: The Indian scenario, J. Mar. Biotechnol., 5, 86-89.
6. Farmer III, J. J. and Brenner, F. W. H. (2006), The genera Vibrio and Photobacterium, dalam Prokaryotes, Bab 3.3.18, Dworkin, M., Falkow, S., Rosenberg, E., Schleifer, K.H., Stackebrandt, E., Editor, Springer, Singapura,
6, 508-563.
7. Feller, G., Lonhienne, T., Deroanne, C., Van Beeumen, J., Gerday, C. (1992), Purification, characterization and nucleotide sequence of the thermolabile α-amylase from the Antarctic psychrotroph Alteromonas haloplanctis A23, J. Biol. Chem., 267, 5217-5221.
8. Godfrey, T. and West, S. (1996), Industrial applications, dalam Industrial Enzymology, Bab 2, Godfrey, T., West, S., Editor, Stockton Press, New York,
2, 91, 105-131, 192, 339-356, 361-371.
9. Goyal, N., Gupta, J.K., Soni, S.K. (2005), A novel raw-starch digesting thermostable α-amylase from Bacillus sp. I-3 and its use in the direct hydrolysis of raw potato starch, Enzyme and Microbial Tech., 37, 723-734.
10. Gupta, R., Gigras, P., Mohapatra, H., Goswami, V.K., Chauhan, B. (2003), Microbial α-amylases: a biotechnological perspective, Process Biochem., 38, 1599-1616.
11. Guzman-Maldonado, H. and Paredes-Lopez, O. (1995), Amylolytic enzymes and products derived from starch, Crit. Rev. Food Sci. Nutr., 35, 373–403.
12. Hormansdorfer, S., Wentaes, H., Neugebaur-Buchler K., Bauer, J. (2000), Isolation of Vibrio alginolyticus from seawater aquaria, Int. J. Hyg. Environ. Health, 203, 169-175.
13. Henrissat, B. (1991), A classification of glycosyl hydrolases based on amino acid sequence similarities, Biochem. J., 280, 309-316.
14. Ichige A., Oishi, K., Mizushima, S. (1988), Isolation and characterization of mutants of a marine Vibrio strain that are defective in production of extracellular proteins, J. Bacteriol., 170, 3537-3542.
15. Judoamidjojo, M., Darwis, A. A., Sa’id, E. G. (1990), Teknologi Fermentasi, PAU Bioteknologi IPB, Bogor.
16. Kandra, L. (2003), α-Amylases of medical and industrial importance, Theochem, 666-667, 487-498.
17. Kelman, D., Kashman, Y., Rosenberg, E., Kushmaro, A., Loya, Y. (2006), Antimicrobial activity of Red Sea Corals, Marine Biology, 149, 357-363.
18. Kim, U. O., Hahm, K. S., Park, Y. H., Kim, Y. J. (1999), CAMP-mediated catabolite repression and electrochemical potential-dependent production of an extracellular amylase in Vibrio alginolyticus, Biosci. Biotechnol. Biochem., 63, 288-292.
19. Koolman, J. and Roehm, K. H. (2005), Color Atlas of Biochemistry, Thieme, New York, 2, 42.
20. Koshland, D.E. (1953), Stereochemistry and the mechanism of enzymatic reactions, Biol. Rev., 28, 416–436.
21. Kuriki, T. and Imanaka, T. (1999), The concept of the α-amylase family: Structural similarity and common catalytic mechanism, J. Biosci. Bioeng., 87, 557-565.
22. Machovic, M. and Janecek, S. (2007), Amylolytic enzymes: types, structures and specificities, dalam Industrial Enzymes: Structure, Function and Applications, Bab 1, Polaina, J., MacCabe, A. P., Editor, Springer, Netherlands, 1, 3-18.
23. MacGregor, E.A. (2005), An overview of clan GH-H and distantly-related families, Biologia Bratislava, 60, 5-12.
24. Martinez, J. M. H., Schoenmakers, P. J., Kok, W. Th. (2004), Determination of the amylose–amylopectin ratio of starches by iodine-affinity capillary electrophoresis, J. Chromatogr. A., 1053, 227-234.
25. Mitsuiki , S., Mukae, K., Sakai, M., Goto, M., Hayashida, S., Furukawa, K. (2005), Comparative characterization of raw starch hydrolyzing α-amylase from various Bacillus strains, Enzyme and Microbial Tech., 37, 410-416.
26. Najafi, M. F., Deobagkar, D., Deobagkar, D. (2005), Purification and characterization of an extracellular α-amylase from Bacillus subtilis AX20, Protein Expression and Purification, 41, 349-354.
27. Najafi, M. F. and Kembhavi, A. (2005), One step purification and characterization of an extracellular α-amylase from marine Vibrio sp., Enzyme and Microbial Technology, 36, 535-539.
28. Nielsen, J. E., Borchert, T. V., Vriend, G. (2001), The determinants of α-amylase pH-activity profiles, Prot. Eng., 14, 505-512.
29. Pandey, A., Nigam, P., Soccol, C.R., Soccol, V.T., Singh, D., Mohan, R. (2000), Advances in microbial amylases, Biotechnol. Appl. Biochem., 31, 135-152.
30. Ratcliffe, C., Sanders, R. L., Tittel, L., O’Brien, R. W. (1982), Protease secretion by the marine bacterium Vibrio gazogenes, J. Biol. Sci., 35, 457-467.
31. Rohwer, F., Seguritan, V., Azam, F., Knowlton, N. (2002), Diversity and distribution of coral-associated bacteria, Marine Ecology Progress Series, 243, 1-10.
32. Sambrook, J. and Russel, D. W. (2001), Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, New York, 3, A8.46-47.
33. Saramma, A.V. and Babu, P. (1994), Regulation of α-amylase biosynthesis in Vibrio alginolyticus (MB1), Indian J. Exp. Biol., 32, 515-516.
34. Siddiqui, K.S., Poljak, A., Guilhaus, M., Feller, G., D’Amico, S., Gerday, C., Cavicchioli, R. (2005), Role of disulfide bridges in the activity and stability of a cold-active α-amylase, J. Bacteriol. 187, 6206-6212.
35. Sivaramakrishnan, S., Gangadharan, D., Madhavan K., Nampoothiri, Soccol, C.R., Pandey, A. (2006), α-Amylases from Microbial Sources – An Overview on Recent Developments, Food Technol. Biotechnol., 44, 173-184.
36. Tolan, J.S. (1996), Industrial applications, dalam Industrial Enzymology, Godfrey, T., West, S., Editor, Stockton Press, New York, 2, 336-337.
37. Uitdehaag, J.C.M., Mosi, R., Kalk, K.H., van der Veen, B.A., Dijkhuizen, L., Withers, S.G., Dijkstra, B.W. (1999), X-ray structures along the reaction pathway of cyclodextrin glycosyltransferase elucidate catalysis in the α-amylase family, Nature Struct. Biol., 6, 432–436.
38. Van der Maarel, M.J.E.C., van der Veen, B., Uitdehaag, J.C.M., Leemhuis, H., Dijkhuizen, L. (2002), Properties and applications of starch-converting enzymes of the α-amylase family, J. Biotechnol. 94, 137-155.
39. Wanderley, K.J., Torres, F.A.G., Moraes, L.M.P., Ulhoa, C.J. (2004), Biochemical characterization of α-amylase from the yeast Cryptococcus flavus. FEMS Microbiology Letters, 231, 165-169.
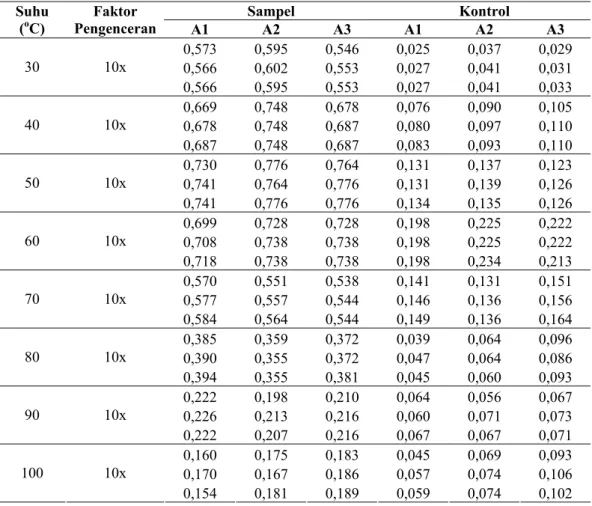
Lampiran 1 Kurva Standar Glukosa
Kurva standar glukosa dibuat dari larutan stok glukosa 0,5% (v/v). 100 µL sampel yang berisi larutan glukosa pada berbagai konsentrasi direaksikan dengan 1 mL reagen DNS, diinkubasi pada 100oC selama 10 menit dan kemudian didinginkan pada suhu ruang selama 20 menit. Setelah itu sampel diukur absorbansinya pada λ 500 nm. Sebagai blanko digunakan 100 µL aqua milipore.
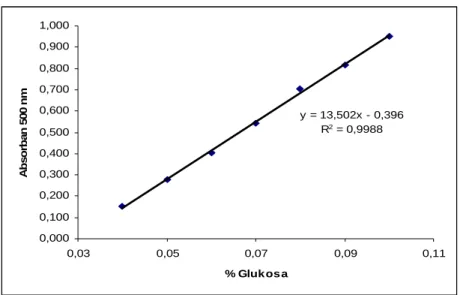
Kurva standar glukosa dibuat untuk tiap reagen DNS yang baru. Berikut adalah contoh dari salah satu kurva standar yang digunakan di dalam penelitian ini.
Tabel L.1. Absorban standar glukosa.
% (w/v) glukosa A1 A2 A3 Rerata A 0,04 0,05 0,06 0,07 0,08 0,09 0,10 0,145 0,277 0,403 0,536 0,698 0,810 0,940 0,155 0,271 0,398 0,542 0,703 0,816 0,952 0,153 0,283 0,406 0,550 0,710 0,823 0,961 0,151 0,277 0,402 0,543 0,704 0,816 0,951
Kurva standar glukosa dari data diatas diberikan sebagai berikut.
y = 13,502x - 0,396 R2 = 0,9988 0,000 0,100 0,200 0,300 0,400 0,500 0,600 0,700 0,800 0,900 1,000 0,03 0,05 0,07 0,09 0,11 % Gluk os a A b so rb a n 50 0 n m
Lampiran 2 Langkah Perhitungan Aktivitas α-Amilase
Perhitungan aktivitas α-amilase dilakukan sebagai berikut.
1. Gram gula pereduksi per mL yang dihasilkan dari reaksi enzimatis diatas didapatkan dengan mengintrapolasikan selisih absorban sampel dengan absorban kontrol yang berada dalam kisaran absorban standar glukosa ke dalam persamaan regresi linier kurva standar glukosa, kemudian mengalikannya dengan faktor pengenceran dan membaginya dengan 100 mL. 2. Selanjutnya gram gula pereduksi per mL dikonversi ke mikromol per mL.
Setelah itu mikromol gula pereduksi per mL dikalikan dengan volume reaksi uji dan dibagi dengan volume enzim yang digunakan dalam reaksi uji.
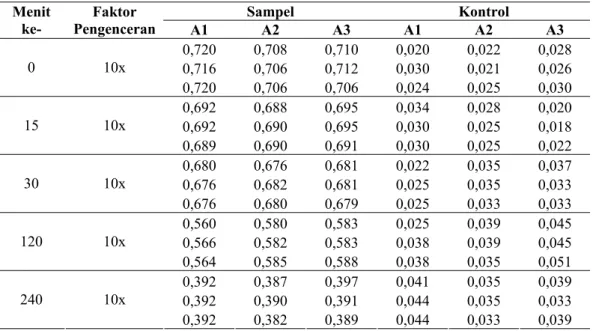
Lampiran 3 Kurva Standar BSA
Kurva standar BSA dibuat dari larutan stok BSA 0,1% (w/v). 800 µL sampel yang berisi larutan BSA pada berbagai konsentrasi direaksikan dengan 200 µL reagen Bradford dan didiamkan pada suhu ruang selama 10 menit. Setelah itu, sampel diukur absorbansinya pada λ 595 nm. Sebagai blanko digunakan 800 µL aqua milipore.
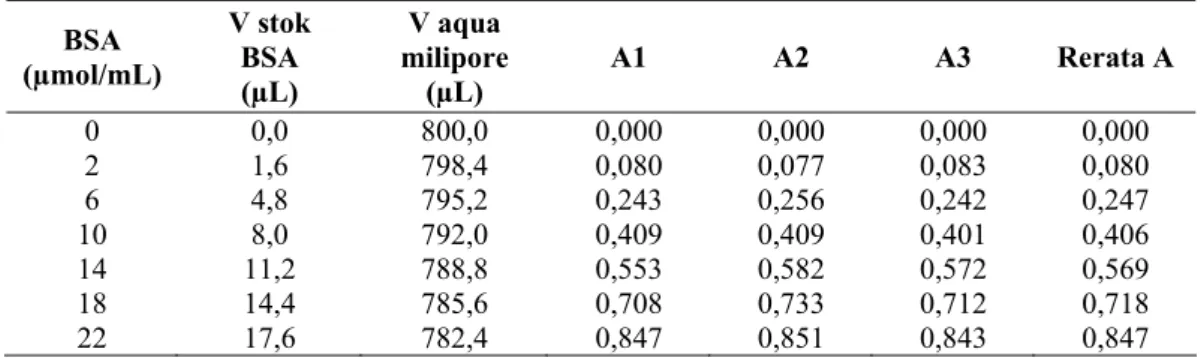
Tabel L.2. Absorban standar BSA.
BSA (µmol/mL) V stok BSA (µL) V aqua milipore (µL) A1 A2 A3 Rerata A 0 0,0 800,0 0,000 0,000 0,000 0,000 2 1,6 798,4 0,080 0,077 0,083 0,080 6 4,8 795,2 0,243 0,256 0,242 0,247 10 8,0 792,0 0,409 0,409 0,401 0,406 14 11,2 788,8 0,553 0,582 0,572 0,569 18 14,4 785,6 0,708 0,733 0,712 0,718 22 17,6 782,4 0,847 0,851 0,843 0,847
Kurva standar BSA dari data diatas diberikan berikut.
y = 0,039x + 0,0084 R2 = 0,9986 0,000 0,100 0,200 0,300 0,400 0,500 0,600 0,700 0,800 0,900 1,000 0 5 10 15 20 25
Konsentrasi BSA (m ikrogram /m L)
Ab so rb an s i 595 n m
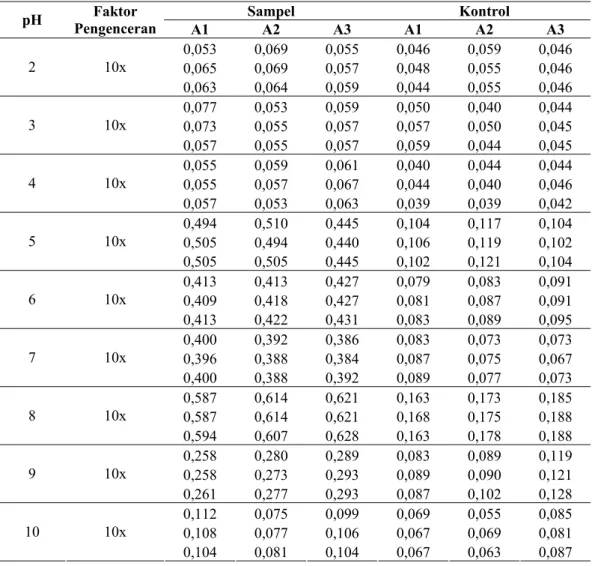
Lampiran 4 Data OD Sel Kurva Pertumbuhan Vibrio sp. SFNB 3 dan Uji DNS Aktivitas α-Amilase
Tabel L.3. Data OD sel kurva pertumbuhan Vibrio sp. SFNB 3.
Jam ke- Pengenceran Faktor OD1 OD2 OD3 Rerata OD Standar Deviasi
0 1x 0,000 0,000 0,000 0,000 0,000 2 1x 0,210 0,214 0,215 0,213 0,003 4 2x 1,554 1,554 1,554 1,554 0,000 6 5x 2,415 2,425 2,525 2,455 0,061 8 8x 2,472 2,520 2,504 2,499 0,024 10 8x 2,048 2,040 2,120 2,069 0,044 12 8x 1,888 1,920 1,952 1,920 0,032 14 8x 1,712 1,744 1,752 1,736 0,021 16 8x 1,744 1,760 1,856 1,787 0,061 24 8x 1,792 1,792 1,864 1,816 0,042
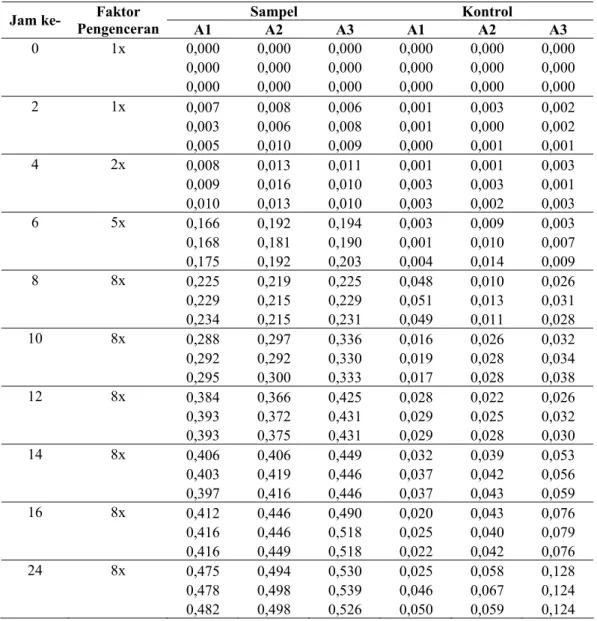
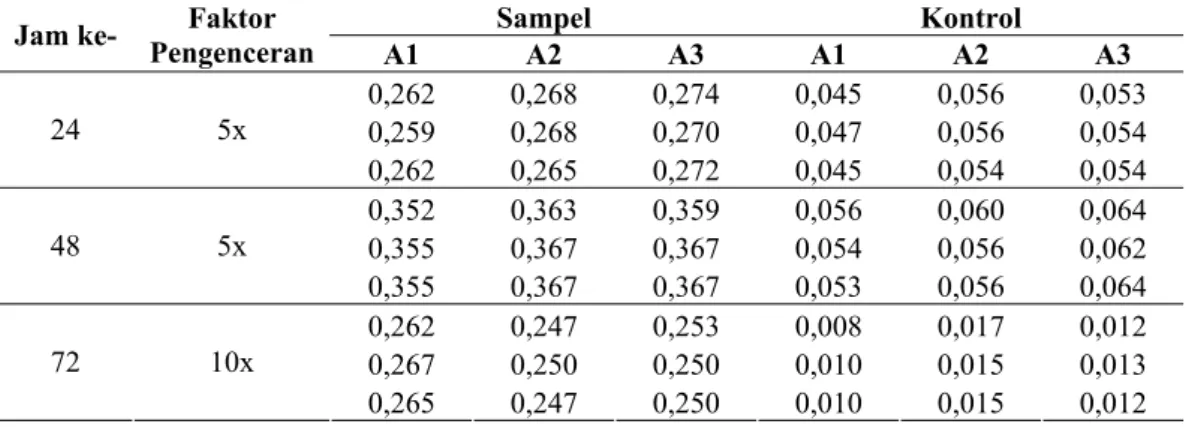
Tabel L.4. Data uji DNS aktivitas α-amilase Vibrio sp. SFNB 3.
Sampel Kontrol
Jam ke- Pengenceran Faktor A1 A2 A3 A1 A2 A3
0 1x 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 2 1x 0,007 0,008 0,006 0,001 0,003 0,002 0,003 0,006 0,008 0,001 0,000 0,002 0,005 0,010 0,009 0,000 0,001 0,001 4 2x 0,008 0,013 0,011 0,001 0,001 0,003 0,009 0,016 0,010 0,003 0,003 0,001 0,010 0,013 0,010 0,003 0,002 0,003 6 5x 0,166 0,192 0,194 0,003 0,009 0,003 0,168 0,181 0,190 0,001 0,010 0,007 0,175 0,192 0,203 0,004 0,014 0,009 8 8x 0,225 0,219 0,225 0,048 0,010 0,026 0,229 0,215 0,229 0,051 0,013 0,031 0,234 0,215 0,231 0,049 0,011 0,028 10 8x 0,288 0,297 0,336 0,016 0,026 0,032 0,292 0,292 0,330 0,019 0,028 0,034 0,295 0,300 0,333 0,017 0,028 0,038 12 8x 0,384 0,366 0,425 0,028 0,022 0,026 0,393 0,372 0,431 0,029 0,025 0,032 0,393 0,375 0,431 0,029 0,028 0,030 14 8x 0,406 0,406 0,449 0,032 0,039 0,053 0,403 0,419 0,446 0,037 0,042 0,056 0,397 0,416 0,446 0,037 0,043 0,059 16 8x 0,412 0,446 0,490 0,020 0,043 0,076 0,416 0,446 0,518 0,025 0,040 0,079 0,416 0,449 0,518 0,022 0,042 0,076 24 8x 0,475 0,494 0,530 0,025 0,058 0,128 0,478 0,498 0,539 0,046 0,067 0,124 0,482 0,498 0,526 0,050 0,059 0,124
Lampiran 5 Data Uji DNS dan Uji Bradford α-Amilase Ekstrak Kasar dan Hasil Isolasi dari Vibrio sp. SFNB 3
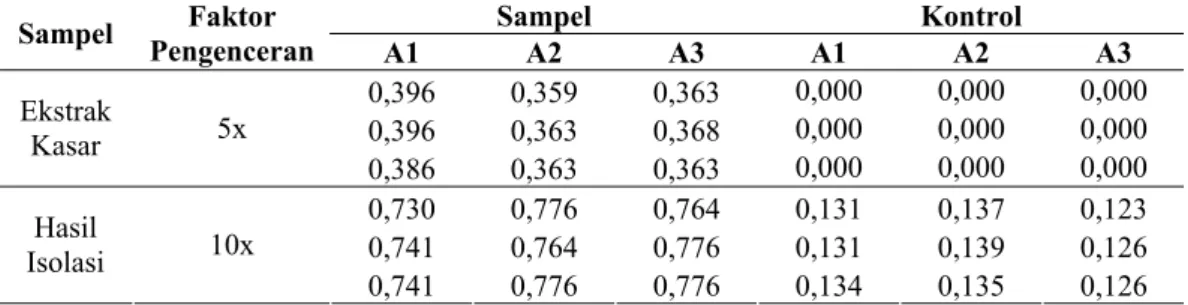
Tabel L.5. Data uji DNS α-amilase ekstrak kasar dan hasil isolasi dari Vibrio sp. SFNB 3.
Sampel Kontrol
Sampel Pengenceran Faktor A1 A2 A3 A1 A2 A3
0,396 0,359 0,363 0,000 0,000 0,000 0,396 0,363 0,368 0,000 0,000 0,000 Ekstrak Kasar 5x 0,386 0,363 0,363 0,000 0,000 0,000 0,730 0,776 0,764 0,131 0,137 0,123 0,741 0,764 0,776 0,131 0,139 0,126 Hasil Isolasi 10x 0,741 0,776 0,776 0,134 0,135 0,126
Tabel L.6. Data uji Bradford α-amilase ekstrak kasar dan hasil isolasi dari Vibrio sp. SFNB 3.
Sampel A1 A2 A3 Rerata A mikrogram/mL mikrogram
Ekstrak Kasar 0,153 0,146 0,160 0,153 3,708 1854
Lampiran 6 Data Uji DNS α-Amilase Hasil Isolasi dari Vibrio sp. SFNB 3 pada Berbagai Suhu
Tabel L.7. Data uji DNS α-amilase hasil isolasi dari Vibrio sp. SFNB 3 pada berbagai suhu. Sampel Kontrol Suhu (oC) Pengenceran Faktor A1 A2 A3 A1 A2 A3 0,573 0,595 0,546 0,025 0,037 0,029 0,566 0,602 0,553 0,027 0,041 0,031 30 10x 0,566 0,595 0,553 0,027 0,041 0,033 0,669 0,748 0,678 0,076 0,090 0,105 0,678 0,748 0,687 0,080 0,097 0,110 40 10x 0,687 0,748 0,687 0,083 0,093 0,110 0,730 0,776 0,764 0,131 0,137 0,123 0,741 0,764 0,776 0,131 0,139 0,126 50 10x 0,741 0,776 0,776 0,134 0,135 0,126 0,699 0,728 0,728 0,198 0,225 0,222 0,708 0,738 0,738 0,198 0,225 0,222 60 10x 0,718 0,738 0,738 0,198 0,234 0,213 0,570 0,551 0,538 0,141 0,131 0,151 0,577 0,557 0,544 0,146 0,136 0,156 70 10x 0,584 0,564 0,544 0,149 0,136 0,164 0,385 0,359 0,372 0,039 0,064 0,096 0,390 0,355 0,372 0,047 0,064 0,086 80 10x 0,394 0,355 0,381 0,045 0,060 0,093 0,222 0,198 0,210 0,064 0,056 0,067 0,226 0,213 0,216 0,060 0,071 0,073 90 10x 0,222 0,207 0,216 0,067 0,067 0,071 0,160 0,175 0,183 0,045 0,069 0,093 0,170 0,167 0,186 0,057 0,074 0,106 100 10x 0,154 0,181 0,189 0,059 0,074 0,102
Lampiran 7 Data Uji DNS α-Amilase Hasil Isolasi dari Vibrio sp. SFNB 3 setelah Inkubasi pada Suhu Optimum selama 0, 15, 30, 60, 120 dan 240 Menit
Tabel L.8. Data uji DNS α-amilase hasil isolasi dari Vibrio sp. SFNB 3 setelah inkubasi pada suhu optimum selama 0, 15, 30, 60, 120 dan 240 menit.
Sampel Kontrol Menit
ke- Pengenceran Faktor A1 A2 A3 A1 A2 A3
0,720 0,708 0,710 0,020 0,022 0,028 0,716 0,706 0,712 0,030 0,021 0,026 0 10x 0,720 0,706 0,706 0,024 0,025 0,030 0,692 0,688 0,695 0,034 0,028 0,020 0,692 0,690 0,695 0,030 0,025 0,018 15 10x 0,689 0,690 0,691 0,030 0,025 0,022 0,680 0,676 0,681 0,022 0,035 0,037 0,676 0,682 0,681 0,025 0,035 0,033 30 10x 0,676 0,680 0,679 0,025 0,033 0,033 0,560 0,580 0,583 0,025 0,039 0,045 0,566 0,582 0,583 0,038 0,039 0,045 120 10x 0,564 0,585 0,588 0,038 0,035 0,051 0,392 0,387 0,397 0,041 0,035 0,039 0,392 0,390 0,391 0,044 0,035 0,033 240 10x 0,392 0,382 0,389 0,044 0,033 0,039
Lampiran 8 Data Uji DNS α-Amilase Hasil Isolasi dari Vibrio sp. SFNB 3 pada Berbagai pH
Tabel L.9. Data uji DNS α-amilase hasil isolasi dari Vibrio sp. SFNB 3 pada berbagai pH. Sampel Kontrol pH Pengenceran Faktor A1 A2 A3 A1 A2 A3 0,053 0,069 0,055 0,046 0,059 0,046 0,065 0,069 0,057 0,048 0,055 0,046 2 10x 0,063 0,064 0,059 0,044 0,055 0,046 0,077 0,053 0,059 0,050 0,040 0,044 0,073 0,055 0,057 0,057 0,050 0,045 3 10x 0,057 0,055 0,057 0,059 0,044 0,045 0,055 0,059 0,061 0,040 0,044 0,044 0,055 0,057 0,067 0,044 0,040 0,046 4 10x 0,057 0,053 0,063 0,039 0,039 0,042 0,494 0,510 0,445 0,104 0,117 0,104 0,505 0,494 0,440 0,106 0,119 0,102 5 10x 0,505 0,505 0,445 0,102 0,121 0,104 0,413 0,413 0,427 0,079 0,083 0,091 0,409 0,418 0,427 0,081 0,087 0,091 6 10x 0,413 0,422 0,431 0,083 0,089 0,095 0,400 0,392 0,386 0,083 0,073 0,073 0,396 0,388 0,384 0,087 0,075 0,067 7 10x 0,400 0,388 0,392 0,089 0,077 0,073 0,587 0,614 0,621 0,163 0,173 0,185 0,587 0,614 0,621 0,168 0,175 0,188 8 10x 0,594 0,607 0,628 0,163 0,178 0,188 0,258 0,280 0,289 0,083 0,089 0,119 0,258 0,273 0,293 0,089 0,090 0,121 9 10x 0,261 0,277 0,293 0,087 0,102 0,128 0,112 0,075 0,099 0,069 0,055 0,085 0,108 0,077 0,106 0,067 0,069 0,081 10 10x 0,104 0,081 0,104 0,067 0,063 0,087
Lampiran 9 Data Uji DNS Hidrolisis Pati Mentah Jagung oleh α-Amilase Hasil Isolasi dari Vibrio sp. SFNB 3 setelah inkubasi pada 37oC, 150 rpm selama 24, 48 dan 72 Jam
Tabel L.10. Data uji hidrolisis pati mentah jagung 5% (w/v) oleh α-amilase hasil isolasi dari Vibrio sp. SFNB 3 setelah inkubasi pada 37oC, 150 rpm selama 24, 48 dan 72 jam dengan metode DNS.
Sampel Kontrol
Jam ke- Pengenceran Faktor
A1 A2 A3 A1 A2 A3 0,262 0,268 0,274 0,045 0,056 0,053 0,259 0,268 0,270 0,047 0,056 0,054 24 5x 0,262 0,265 0,272 0,045 0,054 0,054 0,352 0,363 0,359 0,056 0,060 0,064 0,355 0,367 0,367 0,054 0,056 0,062 48 5x 0,355 0,367 0,367 0,053 0,056 0,064 0,262 0,247 0,253 0,008 0,017 0,012 0,267 0,250 0,250 0,010 0,015 0,013 72 10x 0,265 0,247 0,250 0,010 0,015 0,012
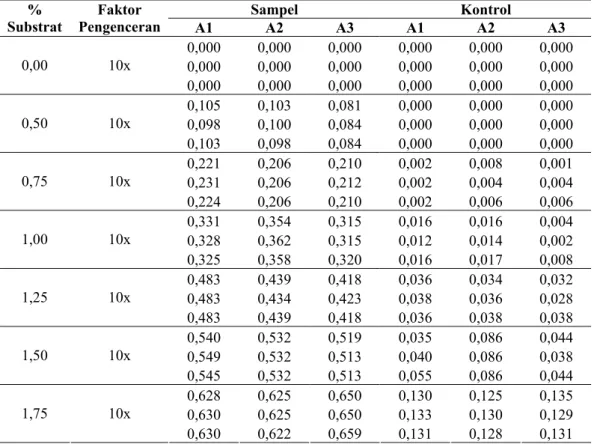
Lampiran 10 Data Uji DNS α-Amilase Hasil Isolasi dari Vibrio sp. SFNB 3 untuk Penentuan Nilai Km, Vmax dan kcat
Tabel L.11. Data uji DNS α-amilase hasil isolasi dari Vibrio sp. SFNB 3 untuk penentuan waktu reaksi enzimatis.
Sampel Kontrol Menit
ke- Pengenceran Faktor A1 A2 A3 A1 A2 A3
0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0 10x 0,000 0,000 0,000 0,000 0,000 0,000 0,253 0,250 0,256 0,029 0,027 0,033 0,253 0,256 0,256 0,025 0,029 0,031 5 10x 0,259 0,250 0,256 0,027 0,031 0,031 0,535 0,548 0,548 0,012 0,022 0,025 0,541 0,548 0,541 0,018 0,023 0,027 10 10x 0,535 0,541 0,554 0,022 0,027 0,025 0,675 0,668 0,677 0,027 0,029 0,033 0,678 0,668 0,677 0,022 0,033 0,037 15 10x 0,678 0,660 0,679 0,027 0,029 0,033 0,739 0,730 0,730 0,027 0,022 0,018 0,737 0,725 0,733 0,027 0,023 0,020 20 10x 0,735 0,728 0,735 0,025 0,020 0,027
Tabel L.12. Data uji DNS α-amilase hasil isolasi dari Vibrio sp. SFNB 3 untuk penentuan Km dan Vmax.
Sampel Kontrol % Substrat Faktor Pengenceran A1 A2 A3 A1 A2 A3 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,000 0,00 10x 0,000 0,000 0,000 0,000 0,000 0,000 0,105 0,103 0,081 0,000 0,000 0,000 0,098 0,100 0,084 0,000 0,000 0,000 0,50 10x 0,103 0,098 0,084 0,000 0,000 0,000 0,221 0,206 0,210 0,002 0,008 0,001 0,231 0,206 0,212 0,002 0,004 0,004 0,75 10x 0,224 0,206 0,210 0,002 0,006 0,006 0,331 0,354 0,315 0,016 0,016 0,004 0,328 0,362 0,315 0,012 0,014 0,002 1,00 10x 0,325 0,358 0,320 0,016 0,017 0,008 0,483 0,439 0,418 0,036 0,034 0,032 0,483 0,434 0,423 0,038 0,036 0,028 1,25 10x 0,483 0,439 0,418 0,036 0,038 0,038 0,540 0,532 0,519 0,035 0,086 0,044 0,549 0,532 0,513 0,040 0,086 0,038 1,50 10x 0,545 0,532 0,513 0,055 0,086 0,044 0,628 0,625 0,650 0,130 0,125 0,135 0,630 0,625 0,650 0,133 0,130 0,129 1,75 10x 0,630 0,622 0,659 0,131 0,128 0,131