VARIASI GENETIK HASIL PERSILANGAN NILA BEST DENGAN
RED NIFI DAN NIRWANA MENGGUNAKAN PENANDA RAPD
Isk andar iah, Ir in Ir iana Kusm ini, Ot ong Zenal Ar if in, dan Rudhy Gust iano
Balai Riset Perikanan Budidaya Air Tawar Jl. Sem pur No. 1, Bogor 16154 E- m ail: [email protected]
(Naskah diterima: 16 Agustus 2011; Disetujui publikasi: 7 November 2011)
ABST RAK
Penelitian m engenai variasi genetik dari hibridisasi 3 varian ikan nila (BEST, Red NIFI, dan Nirwana) telah dilakukan di Laboratorium Molekuler Biologi, Balai Riset Perikanan Budidaya Air Tawar (BRPBAT) Bogor. Tujuan penelit ian ini adalah unt uk m enget ahui keragam an genetik hasil hibridisasi antara nila BEST, Nirwana, dan Red NIFI dengan m enggunakan m etode Random Amplified Polymorphism DNA (RAPD). Am plifikasi DNA m enggunakan prim er OPA- 02, OPA- 03, dan OPC- 05. Hasil analisis m enunjukkan bahwa p er sen t ase p o l i m o r f i sm e b er k i sar an t ar a 1 8 , 1 8 1 8 %–4 2 , 4 2 4 2 %, d en g an n i l ai het er ozigosit as 0 ,0 7 3 3 - 0 ,1 5 6 4 dan j ar ak genet ik ant ar populasi 0 ,3 7 3 5 –0 ,5 3 0 0 . Keragam an genetik tertinggi diperoleh pada hasil hibridisasi antara nila BEST dengan Nirwana. Nilai polim orfism e m eningkat sebesar 10,6060% dan heterozigositas 0,0352 pada populasi hasil hibridisasi dibandingkan dengan populasi true breeding. Hal ini m enunjukkan bahwa hibridisasi dapat m eningkatkan nilai keragam an genetik dari suatu populasi ikan.
KATA KUNCI: RAPD , genet ik , var ian, ik an nila, Oreochrom is
ABSTRACT : Genet ic var iance of hyb r id s p r od uced f r om t hr ee st r ains of t i l a p i a ( BEST , Re d N I FI , a n d N i r w a n a ) u si n g RAPD . By : I sk a n d a r i a h , I r i n I r i a n a Ku sm i n i , O t o n g Z e n a l Ar i f i n , a n d Rudhy Gust iano
Study on genetic variance of hybrids produced from three strains of tilapia (BEST, Red NIFI, and Nirwana) has been carried out at the Laboratory of Molecular Biology, the Research Institute for Freshwater Aquaculture (RIFA) Bogor. The purpose of this study was to determine the genetic diversity of hybrids produced from crossing among BEST, Nirwana, and RedNIFI tilapias by using Random Amplified Polymorphism DNA (RAPD). DNA amplification used primers OPA-02, OPA-03, and OPC-05. The results showed that the percentage of polymorphism ranged from 18.1818%-42.4242%, with a heterozygosity value of 0.733-0.1564 and the genetic distance between populations was 0.3735-0.5300. The highest genetic diversity was found on hybrid between BEST and Nirwana tilapias. Polymorphism value increased by 10.6060% with heterozygosity of 0.0352 in hybrid populations compared with the true breeding populations. This suggests that hybridization can increase the value of genetic diversity of a fish population.
PENDAHULUAN
Ikan nila (Oreochromis niloticus) m eru-pakan salah sat u kom odit as unggulan budi-daya yang diharapkan dapat memenuhi target pem erint ah dalam m eningk at k an produk si perikanan nasional sebesar 353% pada t ahun 2015. Ikan ini tahan terhadap penyakit, mudah berkembang biak dan toleran terhadap kualitas ai r yan g r en d ah t er m asu k k ad ar ok si g en t erlarut yang rendah.
Penggunaan ikan nila sebagai kom odit as budidaya yang sem ak in populer dan ber -k em bang di m asyar a-k at secar a luas t elah m enyebabkan t erjadinya penurunan kualit as genet ik . Secara um um penurunan k ualit as genet ik ikan dit andai dengan pert um buhan yang lam bat , t ingkat kem at ian t inggi, kem a-t angan gonad pada usia dini, dan uk uran individu yang kecil. Rendahnya keragam an genetik akan mengakibatkan munculnya sifat-sif at negat if , ant ara lain m enurunnya per-t um b uhan, k er agam an uk ur an, k esper-t ab ilan perkem bangan organ, t ingkat sint asan sert a adapt asi t erhadap perubahan lingkungannya (Leary et al., 1985). Terjadinya penurunan k u al i t as g en et i k i n i p ad a ak h i r n ya ak an m enyebabk an m enurunnya produk si m au-pun produkt ivit as para pem budidaya ikan, sehingga perlu adanya suatu upaya perbaikan mutu genetik ikan nila.
Salah sat u upaya yang dapat dilakukan dalam meningkatkan mutu genetik ikan adalah dengan m elakukan hibridisasi unt uk m en-dapat kan sif at unggul yang lebih baik dari populasi asal. Pada dasarnya hibridisasi adalah m em anf aat k an sif at het erosis k arena sif at dom inan dan het erozigot pada banyak lokus, at au int eraksi alela pada lokus. Persilangan u m u m n ya d i l ak u k an an t ar p op u l asi yan g m em iliki keunggulan spesifik (Gustiano et al., 2008). Menurut Ariyant o (2003), persilangan dilakukan untuk mencegah terjadinya kemun-duran genetik yang biasa terjadi akibat silang dalam (inbreeding).
Ik an nila BEST, Red NIFI, d an Nir wana m erupakan st rain unggul yang bisa dijadikan bahan dasar unt uk hibridisasi pada ikan nila. Ikan nila BEST (Bogor Enhanced Strain Tilapia) m er upak an ik an nila hasil pem uliaan dar i generasi ke- 6 nila GIFT melalui program seleksi famili yang dilakukan oleh Balai Riset Perikanan Bud id aya Air Tawar Bogor . Ik an nila BEST memiliki beberapa keunggulan, seperti: mampu m eng hasilk an t elur d an b enih yang leb ih
banyak, larva ikan lebih besar dan mempunyai r at a- r at a p er t u m b u h an yan g t i n g g i p ad a b er b ag ai l i n g k u n g an b u d i d aya. Ik an n i l a Nirwana merupakan hasil seleksi famili dengan bahan dasar ikan nila GIFT (Genetic Improve-ment Farm Tilapia) dan nila GET (Genetically Enchanced Tilapia) dari Philipina yang di-lakukan Balai Pengembangan Benih Ikan (BPBI) Wanayasa, Purwakart a. Pert um buhan bobot ikan nila Nirwana m eningkat sekitar 45% pada g en er asi k e- 3 (F3 ) d i b an d i n g k an d en g an populasi awalnya (Gust iano & Arif in, 2010). Sedangkan Red NIFI merupakan jenis ikan nila m erah yang didat angkan dari Thailand pada tahun 1989 (Gustiano et al., 2008). Balai Riset Perik anan Budidaya Air Tawar Bogor t elah m elakukan hibridisasi antara ketiga jenis ikan unggulan t ersebut .
Un t u k m en g et ah u i sej au h m an a h asi l hibridisasi dapat m eningkatkan m utu genetik dari suat u jenis ikan m aka perlu dilakukan analisis DNA. Ada beberapa metode yang dapat dilakukan, salah satunya menggunakan teknik RAPD.
Tujuan penelitian ini adalah untuk menge-tahui keragaman genetik hasil persilangan nila BEST dengan Nirwana dan Red NIFI dengan menggunakan metode RAPD.
BAHAN DAN METODE
Ikan Uji
Ikan uji yang digunakan adalah ikan hasil persilangan ant ara nila BEST dengan Nirwana dan Red NIFI dengan masing- masing populasi diam bil 10 sam pel. Sam pel ik an nila yang digunakan berupa sirip yang dipot ong dan disim pan dalam larut an alkohol 70% unt uk digunakan dalam proses analisis.
Ekstraksi DNA
DNA ikan diekst raksi dari pot ongan sirip dengan m enggunakan m etode Phenol-Chloro-form (Nugroho et al., 1997). Pot ongan sirip seb an y ak 5 –1 0 m g d i m asu k k an d al am mikrotube 1,5 mL yang telah diisi dengan 500 mL TNES Urea, kemudian ditambah 10 µL Pro-t ein Kinase. SePro-t elah divorPro-tex selam a 1 m eniPro-t sam pel diinkubasi pada suhu 37oC selam a 24
m ikrot ube baru, lalu dit am bah dengan 1000 µL ethanol 90% dan 10 µL CH3COONa. Sampel lalu divortex selam a 1 m enit sam pai t erlihat gum palan berwarna put ih. DNA diendapkan d en g an car a m en sen t r i f i u sn y a d en g an kecepat an 10.000 rpm selam a 10 m enit , lalu cairannya dibuang dan dikeringkan pada suhu kam ar. Pellet DNA dilarut kan dengan 100 µL Tris- EDTA (TE) buffer dan disimpan pada suhu 4oC sebelum digunakan pada tahap selanjutnya.
Random Amplified Polymorphism DNA (RAPD)
Primer yang digunakan adalah OPA 01- 20 dan OPC 01- 20. Proses am plifikasi dilakukan dengan m et ode Polymerize Chain Reaction (PCR) dengan kom posisi reaksi: 1 µL DNA; 1,5 µL primer; 12,5 µL 2X PCR Master Mix; dan 1 0 µ L H2O; d en g an t o t al vo l u m e 2 5 µ L. Selanjut nya dim asukkan dalam thermocycler dengan 1 siklus denat urasi pada suhu 94oC
selam a 2 m enit , 35 siklus penggandaan yang terdiri atas denaturasi pada suhu 94oC selama
1 m enit , annealing pada suhu 36oC selam a 1
m enit dan elongasi pada suhu 72oC selam a
2,5 m enit; dan elongasi akhir pada suhu 72oC
sel am a 7 m en i t . Hasi l PCR k em u d i an d i -elekt rof oresis m enggunakan gel agarose 1% dalam Tris- Boric- EDTA (TBE) buffer 1%. Hasilnya diam at i dengan UV illum inat or dan dicet ak gambarnya dengan Polaroid.
Analisis Data
Nilai polim orf ism e dan het erozigosit as dianalisis dengan m enggunakan descriptive
statistics (Mi l l er , 1 9 9 7 ). Anal i si s st at i st i k m enggunakan exact test for population dif-ferentiation (Raymond & Rousset, 1995 dalam Miller, 1997) dan kekerabat an ant ar populasi dianalisis dengan menggunakan jarak genetik m enur ut Wr ight (1 9 7 8 ) m odif ik asi Roger s (1972) dalam Miller (1997).
HASIL DAN BAHASAN
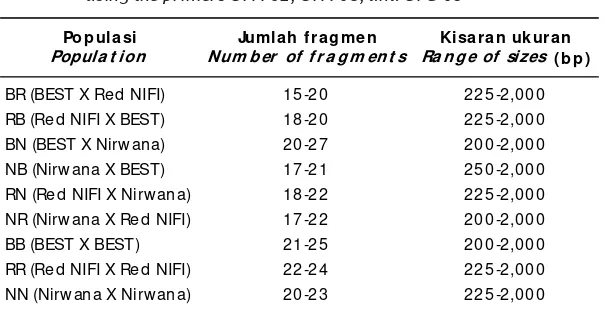
Dari 20 jenis primer yang dicoba, 3 primer yakni: OPA- 02, OPA- 03, dan OPC- 05 dapat menghasilkan amplifikasi dalam jumlah sampel yang banyak. Hasil amplifikasi DNA dari ketiga prim er m enghasilkan jum lah f ragm en ant ara 15–27 buah dengan ukuran berkisar ant ara 2 0 0 –2 0 0 0 b p (T ab el 1 ). Co n t o h h as i l am plif ikasi dari ket iga prim er t ersebut pada p o p u l asi h asi l h i b r i d i sasi d i saj i k an p ad a Gambar 1–3, sedangkan hasil amplifikasi pada populasi true breeding t ersaji pada Gam bar 4- 6.
Persent ase polim orf ism e berkisar ant ara 1 2 ,1 2 1 2 %–4 2 ,4 2 4 2 % d an h et er o z i g o si t as antara 0,0514 – 0,1564. Nilai polimorfisme dan het erozigosit as t ert inggi pada populasi hasil persilangan BEST X Nirwana dan terendah pada populasi true breeding Nirwana X Nirwana (Tabel 2).
Secara st at ist ik dengan m enggunakan uji perbandingan berpasangan Fst m enunjukkan bahwa t erdapat perbedaan genet ik secara n yat a an t ar p op u l asi i k an n i l a yan g d i u j i (P< 0,05), kecuali antara populasi BEST X BEST dengan Red NIFI X Red NIFI (Tabel 3).
Tabel 1. Jum lah f ragm en dan k isaran uk uran hasil am plif ik asi DNA menggunakan primer OPA- 02, OPA- 03, dan OPC- 05
Table 1. Number of fragments and size ranges of DNA amplification using the primers OPA-02, OPA-03, and OPC-05
Gambar 2. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi BEST X Red NIFI Figure 2. Results of amplification of primers OPA-02, OPA-03, and OPA-05 in BEST X Red NIFI
populations
1000 bp
500 bp M BR1 BR2 BR3 BR4 BR5 M BR1 BR2 BR3 BR4 BR5 M BR1 BR2 BR3 BR4 BR5
Gambar 1. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi BEST X Nirwana Figure 1. Results of amplification of primers OPA-02, OPA-03, and OPC-05 in BEST X Nirwana
populations
1000 bp
500 bp M BN1 BN2 BN3 BN4 BN5 M BN1 BN2 BN3 BN4 BN5 M BN1 BN2 BN3 BN4 BN5
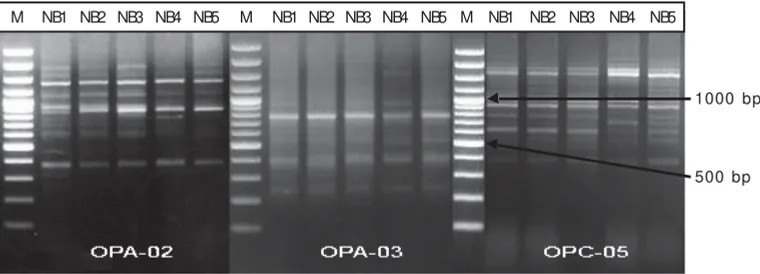
Gambar 3. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi Nirwana X BEST Figure 3. Results of amplification of primers OPA-02, OPA-03, and OPA-05 in Nirwana X BEST
populations
1000 bp
Gambar 5. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi Nirwana X Nirwana Figure 5. Results of amplification of primers OPA-02, OPA-03, and OPA-05 in Nirwana X Nirwana
populations
1000 bp
500 bp Gambar 4. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi BEST X BEST Figure 4. Results of amplification of primers OPA-02, OPA-03, and OPA-05 in BEST X BEST
populations
1000 bp
500 bp M BB1 BB2 BB3 BB4 BB5 M BB1 BB2 BB3 BB4 BB5 M BB1 BB2 BB3 BB4 BB5
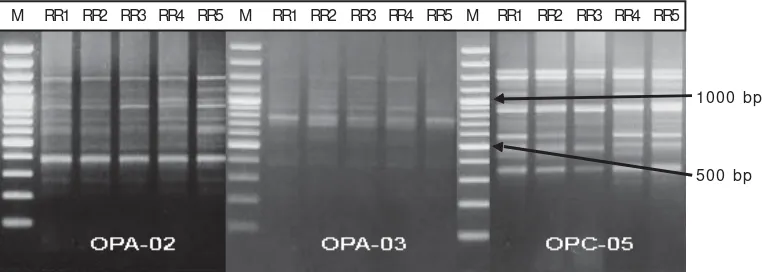
Gambar 6. Hasil amplifikasi primer OPA- 02, OPA- 03, dan OPC- 05 pada populasi Red NIFI X Red NIFI Figure 6. Results of amplification of primers OPA-02, OPA-03, and OPA-05 in Red NIFI X Red
NIFI populations
1000 bp
500 bp M RR1 RR2 RR3 RR4 RR5 M RR1 RR2 RR3 RR4 RR5 M RR1 RR2 RR3 RR4 RR5
Tabel 3. Nilai P dari uji perbandingan berpasangan Fst Table 3. P value of Fst pairwise comparison test
Ket erangan: Sem uanya berbeda nyat a (P< 0,05), kecuali ant ara BB dengan RR Note: All of values are significantly different (P<0.05), except between the BB and RR
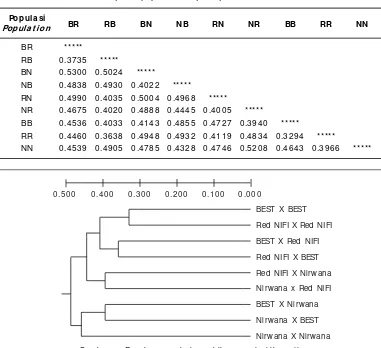
Jarak genet ik t ert inggi ant ara populasi BEST X Red NIFI dengan BEST X Nirwana dan terendah antara populasi BEST X BEST dengan Red NIFI X Red NIFI (Tabel 4). Dendrogram yang dibent uk berdasarkan jarak genet ik t ersebut disajikan pada Gambar 7.
Hasil am plif ikasi dari ket iga prim er yakni OPA- 02, OPA- 03, dan OPC- 05 m enghasilkan jum lah f ragm en ant ara 15–27 buah dengan ukuran berkisar antara 200–2000 bp (Tabel 1). Hasil amplifikasi setiap primer memiliki karakter yang berbeda sehingga jum lah dan ukuran
fragmen yang muncul juga berbeda. Frekuensi allel yang sem ak in besar m engindik asik an bahwa kemunculan fragmen semakin dominan pada lokus t ert ent u (m onom orf ik). Adanya fragmen yang spesifik (polimorfik) berpotensi unt uk m eningk at k an nilai het erozigosit as, yang m eng i nd i k asi k an sem ak i n t i ng g i nya keragaman genetik.
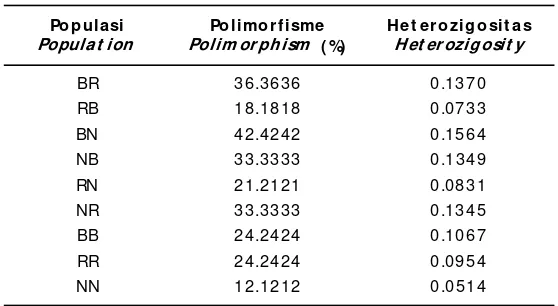
Hasil analisis dengan menggunakan TFPGA menunjukkan bahwa nilai polimorfisme ikan uji b er k i sar an t ar a 1 2 ,1 2 1 2 %–4 2 ,4 2 4 2 % d an heterozigositas 0,0514–0,1564 (Tabel 2). Nilai Po p ulasi
Popula t ion BR RB BN NB RN NR BB RR NN
BR *****
RB 0.0110 *****
BN 0.0000 0.0000 *****
NB 0.0000 0.0000 0.0000 *****
RN 0.0000 0.0000 0.0000 0.0000 *****
NR 0.0000 0.0001 0.0000 0.0000 0.0000 *****
BB 0.0000 0.0003 0.0000 0.0000 0.0000 0.0057 *****
RR 0.0000 0.0042 0.0000 0.0000 0.0000 0.0000 0.3561 *****
NN 0.0001 0.0000 0.0000 0.0001 0.0000 0.0000 0.0000 0.0002 ***** Tabel 2. Persentase polimorfisme dan heterozigositas pada populasi
ikan uji
Table 2. Percentage of polymorphism and heterozigosity of test fish populations
Po p ulasi
Popula t ion
Po limo rf isme
Polim or ph ism ( %)
H et ero zig o sit as
Het er ozig osit y
BR 36.3636 0.1370
RB 18.1818 0.0733
BN 42.4242 0.1564
NB 33.3333 0.1349
RN 21.2121 0.0831
NR 33.3333 0.1345
BB 24.2424 0.1067
RR 24.2424 0.0954
Tabel 4. Jarak genetik sembilan populasi ikan uji
Table 4. Genetic distance of nine populations of test fish
Gambar 7. Dendrogram dari sembilan populasi ikan uji Figure 7. Dendrogram of nine populations of test fish
keragam an genetik tertinggi didapatkan pada populasi hasil hibridisasi ant ara BEST dengan Nirwana, kem udian diikuti oleh populasi BEST d en g an Red NIFI, Ni r wan a d en g an BEST, Nirwana dengan Red NIFI dan t erendah pada Red NIFI dengan Nirwana. Sedangkan pada p o p u l asi true breeding h asi l k er ag am an genet ik t ert inggi dit unjukkan pada populasi BEST, d iik ut i Red NIFI d an t er end ah p ad a populasi Nirwana.
Pada populasi hasil hibridisasi diperoleh rata- rata nilai polimorfisme sebesar 30,8080% dan het erozigosit as 0,1197; sedangkan pada p o p u l asi true breeding r at a- r at a n i l ai polim orf ism e 20,2020% dan het erozigosit as 0,0845. Dengan demikian terjadi peningkatan
n i l ai p o l i m o r f i s m e 1 0 , 6 0 6 0 % d an het erozigosit as 0,0352. Hal ini m enunjukkan bahwa hibridisasi dapat m eningkat kan nilai keragam an genet ik dari suat u populasi ikan. Pada dasarnya hibridisasi adalah memanfaatkan si f at h et er osi s k ar en a si f at d om i n an d an het er oz igosit as p ad a b anyak lok us (Tave, 1993) atau interaksi dari alel pada lokus.
Perbedaan polim orf ism e genet ik dapat d i p eng ar uhi ol eh 4 m od el ut am a p eub ah kom ponen yait u m igrasi, seleksi, m ut asi dan random-metode. Masuknya sum ber genet ik baru dari sum ber lain diduga dapat m enjadi penyebab perubahan genet ik pada populasi ikan melalui hibridisasi. Nila BEST, Nirwana, dan Red NIFI memiliki proses seleksi yang berbeda. Po p ulasi
Popula t ion BR RB BN NB RN NR BB RR NN
BR *****
RB 0.3735 *****
BN 0.5300 0.5024 *****
NB 0.4838 0.4930 0.4022 *****
RN 0.4990 0.4035 0.5004 0.4968 *****
NR 0.4675 0.4020 0.4888 0.4445 0.4005 *****
BB 0.4536 0.4033 0.4143 0.4855 0.4727 0.3940 *****
RR 0.4460 0.3638 0.4948 0.4932 0.4119 0.4834 0.3294 *****
NN 0.4539 0.4905 0.4785 0.4328 0.4746 0.5208 0.4643 0.3966 *****
Nirwana X Nirwana 0.000
Nirwana X BEST BEST X Nirwana Nirwana x Red NIFI BEST X BEST
Red NIFI X Red NIFI
BEST X Red NIFI
Red NIFI X Nirwana Red NIFI X BEST 0.100
0.200 0.300
Ikan nila BEST merupakan ikan hasil pemuliaan untuk karakter keunggulan pertumbuhan yang dihasilk an m elalui proses selam a 4 t ahun penelit ian oleh Tim Penelit i dari Balai Riset Perikanan Budidaya Air Tawar Bogor (Arif in, 2008).
N i l ai h et er o z i g o si t as m en u n j u k k an k er ag am an g en et i k d ar i su at u p o p u l asi . Het erozigosit as yang t inggi m em ungkinkan p er b aik an m ut u g enet ik p op ulasi d eng an m en g ek sp l oi t asi g en g en yan g m en g u n -t u n g k an . Gen o-t i p h e-t er oz i g osi -t as r esesi f m engakibatkan detrim ental yaitu kekurangan enzim penting yang diproduksi oleh alel yang d o m i n an y an g b er h u b u n g an d en g an penurunan fekunditas, penurunan daya hidup atau peningkatan m ortalitas, penurunan daya t ah an t er h ad ap p en y ak i t d an l aj u pert um buhan. Variasi genet ik suat u m akhluk hidup dapat m uncul karena m ut asi, seleksi alam , pengaruh lingkungan, dan perkawinan. A p ab i l a f ak t o r d i at as m u n c u l d ap at m enyebabkan t erjadinya perubahan genet ik su at u i n d i vi d u d an sel an j u t n y a t er j ad i perubahan susunan genet ik populasi yang t erbent uk dari individu t ersebut (Sugam a et al., 1996).
Hasil analisis st at ist ik m enggunakan uji perbandingan berpasangan Fst m enunjukkan bahwa t erdapat perbedaan genet ik secara nyata antar populasi ikan uji (P > 0,05), kecuali ant ara populasi BEST X BEST dan Red NIFI X Red NIFI t i d ak b er b ed a n yat a (Tab el 3 ). Perbedaan yang nyat a m enunjukkan adanya perbedaan karakter genetik maupun asal- usul dari induknya.
Jarak genetik tertinggi ditunjukkan antara p op ul asi BEST X Red NIFI d eng an BEST X Nirwana dan t erendah ant ara populasi BEST X BEST dengan Red NIFI X Red NIFI. Jarak genetik m erupakan ukuran perbedaan genet ik ant ar famili yang dihitung berdasarkan frekuensi alel (Nei, 1987). Sem akin kecil jarak genetik antar individu dalam satu famili, maka famili tersebut sem akin seragam (Koh et al., 1999). Menurut Pandin (2000), tingkat kem iripan genetik dari suat u populasi dapat digam barkan oleh jarak g en et i k d ar i i n d i vi d u - i n d i vi d u an g g o t a populasi. Semakin besar jarak genetik individu d i d al am su at u p o p u l asi , m ak a p o p u l asi t er seb u t m em i l i k i an g g ot a yan g sem ak i n beragam.
Dendrogram yang dibent uk berdasarkan jarak genetik membentuk 2 cluster besar.
Clus-ter pert am a kem udian t erbagi lagi m enjadi 2 cluster yaitu pertam a terdiri atas populasi nila BEST dengan Red NIFI dan hasil hibridisasinya dan kedua terdiri atas populasi hasil hibridisasi Red NIFI dengan Nirwana. Cluster kedua terdiri at as populasi Nirwana dan hasil hibridisasi-nya dengan nila BEST. Terdapat kedekat an hubungan kekerabat an ant ara populasi true breeding BEST X BEST dengan Red NIFI X Red NIFI. Kedekat an hubungan kekerabat an ini m enunj uk k an adanya k esam aan asal- usul induk.
KESIMPULAN
Hasi l an al i si s t er h ad ap p op u l asi h asi l hibridisasi ant ara nila BEST dengan Nirwana dan Red NIFI m enunjukkan bahwa persentase p o l i m o r f i sm e b er k i sar an t ar a 1 8 ,1 8 1 8 %– 4 2 ,4 2 4 2 %; d en g an n i l ai h et er o z i g o si t as 0 ,0 7 3 3 –0 ,1 5 6 4 d an j ar ak g en et i k an t ar populasi 0,3735–0,5300. Keragam an genetik tertinggi diperoleh pada hasil hibridisasi antara nila BEST dengan Nirwana.
Nilai p olim or f ism e m eningk at seb esar 10,6060% dan het erozigosit as 0,0352 pada populasi hasil hibridisasi dibandingkan dengan populasi true breeding. Hal ini m enunjukkan bahwa hibridisasi dapat m eningkat kan nilai keragam an genetik dari suatu populasi ikan.
DAFTAR ACUAN
Arifin, O.Z. 2008. Nila BEST (Bogor Enhanched St r ai n Ti l ap i a). www.b l o g sp o t .co m (8 Agustus 2010).
Ariyanto. 2003. Ikan mas hibrida antara harapan d an k en yat aan . Warta Penelitian dan Perikanan Indonesia, 9(3): 2- 6.
Gustiano, R., Arifin, O.Z., & Nugroho, E. 2008. Per b ai k an p er t u m b u h an i k an n i l a (Oreochromis niloticus) dengan selek si famili. Media Akuakultur, 3(2): 98- 106. Gustiano, R. & Arifin, O.Z. 2010. Menjaring laba
dari budidaya nila BEST. IPB Press. Bogor, 81 hlm.
Leary, R.F., Allendrof, F.W. , & Knudsen, K.L. 1985. Developm ent inst abilit y and high m erist ic count s in int erspecific hybrid of salm onid fishes. Evolution, 39(6): 1318-1326.
Miller, M.P. 1997. Tools For Population Genetic Analysis (TFPGA) version 1.3. Departm ent of biological science. Nort hern Arizona University, Arizona, USA, 30 pp.
Nei, M. 1987. Molecular evolut ionary genet -ics. Colum bia University Press. New York, 512 pp.
Nugroho, E., Takagi, M., & Taniguchi, N. 1997. Practical manual on detection of DNA
poly-morphism in fish population study. Bulletin of marine sciences and fisheries, Kochi University, 17: 109- 129.
Pandin, D.S. 2000. Kemiripan Genetik Populasi Kelapa dalam Mapanget Tenga, Bali, Palu dan Sawarna Berdasarkan Penanda RAPD. Tesis. Program Pasca Sarjana IPB. Bogor. Sugam a, K., Haryant i, & Cholik, F. 1996.
Bio-chemical genetics of tiger shrimp Penaeus monodon. Description electrophoresis de-tectable loci. Indonesian Fish. Res. J., II (1): 19–28.