1
STUDI FILOGENETIK ELANG LAUT PERUT PUTIH (Haliaeetus leucogaster) YANG HIDUP DI PULAU JAWA BERDASARKAN GEN Cytochrome-b (Cyt-b)
Riri Wiyanti Retnaningtyas, Nuning Wulandari, Dwi Listyorini
Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Negeri Malang Email: riri.suryadinata@gmail.com
ABSTRAK
Elang Laut Perut Putih merupakan satu-satunya anggota genus Haliaeetus yang dapat ditemukan di Indonesia. Spesies ini hidup di daerah pesisir, hutan pantai dan lepas pantai dan berperan sebagai predator di ekosistem pesisir. Populasi spesies ini, terutama di pesisir selatan Jawa, semakin menurun akibat perburuan liar, perdagangan satwa illegal, deforestasi, dan aktivitas manusia lainnya. Jangkauan distribusi Elang Laut Perut Putih yang sangat luas, dan dengan adanya fragmentasi habitat, memungkinkan terjadinya perubahan pada keanekaragaman genetik dalam satu spesies. Oleh sebab itu, dalam penelitian ini dilakukan studi filogenetik terhadap tiga individu Elang Laut Perut Putih yang hidup di Pulau Jawa berdasarkan gen
Cytochrome-b (Cyt-b). Penelitian ini dilakukan dengan cara mengekstraksi DNA total dari darah Elang
Laut Perut Putih menggunakan High Pure PCR Preparation Kit dari Roche dengan protokol yang dimodifikasi. Amplifikasi gen Cyt-b dengan cara PCR menggunakan sepasang primer hasil desain yaitu HL Cyt-b F1 5’– TAG GAA TCT GCC TGC TGA CA-3’ dan HL Cyt-b R1 5’–TTA GTG GTT GAG AAG TTT GT- 3’. Hasil PCR kemudian dielektroforesis dengan mengguanakan gel agarose 0,8%. Hasil analisis filogenetik berdasarkan gen Cyt-b, dapat disimpulkan bahwa Elang Laut perut putih yang diteliti merupakan spesies Haliaeetus leucogaster yang berkerabat dekat dan membentuk species complex dengan Haliaeetus
sanfordi.
Kata Kunci: Haliaeetus leucogaster, DNA Barcoding, Cytochrome-b, studi filogenetik
PHYLOGENETIC STUDY OF WHITE-BELLIED SEA EAGLE (Haliaeetus leucogaster) LIVING IN JAVA BASED ON Cytochrome-b (Cyt-b) GENE
Riri Wiyanti Retnaningtyas, Nuning Wulandari, Dwi Listyorini
Department of Biology, Faculty of Mathematics and Natural Sciences, Universitas Negeri Malang Email: riri.suryadinata@gmail.com
ABSTRACT
The only species of genus Haliaeetus that can be found in Indonesia is White-bellied Sea Eeagle. As the top order predator in the coastal ecosystem, White-bellied Sea Eagle is sensitive to any threats caused by human activities. In the southern coast of Java, the population of this species is decreasing due to illegal huntings, deforestations, illegal wildlife trades. Regarding its vast distribution in all coastal areas of South East Asia and Australia, the habitat fragmentation caused by deforestation can lead to changes in this species’ genetic diversity. Thus, in this research, a phylogenetic study based on Cytochrome-b (Cyt-b) gene was conducted in order to obtain the Cyt-b gene sequence of three individuals of White-bellied Sea Eagle and to reconstruct a phylogenetic tree that describes their position Haliaeetus genus. This research was conducted by performing the DNA isolation using High Pure PCR Template Preparation Kit from Roche with a modified protocol. PCR was perfomed using a pair of designed primers HL Cyt-b F1 5’– TAG GAA TCT GCC TGC TGA CA-3’ and HL Cyt-b R1 5’–TTA GTG GTT GAG AAG TTT GT- 3’. The PCR amplicons were then visualized with electrophoresis using 0.8% of agarose gel. The phylogenetic analysis
2
based on Cyt-b gene indicates that White-bellied Sea Eagles in this research belong to the species
Haliaeetus leucogaster and are very closely related to Haliaeetus sanfordi.
Keywords: Haliaeetus leucogaster, DNA Barcoding, Cytochrome-b, phylogenetic study
PENDAHULUAN
Kelompok burung elang laut (genus Haliaeetus) merupakan kelompok monofiletik burung pemangsa yang tersebar di seluruh belahan dunia. Genus ini beranggotakan 8 spesies, 4 di antaranya tersebar di belahan bumi utara yaitu Haliaeetus albicilla, Haliaeetus leucocephlus, Haliaeetus pelagicus, Haliaeetus leucoryphus; 4 spesies sisanya, yaitu Haliaeetus vocifer, Haliaeetus vociferoides, Haliaeetus leucogaster, dan Haliaeetus sanfordi tersebar di belahan bumi selatan (Wink et al., 1996; Lerner & Mindell, 2005). Di Asia Selatan dan Australia, terdapat satu spesies Haliaeetus yang tersebar mulai dari pesisir barat India, China, seluruh wilayah pesisir Asia Tenggara termasuk seluruh kepuauan Indonesia, Filipina, Papua Nugini, dan Australia yaitu Haliaeetus leucogaster (Shephard et al., 2005).
Satu-satunya spesies Haliaeetus yang ada di Indonesia adalah Haliaeetus leucogaster (Elang Laut Perut Putih) (McKinnon et al., 2010). Elang Laut Perut Putih atau Haliaeetus leucogaster (Gmelin, 1788) merupakan raptor teritorial berukuran besar yang hidup di daerah pesisir, danau dan sungai perennial (Shephard et al., 2005). Elang Laut Perut Putih dewasa memiliki panjang tubuh sekitar 70 – 80 cm, bentang sayap sepanjang 1,8 – 2 meter dan lebar sayap ± 50 cm. Tubuh Elang Laut Perut Putih dewasa tertutup oleh bulu berwara putih dan abu-abu, iris mata berwarna cokelat gelap dengan pupil berwarna hitam dan memiliki tonjolan pada bagian atas mata. Kulit bagian tarsus hingga cakar tertutup oleh sisik berwarna kekuningan (Retnaningtyas et al., 2013). Perbedaan ciri morfologi antara Elang Laut Perut Putih dengan spesies lain yang tergolong genus Haliaeetus ditunjukkan oleh Gambar 1.
Gambar 1. Perbedaan ciri morfologi Elang Laut Perut Putih (Haliaeetus lecogaster) (A) dengan Elang Ekor Putih (Haliaeetus albicilla) (B), Elang Botak (Haliaeetus
leucocephalus) (C) dan Elang Laut Steller (Haliaeetus pelagicus) (D). Sumber: www.arkive.com
3
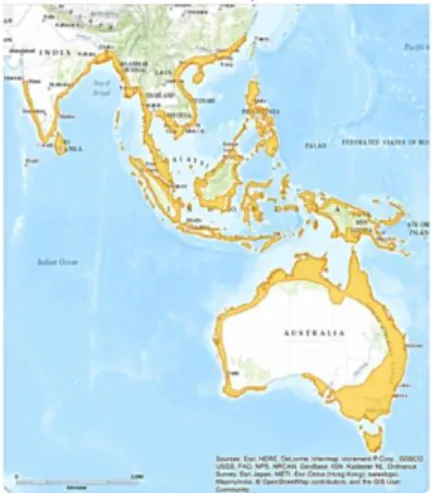
Spesies ini memiliki jangkauan distribusi global yang sangat luas, yaitu meliputi Australia, Bangladesh, Brunei Darussalam, Cambodia; China, Hong Kong, India, Indonesia, Laos, Malaysia, Myanmar, Papua Nugini, Filipina, Singapura, Sri Lanka, Thailand, Timor-Leste, dan Vietnam (Shephard et al., 2005) (Gambar 2). Berdasarkan jangkauan distribusi, maka spesies in dikatakan tidak mendekati ambang batas kategori Rentan (Vulnerable) (BirdLife International, 2015).
Gambar 2 Peta Distribusi global Elang Laut Perut Putih (Haliaeetus leucogaster). Sumber: IUCN Red List, 2012.
Habitat Elang Laut Perut Putih adalah hutan pantai dengan daya jelajah diperkirakan mencapai 13,9 km dan biasanya bersarang di pohon-pohon tinggi seperti Cemara Laut (Casuarina longistifolia) atau Kepuh (Gunawan, 2007), pada tumpukan batuan di tepi laut, tebing curam dan pulau-pulau lepas pantai (Shephard, et al., 2005). Mangsa dari Elang Laut Perut Putih utamanya adalah ikan laut, namun juga dapat memangsa mamalia kecil dan ikan air tawar (Debus, 2008). Di dalam ekosistem pesisir di Indonesia, Elang Laut Perut Putih merupakan salah satu pemangsa puncak dan dapat berperan sebagai indikator kestabilan ekosistem perairan serta untuk mengetahui seberapa besar pengaruh keberadaan manusia terhadap ekosistem tersebut (Romin & Muck, 1999). Elang Laut Perut Putih, selain memiliki peran ekologis, juga memiliki peran dalam kebudayaan seperti yang ada di Sumba Timur, Nusa Tenggara Timur dan Papua Nugini. Di Sumba timur, Elang Laut Perut Putih dianggap sebagai dewa (Forth, 2000), sedangkan di beberapa daerah di Australia dan Papua Nugini, burung ini merupakan salah satu hewan yang dikeramatkan karena dianggap sebagai hewan totemik atau dianggap sebagai penjelmaan roh nenek moyang (Baldwin, 2010).
Populasi spesies ini, terutama di pesisir selatan Jawa, semakin menurun akibat perburuan liar, perdagangan satwa illegal, deforestasi, dan aktivitas manusia lainnya yang menyebabkan burung ini meninggalkan sarang beserta anakan atau telur yang ada di dalamnya (Debus, 2008; Dennis et al., 2011). Banyak spesies elang termasuk Elang Laut Perut Putih sangat sensitive terhadap gangguan terutama selama musim kawin. Gangguan yang ditimbulkan oleh aktivitas manusia dapat menyebabkan pasangan Elang Laut Perut Putih meninggalkan sarangnya dan menelantarkan telur atau juvenilnya. Hal ini menyebabkan turunnya produktivitas sehingga berdampak pada penurunan populasi Elang Laut Perut Putih ( Dennis et al., 2011).
Elang Laut Perut Putih adalah jenis burung pemangsa yang membutuhkan wilayah teritori yang aman dari kompetisi dengan burung pemangsa dan predator lain, predasi, maupun gangguan manusia (Gunawan, 2007). Status konservasi Elang Laut Perut Putih (Haliaeetus leucogaster) ditentukan oleh beberapa parameter, yaitu jangkauan distribusi
4
global, ukuran dan tren populasi (IUCN Red List, 2015). Berdasarkan data IUCN Red List, spesies ini dikategorikan sebagai least concerned (LC) karena populasi globalnya belum mendekati batas ambang kategori vulnerable (Rentan), namun tren populasi global dari Elang Laut Perut Putih cenderung menurun (BirdLife International, 2015). Elang Laut Perut Putih juga menghadapi ancaman kepunahan seperti halnya spesies lain dalam famili Accipitridae. Selain itu, jangkauan distribusi Elang Laut Perut Putih yang sangat luas, dan dengan adanya fragmentasi habitat, memungkinkan terjadinya perubahan pada keanekaragaman genetik dalam satu spesies (Wiley & Lieberman, 2011). Analisis filogenetik yang mendalam perlu dilakukan untuk menggambarkan secara rinci mengenai keanekaragaman genetik spesies ini dan hubungan kekerabatannya dengan spesies Haliaeetus yang lain sebagai salah satu upaya konservasi yang bertujuan untuk melestarikan keanekaragaman genetik dari suatu unit taksonomi (Lerner & Mindell, 2005).
Analisis filogenetik dapat dilakukan dengan menggunakan DNA barcoding. DNA Barcoding adalah teknik identifikasi organisme dengan menggunakan sekuen pendek dari DNA mitokondria dan dapat digunakan untuk mengidentifikasi organisme hingga tingkat spesies dengan cepat dan akurat (Hebert & Gregory, 2005). Data sekuen DNA barcode dapat dijadikan sebagai landasan dalam menyusun pohon filogenetik (Gregory, 2008). DNA barcode dapat merekonstruksi filogeni dengan cara menyeleksi sekuen gen anggota taksa, untuk mengidentifikasi hingga tingkat spesies baik pada spesies yang sudah diidentifikasi maupun spesies yang baru ditemukan. Data sekuen DNA barcode dapat dijadikan sebagai landasan dalam menyusun pohon filogenetik Dalam kaitannya dengan genetika populasi, DNA barcode dapat digunakan sebagai langkah awal dalam menganalisis keanekaragaman genomic dalam satu spesies (Hajibabaei et al., 2007).
Sejauh ini sudah diketahui bahwa Elang Laut Perut Putih membentuk cabang monofiletik dan berkerabat dekat dengan Elang Laut Steller (Haliaeetus pelagicus) dengan jarak genetik 0,104 berdasarkan gen Cytochrome-c Oxydase Subunit I (COI) (Retnaningtyas et al., 2014). Namun demikian, data ini perlu diperkuat dengan parameter dari gen lain untuk menyusun pohon filogenetik yang lebih komprehensif. Untuk memastikan bahwa Elang Laut Perut Putih yang hidup di Jawa Timur tergolong dalam spesies yang sama dengan Elang Laut Perut Putih yang hidup di belahan dunia lain, perlu dilakukan kajian hubungan kekerabatan berdasarkan gen Cytochrome-b (Cyt-b). Gen Cytochrome-b (~1,143 bp) merupakan gen penanda yang paling banyak digunakan untuk mengungkap hubungan kekerabatan di antara taxa yang berkerabat dekat. Untuk burung, sekuen gen Cytochrome b (Cyt-b) efektif untuk merekonstruksi peristiwa filogenetik yang terjadi dalam kurun waktu 20 juta tahun terakhir (Wink, 1995), karena sekuen gen Cyt-b berubah secara lambat selama proses evolusi dan memiliki area dengan variabilitas yang rendah, sehingga berguna untuk menentukan hubungan filogenetik antarspesies (Esposti et al., 1993). Gen Cyt-b memiliki area yang bersifat conserved dan area lain yang menunjukkan adanya variabilitas yang tinggi, dan oleh sebab itu gen ini sangat efektif untuk menentukan jarak filogenetik antar spesies (Esposti, et al., 1993).
Dari data sekuen gen Cyt-b dapat direkonstruksi pohon filogenetik untuk menggambarkan hubungan antar organisme dengan nenek moyang terdekatnya (Gregory, 2008). Dengan membandingkan sekuen gen Cyt-b Elang Laut Perut Putih dengan sekuen gen Cyt-b anggota genus Haliaeetus yang lain dapat diketahui posisi Elang laut Perut Putih pada pohon filogenetik. Kajian filogenetik menggunakan gen tersebut juga dapat menambah informasi mengenai hubungan kekerabatan yang lebih mendalam dan evolusi dari Elang Laut Perut Putih sebagai salah satu upaya konservasi genetik (Lerner &
5
Mindell, 2005; Shephard et al., 2005). Adapun tujuan dari penelitian ini adalah untuk mengetahui sekuen gen Cyt-b Elang Laut Perut Putih (Haliaeetus leucogaster) yang hidup di Jawa Timur dan menyusun topologi pohon filogenetik untuk mengetahui hubungan kekerabatan Elang Laut Perut Putih (Haliaeetus leucogaster) Jawa Timur dengan anggota genus Haliaeetus yang lain berdasarkan sekuen gen Cyt-b.
METODE PENELITIAN
Objek penelitian ini adalah 3 individu Elang Laut Perut Putih (Haliaeetus leucogaster) yang hidup di Pulau Jawa, khususnya di 3 daerah di Jawa Timur, yaitu Tulungagung, Trenggalek, dan Kediri. Untuk memudahkan penelitian, setiap individu diberi kode sampel RR01 untuk individu pertama, RR02 untuk individu kedua, dan RR03 untuk individu ketiga. Sampel darahsebagai material DNA diambil dari vena pectoralis sub clavia yang terdapat di sayap bagian dalam dengan menggunakan syringe 3 ml. Jumlah darah yang diambil sebanyak 1 ml dan meneteskan setiap satu tetes darah yang setara dengan 0,025 gram kedalam microtube yang berisi 1000 μl alkohol absolut. Isolasi DNA murni dari darah dilakukan dengan menggunakan High Pure PCR Template Preparation Kit dari Roche dengan protokol yang dimodifikasi (Hermadhiyanti, et al.,2014). Amplifikasi gen b dengan teknik PCR dilakukan dengan primer hasil desain HL Cyt-b F1 5’– TAG GAA TCT GCC TGC TGA CA-3’ dan HL Cyt-Cyt-b R1 5’–TTA GTG GTT GAG AAG TTT GT- 3’. Kondisi PCR yang digunakan dalam penelitian ini adalah initial denaturaion pada suhu 94°C selama 2,5 menit, kemudian dilanjutkan dengan 32 kali siklus yang terdiri dari denaturasi pada suhu 93°C selama 30 detik, annealing pada suhu 57°C selama 45 detik, dan extension pada suhu 70°C selama 1 menit 30 detik. Selanjutnya adalah final extension pada suhu 70°C selama 5 menit (Lerner & Mindell, 2005). Hasil PCR diperiksa melalui elektroforesis dengan gel agarose 0,8% pada voltase 100 volt 200 A selama 78 menit. Sekuensing dilakukan di First Base Laboratories Malaysia. Analisis filogenetik dilakukan dengan menggunakan software MEGA 6 dengan metode Maximum Likelihood, Neighbour Joining, Minimum Evolution dan Maximum Parsimony.
HASIL DAN PEMBAHASAN
Isolasi DNA Elang Laut Perut Putih (Haliaeetus leucogaster) kode sampel RR01, RR02, dan RR03 dilakukan hingga diperoleh konsentrasi DNA yang cukup dan murni untuk selanjutnya dilanjutkan ke tahap amplifikasi gen dengan teknik Polymerase Chain Reaction (PCR). Pengukuran konsentrasi DNA yang dilakukan menggunakan NanoDrop ND-2000 Spectrophotometer menunjukkan konsentrasi DNA yang diperoleh untuk sampel DNA kode RR01 adalah 252,2 ng/µl; RR02 sebesar 551,3 ng/µl; dan RR03 sebesar 460,6 ng/µl. Ketiga sampel DNA tersebut tidak terkontaminasi RNA maupun protein. Amplifikasi gen Cyt-b menggunakan primer forward HL Cyt-b F1 5’– TAG GAA TCT GCC TGC TGA CA-3’ dan primer reverse HL Cyt-b R1 5’–TTA GTG GTT GAG AAG TTT GT- 3’ menghasilkan fragmen sepanjang ±900 bp. Data hasil sekuensing berupa chromatogram sekuen forward dan reverse masing-masing sampel dibaca menggunakan software Finch TV dan kemudian digabungan dengan menggunakan DNA Baser, menghasilkan sekuen konsensus sekuen forward dan reverse yang diperoleh dari masing-masing sampel adalah RR01 954 bp, RR02 977 bp dan RR03 948 bp.
Masing-6
masing hasil sekuen konsensus kemudian dicocokkan dengan sekuen gen Cyt-b Elang Laut Perut Putih Haliaeetus leucogaster AY987321.1 dari GeneBank. Hasil analisis dengan BLAST menunjukkan bahwa RR01 memiliki kesamaan 99% dengan sekuen query dengan query coverage sebesar 92%; RR02 memiliki kesamaan 99% dengan query coverage sebesar 94%; dan RR03 memiliki kesamaan 99% dengan query coverage sebesar 91%.
Multiple alignment dilakukan menggunakan software ClustalX2 dengan sekuen-sekuen pembanding dari elang laut yang termasuk genus Haliaeetus, yaitu Haliaeetus leucocephalus, Haliaeetus leucogaster, Haliaeetus pelagicus, Haliaeetus sanfordi, Haliaeetus albicilla, Haliaeetus vocifer, Haliaeetus vociferoides, Haliaeetus leucoryphus dan outgrup Cathartes aura dari familia Cathartidae. Hasil multiple alignment menunjukkan bahwa sampel RR01, RR02, dan RR03 memiliki domain-domain sama (conserved) dan domain-domain yang berbeda (variable) dengan spesies lain dalam satu genus.
Pada penelitian sebelumnya berdasarkan gen COI, Elang Laut Perut Putih yang diteliti termasuk dalam spesies Haliaeetus leucogaster dan berada pada clade yang terpisah dari kelompok elang laut dari belahan bumi utara seperti Elang Laut Steller (Haliaeetus pelagicus), Elang Botak (Haliaeetus leucocephalus) dan Elang Ekor Putih (Haliaeetus albicilla) (Retnaningtyas, et al., 2015). Hasil yang sama juga ditunjukkan oleh hasil analisis filogenetik berdasarkan gen Cyt-b.
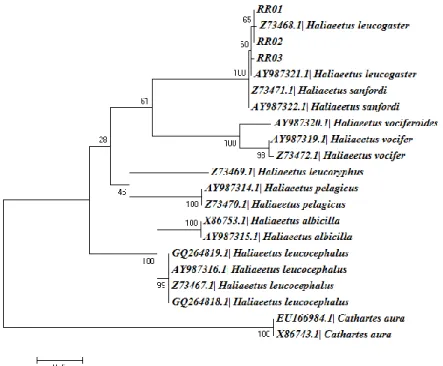
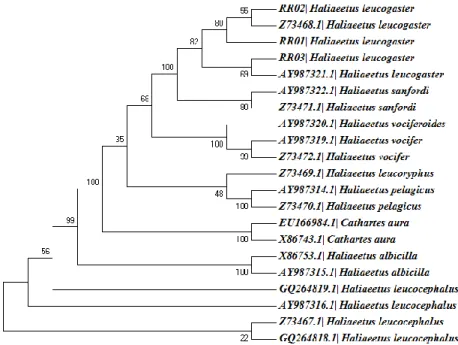
Hasil rekonstruksi pohon filogenetik menggunakan metode Maximum Likelihood (ML) menunjukkan bahwa spesies Haliaeetus leucogaster dan Haliaeetus sanfordi berada pada clade yang sama dengan nilai bootstrap 100. Sampel RR01 membentuk 1 clade dengan RR02 dan Haliaeetus leucogaster Z73468.1 dengan nilai bootstrap 65. Haliaeetus leucogaster RR03 berada pada clade yang sama dengan Haliaeetus leucogaster AY987321.1 dengan nilai bootstrap 60. Hal ini menunjukkan bahwa sampel RR01, RR02 dan RR03 masih tergolong satu spesies dengan Haliaeetus leucogaster AY987321.1 dan Haliaeetus leucogaster Z73468.1 dari GeneBank (Gambanr 3).
Gambar 3. Hasil rekonstruksi pohon filogenetik menggunakan metode Maximum likelihood (ML).
7
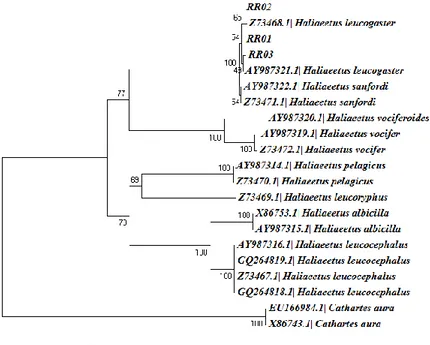
Selain ML, dilakukan pula rekosntruksi pohon filogenetik menggunakan metode Neighbour Joining (NJ) dan Minimum Evolution (ME). Pada pohon filogenetik Neighbour Joining (NJ), spesies Haliaeetus leucogaster dan Haliaeetus sanfordi berada pada satu clade yang sama dengan nilai bootstrap 100. Haliaeetus leucogaster RR01 membentuk 1 clade dengan Haliaeetus leucogaster RR02 dan Haliaeetus leucogaster Z73468.1 dengan nilai bootstrap 65. Haliaeetus leucogaster RR03 dan Haliaeetus leucogaster AY987321. 1 membentuk cabang dengan nilai bootstrap 49. Sampel RR01, RR02, RR03, H. leucogaster Z73468.1 dan AY987321.1 merupakan bagian dari cabang cryptic dengan nilai bootsrap 54. Hal ini juga menunjukkan bahwa sampel RR01, RR02 dan RR03 masih tergolong satu spesies dengan Haliaeetus leucogaster AY987321.1 dan Haliaeetus leucogaster Z73468.1 dari GeneBank (Gambar 4). Hasil rekonstruksi pohon filogenetik menggunakan metode NJ berbeda dengan hasil rekonstruksi menggunakan metode ML. Perbedaannya terletak pada niai bootstrap dan jumlah cabang di dalam clade Haliaeetus leucogaster dan Haliaeetus sanfordi. Pada topologi pohon ML, Haliaeetus sanfordi tidak membentuk cabang yang lebih kecil, sedangkan pada topologi pohon NJ, terlihat bahwa Haliaeetus sanfordi membentuk suatu cabang lebih kecil dengan nilai bootstrap 54. Hal ini disebabkan rekonstruksi menggunakan metode Minimum Evolution (ME) dan Neighbour Joining (NJ) yang didasarkan pada penghitungan jarak genetik (Nei & Saitou, 1987; Peer, 2009), sedangkan metode ML didasarkan pada perhitungan substitusi dan delesi basa nitrogen tiap sekuen DNA yang dianalisis (Felenstein, 1981).
Gambar 4. Hasil rekonstruksi pohon filogenetik menggunakan metode Neighbour Joining. Rekonstruksi pohon filogenetik menggunakan metode Minimum Evolution (ME) menghasilkan topologi yang tidak jauh berbeda dengan Neighbour Joining dan Maximum Likelihood. Pada pohon Mimimum Evolution, sampel RR01, RR02, RR03, Haliaeetus leucogaster AY987321.1 dan Haliaeetus leucogaster Z73468.1 berada pada clade yang sama dengan nilai bootstrap 55. Hal ini berarti, sampel RR01, RR02 dan RR03 termasuk
8
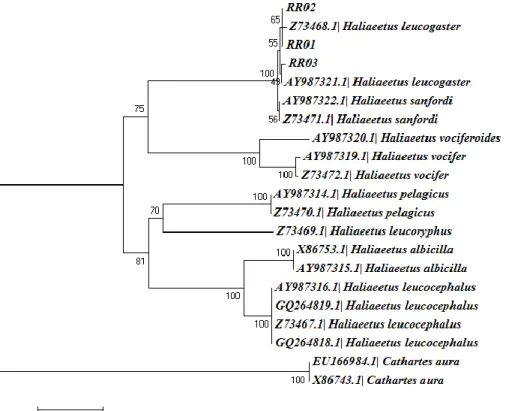
spesies yang sama dengan Haliaeetus leucogaster AY987321.1 dan Haliaeetus leucogaster Z73468.1 (Gambar 5).
Gambar 5. Hasil rekonstruksi pohon filogenetik menggunakan metode Minimum Evolution (ME).
Analisis filogenetik menggunakan metode ML, NJ, dan ME, menunjukkan bahwa dalam clade Haliaeetus leucogaster, terdapat cabang-cabang lebih kecil dengan sampel RR01, RR02 berada pada satu clade dengan Haliaeetus leucogaster Z73468.1 dan sampel RR03 menempati clade yang sama dengan spesies acuan Haliaeetus leucogaster AY987321.1 dan Z73468.1 dari GeneBank dengan nilai bootstrap yang berbeda pada masing-masing pohon. Cabang lebih kecil dalam clade spesies seperti yang terlihat pada ketiga topologi pohon filogenetik ML, NJ dan ME ini menunjukkan bahwa ketiga individu Elang Laut Perut Putih yang diteliti masih tergolong satu spesies dengan spesies acuan dari GeneBank meskipun terdapat variasi dalam sekuen gen Cyt-b-nya. Topologi pohon filogenetik menggunakan metode ML perlu diperjelas dengan pohon Maximum Parsimony (MP) untuk memastikan posisi sampel RR01, RR02 dan RR03 dalam kelompok taksa Haliaeetus leucogaster.
Pada pohon MP terlihat bahwa sampel RR02 secara karakter genetik lebih dekat kekerabatannya dengan Haliaeetus leucogaster Z73468.1 . Sampel RR01 memisah membentuk cabang tersendiri dari RR02 dan H. leucogaster Z73468.1 namun masih tergolong mirip secara karakter genetik dengan kedua sekuen tersebut. Sampel RR03 diketahui memiliki kemiripann terbesar dengan Haliaeetus leucogaster AY987321.1 dan memisah dari Haliaeetus leucogaster RR01, RR02 dan Z73468.1 karena terdapat perbedaan karakter genetik pada sekuen DNA-nya. Namun, perbedaan keempat sekuen DNA tersebut sangat kecil sehingga masih tergolong dalam satu spesies (Gambar 6).
Pohon filogenetik menggunakan metode MP menunjukkan hasil yang berbeda dibandingkan dengan rekonstruksi pohon ML, NJ dan ME. Hal ini disebabkan Maximum
9
Likelihood dan Maximum Parsimony merupakan metode rekonstruksi pohon filogenetk yang didasarkan pada karakter genetik sekuen DNA yang meliputi karakter automorfi, plesiomorfi, dan synapomorfi (Peer, 2009), sedangkan Neighbour Joining dan Minimum Evolution didasarkan pada jarak genetik (Tamura & Nei, 1987). Ide dasar MP adalah hasil rekonstruksi filogenetik yang paling mendekati kebenaran adalah yang menjelaskan data karakter dengan perubahan paling sedikit. Metode Maximum Parsimony digunakan untuk mendeteksi posisi paling mungkin suatu taksa pada pohon filogenetik (Peer, 2009).
Gambar 6. Hasil rekonstruksi pohon filogenetik genus Haliaeetus menggunakan metode Maximum Parsimony (MP).
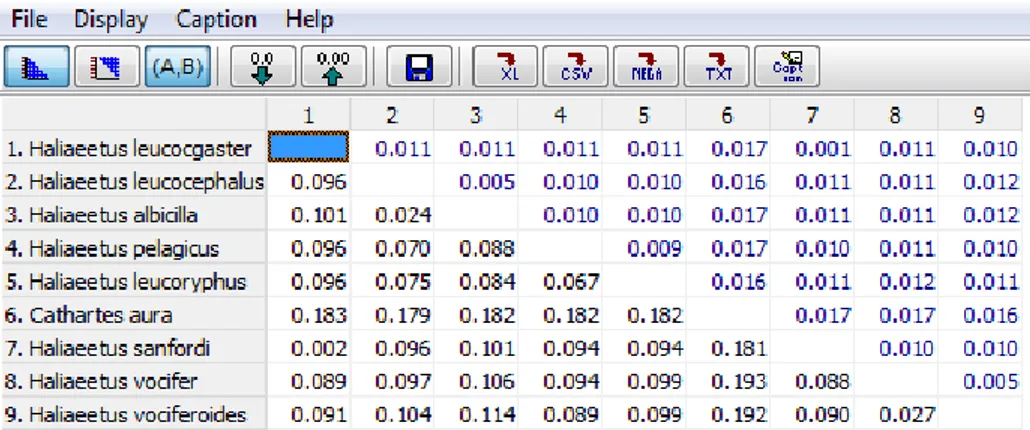
Pernyataan bahwa sampel RR01, RR02 dan RR03 tergolong spesies yang sama dengan spesies acuan didukung oleh hasil analisis jarak genetik pairwise distance antara sampel RR01, RR02 dan RR03 dengan species acuan Haliaeetus leucogaster AY987321.1 dan Z73468.1. Analisis jarak genetik pairwise distance menunjukkan bahwa RR01 merupakan spesies yang sama dengan RR02 karena jarak genetiknya adalah 0,000. Sampel RR02 berkerabat lebih dekat dengan Haliaeetus leucogaster Z73468.1 dengan jarak genetik 0,001 dibandingkan dengan RR03 yang jarak genetiknya adalah 0,002. Sampel RR03 berkerabat lebih dekat dengan Haliaeetus leucogaster AY987321.1 dengan jarak genetik 0,001 dibandingkan dengan RR01 dan RR02 yang berjarak genetik 0,002 (Gambar 7).
Gambar 7. Analisis jarak genetik pairiwise distance antara sampel RR01, RR02 dan RR03 dengan spesies acuan Haliaeetus leucogaster AY987321.1 dan Haliaeetus
10
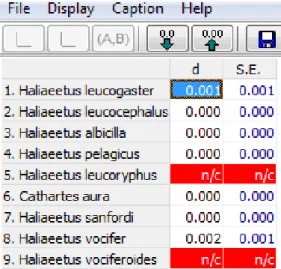
Meskipun ketiga sampel memiliki jarak genetik yang bervariasi dengan spesies acuan, ketiganya masih tergolong satu spesies karena jarak genetiknya kurang dari 0,03. Hal ini didukung oleh hasil analisis jarak genetik intraspesifik yang menunjukkan bahwa kelima sekuen gen Cyt-b Haliaeeetus leucogaster termasuk sampel RR01, RR02 dan RR03 memiliki jarak genetik intraspesifik 0,001 (Gambar 8). Hal ini berarti dalam kelompok spesies Haliaeetus yang dianalisis terdapat variasi genetik sebesar 0,1% dan masih tergolong dalam spesies yang sama.
Gambar 8. Analisis jarak genetik intraspesifik dalam satu kelompok spesies Haliaeetus leucogaster menunjukkan jarak genetik sebesar 0,001.
Hasil rekonstruksi pohon filogenetik menggunakan metode ML, NJ dan ME memiliki kesamaan lain, yaitu ketiga pohon tersebut menghasilkan topologi yang mirip. Ketiganya menunjukkan bahwa kelompok Haliaeetus leucogaster dan Haliaeetus sanfordi berkerabat sangat dekat karena kedua kelompok ini menempati clade yang sama dan membentuk spescies complex, yaitu suatu kelompok spesies yang berkerabat dekat yang sangat mirip secara morfologi sehingga sering dianggap sebagai spesies yang sama (Bickford, et al., 2007), dengan Haliaeetus sanfordi. Jarak genetik interspesifik menunjukkan bahwa dalam satu genus, spesies Haliaeetus leucogaster berkerabat paling dekat dengan Haliaeetus sanfordi dengan jarak genetik 0,02. Jarak genetik 0,02 menunjukkan bahwa Haliaeetus leucogaster dan Haliaeetus sanfordi masih tergolong satu spesies menurut analisis yang dilakukan. Jarak genetik interspesifik terbesar dari spesies Haliaeetus leucogaster dibandingkan dengan spesies lain dalam satu genus adalah dengan Haliaeetus albicilla, yaitu 0,101 (Gambar 9).
11
Gambar 9. Analisis jarak genetik interspesifik dalam genus Haliaeetus menunjukkan bahwa Haliaeetus leucogaster berkerabat paling dekat dengan Haliaeetus
sanfordi dan paling jauh dengan Haliaeetus albicilla.
Berdasarkan rekonstruksi topologi pohon filogenetik menggunakan metode NJ dan ME, diketahui bahwa terdapat kesamaan pada kedua topologi pohon filogenetik yang didapatkan yaitu genus Haliaeetus membentuk suatu cabang monofiletik yang memisah menjadi dua bagian. Kelompok yang pertama terdiri dari spesies Haliaeetus leucogaster termasuk sampel RR01, RR02 dan RR03, Haliaeetus sanfordi, Haliaeetus vociferoides dan Haliaeetus vocifer. Kelompok kedua terdiri dari Haliaeetus pelagicus, Haliaeetus leucoryphus, Haliaeetus albicilla dan Haliaeetus leucocephalus. Kedua kelompok tersebut terpisah oleh faktor distribusi geografis. Kelompok pertama merupakan kelompok spesies yang tersebar di belahan bumi selatan yang memiliki iklim tropis hingga sedang, sedangkan kelompok kedua (H. albicilla, H. leucocephalus, H. leucoryphus dan H. pelagicus) merupakan kelompok yang tersebar di belahan bumi utara (Lerner & Mindell, 2005).
Topologi pohon filogenetik NJ dan ME juga menunjukkan bahwa Haliaeetus leucogaster membentuk species complex dengan Haliaeetus sanfordi. Species complex merupakan suatu kelompok spesies yang berkerabat dekat yang sangat mirip secara morfologi sehingga sering dianggap sebagai spesies yang sama (Bickford, et al., 2007), Hal ini ditunjukkan dengan terbentuknya cabang-cabang kecil dalam satu clade spesies yang sama. Fenomena species complex ini didukung oleh data jarak genetik intraspesies yang menunjukkan bahwa spesies Haliaeetus leucogaster dalam penelitian ini memiliki variasi genetik sebesar 1%. Perbedaan dalam satu spesies ini disebabkan adanya perbedaan asal-usul masing-masing sekuen Elang laut perut putih. Luasnya jangkauan distribusi Elang laut perut putih memungkinkan terjadinya variasi genetik dalam satu populasi.
Selain itu, diketahui pula bahwa spesies Haliaeetus leucogaster dan Haliaeetus sanfordi berada pada clade yang sama walaupun Haliaeetus sanfordi menempati suatu cabang ‘tersembunyi’ atau cryptic dalam clade Haliaeetus leucogaster. Hal ini berarti kedua spesies tersebut memiliki perbedaan secara genetik yang ditunjukkan oleh memisahnya kelompok spesies Haliaeetus leucogaster dari kelompok Haliaeetus sanfordi dengan nilai bootsrap 100 pada topologi pohon MP. Perbedaan interspesifik yang dimiliki oleh kedua spesies tersebut sangat kecil, yaitu 0,2% yang artinya Haliaeetus sanfordi secara genetik masih tergolong satu spesies dengan Haliaeetus leucogaster. Perbedaan 2% diasumsikan dengan divergensi 3-4 juta tahun yang lalu sehingga dapat diperkirakan Haliaeetus sanfordi memisah dari kelompok spesies Haliaeetus leucogaster pada kurang lebih 120.000 tahun yang lalu (Wink et al., 1996).
12
Secara morfologi, Haliaeetus leucogaster dan Haliaeetus sanfordi hampir sama, yang membedakan adalah warna kepala Elang Laut Perut Putih berwarna putih cerah sedangkan warna putih pada kepala Elang Laut Sanfordi lebih gelap dan warna ekor Elang Laut perut putih lebih gelap dibandingkan dengan warna ekor Elang Laut Sanfordi (Wink et al., 1996; Lerner & Mindell, 2005). Kedua spesies ini memiliki perbedaan dalam perilaku dan daya jelajah walaupun mereka hidup pada jenis ekosistem yang sama sehingga dikelompokkan sebagai spesies yang berbeda namun berkerabat dekat (Wink et al., 1996).
KESIMPULAN
Dari penjelasan yang telah disebutkan, dapat disimpulkan bahwa sekuen gen Cytochrome-b (Cyt-b) Elang Laut Perut Putih (Haliaeetus leucogaster) yang hidup di Jawa Timur yang didapatkan pada penelitian ini memiliki panjang ± 980 bp dengan kesamaan dengan sekuen query (Haliaeetus leucogaster AY987321.1) yang ada di GeneBank sebesar 99% dan berdasarkan gen Cyt-b, Elang Laut Perut Putih sampel RR01, RR02 dan RR03 dari Jawa Timur adalah spesies Haliaeetus leucogaster berkerabat sangat dekat dan membentuk species complex dengan Elang Laut Sanfordi (Haliaeetus sanfordi) berdasarkan data sekuen gen Cyt-b.
DAFTAR RUJUKAN
Baldwin, A. 2010. The White-bellied Sea Eagle in the Jervis Bay region: an exploration of the cultural, ecological and conservation significance. Tesis. School of Earth & Environmental Sciences, University of Wollongong.
Bickford, D., D. J. Lohman, N. S. Sodhi, P. K. L. Ng, R. Meier, K. Winker, K. K. Ingram,
& I. Das. 2007. Cryptic Species as A Window on Diversity and Conservation. Trends in Ecology and Evolution 22 (3) : 148-155.
BirdLife International. 2015. Species factsheet: Haliaeetus leucogaster. The IUCN Red List of Threatened Species. (Online) http://www.birdlife.org diakses tanggal 17 Maret 2015.
Brazil, M. 2009. Birds of East Asia: eastern China, Taiwan, Korea, Japan, eastern Russia. Christopher Helm, London.
Debus, S.J.S. 2008. Biology and Diet of the White-bellied Sea-Eagle Haliaeetus leucogaster Breeding in Northern Inland New South Wales. Australian Field Ornithology 2008 (25): 165–193.
Dennis, T. E., McIntosh, R. R. & Shaughnessy, P. D. 2011. Effects of human disturbance on productivity of White-bellied Sea-Eagles (Haliaeetus leucogaster). Emu (111):179–185
13
Esposti, M. D., De Vries, S., Crimi, M., Ghelli, A., Patarnello, T., Meyer, A. 1993. Review Mitochondrial cytochrome b: evolution and structure of the protein. Biochimica et Biophysica Acta, 1143: 243-271
Felsenstein, J. 1981. Evolutionary trees from DNA sequences: A maximum likelihood approach. J. Mol. Evol., 17:368–376.
Ferguson-Lees, J.; Christie, D. A. 2001. Raptors of the world. Christopher Helm, London. Forth, G. 2000. Eastern Sumbanese Bird Classification. Journal of Ethnobiology 20 (2):
161-192
Gegory, T. R. 2008. Understanding Evolutionary Trees. Evo Edu Outreach (1): 121–137. Gunawan & Purwanto, A. A.. 2007. Distribution And Home Range Of White Bellied Sea Eagle (Haliaeetus leucogaster J.M.Gmelin, 1788) at The National Marine Park Of Kepulauan Seribu, Jakarta, Indonesia. Prosiding.
Hajibabaei, M., Singer, G. A. C., Hebert, P. D. N. Hickey, D. A. 2007. DNA Barcoding: How It Complements Taxonomy, Molecular Phylogenetics And Population Genetics. Trends In Genetics Vol.23 (4) : 167-172.
Hebert, P.D. N. & Gregory, T. R. 2005. The Promise of DNA Barcoding for Taxonomy. Systematic Biology 54 (5) :852–859
Hermadhiyanti, W. 2014. Studi Filogenetik Elang Jawa (Nisaetus bartelsi) diantara Elang Genus Nisaetus Berdasarkan Cytochrome-C Oxidase Subunit-1 (COI) DNA Barcode. Skripsi, Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Negeri Malang.
Lerner, H. R.L. & Mindell, D. P. 2005. Phylogeny of eagles, Old World vultures, and other Accipitridae based on nuclear and mitochondrial DNA. Molecular Phylogenetics and Evolution 37 : 327–346
Mirabella, M. F. 2011. Pendekatan Pohon dalam Filogenetik. Bandung: Institut Teknologi Bandung
Nei, M., & N. Saitou. 1987. The Neighbor-joining Method: A New Method for Reconstructing Phylogenetic Trees. Molecular Biology and Evolution 4(4):406-425.
Olsen, P. 2015. Australian Predators of the Sky. Canberra: National Library of Australia Panday, R., Jha, D.K., Thapa, N., Pokharel, B. R. & Aryal, N. K. 2014. Forensic Wildlife Parts and their Product Identification and Individualization Using DNA Barcoding. The Open Forensic Science Journal 7: 6-13
Patwardhan A., Ray S., Roy A. 2014. Molecular Markers in Phylogenetic Studies – A Review. Journal of Phylogenetics and Evolutionary Biology 2: 131. doi:10.4172/2329-9002.1000131
14
Retnaningtyas, R.W., Hermadhiyanti, W., Listyorini, D. 2015. Phylogenetic Study of the White-bellied Sea Eagle (Haliaeetus leucogaster) Based on DNA Barcode Cytochrome-c Oxydase Subunit I (COI). Proceeding of the 9th Asian Raptor Research and Conservation Network (ARRCN) Symposium, Thailand.
Retnaningtyas, R. W., W. Hermadhiyanti, D. A. Rahayu, D.Listyorini. 2015. The Identification of The White-bellied Sea Eagle (Haliaeetus leucogaster) Based on Morphological Characteristics. KnE Proceeding of International Conference on Biological Sciences (ICBS) UGM 2013.
Romin, L. A. & Muck, J. A. (1999). Guidelines For Raptor Protection From Human And Land Use Disturbances. US Fish and Wildlife Service, Salt Lake City, UT. Saitou, N., & M. Nei. 1986. The Number Of Nucleotides Required To Determine The
Branching Order Of Three Species With Special Reference To The Human-Chimpanzee-Gorilla Divergence. J. Molucar Evol. 24: 189-204.
Seibold, I., Helbig, A.J., 1996. Phylogenetic relationships of the sea eagles (genus Haliaeetus): reconstructions based on morphology, allozymes and mitochondrial DNA sequences. Journal of Zoological Systematics Evolutionary Research 34: 103–112.
Shephard, Jill M., Catterall, C. P., Hughes, J. M. 2005. Long-Term Variation in The Distribution of The White-Bellied Sea-Eagle (Haliaeetus leucogaster) Across Australia. Austral Ecology (30): 131–145
Sorenson, M.D., Ast, J.C., Dimcheff, D.E., Yuri, T., Mindell, D.P., 1999. Primers for a PCR-based approach to mitochondrial genome sequencing in birds and other vertebrates. Molecular Phylogenetic and Evolution 12 (2): 105–114.
The IUCN Red List of Threatened Species: Haliaeetus leucogaster. 2012. http://dx.doi.org/10.2305/IUCN.UK.20121.RLTS.T22695097A40326383.en. (Online) Diakses tanggal 23 Januari 2016
Van de Peer, Y., P. Lemey, M. Salemi, & A. M. Vandamme (Eds.). 2009. The Phylogenetic Handbook: A Practical Approach To Phylogenetic Analysis And Hypothesis Testing. London: Cambridge University Press.
Wiley, E. O. & Bruce S. Lieberman. 2011. Phylogenetics: Theory and Practice of Phylogenetic Systematics 2nd Edition. New Jersey: John Wiley and Sons, Inc.
Wink, M. 1995. Phylogeny of Old and New World Vultures (Aves: Accipitridae and Cathartdae) Inferred from Nucleotide Sequences of Mitochondrial Cytochrome-b Gene. Zeitschirft fur Naturforschung 50c : 868-882.
Wink, M., P. Heidrich, & C. Fentzloff. 1996. A mtDNA Phylogeny of Sea Eagles (genus Haliaeetus) Based on Nucleotide Sequences of Cytochrome-b Gene. Biochemical Systematics and Ecology 24 (7/8): 783-791.
15
Zein, M. S. A., & Prawiradilaga, D. M. 2013. DNA Barcode Fauna Indonesia. Jakarta: Kencana Prenadamedia Group.