LAPORAN AKHIR
PENELITIAN DOKTOR BARU
DANA ITS 2020
(Eksplorasi dan Uji Anti Diabetes Senyawa Turunan Oksindola Dalam
Rangka Kemandirian Bahan Baku Obat)
Tim Peneliti :
Arif Fadlan, S.Si., M.Si., D.Sc. (Kimia/FSAD)
Prof. Dr. Mardi Santoso (Kimia/FSAD)
DIREKTORAT RISET DAN PENGABDIAN KEPADA MASYARAKAT
INSTITUT TEKNOLOGI SEPULUH NOPEMBER
SURABAYA
Daftar Isi
Daftar Isi ... i
Daftar Tabel ... ii
Daftar Gambar ... iii
Daftar Lampiran ... iv
BAB I RINGKASAN ... 1
BAB II HASIL PENELITIAN ... 2
BAB III STATUS LUARAN ... 20
BAB IV PERAN MITRA ... 21
BAB V KENDALA PELAKSANAAN PENELITIAN ... 22
BAB VI RENCANA TINDAK LANJUT PENELITIAN ... 23
BAB VII DAFTAR PUSTAKA ... 24
BAB VIII LAMPIRAN ... 25
Daftar Tabel
Tabel 2.1 Data energi minimalisasi (EM) ligan ko-kristal PIM dan ligan 14-35 5 Tabel 2.2 Nilai binding affinity dan RMSD penambatan ulang ko-kristal PIM
terhadap makromolekul 2D0T menggunakan Autodock Vina 8 Tabel 2.3 Nilai RMSD hasil penambatan ulang ligan ko-kristal PIM menggunakan
PyMol 9
Tabel 2.4 Hasil penambatan silang ligan 14-35 11 Tabel 4.5 Hasil penambatan silang senyawa-senyawa pembanding
(11, 12, 13, 40, 41, 42, L-1MT) 13 Tabel 4.6 Interaksi ligan 14-35 terhadap residu makromolekul 2D0T 15
Daftar Gambar
Gambar 2.1 Struktur senyawa turunan oksindola 14-35 2 Gambar 2.2 Struktur kristal IDO1-4PI kompleks (2D0T) 3 Gambar 2.3 Struktur senyawa pembanding 11, 12, 13, 40, 41, 42, dan L-1MT 3 Gambar 2.4 (A) Struktur tiga dimensi (3D) IDO-1 (2D0T); (B) 2D0T
tanpa molekul air; (C) Rantai A 2D0T hasil preparasi 4 Gambar 2.5 Struktur 3D ligan ko-kristal PIM dan ligan 14-35 hasil preparasi 6 Gambar 2.6 Situs dan ukuran grid area penambatan 7 Gambar 2.7 Visualisasi ligan ko-kristal PIM hasil penambatan ulang menggunakan
Autodock Vina 8
Gambar 2.8 Visualisasi ligan ko-kristal PIM (biru) dan ligan ko-kristal PIM hasil
penambatan ulang (magenta) menggunakan PyMol 10 Gambar 2.9 Visualisasi interaksi ligan 14-35 terhadap makromolekul 2D0T 19
Daftar Lampiran
BAB I RINGKASAN
Latar belakang: Beberapa penyakit menjadi penyebab utama kematian di seluruh dunia dan
diabetes menjadi salah satu penyebab kematian yang cukup berbahaya. Di Indonesia, diabetes berada pada peringkat ke-6 sebagai penyakit penyebab kematin pada tahun 2007, namun kemudian melesat menjadi peringkat ke-3 pada tahun 2017 dengan perubahan sebesar 50,1%. Pengembangan agen-agen baru untuk pengobatan diabetes merupakan salah satu fokus utama dalam penanganan diabetes. Oksindola yang memiliki berbagai aktivitas biologis seperti antifungi, antibakteri, antiproliferatif, antikanker, antiinflamatori, antihipertensif, antikonvulsan, antioksidan, antiHIV, dan antidiabetes telah banyak digunakan dalam bidang kesehatan, dan beberapa diantaranya telah dijual bebas (Sunitinib, obat antikanker).
Tujuan: Penelitian bertujuan untuk sintesis senyawa-senyawa berkerangka oksindola dengan
substituen golongan halogen (fluorin, klorin, dan bromin), metil, dan nitro yang dilanjutkan dengan karakterisasi struktur senyawa-senyawa tersebut, uji aktivitas antidiabetes, dan studi penambatan molekular.
Tahapan metode penelitian: Senyawa-senyawa berkerangka oksindola dengan substituen
golongan halogen (fluorin, klorin, dan bromin), metil, dan nitro disintesis melalui reaksi berbagai isatin dengan aseton menggunakan metoda refluks. Reaksi kemudian dilanjutkan dengan eliminasi air menggunakan campuran asam klorida pekat dan asam asetat glasial dengan refluks. Karakterisasi senyawa-senyawa yang terbentuk dilakukan dengan metoda spektroskopi yang meliputi analisis IR, NMR, dan MS. Selanjutnya, uji aktivitas antidiabetes dilakukan melalui evaluasi penghambatan senyawa-senyawa oksindola hasil sintesis terhadap enzim α-glukosidase menggunakan akarbosa sebagai kontrol positif. Lebih lanjut, penambatan molekular dipelajari dengan Autodock Vina dalam PyRx dan visualisasi menggunakan PyMOL.
Luaran yang ditargetkan: Penelitian ini menargetkan sebuah artikel ilmiah yang dipublikasikan
pada jurnal internasional terindeks (Q2) dan diseminasi hasil penelitian pada forum ilmiah internasional.
Ringkasan penelitian berisi latar belakang penelitian,tujuan dan tahapan metode penelitian, luaran yang ditargetkan, kata kunci
BAB II HASIL PENELITIAN
Penelitian dilaksanakan melalui eksperimental laboratorium. Akan tetapi, pelaksanaan penelitian terkendala oleh penutupan kampus dan laboratorium akibat pandemi covid-19. Bahan kimia yang umumnya dapat diperkirakan tiga hingga enam bulan kedatangan juga menjadi tidak dapat diperkirakan akibat pandemi covid-19. Penelitian dilaksanakan di laboratorium sesuai rencana awal sambil menunggu kedatangan bahan kimia dan juga dilaksanakan dengan berbasis teknologi informasi (elektronik/virtual/daring) sesuai Surat Keputusan Kuasa Pengguna Anggaran Deputi Bidang Penguatan Riset dan Pengembangan Kementerian Riset dan Teknologi/Badan Riset dan Inovasi Nasional Republik Indonesia Nomor 31 / EI /KPT / 2020 Tentang Suplemen Panduan Penelitian dan Pengabdian Kepada Masyarakat Pada Masa Pandemi Corona Virus Disease 2019 (Covid-19) perihal Penelitian poin 1 huruf e. Penelitian selanjutnya dilaksanakan dengan penapisan virtual (virtual screening) melalui penambatan molekular (molecular docking) untuk mencapai luaran yang dijanjikan.
2.1 Penambatan Molekular
Penemuan inhibitor potensial IDO1 pada penelitian ini dilakukan melalui studi penambatan molekular berdasarkan konformasi, afinitas ikatan, dan interaksi yang terjadi antara suatu senyawa dan makromolekul target. Studi penambatan dilakukan menggunakan senyawa turunan oksindola
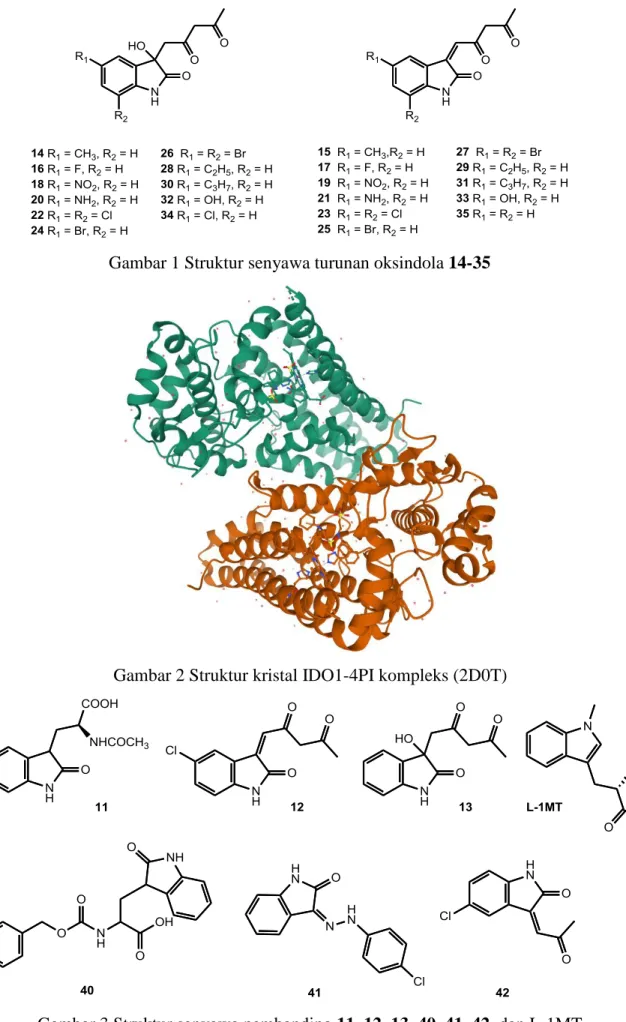
14-35 (Gambar 1) sebagai spesi ligan terhadap enzim IDO1-4PI kompleks dengan identitas 2D0T
(Gambar 2). Pemilihan turunan oksindola 14-35 sebagai spesi ligan didasarkan pada penelitian sebelumnya yang dilaporkan oleh Paul dkk. pada tahun 2017. Hasil penelitian sebelumnya melaporkan beberapa senyawa turunan oksindola dengan potensi terbaik sebagai inhibitor IDO1, yaitu asam (2S)-2-asetamida-3-(2-oksoindolin-3-il)propanoat 11, (Z)-1-(5-kloro-oksoindolin-3-iliden)pentana-2,4-dion 12, 1-(3-hidroksi-oksoindolin-3-il)pentana-2,4-dion 13, asam 2-(((benziloksi)karbonil)amino)-3-(2-oksoindolin-3-il)propanoat 40, (Z)-3-(2-(4-klorofenil)hidra-zono)indolin-2-one 41, (Z)-5-kloro-3-(2-oksopropilidin)indolin-2-one 42 (Gambar 3). Selain itu, penelitian tersebut juga melaporkan penggunaan senyawa 1-metil-L-triptophan (L-1MT) sebagai senyawa pembanding (Gambar 3) [1]. Berdasarkan penelitian tersebut, maka penelitian ini menggunakan senyawa 11, 12, 13, 40, 41, 42, dan L-1MT sebagai senyawa pembanding pada penelitian ini.
Gambar 1 Struktur senyawa turunan oksindola 14-35
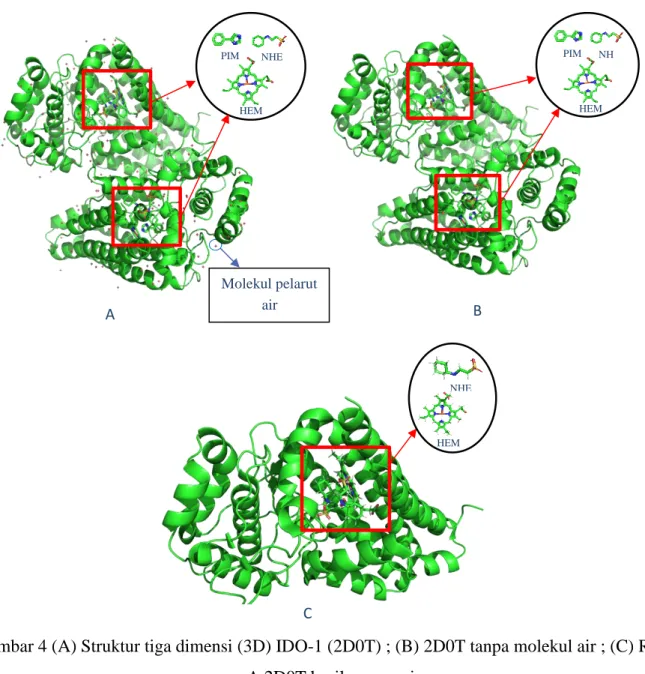
Gambar 2 Struktur kristal IDO1-4PI kompleks (2D0T)
2.2 Preparasi makromolekul 2D0T
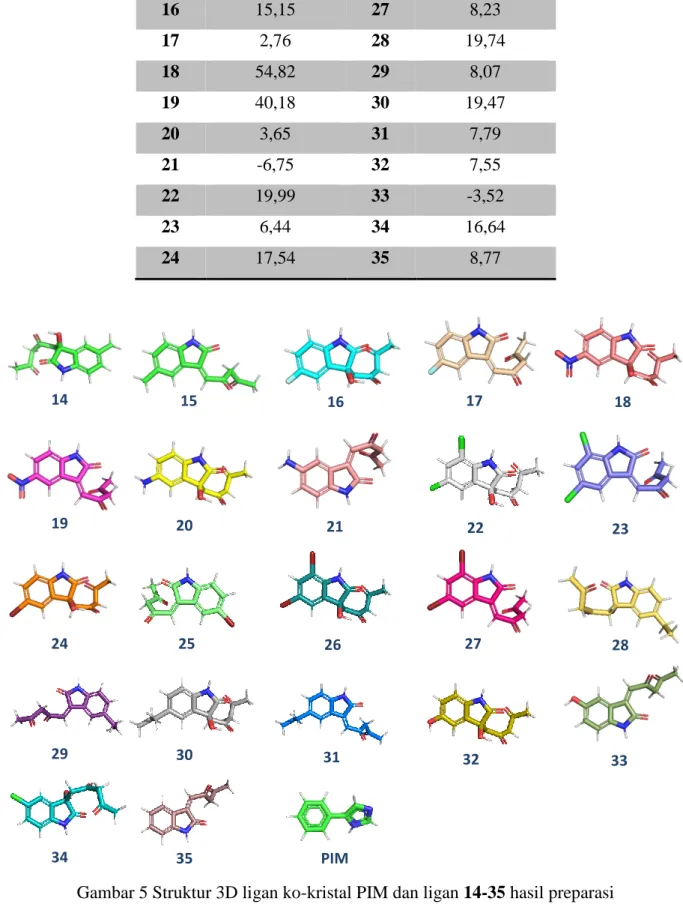
Pada penelitian ini dilakukan penambatan molekular terhadap enzim indolamina 2,3-dioksigenase 1 dengan identitas 2D0T terdiri atas rantai A dan B serta terkompleks dengan tiga ligan ko-kristal, yaitu 4-fenilimidazola (PIM), asam 2-(N-sikloheksilamino) etana sulfonat (NHE), dan protoporfirin IX yang mengandung besi (HEM) (Gambar 4A) [2]. Sebelum dilakukan penambatan, makromolekul terlebih dahulu dipreparasi untuk mengoptimasi dan menciptakan sisi aktif penambatan.
Gambar 4 (A) Struktur tiga dimensi (3D) IDO-1 (2D0T) ; (B) 2D0T tanpa molekul air ; (C) Rantai A 2D0T hasil preparasi
Preparasi makromolekul 2D0T meliputi penghapusan molekul air, penghapusan rantai, penambahan atom-atom hidrogen, dan ekstraksi ligan PIM. Penghapusan molekul air bertujuan untuk mempercepat dan menghindari gangguan pada proses penambatan, dimana molekul air dapat
PIM NHE HEM Molekul pelarut air A PIM NH E HEM B NHE HEM C
menyebabkan variabel algoritma yang diproses menjadi lebih kompleks (Gambar 4B) [3]. Selanjutnya dilakukan pemisahan rantai A dan B dan dilakukan pengukuran root mean square
deviation (RMSD) untuk mengetahui kemiripan rantai A dan B, sehingga proses penambatan dapat
dilakukan pada salah satu rantai saja. RMSD antara dua molekul yang dibandingkan dapat dikatakan identik apabila nilai RMSD ≤ 2 Å [4]. Hasil pengukuran RMSD rantai A dan B menghasilkan nilai sebesar 0,135892 Å, sehingga mengindikasikan bahwa rantai A dan B identik dan proses penambatan dapat dilakukan pada salah satu rantai saja. Selanjutnya, penelitian ini menggunakan rantai A sebagai reseptor penambatan dan dilakukan penghapusan rantai B. Selanjutnya dilakukan penambahan atom hidrogen terhadap rantai A. Preparasi dilanjutkan melalui ekstraksi ligan ko-kristal PIM untuk menciptakan situs ikat proses penambatan, yaitu pada situs ikatan PIM berpusat pada koordinat x = 60 ; y = 53 ; dan z = 18 Å. Ligan PIM yang diekstrak disimpan dalam format file .sdf untuk proses penambatan ulang dan makromolekul 2D0T yang telah dipreparasi disimpan dalam format file .pdb (Gambar 4C).
2.3 Preparasi dan optimasi Ligan Ko-Kristal PIM dan Ligan 14-35
Preparasi dan optimasi ligan ko-Kristal PIM dan ligan 14-35 dilakukan mula-mula melalui penggambaran struktur dua dimensi (2D) ligan 14-35. Selanjutnya, dilakukan penambahan atom-atom hidrogen dan konversi struktur 2D ligan 14-35 menjadi struktur tiga dimensi (3D). Preparasi dan optimasi ligan 14-35 dan ligan ko-kristal PIM dilakukan melalui minimisasi energi dan protonasi pada kondisi pH 7,40. Minimisasi energi dilakukan menggunakan medan gaya molekular
Merck Molecular Force Field 94 (MMFF94), dimana medan gaya MMFF94 dilaporkan lebih
presisi dan intensif dibandingkan dengan medan gaya lain [5]. Minimisasi energi dilakukan untuk menghasilkan ligan dengan konformasi paling stabil untuk proses penambatan molekular selanjutnya [6]. Hasil minimisasi energi dapat diamati pada Tabel 1. Ligan diprotonasi pada kondisi pH 7,40 untuk mengkondisikan ligan pada karakteristik pH subseluler tertentu sebagaimana penelitian yang dilaporkan oleh Kiank dkk. pada tahun 2010 [7]. Data ligan ko-kristal PIM dan ligan 14-35 yang telah dipreparasi selanjutnya disimpan dalam format file .sdf (Gambar 5).
Tabel 1 Data energi minimalisasi (EM) ligan ko-kristal PIM dan ligan 14-35
Ligan EM (Kkal/mol) Ligan EM (Kkal/mol)
PIM 52,1
14 21,42 25 5,37
16 15,15 27 8,23 17 2,76 28 19,74 18 54,82 29 8,07 19 40,18 30 19,47 20 3,65 31 7,79 21 -6,75 32 7,55 22 19,99 33 -3,52 23 6,44 34 16,64 24 17,54 35 8,77
Gambar 5 Struktur 3D ligan ko-kristal PIM dan ligan 14-35 hasil preparasi
14 15 16 17 18
19 20 21 22 23
24 25 26 27 28
29 30 31 32 33
2.4 Penambatan Ulang (Self-docking/Redocking) Ligan Ko-kristal PIM
Gambar 6 Situs dan ukuran grid area penambatan
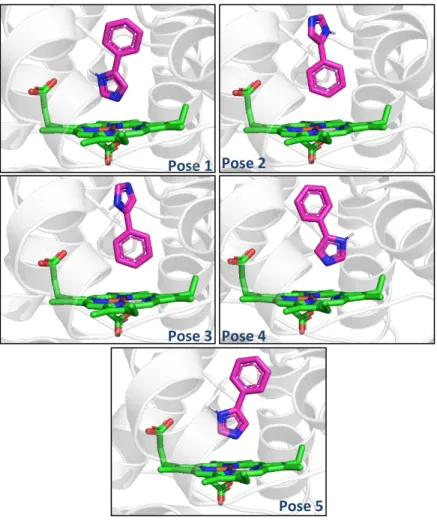
Prosedur penelitian dilanjutkan melalui penambatan ulang ligan ko-kristal PIM yang telah diekstrak dan dioptimasi. Proses penambatan ulang mula-mula dilakukan melalui konversi format data ligan ko-kristal PIM dan data makromolekul 2D0T menjadi format .pdbqt menggunakan aplikasi Pryx sehingga data ligan dan reseptor dapat dibaca dan diproses pada saat penambatan dilakukan oleh program Autodock Vina. Penambatan ulang ligan ko-kristal PIM terhadap makromolekul 2D0T dilakukan menggunakan aplikasi Pryx melalui tools AutoDock Vina dengan ukuran grid dan target penambatan didefinisikan sebesar 12 x 12 x 12 Å pada dimensi x, y, z berturut-turut 60, 53, dan 18 Å (Gambar 6). Hal ini dilakukan dengan tujuan untuk meningkatkan akurasi dan presisi posisi penambatan molekular pada sisi aktif makromolekul 2D0T. Hasil penambatan ulang ligan ko-kristal PIM menghasilkan lima pose penambatan (Gambar 7) dengan hasil afinitas ikatan dapat diamati pada Tabel 2.
Gambar 7 Visualisasi ligan ko-kristal PIM hasil penambatan ulang menggunakan Autodock Vina Tabel 2 Nilai binding affinity dan RMSD penambatan ulang ko-kristal PIM terhadap makromolekul 2D0T menggunakan Autodock Vina
Hasil penambatan ulang menunjukkan pose-1 memiliki nilai binding affinity paling rendah sebesar -6,4 Kkal/mol dengan nilai RMSD sebesar 0 untuk upper bound maupun lower bound. Program AutoDock Vina menyusun ulang atom-atom ligan ko-kristal PIM hasil penambatan sesuai dengan kerangka torsional dan sudut ikatan struktur ligan tersebut sehingga semua pose yang dihasilkan (pose-1 hingga pose-5) memiliki korespondensi langsung satu sama lain, tetapi tidak
Pose Binding Affinity (Kkal/mol) RMSD/ub RMSD/lb
1 -6,4 0 0 2 -6,1 4,645 2,368 3 -6 4,389 2,311 4 -5,4 1,826 1,333 5 -3,9 0,863 0,863 Pose 1 Pose 2 Pose 3 Pose 4 Pose 5
dengan struktur ligan ko-kristal PIM dalam keadaan sebenarnya dalam kristal makromolekul [4]. Hal ini terbukti pada data RMSD yang bernilai 0 pada pose-1. Sehingga, data hasil penambatan ulang ligan ko-kristal PIM dianalisa lebih lanjut menggunakan aplikasi PyMol untuk mengetahui
pose terbaik hasil penambatan yang memiliki posisi dan interaksi paling mendekati dengan ligan
ko-kristal PIM sebenarnya dalam kristal makromolekul 2D0T (Tabel 3).
Tabel 3 Nilai RMSD hasil penambatan ulang ligan ko-kristal PIM menggunakan PyMol
Pose RMSD 1 0,019 2 0,337 3 0,341 4 1,435 5 0,449
Tabel 3 menunjukkan bahwa semua pose hasil penambatan ulang ligan ko-kristal PIM memiliki nilai RMSD < 2 Å, dengan pose-1 memiliki nilai RMSD terendah. Selain itu, hasil analisis secara visualisasi menujukkan pose 1 memiliki orientasi dan posisi yang paling mendekati dengan ligan ko-kristal PIM sebenarnya dalam kristal makromolekul 2D0T (Gambar 8).
Gambar 8 Visualisasi ligan ko-kristal PIM (biru) dan ligan ko-kristal PIM hasil penambatan ulang (magenta) menggunakan PyMol
Berdasarkan hal ini, metode penambatan ulang yang dilakukan dapat menghasilkan pose dengan interaksi dan konformasi yang identik dengan ligan ko-kristal PIM sebenarnya dalam makromolekul 2D0T. Hasil ini mengonfirmasi bahwa metode penambatan ulang dapat diterima dan valid sehingga prosedur penambatan molekular ini dapat digunakan pada tahap berikutnya, yaitu penambatan molekular silang dengan ligan 14-35.
2.5 Penambatan Silang Ligan 14-35
Penambatan silang ligan 14-35 dilakukan sebagaimana penambatan ulang ligan ko-kristal PIM, yaitu menggunakan aplikasi Pryx melalui tools AutoDock Vina dengan pengaturan ukuran
grid target sebesar 12 x 12 x 12 Å pada dimensi x, y, z berturut-turut 60, 53, dan 18 Å. Penambatan
silang berjalan dalam waktu sekitar 1-3 menit. Hasil penambatan silang ligan 14-35 dapat dilihat pada Tabel 4.
Pose 1 Pose 2
Pose 3 Pose 4
Tabel 4 Hasil penambatan silang ligan 14-35
Ligan Pose Binding Affinity(Kkal/mol) rmsd/ub rmsd/lb
14 1 -0,4 0 0 2 2,3 2,916 1,661 3 2,4 4,622 2,067 15 1 -0,4 0 0 2 0,5 5,972 3,087 3 0,8 1,391 1,25 4 2,2 2,239 1,554 16 1 -1,1 0 0 2 1 5,121 2,559 17 1 -1,5 0 0 2 -1 0,174 0,174 3 -0,1 5,939 3,53 18 1 3,1 0 0 2 3,8 4,146 2,025 3 5,4 4,283 1,962 4 5,4 5,107 1,917 19 1 2,6 0 0 2 2,8 0,827 0,806 3 3,1 6,258 2,227 20 1 -0,6 0 0 2 2 4,852 2,249 3 2,1 5,133 2,645 21 1 -0,4 0 0 2 0,7 1,358 1,24 3 0,7 5,944 3,39 4 0,8 1,331 1,152 5 2,2 2,482 1,752 22 1 1,9 0 0 2 3 2,87 1,493 23 1 2,7 0 0
2 4,5 5,995 3,816 24 1 0,7 0 0 2 3,3 1,914 1,068 3 3,5 4,643 2,446 25 1 2,4 0 0 2 2,6 5,983 3,674 3 4,5 5,562 3,369 26 1 5,1 0 0 27 1 5,6 0 0 2 7,5 2,767 1,816 3 7,7 6,068 3,946 28 1 -0,1 0 0 29 1 0,4 0 0 2 1,6 6,052 2,918 3 1,9 0,996 0,915 30 1 1,4 0 0 31 1 1,4 0 0 2 2,5 6,339 2,94 32 1 -0,9 0 0 2 1,5 5,119 2,324 3 1,6 2,802 1,948 4 1,9 4,843 1,988 33 1 -0,5 0 0 2 -0,4 0,93 0,87 3 -0,2 5,919 1,902 4 2 2,553 2,129 5 2,3 5,851 1,921 34 1 -0,4 0 0 2 2,4 2,936 2,11 3 2,4 4,658 2,435 35 1 -0,7 0 0 2 -0,6 5,709 2,974 3 -0,4 1,671 1,293
4 0,3 5,991 2,864 5 0,5 1,372 1,183 6 0,8 5,709 2,748 7 1,1 2,505 1,685 8 1,6 5,978 2,919 9 1,7 2,194 1,698
Data penambatan pada Tabel 4 menunjukkan rentang nilai binding affinity pose terbaik (pose-1) masing-masing ligan 14-35 sebesar -1,5 hingga 5 Kkal/mol, dimana terdapat 11 ligan dengan nilai binding affinity negatif yaitu ligan 14, 15, 16, 17, 20, 21, 28, 32, 33, 34, dan 35. Binding affinity menunjukkan kekuatan dan afinitas interaksi antara ligan dan makromolekul protein, dimana semakin rendah energi afinitas pengikatan maka semakin kuat interaksi antara ligan dan makromolekul protein [8].
Selanjutnya, penelitian ini melakukan penambatan silang dengan ligan pembanding, yaitu ligan asam (2S)-2-asetamida-3-(2-oksoindolin-3-il)propanoat 11, (Z)-1-(5-kloro-oksoindolin-3-iliden)pentana-2,4-dion 12, 1-(5-kloro-3-hidroksi-oksoindolin-3-il)pentana-2,4-dion 13, asam 2-(((benziloksi)karbonil)amino)-3-(2-oksoindolin-3-yl)propanoat 40, (Z)-3-(2-(4-klorofenil) hidrazono) indolin-2-one 41, (Z)-5-kloro-3-(2-oksopropilidin)indolin-2-one 42, dan 1-metil-L-triptophan (L-1MT). Hasil penambatan ligan pembanding dapat diamati pada Tabel 5.
Tabel 5 Hasil penambatan silang senyawa-senyawa pembanding (11, 12, 13, 40, 41, 42, L-1MT)
Ligan Energi Minimalisas(Kkal/mol) Pose Binding Affinity(Kkal/mol) rmsd/ub rmsd/lb 11 -8, 87 1 -0.5 0 0 2 0.4 2.105 1.671 3 0.8 2.473 1.11 4 1.4 5.323 2.484 5 2.2 5.076 2.183 12 4,37 1 1,1 0 0 2 1,2 5,935 3,586 3 1,7 1,025 0,965 4 1,7 1,023 1,002 13 20,86 1 -0,5 0 0
2 0,8 4,975 2,125 3 2,2 3,456 2,025 4 2,2 2,457 1,684 5 2,3 1,949 1,555 6 2,4 5,448 1,971 40 -31,94 1 10,1 0 0 2 11,1 2,436 1,531 3 12,3 6,052 1,92 41 101, 87 1 7,4 0 0 2 8 6,189 2,786 3 8,5 6,085 3,162 4 8,9 5,83 2,621 5 9,1 2,741 1,972 42 15,26 1 -4,6 0 0 2 -3 0,559 0,559 3 -2,9 4,991 3,088 L-1MT 73,9 1 -2,8 0 0 2 -2,1 4,657 2,736 3 -1,3 3,037 1,729 4 -0,9 5,144 2,88 5 -0,7 1,802 1,569
Hasil penambatan ligan pembanding pada Tabel 5 menunjukkan energi afinitas pengikatan pada masing-masing pose terbaik (pose nomor 1) pada rentang nilai -4,6 hingga 10,1 Kkal/mol. Berdasarkan hasil penambatan pada Tabel 4 dan Tabel 5 dapat disimpulkan bahwa ligan 14-35 berpotensi sebagai inhibitor IDO-1 karena nilai energi afinitas pengikatan ligan 14-35 berada dalam rentang nilai energi afinitas pengikatan senyawa-senyawa pembanding (11, 12, 13, 40, 41, 42, L-1MT). Selain itu, ligan 16 dan 17 memiliki energi afinitas pengikatan mendekati energi afinitas pengikatan L-1MT dan memiliki energi afinitas pengikatan yang lebih rendah dari yang lainnya pada penambatan silang ligan 14-35, sehingga dapat dikembangkan dan diteliti lebih lanjut.
2.6 Visualisasi Hasil Penambatan Silang Ligan 14-35
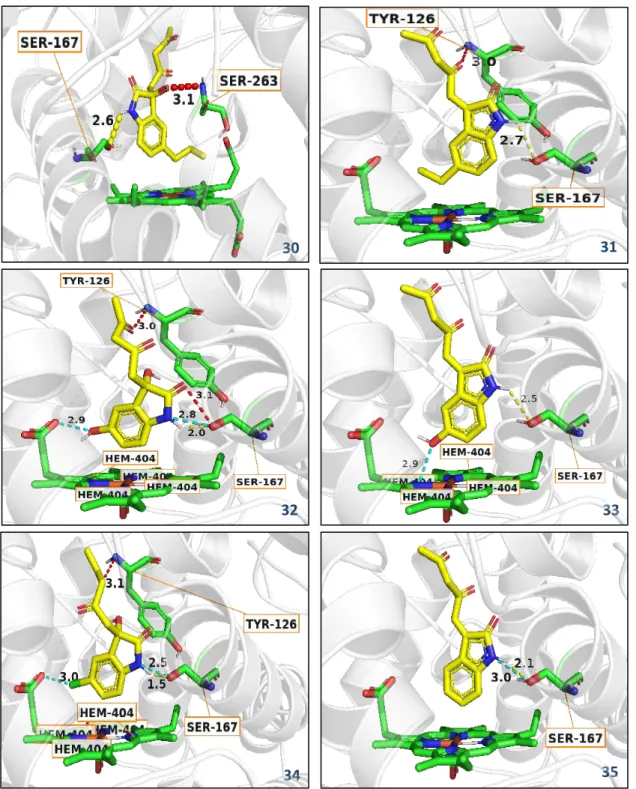
Penelitian kemudian dilanjutkan untuk menganalisis interaksi antara ligan 14-35 hasil penambatan dengan makromolekul 2D0T pada jarak ≤ 3,2 Å. Pemilihan jarak ini didasarkan pada penggolongan kekuatan ikatan hidrogen berdasarkan jarak, yaitu antara 2,2 – 2,5 Å yang berarti kuat dan sebagian besar kovalen, antara 2,5 – 3,2 Å yang merupakan sedang dan sebagian besar elektrostatik, dan 3,2 – 4,0 Å yang merupakan ikatan yang lemah dan elektrostatik [9].
Tabel 6 Interaksi ligan 14-35 terhadap residu makromolekul 2D0T
Ligan Interaksi ligan-residu pada jarak ≤ 3,2 Å (jarak dalam Å)
14 Ser167 (1,5 & 2,5), Tyr126 (3,1)
15 Ser167 (2,6), Tyr126 (3,2)
16 Ser167 (2,0 ; 2,8 ; 3,1), Tyr126 (3,1)
17 Ser167 (2,3 & 3,1)
18 Ser167 (2,5 ; 2,7 ; 2,9), Ser263 (2,8), Ala264 (3,2)
19 Ser167 (2,4 & 3,2), Ser263 (2,4), Val125 (2,6), Tyr126 (3,1), Ala264 (1,8 & 2,8)
20 Ser167 (2,0 ; 2,8 ; 3,1), Ser263 (2,8), Tyr126 (3,0)
21 Ser167 (2,4 & 3,1)
22 Ser167 (2,3 & 2,4), Ser263 (3,1)
23 Ser167 (2,3 ; 2,3 ; 3,2), Val125 (3,1), Tyr126 (3,1)
24 Ser167 (1,4 & 2,4), Tyr126 (3,1)
25 Ser167 (2,6 ; 2,7 ; 3,2), Ser263 (1,8 & 2,7), Ala264 (2,7), Gly265 (2,3 & 3,0)
26 Ser167 (2,1 & 2,4), Ser263 (3,2)
27 Ser167 (1,9 & 2,6), Val125 (3,1), Tyr126 (3,1)
28 Ser167 (2,5 & 3,2), Ser263 (3,1)
29 Ser167 (2,4 & 3,1), Tyr126 (3,0)
30 Ser167 (2,6), Ser263 (3,1)
31 Ser167 (2,7), Tyr126 (3,0)
32 Ser167 (2,0 ; 2,8 ; 3,1) , Tyr126 (3,0)
33 Ser167 (2,5)
34 Ser167 (1,5 & 2,5), Tyr126 (3,1)
Tabel 6 melampirkan data interaksi ligan 14-35 dengan residu asam amino pada makromolekul 2D0T. Hasil analisis menunjukkan bahwa gugus amida indola pada ligan 14-35 secara umum berinteraksi dengan gugus hidroksil residu Ser167, kecuali untuk ligan 16 dan 18. Pada ligan 16 terjadi dua interaksi antara gugus karbonil pada rantai pentana dan gugus amida terhadap residu Ser167. Ligan 18 menunjukkan adanya interaksi antara residu Ser167 dengan gugus nitro pada posisi-5 cincin benzena. Paul dkk pada tahun 2017 melaporkan bahwa interaksi ligan dengan residu Ser167 dan Tyr126 sangat penting dan krusial untuk meningkatkan afinitas terhadap makromolekul 2D0T [1]. Interaksi ligan 14-35 terjadi melalui gugus karbonil rantai pentana ligan dengan gugus hidroksil residu Tyr126. Ligan-ligan yang menunjukkan interaksi dengan residu Tyr126 adalah 14, 15, 16, 19, 20, 23, 24, 27, 29, 31, 32, 34. Hal ini mengindikasikan bahwa residu Ser167 dan Tyr126 memiliki peranan paling penting pada interaksi ligan dengan makromolekul 2D0T. Residu lain yang berinteraksi dengan ligan 14-35 adalah residu Ser263, Ala264, Val125, Gly265. Selain interaksi dengan residu asam amino, ligan 16, 20, 32, dan 34 menunjukkan interaksi dengan 7-propanoat pada lengan heme. Selain itu, beberapa ligan juga menunjukkan interaksi dengan atom N-heme yaitu ligan 17, 18, 21, 22, 23, 26, 27, dan 33. Visualisasi interaksi ligan
14-35 dengan makromolekul 2D07 dapat dilihat pada Gambar 9.
20 21
14 15
18 19
22 23
24 25
26 27
Gambar 9 Visualisasi interaksi ligan 14-35 terhadap makromolekul 2D0T
30 31
32 33
BAB III STATUS LUARAN
Luaran penelitian ini yaitu satu artikel ilmiah yang dipublikasikan pada jurnal internasional terindeks (Q2) dan diseminasi hasil penelitian pada forum ilmiah internasional. Naskah artikel ilmiah telah disusun dan siap didaftarkan. Hasil penelitian telah disampaikan dalam The 1st International Symposium On Health Sciences pada 22-23 September 2020 yang diselenggarakan oleh Fakultas Ilmu Kesehatan Universitas Jenderal Soedirman.
BAB IV PERAN MITRA
BAB V KENDALA PELAKSANAAN PENELITIAN
1. Akses terbatas kampus dan laboratorium sejak pertengahan maret hingga saat ini membatasi pelaksanaan penelitian melalui eksperimen di laboratorium.
2. Pelaksanaan protokol kesehatan dalam eksperimen di laboratorium mengurangi efektifitas penyelesaian penelitian.
3. Pengadaan bahan kimia yang umumnya dapat diperkirakan tiga hingga enam bulan kedatangan menjadi tidak dapat diperkirakan akibat pandemi covid-19.
BAB VI RENCANA TINDAK LANJUT PENELITIAN
Rencana tindak lanjut penelitian ini selanjutnya adalah:
1. Mendaftarkan (submit) naskah (manuskrip) hasil penambatan molekular pada jurnal internasional bereputasi Q2.
2. Melanjutkan penelitian sesuai dengan rencana awal berupa eksperimental di laboratorium. Studi korelasi hasil penambatan molekular selanjutnya dapat dilakukan terhadap hasil eksperimental laboratorium.
BAB VII DAFTAR PUSTAKA
[1] S. Paul et al., “Synthesis and evaluation of oxindoles as promising inhibitors of the immunosuppressive enzyme indoleamine 2,3-dioxygenase 1,” Medchemcomm, vol. 8, no. 8, pp. 1640–1654, 2017, doi: 10.1039/c7md00226b.
[2] H. Sugimoto, S. -i. Oda, T. Otsuki, T. Hino, T. Yoshida, and Y. Shiro, “Crystal structure of human indoleamine 2,3-dioxygenase: Catalytic mechanism of O2 incorporation by a heme-containing dioxygenase,” Proc. Natl. Acad. Sci., vol. 103, no. 8, pp. 2611–2616, Feb. 2006, doi: 10.1073/pnas.0508996103.
[3] J. C. Cole, J. W. M. Nissink, and R. Taylor, “Protein-Ligand Docking and Virtual Screening with GOLD,” In J. Alvarez & B. Shoichet (Eds.), Virtual Screening in Drug
Discovery (pp. 379–415). CRC Press, 2005.
[4] E. W. Bell, and Y. Zhang, “DockRMSD: An Open-source Tool for Atom Mapping and RMSD Calculation of Symmetric Molecules Through Graph Isomorphism," J. of
ChemInfor.., vol. 11, no. 40, pp 1-9, 2019, doi: 10.1186/s13321-019-0362-7.
[5] A. Jász, A. Rák, I. Ladjánszki, and G. Cserey, “Optimized GPU Implementation of Merck Molecular Force Field and Universal Force Field,” J.Mol. Struc., vol. 1188, pp. 227–233, 2019, doi: 10.1016/j.molstruc.2019.04.007.
[6] J. Mcmurry, Organic Chemistry, Enhanced Edition.. Cengage Learning., 2010. [7] C. Kiank, J. P. Zeden, S. Drude, G. Domanska, G. Fusch, W. Otten, and C. Schuett,
“Psychological Stress-Induced, IDO1-Dependent Tryptophan Catabolism: Implications on Immunosuppression in Mice and Humans,"PLoS ONE, vol. 5, no. 7, pp 1-12, 2010, doi: 10.1371/journal.pone.0011825.
[8] C. M. Nisha, A. Kumar, P. Nair, N. Gupta, C. Silakari, T. Tripathi, and A. Kumar,
“Molecular Docking and in silico ADMET Study Reveals Acylguanidine 7a as A Potential Inhibitor of β -secretase.," Adv. in Bioinfor., pp 1-6, 2016, doi: 10.1155/2016/9258578. [9] J.Yu, W. Chen, C. Wu, and H. Chen, “PEG-Protein Interaction Induced Contraction of NalD
BAB VIII LAMPIRAN
LAMPIRAN 1 Tabel Daftar Luaran
Program : Penelitian Doktor Baru
Nama Ketua Tim : Arif Fadlan, S.Si., M.Si., D.Sc.
Judul : Eksplorasi Dan Uji Anti Diabetes Senyawa Turunan Oksindola Dalam Rangka Kemandirian Bahan Baku Obat
1.Artikel Jurnal
No Judul Artikel Nama Jurnal Status Kemajuan*) 1 Molecular Docking and ADME/T
Study on Oxindole Derivatives Against Indoleamina 2,3-dioksigenase 1
Journal of King Saud University Science
Persiapan
*) Status kemajuan: Persiapan, submitted, under review, accepted, published 2. Artikel Konferensi
No Judul Artikel Nama Konferensi (Nama Penyelenggara, Tempat,
Tanggal)
Status Kemajuan*)
1 A Study of Energy Minimization Infuence on The Molecular Docking of Acetylacetone-Based Oxindole Derivatives
(Fakultas Ilmu kesehatan Universitas Jenderal Soedirman, Puwokerto, 22-23 September 2020)
presented
*) Status kemajuan: Persiapan, submitted, under review, accepted, presented 3. Paten
No Judul Usulan Paten Status Kemajuan
- - -
*) Status kemajuan: Persiapan, submitted, under review 4. Buku
No Judul Buku (Rencana) Penerbit Status Kemajuan*)
- - - -
*) Status kemajuan: Persiapan, under review, published 5. Hasil Lain
No Nama Output Detail Output Status Kemajuan*)
- - - -
6. Disertasi/Tesis/Tugas Akhir/PKM yang dihasilkan
No Nama Mahasiswa NRP Judul Status*) 1 Frans Josaphat 01211640000051 Penambatan Molekular Turunan
Oksindola Terhadap
Indoleamina 2,3-dioksigenase 1 (IDO-1) dan Analisis
Farmakofornya
Lulus 2020