commit to user
i
DIVERSITAS GENETIK INTRA DAN INTER SPESIES SAPI BALI DARI
SUMBAWA DAN SAPI ACEH BERDASARKAN ANALISIS
MIKROSATELIT
Skripsi
Untuk memenuhi sebagian persyaratan guna memperoleh gelar Sarjana Sains
Oleh:
Prasasti Wahyu Haryati
M0406048
JURUSAN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
UNIVERSITAS SEBELAS MARET
SURAKARTA
commit to user
commit to user
iii
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi ini adalah hasil penelitian saya sendiri
dan tidak terdapat karya yang pernah diajukan untuk memperoleh gelar
kesarjanaan di suatu perguruan tinggi, serta tidak terdapat karya atau pendapat
yang pernah ditulis atau diterbitkan oleh orang lain, kecuali secara tertulis diacu
dalam naskah ini dan disebutkan dalam daftar pustaka.
Apabila dikemudian hari dapat ditemukan adanya unsur penjiplakan maka gelar
kesarjanaan yang telah diperoleh dapat ditinjau dan /atau dicabut.
Surakarta, Juli 2011
Prasasti Wahyu H
commit to user
iv
Diversitas Genetik Intra dan Inter Spesies Sapi Bali dari Sumbawa dan Sapi Aceh Berdasarkan Analisis Mikrosatelit
Prasasti Wahyu Haryati
Jurusan Biologi, Fakultas Matematika Dan Ilmu Pengetahuan Alam, Universitas Sebelas Maret, Surakarta
ABSTRAK
Sapi-sapi yang terdapat di Indonesia mempunyai karakteristik yang berbeda-beda. Perbedaan karakteristik tersebut salah satunya disebabkan karena adanya diversitas genetik. Salah satu penanda genetik untuk mengetahui diversitas genetik adalah mikrosatelit. Tujuan dari penelitian ini adalah untuk mengetahui diversitas genetik intra dan inter spesies sapi Bali dari Sumbawa dan sapi Aceh berdasarkan analisis mikrosatelit.
Penelitian ini menggunakan sampel darah sapi Bali dari Sumbawa dan sapi Aceh yang di ambil menggunakan metode venopuncture, masing-masing sebanyak 20 individu. DNA diekstrak dari total darah dengan menggunakan teknik Wizard Genomic Purification System. Fragmen DNA diamplifikasi dengan PCR menggunakan primer berturut-turut ETH225, TGLA227, INRA005, BM1824 dan MM12. DNA produk PCR dideteksi menggunakan teknik elektroforesis gel poliakrilamid 12% yang dilanjutkan dengan metode pewarnaan ethidium bromida. Data yang diperoleh dianalisis dengan menggunakan program POPGENE version 1.31.
Hasil analisis alel-alel mikrosatelit menunjukkan bahwa jumlah alel setiap lokus pada intra maupun inter spesies sapi Bali dari Sumbawa dan sapi Aceh bervariasi antara 1 sampai 3 alel. Rata-rata nilai Shannon's Information index pada intra spesies sapi Bali dari Sumbawa adalah 0.45, intra spesies sapi Aceh 0.75, sedangkan pada inter spesies sapi Bali dari Sumbawa dan Aceh 0.75. Rata-rata nilai heterozigositas pada intra spesies sapi Bali dari Sumb awa 0.46, intra spesies sapi Aceh 0 68, sedangkan pada inter spesies sapi Bali dari Sumbawa dan Aceh 0.57. Hasil ini menunjukkan bahwa keragaman genetik pada intra spesies sapi Aceh lebih tinggi dibandingkan intra spesies sapi Bali dari Sumbawa dan inter spesies sapi Bali dari Sumbawa dan Aceh.
Kata kunci : Sapi Bali dari Sumbawa, sapi Aceh, mikrosatelit, diversitas
commit to user
v
Genetic Diversity Intra and Inter Species Bali Cattle from Sumbawa and
Aceh Cattle BasedMicrosatellite Analysis
Prasasti Wahyu Haryati
Department of Biology, Faculty of Mathematics and Natural Sciences, Sebelas Maret University, Surakarta
ABSTRACT
The cattle were found in Indonesia have different characteristics. One of the difference characteristics due to genetic diversity. One of the genetic markers to determine genetic diversity is a microsatellite. The purpose of this study was to investigate intra and inter genetic diversity of Bali cattle from Sumbawa and Aceh cattle based microsatellite analysis.
This study used blood samples from Bali cattle from Sumbawa and Aceh cattle took with venopuncture methods, each of 20 individuals. Total DNA extracted from blood by using the Wizard Genomic Purification System. DNA fragment was amplified by PCR using the primers are ETH225, TGLA227, INRA005, BM1824 and MM12. DNA PCR product was detected using of 12% polyacrylamide gel electrophoresis followed by ethidium bromide staining method. Data were analyzed using the program POPGENE version 1:31.
Results of analysis of microsatellite alleles indicated that the number of alleles per locus in the intra and inter species Bali cattle from Sumbawa and Aceh cattle varies between 1 to 3 alleles. The average value of Shannon's Information index on intra species Bali cattle from Sumbawa was 0,45, intra species Aceh cattle 0,75, while the inter species of Bali cattle from Sumbawa and Aceh cattle were 0,75. The average value of heterozygosity in intra species of Bali cattle from Sumbawa was 0,46, intra-species of Aceh cattle was 0,68, while the inter species of Bali cattle from Sumbawa and Aceh cattle were 0,57. These results suggest that genetic diversity intra species in Aceh cattle was higher than intra species Bali cattle from Sumbawa and inter-species Bali cattle from Sumbawa and Aceh cattle.
Key words: Bali cattle from Sumbawa, Aceh cattle, microsatellite, genetic
commit to user
vi
MOTTO
Belajarlah dari kesalahan orang lain. Anda tak dapat hidup lebih lama untuk melakukan semua kesalahan itu sendiri.
(Martin Vanbee)
Orang-orang yang sukses telah belajar membuat diri mereka melakukan hal yang harus dikerjakan ketika hal itu memang harus dikerjakan, entah mereka
menyukainya atau tidak.
(Aldus Huxley)
Bersikaplah kukuh seperti batu karang yang tidak putus-putusnya dipukul ombak, ia tidak saja berdiri kukuh, bahkan ia menenteramkan amarah ombak dan
gelombang itu
(Marcus Aurelius)
Jadilah kamu manusia yang pada kelahiranmu semua orang tertawa bahagia, tetapi hanya kamu sendiri yang menangis, dan pada kematianmu semua orang menangis
sedih, tetapi hanya kamu sendiri yang tersenyum.
(Mahatma Gandhi)
Kita melihat kebahagiaan itu seperti pelangi, tidak pernah berada diatas kepala kita sendiri, tetapi selalu berada diatas kepala orang lain.
commit to user
vii
PERSEMBAHAN
Dengan hati yang tulus dan penuh rasa syukur kupersembahkan skripsi ini untuk:
Allah SWT yang menjadikan aku lebih sabar, lebih semangat menjalani
hidup dan selalu yakin bahwa Engkau selalu memberiku yang terbaik.
Bapak Dwijo Martono dan ibu Partini yang selalu memberikan motivasi,
nasehat, kasih sayang serta do’a yang tiada hentinya.
Adikku Darmadi yang selalu memberikan motivasi
Mohammad Syaiful yang selalu memberikan motivasi dan kasih sayang
commit to user
viii
KATA PENGANTAR
Puji syukur kehadirat Allah SWT atas segala rahmat, taufiq, hidayah, serta
inayah-Nya sehingga penulis dapat menyelesaikan penulisan skripsi ini dengan
judul “Diversitas Genetik Intra Dan Inter Jenis Sapi Bali dari Sumbawa Dan Sapi Aceh Berdasarkan Analisis Mikrosatelit”, yang merupakan salah satu syarat untuk
memperoleh gelar kesarjanaan strata 1 (S1) pada jurusan Biologi, Fakultas
Matematika Dan Ilmu Pengetahuan Alam Universitas Sebelas Maret Surakarta.
Shalawat dan salam semoga tercurahkan kepada Nabi Muhammad SAW,
keluarga, para sahabat dan para pengikut yang diridhoi-Nya.
Penulis menyadari bahwa dalam penyelesaian penulisan skripsi ini adalah
atas sumbangsih materi maupun pemikiran dari berbagai pihak. Oleh karena itu,
dalam kesempatan ini penulis ingin menghaturkan terimakasih yang
sebesar-besarnya dan penghargaan yang setinggi-tingginya kepada semua pihak yang telah
membantu penulis, terutama kepada Yang Terhormat :
Ir. Ari Handono Ramelan, M.Sc. Ph.D., selaku dekan FMIPA Universitas
Sebelas Maret Surakarta, yang telah memberikan ijin penelitian untuk keperluan
skripsi.
Dra. Endang Anggarwulan, M.Si., selaku ketua jurusan Biologi FMIPA
Universitas Sebelas Maret Surakarta yang telah memberikan arahan serta ijin
penelitian skripsi .
Prof. Dr. Okid Parama Astirin, MS., selaku pembimbing akademik yang
telah memberikan bimbingan, arahan serta dukungan.
Prof. Drs. Sutarno, M.Sc. Ph.D., selaku dosen pembimbing I yang telah
memberikan bimbingan, arahan serta dukungan selama penelitian hingga
commit to user
ix
Dr. Artini Pangastuti, M.Si., selaku dosen pembimbing II yang telah
memberikan bimbingan, arahan serta dukungan selama penelitian hingga
selesainya penyusunan skripsi.
Prof. Drs. Suranto, M.Sc.Ph.D., selaku dosen penelaah I yang telah
memberikan bimbingan dan petunjuk selama penelitian hingga selesainya
penyusunan skripsi.
Dr. Sugiyarto, M.Si., selaku dosen penelaah II yang telah memberikan
bimbingan dan petunjuk selama penelitian hingga selesainya penyusunan skripsi.
Segenap dosen dan staf jurusan Biologi, FMIPA UNS atas segala bentuk
dukungan yang diberikan.
Keluarga besar Dwijo Martono, atas dukungan dan doanya. Keluarga besar
biologi 2006 FMIPA UNS untuk semangat, kebersamaan, dan persaudaraan yang
luar biasa.
Dengan kerendahan hati penulis menyadari bahwa dalam penelitian dan
penyusunan skripsi ini masih jauh dari sempurna, oleh karena itu saran dan kritik
yang membangun dari para pembaca akan sangat membantu. Semoga skripsi ini
dapat bermanfaat bagi kita semua.
Surakarta, Juli 2011
commit to user
A. Latar Belakang Masalah……….……...
commit to user
xi
BAB III. METODE PENELITIAN ………...
A. Waktu dan tempat penelitian ………...…………...
B. Bahan dan Alat ……….……...…….
1. Bahan……….………....
2. Alat ……….………..…………....
C. Cara Kerja…..………...…………...
1. Pengambilan Sampel Darah………...………..…... 2. Ekstraksi DNA………...…………..…. 3. Uji Kualitas Dan Kuantitas DNA………...………....
4. Amplifikasi DNA mikrosatelit dengan Polymerase Chain
Reaction(PCR)…..………..…….
5. Analisis produk PCR dan deteksi alel DNA………... D. Analisa Data………...………...……....
BAB IV. HASIL DAN PEMBAHASAN ……….
A.Diversitas Genetik Intra Spesies ... .………...……… B.Diversitas Genetik Inter Spesies ... .………...………
commit to user
xii
DAFTAR TABEL
Halaman Tabel 1.
Tabel 2.
Tabel 3.
Tabel 4.
Tabel 5.
.
Sekuen nukleotida primer mikrosatelit...
Keragaman alel mikrosatelit tiap individu pada setiap lokus mikrosatelit yang dianalisis...
Jumlah dan frekuensi alel intra spesies sapi Bali dari Sumbawa dan Aceh pada 5 lokus mikrosatetlit...
Parameter keragaman genetik intra spesies sapi Bali dari Sumbawa dan sapi Aceh pada 5 lokus mikrosatelit...
Parameter diversitas genetik inter spesies sapi Balidari Sumbawa dan sapi Aceh pada 5 lokus mikrosatelit...
17
30
31
33
commit to user
xiv
DAFTAR LAMPIRAN
Halaman
Lampiran 1.
Lampiran 2.
Hasil elektroforesis sampel sapi Bali dari Sumbawa pada gel
poliakrilamid 12% dengan pewarnaan ethidium
bromida………...
Pola elektroforesis sampel Sapi Aceh pada gel poliakrilamid
12% dengan pewarnaan ethidium bromida... 50
commit to user
xv
DAFTAR SINGKATAN
Singkatan Keterangan
AFLP Amplified fragment length polymorphism
APS Ammonium Peroxodisulfate Solution
BPTU Balai Pembibitan Ternak Unggul
dNTP deoksiribonukleotida trifosfat
ETBR ethidium bromide
He heterozigositas
Ho Homozigositas
I Shannon's Information index
Na observed number of alleles
Ne Effective number of alleles
PCR Polymerase Chain Reaction
PIC Polymorphic Information Content
PO Peranakan Ongole
RAPD Random Amplified Polymorphism DNA
RFLP Restriction Length polymorphisms
SSR simple sequence repeat
TAE Tris Acetic Acid
TEMED N,N,N',N' Tetramethylethylene - diamine
commit to user BAB I
PENDAHULUAN
A. Latar Belakang Masalah
Sapi-sapi yang terdapat di Indonesia mempunyai karakteristik yang
berbeda-beda. Perbedaan karakteristik tersebut salah satunya disebabkan karena
adanya diversitas genetik, yang dimungkinkan oleh adanya program-program
introduksi sapi impor yang dilakukan pemerintah (Johari et al., 2007). Di
Indonesia terdapat banyak sapi lokal seperti sapi Bali, sapi Aceh, sapi Madura,
sapi Ongole, sapi Peranakan Ongole (PO) dan sapi Pesisir. Di antara sapi-sapi
lokal di Indonesia, sapi Bali dari Sumbawa dan sapi Aceh merupakan bangsa sapi
yang mempunyai potensi dan nilai ekonomi untuk dikembangkan sebagaimana
sapi unggulan lain yang terdapat di Indonesia.
Sapi Bali dari Sumbawa adalah sapi hasil domestikasi dari banteng yang
mempunyai keunggulan dalam daya reproduksi, daya adaptasi dan persentase
karkas yang tinggi. Sapi Bali memiliki ciri khas yang tidak dimiliki oleh sapi dari
bangsa lainnya dan merupakan sumber daya genetik asli Indonesia (Riasari,
2010). Sapi Aceh mempunyai keunggulan antara lain daya reproduksi tinggi,
mampu berkembang pada kondisi lahan marginal, kualitas karkas tinggi, dan rasa
lebih enak (Basri, 2010). Berdasarkan keunggulan yang dimiliki oleh sapi Bali
dari Sumbawa dan sapi Aceh tersebut, maka apabila kedua jenis sapi disilangkan
akan menghasilkan breed sapi yang unggul baik dari segi kualitas maupun
commit to user
Diversitas genetik pada sapi dan juga pada hewan-hewan ternak lainnya
mengalami penurunan sangat cepat (Hall dan Bradley,1995; Hammond dan
Leitch, 1995). Pemilihan suatu jenis sapi tertentu karena
pertimbangan-pertimbangan keunggulan ekonomis dalam hal produksi telah menurunkan
diversitas genetik, dan bahkan menjadi salah satu mekanisme utama yang sangat
potensial menurunkan diversitas genetik.
Diversitas genetik merupakan dasar perkawinan silang bagi hewan ternak
(Buis et al., 1994) karena informasi ini dapat digunakan sebagai titik awal untuk
meningkatkan kuantitas dan kualitas jenis melalui seleksi buatan. Pengetahuan
mengenai pola-pola variabilitas genetik dari masing-masing jenis akan membantu
pengembangan program persilangan, dan merupakan pengetahuan awal yang
diperlukan dalam konservasi sumber genetik (Kidd et al., 1974).
Analisis diversitas genetik dalam organisme tingkat tinggi telah banyak
digunakan dalam memperkirakan keterkaitan genetik dan keanekaragaman dalam
dan di antara spesies. Penanda molekuler adalah molekul yang dapat digunakan
untuk melacak gen yang diinginkan dalam genotipe yang diperiksa. Identifikasi
populasi hewan dapat dilakukan berdasarkan sifat-sifat morfologi, DNA, dan
protein. Marka DNA merupakan kandidat terbaik untuk efisiensi evaluasi dan
identifikasi populasi hewan memisahkan marka DNA sebagai gen tunggal dan
mereka tidak terpengaruh oleh lingkungan sebagai penanda morfologis (Chambers
dan MacAvoy, 2000).
Aplikasi penanda genetik dapat digunakan untuk seleksi, pemuliaan dan
commit to user
berbagai teknik meliputi: teknik Restriction Length polymorphisms (RFLP),
Random Amplified Polymorphism DNA (RAPD), Amplified fragment length
polymorphism (AFLP) , dan mikrosatelit (Sumantri, 2007). Analisis mikrosatelit
merupakan salah satu penciri genetik yang sudah diaplikasikan secara meluas
dalam bidang peternakan, uji keturunan, mengidentifikasi sejumlah sifat dengan
nilai ekonomis tinggi pada banyak spesies. Hal ini dikarenakan DNA mikrosatelit
sangat polimorfik dan terdapat banyak dalam DNA genom (Bawden dan Nicholas,
1999). Informasi tentang diversitas genetik dan kekerabatan genetik pada hewan
ternak termasuk sapi sangat penting dalam usaha meningkatkan mutu genetik sapi
untuk memperoleh bibit unggul. Berdasarkan uraian tersebut, penelitian ini
dilakukan untuk mengidentifikasi diversitas genetik pada populasi sapi Bali
-Sumbawa dan sapi Aceh.
B. Perumusan Masalah
Permasalahan yang diangkat dalam penelitian ini adalah adakah diversitas
genetik intra dan inter spesies sapi Bali dari Sumbawa dan sapi Aceh berdasarkan
commit to user
C. Tujuan Penelitian
Tujuan yang akan dicapai dalam penelitian ini adalah mengidentifikasi
diversitas genetik intra dan inter spesies sapi Bali dari Sumbawa dan sapi Aceh
berdasarkan analisis mikrosatelit.
D. Manfaat Penelitian
Penelitian ini bermanfaat untuk mengetahui adanya diversitas genetik
antara sapi Bali dari Sumbawa dan sapi Aceh yang dapat digunakan sebagai dasar
dalam upaya lanjut untuk peningkatan kualitas maupun kuantitas produksi ternak
commit to user BAB II
LANDASAN TEORI
A. Tinjauan Pustaka
1. Sapi Bali dari Sumbawa
Sapi Bali merupakan ternak asli Indonesia yang mempunyai potensi
genetik dan nilai ekonomis yang cukup potensial untuk dikembangkan sebagai
ternak potong. Sapi Bali tersebar di berbagai wilayah Indonesia seperti Bali,
Lombok, Sumbawa, Flores, Sulawesi, Jawa Tengah, Sumatera dan Kalimantan
(Wiliamson dan payne, 1993). Namun, sapi Bali sebagian besar diternakkan
oleh petani-peternak dengan sistem pertanian yang masih konvensional
sehingga belum memberikan hasil yang optimal. Pulau Sumbawa merupakan
bumi sejuta sapi. Disana sapi-sapi lokal Indonesia khususnya sapi Bali banyak
di kembang biakkan dan di pelihara. Sapi Bali yang berkembang biak di pulau
Sumbawa mempunyai bentuk dan karakteristik sama dengan sapi Bali yang
ada di Bali (Entwistle et al., 2001). Berikut ini adalah gambar morfologi sapi
Bali dari Sumbawa.
commit to user
Sapi Bali adalah domestikasi dari banteng (Bibos banteng Syn. Bos
sondaicus) yang telah terjadi sejak zaman prasejarah. Namun, ada juga yang
menduga bahwa sapi Bali adalah asli berasal dari pulau Bali yang dalam
perkembangan selanjutnya dapat mempertahankan kemurniaannya (Gunawan
et al, 2004).
Sapi Bali memiliki ciri-ciri sebagai berikut warna putih dan pada
bagian belakang paha, pinggiran bibir atas kuku, bulu pada ujung ekor hitam,
bulu pada bagian dalam telinga putih, terdapat garis belut (garis hitam) yang
jelas pada bagian atas punggung, bentuk tanduk pada jantan yang paling ideal
disebut tanduk silak congklok yaitu jalannya pertumbuhan tanduk satu garis
dengan dahi mengarah kebelakang sedikit melengkung ke bawah dan pada
ujungnya mengarah kebawah dan kedalam, tanduk berwarna hitam. Ciri lain
dari sapi Bali yaitu bulunya ketika dilahirkan berwarna merah bata pada jantan
dan betina. Pada saat dewasa, pada sapi Bali jantan warna bulu berubah
menjadi hitam, sedangkan sapi Bali betina tetap berwarna merah bata
(Hardjosubroto, 1994). Bentuk tubuh sapi Bali menyerupai banteng, tetapi
ukuran tubuh lebih kecil akibat proses domestikasi, warna bulu untuk betina
merah bata sedangkan jantan dewasa kehitam-hitaman dan pada
tempat-tempat tertentu baik jantan maupun betina di bagian keempat kakinya dari
sendi kaki sampai kuku dan dibagian pantatnya berwarna putih (Sugeng,
1992). Sapi Bali dari Sumbawa rata-rata lingkar dadanya kecil dan agak pipih
commit to user
Sapi Bali merupakan ternak yang mempunyai dua peranan penting di
masyarakat yaitu sebagai sapi potong dan sapi kerja. Sapi Bali memiliki daya
toleransi / adaptasi yang tinggi terhadap lingkungan (Djagra et al., 2002).
Adaptabilitas sapi Bali terhadap lingkungan baik, baik secara langsung (suhu,
kelembaban, angin) dan yang tidak langsung (lahan, pakan, hama penyakit)
lebih baik dibanding breed sapi lain yang ada di indonesia (Darmadja, 1980).
Oleh karena itu, sapi bali harus dipertahankan keberadaannya dengan cara
meningkatkan populasi serta mutu genetiknya.
2. Sapi Aceh
Sapi Aceh merupakan sapi lokal yang terdapat di Aceh Sumatera dan
diminati sebagai ternak potong. Sapi tersebut masih terdapat beberapa variasi
warna tubuh (Namikawa et al., 1982). Berikut ini adalah gambar morfologi
sapi Aceh.
Gambar 2. Sapi Aceh (Abdullah, 2008).
Sapi Aceh merupakan turunan dari sapi ongole dengan sapi setempat.
Pada umumnya, sapi Aceh mempunyai pola dasar warna rambut coklat merah,
coklat hitam, hitam, putih, dan warna menjangan. Umumnya sapi Aceh
commit to user
dan bertanduk. Bobot badan sapi jantan berumur 3 - 4 tahun berkisar antara
300 - 400 kg, sedangkan sapi betina pada umur yang sama beratnya berkisar
antara 200 - 300 kg (Gunawan, 1998). Sapi Aceh secara fenotipik terdapat
perbedaan dengan sapi PO meski sama-sama keturunan dari sapi Ongole. Sapi
PO mempunyai warna rambut kelabu kehitaman, sedangkan sapi Aceh
memiliki pola dasar warna rambut bervariasi. Bobot badan sapi-sapi Aceh
pada semua tingkat umur lebih rendah daripada bobot badan sapi PO pada
tingkat umur yang sama. Demikian juga dengan semua ukuran tubuh sapi
Aceh lebih rendah dari ukuran-ukuran tubuh sapi PO (Abdullah, 2008).
Sapi Aceh mempunyai daya tahan terhadap lingkungan yang buruk
seperti krisis pakan, air dan pakan berserat tinggi, penyakit parasit, temperatur
panas dan sistem pemeliharaan ekstensif tradisional (Gunawan, 1998). Sapi
Aceh tersebar luas di daerah Aceh dan daerah Sumatera utara, terutama pada
daerah-daerah yang berbatasan dengan Aceh Timur (Siregar, 2003).
3. Diversitas Genetik
Diversitas genetik terjadi tidak hanya antar bangsa tetapi juga di dalam
satu bangsa yang sama, antar populasi maupun di dalam populasi, atau di
antara individu dalam populasi. Pada spesies suatu identifikasi tingkat
keragaman, terutama pada lokus-lokus yang mempunyai sifat bernilai penting
mempunyai keterkaitan dengan seleksi dalam program pemuliaan
(Handiwirawan dan Subandriyo, 2004).
Diversitas genetik merupakan suatu keanekaragaman genetik yang
commit to user
adanya keanekaragaman morfologis, sehingga dapat dikatakan bahwa tidak
ada individu-individu yang sama didunia ini. Keanekaragaman ini dikarenakan
oleh adanya variasi gen-gen yang mengkodekan morfologi makhluk hidup
tersebut, serta adanya pengaruh lingkungan. Keanekaragaman genetik dapat
terjadi karena adanya perubahan nukleotida penyusun DNA. Perubahan ini
mungkin dapat mempengaruhi fenotipe suatu organisme yang dapat dilihat
dengan mata telanjang, atau mempengaruhi reaksi individu terhadap
lingkungan tertentu. Secara umum keanekaragaman genetik dari suatu
populasi dapat terjadi karena adanya mutasi, rekombinasi, atau migrasi gen
dari satu tempat ke tempat lain (Suryanto, 2003).
Diversitas genetik dalam populasi merupakan modal dasar aplikasi
teknologi pemuliaan dalam pemanfaatan hewan. Diversitas genetik populasi
yang digambarkan dalam keragaman penampilan hewan adalah refleksi
informasi genetik yang dimilikinya. Sebagai ilustrasi sapi Bali yang hidup di
pulau Bali memiliki kontruksi gen-gen yang berbeda dengan populasi sapi
pesisir yang ada di Sumatera Barat. Perbedaan-perbedaan tersebut dapat
dinyatakan dalam kemampuan adaptasi, besarnya tubuh, dan ketahanan
penyakit. Komponen ini sangat berpengaruh terhadap kemampuan
menyesuaikan diri (adaptasi) terhadap perubahan lingkungan seperti degradasi
kualitas lingkungan sebagai media tumbuh hewan. Perbedaan penampilan ini
disebabkan selama domestifikasi, tipe-tipe atau bangsa-bangsa hewan terpisah
secara genetik karena adanya penyesuaian (adaptasi) dengan masing-masing
commit to user
berbeda (Muladno 2006). Noor (2008) mengatakan adanya kemampuan
adaptasi hewan disebabkan hewan memiliki kemampuan menghasilkan lebih
dari satu alternatif bentuk morfologi, status fisiologi, dan tingkah laku sebagai
reaksi terhadap perubahan lingkungan (pengaturan ekspresi gen).
Perkembangan sejumlah penanda molekuler dewasa ini telah
memungkinkan untuk melakukan identifikasi terhadap perubahan-perubahan
genetik yang terjadi dalam suatu persilangan serta hubungannya dengan
perubahan sifat kuantitatif dan sifat kualitatif ternak. Selain itu, penanda
molekuler juga dapat digunakan untuk membedakan antara suatu ras ternak
dengan lainya terutama dalam kaitannya dengan upaya pelestarian dan
menjaga kemurnian dari ras tersebut (Maskur et al., 2007).
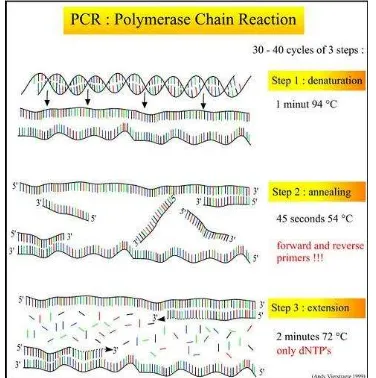
4. Polymerase Chain Reaction (PCR)
Polymerase Chain Reaction (PCR) adalah suatu metode enzimatis
untuk melipatgandakan secara eksponensial suatu sekuen nukleotida tertentu
dengan cara in vitro. Metode ini pertama kali dikembangkan pada tahun 1985
oleh Kary B. Mullis. Metode PCR tersebut sangat sensitif. Sensitivitas tersebut
menjadikan PCR dapat digunakan untuk melipatgandakan satu molekul DNA.
Metode ini juga sering digunakan untuk memisahkan gen-gen berkopi tunggal
dari sekelompok sekuan genom (Mullis dan Faloona, 1989). Kelebihan lain
metode PCR adalah bahwa reaksi ini dapat dilakukan dengan menggunakan
komponen dalam jumlah sangat sedikit (Yuwono T, 2006).
Empat komponen utama pada proses PCR adalah (1) DNA cetakan,
commit to user
yaitu sepasang DNA utas tunggal atau sekuen oligonukleotida pendek (15 – 25
basa nukleotida) yang digunakan untuk mengawali sintesis rantai DNA, (3)
deoksiribonukleotida trifosfat (dNTP), terdiri atas dATP, dCTP, dGTP, dTTP,
dan (4) enzim DNA polimerase, yaitu enzim yang melakukan katalisis reaksi
sintesis rantai DNA. Komponen lain yang juga penting adalah buffer.
Primer dirancang untuk memiliki sekuen yang komplemen dengan
DNA template, jadi dirancang agar menempel mengapit daerah tertentu yang
diinginkan (Yepyhardi, 2009). Primer – primer yang digunakan (primer sense
dan primer antisense) sebaiknya mempunyai nilai Tm (melting temperatur)
yang serupa. Tm adalah suhu pada saat setengah dari molekul DNA
mengalami denaturasi. Nilai Tm oligonukleotida dapat dihitung dengan
menggunakan rumus Tm = 20C x (A+T) + 40C x (G+C) (Yuwono, 2006).
Reaksi pelipatgandaan suatu fragmen DNA dimulai dengan melakukan
denaturasi DNA template sehingga rantai DNA yang berantai ganda (double
stranded) akan terpisah menjadi rantai tunggal (single stranded). Denaturasi
DNA dilakukan dengan menggunakan panas (950C) selama 1-2 menit,
kemudian suhu diturunkan menjadi 550C sehingga primer akan menempel
(annealing) pada cetakan yang telah terpisah menjadi rantai tunggal. Primer
akan membentuk jembatan hidrogen dengan cetakan pada daerah sekuen yang
komplementer dengan sekuen primer. Amplifikasi akan lebih efisien jika
dilakukan pada suhu yang lebih rendah (370C), tetapi biasanya akan terjadi
mispriming yaitu penempelan primer pada tempat yang salah. Pada suhu yang
commit to user
secara keseluruhan efisiensinya akan menurun.
Primer yang digunakan dalam PCR ada dua yaitu oligonukleotida yang
mempunyai sekuen yang identik dengan salah satu rantai DNA cetakan pada
ujung 5’-fosfat, dan oligonukleotida yang kedua identik dengan sekuen pada
ujung 3’-OH rantai DNA cetakan yang lain. Proses annealing biasanya
dilakukan selama 1–2 menit. Setelah dilakukan annealing oligonukleotida
primer dengan DNA cetakan, suhu inkubasi dinaikkan menjadi 720C selama
1,5 menit. Pada suhu ini DNA polimerase akan melakukan proses polimerasi
rantai DNA yang baru berdasarkan informasi yang ada pada DNA cetakan.
Setelah terjadi polimerasi, rantai DNA yang baru akan membentuk jembatan
hidrogen dengan DNA cetakan. DNA rantai ganda yang terbentuk dengan
adanya ikatan hidrogen antara rantai DNA cetakan dengan rantai DNA baru
hasil polimerasi selanjutnya akan di denaturasi lagi dengan menaikkan suhu
inkubasi menjadi 950C. Rantai DNA yang baru tersebut selanjutnya akan
berfungsi sebagai cetakan bagi reaksi polimerasi berikutnya.
commit to user
Reaksi – reaksi seperti yang sudah dijelaskan tersebut diulangi lagi
sampai 25 – 30 siklus sehingga pada akhir siklus akan didapatkan molekul –
molekul DNA rantai ganda yang baru hasil polimerasi dalam jumlah yang jauh
lebih banyak dibandingkan dengan jumlah DNA cetakan yang digunakan.
Banyaknya siklus amplifikasi tergantung pada konsentrasi DNA target di
dalam campuran reaksi. Paling sedikit, diperlukan 25 siklus untuk
melipatgandakan satu kopi sekuen DNA target di dalam DNA genom mamalia
agar hasilnya dapat di lihat secara langsung, misalnya dengan elektroforesis
gel agarose (Sambrook et al., 1989).
5. DNA Mikrosatelit
Salah satu penanda molekuler yang sangat populer dewasa ini adalah
mikrosatelit. Mikrosatelit adalah runutan DNA pendek berulang 2 sampai 6
basa nukleotida dan dapat berulang 10-100 kali, runutan yang paling banyak
ditemukan pada mamalia adalah (dC-dA)n dan (dT-dG) n (Moore et al.,
1991). Mikrosatelit merupakan salah satu penanda genetik yang sudah
diaplikasikan secara meluas dalam bidang peternakan. Selain untuk uji test
keturunan, dapat pula digunakan untuk mengidentifikasi sejumlah sifat dengan
nilai ekonomi tinggi dikarenakan DNA mikrosatelit sangat polimorfik dan
terdapat banyak dalam DNA genom (Bawden dan Nicholas, 1999).
Mikrosatelit (SSR = simple sequence repeat) merupakan salah satu
marka molekuler yang berupa urutan di-nukleotida sampai tetra-nukleotida
yang berulang dan berurutan. Mikrosatelit merupakan marka genetik yang
commit to user
tingkat tinggi, serta mudah dan tidak terlalu mahal untuk dianalisis dengan
menggunakan PCR (Muljopawiro, 2007).
Aplikasi penanda genetik molekuler untuk seleksi dan pemuliaan dapat
meningkatkan mutu genetik ternak (Bawden dan Nicholas, 1999). Mikrosatelit
merupakan penanda genetik yang sering digunakan untuk mempelajari sistem
perkawinan dan struktur populasi (Steffen et al., 1993), pautan (linkage),
pemetaan kromoson, dan analisa populasi (Silva et al., 1999). Mikrosatelit
banyak digunakan oleh peneliti sebagai marka karena keberadaanya
melimpah, bersifat kodominan dan sangat polimorfik (Bennett, 2000). Berikut
hasil penelitian terdahulu mengenai amplifikasi DNA mikrosatelit sapi dengan
primer ILSTS068, yang identik dengan kerbau :
Gambar 4. Urutan nukleotida sapi marker mikrosatelit (ILSTS068) dan identik dengan kerbau. garis tebal menunjukkan urutan primer (Navani et al, 2002). Marka mikrosatelit merupakan sekuen DNA yang bermotif pendek dan
commit to user
meliputi seluruh genom, terutama pada organisme eukariotik. Pasangan primer
mikrosatelit (forward dan reverse) diamplifikasi dengan PCR berdasarkan
hasil konservasi daerah yang diapit (flanking-region) marka untuk suatu gen
pada kromosom. Menurut Powell et al. (1996), beberapa pertimbangan untuk
penggunaan marka mikrosatelit dalam studi genetik di antaranya (1) marka
terdistribusi secara melimpah dan merata dalam genom, variabilitasnya sangat
tinggi (banyak alel dalam lokus), sifatnya kodominan dan lokasi genom dapat
diketahui; (2) merupakan alat uji yang memiliki reproduksibilitas dan
ketepatan yang sangat tinggi; (3) merupakan alat bantu yang sangat akurat
untuk membedakan genotipe, evaluasi kemurnian benih, pemetaan, dan seleksi
genotip untuk karakter yang diinginkan; (4) studi genetik populasi dan analisis
diversitas genetik. Kelemahan teknik ini adalah marka SSR tidak tersedia pada
semua spesies tanaman, sehingga untuk merancang primer baru membutuhkan
waktu yang lama dan biaya yang cukup mahal.
Beberapa laporan menunjukkan adanya asosiasi yang signifikan antara
lokus mikrosatelit dengan sifat kuantitatif seperti sifat produksi susu
(Kantanen et al., 2000), lemak karkas (Fitzsimmons et al., 1998), perbedaan
tingkat fertilitas (Oliveira et al., 2002) dan efisiensi reproduksi (João et al.,
commit to user
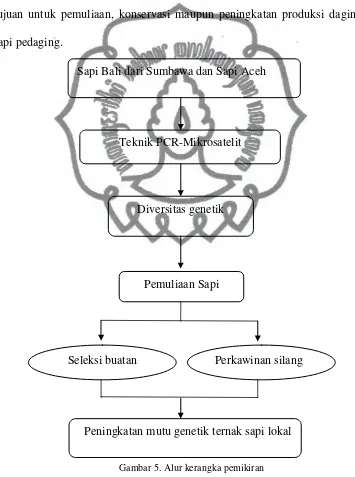
B. Kerangka Pemikiran
Penelitian tentang variasi genetik terhadap sapi pedaging di Indonesia
terutama sapi Bali dari Sumbawa dan sapi Aceh belum banyak dilakukan.
Berawal dari penelitian mengenai diversitas genetik pada sapi Bali dari Sumbawa
dan sapi Aceh, maka akan dapat dilakukan suatu penelitian lebih lanjut dengan
tujuan untuk pemuliaan, konservasi maupun peningkatan produksi daging pada
sapi pedaging.
Gambar 5. Alur kerangka pemikiran Sapi Bali dari Sumbawa dan Sapi Aceh
Teknik PCR-Mikrosatelit
Diversitas genetik
Pemuliaan Sapi
Seleksi buatan Perkawinan silang
commit to user BAB III
METODE PENELITIAN
A. Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan di Laboratorium Pusat Universitas Sebelas Maret
Surakarta dan Laboratorium Biologi MIPA Universitas Sebelas Maret Surakarta.
Waktu penelitian dilaksanakan pada bulan Juli 2010 sampai Januari 2011.
B. Bahan dan Alat
1. Bahan
Bahan yang digunakan dalam penelitian ini adalah sampel darah sapi
Bali dari Sumbawa dan sapi Aceh. Pengambilan sampel darah sapi Bali dari
Sumbawa di Kecamatan Marongge, Kabupaten Sumbawa, sedangkan
pengambilan sampel darah sapi Aceh di BPTU (Balai Pembibitan Ternak
Unggul) Sapi Aceh Indrapuri – Nanggroe Aceh Darussalam. Masing-masing
sampel darah sebanyak 20 individu yang disimpan dalam venoject berisi
EDTA.
Bahan kimia yang digunakan untuk penelitian dibagi dalam beberapa
bagian sesuai tahapan penelitian, yaitu:
Ekstraksi DNA yaitu: Wizard Genomic DNA Purification Kit dari Promega
(cell lysis solution, nuclei lysis solution, protein presipitation solution,
DNA rehydration), isopropanol, etanol 70%, sampel darah Sapi Bali dari
commit to user
Uji Kuantitas DNA yaitu: DNA hasil lisis dan aquades sebagai blangko.
Uji kualitas DNA yaitu: DNA hasil lisis, ultra pure DNA Grade agarose
1% dari Bio Rad, 1X TAE buffer (Tris Acetic Acid), blue loading dye dan
ethidium bromida.
PCR MIX yaitu: Go Tag Green master mix (Promega, USA), Nuclease
Free Water dari Ambion, 5 primer mikrosatelit (BM1824, ETH225,
INRA005, MM12, dan TGLA227) dari SIGMA, DNA hasil lisis
diencerkan 10X.
Analisis Mikrosatelit yaitu: 10X TBE buffer (Tris Borate), 10% APS
(Ammonium Peroxodisulfate Solution), N,N,N',N' Tetramethylethylene -
diamine (TEMED), aquades steril, Blue loading dye, acrylamid:bis (19:1)
30%, Urea, 100 - 1500 base pare DNA Moleculare Weight Marker, buffer
TBE 1X dan ethidium bromida (EtBr).
2. Alat
Alat yang digunakan dalam penelitian ini adalah sentrifuge Hettich
Mikro 22R (Jerman), satu set micropipet (ukuran 10 , 200 , 1.000 ),
satu set tips (10 , 200 , 1.000 , tabung mikro 1,5 ml (Axygen), tabung
mikro 0,6 ml, satu set alat elektroforesis horisontal dan power supply (Bio
Rad), satu set alat elektroforesis vertikal dan power supply (Bio Rad),
microwave, inkubator, waterbath (Julabo), Gene Amplification PCR system
9700 Thermo Cycler (Applied Biosystem), Autoclave (Ogawa Saiki Co.),
Erlenmeyer, gelas ukur, tabung venoject, vortex mixer (Gemmy Industrial
commit to user
elektrik (Denver Instrumen), UV transluminator , kamera digital dan geldoc
(Vilber Lourmat).
C. Cara Kerja
1. Pengambilan Sampel Darah
Sampel darah diambil sebanyak 3 ml dari masing – masing individu
(20 individu sapi Bali dari Sumbawa dan 20 individu sapi Aceh) secara
venopuncture menggunakan venoject dengan ukuran 10 ml yang berisi EDTA
untuk mencegah terjadinya pembekuan darah . Darah disimpan pada suhu
-200C untuk digunakan langsung dalam penelitian ini dan sebagai referensi di
kemudian hari.
2. Ekstraksi DNA
DNA diekstrak dari total darah dengan menggunakan teknik Wizard
Genomic Purification System tahun 2010 (Promega, Madison USA). DNA
diekstrak langsung dari total darah. Sebanyak 300 mikroliter total darah
dimasukkan kedalam 1,5 tabung mikrosentrifuse yang steril dan berisi 450
larutan pelisis sel (cell lysis solution), dicampur dan diinkubasi pada suhu
kamar selama 10 menit untuk melisis sel darah merah yang mungkin masih
tercampur. Sel darah putih kemudian di sentrifugasi pada kecepatan 14.000g
(gravitasi) selama 20 detik untuk memperoleh endapan sel darah putih.
Supernatan (bening bagian atas) yang terbentuk diambil dan dibuang,
kemudian tabung mikrosentrifuse yang berisi endapan sel darah putih divortek
selama 3–5 menit agar sel–sel darah putih memisah secara sempurna.
commit to user
mikrosentrifuse yang berisi suspensi tersebut, kemudian dicampur dengan
menggunakan pipet sebanyak 5 – 6 kali untuk melisis sel–sel darah putih.
Kemudian di inkubasi selama 15 menit pada suhu 370C. Setelah itu sampel
didinginkan pada suhu kamar selama 5 menit, yang dilanjutkan dengan
penambahan 60 protein precipitation solution untuk membentuk precipitat
protein kedalam lisat, kemudian dihomogenkan dengan vortek selama 10 – 20
detik dan disentrifugasi pada 14.000g selama 3 menit untuk membentuk
endapan protein.
Supernatan diambil dan dipindahkan kedalam tabung mikrosentrifuse
steril yang baru yang sebelumnya telah diisi dengan 150 isopropanol.
Campuran yang diperoleh dicampur dengan sempurna dengan membolak –
balikkan tabung mikrosentrifuse sampai terbentuknya materi seperti benang
berwarna putih. DNA kemudian disentrifugasi pada kecepatan 14.000g selama
1 menit pada suhu kamar. Supernatan dibuang kemudian ditambah 300
ethanol 70% dan tabung yang berisi larutan DNA dan ethanol ini dibolak–
balik untuk mencuci endapan DNA dan juga sisi–sisi tabung mikrosentrifuse.
DNA kemudian diendapkan dengan sentrifugasi pada kecepatan 14.000g
selama 1 menit. Ethanol diambil dengan hati-hati, kemudian tabung
mikrosentrifuse dibalik dan dibiarkan dibuka untuk mengeringkan DNA.
Setelah kering, 100 larutan DNA rehydration solution ditambahkan
kedalam tabung dan DNA direhidrasi dengan cara inkubasi pada waterbath
commit to user
suhu 2- 80C sampai penggunaan berikutnya (Certificate of Analysis; WizardTM
Genomic DNA Purification System, Promega Corporation, 2010).
3. Uji Kualitas dan Kuantitas DNA
DNA yang diperoleh dari hasil ekstraksi di uji kualitas dan
kuantitasnya. Uji kualitas DNA dilakukan dengan elektroforesis horisontal
menggunakan gel agarose 1% dalam buffer TAE (Tris-EDTA). Uji dilakukan
dengan mencampur 5 DNA yang akan dicek dan 2 loading dye. 1% gel
agarose dibuat dengan melarutkan 0,48 mg serbuk agarose dalam 48 ml 1 x
buffer TAE (Tris-EDTA) . Selanjutnya campuran buffer tersebut dipanaskan
dalam microwave pada suhu sedang (medium) selama 2 menit. Setelah itu
Erlenmeyer dikeluarkan dari microwave dan ditunggu sampai suhu turun
menjadi suhu kamar (hangat), selanjutnya larutan gel agarose dituangkan ke
dalam cetakan yang sebelumnya telah dipasangi sisir dan ditunggu hingga gel
mengeras. Setelah gel mengeras sisir diangkat dengan hati-hati untuk
menghindari rusaknya gel agarose, maka akan terbentuk sumuran – sumuran
kecil. Selanjutnya gel dimasukkan ke dalam tangki yang berisi 1X buffer TAE
(Tris-EDTA) sampai terendam, kemudian sampel (DNA dan Loading dye) di
masukkan ke dalam sumuran-sumuran kecil pada gel agarose. Proses
elektroforesis berlangsung selama 30 menit pada 85 volt, setelah proses selesai
gel agarose direndam dalam ethidium bromida. Ethidium bromida dibuat
dengan mencampurkan 2 tetes EtBr yang dilarutkan dengan 100 ml aquades.
commit to user
kemudian difoto dengan menggunakan kamera digital. Apabila terdeteksi
adanya pita berarti ekstraksi DNA dari sampel berhasil.
Uji kuantitas DNA dilakukan dengan metode spektrofotometri pada
panjang gelombang 260 nm. Menurut formula yang digunakan dalam
perhitungan menggunakan alat RNA/DNA kalkulator menyatakan bahwa
absorbansi A260 1,0 sesuai untuk 50 µg/mL DNA murni untai ganda, maka
kadar DNA sampel dapat dicari melalui perhitungan
berikut :
X(µg/mL) =
Keterangan :
X : kadar DNA yang dicari
A260 : absorbansi sampel DNA pada panjang gelombang 260 nm
DNA dikatakan murni jika rasio kedua nilai tersebut berkisar antara 1,8 – 2,0.
Jika nilai rasio lebih kecil dari 1,8 maka masih ada kontaminasi protein
didalam larutan (Ratnayani K et al, 2009).
4. Amplifikasi DNA mikrosatelitdengan Polymerase Chain Reaction (PCR)
DNA yang diperoleh langsung digunakan untuk reaksi PCR yang
dilakukan dalam mesin PCR (Applied Biosystem). Fragmen DNA
diamplifikasi dengan PCR menggunakan primer berturut-turut ETH225,
TGLA227, INRA005, BM1824 dan MM12.
Berikut sekuen nukleotida dari masing-masing primer yang akan
commit to user
Tabel 1. Sekuen nukleotida primer mikrosatelit
Marker Sekuen primer Letak
kromosom
oligonukleotida primer (primer forward dan reserve), 6 l Go Taq Green dan
5 l nuclease free water dalam 0,6 ml tabung effendorf. Kondisi reaksi
amplifikasi PCR amplifikasi DNA sebagai berikut: satu tahap reaksi
denaturasi awal pada suhu 950C selama 5 menit, diikuti dengan 30 siklus
amplifikasi yang masing-masing siklus terdiri dari: denaturasi pada suhu 950C
selama 60 detik, annealing dengan suhu berdasarkan Tm primer selama 60
detik, dan exstention pada suhu 720C selama 1 menit; diikuti dengan satu
commit to user
5. Analisis produk PCR dan deteksi alel DNA
Hasil amplifikasi dengan PCR difraksinasi dengan elektroforesis gel
poliakrilamid 12% yang dilanjutkan dengan pewarnaan dengan ethidium
bromida. Berdasarkan Qiagen Supplemantary Ptotocol Gel Poliakrilamid 12%
dibuat dengan cara mencampurkan bahan-bahan berikut : 7,2 gram urea,
buffer TBE 10X 1,5 ml dan 5,4 ml 30% akrilamid : bis akrilamid (19 : 1) pada
erlenmeyer, selanjutnya ditambahkan dengan aquabides sampai 15 ml.
Selanjutnya larutan dipanaskan dalam microwave selama 9 detik dengan
power level tinggi sampai seluruh larutan terlarut dengan sempurna. Setelah
larutan tercampur sempurna kemudian ditambahkan 75 µ l 10% ammonium
persulfat (APS) kemudian ditambahkan dengan segera 7,5 µ l N,N,N’,N’
Tetramethylethylenediamine (TEMED) dan tetap diaduk perlahan. Setelah
penambahan APS dan TEMED, larutan poliakrilamid 12% segera dituangkan
kedalam cetakan agar larutan tidak mengalami polimerisasi. Untuk resep
dengan volume 15 ml digunakan cetakan dengan ukuran 7x10 cm.
Tahap selanjutnya adalah persiapan sampel. Denaturasi sampel
dilakukan pada suhu 950C selama 2 menit dan dilanjutkan dengan
memasukkan dalam pecahan es. Selanjutnya gel dipasang pada alat dan
dilanjutkan dengan memasukkan sampel hasil PCR dan marker ke dalam
sumuran. Harus dipastikan bahwa dalam tahapan ini tidak lebih dari 20 menit
supaya sampel tetap berada pada keadaan dingin. Selanjutnya power supply
dinyalakan dan diatur pada 120 volt dan 400 miliamper. Running dilakukan
commit to user
mencapai daerah munculnya pita maka running dapat dihentikan dan tangki
elektroforesis dapat dikosongkan dari buffer. Selanjutnya pindahkan gel
perlahan-lahan dari plate dan siap dilakukan pewarnaan dengan ethidium
bromide. Gel poliakrilamid direndam dalam ethidium bromida selama 15
menit. Ethidium bromida dibuat dengan mencampurkan 1 µl EtBr yang
dilarutkan dengan 100 ml aquades.
Elektroforesis gel poliakrilamid mampu memisahkan DNA lebih
sempurna dengan jumlah sampel yang dibutuhkan lebih sedikit (Allen et al.,
1984). Hasil pemisahan fragmen DNA dideteksi dengan geldoc. Pita hasil
amplifikasi kemudian dicatat dan diberi nilai. Setiap pita DNA yang muncul
dibandingkan ke marker untuk mengetahui panjangnya. Satu posisi migrasi
yang sama dianggap sebagai satu tipe atau alel.
D. Analisis Data
Analisis data molekuler dilakukan berdasarkan hasil skoring pita DNA
yang muncul pada plate. Skoring dilakukan dengan cara: pita yang paling bawah
diberi sandi A dan selanjutnya B, C, dan seterusnya sampai pita paling atas.
Keragaman genotipe tiap-tiap individu dapat ditentukan dari pita-pita DNA yang
ditemukan. Masing-masing sampel dibandingkan berdasarkan ukuran (marker)
yang sama dan dihitung frekuansi alelnya. Frekuensi alel dihitung berdasarkan
rumus Nei (1987) :
Dimana : Xi = frekuensi alel ke-i
nij = jumlah individu yang bergenotipe ii
commit to user
n
PIC = 1 -
∑ Pij
2j=1
nii = jumlah individu yang bergenotipe ij
n = jumlah sampel
Derajat heterozigositas (ĥ) dihitung berdasarkan frekuensi alel pada tiap lokus
DNA dengan rumus Nei (1987) :
Keterangan :
Xi = frekuensi alel
ĥ = nilai heterozigositas lokus
Tingkat polimorfisme (PIC = Polimorphism Information Content) dari
primer yang digunakan dihitung untuk masing-masing marka SSR (Botstein et al,
1980) dengan formula:
dimana ij merupakan frekuensi alel i dan j.
Untuk jumlah alel, frekuensi alel, Effective number of alleles, derajat
homozigositas, dan derajat heterozigositas antara sapi Bali - Sumbawa dan sapi
Aceh dapat dianalisis dengan menggunakan program POPGENE version 1.31 (Yeh
dan Yang, 1999).
commit to user
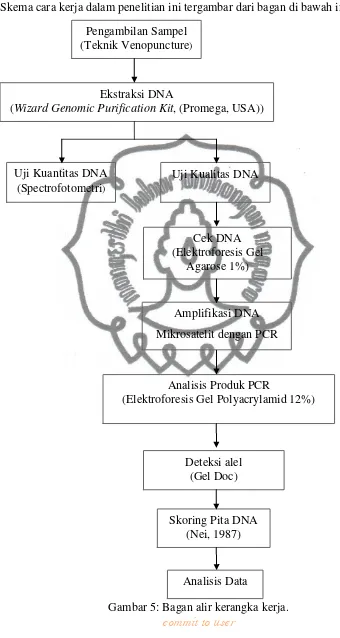
Skema cara kerja dalam penelitian ini tergambar dari bagan di bawah ini :
Gambar 5: Bagan alir kerangka kerja. Skoring Pita DNA
(Nei, 1987) Deteksi alel (Gel Doc) Uji Kualitas DNA Uji Kuantitas DNA
(Spectrofotometri)
Ekstraksi DNA
(Wizard Genomic Purification Kit, (Promega, USA))
Cek DNA (Elektroforesis Gel
Agarose 1%) )
Amplifikasi DNA
Mikrosatelit dengan PCR
Analisis Produk PCR
(Elektroforesis Gel Polyacrylamid 12%)
Analisis Data Pengambilan Sampel
commit to user BAB IV
HASIL DAN PEMBAHASAN
A. Diversitas Genetik Intra Spesies
Polimorfisme dari kedua genom spesies sapi Bali dari Sumbawa dan sapi
Aceh ditetapkan dari hasil amplifikasi DNA dengan primer pengapit mikrosatelit
yang diseparasi dengan elektroforesis gel poliakrilamid dan dilanjutkan dengan
teknik pewarnaan ethidium bromide. Elektroforesis gel poliakrilamid mampu
memisahkan DNA lebih sempurna dengan jumlah sampel yang dibutuhkan lebih
sedikit (Allen et al., 1984). Adapun untuk menentukan jumlah alel dilakukan
sesuai petunjuk Leung et al (1993) dengan asumsi bahwa semua pita DNA dengan
laju migrasi yang sama, diasumsikan sebagai lokus yang homolog. Pada studi ini,
dilakukan terhadap dua populasi sapi Bali dari Sumbawa dan sapi Aceh
berdasarkan 5 lokus mikrosatelit (TGLA227, ETH225, BM1824, INRA005 dan
MM12). Semua lokus mikrosatelit yang dipergunakan berhasil teramplifikasi.
Dalam penentuan jenis alel, meskipun setiap lokus menunjukkan jenis alel yang
sama (misal alel A pada mikrosatelit ETH225 dengan INRA 005), alel tersebut
sebenarnya berbeda karena masing-masing lokus mikrosatelit mencirikan genotip
berbeda. Jadi, dalam hal ini alel A pada mikrosatelit ETH225 berbeda dengan alel
A pada mikrosatelit INRA005 demikian seterusnya. Akan tetapi, untuk antar
spesies maka ketentuan alel berlaku sama, artinya alel A dengan ukuran tertentu
dari mikrosatelit ETH225 yang terdapat pada spesies sapi Bali dari Sumbawa
commit to user
ditunjukkan contoh hasil amplifikasi mikrosatelit dengan primer pengapitnya pada
salah satu spesies sapi (sapi Bali dari Sumbawa).
Gambar 5. Hasil PCR dengan primer mikrosatelit ETH225 pada genom spesies sapi Bali dari Sumbawa individu no 1-9 secara berurutan bergenotipe AC, AC, AC, AC, AC, AC, AA, AB, dan AB
Keragaman genotipe dari setiap lokus pada dasarnya mencerminkan
polimorfisme alel dari setiap lokus yang bersesuaian. Pengukuran keragaman
genetik 40 sampel spesies sapi Bali dari Sumbawa dan sapi Aceh dengan
menggunakan 5 lokus mikrosatelit, menunjukkan semua lokus pada spesies
sapi Bali dari Sumbawa dan sapi Aceh adalah polimorfik, sedangkan lokus
BM1824 dan INRA005 pada sapi Bali dari Sumbawa dan lokus TGLA227 pada
sapi Aceh adalah monomorfik (satu alel). Setiap lokus mempunyai genotip dari
satu sampai tiga macam. Lokus yang mempunyai tiga macam genotip adalah
lokus ETH225 dan MM12 pada sapi Bali dari Sumbawa, lokus BM1824,
INRA005 dan MM12 pada sapi Aceh. Sedangkan lokus yang hanya mempunyai
satu macam genotip adalah lokus BM1824 dan INRA005 pada sapi Bali dari
commit to user
macam genotip adalah lokus TGLA227 pada sapi Bali dari Sumbawa, lokus
ETH225 pada sapi Aceh. Dilihat dari macam genotip yang dihasilkan, setiap lokus
mempunyai jumlah alel bervariasi antara satu sampai tiga alel. Jumlah alel
sebagian besar lokus adalah tiga alel. Jumlah alel terendah terdapat pada lokus
BM1824 dan INRA005 pada sapi Bali dari Sumbawa serta lokus TGLA227 pada
sapi Aceh.
Table 2. keragaman alel mikrosatelit tiap individu pada setiap lokus mikrosatelit yang dianalisis
Hasil amplifikasi PCR terhadap DNA mikrosatelit menunjukkan bahwa
jumlah alel bervariasi antara 1 sampai 3. Jumlah alel dan frekuensi alel yang
terdeteksi untuk setiap lokus pada sapi Bali dari Sumbawa dan sapi Aceh
commit to user
Table 3. Jumlah dan frekuensi alel intra spesies Bali dari Sumbawa dan Aceh pada 5 lokus mikrosatelit
lokus Sapi Bali dari Sumbawa Sapi Aceh
na f na f
Keterangan : Na.: Observed number of alleles ; f = Allele Frequency
Frekuensi alel adalah proporsi alel yang ada dalam suatu populasi (Hartl,
1988). Masing-masing lokus memiliki alel dengan jumlah berbeda-beda. Pada
sapi Bali dari Sumbawa untuk lokus TGLA227 terdapat 2 alel dengan frekuensi
alel A (0,53) dan alel B (0,47). Pada lokus ETH225 terdapat 3 alel dengan
frekuensi dari yang tertinggi alel A (0,75), C (0,15) dan yang terendah B (0,10).
lokus BM1824 dan INRA005 hanya mempunyai 1 alel dengan frekuensi alel A
(1,00) dalam keadaan homozigot. Lokus MM12 memiliki 3 alel dengan
masing-masing frekuensi alel A (0,53), B (0,42), dan C (0,05). Secara keseluruhan jumlah
alel yang terdeteksi pada sapi Bali dari Sumbawa berkisar antara 1 - 3 dengan
rata- rata 2. Frekuensi alel tertentu yang lebih besar atau sama dengan 0,95,
maka alel tersebut digolongkan monomorfik (Hartl, 1988). Dari kelima lokus
tersebut terdapat dua lokus yang monomorfik yaitu lokus BM1824 dan INRA005
dengan frekuensi alel 1,00. Pada sapi Aceh untuk lokus TGLA227 hanya memiliki
commit to user
dan MM12 masing- masing terdapat 3 alel tetapi terdapat perbedaan frekuensi.
Lokus ETH225 dengan frekuensi alel dari yang tertinggi alel A (0,50), B (0,40)
dan terendah alel C (0,10). Lokus BM1824 dengan frekuensi alel masing-masing
A (0,68), B (0,12), dan C (0,20). Lokus INRA005 dengan frekuensi alel
masing-masing A (0,58), B (0,20), dan C (0,22). Lokus MM12 dengan frekuensi alel
masing-masing A (0,17), B (0,27), dan C (0,55). Secara keseluruhan jumlah alel
yang terdeteksi pada sapi Aceh berkisar antara 1 - 3 dengan rata-rata 2,6. Namun
jumlah alel yang terdeteksi pada sapi Aceh lebih banyak dibanding pada sapi Bali
dari Sumbawa, untuk lokus yang monomorfik hanya pada lokus TGLA227.
Hasil pada studi ini berbeda dengan hasil penelitian terdahulu, pada sapi
Bali untuk lokus INRA005 terdapat 2 alel, ETH225 4 alel dan BM1824 5 alel,
sedangkan pada sapi Aceh untuk lokus INRA005 terdapat 6 alel, ETH225 12 alel
dan BM1824 7 alel (Abdullah et al., 2008). Pada sapi Hariana dan Hissar untuk
lokus TGLA227 5 alel, MM12 3 alel (Rehman dan Khan, 2009). Rendahnya
jumlah alel yang terdeteksi pada penelitian ini mungkin disebabkan antara setiap
individu pada populasi sampel yang digunakan masih ada hubungan kekerabatan
yang dekat.
Analisis sederhana untuk mengetahui diversitas genetik intra spesies sapi
bali dari Sumbawa dan sapi Aceh dapat dilakukan dengan menghitung nilai
jumlah alel, frekuensi alel, Effective number of alleles, homozigositas,
heterozigositas, Shannon's Information index dan PIC pada ke 5 lokus pada kedua
populasi tersebut. Beberapa parameter diversitas genetik pada spesies sapi Bali
commit to user
Tabel 4. Parameter keragaman genetik intra spesies Bali dari Sumbawa dan Aceh pada 5 lokus mikrosatelit. Keterangan: Ne.: Effective number of alleles [Kimura and Crow (1964)]; I.: Shannon's Information index [Lewontin (1972)]; Ho.: Observed Homozigocity; He.: Observed Heterozigocity,. PIC.: Polymorphism Information Content.
Effective number of alleles (Ne) merupakan perkiraan jumlah alel dengan
menyesuaikan frekuensinya pada nilai PIC. Ne terendah yaitu 1,00 pada spesies
Bali dari Sumbawa (BM1824; INRA005) dan Aceh (TGLA227) sedangkan nilai
tertinggi terdapat pada spesies Aceh yaitu sebesar 2,45 (MM12). Nilai Ne rata-rata
yaitu sebesar 1,57 untuk spesies Sapi Bali dari Sumbawa dan 2,03 untuk Sapi
Aceh. Nilai Ne rata-rata yang diperoleh dalam penelitian ini lebih rendah dari nilai
Ne rata-rata pada Sapi Hariana dan Sapi Hissar yang secara berturut-turut 2,87
dan 2,89 yang diteliti oleh Rehman dan Khan tahun 2009. Penelitian Pandey et al
(2006) pada Sapi Kherigarh dari India memiliki nilai Ne rata-rata sebesar 3,77.
Nilai effective number of alleles secara keseluruhan lebih kecil dibandingkan nilai
observed number of alleles (Ne < Na) adalah karena terjadi fluktuasi jumlah
populasi pada masa lalu. Juga terdapat kontribusi dari mutasi (delesi) yang terjadi
pada populasi yang diakibatkan oleh mutasi-keseimbangan seleksi (Crow dan
commit to user
hewan berhubungan dengan variabilitas genetik yang diperlukan untuk
memperkirakan nilai koefisien inbreeding. Karena peningkatan koefisien
inbreeding untuk satu generasi F dapat diperkirakan dari nilai Ne (F=1/2 Ne) .
Untuk mengetahui ukuran diversitas gen dapat dilihat berdasarkan
Shannon's Information index. Pada spesies sapi Bali dari Sumbawa, nilai
Shannon's Information index terbesar terdapat pada lokus MM12 (0,85)
sedangkan yang terendah terdapat pada lokus BM1824 dan INRA005 (0).
Rata-rata nilai Shannon's Information index pada sapi bali dari Sumbawa adalah 0,45.
Sedangkan pada sapi Aceh nilai Shannon's Information index terbesar terdapat
pada lokus MM12 (0,99) sedangkan yang terendah terdapat pada lokus TGLA227
(0). Rata-rata nilai Shannon's Information index pada sapi Aceh adalah 0,75. Nilai
I pada penelitian ini jauh lebih kecil apabila dibandingkan dengan penelitian
Pandey et al (2006) terhadap sapi Kherigarh di India, memiliki nilai I sebesar
1,46.
Analisis sederhana untuk mengetahui heterozigositas genetik dari spesies
sapi Bali-Sumbawa dapat dilakukan dengan menghitung heterozigositas pada ke 5
lokus yang kemudian didapatkan pula nilai tengah atau rata-rata heterozigositas
populasi tersebut. Nilai heterozigositas tiap lokus pada spesies sapi Bali dari
Sumbawa sangat bervariasi yaitu antara 0 sampai 1. Pada spesies sapi Bali dari
Sumbawa nilai heterozigositas terbesar terdapat pada lokus TGLA227 (He= 0.95),
sedangkan yang tidak menunjukkan adanya heterozigositas terdapat pada lokus
BM1824 dan INRA005 (He= 0). Hal ini berbanding terbalik dengan nilai
commit to user
Bali dari Sumbawa adalah pada lokus BM1824 dan INRA005 (Ho= 1,00),
sedangkan yang terendah terdapat pada lokus TGLA227 (Ho=0,05). Rata-rata
nilai heterozigositas pada sapi Bali dari Sumbawa adalah 0,46, sedangkan
rata-rata homozigositasnya adalah 0,54 . Gambaran nilai heterozigositas lebih rendah
daripada nilai homozigositas (He < 50%) yang masih ditemukan pada populasi
sampel sapi Bali dari Sumbawa berdasarkan marker mikrosatelit ini dapat
mengindikasikan bahwa bangsa sapi Bali dari Sumbawa diduga mengalami
seleksi ke arah peningkatan silang dalam (inbreeding), dan ada kecenderungan
pula menurunnya beberapa karakter fenotipik dibandingkan sebelumnya.
Nilai heterozigositas tiap lokus pada spesies sapi Aceh berbeda dengan
nilai heterozigositas pada spesies sapi Bali dari Sumbawa, pada sapi Aceh untuk
nilai heterozigositas terbesar terdapat pada lokus ETH225 (He= 1) ), sedangkan
yang terendah terdapat pada lokus TGLA227 (He= 0). Rata-rata nilai
heterozigositas pada sapi Aceh adalah 0,68, sedangkan rata-rata nilai
homozigositas pada sapi Aceh adalah 0,32. Nilai heterozigositas pada penelitian
ini tidak jauh berbeda dengan penelitian Abdullah et al. (2008) dengan
menggunakan 16 lokus mikrosatelit, rata-rata heterozigositas sapi Aceh He (0,62)
dan sapi Bali He (0,49). Rehman dan Khan (2009) melaporkan rata-rata
heterozigositas pada Sapi Hariana 0,51 dan and sapi Hissar 0,47 sedangkan pada
sapi Kherigarh 0,574 (Pandey et al., 2006).
Data yang diperoleh menunjukkan bahwa heterozigositas pada sapi Aceh
cukup tinggi. Laju heterozigositas yang tinggi diakibatkan oleh adanya silang luar
commit to user
(2008), menjelaskan bahwa out breeding berpengaruh dalam meningkatkan
proporsi gen-gen yang heterozigot (individu yang genotipnya memiliki dua gen/
alel yang berbeda) dan menurunkan proporsi gen yang homozigot (individu yang
genotipnya memiliki dua gen/ alel yang sama). Faktor-faktor yang mempengaruhi
tingginya heterozigot antara lain overdominan (heterosis positif), perbedaan
frekuensi gen antara jantan dan betina, perkawinan yang tidak terpilih atau
assortative mating (Baker, 1994). Kemungkinan lain adalah ada aliran gen dari
bangsa-bangsa sapi lain yang masuk ke dalam populasi sapi Aceh karena
program inseminasi buatan yang diterapkan di Aceh telah menimbulkan
segregasi gen-gen sapi lain yang beragam dan meluas dalam populasi sapi Aceh
dan dapat juga karena belum ada seleksi yang dilakukan. Menurut Muladno
(2006), DNA mikrosatelit memiliki laju perubahan basa nukleotida tinggi yang
disebabkan adanya perubahan jumlah ulangan dari urutan basa bergandengan
mencapai 10-3/gamet/generasi. Laju perubahan mikrosatelit dipengaruhi oleh
motifnya. Mikrosatelit dengan motif dinukleotida memiliki laju mutasi 1,5-2 kali
lebih cepat dibandingkan dengan motif tetra-nukleotida.
Frekuensi alel digunakan pula untuk menentukan nilai PIC, yaitu suatu
nilai yang dapat digunakan sebagai penentu derajat informasi tingkat polimorfik
dari suatu marker yang digunakan (Botstein et al., 1980). Berdasarkan kriteria
Botstein et al nilai PIC yang terletak antara (0,25 < PIC < 0,5) dikategorikan
sebagai lokus yang cukup informatif, sedangkan PIC > 0,5 sangat informatif
untuk analisis genetika populasi. Pada penelitian ini nilai Polymorphic
commit to user
lokus TGLA227 sebesar 0,50, lokus ETH225 sebesar 0,41, lokus BM1824 sebesar
0, lokus INRA005 sebesar 0, dan untuk lokus MM12 sebesar 0,54. Pada Sapi
Aceh untuk lokus TGLA227 sebesar 0, lokus ETH225 sebesar 0,58, lokus
BM1824 sebesar 0,48, lokus INRA005 sebesar 0,57 dan pada lokus MM12
sebesar 0,59. Pada spesies sapi Bali dari Sumbawa terdapat dua lokus yang tidak
menunjukkan adanya polimorfik yaitu lokus BM1824 dan INRA005 dengan nilai
PIC 0, namun berbeda pada spesies sapi Aceh untuk kedua lokus tersebut
menunjukkan adanya polimorfik. Lokus TGLA227 pada spesies sapi Aceh yang
tidak menunjukkan adanya polimorfik, namun polimorfik pada spesies sapi Bali
dari Sumbawa. Hasil yang diperoleh dalam penelitian ini berbeda dengan hasil
penelitian Zhou et al. (2005) terhadap lokus mikrosatelit yang sama pada lima
jenis ternak lokal di Cina yaitu ETH225, BM1824, TGLA227 pada penelitian
yang dilakukan Zhou et al. (2005) lokus ETH225, BM1824 dan TGLA227
ketiganya memiliki nilai PIC > 0,50. Penelitian yang dilakukan oleh Pandey et al.
(2006), terhadap sapi Kherigarh di India menyebutkan bahwa nilai PIC untuk
lokus INRA005 dan ETH225 > 0,50. Penelitian yang dilakukan Sodhi et al.
(2006), terhadap Sapi Zebu di India menyebutkan hal yang sebaliknya, yaitu
bahwa nilai PIC untuk lokus ETH225 < 0,50. Berdasarkan perbedaan data-data
tersebut maka dapat disimpulkan bahwa lokus mikrosatelit yang sama memiliki
optimasi kerja yang berbeda untuk jenis ternak yang berbeda.
Adapun rata-rata polimorfisme alel yang rendah pada sapi lokal spesies
sapi Bali dari Sumbawa 0,29 dan sapi Aceh 0,44 dalam studi ini kemungkinan
commit to user
yang diduga masih belum maksimal mewakili populasi yang ada serta memiliki
diversitas letak geografis yang sangat jauh sehingga memungkinkan terjadinya
keragaman genetik akibat proses seleksi. Menurut Meadows et al. (2006), hal ini
disebabkan adanya beberapa faktor potensial, diantaranya faktor seleksi, sistem
perkawinan, atau pola migrasi, serta mekanisme lain yang berakibat pada
rendahnya jumlah alel efektif untuk ukuran populasi.
Hal lain adalah budaya masyarakat setempat, yang sangat membatasi
ternak jantannya digunakan sebagai sumber genetik sehingga penggunaan
pejantan sangat terbatas. Hal ini dimungkinkan karena selama ini peternak cukup
protektif terhadap perkawinan silang sehingga sumbangan keragaman genetik
dari luar cukup rendah. Namun, juga tidak diikuti dengan transfer atau
perpindahan pejantan dari breed yang sama antar peternak yang secara geografis
letaknya cukup jauh, sehingga memungkinkan terjadinya pemanfaatan antar
pejantan. Kenyataan lain, peternak rakyat lebih senang memelihara induk
daripada pejantan dengan tujuan reproduksi, yakni mendapatkan pedet daripada
penggemukan pejantan. Namun, dengan pertimbangan bahwa kualitas sapi Bali
sebagai ikon sapi lokal Indonesia yang saat ini kenyataannya cenderung menurun
secara genetis, maka sebenarnya dengan memanfaatkan beberapa marka
mikrosatelit maupun gen yang telah diketahui, maka masih dapat dilakukan
seleksi lebih lanjut untuk mendapatkan kualitas sapi lebih baik (Winaya, 2010).
Namun dari studi ini yang menunjukkan bahwa semua lokus teramplifikasi
pada semua populasi sampel, maka mikrosatelit ini dapat diuji lebih lanjut untuk
commit to user
breed lokal. Untuk menjamin akurasi serta kestabilan alel yang dihasilkan, maka
penggunaan marka mikrosatelit tersebut sebaiknya perlu dilakukan uji lebih
lanjut atau berulang kali, misal dengan uji keturunan (pedigree) dan segregasi
Mendel dengan menggunakan jumlah individu lebih banyak serta cakupan
geografis yang luas sehingga dapat digunakan untuk estimasi evaluasi genetik
maupun filogenetik bagi ternak-ternak domestik di Indonesia ataupun yang
terkait dapat ditetapkan berdasarkan penanda ini.
B. Diversitas Inter Spesies
Pada diversitas inter spesies sapi Bali dari Sumbawa dan sapi Aceh ini
memiliki jumlah alel antara 2 - 3 dengan rata-rata jumlah alel lebih besar
dibanding pada intra spesies yaitu sebesar 0,28. Frekuensi alel bervariasi antara
0,06 – 0,84, frekuensi alel tertinggi terdapat pada lokus BM1824 alel A (0,84) dan
terendah pada lokus BM1824 alel B (0,06). Beberapa parameter diversitas genetik
inter spesies sapi Bali dari Sumbawa dan sapi Aceh dapat dilihat pada tabel 5.
Table 5. Parameter keragaman genetik inter spesies Bali dari Sumbawa dan Aceh pada 5 lokus mikrosatelit.