Teknologi Genome Scanning Menggunakan 1536- dan 384-SNP Chip untuk
Mendukung Program Pemuliaan Padi
Rice Genome Scanning using 1536- and 384- SNP Chip Technology for
Supporting Rice Breeding Programs
Dwinita W. Utami
Indonesian Centre of Agricultural Biotechnology and Genetic Resources R&D, Jl. Tentara Pelajar 3A, Bogor 16111.
I N F O A R T I K E L A B S T R A C T
Keywords:
keyword1 : Indonesian rice germplasm keyword2 : 1536-and 384-SNP set keyword3 : genotyping and phenotyping
keyword4 : association analysis
Kata Kunci:
kata Kunci1 : Plasma nutfah padi Indonesia
kata Kunci2 : 1536-dan 384-SNP set kata kunci3 : genotyping dan phenotyping
kata kunci4 : analisis asosiasi
Rice is a staple crop in Indonesia whic have the stategic impact. Increasing and stabilizing rice production as main target in agriculture development. Utilization of molecular markes technology gain prominance in rice breeding, particularly to speed up and precission increasing of selection step. Towards this technology, utilization of rice genomic are now being widely used to provide accelerate the rice breeding program particularly on genetic relatedness, linkage to important traits, and detection of donor introgressions in segregating population. Finally, all of these programs, were targeted to support the attainment of food self-sufficiency in Indonesia. This paper summarizes the genome research status of recent developments in ICABIOGRAD, R & D Agency, The Ministry of Agriculture. Rice genome research was initiated in ICABIOGRAD, IAARD, Ministry of Agriculture, since 2011. Currently, the total of 851 accessions of Indonesian rice germplasm were scanned their genome using 2 sets of SNP (Single Nucleotide Polymorphism) markers chip. To acces the expression, in parallel, has also carried out some phenotyping analysis in across conditions, dry and rainny seassons. Some important traits which were targetted in these analysis, as : date to flowering, date to harvesting, yield components and also respons to biotics and abiotics stress. A large number of total data both genotyping and phenotyping were analyzed on their association to obtaine a set of SNP markers and selected genotypes which significant associated with the target traits. The selected SNP markers could be as markers assited for selection while the selected genotypes could be a new promising rice lines or to to be as a source of potential genes for breeding. In other way, the genotypes and phenotypes data have completed were also applicable for DNA fingerprints detections as a barcode of commercial variety. These rice genome research achieved will packaging as the inovation technology which advantage provide for rice stakholders in Indonesia.
S A R I K A R A N G A N
Padi adalah tanaman pangan utama Indonesia yang bernilai strategis. Peningkatan dan stabilitas produksi padi menjadi target utama pembangunan pertanian. Perkembangan teknologi marka molekular dalam pemuliaam padi berpotensi mempercepat dan meningkatkan presisi seleksi dalam program pemuliaan padi. Melalui pengembangan teknologi marka molekuler, saat ini informasi genome telah diaplikasikan secara luas terutama dalam hal untuk menganalisis kekerabatan genetik antar aksesi, keterpautan dengan sifat penting dan introgresi pada populasi segregan. Semua program pemuliaan tersebut pada akhirnya bermuara untuk mendukung pencapaian ketahanan dan kemandirian pangan di Indonesia. Makalah ini merangkum status perkembangan terkini penelitian genom padi di BB Biogen, Badan Litbang, Kementerian Pertanian. Penelitian genom padi di BB Biogen, Balitbang Kementan, telah diinisiasi mulai tahun 2011 dan sampai saat ini telah dianalisis genotyping pada 851 aksesi plasma nutfah padi menggunakan 2 set marka SNP chip, 1536 dan 384. Secara paralel juga telah dianalisis phenotyping sejumlah aksesi yang sama lintas musim dan lintas agroekosistem, untuk beberapa karakter agronomi penting terkait dengan umur tanaman komponen hasil dan ketahanan terhadap cekaman biotik ataupun abiotik. Sejumlah besar total data baik data genotyping ataupun phenotyping di atas selanjutnya dianalisis asosiasi keduanya untuk mendapatkan set marka-SNP chip dan set genotipe terpilih yang terkait dengan karakter target. Pengembangan data genotipe yang diperoleh, dilakukan untuk deteksi sidik jari DNA beberapa aksesi plasma nutfah penting sebagai penciri spesifik varietas (sebagai barcode variety). Keluaran hasil penelitian berbasis genome ini mulai dikemas sebagai paket inovasi teknologi yang dimanfaatkan oleh para stakeholder padi di Indonesia.
© Forum Tahunan Pengembangan Iptek dan Inovasi Nasional IV, Tahun 2014
Corresponding author : E-mail address: [email protected]; [email protected]
Forum Tahunan Pengembangan Iptek dan Inovasi Nasional IV, Tahun 2014
PENDAHULUAN
Pertumbuhan penduduk yang terus terjadi dengan luas lahan pertanian yang cenderung tetap
bahkan berkurangmenyebabkan terjadinya ancaman
krisis pangan. Hal ini diperparah dengan pengaruh perubahan iklim global yang menekan produktifitas tanaman pangan. Pemenuhan kebutuhan pangan melalui peningkatan produktivitas pada saatnya tidak akan mampu memenuhi permintaan pangan. Perluasan lahan (ekstensifikasi) pertanian adalah pendekatan utama yang harus ditempuh untuk menggenjot produksi pangan nasional.
Pengembangan teknologi dapat diaplikasikan untuk mendukung peningkatan produksi tanaman pangan. Teknologi genom salah satu teknologi yang dapat diaplikasikan mulai dari hulu sampai hilir dari serangkaian perakitan varietas unggul tanaman pangan. Teknologi genom dapat diaplikasikan mulai dari pengenalan keragaman genetik sampai dengan pengembangan plasma nutfah sebagai sumber genetik bagi galur unggul baru. Pengetahuan tentang genetika sifat-sifat unggul tanaman padi yang
didukung teknologi genom memungkinkan
dilakukannya percepatan program pemuliaan dan perakitan varietas baru dengan akurasi dan efisiensi yang lebih tinggi, baik melalui seleksi berbasis marka DNA maupun perakitan tanaman transgenik.
Saat ini telah dilakukan pengembangan teknologi high-throughput genotyping dan genome
sequencing sebagai bagian dari pengembangan
lanjut teknologi genomik. Melalui teknologi ini dimungkinkan untuk membuka lembaran baru dalam pemanfaatan sejumlah besar plasma nutfah padi untuk diungkap factor genetic (gen-gen)
penting yang dimilikinya sehingga dapat
mempercepat proses pemuliaan dan pencapaian swasembada pangan.
Pemetaan asosiasi lintas genom (Genome
Wide Association Mapping/GWAS), saat ini telah
banyak dimanfaatkan, diantaranya dalam hal : a. Analisis kandidat gen penting dimungkinkan
untuk melacak keberadaan ‘alamat’ gen target dalam genom beserta fungsinya terkait dengan proses biokimia atau regulation pathway yang berhubungan dengan sifat unggul tanaman padi (Yu and Buckler, 2006).
b. Ketersediaan sekuen genom lengkap pada beberapa spesies selain padi dan dukungan
beberapa teknologi genomika, seperti
sequencing, genotyping, gene expression
profiling, comparative genomics,
bioinfotmatics, linkage analysis, mutagenesis dan biokimia, memungkinkan diketahuinya
gen-gen fungsional untuk beragam karakter penting yang bersifat kuantitatif, seperti komponen hasil pada beragam plasma nutfah (Zhu et al, 2008). c. Ketersediaan sekuen genom lengkap dan
teknologi resequencing juga memungkinkan untuk menemukan ratusan bahkan ribuan marka SNP (Single Nucleotide Polymorphism) yang fungsional untuk dikembangkan sebagai marka molekuler fungsional untuk membantu seleksi pada beragam plasma nutfah terkait dengan karakter yang diinginkan. Penggunaan marka SNP yang bersifat bi-allelic lebih mudah untuk analisis genotyping dan asosiasi (McNally et al, 2009).
Dengan mengacu pada pemanfaatan teknologi berbasis genomika, maka penelitian genom padi yang dilakukan di BB Biogen mencoba menjawab beberapa masalah untuk peningkatan produksi padi
nasional. Makalah ini merangkum status
perkembangan terkini penelitian genom padi di BB Biogen, Badan Litbang, Kementerian Pertanian.
KERANGKA TEORI
Indonesia memiliki keragaman genetik tanaman padi yang melimpah. Kekayaan keragaman genetik ini merupakan bahan dasar untuk memperbaiki berbagai karakter padi, seperti karakter komponen produksi dan umur genjah. Padi lokal adalah salah satu sumber genetik padi yang saat ini sudah terdesak oleh varietas unggul (Crowder, 1997).
Pemanfaatan teknologi dapat mendukung
peningkatan produksi pangan nasional.
Perkembangan teknologi genomik, telah
memungkinkan untuk mengidentifikasi alel-alel dari gen-gen potensial dalam koleksi plasma nutfah padi
yang melimpah untuk dimanfaatkan dalam
perbaikan varietas. Percepatan program pemuliaan dengan memanfaatkan informasi genom memiliki manfaat yang besar dalam penciptaan galur unggul baru.
Pengembangan penelitian Genom dapat
diibaratkan seperti teknologi penginderaan jarak jauh menggunakan citra satelit yang digunakan untuk mendeteksi potensi sumber daya alam di suatu titik lokasi, maka teknologi genom padi ini ditujukan untuk ‘memotret’ potensi genetik terkait dengan karakter unggul pada ratusan koleksi aksesi plasma nutfah, sehingga aksesi-aksesi plasma nutfah tersebut dapat lebih termanfaatkan secara optimal. Secara lebih jauh, dengan termanfaatkannya plasma nutfah padi kita, terutama padi-padi lokal secara lebih optimal diharapkan dapat mendukung
molecular genetic, dan physical) sampai dengan
sekuen lengkap genom padi yang merupakan terobosan penting untuk mengungkap bagian-bagian fungsional dari genom padi (rice functional
genomic). Teknologi ini berperan dalam beberapa
karakter kompleks padi, seperti karakter produksi dan umur berbunga pendek (Tyagi et al, 2004; Yamamoto et al, 2009). Identifikasi genomik dari beragam variasi genetik padi diharapkan dapat mendukung percepatan program pemuliaan yang pada akhirnya dapat mendukung ketahanan pangan nasional.
METODE PENELITIAN
Disain dan seleksi sub set marka SNP-chip
Pada tahap awal disain 1536 dan 384 SNP chip dilakukan dengan berkoordinasi dengan beberapa institusi internasional yang sudah mengembangkan illumina-SNP-chip lebih dahulu, yaitu IRRI dan Cornell University (Zhao et al. 2010). Disamping itu disain SNP chip juga dilakukan dengan menganalisis sekaligus mengkompilasi marka SNP polimorfis tinggi yang dapat diakses di website Oryza SNP Database. Setiap marka SNP dirunut sekuen lengkapnya dan disain primer dibuat dengan memposisikan SNP tepat di tengah sikuen dan diapit oleh 50-60 pasang basa nukleotida. Jumlah marka SNP yang dipilih harus 2 kali dari target SNP-chip yang akan didisain. Validasi masing-masing marka SNP yang terdisain dilakukan dengan sistem deteksi
GoldenGate Illlumina-Align Disaign Tool/ADT.
Setelah melalui sistem skoring sesuai persyaratan untuk assay Golden Gate Illumina, maka terpilih 1536 atau 384 marka SNP yang selanjutnya akan disintesis oleh Illumina sebagai 384 atau 1536 SNP chip. Hasil sintesis SNP chip ini dinamakan sebagai custom OPA dan dilengkapi dengan assay kit
custom synt 1536 atau 384 SNP bead chip telah siap
untuk digunakan dalam analisis genotyping
menggunakan metode GoldenGate reader dengan mesin iScan.
Pemilihan marka SNP yang akan didisain sebagai SNP-chip dapat dikembangkan sesuai dengan target tujuan pemuliaan tertentu. Seperti misalnya untuk tujuan identifikasi profile genotipe (sidikjari DNA) plasma nutfah padi sebagai penciri varietas, maka dipilih SNP yang menyebar di total 12 kromosom genom padi dengan rata-rata jarak per SNP bervariasi untuk masing-masing kromosom. Marka SNP penciri genotipe tersebut dipilih tanpa
karakter tertentu, maka seleksi subset SNP dilakukan pada posisi genetik dari gen/QTL karakter target .
Analisis data
a. Studi keragaman genetik
Untuk mengurangi kesalahan terjadinya asosiasi yang disebabkan oleh struktur populasi, data struktur populasi padi yang dipelajari dikalkulasi
menggunakan software Structure
(http://pritch.bsd.uchicago.edu/structure.html)
dengan menggunakan data genotipe SNP sebagai input, untuk digunakan sebagai covariate dalam analisis asosiasi menggunakan Tassel.
b. Pemetaan gen/QTL : Teknologi Pemetaan dengan populasi persilangan LE
Untuk memahami mekanisme yang
menyebabkan sebuah populasi berevolusi, perlu memperhatikan kondisi-kondisi apa saja yang diperlukan oleh suatu populasi untuk tidak berevolusi. Asas Hardy-Weinberg menyatakan bahwa frekuensi alel (variasi pada sebuah gen) pada sebuah populasi yang cukup besar akan tetap konstan jika gaya dorong yang terdapat pada populasi tersebut hanyalah penataan ulang alel secara acak selama pembentukan gamet dan kombinasi acak sel genetik ini selama penyerbukan. Populasi seperti ini tidak berevolusi dan dikatakan sebagai dalam populasi kesetimbangan Hardy-Weinberg atau populasi kesetimbangan pautan (Linkage equilibrium / LE) (Kerry, 2007). Dalam kaitan dengan pemetaan kelompok pautan maka populasi LE terbentuk dari populasi pemetaan hasil persilangan 2 tetua. Pada kondisi ini alel / lokus bersifat random (random association). Jika 2 alel / lokus berada dalam kondisi LE , maka kedua alel / lokus tersebut diwariskan secara bebas (independent
inheritad) (Bovenhuis dan Meuwissen, 1996). d. Pemetaan LD dan analisis asosiasi : Teknologi
pemetaan dengan populasi alami
Perkembangan teknologi genomik, yang meliputi
pemetaan QTL (Quantitative Trait Locus),
penemuan gene (gene discovery), dan tersedianya sekuen genom secara lengkap telah memungkinkan untuk mencari alel-alel yang berguna dalam koleksi plasma nutfah padi untuk perbaikan varietas melalui suatu strategi yang disebut dengan allele mining. Pemetaan LD (linkage disequilibrium) dilakukan untuk mendapatkan daerah genom yang sempit yang mengandung alel yang diinginkan (Morton, 2005).
Berlainan dengan pemetaan QTL, yang
Forum Tahunan Pengembangan Iptek dan Inovasi Nasional IV, Tahun 2014
tetua saja, pemetaan LD dapat menggunakan secara langsung beberapa sampai ratusan aksesi yang berbeda untuk membuat peta tanpa harus melakukan persilangan (Flint-Garcia et al. 2003). Jika dua alel/lokus berada dalam LD, maka kedua alel/lokus diwariskan secara bersama (dependent inherited) (Bovenhuis dan Meuwissen, 1996).
Analisis asosiasi dilakukan menggunakan
software Tassel (Bradbury et al., 2007). Data marka
dan fenotipe digunakan sebagai input bagi software Tassel untuk mencari asosiasi antara marka dengan karakter fenotipe yang diukur. Marka yang secara signifikan memiliki asosiasi dengan karakter yang diukur diidentifikasi melalui pendekatan general
linear model (GLM) dan mixed linear model
(MLM). Marka-marka dianggap memiliki asosiasi yang signifikan apabila berdasarkan analisis GLM atau MLM memiliki p-value lebih kecil dari 0,001.
HASIL DAN PEMBAHASAN
Filterring kualitas SNPs terbaik
Dari total data genotipe yang diperoleh tidak semuanya dapat diikutsertakan dalam analisis. Beberapa tahapan seleksi (filtering) secara bertahap dilakukan menggunakan beberapa program. Seleksi
pertama dilakukan menggunakan program
ALCHEMY dengan parameter koefisien inbreeding (inbreeding coefficient) 0.9 dan batas kepercayaan seleksi (confidence cut off threshold) 0.80.
Selanjutnya filtering dilakukan menggunakan
program PowerMarker untuk mendapatkan informasi skor minor allele frequency (MAF) dan dipilih marka SNP yang memiliki nilai MAF ≥ 0,7. Hal ini menunjukkan bahwa marka-marka ini memiliki kemampuan membedakan (polimorfis) diantara aksesi-aksesi plasma nutfah yang dianalisis.
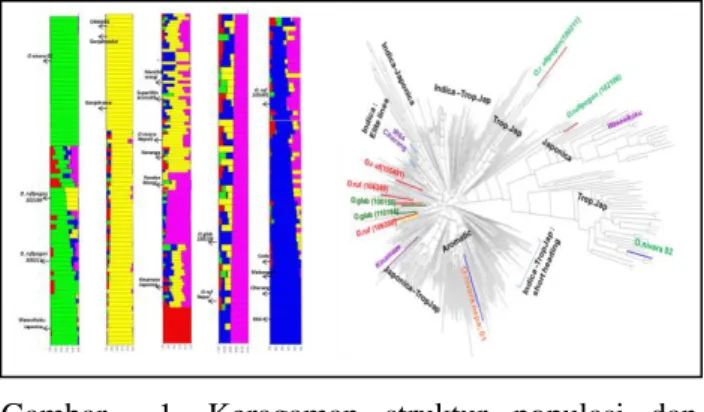
Struktur populasi dan keragaman genetik
Analisis struktur populasi menggunakan
program STRUCTURE dengan nilai variasi K=5 (Gambar 3A) menunjukkan adanya 5 subpopulation utama, yaitu : I. Temperate Japonica (Japonica);
II.Tropical Japonica (Javanica); III. Aromatic, IV.Kelompok introgresi luas dan V. Indica.
Introgresi dari subpopulation Indica (warna biru) tampak menyebar paling luas lintas subpopulasi yang lain, seperti Temperate Japonica, Tropical Japonica dan bahkan introgres ke spesies padi liar
Oryza rufipogon. Hasil ini sesuai dengan fakta
bahwa sebagian besar varietas padi komersial di Indonesia (released varieties) dirakit menggunakan
latar belakang genetik Indica. Diantara Introgresi luas antara Indica dengan O. rufipogon telah menghasilkan 2 varietas yang dilepas yaitu: Blas (SK No:3920/Kpts/SR.120/3/2013) dan Inpari-HDB (SK No :3916/Kpts/SR.120/3/2013).
Gambar 1. Keragaman struktur populasi dan dendrogram keragaman genetik dari 467 aksesi plasma nutfah padi.
Dendrogram keragaman genetik tergambar adanya hubungan kekerabatan antar kelompok subspecies plasma nutfah padi. Beberapa aksesi spesies padi liar terlihat menyebar pada kelompok-kelompok yang terpisah. Spesies padi liar diyakini sebagai ancestor dari padi budidaya (O.sativa).
Melalui beragam proses domestikasi yang
dipengaruhi oleh cara budidaya manusia di areal geographical yang berbeda-beda, terbentuklah padi budidaya dari spesies padi liar (Londo,et al, 2006). Dendrogram keragaman genetik dapat memberikan informasi kedekatan genetic satu aksesi plasma nutfah dengan aksesi atau kelompok genetic yang lain.
SidikJari DNA sebagai penciri plasma nutfah
Untuk tujuan mengidentifikasi penciri genetik suatu aksesinutfah maka teknologi sidik jari DNA
menggunakan marka-marka SNP terpilih
memungkinkan untuk mendapatkan penciri genetik yang bersifat unik yang dapat diaplikasikan pada sejumlah genotype tertentu. Disain marka SNP spesifik untuk setiap subspesies padi yang berbeda (Indica/Japonica/Tropical Japonica) dapat berperan penting dalam deteksi identitas varietas dan kemurnian benih.
mendapatkan penciri unik dari setiap varietas.
Pemetaan QTL dan GWAS (Genome Wide Association Mapping)
1. Pemetaan QTL ketahanan blas pada padi lokal Penggunaan 1536 atau 384 SNP lebih efektif dan efisien untuk menentukan polimorfisme diantara
genepool yang berbeda. Oleh karena itu untuk
tujuan pemetaan gen / QTL tertentu harus diperhatikan beberapa marka SNP yang polimorfis untuk kedua tetua persilangan.
Gambar 3. Seleksi populasi dilakukan menggunakan marka SNP yang bersifat polimorfis untuk kedua tetua persilangan, yaitu : Padai Emas/US2; Gampai/US2 dan Grogol/US2.
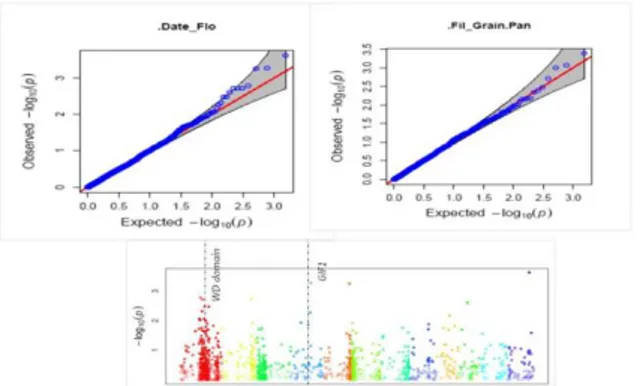
2. Pemetaan asosiasi (GWAS) untuk karakter umur genjah dan komponen hasil unggul padi.
Pendekatan GWAS dilakukan untuk
mendapatkan asosiasi antara data genotipe dan fenotipe pada sejumlah besar aksesi plasma nutfah. Beberapa karakter penting yang menjadi target penelitian GWAS diantaranya adalah umur genjah dan komponen hasil unggul.
Gambar 4. Manhattan Quantile-Quantile Plot (Q-Q plots), hasil analisis GWAS untuk karakter unggul. X axis : sebaran SNP per kromosom dan Y axis : –log (P-value) sebagai skor asosiasi.
3. GWAS untuk sifat toleran keracunan Al
Pendekatan GWAS juga telah diaplikasikan pada sejumlah aksesi hasil persilangan beragam untuk mendapatkan alel potensial yang berperan dalam membentuk sifat toleran keracunan Al pada padi.
Berdasarkan profil genotipe akan diketahui
alel/QTL mana yang berperan dalam membentuk sifat toleran terhadap keracunan Al dari galur-galur terpilih.
Gambar 5. Diantara hasil analisis profile genotipe yang memiliki karakter kontras untuk sifat toleran keracunan Al. Alel potensial dapat dilacak dengan membandingkan profile genotype toleran dengan yang peka.
Pengembangan marka untuk proses seleksi (Marker assisted selection / MAS)
Sebagai langkah untuk validasi hasil GWAS maka perlu dilakukan analisis kandidat gen terkait karakter target (umur genjah dan komponen hasil). Pengembangan dari hasil analisis asosiasi di atas adalah analisis kandidat gen, yaitu dengan menelusuri gen/lokus yang terkait dengan karakter target berdasarkan posisi genetik dari marka SNP yang signifikan tersebut. Sebagai contoh salah satu marka SNP signifikan adalah TBGI068738 dan kandidat gen yang terdeteksi adalah LOC-Os01g72220, yang terpetakan di kromosom 1 pada posisis genetik: 41,837,949-41,880,601, yaitu gen
WD domain, G-beta repeat domain. Gen ini
berfungsi dalam pengaturan waktu pembungaan sampai pemasakan (fase generatif lanjut) (Ouyang
Forum Tahunan Pengembangan Iptek dan Inovasi Nasional IV, Tahun 2014
et al, 2012). Untuk mengidentifikasi gen ini maka
dilakukan disain primer dan digunakan untuk analisis PCR pada sampel yang memiliki karakter kontras. Primer ini ditujukan sebagai marker yang dapat diaplikasikan dalam proses seleksi galur-galur hasil pemuliaan (sebagai marka MAS/ Molecular
Assited Selection).
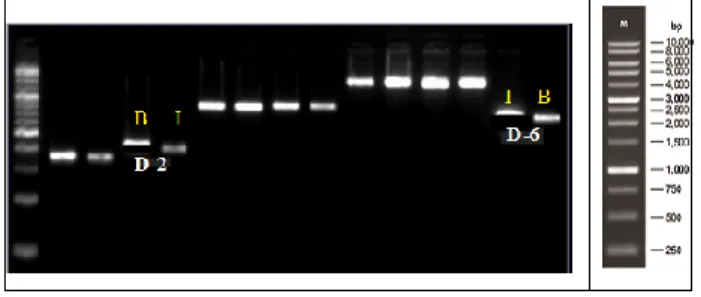
Gambar 6. Hasil amplifikasi menggunakan primer D-2 pada sampel B (BMIP22) dan I (IRBL2) dan primer D-6 pada sampel I (IRBLTa2) dan B (Bio110). Dirunning pada 2% TAE agarose gel pada 100V selama 3 jam.
Hasil pada Gambar 6 di atas menunjukkan bahwa primer D-2 terindikasi sebagai marka MAS karena bersifat polimorfis pada sampel yang memiliki umur yang berbeda. Demikian juga untuk primer D-6 karena bersifat polimorfia pada sampel yang memiliki jumlah gabah isi yang berbeda.
Pengembangan ’Database Variasi Genetis Plasma Nutfah Padi’
Pembuatan dan pengembangan database padi ditujukan untuk mendokumentasikan sekaligus mempublikasikan hasil penelitian genom padi sehingga dapat dimanfaatkan secara lebih optimal bagi para stakeholder padi di Indonesia. Secara bertahap Database Genom-Padi ini ditampilkan dalam bentuk WebSite sehingga dapat diakses secaa terbuka.
PENUTUP
Pengembangan teknologi high-throughput
genotyping menggunakan 1536 atau 384 SNP chip
dapat mengungkap gen-gen penting yang dimiliki sejumlah besar plasma nutfah padi sehingga dapat mempercepat proses pemuliaan yang selanjutnya dapat mendukung ketahanan pangan nasional. Namun demikian teknologi ini perlu didukung dengan pengembangan sistem kerjasama dalam bentuk konsorsium nasional mengingat besarnya dukungan dana yang diperlukan.
UCAPAN TERIMA KASIH
Penelitian ini terlaksana atas dukungan dan koordinasi antara tim genom padi dan tim penelitian lain. Penelitian genom padi didanai oleh DIPA BBIOGEN-Badan Litbang Pertanian, Kementerian Pertanian, tahun anggaran 2010-2014.
DAFTAR PUSTAKA
Bovenhuis, H., & Meuwissen,T. (1996). Detection and mapping of quantitative trait loci. Animal Genetics and Breeding Unit. UNE. Armidale. Australia. ISBN1863893237.
Bradbury P.J., Z.Zhang, D.E.Kroon,
T.M.Casstevens, Y Ramdos, E.D.Buckler. 2007. Tassel : software for association mapping
of complex traits in diverse samples.
Bioinformatics Appl. Note. Vol23, No19 : 2633-2635.
Crowder, L.V. (1997). Genetika Tumbuhan. Gadjah Mada University Press.
Flint-Garcia, S.A., Thornsberry, J.M., Buckler, E.S. (2003). Structure of linkage disequilibrium in plants. J. Annu.Rev. Plant.Biol. 54:357-374. Kerry, B. (2007). Cause of evolution. Teach
Evalution and Make It Relevant. National
Science Foundation
(http://www.evolved.org/lessons/speciation.ht m). Diakses tanggal 30 Desember 20012 McNally, K.L.,Childs,K.L.,Bohert, R.,Davidson,
R.M.,Zhao, K., Ulat, B.J., Zeller, G.G., Clark, R.M., Hoen, D.R., Bureau, T.E. (2009).
Genome-wide SNP variation reveals
relationships among landraces and modern varieties of rice. Proc. Natl. Acad. Sci. USA 106:12273–12278.
Morton, N.E. (2005). Linkage disequilibrium and
association pemetaan. J. Clin. Invest.
115:1425-1430.
Ouyang, Y., Huang, X., Lu, Z.,Yao, J. (2012). Genomic survey, expression profile and co-expression network analysis of OsWD40 family in rice. BMC Genomics 2012, 13:100.http://www.biomedcentral.com/1471-2164/13/100.
Tyagi, A., J.P. Khurana, P. Khurana, S. Raghuvanski, A. Gaur, A. Kapur, V. Gupta, D. Kumar, V. Ravi, S.Viji, P. Khurana, and S. Sharma. 2004. Structural & Functional analysis of rice genome. J. Genetics : 83, 79-99.
Yu, J. and E.S. Buckler. 2006. Genetic association mapping and genome organization of maize. Curr.Opin. Biotechnol, 17:155-160.
Zhao, K., Wright, M., Kimball, K.J., G. Eizenga, A. McClung, M. Kovach, W. Tyagi, L. Ali, C.-W. Tung, A. Reynolds et al. (2010) Genomic diversity and introgression in O. sativa reveal the impact of domestication and breeding on the rice genome. PLOS One 5: e10780.
Zhu, C., Gore, M., Buckler, E.S., Yu, J. (2008). Status and prospects of association mapping in plants. The Plant Genome, 1: 5-20.
Dwinita W. Utami
Lahir di Yogyakarta, 22 Maret 1969. Penulis
menamatkan pendidikan Pascasarjana bidang
aplikasi marka molekuler dalam pemuliaan serta penelitian genomik padi telah dihasilkan. Berbagai
seminar sebagai pembicara maupun visiting
research mengenai Bioteknologi, khususnya tentang
marka molekuler dan genomik telah dilakukannya baik di dalam maupun luar negeri.