KARAKTERISASI MOLEKULER PADI TRANSGENIK
DENGAN BEBERAPA METODE ISOLASI DNA
RIZKI AYU KARTINI
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2012
ABSTRAK
RIZKI AYU KARTINI. Karakterisasi Molekuler Padi Transgenik dengan
Beberapa Metode Isolasi DNA. Dibimbing oleh EMAN KUSTAMAN dan
KURNIAWAN RUDI TRIJATMIKO.
Tanaman padi merupakan penghasil beras sehingga menjadi salah satu
komoditas pangan utama. Ukuran butir merupakan kualitas yang sangat penting
dalam beras. Salah satu usaha yang dilakukan dalam penelitian guna
meningkatkan produksi padi adalah melalui perakitan tanaman transgenik yang
disisipkan gen Os-GS3 (Oryza sativa Grain Size 3). Materi yang digunakan dalam
penelitian ini adalah padi varietas T-309. Padi yang telah disisipi gen Os-GS3
ditanam dan dianalisis keberadaan gennya melalui PCR serta karakterisasi
molekulernya melalui hibridisasi Southern. Metode isolasi DNA yang digunakan
dalam penelitian ini adalah skala cepat, skala miniprep, dan skala besar, sehingga
diperoleh metode yang efisien dari segi waktu penyelesaian dan sumberdaya yang
digunakan untuk PCR dan hibridisasi Southern. Analisis PCR dari ketiga metode
menghasilkan ukuran pita yang sama, yaitu 500 bp. Pengujian hibridisasi
Southern menggunakan metode skala besar menghasilkan pita atau salinan gen,
sedangkan pada metode miniprep tidak ada. Metode skala cepat adalah metode
paling efisien untuk analisis PCR, sedangkan metode skala besar paling efisien
untuk hibridisasi Southern. Hasil pengujian pada pembungkaman ekspresi gen
Os-GS3 dengan antisense RNA berhasil ketika 4 transgen yang terintegrasi ke dalam
genom tanaman.
ABSTRACT
RIZKI AYU KARTINI. Molecular Characterization of Transgenic Rice Plants
with of DNA Isolated using Multiple Methods. Under the direction of EMAN
KUSTAMAN and KURNIAWAN RUDI TRIJATMIKO.
Rice is a major food commodity for agroindustrial country. Grain size is a
major determinant of grain quality. One attempt that has been made in research to
increase rice production is assembly of transgenic plants inserted with Os-GS3
(Oryza sativa Grain Size 3) gene. The material used in this study is cultivar Taipei
309. Rice plants that have been inserted with Os-GS3 gene were grown and the
presence of the genes was analyzed through molecular characterization by PCR
and Southern hybridization. DNA isolation methods used in this study were the
simple method, miniprep scale, and large scale. They were compared to obtain the
most efficient method in terms of turnaround time and resources used for PCR
and Southern hybridization. PCR analysis of the three methods produced bands of
the same size, in that 500 bp. Southern hybridization test of the DNA isolated
using the large scale method produced bands or copy genes, where as that isolated
using miniprep method did not produce band. The fast scale method is the most
efficient DNA isolation method for PCR analysis, where as the large scale method
is the most efficient DNA isolation method for Southern hybridization. The results
in the silencing of gene expression of Os-GS3 with antisense RNA was successful
when 4 transgenes integrated into the plant genome.
KARAKTERISASI MOLEKULER PADI TRANSGENIK
DENGAN BEBERAPA METODE ISOLASI DNA
RIZKI AYU KARTINI
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biokimia
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
BOGOR
2012
Judul Skripsi : Karakterisasi Molekuler Padi Transgenik dengan Beberapa
Metode Isolasi DNA
Nama
: Rizki Ayu Kartini
NIM
: G84080061
Disetujui
Komisi Pembimbing
Ir. H. Eman Kustaman
Dr. Kurniawan Rudi Trijatmiko
Ketua
Anggota
Diketahui
Dr. I Made Artika, M. App. Sc
Ketua Departemen Biokimia
PRAKATA
Syukur Alhamdulillah penulis panjatkan kehadirat Allah SWT yang telah
melimpahkan rahmat dan hidayah-Nya sehingga penulis dapat menyelesaikan
penelitian dan penulisan skripsi ini dengan baik yang berjudul “Karakterisasi
Molekuler Padi Transgenik dengan Beberapa Metode Isolasi DNA”. Kegiatan
penelitian yang merupakan salah satu syarat memperoleh gelar Sarjana Sains pada
Departemen Biokimia ini dilakukan dari bulan Februari 2012 sampai dengan Juli
2012 di Laboratorium Balai Besar Penelitian dan Pengembangan Bioteknologi
dan Sumber Daya Genetik Pertanian, Jalan Tentara Pelajar 3A, Bogor, Jawa
Barat.
Penulis mengucapkan terima kasih kepada semua pihak yang telah
membantu dalam penyelesaian penelitian ini baik secara langsung maupun tidak
langsung. Ucapan terimakasih penulis sampaikan kepada Ir. H. Eman Kustaman
selaku ketua pembimbing dan Dr. Kurniawan Rudi Trijatmiko selaku anggota
pembimbing yang telah memberikan bimbingan, motivasi, dan masukan.
Terimakasih penulis persembahkan kepada kedua orang tua tercinta, kakak,
abang, M. Yuda Ramdani atas segala kasih sayang, perhatian, doa dan
dukungannya dalam setiap langkah penyusunan karya ilmiah ini baik semangat
maupun motivasi untuk selalu berusaha agar menjadi lebih baik. Tidak lupa pula
penulis sampaikan ucapan terima kasih kepada mba Dewi, kak Falin, kak Indra,
dan segenap staf di Laboratorium Biologi Molekuler, Balai Besar Penelitian dan
Pengembangan Bioteknologi dan Sumber Daya Genetik Pertanian atas peran dan
kerjasamanya yang telah banyak membantu dalam menyelesaikan penelitian ini.
Penulis juga mengucapkan terima kasih kepada rekan-rekan selama
penelitian Riris, Ihsan, Naso, Restu, Andi, mas Nazar atas motivasi dan saran
yang diberikan kepada penulis. Selain itu penulis ucapkan terimakasih kepada
teman-teman seperjuangan Biokimia 45, Kenyar, Silvi, Yoan, Titus, Devid, Edo,
dan juga sahabat-sahabat yang tidak dapat disebutkan namanya satu persatu atas
segala bantuan, masukan, kritik, dan motivasi yang diberikan.
Penulis menyadari bahwa karya ilmiah ini jauh dari kesempurnanaan. Untuk
itu penulis mengharapkan saran dan kritik yang membangun untuk perbaikan di
masa mendatang. Akhir kata penulis berharap agar karya ilmiah ini dapat
bermanfaat bagi semua pihak.
Bogor, Agustus 2012
RIWAYAT HIDUP
Penulis dilahirkan di Jakarta Timur pada tanggal 21 April 1990 dari ayah
bernama Freddy Latief dan ibu bernama Marni Lusia. Penulis merupakan anak
keempat dari empat bersaudara.
Pendidikan penulis dimulai dari TK At-Taqwa dan Sekolah Dasar di SD
negeri Harapan Jaya XIII kemudian melanjutkan pendidikan ke jenjang Sekolah
Menengah Pertama di SMP Negeri 5 Bekasi. Tahun 2008 penulis menyelesaikan
pendidikan Sekolah Menengah Atas di SMA Negeri 4 Bekasi dan pada tahun
yang sama lolos seleksi masuk dan melanjutkan studi di Institut Pertanian Bogor
melalui jalur Undangan Seleksi Masuk IPB (USMI). Di IPB penulis mengambil
mayor Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengikuti perkuliahan penulis pernah menjadi asisten praktikum
mata kuliah Biokimia Umum untuk mahasiswa S1 Departemen Biologi tahun
ajaran 2011/2012. Penulis juga aktif dalam kegiatan organisasi kemahasiswaan di
IPB dan organisasi mahasiswa daerah, diantaranya penulis pernah aktif sebagai
staf keilmuan Bioanalisis
Community Research and Educatioan of Biochemistry
periode 2010/2011, dan staf Himpunan Keluarga Mahasiswa Bekasi di Bogor
selama dua periode tahun 2009-2011.
Penulis juga pernah aktif dalam beberapa kepanitiaan seperti panitia
Green
Festival IPB 2009, Masa perkenalan Kampus Mahasiswa Biokimia tahun 2009,
Pesta Sains Nasional 2009 dan 2010, Biokimia Expo 2010, Seminar kesehatan
2011, dan beberapa kepanitiaan lainnya. Penulis melakukan Praktik Lapang di
Laboratorium Mikrobiologi PT. Frisian Flag Indonesia, Jalan Raya Bogor Km 5,
Pasar Rebo, Jakarta Timur dengan judul Analisis Cemaran Mikroba pada Susu
Bubuk dengan Metode Enumerasi dan Deteksi. Penulis dalam bidang karya ilmiah
pernah mendapat hibah dana bersaing dari Direktorat Jenderal Pendidikan Tinggi
(DIKTI) dalam Pekan Kreativitas Mahasiswa (PKM) untuk kategori Bidang
Gagasan tertulis pada tahun 2009 dan Bidang Penelitian pada tahun 2010.
DAFTAR ISI
Halaman
DAFTAR TABEL ………...
x
DAFTAR GAMBAR ………...
xi
DAFTAR LAMPIRAN ………...
xii
PENDAHULUAN ………...
1
TINJAUAN PUSTAKA ………..
2
Tanaman Padi………
2
Isolasi DNA………...
3
Gen Os-GS3………...
3
Polymerase Chain Reaction (PCR)………...
4
Hibridisasi Southern………..
4
Elektroforesis Gel Agarosa………
5
BAHAN DAN METODE ………...……….
6
Bahan dan Alat………..
6
Metode Penelitian …...…..………...………..
6
HASIL DAN PEMBAHASAN ...……….……...
10
DNA Hasil Isolasi Menggunakan Metode Cepat, Miniprep, dan Skala
Besar………..
10
DNA Hasil
Polymerase Chain Reaction Menggunakan Metode
Cepat, Miniprep, dan Skala Besar……….
11
Karakterisasi Molekuler Berdasarkan Hibridisasi Southern ………
12
Karakterisasi Molekuler dengan Fenotipe Tanaman Padi Transgenik
Kultivar Taipei 309 ………
14
SIMPULAN DAN SARAN ……….
15
Simpulan ………...
15
Saran ……….
15
DAFTAR PUSTAKA ………..
15
DAFTAR TABEL
Halaman
1 Perbedaan morfologi dan fisiologi tanaman padi subspecies Indica,
Japonica, dan Javanica………. ………
3
2 Hasil pengukuran konsentrasi dan kemurnian DNA daun padi …………..
11
3 Hasil analisis dan pola integrasi gen sisipan Os-GS3) pada generasi
pertama (T0) pada kultivar Taipe 309 menggunakan primer hpt untuk
PCR dan pelacak hpt untuk hibridisasi Southern ………
13
4 Jumlah salinan gen menggunakan DNA yang diisolasi metode skala besar
dengan fenotipe tanaman padi………..
14
DAFTAR GAMBAR
Halaman
1 Perbedaan butir padi Indica dan Japonica ………....
3
2 Proses umum PCR ………..………..
4
3 Susunan perangkat blotting Membran ………
10
4 Elektroforegram hasil PCR menggunakan primer Hpt………. 11
5 Hasil hibridisasi Southern………. 13
DAFTAR LAMPIRAN
Halaman
1
Tahapan penelitian ………...…...
19
2
DNA genom yang telah di potong enzim BamHI ………. 20
3
DNA pelacak yang telah diberi label DIG-11-dUTP ………
20
4
DNA Genom setelah proses transfer ke membran ….………... 20
5 Tabel jumlah salinan gen dengan fenotipe tanaman padi………..……...
21
6 Tabel jumlah salinan gen dengan fenotipe tanaman padi………...
22
1
PENDAHULUAN
Tanaman padi (Oryza sativa L.) merupakan penghasil beras sehingga menjadi salah satu komoditas pangan utama dan saat ini hingga beberapa tahun mendatang, beras tetap menjadi sumber utama gizi dan energi bagi lebih dari 90% penduduk Indonesia (BPS 2011). Oleh karena itu, produksi beras nasional harus ditingkatkan untuk memenuhi kebutuhan pangan nasional. Beberapa langkah untuk meningkatkan produksi pangan dapat diusahakan melalui intensifikasi, ekstensifikasi, dan diversifikasi (Yuniati 2004). Salah satu cara intensifikasi pertanian dapat dilakukan melalui perbaikan genetika tanaman dengan pendekatan bioteknologi atau teknologi molekuler untuk meningkatkan produktivitas tanaman padi. Bioteknologi atau teknologi molekuler memiliki peranan penting dalam menghasilkan kultivar dengan sifat unggul baru (Soedarini & Patricia 2006). Beberapa varietas baru dapat direkayasa melalui transfer gen dari organisme lain ke dalam tanaman padi, sehingga dihasilkan padi transgenik yang memiliki sifat unggul.
Ukuran butir merupakan penentu utama dari bobot biji. Dalam aplikasi pemuliaan, ukuran butir biasanya dievaluasi oleh bobot biji, yang berhubungan positif dengan beberapa karakter termasuk panjang, lebar, dan ketebalan butiran biji. Ukuran butir juga merupakan sifat kualitas yang sangat penting dalam beras. Dengan demikian, pemahaman dasar genetik dan molekuler ukuran biji sangat penting bagi program perbaikan beras. Salah satu usaha yang dilakukan dalam penelitian guna meningkatkan produksi padi adalah melalui perakitan tanaman transgenik yang disisipi gen antisense Os-GS3 (Oryza sativa Grain Size 3) (Jiang et al. 2005; Fan et al. 2006).
Gen merupakan salah satu komponen penting untuk riset biologi molekuler dan pengembangan bioteknologi. Usaha untuk memahami ataupun memodifikasi berbagai proses biologi pada tingkat molekuler, memerlukan tersedianya gen-gen yang terlibat di dalam proses tersebut termasuk informasi yang terkait dengan gen-gen tersebut. Identifikasi dan karakterisasi gen seringkali diperlukan dalam berbagai percobaan molekuler antara lain isolasi ataupun mempelajari ekspresinya (Santoso 2001).
Identifikasi gen dapat dilakukan antara lain dengan analisis PCR untuk melihat ada atau tidaknya gen dan hibridisasi Southern
untuk mengetahui jumlah salinan transgen pada tanaman transgenik. Dalam analisis gen menggunakan metode hibridisasi Southern dibutuhkan DNA dalam jumlah banyak dan murni. PCR membutuhkan DNA dalam jumlah sedikit dan tidak perlu murni, metode ini dapat dilakukan lebih cepat dibandingkan dengan hibridisasi Southern namun lebih mudah terkontaminasi oleh DNA lain (Padmadi 2009).
Materi yang digunakan dalam penelitian ini adalah padi varietas T-309. Padi yang telah disisipi gen Os-GS3 ditanam dalam tanah dan dianalisis keberadaan gennya melalui PCR serta dikarakterisasi molekulernya melalui hibridisasi Southern. Metode isolasi DNA yang digunakan dalam penelitian ini adalah metode skala cepat, metode skala miniprep, dan metode skala besar. Permasalahan yang dihadapi ketika isolasi DNA antara lain jumlah sumber daya dan efisiensi waktu pengerjaan sampel. Metode isolasi DNA skala miniprep memerlukan sekitar 200 mg jaringan daun dan dalam 1 hari (7 jam) seorang analis dapat mengerjakan maksimal 24 sampel karena proses menghancurkan daun dibantu dengan menggunakan mesin (tissue lyser), sedangkan metode skala besar memerlukan sekitar 2 g jaringan daun dan dalam 1 hari (7 jam) seorang analis dapat mengerjakan 8 sampel karena proses menghancurkan daun harus dikerjakan secara manual menggunakan mortar. Pada situasi tertentu sejumlah besar tanaman transgenik harus dianalisis PCR dalam waktu singkat, sehingga perlu eksplorasi metode isolasi DNA cepat. Demikian juga perlu dieksplorasi kemungkinan penggunaan DNA yang diisolasi dengan skala miniprep dalam analisis hibridisasi Southern sehingga dapat dikerjakan ketika tanaman masih dalam fase bibit untuk menghemat tempat di rumah kaca. Dibutuhkan juga analisis lanjutan untuk mengetahui jumlah salinan gen antisense Os -GS3 yang efektif untuk menaikkan ukuran biji.
Penelitian ini bertujuan melakukan isolasi DNA, perbanyakan DNA, hibridisasi Southern, dan karakterisasi padi transgenik. Isolasi DNA dilakukan dengan 3 metode, yaitu metode skala cepat, miniprep, dan besar. Perbanyakan DNA dilakukan dengan PCR menggunakan DNA yang diisolasi dengan metode skala cepat, miniprep, dan skala besar. Analisis lebih lanjut dengan hibridisasi Southern dari sampel DNA yang diisolasi dengan metode skala besar dan miniprep. Karakterisasi padi transgenik diamati melalui
2
salinan transgen antisense Os-GS3 dengan keefektifan dalam menaikkan bobot biji. Hipotesis yang dikemukakan dalam penelitian ini yaitu hasil isolasi DNA dan analisis PCR dalam mengidentifikasi tanaman padi yang positif transgenik diharapkan sama menggunakan sampel DNA yang diisolasi dengan metode cepat, miniprep, dan skala besar, jumlah salinan transgen yang tersisipkan di dalam genom tanaman padi transgenik pada hibridisasi Southern menggunakan DNA yang diisolasi dengan metode miniprep sama dengan metode skala besar, semakin besar jumlah transgen
antisense Os-GS3 maka akan semakin efektif menaikkan bobot biji. Penelitian ini diharapkan dapat menganalisis metode isolasi DNA yang efisien dari segi waktu penyelesaian dan sumberdaya yang digunakan untuk PCR dan hibridisasi Southern dari tanaman padi transgenik. Selain itu, mengidentifikasi jumlah salinan transgen
antisense Os-GS3 untuk menaikkan ukuran biji.
TINJAUAN PUSTAKA
Tanaman Padi
Tanaman padi (Oryza sativa L.) diklasifikasikan dalam divisi Spermathopyta, subdivisi Angiospermae, kelas Monocotyledone, bangsa Gramineae (Poales), genus Oryza, dan jenis Oryza sativa
L (Remelia 2008). Secara umum padi dapat tumbuh di daerah tropis/subtropis dengan curah hujan yang baik adalah 200 mm/bulan atau 1500-2000 mm/tahun. Di dataran rendah padi tumbuh pada ketinggian 0-650 m di atas permukaan laut dengan temperatur 22-27 sedangkan di dataran tinggi 650-1500 m di atas permukaan laut dengan temperatur 19-23 (Padmadi 2009).
Padi merupakan tanaman semusim yang berumpun kuat dengan tinggi tanaman 0.5-2 m, akar padi adalah akar serabut yang efektif dalam penyerapan hara, tetapi peka terhadap kekeringan. Akar padi terkonsentrasi pada kedalaman antara 10-20 cm. Padi berumur pendek yaitu kurang dari satu tahun dan satu kali berproduksi, helai daun berbentuk garis, sebagian besar bertepi kasar dan panjangnya 15-80 cm, serta memiliki malai dengan panjang 14-40 cm yang tumbuh ke atas dan ujungnya menggantung. Malai padi berupa bulir yang beraneka ragam, kadang berjarum pendek atau panjang, licin atau kasar berwarna hijau atau coklat. Bulir yang masak
akan menghasilkan buah yang kaya akan pati amilosa dan amilopektin (Departemen Pertanian Badan Penelitian dan Pengembangan Pertanian 2003; Remelia 2008).
Pertumbuhan padi dari pembibitan hingga panen dibagi menjadi 3 fase yaitu vegetatif, reproduktif, dan pematangan. Fase vegetatif yaitu awal pertumbuhan sampai pembentukan malai, fase reproduktif yaitu pembentukkan malai sampai pembungaan, dan fase pematangan yaitu pembungaan sampai gabah matang. Organ tanaman padi secara keseluruhan dibedakan menjadi vegetatif meliputi akar, batang, dan daun sedangkan organ generatif meliputi malai, bunga, dan gabah (Banasiak 2010).
Tanaman padi yang ditanam di Asia dikelompokkan menjadi 3 subspesies berdasarkan pada perbedaan morfologi, fisiologi, biokimia dan molekuler, yaitu Oryza sativa Indica, Oryza sativa Japonica dan
Oryza sativa Javanica. Ketiga jenis subspesies tersebut biasanya memiliki hubungan dari perbedaan pada habitat tumbuhnya dan produk yang dihasilkan (Purmaningsih 2006). Padi kultivar Taipei 309 (T-309) adalah salah satu varietas padi yang termasuk dalam subspesies Japonica. Berdasarkan pada karakteristik morfologi dan fisiologinya, padi dari subspesies Japonica memiliki daun yang berwarna hijau pekat dan ukurannya lebih sempit, butir padi pendek dan membulat dibandingkan dengan butir padi Indica yang lebih panjang dan ramping (Gambar 1), pada lemma dan palea memiliki bulu yang panjang dan struktur butir yang padat, serta kadar amilum sekitar 10—20% (Smith & Dilday 2002). Perbedaan morfologi dan fisiologi antara tanaman padi yang berasal dari subspesies Indica, Japonica dan Javanica dapat dilihat pada Tabel 1.
Tanaman padi kultivar T-309 sering digunakan sebagai padi model penelitian bagi tanaman monokotil dan mudah ditransformasi. Dalam bidang kultur in vitro, tanaman padi kultivar T-309 memiliki beberapa kelebihan dibandingkan dengan varietas lain dari subspesies japonica. Menurut Purnamaningsih (2006), padi T-309 memiliki respon yang lebih mudah dan responsif dalam induksi kalus. Padi T-309 juga memiliki regenerasi kalus yang lebih cepat, hal tersebut dikarenakan kandungan poliamin pada padi Japonica lebih tinggi daripada Indica. Poliamin tersebut akan berperan dalam proses pembelahan, pemanjangan dan proliferasi sel. Semakin tinggi kandungan poliamin maka
3
Gambar 1 Perbedaan butir padi Indica dan Japonica (Sumber IRRI 2010) Tabel 1 Perbedaan morfologi dan fisiologi
tanaman padi subspesies Indica, Japonica, dan Javanica (Sumber Smith & Dilday 2002)
Karakteristik Indica Japonica Javanica
Daun Lebar hingga sempit Sempit Lebar dan kaku Warna daun Hijau
muda
Hijau tua Hijau muda Butir padi Panjang
hingga pendek, ramping dan sedikit rata Pendek dan membulat Panjang, lebar, dan tebal
Jaringan Lunak Keras Keras Kadar
amilum
23-31% 10-20% 20-25%
semakin responsif apabila ditumbuhkan secara in vitro.
Isolasi DNA
Sel makhluk hidup memiliki dinding sel pada bagian luarnya, sehingga untuk mengeluarkan materi DNA dari inti, dinding sel harus dipecah terlebih dahulu dengan cara mekanik ataupun enzimatik yang biasa disebut sebagai proses isolasi DNA. DNA merupakan bahan penyusun gen dan makromolekul beruntai ganda berbentuk heliks yang berfungsi sebagai pewarisan sifat yang menyimpan beragam materi genetik. Tiap molekul DNA terdiri atas dua rantai panjang yang masing-masing tersusun dari empat jenis penyusun kimiawi yang disebut nukleotida (Campbell et al.2002).
Masing-masing nukleotida itu sendiri terdiri atas tiga bagian, yaitu suatu molekul organik yang disebut basa nitrogen, suatu pentose (gula berkarbon lima), dan gugus
fosfat. Terdapat dua keluarga basa nitrogen, yaitu basa purin dan pirimidin (Campbell et al. 2002). Basa purin meliputi adenin dan guanin, sedangkan basa pirimidin meliputi timin dan sitosin (Manz et al. 2004). Masing-masing basa tersebut tersusun atas basa nitrogen, molekul gula, dan fosfat (Campbell
et al. 2002).
Isolasi DNA memiliki beberapa tahapan, yaitu: (1) Isolasi sel; (2) Lisis dinding dan membran sel; (3) Ekstraksi dalam larutan; (4) Pemurnian; dan (5) Pemekatan. Prinsip-prinsip dalam melakukan isolasi DNA ada 2, yaitu sentrifugasi dan pemekatan (Sambrook & Russell 2001). Isolasi DNA/RNA merupakan langkah awal yang harus dikerjakan dalam karakterisasi molekuler tanaman padi transgenik sebelum melangkah ke proses selanjutnya. Prinsip dasar isolasi total DNA/RNA dari jaringan adalah dengan memecah dan mengekstraksi jaringan tersebut sehingga akan terbentuk ekstrak sel yang terdiri atas sel-sel jaringan, DNA, dan RNA. Kemudian ekstrak sel dipurifikasi sehingga dihasilkan pelet sel yang mengandung DNA/RNA total (Faatih 2009).
Gen Os-GS3
Definisi gen secara umum adalah unit fisik dan fungsional dari pewarisan sifat yang mengandung informasi untuk sintesis protein atau keseluruhan sekuens asam nukleat yang dapat ditranskripsikan menjadi RNA fungsional dan protein, pada waktu dan tempat yang tepat selama pertumbuhan dan perkembangan organisme. Gen berperan dalam proses ekspresi gen (Qu et al. 2008). Ekspresi gen dipengaruhi oleh lingkungan internal dan eksternal seperti perkembangan fisik atau perilaku dari organisme itu. Gen tersusun atas urutan basa nukleotida, yang terdiri dari daerah yang mengkode suatu informasi genetik (ekson), daerah yang tidak mengkode informasi genetik (intron), dan bagian yang mengatur ekspresi gen yaitu sekuens pengontrol ekspresi gen (Campbell et al. 2002; Nelson & Cox 2008).
Gen antisense Os-GS3 (Oryza sativa- Grain Size 3) merupakan gen untuk meningkatkan produktivitas dan salah satunya adalah pengendali ukuran butir gabah. Ukuran butir merupakan faktor penentu utama dari bobot biji. Selain itu, jumlah malai per tanaman dan jumlah butir padi per malai juga dapat digunakan sebagai faktor penentu utama dari ukuran butir. Dalam aplikasi pemuliaan, ukuran butir biasanya dievaluasi oleh bobot
4
biji, yang berkorelasi positif dengan beberapa karakter termasuk panjang biji padi, lebar biji padi, dan ketebalan butiran biji padi (Jiang et al. 2005; Fan et al. 2006).
Ukuran butir juga merupakan faktor yang penting terhadap kualitas pada padi. Meskipun terdapat ukuran karakteristik gabah beragam, namun mayoritas konsumen di China, Amerika Serikat, dan sebagian besar negara Asia lebih menyukai padi yang ramping dan panjang. Sebagai contoh, rasio panjang atau lebar 2.8 cm merupakan standar kualitas nasional padi indica di Cina. Dengan demikian, pemahaman dasar genetik dan molekuler dari ukuran butir padi sangat penting bagi program peningkatan beras (Jiang et al. 2005; Fan et al. 2006).
Polymerase Chain Reaction (PCR)
Polymerase chain reaction adalah teknik cepat untuk mengamplifikasi fragmen DNA spesifik secara in vitro dengan menggunakan sepasang primer untai tunggal pendek (primer reverse dan forward). Sejumlah kecil fragmen DNA yang diinginkan dapat di amplifikasi secara berulang sampai jutaan kali dalam beberapa jam menggunakan teknik ini.
Polymerase chain reaction merupakan metode yang sensitif, selektif, dan cepat dalam menggandakan DNA target yang diinginkan (Murray et al. 2003), sehingga dari satu pasag molekul DNA dapat diperbanyak menjadi jutaan kali lipat setelah 30-40 siklus PCR (Campbell et al. 2002). Komponen-komponen yang dibutuhkan untuk PCR yaitu fragmen DNA yang akan diamplifikasi (DNA cetakan), sepasang primer oligonukleotida, enzim DNA
polymerase yang tahan panas, empat macam nukleotida (dATP, dGTP, dCTP, dan dTTP), serta bufer reaksi yang mengandung MgCl2. Alat ini mampu secara cepat mengubah temperatur yang dibutuhkan untuk siklus berulang (Berg et al. 2007; Nelson & Cox 2008).
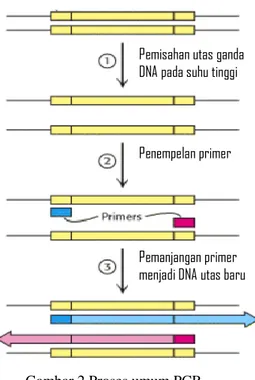
Siklus PCR dibagi menjadi 3 tahap yaitu pemisahan utas DNA pada suhu tinggi (denaturasi), penempelan primer, dan pemanjangan primer menjadi utas baru DNA oleh enzim DNA polimerase (Gambar 2). Tiap-tiap tahapan memerlukan suhu yang berbeda. Tahap denaturasi merupakan tahap awal reaksi yang berlangsung pada suhu tinggi, yaitu 94oC hingga 96 oC, dilakukan sampai 5 menit untuk memastikan semua utas DNA terpisah. Tahap denaturasi bertujuan untuk memisahkan utas ganda DNA menjadi utas tunggal sebagai DNA cetakan dengan memutuskan ikatan hidrogen antar pasangan
basa. Semakin panjang untaian rantai DNA, semakin lama waktu yang diperlukan untuk tahap denaturasi (Berg et al. 2007; Sambrook & Russell 2001).
Tahap kedua adalah penempelan primer atau annealing pada suhu sekitar 42 oC hingga 65 oC. Suhu penempelan ini bersifat spesifik yang merupakan rata-rata dari nilai Tm yang dimiliki masing-masing primer, yaitu forward
(5’-end) dan reverse (3’-end). Perbanyakan fragmen DNA dilakukan secara selektif dan spesifik oleh sepasang oligonukleotida yang disebut primer. Primer merupakan sekuen DNA pendek dengan frekuensi 15 hingga 25 panjang basa dan berutas tunggal. Primer menempel pada bagian DNA cetakan yang memiliki urutan basa komplementer dengan urutan basa primer. Tahap ini di dalam replikasi sel berfungsi sebagai inisiasi sintesis DNA oleh primase untuk membentuk RNA primer pada situs ori (Sambrook & Russell 2001).
Tahap ketiga adalah perpanjangan primer yang bertujuan memberikan kondisi optimum bagi kerja enzim Taq polimerase dalam memanjangkan primer guna membentuk utas DNA baru (Clark & Pazdemik 2009; Handoyo 2001). Enzim polimerase yang digunakan untuk mengaatalisis penempelan dua buah primer pada proses PCR salah satu jenisnya adalah Taq polimerase. Taq DNA polimerase yang diisolasi dari bakteri Thermus aqutiqus
(Taq) dikembangkan pada tahun 1988. Taq
Gambar 2 Proses umum PCR (Berg et al. 2007)
Pemisahan utas ganda DNA pada suhu tinggi
Penempelan primer
Pemanjangan primer menjadi DNA utas baru
5
polimerase memiliki stabilitas termal yang tinggi atau tahan terhadap suhu mendidih 100 oC, aktivitas maksimalnya berlangsung pada suhu 92-95oC (Sambrook & Russell 2001).
Hibridisasi Southern
Hibridisasi merupakan proses identifikasi gen-gen hasil analisis DNA dengan menggunakan enzim restriksi yang sesuai dan diidentifikasi dengan fragmen gen target yang spesifik yang telah diberi label dengan radioisotop seperti 32P, fosfat radioaktif atau dengan substansi nonradioisotop. Substansi nonradiosotop yang sering digunakan sebagai label adalah biotin dan digoksigenin (DIG). Proses hibridisasi dan visualisasi diawali dengan transfer DNA dari gel agarosa ke bahan padat seperti nilon berpori atau membran nitroselulosa. Transfer DNA disebut dengan ‘Southern blotting’, mengacu pada
penemu teknik tersebut yaitu E.M. Southern (1975). Southern transfer dan hibridisasi DNA digunakan untuk mempelajari bagaimana peran gen dalam genom dengan pemetaan titik-titik restriksi dan segmen DNA genomik (Berg et al. 2007).
Southern blot telah lama dikenal sebagai salah satu landasan analisis DNA. Transfer Southern dan teknik hibridisasi yang berkaitan telah dimanfaatkan untuk memperoleh informasi tentang organisasi fisik sekuen tunggal dan ganda pada kompleks genom, mempercepat suksesnya percobaan kloning pada gen eukariot (Primrose & Tywman 2006). Teknik Southern blot dapat digunakan dalam menganalisis keberadaan dan jumlah salinan dari gen hasil transformasi pada tanaman transgenik. Apabila jumlah salinan gen transforman terlalu banyak pada genom maka kemungkinan akan terjadi proses membloking pada gen tertentu (Tang et al.
2007), namun apabila gen terkopi antara 1-10 kemungkinan tidak akan menimbulkan gene silencing pada gen-gen lain (Santoso 2008).
Istilah Southern blot sekarang digunakan untuk mengambarkan transfer DNA dari gel ke membran, yang pada mulanya dilakukan secara kapiler ke membran nitroselulosa (Kumar et al. 2010). Transfer DNA dalam Southern blot dapat dilakukan pada beberapa tipe membran, bufer transfer dan metode transfer. Membran yang paling populer digunakan adalah membran nitroselulosa, nilon bermuatan positif dan nilon tidak bermuatan. Walaupun ketiga membran ini mempunyai sifat yang berbeda tetapi ketiganya dapat digunakan untuk berbagai macam aplikasi. Keuntungan utama membran
nilon adalah relatif kuat dan dapat digunakan untuk hibridisasi lebih dari 10X, sedangkan membran nitroselulosa mudah robek tetapi memberikan background yang lebih bersih pada beberapa proses hibridisasi (Sambrook & Russell 2001).
Transfer DNA dari gel ke membran dapat dilakukan dengan menggunakan metode transfer kapiler, elektroforesis dan vakum. Transfer kapiler seperti yang digunakan oleh Southern (1975), menggunakan aliran larutan garam untuk menarik DNA ke membran dengan adanya gaya kapiler yang ditimbulkan bahan penyerap seperti kertas dan tisu. Transfer kapiler hingga saat ini masih populer digunakan karena selain caranya cukup mudah, biaya relatif murah, dan efisien. Metode ini memerlukan waktu transfer yang relatif lama (12-16 jam). Transfer elektroforesis pada mulanya dikembangkan untuk transfer protein, dapat menekan waktu kerja tetapi tidak berkerja dengan baik pada konsentrasi bufer garam yang tinggi sehingga kurang cocok dengan bufer nitroselulosa. Transfer vakum mungkin merupakan cara yang paling ideal dengan waktu yang relatif cukup singkat (1-2 jam) mampu mentransfer DNA lebih efisien dan memberikan sinyal hibridisasi dua sampai tiga kali lipat dibandingkan transfer DNA kapiler (Coleman & Gregory 2006).
Elektroforesis Gel Agarosa
Elektroforesis gel agarosa digunakan untuk mengetahui keberadaan dan membedakan jenis asam nukleat yang didapat dari hasil ekstraksi serta digunakan untuk menganalisis produk hasil pemotongan dengan enzim retriksi. Prinsip dasar elektroforesis adalah berdasarkan laju perpindahan suatu molekul oleh gaya gerak listrik di dalam matriks gel. Laju perpindahan tersebut bergantung pada ukuran molekulnya, semakin kecil ukurannya maka molekul akan semakin cepat lajunya, begitu pula sebaliknya. Sampel molekul ditempatkan ke dalam sumur pada gel yang berada di dalam larutan penyangga dan dialirkan listrik pada tegangan tertentu. Molekul-molekul sampel akan bergerak di dalam matriks gel ke arah salah satu kutub listrik sesuai muatannya. Arah pergerakan untuk RNA dan DNA adalah menuju elektroda positif karena adanya muatan negatif pada rangka gula-fosfat yang dimilikinya (Berg et al. 2007).
Gel agarosa umumnya digunakan untuk menganalisis fragmen-fragmen berukuran besar (lebih dari 200 bp). Agarosa bersifat
6
tidak toksik, kompleks berupa bubuk yang terdiri atas campuran dua unit dasar galaktosa, agarosa, dan agaropektin (Sambrook & Russell 2001). Konsentrasi dari agarosa di dalam gel menentukan ukuran pori-pori rata-rata. Ukuran pori-porinya lebih besar dari pori-pori pada gel poliakrilamid sehingga asam nukleat dan protein yang terlalu besar untuk dipisahkan oleh gel poliakrilamid dapat dipisahkan menggunakan gel ini. Gel agarosa mengandung gugus-gugus bermuatan terutama sulfat dan beberapa gugus karboksilat. Gugus-gugus ini berinteraksi dengan gugus-gugus bermuatan pada protein sehingga menimbulkan efek pertukaran ion (Manz et al. 2004).
Elektroforesis gel biasanya dilakukan untuk tujuan analisis, namun dapat digunakan sebagai teknik preparatif untuk memurnikan molekul sebelum digunakan untuk teknik lebih lanjut, seperti kloning. Ukuran DNA dapat ditentukan dengan menyertakan marka atau penanda yang digunakan pada proses elektroforesis. Setelah tahap elektroforesis selesai, dilakukan metode pewarnaan dan penghilangan warna. Metode pewarnaanpada DNA atau RNA merupakan pewarnaan gel agarosa yang dilakukan dengan menggunakan larutan etidium bromida selama 15 menit. Hal ini dilakukan dengan tujuan agar molekul sampel berpendar dalam sinar ultraviolet. Penghilangan warna dilakukan dengan cara gel dimasukkan ke dalam aquades selama 5 hingga 7 menit (Manz et al. 2004).
BAHAN DAN METODE
Bahan dan AlatBahan-bahan yang digunakan pada isolasi DNA terdiri atas daun padi T-309 tipe liar dan padi transgenik GS3 (GS (2), GS (5), T-GS (9), T-T-GS (12), T-T-GS (13)) natrium bisulfit, bufer ekstraksi (10 mL Tris-HCl pH 8 M, 10 mL EDTA0.5 M, 12.5 mL NaCl 4M, 67.5 mL akuades), nitrogen cair, 20% SDS, natrium asetat, kalium asetat, isopropanol dingin, etanol 70%, bufer TE (Tris-EDTA), RNAse, 0.5 M NaOH. Bahan-bahan yang digunakan pada tahap PCR terdiri atas bufer PCR, MgCl 25 mM, dNTP set 10 mM, primer
hpt Forward dan Reverse 10 mM, Taq DNA polymerase (5 U/ L), DNA hasil isolasi 10 ng/ L, dan akuades steril. Bahan-bahan yang digunakan untuk elektroforesis yaitu loading dye, bufer TAE 1x, agarosa, DNA hasil PCR, marker, etidium bromide 2 mg/L, dan akuades.
Bahan-bahan yang digunakan untuk hibridisasi southern adalah pelacak hpt, DIG-11-dUTP, anti-Digoxigenin, enzim restriksi
BamHI, kertas blotting, kertas Whatmann 3MM, membran nilon Hybond N+, Amersham Hyperfilm (GE Healthcare), dan substrat CDP-Star. Bahan lain yang digunakan adalah larutan developer dan larutan fixer, Tween 20, plasmid (100 pg/ L), LiCl, heksanukleotida, klenow enzim, reagen
blocking, larutan depurinasi (HCl dan akuades), larutan denaturasi (NaOH, NaCl, dan akuades), larutan netralisasi (Tris base, dan akuades), larutan 20x SSC (NaCl, Tri natrium sitrat dihidrat, dan akuades), larutan
washing 1 (20x SSC, 10% SDS, dan akuades), larutan washing 2 (20X SSC, 10% SDS, dan akuades), larutan bufer 1 (tris hidroksimetil, NaCl, dan akuades), larutan washing buffer
(larutan bufer 1 dan tween 20), larutan bufer 2 (larutan bufer 1 dan reagen blocking),larutan antibodi (larutan bufer 2 dan Anti-DIG AP), larutan bufer 3 (Tris base, NaCl, dan akuades), larutan stripping buffer (NaOH, 10% SDS, dan akuades), HCl 1N, dan HCl 0.1N.
Alat- alat yang digunakan dalam penelitian ini terdiri atas timbangan analitik, shaker,
tissue lyser, mortar, gunting, kertas tisu, pensil, sendok, bak, stopwatch, botol 250 mL, botol 500 mL, botol 1000 mL, gelas kimia 500 mL, gelas kimia 1000 mL, strirrer, gelas ukur 500 mL, labu Erlenmeyer 250 mL, oven,
microwave, vortex, gelas piala, tabung
eppendorf (0.2 mL, 1.5 mL, dan 2 mL),
grinding balls, pipet mikro, tip, mortar, penangas air, bak es, dan mesin sentrifus IEC-Centra 4B. Selain itu, digunakan pula elektroforesis Hoefer scientific instruments, UV cross linker, UV illuminator Chemidoc EQ Biorad, nanodrop Thermo scientific, mesin PCR Thermal Cycle Tetrad, kaset, dan sentrifus mikro.
Metode Penelitian
Isolasi DNA Padi Skala Besar (Modifikasi Dellaporta et al. 1983; Wang et al. 1993)
Daun tanaman padi tipe liar Taipei 309 (T-309), Taipei Grain Size-2 (T-GS (2)), T-GS (5), T-GS (9), T-GS (12), dan T-GS (13) ditimbang menggunakan timbangan analitik sebanyak 2 gram dimasukkan ke dalam mortar dan disiram dengan nitrogen cair, kemudian digerus hingga menjadi serbuk. Serbuk tersebut kemudian dimasukkan ke dalam tabung berukuran 50 mL dan diberi label
7
nama sampel untuk masing-masing tabung. Tabung yang telah berisi serbuk daun padi kemudian disimpan ke dalam bak yang berisi nitrogen cair. Setelah sampel disimpan, dibuat larutan 1 dengan komposisi 100 mL bufer ekstraksi yang dimasukkan ke dalam botol 250 mL yang selanjutnya dicampur dengan 0.38 gram natrium bisulfit. Sebelum digunakan, larutan 1 tersebut diinkubasi di dalam penangas air pada suhu 650C.
Sampel daun padi masing-masing tersebut ditambahkan 15 mL larutan 1 dan 1 mL SDS 20%, dikocok hingga homogen, dan diinkubasi pada suhu 650C selama 15 menit (setiap 5 menit dikocok) lalu diangkat. Setelah itu, ditambahkan sebanyak 5 mL kalium asetat 5 M, lalu tabung dibolak-balik hingga homogen dan diinkubasi di dalam bak es selama 20 menit. Suspensi tersebut selanjutnya disentrifugasi dengan kecepatan 4000 rpm selama 20 menit. Kemudian supernatan (cairan) di saring menggunakan kertas tisu Kimwipes dan dipindahkan ke dalam tabung 50 mL yang baru.
Isopropanol dingin ditambahkan sebanyak 10 mL ke dalam cairan dan diinkubasi ke dalam freezer dengan suhu (-200C) selama 30 menit. Kemudian sampel disentrifugasi pada kecepatan 4000 rpm selama 20 menit lalu cairan di buang. Kemudian sampel tersebut disentrifugasi lagi pada kecepatan 4000 rpm selama 2 menit dan sisa dari cairan di pipet menggunakan pipet mikro sehingga cairan tidak ada sama sekali di dalam tabung tersebut. Setelah itu, tabung sampel yang berisi pelet di keringkan ke dalam oven dengan suhu 65 selama 10 menit.
Pelet yang telah kering dilarutkan ke dalam 700 akuades dan 5 RNAse dan diinkubasi pada suhu 65 selama 30 menit dengan di kocok setiap 10 menit. Suspensi tersebut dipindahkan ke dalam tabung 1.5 mL dan disentrifugasi dengan kecepatan 10000 rpm selama 10 menit. Kemudian supernatan dipindahkan ke dalam tabung dengan ukuran 1.5 mL yang baru. Sebanyak 75 natrium asetat 3 M dan 500 isopropanol dingin ditambahkan dan dicampur serta diinkubasi pada 28 selama 5 menit. Sampel disentrifugasi dengan kecepatan 10000 selama 5 menit dan supernatan dibuang.
Pelet yang diperoleh dicuci dengan 500 µL etanol 70%. Campuran disentrifugasi kembali selama 2 menit pada kecepatan 10000 rpm dan cairan dibuang. Kemudian disentrifugasi kembali pada kecepatan 10000 selama 30 detik dan cairan dibuang dengan pipet. Pelet
yang diperoleh dikeringkan dalam oven selama 10 menit. Pelet yang telah kering dilarutkan dengan 150 µL larutan TE. Tahap selanjutnya adalah diukur kemurnian dan konsentrasinya dengan spektrofotometer.
Isolasi DNA Padi Miniprep (Modifikasi Dellaporta et al. 1983; Wang et al. 1993)
Daun tanaman padi tipe liar Taipei 309 (T-309), Taipei Grain Size-2 (T-GS (2)), T-GS (5), T-GS (9), T-GS (12), dan T-GS (13) ditimbang menggunakan timbangan analitik sebanyak 0.1 gram lalu daun padi tersebut dipotong kecil-kecil dan dimasukkan ke dalam tabung 1.5 mL serta diberi label untuk masing-masing sampel. Setelah itu dimasukkan satu grinding ball ke dalam tabung 1.5 mL yang sudah berisi potongan daun padi. Kemudian tabung yang sudah berisi potongan padi dan grinding ball
tersebut disusun di dalam rak tissue lyser dan disiram dengan nitrogen cair, kemudian sampel tersebut dihancurkan oleh alat tissue lyser tersebut hingga menjadi serbuk. Tabung yang telah berisi serbuk daun padi kemudian disimpan ke dalam bak yang berisi nitrogen cair. Setelah sampel disimpan, larutan 1 dibuat dengan komposisi 10 mL bufer ekstraksi yang dimasukkan ke dalam tabung 50 mL yang selanjutnya dicampur dengan 0.038 gram natrium bisulfit. Sebelum digunakan larutan 1 tersebut diinkubasi di dalam penangas air pada suhu 650C.
Dimasukkan larutan 1 sebanyak 1 mL ke dalam masing-masing tabung yang berisi serbuk sampel daun padi. Setelah itu, larutan 1 dipipet ke dalam tabung lalu ditambahkan 1000 L dan 68 L SDS 20% kemudian diinkubasi selama 15 menit dengan suhu 650C di dalam penangas air dan setiap 2 menit tabung dibolak-balikan agar tercampur dengan rata. Kemudian ditambahkan sebanyak 334 L kalium asetat 5 M, tabung dibolak-balik kembali hingga tercampur dengan rata, dan dan diinkubasi di dalam bak es selama 20 menit Tabung-tabung yang telah diinkubasi kemudian disentrifugasi selama 10 menit dengan kecepatan 12000 rpm, pindahkan cairan sebanyak 800 L dipindahkan ke dalam tabung 1.5 mL baru. Dipipet isopropanol dingin sebanyak 600 L ke dalam tabung yang berisi cairan dan tabung dibolak-balik hingga tercampur rata lalu diinkubasi selama 30 menit ke dalam freezer (-200C).
Setelah diinkubasi selama 30 menit lalu disentrifugasi selama 10 menit dengan
8
kecepatan 12000 rpm kemudian cairan dibuang. Untuk memastikan bahwa cairan sudah tidak ada maka disentrifugasi 30 detik dengan kecepatan 12000 rpm lalu sisa dari cairan tersebut dipipet menggunakan pipet mikro. Kemudian tabung yang berisi pelet dioven selama 10 menit dengan suhu 65 . Tabung yang berisi pelet tersebut ditambahkan 200 L ddH2O dan 2 L RNAse lalu diinkubasi ke dalam penangas air dengan suhu 65 selama 30 menit dan tabung dibolak-balik setiap 10 menit. Tahap selanjutnya, tabung tersebut disentrifugasi selama 10 menit dengan kecepatan 12000 rpm. Cairan dipindahkan ke dalam tabung baru dengan ukuran 1.5 mL. Ditambahkan natrium asetat 3 M sebanyak 25 dan 150 isopropanol dingin lalu dikocok dan diinkubasi ke dalam freezer -20 selama 30 menit. Sampel disentrifugasi dengan kecepatan 12000 selama 5 menit dan supernatan dibuang.
Pelet yang diperoleh dicuci dengan 500 µL etanol 70%. Campuran disentrifugasi kembali selama 10 menit pada kecepatan 12000 rpm dan cairan dibuang. Kemudian disentrifugasi kembali pada kecepatan 12000 selama 30 detik. Pelet yang diperoleh dikeringkan dalam oven selama 10 menit. Pelet yang telah kering dilarutkan dengan 30 µL larutan TE. Tahap selanjutnya adalah diukur kemurnian dan konsentrasinya dengan spektrofotometer.
Isolasi DNA Padi Skala Cepat (Modifikasi Collard et al. 2007)
Daun tanaman padi tipe liar Taipei 309 (T-309), Taipei Grain Size-2 (T-GS (2)), T-GS (5), T-GS (9), T-GS (12), dan T-GS (13) ditimbang menggunakan timbangan analitik sebanyak 0.1 gram lalu daun padi tersebut dipotong kecil-kecil dan dimasukkan ke dalam tabung 1.5 mL serta diberi label untuk masing-masing sampel. Setelah itu dimasukkan satu grinding ball ke dalam tabung 1.5 mL yang sudah berisi potongan daun padi. Kemudian tabung yang sudah berisi potongan padi dan grinding ball
tersebut disusun di dalam rak tissue lyser dan disiram dengan nitrogen cair, kemudian sampel tersebut dihacurkan oleh alat tissue lyser tersebut hingga menjadi serbuk. Tabung yang telah berisi serbuk daun padi kemudian disimpan ke dalam bak yang berisi es.
Dimasukkan 0.5 M NaOH sebanyak 50 L dengan menggunakan pipet mikro ke dalam tabung yang berisi sampel tetapi perlakuan
tersebut tetap dilakukan di dalam bak es. Kemudian tabung-tabung sampel yang berada di dalam bak es tersebut digoyang menggunakan shaker selama 10 menit. Setelah itu, ditambahkan 0.1 M Tris-HCl pH 8.0 sebanyak 400 L ke dalam tabung sampel dan dilanjutkan dengan digoyang-goyangkan kembali menggunakan shaker agar semua larutan tercampur dengan rata. Sampel tersebut disentrifugasi selama 10 menit dengan kecepatan 12000 rpm dan sebanyak 250 L supernatan di pipet ke dalam tabung 1.5 mL baru. Supernatan disentrifugasi kembali selama 5 menit dengan kecepatan 12000 rpm. Tahap selanjutnya adalah diukur kemurnian dan konsentrasinya dengan spektrofotometer.
Analisis Kuantitatif DNA Genomik dengan Nanodrop (Thermo Fisher Scientific 2009)
Sebanyak 2 µL larutan TE dimasukkan ke dalam lubang ukur. Setelah itu, tutup nanodrop dan tekan tombol read blank pada komputer. Kertas tissue digunakan untuk membersihkan sisa buffer TE. Kemudian sampel DNA dimasukkan sebanyak 2 µL ke dalam lubang ukur, kemudian pilih menu read sample. Hasil pengukuran berupa nilai kemurnian sampel akan muncul dalam satuan konsentrasi ng/ µL. DNA yang murni dapat dilihat pada 260/Å280.
Amplifikasi DNA dengan PCR (Trijatmiko et al. 2011)
Hasil isolasi DNA tanaman T-309 yang telah disamakan konsentrasinya selanjutnya diamplifikasi dengan mesin PCR. Amplifikasi DNA padi mula-mula disiapkan sebanyak 20 buah tabung mikro. Setiap tabung diisi dengan reaksi amplifikasi yang terdiri 2 µL 10x bufer PCR, 1.2 µL MgCl2 25 mM, 0.4 µL dNTP mix 10 mM, 1 µL primer forward hpt 10 µM, 1 µL primer reverse hpt 10 µM, 0.16 µL Taq
DNA polymerase 5 U/µL, 5 µL DNA 10 ng/ µL, dan 9.24 µL ddH2O. Sekuen primer yang digunakan adalah Forward 5” GAT GCC TCC GCT CGA AGT AGC G 3” dan Reverse 5” GCA TCT CCC GCC GTG CAC 3”. Total volume reaksi PCR adalah 20 µL. Kemudian dijalankan program pada mesin PCR. Profil PCR yang digunakan adalah pre-denaturasi 94˚C selama 5 menit, denaturasi 94˚C selama 30 detik, penempelan primer 65˚C selama 30 detik, dan pemanjangan primer 72˚C selama 30 detik. Proses tersebut diulang sebanyak 30 siklus.
9
Analisis Kualitas dengan Elektroforesis Gel Agarosa (Sambrook & Russell 2001)
Disiapkan terlebih dahulu 1% gel agarosa dengan 1x bufer TAE pada cetakan. Sebanyak 1 g agarosa ditimbang dan dicampur dengan 100 mL TAE. Larutan tersebut dimasukkan ke dalam microwave selama 2 menit. Larutan yang sudah tercampur dimasukkan ke dalam cetakan agar yang sudah diberi cetakan sumur. Setelah gel agarosa memadat, gel dimasukkan ke dalam tangki elektroforesis yang berisi 1x bufer TAE. Sebanyak 10 µL produk PCR ditambahkan dengan 2 µL loading dye dan dicampur sempurna, kemudian dimasukkan ke dalam sumur gel. Disertakan 1 kb ladder sebagai marker untuk melihat ukuran DNA. Tahap selanjutnya sampel DNA dialiri arus dengan voltase 80 volt selama 35 menit. Gel agarosa diwarnai dengan larutan etidium bromida (20 mg/L) selama 10 menit, kemudian dihilangkan pewarnaannya dengan air selama 10 menit. Gel Pita-pita DNA selanjutnya ditampakkan dengan chemidoc gel system.
Hibridisasi Southern (Trijatmiko et al. 2011)
Analisis Integrasi gen Os-GS3 (Oryza sativa Grain Size3) di dalam tanaman padi transgenik. Hibridisasi Southern yang mengacu pada prosedur yang telah dilakukan oleh Trijatmiko et al. (2011) menggunakan pelabelan Digoxigenin -11-dUTP (DIG-11-dUTP). Kegiatan tersebut terdiri atas beberapa tahap, yaitu pemotongan DNA menggunakan enzim restriksi BamHI, pelabelan marker, transfer DNA yang sudah dipotong dengan enzim restriksi ke membran Hybond N+ (GE Healthcare), pelabelan pelacak hibridisasi membran dengan pelacak yang telah di label, dan deteksi sinyal dengan larutan CDP star.
Lima DNA sampel padi transgenik dan padi tipe liar generasi T0 dipotong menggunakan enzim restriksi BamHI. Pemotongan dilakukan dengan total volume 150 l pada masing-masing DNA. Sebanyak 33 L akuades dan 15 L larutan buffer 10x dimasukkan ke dalam tabung steril 1.5 mL, lalu dilanjutkan dengan ditambahkannya enzim restriksi BamHI (10 U/ L) sebanyak 2 L. Tahap selanjutnya dimasukkan DNA hasil isolasi sebanyak 100 L ke dalam tabung 1.5 mL. Tabung diinkubasi pada suhu 37 oC selama 16 jam. Ditambahkan sebanyak 0.1 x volume natrium asetat (3M) dan sebanyak 2.5
x volume etanol 96%. Kemudian tabung diinkubasi pada freezer (-20oC) selama 2 jam. Tabung disentrifugasi dengan kecepatan 12000 rpm selama 10 menit pada suhu 4 . Cairan dibuang kemudian ditambahkan etanol 70% sebanyak 500 L, lalu disentrifugasi kembali dengan kecepatan 12000 rpm selama 2 menit. Cairan kembali dibuang menggunakan pipet 20 L dan tabung dikeringkan selama 2 menit. Tahap selanjutnya, sebanyak 15 L 0.1x TE dan 3 L loading dye ditambahkan ke masing-masing tabung tersebut. Setelah itu, tabung diinkubasi pada suhu 65 oC selama 15 menit.
Pelabelan marker 1 Kb dilakukan dengan menyiapkan 10x dNTP mix didalam tabung 0.5 mL dengan komposisi 1 mM dATP sebanyak 2 L, 1 mM dCTP sebanyak 2 L, 1 mM dGTP sebanyak 2 L, 0.65 mM dTTP sebanyak 1.3 L, dan 0.35 mM DIG-11-dUTP sebanyak 7 L. Tahap selanjutnya, sebanyak 3 L 1 Kb, 10x heksanukleotida sebanyak 2 L, 10x dNTP mix sebayak 2 L, klenow enzim sebanyak 1 L, dan nucleid free water (NF) sebanyak 12 L dimasukkan ke dalam tabung 1.5 mL. Setelah itu, tabung yang berisi campuran larutan tersebut dipanaskan pada suhu 100 oC selama 10 menit lalu dimasukkan ke dalam es selama 2 menit, kemudian diinkubasi pada suhu 37 oC selama 3 jam. Ditambahkan EDTA 0.2 mol/L sebanyak 2 L dan 2.5 L LiCl 4 N serta etanol absolut sebanyak 75 L. Tahap berikutnya, tabung disimpan pada frezzer selama 1 jam dan disentrifugasi dengan kecepatan 12000 rpm selama 5 menit. Cairan dibuang lalu pelet dicuci dengan etanol 70%. Tahap terakhir, etanol 70% dibuang dan pelet dikeringkan menggunakan oven dengan suhu 50 oC atau dikering anginkan kemudian pelet dilarutkan pada bufer TE 1x sebanyak 50 L.
Proses transfer DNA tanaman padi transgenik yang dipotong dengan menggunkan enzim restriksi BamHI ke membran Hybond -N+ diawali dengan dibuat gel agarosa dengan konsentrasi 0.7% dalam 1x buffer TAE (Tris Acetat acid-EDTA) dan didiamkan hingga memadat. DNA yang telah di potong kemudian dimasukkan sebanyak 18 L dan dimasukkan sebanyak 5 L marker yang telah dilabel oleh DIG ke dalam masing-masing sumur selama 16 jam (semalam) pada tegangan 20 volt. Di hari berikutnya, tegangan diubah menjadi 15 volts dan ditunggu hingga 5 jam. Gel kemudian direndam dalam larutan
10
depurinasi selama 10 menit pada suhu ruang, kemudian dibilas dengan menggunakan akuades, diikuti dengan perendaman dalam larutan denturasi selama 2 x 15 menit pada suhu ruang, dan terakhir direndam dalam larutan netralisasi selama 2 x 15 menit pada suhu ruang. Kemudian gel direndam dengan larutan 20x SSC selama 10 menit pada suhu ruang. Semua proses dilakukan dengan menggoyang gel agarose di dalam larutan tersebut. Transfer DNA ke membran Hybond
–N+
dilakukan dengan proses kapiler menggunakan 20x bufer SSC selama 20 jam (Gambar 3). Setelah DNA berpindah ke membran, DNA pada membran difiksasi menggunakan UV selama 5 menit pada 302 nm.
Pelabelan pelacak gen dilakukan menggunakan teknik amplifikasi PCR. Reaksi PCR dengan total volume 50 L menggunakan primer forward dan reverse
masing-masing 1 L (5 mM), 5 L 10x buffer PCR, 3 L MgCl2 (25 mM), 0.5 L dATP (10 mM), 0.5 L dGTP (10 mM), 0.5 L dCTP (10 mM), 0.44 L dTTP (10 mM), enzim Taq polymerase (5 U/ L) sebanyak 0.4 L, dan 5 L pelacak Hpt (100 pg/ L). Profil suhu yang digunakan adalah predenaturasi 95 selama 2 menit, dilanjutkan dengan 30 siklus denaturasi pada suhu 95 selama 30 detik, penempelan primer pada suhu 60 selama 30 detik, dan pemanjangan pada suhu 72 selama 40 detik. Tahapan akhir PCR dilakukan pemanjangan akhir pada suhu 72 selama 7 menit. Untuk mengetahui keberhasilan pelabelan pelacak, sebanyak 5 L produk PCR labeling dengan Dig-11-dUTP berdampingan dengan produk PCR tanpa DIG-11-dUTP dengan teknik elektroforesis pada gel agarosa 1% (w/v) dan
Gambar 3 Susunan perangkat blotting
membran
hasilnya dianalisis dengan menggunakan CEMIDOC.
Membran yang mengandung DNA kemudian dihibridisasi dengan pelacak yang sudah terlabel. Proses ini dimulai dengan memanaskan larutan DIG Easy Hyb pada suhu 42 . Membran direndam dengan larutan DIG Easy Hyb sebanyak 50 L yang diletakkan dalam kotak plastik dan digoyang pada suhu
42 selama 3 jam. Setelah itu larutan DIG Easy Hyb dimasukkan ke dalam tabung 50 mL dan ditambahkan dengan larutan pelacak yang telah dilabel setelah itu campuran larutan tersebut dimasukkan kembali ke dalam kotak plastik dan digoyang kembali selama 16 jam pada suhu 42 .
Membran yang sudah dihibridisasi kemudian dicuci dengan larutan Wash 1 pada suhu kamar selama 2 x 5 menit, diikuti dengan pencucian dengan larutan Wash 2 pada suhu 68 selama 2 x 15 menit. Membran diseimbangkan dengan larutan washing buffer
selama 2 menit, diikuti dengan inkubasi membran dalam larutan bufer 2 selama 2 jam. Membran diinkubasi selama 30 menit dalam 40 mL larutan Anti-Digoxigenin-Alkalin Fosfataseatau Antibody conjugate (Anti-DIG)
(Roche).
Membran dipindahkan ke dalam kotak plastik baru dan untuk menghilangkan kelebihan antibody conjugate, membran diinkubasi selama 2 x 15 menit dalam
washing buffer. Membran direndam dengan larutan bufer 3 selama 3 menit (Lampiran 8). Untuk pendeteksian sinyal dilakukan penambahan substrat CDP-Star ke membran. Kemudian membran ditutup dengan plastik
wrap dan diletakkan ke dalam kaset. Film X-Ray diletakkan di atas membran dan didiamkan hingga 6 jam. Proses selanjutnya film X-Ray direndam dengan larutan
developer selama 45 detik, air 30 detik, dan larutan fixer selama 30 detik maka jejak dari cahaya yang dihasilkan bisa dilihat sebagai pita-pita pada film X-Ray.
Analisis fenotipe tanaman padi kultivar Taipei-309
Analisis fenotipe tanaman padi kultivar Taipei-309 dilakukan dengan cara mengamati tinggi tanaman padi dan jumlah anakan tanaman padi. Setelah itu dilakukan pemanenan dan diamati panjang malai, jumlah cabang malai dalam satu malai, jumlah gabah hampa dalam satu malai, jumlah gabah isi
11
dalam satu malai, dan berat 100 bulir padi isi dari setiap sampel (Jiang et al. 2005).
Tinggi tanaman padi dan panjang malai diamati menggunakan penggaris. Berat 100 bulir padi isi dari setiap sampel ditimbang menggunakan timbangan analitik. Tahap selanjutnya dilakukan analisis antara jumlah salinan gen dari masing-masing sampel padi baik tansgenik maupun tipe liar dengan hasil data fenotipe.
HASIL DAN PEMBAHASAN
DNA Hasil Isolasi Menggunakan Metode Cepat, Miniprep, dan Skala Besar
Isolasi DNA merupakan tahap yang penting dalam analisis gen dengan PCR. Isolasi DNA dilakukan melalui empat tahap yang meliputi pemanenan sampel padi, lisis, pemurnian atau penghilangan dari komponen selain DNA, dan pemekatan DNA (Sambrook & Russell 2001). Daun padi yang digunakan adalah daun yang masih muda (Ardiana 2009) dikarenakan teksturnya yang masih lunak dan sedikit mengandung serat sehingga mudah dalam proses penggerusan. Selain itu daun muda banyak mengandung DNA karena aktif dalam proses pembelahan dan pertumbuhan sel.
Metode isolasi yang digunakan pada penelitian ini adalah metode skala cepat, miniprep, dan skala besar. Masing-masing metode tersebut memiliki kelebihan dan kekurangan. Pada penelitian ini akan dipilih metode yang paling efisien dari segi waktu pengerjaan tetapi tetap memenuhi syarat untuk digunakan dalam analisis PCR. Kelebihan metode cepat dan miniprep adalah penggunaan sampel daun padi dalam jumlah sedikit (0.5-1 gram), sedangkan pada metode skala besar memerlukan sampel dalam jumlah banyak (2 gram). Kekurangan dari isolasi DNA miniprep adalah tahapan prosedur yang digunakan harus mengikuti 20 langkah sehingga seorang analis dapat mengerjakan maksimal 24 sampel dalam 1 hari (7 jam) dengan menggunakan mesin penggerus jaringan daun padi (tissue lyser). Demikian halnya pada tahapan isolasi DNA skala besar, seorang analis hanya mampu menyelesaikan 8 sampel karena proses pelembutan daun harus dikerjakan secara manual tanpa dibantu mesin penggerus (mortar). Pada tahapan isolasi DNA skala cepat hanya mengikuti 8 langkah sehingga seorang analis dapat mengerjakan 100 sampel dalam 1 hari (7 jam).
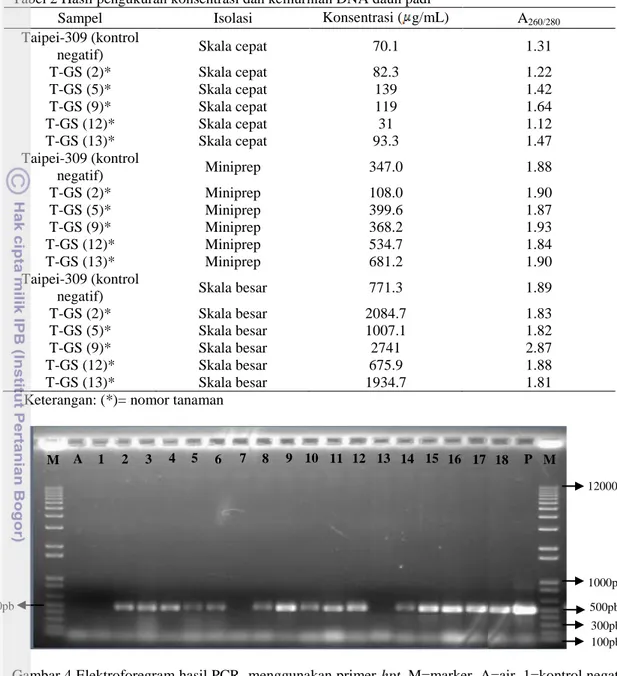
Parameter keberhasilan isolasi DNA dapat dilihat secara kuantitatif. Uji kuantitatif DNA
dilakukan melalui penentuan konsentrasi dan kemurnian DNA dengan menggunakan nanodrop. Metode isolasi skala miniprep dan skala besar menghasilkan kemurnian yang baik jika dibandingkan dengan metode skala cepat. Hal ini terlihat dari hasil nanodrop sampel DNA (Tabel 2). Kemurnian DNA padi menggunakan isolasi DNA miniprep dan skala besar yang diperoleh berkisar antara 1.81-1.93. Namun kemurnian DNA padi menggunakan isolasi DNA skala cepat berkisar antara 1.12-1.64 yang mengindikasikan masih terkandungnya protein, kemungkinan karena pada isolasi skala cepat tidak digunakan larutan untuk mengendapkan protein dan hanya menggunakan larutan NaOH sebagai bufer ekstraksinya.
Menurut Brown (2001) DNA yang murni mempunyai nilai absorbansi 260 dibagi dengan nilai absorbansi 280 ( 260/Å280) berkisar 1,8 hingga 2,0. Apabila nilainya kurang dari 1,8 maka sampel DNA masih mengandung kontaminan protein dan untuk menghilangkannya ditambahkan proteinase. Apabila nilainya lebih dari 2,0 maka sampel DNA masih mengandung kontaminan RNA, dan untuk menghilangkannya ditambahkan ribonuklease.
DNA Hasil Polymerase Chain Reaction Menggunakan Metode Cepat, Miniprep dan Skala Besar
Sampel DNA padi yang telah diisolasi dan di analisis kuantitatif selanjutnya dianalisis lebih lanjut untuk mengetahui secara tepat suatu tanaman mengandung gen target yang diintroduksikan ke dalam jaringan tanaman, dapat dilakukan analisis menggunakan PCR. Teknik PCR adalah suatu teknik untuk mengamplifikasi sekuen DNA tertentu dengan menggunakan primer spesifik secara in vitro
(Apriana 2008) . Sampel yang digunakan adalah 5 tanaman transgenik dan 1 tanaman tipe liar sebagai kontrol negatif. Analisis molekuler dengan PCR dilakukan menggunakan primer hpt. Suhu annealing yang digunakan adalah 65 . Plasmid pCAMBIA 1301 35S Os-GS3 digunakan sebagai kontrol positif .
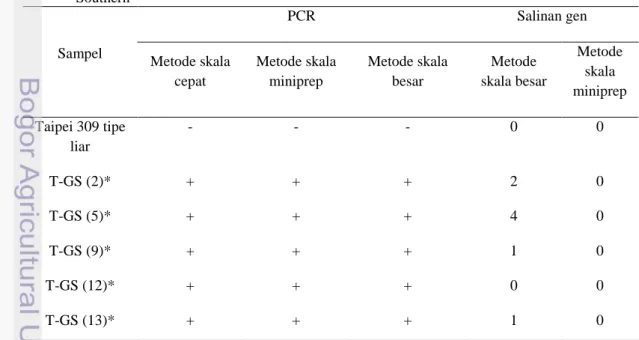
Analisis elektroforesis dari produk PCR dari 5 tanaman transgenik yang masing-masing diisolasi dengan ketiga metode menunjukkan hasil yang sama (Gambar 4). Semua sampel transgenik menghasilkan pita yang ukurannya sama dengan kontrol positif plasmid, yaitu 500 pb (pasangan basa). Penelitian sebelumnya yang dilakukan oleh
12
Tabel 2 Hasil pengukuran konsentrasi dan kemurnian DNA daun padi
Sampel Isolasi Konsentrasi ( g/mL) A260/280
Taipei-309 (kontrol
negatif) Skala cepat 70.1 1.31
T-GS (2)* Skala cepat 82.3 1.22 T-GS (5)* Skala cepat 139 1.42 T-GS (9)* Skala cepat 119 1.64 T-GS (12)* Skala cepat 31 1.12 T-GS (13)* Skala cepat 93.3 1.47 Taipei-309 (kontrol negatif) Miniprep 347.0 1.88 T-GS (2)* Miniprep 108.0 1.90 T-GS (5)* Miniprep 399.6 1.87 T-GS (9)* Miniprep 368.2 1.93 T-GS (12)* Miniprep 534.7 1.84 T-GS (13)* Miniprep 681.2 1.90 Taipei-309 (kontrol
negatif) Skala besar 771.3 1.89
T-GS (2)* Skala besar 2084.7 1.83
T-GS (5)* Skala besar 1007.1 1.82
T-GS (9)* Skala besar 2741 2.87
T-GS (12)* Skala besar 675.9 1.88
T-GS (13)* Skala besar 1934.7 1.81
Gambar 4 Elektroforegram hasil PCR menggunakan primer hpt. M=marker, A=air, 1=kontrol negatif skala cepat, 2-6=sampel, 7=kontrol negatif miniprep, 8-12=sampel, 13=kontrol negatif skala besar, 14-18=sampel, P=plasmid
Mulyaningsih (2011) juga menunjukkan bahwa pita yang dihasilkan dengan menggunakan primer hpt adalah 500 pb. Hasil ini menunjukkan bahwa dalam 5 tanaman transgenik telah mengandung transgen. Ketiadaan perbedaan hasil PCR antara DNA yang diisolasi dengan ketiga metode dan menunjukkan bahwa metode skala cepat merupakan metode yang paling efisien apabila hanya digunakan dalam tahap mengidentifikasi keberadaan gen target pada tanaman padi transgenik. Padmadi (2009) menyebutkan bahwa sampel DNA yang digunakan pada analisis PCR tidak harus merupakan DNA
murni.
Karakterisasi Molekuler menggunakan Hibridisasi Southern
Persiapan DNA genom merupakan hal yang utama sebelum melakukan hibridisasi Southern. Sampel DNA genom yang diisolasi harus memiliki kualitas yang baik yaitu murni dan utuh (tidak mengalami degradasi) (Nafari 2006). Pada penelitian ini persiapan DNA genom tanaman dilakukan menggunakan dua metode yaitu skala besar dan skala miniprep. Metode skala cepat tidak digunakan untuk hibridisasi Southern karena dalam isolasi DNA
M A 1 2 3 4 5 6 6 7 8 9 10 11 12 13 14 15 16 17 18 P M 500pb 300pb 100pb 1000pb 12000pb 500pb
13
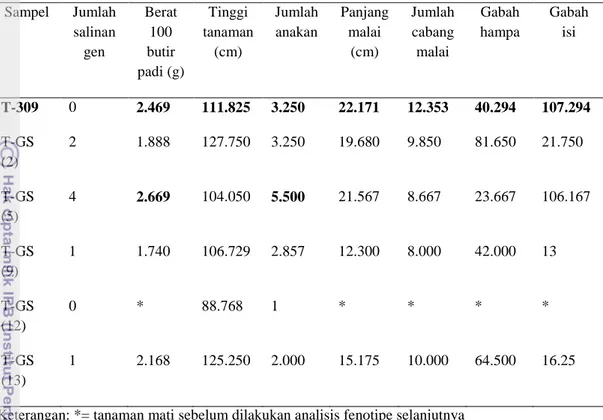
hanya menggunakan larutan NaOH sebagai bufernya, sehingga kemurnian sampel yang dihasilkan sangat buruk apabila digunakan dalam analisis ini. Hasil analisis DNA menggunakan PCR dan hasil analisis salinan gen yang dihasilkan pada metode isolasi miniprep dan skala besar dapat dilihat pada Tabel 3.
Analisis Southern dimulai dengan pemotongan sampel DNA genom menggunakan enzim restriksi BamHI. Ezim ini akan memotong satu kali pada daerah T-DNA sehingga pita DNA yang dihasilkan akan menggambarkan jumlah salinan transgen yang terintegrasi (Berg et al. 2007). Hasil pemotongan DNA genom dengan enzim restriksi menghasilkan jutaan fragmen yang rapat dan ukurannya yang bervariasi baik berat molekul yang tinggi maupun berat molekul rendah, sehingga fragmen-fragmen ini tidak dapat dilihat secara visual sebagai pita yang terpisah. Oleh karena itu, dihasilkan smear
merata dari atas ke bawah yang menunjukkan bahwa pemotongan telah sempurna (Lampiran 2).
Pelacak yang digunakan dalam analisis Southern adalah gen hpt. Pelacak hpt dilabel dengan digoxigenin menggunakan metode amplifikasi PCR dengan pCAMBIA 1301 35S Os-GS3 sebagai cetakan. Analisis elektroforesis menunjukkan bahwa produk PCR yang telah di label memiliki laju migrasinya lebih lambat jika dibandingkan dengan produk amplifikasi tanpa senyawa label (kontrol), karena DIG memiliki berat molekul yang tinggi dari hasil amplifikasi
yang mengandung DIG, sehingga akan memiliki berat molekul yang lebih tinggi dari hasil amplifikasi kontrol yang tidak mengandung DIG. Hal ini menunjukkan bahwa proses label sudah berhasil (Lampiran 3).
Proses transfer DNA ke membran juga merupakan hal penting dalam menentukan keberhasilan hibridisasi Southern. Hal yang perlu diperhatikan dinataranya harus terjadi penempelan sempurna antara gel dan membran tanpa adanya gelembung udara diantara kedunya. Dalam penelitian ini setelah proses transfer, gel direndam dalam Et-Br dan divisulalisasi menggunakan sinar UV. Hasil pengecekan menunjukkan bahwa transfer DNA ke membran terjadi secara sempurna karena tidak ada DNA yang tertinggal di gel agarosa (Lampiran 4). Hal ini sesuai dengan penelitian Nafari (2006) yang menggambarkan keberhasilan transfer DNA dari gel ke membran.
Proses hibridisasi juga merupakan hal penting dalam melakukan hibridisasi Southern. Hibridisasi merupakan pembentukan utas ganda antara DNA genom dengan utas tunggal dengan DNA pelacak utas tunggal yang telah diberi label DIG-11-dUTP (Coleman & Gregory 2006; Roche 2009). Pre-hibridisasi sangat diperlukan sebelum dilakukannya hibridisasi untuk memblok bagian membran yang tidak mengandung fragmen-fragmen DNA agar dapat meminimalisir background pada hasil foto, sehingga diharapkan hanya DNA sampel yang berpasangan dengan pelacak yang muncul saat deteksi.
Tabel 3 Hasil analisis dan pola integrasi gen sisipan (Os-GS3) pada generasi pertama (T0) pada kultivar Taipei 309 menggunakan primer hpt untuk PCR dan pelacak hpt untuk hibridisasi Southern Sampel PCR Salinan gen Metode skala cepat Metode skala miniprep Metode skala besar Metode skala besar Metode skala miniprep Taipei 309 tipe liar - - - 0 0 T-GS (2)* + + + 2 0 T-GS (5)* + + + 4 0 T-GS (9)* + + + 1 0 T-GS (12)* + + + 0 0 T-GS (13)* + + + 1 0