ABSTRACT
Genetically characterized isozyme is useful for taxonomy studies. The objective of this research was to know banding pattern variabilities of some pineapple accessions based on isozyme analyses. The research was conducted in September-November 2001 on 30 pineapple accessions. Polyacrylamide gel with vertical type was used to electrophoresis with six enzymes: peroxidase (PER), phosphoglucomutase (PGM), al-cohol dehydrogenase (ADH), malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), and glucose phosphate isomerase (GPI). The results showed that PER, PGM, ADH, MDH, and SKDH were polymorphic and had 6, 3, 3, 5, and 3 banding patterns, respectively, whereas, GPI was mono-morphic. Dendogram based on PER, PGM, ADM, MDH, and SKDH enzyme analyses on 30 pineapple accessions could be grouped into four clusters at 0.63 coefficient of similarity. [Keywords: Ananas comosus, pineapple, isoenzymes, poly-acrylamide]
ABSTRAK
Karakterisasi secara genetik berdasarkan isozim sangat ber-guna dalam bidang taksonomi. Penelitian ini bertujuan untuk mengetahui keragaman pola pita beberapa aksesi nenas ber-dasarkan analisis isozim. Penelitian dilakukan pada bulan September-November 2001 terhadap 30 aksesi nenas. Me-tode analisis yang digunakan adalah elektroforesis gel poli-akrilamid tipe vertikal dengan enam sistem enzim, yaitu peroksidase (PER), phosphoglucomutase (PGM), alcohol dehydrogenase (ADH), malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), dan glucose phosphate isomerase (GPI). Hasil analisis menunjukkan bahwa sistem enzim PER, PGM, ADH, MDH, dan SKDH adalah polimorfik dan masing-masing mempunyai berturut-turut 6, 3, 3, 5, dan 3 pola pita, sedangkan GPI adalah monomorfik. Dendogram berdasarkan enzim PER, PGM, ADM, MDH, dan SKDH pada 30 aksesi yang diuji terbagi menjadi empat kelompok pada kemiripan genetik 0,63.
[Kata kunci: Ananas comosus, nenas, isoenzim, poliakrilamid]
PENDAHULUAN
Langkah awal dalam pemuliaan nenas adalah mengoleksi genotipe dari populasi yang terdapat di masyarakat atau petani, kemudian mengkarakterisasi dan mengidentifikasinya. Hasilnya dapat digunakan sebagai tetua persilangan, evaluasi terhadap kultivar yang memiliki karakter yang diinginkan, serta untuk perbanyakan berbagai tanaman.
Di Indonesia terdapat berbagai kultivar nenas dengan nama daerah yang berbeda-beda dan klasifikasi botani yang belum jelas. Hume dan Miller (1904) dalam Aradya et al. (1994) membagi kultivar nenas ke dalam tiga kelompok, yaitu Cayenne, Queen, dan Spanish. Pembagian tersebut didasarkan pada kesamaan morfologi daun, ada/tidaknya duri daun, warna bunga, serta bentuk dan ukuran buah. Py et al. (1987) mengelompokkan nenas ke dalam lima kelompok dengan menambahkan pada kelompok yang sudah ada dengan Abacaxi atau Pernambuco dan Perola. Sementara Pracaya (1982) dan Muljohardjo (1984) membagi Cayenne menjadi dua subkelompok, yaitu Hilo dan Hawaian Smooth Cayenne. Hilo tidak mempunyai tunas tangkai buah, tetapi Hawaian Smooth Cayenne mempunyai tunas tangkai buah. Klasifikasi berdasarkan karakter morfologi mem-punyai beberapa kelemahan, antara lain penampilan karakternya sering rancu karena dipengaruhi oleh faktor lingkungan, karakter yang dapat digunakan untuk mengidentifikasi fase vegetatif sedikit, dan waktu yang dibutuhkan sampai tanaman mencapai dewasa cukup lama.
Penanda biokimia seperti isozim merupakan salah satu alternatif yang dapat digunakan untuk meng-karakterisasi dan mengklasifikasi koleksi plasma nutfah, karena isozim relatif stabil terhadap ling-kungan dan umumnya polimorfik (Aradya et al., 1994). Isozim memiliki beberapa karakteristik dan keuntungan
Keragaman pola pita beberapa aksesi nenas
berdasarkan analisis isozim
Banding pattern variabilities of some pineapple accessions based on isozyme analyses
Sri Hadiati1 dan Deden Sukmadjaja2
1Balai Penelitian Tanaman Buah, Jalan Raya Solok-Aripan km. 8, Solok, Sumatera Barat, Indonesia
(Brown dan Weir, 1983; Pasteur et al., 1988; Brar, 1992), antara lain: (1) produk dari alel yang berbeda bergerak pada posisi yang berbeda dalam gel, (2) alel yang berbeda biasanya diwariskan secara kodominan, bebas dari epistasis, sehingga individu homozigot dapat dibedakan dari heterozigot, (3) sering kali posisi pita merupakan produk dari suatu lokus sehingga me-mungkinkan untuk mendeteksi jumlah gen yang mengkode suatu enzim dengan menganalisis pola pita dari enzim tersebut, (4) peralatan dan bahan yang diperlukan relatif murah dan percobaan dapat di-lakukan dengan mudah di laboratorium, (5) jumlah sampel yang banyak dapat dianalisis dalam waktu singkat, serta (6) dapat dilakukan pada fase bibit sehingga menghemat waktu, tempat, dan biaya.
Berdasarkan karakteristik tersebut maka isozim telah banyak digunakan untuk mengidentifikasi dan mengklasifikasi beberapa spesies tanaman, seperti identifikasi triploid pada jeruk dengan empat sistem enzim (MDH, 6-PGD, SKDH, dan PGI) (King et al., 1996), identifikasi kultivar nenas dengan enzim
phosphoglucomutase (PGM) dan peroksidase (PER)
(de Wald et al., 1988), serta karakterisasi, klasifikasi, dan hubungan kekerabatan plasma nutfah nenas di Hawai (ADH, GPI, PGM, SKDH, TPI, UGPP) (Aradya
et al., 1994).
Kebun percobaan Balai Penelitian Tanaman Buah, Solok, mempunyai koleksi beberapa aksesi nenas yang berasal dari hasil eksplorasi di beberapa daerah di Jawa dan Sumatera. Koleksi nenas tersebut mem-punyai penampilan fenotipik yang beragam. Tepi daun bervariasi mulai tidak berduri sampai berduri, panjang tangkai buah 15-35 cm, warna daun hijau sampai hijau kemerahan, bentuk buah silindris hingga bulat, warna kulit buah kuning kehijauan sampai kuning keemasan, dan sebagainya. Perbedaan fenotipik tersebut kemungkinan disebabkan oleh perbedaan genetik, adanya mutasi, dan perbedaan daya adaptasi pada lingkungan tumbuh yang baru. Menurut Collins (1968), walaupun tanaman nenas diperbanyak secara vegetatif, tetapi terdapat ke-ragaman karakter yang disebabkan oleh mutasi. Broertjes dan Harten (1988) telah menemukan 30 mutan dari kultivar Cayenne. Penelitian ini bertujuan untuk mengetahui keragaman pola pita beberapa nomor aksesi nenas berdasarkan analisis isozim.
BAHAN DAN METODE
Penelitian dilaksanakan di Balai Penelitian Tanaman Buah pada bulan September-November 2001. Bahan
tanaman yang digunakan adalah daun muda dari 30 nomor aksesi nenas yang diambil sebelum tanaman memasuki fase generatif. Berdasarkan data deskripsi dan pengamatan morfologi, aksesi tersebut di-kelompokkan menjadi lima, yaitu Merah (no. 10, 33, 35, 44), Hijau (no. 3, 30, 32, 46), Merah Pagar (no. 1, 45), Queen (no. 2, 7, 11, 16, 17, 18, 22, 24, 28, 31), dan Cayenne (no. 4, 5, 8, 20, 26, 27, 34, 37, 38, 39) (Lampiran 1).
Elektroforesis isozim dilakukan dengan mengguna-kan dua lapis gel poliakrilamid, yaitu stacking gel (4% akrilamid, 0,5 M Tris-HCl pH 6,8, amonium persulfat, TEMED) dan separating gel (6-7,5% akrilamid, 1,5 M Tris-HCl pH 8,8) (Triest dan Kabir, 2000). Bufer elektroda yang digunakan adalah Tris-glicine pH 8,3. Daun muda sebanyak 0,5 g digerus pada kondisi dingin, kemudian ditambahkan 1 ml bufer ekstraksi menurut Wendel dan Weeden (1989). Ekstrak sampel disentrifuse dalam suhu dingin pada 10 000 rpm, kemudian pelet dibuang dan supernatan siap gunakan untuk elektroforesis. Elektroforesis di-lakukan pada suhu 4-10o C, voltase 125-175 V, dan arus 18-34 mA selama 4-5 jam. Selesai elektroforesis, gel dipindahkan ke baki dan diberi larutan pewarna sesuai dengan sistem enzim yang akan dianalisis. Pewarnaan dilakukan di atas shaker pada suhu ruang selama 30-60 menit. Komposisi larutan pewarna dibuat menurut prosedur Arulsekar dan Parfitt (1986) serta Wendel dan Weeden (1989) dengan beberapa modifikasi. Enzim yang dianalisis adalah peroksidase (PER : EC 1.11.1.7), phosphoglucomutase (PGM : EC 2.7.2.1), alcohol dehydrogenase (ADH : EC 1.1.1.1),
malate dehydrogenase (MDH: EC 1.1.1.37), shikimate dehydrogenase (SKDH: EC 1.1.1.25), dan glucose phosphate isomerase (GPI : EC 5.3.1.9).
Pola pita yang terbentuk digambar sebagai zimogram. Polimorfisme dari pola pita yang terbentuk diukur kemiripan genetiknya berdasarkan ada tidak-nya pita. Rumus kemiripan genetik yang digunakan adalah kemiripan genetik Dice (Sakal dan Sneath, 1963. dalam Lamadji, 1998), kemudian dibuat matrik data untuk analisis kelompok dengan menggunakan program NTSys. Tingkat kepercayaan dari dendo-gram ditentukan melalui analisis bootstrap dengan pe-ngulangan 200 kali dengan program win boot.
HASIL DAN PEMBAHASAN
Hasil analisis terhadap enam sistem enzim pada 30 nomor aksesi nenas menunjukkan bahwa tidak semua enzim mempunyai keragaman pola pita. Enzim yang
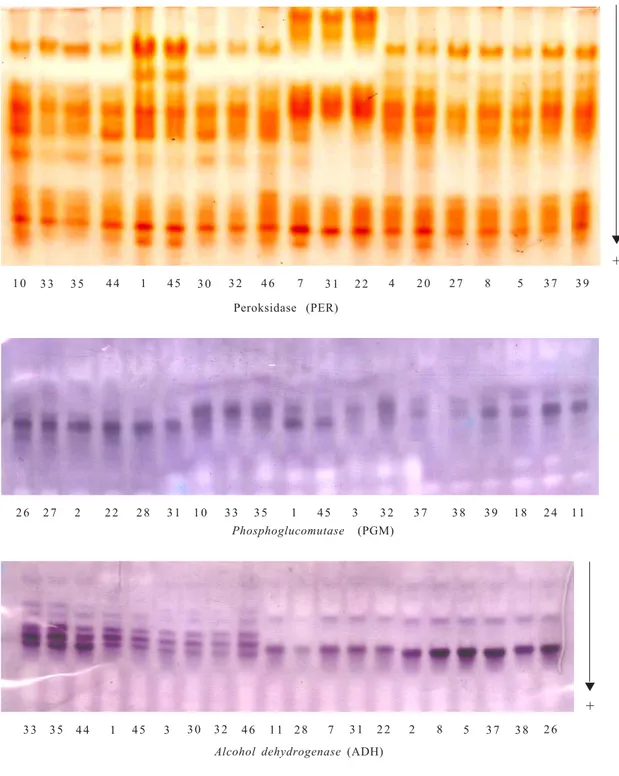
menunjukkan keragaman pola pita adalah PER, PGM, ADH, MDH, dan SKDH, sedangkan GPI tidak me-nunjukkan keragaman pola pita (Gambar 1 dan 2).
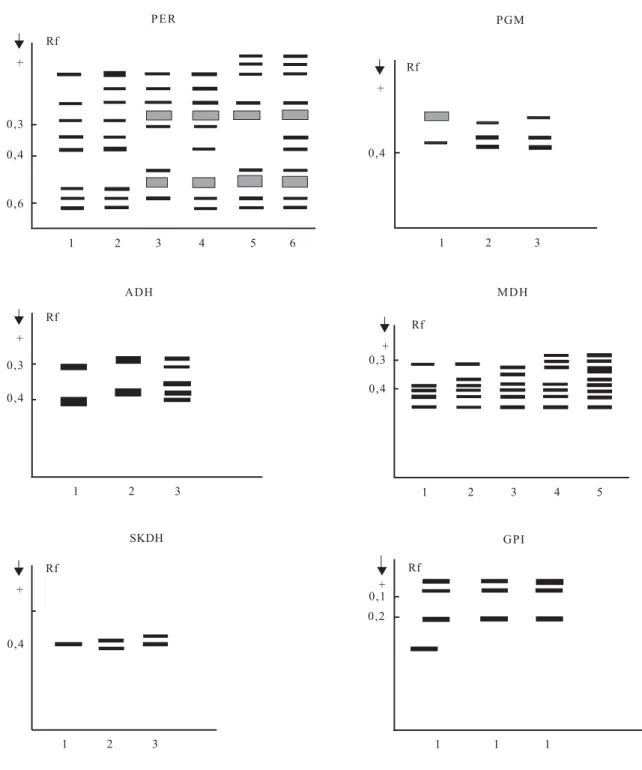
Pada sistem enzim PER, terdapat tiga daerah aktif yang dikendalikan oleh tiga lokus (Gambar 1). Hasil penelitian de Wald et al. (1988; 1992) juga
mem-perlihatkan bahwa enzim PER mempunyai tiga daerah aktif, tetapi hanya dua daerah aktif yang jelas dan dapat digunakan untuk identifikasi nenas. Ketiga daerah aktif tersebut digambar pola pitanya, dan terdapat enam pola pita (Gambar 3). Pola pita satu ditemukan pada klon Merah (no. 10, 33, 35, 44) dan
Gambar 1. Variasi pola pita isozim peroksidase (PER), phosphoglucomutase (PGM), dan alcohol dehydrogenase (ADH) pada beberapa aksesi nenas.
Fig 1. Variation of isozyme banding pattern of peroxidase (PER), phosphoglucomutase (PGM), and alcohol dehydrogenase (ADH)
of some pineapple accesions.
Alcohol dehydrogenase (ADH)
▼ 1 0 + Peroksidase (PER) 2 6 2 7 2 2 2 2 8 3 1 1 0 3 3 3 5 1 4 5 3 3 2 3 7 3 8 3 9 1 8 2 4 1 1 Phosphoglucomutase (PGM) 3 3 3 5 4 4 1 4 5 3 3 0 3 2 4 6 1 1 2 8 7 3 1 2 2 2 8 5 3 7 3 8 2 6 3 3 3 5 4 4 1 4 5 3 0 3 2 4 6 7 3 1 2 2 4 2 0 2 7 8 5 3 7 3 9 + ▼
Malate dehydrogenase (MDH)
▼
+
Shikimate dehydrogenase (SKDH)
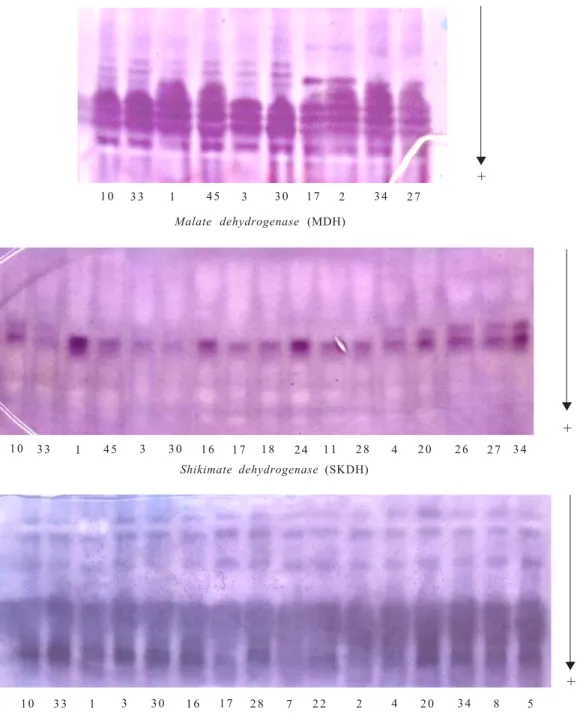
Gambar 2. Variasi pola pita isozim malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), dan glucose phosphate isomerase (GPI) pada beberapa aksesi nenas.
Fig 2. Variation of isozyme banding pattern of malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), and
glucose phosphate isomerase (GPI) of some pineapple accesions.
1 0 3 3 1 4 5 3 3 0 1 7 2 3 4 2 7
1 0 3 3 1 4 5 3 3 0 1 6 1 7 1 8 2 4 1 1 2 8 4 2 0 2 6 2 7
▼
+
3 4
Glucose phosphate isomerase (GPI)
1 0 3 3 1 3 3 0 1 6 1 7 2 8 7 2 2 2 4 2 0 3 4 8 5
▼
+
Hijau (no. 3, 30, 32, 46), dan pola pita dua pada klon Merah Pagar (no. 1, 45). Enzim PER mampu mem-bedakan klon Cayenne menjadi dua kelompok, yaitu aksesi yang mempunyai pola pita tiga (no. 5, 8, 20, 26, 27, 34, 39) dan yang mempunyai pola pita empat (no. 4, 37, 38). Enzim PER juga membedakan klon Queen menjadi dua kelompok, yaitu aksesi yang mempunyai
pola pita lima (no. 2, 16, 17, 18, 22, 24, 28, 31) dan yang berpola pita enam (no. 11, 7) (Tabel 1).
Enzim PGM hanya memperlihatkan satu daerah aktif dengan tiga alel (Gambar 1). Aradya et al. (1994) mendapatkan dua daerah aktif pada tanaman nenas, tetapi hanya satu daerah aktif yang jelas, yaitu pada pita yang migrasinya lambat. Variasi jumlah daerah
Gambar 3. Keragaman pola pita isozim peroksidase (PER), phosphoglucomutase (PGM), alcohol dehydrogenase (ADH), malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), dan glucose phosphate isomerase (GPI).
Fig. 3. Variabilities of isozyme banding pattern of peroxidase (PER), phosphoglucomutase (PGM), alcohol dehydrogenase (ADH),
malate dehydrogenase (MDH), shikimate dehydrogenase (SKDH), and glucose phosphate isomerase (GPI). 0,3 0,4 0,6 Rf + 1 2 3 4 5 6 ▲ 0,3 0,4 Rf + ▲ 1 2 3 Rf + ▲ 0,4 1 2 3 Rf + ▲ 0,4 1 2 3 Rf + ▲ 0,3 0,4 1 2 3 4 5 Rf + ▲ 0,2 0,1 1 1 1 PER PGM ADH MDH SKDH GPI
aktif/lokus ini sangat dipengaruhi oleh pH (Soltis et
al., 1983). Seluruh nomor aksesi dapat digambarkan
menjadi tiga pola pita (Gambar 3 ). Pola pita satu ditemukan pada klon Merah (no. 10, 33, 35, 44 ) dan Hijau (no. 3, 30, 32, 46), pola pita dua pada klon Cayenne (no. 4, 5, 8, 20, 26, 27, 34, 37, 38, 39), serta pola pita tiga pada klon Merah Pagar (no. 1, 45) dan klon Queen (no. 2, 7, 11, 16, 17, 18, 22, 24, 28, 31) (Tabel 1).
Enzim PGM belum mampu membedakan antara klon Merah dan Hijau dan antara klon Merah Pagar dan Queen. Dengan menggunakan enzim PGM ini, keragaman pola pita dalam klon tidak terlihat.
Alcohol dehydrogenase (ADH) mempunyai dua
daerah aktif (Gambar 1) dan dari kedua daerah tersebut diperoleh tiga pola pita (Gambar 3). Pola pita satu ditemukan pada klon Cayenne (no. 4, 5, 8, 20,
(Tabel 1). Enzim MDH belum dapat membedakan antara klon Merah dan Hijau.
Shikimate dehydrogenase (SKDH) mempunyai satu
daerah aktif dengan satu sampai dua alel (Gambar 2). Dari seluruh aksesi dapat diperoleh tiga pola pita (Gambar 3). Pola pita satu ditemukan pada klon Queen (no. 2, 7, 11, 16, 17, 18, 22, 24, 28, 31), pola pita dua pada klon Merah Pagar (no. 1, 45), dan pola pita tiga pada klon Hijau (no. 3, 30, 32, 46), Merah (no. 10, 33, 35, 44), dan Cayenne ( no. 4, 5, 8, 20, 26, 27, 34, 37, 38, 39) (Tabel 1).
Glucose phosphate isomerase (GPI) mempunyai dua
daerah aktif (Gambar 2). Pada daerah aktif pertama, yaitu pita yang migrasinya cepat, pola pitanya tidak jelas sehingga sulit dalam melakukan penyekoran. Pada daerah aktif kedua, yaitu pita yang migrasinya lambat, pola pita terdiri atas tiga alel dan tidak menunjukkan keragaman dalam klon maupun antar-klon atau monomorfik. Di Hawai, dari 161 aksesi nenas yang diamati, 128 aksesi mempunyai pola pita GPI yang sama dan 33 aksesi mempunyai enam pola pita GPI yang berbeda (Aradya et al., 1994). Perbedaan hasil tersebut disebabkan jumlah spesies dan aksesi yang digunakan di Hawai lebih banyak dibandingkan dengan yang digunakan dalam penelitian ini. Selain itu, tidak munculnya polimorfisme pola pita isozim pada aksesi yang dideteksi dengan elektroforesis disebabkan pola pita yang terdeteksi hanya didasar-kan pada perbedaan muatan pada protein terlarut saja. Padahal dari 30% protein yang terlarut pada sel tanaman, hanya 25% yang memiliki muatan berbeda yang dapat terdeteksi dengan elektroforesis (Weir, 1990).
Semua enzim yang digunakan, yaitu PER, PGM, ADH, MDH, SKDH, dan GPI, tidak dapat membedakan klon Merah (no. 10, 33, 35, 44) dengan klon Hijau (no. 3, 30, 32, 46). Menurut asal usulnya, diduga nenas Hijau merupakan hasil mutasi dari nenas Merah. Hal ini diperkuat oleh Leal dan Coppens (1996), bahwa Green Selangor dan Green Spanish merupakan mutan yang terjadi dari Red Spanish. Hal serupa juga terjadi pada mutan kaktus. Mutan Dark Marie mempunyai pola pita yang sama dengan Marie sebagai tetuanya berdasar-kan enzim GPI, MDH, PGM, SKDH, TPI, dan LAP, padahal kedua kultivar tersebut mempunyai bunga dan pigmen phylloclade yang berbeda (Leary dan Boyle, 2000). Agar diperoleh keragaman pola pita yang dapat membedakan klon Merah dan Hijau, maka perlu dilakukan penambahan jenis enzim lainnya. Menurut Purnomo (1994), untuk menelaah perbedaan individu dalam populasi, disarankan paling sedikit menggunakan delapan macam enzim.
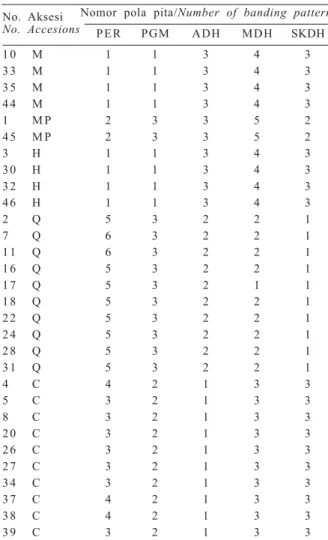
Tabel 1. Keragaman pola pita isozim pada 30 nomor aksesi nenas.
Table 1. Variabilities of isozyme banding pattern of 30 pineapple accesions.
No. Aksesi Nomor pola pita/Number of banding pattern
No. Accesions PER PGM ADH MDH SKDH
1 0 M 1 1 3 4 3 3 3 M 1 1 3 4 3 3 5 M 1 1 3 4 3 4 4 M 1 1 3 4 3 1 M P 2 3 3 5 2 4 5 M P 2 3 3 5 2 3 H 1 1 3 4 3 3 0 H 1 1 3 4 3 3 2 H 1 1 3 4 3 4 6 H 1 1 3 4 3 2 Q 5 3 2 2 1 7 Q 6 3 2 2 1 1 1 Q 6 3 2 2 1 1 6 Q 5 3 2 2 1 1 7 Q 5 3 2 1 1 1 8 Q 5 3 2 2 1 2 2 Q 5 3 2 2 1 2 4 Q 5 3 2 2 1 2 8 Q 5 3 2 2 1 3 1 Q 5 3 2 2 1 4 C 4 2 1 3 3 5 C 3 2 1 3 3 8 C 3 2 1 3 3 2 0 C 3 2 1 3 3 2 6 C 3 2 1 3 3 2 7 C 3 2 1 3 3 3 4 C 3 2 1 3 3 3 7 C 4 2 1 3 3 3 8 C 4 2 1 3 3 3 9 C 3 2 1 3 3
Keterangan: M = Merah; MP = Merah Pagar; H = Hijau; Q = Queen; C = Cayenne; Nomor pola pita sesuai dengan Gambar 3.
Notes: M = Merah; MP = Merah pagar; H = Hijau; Q = Queen; C = Cayenne; The band number of banding pattern is same as the Figure 3.
26, 27, 34, 37, 38, 39), pola pita dua pada klon Queen (no. 2, 7, 11, 16,17, 18, 22, 24, 28, 31) dan pola pita tiga pada klon Merah (no. 10, 33, 35, 44 ), Hijau (no. 3, 30, 32, 46), dan Merah Pagar (no. 1, 45) (Tabel 1).
Malate dehydrogenase (MDH) mempunyai tiga
daerah aktif dengan lima pola pita (Gambar 2). Enzim MDH dapat membedakan klon Queen menjadi dua kelompok, yaitu yang berpola pita satu (no. 17) dan yang berpola pita dua (no. 2, 7, 11, 16, 18, 22, 24, 28, 31). Pola pita tiga ditemukan pada klon Cayenne (no. 4, 5, 8, 20, 26, 27, 34, 37, 38, 39), pola pita empat pada klon Merah (no. 10, 33, 35, 44) dan Hijau (no. 3, 30, 32, 46), serta pola pita lima pada klon Merah Pagar (no. 1, 45)
yang digunakan hanya sesuai untuk pengelompokan antarklon, sedangkan untuk pengelompokan dalam klon memerlukan jumlah enzim yang lebih banyak lagi.
KESIMPULAN
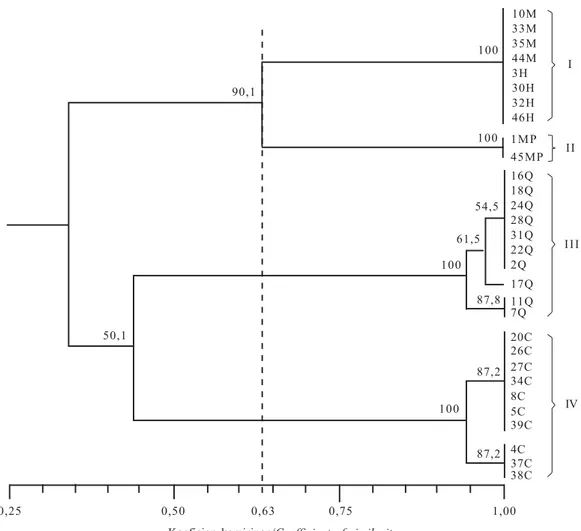
Hasil analisis isozim terhadap 30 nomor aksesi nenas menunjukkan bahwa sistem enzim PER, PGM, ADH, MDH, dan SKDH adalah polimorfik dengan jumlah pola pita masing-masing sebanyak 6, 3, 3, 5, dan 3 pola pita. Enzim GPI tidak menunjukkan pola pita yang berbeda atau monomorfik. Pada kemiripan genetik 0,63, dendogram terdiri atas empat kelompok, yaitu (I) klon Merah dan klon Hijau, (II) klon Merah Pagar, (III) klon Queen, dan (IV) klon Cayenne.
Dendogram pada Gambar 4 menunjukkan penge-lompokan dan kemiripan genetik dari semua aksesi yang dianalisis berdasarkan variasi pola pita dari enzim PER, PGM, ADH, MDH, dan SKDH. Dari dendogram tersebut, pada kemiripan 0,63 (63%) 30 aksesi nenas terbagi menjadi empat kelompok, yaitu (I) klon Merah dan klon Hijau, (II) klon Merah Pagar, (III) klon Queen, dan (IV) klon Cayenne.
Analisis bootstrap dilakukan untuk mengetahui tingkat kepercayaan pengelompokan. Hasil analisis menunjukkan bahwa keempat kelompok mempunyai tingkat kepercayaan yang tinggi, yaitu masing-masing 100%, sedangkan pengelompokan aksesi di dalam kelompok Queen dan Cayenne mempunyai tingkat kepercayaan yang rendah, yaitu di bawah 90% (Gambar 4). Hasil ini menunjukkan bahwa jumlah enzim
50,1 90,1 0,50 0,75 100 100 87,2 87,2 87,8 61,5 1MP 45MP 16Q 18Q 24Q 28Q 31Q 22Q 2Q 17Q 11Q 7Q 20C 26C 27C 34C 8C 5C 39C 4C 38C 37C
Koefisien kemiripan/Coefficient of similarity
0,25 1,00 10M 33M 35M 44M 3H 30H 32H 46H 100 100 54,5
Gambar 4. Dendogram 30 nomor aksesi nenas berdasarkan analisis enzim PER, PGM, ADH, MDH, dan SKDH. Angka-angka pada garpu merupakan persentase tingkat ketelitian hasil analisis bootstrap 2000 kali.
Fig 4. Dendogram of 30 pineapple accessions based on PER, PGM, ADH, MDH, and SKDH analysis. Numbers in the fork are
percentage of signifance of the bootstrap analysis at 2000 times.
I
II
III
IV
UCAPAN TERIMA KASIH
Penulis mengucapkan terima kasih kepada Prof. Dr. Ir. Murdaningsih Haeruman Karmana, M.Sc, Prof. Ir. Achmad Baihaki, MSc, PhD, dan Dr. Ir. Neni Rostini, MSc atas segala saran-saran dalam penelitian maupun penulisan ini, serta kepada Kartono, SP yang telah membantu dalam penyediaan materi penelitian.
DAFTAR PUSTAKA
Aradya, K.M., F. Zee, and R.M. Manshardt. 1994. Isozyme variation in cultivated and wild pineapple. Euphytica 79: 87-99.
Arulsekar, S. and D.E. Parfitt. 1986. Isozyme analysis procedures for stone fruits, almond, grape, walnut, pistachio and fig. Hort. Sci. 21 (4): 928-933.
Brar, D.S. 1992. Isozyme: Technique and aplications in rice improvement. Third Rice Biotechnology Training Course, Los Banos, 5 October-27 November 1992. 18 p. Broertjes, C. and A.M. van Harten. 1988. Applied mutation
breeding for vegetatively propagated crops. Development in Crops Science 12. Elsevier, London. p. 286-287. Brown, A.D.H. and B.S. Weir. 1983. Measuring genetic
variability in plant population. p. 219-240. In S.D. Tanksley and T.J. Orton (Eds.). Isozymes in Plant Genetics and Breeding. Part A. Elsevier, Amsterdam.
Collins, J.L. 1968. The pineapple, botany, cultivation and utilization. Leonard Hill, London. 293 p.
De Wald, M., G.A. Moore, and W.B. Sherman. 1988. Identification of pineapple cultivars by isozyme genotype. J. Amer. Soc. Hort. Sci. 113 (6): 935-938.
De Wald, M., G.A. Moore, and W.B. Sherman. 1992. Isozymes in Ananas (pineapple): Genetics and usefulness in taxonomy. J. Amer. Soc. Hort. 117 (3): 491-496.
King, B.J., L.S. Lee, and P.T. Scott. 1996. Identification of triploid Citrus by isozyme analysis. Euphytica 99: 223-231.
Lamadji, S. 1998. Pemberdayaan sifat morfologi untuk analisis kekerabatan plasma nutfah tebu. Buletin P3GI 148: 17-31.
Leal, F. and G. Coppens. 1996. Pineapple. p. 515-557. In J. Janick and J.N. Moore (Eds.). Fruit Breeding Volume I. Tree and Tropical Fruit. John Wiley and Son Inc., New York. Leary, M.C.O. and T.H. Boyle. 2000. Diversity and distribution
of isozymes in a Schlumbergera (Cactaceae) clonal germ-plasm collection. J. Amer. Soc. Hort. Sci. 125 (1): 81-85. Muljohardjo, M. 1984. Nenas dan teknologi pengolahannya
(Ananas comosus). Penerbit Liberty, Yogyakarta. hlm. 12-30. Pasteur, N., G. Pasteur, F. Bonhomme, J. Catalan, and J. Britton-Davidian. 1988. Practical isozyme genetics. Ellis Horwood Limited, England. 212 p.
Pracaya. 1982. Bertanam nenas. Penebar Swadaya, Jakarta. 94 hlm.
Purnomo, S. 1994. Kaitan aktivitas beberapa enzim dengan sifat buah dan pola pewarisannya pada persilangan dialil salak Bali dan Pondoh. Disertasi. Universitas Padjadjaran, Bandung.
Py, C., J.J. Lacoeuilhe, and C. Teisson. 1987. The pineapple, cultivation and uses. G.P. Maisonneuve and Larose, Paris. 568 p.
Soltis, D.E., C.H. Haufler, D.C. Darrow, and G.J. Gastory. 1983. Starch gel electrophoresis of ferns: A compilation of grinding buffers, gels and electrode buffers, and staining schedules. Amer. Fern J. 73: 9-27.
Triest, L. and S.M.G. Kabir. 2000. The use of allozymes at three life stages influences genetic variation analysis and clustering in Cicer arietinum germplasm collections. Euphytica 112: 109-115.
Weir. 1990. Genetic data analysis: Methods for discrete population genetic data. Sunderland, Massachusetts. p. 1-2 0 .
Wendel, J.F. and N.F. Weeden. 1989. Visualization and interpretation of plant isozymes. In D.E. Soltis and P.S. Soltis. Isozymes in Plant Biology. Dioscorides Press, Portland, Oregon, vol. 4: 5-45.
Lampiran 1. Daftar aksesi tanaman nenas yang digunakan dalam penelitian.
Appendix 1. List of pineapple accessions used in the study.
Nomor aksesi/
Klon/Clone Nama/Name Asal/Origin
Accesion number
10 M Merah Nenas Merah 2 Deli Serdang
33 M Merah Nenas Merah 3 Paninjauan
1 MP Merah pagar Merah -1 Aripan - Solok
45 MP Merah pagar Merah-2 Payakumbuh
3 H Hijau Nenas Hijau Aripan - Solok
32 H Hijau Nenas Hijau Panjaran
30 H Hijau Nenas Hijau Kiliranjau
16 Q Queen Nenas Palembang 4 Sukananti - Ogan Komering Ilir
17 Q Queen Nenas Palembang 1 Tbg Kelekar - Muara Enim
18 Q Queen Nenas Palembang 2 Tbg Kelekar - Muara Enim
24 Q Queen Nenas Palembang 6 Fitotek - Lampung
11 Q Queen Nenas Palembang 3 Sekayu - Palembang
28 Q Queen Nenas berduri Kiliranjau
7 Q Queen Nenas Bangkinang 1 Bangkinang - Pekanbaru
31 Q Queen Nenas Bangkinang 3 Bangkinang - Pekanbaru
22 Q Queen Nenas Blitar Blitar
2 Q Queen Bangka Bangka
4 C Cayenne Nenas Smooth Cayenne Payakumbuh
20 C Cayenne Nenas Smooth Cayenne 1 Terbanggi Besar- Lampung
26 C Cayenne Nenas Smooth Cayenne Paninjauan
27 C Cayenne Nenas Smooth Cayenne Payakumbuh
34 C Cayenne Nenas Smooth Cayenne Pariaman
8 C Cayenne Nenas Smooth Cayenne 4 Simalungun
5 C Cayenne Nenas Smooth Cayenne Bogor
37 C Cayenne Nenas Smooth Cayenne Subang
38 C Cayenne Nenas SC Bogor Subang
39 C Cayenne Nenas Hutan Tiga Tanah Karo
44 M Merah Nenas Merah 4 Aripan - Solok
35 M Merah Pariaman Pariaman