EVALUASI STRUKTUR KRISTAL 4XCT SEBAGAI TARGET VIRTUAL ENZIM MATRIX METALLOPROTEINASE 9
PADA PENAPISAN VIRTUAL BERBASIS STRUKTUR
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh: Donny Suparto NIM: 138114094
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA YOGYAKARTA
i
EVALUASI STRUKTUR KRISTAL 4XCT SEBAGAI TARGET VIRTUAL ENZIM MATRIX METALLOPROTEINASE 9
PADA PENAPISAN VIRTUAL BERBASIS STRUKTUR
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh: Donny Suparto NIM: 138114094
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA YOGYAKARTA
iv
HALAMAN PERSEMBAHAN
“Tuhan akan berperang untuk kamu, dan kamu akan diam saja“
(Keluaran 14:14)
Karya ini kupersembahkan untuk Papa, Mama
viii DAFTAR ISI
HALAMAN JUDUL ... i
HALAMAN PERSETUJUAN ... ii
HALAMAN PENGESAHAN ... iii
HALAMAN PERSEMBAHAN ... iv
PERNYATAAN KEASLIAN KARYA ... v
LEMBAR PERNYATAAN PERSETUJUAN PUBLIKASI KARYA ILMIAH UNTUK KEPENTINGAN AKADEMIS ... vi
PRAKATA ... vii
DAFTAR ISI ... viii
DAFTAR TABEL ... x
DAFTAR GAMBAR ... xi
DAFTAR LAMPIRAN ... xii
ABSTRAK ... xiii ABSTRACT ... xiv PENDAHULUAN ... 1 METODE ... 2 Bahan ... 2 Alat ... 2 Metode ... 3
Preparasi Ligan EN73 ... 3
Penambatan Molekul Ligan EN73 ... 3
Konversi Seribu Pose Terbaik Menjadi PDB ... 3
Penyesuaian Koordinat Ligan ... 3
Perhitungan RMSD dan Visualisasi ... 3
Analisis Statistik ... 4
HASIL DAN PEMBAHASAN ... 4
KESIMPULAN ... 7
SARAN ... 7
DAFTAR PUSTAKA ... 8
ix
x
DAFTAR TABEL
Tabel I. Jumlah Pose dan Median Tiap Klaster ... 5 Tabel II. Jarak Interaksi Ligan dengan Residu Pada Tiap Pose ... 6
xi
DAFTAR GAMBAR
Gambar 1. Gambar 3D setelah penyesuaian koordinat atom ligan referensi dengan ligan EN73 beserta gambar 2D ... 4 Gambar 2. Histogram Seribu Pose dengan Tiga Klaster... 5 Gambar 3. Pose Ligan Ref, K1, K2 dan K3 dengan Residu Pada Kantung Ikatan
xii
DAFTAR LAMPIRAN
Lampiran 1. Script yang digunakan untuk preparasi ligan EN73 ... 11 Lampiran 2. Script yang digunakan untuk penambatan ulang ligan EN73 ... 12 Lampiran 3. Script yang digunakan untuk membuat daftar
1000 pose terbaik ... 13 Lampiran 4. Script untuk menyalin 1000 pose terbaik menjadi pdb ... 14 Lampiran 5. Script yang digunakan untuk menghitung nilai RMSD ... 15
xiii ABSTRAK
Diabetic Foot Ulcer (DFU) merupakan salah satu komplikasi dari
diabetes yang dapat berdampak buruk hingga berakibat pada amputasi. Ekspresi berlebihan matrix metalloproteinase 9 (MMP-9) pada saat diabetes terbukti dapat menunda penyembuhan luka pada penderita DFU. N~2~-[biphenyl-4-yl(dihydroxy)-lambda~4~-sulfanyl]-N-oxo-N~2~-(propan-2-yloxy)-D-valinamide dengan kode ligan EN73 diketahui dapat menghambat aktivitas MMP-9. Penambatan ulang ligan EN73 perlu dilakukan untuk pengembangan protokol Penapisan Virtual Berbasis Struktur (PVBS). Protokol penapisan virtual berbasis struktur bisa diterima apabila penambatan ulang ligan EN73 dan MMP-9 menghasilkan nilai RMSD tidak lebih besar dari 2,0 Å. Evaluasi struktur kristal 4XCT dilakukan dengan penambatan molekuler menggunakan PLANTS 1.2 dan konfigurasi PLANTS yang dikembangkan oleh Anita et al. (2012). Hasil penambatan seribu kali dengan lima kali iterasi hanya mampu menghasilkan 19 pose dengan RMSD tidak lebih dari 2,0 Å. Median tiga klaster berturut-turut yaitu K1 1,722 Å ; K2 3,475 Å ; K3 4,299 Å. Banyaknya ikatan hidrogen pada setiap klaster yaitu K1 2 ikatan, K2 1 ikatan dan K3 1 ikatan. Berdasarkan uji statistik
one sample t-test nilai p-value 2,2 x 10-16 dapat disimpulkan RMSD lebih besar dari 2,0 Å sehingga tidak sesuai digunakan sebagai target virtual MMP-9 dengan PLANTS konfigurasi Anita et al. (2012).
xiv ABSTRACT
Diabetic Foot Ulcer (DFU) is one of complications in diabetes that can adversely affect the possibility of amputation. Excessive expression of Matrix Metalloproteinase 9 (MMP-9) during diabetes can delay wound healing in patients with DFU. N ~ 2 ~ - [biphenyl-4-yl (dihydroxy) -lambda ~ 4 ~ -sulfanyl] -N-oxo-N ~ 2 ~ - (propane-2-yloxy) -D-valinamide coded as E-N-oxo-N73 ligand is known to inhibit MMP-9 activity. Anchoring the ligand EN73 is necessary for a protocol development in Structure-Based Virtual Screening (SBVS). Structure-based virtual screening protocols can be accepted if the interaction of ligand EN73 and MMP-9 can produce RMSD value not greater than 2.0 Å. Evaluation of 4XCT crystal structure is carried out by docking using PLANTS 1.2 and PLANTS configuration developed by Anita et al. (2012). Results with 1000 times to five times iteration can produce 19 poses with RMSD not more than 2.0 Å. Median of three clusters are K1 1,722 Å; K2 3.475 Å; K3 4.299 Å, respectively. The observed hydrogen bonds interaction are: K1 has two hydrogen bonds, both K2 and K3 only have 1 hydrogen bond. Based on statistical test using one sample t-test result p-value of 2.2 x 10-16 thus concluded the RMSD is greater than 2.0 Å therefore it is unsuitable used as virtual target using PLANTS docking method and configuration developed by Anita et al. (2012).
1
Diabetes melitus adalah kelompok penyakit metabolik yang ditandai dengan hiperglikemia akibat gangguan pada sekresi insulin, kerja insulin, atau keduanya (American Diabetes Association, 2014). Indonesia masuk ke dalam salah satu dari 10 negara dengan penderita diabetes tertinggi di dunia (Guariguata et al., 2014). Setiap tahunnya penderita diabetes di Indonesia mengalami peningkatan. Pada tahun 2007 prevalensi penderita diabetes di Indonesia mencapai 5,7% (Mihardja et al., 2009) dan pada tahun 2030 diprediksi akan mencapai 6,0% (Shaw et al., 2010) atau setara 8,5 juta pada tahun 2013 dan akan meningkat menjadi 14,1 juta pasien pada tahun 2035 (Guariguata et
al., 2014).
Diabetes yang mengalami komplikasi dapat menyebabkan peripheral neuropathy dengan resiko terjadinya diabetic foot ulcer (DFU) dan berdampak pada kemungkinan amputasi (American Diabetes Association, 2014). Penderita diabetes yang mengalami DFU sebanyak 7,3% - 24% dari total penderita diabates di Indonesia (Soewondo et al., 2013). DFU ini sering menjadi infeksi, menyebabkan morbiditas yang besar, membutuhkan biaya pengobatan yang cukup besar, dan langkah terakhir adalah amputasi (Singh et al., 2015).
Diabetes dikaitkan dengan terjadinya kerusakan sel yang dapat menghambat fibroblas membentuk extracellular matrix (ECM) dan keratinosit untuk mengepitelisasi ulkus (Hamed et al., 2014). Penderita DFU membutuhkan waktu rata rata penyembuhan ulkus kaki tanpa amputasi yaitu 63 hari (Ince et al., 2007). Gangguan re-epitalisasi pada ulkus dikarenakan ekspresi berlebihan dari matrix metalloproteinase 9 (MMP-9) (Mohan
et al., 2002). Diketahui bahwa kelompok matrix metalloproteinases (MMPs) adalah
protease endopeptidase zinc-dependent yang dapat mendegradasi semua komponen ECM (Lobmann et al., 2002). Maka dari itu, penghambatan ekspresi dan aktivitas MMP-9 yang berlebihan diharapkan bisa mempercepat proses penyembuhan luka bagi penderita DFU.
Penapisan virtual berbasis struktur merupakan pilihan pada tahap awal proses penemuan obat baru (Cavasotto and Orry, 2007). Keuntungan penggunaan komputerisasi dalam proses penemuan obat baru yaitu mendapatkan kandidat obat baru dalam waktu yang lebih cepat dan biaya yang lebih efisien (Lionta et al., 2014). Faktor yang menentukan ligan dan protein diterima sebagai protokol penapisan virtual berbasis struktur yaitu bila dapat menghasilkan mode ikatan yang reprodusibel (Verdonk et al., 2004). Selain itu protokol penapisan virtual berbasis struktur bisa diterima apabila menghasilkan
2
berbasis struktur dapat menyeleksi senyawa aktif dari berbagai senyawa yang berpotensi (Bissantz et al., 2000).
N~2~-[biphenyl-4-yl(dihydroxy)-lambda~4~-sulfanyl]-N-oxo-N~2~-(pro pan-2-yloxy)-D-valinamide dengan kode ligan EN73 telah berhasil dikristalkan dengan MMP-9 (Nuti et al. 2015). Dengan X-ray crystallography, diketahui EN73 memiliki resolusi sebesar 1,3 Å (Nuti et al., 2015). Meskipun telah diuji secara in vitro dan in vivo, serta diketahui jenis ikatannya dengan MMP-9, untuk keperluan computational based drug
design perlu dilakukan penambatan ulang untuk mengetahui apakah EN73 akan
memberikan nilai RMSD tidak lebih besar dari 2,0 Å. Apabila menghasilkan RMSD yang tidak lebih besar dari 2,0 Å maka ligan EN73 dapat diterima sebagai target virtual dalam penapisan virtual berbasis struktur. Penelitian ini menggunakan konfigurasi PLANTS 1.2 oleh Anita et al. (2012) untuk mengetahui pose EN73 pada kantung ikatan MMP-9.
METODE PENELITIAN Bahan
Bahan yang digunakan dalam penelitian ini adalah konfigurasi PLANTS dari Anita
et al. (2012), struktur ko-kristal MMP-9 (didapatkan dari http://www.rscb.org dengan kode 4XCT), SPORES (Brink dan Exner, 2009) untuk menyiapkan senyawa dengan format .mol2 supaya dapat digunakan oleh perangkat lunak PLANTS 1.2 (Korb et al., 2009), Open Babel untuk mengkonversi file .mol2 menjadi .pdb, docking software PLANTS 1.2 (Korb et al., 2009) untuk melakukan simulasi penambatan ulang molekuler dan didapatkan skor ChemPLP, PyMOL 1.8.2 untuk menghitung nilai RMSD dan visualisasi pose, serta R
statistical software 3.3.0 untuk analisis statistik.
Alat
Peralatan yang digunakan adalah server Fakultas Farmasi Universitas Sanata Dharma dengan nomor alamat IP 103.247.10.66 (pharcomp.web.id), HP V201TX dengan spesifikasi: prosesor Intel Core i5 5200U @2,2GHz, RAM 4,00 GB, 64-bit Operating System, Linux Ubuntu 14.04
3 Preparasi Ligan EN73
Berdasarkan inspeksi visual pada ko-kristal ligan EN73 pada kantung ikatan MMP-9 dengan menggunakan PyMOL 1.8.2, hanya terdapat rantai A. Kemudian dilakukan preparasi ligan dan protein menggunakan perangkat lunak SPORES dan menghasilkan luaran ligand.mol2 dan protein.mol2. File ligand.mol2 diubah menjadi ligand.smi kemudian diubah menjadi gen3d menjadi ligand.mol2 menggunakan Babel. File ligand.mol2 diprotonasi ulang menggunakan SPORES. Kemudian nama file diubah menjadi ligand_input.mol2
Penambatan Molekul Ligan EN73
Hasil luaran dari preparasi SPORES berupa ligand.mol2 ditambatkan dengan protein.mol2 menggunakan perangkat lunak PLANTS 1.2 (Korb et al. 2007) pada server dengan konfigurasi yang mengacu pada Anita et al. (2012). Setiap kali penambatan (1 run) dilakukan lima kali iterasi penambatan molekuler. Luaran dari setiap run adalah lima pose berupa skor ChemPLP yang kemudian diambil satu pose dengan nilai ChemPLP terbaik. Dilakukan 1000 kali run sehingga diperoleh data 1000 pose terbaik dari masing masing
run.
Konversi Seribu Pose Terbaik Menjadi PDB
Hasil 1000 pose terbaik dari masing-masing run berupa file .mol2 dikonversi menggunakan Babel menjadi file .pdb juga dihilangkan atom hidrogen.
Penyesuaian Koordinat Ligan
File ligand_input.mol2 dan ligand.mol2 diubah formatnya menjadi .pdb juga dihilangkan atom hidrogen. Koordinat atom-atom pada ligand_input.pdb disesuaikan dengan koordinat atom-atom pada ligand.pdb. Nama file ligand_input.pdb diubah menjadi ref.pdb
Perhitungan RMSD dan Visualisasi
Hasil penambatan berupa 1000 pose dengan skor ChemPLP terbaik kemudian dihitung nilai RMSD menggunakan peragkat lunak PyMOL 1.8.2. Perhitungan antara
4
berdasarkan nilai RMSD kemudian ditentukan pose median setiap klaster.
Perangkat lunak PyMOL 1.8.2 digunakan untuk memvisualisasikan interaksi ligan referensi, pose median klaster pertama (K1), pose median klaster kedua (K2) dan pose median klaster ketiga (K3) dengan residu pada kantung ikatan MMP-9.
Analisis Statistik
Data nilai RMSD yang didapat melalui perangkat lunak PyMOL 1.8.2 diuji statistik dengan metode one sample t-test untuk mengetahui apakah RMSD tidak lebih besar dari 2,0 Å menggunakan R statistical software 3.3.0.
HASIL DAN PEMBAHASAN
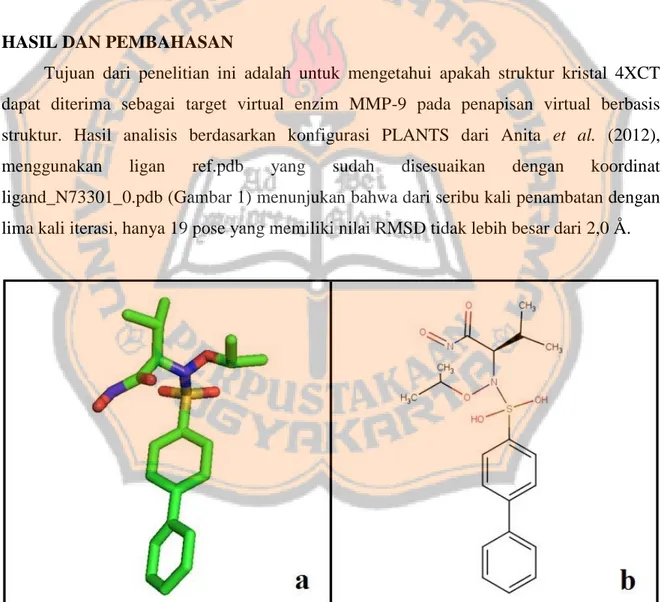
Tujuan dari penelitian ini adalah untuk mengetahui apakah struktur kristal 4XCT dapat diterima sebagai target virtual enzim MMP-9 pada penapisan virtual berbasis struktur. Hasil analisis berdasarkan konfigurasi PLANTS dari Anita et al. (2012), menggunakan ligan ref.pdb yang sudah disesuaikan dengan koordinat ligand_N73301_0.pdb (Gambar 1) menunjukan bahwa dari seribu kali penambatan dengan lima kali iterasi, hanya 19 pose yang memiliki nilai RMSD tidak lebih besar dari 2,0 Å.
Gambar 1. Gambar 3D setelah penyesuaian koordinat atom ligan referensi dengan ligan EN73 (a) beserta gambar 2D (b)
5
menghasilkan pose yang memiliki nilai RMSD tidak lebih dari 2,0 Å sebanyak 19 pose atau hanya 1,9%. Jumlah pose dengan nilai RMSD antara 2,1 Å - 4,0 Å sebanyak 940 pose atau 94% dan RMSD lebih dari 4,0 Å sebanyak 41 pose atau 4,1%. Hasil tersebut kurang baik karena lebih dari 90% pose memiliki RMSD di atas 2,0 Å.
Hasil data seribu pose terbaik dibagi menjadi 3 klaster (Gambar 2), klaster pertama dengan nilai RMSD tidak lebih besar dari 2,0 Å, klaster kedua dengan nilai RMSD antara 2,01 Å hingga 4,0 Å dan klaster ketiga dengan nilai RMSD lebih besar dari 4,0 Å dan setiap klasternya dihitung nilai median setiap klasternya (Tabel I). Setiap klaster diambil nilai median ditujukan untuk menggambarkan suatu nilai yang mewakili kumpulan data pada klaster tersebut. Nilai RMSD terendah dari seribu pose terbaik tersebut adalah 1,267 Å dan tertinggi 4,333 Å.
Gambar 2. Histogram seribu pose dengan tiga klaster
Tabel I. Jumlah pose dan median tiap klaster
Klaster 1 Klaster 2 Klaster 3
Jumlah Pose 19 940 41 Median 1,722 Å (pose ke 813) 3,475 Å (pose ke 2) 4,299 Å (pose 246)
Faktor yang menentukan ligan dan protein diterima sebagai protokol penapisan virtual berbasis struktur yaitu bila dapat menghasilkan mode ikatan yang reprodusibel
6
apabila menghasilkan nilai RMSD 2,0 Å (Marcou and Rognan 2007). Berdasarkan nilai RMSD hasil dari penambatan ulang, struktur kristal 4XCT tidak dapat diterima sebagai target PVBS karena konfigurasi PLANTS dari Anita et al. (2012) menghasilkan RMSD lebih besar dari 2,0 Å.
Ligan referensi memiliki empat ikatan hidrogen dengan residu Ala 189-CO, Leu 188-NH dan Glu 227-OH. Berdasarkan perhitungan jarak (Tabel II) interaksi antara ligan median setiap klaster dengan residu pada kantung ikatan MMP-9, terjadi kehilangan ikatan hidrogen. Ikatan hidrogen terjadi bila jarak antara donor dan aseptor tidak lebih dari 3,5 Å (Berg et al., 2002). Pose hasil penambatan klaster 1 (K1) hanya memiliki dua ikatan hidrogen sedangkan klaster 2 (K2) dan klaster 3 (K3) hanya memiliki satu ikatan hidrogen. Hilangnya ikatan hidrogen akan menghasilkan hasil prediksi skoring interaksi ligan-protein tidak baik (Zhou et al., 2012).
Tabel II. Jarak interaksi ligan dengan residu pada tiap pose
K1 K2 K3
Glu 227-OH H01-O - - -
H01-N - - -
Ala 189-CO O-H24 3,2 Å 3,2 Å 3,2 Å
Leu 188-NH H02-O 3,2 Å - -
Ikatan yang penting dan harus dipertahankan adalah ikatan hidrogen antara HO-sulfonamida dengan Leu 188-NH. Hal ini dibuktikan dari nilai RMSD K1 sebesar 1,722 Å karena adanya ikatan hidrogen dengan Ala 189-CO dan Leu 188-NH. Berbeda dengan K2 dan K3 dengan nilai RMSD 3,475 Å dan 4,299 Å, hanya terdapat ikatan hidrogen dengan Ala 189-CO. Hilangnya ligan dengan Leu 188-NH membuat nilai RMSD lebih besar dari 2,0 Å.
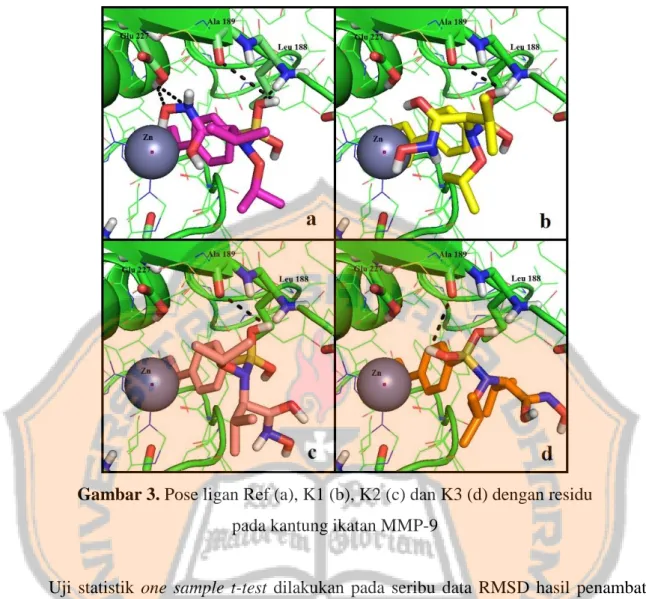
Visualisasi pose dilakukan dengan menggunakan perangkat lunak PyMOL. Hasil visualisasi menggambarkan interaksi ligan EN73 dengan residu yang terdapat di kantung ikatan MMP-9. Pose yang divisualisasikan (Gambar 3) yaitu pose ligan Ref (Gambar 3.a), median K1 (Gambar 3.b), median K2 (Gambar 3.c) dan median K3 (Gambar 3.d) dengan residu pada kantung ikatan MMP-9.
7
Gambar 3. Pose ligan Ref (a), K1 (b), K2 (c) dan K3 (d) dengan residu pada kantung ikatan MMP-9
Uji statistik one sample t-test dilakukan pada seribu data RMSD hasil penambatan untuk mengetahui apakah RMSD tidak lebih besar dari 2,0 Å menggunakan R statistical
software 3.3.0. Nilai p-value 2,2 x 10-16 sehingga dapat disimpulkan RMSD lebih besar
dari 2,0 Å.
KESIMPULAN
Kesimpulan dari penelitian ini adalah struktur kristal 4XCT tidak dapat diterima sebagai target virtual enzim MMP-9 pada penapisan virtual berbasis struktur berdasarkan konfigurasi PLANTS dari Anita et al. (2012) karena menghasilkan RMSD lebih besar dari 2,0 Å. Saran untuk penelitian selanjutnya adalah perlu dilakukan pengembangan pada konfigurasi PLANTS oleh Anita et al. (2012) agar dapat digunakan untuk penapisan virtual berbasis struktur enzim MMP-9.
8
American Diabetes Association, 2014. Diagnosis and Classification of Diabetes Mellitus,
Diabetes Care, 37(1), 81-90.
Anita, Y., Radifar, M., Kardono, L.B., Hanafi, M., and Istyastono, E.P., 2012. Structure-Based Design Of Eugenol Analogs As Potential Estrogen Receptor Antagonists.
Bioinformation, 8 (819), 901–906.
Barril, X., Hubbard, R.E., and Morley, S.D., 2004. Virtual Screening in Structure-Based Drug Discovery. Mini-Reviews in Medicinal Chemistry, 4, 779–791.
Berg, Jeremi M., Tymoczko, Jhon L., and Stryer L., 2002. Biochemistry Fifth Edition. W.H. Freeman and Company.
Bissantz, C., Folkers, G., and Rognan, D., 2000. Protein-Based Virtual Screening of Chemical Databases. 1. Evaluation of Different Docking / Scoring Combinations.
Nature, 43 (25), 4759–4767.
Brink, T. Ten and Exner, T.E., 2009. Influence of protonation, tautomeric, and stereoisomeric states on protein-ligand docking results. Journal of Chemical
Information and Modeling, 49, 1535-1546.
Cavasotto, C.N. and Orry, A.J., 2007. Ligand Docking And Structure-Based Virtual Screening In Drug Discovery. Current Topics in Medicinal Chemistry, 7 (10), 1006– 1014.
Guariguata, L., Whiting, D.R., Hambleton, I., Beagley, J., Linnenkamp, U., and Shaw, J.E., 2014. Global Estimates Of Diabetes Prevalence For 2013 And Projections for 2035.
Diabetes Research and Clinical Practice, 103 (2), 137–149.
Hamed, S., Bennett, C.L., Demiot, C., Ullmann, Y., Teot, L., and Desmoulière, A., 2014. Erythropoietin, a novel repurposed drug: An innovative treatment for wound healing in patients with diabetes mellitus. Wound Repair and Regeneration, 22 (1), 23–33. Ince, P., Game, F.L., and Jeffcoate, W.J., 2007. Rate of Healing of Neuropathic Ulcers of
The Foot in Diabetes and Its Relationship to Ulcer Duration And Ulcer Area.
Diabetes Care, 30 (3), 660–663.
Korb, O., Stutzle, T., and Exner, T.E., 2009. Empirical Scoring Functions For Advanced Protein-Ligand Docking with PLANTS. Journal of Chemical Information and
Modeling, 49 (1), 84–96.
Lionta, E., Spyrou, G., Vassilatis, D.K., and Cournia, Z., 2014. Structure-Based Virtual Screening for Drug Discovery: Principles, Applications and Recent Advances.
Current Topics in Medicinal Chemistry, 14 (16), 1923–38.
Lobmann, R., Ambrosch, A., Schultz, G., Waldmann, K., Schiweck, S., and Lehnert, H., 2002. Expression of Matrix-Metalloproteinases and Their Inhibitors in The Wounds of Diabetic and Non-Diabetic Patients. Diabetologia, 45 (7), 1011–1016.
Marcou, G. and Rognan, D., 2007. Optimizing Fragment and Scaffold Docking by Use of Molecular Interaction Fingerprints. Journal of Chemical Information and Modeling, 47 (1), 195–207.
Mihardja, L., Delima, Siswoyo, H., Ghani, L. and Soegondo, S., 2009. Prevalence and Determinants of Diabetes Mellitus and Impaired Glucose Tolerance in Indonesia.
Acta Medica Indonesiana, 41, 169-74.
Mohan, R., Chintala, S.K., Jung, J.C., Villar, W.V.L., McCabe, F., Russo, L.A., Lee, Y., McCarthy, B.E., Wollenberg, K.R., Jester, J. V., Wang, M., Welgus, H.G., Michael Shipley, J., Senior, R.M., and Elizabeth Fini, M., 2002. Matrix Metalloproteinase Gelatinase B (MMP-9) Coordinates and Effects Epithelial Regeneration. Journal of
9
and Rossello, A., 2015. N-O-Isopropyl Sulfonamido-Based Hydroxamates as Matrix Metalloproteinase Inhibitors: Hit Selection and in Vivo Antiangiogenic Activity.
Journal of Medicinal Chemistry, 58 (18), 7224–7240.
Shaw, J.E., Sicree, R.A., and Zimmet, P.Z., 2010. Global Estimates of The Prevalence of Diabetes For 2010 and 2030. Diabetes Research and Clinical Practice, 87 (1), 4–14. Singh, N., Armstrong, D.G., and Lipsky, B.A., 2015. Preventing Foot Ulcers in Patients
With Diabetes. The Journal of the American Medical Association (JAMA), 8 (2), 217– 228.
Soewondo, P., Ferrario, A., and Tahapary, D.L., 2013. Challenges in Diabetes Management in Indonesia: A Literature Review. Globalization and Health, 9 (63), 1-17.
Verdonk, M.L., Berdini, V., Hartshorn, M.J., Mooij, W.T.M., Murray, C.W., Taylor, R.D., and Watson, P., 2004. Virtual Screening Using Protein - Ligand Docking : Avoiding Artificial Enrichment. Journal of Chemical Information and Modeling, 44(3), 793– 806.
Zhou, W., Yan, H., Hao, Quan., 2012. Analysis Of Surface Structures Of Hydrogen Bonding In Protein–Ligand Interactions Using The Alpha Shape Model. Chemical
10
11
> wget https://files.rcsb.org/download/4XCT.pdb.gz > gunzip 4XCT.pdb.gz
> /home/enade/program/PLANTS/SPORES1.3 --mode splitpdb 4XCT.pdb
> /home/enade/program/PLANTS/PLANTS1.2 --mode bind ligand_N7301_0.mol2 5 protein.mol2
> cp
/home/enade/program/master_ER/Bioinformation_Anita2012_ER -ANT/validated/WAT/VS/plants.config .
> grep -EV bindingsite_plants.config | grep -Ev water > plants.config.tmp
> cat plants.config.tmp bindingsite.def > plants.config > mv ligand.mol2 ligand_input.mol2
> babel -imol2 ligand_input.mol2 -osmi ligand_input.smi > babel --gen3d -ismi ligand_input.smi -omol2
ligand_input_babel.mol2
> /home/enade/program/PLANTS/SPORES1.3 --mode reprot ligand_input_babel.mol2 ligand_input.mol2
12 File plants.config
# scoring function and search settings scoring_function chemplp search_speed speed2 # input protein_file protein.mol2 ligand_file ligand_input.mol2 # output output_dir results
# write single mol2 files (e.g., for RMSD calculation) write_multi_mol2 0
# cluster algorithm cluster_structures 50 cluster_rmsd 1.0
# binding site definition
bindingsite_center 17.8523 -17.7221 19.1884 bindingsite_radius 7.1593 File file.sh #!/bin/sh alias plants='/home/enade/program/PLANTS/PLANTS1.2' mkdir /home/donnysuparto/pilot/dockingbaru mkdir /home/donnysuparto/pilot/dockingbaru/mmp9 cd /home/donnysuparto/pilot/dokingbaru/ for i in $(seq 1 100); do mkdir /home/donnysuparto/pilot/dockingbaru/mmp9/replikasi_$i cd /home/donnysuparto/pilot/dockingbaru/mmp9/replikasi_$i/ for j in $(seq 1 5); do mkdir dock_$j cd dock_$j/ cp /home/donnysuparto/pilot/plants.config . cp /home/donnysuparto/pilot/protein.mol2 . cp /home/donnysuparto/pilot/ligand_input.mol2 . plants --mode screen plants.config
rm plants.config protein.mol2 ligand_input.mol2 cd ../
done
cd ../../ done
13 > for j in $(seq 1 1000);
do for i in $(seq 1 5); do echo "$i `cat
/home/donny/home/donny/skripsi1000/datadocking1000/mmp9/ replikasi_$j/dock_$i/results/bestranking.csv`,$i";
done | grep -Ev TOTAL_SCORE | sort -t, -k2n | head -1 | awk -F, '{print $12}';
14 > best1000=($(cat best1000.lst )) > n=${#best1000[@]} > for ((j=0; j<=$n; j++)); do echo"cp/home/donny/home/donny/skripsi1000/datadocking100 0/mmp9/replikasi_$[j+1]/dock_${best1000[$j]}/results/4XC T_ligand_0_entry_00001_conf_01.mol2 replikasi_$[j+1].${best1000[$j]}.mol2"; done > copy1000.sh
> chmod u+x copy1000.sh > ./copy1000.sh
15 > mkdir rmsd.pilot
> cd rmsd.pilot
> ls ../best/bestpdb/ | grep replikasi | sort t_ -k2,2n > hasil.pdb.all
> cp ../best/bestpdb/ligand_ref.pdb ref.pdb > gedit all.rmsd.sh &
#!/bin/sh
for i in $(cat hasil.pdb.all); do
cp ../best/bestpdb/$i res.pdb
pymol -c rmsd.pml | grep RMS | awk '{print $4}' > $i.rmsd done rm res.pdb > gedit rmsd.pml & load ref.pdb load res.pdb rms_cur ref,res quit
> chmod u+x all.rmsd.sh > ./all.rmsd.sh
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done (menampilkan 1000 rmsd)
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done | sort -n
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done | awk '{if ($1 <= 2.0) print $0}'
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done | awk '{if ($1 <= 2.0) print $0}' | wc -l
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done > rmsdhistoori.csv
> for i in $(cat hasil.pdb.all); do cat $i.rmsd; done | sort -n > rmsdhisto.csv
16
Penulis skripsi dengan judul “Evaluasi Struktur Kristal 4xct Sebagai Target Virtual Enzim Matrix Metalloproteinase 9 Pada Penapisan Virtual Berbasis Struktur” memiliki nama lengkap Donny Suparto. Penulis lahir di Jember pada tanggal 7 Februari 1995. Pendidikan formal yang ditempuh oleh penulis adalah TK Kartika XII, Jember (2000-2001), SD Negeri Bintoro 1, Jember (2001-2007), SMP Katolik Santo Petrus, Jember (2007-2010), dan SMA Katolik Santo Paulus, Jember (2010-2013). Pada tahun 2013 penulis melanjutkan pendidikan ke jenjang yang lebih tinggi di Fakultas Farmasi Universitas Sanata Dharma Yogyakarta.
Selama menjalani pendidikan di Fakultas Farmasi Universitas Sanata Dharma Yogyakarta, penulis pernah aktif di beberapa kegiatan, yaitu Panitia TITRASI 2014 divisi Dana Dan Usaha, Ketua Panitia Pelepasan Wisuda 2 tahun 2014, koordinator divisi Pengabdian Masyarakat BEM Fakultas Farmasi Universitas Sanata Dharma Yogyakarta periode 2015/2016. Penulis juga pernah berperan aktif sebagai asisten praktikum Kimia Dasar.