EVALUASI PERBANDINGAN NILAI Ct (Cycle threshold) TERHADAP PEMERIKSAAN SARS-CoV-2 MENGGUNAKAN METODE POOLED TEST DI PROVINSI SUMATERA UTARA
SKRIPSI
NUR UTAMI 170805026
PROGRAM STUDI BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM UNIVERSITAS SUMATERA UTARA
MEDAN 2021
EVALUASI PERBANDINGAN NILAI Ct (Cycle threshold) TERHADAP PEMERIKSAAN SARS-CoV-2 MENGGUNAKAN METODE POOLED TEST DI PROVINSI SUMATERA UTARA
SKRIPSI
Diajukan untuk melengkapi tugas dan memenuhi syarat mencapai gelar Sarjana Sains
NUR UTAMI 170805026
PROGRAM STUDI BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM UNIVERSITAS SUMATERA UTARA
MEDAN 2021
PERNYATAAN ORISINALITAS
EVALUASI PERBANDINGAN NILAI Ct (Cycle threshold) TERHADAP PEMERIKSAAN SARS-CoV-2 MENGGUNAKAN METODE POOLED TEST DI PROVINSI SUMATERA UTARA
SKRIPSI
Saya menyatakan bahwa skripsi ini adalah hasil karya saya sendiri, kecuali beberapa kutipan dan ringkasan yang masing-masing disebutkan sumbernya.
Medan, Desember 2021
Nur utami
170805026
ii
EVALUASI PERBANDINGAN NILAI Ct (Cycle threshold) TERHADAP PEMERIKSAAN SARS-CoV-2 MENGGUNAKAN METODE POOLED TEST DI PROVINSI SUMATERA UTARA
ABSTRAK
Severe Acute Respiratory Syndrome Coronavirus-2 (SARS-CoV-2) merupakan jenis coronavirus baru yang menyebabkan pneumonia yaitu kerusakan pada pernapasan ringan maupun berat. Nama penyakit yang disebabkan oleh virus ini adalah Coronavirus Disease-2019 (COVID-19). Transmisi virus ini yang mudah menyebar termasuk di Sumatera utara menyebabkan penularan meningkat dari waktu ke waktu, sehingga perlu dilakukan deteksi pemeriksaan secara molekuler. Salah satu strategi percepatan dalam pemeriksaan virus ini adalah dilakukan penyederhanaan pengerjaan sampel menggunakan metode pooled test. Metode ini dilakukan dengan menggabungkan beberapa sampel sesuai dengan jumlah yang telah di tentukan.
Tujuan penelitian ini adalah untuk mengetahui pengaruh perubahan nilai Ct (Cycle threshold) terhadap perbandingan sampel pooled test. Penelitian ini dilakukan dengan pengambilan sampel swab massal terhadap masyarakat Sumatera Utara di Balai Teknik Kesehatan Lingkungan Pengendalian Penyakit Medan dengan menggunakan perbandingan Pooled 1:4, 1:6 dan 1:9 berdasarkan viral load (high, intermediet dan low). Virus ini dideteksi dengan metode Reverse Transcription- Polymerase Chain Reaction (RT-PCR). Data di analisis menggunakan uji Chi- Square dengan nilai signifikansi p<0,05. Hasil dari penelitian ini didapatkan perubahan nilai Ct pada sampel high viral load dan intermediet viral load dan adanya perubahan yang sangat signifikan terhadap perbandingan sampel pada low viral load dimana sampel berubah hasil positif menjadi negatif. Perubahan nilai Ct yang terjadi sebesar 0,26-0,5 (high viral load), 0,3-0,5 (intermediet viral load) dan perubahan sampel positif menjadi negatif (low viral load). Perubahan hasil pada sampel low viral load menimbulkan ancaman bagi masyarakat yang dapat terinfeksi terutama pada sampel yang tidak terdeteksi.
Kata kunci: pooled test, reverse transcription polymerase chain reaction (RT-PCR), SARS-CoV-2
EVALUATION OF COMPARISONAL Ct VALUE (Cycle threshold) TO WARDS SARS-CoV-2 USING METHOD POOLED TEST IN
PROVINCE OF NORTH SUMATRA
ABSTRACT
Severe Acute Respiratory Syndrome Coronavirus-2 (SARS-CoV-2) is a new type of coronavirus causes pneumonia, which causes mild or severe respiratory damage. The name of the disease caused by this virus is Coronaviurs Disease-2019 (COVID-19). The transmission of this virus is easy to spread, including in North Sumatra, causes transmission to increase from time to time. Therefore it is necessary to detect the disease by molecular tests. One of the accelerated strategies in examining this virus is to simplify sample processing using the pooled test method.
This method is done by combining several samples in accordance with a predetermined amount. The purpose of this study was to determine the effect of changes in the value of Ct (Cycle threshold) on the comparison of samples pooled test. This research was conducted by taking mass swab samples from the people of North Sumatra at the Center for Environmental Health Engineering and Disease Control of Medan using a ratio of pooled 1:4, 1:6 and 1:9 based on viral load (high, intermediate and low). This virus was detected by the Reverse Transcription- Polymerase Chain Reaction (RT-PCR) method. The data were analyzed using the Chi-Square test with a significance value of p<0.05. The results of this study showed a change in the Ct value in the samples of high viral load and intermediate viral load and a very significant change in the comparison of samples at low viral load where the sample changed from positive to negative. Changes in Ct values that occurred were 0.26-0.5 (high viral load), 0.3-0.5 (intermediate viral load) and changes in positive samples to negative (low viral load). Changes in the results in samples with low viral load pose a threat to the public who can be infected, especially in undetected samples.
Keywords: pooled test, reverse transcription polymerase chain reaction (RT-PCR), SARS-CoV-2
iv
PENGHARGAAN
Puji dan syukur terhadap Allah Subhanahu Wa Ta’ala yang telah memberikan rahmat dan hidayah-Nya. Shalawat beriringkan salam kepada Rasulullah Salallahualaihi Wassalam beserta keluarga dan para sahabatnya yang senantiasa mengembangkan ajaran dan ilmu tauhid pada seluruh umatnya. Serta rasa terima kasih kepada diri sendiri yang semangat dan pantang menyerah dalam penulisan hasil penelitian ini yang berjudul “Evaluasi Perbandingan Nilai Ct (Cycle threshold) Terhadap Pemeriksaan SARS-CoV-2 Menggunakan Metode Pooled Test di Provinsi Sumatera Utara”.
Terimakasih dan rasa sayang sebesar-besarnya kepada Ayahanda tercinta Alik Supriadi dan Ibunda tercinta Sri Lestari yang telah membesarkan, mendidik, memberi motivasi, semangat dan nasihat serta rasa sayang yang teramat besar dan senantiasa mendoakan penulis. Terima kasih dan rasa cinta kepada adik Bima wardana selaku teman mengadu yang selalu mendukung dan memberi semangat kepada penulis.
Dalam menyelesaikan penulisan hasil penelitian ini, penulis telah dibantu oleh banyak pihak. Dengan tidak mengurangi rasa hormat, penulis menyampaikan rasa terima kasih kepada Ibu Dra. Deny Supriharti, M.Sc selaku Dosen Pembimbing Tugas Akhir yang telah banyak membantu serta membagi waktunya untuk memberikan arahan, ilmu serta dukungan hingga penulis dapat menyelesaikan tugas akhir. Penulis mengucapkan terimakasih kepada Ibu Dr. Saleha Hannum, S.Si, M.Si selaku dosen penguji I dan Bapak Prof. Dr. Syafruddin Ilyas M.Biomed selaku dosen penguji II yang telah memberikan masukan, saran dan motivasi yang sangat berperan penting dalam penulisan hasil penelitian ini.
Penulis juga mengucapkan terima kasih kepada Ibu Dr. Liana Dwi Srihastuti S.Si, M.Si selaku Dosen Penasehat Akademik yang telah memberikan motivasi dan bimbingan semasa perkuliahan hingga selesai. Terima kasih penulis ucapkan kepada Ibu Dr. Yurnaliza, M.Si selaku ketua program studi Biologi yang telah meluangkan waktu untuk memberikan motivasi bagi para penulis semester akhir untuk tidak menyerah dalam menjenjang sarjana S.Si. Dan juga seluruh dosen program studi Biologi serta para staf yang telah memberikan ilmu pengetahuan serta membantu
proses perkuliahan hingga saat ini.
Terima kasih penulis ucapkan kepada Ibu Dr. R. Lia Kusumawati, M.
Biomed, Sp.MK(K), Ph.D selaku penanggung jawab laboratorium PCR COVID-19 Mikrobiologi Fakultas Kedokteran Universitas Sumatera Utara dan abang Mirzan Hasibuan, S.Si, M.Si, selaku pembimbing dan panutan yang telah berperan penting bagi penulis selama berada di laboratorium PCR COVID-19 Mikrobiologi Fakultas Kedokteran Universitas Sumatera Utara. Serta seluruh staff Laboratorium Mikrobiologi Fakultas Kedokteran yang tidak dapat disebutkan satu persatu, namun peran dan bantuan abang, kakak dan teman teman yang telah banyak membantu dan mengarahkan penulis selama penelitian.
Terima kasih juga penulis ucapkan kepada rekan seperjuangan BIOCHROME 2017 dan Ekstraksi Kita (Kak Rahmi, Laura Agnes, Sheila Syahdani Lubis, Rizkannisa, Nisrina Tari dan Ikhsan Zulfani) dan para Asisten Laboratorium Genetika dan Biologi Molekuler yang tidak memungkinkan untuk disebut satu persatu, namun semangat dan motivasi yang diberikan kepada penulis sangat berharga dalam penyusunan hasil penelitian ini. Terimakasih dan rasa syukur kepada teman-teman sepenelitian Rizkannisa dan Sheila syahdani yang bersedia berjalan bersama-sama hingga akhir dari penelitian ini. Dan teman tercinta Dila Syindi Marpaung, Kak Indah Khairani Putri, Kak Ira Elviana, Kak Eka Nurdiana, Siti Khadijah Marpaung dan Leni Kartika yang telah menemani penulis dari masa perkuliahan hingga saat ini.
Penulis menyadari bahwa hasil penelitian ini masih jauh dari kesempurnaan, sehingga diharapkan kritik dan saran yang membangun demi kesempurnaan laporan penelitian ini. Demikian yang dapat penulis sampaikan, semoga penelitian ini dapat bermanfaat bagi perkembangan ilmu pengetahuan. Atas perhatiannya, penulis ucapkan terima kasih.
Medan, Desember 2021
Nur utami
vi DAFTAR ISI
Halaman
PENGESAHAN SKRIPSI i
ABSTRAK ABSTRACT
ii iii
PENGHARGAAN iv
DAFTAR ISI vi
DAFTAR TABEL viii
DAFTAR GAMBAR ix
DAFTAR LAMPIRAN DAFTAR SINGKATAN
x xi BAB I PENDAHULUAN
1.1 Latar Belakang 1
1.2 Rumusan Masalah 2
1.3 Hipotesis 3
1.4 Tujuan Penelitian 3
1.5 Manfaat Penelitian 3
BAB 2 TINJAUAN PUSTAKA
2.1 Coronavirus 4
2.2 Karakteristik SARS-CoV-2 4
2.3 Epidemologi SARS-CoV-2 6
2.4 Metode Pemeriksaan SARS-CoV-2 7
2.4.1 Gabungan Sampel 8
2.4.2 Reverse Transcription-Polymerase Chain Reaction (RT-PCR)
9
2.5 Nilai Ct (Cycle threshold) 9
BAB 3 METODE PENELITIAN
3.1 Waktu dan Tempat 11
3.2 Rancangan Penelitian 11
3.3 Prosedur Penelitian 11
3.3.1 Sampel Individu 11
3.3.1.1 Ekstraksi RNA Individu 11
3.3.1.2 Amplifikasi cDNA Individu 12
3.3.1.3 Interpretasi Nilai Ct 13
3.3.2 Sampel Pooled Test 13
3.3.2.1 Ekstraksi RNA Pooled Test 13 3.3.2.2 Amplifikasi cDNA Pooled Test 14
3.3.2.3 Interpretasi Nilai Ct 14
3.4 Analisis Data 15
BAB 4 HASIL DAN PEMBAHASAN
4.1 Hasil Nilai Ct Individual 16
4.2 Nilai Ct pada Sampel Individu dan Sampel Pooled test 18 4.3 Hubungan Tingkat Viral load dan Sampel Pooled test dengan
Nilai Ct pada RT-PCR
19 4.3.1 Hubungan Antara Tingkat Viral load dengan Nilai Ct
pada RT-PCR
20 4.3.2 Hubungan Antara Sampel Pooled test dengan Nilai Ct
RT-PCR
20 4.4 Perbedaan Nilai Cycle threshold (Ct) pada Perbandingan
Pooled test
21 4.4.1 Perbandingan Cycle threshold (Ct) pada Sampel
Pooled test Berdasarkan High Viral Load
21 4.4.2 Perbandingan Cycle threshold (Ct) pada Sampel
Pooled test Berdasarkan Intermediet Viral Load
22 4.4.3 Perbandingan Cycle threshold (Ct) pada Sampel
Pooled test Berdasarkan Low Viral Load
24
BAB 5 KESIMPULAN DAN SARAN
5.1 Kesimpulan 27
5.2 Saran 27
DAFTAR PUSTAKA 28
LAMPIRAN 31
viii DAFTAR TABEL
Nomor
Tabel Judul Halaman
3.1 Komposisi master mix RT-PCR 12
3.2 Program RT-PCR untuk mendeteksi SARS-CoV-2 12
3.3 Interpretasi nilai Ct sampel individu 13
3.4 Komposisi master mix RT-PCR 14
3.5 Program RT-PCR untuk mendeteksi SARS-CoV-2 14
3.6 Interpretasi nilai Ct sampel pooled test 15
3.7 Batas cut-off nilai Ct 15
4.1 Hasil nilai Ct sampel individu dan interpretasi hasil positif dan negatif
16 4.2 Nilai Ct pada sampel individu dan sampel pooled test 18 4.3 Hubungan antara tingkat viral load dengan nilai Ct pada
RT-PCR
20 4.4 Hubungan antara sampel pooled test dengan nilai Ct
pada RT-PCR
20 4.5 Perbedaan nilai Ct pada sampel pooled test dengan high
viral load
21 4.6 Perbedaan nilai Ct pada sampel pooled test dengan
intermediet viral load
23 4.7 Perbedaan nilai Ct pada sampel pooled test dengan low
viral load
24
DAFTAR GAMBAR
Nomor
Gambar Judul Halaman
4.1
Perbandingan pooled test 1:4 (1), perbandingan pooled test 1:6 (2), dan perbandingan pooled test 1:9(3) dengan nilai Ct (high viral load).
22
4.2
Perbandingan pooled test 1:4 (1), perbandingan pooled test 1:6 (2), dan perbandingan pooled test 1:9(3) dengan nilai Ct (intermediet viral load).
23
4.3
Perbandingan pooled test 1:4 (1), perbandingan pooled test 1:6 (2), dan perbandingan pooled test
1:9(3) dengan nilai Ct (low viral load). 25
x DAFTAR LAMPIRAN
Nomor Lampiran
Judul Halaman
1. Hubungan antara tingkat viral load dengan nilai Ct pada RT-PCR dengan uji Chi-Square
31 2. Hubungan antara sampel pooled test dengan nilai Ct
pada RT-PCR dengan uji Chi-Square
32
DAFTAR SINGKATAN
ACE2 = Angiotensin-Converting Enzyme-2 cDNA = complementery Deoxyribonucleic Acid COVID-19 = Coronavirus Disease 2019
Ct = Cycle threshold
PCR = Polymerase Chain Reaction
RNA = Ribonucleic Acid
RT-PCR = Reverse Transcription Polymerase Chain Reaction SARS-CoV-2 = Severse Acute Respiratory Syndrome Coronavirus -2
1
BAB 1 PENDAHULUAN
1.1 Latar Belakang
Penyebaran wabah virus corona baru telah menggemparkan dunia karena menjadi wabah pandemi. World Health Organization menetapkan virus baru tersebut Severe Acute Respiratory Syndrome Coronavirus-2 (SARS-CoV-2) dan nama penyakitnya disebut sebagai Coronavirus Disease 2019 (COVID-19) (Yuliana, 2020). SARS-CoV-2 merupakan jenis coronavirus baru yang menyerang saluran pernapasan pada manusia. Virus tersebut bersifat contagious, dimana virus menular dan menyebar secara cepat ke dalam sel inang. Virus SARS-CoV-2 memiliki tingkat transmisi yang tinggi dibandingkan dengan jenis virus corona lainnya. Penyebaran SARS-CoV-2 terus meningkat di seluruh dunia termasuk Indonesia. Berdasarkan laporan Kementerian Kesehatan Republik Indonesia, pada tanggal 15 Mei 2021 tercatat kasus konfirmasi 4.247.320, kasus meninggal 143.519 dan kasus sembuh 4.091.938 (BNPB, 2021). Dan data tersebut masih menunjukkan adanya peningkatan penyebaran SARS-CoV-2.
Peningkatan kasus infeksi SARS-CoV-2 terjadi dalam kurun waktu yang cepat sehingga dibutuhkan penanganan dan pencegahan penularan virus tersebut.
Oleh karena itu, pemerintah menerapkan pembatasan sosial dan peningkatan kesehatan masyarakat untuk mengendalikan penyebaran virus (Nugroho dkk, 2020).
Salah satu cara pengendalian penyebaran virus dengan dilakukannya pemeriksaan laboratorium yang berperan penting dalam mendiagnosis seseorang terkait infeksi SARS-CoV-2. Saat ini, gold standart pemeriksaan laboratorium yang digunakan untuk mendeteksi keberadaan SARS-CoV-2 adalah Real Time-PCR (RT-PCR).
Pengujian diagnosis RT-PCR digunakan untuk mendeteksi keberadaan SARS- CoV-2 dengan cara memperbanyak materi genetik virus di dalam sampel.
Namun, konfirmasi infeksi terus meningkat termasuk di Provinsi Sumatera Utara terutama pada pasien tanpa gejala, sehingga perlu dilakukan strategi peningkatan kapasitas pemeriksaan SARS-CoV-2 guna mempercepat dalam penanganan dan pencegahan secara merata di seluruh wilayah. Salah satu teknik yang dilakukan adalah dengan pengujian sampel gabungan (pooled test) (Deckert et al, 2020).
2
Penggabungan sampel (pooled test) dalam satu tabung dari beberapa orang dapat meningkatkan kapasitas diagnostik untuk mendeteksi SARS-CoV-2 saat angka tes tidak memenuhi kebutuhan di tempat-tempat tertentu. Pada prinsipnya deteksi gabungan (pooled test) dapat ditentukan jika hasil deteksi gabungan negatif, maka seluruh spesimen di dalam gabungan dianggap negatif. Sebaliknya, jika hasil gabungan (pooled test) positif, maka perlu dilakukan ekstraksi ulang hingga diketahui individu yang positif. Hasil deteksi sampel positif tersebut dilanjutkan secara individual untuk mengkonfirmasi spesimen yang positif (WHO, 2020).
Hasil pemeriksaan akhir dari pengujian diagnosis SARS-CoV-2 dapat dinyatakan positif bila terdapat nilai Ct (Cycle threshold). Nilai Ct pada RT-PCR menunjukkan jumlah siklus amplifikasi yang diperlukan gen target agar dapat melewati batas ambang (threshold). Korelasi antara nilai Ct dan SARS-CoV-2 menunjukkan bahwa semakin rendah nilai Ct dalam suatu sampel maka semakin banyak virus yang terdeteksi di dalam sampel tersebut dan sebaliknya, semakin tinggi nilai Ct dalam suatu sampel maka semakin sedikit jumlah virus yang terdeteksi di dalam sampel (Rao et al, 2020).
Hasil diagnosis berdasarkan nilai Ct dan penggunaan metode pooled test sangat penting dalam pemeriksaan laboratorium. Beberapa laboratorium di Indonesia termasuk di Laboratorium Universitas Andalas dan Laboratorium Mikrobiologi Universitas Sumatera Utara serta beberapa negara-negara di dunia telah menggunakan metode tersebut guna meningkatkan pengujian SARS-CoV-2 dengan waktu yang efektif dan penggunaan lebih sedikit reagen serta hasil yang lebih cepat.
Akan tetapi, sejauh ini belum ada evaluasi akurasi mengenai perbandingan nilai Ct dengan menggunakan metode pooled test termasuk di Sumatera Utara. Hal yang sama dilakukan pada penelitian sebelumnya oleh Sawicki et al., (2020) yaitu dalam strategi pooled sampel, kemungkinan ada resiko kehilangan kasus positif rendah dikarenakan viral load dari kasus positif yang diencerkan dengan peningkatan volume dalam sampel yang telah dikumpulkan (pooled). Oleh karena itu, perlu dilakukan penelitian ini untuk mengevaluasi adanya perubahan keakuratan dari perbandingan nilai Ct tersebut dengan metode pooled test terutama di Sumatera Utara, sehingga metode ini layak untuk direkomendasikan pada beberapa laboratorium dalam pemeriksaan SARS-CoV-2.
3
1.2 Rumusan Masalah
Infeksi virus SARS-CoV-2 memiliki tingkat penyebaran yang cepat termasuk di Sumatera Utara. Salah satu strategi percepatan pemeriksaan sampel yang dilakukan yaitu dengan menggunakan metode pooled test. Berdasarkan latar belakang yang telah diuraikan di atas, maka rumusan masalah dalam penelitian ini adalah bagaimana perbandingan nilai Ct terhadap pemeriksaan SARS-CoV-2 dengan menggunakan metode pooled test.
1.3 Hipotesis
Hipotesis pada penelitian ini adalah:
a. Tidak terdapat perubahan yang signifikan terhadap nilai Ct berdasarkan high viral load dan intermediet viral load pada pemeriksaan SARS-CoV-2 dengan menggunakan metode pooled test.
b. Terdapat perubahan yang signifikan terhadap nilai Ct berdasarkan low viral load pada pemeriksaan SARS-CoV-2 dengan menggunakan metode pooled test.
1.4 Tujuan Penelitian
Penelitian ini bertujuan untuk menganalisis perbandingan nilai Ct (Cycle threshold) terhadap sampel pooled test guna peningkatan kapasitas pemeriksaan SARS-CoV-2.
1.5 Manfaat Penelitian
Manfaat penelitian ini dapat meningkatkan kapasitas pemeriksaan SARS- CoV-2 dengan metode pooled test sehingga dapat direkomendasikan ke seluruh Laboratorium penanganan COVID-19.
4
BAB 2
TINJAUAN PUSTAKA
2.1 Coronavirus
Coronavirus merupakan salah satu famili virus yang dapat menyerang saluran pernapasan. Nama virus ini berasal dari kata Latin “Corona” yang berarti mahkota, karena bagian luar virus ini runcing seperti mahkota. Secara umum, coronavirus diklasifikasikan ke dalam empat genus utama yang diantaranya Alphacoronavirus, Betacoronavirus, Gammacoronavirus dan Deltacoronavirus (Amin dkk, 2020).
Diantaranya terdapat tujuh jenis coronavirus yang secara spesifik dapat menyerang pernapasan manusia seperti Human Coronavirus 22gE, Human Coronavirus-NL63, Human Coronavirus OC43, Human Coronavirus-HKU1, SARS-CoV-1 (Severe Acute Respiratory Syndrome Coronavirus-1), MERS-CoV (Middle East Respiratory Syndrome Coronavirus) dan SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus-2) (Chohan et al., 2020).
Coronavirus termasuk dalam golongan virus RNA yang paling banyak menginfeksi hewan dan manusia. Virion-virion virus ini mengandung single-strand positive sense RNA (ssRNA) terbesar yang membedakannya dengan virus RNA lainnya. Coronavirus ditandai dengan keragaman genetik yang tinggi akibat dari mutasi dan rekombinasi yang menyebabkan munculnya virus baru. Selain itu, coronavirus memiliki sifat zoonosis yang artinya dapat menular dari hewan ke manusia. Salah satu contohnya adalah SARS-CoV (Severe Acute Respiratory Syndrome Coronavirus) dan MERS-CoV (Middle East Respiratory Syndrome Coronavirus) (Ridwan, 2020).
2.2 Karakteristik SARS CoV-2
SARS-CoV-2 (Severe Acute Respiratory Syndrome Coronavirus strain-2) merupakan virus penyebab penyakit Coronavirus Disease 2019 (COVID-19). SARS- CoV-2 diklasifikasikan ke dalam genus Betacoronavirus dari famili Coronaviridae.
Virus ini bersifat monopartit atau asam nukleat untai tunggal dengan genom 29.903 nukleotida yang ditetapkan sebagai genom virus RNA terbesar yang telah diketahui (Mashuri dkk, 2020). Genom tersebut memiliki kode berupa Spike (S) glikoprotein,
5
Envelope (E) glikoprotein, Membrane (M) glikoprotein, Nucleocapsid (N) glikoprotein dan beberapa protein tambahan (WHO, 2020).
Menurut Prastyowati (2020), berikut taksonomi SARS-CoV-2 yang dijelaskan dalam jurnal Nature Microbiology yaitu sebagai berikut:
Kingdom : Riboviria
Ordo : Nidoverales
Subordo : Cornidovirineae Famili : Coronaviridae Subfamili : Orthocoronavirinae Genus : Betacoronavirus Subgenus : Sarbecovirus
Spesies : Severe acute respiratory syndrome coronavirus strain ke -2 SARS-CoV-2 termasuk genus Betacoronavirus yang berbentuk pleomorfik (tidak beraturan). Virus ini sensitif terhadap panas dan sinar ultraviolet serta dapat di nonaktifkan dengan desinfektan kecuali klorheksidin. Oleh karena itu, bahan klorheksidin tidak direkomendasikan sebagai sterilisasi tangan. Beberapa penyakit yang disebabkan oleh SARS-CoV-2 berupa penyakit pada saluran pernapasan, pencernaan dan sistem saraf pusat pada hewan dan manusia. Namun secara spesifik, virus ini dapat menyebabkan pneumonia yaitu kerusakan saluran pernapasan baik ringan maupun berat pada manusia (Safrizal dkk, 2020).
Invasi SARS-CoV-2 ke dalam tubuh sel inang dimulai dari penempelan glikoprotein Spike (S) pada reseptor Angiotensin-Converting Enzyme-2 (ACE-2) dengan bantuan enzim Transmembrane Protease Serine 2 (TMPRSS-2). Kemudian terjadi proses fusi membran virus dengan membran sel inang. RNA virus dilepaskan ke dalam sitoplasma sel inang dan berlangsungnya proses replikasi materi genetik virus. RNA yang sudah diperbanyak bersama dengan protein E (Envelope) dan N (Nucleocapsid) membentuk vesikel yang mengandung virion. Kemudian menembus membran sel dan akhirnya keluar dari sel inang yang selanjutnya menginfeksi sel lainnya (Ikawaty, 2020).
Inkubasi virus di dalam sel inang terjadi selama 3-7 hari hingga muncul gejala penyakit yang disebabkan oleh virus tersebut (PDPI, 2020). Hal ini ditandai dengan kadar leukosit dan limfosit yang sedikit menurun, akan tetapi pasien masih belum
6
merasakan gejala-gejala apapun. Kemudian virus menyebar melalui aliran darah menuju ke organ yang dapat mengekspresikan ACE2 hingga muncul gejala ringan pada pasien. Kemudian 4-7 hari, kondisi pasien semakin memburuk hingga adanya rasa sesak, menurunnya limfosit dan perburukan lesi di paru-paru. Fase ini bertambah parah jika tidak diatasi, hal ini dapat menyebabkan terjadinya Acute Respiratory Distress Syndrome (ARSD), sepsis dan penyakit komplikasi lainnya (Fitriani, 2020).
2.3 Epidemologi SARS-CoV-2
Inang penyebaran SARS-CoV-2 berasal dari kalelawer. Kasus penularan pertama antar manusia terjadi di Vietnam dan United State yang terinfeksi selama berada di Wuhan tanpa kontak fisik dengan Seafood Wholesale Market yang merupakan awal terjadinya penyebaran virus SARS-CoV-2. Akhirnya dapat dikonfirmasi bahwa virus ini dapat menular dari manusia ke manusia (Wahjudi, 2020). Transmisi SARS-CoV-2 terjadi melalui kontak droplet dan udara. Transmisi melalui droplet dapat terjadi melalui kontak langsung, kontak tidak langsung atau kontak erat dengan orang yang terinfeksi melalui sekresi seperti air liur dan sekresi pernapasan atau droplet yang keluar saat orang yang terinfeksi batuk ataupun bersin, sedangkan transmisi melalui udara terjadi jika penyebaran agen terinfeksi (aerosol) yang tetap terinfeksi saat melayang di udara dan bergerak hingga jarak yang jauh.
Transmisi lainnya yang dapat terjadi jika sekresi saluran pernapasan atau droplet yang dikeluarkan oleh pasien yang terinfeksi virus menimbulkan kontaminasi pada permukaan benda tersebut. Sehingga permukaan benda yang berkontak langsung dengan pasien akan terinfeksi. Hal ini menyebabkan terjadinya penyebaran virus SARS-CoV-2 secara tidak langsung (WHO, 2020).
Beberapa faktor yang menyebabkan penyebaran SARS-CoV-2 terus meningkat pesat yaitu kecepatan transmisi virus yang sangat tinggi, penyebaran terjadi ketika pasien positif tanpa gejala yang tidak sadar akan adanya penularan virus, kurangnya sistem imun tubuh sebagai pertahanan awal pencegahan infeksi virus dan sistem kesehatan masyarakat yang buruk atau terlambatnya penanganan pada saat awal pandemi (Wardiana, 2020). Manifetasi klinis yang diakibatkan oleh COVID-19 sangat beragam, dimulai dari pasien yang bergejala maupun tidak bergejala hingga bergejala pneumonia ringan. Penyebar virus tanpa gejala
7
(asimptomatik) ini cukup berbahaya, hal ini dapat menyebabkan penularan virus tanpa diketahui orang lain bahkan pasien tersebut (Prastyowati, 2020).
Panduan surveilans menurut WHO dalam mendiagnosis pasien dapat dilihat berdasarkan bentuk infeksi pada pasien SARS-CoV-2. Pertama, kasus terduga (Suspect case) merupakan pasien dengan gangguan napas akut dan berkontak erat dengan pasien yang terkonfirmasi pasien COVID-19. Kedua, kasus probable (probable case) dengan kasus terduga hasil tes bersifat inkonklusif dan perlu dilakukan pengerjaan ulang. Ketiga, kasus terkonfirmasi dimana pasien tersebut terkonfirmasi adanya infeksi COVID-19, baik bergejala maupun tidak bergejala (Handayani, 2020).
Klasifikasi infeksi COVID-19 di Indonesia memiliki perbedaan dengan klasifikasi infeksi menurut WHO. Berdasarkan panduan tatalaksana Pneumonia COVID-19 Kementerian Kesehatan Republik Indonesia bentuk infeksi pada pasien SARS-CoV-2 tersebut adalah yang pertama, Pasien dalam Pengawasan (PDP) dimana terjadi kasus pasien dengan Infeksi Saluran Pernapasan Akut (ISPA) seperti demam (≥38°C) disertai dengan batuk, sesak nafas, sakit tenggorokan, pilek, pneumonia ringan maupun berat, memiliki kontak erat dengan pasien terkonfirmasi dan orang yang membutuhkan perawatan di rumah sakit. Kedua, Orang dalam Pemantauan (ODP) dimana pasien mengalami demam (≥38°C) disertai dengan batuk, sesak nafas, sakit tenggorokan, pilek dan pneumonia ringan maupun berat serta memiliki kontak erat dengan pasien konfirmasi infeksi COVID-19. Ketiga, pasien yang tidak memiliki gejala dan resiko tertular dari pasien terkonfirmasi serta adanya kontak erat dengan pasien infeksi COVID-19. Kontak erat yang dimaksud adalah seseorang yang terinfeksi virus dengan melakukan kontak fisik ataupun berada di suatu ruangan yang sama pada pasien terkonfirmasi (Direktorat Jenderal Pencegahan dan Pengendalian Penyakit, 2020).
2.4 Metode Pemeriksaan SARS-CoV-2
Identifikasi virus SARS-CoV-2 secara molekuler pada pasien terinfeksi saat ini masih menjadi pemeriksaan standar dalam menentukan pasien positif yang terinfeksi atau negatif yang tidak terinfeksi COVID-19. Pemeriksaan yang dilakukan untuk identifikasi virus membutuhkan strategi efektif agar dapat meningkatkan
8
kapasitas pemeriksaan SARS-CoV-2. Berikut metode yang digunakan dalam pemeriksaan SARS-CoV-2, yaitu sebagai berikut:
2.4.1 Gabungan Sampel (Pooled test)
Organisasi Kesehatan Dunia (WHO) merekomendasikan pengujian diagnostik untuk membedakan SARS-CoV-2 dari infeksi pernapasan lainnya. Sejak pandemi, pemeriksaan SARS-CoV-2 semakin terus meningkat dan membutuhkan banyak sumber daya. Sehingga WHO memutuskan untuk meningkatkan pemeriksaan sekitar 10-30 tes kasus positif. Sehingga diperlukan strategi pengujian yang memadai dengan rekomendasi tingkat positif lebih rendah dari 10%. Dalam situasi ini, metode gabungan sampel (Pooled test) menjadi strategi penting untuk pemeriksaan dalam skala besar, dimana setiap sampel dari suatu populasi digabungkan menjadi dalam satu tabung (Mahmoud et al., 2021).
Gabungan sampel (Pooled test) merupakan strategi yang digunakan untuk skrining komprehensif awal dari virus Influenza dan Human Immunodeficiency Virus (HIV), namun saat ini direkomendasikan sebagai salah satu strategi dalam pemeriksaan SARS-CoV-2. Adanya kekurangan bahan habis pakai terutama pada peralatan ekstraksi RNA karena peningkatan global untuk pengujian virus, maka menyebabkan terjadinya penundaan dan efisiensi pemeriksaan SARS-CoV-2, sehingga metode ini dapat diaplikasikan untuk menghemat biaya peningkatan kapasitas deteksi virus serta mempermudah skrinning skala komunitas tanpa harus mengurangi keakuratan pengujian (Lim, 2020).
Hasil dari penggabungan sampel (Pooled test) menunjukkan jika hasil gabungannya negatif, maka semua spesimen yang di dalam gabungan dianggap negatif. Kemudian jika hasil dari gabungan positif, maka perlu dilakukan tes individual (penguraian gabungan) untuk mengidentifkasi spesimen yang positif.
Protokol penggabungan sampel hanya dilakukan dalam suatu populasi homogen, dimana hanya terdiri dari satu wilayah dalam skala yang besar. Kemudian dilakukan pemeriksaan laboratorium dengan perbandingan-perbandingan yang telah disesuaikan. Strategi tes yang tidak sesuai dapat menimbulkan terlewatnya kasus atau kekeliruan laboratorium yang dapat berdampak negatif bagi tatalaksana pasien dalam pengendalian kesehatan masyarakat (WHO, 2020).
9
2.4.2 Reverse Transcription Polymerase Chain Reaction (RT-PCR)
Teknik RT-PCR (Reverse Transcription Polymerase Chain Reaction) merupakan metode umum yang digunakan dalam mendeteksi infomasi genetik, terutama materi genetik SARS-CoV-2. Teknik ini dilakukan dengan menggunakan mRNA (messenger RNA) sebagai cetakan (template). Pada tahap pertama, teknik ini menggunakan enzim reverse transcription untuk mengubah template mRNA sehingga menghasilkan DNA untai tunggal komplementer yang disebut dengan cDNA (complementary DNA). Tahapan ini dikenal dengan transkripsi balik.
Kemudian tahap selanjutnya mengubah cDNA untai tunggal menjadi DNA untai ganda dengan menggunakan enzim DNA polimerase. Molekul yang dihasilkan akan digunakan sebagai template untuk reaksi Real Time PCR (Herawati, 2020).
Proses PCR konvensional hanya memperbanyak DNA target dan tidak dapat mengamplifkasi RNA sehingga deteksi materi genetik berupa RNA diperlukan tahapan tambahan sebelum proses PCR. Pada proses RT-PCR, material genetik RNA diubah menjadi cDNA melalui proses reverse transcription (RT). Formasi hibridisasi RNA akan membentuk untai pertama cDNA untuk kemudian akan diamplifkasi pada proses PCR. Teknik RT-PCR umumnya digunakan untuk mendeteksi RNA spesifk, antara lain, adalah diagnosis penyakit, baik maupun deteksi mRNA dalam studi ekspresi gen (Fraga, 2014).
Real Time PCR mendeteksi adanya RNA virus yang terdeteksi pada sampel pasien. Teknik ini berjalan untuk memperjelas materi genetik pada SARS-CoV-2 seperti protein Spike (S), protein Nucleic (N) dan Envelope (E) dari virus. Hingga teknik ini membutuhkan marker fluorescent yang membantu mendeteksi virus. Jika nilai fluoresen ini mencapai level tertentu, maka dapat mengkonfirmasi presensi dari virus. Hasil positif maka menginterpretasikan pasien terinfeksi virus, sedangkan hasil negatif menginterpretasikan pasien tidak sedang terinfeksi oleh virus. Nilai fluorensen ini dikenal dengan nilai Ct (Cycle threshold) (Yanti, 2020).
2.5 Nilai Ct (Cycle threshold)
Nilai Ct (Cycle Threshold) adalah jumlah siklus yang diperlukan agar sinyal fluoresen pada tahap RT-PCR dapat melewati ambang (yaitu melebihi level latar belakang). Nilai Ct berbanding terbalik dengan jumlah target asam nukleat dalam
10
sampel, dimana semakin rendah tingkat Ct maka semakin besar jumlah asam nukleat target dalam sampel (Herawati, 2020). Tinggi atau rendahnya nilai Ct sangat bergantung pada teknis pengerjaan, jumlah RNA di dalam sampel, metode pengambilan sampel, metode ekstraksi RNA yang digunakan, reagen dan primer yang digunakan dalam reaksi PCR (PAMKI, 2020).
Kurva amplifkasi PCR yang berbentuk sigmoid terdiri atas 3 fase, yaitu eksponensial, linear, dan plateu. Hasil akhir RT-PCR berupa nilai Ct yang merupakan perpotongan antara garis threshold dan kurva amplifkasi. Garis threshold merupakan garis yang berada di atas baseline sinyal fluorescenc sehingga sinyal fluorescence yang terdeteksi di atas threshold adalah sinyal yang digunakan dalam menentukan nilai Ct (Cao et al., 2020). Teknik RT-PCR memiliki nilai Ct maksimum 40. Nilai Ct tersebut ditetapkan untuk memastikan hasil yang spesifik. Hasil nilai Ct ini memiliki nilai yang sama dengan pasien bergejala maupun tanpa gejala. Pada orang tanpa gejala, seringkali tidak diketahui kapan orang tersebut terinfeksi virus dan menyebabkan resiko infektivitasnya tinggi dibandingkan dengan orang yang bergejala (Public Health England, 2020).
Interpretasi hasil pada pemeriksaan deteksi virus ditentukan berdasarkan nilai gen target. Gen target dari nilai Ct berupa ORF1ab, E-gene dan N-gene. Gen ORF1ab merupakan 1/3 bagian dari genom virus yang menyandikan 16 protein non-strutural (NSPs), kemudian sepertiga sisanya dari genom mengkodekan 4 protein struktural dan 6 protein pelengkap. Protein struktural berupa spike glikoprotein (S), protein matriks (M), protein amplop (E) dan protein nukleokapsid (N). Protein pelengkap berupa ORF3a, ORF6, ORF7a, ORF7b dan ORF10 (Mashuri dkk, 2020).
Gen ORF1ab berperan penting dalam proses transkripsi dan replikasi virus.
Sehingga daerah ORF1ab ini sering digunakan sebagai target untuk deteksi/konfirmasi virus SARS-CoV-2. Gen S bertugas dalam mengkode pembentukan protein spike pada permukaan sel host. Gen N mengkode pembentukan protein nukleokapsid dan merupakan gen target yang paling sensitif dibandingkan gen lainnya, hal ini dikarenakan gen N memiliki peluang yang sangat kecil untuk bermutasi. Gen E bertugas dalam mengkode pembentukan protein E (Envelope) yang merupakan protein selubung bagi virus yang berperan penting dalam siklus kehidupan virus serta patogenesis (PAMKI, 2020).
11
BAB 3
METODE PENELITIAN
3.1 Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Juli-Agustus 2021 di Laboratorium Mikrobiologi, Fakultas Kedokteran, Universitas Sumatera Utara, Medan.
3.2 Rancangan Penelitian
Pemeriksaan SARS-CoV-2 dimulai dengan pengambilan sampel swab pada masyarakat yang dilakukan oleh Satuan Tugas Balai Teknik Lingkungan dan Pengendalian Penyakit (BTKLPP) Medan. Sampel diambil secara purposive sampling yang merupakan salah satu teknik pengambilan sampel berdasarkan dengan kriteria-kriteria tertentu (Sugiyono, 2008). Kriteria yang digunakan dalam penelitian ini adalah masyarakat yang melakukan swab dengan kategori swab pertama sebanyak 30 orang, diuji secara individual dengan metode RT-PCR untuk menentukan hasil negatif dan positif. Selanjutnya, sampel positif dan negatif diuji kembali berdasarkan tiga kategori viral load yaitu high (<20), intermediet (>20<30) dan low (>35) (Meletis, 2020) dengan metode pooled test dengan perbandingan 1:4, 1:6 dan 1:9 (Mulu, 2021). Dilakukan tiga kali pengulangan pada setiap perbandingan.
3.3 Prosedur Penelitian 3.3.1 Sampel Individu
Pemeriksaan SARS-CoV-2 dilakukan secara swab (nasofaring dan orofaring) dengan kategori swab pertama. Sampel dimasukkan ke dalam cryotube yang berisi Viral Media Transport (VTM) sebanyak 30 sampel. Setiap spesimen diberi tanda sesuai dengan kode yang telah ditentukan dan disusun ke dalam rak sampel dan dilakukan pengujian ekstraksi RNA.
3.3.1.1 Ekstraksi RNA Individu
Ekstraksi RNA dilakukan sesuai metode Maccura Nucleic Acid Extraction Kit, Fast version (Maccura Biotechnology, 2020). Kit tersebut terdiri dari well extraction Kit, 320µl Mag-Bind RNA Extraction Kit, internal control, control positif
12
dan control negative. Penambahan internal control pada Mag-Bind RNA Extraction Kit sebanyak 64µl lalu di vortex dan dilakukan centrifuge spin down selama 15 detik.
Sediakan well Extraction Kit yang terdiri dari 12 well, dimana pada well 1 dan 7 berisi Magnetic nanoparticles; well 2 dan 8 berisi Lysis Buffer; well 3 dan 9 berisi Wash Buffer 1; well 4 dan 10 berisi Wash Buffer 2; well 6 dan 12 berisi Elution Buffer. Sebanyak 10µl Mag-Bind; 2µl Internal control; 200µl sampel; 200µl control negative dan control positif dimasukkan kedalam well 2 dan 8. Running well extraction kit selama 16 menit. RNA hasil ekstraksi tersebut diambil sebanyak 35µl dan dipindahkan kedalam collection tube. Selanjutnya RNA di amplifikasi.
3.3.1.2 Amplifikasi cDNA Individu
Amplifikasi cDNA dilakukan secara RT-PCR dengan Maccura SARS-CoV-2 Fluorescent PCR Kit (Maccura Biotechnology, 2020). Persiapan master-mix dihitung sesuai dengan jumlah sampel yang akan di amplifikasi yang dapat dilihat pada Tabel 3.1. Sebanyak 17µl qRT-PCR Reaction Mix; 3µl qRT-PCR Enzyme Mix; 10µl RNA;
10µl control negative dan control positif dimasukkan ke dalam plate PCR.
Homogenkan dengan mix-mate dengan kecepatan 3000 Rpm selama 15 detik. Plate PCR diletakkan ke dalam mesin RT-PCR dengan menekan tombol Run sesuai dengan tamplate yang sudah diatur. Program RT-PCR untuk mendeteksi SARS-CoV-2 dapat dilihat pada Tabel 3.2.
Tabel 3.1 Komposisi master-mix RT-PCR
Master Mix Reaktan Komponen Volume (µl)/sampel
qRT-PCR Mix
qRT-PCR Reaction Mix
Primer spesifik, Probe, dNTP,
Mg2+, Buffer 17
qRT-PCR Enzyme Mix
Taq Polymerase, Uracil-DNA
Glycosylase 3
Sumber: (Maccura Biotechnology, 2020).
Tabel 3.2 Program RT-PCR untuk mendeteksi SARS-CoV-2
No. Tahapan Suhu Waktu Siklus
1. Reverse transcription 55°C 15 min 1
2. Aktivasi Taq Polymerase , Pre-denaturasi 95°C 2 min 1
3.
4.
5.
Denaturasi 95°C 15 sec
40 Annealing
Ekstensi
58°C 72°C
35 sec 60 sec
6. Instrument cooling 40°C 10 sec 1
Sumber: (Maccura Biotechnology, 2020)
13
3.3.1.3 Interpretasi Nilai Ct
Keterangan hasil amplifikasi cDNA diinterpretasikan berdasarkan nilai Ct yang mencakup tiga gen target yang diantaranya E-gene, ORF1ab, N-gene dan IC sebagai kontrol ekstraksi. Berikut interpretasi nilai Ct dalam mendiagnosa SARS- CoV-2 dapat dilihat pada Tabel 3.3 sebagai berikut.
Tabel 3.3 Interpretasi nilai Ct sampel individu
No. Gen Target
Interpretasi Keterangan E-gene ORF1ab N-gene IC
1. + + + + Positif Non repeat
2. + - + + Positif Repeat PCR
3. + + - + Positif Non repeat
4. + - - + Negatif Non repeat
5. - - - - Invalid Repeat ekstraksi
Keterangan :
Tanda - : tidak terdeteksi Tanda + : terdeteksi
Sumber: (Maccura Biotechnology, 2020) 3.3.2 Sampel Pooled Test
Sampel individu yang telah dideteksi, didapatkan hasil pengujian berupa sampel negatif dan positif. Sampel diseleksi untuk diuji kembali dengan menggunakan metode pooled test dengan perbandingan 1:4 (1 sampel positif dan 4 negatif), 1:6 (1 sampel positif dan 6 sampel negatif) dan 1:9 (1 sampel positif dan 9 sampel negatif). Kategori Nilai Ct yang digunakan dapat dilihat berdasarkan tingkat viral load dimana high (<25), intermediet (>25<30) dan low (>30) (Meletis et al., 2021). Diambil 100µl sampel negatif dan positif yang akan digabungkan, kemudian dimasukkan ke dalam tube 2 ml dan di vortex hingga homogen. Selanjutnya, dilakukan pengujian ekstraksi RNA pooled test.
3.3.2.1 Ekstraksi RNA Pooled test
Ekstraksi RNA dilakukan sesuai metode Maccura Nucleic Acid Extraction Kit, Fast version (Maccura Biotechnology, 2020). Kit tersebut terdiri dari well extraction Kit, 320µl Mag-Bind RNA Extraction Kit, internal control, control positif dan control negative. Penambahan internal control pada Mag-Bind RNA Extraction Kit sebanyak 64µl lalu di vortex dan dilakukan centrifuge spin down selama 15 detik.
14
Sediakan well Extraction Kit yang terdiri dari 12 well, dimana pada well 1 dan 7 berisi Magnetic nanoparticles; well 2 dan 8 berisi Lysis Buffer; well 3 dan 9 berisi Wash Buffer 1; well 4 dan 10 berisi Wash Buffer 2; well 6 dan 12 berisi Elution Buffer. Sebanyak 12µl (10µl Mag-Bind + 2µl Internal control); 200µl sampel; 200µl control negative dan control positive dimasukkan kedalam well 2 dan 8. Running well extraction kit selama 16 menit. RNA hasil ekstraksi tersebut diambil sebanyak 35µl dan dipindahkan kedalam collection tube. Selanjutnya RNA di amplifikasi.
3.3.2.2 Amplifikasi cDNA Pooled test
Amplifikasi cDNA pooled test dilakukan secara RT-PCR dengan Maccura SARS-CoV-2 Fluorescent PCR Kit (Maccura Biotechnology, 2020). Persiapan master-mix dihitung sesuai dengan jumlah sampel yang akan di amplifikasi yang dapat dilihat pada Tabel 3.4. Sebanyak 17µl qRT-PCR Reaction Mix; 3µl qRT-PCR Enzyme Mix; 10µl RNA; 10µl control negative dan control positive dimasukkan ke dalam plate PCR. Homogenkan dengan mix-mate dengan kecepatan 3000 Rpm selama 15 detik. Plate PCR diletakkan ke dalam mesin RT-PCR dengan menekan tombol Run sesuai dengan tamplate yang sudah diatur. Program RT-PCR untuk mendeteksi SARS-CoV-2 dapat dilihat pada Tabel 3.5 berikut.
Tabel 3.4 Komposisi master-mix RT-PCR
Master Mix Reaktan Komponen Volume (µl)/sampel
qRT-PCR Mix
qRT-PCR Reaction Mix
Primer spesifik, Probe, dNTP,
Mg2+, Buffer 17
qRT-PCR Enzyme Mix
Taq Polymerase, Uracil-DNA
Glycosylase 3
Sumber: (Maccura Biotechnology, 2020).
Tabel 3.5 Program RT-PCR untuk mendeteksi SARS-CoV-2
No. Tahapan Suhu Waktu Siklus
1. Reverse transcription 55°C 15 min 1
2. Aktivasi Taq Polymerase , Pre-denaturasi 95°C 2 min 1
3.
4.
5.
Denaturasi 95°C 15 sec
40 Annealing
Ekstensi
58°C 72°C
35 sec 60 sec
6. Instrument cooling 40°C 10 sec 1
Sumber: (Maccura Biotechnology, 2020).
15
3.3.2.3 Interpretasi Nilai Ct
Keterangan hasil amplifikasi cDNA diinterpretasikan berdasarkan nilai Ct yang mencakup tiga gen target yang diantaranya E-gene, ORF1ab, N-gene dan IC sebagai kontrol ekstraksi. Berikut interpretasi nilai Ct dalam mendiagnosa SARS- CoV-2 dapat dilihat pada Tabel 3.6 sebagai berikut.
Tabel 3.6 Interpretasi nilai Ct sampel pooled test
No. Gen Target
Interpretasi Keterangan E-gene ORF1ab N-gene IC
1. + + + + Positif Non repeat
2. + - + + Positif Repeat PCR
3. + + - + Positif Non repeat
4. + - - + Negatif Non repeat
5. - - - - Invalid Repeat ekstraksi
Keterangan:
Tanda - : tidak terdeteksi Tanda + : terdeteksi
Sumber: (Maccura Biotechnology, 2020)
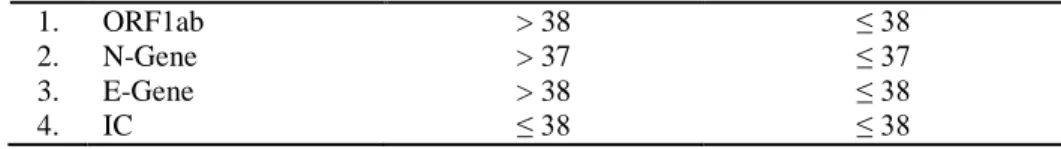
Adapun batas ambang siklus dalam amplifikasi (Cut-off) yang telah disesuaikan berdasarkan dengan Maccura SARS-CoV-2 Fluorescent PCR Kit (Maccura Biotechnology, 2020) yang dapat dilihat pada Tabel 3.7 berikut.
Tabel. 3.7 Batas cut off nilai Ct
No. Gen target Negatif Positif
1. ORF1ab > 38 ≤ 38
2. N-Gene > 37 ≤ 37
3. E-Gene > 38 ≤ 38
4. IC ≤ 38 ≤ 38
Sumber: (Maccura Biotechnology, 2020).
3.4 Analisis Data
Data hasil penelitian yang telah diperoleh, dianalisis secara statistik dengan menggunakan software SPSS versi 22.0 dengan menggunakan uji Chi-square.
Interpretasi nilai signifikansi p<0,05 maka terdapat perubahan yang signifikan dan jika nilai p>0,05 maka hasil tidak signifikan.
16
BAB 4
HASIL DAN PEMBAHASAN
Berdasarkan penelitian yang telah dilakukan dengan menggunakan sampel swab SARS CoV-2 masyarakat oleh satuan tugas BTKL-PP Kelas I Medan didapatkan hasil sebagai berikut:
4.1 Hasil Nilai CT Individual
Dari hasil pemeriksaan RT-PCR yang telah dilakukan pada sampel swab sebanyak 30 sampel, terdapat 24 sampel negatif dan 6 sampel positif yang terinfeksi SARS-CoV-2 dan dapat dilihat pada Tabel 4.1 sebagai berikut.
Tabel 4.1 Hasil nilai Ct sampel individu dan interpretasi hasil positif dan negatif
No. Kode sampel Hasil qRT-PCR (Nilai Ct)
Hasil IC ORF1ab E-gene N-gene
1. 01 29 - - - Negatif
2. 02 28 - - - Negatif
3. 03 28 - - - Negatif
4. 04 29 - - - Negatif
5. 05 28 35,4 33,3 34,8 Positif
6. 06 29 - - - Negatif
7. 07 28 39 - 39 Negatif
8. 08 28 38 - - Negatif
9. 09 28 - - - Negatif
10. 10 28 - - 39,1 Negatif
11. 11 28 - - - Negatif
12. 12 28 24,8 25,1 26,7 Positif
13. 13 29 - - - Negatif
14. 14 28 - - - Negatif
15. 15 29 - - - Negatif
16. 16 28 39,2 37,9 - Negatif
17. 17 28 - - 38,7 Negatif
18. 18 28 17,7 18,1 19,5 Positif
19. 19 28 - - - Negatif
20. 20 28 - - - Negatif
21. 21 28 - - - Negatif
22. 22 28 - - - Negatif
23. 23 28 34,9 - 37,1 Positif
24. 24 28 - - 38,4 Negatif
25. 25 28 - - - Negatif
26. 26 29 27,9 28,3 29,2 Positif
27. 27 29 - - - Negatif
28. 28 28 - - - Negatif
29. 29 28 - - - Negatif
30. 30 29 27,2 27,5 27,8 Positif
Keterangan:
Tanda - : tidak terdeteksi IC : Internal control
17
Hasil qRT-PCR berupa nilai Ct didapatkan 6 sampel positif yang dapat dilihat berdasarkan dari tiga gen target diantaranya ORF1ab, E-gene dan N-gene.
Sedangkan interpretasi sampel negatif didapat sebanyak 24 sampel, sementara internal control menunjukkan nilai Ct antara 28-29. Hal ini menunjukkan bahwa sampel yang di running dengan RT-PCR berjalan dengan baik, dimana internal control tersebut digunakan sebagai kontrol RT-PCR. Costa et al (2021) menyatakan bahwa sampel yang dapat di deteksi dengan nilai Ct lebih besar dari batas Cut-off Ct yang telah ditentukan dapat diinterpretasikan sebagai negatif. Maccura Biotechnology (2020) menyatakan bahwa batas ambang pada siklus amplifikasi (cut- off) berpengaruh dalam penentuan gen target pada sampel. Cut-off pada ORF1ab (≤38), E-gene (≤37) dan N-gene (≤38). Jika cut off melewati batas ambang nilai Ct, maka hasil interpretasi dinyatakan negatif, sedangkan cut-off dibawah batas ambang siklus maka hasil interpretasi dianggap positif.
Menurut PAMKI (2020) untuk mempertimbangkan hasil nilai Ct pada pemeriksaan SARS-CoV-2 harus disertai dengan nilai rujukan berupa range atau konversi ke nilai viral load atau dikelompokkan menjadi viral load rendah, medium maupun tinggi. Menurut Zein et al., (2021), viral load menunjukkan jumlah konsentrasi materi genetik virus dalam suatu sampel. Pada Tabel 4.1 viral load tertinggi terdapat pada sampel 18 dengan nilai Ct pada gen target ORF1ab (17,7), E- gene (18,1) dan N-gene (19,5), viral load sedang terdapat pada kode sampel 26 dengan nilai Ct pada gen target ORF1ab (27,9), E-gene (28,3), N-gene (29,2) dan IC (29,0); kode sampel 12 dengan nilai Ct pada gen target ORF1ab (24,8), E-gene (25,1), N-gene (26,7) dan IC (28,0); kode sampel 30 dengan nilai Ct pada gen target ORF1ab (27,2), E-gene (27,5), N-gene (27,8) dan IC (29), sedangkan viral load terendah terdapat pada sampel 05 dengan nilai Ct pada gen target ORF1ab (35,4), E- gene (33,3), N-gene (34,8) dan IC (28,0); kode sampel 23 dengan nilai Ct pada gen target ORF1ab (34,9), N-gene (37,1) dan IC (28,0).
Public Health England (2020) menyatakan bahwa jumlah viral load berbanding terbalik dengan nilai Ct, dimana semakin rendah nilai Ct maka viral load pada virus semakin tinggi dan beresiko dalam infektivitas. Sedangkan semakin tinggi nilai Ct maka konsentrasi materi genetik virus (viral load) semakin rendah dan resiko infektivitas lebih rendah. Menurut Calle et al., (2020) kategori viral load ditentukan
18
menjadi tiga kategori yaitu viral load tinggi (high viral load) dengan nilai Ct<20, viral load sedang (intermediet viral load) dengan nilai Ct >20<30 dan viral load rendah (low viral load) dengan nilai Ct >30.
Keberhasilan proses amplifikasi cDNA pada pemeriksaan SARS-CoV-2 sangat bergantung pada kit RT-PCR yang dipakai yaitu dengan menggunakan qRT- PCR Mix dari Maccura SARS-CoV-2 Fluorescent PCR Kit. qRT-PCR Mix yang digunakan terdiri dari qRT-PCR Reaction Mix dan qRT-PCR Enzyme Mix.
Komponen qRT-PCR Reaction Mix berupa Primer spesifik, Probe, dNTP, Mg2+ dan Buffer. Sedangkan komponen qRT-PCR Enzyme Mix berupa Taq Polymerase dan Uracil-DNA Glycosylase. Suhu program RT- PCR yang digunakan untuk mendeteksi virus tersebut berkaitan dengan proses Reverse transcription (55°C), Aktivasi Taq Polymerase dan Predenaturasi (95°C), Denaturasi (95°C), Annealing (58°C), ekstensi (72°C), Instrument cooling (40°C) (Maccura Biotechnology, 2020).
4.2 Nilai Ct pada Sampel Individu dan Sampel Pooled Test
Nilai Ct dari sampel positif yang telah diuji sebelumnya, kemudian diuji kembali secara pooled test dengan sampel negatif yang telah diketahui hasilnya dan dapat dilihat pada Tabel 4.2 berikut.
Tabel. 4.2 Nilai Ct pada sampel individu dan sampel pooled test
Viral load Sampel individu
Nilai Ct sampel Pooled test
Hasil
1:4 ΔCt 1:6 ΔCt 1:9 ΔCt
High Orf1ab N-gene E-gene IC
19,4 18,4 20,2 34
21,04 21,09 21,70 34
1,64 2,69 1,50 -
21,1 21,02 21,90 33
1,70 2,62 1,70 1
21,3 21,4 22,2 35
1,9 3,0 2,0 1
Positif
Intermediet Orf1ab N-gene E-gene IC
26,7 26,2 28,2 35
28,9 29,1 30,1 34
2,2 2,9 1,9 -1
28,7 29,1 30 33
2,0 2,9 1,8 -2
29,2 29,4 30,5 33
2,5 3,2 2,3 -2
Positif
Low Orf1ab N-gene E-gene IC
35,7 36,8 37,4 33
- - - 34
-35,7 -36,8 -37,4 1
39,05 39,50
- 34
3,35 2,70 - 1
- - - 33
-35,7 -36,8 -37,4 -
Negatif
Keterangan:
ΔCt : perbedaan nilai Ct (Ctpooled - Ctindividual) (Deka et al., 2020).
Tanda - : tidak terdeteksi
19
Nilai Ct pada pengujian sampel individu dan sampel pooled test dapat dilihat berdasarkan high viral load, intermediet viral load dan low viral load. Terdapat perubahan nilai Ct antara sampel individu (high viral load dan intermediet viral load) dan sampel pooled test namun masih dalam batas ambang (cut-off) positif yaitu (<38), sedangkan perubahan nilai Ct pada sampel individu low viral load dengan sampel gabungan mengalami pergeseran nilai Ct hingga sampel positif berubah menjadi sampel negatif. Griesemer et al., (2021) menyatakan bahwa tingkat sampel positif bergantung pada ukuran pooled yang digunakan. Semakin besar jumlah pooled maka kualitas sampel positif akan menjadi lemah. Kualitas sampel menurun dapat disebabkan oleh adanya pembawa materi genetik dari sampel sedikit, sehingga kondisi sampel yang melemah dapat menyebabkan perubahan hasil menjadi sampel negatif.
Uji pooled test dengan perbandingan 1:4, 1:6 dan 1:9 memiliki perubahan nilai Ct berdasarkan high viral load, intermediet viral load dan low viral load. Akan tetapi perubahan nilai Ct pada masing-masing perbandingan dengan sampel high viral load dan intermediet viral load masih dalam ambang batas nilai Ct yang tidak menyebabkan perubahan hasil positif yang telah digabungkan (pooled), sedangkan pada sampel low viral load mengalami perubahan hasil positif menjadi negatif.
Namun pada uji low viral load dengan pooled 1:6 masih terdeteksi gen target berupa ORF1ab dan N-gene. Hal ini menunjukkan munculnya dugaan jumlah virus yang sangat kecil pada sampel positif yang telah digabungkan sehingga pada perbandingan 1:6 gen target virus masih dapat terdeteksi. Berdasarkan penelitian yang sebelumnya Meletis (2021) menyatakan bahwa pergeseran nilai Ct tersebut disebabkan oleh adanya efek pembawa dari kandungan RNA yang lebih tinggi jika sampel di pooled, sehingga ketika sampel digabungkan masih terdapat kemunculan gen target virus pada sampel dalam jumlah yang sedikit.
4.3 Hubungan Tingkat Viral Load dan Sampel Pooled Test dengan Nilai Ct pada RT-PCR
Hubungan tingkat viral load dan perbandingan pooled test dengan interpretasi nilai Ct pada RT-PCR di analisis dengan Statical Pakage of Social Sciense (SPSS) versi 22.0 menggunakan uji Chi-square dengan nilai signifikansi p<0.05. Menurut
20
Szentesi et al., (2021) jika nilai (p<0,05) maka terdapat hubungan yang signifikan, dan sebaliknya jika (p>0,05) maka tidak ada perbedaan yang signifikan antara hubungan tingkat viral load dan perbandingan pooled test dengan interpretasi nilai Ct pada RT-PCR.
4.3.1 Hubungan Antara Tingkat Viral Load dengan Nilai Ct pada RT-PCR Hubungan antara tingkat viral load dengan nilai Ct pada RT-PCR dapat di lihat pada Tabel 4.3 berikut.
Tabel 4.3 Hubungan antara tingkat viral load dengan nilai Ct pada RT-PCR
Variabel Nilai Ct
P value n Positif n Negatif
High viral load 9 -
0,000
Intermediet viral load 9 -
Low viral load - 9
Keterangan :
Tanda - : tidak terdeteksi Tanda n : jumlah sampel
Pada Tabel 4.3 di atas menunjukkan nilai signifikansi p<0,05 yang menjelaskan hubungan antara tingkat viral load dengan nilai Ct pada RT-PCR memiliki perbedaan nilai yang signifikan (Lampiran 1). Perbedaan nilai Ct tersebut terjadi dikarenakan adanya perubahan hasil positif pada sampel low viral load menjadi negatif. Dalam penelitian Manurung (2021) menjelaskan bahwa strategi pengumpulan sampel untuk qRT-PCR memiliki sensitivitas yang lebih rendah daripada qRT-PCR individu standar dan sensitivitasnya menurun seiring dengan peningkatan ukuran kumpulan (pooled).
4.3.2 Hubungan Antara Perbandingan Pooled Test dengan Nilai Ct RT-PCR Hubungan antara perbandingan pooled test dengan nilai Ct RT-PCR dapat dilihat pada Tabel 4.4 berikut.
Tabel 4.4 Hubungan antara sampel pooled test dengan nilai Ct pada RT-PCR
Variabel Nilai Ct
P value n Positif n Negatif
Pooled 1:4 6 3
1,000
Pooled 1:6 6 3
Pooled 1:9 6 3