1
PENERAPAN ALGORITMA
SINGULAR VALUE DECOMPOSITION
(SVD) UNTUK PENGURANGAN DIMENSI PADA HIGH-DIMENTIONAL
BIOMEDICAL DATA SET
Irfan Abbas STMIK Ichsan Gorontalo Email: [email protected]
Abstrak: Dewasa ini data high dimentional biomedical data set telah menunjukkan peningkatan popularitas dan perhatian, serta telah berkembang dengan cepat selama dua dekade. Data High dimentional biomedical data set memiliki ribuan atribut serta ribuan jumlah record yang saling berkorelasi (multicolinearity), berskala besar dan kompleks. Teknik pengurangan dimensi merupakan bagian dari pengolahan awal data dan sangat tepat digunakan untuk memecahkan data yang kompleks. Algoritma Singular Value Decomposition (SVD adalah metode teknik pengurangan dimensi yang tergolong dalam kelompok ekstraksi atribut. Penelitian ini menggunakan Algoritma Singular Value Decomposition (SVD untuk mengurangi dimensi pada data high dimentional biomedical data set yang memiliki ribuan atribut
Keywords: Algoritma Singular Value Decomposition (SVD), Dataset High dimentional biomedical.
1. PENDAHULUAN
Data saat ini cenderung multidimensi dan berdimensi tinggi serta lebih kompleks daripada data konvensional . Data yang berdimensi tinggi dan multidimensi memerlukan teknik pengurangan dimensi yang merupakan teknik penting dan menjadi teknik mendasar dalam sebagian besar proses data mining ketika menghadapi data yang kompleks [1] [2]. Tujuan dari teknik pengurangan dimensi adalah untuk mendapatkan representasi data baru yang dikelola menjadi dimensi lebih rendah [3]. Representasi data baru ditinjau dari sudut waktu dan kompleksitas komputasi yang jauh lebih efektif untuk pengolahan data berikutnya, misalnya untuk klasifikasi, assosiasi, prediksi, estimasi dan analisis pengelompokan [4]. Algoritma pengurangan dimensi yang ada seperti ISOMAP (Isometric Feature Mapping) LLE (Local Linear Embedded) [5] kernelPCA, Generalised Singular Value Decomposition (GSVD), Generaised Partial Leat Square (GPLS) dan Maximum Variancesce Unfolded (MVU) tergolong algoritma nonlinier [6] [7].
Principal component analysis (PCA), linear discriminant analysis (LDA), independent component analysis (ICA) singular value decomposition (SVD) [8] Canonical Correletion Analysis (CCA), Partial Least Square (PLS) dan Latent Semantic Indexing (LSI) tergolong algoritma linier [9] [10] . Algoritma Latent Semantic Indexing (LSI), adalah teknik aljabar linier yang menggunakan algoritma Singular value decomposition (SVD) untuk menguraikan matriks [6] [7]. Algoritma Canonical Correlation Analysis (CCA) adalah eksplorasi korelasi antara dua set sampel variabel kuantitatif, namun kekurangan algoritma CCA kurang akurat ketika jumlah unit data kurang dari jumlah variable terbesar dari kedua data set [10]. Algoritma Partial Least Squares (PLS) adalah teknik yang terkenal untuk ektraksi atribut, metode untuk menganalisis hubungan antara data set melalui variabel laten, namun lemah di proyeksi linier [11]. Algoritma Linear Discriminant Analysis (LDA) termasuk teknik ekstraksi atribut yang banyak digunakan [9] namun LDA tidak stabil pada data yang berdimensi tinggi pada kasus class scetter matrix untuk dijadikan nlai matrix singular (matriks tunggal) [12]. Algoritma Independent Component Analysis (ICA) dan FastICA (extension of ICA) termasuk teknik analisa komponen dasar sama seperti PCA, namun lemah di teknik pengurangan dimensi [13]. Teknik aljabar linier secara signifikan lebih sederhana dari Canonical Correlation Analysis (CCA), Partial Least Square (PLS) dan Linear Discriminant Analysis (LDA) [1]. Algoritma yang termasuk teknik aljabar linier adalah algoritma Singular Value Decomposition (SVD) dan Principal Component Analysis (PCA).
2
matematis untuk menguraikan matriks tunggal [16], dengan mengkompres menjadi tiga matriks yang lebih kecil dengan ukuran yang sama [17]. Cara kerjanya mengurangi data pada kolom dan baris [18] [19]. Salah satu alat analisis numerik yang efektif digunakan untuk menganalisis matriks [16] beroperasi di jantung algoritma seperti pada algoritma PCA dan LSI dan tergolong metode machine learning yang banyak dipakai [5] dan banyak diterapkan pada berbagai bidang, termasuk clustering pada data klinik [20], image compresing [21], wetermarking, [22] [23], klasifikasi dokumen [24], mapping gen [25] dan pencarian data series [26]. Algoritma Singular Value Decomposition (SVD) mempunyai kelebihan pada efisiensi waktu proses [15] untuk digunakan pada dataset yang berskala besar [27].Masalah pada data dibidang high dimentional biomedis disebabkan karena data tersebut memiliki ribuan atribut serta ribuan jumlah record yang saling berkorelasi (multicolinearity) dan menghasilkan data yang kompleks [25]. Data yang kompleks memiliki banyak kebisingan (noise), anomali (outlier), elemen yang hilang (missing value), tidak konsisten dan juga entitas data tidak berhubungan [28] [29]. Pada Penelitian ini menggunakan algoritma singular value decomposition untuk mengurangi dimensi pada high-dimentional biomedical data set
2. PENELITIAN TERKAIT
Beberapa penelitian yang terkait seperti pada penelitian. Shameek Biswas et al [25] pada penelitian ini menggabungkan keunggulan dua teknik multivariate yang berbeda pada pengurangan dimensi, yaitu algoritma Singular value decomposition (SVD) dengan Independent Component Analisys (ICA) hasil menunjukkan bahwa metode pengurangan dimensi berguna untuk menyelidiki arsitektur genetik variasi ekspresi gen. Taufik Fuadi Abidin [30] menggunakan algoritma Singular value decomposition untuk mengurangi dimensi pada dokumen. Hasil penelitian menunjukkan bahwa kualitas cluster sangat baik ketika dimensi telah dikurangi serta waktu proses dapat dikurangi secara signifikan, clustering dilakukan pada dimensi yang telah dikurangi serta hasil empiris menunjukkan bahwa SVD adalah teknik yang bagus untuk pengurangan dimensi. Rahmat Widia Sembiring et al [31] mengusulkan sebuah model untuk memproses data multidimensi untuk pengelompokan. Pada
penelitian ini menerapkan empat algoritma untuk pengurangan dimensi antara lain Algoritma SVD, principal component analysis (PCA), Self Organizing Map (SOM) dan FastICA dan membandingkan hasilnya dengan mengunakan atau tanpa algoritma pengurangan dimensi pada pengelompokan data (cluster). Hasil penelitian menunjukkan bahwa pengurangan dimensi secara signifikan mengurangi dimensi dan memperpendek waktu proses dan juga meningkatkan kinerja, pada hasil clister DBSCAN dengan SVD memiliki waktu proses tercepat pada beberapa dataset.
2.1Kontribusi penelitian
Perbedaan penelitian ini pada penelitian Shameek Biswas et al [25], Taufik Fuadi Abidin [30] dan pada penlitian Rahmat Widia Sembiring et al [31] kontribusinya pada metode yang diusulkan dan pada dataset yang diuji, pada penelitian ini dataset yang duji diambil dari:
http://datam.i2r.astar.edu.sg/datasets/krbd/.
3. METODE YANG DIUSULKAN
3
Gambar 1. Metode yang diusulkanGambar 2. Flowchart model yang diusulkan 3.1 Data Cleansing
Pembersihan data dimulai dengan proses keterpusatan untuk mengurangi data dengan mencari rata-rata setiap atribut, dengan menggunakan formula 𝑋̂ = 𝑋 − 𝑋̅ dimana 𝑋̂ adalah hasil setelah pemusatan, 𝑋 adalah kolom vektor dan 𝑋̅ adalah rata-rata dari kolom yang sesuai. Proses berpusat dilakukan untuk semua rangka dalam, jika nilai null ditemukan, nilai akan diganti dengan nilai rata-rata pada kolom tersebut, hasil dari proses centering dapat digunakan untuk menemukan scetter dengan menggunakan rumus scetter: 𝑋̂ ′ 𝑋̂ Hasil penyebaran dapat digunakan untuk menemukan nilai kovarian dengan menggunakan rumus 𝑘𝑜𝑣𝑎𝑟𝑖𝑎𝑛 =𝑋̂ ′ 𝑋̂𝑚−1
3.2 Data Denoising
Setelah dibersihkan data kemudian di denoising (menghilangkan noise dari data) pada penelitian ini menggunakan Algorithma Singular Value Decomposition (SVD) yang juga dapat digunakan sebagai alat untuk denosing
3.3 Data Normalisasi
Setelah data di denoising langkah berikutnya dinormalisasi. Pada penelitian ini menggunakan fungsi Min to max gunanya untuk mentransformasi variabel jarak (range) yang baru, seperti dari 0 ke 1. digunakan rumus berikut ini [32] [33]
𝑉𝑎𝑙𝑢𝑒′=
𝑉𝑎𝑙𝑢𝑒−𝑂𝑟𝑖𝑔𝑖𝑛𝑎𝑙𝑀𝑖𝑛
𝑂𝑟𝑖𝑔𝑖𝑛𝑎𝑙𝑀𝑎𝑥−𝑂𝑟𝑖𝑔𝑖𝑛𝑎𝑙𝑀𝑖𝑛(NewMax – 𝑁𝑒𝑤𝑀𝑖𝑛) +
𝑁𝑒𝑤𝑀𝑖𝑛
𝑉𝑎𝑙𝑢𝑒′ adalah hasil nilai normalisasi baru.
Value adalah variabel nilai asli. Original Min adalah nilai minimum dari variabel asli.
Original Max adalah nilai maksimum asli. Nilai Min baru dan Nilai Max baru adalah nilai untuk rentang normal [32] [33].
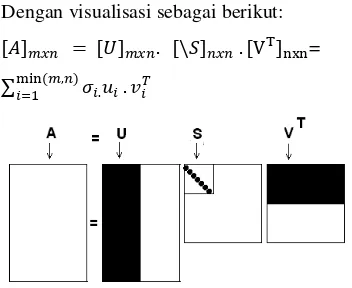
3.4 Algoritma Singular value Decomposition Algoritma Singular Value Decomposition (SVD) adalah faktorisasi nyata pada matriks kompleks, [34] [15] dimana dalam transformasi SVD, matriks asli dapat diuraikan menjadi tiga matriks dengan ukuran yang sama, namun jika dikalikan ketiga matriks yang telah diuraikan, maka akan sama dengan matriks asli [17] [16]. Persamaan algoritma SVD adalah:
𝐴 = 𝑈𝑆𝑉𝑇
Dengan visualisasi sebagai berikut: [𝐴]𝑚𝑥𝑛 = [𝑈]𝑚𝑥𝑛. [\𝑆]𝑛𝑥𝑛 . [VT]nxn=
∑min (𝑚,𝑛)𝑖=1 𝜎𝑖.𝑢𝑖. 𝑣𝑖𝑇
U adalah m x n matriks, S adalah n x n diagonal matriks, dan 𝑉T adalah matriks Kolom n x n, U disebut vektor kiri tunggal, {Uk} untuk membentuk ortonormal dasar, untuk uji ekspresi profil 𝑈𝑖 . 𝑈𝑗= 1 for i = j dan jika tidak 𝑈𝑖 . 𝑈𝑗= 0. Baris-baris 𝑉T mengandung unsur-unsur dari vektor singular kanan {Vk} dan membentuk basis ortonormal untuk tanggapan transkripsi gen. unsur-unsur S hanya nol pada diagonal dan disebut nilai-nilai singular , maka 𝑆 = 𝑑𝑖𝑎𝑛𝑔(𝑆1, ,….,𝑆𝑛) selanjutnya 𝑆𝑘 >0 untuk1 ≤ 𝑘 ≤ 𝑟 dan 𝑆1= 0 𝑓𝑜𝑟 (𝑟 + 1) ≤ 𝑘 ≤ 𝑛 dengan konvensi pemilahan urutan nilai-nilai tunggal dari vektor singular ditentukan oleh tinggi ke rendah, penyortiran nilai tunggal dimulai dengan nilai tunggal tertinggi di kiri atas dari indeks S matriks [35] [15] [17]. Dalam perhitungan SVD pertama-tama kita perlu menghitung nilai eigen dan vektor eigen dari 𝐴𝐴𝑇 𝑑𝑎𝑛 𝐴𝑇𝐴 . Vektor eigen dari 𝐴𝐴𝑇 bentuk
4
4. HASIL PENELITIAN DANPEMBAHASAN
Sumber data eksperimen pada penelitian ini diunduh dari:
http://datam.i2r.astar.edu.sg/datasets/krbd/. Dengan hasil sebagai berikut:
Tabel.1 Eksperiment menggunakan Dataset Breast_Cancer
item Result
Process time 1 menit 1 detik keep_variancesce threshold 0.05 Jumlah Atribut 24482 Dimensionality Reduction
Result
23 attribute
Mean 0.001
Variance 0.001
Korelasi antar atribut 0.01 s/d 0.03
Pada uji dataset high dimentional biomedical data set breast cancer atributnya sebanyak 24482, setelah di kurangi dimensi menjadi 23 atribut dengan presentase treshold 0.05 lama proses 1 menit 1 detik
Tabel. 2 Eksperiment menggunakan Dataset Colon_Tumor
Item Result
Process time 14 menit 16 detik keep_variancesce
threshold
0.05
Jumlah Atribut 2000 Dimensionality Reduction
Result
45 attribute
Mean 0.000 Variance 0.001 Korelasi antar atribut 0.01 s/d 0.03
Pada uji dataset high dimentional biomedical data set colon tumor atributnya sebanyak 2000, setelah di kurangi dimensi menjadi 45 atribut dengan presentase treshold 0.05 lama proses 14 menit 16 detik
Tabel. 3 Eksperiment menggunakan Dataset Lunc_Cancer
item Result
Process time 1 menit 6 detik keep_variancesce
threshold
0.05
Jumlah Atribut 12534 Dimensionality
Reduction Result
8 attribute
Mean 0.032
Variance 0.001 Korelasi antar atribut 0.001 s/d 0.03
Pada uji dataset high dimentional biomedical data set lunch cancer atributnya sebanyak 12534, setelah di kurangi dimensi menjadi 8 atribut dengan presentase treshold 0.95 lama proses 1 menit 6 detik menggunakan bantuan
Tabel. 4 Eksperiment pada Dataset Lunc_Michigan
item Result
process time 20 menit keep_variancesce threshold 0.05
Jumlah Atribut 7130 Dimensionality Reduction
Result
23 attribute
Mean 0.022 Variance 0.001 Korelasi antar atribut 0.01 s/d
0.03
Pada uji dataset high dimentional biomedical data set lunch michigan atributnya sebanyak 7130, setelah di kurangi dimensi menjadi 23 atribut dengan presentase treshold 0.05 lama proses 20 menit 6 detik
5
5. KESIMPULAN.Hasil eksperimen yang dilakukan terbukti bahwa algoritma Singular Value Decomposition (SVD) mampu mengurangi jumlah atribut dengan keep_variancesce threshold 0.05, nilai mean dan varians mendekati angka 0, yang artinya hasil data ektraknya terpusat dan tidak menyebar, serta prosesnya sangat cepat namun korelasi antar atribut menghasilkan nilai 0.01 sampai dengan 0.03 yang artinya korelasi antar atribut masih berkorelasi. Rencana penelitian selanjutnya peneliti akan menggunakan algoritma Principal Component Analysis (PCA) dengan menggunakan dataset yang sama.
REFERENCES:
[1] Shanwen Zhang; Rongzhi Jing;,
“Dimension Reduction Based on
Modified Maximum Margin Criterion for
Tumor Classification,” dalam Fourth
International Conference on Information and Computing, China, 2011.
[2] M. Revathi, “Network Intrusion Detection
System Using Reduced Dimensionality,”
Indian Journal of Computer Science and Engineering (IJCSE), vol. 2 , no. 1, pp. 61-67, 2010.
[3] Longcun Jin; Wanggen Wan; Yongliang Wu; Bin Cui; Xiaoqing Yu; Youyong
Wu;, “A Robust High-dimensional Data
Reduction Method,” The International
Journal of Virtual Reality,, vol. 9, no. 1, pp. 55-60, 2010.
[4] R. Krakovsky; R. Forgac;, “Neural Network Approach to Multidimensional Data Classification via Clustering,” dalam IEEE 9th International Symposium on Intelligent Systems and Informatics , Serbia, 2011.
[5] Panagis Magdalinos; Christos Doulkeridis; Michalis Vazirgiannis;,
“Enhancing Clustering Quality through
Landmark-Based Dimensionality
Reduction,” ACM Transactions on
Knowledge Discovery from Data, vol. 5, no. 2, pp. 1-44, Februari 2011.
[6] Md. Monjurul Islam; A. S. M. Latiful
Hoque;, “Automated Essay Scoring Using Generalized Latent Semantic Analysis,”
Journal of Computer, vol. 7, no. 3, pp. 616-626, March 2012.
[7] Altangerel Chagnaa; Cheol-Young Ock; Chang-Beom Lee; Purev Jaimai;,
“Feature Extraction of Concepts by Independent Component Analysis,”
International Journal of Information Processing Systems, vol. 3, no. 1, pp. 33-37, June 2007.
[8] Ethem Alpaydın, Introduction to Machine Learning, Second penyunt., T. Dietterich, C. Bishop, D. Heckerman dan M. Jorda, Penyunt., London,: Cambridge, Massachusetts, 2010.
[9] Ignacio Gonzalez;Sebastien Dejean;Pascal G. P. Martin;Alain Baccini,
“CCA: An R Package to Extend
Canonical,” Journal of Statistical
Software, vol. 23, no. 12, pp. 1-14, January 2008.
[10] Matthias Scholz; Martin Fraunholz;
Joachim Selbig;, “Nonlinear Principal
Component Analysis Neural Network
Models and Applications,” dalam Federal Ministry of Education and Research (BMBF), German , 2008.
[11] Liang Sun; Shuiwang J; Shipeng Yu;
Jieping Ye;, “On the Equivalence
Between Canonical Correlation Analysis and Orthonormalized Partial Least
Squares,” The International journal of Multimedia & Its Applications (IJMA), vol. 2, no. 3, pp. 1230-1235, Agustus 2010.
[12] Neeta Nain; Prashant Gour; Nitish Agarwal; Rakesh P Talawar; Subhash
Chandra;, “Face Recognition using PCA
and LDA with Singular Value
Decomposition(SVD) using 2DLDA,”
dalam Proceedings of the World Congress on Engineering, London, U.K, 2008. [13] Fangzhou Yao; Jeff Coquery; Kim-Anh
Lê Cao;, “Independent Principal
Component Analysis for biologically meaningful dimension reduction of large
biological data sets,” IEEE Transaction On Computational Biology and Bioinformatics, vol. 13, no. 24, pp. 1-15, 2012.
[14] Stan Lipovetsky;, “PCA and SVD with
nonnegative loadings,” GfK Custom
Research for excelence, vol. 42, no. 1, pp. 1-30, Januari 2009.
6
“Dimension Reduction of Health Data
Clustering,” International Journal on New Computer Architectures and Their Applications (IJNCAA), vol. 3, no. 1, pp. 1041-1050, 2011.
[16] S.M. Rafizul Haque, “Singular Value
Decomposition and Discrete,” Sweden,
2008.
[17] Taufik Fuadi Abidin ; Bustami Yusuf ;
Munzir Umran;, “Singular Value
Decomposition for Dimensionality Reduction in Unsupervised Text Learning
Problems,” dalam 2nd International
Conference on Education Technology and Computer (ICETC), Banda Aceh-Indonesia, 201O.
[18] Taro Konda; Yoshimasa Nakamura;, “A new algorithm for singular value
decomposition and its parallelization,”
Parallel Computing, vol. 02, no. 001, pp. 1-14, 2009.
[19] C.Venkata Narasimhulu; K.Satya Prasad;,
“A Robust Watermarking Technique
based on Nonsubsampled Contourlet
Transform and SVD,” International
Journal of Computer Applications, vol. 16, no. 8, pp. 27-36, February 2011. [20] Kumar, Nishith; Mohammed Nasser ;
Subaran Chandra Sarker;, “A New
Singular Value Decomposition Based Robust Graphical Clustering Technique
and Its Application in Climatic Data,”
Journal of Geography and Geology, vol. 3, no. 1, pp. 227-238, September 2011. [21] Pritha.D.N; L.Savitha; Shylaja.S.S ;,
“Face Recognition by Feedforward
Neural Network using Laplacian of Gaussian filter and Singular Value
Decomposition,” dalam IEEE
International Conference on Data Engineering, India, 2010.
[22] Hu Zhihua, “Binary Image Watermarking
Algorithm Based on SVD,” dalam
International Conference on Intelligent Human-Machine Systems and Cybernetics, China, 2009.
[23] Satyanarayana Murty. P; M.Uday
Bhaskar; P. Rajesh Kumar;, “A Semi -Blind Reference Watermarking Scheme Using DWT-DCT-SVD for Copyright
Protection,” International Journal of Computer Science & Information
Technology (IJCSIT), vol. 4, no. 2, pp. 69-82, April 2012.
[24] Lailil Muflikhah; Baharum Baharudin;,
“Document Clustering using Concept
Space and Cosine Simila rity
Measurement,” dalam 2009 International
Conference on Computer Technology and Development, Malaysia, 2009.
[25] Biswas, Shameek; Storey, John D; Akey,
Joshua M, “Mapping gene expression
quantitativetrait loci by singular value decomposition and independent
component analysis,” IEEE Transaction On Computational Biology and Bioinformatics, vol. 5, no. 7, pp. 1-14, May 2008.
[26] Muhammad Marwan Muhammad Fuad; Pierre-François Marteau, “Speeding-up the Similarity Search in Time Series Databases by Coupling Dimensionality Reduction Techniqueswith a
Fast-and-dirty Filter,” dalam IEEE International Conference on Semantic Computing, Vannes, France, 2010.
[27] Mario Navas; Carlos Ordonez;, “Efficient
computation of PCA with SVD in SQL,”
dalam ACM, Paris, 2009.
[28] Sanga, S; Chou, T.Y; Cristini, V;
Edgerton, M.E;, “Neural Network with K
-Means Clustering via PCA for Gene Expression Profile Analysis,” IEEE - Computer Science and Information Engineering, vol. 3, pp. 670-673, April 2009.
[29] Tajunisha; Saravanan;, “Performance analysis of k-means with different initialization methods for high
dimensional data,” International Journal of Artificial Intelligence & Applications (IJAIA),, vol. 1, no. 4, pp. 44-52, October 2010.
[30] Taufik Fuadi Abidin; Bustami Yusuf;
Munzir Umran;, “Singular Value
Decomposition for Dimensionality Reduction in Unsupervised Text Learning
Problems,” dalam 2nd International
Conference on Education Technology and Computer (ICETC), Banda Aceh - Indonesia, 2010.
[31] Rahmat Widia Sembiring; Jasni Mohamad Zain; Abdullah Embong;,
7
New Computer Architectures and Their Applications (IJNCAA), vol. 1, no. 3, pp. 1041-1050, 2011.[32] Glenn J. Myatt, Making Sense of Data A Practical Guide to Exploratory Data Analysis and Data Mining, Hoboken: John Wiley & Sons, Inc, 2007.
[33] Sembiring , Rahmat Widia; Jasni ,
Mohamad Zain;, “The Design of Pre
-Processing Multidimensional Data Based
on Component Analysis,” Computer and
Information Science, vol. 4, no. 3, pp. 106-115, May 2011.
[34] Luis M. Ledesma-Carrillo; Eduardo Cabal-Yepez; Rene de J. Romero-Troncoso; Roque A. Osornio-Rios; Tobia
D. Carozzi;, “Reconfigurable FPGA
-Based Unit for Singular Value
Decomposition of Large m × n Matrices,”
dalam International Conference on Reconfigurable Computing and FPGAs, Mexico & Sweden, 2011.
[35] Jha, Sunil K.; R. D. S. Yadava;,
“Denoising by Singular Value
Decomposition and Its Application to
Electronic Nose Data Processing,” IEEE
Sensor Journal, vol. 11, no. 1, pp. 35-44, January 2011.
[36] Hsinchun Chen; Mihail C. Roco;, “Global and Longitudinal Patent and Literature
Analysis,” dalam Mapping Nano
Technology Innovations and Knowledge, P. R. Sharda dan P. D. Stefan, Penyunt., Tucson, Arizona, Arlington, Virginia, springer.com, 2009, pp. 1-321.
[37] Jiawei Han ; Micheline Kamber;, Data Mining: Concepts and Techniques, Second penyunt., A. Stephan, Penyunt., San Francisco: Morgan Kaufmann Publishers is an imprint of Elsevier, 2007. [38] Karunaratne, Thashmee ; Boström,
Henrik; Norinder, Ulf;, “Pre-Processing Structured Data for Standard Machine Learning Algorithms by Supervised Graph Propositionalization - a Case Study
with Medicinal Chemistry Datasets,”
dalam International Conference on Machine Learning and Applications, Sweden, 2010.
[39] D.Napoleon; S.Pavalakodi;, “A New Method for Dimensionality Reduction using K Means Clustering Algorithm for
High Dimensional Data Set,”
International Journal of Computer Applications (0975 – 8887), vol. 13, no. 7, pp. 41-46, January 2011.
[40] B. B. Zhao and Y. Q. Chen, “Singular value decomposition (SVD) for extraction of gravity anomaly associated with gold mineralization in Tongshi gold
field,Western Shandong Uplifted Block,
Eastern China,” Beijing- China, 2011. [41] Jun Yan, Benyu Zhang, Ning Liu;
Shuicheng Yan; Qiansheng Cheng; Weiguo Fan; Qiang Yang; Wensi Xi;
Zheng Chen;, “Effective and Efficient
Dimensionality Reduction for
Large-Scale and Streaming Data Preprocessing,”
IEEE Transaction On Knowledge And Data Engineering, vol. 18, no. 2, pp. 1-14, Febbruari 2008.
[42] Ali Shadvar, “Dimension Reduction by Mutual Information discriminant
analysis,” International Journal of
Artificial Intelligence & Applications (IJAIA), vol. 3, no. 3, pp. 23-35, May 2012.
[43] Chang, Cheng-Ding; Wang, Chien-Chih;
Jiang, Bernard C;, “Singular Value
Decomposition Based Feature Extraction Technique for Physiological Signal
Analysis,” Journal of Medical Systems., vol. 36, no. 3, pp. 1769 - 1777, June 2012. [44] Taufik Fuadi Abidin; Bustami Yusuf;
Munzir Umran;, “Singular Value
Decomposition for Dimensionality Reduction in Unsupervised Text Learning
Problems,” dalam 2nd International
Conference on Education Technology and Computer (ICETC), Banda Aceh - Indonesia, 201O.
[45] G.kalaivani; k.aravi;, “dimensionality reduction techniques for document clustering- a survey,” international journal for trends in engineering & technology, vol. 3, no. 3, p. 80=83, 2015. [46] Eko prasetyo, “reduksi dimensi set data dengan drc pada metode klasifikasi svm dengan upaya penambahan komponen