KEANEKARAGAMAN KETAM DI SUNGAI YANG BERHULU
DARI GUNUNG SALAK, BOGOR, JAWA BARAT
Ahmad Nasokha
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
AHMAD NASOKHA. Keanekaragaman ketam di Sungai yang Berhulu dari Gunung Salak, Bogor, Jawa Barat. Dibimbing oleh ACHMAD FARAJALLAH dan YUSLI WARDIATNO.
Ketam merupakan anggota subfilum Crustacean, ordo Decapoda dan infraordo Brachyura yang dapat hidup di air tawar. Ketam merupakan salah satu kelompok makro-invertebrata yang penting dalam ekologi perairan. Tujuan dari penelitian ini adalah untuk mengetahui
keanekaragaman ketam di sungai-sungai yang berhulu dari Gunung Salak, Bogor, Jawa Barat. Ketam diambil di sebelas titik pengambilan. Kepastian spesies dilakukan dengan identifikasi morfologi dan diverifikasi menggunakan DNA barcode. Di daerah Gunung Salak ditemukan tiga spesies ketam, yaitu Geosesarma sp., Parathelphusa bogoriensis dan Parathephusa convexa.
Ketiganya merupakan ketam yang sudah dikenal distribusinya di Jawa. Geosesarma sp. dapat
ditemukan di sela-sela bebatuan di sekitar sungai. Parathelphusa bogoriensis dapat ditemukan di
dasar sungai atau di bawah bebatuan. Parathelphusa convexa dapat ditemukan di pinggir-pinggir
sungai yang bersemak.
Kata kunci : ketam, keanekaragaman, DNA barcode, relung ekologi
ABSTRACT
AHMAD NASOKHA. Diversity of Freshwater Crabs in River of Salak Mountain, Bogor, West Java. Supervised by ACHMAD FARAJALLAH and YUSLI WARDIATNO.
Freshwater crab is decapods crustacean of infraorder Brachyura. Freshwater crabs are one of the most ecologically important macro-invertebrate groups in tropical inland waters. The objective of this research was to assess the diversity of freshwater crabs in rivers of mount Salak, Bogor, West Java. Freshwater crabs were taken from eleven sites. Identification based on morphological characteristic and verified by DNA barcode. Three species of freshwater crabs were found Salak Mountain, that are Geosesarma sp., Parathelphusa bogoriensis and Parathelphusa convexa.Those three species were commonly found in Java. Geosesarma sp. can be found around
rocks on river banks. Parathelphusa bogoriensis can be found at the brook or under rocks. Parathelphusa convexa can be found on river banks.
KEANEKARAGAMAN KETAM DI SUNGAI YANG BERHULU
DARI GUNUNG SALAK, BOGOR, JAWA BARAT
AHMAD NASOKHA
Skripsi
sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biologi
DEPARTEMEN BIOLOGI
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi : Keanekaragaman Ketam di Sungai yang Berhulu dari Gunung
Salak, Bogor, Jawa Barat
Nama
: Ahmad Nasokha
NIM
: G340801020
Disetujui,
Dr. Ir. Achmad Farajallah, M.Si
Dr. Ir. Yusli Wardiatno, M.Sc
Pembimbing I
Pembimbing II
Diketahui,
Dr. Ir. Ence Darmo Jaya Supena, M.Si.
Ketua Departemen Biologi
PRAKATA
Puji dan syukur penulis panjatkan kehadirat Allah SWT atas limpahan rahmat dan hidayah-Nya sehingga penulis dapat menyelesaikan skripsi ini dengan baik. Shalawat serta salam penulis panjatkan kepada Nabi Muhammad SAW. Penelitian yang dilaksanakan pada bulan Januari sampai Maret 2012 yang berjudul Keanekaragaman Ketam Air Tawar di Sungai yang Berhulu dari Gunung Salak, Bogor, Jawa Barat merupakan salah satu syarat untuk memperoleh gelar sarjana di Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Ucapan terima kasih penulis sampaikan kepada Bapak Dr. Ir. Achmad Farajallah, M.Si dan Bapak Dr. Ir. Yusli Wardiatno, M.Sc selaku pembimbing yang telah memberikan ilmu, pengarahan, motivasi dan bimbingannya kepada penulis. Penulis mengucapkan terima kasih kepada Dr. Ir. Sulistyorini M. Si selaku penguji yang telah memberikan kritik dan sarannya dalam penyusunan skripsi ini. Ucapan terima kasih juga penulis sampaikan kepada keluarga besar ayah saya Bapak Koderi dan Ibu Dasuti, kakak (Mu’minatun, Fathurrohman, Nur Khasanah, Mohammad Soffa) dan adik (Achmad Khaerozi, Isna Inayati, Isna Izayati, Khaenur Rofik, Zumratun Nikmah, Naelatus Sa’adah, Fia Maftahurrahmah, Adib Khoerul Masulin) yang telah memberikan kasih sayangnya, perhatian, do’a dan dukungan baik moral maupun material. Penulis mengucapkan kepada pihak Karya Salemba Empat (KSE) yang telah memberikan beasiswa kepada penulis. Ucapan terima kasih penulis sampaikan kepada kelurga besar zoologi (Pak Bambang, Ibu Kanthi, Pak Islamul, Ibu Ana, Mba Puji, Kak Sarah Nila, Kak Nunus, Kak Iqbal, Kak Wildan, Mba Tini, Mba Ani, Pak Adi, Yanti, Lili, Delvi, Dalfit, Agus s, Adit, Agus H, Amar, Shinta Anggraini) dan Ibu Siti selaku laboran Departemen MSP bagian Biologi Mikro 1 yang telah memberikan ilmu dan segala dukungannya. Penulis mengucapkan terima kasih kepada keluarga OWA, khususnya OWA 11 (Shinta Anggraini, Dini, Siti, Titi, Uun, Agil, Ipeh,) dan juga OWA 12 dan 13 yang telah memberikan saran dan dukungannya. Terima kasih penulis ucapkan kepada temen-temen Biologi 45 yang telah memberikan dukungannya. Ucapan terima kasih terakhir penulis sampaikan kepada teman-teman satu kosan (Rama, Tri, Tedi, Arif, Faqih, Agus, Wendhi, Wahyu, Afnan, Santo, Fikri, Bambang) yang telah merelakan waktunya untuk bertukar pikir.
Semoga karya tulis ini dapat bermanfaat dan menambah wawasan, ilmu pengetahuan kita semua. Saran dan kritik yang membangun sangat penulis harapkan untuk penulisan yang lebih baik.
Bogor, September 2012
Ahmad Nasokha
RIWAYAT HIDUP
Penulis dilahirkan di Batang pada tanggal 06 April 1990 sebagai anak kelima dari tiga belas bersaudara dari pasangan Bapak Koderi dan Ibu Dasuti. Penulis menyelesaikan pendidikan, MII Candi Areng pada tahun 2002, dan SMP Negeri 02 Warung Asem lulus tahun 2005. Pada tahun 2008 penulis lulus dari SMA Negeri 1 Kedungwuni dan pada tahun yang sama diterima di Institut Pertanian Bogor (IPB) melalui jalur Seleksi Nasional Mahasiswa Perguruan Tinggi Negeri (SNMPTN).
Penulis aktif sebagai anggota Observasi Wahana Alam (OWA) Himabio pada tahun 2009 sampai sekarang. Penulis juga aktif sebagai anggota Himabio pada tahun 2009-2010 sebagai anggota Bidang OWA, dan 2010-2011 sebagai Ketua Bidang OWA. Penulis aktif dalam beberapa kegiatan kampus diantaranya Pesta Sains, Revolusi Sains II, Exam Bodogol, Wonder-pul, Tatap Muka Beasiwa KSE, Pradiklat dan Diklat OWA angkatan 12, Pradiklat dan Diklat OWA angkatan 13, Pananaman Seribu Pohon
Penulis melakukan Studi Lapang pada tahun 2010 mengenai Keanekaragaman Moluska Laut di Pananjung Pangandaran. Penulis melakukan Praktik Lapang pada tahun 2011 mengenai Budidaya Ikan Mas (Cyprinus carpio) di PT Aldepos Salaca, Desa Tapos, Tenjolaya, Bogor.
DAFTAR ISI
Halaman
DAFTAR TABEL ... viii
DAFTAR GAMBAR ... viii
DAFTAR LAMPIRAN ... viii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 1
BAHAN DAN METODE ... 1
Waktu dan Tempat ... 1
Area sampling ... 2
Amplifikasi DNA barcode ... 2
HASIL ... 3
Koleksi sampel ketam ... 3
Identifikasi morfologi ketam... 3
Analisis DNA barcode ... 4
PEMBAHASAN ... 6
SIMPULAN ... 6
DAFTAR PUSTAKA ... 6
DAFTAR TABEL
Halaman
1 Hasil pengambilan sampel ketam ... 3
2 Jumlah perbedaan nukleotida gen CO1 antara spesies ketam (diatas garis diagonal) dan jarak genetik gen CO1 antara spesies ketam berdasarkan model subtitusi K2P (dibawah garis diagonal) ... 5
DAFTAR GAMBAR
Halaman 1 Peta pengambilan sampel ketam ... 22 Karakteristik morfologi P. convexa ... 4
3 Karakteristik morfologi P. bogoriensis ... 4
4 Karakteristik morfologi Geosesarma sp. ... 5
5 Rekonstruksi pohon filogeni berdasarkan mtCO1 dengan NJ bootstrap1000X ... 5
DAFTAR LAMPIRAN
Halaman 1 Ekstraksi dan isolasi DNA dengan DNA Extraction Kit for Fresh Blood (GeneAid) yang dimodifikasi untuk jaringan otot yang diawetkan dalam etanol ... 92 Daftar pengambilan sampel ketam di sungai-sungai yang berhulu dari Gunung Salak ... 10
PENDAHULUAN
Latar Belakang
Ketam merupakan anggota subfilum Crustaceae, ordo Decapoda dan infraordo Brachyura yang hidup di air tawar. Anggota Brachyura yang hidup di air tawar adalah anggota famili Pseudothelphusidae, Tricho-dactylidae, Potamonautidae, Potamoidae dan Gecarcinucidae (Cumberlidge et al. 2009). Ketam mempunyai lima pasang kaki. Bagian mulut ketam ditutupi oleh maksileped yang rata, dan bagian depan dari karapas tidak membentuk sebuah rostrum yang panjang (Dixon et al. 2004). Insang ketam tersusun
oleh filamen-filamen yang pipih ( phyllobran-chiate) (Taylor & Taylor 1992). Beberapa
karakter morfologi yang biasa dijadikan karakter taksonomi adalah bentuk karapas, bentuk abdomen, pleopod pertama dan kedua, maksileped ketiga, palpus mandibular, dan meri ambulatori (Ng 1990; Ng 1991; Ng 2004; Cai & Ng 2001; Klaus et al. 2006).
Ketam dapat digunakan sebagai indikator perairan dalam ekosistem air tawar. Beberapa ketam hanya dapat ditemukan di perairan bersih. Ketam merupakan salah satu hewan macro-invertebrata yang penting dalam jejaring makanan yaitu sebagai omnivora sekaligus ditritifora. Ketam juga menjalin simbiosis dengan beberapa hewan lainnya, salah satunya adalah simbiosis dengan trematoda dari genus Paragonimus yang
menyebabkan penyakit paragonimiasis. Ketam juga berperan sebagai inang larva Simulium
spp. yang merupakan vektor parasit
Onchocerca volvulus yang menyebabkan
penyakit onchocerciasis (Cumberlidge et al.
2009).
Beberapa anggota Brachyura juga dapat hidup di air laut dan air payau. Famili Portunidae merupakan salah satu anggota Brachyura yang dapat hidup di air laut atau air payau. Anggota Portunidae dicirikan memiliki kaki renang yang fungsional dan insang yang tidak termodifikasi menjadi pelat-pelat insang yang kaku. Anggota Portunidae tidak dapat hidup di luar air dalam jangka waktu yang lama. Anggota Brachyura lainnya dapat hidup di air payau yaitu famili Varunidae dan Sesarmidae (Ng 2004). Anggota Varunidae dan Sesarmidae dicirikan memiliki insang yang dilengkapi dengan filamen-filamen insang yang mengeras dan berbentuk vascular. Filamen-filamen ini berfungsi mempertahan-kan kelembaban ruang insang selama di darat.
Kaki renang pada anggota famili Varunidae dan Sesarmidae tereduksi sehingga menyebabkan anggota famili ini tidak dapat hidup di air laut dalam jangka waktu yang lama dan menyeberangi lautan. Hal ini sama seperti anggota ketam pada umumnya. Perbedaannya terletak pada siklus hidupnya. Anggota Varunidae dan Sesarmidae bersifat migran yaitu siklus hidupnya membutuhkan air payau sedangkan seluruh siklus hidup ketam terdapat di perairan tawar. Anggota Verunidae dan Sesarmidae akan ke perairan payau untuk bereproduksi dan meletakkan telur. Telur akan berkembang menjadi juvenil di perairan payau dan akan ke parairan tawar pada saat dewasa (Morris 2001).
Restriksi habitat ketam didikte oleh proses pembentukan pulau-pulau karena ketam yang tidak dapat menyeberangi lautan. Barrier ini
menyebabkan ketam yang berasal dari satu nenek moyang dapat menjadi beberapa spesies ketam baru. Dengan begitu, jika satu spesies ketam bisa ditemukan di wilayah yang lebih luas maka proses spesiasinya bisa diartikan lebih lama dibanding pembentukan wilayah tersebut, dan sebaliknya. Indonesia merupakan daerah interaksi antara empat lempeng utama, yaitu Eurasia, Pasifik, Filipina dan Indo-Australia (Yugo 1994). Interaksi lempeng-lempeng utama ini menyebabkan Indonesia tersusun atas pulau-pulau sehingga Indonesia memiliki potensi biodiversitas ketam yang tinggi.
Setiap spesies memiliki posisi dan menempati suatu ruang berdasarkan rentang fisik yang ditempatinya dan peranan yang dilakukan dalam komunitasnya yang disebut relung ekologi (Morris 1992). Setiap spesies memiliki relung ekologi yang berbeda sehingga jarang terjadi tumpang tindih antara spesies satu dengan lainnya. Relung ekologi suatu spesies erat kaitannya dengan sumber makanan, predator, dan kompetitor (Leibold 2003).
Tujuan
Penelitian ini bertujuan untuk menge-tahui keanekaragaman ketam di sungai yang berhulu dari Gunung Salak, Bogor, Jawa Barat.
BAHAN DAN METODE
Waktu dan Tempat2
dari Gunung Salak, Bogor, Jawa Barat. Ana-lisis data dilakukan di Bagian Biosistematik dan Ekologi Hewan Departemen Biologi FMIPA IPB.
Area sampling
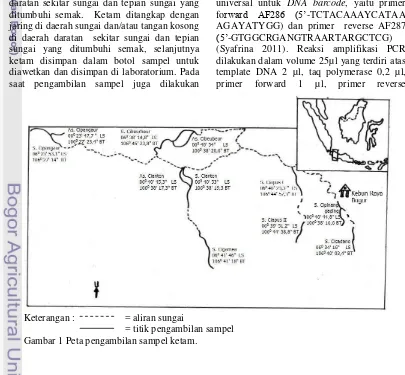
Gunung Salak dengan puncak di titik 6°43' LS dan 106°44' BT mengalirkan aliran sungai ke wilayah Kabupaten Bogor dan Kabupaten Sukabumi, salah satu yang terbesar adalah sungai Cisadane. Pengambilan sampel dilakukan di 11 titik sampling yaitu Sungai Cigamea, Sungai Cianten, Anak sungai Cianten, Sungai Cisadane, Sungai Cipinang Gading, Sungai Ciapus I, Sungai Ciapus II, Sungai Cipangaur, Anak sungai Cipangaur, sungai Cibebeur, dan Anak Sungai Cibebeur (Gambar 1).
Koleksi ketam
Pemetaan lokasi sampling secara global dilakukan sebelum penangkapan dilakukan. Hasil pemetaan digunakan untuk memilih lokasi secara purposive, yaitu memilih lokasi
yang relatif mudah dijangkau. Dari satu titik lokasi kemudian dilanjutkan dengan road sampling, yaitu berjalan di daerah sungai,
daratan sekitar sungai dan tepian sungai yang ditumbuhi semak. Ketam ditangkap dengan jaring di daerah sungai dan/atau tangan kosong di daerah daratan sekitar sungai dan tepian sungai yang ditumbuhi semak, selanjutnya ketam disimpan dalam botol sampel untuk diawetkan dan disimpan di laboratorium. Pada saat pengambilan sampel juga dilakukan
penulisan data pengambilan sampel, diantaranya waktu pengambilan, lokasi pengambilan, jumlah spesimen yang didapat, alat tangkap dan posisi pengambilan.
Identifikasi ketam
Identifikasi ketam dilakukan berdasarkan kunci identifikasi yang disusun oleh Ng (2004). Beberapa karakter taksonomi yang digunakan adalah bentuk karapas, bentuk abdomen, pleopod pertama dan kedua, maksileped ketiga, palpus mandibular, dan meri ambulatori.
Amplifikasi DNA barcode
Beberapa spesimen yang tampak berbeda secara morfologi dan menampilkan bentuk yang berbeda kemudian dipilih untuk dianalisis lebih lanjut menggunakan DNA barcode.
Jaringan yang diambil sebagai sumber DNA adalah jaringan otot dari kaki jalan ke-5. Ekstraksi DNA dilakukan menggunakan DNA Extraction Kit for Fresh Blood (Geneaid) yang
telah dimodifikasi untuk jaringan otot yang disimpan dalam etanol (Lampiran 1).
Ruas gen CO1 genom mitokondria kemudian diamplifikasi menggunakan primer universal untuk DNA barcode, yaitu primer
forward AF286 (5’-TCTACAAAYCATAA AGAYATYGG) dan primer reverse AF287 (5’-GTGGCRGANGTRAARTARGCTCG) (Syafrina 2011). Reaksi amplifikasi PCR dilakukan dalam volume 25µl yang terdiri atas template DNA 2 µl, taq polymerase 0,2 µl, primer forward 1 µl, primer reverse
Keterangan : = aliran sungai
3
1 µl, dNTP 1,5 µl, MgCl2 1,5 µl, buffer 2,5 µl
dan air destilata 15,3 µl. Reaksi PCR dilakukan menggunakan mesin thermocycler ESCO
MX-BLC-7. Reaksi dilakukan pada kondisi pradenaturasi pada suhu 940C selama 5 menit,
selanjutnya dilakukan 30 siklus yang terdiri atas proses denaturasi pada suhu 940 selama 1
menit, penempelan primer pada suhu 550C selama 1,5 menit, pemanjangan pada suhu 720C selama 2 menit dan yang terakhir
pemanjangan akhir pada suhu 720C selama 5
menit.
Kualitas amplikon diuji menggunakan elektroforesis gel poliakrilamide 5% dalam bufer TBE yang dilanjutkan dengan pewar-naan sensitif perak. Pengujian amplikon dijalankan pada tegangan 200 V selama 50 menit atau sampai pelacak warna bromtimol blue mencapai batas bawah gel. Amplikon
yang menunjukkan pita tunggal di atas gel poliakrilamid kemudian dijadikan cetakan dalam reaksi amplifikasi untuk perunutan nukleotida menggunakan primer yang sama dengan amplifikasi awal. Metode perunutan nukleotida dilakukan dengan metode big dye terminator yang dibaca oleh mesin pembaca
otomatis ABI (Macrogen Inc, Korea).
HASIL
Koleksi sampel ketam
Hasil sampling memperoleh 112 spesimen ketam, yang terdiri atas 54 ekor Parathelphusa convexa 28 ekor Parathelphusa bogoriensis,
dan 30 ekor Geosesarma sp. (Tabel 1). Data pengambilan sampel dapat diketahui bahwa P. convexa dapat ditemukan di
semak-semak pinggir sungai, P. bogoriensis dapat
ditemukan di dasar sungai dan di bawah batu sedangkan Geosesarma sp. dapat ditemukan di
sela-sela batu di pinggir sungai (Lampiran 2).
Identifikasi morfologi ketam
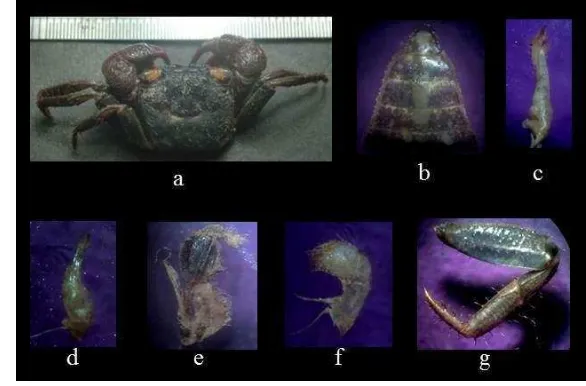
Berdasarkan morfologi luar, ketiga spesies ketam yang ditemukan relatif mudah dibedakan. P. convexa dicirikan dengan karapas berbentuk mirip trapesium, sisi-sisi cembung, permukaan cembung; mata relatif kecil, di samping terdapat tiga duri (Gambar 2a); abdomen menyerupai huruf “T”, tersusun atas 5 segmen (Gambar 2b); pleopod pertama bagian proksimal tidak memiliki celah, pinggir terdapat banyak rambut (Gambar 2c); pleopod kedua memiliki flagellum memanjang (Gambar 2d); maksileped ketiga rata, tanpa celah ditengahnya dan memiliki flagellum yang panjang (Gambar 2e); palpus mandibular dua lobus, di pinggirnya banyak rambut (Gambar 2f); meri ambulatori antara segmen merus dan karpus terdapat celah (Gambar 2g). Ciri-ciri P. bogoriensis hampir sama dengan P. convexa (Gambar 3a-f). Keduanya bisa
dibedakan berdasarkan permukaan karapasnya, yaitu pada P. bogoriensis lebih rata dan
segmen merus dan karpus dari meri ambulatorinya tidak memiliki celah (Gambar 3g).
Geosesarma sp. dicirikan dengan bentuk
karapas yang cenderung segi empat, permukaan rata; mata relatif besar (Gambar 4a); abdomen berbentuk parabola, telson meruncing, tersusun atas 5 segmen (Gambar 4b); pleopod pertama terdapat celah di tengah, ada rambut-rambut di ujungnya (Gambar 4c), ukuran lebih besar dan panjang dibanding pleopod kedua yang tidak berambut diujungnya (Gambar 4d); maksileped ketiga berbentuk cembung, ada celah di tengah, ada flagellum yang membulat kebelakang (Gambar 4e); palpus mandibular memiliki satu lobus, berambut (Gambar 4f); meri ambulatori memiliki duri-duri halus, celah antara segmen merus dan karpus (Gambar 4g).
4
Analisis DNA barcode
Sampel yang secara morfologi berbeda selanjutnya diverifikasi menggunakan DNA barcode. Enam runutan DNA CO1 ketam disejajarkan dengan ruas mtDNA yang homolog dari Nautilothelphusa zimmeri nomor
aksesi FM177605, Parathelphusa pantherina
nomor aksesi FM177630 dan Parathelphusa ferruginea nomor aksesi FM177626 yang
diambil dari GeneBank (Lampiran 3). Runutan
panjang DNA sampel yang dapat dianalisis dan dibandingkan dengan runutan DNA referensi adalah 599 nt.
Perbedaan jumlah nukleotida terkecil terjadi antara sampel bernomor 3a dengan sampel bernomor 2a yaitu berbeda 4 nt dengan jarak genetik 0,007. Keduanya merupakan
spesies P. convexa. Perbedaan terbesar terjadi
antara sampel ketam bernomor 6a dengan P. ferruginea yaitu sebesar 88 dengan jarak
genetik sebesar 0,173 (Tabel 2).
Hasil verifikasi DNA barcode
mengelom-pokkan spesies ketam nomor 1a, 1b, 2a, 3a dan 6a menjadi satu kelompok. Spesies dengan nomor-nomor tersebut teridentifikasi secara morfologi sebagai jenis P. convexa dan dapat
diidentifikasi secara DNA barcode sebagai
spesies P. convexa. Spesies dengan nomor 4a
mengelompok tersendiri. Spesies ini teridentifikasi P. bogorensis secara morfologi
dan dapat diidentifikasi secara DNA barcode
sebagai spesies P.bogoriensis. Hasil ini
menunjukkan konkruensi antara hasil analisis morfologi dengan analisis DNA (Gambar 5).
Gambar 2 Karakteristik morfologi P. convexa.
5
Gambar 4 Karakteristik morfologi Geosesarma sp.
Tabel 2 Jumlah perbedaan nukleotida gen CO1 antar spesies ketam (diatas garis diagonal) dan jarak genetik gen CO1 antar spesies ketam berdasarkan model subtitusi K2P (dibawah garis diagonal)
Sampel 1 2 3 4 5 6 7 8 9
1 Parathelphusa convexa1a 27 15 16 70 26 72 76 77 2 Parathelphusa convexa1b 0.047 28 32 65 42 70 70 71 3 Parathelphusa convexa2a 0.026 0.049 4 65 15 71 73 77 4 Parathelphusa convexa3a 0.027 0.056 0.007 67 14 73 77 80 5 Parathelphusa bogoriensis4b 0.133 0.123 0.123 0.127 74 62 67 63 6 Parathelphusa convexa6a 0.046 0.075 0.026 0.024 0.143 80 85 88 7 Nautilothelphusazimmeri1) 0.137 0.133 0.135 0.139 0.114 0.156 23 18
8 Parathelphusa pantherina2) 0.147 0.134 0.140 0.148 0.125 0.168 0.040 27
9 Parathelphusa ferruginea3) 0.148 0.135 0.145 0.154 0.116 0.173 0.031 0.047
Keterangan : 1) GeneBank Accession Number FM177605 2) GeneBank Accession Number FM177630 3) GeneBank Accession Number FM177626
Gambar 5 Rekonstruksi pohon filogeni berdasarkan mtCO1 dengan NJ bootstrap1000X.
Parathelphusa convexa 3a
Parathelphusa convexa 6a
Parathelphusa convexa 2a
Parathelphusa convexa 1a
Parathelphusa convexa 1b
Parathelphusa bogoriensis 4b
Parathelphusa pantherina
Nautilothelphusa zimmeri
Parathelphusa ferruginea
76 100 100
97
93
98
6
PEMBAHASAN
Hasil analisis secara morfologi dan DNA barcode, didapatkan tiga spesies ketam yaitu Geosesarma sp., P. bogorensis dan P. convexa.
Ketiga spesies ini berbeda dengan ketam yang dilaporkan dari Sri Langka (51 spesies ketam dari famili Parathelphusidae) (Bahir et al.
2005). Di Asia Timur yang meliputi China, Taiwan, Ryukyus, dan daratan Jepang melaporkan terdapat 40 genus dan 311 spesies ketam, bahkan di Provinsi Yunan,China memiliki keanekaragaman yang tinggi yaitu terdapat 48 spesies ketam dan di Taiwan terdapat 42 spesies ketam (Shih et al. 2011). Di Sulawesi dilaporkan terdapat 13 spesies ketam air tawar dari genus Parathelphusa
(Chia & Ng 2006).
Ketiga spesies yang ditemukan memiliki relung ekologi yang berbeda-beda. Relung ekologi ini erat kaitannya dengan sumber makanan, predator dan kompetitor. Geosesar-ma sp. dapat ditemukan di celah-celah
bebatuan di sekitar sungai. Spesies ini cenderung memilih tempat yang tidak terlalu tergenang oleh air karena Geosesarma sp.
hanya membutuhkan sedikit air untuk menjaga kelembaban insangnya. Wowor et al. (2010)
menyatakan bahwa Geosesarma sp.
merupakan ketam yang menghuni lubang-lubang tanah yang lembab.
P. bogoriensis dapat ditemukan di dasar
sungai, di bawah bebatuan yang sungainya beraliran deras. Spesies ini merupakan spesies ketam yang menghuni sungai bagian hulu (Wowor et al. 2010). Relung ekologi P. bogoriensis bersesuaian dengan morfologinya. P. bogoriensis memiliki permukaan karapas
rata yang meminimalkan gesarakan antara arus sungai dangan permukaan karapas.
Relung ekologi P. convexa berbeda dengan P. bogoriensis meskipun masih tergolong
dalam genus yang sama. Keduanya bahkan tidak pernah ditemukan dalam satu posisi pengambilan. P. convexa lebih banyak
ditemukan di sungai yang beraliran lambat dan dipinggir sungai. P. convexa dapat ditemukan di daerah-daerah sungai berarus lambat, daerah-daerah genangan air, selokan dan persawahan. Persebaran spesies ini lebih luas, yaitu dari sungai bagian hulu sampai bagian hilir (Wowor et al. 2010).
Ketam tidak dapat ditemukan di 5 titik pengambilan sampel, yaitu di Sungai Cigamea, Sungai Cisadane, Sungai Ciapus I, Sungai ciapus II dan Sungai Cibebeur. Sungai Cigamea terdapat di hulu Gunung Salak yang memiliki kandungan belerang tinggi sehingga
ketam tidak dapat hidup di daerah tersebut. Sungai Cisadane merupakan salah satu sungai besar yang berarus deras. Sungai ini memiliki debit air yang tinggi sehingga ketam sulit ditemukan. Sungai Ciapus I dan Ciapus II merupakan daerah tambang pasir yang menyebabkan daerah aliran sungai menjadi keruh. Ketam tidak dapat hidup di daerah tersebut karena insang ketam akan tertutupi oleh butiran-butiran pasir sehingga ketam akan mati. Sungai Cibeubeur memiliki kontur yang tidak rata. Sungai ini memiliki debit air yang tidak terlalu besar namun sulit untuk melewatinya karena terdapat banyak bebatuan besar yang menutupi badan sungai.
SIMPULAN
Hasil analisis morfologi dan analisis DNA barcode memperoleh tiga spesies ketam yaitu Geosesarma sp., P. bogoriensis dan P. convexa. Geosesarma sp. ditemukan di
celah-celah bebatuan di sekitar sungai. P. boriensis
dapat ditemukan di dasar sungai atau di bawah batu. P. convexa dapat ditemukan di pinggiran
sungai yang ditumbuhi semak-semak.
DAFTAR PUSTAKA
Bahir MM et al. 2005. A Conservation
Assessment Of The Freshwater Crabs Of Sri Lanka. The Raffles Bulletin Of Zoology
12:121-126.
Bott, R. 1968. Potamiden aus SuÈ d-Asien (Crustacea, Decapoda). Senckenbergiana Biologica 49:119-130.
Cai Y, Ng PKL. 2001. The freshwater Decapod Crustaceans of Halmahera, Indonesia. Journal Of Crustacean Biology
21:665-695.
Cumberlidge N et al. 2009. Freshwater crabs
and the biodiversity crisis: Importance, threats, status, and conservation chal-lenges. Biological Conservation
142:1665-1673.
Dixon CJ, Schram FR, Ahyong ST. 2004. A new hypothesis of decapod phylogeny.
Crustaceana 76:935-975.
Klaus S, Schubart CD, Brandis D. 2006. Phylogeny, biogeography and a new taxonomy for the Gecarcinucoidea Rathbun, 1904 (Decapoda: Brachyura).
Organisms, Diversity & Evolution
6:199-217.
Krebs. 1989. Ecological Methodology. New
7
Leibold, M. (2003). Ecological Niches: Link-ing Classical and Contemporary Appro-aches. Chicago: University of Chicago
Press.
Morris CG. 1992. Academic Press Dictionary of Science and Technology. San Diego:
Academic Press.
Morris S. 2001. Neuroendocrine regulation of osmoregulation and the evolution of air-breathing in decapod crustaceans. The Journal of Experimental Biology
204:979-989.
Ng PKL. 1990. Parathelphusa reticulata spec.
Nov.,a new species of freshwater crab from blackwater swamps in Singapore (Crustacea: Decapoda: Brachyura: Gecar-cinucoidea). Zoologische Mededelingen
63:18.
Ng PKL. 1991. Redescription of Parathel-phusa (liothelParathel-phusa) wirzi roux, 1930 and
deffinition of a new genus parathelphusid (Crustacea: Decapoda: Brachyura) from Nias islands, western Sumatra. Verhan-dlungen der Naturfor-schenden Gesell-schaft Basel 101:35-40.
Ng PKL. 2004. Freshwater invertebrates of the Malaysian region: Crustacea: Decapoda, Brachyura. The Raffles Bulletin of Zoology
53:181.
Ng PKL & Tan SH. 1999. Revision of the Southeast Asian potamid crabs of the genus
Malayopotamon Bott, 1968 (Crustacea:
Decapoda: Brachyura). Journal of Natural History 33:207-231.
Shih HT et al. 2011. Diversity and Biogeo-graphy of Freshwater Crabs (Crustacea: Brachyura: Potamidae, Gecarcinucidae) from East Asia. Systematics and Biodiver-sity 9:1-16.
Syafrina RS. 2011. Penggunaan DNA bar-code sebagai alternative identifikasi spesies
udang mantis [skripsi]. Bogor: Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor.
Taylor HH, Taylor EW. 1992. Gills and Lungs: The Exchange of Gases and Ions.
Microscopic Anatomy of Invertebrates
10:203-293.
Yugo K. 1994. Proses geodinamika zona tumbukan. Di dalam: Prosiding Pertemuan
Ilmiah Tahunan; Bandung, 20 Agu 1994. Bandung: Puslitbang Geoteknologi. hlm 170-177.
8
9
Lampiran 1 Ekstraksi dan isolasi DNA dengan DNA Extraction Kit for Fresh Blood (GeneAid) yang dimodifikasi untuk jaringan otot yang diawetkan dalam etanol
A. Penghancuran jaringan
1. Satu ruas tungkai ketam dalam alkohol 70% dipotong dan dimasukkan ke dalam tube 1,5 ml.
2. Ruas tungkai yang telah dipotong (otot tungkai) kemudian dicuci dengan air destilata sebanyak 2 kali ulangan menggunakan mesin sentrifuse.
3. Sampel yang telah dicuci kemudian dihomogenasi menggunakan grinder dan ditambahkan 200 µl buffer STE (NaCl 1M, Tris-HCL 10mM, EDTA 0.1mM, pH 8) ke dalam tube.
4. Sebanyak 50 mg/ml proteinase K 20 µl ditambahkan ke dalam tabung dan vortex selama beberapa detik.
B. Lysis
1. Inkubasi pada suhu 600C selama 30 menit. Setiap 5 menit sekali tabung dibolak-balik secara perlahan.
2. Sebanyak 200 µl buffer GB ditambahkan ke dalam tabung dan vortex selama 5 detik. 3. Inkubasi pada suhu 700C selama 20 menit. Setiap 5 menit sekali tabung dibolak-balik secara
perlahan. Pada saat yang bersamaan, inkubasi buffer elusi pada suhu yang sama untuk step elusi DNA.
4. Sebanyak 200 µl etanol ditambahkan ke dalam tabung dan vortex selama 10 detik.
C. DNA Binding
1. Sampel dipindahkan ke dalam kolom GD pada tube 2 ml.
2. Sentrifugasi 13.000 rpm selama 2 menit.
3. Kolom GD dipindahkan ke dalam tabung koleksi yang baru dan supernatan dibuang.
D. Pencucian
1. Sebanyak 400 µl buffer W1 ditambahkan ke dalam tabung. 2. Sentrifugasi 13.000 rpm selama 30 detik.
3. Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung.
4. Sebanyak 600 µl wash buffer (mengandung etanol) ditambahkan ke dalam kolom GD.
5. Sentrifugasi 13.000 rpm selama 30 detik.
E. DNA elution
1. Supernatan dibuang dan kolom GD diletakkan kembali ke dalam tabung. 2. Sentrifugasi 13.000 rpm selama 3 menit.
3. Kolom GD dipindahkan ke dalam tube 1,5 ml yang baru.
4. Sebanyak 100 µl buffer elusi yang telah diinkubasi ditambahkan ke dalam kolom GD (tepat bagian tengah matriks kolom GD).
5. Diamkan selama 5 menit.
6. Sentrifugasi 13.000 rpm selama 30 detik.
10
Lampiran 2 Daftar pengambilan sampel ketam di sungai-sungai yang berhulu dari Gunung Salak
Tanggal Titik sampling ∑
spesimen Spesies Posisi penangkapan tangkap Alat
26-01-12 S. Cigamea 0 - - Jaring
23-03-12 0 - - Tangan
26-01-12 S. Cianten 10 P. convexa Semak-semak dipinggir sungai Jaring
27-03-12 0 - - Jaring
26-01-12 As. Cianten 0 - - Jaring
27-03-12 6 P. bogoriensis Dasar sungai, dibawah batu Tangan
28-01-12 S. Cisadane 0 - - Jaring
31-03-12 0 - - Tangan
28-01-12 S.Cipinang
Gading 0
- - Tangan
31-03-12 9 P. convexa Semak-semak dipinggir sungai Jaring
23 P. bogoriensis Dasar sungai,dibawah batu Tangan 11 Geosesarma Sp. Disela-sela batu pinggir sungai Tangan
31-01-12 S. Cipangaur 10 P. convexa Semak-semak dipinggir sungai Jaring
21-03-12 7 P. convexa Semak-semak dipinggir sungai Jaring
31-01-12 As. Cipangaur 4 P. convexa Semak-semak dipinggir sungai Jaring
21-03-12 7 P. convexa Semak-semak dipinggir sungai Jaring
07-02-12 S. Ciapus I 0 - - Jaring
10-03-12 0 - - Tangan
07-02-12 S. Ciapus II 0 - - Tangan
10-03-12 0 - - Jaring
09-02-12 S. Cibeubeur 0 - - Tangan
24-03-12 0 - - Jaring
09-02-12 As. Cibeubeur 2 P. convexa Semak-semak dipinggir sungai Jaring
3 P. bogoriensis Dasar sungai, dibawah batu Tangan
24-03-12 5 P. convexa Semak-semak dipinggir sungai Jaring
11
Lampiran 3 Hasil alignment runutan nukleotida gen CO1 enam spesimen ketam
1. P. convexa1a TAT TCG GGC TGA ACT AAG GCC CCT TTG GAA CTT TAA TTG GAA ATG ATC AGA TTT ATA ATG TAA TTG TGA CTG [72]
2. P. convexa1b ... ... TA. ... .T. ... C.- ... ... .GG ... ... ... ... ... ... .A. ... ... ... ... ... ... ... [72]
3. P. convexa2a ... ... T.. A.. ... ... ..- AGC C.. .G. ... ... ... ... ... ... .A. ... ... ... ... ... ... ... [72]
4. P. convexa3a ... ... C.. AA. ... ... ..- AGC C.. .G. ... ... ... ... ... ... .A. ... ... ..A ... ... ... ... [72]
5. P. bogoriensis4b ... ... T.. ... .T. ... A.- AAC CA. ... ..C ... ... ... ... ... .A. ... ... ... ... ... .A. .C. [72]
6. P. convexa6a .T. ... ..A AA. ... ... ..- AGC C.. .G. ... ... ... ... ... ... .A. ... ... ... ... ... ... ... [72]
7. N. zimmeri ... ... T.. ... .T. ... A.- AAC CA. ... .C. ... ... ... ... ... .A. ... ... ... ... .C. .A. ... [72]
8. P. pantherina ... ... T.. ... .T. ... A.- AAC CA. ... .C. ... ... ... ... ... .A. ... ... ... ... .C. .A. ... [72]
9. P. ferruginea ... ... T.. ... .T. ... A.- AAC CA. ... .CC ... ... ... .C. ... .A. ... ... ... ... .C. ... ... [72]
1. CTC ATG CAT TCA TTA TAA TTT TCT TTA TAG TAA TAC CTA TTA TAA TTG GAG GAT TTG GTA ATT GAA TAT TAC CTT TAA TAT TAG [156]
2. ... ... .T. ..G ... ... .C. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [156]
3. ... ... ... ..G ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [156]
4. ... ... ... ..G ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [156]
5. ... ... .T. .TG .A. ... ... ... ... ... .T. ... ... ... ... ... ... ... ... ... ... ... ... ... ..C ... ... ... [156]
6. ... ... ... ..G ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [156]
7. ... ... ... ..G .A. ... ... .T. ... ... .T. ... ... ... ... ... .G. ... .C. .A. ... ... ... ... ... ... ... ... [156]
8. ... ... ... ..G .A. ... ... .T. ... ... .T. ... ... ... ... ... .G. ... .C. .A. ... ... ... ... .C. ... ... ... [156]
9. ... ... ... ..G .A. ... ... .T. ... ... .T. ... ... ... ... ... .G. ... .C. .A. ... ... ... ... ... ... ... ... [156]
1. GAG CTC CTG ATA TAG CCT TCC CAC GTA TAA ATA ATA TAA GAT TTT GAC TTT TAC CTC CTT CAT TAA CAT TAC TTT TAA TAA GAG [240]
2. ... ... ... ... ... ... ... ... ... ... .C. .C. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [240]
3. ... ... ... ... ... ... ... ... ... ... .C. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [240]
4. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [240]
5. ... ... ... ... ... .A. ... .C. .C. ... ... ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... .G. ... [240]
6. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [240]
7. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... .G. .C. ... ..C ... .C. ... .CC ... ... ... [240]
8. ... ... ... ... .G. ... ... ... ... ... .C. .C. ... ... ... ... ... ... ... ... ... ... .C. ... .CC ... ... ... [240]
9. ... ... .C. ... ... ... ... ... ... ... .C. ... ... ... ... ... ... ... .C. ... ..C ... .T. ... .CC ... ... ... [240]
1. GAA TAG TAG AAA GAG GAG TTG GCA CAG GAT GAA CTG TAT ACC CTC CTC TAG CAG CAG CTA TTG CCC ATG CTG GAG CTT CAG TAG [324]
2. ... ... ... ... ... ... ... .T. ... ... ... ... .G. .T. ... ..T ... ... ... ... ... ... ... ... ... ... ... ... [324]
3. ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... .G. ... ... ... ... ... ... ... ... ... ... ... [324]
4. ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... .G. ... ... ... ... ... ... ... ... ... ... ... [324]
5. ... ... ... ... ... ... ... .T. ... ... ... ... ... .T. ... ..T ... .T. .C. .A. ... ... ... ... ... ... ... .T. [324]
6. ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... .G. ... ... ... ... ... ... ... ... ... ... ... [324]
7. .T. ... ... ... ... ... ... .T. ... .T. ... .C. .T. .T. ... ..T ... .T. .T. .A. ... ... .C. .C. ... ... ... .T. [324]
8. .T. ... ... ... ... ... ... .T. ... .C. .G. .A. .T. .T. ... ..T ... .T. .C. .A. ... ... .C. ... .G. ... ... .T. [324]
9. .T. ... ... ... ... ... ... ... ... .T. .G. .C. .T. .T. ... ..T ... .T. .C. .A. ... ... .C. .C. ... ... ... .T. [324]
12
1. ATA TAG CAA TTT TCT CAT TAC ATT TAG CCG GTG TAT CCT CTA TTC TCG GTG CCG TAA ATT TTA TAA CTA CTG TTA TTA ACA TAC [408]
2. ... ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... ... .T. ... ... ... ... ... .C. ... ... ... ... [408]
3. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [408]
4. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [408]
5. ... ... ... ... .T. .TC ... ... ... .G. .A. .T. ... .A. ..T .A. ... ... ... ... ... ... .C. .C. ... ... .T. ... [408]
6. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [408]
7. ... ... ... ... ... ... ... ... ... .A. ..A .T. ... .A. .C. .T. ... .T. .T. ... ... ... ... ... .A. .C. .T. ... [408]
8. ... ... ... ... ... ... ... ... ... .A. ... .T. ... .A. .C. .T. ... .T. .T. ... ... ... ... ... .A. .C. .T. ... [408]
9. ... ... ... ... ... ... ... ... ... .A. ... .T. .T. .A. .C. .T. ... .T. .T. ... ... ... ... ... .A. .C. .T. ... [408]
1. GCT CTT ATG GTA TAA CAA TAG ATC AAA TAC CAT TAT TTG TCT GAG CAA TTT TCA TTA CAG TAA TCT TAC TTT TAC TTT CCT TAC [492]
2. ... ... ... ... ... ... ... .C. ... ... ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... ... ... [492]
3. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [492]
4. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [492]
5. .T. ... ... ... ... ... ... .C. ... ... ... ... .C. .T. ... ... ... .T. ... .T. ... .T. ..T .A. ... ... .A. ... [492]
6. ... ... ... ... ... ... ... ... ... ... .G. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... [492]
7. ... ... ... ... ... ... ... .C. ... ... ... ... .C. ... ... ... ... .T. ... ... ... .TC ..T .A. ... ... .A. ... [492]
8. ... ... ... .A. ... ... ... .C. ... ... ... ... .C. .T. ... ... ... .T. ... ... ... ..C .TT .A. ... ... .A. ... [492]
9. ... ... ... ... ... ... ... .C. ... ... ... ... .C. .T. ... ... ... .T. ... ... .G. .TC ..T .A. ... ... .A. .G. [492]
1. CAG TGC TCG CTG GTG CTA TTA CTA TAC TTT TAA CAG ACC GAA ATT TAA ATA CAT CAT TCT TTG ACC CTG CTG GTG GGG GAG ACC [576]
2. ... .A. ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... .A. ... .T. [576]
3. ... .A. ... ... ... ... ... .C. ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... [576]
4. ... .A. ... ... ... ... ... .C. ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... ... ... ... ... ... [576]
5. ... .TT .A. .C. ... ... ... .C. ..T .A. ... ... .T. .T. ... ... ... ... .T. .T. ... .T. ... ... .A. .A. ... .T. [576]
6. ... .A. ... ... .G. ... ... .C. ... ... ... ... ... ... ... ... ... ... ... .T. ... ... ... ... .G. ... ... ... [576]
7. .T. .C. .T. ... .C. ... ... ... ..T .A. ... ... ... .T. ... ... .C. .C. .T. ... ... .T. ... ... .A. ... ... .T. [576]
8. .T. .C. .T. ... ... ... ... ... ..T .GC ... ... ... .T. ... ... .C. .C. .C. ... ... .T. ... ... .A. ... .G. .T. [576]
9. .C. .C. .T. ... ... ... ... ... ..T .A. ... ... ... .T. ... ... .C. .C. .T. ... ... .T. ... ... .A. .A. ... .T. [576]
1. CTA TTC TCT ACC AAC ATT TAT TT [599]
2. ... .C. ... .T. ... ... ... .. [599]
3. ... ... ... ... ... ... ... .. [599]
4. ... ... ... ... ... ... ... .. [599]
5. .G. ... .T. .T. ... ... ... .. [599]
6. ... ... .TC TAA .GA ... ... .. [599]
7. ... .C. .T. ... ... ... ... .. [599]
8. ... .C. .T. ... ... ... ... .. [599]
9. ... .C. .T. ... ... ... ... .. [599]
13
Lampiran 4 Spesiasi
A. Pengertian Spesiasi
Spesiasi adalah pembentukan suatu spesies baru. B. Syarat terjadinya spesiasi
1. Adanya perubahan lingkungan 2. Adanya relung ekologi yang kosong 3. Adanya variasi dalam suatu spesies C. Macam-macam spesiasi
1. Spesiasi Sympatrik
Spesiasi sympatrik yaitu suatu spesies terpisah menjadi dua spesies yang berbeda tanpa terjadinya isolasi geografi. Spesiasi ini didasarkan pada dua postulat :
a. Terbentuknya populasi baru dari suatu spesies dalam niche ekologi berbeda-beda di dalam domain normal dari persilangan spesies orang tuanya,
b.Isolasi reproduksi populasi asal dari populasi baru dibandingkan dengan individu-individu dari populasi orang tua.
Dari kedua postulat tersebut aliran gen antara populasi turunan dengan populasi orang tuanya dihambat oleh faktor intrinsik dan bukan oleh faktor ekstrinsik.
2. Spesiasi Allopatrik
Spesiasi allopatrik terjadi jika barrier terbentuk diantara variasi geografik yang bersifat kontinue.
3. Spesiasi Parapatrik