IDENTIFIKASI KERAGAMAN GEN HORMON PERTUMBUHAN
DAN RESEPTORNYA SERTA HUBUNGANNYA DENGAN
PRODUKSI SUSU KUMULATIF PARSIAL PADA SAPI
FRIESIAN HOLSTEIN DI SENTRA PRODUKSI
JAWA BARAT
RESTU MISRIANTI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS
DAN SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis yang berjudul Identifikasi Keragaman Gen Hormon Pertumbuhan dan Reseptornya serta Hubungannya dengan Produksi Susu Kumulatif Parsial pada Sapi Friesian Holstein di Sentra Produksi Jawa Barat adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam daftar pustaka di bagian akhir tesis ini.
Bogor, Mei 2011
Restu Misrianti
RESTU MISRIANTI. Identification of polymorphism of the Growth Hormone Gene and its Receptor and their Association with Partial Cumulative Milk Yield in Holstein Friesian Cattle in West Java Production Center. Under the direction of CECE SUMANTRI, ANNEKE ANGGRAENI.
Growth hormone gene has a critical role in the regulation of lactation, mammary gland development and growth process through its interaction with its specific receptor. Growth Hormone (GH) and Growth Hormone Receptor (GHR) genes have been suggested as candidate genes for traits related to milk production. The purpose of this study was to identify genetic polymorphism of the Growth Hormone (GH) and the Growth Hormone Receptor (GHR) genes, and the association of their genetic variants with milk yield in Holstein Friesian (HF) cattle. A total of 370 HF cattle from six populations of Institute for breeding of dairy cattle Cikole station (88 samples), Pasir Kemis (96 samples), Cilumber (98 samples), Lembang Artificial Insemination Center (LAIC) (17 samples), Livestock Embryo Transfer Center (40 samples) and Singosari Artificial Insemination Center (SAIC) (32 samples) were genotyped by the Polymerase Chain Reaction and Restriction Fragment Length Polymorphism (PCR-RFLP) methods. Genotyping the GH gene with Alu1 restriction enzyme to all DNA samples produced two variant genotypes, i.e LL (89 %) and LV (11%) genotypes. The frequency of L allele was 94 % and V allele was 6%. Genotyping of GHR gene with Alu1 restriction enzymes produced three genotypes and two alleles. The frequencies of AA, AG, and GG genotypes were 58 %, 34%, and 8% respectively. The frequency of A allele was 75% and G allel was 25%. There was no association between GH and GHR polymorphism with milk production.
Reseptornya serta Hubungannya dengan Produksi Susu Kumulatif Parsial pada Sapi Friesian Holstein di Sentra Produksi Jawa Barat. Dibimbing oleh CECE SUMANTRI dan ANNEKE ANGGRAENI.
Perbaikan genetik sapi perah dapat dilakukan melalui seleksi keunggulan pada gen yang berasosiasi kuat dengan sifat produksi susu. Gen hormon pertumbuhan diketahui berperan penting dalam regulator pertumbuhan ternak dan perkembangan kelenjar mammari. Penelitian ini bertujuan untuk mengidentifikasi keragaman genetik gen Growth Hormone (GH) dan Growth Hormone Receptor (GHR) sapi Friesian Holstein (FH) serta menganalisis hubungan antara keragaman genotipe kedua gen dengan produksi susu kumulatif parsial.
Isolasi DNA dilakukan dari darah dan sperma. Total sampel darah dan sperma yang digunakan yaitu sebanyak 363 sampel darah dan 17 sampel sperma. Sampel darah berasal dari populasi Balai Besar Inseminasi Buatan (BBIB) Singosari 32 sampel, Balai Embryo Transfer (BET) Cipelang 34 sampel, Cikole 88 sampel, Cilumber 98 sampel dan Pasir Kemis 95 sampel. Sampel sperma berasal dari BIB Lembang. Isolasi DNA dari sampel darah dan sperma menggunakan metode ekstraksi phenol-chloroform (Sambrook et al. 1989) yang telah dimodifikasi untuk sampel darah dan sperma yang disimpan dalam alkohol. Data produksi susu merupakan data sekunder yang berasal dari sapi FH pada periode laktasi pertama tahun 2008-2010. Data ini diambil dari populasi sapi BPPT SP-Cikole yang merupakan peternakan dengan sistem pemeliharaan intensif. Catatan produksi susu yang digunakan merupakan catatan produksi harian yang diukur setiap minggunya. Data produksi susu dicatat dua kali sehari, yaitu pada pemerahan pagi dan pemerahan sore. Produksi susu total sehari adalah penjumlahan dari produksi susu pada pemerahan pagi dan sore. Data produksi 15 hari diestimasi berdasarkan data harian yang diukur setiap minggu. Data kumulatif parsial dihitung berdasarkan data produksi setiap interval 15 hari, mulai dari 15 hari sampai 180 hari setelah beranak.
Penentuan genotipe masing-masing individu dilakukan dengan pendekatan Restriction Fragment Length Polymorphism (RFLP). Keragaman gen dihitung dengan analisis frekuensi alel, frekuensi genotipe dan nilai heterosigozitas. Keseimbangan genotipe gen GH dan GHR dalam populasi diuji dengan uji χ2 (chi square). Hubungan antara varian genotipe GH dan GHR dengan produksi susu kumulatif parsial dihitung berdasarkan metode General Linear Model (GLM).
sepanjang 131 dan 167 pb. Genotipe AG merupakan gabungan dari keduanya yaitu fragmen sepanjang 50, 81, 131 dan 167 pb.
Frekuensi genotipe LL pada ruas gen GH|Alu1 tinggi di semua lokasi pengamatan, dengan kisaran nilai 0.84-0.95, sebaliknya frekuensi genotipe LV rendah di semua populasi dengan kisaran nilai 0.05-0.16. Genotipe VV tidak ditemukan pada semua sampel yang diamati, hal ini disebabkan tidak adanya genotipe VV pada pejantan IB yang digunakan. Frekuensi genotipe AA pada ruas gen GHR|Alu1 tinggi pada semua populasi dengan kisaran nilai 0.49-0.76, sedangkan frekuensi genotipe AG berkisar antara 0.12-0.43. Sebaliknya frekuensi genotipe GG rendah di semua lokasi yang diamati yaitu berkisar antara 0.05-0.13. Tingginya frekuensi genotipe LL dan AA pada sampel yang diamati, diduga disebabkan oleh tingginya frekuensi genotipe LL dan AA pada pejantan IB yang digunakan.
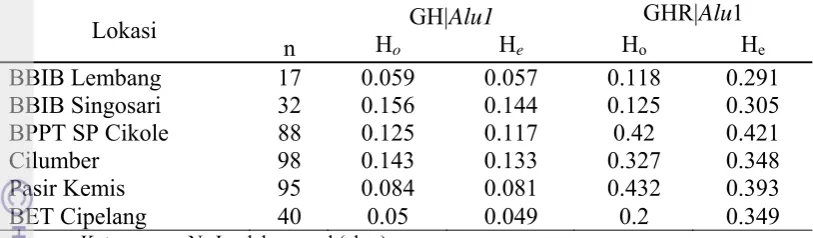
Nilai heterozigositas lokus GH|Alu1 berkisar antara 0.050 – 0.156. Nilai heterozigositas tertinggi ditemukan pada sapi pejantan IB di BBIB Singosari, sedangkan pada lokasi BET Cipelang diperoleh nilai terendah yaitu 0.050. Nilai heterozigositas lokus GHR|Alu1 berkisar antara 0.125-1.432. Nilai tertinggi ditemukan pada sampel yang berasal dari Pasir Kemis, sedangkan pada lokasi BIB Lembang ditemukan nilai heterozigositas terendah yaitu 0.118. Rendahnya heterozigositas pada sampel yang berasal dari BIB Lembang, diduga dikarenakan sapi-sapi yang ada di stasiun bibit ini merupakan sapi hasil seleksi yang intensif. Tingginya nilai heterozigositas pada sampel yang berasal dari peternakan rakyat menunjukkan belum dilakukannya seleksi pada sampel yang berasal dari lokasi tersebut, sehingga masih ada peluang untuk melakukan seleksi.
Hasil uji chi square terhadap genotipe lokus GHR|Alu1 menunjukkan bahwa frekuensi genotipe gen GHR dalam keadaan seimbang (keseimbangan Hardy Weinberg) pada populasi BPPT Cikole, Cilumber dan Pasir Kemis. Frekuensi genotipe pada populasi BET Cipelang, BBIB Lembang dan BBIB Singosari berdasarkan hasil pengujian chi square menyimpang dari keseimbangan Hardy-Weinberg. Keseimbangan Hardy Weinberg untuk lokus GH|Alu1 tidak dianalisis karena jumlah genotipe yang ditemukan tidak memenuhi untuk dianalisis (hanya terdapat dua genotipe yaitu LL dan LV, db=0) pada semua populasi. Pemeriksaan pengaruh genotipe GH|Alu1 dan GHR|Alu1terhadap produksi susu kumulatif parsial pada lokasi BPPT Cikole menunjukkan bahwa secara umum keragaman gen GH dan GHR tidak berpengaruh nyata terhadap tingkat produksi susu sapi FH (P>0.05).
©Hak Cipta Milik IPB, Tahun 2011
Hak Cipta dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan Pengutipan tersebut tidak merugikan kepentingan yang wajar IPB
IDENTIFIKASI KERAGAMAN GEN HORMON PERTUMBUHAN
DAN RESEPTORNYA SERTA HUBUNGANNYA DENGAN
PRODUKSI SUSU KUMULATIF PARSIAL PADA SAPI
FRIESIAN HOLSTEIN DI SENTRA PRODUKSI
JAWA BARAT
RESTU MISRIANTI
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Mayor Ilmu Produksi dan Teknologi Peternakan
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Judul Tesis : Identifikasi Keragaman Gen Hormon Pertumbuhan dan Reseptornya serta Hubungannya dengan Produksi Susu Kumulatif Parsial pada Sapi Friesian Holstein di Sentra Produksi Jawa Barat
Nama : Restu Misrianti
NIM : D151090131
Program Studi/Mayor : Ilmu Produksi dan Teknologi Peternakan
Disetujui
Komisi Pembimbing
Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc. Ir. Anneke Anggraeni, M.Si, Ph.D
Ketua Anggota
Diketahui
Koordinator Mayor Dekan Sekolah Pascasarjana IPB Ilmu Produksi dan Teknologi Peternakan
Dr. Ir. Rarah R. A. Maheswari, DEA Dr. Ir. Dahrul Syah, M.Sc.Agr
sehingga penulis mampu menyelesaikan penelitian dengan judul “Identifikasi
Keragaman Gen Hormon Pertumbuhan dan Reseptornya serta Hubungannya
dengan Produksi Susu Kumulatif Parsial pada Sapi Friesian Holstein di Sentra
Produksi Jawa Barat”. Tesis ini dibuat sebagai salah satu syarat bagi mahasiswa
pascasarjana program S2 untuk meraih gelar magister pada program studi Ilmu
Produksi dan Teknologi Peternakan, Institut Pertanian Bogor.
Penulis menyampaikan penghargaan dan ucapan terimakasih kepada Prof.
Dr. Ir. Cece Sumantri, M.Agr.Sc selaku ketua komisi pembimbing, Ir. Anneke
Anggraeni, M.Si,Ph.D selaku anggota komisi pembimbing atas segala waktu,
arahan, bimbingan, saran serta ketulusan dan kesabaran sehingga penulis bisa
menyelesaikan tugas akhir ini. Kepada Prof. Dr. Ir. Muladno, MSA penulis
menyampaikan terimakasih atas masukan dan kesediannya sebagai penguji luar
komisi pada ujian tesis. Ucapan terimakasih juga penulis sampaikan kepada Prof.
Dr. Ir. Ronny Rachman Noor M.Rur.Sc yang telah memberikan kesempatan
kepada penulis untuk melakukan penelitian di Laboratorium Genetika Molekuler
Ternak, Bagian Pemuliaan dan Genetika Ternak.
Ucapan terimakasih juga penulis sampaikan kepada pihak yang telah
memberikan beasiswa, sehingga sangat membantu penulis dalam menyelesaikan
studi pada program magister Sekolah pascasarjana Institut Pertanian Bogor.
Kepada ayahanda dan ibunda tercinta penulis sampaikan terimakasih atas
doa, kasih sayang dan nasihat yang diberikan. Kepada kakak dan adik tersayang,
penulis sampaikan terimakasih atas doa dan dukungannya. Kepada rekan-rekan
mahasiswa pascasarjana program studi ITP khususnya angkatan 2009 penulis
menyampaikan terimakasih atas segala bantuan dan dukungannya. Kepada
teman-teman Laboratorium Genetika Molekuler Ternak dan teman-teman-teman-teman ABGSCi
penulis mengucapkan terimakasih atas kerjasamanya selama penelitian
berlangsung. Semoga Allah SWT memberikan balasan yang berlipat ganda dan
semoga penelitian ini bermanfaat bagi upaya pembangunan peternakan Indonesia.
Bogor Mei 2011
Penulis dilahirkan di Siberakun (Riau) pada tanggal 23 September 1987,
sebagai anak kedua dari tiga bersaudara dari Ayahanda Syafriadi dan Ibunda
Missiati.
Pendidikan dasar diselesaikan pada tahun 2001 di SD Negeri 006
Siberakun. Pendidikan lanjutan tingkat pertama diselesaikan pada tahun 2003 di
SLTP Negeri 1 Benai. Pendidikan lanjutan menengah tingkat atas diselesaikan
pada tahun 2005 di SMA Negeri 1 Benai. Pada tahun 2005 penulis diterima di
Institut Pertanian Bogor melalui jalur Seleksi Penerimaan Mahasiswa Baru
(SPMB). Pendidikan sarjana ditempuh di departemen Ilmu Produksi dan
Teknologi Peternakan Fakultas Peternakan Institut Pertanian Bogor mulai tahun
2005 dan lulus pada tahun 2009. Pada tahun yang sama penulis melanjutkan studi
pada program studi peternakan dengan mayor Ilmu Produksi dan Teknologi
Peternakan bidang Pemuliaan dan Genetika Ternak.
Selama menjadi mahasiswa pascasarjana IPB penulis aktif mengikuti
berbagai kegiatan seminar dan kompetisi ilmiah. Pada tahun 2009 penulis
mempresentasikan hasil penelitian yang berjudul “Polymorphism Identification of
f Pituitary-Specific Positive Transcription Factor1 (Pit1) Gene in Indonesian
Local Buffalo (Bubalus bubalis) and Holstein-Friesian Cows” pada International
Seminar on Animal Industri (ISAI), dengan tulisan yang sama penulis berhasil
meraih award sebagai second winner pada scientific award yang diadakan Media
Peternakan. Penulis juga menjadi asisten mata kuliah Pemuliaan Ternak dan
Genetika Ternak pada Departemen Ilmu Produksi dan Teknologi Peternakan,
DAFTAR GAMBAR ... iv
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan Penelitian ... 2
Manfaat Penelitian ... 2
Kerangka Pemikiran Penelitian ... 2
TINJAUAN PUSTAKA ... 4
Sapi Perah Friesian Holstein... 4
Seleksi dengan Menggunakan Penanda Molekuler ... 5
Keragaman Genetik ... 6
Growth Hormone (Gen Hormon Pertumbuhan) ... 7
Growth Hormone Receptor (Gen Reseptor Hormon Pertumbuhan) ... 10
Produksi Susu Sapi FH ... 11
Hubungan Varian Genetik Gen Pengontrol Produksi Susu dengan Sifat Produksi Susu ... 14
Penentuan Genotipe dengan Pendekatan PCR-RFLP .... 20
Analisis Data ... 20
Frekuensi Alel dan Genotipe ... 20
Proporsi Hardy-Weinberg ... 21
Heterozigositas ... 21
Analisis Hubungan Genotipe Gen GH dan GHR terhadap Produksi Susu ... 22
HASIL DAN PEMBAHASAN ... 23
Amplifikasi Gen GH (Growth Hormone) ... 23
Keragaman Genotipe gen GH Sapi FH dengan Pendekatan PCR RFLP ... 24
Amplifikasi Ruas Gen GHR ... 27
Keragaman Genotipe Gen GHR Sapi FH dengan Pendekatan PCR-RFLP ... 27
Proporsi Hardy-Weinberg ... 32
Produksi Susu Harian Sapi FH di BPPT Cikole` ... 33
Produksi Susu Kumulatif Parsial Sapi FH di BPPT Cikole ... 35
Hubungan Keragaman Genotipe GH dan GHR dengan Produksi Susu Kumulatif Parsial ... 37
KESIMPULAN DAN SARAN ... 41
Kesimpulan ... 41
Saran ... 41
1 Sampel darah sapi Friesian Holstein ... 17
2 Runutan primer yang digunakan ... 18
3 Frekuensi genotipe dan alel pada ruas gen GH|Alu1 ... 26
4 Frekuensi genotipe dan alel pada ruas gen GHR|Alu1 ... 29
5 Nilai heterozigositas pengamatan (Ho) dan heterozigositas harapan (He) fragmen gen GH|Alu1 sapi FH betina dan pejantan ... 31
6 Hasil pengujian Herdy Weinberg berdasarkan test chi square Lokus GHR|Alu1 pada sapi betina FH dan pejantan IB ... 32
7 Rataan produksi susu harian pada sapi FH di BPPT Cikole ... 34
8 Produksi susu kumulatif parsial sapi FH di BPPT Cikole ... 37
Nomor Halaman
1. Kerangka pemikiran ... 3
2. Diagram pengaturan sekresi hormon pertumbuhan dan kerjanya pada ternak domestik ... 8
3. Rekonstruksi struktur GH berdasarkan sekuen gen GH di GenBank ... 9
4. Rekonstruksi struktur GHR berdasarkan sekuen gen GHR di GenBank ... 11
5. Produksi susu selama masa laktasi dengan tingkat persistensi yang berbeda ... 12
6. Visualisasi hasil amplifikasi ruas gen GH pada gel agarose 2% ... 23
7. Runutan nukleotida ruas Gen GH ... 24
8. Perbedaan sekuen alel L dan V gen GH|Alu1 ... 24
9. Visualisasi PCR-RFLP ruas gen GH|Alu1 pada gel agarose 2% .... 25
10.Visualisasi hasil amplifikasi ruas gen GHR pada gel agarose 2% .. 27
11.Runutan nukleotida ruas Gen GHR ... 28
12.Perbedaan sekuen alel A dan G gen GHR|Alu1 ... 28
13.Visualisasi PCR-RFLP ruas gen GHR|Alu1 pada gel agarose 2% .. 28
14.Rataan produksi harian laktasi periode 1 ... 34
15.Produksi susu kumulatif parsial sapi FH laktasi periode 1 ... 36
16.Produksi susu kumulatif parsial sapi perah berdasarkan genotipe GH|Alu1 ... 38
Nomor Halaman
1. Sekuen gen GH|Alu1 yang di akses di gen bank No J00008 ... 49
2. Sekuen gen GHR|Alu1 yang di akses di gen bank No EF207442 ... 51
3. Hasil analisis ragam pengaruh GH|Alu1 terhadap produksi
susu kumulatif parsial sapi FH di BPPT Cikole ... 56
4. Hasil analisis ragam pengaruh GHR|Alu1 terhadap produksi
Latar Belakang
Populasi sapi perah di Indonesia diperkirakan sebanyak 487.000 ekor yang
sebagian besar adalah bangsa sapi Friesian Holstein (FH) (Direktorat Jenderal
Peternakan 2009) dan tersebar di sentra-sentra produksi sapi perah di pulau Jawa
seperti di Provinsi Jawa Barat, Jawa Tengah dan Jawa Timur. Produksi susu di
Provinsi Jawa Barat, Jawa Timur dan Jawa Tengah pada tahun 2009 adalah
225.215, 312.270 dan 89.748 ton.
Tingkat produksi susu dari peternakan sapi perah di Indonesia masih lebih
rendah dibandingkan laju kebutuhannya, sehingga masih terdapat kesenjangan
antara produksi dan kebutuhan susu. Beberapa hal yang menyebabkan
kesenjangan antara produksi susu dan pemenuhan konsumsi susu tersebut adalah
rendahnya populasi dan potensi genetik sapi perah, serta kondisi manajemen
pemeliharaan yang kurang baik.
Pemilihan pejantan dan betina unggul melalui seleksi bertujuan untuk
mendapatkan sumber materi genetik bagi generasi berikutnya. Sifat produksi susu
biasanya digunakan sebagai dasar seleksi pada sapi perah. Produksi susu
merupakan sifat kuantitatif yang dikendalikan banyak gen dan ekspresinya
merupakan akumulasi dari pengaruh genetik, lingkungan dan interaksi keduanya.
Seleksi dapat diakselerasi melalui pemanfaatan penciri genetik dengan
menggunakan teknik molekuler. Seleksi berdasarkan marker genetik untuk penciri
sifat tertentu akan menjadikan seleksi berlangsung lebih awal, sehingga
pemeliharaan ternak-ternak yang tidak produktif dapat dihindari. Selain itu,
penerapan marker genetik dalam program pemuliaan ternak dapat mempercepat
peningkatan kualitas genetik ternak.
Gen hormon pertumbuhan atau Growth Hormone (GH), gen reseptor
hormon pertumbuhan atau Growth Hormone Receptor (GHR) dan
hormon-hormon lainnya seperti Insulin Like Growth Factor 1 (IGF1) banyak digunakan
dalam studi gen kandidat terhadap sifat-sifat produksi ternak, selanjutnya
digunakan sebagai marker genetik dalam seleksi. Hal ini dikarenakan
hormon-hormon tersebut merupakan hormon-hormon regulator pertumbuhan dan perkembangan
Penelitian mengenai keragaman gen GH dan hubungannya dengan
produksi susu telah dilakukan pada sapi perah di luar negeri, misalnya pada sapi
Friesian Holstein (FH) Hungaria (Balogh et al. 2009), sapi Holstein Iran
(Mohammadabadi et al. 2010) dan Sapi FH Polandia (Olenski et al. 2010).
Berdasarkan hasil beberapa studi tersebut diketahui bahwa GH bersama dengan
GHR berperan penting dalam mengatur pertumbuhan kelenjar mammari dan
produksi susu, metabolisme, laktasi dan komposisi tubuh.
Tujuan
1. Mengidentifikasi keragaman genetik gen Growth Hormone (GH) dan gen
Growth Hormone Receptor (GHR)
2. Menganalisis hubungan antara keragaman genotipe gen Growth Hormone
(GH) dan Growth Hormone Receptor (GHR) dengan produksi susu
kumulatif parsial pada sapi FH.
Manfaat
1. Diperoleh informasi tentang keragaman genetik gen GH dan GHR pada
sapi perah Friesian Holstein di sentra produksi Jawa Barat
2. Didapatkan informasi genetik sapi FH dari pejantan di stasiun bibit
sehingga diketahui sumbangan pejantan IB terhadap frekuensi genotipe
dan alel dari gen GH dan GHR pada sapi FH betina
3. Informasi asosiasi gen GH dan GHR dengan produksi susu kumulatif
parsial.
Kerangka Pemikiran
Produksi susu dipengaruhi oleh faktor genetik, fisiologi dan lingkungan.
Besarnya pengaruh genetik terhadap sifat produksi susu ditentukan melalui
analisis keragaman genetik gen pengontrol sifat produksi susu (gen GH dan
GHR). Pemeriksaan keragaman genetik dilakukan pada sapi FH betina yang
berasal dari dua manajemen pemeliharaan yang berbeda, yaitu sapi FH yang
dipelihara secara intensif di stasiun bibit pemerintah (BET Cipelang, BPPT
Cikole) dan sapi yang berasal dari peternakan semi intensif yaitu di peternakan
peternakan binaan KPSBU Lembang. Sumbangan pejantan IB terhadap
keragaman genetik gen GH dan GHR pada betina turunannya, ditentukan melalui
analisis sampel yang berasal dari BBIB Lembang dan BBIB Singosari.
Besarnya pengaruh keragaman genetik terhadap sifat fenotipik (produksi
susu) dianalisis melalui hubungan antara keragaman gen GH dan GHR dengan
produksi susu kumulatif parsial. Pemeriksaan hubungan ini dilakukan pada
peternakan dengan sistem manajemen pemeliharaan intensif (BPPT Cikole),
dengan tujuan untuk minimalisasi pengaruh lingkungan terhadap ekspresi gen.
Kerangka pemikiran ini diringkas dalam Gambar 1.
Gambar 1 Kerangka pemikiran penelitian.
Sapi Perah di BPPT Cikole, KPSBU Pasir Kemis dan KPSBU Cilumber, BIB Singosari, BIB Lembang dan BET Cipelang
Fenotipe Genotipe
Sifat Produksi Susu
Produksi susu kumulatif parsial
(BPPT Cikole) Gen GH dan GHR
PCR-RFLP
Polimorfisme
Sapi perah termasuk kedalam famili Bovidae, sub famili Bovinae, genus
Bos. Sapi perah yang dikembangkan di berbagai belahan dunia adalah jenis Bos
taurus (sapi Eropa) yang berasal dari daerah sub tropis dan Bos indicus (sapi
berponok di Asia) yang berasal dari daerah tropis, serta hasil persilangan
keturunan Bos taurus dan Bos indicus. Sapi yang berasal dari Bos taurus yang
banyak dikembangkan ada lima bangsa yaitu Holstein, Brown Swiss, Ayshire,
Guernsey dan Jersey. Bangsa yang umum dikembangkan di Indonesia adalah
bangsa Friesian Hosltein (FH). Sapi FH berasal dari propinsi Friesland negeri
Belanda. Bangsa sapi ini adalah bangsa sapi perah yang tertua, terkenal dan
tersebar hampir di seluruh dunia.
Bangsa sapi FH murni memiliki warna bulu Black and White (hitam dan
putih) atau merah dan putih (Red Holstein) dengan batas-batas warna yang jelas,
seperti pada dahi umumnya terdapat warna putih berbentuk segitiga dan bulu
kipas ekor, bagian perut serta kaki dari teracak sampai lutut (knee atau hock)
berwarna putih. Selain itu, sapi FH memiliki tanduk yang pendek dan mengarah
kedepan. Sifat-sifatnya adalah jinak, tidak tahan panas, tetapi sapi ini mudah
menyesuaikan diri dengan keadaan lingkungan dan lambat dewasa. Menurut
Blakely dan Bade (1991), Karakteristik sapi FH adalah memiliki berat induk 675
kg, warna bulu hitam dan putih, temperamen tenang, kemampuan merumputnya
sedang, masak kelamin lambat, kadar lemak susu 3.5-3.7 %, dengan warna lemak
kuning membentuk butiran-butiran (glubola) sehingga aman untuk konsumsi susu
segar, bahan kering tanpa lemak 8.5 %, rata-rata produksi susu per tahun
5750-6250 kg dan berat lahir anak 42 kg.
Populasi sapi perah di Indonesia pada tahun 2009 diperkirakan sebanyak
487.000 ekor. Populasi ini 29.423 ekor lebih tinggi dibandingkan populasi pada
tahun 2008 (457.577 ekor). Produksi susu pada tahun 2009 adalah 646.953 ton.
Seleksi Menggunakan Penanda Molekuler
Metode seleksi sederhana berdasarkan informasi fenotipe telah banyak
dilakukan untuk perbaikan produktivitas ternak, namun terdapat beberapa
untuk diukur dan diamati (Vischer et al. 2000). Salah satu metode untuk
mengatasi kelemahan tersebut adalah dengan melakukan seleksi menggunakan
penanda molekuler.
Penanda molekuler merupakan pemanfaatan dari keragaman pada tingkat
DNA, yaitu molekul yang terdapat dalam inti sel. Molekul DNA terdiri atas dua
untai nukleotida yang saling berkomplemen. Struktur tersebut memungkinkan
terjadinya mekanisme pewarisan sifat (Alberts 2002). Hal tersebut merupakan
salah satu faktor utama yang mendasari terjadinya proses seleksi (Vignal et al.
2002). Tipe dasar dalam perubahan DNA berupa substitusi, delesi, insersi dan
inversi (Nei dan Kumar 2000).
Single nucleotide polymorphisms (SNP) merupakan penanda yang
memiliki perbedaan satu nukleotida dalam sekuen DNA dan diperkirakan bahwa
satu SNP terjadi setiap 250–1000 pb. Perbedaan tersebut disebabkan oleh
terjadinya proses substitusi, sehingga biasanya memiliki dua kemungkinan pada
posisi yang sama dalam sekuen DNA (Vignal et al. 2002). Frekuensi mutasi dan
stabilitas yang tinggi menyebabkan SNP sering digunakan sebagai penanda
molekuler dalam penelitian tentang genetika populasi dan pemetaan gen untuk
penyakit kompleks (Ye et al. 2001). Metode yang umum digunakan dalam
analisis adanya SNP antara lain PCR-RFLP dan PCR-SSCP.
PCR merupakan suatu teknik untuk menggandakan jumlah molekul DNA
pada ruas-ruas tertentu dan monomer-monomer nukleotida secara in vitro (Viljoen
et al. 2005). Proses ini berjalan dengan bantuan primer dan enzim polymerase.
Primer merupakan oligonukleotida spesifik yang menempel pada bagian sampel
DNA yang akan diperbanyak (Williams 2005). Enzim polymerase merupakan
enzim yang dapat mencetak urutan DNA baru. Hasil dari proses PCR dapat
divisualisasikan dengan elektroforesis (Williams 2005).
PCR-RFLP merupakan salah satu metode analisis lanjutan dari produk
PCR. Metode PCR memanfaatkan runutan nukleotida yang bisa dikenali oleh
enzim restriksi yang disebut sebagai situs restriksi. Jika situs restriksi mengalami
mutasi (meskipun pada satu basa) maka enzim restriksi tidak mampu
mengenalinya. Ada tidaknya situs restriksi dapat digunakan untuk penentuan ada
Analisis RFLP biasa digunakan untuk deteksi adanya keragaman pada gen
yang berhubungan dengan sifat ekonomis, seperti produksi dan kualitas susu
(Sumantri et al. 2007). PCR-SSCP juga dapat digunakan untuk analisis
keragaman DNA. PCR-SSCP merupakan metode analisis lebih lanjut yang
memanfaatkan produk PCR. Metode PCR-SSCP merupakan metode yang handal
dalam mendeteksi adanya mutasi secara cepat (Hayashi 1991). Asumsi yang
mendasari metode analisis SSCP adalah perubahan yang terjadi pada nukleotida
meskipun terjadi hanya pada satu basa, akan mempengaruhi conformation
(bentuk) dari fragmen DNA pada kondisi untai tunggal. Perbedaan konformasi
molekul akan menyebabkan perbedaan migrasinya dalam gel poliakrilamid pada
saat elektroforesis (Montaldo et al. 1998).
Metode PCR-RFLP bisa mendeteksi mutasi jika situs restriksi mengalami
perubahan susunan basa. Apabila mutasi terjadi diluar situs restriksi, maka mutasi
tersebut tidak dapat dideteksi. Metode PCR-SSCP dapat mendeteksi perubahan
pada satu basa, tetapi tidak dapat diketahui basa yang berubah (Hayashi 1991).
DNA sekuensing dapat digunakan untuk penyelesaian masalah kelemahan
tersebut. Metode yang biasa digunakan dalam sekuensing DNA adalah metode
Sanger. Metode ini menggunakan pendekatan sintesis molekul DNA baru dan
pemberhentian sintesis tersebut pada basa tertentu. Metode sekuens ini dapat
digunakan untuk perbandingan sekuens dari gen yang sama pada spesies yang
berbeda, sehingga dimungkinkan dibuatnya diagram filogenetik (Muladno 2002).
Keragaman Genetik
Keragaman genetik adalah perbedaan di dalam urutan DNA antara
individu, kelompok atau antara populasi dengan subpopulasi. Sumber keragaman
ini adalah SNP yang berupa adanya pengulangan urutan sekuen, insersi, delesi dan
rekombinasi. Menurut Kirby (1990) keragaman genetik atau polimorfisme genetik
adalah terdapatnya lebih dari satu bentuk atau macam genotipe di dalam populasi.
Keragaman genetik bisa dilihat dari sifat-sifat eksternal sampai urutan asam
amino dan protein lainnya.
Identifikasi keragaman genetik dalam suatu populasi bertujuan untuk
mengetahui dan melestarikan bangsa-bangsa dalam populasi terkait dengan
keragaman genetik yang tinggi. Informasi keragaman genetik suatu bangsa akan
sangat bermanfaat bagi ketahanan dan ketersediaan bahan pangan yang
berkesinambungan (Blott et al. 2003). Populasi dinilai beragam jika memiliki dua
atau lebih alel dalam satu lokus dengan frekuensi yang cukup, biasanya lebih dari
1% (Nei dan Kumar 2000).
Tingkat keragaman dalam populasi dapat digambarkan dari frekuensi alel.
Frekuensi alel merupakan rasio relatif suatu alel terhadap keseluruhan alel yang
ditemukan dalam satu populasi (Nei dan Kumar 2000). Ukuran tinggi rendahnya
keragaman genetik dalam suatu kelompok atau populasi dapat dilihat berdasarkan
nilai heterozigositas (Notter 1999). Nilai heterozigositas yang tinggi menunjukkan
tingginya keragaman dalam populasi sehingga masih dimungkinkan untuk
melakukan seleksi.
Gen Hormon Pertumbuhan
Gen Growth Hormone (GH) memiliki peranan penting dalam
pertumbuhan dan perkembangan longitudinal pascanatal, pertumbuhan jaringan,
laktasi, reproduksi dan metabolisme protein, lipid, juga karbohidrat (Akers 2006;
ThidarMyint et al. 2008). Pengaruh GH terhadap pertumbuhan telah diamati pada
beberapa jaringan, termasuk tulang, otot dan jaringan adiposa. Pada hewan
ruminansia, GH berperanan terhadap pengaturan perkembangan kelenjar mammae
(Akers 2006). Gen GH telah digunakan secara luas sebagai penanda genetik pada
beberapa spesies ternak seperti pada sapi (Bos taurus dan Bos indicus) (Zhou et
al. 2005; Katoh et al. 2008), domba (Ovis aries) (Marques et al. 2006) dan
kambing (Capra hircus) (Boutinaud et al. 2003).
Proses biokimia secara nyata tentang cara kerja GH untuk mengontrol
produksi susu belum diketahui secara penuh. Tidak ada situs reseptor untuk
hormon yang ditemukan dalam kelenjar mammae. Growth Hormone (GH)
menstimulasi kelenjar mammae dengan cara menstimulasi produksi somatomedin,
yaitu hormon yang dihasilkan oleh jaringan tubuh lain seperti hati (Gambar 2).
Berdasarkan hasil penelitian diketahui bahwa injeksi subkutan bGH pada sapi
yang sedang laktasi akan menyebabkan peningkatan produksi susu 10-40%, yaitu
melalui peningkatan konsentrasi plasma IGF1 sebesar 3-4 kali. Aliran darah ke
yang mencapai kelenjar mammae sapi sehari sebelum dibandingkan dengan tujuh
hari setelah perlakuan bGH berturut-turut adalah 24 dan 116 nmol/menit/setengah
ambing.
Gambar 2 Diagram Alir Pengaturan Sekresi Hormon Pertumbuhan dan Kerjanya pada Ternak Domestik (Lawrance dan Fowler 2002).
Hormon pertumbuhan atau growth hormone (GH) merupakan hormon
anabolik yang disintesis dan disekresikan oleh sel somatotrop pada lobe anterior
pituitari (Ayuk dan Sheppard 2006). Gen GH terletak pada kromosom 19 pada
sapi dengan panjang sekitar 2800 pb yang tersusun atas lima ekson dan empat
intron (Gambar 3). Protein GH terdiri atas 191 asam amino dengan berat molekul
22 kDa. Proses sintesis dan sekresinya dipengaruhi oleh umur dan jenis kelamin
Lokus = BOVGH Panjang = 2856 pb
Gen = 649-723, 971-1131, 1359-1475, 1703-1864.2138-2439 Sekuen depan = 648 = 648 bp
Gambar 3 Rekonstruksi struktur gen GH berdasarkan sekuens gen GH di GenBank (Kode Akses J00008).
Polimorfisme pada exon 5 (daerah 5’) gen bGH dilaporkan Zhang et al.
(1992) dan ditemukan dua alel yaitu alel L dan V. Subsitusi Sitosin (C) dengan
Guanin (G) pada posisi 2141 menyebabkan perubahan asam amino dari leusin (L,
kodon CTG) menjadi valin (V, kodon GTC). Transversi ini memungkinkan
genotiping pada lokus spesifik ini menggunakan enzim restriksi Alu1 karena
enzim ini mengenali situs potong AGCT. Keragaman genetik yang disebabkan
perubahan asam amino tersebut dipercaya berhubungan dengan level plasma GH
seperti yang diduga oleh Scanes (2003) bahwa genotipe LL berhubungan dengan
kosentrasi sirkulasi GH yang lebih tinggi jika dibandingkan genotipe LV. Eppard
et al. (1992) menemukan dua bentuk bGH rekombinan sebagai hasil perubahan
asam amino valin menjadi leusin pada posisi 127 pada lokus bGH.
Zakisadeh et al. (2006) melaporkan adanya keragaman gen hormon
pertumbuhan pada tiga bangsa sapi perah Iran dan sapi FH dengan menggunakan
metode PCR-RFLP. Bangsa sapi lokal Iran yang digunakan yaitu bangsa
Mazandarani, Sarabi dan Golpaygani yang selanjutnya dibandingkan dengan sapi
FH. Berdasarkan hasil penelitian didapatkan bahwa terdapat perbedaan signifikan
hasil genotiping pada tiga bangsa sapi Iran, jika dibandingkan dengan hasil
genotiping pada bangsa sapi FH. Didapatkan dua alel pada penelitian ini yaitu alel
(+) dan (-). Perbedaan ini ditunjukkan oleh frekuensi alel (+) pada sapi
Mazandarani, Sarabi, Golpaygani dan sapi FH berturut-turut adalah 0.52, 0.54,
0.47 dan 0.86. Adanya perbedaan yang nyata frekuensi genotipe (+) antara bangsa
Holstein dan sapi lokal Iran diduga dipengaruhi oleh perbedaan bangsa pada
ternak tersebut.
Gen Reseptor Hormon Pertumbuhan
Gen Growth Hormone Receptor (GHR) merupakan anggota dari kelas 1
cytokine receptor super family. Cytokine receptor berasosiasi dengan salah satu
anggota dari Janus Kinase Family (JAK), yang mengaktifkan spesific
transcription factor, signal transducer dan aktivator transkripsi (Scanes 2003).
Gen GHR pada sapi terletak pada kromosom 20 (Moody et al. 1995). Gen GHR
terdiri atas 10 ekson dengan panjang 25.688 pb (Gambar 4). Proses transkripsi gen
GHR pada sapi diinisiasikan oleh tiga promotor utama 1A, 1B dan 1C. Gen GH
1A diekspresikan secara eksklusif pada hati, sedangkan gen GHR 1B dan gen
GHR 1C diekspresikan pada jaringan yaitu pada tahap awal dan akhir
perkembangan setelah setelah kelahiran (Lucy et al. 1998).
Lokus = EF207442
Gambar 4 Rekonstruksi struktur gen GHR berdasarkan sekuens gen GHR di GenBank (Kode Akses EF207442).
Reseptor hormon pertumbuhan atau Growth Hormone Receptor (GHR)
merupakan protein transmembran yang mengikat GH dengan afinitas dan
spesifitas yang tinggi. Ekspresi reseptor diperlukan untuk aktivitas selular
terhadap GH. Hal tersebut mengindikasikan bahwa perubahan fungsi GHR dapat
mempengaruhi kemampuan mengikat GH dan aktivitas GH dalam jaringan target
(DiStasio et al. 2005).
GHR merupakan member dari super family reseptor sitokinin atau
hematopoietin. Ikatan antara hormon pertumbuhan dengan reseptornya
mengakibatkan terjadinya aktivitas enzim forforilasi yang dilakukan oleh enzim
kinase dengan cara menambah gugus fosfat. Hal ini menyebabkan timbulnya
reaksi intrasel yang dapat berpengaruh pada metabolisme dari fungsi sel (Granner
2003).
Gen GHR berperan penting dalam proses pertumbuhan ternak. Mutasi
pada gen GHR telah diasosiasikan sebagai Larontype dwarfism pada manusia, sex
linked dwarfism pada ayam (Burnside et al. 1992), sifat pertumbuhan pada sapi
pedaging dan sifat produksi susu sapi FH (Aggrey et al. 1999). Ge et al. (2000)
menemukan adanya mutasi pada lokus GHR|Alu1. Mutasi yang terjadi merupakan
mutasi subtitusi tipe transisi atau terjadi perubahan basa nukleotida A menjadi G.
Perubahan basa tersebut menyebabkan perubahan asam amino serin (AGC)
menjadi glisin (GGC). Perubahan ini terjadi pada posisi basa ke 3338 (Genebank
kode akses EF207442). Zulkharnaim (2008) mengidentifikasi adanya keragaman
gen GHR|Alu1 pada sapi Bali, Limousin, Simmental dan sapi pesisir. Berdasarkan
hasil sekuen yang dilakukan ditemukan adanya mutasi pada sapi dengan genotipe
AG dan GG, sedangkan genotipe AA tidak terjadi mutasi.
Produksi Susu Sapi FH
Produktivitas sapi perah dapat dievaluasi dengan cara pengukuran
produksi susu selama satu masa laktasi. Produksi susu biasanya cukup tinggi
setelah enam minggu masa laktasi hingga mencapai produksi maksimum, setelah
itu terjadi penurunan produksi secara bertahap sampai akhir masa laktasi.
Penurunan produksi susu yang terjadi setelah mencapai puncak laktasi adalah
sebesar 6% setiap bulannya (Tyler dan Ensminger 2006).
Produksi puncak tergantung pada kondisi induk pada saat melahirkan,
keturunan, terbebasnya induk dari infeksi penyakit, serta pakan setelah
melahirkan. Induk yang mengalami penurunan produksi susu secara cepat setelah
adalah kemampuan induk sapi mempertahankan tingkat produksi selama masa
laktasi. Persistensi ini dipengaruhi oleh umur sapi, kondisi sapi pada saat beranak,
lama masa kering sebelumnya dan jumlah pakan (Akers 2002). Gambar 5
memperlihatkan variasi produksi susu selama masa laktasi dengan tingkat
persistensi yang berbeda.
Gambar 5 Produksi susu selama laktasi dengan tingkat persistensi yang berbeda (Tyler dan Ensminger 2006).
Pada umumnya lama masa laktasi adalah 10 bulan (305 hari) pada
sapi-sapi yang mempunyai selang beranak 12 bulan. Produksi air susu yang tertinggi
diperoleh pada periode laktasi ke tiga (Philips 2002). Produksi susu total setiap
laktasi bervariasi, namun umumnya puncak produksi dicapai pada umur 6-7
tahun, atau pada laktasi ke 3 dan ke 4. Mulai dari laktasi pertama produksi susu
akan meningkat sampai umur dewasa. Umur sapi yang semakin bertambah
menyebabkan penurunan produksi secara perlahan. Produksi susu pada laktasi
pertama adalah 70%, laktasi kedua adalah 80%, laktasi ketiga 90%, laktasi ke
empat 95% dari produksi susu pada umur dewasa dengan selang beranak 12 bulan
dan beranak pertama pada umur dua tahun (Tyler dan Ensminger 2006).
Produksi susu secara umum dikontrol oleh faktor luar (eksternal) dan
ternak seperti iklim, jumlah dan kualitas pakan, penyakit dan parasit (Indrijani
2001), sedangkan faktor internal adalah faktor genetik, periode laktasi, frekuensi
pemerahan, umur dan ukuran tubuh ternak, masa kering, siklus estrus dan
kebuntingan, ketosis dan milk fever (Sudono et al. 2003).
Masa laktasi adalah periode sapi selama menghasilkan air susu yaitu
antara waktu beranak sampai masa kering (Sudono et al. 2003). Umumnya masa
laktasi adalah selama 305 hari dengan 60 hari masa kering . Pada kenyataannya,
masa laktasi seekor sapi perah bervariasi dari 270 hari hingga 400 hari. Rataan
masa laktasi sapi perah di beberapa peternakan sapi perah di Indonesia sangat
bervariasi yaitu 363, 355, 368 dan 348 hari masing-masing pada daerah
Pengalengan, Kertasari, Lembang dan Cisarua (Sudarisman et al. 1996). Setelah
sapi beranak produksi susu akan meningkat, produksi maksimum akan dicapai
sekitar minggu ke empat sampai minggu ke enam, kemudian akan turun
perlahan-lahan sampai akhir laktasi (Tyler and Ensminger 2006).
Selain masa laktasi, masa kering dan masa kosong juga berpengaruh
terhadap produksi susu. Masa kering yaitu periode atau lamanya sapi berhenti
diperah hingga beranak (Sudono et al. 2003). Masa kering yang terbaik adalah 50
sampai 60 hari, karena produksi susu akan lebih tinggi pada masa berikutnya
dibandingkan masa kering yang diperpanjang atau diperpendek. Periode masa
kering berguna untuk memperbaiki tubuh dengan nutrisi yang telah dipakai
selama masa laktasi, memperbaiki dan memperbaharui sistem kelenjar susu dan
saluran-salurannya, serta tambahan stimulasi untuk laktasi berikutnya. Periode
kering memungkinkan untuk glandula mamari dari sapi induk memperkuat diri
kembali dan membentuk cadangan zat-zat makanan dalam tubuh untuk laktasi
berikutnya (Akers 2002).
Masa kosong adalah jarak antara induk beranak hingga bunting kembali.
Masa kosong merupakan faktor yang penting dalam tata laksana sapi perah dalam
hal kebuntingan yang diinginkan. Panjang masa kosong akan berbeda pada setiap
ternak. Rataan masa kosong pada pada sapi perah di Indonesia adalah 133 hari
pada peternakan Taurus Dairy Farm dan 139 hari pada peternakan BPTU
uteri, estrus kembali setelah beranak, interval dikawinkan kembali setelah beranak
dan service per conception (S/C) (Akers 2002).
Hubungan Varian Genetik Gen Pengontrol Produksi Susu dengan Sifat Produksi Susu
Seleksi keunggulan genetik pada sapi FH dapat dilakukan melalui
identifikasi keragaman gen yang terkait dengan sifat produksi dan kualitas susu.
Keragaman genetik bisa digunakan untuk pendugaan adanya seleksi atau tidak
dalam suatu populasi. Beberapa penelitian telah dilakukan untuk penentuan
hubungan antara keragaman genetik dengan sifat produksi susu pada sapi perah.
Kelompok gen hormon pertumbuhan merupakan gen kandidat yang baik untuk
analisis dengan Quantitatif Trait Loci (QTL) dikarenakan pengaruh biologisnya
pada sifat-sifat kuantitatif. Dijelaskan oleh Chung et al. (1996), bahwa keragaman
alelik dalam sekuens struktural atau regulator gen, serta keragaman dalam sekuens
intron atau pengapitnya, secara langsung atau tidak langsung berpengaruh
terhadap produksi susu dan performan pertumbuhan.
Beberapa penelitian dilakukan untuk penentuan keragaman genetik sapi
FH di Indonesia dan hubungannya dengan produksi susu. Rahmani et al. (2004)
melaporkan keragaman genetik sapi FH berdasarkan gen hormon pertumbuhan
pada bagian intron 3 di BPTU Baturraden dan menemukan empat alel yaitu A, B,
C dan D dengan lima tipe genotipe yaitu AA, AD, BC, CD dan DD. Frekuensi
alel A, B, C dan D berturut-turut adalah 21.19, 8,94, 3.97 dan 65.89. Keragaman
genotipe dan alel ini dihubungkan dengan nilai pemuliaan produksi susu dan
protein susu. Nilai pemuliaan produksi susu dan protein susu nyata dipengaruhi
(P<0.05) oleh genotipe gen GH yaitu AD, CD dan DD. Individu dengan genotipe
CD mempunyai nilai pemuliaan lebih kecil dibandingkan dengan genotipe AD.
Individu dengan genotipe CD mempunyai nilai pemuliaan dari produksi protein
susu lebih tinggi dibandingkan dengan genotipe AD dan AA.
Penelitian tentang hubungan keragaman gen GH dengan tingkat produksi
susu juga telah dilakukan pada bangsa sapi perah luar negeri. Dybus et al. (2002)
melaporkan keragaman gen hormon pertumbuhan menggunakan metode
restriction fragment length polymorphism (RFLP) dengan enzim pemotong Alu1
genotipe yaitu LL, LV dan VV dengan frekuensi genotipe tertinggi yaitu genotipe
LL 0.653 (65%). Secara statistik perbedaan genotipe berpengaruh terhadap tingkat
produksi susu (305 hari), kadar lemak dan protein. Pengaruh ini terlihat nyata
pada periode laktasi pertama. Genotipe LL memiliki tingkat produksi susu
(225kg), kadar lemak (7kg) dan protein (7kg) yang lebih tinggi dibandingkan
genotipe LV.
Dybus et al. (2002) melihat pengaruh antara kombinasi genotipe gen GH
lokus GH|Alu1 dan GH|Msp1 dengan produksi susu kumulatif (305 hari), kadar
lemak dan protein pada sapi Black and White Polandia. Sapi dengan genotipe
LL/++ memiliki produksi susu yang lebih tinggi (P≤0.01) sebesar 332, 259 dan
649 kg dibandingkan sapi dengan genotipe LL/+-, LV/++ dan LV/+-
berturut-turut (P≤0.01) dan LV/++ memiliki produksi susu lebih tinggi (390 kg)
dibandingkan sapi dengan genotipe LV/+- (P≤0.05) pada periode laktasi I.
Kombinasi genotipe kedua gen tersebut, tidak berpengaruh signifikan terhadap
produksi susu kumulatif (305 hari) pada laktasi ke II dan III.
Keragaman genotipe juga berpengaruh signifikan pada kadar lemak susu
yang diproduksi pada laktasi 1. Genotipe LL/++ memiliki produksi susu dengan
kadar lemak lebih tinggi dibandingkan sapi dengan genotipe LL/+-,LV/++, dan
LV/+- berturut-turut (9.6 kg, 8.0 kg dan 20.7 kg) (P≤0.01). Susu yang berasal dari
genotipe LL/+- memiliki kadar lemak (11.1 kg) lebih tinggi dibandingkan
genotipe LV/+- (P≤0.05). Protein susu secara nyata juga dipengaruhi oleh
keragaman genotipe GH|Alu1 dan GH|Msp1. Pada laktasi 1, sapi dengan genotipe
LL/++ memiliki kadar protein susu lebih tinggi dibandingkan sapi dengan
genotipe LV/+- dan LL/+- berturut-turut (20.7% dan 10.20%), tetapi keragaman
genotipe tidak berpengaruh nyata terhadap kadar lemak dan protein susu (Dybus
et al. 2002).
Pengamatan yang dilakukan Zhou et al. (2005) menunjukkan adanya
pengaruh varian gen hormon pertumbuhan atau growth hormone (GH) lokus
Msp1 terhadap produksi susu kumulatif 305 hari. Hasil yang didapatkan
menunjukkan bahwa sapi dengan genotipe AA memiliki tingkat produksi susu
lebih tinggi dibandingkan dengan sapi yang bergenotipe AB, dengan selisih 353
protein 9.2 kg lebih tinggi dibandingkan sapi dengan genotipe AB. Perbedaan
pendapat dari berbagai hasil studi masih terdapat dalam melihat pengaruh
keragaman gen GH|Msp1 terhadap produksi susu sapi perah. Berdasarkan hasil
kesepakatan umum dari berbagai peneliti, diketahui bahwa alel A pada gen GH
berkaitan erat dengan sifat tingginya produksi susu, protein dan kadar lemak (Yao
et al. 1996; Sabour et al. 1997), tetapi rendah dalam hal persentase lemak dan
protein (Lee 1993; Hoj 1993; dan Falaki 1996).
Selain pada sapi, hubungan antara keragaman gen GH dengan produksi
susu juga telah diteliti pada kambing perah di luar negeri. Keragaman gen GH
pada kambing Algarvia (Portugis) menggunakan metode Single Strand
Conformation Polymorphism (SSCP) berhubungan dengan produksi susu, kadar
lemak susu dan protein susu (Boutinaud et al. 2003). Keragaman haplotipe gen
GH|HaeIII pada kambing Boer jantan berpengaruh terhadap bobot lahir, bobot
sapih, pertambahan bobot badan perhari sebelum sapih dan bobot 11 bulan (Hua
Waktu dan Tempat
Penelitian ini dilaksanakan selama empat bulan, yaitu dari bulan
September sampai Desember 2010. Penelitian ini dilaksanakan di Laboratorium
Genetika Molekuler Ternak, Bagian Pemuliaan dan Genetika, Departemen Ilmu
Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian
Bogor.
Materi
Sampel Darah Sapi Friesian-Holstein
Sampel darah yang digunakan adalah sampel darah sapi FH yang diambil
dari bagian vena jugularis. Total sampel darah yang digunakan 370 sampel (Tabel
1). Sampel darah diambil dari sapi perah dengan dua kondisi pemeliharaan
berbeda. Lokasi pertama adalah Unit Pengembangan Ternak Daerah (UPTD)
Balai Pengembangan Pembibitan Sapi Perah (BPPT-SP) Cikole (88 sampel) dan
Balai Embrio Transfer (BET) Cipelang (34 sampel). Pemeliharaan sapi dilakukan
dalam skala besar dengan penerapan manajemen pemeliharaan intensif. Lokasi
kedua berasal dari peternakan rakyat Desa Cilumber (98 sampel) dan Pasir Kemis
(95 sampel) yang merupakan binaan KPSBU Lembang dan menerapkan
manajemen pemeliharaan semi intensif. Aliran genetik dari pejantan terhadap
anak betinanya ditentukan dari sampel FH pejantan yang berasal dari lokasi Balai
Besar Inseminasi Buatan (BBIB) Lembang (17 sampel) dan BBIB Singosari (32
sampel).
Tabel 1 Sampel darah sapi Friesian Holstein (FH)
No Populasi Sex Jumlah (ekor) Keterangan
1. BBIB Singosari Jantan 32 UPT Pusat
2. BBIB Lembang Jantan 17 UPT Pusat
3. BET Cipelang Betina 34 UPT Pusat
4. BPPT Cikole Betina 88 UPT Jabar
5. Peternakan Cilumber Betina 98 Peternakan rakyat
6. Peternakan Pasir kemis Betina 95 Peternakan Rakyat
Primer
Primer untuk mengamplifikasi ruas gen GH mengikuti Balogh et al.
(2009) dengan produk hasil amplifikasi sepanjang 432 bp. Primer untuk
mengamplifikasi ruas gen GHR mengikuti Andreas et al. (2010) dengan produk
hasil amplifikasi sepanjang 298 pb. Runutan Primer Forward dan Reverse dapat
dilihat pada Tabel 2.
Tabel 2 Runutan primer yang digunakan
Primer Sekuens primer Ukuran
Produk
Target amplifikasi
Referensi
GH F 5’CGGACCGTGTCTATGAGAAGCTGAAG-3’ 432 pb Ekson 4 dan
Intron 4
Balogh et al. (2009)
GH R 5’-GTTCTTGAGCAGCGCGTCGTCA-3’
GHR F 5’-CGC TTACTTCTGCGAGGTAGACGC-3’ 298 pb Ekson 10 Andreas et
al. (2010)
GHR R 5’-GTCTGTGCTCACAT GCCAC-3’
Data Produksi Susu
Pengambilan data produksi susu dilakukan di peternakan dengan sistem
pemeliharaan intensif (BPPT Cikole), hal ini bertujuan untuk meminimalkan
pengaruh lingkungan terhadap tingkat produksi susu. Data produksi susu
merupakan data sekunder tahun 2008-2010. Data produksi susu dipilih bagi sapi
FH yang telah digenotyping. Data tersebut berasal dari sapi FH periode laktasi
ke-1 (46 ekor), laktasi ke-3 (7 ekor) dan laktasi ke-4 (3 ekor). Data yang diambil
meliputi data produksi susu dan data identitas ternak (nama atau nomor sapi, umur
sapi dan data silsilah silsilah tetuanya (pejantan dan induk). Data yang dianalisis
adalah data dengan catatan produksi minimal 180 hari, yang sebelumnya
dianalisis kenormalannya dengan normality test. Jumlah data yang memenuhi
untuk analisis adalah data yang berasal dari sapi FH periode laktasi I yaitu
sebanyak 40 ekor. Ternak yang memiliki data silsilah yang lengkap hanya
terdapat sekitar 20% dari total ternak dengan data produksi susu, sehingga tidak
memungkinkan untuk dilakukan analisis nilai pemuliaan produksi susu.
Catatan produksi susu yang digunakan merupakan catatan produksi harian
yang diukur pada setiap minggunya. Data produksi susu dicatat dua kali sehari,
yaitu pada pemerahan pagi dan sore. Produksi susu total sehari adalah
hari diestimasi berdasarkan data harian yang diukur setiap minggu. Data kumulatif
parsial dihitung berdasarkan data produksi 15 hari.
Prosedur
Ekstraksi DNA dari Sampel Darah dan Sperma
Ektraksi DNA dilakukan dari darah dan sperma sapi FH yang disimpan
dalam alkohol. Ekstraksi DNA dari darah dilakukan untuk sampel yang berasal
dari peternakan rakyat Desa Pasir Kemis dan Desa Cilumber serta stasiun Bibit
BPPT Cikole, BET Cipelang dan BBIB Singosari. Ekstraksi DNA dari sperma
dilakukan untuk sampel yang berasal dari stasiun bibit BBIB Lembang. Isolasi
DNA dari sampel darah dan sperma menggunakan metode ekstraksi
phenol-chloroform (Sambrook et al. 1989) yang telah dimodifikasi untuk sampel darah
dan sperma yang disimpan dalam alkohol. Tahapan ekstraksi DNA dari darah dan
sperma dijelaskan sebagai berikut:
Preparasi Sampel. Darah dan sperma dalam alkohol sebanyak 200 µl
dimasukkan ke dalam tabung 1,5 ml. Alkohol dihilangkan dari sampel dengan
penambahan air destilasi sampai 1000 µl, lalu dibiarkan selama 20 menit. Sampel
kemudian diendapkan dengan sentrifugasi pada kecepatan 8000 rpm selama 5
menit.
Degradasi Protein. Sampel darah dan sperma yang telah bersih dari alkohol
ditambahkan 1x STE (sodium tris EDTA) sampai volume 340 µl, 40 µl sodium
dosesil sulfat 10% dan 20 µl proteinase K 5 mg/ml. Campuran diinkubasi pada
inkubator kering dengan suhu 50ºC selama semalam sambil digoyang perlahan
Degradasi Bahan Organik. Sampel yang telah diinkubasi ditambahkan 400 µl
larutan phenol, 400 µl choloform:isoamil alkohol (24:1) dan 40 µl NaCl 5M.
Campuran digoyang pada suhu ruang selama satu jam.
Presipitasi DNA. Sampel selanjutnya disentrifugasi pada kecepatan 5000 rpm
selama 10 menit hingga fase air terpisah dengan fase phenol. Fase air dipindahkan
dalam tabung baru dengan volume terukur. Molekul DNA diendapkan melalui
penambahan 2x volume alkohol absolut dan 0,1x volume NaCl 5M. Campuran
kemudian diinkubasi pada suhu -20ºC selama semalam. Pengendapan DNA
selanjutnya dilakukan dengan sentrifugasi pada kecepatan 12000 rpm selama 10
diendapkan lagi. Endapan DNA yang telah bersih dari alkohol dipulihkan dengan
penambahan 100 µl TE (Tris EDTA). Sampel DNA disimpan pada suhu -20ºC
dan siap untuk digunakan.
Amplifikasi Ruas Gen GH dan GHR
Amplifikasi ruas gen GH dan GHR dilakukan dengan metode PCR
(Polymerase Chain Reaction). Pereaksi yang digunakan untuk amplifikasi kedua
ruas gen target adalah 2 µl sampel DNA, masing-masing primer 25 ρmol,
campuran dNTP 200 µM, MgCl2 1 mM dan taq polymerase 0,5 unit dan bufernya
dalam larutan total 25 µl. Amplifikasi in vitro dengan mesin thermal cycler
dilakukan dengan kondisi denaturasi awal pada suhu 94ºC selama 5 menit, 35
siklus yang terdiri atas denaturasi pada suhu 94ºC selama 45 detik, penempelan
primer pada suhu 62ºC selama 45 detik dan pemanjangan DNA baru pada suhu
72ºC selama 1 menit dan pemanjangan akhir pada suhu 72ºC selama 5 menit.
Penentuan Genotipe dengan Pendekatan PCR-RFLP
Penentuan genotipe masing-masing individu dilakukan dengan pendekatan
Restriction Fragment Length Polymorphism (RFLP) yang divisualisasikan pada
gel agarosa 2% dengan bufer 0,5x TBE (tris borat EDTA) yang difungsikan pada
tegangan 100 Volt selama 40 menit yang diwarnai dengan etidium bromida diatas
UV trans iluminator. Enzim pemotong yang digunakan untuk ruas gen GH dan
GHR adalah AluI.
Analisis Data
Frekuensi Genotipe dan Alel
Frekuensi alel merupakan rasio suatu alel terhadap keseluruhan alel pada
suatu lokus dalam populasi. Model matematika frekuensi alel (Nei dan Kumar
2000) dilambangkan sebagai berikut:
Frekuensi genotipe dapat diperkirakan dengan menghitung perbandingan jumlah
genotipe pada populasi. Frekuensi genotipe AiAi (χii) dapat dihitung dengan
n nii
ii =
χ
Keterangan :
χii = frekuensi genotipe ke ii nii = jumlah sampel bergenotipe ii nij = jumlah sampel bergenotipe ij N = jumlah populasi sampel χi = frekuensi alel ke i
Proporsi Hardy-Weinberg
∑
−=
E E
O 2
2 ( )
χ
χ2
= nilai chi-square uji
O = jumlah pengamatan genotipe ke-i E = jumlah harapan genotipe ke-i
Heterozigositas
Heterozigositas merupakan salah satu indikator keragaman gen dalam
populasi yang menunjukkan proporsi heterozigot pada individu dengan
pengambilan secara acak. Jika N1ij adalah jumlah individu heterozigositas pada
lokus ke-1, dan N jumlah individu yang dianalisis, maka heterozigositas
pengamatan dapat dihitung sebagai berikut:
Heterozigositas harapan dihitung sebagai berikut:
Keterangan :
Ho = frekuensi heterozigositas harapan
P1i = frekuensi alel ke-I pada lokus 1
n = jumlah alel pada lokus ke-1
Analisis keragaman dan keseimbangan genetik gen GH dan GHR ini diuji dengan
Analisis Hubungan Genotipe Gen GH dan GHR terhadap Produksi Susu
Data produksi susu merupakan data kumulatif parsial dari 40 ekor sapi FH
periode laktasi 1. Data kumulatif parsial ini didapatkan dari data catatan mingguan
yang dianalisis per-15 hari. Analisis hubungan antara genotipe GH dan GHR
dengan produksi susu dilakukan dengan metode analisis General Linear Model
(GLM) dengan software SAS Ver 9,1. Pengaruh masing-masing varian genotipe
gen GH dan GHR terhadap produksi susu dihitung berdasarkan model linear dari
Mattjik dan Sumertawijaya (2006):
Yijklm = μ + Ai + eijk
Keterangan :
Yijk = nilai pengamatan
μ = komponen aditif dari rataan Ai = pengaruh aditif gen i
Amplifikasi Gen Growth Hormone (GH)
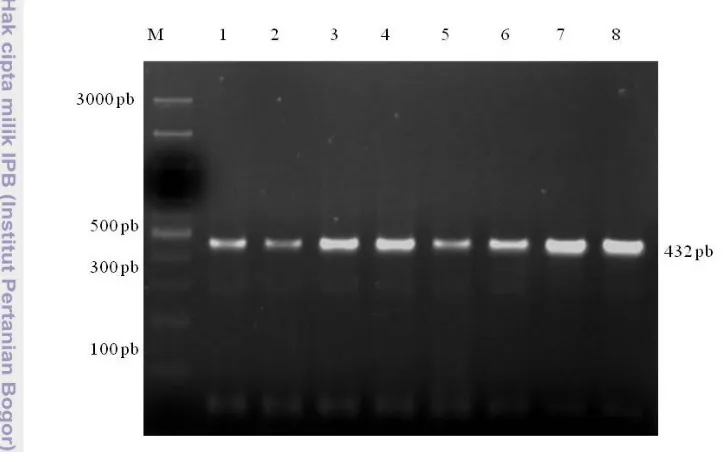
Panjang produk hasil amplifikasi ruas gen GH|Alu1 adalah 432 pb,
meliputi 55 pb dari ekson 4, intron 4 dan 99 pb dari ekson 5. (Genbank Nomor
Akses J00008) (Balogh et al. 2009). Keberhasilan amplifikasi pada ruas gen
GH|Alu1 dalam penelitian ini adalah 100%. Hasil amplifikasi ruas gen GH|Alu1
disajikan pada Gambar 6.
Gambar 6 Visualisasi hasil amplifikasi ruas gen GH pada gel agarose 2% M=Marker 100 pasang basa (pb), 1-8=nomor sampel.
Salah satu faktor penentu tingkat keberhasilan amplifikasi adalah suhu
annealing. Suhu annealing merupakan suhu yang memungkinkan terjadinya
penempelan primer pada DNA cetakan selama proses PCR. Suhu annealing yang
digunakan dalam penelitian ini sesuai dengan suhu annealing yang digunakan
oleh Balogh et al. (2009), Mohammadabadi et al. (2010) dan Andreas et al.
(2010) yaitu pada suhu 60oC selama 1 menit. Jakaria (2008) berhasil mengamplifikasi pada suhu annealing yang lebih rendah yaitu 55oC.
Ge et al. (2000) berhasil mengamplifikasi ruas gen yang sama pada suhu
annealing yang lebih tinggi yaitu 65oC selama 1 menit. Komposisi nukleotida (A, G, T, C) serta panjang primer merupakan faktor utama penentu suhu annealing
yang digunakan. Suhu annealing yang terlalu rendah dapat mengakibatkan
ruas gen GH|Alu1 juga dipengaruhi oleh beberapa faktor lain seperti bahan
pereaksi PCR dan kondisi mesin thermal cylcler yang digunakan dalam PCR.
Keragaman Genotipe Gen GH Sapi FH dengan Pendekatan PCR-RFLP
Enzim Alu1 sebagai enzim restriksi mengenali situs pemotongan AG|CT.
Tiga situs pemotongan Alu1 dihasilkan yaitu fragmen dengan panjang 20, 51, 96
dan 265 pb yang dikenal dengan alel leucine (L). Keragaman pada ruas gen
GH|Alu1 diperoleh karena adanya mutasi pada posisi ke 1758 yaitu dari C
menjadi G (Gambar 7) (Lucy et al. 1993; Balogh et al. 2009). Perubahan ini
menyebabkan perubahan fragmen hasil pemotongan Alu1 menjadi 20, 147 dan
265 pb yang dikenal dengan alel valine (V) (Lucy et al. 1993; Balogh et al. 2009).
Gambar 7 Runutan nukleotida ruas gen GH (Genbank nomor akses J00008). Posisi primer (cetak tebal bergaris bawah), situs pemotongan enzim AluI (cetak tebal) dan titik mutasi (cetak tebal huruf kapital) ( Balogh et al. 2009).
Perbedaan sekuen alel L dan V yang disebabkan mutasi pada posisi ke
I758 (C menjadi G) disajikan pada Gambar 8.
Alel L : 5’---CCCTCCCTGGCAGGAG|CTGGAAGATGGCA---3’
Alel V : 5’---CCCTCCCTGGCAGGAGGTGGAAGATGGCA---3’
Gambar 8 Perbedaan sekuen alel L dan V gen GH|Alu1. Situs potong enzim restriksi Alu1 (cetak tebal) dengan titik mutasi (garis bawah).
Hasil genotyping pada ruas gen GH menghasilkan dua genotipe yaitu LL
dan LV (Gambar 9). Genotipe VV tidak ditemukan pada semua sampel yang
dianalisis. Visualisasi hasil genotyping ruas gen GH|Alu1 disajikan pada Gambar
9. Gen GH|Alu1 genotipe homozigot LL ditunjukkan dengan panjang fragmen 20,
1381 cctcagcaga gtcttcacca acagcttggt gtttggcacc tcggaccgtg tctatgagaa
1441 g|ctgaaggac ctggaggaag gcatcctggc cctgatgcgg gtggggatgg cgttgtgggt 1501 cccttccatg ctgggggcca tgcccgccct ctcctggctt agccaggaga atgcacgtgg 1561 gcttggggag acagatccct gctctctccc tctttctagc agtccagcct tgacccaggg 1621 gaaacctttt ccccttttga aacctccttc ctcgcccttc tccaagcctg taggggaggg
1681 tggaaaatgg agcgggcagg agggag|ctgc tcctgagggc ccttcggcct ctctgtctct
51, 96 dan 265 pb, sedangkan genotipe heterozigot LV ditunjukkan dengan
panjang fragmen 20, 51, 96, 147 dan 265 pb.
Hasil ini sejalan dengan Pereira et al. (2005) yang mendapatkan dua
genotipe (LL dan LV) pada sapi Canchim Brazil. Curi et al. (2006) tidak
menemukan genotipe VV pada sapi Zebu Brasil dan persilangannya. Mattos et al.
(2004) hanya menemukan genotipe LL pada 40 ekor pejantan Dairy Gyr Brazil.
Hasil ini berbeda dengan Dybus et al. (2002), yang mengidentifikasi keragaman
gen GH|Alu1 pada 1086 ekor sapi Black and White Polandia. Genotyping GH|Alu1
pada populasi sapi tersebut menghasilkan tiga genotipe yaitu LL, LV dan VV.
Grochowska et al. (2001) juga mengidentifikasi keragaman gen GH|Alu1 pada
sapi FH Polandia dan ditemukan tiga genotipe yaitu LL, LV dan VV. Perbedaan
hasil genotyping ini disebabkan perbedaan bangsa yang dianalisis dan perbedaan
sistem breeding yang digunakan. Perbedaan jumlah sampel juga mempengaruhi
peluang untuk mendapatkan genotipe yang berbeda.
Gambar 9 Visualisasi PCR-RFLP ruas gen GH|AluI pada gel agarosa 2% M: Marker 100 pb.
Hasil analisis pada ruas GH|Alu1 menunjukkan bahwa frekuensi alel L 88%
lebih tinggi dari alel V. Frekuensi alel L di semua lokasi penelitian adalah antara
0.92-0.98. Tingginya frekuensi alel L di semua lokasi pengamatan menyebabkan
VV tidak ditemukan di semua lokasi yang diamati. Frekuensi alel dan genotipe
ruas gen GH|Alu1 disajikan pada Tabel 3.
Tidak ditemukannya genotipe VV dari sapi betina Friesian Holstein baik di
peternakan rakyat maupun di stasiun bibit dipengaruhi oleh pejantan IB yang
digunakan. Pejantan yang digunakan untuk IB pada betina laktasi umumnya
berasal dari BBIB Lembang dan BBIB Singosari. Berdasarkan hasil analisis pada
sapi pejantan IB yang berasal dari BBIB Lembang dan BBIB Singosari tidak
ditemukan genotipe VV dan frekuensi alel V rendah pada pejantan yang diamati.
Tabel 3 Frekuensi genotipe dan alel pada ruas gen GH|Alu1
Populasi
GH
n Alel Genotipe
L V LL LV VV
BBIB Singosari 32 0.92 0.08 0.84(27) 0.16(5) 0.00
BBIB Lembang 17 0.97 0.03 0.94(16) 0.06(1) 0.00
Sub Total 0.94 0.06 0.88 0.12 0.00
Cikole 88 0.94 0.06 0.88(77) 0.13(11) 0.00
Cilumber 98 0.93 0.07 0.86(84) 0.14(14) 0.00
asir Kemis 95 0.96 0.04 0.92(87) 0.08(8) 0.00
BET Cipelang 40 0.98 0.02 0.95(38) 0.05(2) 0.00
Total 370 0.94 0.06 0.89 0.11 0.00
Keterangan: n dan (...) jumlah sampel (ekor)
Hasil penelitian ini sesuai dengan Sorensen et al. (2002) yang
mengidentifikasi keragaman gen GH|Alu1 pada bangsa Danish Hosltein dan
mendapatkan dua genotipe yaitu LL dan LV. Frekuensi tertinggi ditemukan pada
genotipe LL yaitu 0.85. Hasil ini sejalan dengan penelitian Grochowska et al.
(2009) yang mengidentifikasi keragaman gen GH|Alu1 pada 214 ekor sapi Polish
Friesian populasi Inggris. Hasil genotyping menunjukkan terdapat tiga genotipe
pada ruas gen GH|Alu1 yang diamati, dengan frekuensi genotipe tertinggi yaitu
genotipe LL (51%). Sebaliknya frekuensi genotipe paling rendah ditemukan pada
genotipe VV yaitu 13 %. Sabour et al. (1997) melaporkan keragaman gen
GH|Alu1 pada bangsa Ayrshire, Holstein dan Jersey, dengan frekuensi alel L lebih
tinggi dibandingkan alel V. Frekuensi alel V rendah pada ketiga bangsa tersebut
Amplifikasi Ruas Gen GHR
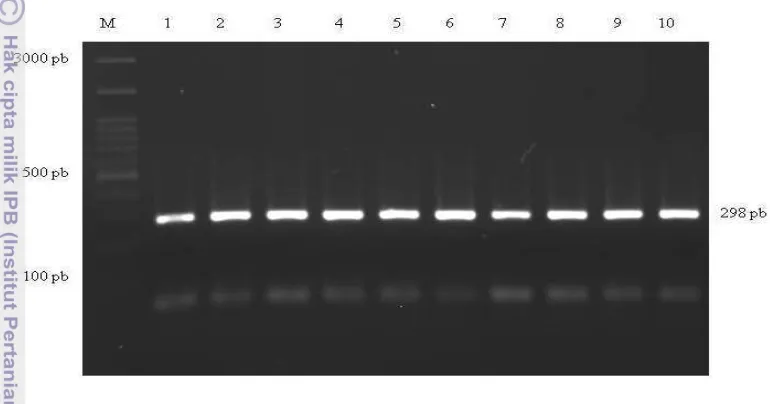
Tingkat keberhasilan amplifikasi gen GHR ekson 10 dalam penelitian ini
adalah 100%. Panjang produk hasil amplifikasi ruas gen GHR adalah 298 pb yang
terletak pada ekson 10 (Genbank Nomor Akses AY053546). Hasil amplifikasi
ruas gen GHR ekson 10 divisualisasikan pada gel agarose 1.5% yang disajikan
pada Gambar 10.
Gambar 10 Visualisasi hasil amplifikasi ruas gen GHR pada gel agarose 1.5%. M=Marker 100 pasang basa (pb), 1-16=nomor sampel.
Suhu annealing yang digunakan dalam penelitian ini sesuai dengan suhu
annealing yang digunakan oleh Andreas et al. (2010), Zulkharnaim et al. (2010)
dan Jakaria (2008) yaitu pada suhu 60oC selama 1 menit. Di stasio et al. (2005) berhasil mengamplifikasi ruas gen yang sama pada suhu annealing yang lebih
rendah yaitu 53oC selama 30 detik. Selain suhu annealing, lama waktu annealing juga menentukan spesifik tidaknya hasil amplifikasi. Faktor lain yang berperan
dalam menentukan keberhasilan amplifikasi adalah kualitas atau tingkat
kemurnian DNA sebagai DNA template.
Keragaman Genotipe Gen GHR Sapi FH dengan Pendekatan PCR-RFLP
Berdasarkan sekuen DNA ruas gen GHR yang diamplifikasi terdapat dua
situs pemotongan Alu1, yaitu menghasilkan fragmen dengan panjang 50, 81 dan
167 pb yang dikenal dengan alel A (Gambar 11). Keragaman pada ruas gen
GHR|Alu1 disebabkan adanya mutasi atau perubahan basa pada posisi ke 256,
dikenali oleh enzim Alu1, sehingga menghasilkan fragmen sepanjang 167 dan 131
pb, yang dikenal dengan alel G (Ge et al. 2000; Di Stasio et al. 2005).
Gambar 11 Runutan nukleotida ruas gen GHR (Genbank nomor akses Kode Akses. EF207442). Posisi primer (cetak tebal miring bergaris bawah), situs pemotongan enzim AluI (cetak tebal) dan titik mutasi (cetak tebal huruf kapital) (Ge et al. 2000).
Perbedaan sekuen alel A dan G yang disebabkan mutasi pada posisi ke
3338 (A menjadi G) pada gen GHR ekson 10 disajikan pada Gambar 12.
Perubahan basa tersebut menyebabkan perubahan asam amino serine menjadi
glicine.
Alel A : 5’---CCCTCCCTGGCAGGAG|CTGGAAGATGGCA---3’
Alel G : 5’---CCCTCCCTGGCAGGGGCTGGAAGATGGCA---3’
Gambar 12 Perbedaan sekuen alel A dan G gen GHR|Alu1. Situs potong enzim restriksi Alu1 (cetak tebal) dengan titik mutasi (garis bawah).
Hasil genotyping pada ruas gen GHR menghasilkan tiga genotipe, yaitu
AA, AG dan GG. Genotipe AA ditunjukkan fragmen sepanjang 50, 81 dan 167
pb. Genotipe GG ditunjukkan fragmen sepanjang 131 dan 167pb. Genotipe AG
merupakan gabungan dari keduanya yaitu fragmen sepanjang 50, 81, 131 dan 167
pb (Gambar 13).
Munculnya tiga fragmen pada individu yang sama dimungkinkan karena
adanya tipe yang berbeda dari kromosom homolog yang diturunkan oleh
masing-masing tetua (jantan dan betina) pada saat fertilisasi. Ternak dengan genotipe
homozigot AA menunjukkan bahwa kedua tetua menyumbangkan alel (gen) yang
sama. Ternak dengan genotipe heterozigot (AG) merupakan kombinasi dua alel
berbeda dari kedua tetuanya. Asumsi yang mendukung dalam penentuan tipe
genotipe ini yaitu semua pita yang memiliki laju migrasi yang sama merupakan
alel yang homolog (Nei dan Kumar 2000).
3241 taacttcatc gtggacaacg cttacttctg cgaggtagac gccaaaaagt acattgccct 3301 ggcccctcac gtcgaggctg aatcacacgt agagccaAgc tttaaccagg aagacattta 3361 catcaccaca gaaagcctta ccactacagc tgggaggtcg gggacagcag aacatgttcc 3421 aagttctgag atacctgtcc cagattatac ctccattcat atagtacagt ctccacaggg 3481 cctcgtactc aatgcgactg ccctgccctt gcctgacaaa gagtttctct catcatatgg
Gambar 13 Visualisasi PCR-RFLP ruas gen GHR|AluI pada gel agarosa 2% . M: Marker 100 pb.
Hasil analisis ruas gen GHR|Alu1 menunjukkan, bahwa frekuensi alel A
50% lebih tinggi dibandingkan frekuensi alel G (Tabel 4). Frekuensi alel A tinggi
di semua lokasi dengan kisaran 0.70-0.82. Tingginya frekuensi alel A
menyebabkan tingginya frekuensi genotipe AA di semua lokasi pengamatan.
Salah satu penyebab tingginya frekuensi alel A pada ruas gen GHR|Alu1 pada sapi
betina FH laktasi disebabkan oleh tingginya frekuensi alel A pada pejantan yang
digunakan untuk IB. Hasil ini sejalan dengan hasil penelitian yang dilakukan pada
sapi perah di luar negeri. Frekuensi alel A lebih tinggi dibandingkan dengan
frekuensi alel G yaitu pada sapi Freisian Holstein Polandia yaitu masing-masing
0.832 dan 0.168 (Olenski et al. 2010). Frekuensi alel G dan frekuensi genotipe
GG yang diperoleh sebaliknya yaitu rendah di semua lokasi yang dianalisis.
Frekuensi alel dan genotipe ruas gen GHR|Alu1 disajikan pada Tabel 4.
Tabel 4 Frekuensi genotipe dan alel pada ruas gen GHR|Alu1
Lokasi
GHR
n Alel Genotipe
A G AA AG GG
BBIB Singosari 32 0.81 0.19 0.75(24) 0.13(4) 0.13(4)
BBIB Lembang 17 0.82 0.18 0.76(13) 0.12(2) 0.12(2)
Sub Total 0.82 0.18 0.76 0.12 0.12
Cikole 88 0.70 0.30 0.49(43) 0.42(37) 0.09(8)
Cilumber 98 0.78 0.22 0.61(60) 0.33(32) 0.06(6)
Pasir Kemis 95 0.73 0.27 0.52(49) 0.43(41) 0.05(5)
BET Cipelang 40 0.78 0.23 0.68(27) 0.20(8) 0.13(5)
Total 370 0.75 0.25 0.58 0.34 0.08