IDENTIFIKASI KERAGAMAN GEN
DGAT1
|
Eae
I
PADA SAPI
FRIESIAN HOLSTEIN
DENGAN METODE PCR-RFLP
DICKY TRI UTAMA
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Identifikasi Keragaman Gen DGAT1|EaeI pada Sapi Friesian Holstein dengan Metode PCR-RFLP adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
ABSTRAK
DICKY TRI UTAMA. Identifikasi Keragaman Gen DGAT1|EaeI pada Sapi Friesian Holstein dengan Metode PCR-RFLP. Dibimbing oleh CECE SUMANTRI dan ANNEKE ANGGRAENI.
Gen Diacylglycerol O-acyltransferase 1 (DGAT1) merupakan gen yang berperan penting dalam mengontrol sintesis lemak susu. Penelitian ini bertujuan mengidentifikasi keragaman gen DGAT1 menggunakan enzim restriksi EaeI dengan metode Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP). Identifikasi dilakukan pada sapi FH dari Balitnak (86 ekor betina dara dan 12 ekor pedet) dan BIB Lembang (21 ekor pejantan). Panjang produk PCR yang diketahui adalah 411 pb. Genotyping menghasilkan dua tipe alel, yaitu K (411 pb) dan A (203 dan 208 pb); serta tiga genotipe, yaitu KK (411 pb), KA (203, 208 dan 411 pb) dan AA (203 dan 208 pb). Frekuensi genotipe KA (0.62) lebih tinggi dibandingkan dengan KK (0.35), sedangkan frekuensi genotipe AA sangat rendah (0.03). Dengan demikian, frekuensi alel K (0.66) lebih tinggi dibandingkan dengan alel A (0.34). Frekuensi gen DGAT1 sapi FH di BIB Lembang diketahui dalam keadaan seimbang (x2 hitung<x2 tabel), hal sebaliknya terjadi pada sapi FH Balitnak. Derajat heterozigositas pada kedua populasi relatif tinggi (Ho>He). Disimpulkan bahwa gen DGAT1 pada sapi FH pengamatan bersifat polimorfik.
Kata kunci: Friesian Holstein, gen DGAT1, keragaman gen, PCR-RFLP
ABSTRACT
DICKY TRI UTAMA. Identification of DGAT1|EaeI Gene Polymorphism in Holstein Friesian Cattle Using PCR-RFLP Method. Supervised by CECE SUMANTRI and ANNEKE ANGGRAENI.
Diacylglycerol O-acyltransferase 1 (DGAT1) gene is one of major genes having an important role in milk fat synthesis. This research was aimed to identify DGAT1 gene polymorphism using EaeI enzyme by PCR-RFLP method. Identification was done in Holstein Friesian cattle from Balitnak (86 cows and 12 calves) and BIB Lembang (21 bulls). The length of PCR product was 411 bp. Genotyping resulted two types of alleles, namely K (411 bp) and A (203 and 208 bp); and three genotypes, namely KK (411 bp), KA (203, 208 and 411 bp) and AA (203 and 208 bp). For both locations, genotype frequency of KA (0.62) was higher than KK (0.35), while genotype frequency of the AA was very low (0.03). The allele frequency of K (0.66) was higher than A (0.34). Gene frequency of HF bulls at BIB Lembang was in Hardy-Weinberg equilibrium (x2 obs<x2 exp), but that of HF cows at Balitnak was disequilibrium. Heterozygosity of HF cattle at both locations was relatively high (Ho>He). DGAT1 gene of the observed HF cattle was polymorphic.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Peternakan
pada
Departemen Ilmu Produksi dan Teknologi Peternakan
IDENTIFIKASI KERAGAMAN GEN
DGAT1
|
Eae
I
PADA SAPI
FRIESIAN HOLSTEIN
DENGAN METODE PCR-RFLP
DICKY TRI UTAMA
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
INSTITUT PERTANIAN BOGOR BOGOR
Judul Skripsi : Identifikasi Keragaman Gen DGAT1|EaeI pada Sapi Friesian Holstein dengan Metode PCR-RFLP
Nama : Dicky Tri Utama NIM : D14090003
Disetujui oleh
Prof Dr Ir Cece Sumantri, MAgrSc Pembimbing I
Ir Anneke Anggraeni, MSi PhD Pembimbing II
Diketahui oleh
Prof Dr Ir Cece Sumantri, MAgrSc Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya, sehingga karya ilmiah ini berhasil diselesaikan. Penelitian yang dilaksanakan sejak bulan Mei sampai Juli 2013 ini, berjudul Identifikasi Keragaman Gen DGAT1|EaeI pada Sapi Friesian Holstein dengan Metode PCR-RFLP. Dana penelitian bersumber dari Balai Penelitian Ternak, Ciawi, Bogor.
Terima kasih penulis ucapkan kepada Bapak Prof Dr Ir Cece Sumantri, MAgrSc dan Ibu Ir Anneke Anggraeni, MSi PhD selaku pembimbing skripsi, serta Bapak Dr Ir Rudy Priyanto selaku pembimbing akademik. Ucapan terima kasih penulis sampaikan kepada Bapak Dr Ir Afton Attabany, MSi, Bapak Dr Iwan Prihantoro, SPt MSi serta Bapak Edit Lesa Aditya, SPt MSc selaku dosen penguji sidang. Penghargaan penulis sampaikan juga kepada LGMT members yang telah berkontribusi dalam penelitian ini. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, saudara, keluarga IPTP 46 dan seluruh kerabat atas segala doa dan dukungannya. Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 1
Ruang Lingkup Penelitian 2
METODE 2
Waktu dan Tempat penelitian 2
Bahan 2
Alat 3
Prosedur 3
HASIL DAN PEMBAHASAN 6
Amplifikasi Gen DGAT1 6
Keragaman Gen DGAT1 7
Frekuensi Genotipe dan Alel Gen DGAT1 9
Keseimbangan Distribusi Genotipe Gen DGAT1 10
Nilai Heterozigositas Gen DGAT1 11
SIMPULAN DAN SARAN 11
DAFTAR PUSTAKA 12
DAFTAR TABEL
1 Nilai frekuensi genotipe dan alel gen DGAT1 sapi FH pengamatan 9 2 Keseimbangan distribusi genotipe gen DGAT1 sapi FH pengamatan 11 3 Nilai heterozigositas pengamatan (Ho) dan heterozigositas harapan
(He) gen DGAT1 sapi FH pengamatan 11
DAFTAR GAMBAR
1 Visualisasi amplikon gen DGAT1 sapi FH pengamatan 6 2 Posisi penempelan primer forward dan reverse serta situs pemotongan
enzim EaeI 7
PENDAHULUAN
Latar Belakang
Produksi susu dalam negeri diketahui hanya mampu memenuhi sekitar 30% dari kebutuhan nasional, sisanya dipenuhi dari impor. Susu segar nasional pada umumnya dihasilkan oleh sapi perah Friesian Holstein (FH). Jumlah sapi perah nasional pada tahun 2012 diestimasikan sedikit, yaitu sebanyak 621 980 ekor (Ditjen PKH 2012). Sapi FH merupakan bangsa sapi perah yang memiliki kemampuan produksi susu lebih tinggi dibandingkan dengan bangsa sapi perah lainnya (Sudono 2003). Salah satu upaya untuk meningkatkan ketersediaan susu nasional adalah dengan meningkatkan populasi sapi FH.
Pertumbuhan ekonomi Indonesia yang semakin meningkat, mendorong sebagian masyarakat menerapkan pola hidup sehat dengan mengkonsumsi susu sapi sebagai sumber protein hewani dan kalsium. Masyarakat yang sudah berusia lanjut mulai menuntut kebutuhan susu sapi dengan kandungan asam lemak jenuh yang lebih rendah. Asam lemak jenuh dapat menjadi salah satu pemicu penyakit kardiovaskuler seperti penyakit jantung koroner dan stroke pada orang berusia lanjut (Micha dan Mozaffarian 2010). Seiring dengan peningkatan populasi sapi perah nasional, perbaikan kualitas susu dapat dilakukan melalui kegiatan perkawinan dan seleksi.
Seleksi akan memberikan perbaikan genetik secara permanen terhadap sifat yang diinginkan. Dengan berkembangnya teknologi molekuler saat ini, maka seleksi pada tingkat Deoxyribonucleic acid (DNA) dapat menjadi alternatif untuk memberikan respon seleksi yang lebih cepat dan akurat. Keragaman genetik dari gen-gen mayor yang memiliki pengaruh langsung terhadap sifat yang diperbaiki, dapat menjadi informasi dasar untuk melakukan kegiatan seleksi pada tingkat DNA. Teknik Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP) dapat digunakan untuk mengidentifikasi keragaman gen yang akan menjadi dasar pertimbangan pada program seleksi (Boichard et al. 2006).
Salah satu sifat yang perlu diperbaiki adalah kadar dan komposisi asam lemak susu. Enzim diacylglycerol O-acyltransferase 1 (DGAT1) berkontribusi dalam mengontrol sintesis trigliserida atau lemak susu di retikulum endoplasma sel kelenjar ambing (Winter et al. 2002). Enzim restriksi EaeI berperan dalam memotong basa-basa nukleotida pada situs mutasi K232A untuk membedakan genotipe dari gen DGAT1 (Kong et al. 2007). Keragaman genotipe berpengaruh terhadap kadar dan komposisi asam lemak susu. Oleh karena itu, identifikasi keragaman gen DGAT1 diperlukan sebagai informasi dasar untuk program seleksi sapi perah FH di dalam negeri.
Tujuan Penelitian
2
Ruang Lingkup Penelitian
Batasan penelitian mencakup metode identifikasi keragaman gen DGAT1 dengan kombinasi teknik PCR danRFLP yang menggunakan enzim restriksi EaeI pada sapi FH pengamatandi Balitnak dan BIB Lembang.
METODE
Waktu dan Tempat Penelitian
Penelitian berlangsung selama tiga bulan, dari Mei sampai Juli 2013. Pengambilan sampel darah sapi FH dilakukan di Balitnak, Ciawi dan BIB Lembang. Ekstraksi DNA, PCR-RFLP dan genotyping dilakukan di Laboratorium Genetika Molekuler Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor.
Bahan
Darah
Sampel darah berasal dari 119 ekor sapi FH, yang terdiri atas 86 ekor dara, 12 ekor pedet (Balitnak) dan 21 ekor pejantan (BIB Lembang).
Ekstraksi DNA
Ekstraksi DNA menggunakan NaCl 0.2%, ethylene diamide tetra acetate (EDTA), sodium diodecyl sulfat 10% (SDS), sodium tris EDTA (STE 1.0x), enzim proteinase k 5.0 mg/ml, fenol, CIAA (24 chloroform : 1 isoamyl alcohol), etanol absolut, etanol 70% dan tris EDTA 80% (TE).
PCR-RFLP
Analisis PCR menggunakan sampel DNA dan premix PCR. Premix PCR terdiri atas 9.35 µl air destilasi steril, 0.3 µl deoksi nukleotida triposfat (dNTP), 3.0 µl buffer (5.0x), 1.0 µl MgCl2, 0.05 µl enzim taq DNA polymerase dan 0.3 µl
primer. Primer yang digunakan mengacu pada penelitian Winter et al. (2002), yaitu forward 5’-GCACCATCCTCTTCCTCAAG-3’ dan reverse 5’-GGAAGCG CTTTCGGATG-3’. Analisis RFLP menggunakan produk PCR dan premix RFLP. Premix tersebut terdiri atas enzim restriksi EaeI yang mengacu pada penelitian Kong et al. (2007), buffer dan air destilasi steril.
Elektroforesis
3
Alat
Peralatan yang digunakan meliputi jarum vaccutainer 21 G, tabung0.2 ml, 0.5 ml dan 1.5 ml, satu set pipet mikro dan tipnya, pengaduk Vortex, mesin PCR, mesin sentrifugasi, refrigerator, freezer, inkubator, timbangan digital, gelas piala, gelas ukur, magnetic stirrer, microwave, cetakan gel, power supplyer, sarung tangan, masker, jas lab dan UV transilluminator yang berintegrasi dengan personal computer.
Prosedur
Pengambilan Sampel Darah
Sampel darah diambil dengan menggunakan jarum vaccutainer nomor 21 G melalui vena jugularis sapi perah FH.
Ekstraksi DNA
Isolasi DNA dilakukan dengan memodifikasi metode phenol-chloroform (Sambrook et al. 1989). Sebanyak 200 µl sampel darah dipindahkan ke dalam tabung 1.5 µl, kemudian ditambahkan NaCl 0.2% sebanyak 1 000 µl untuk menghilangkan alkohol. Sampel dihomogenkan dengan vortex mixer, lalu didiamkan selama lima menit. Sampel disentrifugasi pada kecepatan 8 000 rpm selama lima menit, lalu bagian supernatan dibuang. Sampel kemudian diberi larutan STE (1.0x) sebanyak 200 µl, 40 µl SDS 10% dan 10 µl proteinase k, lalu diinkubasi sambil digoyang pelan pada suhu 55 oC selama dua jam dengan alat pemutar (nutator).Selanjutnya, ditambahkan 400 µl larutan fenol, 40 µl NaCl 5.0 M dan 400 µl CIAA, lalu digoyang pada suhu ruang selama satu jam. Larutan hasil degradasi kemudian disentrifugasi pada kecepatan 12 000 rpm selama lima menit sampai fase DNA terpisah dengan fase fenol. DNA selanjutnya dipindahkan ke dalam tabung 1.5 µl yang baru sebanyak 40 µl, ditambahkan 40 µl NaCl 5.0 M dan 800 µl etanol absolut, lalu dihomogenkan dan disimpan pada suhu -20 oC selama semalam. Larutan kemudian disentrifugasi pada kecepatan 12 000 rpm selama lima menit sampai fase DNA terpisah dari fase etanol absolut. Supernatan kemudian dibuang dan tabung ditiriskan sampai tersisa endapan (pellet) DNA. Sebanyak 100 µl buffer TE 80% ditambahkan ke dalam tabung yang berisi DNA. Sampel DNA tersebut disimpan pada suhu -20 oC untuk digunakan pada tahap amplifikasi.
PCR-RFLP
Amplifikasi gen DGAT1 dengan metode PCR yang dimodifikasi dari Winter et al. (2002) dan Kong et al. (2007) menggunakan tabung 0.2 ml, yang di dalamnya terdiri atas 1.0 µl sampel DNA genom dan 14 µl premix. Premix terlebih dahulu dihomogenkan dengan pengaduk Vortex, lalu diendapkan dengan cara disentrifugasi pada kecepatan 5 000 rpm selama 10 detik. Premix kemudian didistribusikan ke setiap sampel. Sampel PCR kemudian disentrifugasi pada kecepatan 5 000 rpm selama 10 detik.
4
(annealing) pada suhu 60 oC selama 30 detik, pemanjangan (elongasi) pada suhu 72 oC selama 40 detik dan pemanjangan akhir pada suhu yang sama selama lima menit. Denaturasi, annealing dan elongasi dilakukan sebanyak 35 siklus.
Produk PCR selanjutnya dipindahkan ke dalam tabung khusus 0.5 ml untuk RFLP sebanyak 5.0 µl. Sebanyak 1.0 µl enzim restriksi EaeI, 0.7 µl buffer dan 0.3 µl air destilasi steril ditambahkan, kemudian campuran disentrifugasi pada kecepatan 5 000 rpm selama 10 detik. Campuran diinkubasi selama 16 jam pada suhu 37 oC.
Elektroforesis
Elektroforesis produk PCR dilakukan dengan menggunakan 5.0 µl produk PCR pada gel agarose 1.5%, sedangkan produk RFLP menggunakan gel agarose 2.0%. Gel dibuat dengan mencampurkan 0.45 g (1.5%) atau 0.60 g (2.0%) bubuk agarose dengan 20 ml atau 30 ml TBE (0.5x). Campuran kemudian dimasukkan ke dalam microwave selama tiga menit pada suhu medium. Campuran selanjutnya dihomogenkan dengan magnetic stirrer dan ditambahkan EtBr (Etidium bromide) sebanyak 2.5 µl. Larutan gel kemudian diangkat dan dituang ke dalam cetakan gel. Gel selanjutnya dimasukkan ke dalam alat elektroforesis. Larutan TBE (0.5x) dituang ke alat elektroforesis sampai gel tenggelam. Sebanyak 5.0 µl produk PCR dan marker 100 pb, masing-masing dicampur dengan loading dye sebanyak 1.0 µl. Campuran selanjutnya dimasukkan ke dalam sumur-sumur pada gel agarose dan dielektroforesis pada tegangan 100 volt selama 45 menit. Hasil elektroforesis kemudian divisualisasikan dengan mesin UV transilluminator. Setiap sampel dengan pita DNA yang muncul, kemudian dibandingkan dengan marker agar diketahui panjang fragmennya (Winarno dan Agustinah 2007).
Analisis Data
Frekuensi Alel dan Genotipe
Frekuensi genotipe merupakan rasio dari jumlah suatu genotipe terhadap jumlah sampel. Genotipe pada masing-masing ternak dapat ditentukan dari pita-pita DNA yang terlihat. Frekuensi genotipe dapat diketahui dengan menghitung perbandingan jumlah genotipe tertentu pada setiap lokasi. Rumus menghitung frekuensi genotipe menurut Nei dan Kumar (2000) adalah:
Xii = nii
N
Frekuensi alel merupakan frekuensi relatif dari suatu alel dalam suatu lokasi pengambilan sampel atau jumlah suatu alel terhadap jumlah total alel. Frekuensi alel (Xi) gen DGAT1 dapat dihitung dengan rumus menurut Nei dan
Kumar (2000) sebagai berikut:
5
nii = jumlah individu bergenotipe ii
nij = jumlah individu bergenotipe ij
N = jumlah individu sampel
Keseimbangan Hardy-Weinberg
Keseimbangan Hardy-Weinberg diuji dengan rumus menurut Hartl dan Clark (1997) sebagai berikut:
O = jumlah pengamatan genotipe ke-i E = jumlah harapan genotipe ke-i
Derajat bebas (db) yang digunakan merupakan hasil pengurangan jumlah genotipe dengan jumlah alel dan dapat dihitung dengan rumus menurut (Allendrof dan Luikart 2007) sebagai berikut:
db x2 = (jumlah genotipe-1)-(alel-1)
Heterozigositas
Keragaman genetik dapat diketahui dengan mengestimasi frekuensi heterozigositas pengamatan (Ho) yang diperoleh dari setiap lokasi menurut Weir (1996) sebagai berikut:
��=���� �
�=�
Heterozigositas harapan (He) berdasarkan frekuensi alel dapat dihitung dengan menggunakan rumus menurut Nei dan Kumar (2000) sebagai berikut:
��= 1− � ��2
�
�=1
Keterangan :
Ho = nilai heterozigositas pengamatan
nij = jumlah individu heterozigot
N = jumlah individu yang diamati He = nilai heterozigositas harapan
xi = frekuensi alel
6
HASIL DAN PEMBAHASAN
Amplifikasi Gen DGAT1
Amplifikasi gen DGAT1 berhasil dilakukan pada seluruh sampel (100%) sapi FH pengamatan dari Balitnak dan BIB Lembang. Amplifikasi mengacu pada penelitian Winter et al. (2002). Gen tersebut terletak di kromosom autosom ke-14 dengan panjang total 14 117 pb (pasang basa), terdiri atas dua flanking region, 17 ekson dan 16 intron. Produk amplifikasi atau amplikon yang diperoleh, memiliki panjang 411 pb. Amplikon tersebut terletak pada posisi basa ke-10 232 sampai basa ke-10 644. Visualisasi hasil elektroforesis amplikon dengan menggunakan gel agarose 1.5% dapat dilihat pada Gambar 1.
Keterangan: Huruf M = Marker; Nomor 1-7 = amplikon gen DGAT1
Tingkat keberhasilan amplifikasi menggunakan metode PCR dapat diketahui dengan melihat tebal dan terangya pita amplikon. Hal tersebut dipengaruhi oleh konsentrasi dan kemurnian DNA genom hasil ekstraksi, aktivitas taq polymerase, dNTP, Mg2+, buffer, primer dan kesesuaian suhu pada setiap tahap PCR, sehingga EtBr akan menempel pada DNA yang teramplifikasi dan berpendar ketika terkena sinar ultraviolet (UV) (Yuwono 2006). Hasil penelitian menunjukkan bahwa penggunaan bahan-bahan yang sesuai dengan rumusan premix, terjaga suhu penyimpanannya, terhindar dari kontaminasi oleh mikroorganisme dan sesuai dengan pengaturan suhu, waktu dan siklus PCR dapat menghasilkan amplikon dengan kualitas terbaik.
Muladno (2010) menyatakan bahwa tahap perbanyakan (amplifikasi) fragmen DNA di dalam mesin PCR terdiri atas pemisahan untai DNA (denaturasi), penempelan primer (annealing) dan tahap pemanjangan primer (elongasi) dengan suhu dan waktu yang berbeda pada masing-masing tahap, sesuai dengan primer yang digunakan. Primer akan berhasil menempel pada situs target pada suhu dan waktu tertentu, sehingga menghasilkan fragmen amplikon dengan ukuran yang lebih pendek dibandingkan dengan fragmen DNA genom. Suhu annealing yang umum digunakan bervariasi, mulai dari 55 sampai 65 oC.
7 Semakin panjang sekuens primer, maka suhu annealing semakin rendah. Penelitian ini menggunakan suhu 60 oC selama 30 detik untuk masing-masing primer dengan panjang 20 pb (forward) dan 17 pb (reverse), yang dimodifikasi dari penelitian sebelumnya oleh Winter et al. (2002).
Keragaman Gen DGAT1
Enzim restriksi EaeI berhasil memotong situs mutasi gen DGAT1. Posisi mutasi menurut studi Winter et al. (2002), yakni pada ekson 8, posisi basa ke-10 433 yang menyebabkan perubahan adenin menjadi guanin (A→G) atau mutasi transisi dan basa ke-10 434 yang menyebabkan perubahan adenin menjadi sitosin (A→C) atau mutasi transversi. Mutasi tersebut mengakibatkan perubahan asam amino dari lisin (K=AAG) menjadi alanin (A= GCG) atau terjadi perubahan bermakna. Enzim restriksi EaeI menurut Kong et al. (2007) memotong situs mutasi GC*GGCC pada posisi basa ke-10 434. Posisi penempelan primer dan pemotongan oleh enzim EaeI dapat dilihat pada Gambar 2.
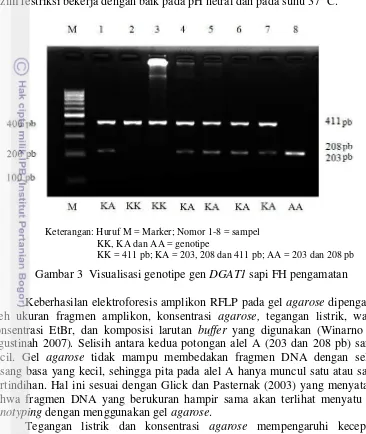
Dua tipe alel gen DGAT1 berhasil diidentifikasi dengan menggunakan enzim restriksi EaeI dan memunculkan tiga tipe genotipe. Dua alel tersebut adalah K dan A. Alel K tidak memiliki situs mutasi GC*GGCC, sehingga tidak terpotong oleh enzim. Panjang fragmennya sama dengan amplikon, yakni 411 pb. Fragmen alel A terpotong oleh enzim karena memiliki situs mutasi GC*GGCC, sehingga terbagi menjadi dua fragmen. Panjang masing-masing fragmennya, yakni 203 dan 208 pb. Tiga genotipe yang dihasilkan meliputi KK (411 pb), KA (203, 208 dan 411 pb) dan AA (203 dan 208 pb). Hal ini sesuai dengan penelitian Winter et al. (2002). Setiap pasangan kromosom homolog memiliki alel masing-masing. Mutasi dapat terjadi baik pada salah satu kromosom (KA) maupun pada keduanya (AA). Visualisasi hasil elektroforesis amplikon yang telah diinkubasi dengan enzim EaeI pada gel agarose 2% dapat dilihat pada Gambar 3.
Keberhasilan RFLP dipengaruhi oleh aktivitas enzim. Pada penelitian ini, aktivitas enzim dipengaruhi oleh kemurnian DNA, buffer, suhu penyimpanan, serta waktu dan suhu inkubasi. Davis et al. (1986) menyatakan bahwa DNA yang Sumber: Winter et al. (2002)
8
terpotong oleh enzim restriksi umumnya bebas dari bahan pengotor. Penggunaan buffer dan prosedur RFLP dalam penelitian ini sesuai dengan protokol standar, yakni diinkubasi pada suhu 37 oC selama 16 jam. Buffer berperan dalam stabilisasi pH medium. Pingoud et al. (1993) menyatakan bahwa hampir semua enzim restriksi bekerja dengan baik pada pH netral dan pada suhu 37 oC.
Keterangan: Huruf M = Marker; Nomor 1-8 = sampel KK, KA dan AA = genotipe
KK = 411 pb; KA = 203, 208 dan 411 pb; AA = 203 dan 208 pb
Keberhasilan elektroforesis amplikon RFLP pada gel agarose dipengaruhi oleh ukuran fragmen amplikon, konsentrasi agarose, tegangan listrik, waktu, konsentrasi EtBr, dan komposisi larutan buffer yang digunakan (Winarno dan Agustinah 2007). Selisih antara kedua potongan alel A (203 dan 208 pb) sangat kecil. Gel agarose tidak mampu membedakan fragmen DNA dengan selisih pasang basa yang kecil, sehingga pita pada alel A hanya muncul satu atau saling bertindihan. Hal ini sesuai dengan Glick dan Pasternak (2003) yang menyatakan bahwa fragmen DNA yang berukuran hampir sama akan terlihat menyatu saat genotyping dengan menggunakan gel agarose.
Tegangan listrik dan konsentrasi agarose mempengaruhi kecepatan fragmen DNA untuk bermigrasi dari kutub negatif ke kutub positif. Penggunaan tegangan 100 volt pada gel agarose 2% menyebabkan waktu elektroforesis yang dibutuhkan sekitar 45 menit. Semakin tinggi konsentrasi agarose, maka semakin lama pula waktu yang dibutuhkan untuk elektroforesis. Penggunaan agarose 2% membutuhkan EtBr sebanyak 2.5 µl untuk menghasilkan visualisasi genotipe yang baik. EtBr yang melekat pada fragmen DNA akan berpendar ketika terkena sinar UV, sehingga pita DNA akan terlihat saat genotyping. Larutan buffer yang sesuai sebagai media penghantar listrik adalah TBE (0.5x) pH netral.
9
Frekuensi Gen DGAT1
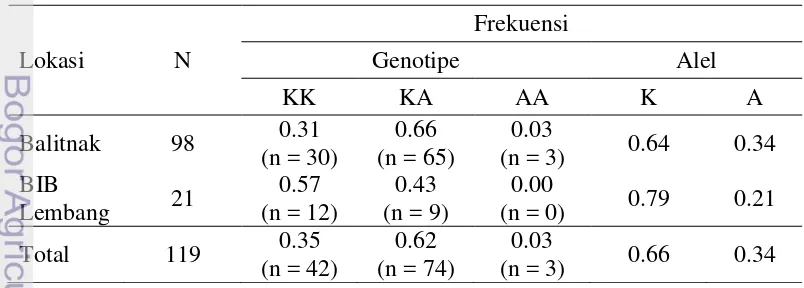
Keragaman gen pada suatu populasi dapat ditentukan dengan menghitung nilai frekuensi alel dan genotipe. Nei dan Kumar (2000) menyatakan bahwa suatu populasi dikatakan beragam jika memiliki dua atau lebih alel dalam satu lokus dengan frekuensi kurang dari satu (100%). Hasil genotyping pada total sampel sapi FH pengamatan menunjukkan bahwa frekuensi genotipe KA paling tinggi, diikuti dengan KK, sedangkan frekuensi genotipe AA sangat rendah. Pola yang sama terjadi pada sampel pengamatan dari Balitnak dengan frekuensi genotipe KA tertinggi, namun frekuensi genotipe KK tertinggi pada sampel dari BIB Lembang. Jumlah sampel dari Balitnak lebih banyak dibandingkan dengan BIB Lembang. Oleh karena itu, frekuensi alel K lebih tinggi dibandingkan dengan frekuensi alel A di kedua lokasi. Gen DGAT1 pada kedua lokasi bersifat beragam (polimorfik) karena tidak ditemukan frekuensi alel yang bernilai satu (100%). Nilai frekuensi genotipe dan alel gen DGAT1 sapi FH pengamatan disajikan pada Tabel 1.
Variasi genotipe muncul akibat adanya seleksi, mutasi, migrasi dan sistem perkawinan silang dalam. Genotipe terbentuk dari kombinasi kromosom homolog yang membawa satu macam alel. Kombinasi tersebut terjadi pada saat fertilisasi atau bertemunya sel gamet jantan dan betina (Hardjosubroto 2001). Tabel 1 menunjukkan bahwa alel K lebih banyak dimiliki oleh sapi FH pejantan di BIB Lembang. Sapi FH di Balitnak tidak terlepas dari program pemuliaan yang menggunakan materi genetik dari pejantan di BIB Lembang. Oleh karena itu, frekuensi alel K juga tinggi pada sapi FH Balitnak. Sapi FH pejantan di BIB Lembang juga turut menyumbangkan alel A yang berasal dari individu heterozigot (KA) kepada sapi FH di Balitnak. Frekuensi genotipe (KA) yang tinggi pada sapi FH di Balitnak dapat disebabkan oleh frekuensi penggunaan semen dari pejantan heterozigot (KA) dari BIB Lembang yang tinggi. Noor (2010) menyatakan bahwa setiap tetua dari makhluk tingkat tinggi mewariskan informasi genetik ke generasi berikutnya melalui perkawinan.
Tabel 1 Nilai frekuensi genotipe dan alel gen DGAT1 sapi FH pengamatan
Lokasi N Keterangan: N = jumlah sampel; (n =) = jumlah sampel per genotipe
10
penelitian. Komisarek et al. (2011) melaporkan bahwa frekuensi genotipe sapi perah Jersey hampir sama dengan sapi perah BIB Lembang, yaitu KK (0.62) tertinggi, diikuti dengan KA (0.33) dan AA (0.05) yang terendah dengan frekuensi alel K (0.79) lebih tinggi dibandingkan dengan frekuensi alel A (0.21).Pareek et al. (2005) melaporkan bahwa frekuensi genotipe sapi perah Polish Black-White hampir sama dengan sapi perah Balitnak, yaitu KK (0.31), KA (0.55) dan AA (0.13) dengan frekuensi alel K (0.59) lebih tinggi dibandingkan dengan frekuensi alel A (0.41). Sebaliknya, Schennink et al. (2008) melaporkan bahwa frekuensi alel A pada populasi sapi Dutch Holstein lebih tinggi (0.60) dibandingkan dengan frekuensi alel K (0.40). Sama halnya dengan Conte et al. (2010) yang melaporkan bahwa frekuensi alel A pada populasi sapi Italian Brown lebih tinggi (0.98) dibandingkan dengan frekuensi alel K (0.02). Hal ini dapat disebabkan oleh proses seleksi berdasarkan gen DGAT1 yang sudah mulai terjadi pada sapi Dutch Holstein dan Italian Brown. Bourdon (1999) menyatakan bahwa seleksi terhadap sifat tertentu akan mengakibatkan tingginya frekuensi dari sifat tersebut pada suatu populasi.
Keragaman gen DGAT1 dapat dijadikan sebagai informasi acuan untuk program seleksi sapi perah karena gen tersebut berperan dalam mengatur kadar dan komposisi asam lemak susu. Duchemin et al. (2013) dan Schennink et al. (2008) melaporkan bahwa alel A dari gen DGAT1 berasosiasi negatif terhadap asam lemak rantai pendek (kurang dari 18 karbon) dan asam lemak jenuh, namun berasosiasi positif terhadap asam lemak C14:0, C18 tak jenuh, conjugated linoleic acid (CLA) dan asam lemak tak jenuh lainnya. Thaller et al. (2003) dan Komisarek et al. (2011) melaporkan bahwa alel K berasosiasi positif terhadap asam lemak jenuh rantai pendek. Dengan kata lain, sapi perah bergenotipe AA menghasilkan susu dengan asam lemak jenuh lebih sedikit dibandingkan dengan sapi perah bergenotipe KK. Tabel 1 menunjukkan bahwa ternak bergenotipe AA terbilang sangat sedikit. Perkawinan antara sapi FH pejantan bergenotipe KA di BIB Lembang dengan sapi FH betina bergenotipe KA dan AA dapat menghasilkan keturunan bergenotipe AA dengan peluang masing-masing sebesar 25% dan 50%.
Keseimbangan Distribusi Genotipe Gen DGAT1
11
Tabel 2 Keseimbangan distribusi genotipe gen DGAT1 sapi FH pengamatan
Lokasi N x2 hitung
Balitnak 98 18.57**
BIB Lembang 21 1.56tn
Keterangan: N = jumlah sampel ** = sangat berbeda nyata tn = tidak berbeda nyata x20.05 = 3.84 dan x20.01 = 6.64
Nilai Heterozigositas Gen DGAT1
Tingkat keragaman gen DGAT1 pada kedua lokasi tidak sama berdasarkan analisis nilai heterozigositas. Hasil analisis disajikan pada Tabel 3. Nilai Ho pada populasi sapi FH di Balitnak (0.66) lebih besar dibandingkan dengan populasi sapi FH di BIB Lembang (0.43), namun keduanya menunjukkan nilai yang lebih tinggi dibandingkan nilai He pada masing-masing populasi. Nilai Ho yang tinggi (Ho>0.50) menunjukkan keragaman yang tinggi pada suatu populasi (Javanmard et al. 2005). Nilai Ho yang lebih tinggi dibandingkan dengan nilai He pada suatu populasi menunjukkan bahwa nilai respon seleksi pada lokasi tersebut relatif tinggi (Machado et al. 2003). Dengan kata lain, seleksi akan efektif baik di Balitnak maupun di BIB Lembang.
Tabel 3 Nilai heterozigositas pengamatan (Ho) dan heterozigositas harapan (He) gen DGAT1 sapi FH pengamatan
Lokasi N Ho He
Balitnak 98 0.66 0.46
BIB Lembang 21 0.43 0.34
Total 119 0.62 0.45
SIMPULAN DAN SARAN
Simpulan
12
Saran
Gen DGAT1 dapat menjadi salah satu gen mayor yang dipertimbangkan dalam program seleksi. Penambahan jumlah sampel serta studi asosiasi genotipe terhadap komposisi asam lemak susu perlu dilakukan untuk memberikan informasi awal dalam program seleksi.
DAFTAR PUSTAKA
Allendrof FW, Luikart G. 2007. Conversation and The Genetics of Populations. Oxford (UK): Blackwell Publishing.
Boichard D, Fritz S, Rossignol MN, Guillaume, Colleau JJ, Druet T. 2006. Implementation of marker-assisted selection: practical lessons from dairy cattle. Di dalam: Proceedings of The 8th World Congress on Genetic Applied to Livestock Production [Internet]. 2006 Agustus 11-16. Belo Horizonte (BR): ORBi. [diunduh 2013 Mei 8]. Tersedia pada: http://hdl.handle.net/2268/84556
Bourdon RM. 1999. Understanding Animal Breeding. Ed ke-2. New Jersey (US): Prentice Hall.
Conte G, Mele M, Chessa S, Castiglioni B, Serra A, Pagnacco G, Secchiari P. 2010. Diacylglycerol acyltransferase 1, stearoyl-CoA desaturase 1, and sterol regulatory element binding protein 1 gene polymorphisms and milk fatty acid composition in Italian Brown cattle. J. Dairy Sci. 93: 753-763. Davis LG, Dibner MD, Battey JF. 1986. Basic Methods in Molecular Biology.
New York (US): Elsevier.
[Ditjen PKH] Direktorat Jenderal Peternakan dan Kesehatan Hewan. 2012. Statistik Peternakan dan Kesehatan Hewan 2012. Jakarta (ID): Kementerian Pertanian RI.
Duchemin S, Bovenhuis H, Stoop WM, Bouwman AC, van Arendonk JAM, Visker MHPW. 2013. Genetic correlation between composition of bovine milk fat in winter and summer, and DGAT1 and SCD1 by season interactions. J. Dairy Sci. 96: 592-604.
Glick BR, Pasternak JJ. 2003. Molecular Biotechnology: Principles and Applications of Recombinant DNA. Washington DC (US): ASM Press. Hardjosubroto W. 2001. Genetika Hewan. Yogyakarta (ID): Gadjah Mada
University Press.
Hartl DL, Clark AG. 1997. Principle of Population Genetics. Sunderland (UK): Sinaver Associates.
Javanmard A, Asadazadeh N, Banahazi MH, Tavakolian J. 2005. The allele and genotype frequencies of bovine pituitary specific transcription factor and leptin genes in Iranian cattle and buffalo populations using PCR-RFLP. Iranian J. Biotech. 3: 104-108.
13 Kong HS, Oh JD, Lee JD, Yoon DH, Choi YH, Cho BW, Lee HK, Jeon GJ. 2007. Association of sequence variations in DGAT1 gene with economic traits in Hanwoo (Korea cattle). Asian-Aust. J. Anim. Sci. 20 (6): 817-820.
Machado MA, Schuster I, Martinez ML, Campos AL. 2003. Genetic diversity of four breed using microsatellite markers. Rev. Bras. De Zool. 32: 93-98. Micha R, Mozaffarian D. 2010. Saturated fat and cardiometabolic risk factors,
coronary heart disease, stroke and diabetes: a fresh look at the evidence. Lipids. 45: 893-905.
Muladno. 2010. Teknologi Rekayasa Genetika. Ed ke-2. Bogor (ID): IPB Press. Nei M, Kumar S. 2000. Molecular Evolution and Phylogenetics. Oxford (UK):
Oxford University Press.
Noor RR. 2010. Genetika Ternak. Ed ke-6. Jakarta (ID): Penebar Swadaya. Pareek CS, Czarnik U, Zabolewicz, Pareek RS, Walawski K. 2005. DGAT1
K232A quantitative trait nucleotide polymorphism in Polish Black-White cattle. J. Appl. Genet. 46 (1): 85-87.
Pingoud A, Alves J, Geiger R. 1993. Restriction enzymes. Di dalam: Burrel MM, editor. Methods in Molecular Biology. Volume ke-16. New Jersey (US): Humana Press.
Sambrook J, Fritsch EF, Maniatis T. 1989. Molecular Cloning: A Laboratory Manual. New York (ID): Cold Spring Harbor Laboratory Press.
Schennink A, Heck JML, Bovenhuis H, Visker MHPW, Van Valenberg HJF, Van Arendonk JAM. 2008. Milk fatty acid unsaturation: Genetic parameters and effects of stearoyl-CoA desaturase (SCD1) and acyl CoA: diacylglycerol acyltransferase 1 (DGAT1). J. Dairy Sci. 91: 2135-2143. Sudono A, Rosdiana RF, Setyawan BS. 2003. Beternak Sapi Perah secara
Intensif. Jakarta (ID): Agromedia Pustaka.
Thaller G, Kramer W, Winter A, Kaupe B, Erhardt G, Fries R. 2003. Effects of DGAT1 variants on milk production traits in German cattle breeds. J. Anim. Sci. 81: 1911-1918.
Weir BS. 1996. Genetic Data Analysis: Method for Discrete Population Genetics Data. Ed ke-2. Sunderland (UK): Sinauer Associates.
Winarno, Agustinah W. 2007. Pengantar Bioteknologi. Bogor (ID): M-Brio Biotekindo Press.
Winter A, Kramer W, Werner FAO, Kollers S, Kata S, Durstewitz G, Buitkamp J, Womack JE, Thaller G, Fries R. 2002. Association of a lysine-232/alanine polymorphism in a bovine gene encoding acyl-CoA: diacylglycerol acyltransferase (DGAT1) with variation at a quantitative trait locus for milk fat content. Di dalam: Womack JE, editor. Proceedings of The National Academy of Sciences of The United States of America 99 [Internet]. [Waktu dan tempat pertemuan tidak diketahui]. Texas (US): PNAS. hlm 9300-9305; [diunduh 2013 Mei 10]. Tersedia pada: http://www.pnas.org/cgi/doi/10.1073/pnas.142293799
14
RIWAYAT HIDUP
Penulis dilahirkan pada tanggal 1 Desember 1991 di Tebing Tinggi, Sumatera Utara. Penulis adalah anak ketiga dari tiga bersaudara dari pasangan Bapak Ir Bambang Setyono dan Ibu Sumijah. Penulis menyelesaikan pendidikan dasar pada tahun 2003 di SD Kartika I-2 Medan. Pendidikan menengah pertama diselesaikan pada tahun 2006 di SMPN 7 Medan. Pendidikan menengah atas diselesaikan pada tahun 2009 di SMA Kartika I-2 Medan. Penulis diterima sebagai mahasiswa Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan, Institut Pertanian Bogor melalui jalur Undangan Seleksi Masuk IPB (USMI) pada tahun 2009.
Selama mengikuti perkuliahan, penulis aktif di berbagai kegiatan kema-hasiswaan seperti pernah menjadi pengurus Badan Eksekutif Mahasiswa Tingkat Persiapan Bersama (BEM TPB) periode 2009-2010 dan Keluarga Mahasiswa (BEM KM) periode 2010-2011 serta Lembaga Dakwah Fakultas FAMM Al Anaam periode 2011-2012. Penulis juga aktif dalam kegiatan keilmiahan mahasiswa seperti Program Kreativitas Mahasiswa Bidang Penelitian (PKM-P) tahun 2012.