RINGKASAN
SEPTYANINGTYAS ANGGIA SARI. D14080028. 2012. Identifikasi Keragaman Gen Follicle Stimulating Hormone Receptor (FSHR|AluI) pada Spesies Sapi Bos javanicus, Bos taurus, dan Bos indicus dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan. Fakultas Peternakan. Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir.Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Dr. Jakaria, S.Pt,M.Si.
Sapi dari spesies Bos javanicus, Bos taurus,dan Bos indicus merupakan sapi-sapi yang tersebar diseluruh wilayah Indonesia. Sumber daya genetik ternak yang harus dipertahankan agar mencegah terjadinya kepunahan menjadi alasan untuk mengkaji keragaman gen FSHR yang berperan dalam fungsi reproduksi. Gen FSHR (Follicle Stimulating Hormone Receptor) akan menghasilkan produk yaitu FSHR yang diperlukan oleh hormone FSH untuk mencapai sel target sehingga dapat melaksanakan fungsi reproduksi (menghasilkan sperma dan ovum). Penelitian ini bertujuan untuk mengetahui keragaman gen Follicle Stimulating Hormone Receptor (FSHR|AluI) dengan menggunakan metode PCR-RFLP pada spesies sapi Bos javanicus, Bos taurus, dan Bos indicus.
Sampel yang digunakan berjumlah 207 sampel yaitu terdiri dari sampel spesies sapi Bos javanicus 59 sampel, spesies Bos taurus 137 sampel, dan spesies Bos indicus 11 sampel. Sampel spesies sapi Bos javanicus berasal dari sapi Bali, sampel sapi Bos taurus terdiri dari sapi Limousin (57 sampel), Simmental (50 sampel), Angus (6 sampel), dan FH (24 sampel), serta sampel spesies sapi Bos indicus terdiri dari sampel Brahman (9 sampel) dan sapi Ongole (2 sampel). Sampel sapi yang digunakan berasal dari BIB Lembang, BIB Singosari, BPTU Bali, BIBD Bali, dan BET Cipelang. Penelitian ini menggunakan metode PCR (Polymerase Chain Reaction) dan RFLP (Restriction Fragment Length Polymorphism) untuk mendeteksi adanya keragaman genetic pada gen FSHR menggunakan enzim rektriksi AluI yang memotong basa AG|CT. Penelitian ini dilakukan di Laboratorium Genetika Molekuler Ternak sejak bulan Oktober 2011 hingga Februari 2012. Keragaman gen FSHR ditentukan dengan analisis frekuensi genotipe, frekuensi alel, nilai heterozigositas pengamatan (Ho), dan
nilai heterozigositas harapan (He).
Amplifikasi gen FSHR menghasilkan fragmen dengan panjang 306 bp yang terletak pada ekson 10 dengan suhu annealing 60oC selama 45 detik. Genotyping gen FSHR|AluI menghasilkan dua jenis alel yaitu alel C dan alel G serta menghasilkan tiga jenis genotipe yaitu CC (243 dan 63 bp), CG (243, 293, 50, 63 bp), dan GG (193, 50, dan 63 bp). Frekuensi genotipe GG tertinggi pada spesies sapi Bos javanicus yaitu 1,00, Bos indicus sebesar 0,46 dan terendah pada spesies sapi Bos taurus yaitu 0,11. Frekuensi genotype CC paling tinggi pada spesies sapi Bos taurus yaitu 0,57, Bos indicus sebesar 0,18, dan terendah pada spesies sapi Bos javanicus. Nilai heterozigositas Ho dan He paling tinggi pada spesies sapi Bos indicus sedangkan nilai
terendah pada spesies sapi Bos javanicus.
ABSTRACT
Identification Polymorphism of The Follicle Stimulating Hormone Receptor (FSHR|AluI) Gene in Species Bos javanicus, Bos taurus, and Bos indicus
Using PCR-RFLP Method Sari, S.A., C. Sumantri, and Jakaria
Follicle Stimulating Hormone Receptor (FSHR) gene is a very important in reproduction trait. FSHR is required by Follicle Stimulating Hormone (FSH) to carry out on target tissues. The purpose of this study was to identify polymorphism of the Follicle Stimulating Hormone Receptor gene in Bos javanicus, Bos taurus, and Bos indicus Cattle. Blood samples used 207 samples consisting of Bos javanicus (Bali Cattle) 59 heads, Bos taurus 137 heads (Limousin 57 heads, Simmental 50 heads, Angus 6 heads, and Holstein Friesian (FH) 24 heads), and Bos indicus 11 heads (Brahman 9 heads and Ongole 2 heads). The polymorphism site analysis from digestion with AluI restriction enzyme allowed the genotype identification. AluI restriction enzyme cut the base position at AG|CT in 193 and 243 bp. Genotyping FSHR gene produced two alleles, namely C and G alleles. These allele produced three genotypes respectively, there are CC (243 and 63 bp), GG (193, 63, and 50 bp), and CG (243, 193, 63, and 50 bp). The highest G allele frequency was found in Bos javanicus (Bali cattle), but the lowest in Bos taurus. The highest C allele frecuency was found in Bos taurus. The value of heterozigosity was the highest in Bos indicus and the lowest in Bos javanicus. The result of this study showed that the allele of FSHR gene in Bos taurus and Bos indicus were polymorphic, while in Bos javanicus was monomorphic.
PENDAHULUAN Latar Belakang
Sapi-sapi yang tersebar luas di Indonesia terbagi menjadi tiga kriteria yaitu
sapi asli Indonesia, sapi impor, dan sapi yang sudah beradaptasi (Sarbaini, 2004).
Secara garis besar, spesies sapi yang tersebar di Indonesia terdiri dari spesies sapi
Bos taurus yang merupakan sapi asal Eropa dan Afrika Barat, spesies Bos indicus
yang berasal dari wilayah Asia dan Afrika Selatan, serta Bos javanicus yang
merupakan spesies asli Indonesia yang berasal dari domestikasi Banteng
(Rahmatullah, 2011). Spesies Bos taurus dan Bos indicus merupakan spesies sapi
yang di impor ke Indonesia dan beberapa diantaranya telah dapat beradaptasi dengan
lingkungan di Indonesia seperti sapi FH. Sapi impor tersebut kemudian disebar
keseluruh wilayah Indonesia untuk disilangkan dengan sapi lokal Indonesia dan
membentuk bangsa sapi baru seperti sapi Peranakan Ongole.
Kebutuhan akan pangan dan produksi dari ternak sapi di Indonesia menjadi
perlu untuk mempertahankan secara optimal sumber daya genetik ternak terutama
ternak asli Indonesia. Departemen Pertanian (2006) menyatakan jika sumber daya
genetik ternak merupakan substansi ternak yang secara genetik unik, terbentuk dari
proses domestikasi yang memiliki nilai potensial serta dapat dimanfaatkan dan
dikembangkan dengan baik untuk menciptakan rumpun atau galur unggul. Sapi Bali
merupakan salah satu sumberdaya genetik ternak asli Indonesia yang menjadi plasma
nutfah nasional. Keberadaan sumberdaya genetik ternak perlu dipertahankan dan
dimanfaatkan. Oleh karena itu, perlu adanya upaya peningkatan mutu genetik ternak
Upaya yang dapat dilakukan untuk meningkatkan mutu genetik ternak sapi
adalah melalui seleksi dan persilangan. Seleksi dapat dilakukan berdasarkan sifat
reproduksi dari masing-masing ternak. Pemanfaatan penerapan teknologi molekuler
dapat digunakan sebagai metode seleksi pada tingkat DNA, dalam hal ini DNA yang
berperan dalam sifat reproduksi. Salah satu gen yang memiliki peranan penting
dalam reproduksi adalah gen FSHR (Follicle Stimulating Hormone Receptor). Gen
ini sangat penting dalam proses pembentukan sperma pada sapi jantan dan ovum
pada sapi betina. Gen ini terletak pada kromosom ke 11 yang terdiri dari 10 ekson
dan 9 intron (Aguirre dan Timossi, 1998). Teknik yang dapat digunakan untuk
2 Chain Reaction) dengan RFLP (Restriction Fragment Length Polymorphism).
Metode RFLP menggunakan enzim pemotong AluI untuk mengidentifikasi
polymorfisme gen FSHR pada ekson 10 dengan menggunakan primer forward dan
reverse berdasarkan Houde et al. (1994). Apabila polymorfisme yang terjadi pada
gen FSHR berhubungan dengan sifat produksi dan reproduksi sapi, maka dapat
dijadikan sebagai Marker Assisted Selection (MAS). Untuk dapat memenuhi kriteria
seleksi MAS maka perlu terlebih dahulu dilakukan identifikasi keragaman gen.
Melalui seleksi MAS diharapkan ketepatan seleksi dapat lebih baik sejak periode
anakan.
Tujuan
Penelitian ini bertujuan untuk mengetahui keragaman gen Follicle
Stimulating Hormone Receptor (FSHR|Alu I) dengan menggunakan metode
TINJAUAN PUSTAKA Asal Usul Sapi di Indonesia
MacHugh (1996) menyatakan jika terdapat dua spesies sapi yang tersebar
diseluruh dunia yaitu spesies tidak berpunuk dari Eropa, Afrika Barat, dan Asia
Utara yang disebut dengan spesies Bos taurus, serta spesies sapi berpunuk dari Asia
Selatan dan Afrika yang disebut dengan Bos indicus. Perbedaan antara keduanya
terletak dari ada/tidaknya punuk, cara beradaptasi (Bos indicus lebih mendiami
daerah kering dan panas, sedangkan Bos taurus di daerah beriklim sedang dengan
curah hujan cukup).
Sapi yang terdapat di Indonesia terbagi menjadi : 1) sapi asli Indonesia, 2)
sapi impor, dan 3) sapi yang telah beradaptasi (Sarbaini, 2004). Kusdiantoro et al.
(2007) menyatakan pejantan sapi Ongole di-impor ke Indonesia pada akhir abad
ke-19 ke pulau Jawa dan beberapa pulau lainnya di Indonesia kecuali pulau Bali dan
Madura. Utoyo (2002) menjelaskan penyebaran sapi Ongole pada abad ke-20
dimaksudkan agar dapat disilangkan dengan sapi asli jawa yang akhirnya
membentuk bangsa sapi peranakan ongole dan sapi Madura. Sapi domestikasi yang
ada di Indonesia merupakan sapi spesies bibos dan sapi silangan yang berbeda
dengan sapi domestikasi di Afrika dan Eropa (Jakaria, 2008).
Menurut Rollinson (1984) sapi Bali (Bibos sondaicus) berasal dari hasil
domestikasi banteng liar (Bibos banteng). Proses domestikasi sapi Bali itu terjadi
sebelum 3.500 SM di Indonesia atau Indochina. Banteng liar saat ini bisa ditemukan
di Jawa bagian Barat dan bagian Timur, di Pulau Kalimantan, serta ditemukan juga
di Malaysia (Payne dan Rollinson 1973). Penyebaran sapi Bali di Indonesia dimulai
pada tahun 1890 dengan adanya pengiriman ke Sulawesi, pengiriman selanjutnya
dilakukan pada tahun 1920 dan 1927. Pada tahun 1964 di Bali terjadi musibah
penyakit jembrana secara besar-besaran yang menyebabkan sapi Bali tidak boleh
dikeluarkan lagi dari pulau Bali sebagai ternak bibit. Mulai periode inilah sumber
bibit sapi Bali bagi daerah lain di Indonesia digantikan oleh NTT, Sulawesi Selatan
dan NTB (Talib, 2002).
Bangsa sapi dari spesies Bos taurus yang banyak dimanfaatkan di Indonesia
4 Bos indicus yang banyak dimanfaatkan adalah sapi Peranakan Ongole dan Brahman.
Karakteristik bangsa sapi dari berbagai spesies tercantum pada Tabel 1.
Tabel 1. Karakteristik Berbagai Bangsa Sapi pada Tiga Spesies
Spesies Bangsa Sapi Karakteristik
Bos javanicus
Sapi Bali Ciri sapi ini yaitu memiliki warna bulu merah bata kecoklatan hingga berumur 6 bulan dan akan berubah warna (bagi jantan) menjadi coklat tua hingga hitam mencapai dewasa. Sapi ini memiliki warna putih pada bagian pantat, bagian kaki (white stocking) hingga di atas kuku (Williamson dan Payne, 1993). Presentase karkas tinggi (55,85-60,80%), tingkat fertilitas tinggi (83-86%), serta angka kelahiran tinggi (72,6-92,6%) (Martodjo, 1990).
Bos taurus Limousin Sapi ini memiliki karakteristik warna bulu merah keemasan, kaki dan lutut kebawah memiliki warna merah agak muda, dan umumnya terdapat lingkaran berwarna merah muda disekeliling mata (Pane, 1986).
Simmental Karakteristik sapi ini memiliki warna bulu kecoklatan hingga sedikit merah, bulu muka berwarna putih dan bagian ekor serta lutut kebawah berwarna putih pula. Ukuran tanduk tidak begitu besar (Pane, 1986).
Angus Sapi Angus memiliki karakteristik kulit berwarna hitam, tidak bertanduk, tubuh rata, lebar dan dalam, seperti balok, padat dengan urat daging yang baik. Berat badan betina dewasa mencapai 1600 pounds sedang jantan dewasa 2000 pounds (Pane, 1986).
FH Sapi perah ini memiliki warna putih dengan belang hitam, hitam belang putih, sampai warna hitam. Sapi ini merupakan bangsa sapi perah dengan produksi susu paling tinggi (Syarif dan Sumopastowo, 1984).
Bos indicus Brahman Sap ini memiliki karakteristik ponok besar dan kulit yang longgar dengan lipatan kulit dibawah leher dan perut yang lebar, telinga menggantung. Warna kulit merah kecoklatan dengan tanduk melengkung keatas (Balai Pembibitan Ternak Potong Nusa Tenggara Timur, 2009).
5 Perbedaan karakteristik fenotipik dari berbagai bangsa sapi dapat terlihat jelas
dengan melihat gambar. Gambar 1 merupakan gambar fenotipik berbagai bangsa sapi
dari spesies sapi Bos javanicus, Bos taurus, dan Bos indicus.
(a) (b)
(c) (d)
(e) (f)
(g)
6
Keragaman Genetik
Sumber daya genetik yang semakin beragam menurut Frankham et al.(2002)
akan semakin tahan populasi tersebut untuk hidup dalam jangka waktu yang lama,
serta semakin tinggi daya adaptasi populasi terhadap perubahan lingkungan. Estimasi
perhitungan keragaman genetik dalam populasi secara kualitatif dapat diperoleh
melalui dua ukuran keragaman variasi populasi yaitu proporsi lokus polimorfisme
dalam populasi dan rata-rata proporsi individu heterozigot dalam lokus (Nei, 1987).
Keragaman genetik dalam antara subpopulasi dapat diketahui dengan melihat
persamaan dan perbedaan frekuensi alel di antara subpopulasi (Li et al., 2000).
Falconer dan Mackay (1996) menyatakan jika suatu alel dinyatakan polimorfik jika
memiliki frekuensi alel sama dengan atau kurang dari 0,99.
Noor (2000) menyatakan jika frekuensi genotipe suatu populasi yang cukup
besar akan selalu dalam keadaan seimbang bila tidak ada seleksi, migrasi, mutasi dan
genetic drift, selain itu silang dalam dan silang luar juga dapat mempengaruhi
frekuensi genotipe. Derajat heterozigositas merupakan rataan presentase lokus
heterozigositas tipe individu atau rataan presentase individu heterozigot dalam
populasi (Nei, 1987). Avise (1994) menyatakan jika semakin tinggi derajat
heterozigositas dalam suatu populasi maka daya tahan populasi tersebut akan
semakin tinggi. Javanmard et al. (2005) menyatakan jika nilai heterozigositas
dibawah 0,5 (50%) mengindikasikan rendahnya variasi suatu gen dalam populasi.
Machado et al. (2003) menyatakan jika nilai Ho lebih rendah dari He
mengindikasikan adanya proses seleksi yang intensif.
Gen Follicle Stimulating Hormone Receptor (FSHR)
Follicle stimulating hormone (FSH) adalah hormon yang disintesa dan
disekresikan oleh gonadotropes di kelenjar anterior pituitary. Fungsi utama FSH
adalah menstimulasi pertumbuhan dan pematangan folikel de graaf di dalam ovarium
dan spermatogenesis di dalam tubuli seminiferi testis. Proses spermatogenesis
meliputi proses spermatocytogenesis (pembentukan spermatosit primer dan
sekunder) dan spermiogenesis (proses pembentukan spermatozoa) (Toelihere, 1979).
Keberadaan hormon FSH sangat dipengaruhi oleh adanya gen FSH dan gen
FSHR. Gen FSH terbagi atas dua subunit yaitu α-subunit dan β-subunit dengan
7 memproduksi FSH reseptor yang membawa FSH ke target jaringan sehingga dapat
berfungsi untuk spermatogenesis pada jantan dan oogenesis pada betina. FSH
reseptor terekspreksi pada sel-sel granulose di ovarium dan sel-sel sertoli di testis.
Gen FSHR terdapat pada kromosom 11 dan terdiri dari 10 ekson dan 9 intron dengan
panjang keseluruhan ekson 2375 bp (Houde et al., 1994). Struktur pada sembilan
ekson pertama mengkodekan bagian terminal-amino ekstra-selular yang besar
sedangkan pada ekson kesepuluh mengkodekan bagian transmembran dan
intraselular dari protein. Ekson kedua hingga kedelapan memiliki panjang yang
hampir sama (68-77 bp) (Heckert and Griswold, 2002). Marson (2008) menyebutkan
jika penentuan genotipe gen FSHR adalah GG (193 dan 63/50 bp), CG (243/193 dan
63/50 bp), dan CC (243 dan 63 bp). Huhtaniemi dan Kristiina (1998) menyatakan
FSHR merupakan produk dari gen FSHR sangat menentukan ukuran testis, jumlah
sperma, dan motilitas sperma bagi pejantan, serta keberhasilan fungsi ovarium pada
betina.
Gambar 2. Struktur gen FSHR (Aguirre dan Timossi, 1998)
Polymerase Chain Reaction-Restriction Fragment Length Polymorphism (PCR-RFLP)
Poymerase Chain Reaction (PCR) menurut Mullis (1986) merupakan suatu
teknologi untuk mengamplifikasi (memperbanyak) fragmen DNA spesifik secara
in-vitro. Williams (2005) menambahkan jika PCR merupakan suatu teknik yang dapat
menggandakan jumlah molekul DNA pada ruas-ruas tertentu dan
monomer-monomer nukleotida yang berjalan dengan bantuan primer dan enzim polymerase.
Proses PCR terdiri dari tiga tahapan yaitu : (1) Denaturasi, yaitu perubahan struktur
DNA utas ganda menjadi utas tunggal, (2) Anealing, yaitu penempelan primer pada
sekuens DNA komplementer yang akan diperbanyak, dan (3) Ekstensi, yaitu
8 menyatakan jika proses PCR berlangsung dalam mesin thermocycler dimana pada
tahap denaturasi, annealing, dan ekstension berlangsung dalam 25-30 siklus. Tahap
DNA berlangsung dalam suhu 94oC sehingga DNA utai ganda dapat terpisah menjadi utai tunggal. Tahap yang paling menentukan adalah tahap penempelan
primer, karena setiap pasang primer memiliki suhu penempelan primer yang spesifik.
Tahap pemanjangan primer berlangsung pada suhu 27oC. Pada tahap ini enzim taq polymerase, buffer, dNTP, dan Mg2+ memulai aktifitasnya memperpanjang primer.
PCR-RFLP merupakan salah satu metode dalam PCR yang dikembangkan
untuk memvisualisasikan perbedaan level DNA yang didasarkan oleh penggunaan
enzim pemotong (restriction enzyme). Enzim restriksi ini dapat memotong DNA
pada sekuens nukleotida spesifik (Montaldo dan Herrera, 1998). Metode PCR
memanfaatkan runutan nukleotida yang dikenali oleh enzim restriksi dan disebut
dengan situs restriksi. Jika situs retriksi mengalami mutasi maka enzim restriksi tidak
mampu mengenalinya. Analisi RFLP biasa digunakan untuk mendeteksi ada atau
tidaknya keragaman pada gen yang berhubungan dengan sifat ekonomis (Sumantri et
MATERI DAN METODE Lokasi dan Waktu
Penelitian ini dilakukan di Laboratorium Genetika Molekuler Ternak, Bagian
Pemuliaan dan Genetik Ternak, Departemen Ilmu Produksi dan Teknologi
Peternakan, Fakultas Peternakan, Institut Pertanian Bogor. Penelitian dilaksanakan
pada bulan Oktober 2011 hingga bulan Februari 2012.
Materi Ternak
Sampel darah yang digunakan berasal dari tiga spesies sapi yaitu Bos
javanicus (sapi Bali), Bos taurus (sapi Limousin, Simmental, Angus, dan FH), dan
Bos indicus (sapi Brahman dan PO) dari berbagai lokasi. Sampel tersebut merupakan
koleksi Laboratorium Genetika Molekuler Ternak dengan jumlah seperti yang tertera
pada Tabel 2.
Tabel 2. Jumlah Sampel yang Digunakan dalam Penelitian
No Spesies Bangsa Asal Jumlah Sampel
Total Keseluruhan Sampel 207
Keterangan : Angka di dalam kurung menunjukkan jumlah sampel setiap bangsa sapi
Pengambilan Sampel
Bahan yang digunakan untuk pengambilan sampel adalah alkohol 70%, es,
dan kapas. Alat yang digunakan adalah jarum venoject, tabung vacutainer 10 ml, dan
10
Ekstraksi DNA
Bahan yang digunakan untuk ekstraksi DNA adalah sampel darah,
Destilation Water (DW), NaCl, Proteinase-K, 1 x STE (5M NaCl, 2M tris HCl, 0,2M
EDTA), SDS 10% (sodium dodesil sulfat), CIAA, etanol absolute, etanol 70%,
phenol, buffer TE 80% (tris EDTA). Peralatan yang digunakan adalah tabung
eppendorf 1,5 ml, satu set mikro pipet, tip, vortexmixer, mikrosentrifuge, inkubator,
dan freezer.
Primer
Primer yang digunakan dalam penelitian untuk mengamplifikasi gen
FSHR|Alu1 berdasarkan Houde et al. (1994) adalah forward : 5’-CTG CCT CCC
TCA AGG TGC CCC TC-3’; dan reverse : 5’-CCC CCT AAG ACA TTT AGC
CAA GAA CT -3’.
Amplifikasi Gen FSHR|Alu1
Bahan yang digunakan dalam amplifikasi metode analisa PCR-RFLP
(Polymerase Chain Reaction-Restriction Fragment Lenght Polymorphism) adalah
sampel DNA, destilated water (DW), buffer, MgCl2, pasangan primer fragmen Gen
FSHR|AluI, enzim Taq polymerase, dan dNTP (deoxy Nukleotida Triposfat). Alat
yang digunakan adalah satu set pipet mikro, sentrifuge, mesin thermocycler, rak dan
tabung eppendorf, tip pipet dan vortex.
Elektroforesis
Bahan yang digunakan adalah produk PCR, agarose, loading dye, marker
100 pb, 0,5 x TBE, dan Ethidium Bromide. Alat yang digunakan adalah tip pipet,
mikropipet, gelas ukur, stirrer, microwave, gel tray, pencetak untuk sumur (comb),
power supply electrophoresis 100 volt, alat foto UV trans-iluminator, dan sarung
tangan.
Genotyping (Penentuan Genotipe)
Bahan yang digunakan untuk genotyping adalah produk PCR, agarose,
loading dye, marker 100pb, enzim restriksi AluI, DW, buffer tango, 0,5 x TBE, dan
Ethidium Bromide (EtBr). Alat yang digunakan adalah tip pipet, mikropipet 10 P
11 (comb), power supply electrophoresis 100 volt, alat foto UV trans iluminator, dan
sarung tangan.
Prosedur Pengambilan Sampel
Sampel darah diambil melalui vena jugularis dengan menggunakan jarum
venoject. Sampel darah ditaruh pada tabung vaccutainer yang ditambahkan etanol
absolute dengan perbandingan 1:2 dan disimpan dalam lemari es hingga akan
digunakan lebih lanjut.
Ekstraksi DNA
Ekstraksi DNA dilakukan dengan menggunakan metode yang dikemukakan
oleh Sambrook et al. (1989). Ekstraksi DNA dimulai dengan preparasi sampel.
Sampel darah sebanyak 200 µl dimasukkan ke dalam tabung 1,5 ml dan ditambahkan
1000 µl DW (destilation water). Campuran sampel darah dan DW tersebut
selanjutnya dikocok atau di vortex dan didiamkan selama lima menit, setelah itu
disentrifugasi dengan kecepatan 8000 rpm selama lima menit. Bagian supernatannya
dibuang selanjutnya diulangi seperti proses sebelumnya. Tahap selanjutnya dari
ekstraksi DNA adalah degradasi protein. Proteinase-K sebanyak 10 µl, 350 µl
1xSTE (sodium tris-EDTA) serta 40 µl 10% SDS (sodium dodesil sulfat)
ditambahkan. Campuran tersebut kemudian diinkubasi pada suhu 55oC selama 2 jam sambil dikocok perlahan menggunakan alat pemutar (nutator).
Degradasi bahan organik dilakukan dengan menambahkan 40 µl 5M NaCl,
400 µl larutan fenol, dan 400 µl CIAA (chloroform iso amil alcohol), lalu dikocok
pelan pada suhu ruang selama satu jam. Tahap terakhir dari ekstraksi DNA adalah
presipitasi DNA yakni dengan larutan hasil degradasi bahan organik disentrifugasi
dengan kecepatan 12.000 selama lima menit sehingga terbentuk fase DNA. Fase
DNA tersebut selanjutnya diambil sebanyak 40 µl dan dipindahkan ke tabung baru
1,5 ml. NaCl 5M sebanyak 40 µl dan etanol absolut sebanyak 800 µl ditambahkan,
kemudian dihomogenkan, dan didiamkan over night pada suhu -20oC. Molekul DNA kemudian dipisahkan daru etanol absolute dengan cara disentrifugasi pada kecepatan
12.000 rpm selama lima menit.Supernatan yang terbentuk dibuang sehingga
12 disuspensikan dalam 100 µl 80% buffer TE (tris EDTA) lalu di spin down. Sampel
selanjutnya dielektroforesis untuk mengetahui ada atau tidaknya DNA menggunakan
agar 1,5%.
Amplifikasi DNA
Amplifikasi gen FSHR dilakukan dengan menggunakan metode PCR-RFLP.
Pereaksi yang dicampurkan untuk melakukan amplifikasi terdiri dari 1 µl sampel
DNA, 10,85 µl DW, 0,3 µl primer, 0,05 µl taq polymerase, 1,5 µl buffer, 0,3 µl
dNTP, dan 1 µl MgCl2. Amplifikasi invitro ini menggunakan mesin thermocycler.
Kondisi suhu yang terjadi pada setiap tahapan PCR yaitu pra-denaturasi 95oC selama lima menit dalam satu siklus, 35 siklus untuk tahapan denaturasi 95oC selama 45 detik, annealing pada suhu 60oC selama 45 detik, dan elongasi pada suhu 72oC selama satu menit. Tahap selanjutnya adalah elongasi akhir 72oC selama lima menit dalam satu siklus.
Elektroforesis
Elektroforesis produk PCR dilakukan dengan terlebih dahulu membuat gel
agarose 1,5% dengan cara agarose 0,45 g dilarutkan dalam larutan 0,5 x TBE
sebanyak 30 ml lalu dipanaskan dalam microwave selama lima menit dan distirer
dengan menambahkan 2,5 µl EtBr. Larutan yang masih cair dituangkan ke dalam
pencetak gel serta sisir ditempatkan di dekat tepian gel dan dibiarkan mengeras. Sisir
dicabut setelah gel mengeras sehingga terbentuk sumur-sumur. Gel selanjutnya
ditempatkan ke dalam gel tray elektroforesis yang sudah berisi larutan buffer. Produk
PCR sebanyak 5 µl dicampur dengan loading dye (bromothymol blue 0,01%, xylene
cyanol 0,01%, dan gliserol 50%) sebanyak 1 µl dengan menggunakan mikropipet
dimasukkan dalam sumur-sumur gel. Marker sebanyak 2 µl ditaruh dalam sumur
paling kiri sebagai penanda. Gel tray selanjutnya dialiri listrik 100 volt selama 30
menit, molekul DNA yang bermuatan negatif pada pH netral akan bergerak
(bermigrasi) ke arah positif. Setelah elektroforesis selesai, gel agarose diambil untuk
dilihat panjang pita DNA dengan menggunakan sinar ultraviolet dalam mesin
UV-transiluminator. Pembacaan fragmen DNA dilakukan dengan menarik garis lurus
13
Genotyping
Tahapan RFLP dilakukan dengan memindahkan 5 µl produk PCR kedalam
tabung 0,5 ml dan ditambahkan 0,3 µl enzim restriksi AluI, buffer tango 0,7 µl, dan
DW 1 µl. Campuran tersebut kemudian diinkubator pada suhu 37oC selama 16 jam.
Sampel DNA yang telah dipotong dengan enzim restriksi ditambahkan
loading dye sebanyak 1 µl, dielektroforesis pada gel agarose 2% (0,6 agarose dalam
30 ml 0,5 x TBE) dengan tegangan 100 volt selama 45 menit. Setelah
dielektroforesis, gel agarose diambil untuk dilihat panjang pita DNA dengan
menggunakan sinar ultraviolet dan dibandingkan dengan marker untuk mengetahui
panjangnya. Gambar 3 merupakan posisi penempelan primer gen FSHR dan posisi
pemotongan oleh enzim AluI.
forward
1 ctgcctccct caaggtgccc ctcatcactg tgtccaagtc aaagatcctc ctggtcctgt
61 tctaccccat caactcctgt gccaacccct tcctctatgc catcttcacc aagaacttcc
121 gcagggattt cttcattctg ctgagcaagt ttggctgcta tgaagtgcaa gcccagacct
181 ataggtcaga aACCTcatcc actgcccaca actttcatcc aaggaatggc cactgccccc
241 cAG|CTcccag ggttactaat ggttccaatt acacccttat ccccctaaga catttagcca
301 agaact
reverse
Alel C : 5’---tcagaaACCTcatcc---3’ Alel G : 5’---tcagaaAGCTcatcc---3’ Keterangan : Alel C mempunyai basa C pada posisi basa ke 193
Alel G mempunyai basa G pada posisi basa ke 193
Gambar 3. Fragmen gen Follicle Stimulating Hormone Reseptor (FSHR|AluI) berdasakan sekuens gen FSHR di genbank (kode akses: L22319.1)
Analisis Data Frekuensi Alel dan Genotipe
Frekuensi genotipe adalah rasio dari jumlah suatu genotipe terhadap suatu
populasi dengan menghitung perbandingan antara jumlah genotipe tertentu pada
setiap populasi. Rumus menghitung frekuensi genotipe menurut Nei dan Kumar
(2000) adalah sebagai berikut :
�ii =
� Keterangan :
�ii = frekuensi genotipe ke-ii
nii = jumlah individu bergenotipe ii
14 Frekuensi alel adalah rasio suatu alel terhadap keseluruhan alel pada satu
lokus dalam populasi. Frekuensi alel (�i) gen FSHR dapat dihitung berdasarkan
rumus Nei dan Kumar (2000) seperti di bawah ini :
�i = +∑
�
Keterangan :
�i = frekuensi alel ke-i
nii = jumlah individu bergenotipe ii
nij = jumlah individu bergenotipe ij
N = jumlah individu sampel
Heterozigositas
Keragaman genetik dapat diketahui melalui estimasi frekuensi heterozigositas
pengamatan yang diperoleh dari masing-masing populasi dengan menggunakan
rumus Weir (1996) sebagai berikut :
� = � �
≠ Keterangan :
Ho = heterozigositas pengamatan (populasi) nij = jumlah individu heterozigot
N = jumlah individu yang diamati
Heterozigositas harapan (He) berdasarkan frekuensi alel dihitung dengan
menggunakan rumus Nei dan Kumar (2000) :
��= − � �
�
= Keterangan :
He = nilai heterozigositas harapan �i = frekuensi alel
HASIL DAN PEMBAHASAN Amplifikasi Gen FSHR
Ruas gen Follicle Stimulating Hormone Receptor (FSHR) diamplifikasi
dengan metode Polymerase Chain Reaction (PCR) menggunakan primer yang
dirancang oleh Houde et al. (1994). Suhu annealing yang digunakan dalam
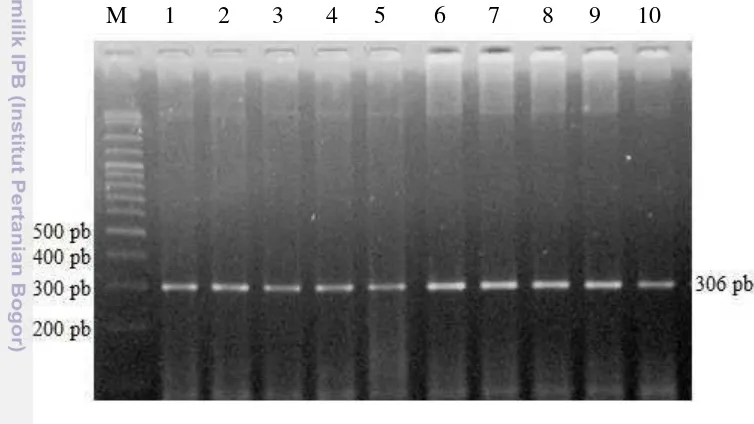
penelitian ini adalah 60oC selama 45 detik. Produk amplifikasi ruas gen FSHR adalah sebesar 306 pb dengan presentase keberhasilan amplifikasi mencapai 100%
(207/207). Hasil amplifikasi gen FSHR yang divisualisasikan pada gel agarose 1,5%
ditampilkan dalam Gambar 4.
M 1 2 3 4 5 6 7 8 9 10
Gambar 4. Visualisasi Amplifikasi PCR Fragmen Gen FSHR Menggunakan Gel Agarose 1,5 %. Ket : M=marker (100 pb); pb= pasang basa; 1-10= beberapa sampel sapi yang digunakan
Suhu annealing merupakan suhu yang memungkinkan terjadinya penempelan
primer pada DNA yang merupakan proses paling penting dalam amplifikasi. Viljoen
et al. (2005) menyatakan jika keberhasilan amplifikasi gen ditentukan oleh
keberhasilan penempelan primer pada gen target. Loss et al. (2008); Marson et al.
(2008); dan Hernandez et al. (2009) menggunakan suhu annealing 58 oC selama 30 detik dalam mengamplifikasi ruas gen FSHR dan menghasilkan produk PCR yang
baik. Perbedaan suhu annealing dengan hasil penelitian ini dapat disebabkan adanya
perbedaan komposisi mix sebagai komponen PCR. Faktor-faktor yang menentukan
keberhasilan penempelan primer pada gen target yakni kondisi suhu PCR
16 Rychilk (1995) menambahkan jika konsentrasi sampel DNA, konsentrasi primer,
garam, dan konsentrasi pelarut dalam Premix yang digunakan juga mempengaruhi
keberhasilan proses amplifikasi.
. Gen FSHR merupakan gen yang berperan dalam proses reproduksi. Produk
dari gen FSHR yaitu FSHR berperan untuk membawa hormon FSH menuju sel target
sehingga dapat melakukan fungsi reproduksinya (pembentukan ovum pada betina
dan sperma pada jantan). Gen FSHR pada sapi terletak di kromosom 11 dan terdiri
atas 10 ekson dan 9 intron (Aguirre dan Timossi, 1998). Panjang produk amplifikasi
ruas gen FSHR pada sapi-sapi Bos Javanicus, Bos Taurus, dan Bos Indicus dihitung
dari awal primer forward menempel hingga akhir penempelan primer reverse.
Penempelan primer dalam ruas gen FSHR ini terletak pada ekson 10 dengan panjang
306 pb. Loss et al. (2008), Marson et al. (2008), dan Hernandez et al. (2009) juga
menemukan panjang produk amplifikasi gen FSHR sepanjang 306 pb.
Keragaman Gen FSHR|AluI
Pendeteksian keragaman genetik gen FSHR dilakukan dengan metode RFLP.
Metode ini menggunakan enzim pemotong AluI (FSHR|AluI) yang mengenali situs
pemotongan AG|CT. Pemotongan ruas gen hasil amplifikasi ini menentukan genotipe
dan alel masing-masing individu sapi yang berperan dalam pendeteksian keragaman
genetiknya. Hasil pemotongan sekuens DNA gen FSHR pada sapi-sapi Bos taurus
dan Bos indicus ditemukan dua alel (alel C dan G) dalam ruas gen FSHR, sedangkan
pada Bos javanicus hanya ditemukan satu alel yaitu alel G. Alel C dalam ruas gen
FSHR diakibatkan oleh adanya satu titik potong yang dikenali enzim AluI pada
posisi basa ke 243 pb. Pemotongan tersebut menghasilkan dua fragmen yang
panjangnya 243 pb dan 63 pb. Sedangkan alel G diakibatkan oleh adanya dua titik
potong yaitu pada posisi basa ke 193 pb dan 243 pb. Pemotongan tersebut
menghasilkan tiga fragmen yang panjangnya 193, 63, dan 50 pb.
Hasil PCR-RFLP ruas gen FSHR|AluI menunjukkan adanya pola pita
beragam dengan tiga macam genotipe. Genotipe CC terdiri dari dua pita (243 pb dan
63 pb), genotipe CG terdiri dari empat pita (243, 193, 63, dan 50 pb), dan genotipe
GG terdiri dari tiga pita (193, 63, dan 50 pb). Individu CC dan GG disebut sebagai
individu homozigot karena kedua tetuanya menyumbangkan alel yang sama,
17 menyumbangan alel yang berbeda. Hasil visualisasi produk PCR dengan pada gel
agarose 2% ditampilkan pada Gambar 4. Hasil visualisasi dalam Gambar 5, pita
dengan panjang 50 bp dan 63 bp tidak terlihat dalam agarose 2% karena panjang pita
tersebut terlalu rendah (terjadi running out) sehingga butuh konsentrasi agarose yang
lebih tinggi sehingga dapat memunculkannya. Muladno (2002) menyatakan jika
dalam konsentrasi agarose 2% kisaran ukuran DNA yang dapat dianalisis berada
diantara 100 hingga 2000 pb.
M 1 2 3 4 5 6 7 10 11 12 13 14
GG GG CG CG CC CC GG GG CG CG CC CC
Gambar 5. Hasil Visualisasi PCR-RFLP Gen FSHR|AluI dalam Gel Agarose 2%
Perbedaan alel yang ditemukan dalam ruas gen FSHR dari individu sapi yang
satu dengan lainnya disebabkan adanya perubahan basa sehingga dapat dikenali oleh
enzim AluI. Mutasi yang ditemukan dalam ruas gen FSHR termasuk dalam tipe
mutasi subtitusi transversi karena basa Cytosine berubah menjadi basa Guanin.
Sebagaimana yang dinyatakan Li dan Graur (1991) mutasi tipe transversi terjadi
akibat adanya pertukaran basa antara basa purin (A=Adenin, G=Guanin) dengan basa
pirimidin (C=Cytosine, T=Timin). Mutasi tersebut menyebabkan terjadinya
perubahan susunan asam amino yaitu asam amino Asparagin berubah menjadi asam
amino lysin (AAC – Asparagin; AAG – Lysin). Akan tetapi dalam penelitian ini,
mutasi hanya terjadi pada sapi-sapi Bos taurus dan Bos indicus saja. Pada sapi Bali
(Bos javanicus) meskipun alel yang ditemukan dalam ruas gen FSHR ini hanya alel
G saja namun hal tersebut bukan disebabkan oleh mutasi melainkan karena adanya
perbedaan nukleotida pada sapi Bali dengan sapi Bos taurus dan Bos indicus. Hasil
pemotongan tersebut dapat digunakan sebagai penciri genetik gen FSHR|AluI untuk
18 Genotipe yang ditemukan pada sapi Bos taurus dan sapi Bos indicus adalah
CC, CG, dan GG, sedangkan pada sapi Bos javanicus (sapi Bali) hanya ditemukan
satu genotipe saja yaitu genotipe GG. Sapi limousin, simmental, FH, yang
merupakan sapi Bos taurus muncul ketiga genotipe tersebut, akan tetapi pada sapi
Angus hanya muncul dua genotipe saja yaitu CC dan CG. Jumlah sampel sapi angus
yang digunakan dalam penelitian ini hanya enam sampel. Jumlah tersebut harus
diperbanyak agar mendapatkan hasil yang lebih akurat. Sapi Bos indicus yaitu sapi
Brahman yang digunakan dalam penelitian ini muncul ketiga genotipe CC, CG, dan
GG, akan tetapi pada sapi PO hanya menunjukkan dua genotipe yakni CC dan GG.
Frekuensi Genotipe dan Frekuensi Alel Gen FSHR
Frekuensi genotipe adalah rasio dari jumlah suatu genotipe dengan
menghitung perbandingan antara jumlah genotipe tertentu. Sedangkan frekuensi alel
adalah rasio suatu alel terhadap keseluruhan alel pada satu lokus dalam populasi.
Frekuensi genotipe dan frekuensi alel dari penelitian yang dilakukan ditampilkan
pada Tabel 3.
Tabel 3. Frekuensi Genotipe dan Frekuensi Alel dari Gen FSHR|AluI
Spesies Bangsa n Genotipe Alel
Keterangan : angka di dalam kurung menunjukkan jumlah sampel
Hasil analisis frekuensi genotipe dan frekuensi alel dalam Tabel 3
menunjukkan jika pada sapi Limousin, Simmental, dan Angus memiliki frekuensi
genotipe CC yang lebih besar dibandingkan dua genotipe lainya dengan nilai
19 dan sapi Simmental memiliki nilai frekuensi genotipe GG yang paling kecil yaitu
0,05 dan 0,18. Pada sapi Angus hanya ditemukan genotipe CC dan CG saja dengan
nilai frekuensi genotipe CG sebesar 0,17. Sapi FH memiliki frekuensi genotipe CG
yang lebih besar dibandingkan kedua genotipe lainnya dengan nilai 0,46. Sapi
Brahman memiliki frekuensi genotipe CC yang paling rendah yaitu 0,12 sedangkan
genotipe CG dan GG memiliki nilai frekuensi genotipe yang sama yaitu 0,44. Sapi
PO yang diamati dalam penelitian hanya menunjukkan dua genotipe saja yaitu CC
dan GG dengan nilai yang sama masing-masing yaitu 0,50. Sapi Bali yang
merupakan sapi lokal Indonesia hanya menunjukkan satu buah genotipe saja dari
keseluruhan sampel yaitu genotipe GG. Hal ini menyebabkan frekuensi genotipe GG
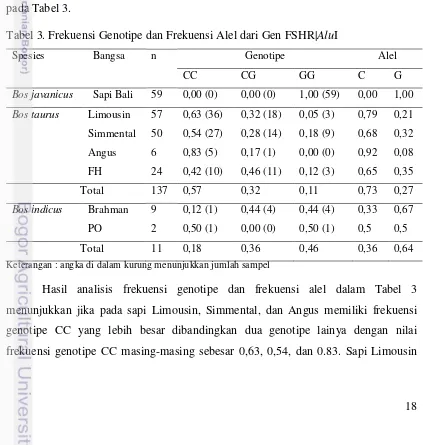
pada sapi Bali sangat tinggi yaitu 1,00. Gambar 6 menunjukkan grafik frekuensi
genotipe pada spesies sapi Bos javanicus, Bos taurus, dan Bos indicus.
Gambar 6. Frekuensi Genotipe FSHR|AluI di Berbagai Spesies Sapi (dalam Persen)
Gambar 6 menunjukkan dengan jelas perbedaan antara spesies sapi Bos
taurus dengan spesies sapi Bos indicus. Spesies sapi Bos taurus memiliki genotipe
CC yang terbesar, sedangkan pada spesies sapi Bos indicus genotipe yang terbesar
adalah genotipe GG. Genotipe GG yang merupakan genotipe satu-satunya yang 0
Bos javanicus Bos taurus Bos indicus
20 muncul pada sapi Bali (Bos javanicus) menjadikan frekuensi genotipe GG pada
spesies sapi Bos javanicus adalah sebesar 1,00, sedangkan frekuensi genotipe CC dan
CG adalah 0,00. Sapi Bos taurus memiliki frekuensi genotipe CC yang lebih besar
dari dua genotipe lainnya yaitu sebesar 0,57. Genotipe GG merupakan genotipe
dengan frekuensi yang paling rendah dari spesies sapi Bos taurus yaitu sebesar 0,11.
Hasil ini berbanding terbalik dengan spesies sapi Bos indicus dengan frekuensi
genotipe GG paling besar yaitu 0,46 dan frekuensi genotipe CC paling rendah yaitu
0,18. Frekuensi genotipe CG pada sapi Bos taurus dan Bos indicus secara berurutan
adalah 0,32 dan 0,36.
Tipe alel yang ditemukan dalam penelitian ini adalah alel C dan alel G.
Frekuensi alel C pada sapi Simmental, Limousin, Angus, dan FH lebih tinggi
dibandingkan frekuensi alel G. Nilai frekuensi alel C dan G pada sapi Simmental,
Limousin, Angus, dan FH berturut-turut adalah 0,680 dan 0,320, 0,789 dan 0,211,
0,917 dan 0,083, serta 0,646 dan 0,354. Pada sapi Bali dan Brahman nilai frekuensi
alel G lebih besar dibandingkan frekuensi alel C yaitu sebesar 1,000 dan 0,000 pada
sapi Bali serta 0,667 dan 0,333 pada sapi Brahman. Sapi PO memiliki nilai frekuensi
alel C dan G yang sama yaitu 0,500. Hal ini disebabkan hanya ditemukan 2 genotipe
saja pada sapi PO yaitu CC dan GG.
Frekuensi alel C pada spesies Bos taurus lebih besar dibandingkan dengan
frekuensi alel G. Frekuensi alel C Bos taurus 0,73 sedangkan frekuensi alel G
sebesar 0,27. Hasil tersebut sesuai dengan penelitian Ishak (2012) yang menunjukkan
frekuensi alel C lebih besar dibandingkan frekuensi alel G pada sapi Bos taurus.
Hasil tersebut berbanding terbalik dengan spesies Bos javanicus dan Bos indicus.
Kedua spesies ini menujukkan jika frekuensi alel G lebih besar dibandingkan
frekuensi alel C. Frekuensi alel G pada spesies Bos javanicus adalah 1,00 dan pada
spesies Bos indicus adalah 0,64. Penelitian yang dilakukan Ishak (2012)
menunjukkan jika sapi dari spesies Bos javanicus (sapi Bali) memiliki nilai frekuensi
alel G 0,951 dan alel C 0,049. Terdapatnya alel G dalam hasil tersebut dikarenakan
sapi Bali yang diteliti oleh Ishak (2012) berasal dari NTT, NTB dan Sulawesi yang
diduga telah mengalami pencemaran. Penelitian yang dilakukan oleh Loss et al.
21 dengan penelitian yaitu memiliki frekuensi alel G yang besar dibandingkan dengan
alel C.
Nilai frekuensi alel dalam penelitian ini pada spesies sapi Bos taurus dan Bos
indicus bersifat polimorfik. Nilai frekuensi alel pada spesies sapi Bos javanicus
hanya ditemukan genotipe GG dan hanya memiliki satu alel yaitu alel G dengan nilai
1,000. Oleh karena itu Gen FSHR yang ditemukan pada sapi Bos javanicus bersifat
monomorfik atau seragam. Hal ini sesuai dengan pendapat Nei (1987) yang
menyatakan bahwa suatu alel dikatakan polimorfik atau beragam jika memiliki
frekuensi alel sama dengan atau kurang dari 0,99, akan tetapi jika terjadi lebih dari
0,99 dikatakan monomorfik atau seragam. Keragaman yang bersifat monomorfik
pada sapi Bos javanicus menunjukkan jika adanya manajemen perkawinan yang
tidak acak terlebih sampel sapi Bali yang digunakan bersasal dari daera Bali saja,
selain itu adanya seleksi dalam sifat tertentu, serta tingkat silang dalam yang tinggi.
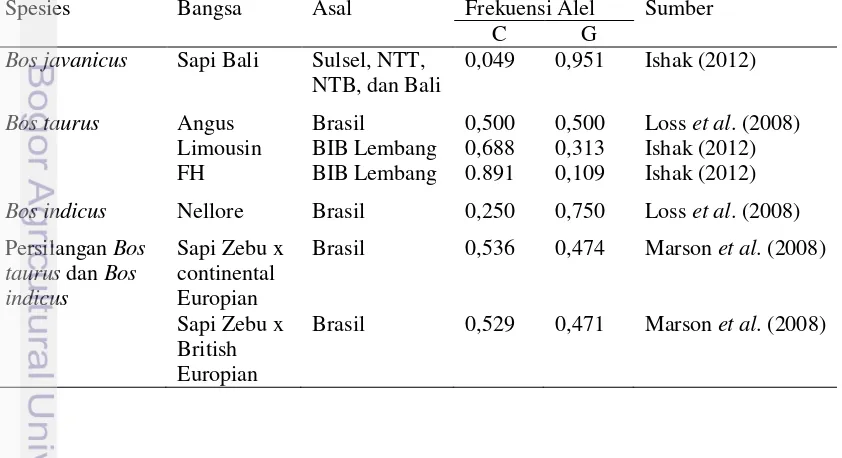
Tabel 4 menunjukkan beberapa pembanding hasil frekuensi alel gen
FSHR|AluIdari beberapa literatur yang juga menggunakan metode PCR-RFLP. Pada
tabel tersebut terlihat jika Marson et al (2008) mengidentifikasi keragaman gen
FSHR menggunakan sapi persilangan spesies Bos taurus dan Bos indicus. Hasil dari
penelitian tersebut adalah nilai frekuensi alel C lebih tinggi dibandingkan alel G.
Antara nilai frekuensi alel C dan alel G juga tidak berbeda jauh. Hasil penentuan
genotipe sapi persilangan ini memiliki frekuensi genotipe CG yang lebih tinggi
dibandingkan genotipe CC dan GG.
Tabel 4.Frekuensi alel gen FSHR|AluI pada Beberapa sumber
Spesies Bangsa Asal Frekuensi Alel Sumber
C G
Bos javanicus Sapi Bali Sulsel, NTT,
22 Gen FSHR berperan dalam fungsi reproduksi sangat menentukan kualitas
sperma sapi pada sapi jantan. Huhtaniemi dan Kristiina (1998) menyatakan jika
FSHR yang merupakan produk dari gen FSHR sangat menentukan ukuran testis,
jumlah sperma, dan motilitas sperma bagi pejantan, serta keberhasilan fungsi
ovarium pada betina. Ishak (2012) dalam penelitianya menunjukkan jika sapi Bos
taurus dan Bos indicus dengan genotipe GG memiliki tingkat keabnormalitas yang
lebih tinggi dibandingkan dua genotipe lainnya. Ishak (2012) juga menyatakan
bangsa sapi Bali (Bos javanicus) memiliki kualitas sperma yang paling baik
dibandingkan dengan bangsa sapi dari dua spesies lainnya. Arifianti et al. (2010)
menyatakan bahwa presentase abnormanilitas sapi Bali (Bos javanicus) sebesar 6%
dengan 2% kecatatan pada kepala. Kualitas sperma sapi tidak hanya dipengaruhi oleh
faktor genetik saja. Faktor lingkungan juga memiliki pengaruh besar pada kualitas
sperma sapi seperti kandungan nutrisi pakan, manajemen pemeliharaan, dan cuaca
lingkungan.
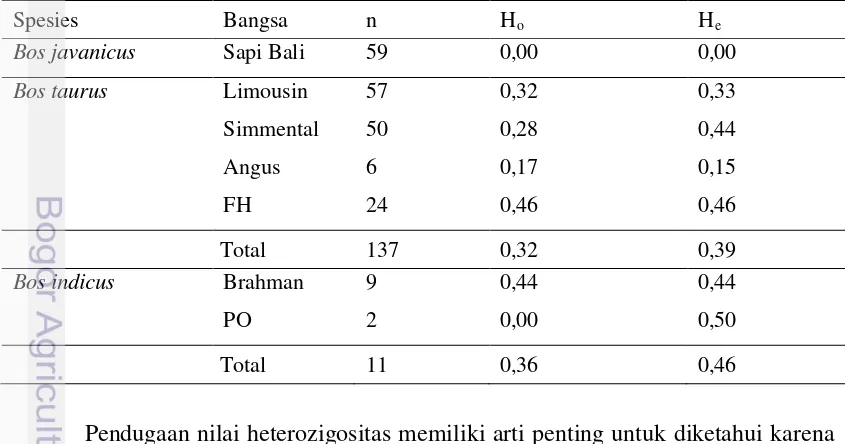
Heterozigositas
Nilai heterozigositas dihitung untuk mengetahui keragaman genetik gen
FSHR|AluI pada suatu populasi. Analsisis hetorozigositas pada berbagai sapi
ditampilkan pada Tabel 5.
Tabel 5. Nilai Heterozigositas Pengamatan (Ho) dan Harapan (He) Gen FSHR
Spesies Bangsa n Ho He
Pendugaan nilai heterozigositas memiliki arti penting untuk diketahui karena
dapat memberikan informasi mengenai tingkat polimorfisme suatu alel, serta prospek
23 (2005) menambahkan jika pendugaan nilai heterozigositas diperoleh untuk
mendapatkan kergaman genetik dalam populasi sehingga dapat dimanfaatkan untuk
program seleksi dan menjadi sumber genetik pada generasi berikutnya. Pendugaan
nilai heterozigositas pada penelitian ini berdasarkan nilai heterozigositas pengamatan
(Ho) dan nilai heterozigositas harapan (He). Heterosigositas harapan (He) merupakan
penduga keragaman genetik yang tepat pada populasi hewan ternak karena
perhitungannya langsung berdasarkan pada frekuensi alel (Moioli et al., 2004).
Heterozigositas pengamatan (Ho) dalam penelitian ini paling besar adalah
pada spesies sapi Bos indicus yaitu 0,36 dan yang paling terendah adalah spesies Bos
javanicus yaitu 0,00. Pada spesies sapi Bos taurus, sapi FH memiliki nilai
Heterozigositas pengamatan (Ho) yang paling tinggi yaitu 0,46 dan pada spesies sapi
Bos indicus sapi Brahman memiliki nilai yang paling tinggi yaitu 0,44.
Javanmard et al. (2005) menyatakan jika nilai heterozigositas dibawah 0,5
(50%) mengindikasikan rendahnya variasi suatu gen dalam populasi.Hal demikian
menunjukkan jika rendahnya keragaman gen FSHR|AluI pada sapi-sapi spesies Bos
javanicus, Bos taurus, dan Bos indicus. Nilai heterozigositas dipengaruhi oleh
banyaknya sampel, jumlah alel, dan frekuensi alel dalam suatu populasi (Nei, 1987).
Jumlah sampel pada spesies Bos indicus yang sangat rendah (11 sampel) mengurangi
keakuratan nilai heterozigositas.
Nilai heterozigositas harapan (He) pada spesies sapi Bos javanicus dan Bos
taurus yang diamati tidak menunjukkan perbedaan yang besar dengan nilai
heterozigositas pengamatannya (Ho). Akan tetapi, pada spesies sapi Bos indicus
memiliki perbedaan yang cukup tinggi. Tambasco et al. (2003) menyatakan jika
terjadi pebedaan yang besar antara heterozigositas harapan (He) dan heterozigositas
pengamatannya (Ho) maka terjadi ketidakseimbangan genotipe dalam populasi yang
diamati. Hal yang serupa juga terjadi pada nilai heterozigositas sapi Simmental dari
spesies Bos tarus. Nilai Ho pada keseluruhan spesies sapi memiliki nilai yang lebih
rendah dari nilai He. Machado et al. (2003) menyatakan jika nilai Ho lebih rendah
dari He mengindikasikan adanya proses seleksi yang intensif. Berdasarkan hal
tersebut maka deiperkirakan telah terjadi proses seleksi intensif berdasarkan gen
KESIMPULAN DAN SARAN Kesimpulan
Gen FSHR|AluI pada spesies sapi Bos taurus dan Bos indicus bersifat
polimorfik dengan ditemukannya dua alel (alel C dan G) dan tiga genotipe (genotipe
CC, CG, dan GG), sedangkan pada spesies Bos javanicus (sapi Bali) bersifat
monomorfik dengan ditemukan satu alel (G) dan satu genotipe (GG). Derajat
heterozigositas pada spesies Bos taurus (Limousin, Simmental, Angus, dan FH) dan
Bos indicus (Brahman dan Peranakan Ongole) memiliki nilai heterozigositas tinggi
dibandingkan dengan sapi yang termasuk dalam spesies Bos javanicus (sapi Bali).
Saran
Penelitian terhadap gen FSHR|AluI perlu dilakukan dengan jumlah sampel
yang banyak terutama pada sampel sapi yang termasuk spesies Bos indicus. Selain
IDENTIFIKASI KERAGAMAN GEN
FOLLICLE STIMULATING
HORMONE RECEPTOR
(FSHR|
Alu
I) PADA SPESIES SAPI
Bos javanicus
,
Bos taurus
, DAN
Bos indicus
DENGAN METODE PCR-RFLP
SKRIPSI
SEPTYANINGTYAS ANGGIA SARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
IDENTIFIKASI KERAGAMAN GEN
FOLLICLE STIMULATING
HORMONE RECEPTOR
(FSHR|
Alu
I) PADA SPESIES SAPI
Bos javanicus
,
Bos taurus
, DAN
Bos indicus
DENGAN METODE PCR-RFLP
SKRIPSI
SEPTYANINGTYAS ANGGIA SARI
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
RINGKASAN
SEPTYANINGTYAS ANGGIA SARI. D14080028. 2012. Identifikasi Keragaman Gen Follicle Stimulating Hormone Receptor (FSHR|AluI) pada Spesies Sapi Bos javanicus, Bos taurus, dan Bos indicus dengan Metode PCR-RFLP. Skripsi. Departemen Ilmu Produksi dan Teknologi Peternakan. Fakultas Peternakan. Institut Pertanian Bogor.
Pembimbing Utama : Prof. Dr. Ir.Cece Sumantri, M.Agr.Sc. Pembimbing Anggota : Dr. Jakaria, S.Pt,M.Si.
Sapi dari spesies Bos javanicus, Bos taurus,dan Bos indicus merupakan sapi-sapi yang tersebar diseluruh wilayah Indonesia. Sumber daya genetik ternak yang harus dipertahankan agar mencegah terjadinya kepunahan menjadi alasan untuk mengkaji keragaman gen FSHR yang berperan dalam fungsi reproduksi. Gen FSHR (Follicle Stimulating Hormone Receptor) akan menghasilkan produk yaitu FSHR yang diperlukan oleh hormone FSH untuk mencapai sel target sehingga dapat melaksanakan fungsi reproduksi (menghasilkan sperma dan ovum). Penelitian ini bertujuan untuk mengetahui keragaman gen Follicle Stimulating Hormone Receptor (FSHR|AluI) dengan menggunakan metode PCR-RFLP pada spesies sapi Bos javanicus, Bos taurus, dan Bos indicus.
Sampel yang digunakan berjumlah 207 sampel yaitu terdiri dari sampel spesies sapi Bos javanicus 59 sampel, spesies Bos taurus 137 sampel, dan spesies Bos indicus 11 sampel. Sampel spesies sapi Bos javanicus berasal dari sapi Bali, sampel sapi Bos taurus terdiri dari sapi Limousin (57 sampel), Simmental (50 sampel), Angus (6 sampel), dan FH (24 sampel), serta sampel spesies sapi Bos indicus terdiri dari sampel Brahman (9 sampel) dan sapi Ongole (2 sampel). Sampel sapi yang digunakan berasal dari BIB Lembang, BIB Singosari, BPTU Bali, BIBD Bali, dan BET Cipelang. Penelitian ini menggunakan metode PCR (Polymerase Chain Reaction) dan RFLP (Restriction Fragment Length Polymorphism) untuk mendeteksi adanya keragaman genetic pada gen FSHR menggunakan enzim rektriksi AluI yang memotong basa AG|CT. Penelitian ini dilakukan di Laboratorium Genetika Molekuler Ternak sejak bulan Oktober 2011 hingga Februari 2012. Keragaman gen FSHR ditentukan dengan analisis frekuensi genotipe, frekuensi alel, nilai heterozigositas pengamatan (Ho), dan
nilai heterozigositas harapan (He).
Amplifikasi gen FSHR menghasilkan fragmen dengan panjang 306 bp yang terletak pada ekson 10 dengan suhu annealing 60oC selama 45 detik. Genotyping gen FSHR|AluI menghasilkan dua jenis alel yaitu alel C dan alel G serta menghasilkan tiga jenis genotipe yaitu CC (243 dan 63 bp), CG (243, 293, 50, 63 bp), dan GG (193, 50, dan 63 bp). Frekuensi genotipe GG tertinggi pada spesies sapi Bos javanicus yaitu 1,00, Bos indicus sebesar 0,46 dan terendah pada spesies sapi Bos taurus yaitu 0,11. Frekuensi genotype CC paling tinggi pada spesies sapi Bos taurus yaitu 0,57, Bos indicus sebesar 0,18, dan terendah pada spesies sapi Bos javanicus. Nilai heterozigositas Ho dan He paling tinggi pada spesies sapi Bos indicus sedangkan nilai
terendah pada spesies sapi Bos javanicus.
ABSTRACT
Identification Polymorphism of The Follicle Stimulating Hormone Receptor (FSHR|AluI) Gene in Species Bos javanicus, Bos taurus, and Bos indicus
Using PCR-RFLP Method Sari, S.A., C. Sumantri, and Jakaria
Follicle Stimulating Hormone Receptor (FSHR) gene is a very important in reproduction trait. FSHR is required by Follicle Stimulating Hormone (FSH) to carry out on target tissues. The purpose of this study was to identify polymorphism of the Follicle Stimulating Hormone Receptor gene in Bos javanicus, Bos taurus, and Bos indicus Cattle. Blood samples used 207 samples consisting of Bos javanicus (Bali Cattle) 59 heads, Bos taurus 137 heads (Limousin 57 heads, Simmental 50 heads, Angus 6 heads, and Holstein Friesian (FH) 24 heads), and Bos indicus 11 heads (Brahman 9 heads and Ongole 2 heads). The polymorphism site analysis from digestion with AluI restriction enzyme allowed the genotype identification. AluI restriction enzyme cut the base position at AG|CT in 193 and 243 bp. Genotyping FSHR gene produced two alleles, namely C and G alleles. These allele produced three genotypes respectively, there are CC (243 and 63 bp), GG (193, 63, and 50 bp), and CG (243, 193, 63, and 50 bp). The highest G allele frequency was found in Bos javanicus (Bali cattle), but the lowest in Bos taurus. The highest C allele frecuency was found in Bos taurus. The value of heterozigosity was the highest in Bos indicus and the lowest in Bos javanicus. The result of this study showed that the allele of FSHR gene in Bos taurus and Bos indicus were polymorphic, while in Bos javanicus was monomorphic.
IDENTIFIKASI KERAGAMAN GEN
FOLLICLE STIMULATING
HORMONE RECEPTOR
(FSHR|
Alu
I) PADA SPESIES SAPI
Bos javanicus
,
Bos taurus
, DAN
Bos indicus
DENGAN METODE PCR-RFLP
SEPTYANINGTYAS ANGGIA SARI D14080028
Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada
Fakultas Peternakan Institut Pertanian Bogor
DEPARTEMEN ILMU PRODUKSI DAN TEKNOLOGI PETERNAKAN FAKULTAS PETERNAKAN
KATA PENGANTAR
Puji dan syukur senantiasa penulis dipanjakan kepada Allah SWT atas segala
rahmat dan karunia-Nya sehingga penelitian dan penulisan skripsi ini dapat
terselesaikan. Skripsi yang berjudul Identifikasi Keragaman Gen Follicle Stimulating Hormone Receptor (FSHR|AluI) pada Spesies Sapi Bos javanicus, Bos taurus, dan Bos indicus dengan Metode PCR-RFLP ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Peternakan pada Fakultas Peternakan, Institut Pertanian
Bogor.
Upaya perbaikan produktivitas ternak khususnya dalam reproduksi dapat
dilakukan melalui seleksi menggunakan penciri genetik pada level DNA. Sifat
reproduksi ternak dikontrol oleh banyak gen salah satunya adalah gen FSHR. Gen
FSHR memiliki produk bernama FSHR yang berfungsi membawa hormon FSH ke sel
target. Interaksi antara keduanya berfungsi dalam proses spermatogenesis dan oogenesis
pada sapi yang berhubungan dengan reproduksi ternak. Teknik PCR-RFLP dapat
digunakan untuk mendeteksi adanya keragaman gen FSHR tersebut.
Manfaat dari penelitian ini untuk memberikan informasi mengenai identifikasi
keragaman gen FSHR menggunakan metode PCR-RFLP dengan enzim rektriksi AluI
pada spesies sapi Bos javanicus, Bos taurus, dan Bos indicus. Informasi mengenai
keragaman gen FSHR diharapkan menjadi dasar seleksi pada level DNA. Penulis sangat
menyadari penulisan skripsi ini masih jauh dari sempurna, namun penulis berharap
semoga skripsi ini dapat memberikan manfaat bagi dunia peternakan. Amin.
Bogor, Juli 2012
RIWAYAT HIDUP
Penulis dilahirkan di kota Depok pada tanggal 25 September 1990. Penulis
adalah anak pertama dari dua bersaudara pasangan Bapak Imam Sudarmo dan Ibu Sri
Indrayati Faidah. Pendidikan taman kanak-kanak penulis diselesaikan di TK Panca
Daya Depok pada tahun 1996. Sekolah dasar diselesaikan pada tahun 2002 di SD
Negeri Mekarjaya 8 Depok. Pendidikan menengah pertama diselesaikan pada tahun
2005 di SMP Negeri 4 Depok dan pendidikan menengah atas diselesaikan pada tahun
2008 di SMA Al-Ma’soem Bandung.
Penulis diterima sebagai mahasiswa Fakultas Peternakan, Departemen Ilmu
Produksi dan Teknologi Peternakan, Institut Pertanian Bogor melalui jalur Undangan
Seleksi Masuk IPB (USMI) pada tahun 2008. Selama mengikuti pendidikan, penulis
aktif di berbagai kepanitiaan dan organisasi. Penulis aktif dalam Tim Manajerial
Olahraga Fakultas Peternakan sebagai Manajer Badminton Fapet IPB periode
2010-2012, penulis juga menjadi pengurus Badan Eksekutif Mahasiswa (BEM) Fakultas
Peternakan sebagai sekertaris departemen Informasi dan Komunikasi (Infokom)
periode 2010-2011, penulis menjadi anggota Himaproter, anggota Animal Breeding
and Genetic Student Community (ABGSCi) periode 2011-2012, serta menjadi
anggota paguyuban mahasiswa Bandung (Pamaung).
Penulis pernah mengikuti Program Kreativitas Mahasiswa (PKM) di bidang
penelitian yang dibiayai Dikti dengan judul “Penggunaan Gen Cyt-b sebagai
Pendeteksi Adanya Kandungan Gelatin Babi pada Produk Permen Jelly”. Penulis
juga menjadi asisten praktikum mata kuliah Teknologi Pengolahan Telur dan Daging
Unggas (TPTDU) tahun ajaran 2011-2012. Penulis berkesempatan menjadi penerima
beasiswa PPA (Peningkatan Prestasi Akademik) pada tahun 2010-2011 dan
DAFTAR ISI
Halaman
RINGKASAN………. ii
ABSTRAK ……… iii
LEMBAR PERNYATAAN ……….... iv
LEMBAR PENGESAHAN ………. v
Asal Usul Sapi di Indonesia ……… Keragaman Genetik ……….. Gen Follicle Stimulating Hormone Receptor (FSHR) ……… Polymerase Chain Reaction – Restrictiom Fragment Length Polymorphism (PCR-RFLP) ……….... Amplifikaasi Gen FSHR|AluI ……… Elektroforesis ………
Rancangan dan Analisis Data……… Frekuensi Alel dan Genotipe ………
ix
Heterozigositas ……… 14
HASIL DAN PEMBAHASAN ……… 15
Amplifikasi Gen FSHR ………. Keragaman Gen FSHR|AluI ………... Frekuensi Genotipe dan Frekuensi Alel Gen FSHR|AluI ………… Heterozigositas ………
15 16 18 22
KESIMPULAN DAN SARAN ………. 24
Kesimpulan ……… Saran ………...
24 24
UCAPAN TERIMAKASIH ………. 25
DAFTAR PUSTAKA ……… 26
DAFTAR TABEL
Nomor Halaman
1. Karakteristik Bangsa Sapi pada Beberapa Spesies……… 4
2. Jumlah Sampel yang Digunakan dalam Penelitian ……….. 9
3. Frekuensi Genotipe dan Frekuensi Alel Gen FSHR|AluI ………….... 18
4. Frekuensi Alel Gen FSHR|AluI dari Beberapa Sumber ………. 21
5. Nilai Heterozigositas Pengamatan (Ho) dan Harapan (He)
xi
DAFTAR GAMBAR
Nomor Halaman
1. Fenotipik Berbagai Bangsa Sapi (a) sapi Bali, (b) sapi Limousin,
(c) sapi Simmental (d) sapi Angus, (e) sapi FH, (f) sapi Brahman
dan (g) sapi PO ………... 5
2. Struktur Gen FSHR ……….………. 7
3. Fragmen Gen FSHR|AluI berdasarkan GenBank ……… 13
4. Visualisasi Amplifikasi PCR Fragmen Gen FSHR menggunakan Gel Agarose 1,5% ………..
15
5. Hasil Visualisasi PCR-RFLP Gen FSHR|AluI dalam Gel
Agarose 2% ……… 17
DAFTAR LAMPIRAN
Nomor Halaman
xiii
PENDAHULUAN Latar Belakang
Sapi-sapi yang tersebar luas di Indonesia terbagi menjadi tiga kriteria yaitu
sapi asli Indonesia, sapi impor, dan sapi yang sudah beradaptasi (Sarbaini, 2004).
Secara garis besar, spesies sapi yang tersebar di Indonesia terdiri dari spesies sapi
Bos taurus yang merupakan sapi asal Eropa dan Afrika Barat, spesies Bos indicus
yang berasal dari wilayah Asia dan Afrika Selatan, serta Bos javanicus yang
merupakan spesies asli Indonesia yang berasal dari domestikasi Banteng
(Rahmatullah, 2011). Spesies Bos taurus dan Bos indicus merupakan spesies sapi
yang di impor ke Indonesia dan beberapa diantaranya telah dapat beradaptasi dengan
lingkungan di Indonesia seperti sapi FH. Sapi impor tersebut kemudian disebar
keseluruh wilayah Indonesia untuk disilangkan dengan sapi lokal Indonesia dan
membentuk bangsa sapi baru seperti sapi Peranakan Ongole.
Kebutuhan akan pangan dan produksi dari ternak sapi di Indonesia menjadi
perlu untuk mempertahankan secara optimal sumber daya genetik ternak terutama
ternak asli Indonesia. Departemen Pertanian (2006) menyatakan jika sumber daya
genetik ternak merupakan substansi ternak yang secara genetik unik, terbentuk dari
proses domestikasi yang memiliki nilai potensial serta dapat dimanfaatkan dan
dikembangkan dengan baik untuk menciptakan rumpun atau galur unggul. Sapi Bali
merupakan salah satu sumberdaya genetik ternak asli Indonesia yang menjadi plasma
nutfah nasional. Keberadaan sumberdaya genetik ternak perlu dipertahankan dan
dimanfaatkan. Oleh karena itu, perlu adanya upaya peningkatan mutu genetik ternak
Upaya yang dapat dilakukan untuk meningkatkan mutu genetik ternak sapi
adalah melalui seleksi dan persilangan. Seleksi dapat dilakukan berdasarkan sifat
reproduksi dari masing-masing ternak. Pemanfaatan penerapan teknologi molekuler
dapat digunakan sebagai metode seleksi pada tingkat DNA, dalam hal ini DNA yang
berperan dalam sifat reproduksi. Salah satu gen yang memiliki peranan penting
dalam reproduksi adalah gen FSHR (Follicle Stimulating Hormone Receptor). Gen
ini sangat penting dalam proses pembentukan sperma pada sapi jantan dan ovum
pada sapi betina. Gen ini terletak pada kromosom ke 11 yang terdiri dari 10 ekson
dan 9 intron (Aguirre dan Timossi, 1998). Teknik yang dapat digunakan untuk
2 Chain Reaction) dengan RFLP (Restriction Fragment Length Polymorphism).
Metode RFLP menggunakan enzim pemotong AluI untuk mengidentifikasi
polymorfisme gen FSHR pada ekson 10 dengan menggunakan primer forward dan
reverse berdasarkan Houde et al. (1994). Apabila polymorfisme yang terjadi pada
gen FSHR berhubungan dengan sifat produksi dan reproduksi sapi, maka dapat
dijadikan sebagai Marker Assisted Selection (MAS). Untuk dapat memenuhi kriteria
seleksi MAS maka perlu terlebih dahulu dilakukan identifikasi keragaman gen.
Melalui seleksi MAS diharapkan ketepatan seleksi dapat lebih baik sejak periode
anakan.
Tujuan
Penelitian ini bertujuan untuk mengetahui keragaman gen Follicle
Stimulating Hormone Receptor (FSHR|Alu I) dengan menggunakan metode
TINJAUAN PUSTAKA Asal Usul Sapi di Indonesia
MacHugh (1996) menyatakan jika terdapat dua spesies sapi yang tersebar
diseluruh dunia yaitu spesies tidak berpunuk dari Eropa, Afrika Barat, dan Asia
Utara yang disebut dengan spesies Bos taurus, serta spesies sapi berpunuk dari Asia
Selatan dan Afrika yang disebut dengan Bos indicus. Perbedaan antara keduanya
terletak dari ada/tidaknya punuk, cara beradaptasi (Bos indicus lebih mendiami
daerah kering dan panas, sedangkan Bos taurus di daerah beriklim sedang dengan
curah hujan cukup).
Sapi yang terdapat di Indonesia terbagi menjadi : 1) sapi asli Indonesia, 2)
sapi impor, dan 3) sapi yang telah beradaptasi (Sarbaini, 2004). Kusdiantoro et al.
(2007) menyatakan pejantan sapi Ongole di-impor ke Indonesia pada akhir abad
ke-19 ke pulau Jawa dan beberapa pulau lainnya di Indonesia kecuali pulau Bali dan
Madura. Utoyo (2002) menjelaskan penyebaran sapi Ongole pada abad ke-20
dimaksudkan agar dapat disilangkan dengan sapi asli jawa yang akhirnya
membentuk bangsa sapi peranakan ongole dan sapi Madura. Sapi domestikasi yang
ada di Indonesia merupakan sapi spesies bibos dan sapi silangan yang berbeda
dengan sapi domestikasi di Afrika dan Eropa (Jakaria, 2008).
Menurut Rollinson (1984) sapi Bali (Bibos sondaicus) berasal dari hasil
domestikasi banteng liar (Bibos banteng). Proses domestikasi sapi Bali itu terjadi
sebelum 3.500 SM di Indonesia atau Indochina. Banteng liar saat ini bisa ditemukan
di Jawa bagian Barat dan bagian Timur, di Pulau Kalimantan, serta ditemukan juga
di Malaysia (Payne dan Rollinson 1973). Penyebaran sapi Bali di Indonesia dimulai
pada tahun 1890 dengan adanya pengiriman ke Sulawesi, pengiriman selanjutnya
dilakukan pada tahun 1920 dan 1927. Pada tahun 1964 di Bali terjadi musibah
penyakit jembrana secara besar-besaran yang menyebabkan sapi Bali tidak boleh
dikeluarkan lagi dari pulau Bali sebagai ternak bibit. Mulai periode inilah sumber
bibit sapi Bali bagi daerah lain di Indonesia digantikan oleh NTT, Sulawesi Selatan
dan NTB (Talib, 2002).
Bangsa sapi dari spesies Bos taurus yang banyak dimanfaatkan di Indonesia
4 Bos indicus yang banyak dimanfaatkan adalah sapi Peranakan Ongole dan Brahman.
Karakteristik bangsa sapi dari berbagai spesies tercantum pada Tabel 1.
Tabel 1. Karakteristik Berbagai Bangsa Sapi pada Tiga Spesies
Spesies Bangsa Sapi Karakteristik
Bos javanicus
Sapi Bali Ciri sapi ini yaitu memiliki warna bulu merah bata kecoklatan hingga berumur 6 bulan dan akan berubah warna (bagi jantan) menjadi coklat tua hingga hitam mencapai dewasa. Sapi ini memiliki warna putih pada bagian pantat, bagian kaki (white stocking) hingga di atas kuku (Williamson dan Payne, 1993). Presentase karkas tinggi (55,85-60,80%), tingkat fertilitas tinggi (83-86%), serta angka kelahiran tinggi (72,6-92,6%) (Martodjo, 1990).
Bos taurus Limousin Sapi ini memiliki karakteristik warna bulu merah keemasan, kaki dan lutut kebawah memiliki warna merah agak muda, dan umumnya terdapat lingkaran berwarna merah muda disekeliling mata (Pane, 1986).
Simmental Karakteristik sapi ini memiliki warna bulu kecoklatan hingga sedikit merah, bulu muka berwarna putih dan bagian ekor serta lutut kebawah berwarna putih pula. Ukuran tanduk tidak begitu besar (Pane, 1986).
Angus Sapi Angus memiliki karakteristik kulit berwarna hitam, tidak bertanduk, tubuh rata, lebar dan dalam, seperti balok, padat dengan urat daging yang baik. Berat badan betina dewasa mencapai 1600 pounds sedang jantan dewasa 2000 pounds (Pane, 1986).
FH Sapi perah ini memiliki warna putih dengan belang hitam, hitam belang putih, sampai warna hitam. Sapi ini merupakan bangsa sapi perah dengan produksi susu paling tinggi (Syarif dan Sumopastowo, 1984).
Bos indicus Brahman Sap ini memiliki karakteristik ponok besar dan kulit yang longgar dengan lipatan kulit dibawah leher dan perut yang lebar, telinga menggantung. Warna kulit merah kecoklatan dengan tanduk melengkung keatas (Balai Pembibitan Ternak Potong Nusa Tenggara Timur, 2009).
5 Perbedaan karakteristik fenotipik dari berbagai bangsa sapi dapat terlihat jelas
dengan melihat gambar. Gambar 1 merupakan gambar fenotipik berbagai bangsa sapi
dari spesies sapi Bos javanicus, Bos taurus, dan Bos indicus.
(a) (b)
(c) (d)
(e) (f)
(g)
6
Keragaman Genetik
Sumber daya genetik yang semakin beragam menurut Frankham et al.(2002)
akan semakin tahan populasi tersebut untuk hidup dalam jangka waktu yang lama,
serta semakin tinggi daya adaptasi populasi terhadap perubahan lingkungan. Estimasi
perhitungan keragaman genetik dalam populasi secara kualitatif dapat diperoleh
melalui dua ukuran keragaman variasi populasi yaitu proporsi lokus polimorfisme
dalam populasi dan rata-rata proporsi individu heterozigot dalam lokus (Nei, 1987).
Keragaman genetik dalam antara subpopulasi dapat diketahui dengan melihat
persamaan dan perbedaan frekuensi alel di antara subpopulasi (Li et al., 2000).
Falconer dan Mackay (1996) menyatakan jika suatu alel dinyatakan polimorfik jika
memiliki frekuensi alel sama dengan atau kurang dari 0,99.
Noor (2000) menyatakan jika frekuensi genotipe suatu populasi yang cukup
besar akan selalu dalam keadaan seimbang bila tidak ada seleksi, migrasi, mutasi dan
genetic drift, selain itu silang dalam dan silang luar juga dapat mempengaruhi
frekuensi genotipe. Derajat heterozigositas merupakan rataan presentase lokus
heterozigositas tipe individu atau rataan presentase individu heterozigot dalam
populasi (Nei, 1987). Avise (1994) menyatakan jika semakin tinggi derajat
heterozigositas dalam suatu populasi maka daya tahan populasi tersebut akan
semakin tinggi. Javanmard et al. (2005) menyatakan jika nilai heterozigositas
dibawah 0,5 (50%) mengindikasikan rendahnya variasi suatu gen dalam populasi.
Machado et al. (2003) menyatakan jika nilai Ho lebih rendah dari He
mengindikasikan adanya proses seleksi yang intensif.
Gen Follicle Stimulating Hormone Receptor (FSHR)
Follicle stimulating hormone (FSH) adalah hormon yang disintesa dan
disekresikan oleh gonadotropes di kelenjar anterior pituitary. Fungsi utama FSH
adalah menstimulasi pertumbuhan dan pematangan folikel de graaf di dalam ovarium
dan spermatogenesis di dalam tubuli seminiferi testis. Proses spermatogenesis
meliputi proses spermatocytogenesis (pembentukan spermatosit primer dan
sekunder) dan spermiogenesis (proses pembentukan spermatozoa) (Toelihere, 1979).
Keberadaan hormon FSH sangat dipengaruhi oleh adanya gen FSH dan gen
FSHR. Gen FSH terbagi atas dua subunit yaitu α-subunit dan β-subunit dengan