DINAMIKA PERTUMBUHAN BAKTERI DAN IDENTIFIKASI
BAKTERI ASAM LAKTAT TERKULTUR YANG DOMINAN
SELAMA FERMENTASI TEMPE PADA DUA INDUSTRI
RUMAH TANGGA YANG BERBEDA
ALLIA LAKSMI NURDINI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Dinamika Pertumbuhan Bakteri dan Identifikasi Bakteri Asam Laktat Terkultur yang Dominan selama Fermentasi Tempe pada Dua Industri Rumah Tangga yang Berbedaadalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Februari 2015
Allia Laksmi Nurdini
RINGKASAN
ALLIA LAKSMI NURDINI. Dinamika Pertumbuhan Bakteri dan Identifikasi Bakteri Asam Laktat Terkultur yang Dominan selama Fermentasi Tempe pada Dua Industri Rumah Tangga yang Berbeda. Dibimbing oleh LILIS NURAIDA dan ANTONIUS SUWANTO.
Tempe merupakan makanan fermentasi tradisional Indonesia yang telah dikenal secara global. Kultur starter utama pada fermentasi tempe ialah Rhizopus oligosporus, namun beberapa penelitian telah mendeteksi adanya bakteri asam laktat (BAL) pada tempe dan mikroorganisme lainnya. Pada umumnya, tempe diproduksi pada industri rumah tangga dengan skala kecil dan proses fermentasi yang kurang terkontrol, sehingga pada fermentasi tempe terdapat berbagai jenis mikroorganisme.
Penelitian ini bertujuan untuk mengevaluasi keberadaan, pertumbuhan dan interaksi bakteri asam laktat, spora bakteri dan Enterobacteriaceae; serta untuk mengidentifikasi keberadaan bakteri asam laktat (BAL) terkultur dominan dengan T-RFLP pada dua industri rumah tangga di Bogor dengan metode produksi yang berbeda. Sampel diambil dari tujuh tahap produksi dari dua metode produksi tempe yang berbeda yaitu produksi dengan satu kali perebusan sebelum perendaman kedelai dan dua kali perebusan sebelum dan sesudah proses perendaman kedelai. Analisis dilakukan dengan metode perhitungan koloni pada media sesuai dengan bakteri yang akan dianalisis. Identifikasi BAL dilakukan dengan terminal restriction fragment length polymorphism (T-RFLP) terhadap koloni BAL dominan yang diisolasi dari media MRSA natrium azida dari tiap tahap fermentasi. Analisis T-RFLP menggunakan primer 16S rRNA dan enzim restriksi HhaI.
Proses yang menerapkan perebusan kedua sebelum inokulasi memiliki jumlah BAL pada tahap awal fermentasi jauh lebih rendah daripada proses yang menerapkan satu kali perebusan sebelum perendaman kedelai. Hasil ini mengindikasikan BAL telah terlibat sejak proses perendaman dan terbawa ke tahap fermentasi kapang. Hasil analisis T-RFLP juga menunjukkan pada proses dengan satu kali perebusan sebelum perendaman, BAL pada tahap setelah perendaman kedelai terbawa selama fermentasi tempe dan menjadi dominan.
Jumlah BAL yang tinggi pada tahap awal fermentasi berkorelasi dengan pH yang rendah pada tahap awal fermentasi dan dapat berkontribusi terhadap penghambatan pertumbuhan Enterobacteriaceae dan spora bakteri. Keberadaan BAL hingga 8 log cfu/g selama fermentasi mengindetifikasikan bakteri tersebut berkontribusi terhadap proses fermentasi tempe.
Hasil analisis T-RFLP mengindikasikan keberadaan BAL terkultur yang dominan yang sama selama proses fermentasi tempe pada industri rumah tangga dengan perebusan satu kali sebelum perendaman. BAL tersebut diprediksi sebagai
Lactobacillus reuteri, Lactobacillus fermentum dan Lactobacillus sp. Pada industri rumah tangga yang melakukan perebusan kedua setelah perendaman, jenis BAL terkultur yang dominan memiliki variabilitas yang lebih tinggi selama fermentasi tempe yaitu Enterococcus pseudoavium, Enterococcus sulfureus dan
ke-24 dan 72; Lactobacillus sp. pada fermentasi jam ke-0; P. pentosaceus pada fermentasi jam ke-12; Pediococcus pentosaceus dan Weissella kandleri pada fermentasi jam ke-48.
Hasil penelitian menunjukkan bahwa dua persiapan fermentasi tempe yang berbeda menyebabkan komposisi mikroba yang berbeda dan juga berkontribusi terhadap perbedaan BAL terkultur yang mendominasi selama fermentasi sebagaimana yang diidentifikasi dengan T-RFLP. Penelitian ini merupakan penelitian awal untuk mengidentifikasi BAL terkultur dominan selama fermentasi tempe dengan T-RFLP. Untuk studi selanjutnya, diperlukan konfirmasi metode identifikasi untuk mendapatkan hasil yang optimal.
SUMMARY
ALLIA LAKSMI NURDINI. Bacterial Growth Dynamics and Identification of Culturable Dominant Lactic Acid Bacteria during Tempe Fermentation in Two Different Home Industries. Supervised by LILIS NURAIDA dan ANTONIUS SUWANTO.
Tempe is a traditional fermented food of Indonesia that has been recognised globally. The main starter culture for tempe fermentation is Rhizopus oligosporus. However, several researchers have detected lactic acid bacteria (LAB) and other microorganisms from tempe. In general, tempe is produced by small scale at home industries with poorly controlled fermentation process, therefore there were varied kind of microrganisms in tempe fermentation.
The present research aimed to evaluate the presence, growth dynamics and interactions of bacteria; and to identify the culturable dominant LAB by T-RFLP during tempe fermentation in two different home industries in Bogor. Samples were collected from seven production stages of two different tempe production method applied only one boiling step before soaking, and applied two boiling steps before and after soaking process.
Observations were carried out by employing colony counting on media correspond with the microbes that would be enumerated. Identification of LAB performed by terminal restriction fragment length polymorphism (T-RFLP) of the dominant LAB colonies isolated from MRSA sodium azide medium from each fermentation stage. T-RFLP analysis was performed using 16S rRNA primer pairs and HhaI restriction enzyme.
In process applying second boiling step before inoculation, the number of LAB at initial step was much lower than that of applying one boiling step before soaking. The results indicated that LAB has involved since soaking process and they may be carried over into mould fermentation stage. The result of T-RFLP analysis also showed in process applying one boiling steps before soaking, LAB from after soaking steps were carried over and become dominant during tempe fermentation.
High number of LAB at initial step also correlated with low pH at initial step and this may also contribute to inhibition of the growth of
Enterobacteriaceae and bacterial spores. The presence of LAB up 8 log cfu/g during fermentation indicated that those microorganisms contribute to fermentation process of tempe.
T-RFLP analysis results indicated the presence of similar culturable dominant LAB throughout tempe fermentation in the home industry applying one boiling step before soaking identified as L. reuteri, L. fermentum, Lactobacillus
sp.. In home industry applying boiling step after soaking, higher variability of culturable dominant LAB was found during tempe fermentation. T-RFLP analyses showed that Enterococcus pseudoavium, Enterococcus sulfureus, and
The results revealed that two different preparation of tempe fermentation caused different microbial composition and lead to different culturable LAB dominated fermentation process as identified using T-RFLP. This was a preliminary research for identification culturable dominant LAB during tempe fermentation. For future study the identification methods were needed to be confirmed to obtain optimum results.
Keywords: bacterial spores, Enterobacteriaceae, lactic acid bacteria, T-RFLP, tempe
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Pangan
DINAMIKA PERTUMBUHAN BAKTERI DAN IDENTIFIKASI
BAKTERI ASAM LAKTAT TERKULTUR YANG DOMINAN
SELAMA FERMENTASI TEMPE PADA DUA INDUSTRI
RUMAH TANGGA YANG BERBEDA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
Judul Tesis : Dinamika Pertumbuhan Bakteri dan Identifikasi Bakteri Asam Laktat Terkultur yang Dominan selama Fermentasi Tempe pada Dua Industri Rumah Tangga yang Berbeda
Nama : Allia Laksmi Nurdini NIM : F251114091
Disetujui oleh Komisi Pembimbing
Prof Dr Ir Lilis Nuraida, MSc Ketua
Prof Dr Ir Antonius Suwanto, MSc Anggota
Diketahui oleh
Ketua Program Studi Ilmu Pangan
Prof Dr Ir Ratih Dewanti-Hariyadi, MSc
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis ucapkan kepada Allah SWT atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tesis dengan judul “Dinamika Pertumbuhan Bakteri dan Identifikasi Bakteri Asam Laktat Terkultur yang Dominan selama Fermentasi Tempe pada Dua Industri Rumah Tangga yang Berbeda” ini dilaksanakan pada bulan Juni 2013 hingga Agustus 2014.
Dalam kesempatan ini, penulis mengucapkan terima kasih dan penghargaan kepada Prof Dr Ir Lilis Nuraida, MSc selaku ketua komisi pembimbing dan Prof Dr Ir Antonius Suwanto, MSc selaku anggota komisi pembimbing atas bantuannya serta waktu dan kesempatan yang telah diluangkan dalam memberikan bimbingan, ilmu, arahan, motivasi dan masukan selama penulis mengikuti pendidikan, penyusunan proposal, pelaksanaan penelitian, pembuatan artikel jurnal hingga penyusunan tesis. Di samping itu, terima kasih kepada Dr Ir Harsi D. Kusumaningrum atas masukannya selaku penguji luar komisi pembimbing pada ujian tesis dan Dr Suliantari, MSc atas saran dan kontribusi beliau pada awal penelitian ini.
Penghargaan penulis sampaikan kepada Direktorat Jenderal Pendidikan Tinggi melalui Hibah Kompetensi atas nama Prof Dr Ir Lilis Nuraida, MSc pada tahun 2013-2014, yang telah mendanai sebagian besar penelitian ini. Ucapan terima kasih ditunjukkan kepada Lembaga Pengelola Dana Pendidikan (LPDP) Kementrian Keuangan yang mendanai sebagian penelitian ini dan Direktorat Pendidikan Tinggi yang telah memberikan beasiswa kepada penulis dari September 2012 hingga Agustus 2013 melalui program Beasiswa Unggulan. Ucapan terima kasih juga ditunjukkan kepada SEAFAST (Southeast Asian Food and Agricultural Science and Technology) Center untuk fasilitas penelitian.
Penulis menyampaikan terima kasih kepada Mbak Ari dan Mas Yeris atas bantuan dan kerjasama pada penelitian ini, serta pengrajin industri tempe Sindang Barang dan Warung Jambu atas keramahan selama proses pengambilan sampel. Di samping itu, terima kasih penulis sampaikan kepada Ibu Esti Puspitasari, Redi dan CSR PT Wilmar Benih Cikarang untuk dukungannya terhadap analisis fragmen DNA yang dipotong dengan enzim HaeIII. Terima kasih kepada Pak Yepy dari PT Wilmar Benih Cikarang untuk masukan terhadap analisis molekukar serta Ibu Efriwati, Ibu Nurjana dan Ibu Cecil dari Program Studi Mikrobiologi, IPB untuk masukan penelitian.
Di samping itu, terima kasih penulis sampaikan kepada Mbak Desty dan Teh Asih dari SEAFAST IPB, Ibu Emi dan Sepri dari IPB-CC, Dini, Tika, serta seluruh rekan-rekan yang telah membantu selama proses penelitian ini berlangsung yang tidak bisa disebutkan satu per satu.
Ungkapan terima kasih yang sebesar-besarnya juga penulis ucapkan kepada Ibunda Ibu Herlina dan ayahanda Bambang Mulyanto dan juga adik penulis M. Wira Baskoro. Terima kasih tak lupa penulis ucapkan kepada seluruh keluarga dan sahabat atas segala doa, dukungan, serta kasih sayang yang telah diberikan.
Semoga karya ilmiah ini bermanfaat bagi ilmu pengetahuan.
Bogor, Februari 2015
DAFTAR ISI
PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 3
Manfaat Penelitian 3
Hipotesis 3
TINJAUAN PUSTAKA 4
Proses Produksi Tempe 4
Mikroorganisme yang Berasosiasi selama Fermentasi Tempe 4
Bakteri Asam Laktat pada Tempe 5
Identifikasi BAL pada Tempe 7
Terminal Restriction Fragment Length Polymorphism (T-RFLP) 8
METODE 10
Waktu dan Tempat 10
Bahan 10
Alat 10
Tahapan Penelitian 11
Prosedur Kerja 12
HASIL DAN PEMBAHASAN 20
Perbedaan Proses Produksi Tempe pada Industri Rumah Tangga 20
SDBR dan WJB 20
Populasi Mikroba pada Laru Tempe Proses SDBR dan WJB 21
Total Bakteri 22
Bakteri Asam Laktat 23
Total Spora Bakteri dan Enterobacteriaceae 25
Nilai pH dan Total Asam Tertitrasi (TAT) 27
Amplifikasi Gen 16S rRNA dan Purifikasi Produk PCR 28 Seleksi Enzim Restriksi Berdasarkan Analisis T-RFLP BAL Referensi 29 Dinamika BAL Terkultur Dominan selama Fermentasi Tempe 31
Identifikasi TRF BAL Terkultur Dominan 34
KESIMPULAN DAN SARAN 36
Kesimpulan 36
Saran 37
DAFTAR PUSTAKA 37
LAMPIRAN 43
DAFTAR TABEL
1 Akumulasi asam organik (% b/v) pada air perendaman dengan fermentasi alami, inokulasi kultur murni dan back-sloppinga 6 2 Identifikasi dan keberadaan BAL terkultur selama proses fermentasi
tempe SDBR dan WJB 35
DAFTAR GAMBAR
1 Prinsip terminal fragment length polymorphism (Gruntzig et al. 2002) 8
2 Tahapan kerja penelitian 11
3 Titik pengambilan sampel kedelai dan tempe pada dua industri tumah
tangga 12
4 Langkah kerja identifikasi BAL terkultur dominan 15 5 Persiapan sampel DNA untuk identifikasi BAL terkultur domiman 16 6 Alur produksi tempe pada proses SDBR (a) dan (b) proses WJB 20 7 Persiapan laru onggok yang digunakan proses SDBR 21 8 Populasi bakteri pada laru dan onggok. Rataan dengan huruf yang
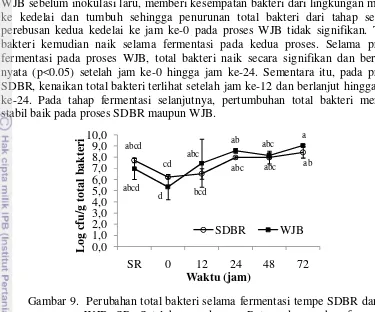
berbeda menunjukkan jumlah mikroba yang berbeda nyata (p<0.05). 22 9 Perubahan total bakteri selama fermentasi tempe SDBR dan WJB.
SR: Setelah perendaman. Rataan dengan huruf yang berbeda menunjukkan jumlah total bakteri yang berbeda nyata (p<0.05). 23 10 Perubahan bakteri asam laktat selama fermentasi tempe SDBR dan
WJB. SR: setelah perendaman. Rataan dengan huruf yang berbeda menunjukkan jumlah BAL yang berbeda nyata (p<0.05). 24 11 Perubahan spora bakteri selama fermentasi tempe SDBR dan WJB.
SR: setelah perendaman. Rataan dengan huruf yang berbeda menunjukkan jumlah total spora bakteri yang berbeda nyata (p<0.05). 25 12 Perubahan Enterobacteriaceae selama fermentasi tempe SDBR dan
WJB. SR: setelah perendaman. Rataan dengan huruf yang berbeda menunjukkan jumlah Enterobacteriaceae yang berbeda nyata
(p<0.05). 26
13 Perubahan pH dan TAT selama fermentasi tempe (a) SDBR dan (b) WJB. SR: setelah perendaman. Rataan dengan huruf yang berbeda pada parameter yang sama menunjukkan nilai pH atau TAT yang
berbeda nyata (p<0.05). 28
14 Hasil amplifikasi gen 16S rRNA BAL terkultur dominan proses (a) SDBR dan (b) WJB. M: DNA ladder, 1: Setelah perendaman, 2-6: fermentasi jam ke -0 hingga jam ke-72, 7: laru onggok, K: BAL
referensi. 29
16 Profil T-RFLP dari BAL terkultur selama proses fermentasi tempe SDBR. L: laru onggok, SR: setelah perendaman, 0-72: waktu
fermentasi (jam). 32
17 Distribusi dan kelimpahan relatif TRF bakteri selama proses fermentasi tempe SDBR. S: SDBR, SR: setelah perendaman, SL: laru
onggok, 0-72: waktu fermentasi (jam). 32
18 Distribusi dan kelimpahan relatif TRF bakteri selama proses fermentasi tempe WJB. W: WJB, SR: setelah perendaman, 0-72:
waktu fermentasi (jam). 33
19 Profil T-RFLP dari BAL terkultur selama proses fermentasi tempe WJB. SR: Setelah perendaman, 0-72: waktu fermentasi (jam). 34
DAFTAR LAMPIRAN
1 Hasil analisis statistika perhitungan bakteri pada laru dan onggok 44 2 Hasil analisis statistika perhitungan total bakteri selama fermentasi
tempe SDBR dan WJB 44
3 Hasil analisis statistika perhitungan BAL selama fermentasi tempe
SDBR dan WJB 45
4 Hasil analisis statistika perhitungan spora bakteri selama fermentasi
tempe SDBR dan WJB 45
5 Hasil analisis statistika perhitungan Enterobacteriaceae selama
fermentasi tempe SDBR dan WJB 46
6 Hasil analisis statistika perhitungan pH selama fermentasi tempe
SDBR dan WJB 47
7 Hasil analisis statistika perhitungan TAT selama fermentasi tempe
1
PENDAHULUAN
Latar Belakang
Tempe merupakan pangan tradisional Indonesia yang terbuat dari kedelai, memiliki flavor campuran kapang dan kedelai, serta tekstur dan daya cerna yang baik. Selama fermentasi tempe, kapang tumbuh menjalin kedelai menjadi bentuk kompak yang dapat dipotong. Tempe diketahui berasal dari Jawa dan telah menjadi konsumsi sehari-hari masyarakat di Indonesia selama ratusan tahun. Kini tempe telah menjadi pangan eksotis yang dikenal luas di mancanegara. Selain nutrisi yang tinggi, tempe juga diketahui sebagai sumber komponen bioaktif seperti antioksidan. Karakter tempe yang unggul ini menjadikan tempe sebagai pangan fermentasi yang disukai oleh banyak kalangan, terutama oleh vegetarian.
Pada umumnya, tempe diproduksi dalam skala kecil pada industri rumah tangga dengan proses fermentasi yang kurang terkontrol sehingga proses fermentasi tidak dilakukan di bawah kondisi aseptik walaupun kultur starter yang mengandung kapang telah ditambahkan pada awal fermentasi. Oleh karena itu, pada fermentasi tempe terdapat berbagai jenis mikroorganisme yang dapat berpartisipasi selama proses fermentasi tempe (Barus et al. 2008; Seumahu et al. 2013). Hal ini menyebabkan variasi pada flavor tempe, contohnya rasa pahit yang terkadang muncul pada tempe (Barus et al. 2008).
Saat ini, keragaman bakteri selama fermentasi tempe telah dipelajari secara ekstensif. Selama perendaman kedelai, BAL telah terdeteksi dalam jumlah yang tinggi (Nuraida et al. 2008; Efriwati et al. 2013). BAL yang terdapat dalam jumlah tinggi selama perendaman kedelai dapat mengasamkan kedelai melalui proses fermentasi alami (Mulyowidarso et al. 1989). Perendaman kedelai diketahui dapat membantu mengendalikan populasi mikroba pada tempe. Saat akhir proses perendaman kedelai, Enterobacteriaceae tidak bisa beradaptasi dengan kondisi asam karena asam organik yang dihasilkan oleh BAL sehingga mengakibatkan jumlah Enterobacteriaceae menurun (Mulyowidarso et al. 1989). Di samping itu, Seumahu (2012) telah mendeteksi Enterobacteriaceae pada tempe yang diproduksi pada industri rumah tangga. Salah satu jenis Enterobacteriaceae
yaitu Salmonella, juga dapat mengontaminasi kultur starter tempe seperti yang telah dilaporkan oleh FDA (2012).
Nuraida et al. (2008) melaporkan bahwa BAL meningkat selama fermentasi tempe dan mencapai jumlah sebesar 106 cfu/g. Selama fermentasi tempe, peran mikroorganisme selain kapang masih perlu dipelajari lebih lanjut. BAL dilaporkan memiliki peran dalam mengontrol bakteri patogen selama produksi tempe. Suatu penelitian menunjukkan bahwa keberadaan Lactobacillus plantarum pada kedelai yang sengaja diinokulasi dengan bakteri patogen dapat menghambat pertumbuhan
Listeria monocyotgenes dan produksi toksin stafilokokal saat fermentasi tempe (Nout dan Kiers 2005). Seumahu (2012) juga melaporkan bahwa
2
Keragaman bakteri yang berbeda dalam fermentasi tempe tradisional akan menyebabkan inkonsistensi mutu tempe. Hal ini akan menyebabkan hambatan dalam pemenuhan standar dan produksi tempe dalam skala besar. Studi dinamika bakteri selama fermentasi tempe tradisional akan membantu pemetaan mikroorganisme yang diinginkan selain kapang selama fermentasi tempe. Sebelumnya, Mulyowidarso et al. (1990) telah melaporkan dinamika mikroba selama fermentasi tempe, namun fermentasi dilakukan di bawah kondisi laboratorium. Penelitian ini melaporkan dinamika bakteri selama fermentasi tempe pada dua industri rumah tangga yang berbeda. Profil mikroorganisme tersebut selama fermentasi tempe akan memberikan gambaran apakah BAL memiliki peran penting selama fermentasi tempe. Informasi ini diperlukan untuk mengembangkan starter tempe multikultural yang diharapkan dapat meningkatkan mutu dan keamanan tempe secara konsisten. Selama proses fermentasi tradisional, jumlah bakteri yang muncul dapat tidak konsisten, menyebabkan beberapa data memiliki standar deviasi yang tinggi.
Sampai saat ini profil BAL terkultur dominan selama fermentasi tempe belum dilaporkan. Sebelumnya Efriwati et al. (2013) telah mengidentifikasi BAL selama fermentasi tempe. BAL tersebut diidentifikasi langsung dari tempe tanpa pengkulturan maupun terlebih dahulu dikulturkan pada media laboratorium. Akan tetapi BAL terkultur yang telah teridentifikasi tersebut belum tentu dominan selama fermentasi tempe. Pada penelitian ini, digunakan teknik T-RFLP untuk mengidentifikasi BAL terkultur dominan pada tempe. TRFLP merupakan metode yang sensitif, mudah, relatif murah dan cepat untuk menentukan profil komunitas mikroba pada pangan fermentasi (Bokulich dan Mills 2012). Sebelumnya Efriwati
et al. (2008) juga melakukan analisis T-RFLP untuk mengidentifikasi BAL terkultur pada tempe. Penggunaan teknik ini dapat mempercepat proses identifikasi karena isolat-isolat BAL yang berasal dari tahap fermentasi yang sama dapat dianalisis sebagai satu sampel.
Perumusan Masalah
Berdasarkan penelitian sebelumnya, selain kapang mikroorganisme lainnya seperti bakteri ikut tumbuh selama fermentasi tempe baik bakteri yang tidak diinginkan seperti Enterobacteriaceae dan spora bakteri, maupun bakteri yang diinginkan seperti bakteri asam laktat. Keberadaan mikroorganisme yang bervariasi selama fermentasi tempe dapat menimbulkan mutu tempe yang tidak konsisten sehingga hal ini dapat menjadi hambatan pada produksi tempe dalam skala besar. Terdapat penelitian yang melaporkan peran BAL terhadap penghambatan bakteri yang tidak diinginkan pada tempe. Oleh karena itu penelitian dinamika bakteri dapat melihat apakah BAL memiliki peran yang signifikan selama fermentasi tempe terutama dalam menekan pertumbuhan
Enterobacteriaceae dan spora bakteri. Informasi ini diperlukan untuk pengembangan kultur starter multikultural. BAL terkultur dominan asal tempe diharapkan dapat dijadikan salah satu komposisi kultur starter multikultural yang dapat memperbaiki mutu dan keamanan tempe.
3 pengaruh terhadap asal, komposisi, dinamika bakteri dan BAL dominan yang berbeda selama fermentasi tempe. Oleh karena itu penelitian ini akan menjawab: 1) apakah perbedaan metode produksi dapat mengakibatkan perbedaan dinamika pertumbuhan BAL, spora bakteri dan Enterobacteriaceae selama fermentasi tempe; 2) Apakah keberadaan BAL dapat berpengaruh terhadap keberadaan
Enterobacteriaceae dan spora bakteri; 3) apakah ada tahap yang berkontribusi terhadap penambahan bakteri ke dalam tempe; dan 4) identitas BAL yang mendominasi selama fermentasi tempe pada kedua metode produksi.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengevaluasi keberadaan dan pertumbuhan bakteri asam laktat, spora bakteri dan Enterobacteriaceae; serta untuk mengetahui identitas bakteri asam laktat (BAL) terkultur dominan dengan teknik T-RFLP pada berbagai tahap produksi tempe pada dua industri rumah tangga di Bogor dengan metode produksi yang berbeda.
Manfaat Penelitian
Penelitian ini memberikan informasi dan data tentang dinamika bakteri yang dapat menjelaskan peran BAL selama fermentasi tempe pada industri rumah tangga. Penelitian ini memberikan informasi awal mengenai identitas BAL terkultur dominan selama fermentasi tempe. Isolat BAL yang dominan tersebut diharapkan dapat diaplikasikan untuk formulasi kultur starter campuran yang dapat berperan untuk meningkatkan mutu tempe.
Hipotesis
1. Metode produksi tempe yang berbeda akan memberikan profil pertumbuhan bakteri yang berbeda serta komposisi BAL terkultur dominan yang berbeda selama fermentasi tempe.
2. Pertumbuhan BAL diduga menurunkan pertumbuhan Enterobacteriaceae dan spora bakteri pada tempe.
4
TINJAUAN PUSTAKA
Proses Produksi Tempe
Berdasarkan SNI (BSN 2009) tempe merupakan produk fermentasi yang terbuat dari kedelai dan kapang Rhizopus oligosporus, memiliki bentuk kompak, berwarna putih keabu-abuan dan memiliki aroma seperti tempe. Standar tempe juga telah ditetapkan pada Codex Alimentarius (CAC 2013) yaitu tempe merupakan produk berbentuk kompak, berwarna putih, membentuk cake, dipreparasi dengan memfermentasikan keledai yang telah dikupas kulit arinya dengan Rhizopus spp. melalui fermentasi substrat padat. Standar mutu organoleptik tempe yang telah ditetapkan oleh Codex Alimentarius antara lain: 1) memiliki tekstur yang kompak dan tidak mudah hancur ketika dipotong dengan pisau, 2) memiliki warna putih dari pertumbuhan Rhizopus spp., 3) memiliki flavor tempe yaitu nutty, meaty dan seperti jamur, 4) memiliki aroma dari tempe segar tanpa bau amonia.
Di Indonesia, tempe biasanya diproduksi dengan metode yang bervariasi. Salah satu metode yang dilakukan meliputi beberapa langkah berikut: (1) perebusan dan perendaman kedelai; (2) pengupasan kulit ari dan pencucian kedelai; (3) perebusan kedua (opsional); (4) inokulasi kultur starter; (5) pengemasan kedelai pada daun pisang atau plastik yang telah dilubangi; (6) dan inkubasi kedelai hingga menjadi tempe (Hermana dan Karmini 1996). Proses sebelum tahap fermentasi dapat mendukung pertumbuhan kapang pada substrat kedelai. Proses perebusan dan perendaman menyebabkan hidrasi kedelai sehingga kotiledon kedelai menjadi lunak. Hidrasi juga menyediakan aw 0.99 yang optimal untuk pertumbuhan kapang. Pengupasan kulit ari dapat melepaskan kulit ari kedelai yang keras sehingga keping kedelai lebih mudah ditembus miselium kapang. Proses pencucian dapat membuang kulit ari yang tertinggal dan mengurangi kelebihan asam dan lendir (Hermana dan Karmini 1996).
Sebelum penambahan kultur starter, kedelai terlebih dahulu mengalami fermentasi spontan selama tahap perendaman. Selama proses perendaman terdapat pertumbuhan mikroba secara cepat yang dapat mengasamkan air perendaman dan kedelai yang direndam. Setelah inokulasi kultur starter, Rhizopus sp. akan berperan sebagai mikroorganisme utama yang memfermentasi tempe. Selama fermentasi, miselium akan tumbuh dan menjalin ikatan pada keping-keping kedelai (Nout dan Kiers 2005). Fermentasi ini dapat berlangsung 18 hingga 48 jam (Mulyowidarso et al. 1989).
Mikroorganisme yang Berasosiasi selama Fermentasi Tempe
Mikroorganisme yang berperan utama selama fermentasi tempe ialah kapang. Menurut Purwijatiningsih et al. (2005), jenis kapang yang terdapat pada kultur starter komersil (laru Raprima) adalah R. oligosporus. Kapang lain dapat tumbuh pada kultur starter lokal seperti A. niger, R. oryzae, Rhizomucor pusillus
5 selama perendaman, proses perebusan, kontaminasi silang selama proses pendinginan, komposisi kultur starter dan lingkungan ketika tempe difermentasikan (Nout dan Kiers 2005). Pertumbuhan cepat bakteri telah dimulai selama tahap perendaman kedelai dan dapat mencapai 4.4x109 cfu/g (Nuraida et al. 2008). Air perendaman kedelai kaya akan gula sederhana seperti glukosa, fruktosa dan galaktosa. Glukosa merupakan substrat utama untuk pertumbuhan mikroba pada air perendaman. Aktivitas enzim invertase dan α-galaktosidase yang dimiliki kedelai menghasilkan gula-gula sederhana pada air perendaman. Mikroba yang dapat ditemukan pada air perendaman ialah bakteri dan khamir (Mulyowidarso et al. 1991).
Kedelai mengandung oligosakarida, lemak dan protein yang dapat didegradasi oleh Rhizopus sp.. Rhizopus sp. memiliki enzim untuk mendagradasi substrat kedelai seperti karbohidrase (poligalakturonase, endoselulase, xilanase dan arabinase), lipase dan protease (Nout dan Kiers 2005). Selama fermentasi tempe dengan kultur tunggal R. oligosporus, terjadi reduksi rafinosa sebesar 60% dan stakiosa sebesar 10%; peningkatan gula tereduksi sebesar 700 mg/100 g; dan peningkatan asam amino bebas sebesar 70 mg/100 g (Egounlety dan Aworh 2003). Diduga, nutrisi hasil metabolisme Rhizopus sp. dapat mendukung pertumbuhan bakteri selama fermentasi tempe.
Bakteri yang muncul selama produksi tempe telah diidentifikasi pada penelitian sebelumnya. Bakteri tersebut berasal dari air perendaman, fermentasi kapang dan kultur starter (Barus 2008; Efriwati 2013; FDA 2012; Keuth dan Bisping 1993; Kim et al. 2006; Klus et al. 1993; Klus dan Barz 1998; Moreno et al. 2002; Mulyowidarso et al. 1989, 1990; Roubos-van den Hil et al. 2010; Seumahu 2012; Touw 2014). Aktivitas bakteri selama fermentasi tempe telah dilaporkan oleh beberapa peneliti baik merupakan bakteri yang diinginkan atau tidak diinginkan. Citrobacter freundii dan Klebsiella pneumoniae yang merupakan kelompok Enterobacteriaceae memiliki kemampuan untuk mensintesis vitamin B12 selama fermentasi tempe. C. freundii dilaporkan dapat mensitesis thiamin pada tempe (Keuth dan Bisping 1993). BAL yang diisolasi dari tempe dilaporkan dapat menghasilkan bakteriosin (Moreno et al. 2002). Aktivitas bakteri tersebut berguna untuk meningkatkan nilai gizi dan keamanan tempe. Akan tetapi, terdapat bakteri yang tidak diinginkan pada tempe seperti Salmonella
dan Bacillus. Salmonella yang termasuk ke dalam kelompok Enterobacteriaceae
diketahui mengontaminasi laru yang diimpor dari Indonesia (FDA 2012). Kasus kontaminasi yang disebabkan oleh Salmonella paratyphi B telah menyebabkan
outbreak di salah negara bagian North Carolina, USA akibat konsumsi tempe yang dibuat dari laru yang terkontaminasi (Griese et al. 2013). Bacillus sp. yang merupakan bakteri pembentuk spora memiliki aktivitas proteolitik yang tinggi (Roubos-van den Hill et al. 2010) yang dapat meningkatkan pH kedelai sehingga menghambat pertumbuhan kapang (Ashenafi dan Busse 1991). Bacillus juga diduga menyebabkan rasa pahit pada tempe (Barus et al. 2008).
Bakteri Asam Laktat pada Tempe
6
Efriwati et al. 2013). Moreno et al. (2002) melaporkan pertumbuhan bakteri asam laktat selama produksi tempe di Malaysia. Bahan baku kedelai memiliki BAL dengan jumlah rendah (< 102 cfu/g). Pada akhir proses perendaman, jumlah BAL meningkat hingga 7.9-9.3 log cfu/ml pada air perendaman dan 7.8-9.2 log cfu/g pada kedelai yang direndam. Perebusan kedua kedelai mereduksi jumlah BAL hinggga menjadi kurang dari 4 log cfu/g. Jumlah ini meningkat tajam hingga 6.8-9.9 log cfu/g pada tempe (Moreno et al. 2002).
Nuraida et al. (2008) dan Efriwati et al. (2013) melaporkan perubahan jumlah BAL selama produksi tempe di Indonesia. Nuraida et al. (2008) melaporkan BAL pada kedelai yang telah mengalami proses perendaman memiliki jumlah 9 log cfu/g. Akan tetapi, jumlah BAL pada tempe berkurang menjadi 6 log cfu/g. Efriwati et al. (2013) melaporkan BAL pada tempe yang diproduksi dengan dua metode yang berbeda yaitu metode dengan satu kali dan dua kali perebusan kedelai. Jumlah BAL pada akhir perendaman pada kedua metode memiliki jumlah 6 log cfu/g. Akan tetapi, jumlah BAL pada tempe dengan metode satu kali perebusan kedelai ialah 8 log cfu/g, lebih besar dibandingkan BAL pada tempe dengan metode dua kali perebusan kedelai yaitu 6.5 log cfu/g.
Menurut Ashenafi dan Busse (1991b), BAL berkontribusi meningkatkan keamanan pada tempe melalui mekanisme penurunan pH, produksi asam organik dan produksi metabolit penghambat lain. Selama proses perendaman, BAL akan memproduksi asam laktat sebagai produk fermentasi utama dan asam organik lainnya dan menurunkan pH air perendaman (Tabel 1) (Nout dan Kiers 2005). Pengasaman alami tersebut akan menurunkan pH kedelai yang dapat mengontrol populasi bakteri (Mulyowidarso et al. 1991b). Pengasaman juga dapat menyediakan lingkungan yang baik untuk germinasi spora kapang dan menurunkan waktu lag R. oligosporus (Nour dan Kiers 2005). Selain asam organik, terdapat laporan oleh Moreno et al. (2002) mengenai BAL penghasil bakteriosin,Enterococcus faecium yang diisolasi dari tempe yang lewat matang (over ripe). Bakteriosin yang dihasilkan E. faecium dapat menghambat pertumbuhan Lysteria monocytogenes, Bacillus pumilus dan Clostridium sporogenes pada media pertumbuhan.
Tabel 1. Akumulasi asam organik (% b/v) pada air perendaman dengan fermentasi alami, inokulasi kultur murni dan back-sloppinga
Fermentasib pH Asam laktat Asam malat Asam asetat
Spontan 4.5 0.6 0.2 0.07
Sumber: Nout dan Kiers (2005), bFermentasi dilakukan dengan perendaman 300 g kedelai di
dalam 900-1000 ml air pada suhu 30oC selama 24 jam tanpa inokulum (fermentasi spontan),
dengan inokulasi 104 cfu/ml kultur murni BAL, atau dengan penambahan 3% v/v air
7 Interaksi antara BAL dan bakteri patogen pada tempe telah dilaporkan pada beberapa penelitian. L. plantarum dan L. brevis yang diisolasi dari air perendaman dapat mereduksi pertumbuhan S. aureus serta produksi toksin stafilokokal pada tempe (Tuncel dan Goktan 1990). Ko-kultur L. plantarum dapat menurunkan jumlah B. cereus sebesar 1 log cfu/g (Ashenafi dan Busse 1991); menghambat pertumbuhan Listeria monocytogenes secara signifikan (Ashenafi 1991a); serta
Salmonella infantis dan E. coli pada tempe (Ashenafi 1991b). Akan tetapi pada laporan tersebut tidak dijelaskan asal isolat L. plantarum yang digunakan.
Identifikasi BAL pada Tempe
Identifikasi BAL pada tempe telah dilakukan beberapa tahun terakhir dengan analisis fenotipik maupun genotipik. Analisis fenotipik meliputi pengamatan morfologi, fisiologis dan biokimia BAL (Touw 2014) sedangkan analisis genotipik untuk mempelajari identitas BAL pada tempe ialah sekuensing gen 16S rRNA (Seumahu 2012) dan T-RFLP (Efriwati 2013). Seumahu (2012) mempelajari keanekaragaman bakteri secara umum pada tempe menggunakan teknik pengklonan gen 16S rRNA dari DNA bakteri tempe yang diekstraksi langsung dari lingkungan. Hasil sekuensing gen 16S rRNA menunjukkan dari 60 jenis bakteri, teridentifikasi empat spesies BAL yaitu Lactobacillus fermentum,
Lactobacillus delbrueckii subsp. delbrueckii, Lactobacillus delbrueckii subsp. bulgaricus dan Lactobacillus mucosae. Barus (2008) mempelajari keanekaragaman bakteri pada tempe menggunakan teknik T-RFLP pada sampel DNA yang diekstraksi langsung dari tempe. Akan tetapi, hasil identifikasi T-RFLP menunjukkan hanya satu spesies yang diduga sebagai Lactobacillus. BAL juga tidak terdeteksi melalui metode sekuensing gen 16S rRNA dari bakteri yang ditumbuhkan pada media umum (Barus 2008).
Touw (2014) telah mengidentifikasi BAL terkultur dominan asal tempe. BAL diisolasi dari medium MRSA (Man de Rogosa Agar) dengan penambahan natrium azida 0.02% (Reina et al. 2005). BAL yang telah diisolasi kemudian diuji secara konvensional dengan pewarnaan Gram, identifikasi morfologi, produksi katalase dan pola fermentasi glukosa (Touw 2014). Identifikasi morfologi dapat mengelompokkan BAL menjadi bentuk batang atau kokus. Cara fermentasi glukosa dapat mengelompokkan BAL menjadi BAL homofermentatif atau heterofermentatif. Beberapa isolat BAL kemudian dipilih dari pengelompokkan bentuk dan pola fermentasi glukosa untuk diidentifikasi secara biokimia menggunakan API 50 CH (Biomerieux, Perancis). API 50 mengidentifikasi BAL berdasarkan kemampuan BAL untuk memfermentasi 49 jenis karbohidrat dan satu kontrol.
8
Terminal Restriction Fragment Length Polymorphism (T-RFLP)
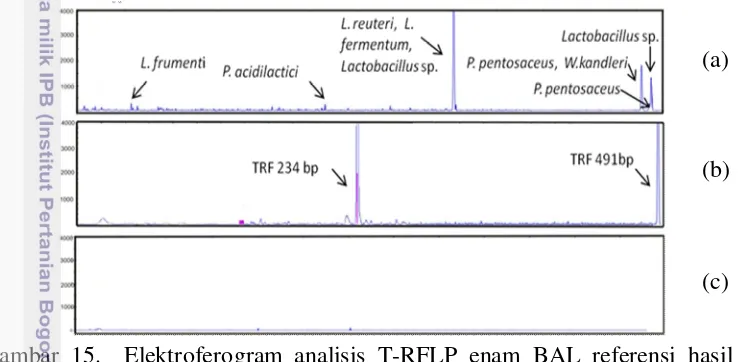
Terminal restriction fragment length polymorphism (T-RFLP) adalah metode sidik jari untuk menganalisis komunitas mikroba. Pada teknik ini, gen target (biasanya gen 16S rRNA) diamplifikasi dengan PCR menggunakan pasangan primer yang diberi label dengan pewarna fluoresens pada bagian terminal salah satu atau kedua primer. Produk PCR kemudian dipotong dengan satu (atau lebih) enzim restriksi yang memotong DNA pada urutan tertentu. Enzim restriksi yang digunakan akan mengenali situs pemotongan spesifik pada 4 pasangan basa gen yang telah diamplifikasi. Hasilnya adalah fragmen DNA dengan berbagai ukuran atau disebut juga terminal restriction fragment (TRF). Hasil pemotongan kemudian dipisahkan dengan alat sekuensing DNA untuk mendeteksi fragmen yang berlabel fluoresens (Rastogi dan Sani 2011). Prinsip kerja T-RFLP dapat dilihat pada Gambar 1.
Data T-RFLP disajikan dalam bentuk elektroferogram yang menunjukkan ukuran fragmen terminal (TRF) dari DNA yang berlabel. Tinggi puncak menunjukkan intensitas fluoresens TRF (Gambar 1). Keragaman komunitas mikroba kemudian dianalisis berdasarkan ukuran, jumlah dan ketinggian TRF. Setiap TRF diasumsikan sebagai OTU (operational taxonomic unit) tunggal atau ribotipe (Rastogi dan Sani 2011).
Data T-RFLP dapat dianalisis menggunakan database dari sekuens DNA yang telah diketahui untuk membuat profil komunitas mikroba. Shyu et al. (2007) telah mengembangkan database berbasis web yang disebut MiCA (Microbial Analysis Community). MiCA merupakan program perangkat lunak yang memungkinkan untuk membandingkan fragmen observasi hasil keluaran mesin sekuensing dengan fragmen prediksi hasil pemotongan DNA secara virtual. Dari fragmen prediksi tersebut kemudian dapat diketahui identitas bakteri dari
database. Dengan program MiCA, juga dapat dilakukan simulasi amplifikasi DNA dan digesti hasil amplifikasi secara virtual.
Gambar 1. Prinsip terminal fragment length polymorphism (Gruntzig
et al. 2002) Ekstrak DNA dari komunitas mikroba
PCR dengan primer forward 16S rRNA yang berlabel fluoresens
Pemotongan produk amplifikasi
9 Keunggulan T-RFLP ialah sensitif, mudah, murah dan cepat untuk menentukan profil mikrobiota pada lingkungan fermentasi (Bokulich dan Mills 2012). Metode T-RFLP dapat mengurangi beban kerja untuk mengidentifikasi koloni pada konsorsium mikroba. Sebagai contoh, 50 sampel DNA dari 50 komunitas mikroba dapat dianalisis pada selama dua hingga tiga hari. Jika setiap komunitas diambil sebanyak 20 koloni, koloni yang diperoleh adalah 1.000 koloni sehingga identifikasi membutuhkan waktu yang lama (Nieminen et al. 2011). Kekurangan metode ini adalah kurang akurasinya dalam mendeteksi komunitas mikroba yang sangat beragam (Li et al. 2007) dan adanya T-RF yang sama yang dapat dihasilkan oleh populasi dengan kekerabatan yang berbeda (Marsh 2000).
Teknik T-RFLP untuk mengidentifikasi struktur BAL pada pangan fermentasi telah dilakukan beberapa peneliti seperti pada tempe (Barus 2008; Efriwati 2013), bir dan wine (Bokulich dan Mills 2012) serta barley selama proses
10
METODE
Waktu dan Tempat
Penelitian ini dilakukan di Laboratorium Mikrobiologi dan Bioteknologi SEAFAST (Southeast Asian Food and Agricultural Science and Technology)
Center, Institut Pertanian Bogor, Juni 2013-Agustus 2014. Analisis T-RFLP dilakukan pada layanan jasa Fragment analysis (1st Base, Singapore) dan pada Laboratorium Bioteknologi, PT Wilmar Benih Indonesia (Cikarang).
Bahan
Bahan utama dalam penelitian ini adalah kedelai dan sampel tempe yang diambil dari berbagai tahapan fermentasi, kultur starter (laru) komersial dengan merk Raprima (laru Raprima), kultur starter subkultur (laru onggok) dan limbah padat hasil produksi tapioka (onggok) yang diambil dari dua industri rumah tangga yang berbeda di Bogor yang selanjutnya disebut sebagai SDBR dan WJB. Bahan untuk analis kimia dan mikrobiologi antara lain 0.1% peptone water
(Oxoid, Bakingstoke, UK), 0.1 N NaOH, asam oksalat, standar fenolftalein, bufer pH 4, 7 dan 10. Media pertumbuhan bakteri: media MRSA (Oxoid, Bakingstoke, UK), 0.02% natrium azida, nutrient agar (NA) (Merck, Darmstadt, Germany) dan violet red-bile agar (VRBA) (Oxoid, Bakingstoke, UK).
Untuk identifikasi BAL digunakan bahan-bahan sebagai berikut: agarosa (Thermo Scientific, USA), etidium bromida, bufer TAE (tris asetat EDTA) pH 8.0, bufer TE (tris EDTA) pH 8.0, bufer Tris-Cl, nuclease free water (Amresco, USA), lisozim (Biobasic, Canada), KAPA universal ladder (KAPA Biosystems, USA), Wizard Genomic DNA Purification Kit (Promega, USA), Gotaq Green Master Mix (Promega, USA), Wizard SV Gel and PCR Clean-Up System (Promega, USA), enzim restriksi RsaI, HhaI dan HaeIII (NEB, USA), primer 8f berlabel 6-FAM (6-carboxyfluorescein) (5'-6-FAM-AGAGTTTGATCMTGG CTCAG-3 ') dan 926r (5'-CCGTCAATTCCTTTRAGTTT-3') (Macrogen, Korea) dan standar internal 500 ROX™ (Applied Biosystem, Foster City, CA).
Untuk bakteri referensi, digunakan BAL yang diisolasi selama fermentasi tempe SDBR dan WJB yang telah diidentifikasi dengan API 50 CH (Touw 2014), yaitu: Lactobacillus fermentum, Weissella confusa, Lactobacillus plantarum,
Pediococcus acidilactici, Pediococcus pentosaceus dan Lactobacillus delbrueckii
ssp. pentosaeus delbrueckii.
Alat
11
Tahapan Penelitian
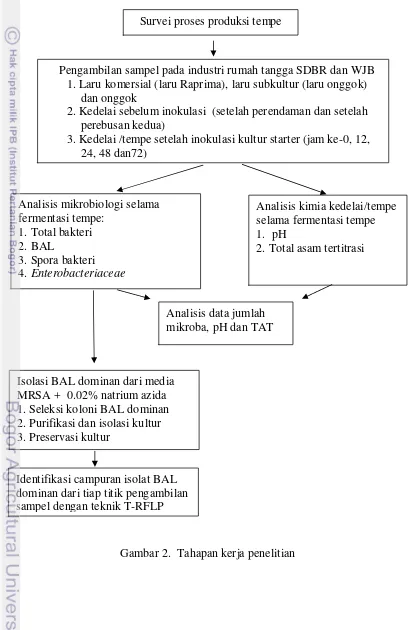
Prosedur penelitian ini dilakukan dalam lima tahap, yaitu sebagai berikut: pengambilan sampe laru, onggok, kedelai dan tempe; analisis kimia kedelai dan tempe selama fermentasi; analisis perhitungan mikroba selama fermentasi tempe; isolasi BAL dominan dan identifikasi BAL dominan (Gambar 2).
Gambar 2. Tahapan kerja penelitian Analisis mikrobiologi selama
fermentasi tempe: 1. Total bakteri 2. BAL
3. Spora bakteri 4. Enterobacteriaceae
Isolasi BAL dominan dari media MRSA + 0.02% natrium azida 1. Seleksi koloni BAL dominan 2. Purifikasi dan isolasi kultur 3. Preservasi kultur
Analisis kimia kedelai/tempe selama fermentasi tempe 1. pH
2. Total asam tertitrasi
Analisis data jumlah mikroba, pH dan TAT Survei proses produksi tempe
Pengambilan sampel pada industri rumah tangga SDBR dan WJB 1. Laru komersial (laru Raprima), laru subkultur (laru onggok)
dan onggok
2. Kedelai sebelum inokulasi (setelah perendaman dan setelah perebusan kedua)
3. Kedelai /tempe setelah inokulasi kultur starter (jam ke-0, 12, 24, 48 dan72)
12
Prosedur Kerja Survei Proses Produksi Tempe
Survei dilakukan pada dua industri rumah yang memproduksi tempe di Bogor dengan kode SDBR dan WJB. Kemudian diamati perbedaan proses produksi pada kedua industri tersebut. Setelah itu, ditentukan titik pengambilan sampel.
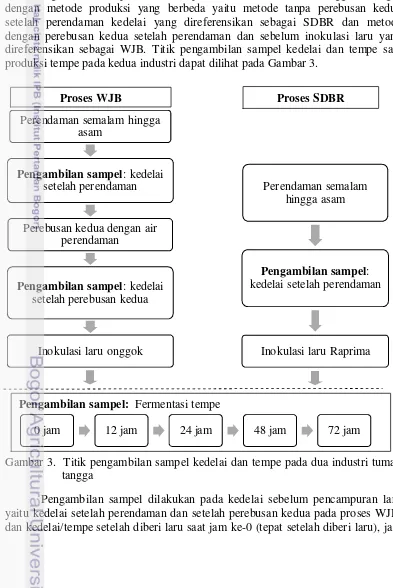
Pengambilan Sampel
Pengambilan sampel dilakukan pada dua industri rumah tangga di Bogor dengan metode produksi yang berbeda yaitu metode tanpa perebusan kedua setelah perendaman kedelai yang direferensikan sebagai SDBR dan metode dengan perebusan kedua setelah perendaman dan sebelum inokulasi laru yang direferensikan sebagai WJB. Titik pengambilan sampel kedelai dan tempe saat produksi tempe pada kedua industri dapat dilihat pada Gambar 3.
Gambar 3. Titik pengambilan sampel kedelai dan tempe pada dua industri tumah tangga
Pengambilan sampel dilakukan pada kedelai sebelum pencampuran laru yaitu kedelai setelah perendaman dan setelah perebusan kedua pada proses WJB; dan kedelai/tempe setelah diberi laru saat jam ke-0 (tepat setelah diberi laru), jam
Perendaman semalam hingga asam
Pengambilan sampel: kedelai setelah perendaman
Perebusan kedua dengan air perendaman
Pengambilan sampel: kedelai setelah perebusan kedua
Inokulasi laru onggok
Perendaman semalam hingga asam
Pengambilan sampel: kedelai setelah perendaman
Inokulasi laru Raprima
Proses WJB Proses SDBR
Pengambilan sampel: Fermentasi tempe
13 ke-12, 24, 48 dan 72 (Gambar 3). Pengambilan sampel juga dilakuan pada laru komersial (laru Raprima), laru subkultur (laru onggok), limbah padat hasil samping produksi tapioka (onggok). Laru yang digunakan untuk proses SDBR merupakan laru komersial yang disubkulturkan ke onggok (laru onggok) sedangkan proses WJB menggunakan laru komersial.
Sekitar 500 g sampel tempe/kedelai diambil secara aseptis dari tiap industri rumah tangga, dikirim ke laboratorium menggunakan ice box dan dianalisis langsung untuk perhitungan koloni bakteri. Sisa sampel kedelai dan tempe disimpan pada suhu-20 oC untuk analisis kimia. Sampel tempe dan kedelai untuk analisis kimia proses SDBR dan WJB; analisis mikrobiologi proses WJB; dan analisis Enterobacteriaceae proses SDBR berasal dari dua kali ulangan pengambilan sampel. Untuk analisis mikrobiologi (selain Enterobacteriaceae) proses SDBR, sampel kedelai dan tempe diambil sebanyak tiga kali ulangan. Laru onggok, laru Raprima dan onggok berturut-turut diambil sebanyak tiga kali ulangan, dua kali ulangan dan satu kali ulangan. BAL diisolasi dari dua kali ulangan pengambilan sampel tempe dan kedelai pada proses WJB. BAL pada proses SDBR diisolasi dari tiga kali ulangan pengambilan sampel kedelai, tempe dan laru onggok.
Analisis Mikrobiologi Kultur Stater Tempe (Laru) dan Onggok
Sebanyak 50 gram sampel laru Raprima, laru onggok dan onggok ditimbang dengan plastik steril, diencerkan dengan 450 ml 0.1% peptone water steril. Setelah itu, campuran sampel diencerkan berseri. Sebanyak 1 ml sampel dipipet ke dalam cawan petri steril (duplo) dan dituang dengan media sebagai berikut: Nutrient Agar (NA, Merck) untuk total bakteri; Violet Red-Bile Agar (VRBA, Oxoid) untuk Enterobacteriaceae; dan media Man Rogosa and Sharpe Agar (MRSA, Oxoid, Bakingstoke, UK) yang ditambah 0.02% natrium azida (Reina et al. 2005) yang bertujuan mencegah pertumbuhan khamir (Plengvidhya et al. 2007) untuk BAL. Untuk analisis Enterobacteriaceae agar dituang dengan metode
double layer. Untuk menghitung spora bakteri, sebelum dilakukan pengenceran serial, 150 ml sampel yang telah dihomogenisasi dipanaskan pada suhu 80 °C selama 10 menit menggunakan autoklaf untuk membunuh sel vegetatif (Harrigan 1998). Kemudian sampel dienumerasi pada media NA. Total bakteri dan spora bakteri diinkubasi selama 48 jam, BAL selama 5-7 hari dan Enterobacteriaceae selama 24 jam pada suhu 30 oC.
Analisis Mikrobiologi Kedelai dan Tempe selama Fermentasi
Lima puluh gram sampel yang diambil dari tahap fermentasi yang berbeda yang telah dijelaskan sebelumnya dicampur dengan 450 ml 0.1% peptone water
dan dihomogenisasi dengan stomacher (Bagmixer, Interscience). Campuran sampel diencerkan berseri. Analisis mikrobiologi kedelai dan tempe dilakukan sama dengan analisis mikrobiologi kultur starter tempe.
Analisis pH dan Total Asam Tertitrasi (TAT)
14
kedelai yang telah dihomogenisasi disentrifugasi dengan kecepatan 3100 rpm selama 10 menit pada suhu 4 oC (Ko et al. 2012). Analisis pH dan asam tertitrasi supernatan sampel dilakukan triplo.
1. Analisis pH
Derajat keasaman atau pH diukur dengan instrumen pH meter (Eutech instrument, Singapore). pH meter dihidupkan selama 15 sampai 30 menit. Sebelum dilakukan pengukuran pH sampel, pH meter distandarkan menggunakan larutan bufer pH 4, 7 dan 10. Nilai pH dari 50 ml supernatan sampel diukur dan dibaca pada saat nilai stabil
2. Analisis Total Asam Tertitrasi (AOAC 1995)
TAT diukur menggunakan metode titrasi menggunakan 0.1 N NaOH dan indikator fenolftalein. Sebelumnya dilakukan standardisasi NaOH yang akan digunakan. Standarisasi NaOH dilakukan dengan menimbang 0.1 g asam oksalat (MW = 216) dan dimasukkan ke dalam erlenmeyer 250 ml. Asam oksalat kemudian ditambah 50 ml aquades dan 2-3 tetes indikator fenolftalein. Larutan dititrasi dengan larutan NaOH sampai terbentuk warna merah muda yang berlangsung selama 15 detik. Normalitas NaOH dihitung dengan rumus berikut:
( ) = 2
0.126
Untuk menentukan TAT, sebanyak 50 ml supernatan sampel diambil dan dimasukkan ke dalam labu erlenmeyer kemudian ditambahkan tiga tetes indikator fenolftalein. Sampel dititrasi dengan NaOH 0.1 N untuk membentuk warna merah muda. TAT dihitung sebagai persentase ke dalam rumus berikut:
% = x 100%
Analisis Data Jumlah Mikroba, pH dan TAT
Untuk mengevaluasi data perubahan jumlah mikroba, pH dan TAT selama waktu fermentasi pada dua proses yang berbeda, dilakukan analisis sidik ragam menggunakan General Linear Model (GLM) dengan perangkat lunak Minitab 16 pada taraf nyata 5% kemudian dilakukan uji lanjut Tukey (p<0.05). Sejumlah data yang tidak terdistribusi normal diperbaiki dengan transformasi data.
Seleksi, Purifikasi dan Isolasi BAL dominan
Sebanyak 10 sampai 15 koloni BAL dominan diisolasi dari cawan MRSA-natrium azida yang dipakai untuk analisis mikroba dengan pengenceran tertinggi yang jumlah koloninya dapat dihitung (25-250) (Kostinek et al. 2005). Pemurnian dilakukan pada media MRSA tanpa natrium azida. Koloni yang diambil digores berulang dengan metode kuadran sehingga menjadi koloni seragam terpisah. Inkubasi dilakukan pada suhu 30 °C selama dua hari. Koloni diisolasi ke agar tegak MRSA dan disimpan pada suhu 4 oC.
Keterangan:
V : Volume NaOH untuk titrasi (ml) N : normalitas NaOH
P : jumlah pengenceran
15
Preservasi Kultur
Kultur sel diimobilisasi pada manik-manik dengan gliserol sebagai agen krioprotektan. Suspensi BAL yang telah ditumbuhkan semalam pada media cair MRS pada suhu 30 oC dimasukkan ke dalam cryotube yang berisi gliserol steril dan manik-manik. Rasio kultur dengan gliserol ialah 4:1. Tabung dikocok dan didiamkan selama 2-3 jam. Cairan residu dibuang hingga sisa cairan sebatas tinggi manik-manik. Cryotube disimpan pada suhu -20 °C.
Identifikasi BAL
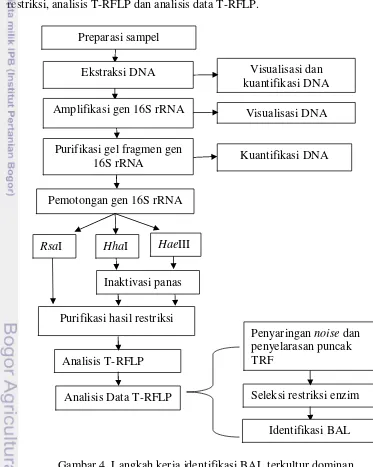
Identifikasi BAL dilakukan dalam beberapa langkah (Gambar 4). Tahapan prosedur identifikasi BAL antara lain preparasi sampel, ekstraksi DNA, amplifikasi gen 16S rRNA, restriksi gen 16S rRNA, purifikasi hasil PCR dan hasil restriksi, analisis T-RFLP dan analisis data T-RFLP.
Gambar 4. Langkah kerja identifikasi BAL terkultur dominan Preparasi sampel
Ekstraksi DNA
Amplifikasi gen 16S rRNA
Purifikasi gel fragmen gen 16S rRNA
Pemotongan gen 16S rRNA
RsaI HhaI HaeIII
Inaktivasi panas
Purifikasi hasil restriksi
Visualisasi dan kuantifikasi DNA
Kuantifikasi DNA
Analisis T-RFLP
Seleksi restriksi enzim Penyaringan noise dan penyelarasan puncak TRF
Analisis Data T-RFLP
Visualisasi DNA
16
Preparasi Kultur Sebanyak 342 koloni BAL yang berasal dari penyimpanan suhu 4 oC disegarkan kembali pada agar tusuk MRS dan diinkubasi pada suhu 30 o
C selama 48 jam. Setelah tumbuh, sebanyak 10-15 isolat pada tahap fermentasi yang sama diinokulasikan sebagai satu titik pada cawan MRSA sehingga ada 7 sampel campuran kultur yang berasal dari proses SDBR (laru onggok, fermentasi 0, 12, 24, 48, 72 jam) dan 6 sampel campuran kultur dari proses WJB (fermentasi 0, 12, 24, 48 dan 72 jam) (Gambar 5). Kultur BAL tersebut diinkubasi pada suhu 30 oC selama 18 jam, kemudian dipanen terpisah dari tiap cawan. Campuran kultur BAL dari satu tahap fermentasi yang sama diusap dengan cotton bud yang telah dibasahi dengan larutan fisiologis (0.85% NaCl) steril kemudian kultur BAL disuspensikan ke dalam larutan fisiologis steril pada mikrotube 1.5 ml. Suspensi BAL disentrifugasi dengan kecepatan 14000 rpm selama 2 menit sehingga didapat campuran pelet sel BAL yang berasal dari tahap fermentasi yang sama. Pelet sel tersebut akan digunakan untuk ekstraksi DNA
Untuk bakteri referensi digunakan BAL dominan tempe yang telah diidentifikasi dengan API 50 CH (Touw 2014) yaitu Lactobacillus fermentum,
Weissella confusa, Lactobacillus plantarum, Pediococcus acidilactcii,
Pediococcus pentosaeus, Weissella confusa, Lactobacillus plantarum dan
Lactobacillus delbrueckii ssp delbrueckii. Bakteri tersebut juga diinokulasikan sebagai satu titik pada satu cawan MRSA dan dipreparasi dengan cara yang sama seperti preparasi sampel.
Gambar 5. Persiapan sampel DNA untuk identifikasi BAL terkultur domiman
Ekstraksi DNA Genom BAL Ekstraksi DNA dilakukan dengan Wizard Genomic DNA Purification Kit (Promega, USA) dengan penambahan tiga siklus freeze-thaw (Ulrich dan Hughens 2001). Pelet dicuci dengan 200 μl 50 mmol EDTA dan disentrifugasi pada 14000 rpm, selama 2 menit. Pelet ditambah dan diresuspensi dengan 500 μl 50 mmol EDTA dan 100 μl 40 mg/ml lisozim (20000 U/mg) (Biobasic, Canada). Campuran diinkubasi selama 60 menit pada suhu 37 oC dan disentrifugasi pada 14000 rpm, selama 1 menit. Pelet diresuspensi dengan 850 μl
nuclei lysis solution. Setelah itu dilakukan tiga kali siklus freeze-thaw, dengan satu siklus sebagai berikut: sampel diinkubasi dengan dry ice atau nitrogen cair
Kultur diinokulasi sebagai satu titik pada satu cawan
Pencampuran kultur BAL dari tahap fermentasi yang sama
Ekstraksi DNA
Satu sampel DNA dari kultur campuran 10-15 koloni dari tahap
17 selama 60 detik, kemudian diinkubasi pada suhu 80 oC dengan penangas air selama 2 menit. Setelah didinginkan pada suhu ruang, campuran ditambah 4 μl (4 mg/ml) RNAse dan diinkubasi pada suhu 37 oC selama 30 menit. Setelah didinginkan pada suhu ruang, campuran ditambah 300 μl protein precipitation solution. Tabung yang berisi larutan campuran dibolak-balik selama 20 kali dan disentrifugasi pada 14000 rpm selama 10 menit. Supernatan dimasukkan ke dalam mikrotube baru dan disentrifugasi kembali pada 14000 rpm, 25 oC selama 5 menit untuk memisahkan residu protein dari ekstrak. Supernatan dipindahkan ke dalam tabung baru yang berisi 600 μl isopropanol. Tabung dibolak-balik perlahan hingga terlihat helaian putih yang merupakan DNA kemudian tabung disentrifugasi pada 14000 rpm, selama 10 menit. Pelet dicuci dengan 600 μl etanol 70% dan disentrifugasi kembali. Pelet DNA dikeringkan di udara terbuka selama 15 menit kemudian ditambah dengan 100 μl DNA rehydration solution. DNA diinkubasi semalam pada suhu 4 oC kemudian disimpan pada suhu -20 oC. Konfirmasi keberhasilan isolasi DNA dilakukan dengan elektroforesis gel agarosa. Kemurnian dan kuantitas DNA diukur dengan spektrofotometer.
Visualisasi DNA Visualiasasi DNA dilakukan dengan elektroforesis gel agarosa. Sebanyak 2 ml DNA diencerkan dengan 8 ml ddH2O dan 2 ml loading dye. Campuran dipindahkan ke sumur agarosa (0.8%) yang sudah direndam dengan 1x bufer TAE. Penanda berat molekul DNA yang digunakan ialah universal ladder
(KAPA Biosystems, USA) yang mengandung 100 bp - 1 kb DNA ladder. Elektroforesis dijalankan pada tegangan 90 volt selama 45 menit dengan mesin elektroforesis (BioRad, Singapore).
Kuantifikasi DNA Penentuan konsentrasi dan kemurnian DNA dilakukan dengan spektrofotometri menggunakan NanoDrop 2000 Spectrophotemer (Thermo Scientfic) atau Spektofotometer UV 2450 (Shimadzu, Japan). Absorbansi DNA diukur pada panjang gelombang 260 nm dan 280 nm. Rasio A260/A280 1.8-2.0 dinyatakan sebagai DNA yang murni. Konsentrasi DNA dihitung dengan rumus berikut:
Konsentr asi DNA ( ng/ ml) = A260 x 50 x faktor pengencer an
18
Purifikasi Produk PCR Fragmen produk PCR pada gel agarosa dipurifikasi dengan Wizard SV-Gel and Clean Up System (Promega, USA). Dengan bantuan sinar UV pada panjang gelombang tinggi, gel agarosa yang mengandung DNA target dipotong sekecil mungkin dengan cepat. Potongan gel yang berisi DNA sampel dimasukkan ke dalam mikrotube kemudian ditambah dengan 10µ l
membrane binding solution tiap 10 mg potongan gel, divortex dan diinkubasi pada 50-65 °C hingga gel larut. Untuk mengikat DNA, gel yang telah larut dimasukkan ke dalam SV minicolumn yang telah diletakkan pada tabung koleksi kemudian sampel diinkubasi pada suhu ruang selama 1 menit. Kolom disentrifugasi pada 14000 rpm selama 1 menit kemudian supernatan dibuang. Untuk pencucian DNA, 700 μl membrane wash solution ditambahkan ke dalam kolom dan disentrifugasi pada 14000 rpm selama 1 menit kemudian supernatan dibuang. Sebanyak 500 μl
membrane wash solution ditambahkan kembali ke kolom kemudian kolom disentrifugasi pada 14000 rpm selama 5 menit. Setelah itu, untuk evaporasi residu etanol, supernatan dibuang dan kolom disentrifugasi kembali pada 14000 rpm selama 5 menit, kemudian kolom diletakkan pada tabung baru. Untuk elusi DNA, sebanyak 50 μl nuclease free water ditambahkan ke dalam kolom. Kolom diinkubasi pada suhu ruang kemudian disentrifugasi pada 14000 selama 1 menit. Kolom dibuang kemudian DNA disimpan pada suhu -20 oC. Untuk mengetahui kualitas dan kuantitas DNA, hasil purifikasi dikonfirmasi dengan spektrofotometri seperti yang dijelaskan pada prosedur sebelumnya.
Pemotongan Produk PCR Produk PCR yang telah dipurifikasi dipotong dengan masing-masing enzim restriksi HhaI, HaeIII dan RsaI (New England Biolab, Beverly, MA). Kondisi reaksi dari masing-masing enzim disesuaikan dengan petunjuk perusahaan yaitu 1.0 μl enzim, 2 μl 10X NEB buffer, sekitar 300 ng amplikon dan nuclease free water hingga volume reaksi menjadi 20 μl. Reaksi dilakukan pada suhu 37 oC selama 18 jam. Inaktivasi enzim dilakukan dilakukan sesuai petunjuk perusahaan yaitu inaktivasi panas untuk HhaI dan HaeIII; dan inaktivasi dengan presipitasi EDTA/etanol untuk RsaI. HhaI diinaktivasi pada suhu 65 oC sedangkan HaeIII diinaktivasi pada suhu 80 oC selama 20 menit menggunakan 2720 Thermal Cycler (Applied Biosystems, Foster City, CA).
Purifikasi Hasil Restriksi Hasil restriksi dipurifikasi dengan metode presipitasi EDTA/etanol sesuai protokol BigDye v3.1. Sebanyak 5 μl 125 mM EDTA dan 60 μl etanol absolut dingin ditambahkan ke dalam 20 μl sampel. Tabung yang berisi larutan campuran dibolak-balik, diinkubasi pada suhu ruang selama 15 menit kemudian disenstrifugasi pada 6000 rpm selama 30 menit pada suhu 4 oC. Supernatan dibuang, pelet ditambah 60 μl etanol 70% dan disentrifugasi 4000 rpm selama 15 menit pada suhu 4 oC. Supernatan dibuang, dikeringkan di udara terbuka selama 15 menit kemudian ditambah 20 μl nuclease free water (Amresco, USA). Hasil purifikasi disimpan pada suhu -20 oC.
19 Wilmar Benih Indonesia, JABABEKA Bekasi Jawa Barat, Indonesia menggunakan ABI PRISM 3130 (Applied Biosystems).
20
HASIL DAN PEMBAHASAN
Perbedaan Proses Produksi Tempe pada Industri Rumah Tangga SDBR dan WJB
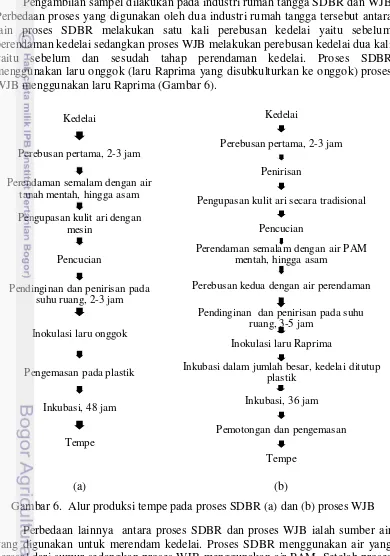
Pengambilan sampel dilakukan pada industri rumah tangga SDBR dan WJB. Perbedaan proses yang digunakan oleh dua industri rumah tangga tersebut antara lain proses SDBR melakukan satu kali perebusan kedelai yaitu sebelum perendaman kedelai sedangkan proses WJB melakukan perebusan kedelai dua kali yaitu sebelum dan sesudah tahap perendaman kedelai. Proses SDBR menggunakan laru onggok (laru Raprima yang disubkulturkan ke onggok) proses WJB menggunakan laru Raprima (Gambar 6).
Gambar 6. Alur produksi tempe pada proses SDBR (a) dan (b) proses WJB Perbedaan lainnya antara proses SDBR dan proses WJB ialah sumber air yang digunakan untuk merendam kedelai. Proses SDBR menggunakan air yang berasal dari sumur sedangkan proses WJB menggunakan air PAM. Setelah proses perendaman dan sebelum inokulasi laru, kedelai pada proses SDBR hanya
Pendinginan dan penirisan pada suhu ruang, 3-5 jam
Inokulasi laru Raprima
Inkubasi dalam jumlah besar, kedelai ditutup plastik
Inkubasi, 36 jam
Pemotongan dan pengemasan
21 didinginkan selama 3 jam sedangkan kedelai pada proses WJB didinginkan selama 5 jam. Selain itu, pengupasan kulit ari kedelai pada proses WJB menggunakan cara tradisional sedangkan kulit ari kedelai pada proses SDBR dikupas dengan mesin. Setelah pemberian laru pada proses SDBR, kedelai dikemas dalam plastik berlubang kemudian diinkubasi selama 48 jam. Sementara itu pada proses WJB, kedelai yang telah diberi laru dipadatkan dan difermentasi dengan jumlah besar. Selama proses fermentasi, kedelai/tempe dialasi plastik dan ditutup dengan plastik berlubang yang telah digunakan untuk menginkubasi tempe
batch sebelumnya. Setelah itu, kedelai yang telah ditutup plastik ditindih dengan ubin atau batu bata. Inkubasi tempe proses WJB berlangsung selama 36 jam kemudian tempe dipotong dan dikemas dengan daun pisang.
Populasi Mikroba pada Laru Tempe Proses SDBR dan WJB
Pada penelitian ini, dua produsen tempe menggunakan jenis laru yang berbeda. Proses SDBR menggunakan laru onggok yang diperoleh dengan mengsubkulturkan laru komersial dengan merk Raprima ke onggok yang kemudian disebut sebagai laru onggok, sedangkan proses WJB menggunakan laru Raprima tanpa pengsubkulturan lebih lanjut. Laru Raprima diproduksi secara komersial oleh PT Aneka Fermentasi Industri (AFI) (Bandung, Indonesia) dan merupakan kultur starter yang biasanya dipakai untuk fermentasi tempe di Indonesia (Seumahu 2012). Laru onggok dibuat sendiri oleh industri rumah tangga bertujuan mengurangi biaya produksi. Proses pembuatan laru onggok dapat dilihat pada Gambar 7.
Gambar 7. Persiapan laru onggok yang digunakan proses SDBR
Gambar 8 menunjukkan populasi mikroba pada laru Raprima, laru onggok dan onggok. Pada laru dan onggok, terdapat spora bakteri dengan jumlah yang tinggi. Perbedaan profil mikroba yang besar terdapat pada laru onggok dan laru Raprima. BAL hadir pada laru onggok yang digunakan oleh proses SDBR sebesar 3.9 log cfu/g dan pada onggok sebesar 2.2 log cfu/g, tetapi BAL tidak ditemukan pada laru Raprima. Enterobacteriaceae hadir dalam jumlah yang rendah baik pada
Pencampuran laru Raprima dengan onggok dan air hangat Pengemasan dengan
plastik Inkubasi semalam
Pemotongan Pengeringan dengan
22
laru onggok dan laru Raprima namun tidak ditemukan pada onggok. Laru onggok memiliki Enterobacteriaceae yang lebih tinggi (1.6 log cfu/g) dibandingkan pada laru Raprima (0.7 log cfu/g). Hal ini dapat disebabkan oleh penambahan air yang hanya dimasak hingga suhunya hangat ketika proses produksi laru onggok. Proses pengsubkulturan laru Raprima ke onggok berkontribusi terhadap bertambahnya jumlah Enterobacteriaceae pada laru onggok. Keberadaan
Enterobacteriaceae pada laru mungkin menjadi salah satu sumber keberadaan bakteri ini pada tahap awal fermentasi tempe. BAL ditemukan dalam onggok dan laru onggok, namun tidak ditemukan pada laru Raprima. Oleh karena itu onggok diduga berkontribusi terhadap keberadaan BAL pada laru onggok. Proses pengsubkulturan laru Raprima ke onggok menyumbangkan keberadaan BAL pada laru onggok.
Onggok merupakan produk samping produksi tapioka. Selama pengolahan singkong menjadi tapioka, peningkatan keasaman limbah singkong terjadi akibat aktivitas bakteri. Arotupin (2007) melaporkan bahwa limbah singkong memiliki nilai pH rendah (3.55-4.02) yang diakibatkan aktivitas mikroba yang telah diisolasi dan mikroba tersebut mungkin memainkan peran selama pengolahan singkong menjadi produk lain.
Total Bakteri
Total bakteri setelah perendaman kedelai memiliki jumlah yang cukup tinggi pada proses SDBR dan WJB (Gambar 9). Total bakteri setelah perendaman kedelai berjumlah 8 log cfu/g pada proses SDBR dan 7 log cfu/g pada proses WJB. Pengupasan kulit ari dan pencucian pada proses SDBR menurunkan total bakteri. Perebusan kedua sebelum inokulasi laru pada proses WJB juga menurunkan total bakteri. Preparasi setelah proses perendaman menurunkan jumlah total bakteri pada proses SDBR dan WJB sebesar 1.5 log cfu/g yang dapat dilihat pada jam ke-0, namun penurunannya tidak signifikan dan tidak berbeda nyata (p<0.05). Proses pendinginan setelah perebusan kedua kedelai pada proses
Gambar 8. Populasi bakteri pada laru dan onggok. Rataan dengan
23 WJB sebelum inokulasi laru, memberi kesempatan bakteri dari lingkungan masuk ke kedelai dan tumbuh sehingga penurunan total bakteri dari tahap setelah perebusan kedua kedelai ke jam ke-0 pada proses WJB tidak signifikan. Total bakteri kemudian naik selama fermentasi pada kedua proses. Selama proses fermentasi pada proses WJB, total bakteri naik secara signifikan dan berbeda nyata (p<0.05) setelah jam ke-0 hingga jam ke-24. Sementara itu, pada proses SDBR, kenaikan total bakteri terlihat setelah jam ke-12 dan berlanjut hingga jam ke-24. Pada tahap fermentasi selanjutnya, pertumbuhan total bakteri menjadi stabil baik pada proses SDBR maupun WJB.
Total bakteri pada tempe proses SDBR dan WJB hadir dengan jumlah yang tinggi (≥ 8 log cfu/g) (Gambar 9) dan melebihi jumlah kapang yang telah dilaporkan pada tempe proses WJB yaitu sebesar 4.2 log cfu/g dan proses SDBR yaitu sebesar 6.7 log cfu/g (Nurdini et al. 2015). Hal ini sesuai dengan hasil yang dilaporkan oleh Mulyowidarso et al. (1990) bahwa total bakteri pada tempe memiliki jumlah yang lebih tinggi 1 log dibandingkan dengan jumlah kapang.
Peran bakteri dalam fermentasi tempe belum diketahui secara jelas. Akan tetapi, beberapa peneliti telah melaporkan peran bakteri dalam pembentukan vitamin B12 oleh Citrobacter freundii dan Klebsiella pneumoniae (Keuth dan Bisping 1993) dan isoflavon oleh Micrococcus dan Arthrobacter (Klus et al. 1993).
Bakteri Asam Laktat
Proses pengupasan kulit ari dan pencucian kedelai setelah perendaman kedelai dapat mengurangi BAL pada proses SDBR sebesar 1 log yang dapat dilihat pada perubahan jumlah BAL setelah perendaman kedelai hingga fermentasi jam ke-0 (Gambar 10). Penurunan jumlah BAL yang lebih besar terjadi pada proses WJB setelah perebusan kedua kedelai dan sebelum inokulasi laru yang dapat dilihat pada fermentasi jam ke-0. Pada proses WJB, jumlah BAL setelah kedelai direndam berbeda nyata (p<0.05) dengan BAL pada jam ke-0 yang