KONSTRUKSI DAN ANALISIS KUALITAS PUSTAKA GENOM
KEDELAI (
Glycine max
(L.) Merr.
)
UNTUK SEKUENSING
GENOM TOTAL
ANDI KOSASIH
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
ABSTRAK
ANDI KOSASIH. Konstruksi dan Analisis Kualitas Pustaka Genom Kedelai
(Glycine max
(L.) Merr.) untuk Sekuensing Genom Total. Dibimbing oleh I
MADE ARTIKA dan I MADE TASMA.
Kedelai merupakan salah satu dari komoditas pertanian terpenting dalam
penyediaan bahan pangan di Indonesia. Kendala dalam budidaya kedelai di
Indonesia yaitu rendahnya produktivitas kedelai nasional. Usaha untuk
meningkatkan produktivitas kedelai nasional dapat dilakukan dengan perbaikan
genetik tanaman. Sekuensing genom total dan analisis genom kedelai merupakan
salah satu cara untuk mempercepat peningkatan pemuliaan kedelai serta untuk
lebih meningkatkan pemahaman mengenai informasi genetik dan genomik
kedelai. Tujuan penelitian ini adalah mengkonstruksi pustaka genom dari tiga
genotipe kedelai di Indonesia (Tambora, B3293 dan Grobogan) dan menganalisis
kualitas pustaka genom hasil sekuensing genom total. Konstruksi pustaka genom
dilakukan secara in vitro dengan menempelkan adapter pada ujung fragmen DNA.
Pustaka genom selanjutnya diklasterisasi dan disekuensing dengan menggunakan
cBOT claster generation
dan
Next Generation Sequencing HiSeq2000. Pustaka
genom yang berhasil dikonstruksi berukuran 400 bp dengan konsentrasi 23.20
ng/µL (Tambora), 64.50 ng/µL (B3293), dan 21.20 ng/µL (Grobogan). Ukuran
dan konsentrasi pustaka genom yang dikonstruksi telah memenuhi persyaratan
untuk sekuensing genom total. Jumlah basa yang dihasilkan dari proses
sekuensing yaitu sebanyak 50.1 x 10
9bp. Klaster pustaka genom dengan nilai
Q
scores di atas
30 yang dihasilkan sebesar 88.6%. Tingkat kesalahan pembacaan
basa selama proses sekuensing sebesar 0.97%. Nilai densitas klaster, persen
klaster PF, intensitas basa, persen phasing, dan persen prephasing yang dihasilkan
dari penelitian ini menunjukkan bahwa kualitas klaster pustaka genom termasuk
kategori ideal.
ABSTRACT
ANDI KOSASIH. Construction and Quality Analysis of Soybean (Glycine max
(L.) Merr.) Genomic Library for Whole Genome Sequencing. Under the direction
of I MADE ARTIKA and I MADE TASMA.
Soybean is one of the most important agricultural commodities in the supply
of food in Indonesia. However, the low of the national soybean productivity is a
constraint in soybean cultivation in Indonesia. National soybean productivity can
be increased by genetic improvement of plants. Whole genome sequencing and
genomic analysis of soybean is one way to accelerate soybean breeding progress
and improve understanding of genetic and genomic information of soybean.
Purposes of this research were to construct genomic library of three Indonesian
soybean genotypes (Tambora, B3293 and Grobogan) and analyze the quality of
the genomic library on whole genome sequencing result. Construction of genomic
libraries was carried out in vitro by attaching an adapter to the end of DNA
fragments. Clasterization and sequencing of genomic library in this research were
conducted by using cBOT claster generation dan Next Generation Sequencing
HiSeq2000. Genomic libraries were constructed successfully sized 400 bp with a
concentration of 23.20 ng/µL (Tambora), 64.50 ng/µL (B3293), and 21.20 ng/µ L
(Grobogan). Size and concentration of the constructed genomic libraries were
suitable for whole genome sequencing. The total yield of bases generated from the
sequencing was 50.1 x 10
9bp. Cluster library produced during the process of
genome sequencing was Q scores value above 30 was 88.6%. Error rate of base
reading during the sequencing was 0.97%. Values of the cluster density, percent
cluster PF, the intensity of the base, phasing percent, and prephasing percent
generated from this research showed that the cluster quality of genomic libraries
was of ideal category.
KONSTRUKSI DAN ANALISIS KUALITAS PUSTAKA GENOM
KEDELAI (
Glycine max
(L.) Merr.
)
UNTUK SEKUENSING
GENOM TOTAL
ANDI KOSASIH
G84080043
Skripsi
sebagai salah satu syarat memperoleh gelar
Sarjana Sains pada
Departemen Biokimia
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul skripsi
: Konstruksi dan Analisis Kualitas Pustaka Genom Kedelai
(Glycine max (L.) Merr.) untuk Sekuensing Genom Total
Nama
: Andi Kosasih
NIM
: G84080043
Disetujui,
Komisi Pembimbing
Dr. Ir. I Made Artika, M.App.Sc
Ketua
Dr. Ir. I Made Tasma, M.Sc
Anggota
Diketahui,
Dr. Ir. I Made Artika, M.App.Sc.
Ketua Departemen Biokimia
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala rahmat
dan karunia-Nya. Shalawat dan salam semoga selalu tercurah kepada Nabi
Muhammad SAW beserta seluruh keluarga, sahabat dan kepada para pengikutnya
sampai akhir zaman sehingga penulis dapat menyelesaikan karya ilmiah ini
sebagai salah satu persyaratan untuk memperoleh gelar Sarjana Sains pada
Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut
Pertanian Bogor. Tema yang dipilih pada penelitian ini adalah biologi molekuler,
dengan judul
“Konstruksi dan Analisis Kualitas Pustaka Genom Kedelai (Glycine
max
(L.) Merr.) untuk Sekuensing Genom Total”. Penelitian ini dilaksanakan
selama lima bulan sejak bulan Februari sampai Juni 2012, bertempat di
Laboratorium Biologi Molekuler, Balai Besar Penelitian dan Pengembangan
Bioteknologi dan Sumber Daya Genetik Pertanian (BB Biogen), Bogor.
Terima kasih penulis ucapkan kepada Dr. Ir. I Made Artika, M.App.Sc dan
Dr. Ir. I Made Tasma, M.Sc atas semua ilmu, bimbingan dan dukungan yang
diberikan kepada penulis selama penelitian dan penyusunan karya ilmiah. Ucapan
terima kasih juga penulis sampaikan kepada Bapak Dani Setyawan, M.Si yang
telah banyak membantu dalam teknis pelaksanaan penelitian, kepada kedua orang
tua dan seluruh keluarga atas segala doa, dukungan, dan kasih sayangnya yang
selalu memberi inspirasi kepada penulis untuk selalu berjuang keras dan menjadi
lebih baik.
Penulis juga menyampaikan terima kasih kepada Esti Sahifah, Riris,
Nanda, Wulan, rekan-rekan Biokimia 45 dan seluruh penghuni Wisma Badenten
atas dukungan, kritik dan bantuannya selama penelitian dan penyusunan karya
ilmiah. Semoga karya ilmiah ini dapat bermanfaat untuk perkembangan ilmu
pengetahuan dan mampu memberikan informasi bagi yang memerlukan.
Bogor, November 2012
RIWAYAT HIDUP
Penulis dilahirkan di Ciamis pada tanggal 6 Mei 1990 dari ayah Drs.
Kartono Kosasih dan ibu Yeyet Nurhayati. Penulis merupakan putra kedua dari
dua bersaudara.
Pendidikan penulis dimulai dari SD Negeri Cibodas dan melanjutkan
pendidikan ke SMP Negeri 1 Banjar. Penulis lulus tahun 2008 dari SMA Negeri 1
Banjar dan pada tahun yang sama lulus seleksi masuk IPB melalui jalur Undangan
Seleksi Masuk IPB (USMI). Penulis memilih mayor Biokimia, Departemen
Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam dan minor
Pengolahan Pangan, Departemen Ilmu dan Teknologi Pangan, Fakultas Teknologi
Pertanian.
Selama mengikuti perkuliahan, penulis pernah menjadi asisten praktikum
Biokimia Umum untuk mahasiswa S1 Biologi tahun ajaran 2011/2012 dan asisten
praktikum Struktur dan Fungsi Biomolekul untuk mahasiswa S1 Biokimia tahun
ajaran 2011/2012. Penulis juga aktif dalam kegiatan kemahasiswaan di IPB dan
organisasi mahasiswa daerah, di antaranya penulis pernah aktif sebagai anggota
Unit Kegiatan Mahasiswa Koperasi Mahasiswa IPB pada tahun 2008/2009, staf
Divisi Human Resource Development Himpunan Profesi
Community of Research
and Education in Biochemistry
(CREBs) periode 2009/2010, staf divisi Budaya,
Olahraga dan Seni Paguyuban Mahasiswa Galuh Ciamis periode 2009/2010,
Anggota Forum Silaturahmi Ketua Lembaga Kemahasiswaan FMIPA IPB periode
2010/2011 dan Ketua Himpunan Profesi Biokimia Community of Research and
Education in Biochemistry (CREBs) periode 2010/2011.
DAFTAR ISI
Halaman
DAFTAR GAMBAR ... ix
DAFTAR TABEL ... ix
DAFTAR LAMPIRAN ...
x
PENDAHULUAN ...
1
TINJAUAN PUSTAKA
Kedelai ...
1
Pustaka Genom ...
3
Sekuensing DNA ...
4
Next Generation Sequencing ...
5
Klasterisasi Pustaka Genom ...
6
BAHAN DAN METODE
Alat dan Bahan ...
6
Metode ...
7
HASIL DAN PEMBAHASAN
Hasil Isolasi DNA Genom Kedelai ... 11
Hasil Fragmentasi DNA Genom Kedelai ... 12
Hasil Konstruksi Pustaka Genom Kedelai ... 13
Analisis Kualitas Pustaka Genom Hasil Sekeunsing Genom Total ... 17
SIMPULAN DAN SARAN
Simpulan ... 20
Saran ... 20
DAFTAR PUSTAKA ... 20
DAFTAR TABEL
Halaman
1 Kualitas dan kemurnian DNA genom total kedelai ... 12
2 Kuantitas dan kemurnian DNA genom kedelai hasil fragmentasi ... 13
3 Kuantitas dan kemurnian DNA cetakan untuk preparasi konstruksi pustaka
genom setelah dilarutkan dalam resuspension buffer ...
14
4 Kuantitas dan kemurnian fragmen DNA setelah tahapan modifikasi ujung
fragmen DNA pada preparasi pustaka genom kedelai ... 15
5 Kuantitas dan kemurnian DNA cetakan setelah tahapan ligasi adapter ... 15
6 Kuantitas dan kemurnian DNA hasil ekstraksi gel ... 16
7 Kuantitas dan rasio kemurnian pustaka genom kedelai ... 17
8 Jumlah basa dan kualitas klaster pustaka genom hasil sekuensing ... 17
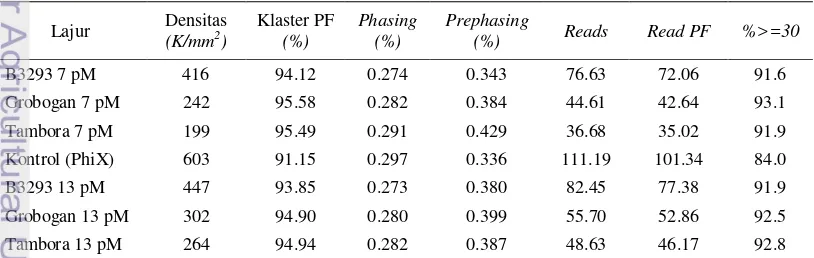
9 Densitas dan kualitas pustaka genom hasil sekuensing ... 18
10 Tingkat kesalahan (error rate), nilai penyejajaran (aligned), dan intensitas
basa hasil sekuensing ... 20
DAFTAR GAMBAR
Halaman
1 Tanaman kedelai (Glycine max (L.) Merr.) ... 2
2
Next Generation Sequencing HiSeq 2000 ... 5
3 Elektroforegram DNA genom total kedelai ... 11
4 Elektroforegram DNA genom kedelai hasil fragmentasi ... 13
5 Elektroforegram tahapan purifikasi DNA hasil ligasi... 15
6 Elektroforegram pustaka genom tiga genotipe kedelai ... 16
7 Histogram jumlah klaster berdasarkan kualitas sekuens yang dihasilkan.... 19
DAFTAR LAMPIRAN
Halaman
1
Strategi Penelitian ... 25
2
Flow cell Illumina HiSeq2000 ... 26
3
Prinsip sequencing by synthesis next generation sequencing Illumina HiSeq
2000 ... 27
4
Tahapan klasterisasi pustaka genom ... 28
5
Tabung nebulisasi ... 29
6
Ilustrasi phasing dan prephasing yang dapat terjadi saat proses
sekuensing ... 30
7
Gambar intensitas basa adenin pada siklus 1 proses sekuensing ... 31
8
Grafik jumlah densitas klaster setiap lajur dalam flow cell ... 32
PENDAHULUAN
Kedelai (Glycine max (L.) Merr.)
merupakan komoditas pertanian terpenting ketiga setelah padi dan jagung dalam penyediaan bahan pangan di Indonesia. Kedelai berperan sebagai sumber protein nabati yang sangat penting dalam rangka peningkatan gizi masyarakat karena harganya yang terjangkau dan aman bagi kesehatan
(Kementerian Pertanian 2007).
Shanmugasundaram dan Sumarno (1993) melaporkan bahwa dalam 100 g kedelai mengandung 10 g air, 35 g protein, 18 g lemak dan 32 g karbohidrat.
Kebutuhan kedelai meningkat seiring dengan pertumbuhan jumlah penduduk di
Indonesia. Hal tersebut terlihat dari
meningkatnya nilai konsumsi kedelai per kapita pada tahun 2004 sebesar 8.97 kg meningkat menjadi 10.10 kg pada tahun 2011 (Kementerian Pertanian 2007, Sari 2011).
Direktorat Jenderal Tanaman Pangan
Republik Indonesia (2010) memperkirakan konsumsi kedelai per kapita di Indonesia akan semakin meningkat dan dapat mencapai 10.20
kg pada tahun 2012 dengan tingkat
pertumbuhan konsumsi sebesar 0.24%.
Namun, meningkatnya tingkat konsumsi berbanding terbalik dengan produksi kedelai nasional. Tahun 2011, produksi kedelai nasional hanya sebesar 843 838 ton dengan luas panen 620 928 ha dan produktivitas 1.36
ton/ha. Nilai produksi tersebut sangat
menurun dibandingkan produksi kedelai nasional tahun 1992 yang mencapai 1.8 juta ton dengan luas panen 1.6 juta ha dan
produktivitas 1.12 ton/ha. Rendahnya
produktivitas kedelai nasional menyebabkan
kebutuhan kedelai dalam negeri tidak
tercukupi. Produksi kedelai nasional hanya memenuhi 48% dari total kebutuhan kedelai dalam negeri yang mencapai 2 juta ton (Dirjen Tanaman Pangan RI 2010, BPS 2011, Sari 2011).
Usaha untuk meningkatkan produktivitas kedelai nasional dapat dilakukan melalui perbaikan genetik tanaman. Salah satu faktor yang penting dalam perbaikan genetik
tanaman adalah tahapan isolasi dan
karakterisasi gen. Tahapan tersebut dapat dilakukan dengan memanfaatkan informasi genomik dan genetik kedelai yang diperoleh melalui sekuensing genom total kedelai.
Sekuensing genom total (whole genome
sequensing) adalah proses penentuan urutan genom suatu organisme yang dilakukan dalam satu waktu. Argout et al (2010) menyatakan
bahwa sekuensing genom total penting
dilakukan terutama untuk mengetahui
informasi genomik dan genetik suatu
organisme secara menyeluruh. Sekuensing
genom total dapat dilakukan dengan
teknologi sekuensing yang disebut next
generation sequensing (NGS). Panjang untai DNA yang disekuens menggunakan metode NGS yaitu 25-500 bp namun dapat dibaca ratusan bahkan ribuan kali. Jika dibandingkan dengan menggunakan metode sekuensing
genom total yang lainnya seperti metode
shotgun sequencing, metode NGS menghasilkan data yang lebih banyak dengan waktu yang lebih singkat (Voelkerding et al.
2009, Metzker 2010). Instrumen NGS yang
digunakan pada penelitian ini adalah Illumina
HiSeq2000. Keunggulannya yaitu dapat
membaca untai DNA sekitar 2x100 bp dan menghasilkan data sekitar 200 Gbp dalam satu kali sekuens dengan akurasi data yang dihasilkan dapat mencapai 99.5% (Zhang et al. 2011).
Tahapan sekuensing genom total diawali dengan mengkonstruksi pustaka genom. Konstruksi pustaka genom untuk sekuensing genom total menggunakan NGS Illumina HiSeq2000 membutuhkan waktu yang relatif singkat dan efisien. Hal tersebut terjadi karena dalam tahapan konstruksi pustaka genom tidak membutuhkan vektor untuk menyisipkan fragmen DNA dan tidak memerlukan sebuah ruang penyimpanan khusus untuk menyimpan vektor yang mengandung fragmen DNA.
Penelitian ini bertujuan untuk
mengkonstruksi pustaka genom kedelai dan menganalisis kualitas pustaka genom hasil sekuensing genom total. Hipotesis penelitian ini adalah pustaka genom tiga genotipe kedelai Indonesia dapat dikonstruksi untuk sekuensing genom total. Penelitian ini diharapkan dapat membantu meningkatkan informasi genomik dan genetik kedelai yang pada tahapan selanjutnya dapat digunakan untuk membantu pemuliaan kedelai
TINJAUAN PUSTAKA
Kedelai
Kedelai (Glycine max (L.) Merr)
2
makanan. Kedelai berasal dari daerah
Manshukuo (Cina Utara) dan merupakan salah satu tanaman yang penting dalam kehidupan masyarakat di daratan Cina dan telah
dibudidayakan sejak tahun 2500 SM.
Tanaman ini kemudian tersebar luas sampai ke Cina bagian selatan, Korea, Jepang dan negara-negara di Asia Tenggara termasuk Indonesia. Di samping itu, kedelai juga tersebar hingga ke luar benua Asia seperti Amerika dan Eropa (Poehlman dan Sleper 1995, Purwono dan Purnamawati 2008).
Pertumbuhan kedelai ditentukan oleh ketinggian tempat. Kedelai tumbuh pada daerah yang memiliki ketinggian 0-900 m di atas permukaan laut (dpl) dan akan tumbuh baik pada ketinggian dibawah 500 m dpl. Kondisi curah hujan yang ideal bagi pertumbuhan kedelai adalah lebih dari 1500 mm/tahun dengan curah hujan optimal antara 100-200 mm/bulan. Pertumbuhan terbaik juga
diperoleh pada kisaran suhu 20-30 0C dengan
suhu optimal 25-27 0C dan kelembaban udara
rata-rata sebesar 50%. Intensitas cahaya matahari penuh selama 12 jam sehari merupakan intensitas yang dikehendaki bagi pertumbuhan kedelai (Suprapto 2002, Pitojo 2007).
Pitojo (2007) mengklasifikasikan kedelai (Gambar 1) ke dalam Divisi Spermatophyta,
Subdivisi Angiospermae, Kelas
Dicotyledoneae, Ordo Polypetales, Famili
Leguminosae, Subfamili Papilionoideae,
Genus Glycine, Spesies Glycine max. Kedelai yang dibudidayakan saat ini termasuk dalam spesies Glycine max (L.) Merr. yang berasal
dari kedelai liar yang disebut Glycine
usuriensis (Sumarno dan Harnoto 1983). Secara morfologi, kedelai memiliki akar tunggang yang dapat mencapai kedalaman 150 cm pada tanah yang gembur (Suprapto 2002). Perakaran kedelai dapat membentuk hubungan simbiosis mutualisme dengan bakteri pengikat nitrogen bebas dari udara dengan membentuk bintil akar. Bintil akar tersebut terbentuk setelah tanaman kedelai muda memiliki akar rambut pada akar utama atau cabang (Hidajat 1985).
Hidajat (1985) menyatakan bahwa kedelai memiliki empat tipe daun yang berbeda dalam masa hidupnya. Empat tipe daun tersebut yaitu kotiledon, daun primer sederhana, daun trifoliate dan profilia. Daun primer sederhana
berbentuk oval, berupa daun tunggal
(unfoliate) serta memiliki tangkai sepanjang 1-2 cm dan terletak bersebrangan pada buku pertama di atas kotiledon. Daun trifoliate adalah daun-daun yang terbentuk pada batang
utama dan cabang. Bentuk anak daun dapat berbentuk oval hingga lancip.
Bunga kedelai termasuk bunga sempurna dan memiliki sifat penyerbukan sendiri. Kedelai mulai berbunga pada umur 30-50 hari setelah ditanam dan sekitar 60% bunga rontok sebelum membentuk polong. Buah kedelai berbentuk polong dengan 1-4 biji di dalamnya. Jumlah polong per tanaman bervariasi tergantung varietas, kesuburan tanah dan jarak tanam. Setiap tanaman mampu menghasilkan 100-250 polong, namun pada jarak tanam yang rapat, kedelai hanya mampu menghasilkan 30 polong. Waktu yang dibutuhkan polong sampai masak bervariasi tergantung varietas dan berkisar antara 75-100 hari setelah ditanam (Rukmana dan Yunarsih 1996, Suprapto 2002)
Tanaman kedelai berbatang pendek (30-100 cm), memiliki 3-6 percabangan dan berbentuk tanaman perdu. Batang tanaman kedelai berasal dari poros embrio yang terdapat pada biji masak (Pitojo 2007, Adie dan Krisnawati 2007). Pola pertumbuhan kedelai berdasarkan batang dan bunga
dibedakan menjadi determinate dan
indeterminate. Pola pertumbuhan determinate dicirikan dengan fase vegetatif terhenti setelah fase berbunga, sedangkan fase indeterminate dicirikan dengan pertumbuhan vegetatif berlanjut setelah berbunga. Di samping kedua pola pertumbuhan tersebut dikenal juga pola pertumbuhan semideterminate yang memiliki pola pertumbuhan diantara kedua tipe tersebut. Kedelai di Indonesia pada umumnya
memiliki pola pertumbuhan determinate (Adie
dan Krisnawati 2007). Contoh genotipe kedelai Indonesia yang memiliki pola
pertumbuhan determinate yaitu genotipe
Tambora dan grobogan.
Genotipe kedelai yang digunakan pada penelitian ini adalah genotipe Tambora, grobogan dan B3293. Genotipe Tambora merupakan kedelai yang dihasilkan melalui
tetua yang berasal dari IRRI Philipina dan dimuliakan oleh Sumarno, Rodiah, Ono Sutrisno dan Cheppy Syukur. Ciri-ciri genotipe ini yaitu hipokotil berwarna unggu, bunga berwarna ungu, kulit polong tua berwarna coklat tua, dan biji berwarna kuning mengkilat. Rataan hasil kedelai tambora yaitu sekitar 1.5 ton/ha. Genotipe ini memiliki keunggulan toleran terhadap penyakit karat. Sedangkan, genotipe Grobogan merupakan kedelai yang berasal dari pemurnian populasi lokal Malabar Grobogan dan dimuliakan oleh Suhartina dan M. Muclisch Adie. Potensi hasil yang dapat diperoleh dari varietas ini yaitu sebanyak 3.4 ton/ha dengan keunggulan umur panen lebih pendek, polong lebih besar dan
tingkat kematangan polong dan daun
bersamaan (Pusat Penelitian dan
pengembangan Tanaman Pangan 2012). Genotipe B3293 merupakan kedelai yang berasal dari Kediri, termasuk kedelai yang bersifat genjah dan sensitif terhadap toksisitas aluminium. Genotipe ini digunakan sebagai tetua dalam persilangan untuk pemuliaan kedelai yang toleran terhadap toksisitas
aluminium (BB-Biogen 2004, Tasma et al.
2008).
Pustaka Genom
Pustaka genom diartikan sebagai
kumpulan sekuens (urutan) DNA dari suatu organisme (Wulandari 2009). Suharsono (2002) menyatakan bahwa pustaka genom mengandung semua gen yang dipunyai oleh suatu organisme, termasuk daerah penyandi sehingga sangat penting untuk menyimpan seluruh informasi genetika yang dipunyai oleh suatu organisme. Pustaka genom sangat
bermanfaat dalam usaha isolasi dan
karakterisasi suatu gen dan dapat digunakan untuk pemetaan gen secara fisik. Peta genetik ini sangat penting di dalam program pemuliaan tanaman secara konvensional maupun secara molekuler. Oleh karena itu, pustaka genom merupakan bagian yang sangat penting dalam program perbaikan genetika tanaman secara molekuler maupun melalui pemuliaan konvensional.
Berdasarkan sumber DNA yang
digunakan, pustaka gen dapat dibedakan menjadi dua jenis yaitu pustaka genom dan pustaka cDNA. Pustaka genom menggunakan DNA genomik/kromosom sebagai sumber DNA yang digunakannya. Sementara itu, pustaka cDNA menggunakan DNA hasil transkripsi balik dari suatu populasi mRNA. Hal yang perlu diperhatikan dalam melakukan konstruksi pustaka genom yaitu bahwa
pustaka yang dibuat harus merepresentasikan semua gen yang ada di dalam sumber DNA asalnya. Suatu pustaka dikatakan representatif jika mengandung semua sekuens aslinya. Di samping itu, jika suatu pustaka tidak mengandung klon dalam jumlah yang
mencukupi maka sangat dimungkinkan
hilangnya beberapa gen tertentu. Pustaka genom yang representatif diperoleh dengan memurnikan DNA genom yang kemudian
dipotong-potong secara acak menjadi
fragmen-fragmen dengan ukuran yang
diperlukan. Fragmentasi DNA dapat
dilakukan dengan dua cara yaitu secara fisik contohnya dengan sonikasi atau nebulisasi dan
fragmentasi secara enzimatik dengan
menggunakan enzim restriksi (Wahyudi 2001).
Pustaka genom dapat dikonstruksi dengan menggunakan vektor yang disisipkan pada sel
inang. Vektor yang diperlukan dalam
pembuatan pustaka genom dapat berupa
bakteriofage, P1 artificial chromosome
(PAC), yeast artificial chromosome (YAC),
bacterial artificial chromosome (BAC) atau kosmid. Pemilihan vektor-vektor tersebut tergantung pada ukuran panjang fragmen
DNA (Wahyudi 2001). Menurut Woo et al.
(1994), pustaka DNA sebagian besar
dikonstruksi melalui vektor bakteriofage, kosmid dan YAC. Ukuran panjang fragmen DNA yang dapat disisipkan dalam vektor-vektor tersebut yaitu maksimal 17 kb untuk bakteriofage, 46 kb untuk kosmid, 100 kb untuk PAC dan 2000 kb untuk YAC.
Penggunaan vektor dalam konstruksi pustaka genom memiliki beberapa kendala. Ukuran fragmen DNA yang dapat disisipi hanya dapat dilakukan untuk fragmen berukuran kecil. Walaupun, yeast artificial chromosome (YAC) dapat disisipkan ukuran fragmen yang cukup besar berkisar 2000 kb
namun dalam aplikasinya mengalami
beberapa kendala seperti banyaknya DNA khimera yang tidak diinginkan dan sulitnya mengisolasi DNA yang telah disisipkan dalam YAC (Woo et al. 1994).
Konstruksi pustaka genom untuk
sekuensing genom total menggunakan Next
Generation Sequencing (NGS) memiliki metode yang berbeda. Metode konstruksi pustaka genom yang digunakan memiliki prinsip yang sama dengan konstruksi genom
dengan menggunakan metode shotgun
4
fragmentasi DNA berbeda antara NGS dengan shotgun sequencing. Pada metode shotgun sequencing, DNA yang telah difragmentasi tidak dilakukan ligasi adapter tetapi langsung dilakukan sekuensing. Hal tersebut berbeda
dengan konstruksi pustaka genom
menggunakan NGS, fragmen DNA yang dihasilkan sebelum disekuens dipreparasi dengan menempelkan adapter pada ujung fragmen DNA. Adapter tersebut pada tahapan selanjutnya (klasterisasi pustaka genom) akan
menempel pada sebuah flow cell (Lampiran 2)
untuk amplifikasi fragmen DNA melalui metode bridge amplification (Mardis 2008, Metzker 2010, Commins et al. 2011).
Sekuensing DNA
Sekuensing DNA atau pengurutan basa DNA merupakan teknik kunci untuk berbagai perkembangan ilmu pengetahuan di antaranya
arkeologi, antropologi, ilmu genetika,
bioteknologi, biologi molekular dan ilmu forensik (Franca et al. 2002). Sekuensing DNA digunakan untuk menentukan urutan basa nitrogen (adenin, guanin, sitosin dan timin) dalam suatu DNA. Salah satu contoh aplikasi sekuensing DNA yaitu dalam pengurutan genom manusia melalui proyek
genom manusia atau Human Genome Project.
Contoh lain yaitu dalam ilmu pengobatan, sekuensing DNA dapat digunakan untuk
mengidentifikasi, mendiagnosis, dan
mengembangkan pengobatan penyakit
genetik. Awal tahun 1970 merupakan
perkembangan awal dari sekuensing DNA dengan metode yang digunakan yaitu metode
kromatografi. Perkembangan selanjutnya
mulai diperkenalkan metode sekuensing DNA
dengan menggunakan metode dye based
sequencing (Olsvik 1993).
Metode sekuensing DNA yang dikenal sekarang adalah metode enzimatik yang diperkenalkan oleh Sanger dan metode kimiawi yang diperkenalkan Maxam-Gilbert. Sebelum dikenal metode enzimatik atau metode terminasi rantai, Sanger dan Coulson (1975) terlebih dahulu memperkenalkan sekuensing DNA yang disebut metode plus-minus. Metode ini menggunakan DNA polimerase I yang berasal dari Escherichia coli dan DNA polimerase dari bakteriofage T4
(Englund 1971,1972). Kedua DNA
polimerase tersebut mempunyai perbedaan dalam urutan nukleosida trifosfat. Produk yang dihasilkan dari tahapan polimerase kemudian diselesaikan dengan menggunakan elektroforesis dalam gel akrilamid. Dua tahun setelah memperkenalkan metode plus-minus,
Sanger kemudian memperkenalkan metode sekuensing yang lain dengan menggunakan polimerisasi enzimatik atau dikenal juga dengan nama metode terminasi rantai (Sanger dan Coulson 1975).
Perpanjangan atau ekstensi rantai DNA pada metode enzimatik Sanger, dimulai dari situs spesifik pada DNA cetakan dengan
menggunakan sebuah primer atau
oligonukleotida pendek yang komplementer terhadap DNA pada daerah situs tersebut. Primer tersebut diperpanjang menggunakan DNA polimerase (enzim yang mereplikasi DNA). Di samping itu, dalam metode enzimatik juga ditambahkan empat jenis basa deoksinukleotida dan nukleotida pemutus atau penghenti rantai (terminator rantai) yang biasanya dalam bentuk di-deoksinukleotida. Pengikatan nukleotida pemutus rantai dengan rantai DNA yang dibantu oleh DNA polimerase menghasilkan fragmen-fragmen DNA yang berhenti memanjang hanya pada posisi DNA tempat nukleotida tersebut berikatan. Fragmen-fragmen DNA tersebut dideteksi dengan menandai (labelling) primer yang digunakan dengan fosfor radioaktif sebelum reaksi sekuensing dimulai. Keempat hasil reaksi tersebut kemudian dielektroforesis pada empat lajur yang berdekatan pada gel akrilamid. Perkembangan selanjutnya dari sekuensing DNA metode sanger adalah
penggunaan flourescent dye sebagai penanda
ujung primer. Hal ini memiliki kelebihan karena tidak menggunakan bahan radioaktif serta menambah keamanan dan kecepatan karena keempat hasil reaksi dapat dicampur dan dielektroforesis pada satu lajur pada gel. Metode ini dikenal juga sebagai metode dye primer sequencing (Franca et al. 2002).
Pada waktu yang hampir bersamaan dengan dikenalkannya metode sekuensing Sanger, Allan Maxam dan Walter Gilbert
tepatnya pada tahun 1976-1977
mengembangkan metode sekuensing DNA yang didasarkan pada modifikasi kimiawi DNA yang dilanjutkan dengan pemotongan DNA (Pettersson et al. 2009). Metode ini awalnya cukup populer karena dapat langsung
menggunakan DNA hasil pemurnian.
Sedangkan, metode Sanger pada waktu itu memerlukan kloning untuk membentuk DNA
untai tunggal. Seiring dengan
Metode lain yang dapat digunakan dalam
sekuensing DNA adalah metode
pyrosequencing yakni teknik pemetaan DNA yang berdasarkan deteksi terhadap pirofosfat (PPi) yang dilepaskan selama sintesis DNA. Teknik ini memanfaatkan reaksi enzimatik yang dikatalisis oleh ATP sulfurilase dan luciferase untuk pirofosfat anorganik yang dilepaskan selama penambahan nukleotida (Poirel et al. 2006).
Sekuens DNA skala besar dibutuhkan dalam sekuensing genom. Berbagai strategi telah dikembangkan untuk sekuensing DNA skala besar, termasuk strategi primer walking dan shotgun sequencing. Kedua strategi tersebut melibatkan pembacaan banyak bagian DNA dengan metode Sanger dan selanjutnya menyusun hasil pembacaan tersebut menjadi sekuens yang runut. Masing-masing strategi memiliki kelemahan sendiri dalam hal kecepatan dan ketepatan, contohnya metode shotgun sequencing merupakan metode yang paling praktis untuk sekuensing genom ukuran besar, namun proses penyusunannya rumit
dan rentan kesalahan. Perkembangan
sekuencing skala besar selanjutnya disebut sebagai Next Generation Sequencing (Mardis 2008).
Next Generation Sequencing Next Generation Sequencing (NGS) merupakan istilah yang digunakan untuk
menyebutkan perkembangan metode
sekuensing DNA setelah metode yang diperkenalkan oleh Sanger dan Maxam-Gilbert pada tahun 1977. Metode yang diperkenalkan dalam NGS memungkinkan sekuensing dapat dilakukan dalam skala besar dengan waktu yang relatif singkat dan tingkat kesalahan yang lebih kecil daripada metode sekuensing sebelumnya (Schuster 2008). Metzker (2010) menyatakan bahwa metode sekuensing Sanger disebut juga sebagai generasi sekuensing yang pertama.
Instrumentasi NGS mulai diperkenalkan
tahun 1990-an melalui Massive Parallel
Signature Sequencing (MPSS) yang
dikembangkan oleh Lynx Therapeutics,
sebuah perusahaan yang didirikan oleh Sydney Brenner dan Sam Eletr. Perusahaan tersebut pada tahun 2004 bergabung dengan Solexa dan berganti nama menjadi Illumina.
Metode MPSS adalah metode bead-based
yang menggunakan pendekatan kompleks penempelan adaptor yang selanjutnya akan dibaca melalui adaptor decoding (Brenner et al. 2000). Instrumen NGS yang lain yaitu
Polony Sequencing, sebuah instrumen yang diperkenalkan pada tahun 2005 oleh Laboratory of George Chruch, Harvard.
Metode NGS dengan Polony Sequencing
merupakan NGS pertama yang dapat
mengurutkan genom pertama kali melalui kombinasi in vitro paired-tag library dengan emulsi PCR dan ligasi yang berdasarkan pada sekuensing kimiawi. Genom yang berhasil disekuens adalah genom yang berasal dari Escherichia coli. Apabila dibandingkan dengan metode sekuensing yang dilakukan
dengan menggunakan metode Sanger,
sekuensing genom total menggunakan NGS memiliki tingkat akurasi yang tinggi yaitu 99,99%. Biaya yang dibutuhkan juga lebih murah yaitu satu per sepuluh dari biaya yang
dibutuhkan untuk mengurutkan genom
melalui metode sanger (Schuster 2008).
Instrumen NGS yang lain yaitu
pyrosequencing (454 Life Sciences), SOLiD sequencing ( Applied Biosystem) , ion semicunductor sequencing (Ion Torrent System Inc.), DNA nanoball sequencing, dan helioscope single molecule sequencing, (Margulies et al 2005, Valouev et al 2008,
Rusk 2011). Selain yang disebutkan
sebelumnya, instrumen NGS yang lain yaitu HiSeq2000 (Gambar 2) yang dikembangkan
oleh Illumina. HiSeq2000 mempunyai
kemampuan untuk mengurutkan lebih dari lima genom manusia dalam 30 kali cakupan secara bersamaan, serta mengurutkan lebih dari 192 ekspresi gen. Instrumen ini menghasilkan 540-600 Gb dengan panjang fragmen 2 x 100 bp selama 11 hari. Secara
umum, terdapat empat tahapan yang
dilakukan dalam sekuensing DNA
menggunakan HiSeq2000 yaitu preparasi sampel pustaka genom, klasterisasi pustaka genom, sekuensing dan analisis data sekuens (Illumina 2010a).
Gambar 2 Next Generation Sequencing
6
Prinsip sekuensing menggunakan NGS HiSeq2000 adalah Sequencing by Synthesis
(SBS) (Lampiran 3). Selama proses
sekuensing keempat basa nitrogen penyusun DNA (adenin, timin, guanin dan sitosin) dialirkan secara simultan ke dalam flow cell dan akan berikatan dengan fragmen DNA pada klaster dengan dibantu oleh DNA polimerase. Secara spesifik, keempat basa tersebut membawa penanda berupa flourescen yang akan berpendar ketika basa-basa tersebut berikatan membentuk utas DNA. Utas DNA yang terbentuk merupakan utas DNA yang komplemen dengan fragmen DNA pada klaster. Gugus 3’OH pada basa-basa tersebut telah mengalami modifikasi sehingga tidak dapat berikatan dengan basa penyusun yang lain sebelum satu siklus sintesis telah selesai dilakukan. Satu siklus telah selesai jika pendaran flourescen yang dihasilkan dari basa yang telah disintesis dideteksi oleh sebuah kamera CCD yang terdapat pada NGS HiSeq2000. Kamera CCD tersebut akan menangkap pendaran flourescen hasil reaksi pembentukan utas DNA dan akan mencitrakan
hasilnya melalui tampilan Real Time Analysis
pada monitor NGS HiSeq2000. Di samping
itu, berakhirnya satu siklus sintesis juga ditandai dengan putusnya ikatan kimia penanda flourescen dan gugus 3’OH termodifikasi yang selama sintesis berikatan dengan fragmen DNA dalam klaster (Mardis 2008, Ansorge 2009, Metzker 2010).
Klasterisasi Pustaka Genom Klasterisasi pustaka genom merupakan tahapan yang harus dilakukan sebelum
sekuensing menggunakan Next Generation
Sequencing Hiseq2000. Klasterisasi pustaka genom dilakukan melalui lima tahapan yaitu denaturasi, immobilisasi dan perpanjangan DNA, amplifikasi, linearisasi dan hibridisasi (Lampiran 3). Klasterisasi bertujuan untuk menghasilkan klaster atau kelompok salinan dari pustaka genom yang akan disekuen (Mardis 2008). Tahap awal klasterisasi pustaka genom yaitu denaturasi utas ganda
DNA menjadi utas tunggal dengan
menambahkan NaOH pada larutan pustaka genom yang akan disiapkan untuk proses klasterisasi. Utas tunggal yang telah terbentuk akan mengalami immobilisasi dengan cara menempel pada oligonukleotida di permukaan flow cell. Setelah terimmobilisasi, utas tunggal akan membentuk salinan pustaka genom dengan oligonukleotida yang menempel pada permukaan flow cell berperan sebagai primer dalam proses penyalinan tersebut. Setelah
salinan terbentuk, utas tunggal yang berperan sebagai cetakan akan terdenaturasi kembali dan menyisakan utas salinan pustaka genom yang berikatan dengan oligonukleotida pada flow cell.
Tahapan selanjutnya yaitu amplifikasi
pustaka genom dengan metode bridge
amplification. Dalam metode amplifikasi tersebut, adapter pada pustaka genom akan menempel pada olinukleotida yang lain di
permukaan flow cell sehingga membentuk
lengkungan seperti jembatan (Lampiran 4). Setelah proses bridge amplification selesai, salinan pustaka genom akan kembali ke bentuk awal dan tidak membentuk lengkungan kembali. Hasil akhirnya akan terbentuk klaster salinan dari pustaka genom yang akan disekuen. Namun, beberapa salinan pustaka genom dalam klaster akan mengalami denaturasi secara acak sehingga menyebabkan oligonukleotida pada flow cell terblokir dan tidak dapat digunakan kembali. Hal tersebut bertujuan untuk mencegah gangguan dan mengurangi tingkat kesalahan pada proses sekuensing (Illumina 2010b).
BAHAN DAN METODE
Alat dan Bahan
Alat-alat yang digunakan meliputi mortar, alu, tabung 15 mL, tabung 2 mL, tabung 1.5
mL, penangas air, inkubator thermostat,
sentrifus 5810X effendorf, thermolyne vortex mixer maxi II, mikropipet, tip pipet,
spektrofotometer nanodrop thermoscientific
2000c, tabung nebulisasi invitrogen, selang nebulisasi, tabung gas nitrogen, microwave sanyo, mikrosentrifus VWR minifuge galaxy minister C1413, kotak es, sarung tangan karet, termos, neraca analitik, cetakan gel, sisir, alat pengering, thermal cycler biometra T1, sudip, gelas piala, parafilm, power supply, perangkat elektroforesis, kolom elusi gel minElute Spin, UV Transilluminator DigiDoc-lt Imaging System, magnetic stand, mesin pendingin tropicalized Sansio, Illumina cBot Cluster Generation, Illumina Hiseq 2000.
Bahan-bahan yang digunakan untuk isolasi DNA adalah daun kedelai, es batu, etanol, nitrogen cair, bufer ekstraksi CTAB (Cetil
Trimetil Amonium Bromida), NaOAc
gliserol 50%, 1 x TAE, akuades, isolat DNA, resuspension buffer, etanol absolut, etanol 70%, NaOAc, serbuk agarosa. Bahan-bahan yang digunakan untuk modifikasi ujung
fragmen DNA adalah end repair control, end
repair mix, AMPure XP Beads, etanol 80%, dan resuspension buffer. Bahan-bahan yang digunakan untuk adenilasi ujung 3’OH DNA adalah A-tailing control dan A-tailing mix. Bahan-bahan yang digunakan untuk ligasi adapter adalah ligase control, DNA ligase mix, DNA adapter index, stop ligase mix, AMPure XP Beads, etanol 80%, dan resuspension buffer. Bahan-bahan yang digunakan untuk purifikasi DNA hasil ligasi adalah serbuk agarosa, 1 x TAE, SyBr Gold, loading dye, DNA ladder, bufer QG, bufer PE, dan bufer EB. Bahan-bahan yang digunakan untuk
amplifikasi DNA adalah PCR Primer
Cocktail, PCR Master Mix, AMPure XP Beads, etanol 80%, dan Resuspension Buffer.
Bahan-bahan yang digunakan untuk
klasterisasi dan sekuensing genom kedelai adalah 2 N NaOH, bufer hibridisasi, Tris-Cl, buffer library, TruSeq SR Cluster Kit v3-cBot-HS, Truseq SBS Kit-HS (200 cycle).
Metode
Isolasi DNA Genom Kedelai (Modifikasi Michiels et al.. 2002)
Sampel daun muda ditimbang sebanyak
300 mg dan digerus sampai halus
menggunakan mortar yang sebelumnya telah dibasuh dengan nitrogen cair. Bufer ekstraksi yang telah dipanaskan sebelumnya pada suhu 600C ditambahkan sebanyak 5 mL pada daun yang telah halus dan dicampurkan secara perlahan dalam tabung 15 mL. Sampel
diinkubasikan pada suhu 600C selama 60
menit dan secara berkala dilakukan
pencampuran untuk menghindari
penggumpalan. Sebanyak 5 mL kloroform :isoamilalkohol (24 :1) ditambahkan ke dalam sampel dan dicampurkan dengan cara dibolak-balik secara perlahan selama 5 menit. Campuran tersebut kemudian disentrifus selama 5 menit pada 2500 g (suhu 200C).
Kemudian lapisan atas hasil sentrifus
dipindahkan ke dalam tabung baru. Langkah
penambahan kloroform:isoamilalkohol
diulangi kembali dan tabung yang berisi sampel dicampur dengan cara dibolak-balik secara perlahan selama 5 menit sebelum
disentrifus sebanyak 2 kali untuk
menjernihkan supernatan. Sebanyak 3.5 mL isopropanol ditambahkan ke dalam tabung yang berisi supernatan. Sampel diinkubasi
pada suhu 250C selama satu malam untuk mengendapkan DNA. Kemudian, sampel disentrifus selama 10 menit pada 5000g
dengan suhu 200C. Supernatan yang
dihasilkan dibuang dan ditambahkan 5 mL larutan pencuci ke dalam tabung yang berisi pelet. Sampel diinkubasikan selama 15 menit pada suhu ruang. Setelah 15 menit, sampel disentrifus kembali pada 2500 g selama 6
menit dengan suhu 200C dan tahapan
pencucian diulangi satu kali lagi. Supernatan hasil sentrifus dan pencucian kemudian dibuang dan pelet yang dihasilkan dikeringkan pada udara terbuka sebelum ditambahkan 300 µL bufer TE.
Uji Kualitatif DNA Genom Kedelai
Uji kualitatif DNA dilakukan untuk mengetahui kualitas DNA genom kedelai yang telah berhasil diisolasi. Uji kualitatif DNA menggunakan elektroforesis gel agarosa 1%. Gel agarosa dibuat dengan menambahkan agarosa sebanyak 0.55 g dalam 55 mL larutan TAE 1X. Kemudian dipanaskan hingga larut dan didinginkan pada suhu kamar hingga hangat. Selanjutnya ditambahkan 5.5 µL SyBr Gold dan dituang ke dalam cetakan gel elektroforesis yang telah dipasangi sisir (cetakan sumur) hingga gel memadat. Gel yang sudah padat dipindahkan ke dalam bak elektroforesis yang berisi TAE 1X. Sampel yang akan dielektroforesis dicampur dengan loading buffer dengan perbandingan 1:10 pada parafilm. Setelah tercampur maka campuran sampel diinjeksi ke dalam sumur gel agarosa. Marker yang digunakan adalah 100 bp plus DNA ladder sebanyak 6 µL. Setelah semua
sampel selesai diinjeksi maka alat
elektroforesis dihubungkan pada power supply
yang dialiri tegangan listrik 100 volt selama 30 menit. Hasil elektroforesis diamati dengan bantuan lampu UV dalam trasnsilluminator. Uji Kuantitatif DNA Genom Kedelai
Uji kuantitatif dilakukan dengan
menggunakan spektrofotometer nanodrop
thermoscientific, USA. Blanko yang digunakan adalah larutan TE. Sebelum digunakan lubang optik dibersihkan terlebih dahulu. Larutan TE dimasukkan ke dalam lubang optik sebanyak 2 µL. Nanodrop thermoscientific ditutup kemudian dipilih
menu measure blank pada komputer.
8
langsung dalam A280 dan A260. Uji kuantitas DNA dilakukan untuk mengetahui konsentrasi dan kemurnian DNA dengan perbandingan A260/280 yang baik sekitar 1.8 hingga 2.0. Fragmentasi DNA (Invitrogen 2004)
Fragmentasi DNA dilakukan dengan
menggunakan metode nebulisasi DNA.
Sebanyak 700 µL bufer nebulisasi
ditambahkan ke dalam tabung yang berisi 25 µg DNA dan dicampurkan sampai homogen. Semua campuran sampel yang telah homogen dipindahkan ke dalam tabung nebulisasi yang disimpan dalam es dan diberikan gas dengan tekanan 40 psi selama 6 menit. Kemudian, disentrifus pada 450 rpm selama 2 menit dengan suhu 250C. Sampel sebanyak 360 µL dipindahkan ke dalam tabung yang baru dan disimpan dalam es. Sebanyak 40 µL natrium asetat dan 1000 µL etanol absolut ditambahkan ke dalam setiap tabung dan
dicampurkan sampai homogen sebelum
diinkubasi dalam es selama 30 menit. Kemudian, sampel disentrifus pada 12000
rpm selama 10 menit dengan suhu 150C.
Supernatan yang dihasilkan dibuang dan 500 µL etanol 70% ditambahkan ke dalam tabung yang berisi pelet. Sampel disentrifus kembali pada 12000 rpm selama 10 menit dengan suhu
150C. Supernatan dibuang dan pelet
dikeringkan. Resuspension Buffer
ditambahkan sebanyak 50 µL dan
dicampurkan sampai homogen. Sampel DNA diambil sebanyak 5 µL untuk mengetahui kualitas fragmen DNA yang dihasilkan dengan elektroforesis gel agarosa. Untuk mengetahui kuantitas DNA yang dihasilkan, sebanyak 2 µL diambil dan dianalisis dengan menggunakan nanodrop thermoscientific. Konstruksi Pustaka Genom (TruSeq DNA low throughput protocol 2010c)
Preparasi Fragmen DNA cetakan. Sebanyak 1 µg DNA hasil fragmentasi DNA
dilarutkan dalam 52.5 µL Resuspension
Buffer. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Untuk mengetahui kuantitas fragmen DNA cetakan, sebanyak 2 µL diambil dan dianalisis dengan menggunakan nanodrop thermoscientific.
Modifikasi Ujung Fragmen DNA. Modifikasi ujung fragmen DNA diawali dengan pembuatan Insert Modification Plate (IMP). Sebanyak 10 µL End repair control
dan 40 µL End Repair Mix ditambahkan
dalam tabung yang mengandung 50 µL fragmen DNA cetakan. Sampel dicampurkan
menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Tabung yang berisi sampel ditutup dan diinkubasi dalam thermal cycler selama 30 menit pada suhu 300C.
Langkah selanjutnya yaitu purifikasi IMP yang diawali dengan penambahan 160 µL AMPure XP Beads ke dalam sampel. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali.
Campuran sampel dan AMPure XP Beads
diinkubasi selama 15 menit pada suhu ruang.
Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 15 menit. Sebanyak 127.5 µL supernatan dibuang dengan hati-hati dan jangan sampai mengenai pelet yang terletak pada dinding tabung. Tahapan sebelumnya diulangi untuk menghilangkan supernatan yang masih tersisa. Untuk pencucian pelet, sebanyak 200 µL etanol 80% ditambahkan dan diinkubasi selama 30 detik. Selanjutnya, etanol 80% yang telah ditambahkan dibuang dan tahapan penambahan etanol 80% diulangi kembali satu kali. Pelet kemudian dikeringkan selama 15 menit pada suhu ruang dan
dipindahkan dari magnetic stand.
Resuspension buffer sebanyak 17.5 µL ditambahkan ke dalam tabung yang berisi pelet. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Selanjutnya, sampel diinkubasi selama 2 menit pada suhu ruang.
Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 5 menit untuk memisahkan supernatan dengan pelet. Sebanyak 15 µL supernatan dipindahkan ke dalam tabung yang baru dan disimpan dalam mesin pendingin.
Adenilasi Ujung 3’ DNA. A-tailing control 2.5 µL dan A-tailing mix 12.5 µL ditambahkan ke dalam tabung yang berisi sampel hasil tahapan sebelumnya. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Tabung yang berisi sampel ditutup dan diinkubasi dalam thermal cycler selama 30 menit pada suhu 370C.
yang berisi sampel. Kemudian, sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Langkah selanjutnya yaitu pemurnian sampel yang diawali dengan penambahan 160 µL AMPure XP Beads ke dalam sampel. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak
10 kali. Campuran sampel dan AMPure XP
Beads diinkubasi selama 15 menit pada suhu ruang. Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 2 menit. Sebanyak 127,5 µL supernatan dibuang dengan hati-hati agar tidak sampai mengenai pelet yang terletak pada dinding tabung. Tahapan sebelumnya diulangi untuk menghilangkan supernatan yang masih tersisa. Untuk pencucian pelet, sebanyak 200 µL etanol 80% ditambahkan dan campuran diinkubasi selama 30 detik.
Selanjutnya, etanol 80% yang telah
ditambahkan dibuang dan tahapan
penambahan etanol 80% diulangi kembali satu kali. Pelet kemudian dikeringkan selama 15 menit pada suhu ruang dan dipindahkan
dari magnetic stand. Resuspension buffer
sebanyak 22.5 µL ditambahkan ke dalam tabung yang berisi pelet. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Selanjutnya, sampel diinkubasi selama 2 menit pada suhu ruang. Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 2 menit untuk memisahkan supernatan dengan pelet. Sebanyak 20 µL supernatan dipindahkan ke dalam tabung yang baru dan disimpan dalam mesin pendingin.
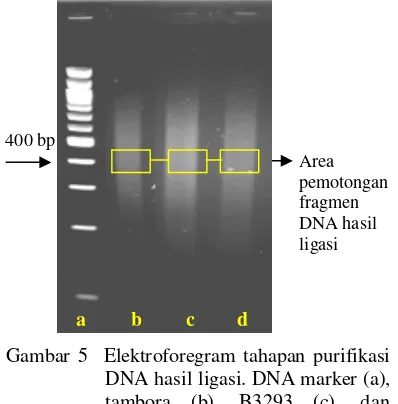
Purifikasi DNA Hasil Ligasi. Purifikasi hasil ligasi terdiri dari dua tahap yaitu pemisahan ukuran fragmen DNA melalui elektroforesis dan pemisahan fragmen DNA yang telah dielektroforesis melalui proses ekstraksi gel DNA. Pemisahan ukuran fragmen DNA diawali dengan pembuatan gel agarosa 2%. Sebanyak 1.1 gram bubuk agarosa ditimbang dan ditambahkan larutan TAE 1x sebanyak 55 mL. Larutan kemudian
dipanaskan dalam microwave sampai semua
agarosa larut. Setelah suhu larutan hangat,
kemudian tambahkan 5,5 uL pewarna SyBr
Gold dan dicampurkan hingga homogen.
Kemudian dituang larutan ke dalam cetakan yang telah disiapkan.
Elektroforesis diawali dengan
disiapkannya seperangkat bak elektroforesis, kemudian larutan TAE 1x dituangkan ke dalam bak hingga gel terendam penuh. Selanjutnya, ditempat terpisah diteteskan 1 µL
larutan loading dye pada microfilm. Sebanyak 5 µL sampel DNA dicampurkan dengan loading dye. Untuk penanda disiapkan 6 µL DNA ladder. Sampel kemudian dimasukkan ke dalam sumur gel yang tersedia. Sampel tersebut kemudian di elektroforesis pada kuat arus 120 volt selama 120 menit. Hasil elektroforesis dilihat di atas lampu UV
transiluminator dan didokumentasikan.
Ukuran fragmen DNA antara rentang 200-500 bp yang memiliki intensitas yang lebih tinggi diiris dari gel agarosa dan disimpan dalam tabung untuk digunakan pada tahapan selanjutnya.
Tahapan ekstraksi gel hasil elektroforesis diawali dengan penimbangan irisan gel. Tiga volume Bufer QG ditambahkan untuk satu volume gel (100 mg = 100 µL). Tabung yang berisi campuran gel dan bufer QG diinkubasi pada suhu 50 0C atau sampai gel agarosa mencair seluruhnya. Setelah gel larut, warna campuran dicek untuk menyamakan dengan warna bufer QG yang berwarna kuning. Jika campuran berwarna jingga atau violet, sebanyak 10 µL natrium asetat 3 M, pH 5 ditambahkan ke dalam campuran sampel. Selanjutnya, isopropanol sebanyak 1 volume gel ditambahkan ke dalam sampel dan
dicampurkan sampai homogen. Sampel
dipindahkan dalam MinElute Spin Colomn
dan disentrifus selama 1 menit pada 13000 rpm. Supernatan yang dihasilkan dibuang dan
ke dalam MinElute Spin Colomn ditambahkan
500 µL bufer QG. Sampel kemudian disentrifus kembali selama 1 menit pada 13000 rpm. Supernatan yang dihasilkan dibuang. Untuk pencucian, sebanyak 750 µL
bufer PE ditambahkan ke dalam MinElute
Spin Colomn. Supernatan yang dihasilkan
dibuang dan MinElute Spin Colomn
disentrifus kembali selama 1 menit pada
13000 rpm. Selanjutnya, MinElute Spin
Colomn disimpan dalam tabung mikrosentrifus 1.5 mL. Untuk mengelusi DNA, sebanyak 25 µL bufer EB (10 mM
Tris-HCl, pH 8.5) ditambahkan ke dalam MinElute
Spin Colomn dan disimpan dalam suhu ruang selama 1 menit. Selanjutnya, MinElute Spin Colomn disentrifus selama 1 menit pada 13000 rpm. Sebanyak 20 µL dipindahkan ke dalam tabung dan disimpan dalam mesin pendingin untuk tahapan selanjutnya.
10
sebanyak 10 kali. Tabung kemudian disimpan dalam mesin PCR. Proses amplifikasi terdiri dari 16 siklus selama 1 jam dengan rincian reaksi predenaturasi pada suhu 98°C selama 30 detik, denaturasi pada suhu 98°C selama 10 detik, annealing pada suhu 60°C selama 30 detik, extension pada suhu 72°C selama 30 detik, dan pasca pemanjangan primer pada suhu 72°C selama 5 menit.
Tahapan selanjutnya yaitu pemurnian hasil PCR yang diawali dengan penambahan 50 µL AMPure XP Beads ke dalam sampel. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali.
Campuran sampel dan AMPure XP Beads
diinkubasi selama 15 menit pada suhu ruang.
Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 2 menit. Sebanyak 95 µL supernatan dibuang dengan hati-hati jangan sampai mengenai pelet yang terletak pada dinding tabung. Ulangi tahapan sebelumnya untuk menghilangkan supernatan yang masih tersisa. Untuk pencucian pelet, sebanyak 200 µL etanol 80% ditambahkan dan diinkubasi selama 30 detik. Selanjutnya, etanol 80% yang telah ditambahkan dibuang dan tahapan penambahan etanol 80% diulangi kembali satu kali. Pelet kemudian dikeringkan selama 15 menit pada suhu ruang dan dipindahkan
dari magnetic stand. Resuspension buffer
sebanyak 32.5 µL ditambahkan ke dalam tabung yang berisi pelet. Sampel dicampurkan menggunakan pipet mikro dengan cara dinaik turunkan sebanyak 10 kali. Selanjutnya, sampel diinkubasi selama 2 menit pada suhu ruang. Tabung yang berisi sampel kemudian
dipindahkan dalam magnetic stand pada suhu
ruang selama 2 menit untuk memisahkan supernatan dengan pelet. Sebanyak 30 µL supernatan dipindahkan ke dalam tabung yang baru dan disimpan dalam mesin pendingin.
Validasi pustaka genom. Validasi pustaka genom dilakukan melalui uji kualitatif dan kuantitatif untuk mengetahui ukuran dan konsentrasi dari pustaka genom sebelum
disekuens menggunakan next generation
sequencing HiSeq2000. Pustaka genom yang akan disekuens harus memenuhi beberapa persyaratan yaitu memiliki panjang fragmen sekitar 300-500 bp dengan konsentrasi 10 nM. Uji kualitatif dilakukan dengan metode elektroforesis gel agarosa. Sampel DNA diambil sebanyak 5 µL dan dielektroforesis pada gel agarosa dengan konsentrasi 1% selama 40 menit pada kuat arus 120 volt. Untuk mengetahui kuantitas pustaka genom yang dihasilkan, sebanyak 2 µL sampel
diambil dan dianalisis dengan menggunakan spektrofotometer nanodrop thermoscientific. Klasterisasi Pustaka Genom Kedelai (cBot cluster generation protocol 2010b)
Klasterisasi DNA diawali dari preparasi sampel yang terdiri dari dua tahapan, yaitu denaturasi dengan menggunakan 2 N NaOH dan pelarutan DNA dalam bufer hibridisasi. Denaturasi diawali dengan penambahan 17 µL Tris-Cl 10 mM, pH 8.5 dan 1 µL 2 N NaOH ke dalam 2 µL sampel DNA sehingga diperoleh konsentrasi DNA sebesar 1 nM. Campuran tersebut kemudian dicampurkan
sampai homogen dengan menggunakan
vortek. Sampel diinkubasi selama 5 menit pada suhu ruang untuk mendenaturasi utas ganda DNA menjadi utas tunggal. DNA yang telah didenaturasi disimpan dalam mesin pendingin sampai proses pelarutan DNA akan dilakukan.
Tahapan preparasi sampel selanjutnya yaitu pelarutan DNA dalam bufer hibridisasi. Tahapan ini diawali dengan pengenceran 1 nM DNA hasil denaturasi menjadi DNA yang memiliki konsentrasi 7 pM dan 13 pM. Konsentrasi tersebut didapat dari pengenceran 7 µL sampel DNA dalam 993 µL bufer hibridisasi (7 pM) dan 13 µL sampel DNA dalam 987 µL bufer hibridisasi (13 pM). Sebanyak 120 µL control library disiapkan dalam tabung pertama dalam suatu lajur yang memiliki 8 tabung untuk digunakan sebagai kontrol dalam flow cell. Sampel DNA yang
telah diencerkan sebelumnya kemudian
dipindahkan dalam tabung yang lain dalam lajur tersebut. Posisi sampel dicatat dan tabung disimpan dalam mesin pendingin sampai sampel akan dimasukkan ke dalam cBot Cluster Generation.
Sekuensing Pustaka Genom Kedelai (HiSeq2000 protocol 2011)
Sekuensing pustaka genom menggunakan Illumina HiSeq2000 terdiri dari beberapa tahap yaitu persiapan reagen, pemilihan
parameter untuk sekuensing, pemasangan flow
cell, sekuensing, dan post-run sequencing (maintenance wash). Reagen yang digunakan selama sekuensing di antaranya SBS reagent (TruSeq SBS kit), multiplexing reagent dan paired end reagent. Reagen-reagen tersebut dimasukkan ke dalam rak dan disimpan dalam
ruang reagen compartment yang terdapat
dalam Illumina Hiseq2000. Pemilihan
experiment, user name, flow cell type, control lane, output folder,confirm first base, keep intensity files, existing recipe, save image dan align lanes. Tahapan selanjutnya yaitu pemasangan flow cell yang telah diklasterisasi melalui cBotcluster generation pada flow cell stage. Sekuensing dilakukan selama sekitar 11 hari dengan panjang pembacaan DNA 2 x 101 siklus. Tahapan terakhir setelah proses sekuensing selesai dilakukan yaitu dengan dipindahkannya flow cell dari flow cell stage dan dilakukannya post-run sequencing yaitu maintenance wash. Tahapan maintenance wash terdiri dari pembilasan dengan menggunakan air dan NaOH.
HASIL DAN PEMBAHASAN
Hasil Isolasi DNA Genom Kedelai DNA genom ketiga genotipe kedelai diisolasi menggunakan metode Michiels et
al. (2003). Metode tersebut merupakan
modifikasi dari metode Doyle&Doyle (1990). Metode Michiels et al. (2003) dan Metode Doyle & Doyle (1990) pada prinsipnya menggunakan cetil trimetil amonium bromida (CTAB) yang berfungsi untuk mendegradasi senyawa-senyawa metabolit sekunder yang
terdapat dalam tanaman. CTAB akan
membentuk kompleks tak larut dengan asam nukleat sehingga dapat memisahkan kontaminan polisakarida dan polifenol dari asam nukleat. Namun, perbedaan kedua metode tersebut di antaranya terletak pada penambahan polivinil pirolidon (PVP) saat proses ekstraksi yang berfungsi sebagai antioksidan untuk mencegah terbentuknya warna coklat polifenol pada DNA. PVP pada metode Michiels et al. (2003) ditambahkan langsung ke dalam bufer ekstraksi yang telah mengandung CTAB. Hal tersebut, berbeda dengan metode isolasi Doyle&Doyle (1990)
yang menambahkan PVP pada saat
penggerusan sampel DNA yang akan
diisolasi. Perbedaan lainnya yaitu terletak pada suhu yang digunakan saat presipitasi isopropanol selama satu malam. Metode
Michiels et al. (2003) menggunakan suhu
250C saat presipitasi isopropanol sedangkan
suhu yang digunakan pada metode
Doyle&Doyle (1990) adalah suhu 40C.
Melalui kedua modifikasi tersebut, Michiels et al. (2003) melaporkan bahwa hasil isolasi DNA genom yang diperoleh memiliki kualitas yang lebih baik dibandingkan metode Doyle&Doyle (1990).
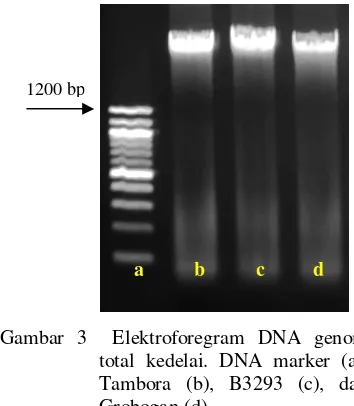
DNA genom tiga genotipe kedelai berhasil diisolasi dengan ditandai terbentuknya pita DNA pada elektroforegram hasil isolasi DNA
(Gambar 3). Elektroforegram tersebut
merupakan hasil uji kualitatif terhadap DNA yang telah berhasil diisolasi. Uji kualitatif bertujuan untuk mengetahui kualitas dan kemurnian DNA hasil isolasi. Uji kualitatif dilakukan dengan menggunakan elektroforesis gel agarosa 1%. Elektroforegram hasil isolasi DNA genom total dari tiga genotipe kedelai menunjukkan hasil yang baik, terlihat dari intensitas pita DNA yang tampak jelas (Gambar 3). Hasil elektroforesis juga menunjukkan bahwa ketiga pita DNA yang dihasilkan merupakan DNA genom. Hal tersebut terlihat dari ukuran pita DNA yang cukup besar (lebih dari 1200 bp) dan posisi pita DNA yang dekat dengan sumur gel.
Brown (2007) menyatakan bahwa
elektroforesis gel akan memisahkan molekul DNA sesuai dengan ukurannya dan semakin besar molekul DNA maka pita DNA yang dihasilkan akan semakin dekat dengan sumur gel.
Selain diuji secara kualitatif, DNA hasil isolasi juga diuji secara kuantitatif. Uji
kuantitatif bertujuan untuk mengetahui
konsentrasi DNA dan tingkat kemurnian DNA dari kontaminan polisakarida dan protein. Uji kuantitatif dilakukan dengan menggunakan
spektrofotometer nanodrop thermoscientific
pada panjang gelombang 230, 260 dan 280 nm. Panjang gelombang 230 nm merupakan
serapan maksimum untuk polisakarida.
Sedangkan, panjang gelombang 260 nm merupakan serapan maksimum untuk asam nukleat dan panjang gelombang 280 nm
1200 bp
Gambar 3 Elektroforegram DNA genom total kedelai. DNA marker (a), Tambora (b), B3293 (c), dan Grobogan (d).
12
merupakan serapan maksimum untuk protein.
Kemurnian DNA ditentukan melalui
perbandingan nilai absorbansi 260 nm dengan 280 nm dan perbandingan nilai absorbansi 260 nm dengan 230 nm. Rasio A260/A280
menunjukkan kemurnian terhadap proteindan
rasio A260/A230 menunjukan kemurnian
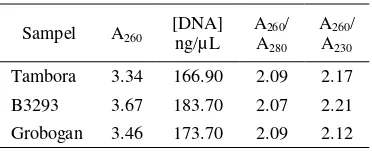
terhadap polisakarida. Hasil uji kuantitatif menunjukkan bahwa DNA genom total kedelai yang dihasilkan memiliki konsentrasi pada kisaran 902.4 – 1485.7 ng/µL dengan rasio A260/A280 dan A260/A230 berkisar 2.03 –
2.06 dan 1.90 - 2.03 (Tabel 1). Hasil tersebut menunjukkan bahwa DNA genom total yang diisolasi memiliki tingkat kemurnian yang baik. Walker & Wilson (2000) menyatakan bahwa nilai kemurnian yang lebih dari 2.0 menunjukan bahwa sampel mengandung
kontaminasi RNA. Sedangkan, nilai
kemurnian yang kurang dari 1.8 menunjukkan bahwa sampel mengandung kontaminasi protein dan polisakarida (Yuwono 2005). Berdasarkan uji kuantitatif dan kualitatif, DNA genom kedelai yang berhasil diisolasi telah memenuhi persyaratan untuk digunakan sebagai bahan konstruksi pustaka genom.
Michiels et al. (2003) menyatakan bahwa
DNA genom yang dibutuhkan untuk
konstruksi pustaka genom harus memiliki kualitas dan kuantitas yang baik. DNA genom
yang digunakan harus utuh, sedikit
mengandung fragmen DNA yang berukuran kecil serta memiliki konsentrasi yang tinggi. Tabel 1 Kuantitas dan kemurnian DNA
genom total kedelai
Hasil Fragmentasi DNA Genom Kedelai Tahapan yang harus dilakukan sebelum konstruksi pustaka genom adalah fragmentasi DNA genom. Pada tahapan ini, DNA genom tiga genotipe kedelai yang telah diisolasi pada tahapan sebelumnya difragmentasi menjadi fragmen-fragmen DNA yang lebih kecil
ukurannya. Fragmen DNA yang dibutuhkan
untuk konstruksi pustaka genom yaitu berukuran kurang dari 800 bp. Ukuran tersebut dipilih untuk memudahkan dalam mendapatkan ukuran pustaka genom yang optimum untuk sekuensing genom total
menggunakan Next Generation Sequencing
HiSeq2000.
Metode fragmentasi DNA yang digunakan pada penelitian ini adalah metode fragmentasi secara fisik dengan cara nebulisasi. Pemilihan metode ini karena nebulisasi merupakan metode yang efektif untuk fragmentasi DNA genom. Surzcky (1990) menyatakan bahwa fragmentasi DNA dengan cara nebulisasi menghasilkan fragmen DNA yang lebih acak dengan ukuran yang lebih seragam sehingga cocok digunakan sebagai cetakan dalam pembuatan pustaka genom. Di samping itu, metode nebulisasi merupakan metode yang mudah dalam pengontrolan ukuran
fragmen DNA yang diinginkan dan
membutuhkan waktu yang lebih singkat jika dibandingkan dengan metode fragmentasi yang lain.
Prinsip fragmentasi DNA dengan cara nebulisasi yaitu melalui pemutusan ikatan fosfodiester pada DNA. Hal tersebut terjadi karena pengaruh tekanan gas yang diberikan
selama proses nebulisasi berlangsung.
Tekanan gas yang ditambahkan ke dalam tabung nebulisasi menyebabkan larutan DNA masuk ke dalam saluran yang disebut sebagai saluran atomizer (Lampiran 5). Larutan DNA kemudian terpompa ke bagian atas saluran atomizer dengan pola aliran laminar dan keluar dengan melewati sejenis kerucut
penghambat yang dapat mengakibatkan
perubahan aliran larutan DNA. Perubahan aliran tersebut berdampak pada kecepatan aliran yang semakin cepat dan menghasilkan tegangan pada struktur DNA pada saat larutan DNA akan keluar dari saluran atomizer. Ketika tegangan pada DNA mencapai maksimal maka ikatan fosfodiester akan terputus dan menghasilkan fragmen-fragmen dengan ukuran yang lebih kecil (Sambrook & Russell 2001, Surzycki 2003, Illumina 2010c)
DNA yang akan dinebulisasi dilarutkan terlebih dahulu ke dalam bufer nebulisasi. Bufer nebulisasi mengandung gliserol dan bufer TE. Bufer TE mengandung Tris-HCl dan EDTA yang berfungsi untuk menjaga kestabilan pH optimum pada larutan DNA dan berperan sebagai pengkelat ion magnesium untuk mencegah denaturasi (Horison et al. 2003, Barnum 2005). Gliserol dalam bufer
tersebut berfungsi untuk meningkatkan
viskositas larutan DNA yang akan
dinebulisasi. Viskositas merupakan salah satu faktor yang mempengaruhi ukuran fragmen
DNA yang dihasilkan selama proses
nebulisasi (Sambrook&Russell 2001). Selain dipengaruhi oleh viskositas, ukuran fragmen DNA juga dipengaruhi oleh besarnya tekanan yang diberikan selama proses nebulisasi.
Sampel A260
Surzcky (2000) menyatakan bahwa semakin besar tekanan gas maka ukuran fragmen DNA yang dihasilkan selama proses nebulisasi akan semakin kecil.
Fragmentasi DNA yang telah dilakukan menunjukkan bahwa DNA genom tiga
genotipe kedelai berhasil difragmentasi
menjadi fragmen DNA yang lebih kecil ukurannya. Hasil tersebut diketahui melalui uji kualitatif terhadap fragmen DNA yang dihasilkan. Sebelum uji kualitatif dilakukan, DNA genom ketiga genotipe kedelai yang
telah difragmentasi dimurnikan terlebih
dahulu untuk menghilangkan gliserol yang masih menempel pada fragmen DNA. Hasil uji kualitatif menunjukkan visualiasi DNA
genom yang terlihat smear dengan ukuran
fragmen DNA yang dihasilkan antara 100 -800 bp (Gambar 4). Berdasarkan hasil
tersebut, ukuran fragmen DNA yang
dihasilkan telah sesuai dengan fragmen DNA yang dibutuhkan dalam konstruksi pustaka genom.
Uji kuantitatif juga dilakukan pada DNA genom yang telah berhasil difragmentasi. Hal
tersebut dilakukan untuk mengetahui
konsentrasi dan tingkat kemurnian fragmen DNA ketiga genotipe kedelai. Uji kuantitatif yang dilakukan setelah fragmentasi DNA sama seperti uji kuantitatif pada tahap isolasi DNA genom yaitu dengan menggunakan spektrofotometer nanodrop thermoscientific. Hasil uji kuantitatif menunjukkan bahwa DNA genom hasil fragmentasi memiliki rasio A260/A280 berkisar 2.07 – 2.09 (Tabel 2). Hasil
tersebut menunjukkan bahwa DNA genom hasil fragmentasi memiliki tingkat kemurnian yang baik karena rasio A260/A280 yang dimiliki
masih termasuk ke dalam kisaran 1.8 – 2.0 (Walker & Wilson 2000).
Hasil Konstruksi Pustaka Genom Kedelai Fragmen DNA genom ketiga genotipe kedelai telah berhasil dikonstruksi menjadi pustaka genom dengan ukuran 400 bp. Pustaka genom tersebut akan digunakan untuk sekuensing genom total dengan menggunakan Next Generation Sequencing (NGS) HiSeq2000. Pustaka genom diperoleh melalui beberapa tahapan konstruksi pustaka genom yaitu preparasi fragmen DNA cetakan, modifikasi ujung fragmen DNA, adenilasi ujung 3’OH, ligasi adapter, purifikasi DNA hasil ligasi dan amplifikasi fragmen DNA.
Prinsip konstruksi pustaka genom untuk sekuensing genom total menggunakan NGS Illumina HiSeq2000 adalah menempelkan adapter pada kedua ujung fragmen DNA. Adapter tersebut berfungsi sebagai tempat penempelan primer pada tahap amplifikasi pustaka genom dengan menggunakan PCR (Polymerase Chain Reaction). Pada tahap klasterisasi pustaka genom, adapter juga berfungsi membantu pustaka genom untuk berikatan dengan oligonukleotida pada flow
cell (Mardis 2008, Metzker 2011).
Keberhasilan konstruksi pustaka genom dipengaruhi oleh ketepatan dalam penggunaan mikropipet dalam pencampuran fragmen
DNA dengan pereaksi-pereaksi yang
digunakan dalam setiap tahapannya. Selain itu, faktor yang mempengaruhi keberhasilan konstruksi pustaka genom adalah kualitas DNA yang digunakan sebagai bahan DNA cetakan dan keefektifan pemisahan fragmen DNA dengan pereaksi pada tahap purifikasi. Fragmen DNA Cetakan untuk Konstruksi Pustaka genom
Preparasi fragmen DNA cetakan
meru