SELEKSI PRIMER
Simple Sequence Repeat
(SSR) UNTUK
IDENTIFIKASI 17 KLON KARET (
Hevea brasiliensis
)
FARIZA ITSNA QURROTA A’YUN
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Seleksi Primer Simple Sequence Repeat (SSR) untuk Identifikasi 17 Klon Karet (Hevea brasiliensis) adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2014 Fariza Itsna Qurrota A’yun
ABSTRAK
FARIZA ITSNA QURROTA A’YUN. Seleksi Primer Simple Sequence Repeat (SSR) untuk Identifikasi DNA 17 Klon Karet (Hevea brasiliensis). Dibimbing oleh EDY DJAUHARI PUWAKUSUMAH dan ASMINI BUDIANI.
Upaya pemuliaan karet mengalami kendala karena karet merupakan tanaman tahunan sehingga diperlukan waktu seleksi yang sangat lama dan areal seleksi yang luas. Di sisi lain, upaya peningkatan penanaman karet tidak diikuti dengan ketersediaan jumlah bibit unggul yang memadai sehingga memicu peredaran bibit palsu di masyarakat. Identifikasi secara cepat dan akurat dibutuhkan untuk mengatasi kendala ini. Salah satu strategi yang dapat digunakan untuk mengatasi kendala ini adalah dengan menggunakan teknik marka molekuler Simple Sequence Repeat (SSR). Penelitian ini bertujuan melakukan seleksi primer SSR yang secara spesifik dapat mengidentifikasi klon tanaman karet berdasarkan pola pita DNA yang dihasilkan dan tingkat polimorfismenya. Sebanyak sembilan primer SSR digunakan untuk mengamplifikasi 17 DNA klon karet. Hasil SSR dievaluasi menggunakan gel agarosa 3% dan gel poliakrilamid 6%. Sebanyak delapan primer yaitu primer SSRH403, SSRH548, gSSR165, gSSR212, gSSR268, EHB109, EHB169, dan HBE280 dapat mengidentifikasi beberapa klon karet. Primer paling informatif yaitu SSRH403 (PIC: 0.7069) dapat mengidentifikasi lima klon karet, sedangkan primer HBE280 (PIC: 0) hanya dapat mengidentifikasi satu klon karet.
Kata kunci: tanaman karet, Hevea brasiliensis, SSR, PIC
ABSTRACT
FARIZA ITSNA QURROTA A’YUN. Primer Simple Sequence Repeat (SSR) Selection for Identification of 17 Rubber (Hevea brasiliensis) Clones. Supervised by EDY DJAUHARI PURWAKUSUMAH and ASMINI BUDIANI.
Rubber tree (Hevea brasiliensis) breeding have problem because it takes long time and wide area to select new superior clones. On the other hand, the effort to increase rubber trees planting is not followed by availability of original clone seeds. Quick and accurate identification is needed to solve these problems. One of strategy that can be used to solve these problems is by using Simple Sequence Repeats (SSR) molecular marker. The aim of this research was to select SSR primer that identify the 17 rubber clones specifically by its DNA pattern and polymorphism degree. Nine primers used to amplified the 17 DNA of rubber clones. SSR results was evaluated by 3% agarose and 6% polyacrilamide. Eight of nine primers in this research can identify some rubber clones. Those primers are SSRH403, SSRH548, gSSR165, gSSR212, gSSR268, EHB109, EHB169, and HBE280. The most informative primer that is SSRH403 (PIC: 0.7069) can identify five clones, while the weakest primer that is HBE280 (PIC: 0) can identify just one clone.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biokimia
SELEKSI PRIMER
Simple Sequence Repeat
(SSR) UNTUK
IDENTIFIKASI 17 KLON KARET (
Hevea brasiliensis
)
FARIZA ITS
NA QURROTA A’YUN
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
Judul Skripsi: Seleksi Primer Simple Sequence Repeat (SSR) untuk Identifikasi 17 Klon Karet (Hevea brasiliensis)
Nama : Fariza Itsna Qurrota A’yun NIM : G84100009
Disetujui oleh
Drs Edy Djauhari P, MSi Pembimbing I
Dr Asmini Budiani, MSi Pembimbing II
Diketahui oleh
Dr Ir I Made Artika, MAppSc Ketua Departemen
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul penelitian yang dilaksanakan sejak bulan Januari hingga April 2014 ini adalah Seleksi Primer Simple Sequence Repeat (SSR) untuk Identifikasi 17 Klon Karet (Hevea brasiliensis). Penelitian ini didanai oleh proyek penelitian atas nama Dr Asmini Budiani, MSi di Balai Penelitian Bioteknologi Perkebunan Indonesia (BPBPI).
Terima kasih penulis ucapkan kepada Drs Edy Djauhari Purwakusumah, MSi dan Dr Asmini Budiani, MSi selaku pembimbing yang telah banyak memberikan pengarahan dan saran. Penulis juga menyampaikan terima kasih kepada orang tua, adik, dan keluarga atas segala doa, kasih sayang, dan motivasinya. Ucapan terima kasih juga penulis sampaikan kepada Teh Niyyah, Emi, Teh Rini, Teh Nida, Akhmad Aminullah, rekan-rekan Biokimia 47, keluarga Pinus merkusii atas bimbingan, doa dan dukungannya sehingga penelitian ini dapat diselesaikan dengan baik.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL viii
DAFTAR GAMBAR viii
DAFTAR LAMPIRAN viii
PENDAHULUAN 1
METODE 2
Alat dan Bahan 2
Metode Penelitian 3
HASIL 6
Kuantitas DNA Hasil Isolasi 6
Kualitas DNA Hasil Isolasi 7
Hasil Evaluasi Sembilan Primer SSR 7
Hasil Analisis Molekuler 11
PEMBAHASAN 13
Hasil Isolasi DNA Tanaman Karet 13
Evaluasi Sembilan Primer SSR 14
Profil Marka SSR 15
SIMPULAN DAN SARAN 17
Simpulan 17
Saran 17
DAFTAR PUSTAKA 17
LAMPIRAN 21
DAFTAR TABEL
1 Klon-klon karet yang digunakan dalam penelitian 2
2 Pasangan primer yang digunakan 4
3 Kemurnian dan konsentrasi DNA tanaman karet hasil isolasi yang
akan digunakan untuk SSR secara spektrofotometri 6
4 Deteksi hasil PCR dengan metode gel agarosa 7
5 Deteksi hasil PCR dengan metode gel poliakrilamid 8 6 Scoring hasil PCR dengan metode gel poliakrilamid 12 7 Hasil analisis primer menggunakan software PowerMarker V3.25 13
DAFTAR GAMBAR
1 Elektroforegram DNA karet hasil isolasi 7
2 Elektroforegram hasil amplifikasi DNA karet dengan primer SSRH403 (a) dengan metode elektroforesis gel agarosa (b) dengan metode
elektroforesis gel poliakrilamid 8
3 Elektroforegram hasil amplifikasi DNA karet dengan primer (a) SSRH358 (b) SSRH403 (c) SSRH548 (d) gSSR165 pada gel
poliakrilamid 9
4 Elektroforegram hasil amplifikasi DNA karet dengan primer (a)
gSSR212 (b) gSSR268 pada gel poliakrilamid 10
5 Elektroforegram hasil amplifikasi DNA karet dengan primer (a) EHB109 (b) EHB169 (c) HBE280 pada gel poliakrilamid 11
DAFTAR LAMPIRAN
1 Diagram alir penelitian 21
2 Elektroforegram hasil amplifikasi DNA karet menggunakan gel
PENDAHULUAN
Karet (Hevea brasiliensis) merupakan salah satu komoditas perkebunan yang penting bagi Indonesia baik dari segi ekonomi, dilihat dari devisa yang dihasilkan, maupun dari banyaknya petani, tenaga kerja dan pengusaha yang terlibat. Jumlah petani yang terlibat dalam usaha karet alam pada tahun 2006 mencapai 1.907 juta kepala keluarga (Ditjenbun 2006). Sumbangan devisa dari komoditas ini mencapai US$ 5.23 milyar pada tahun 2013 (Pusdatin 2013) dengan produksi karet sebanyak 3.18 juta ton dan luas areal perkebunan 3.49 juta hektar (BPS 2012).
Salah satu cara untuk meningkatkan produksi karet adalah dengan penggunaan klon unggul yang berproduksi tinggi. Para pemulia tanaman karet selama ini terus berupaya untuk mendapatkan klon-klon baru yang mempunyai potensi hasil yang tinggi. Upaya pemuliaan karet untuk memperoleh klon-klon unggul baru mengalami kendala karena karet merupakan tanaman tahunan sehingga untuk mendapatkan klon unggul baru diperlukan waktu seleksi yang sangat lama (20 hingga 25 tahun) dan areal seleksi yang luas (Novalina 2009).
Masalah lainnya yaitu usaha peningkatan penanaman karet tidak diikuti dengan ketersediaan bibit unggul bermutu dalam jumlah yang memadai. Hal ini mengakibatkan tersebarnya bibit palsu di masyarakat (Boerhendhy 2009). Dinas Perkebunan Kalimantan Timur pada tahun 2013 menemukan sebanyak 28300 bibit karet palsu beredar di wilayah Kalimantan Timur dan Kalimantan Utara (Diskominfo Prov. Kaltim 2014). Identifikasi bibit karet asli dan palsu membutuhkan waktu cukup lama. Bibit karet harus ditanam hingga dewasa untuk mengetahui jumlah lateks dan morfologinya sesuai dengan seharusnya atau tidak.
Salah satu strategi yang dapat digunakan untuk mengatasi kendala tersebut yaitu dengan menggunakan teknik marka molekuler (Novalina 2009). Data molekuler yang diperoleh dapat dijadikan sebagai pangkalan data (database) sidik jari DNA. Upaya pembuatan database sidik jari DNA untuk keperluan identifikasi dan diskriminasi varietas telah dilakukan pada beberapa tanaman (Septiningsih et al. 2004). Marka molekuler dapat memberikan hasil yang lebih cepat, efektif, dan akurat dibandingkan dengan identifikasi secara morfologi. Identifikasi menggunakan marka molekuler dapat dilakukan pada stadium awal (dapat dilakukan pada benih) dan tidak merusak karena hanya membutuhkan sedikit sampel serta tidak dipengaruhi oleh faktor lingkungan (Septiningsih et al. 2004).
2
sehingga menghasilkan pola pita DNA tertentu untuk setiap klon sehingga dapat digunakan sebagai marka molekuler untuk identifikasi klon tanaman karet.
METODE
Alat dan Bahan
Peralatan yang digunakan adalah gunting, timbangan, mortar, sudip, gelas piala, bulp, pipet volumetrik, gelas ukur, tabung sentrifus, vortex, penangas, sentrifus Beckman Coulter Allegra 64R, pipet mikro, kaca dengan ukuran 38x32.5x0.5 cm, lemari asam, lemari es, freezer, autoclave, Mutiskan Go Thermo Scientific, perangkat elektroforesis Toylab, power supply, laminar air flow cabinet, PCR Applied Biosystem Veriti, dan Sequi-Gen GT Nucleic Acid Electrophoresis Cell BIORAD.
Bahan-bahan yang digunakan dalam penelitian ini adalah daun tanaman karet yang berasal dari 17 klon tanaman karet (Tabel 1), nitrogen cair, akuades, poly(1-ethenylpyrrolidin-2-one) (PVPP), β merkaptoetanol, larutan CTAB 10%, bufer Tris-HCl 1M pH 8, larutan EDTA 0.5M pH 8, NaCl 5M, akuades steril, bufer ekstraksi, etanol 70%, isopropanol dingin, etanol absolut dingin, bufer TE (Tris-HCl:EDTA), bufer TBE 0.5x, loading dye, gel red, larutan kloroform:isoamilalkohol (24:1), Na-Asetat 3M pH 5.2, nuclease free water (NFW), RNAse, urea, akuades, buffer TBE 10x, akrilamid/bis 40% (19:1), APS 10%, TEMED, asam asetat 10%, AgNO3, formaldehida, Na2CO3, Natrium tiosulfat, agarosa, bufer TBE 5x, 10x DreamTaq buffer, DreamTaq DNA polimerase, dNTPs 10 mM, 9 primer forward-reverse yaitu SSRH403, SSRH358, SSRH548, gSSR165, gSSR212, gSSR268, EHB109, EHB169, dan HBE280 (susunan basanya dapat dilihat pada Tabel 2).
Tabel 1 Klon-klon karet yang digunakan dalam penelitian
No. Klon Tetua Asal Keterangan
1 GT 1 Klon primer Indonesia Penghasil lateks
2 IRR 220 PB 260 x IAN 873 Indonesia Penghasil lateks kayu
3 PR 261 TJIR 1 x PR 107 Indonesia Penghasil lateks
4 IRR 112 IAN 873 x RRIC 110 Indonesia Penghasil lateks kayu
5 PB 5/51 Klon primer Malaysia Penghasil lateks
6 IRR 118 LCB 1320 x FX 2784 Indonesia Penghasil lateks kayu
7 PR 107 Klon primer Indonesia Penghasil lateks kayu
8 IRR 104 BPM 101 x RRIC 110 Indonesia Penghasil lateks
9 PB 260 PB 5/51 x PB 49 Malaysia Penghasil lateks
10 IAN 873 PB 86 x F 1717 Brazil Penghasil lateks kayu
11 IRR 5 PBIG seedling Indonesia Penghasil lateks kayu
12 BPM 101 Klon primer Indonesia Penghasil lateks kayu
13 RRIC 110 LCB 1320 x RRIC 7 Sri Lanka Penghasil lateks kayu
14 PB 330 PB 5/51 x PB 32/36 Malaysia Penghasil lateks kayu
15 PB 217 PB 5/51 x B 5/9 Malaysia Penghasil lateks
16 PB 340 PB 235 x PR 107 Malaysia Penghasil lateks kayu
17 RRIM 712 RRIM 605 x RRIM 701 Malaysia Penghasil lateks
3 Metode Penelitian
Pengembangan metode seleksi klon karet dengan teknik SSR ini terbagi menjadi 6 tahapan penting, yaitu isolasi DNA dari daun karet, uji kualitatif dan kuantitatif DNA hasil isolasi, PCR Simple Sequence Repeat (SSR), elektroforesis gel agarosa 3%, elektroforesis gel poliakrilamid 6% dan analisis marka mikrosatelit atau SSR.
Isolasi DNA (Castillo et al. 1994)
Daun karet dicuci bersih dan dikeringkan dengan kertas tisu lalu dibuang tulang daunnya dan dipotong kecil-kecil. Daun karet digerus menggunakan mortar sambil ditambahkan nitrogen cair dan 0.1 gram poly(1-ethenylpyrrolidin-2-one) (PVPP). Setelah halus, sebanyak 1 gram sampel ditimbang dan dimasukkan ke dalam tabung sentrifus yang telah direndam pada nitrogen cair. Sebanyak 5 mL bufer ekstraksi yang telah dipanaskan dan β merkaptoetanol 1% ditambahkan kemudian dikocok dengan vortex dan kemudian diinkubasi selama 30 menit pada suhu 650C. Sampel dibiarkan dingin dalam lemari asam, kemudian ditambahkan 5 mL larutan kloroform:isoamilalkohol (24:1).
Sampel disentrifus dengan kecepatan 11.000 rpm selama 10 menit pada suhu 250C. Supernatan yang diperoleh dipindahkan, lalu ditambahkan 5 mL larutan kloroform:isoamilalkohol (24:1), dikocok dengan vortex dan disentrifus kembali dengan kecepatan 11.000 rpm selama 10 menit pada suhu 250C. Supernatan yang diperoleh dipindahkan lalu ditambahkan isopropanol dingin sebanyak 1x volume. Sampel dihomogenkan dengan cara tabung dibolak-balik lalu disimpan dalam lemari es (40C) selama 30 menit kemudian disentrifus kembali dengan kecepatan 11.000 rpm selama 10 menit pada suhu 250C. Supernatan yang diperoleh dibuang sedangkan pelet yang diperoleh dikeringkan. Setelah kering, pelet dilarutkan dengan bufer TE sebanyak 1 mL kemudian dikocok. Sebanyak 1/10 volume Na-Asetat 3M pH 5.2 dan 2.5 mL etanol absolut ditambahkan dan dikocok hingga terlihat sekumpulan serabut putih DNA. Sampel disimpan dalam freezer -200C selama 30 menit atau semalam.
Sampel disentrifus dengan kecepatan 12.000 rpm selama 10 menit pada suhu 40C. Supernatan yang diperoleh dibuang dan pelet dikeringkan. Pelet dicuci dengan etanol 70% sebanyak 100 µL. Campuran disentrifus kembali dengan kecepatan 8.000 rpm selama 5 menit pada suhu 250C. Supernatan dibuang dan pelet dikeringkan dalam laminar air flow cabinet. Pelet yang sudah kering dilarutkan dalam 100 µL nuclease free water (NFW) lalu ditambahkan RNAse sebanyak 3 µL. Sampel diinkubasi selama 30 menit pada suhu 370C.
Uji Kuantitas DNA dengan Multiskan Go (Thermo Scientific)
4
Uji Kualitas DNA dengan Elektroforesis Gel Agarosa
Gel agarosa 0.8 % dibuat dengan cara 0.32 gr serbuk agarosa dilarutkan dengan 40 mL bufer TBE 0.5x. Campuran dididihkan dalam microwave selama 2 menit lalu ditambahkan 2 µL gel red, dikocok perlahan dan dituang ke dalam cetakan gel yang telah disiapkan. Gel dibiarkan memadat.
Gel diletakkan ke dalam perangkat elektroforesis dan direndam dalam bufer TBE 0.5x hingga setinggi 1-2 mm di atas gel. Sebanyak 1 µL larutan stok DNA sampel ditanbahkan dengan 1 µL loading buffer lalu dimasukkan ke dalam sumur gel. Perangkat elektroforesis disambungkan pada power supply. Tegangan yang digunakan sebesar 60 volt. Elektroforesis dilakukan selama 60 menit. Hasil elektroforesis divisualisasikan dengan gel documentation.
Metode PCR Simple Sequence Repeat (SSR)
Larutan mix untuk satu kali reaksi dibuat dari campuran nuclease free water (NFW) 14.2 µL, 10x DreamTaq buffer (mengandung MgCl2) 2.5 µL, 10 mM dNTPs 0.5 µL, primer forward dan reverse masing-masing 1.25 µL, Taq polimerase 0.3 µL, dan DNA sampel 5 µL. Proses amplifikasi dilakukan menggunakan mesin PCR Applied Biosystem Veriti. Program running PCR yaitu pradenaturasi 940 C selama 3 menit, dilanjutkan 35 siklus meliputi denaturasi 940 C selama 45 detik, annealing dengan suhu annealing masing-masing primer (Tabel 2) selama 45 detik, extention 720 C selama 1 menit, dan terakhir post extention 720 C selama 5 menit. Informasi mengenai sembilan primer yang digunakan dalam penelitian ditampilkan secara jelas pada Tabel 2.
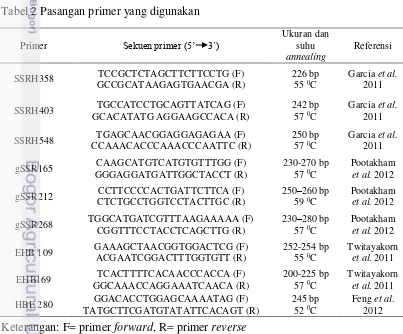
Tabel 2 Pasangan primer yang digunakan
5 Elektroforesis Gel Agarosa
Elektroforesis pada gel agarosa dilakukan dengan cara melakukan migrasi pada gel agarosa 3% dengan pewarna gel red. Sampel hasil PCR sebanyak 3 µL ditambahkan dengan 1 µL loading buffer kemudian dilakukan running pada tegangan 50 volt selama 4-5 jam. Hasil elektroforesis divisualisasikan dengan gel documentation.
Elektroforesis Gel Poliakrilamid (modifikasi Diputra 2013)
Gel poliakrilamid 6% dibuat sebanyak 60 mL. Sebanyak 25.2 gram urea, 29 mL aquabidest steril, 3 mL buffer TBE 10x dan 9 mL akrilamid/bis 40% (19:1) dihomogenkan sampai larutan berwarna bening. Sebanyak 300 µL APS 10% dan 40 µL TEMED ditambahkan ke dalam larutan yang sudah homogen kemudian dimasukkan ke dalam chamber dan ditunggu hingga mengeras. Gel yang sudah mengeras dipanaskan terlebih dahulu dengan cara pre-run pada daya 75 Watt selama 1 jam.
Produk PCR berkualitas baik ditambahkan 20 µL loading dye formamide dan selanjutnya didenaturasi pada suhu 940C selama 3 menit di dalam mesin PCR. Sebanyak 6 µL sampel kemudian dimasukkan ke dalam sumur gel, diatur sehingga suhu gel berkisar 500C. Gel dielektroforesis pada daya 65 Watt selama 1 jam 15 menit.
Visualisasi hasil elektroforesis gel poliakrilamid dilakukan dengan cara gel dikeluarkan dari chamber kemudian dimasukkan ke dalam tray pewarnaan yang telah ditambahkan 1 L larutan fiksasi (asam asetat 10%) dan dibiarkan selama 20 menit di atas shaker. Larutan fiksasi yang telah digunakan ditampung sejenak dan gel dicuci dengan 1 L aquadest sebanyak 2 kali masing-masing selama 2 menit. Sebanyak 1 L larutan pewarna atau stainning (0.8 gram serbuk AgNO3 dan 1.2 mL formaldehida dilarutkan dengan aquadest hingga volume 1 L) ditambahkan dan ditempatkan kembali dalam shaker selama 30 menit. Larutan pewarna dibuang, gel dicuci dengan 1 L aquadest selama 10 detik kemudian direndam dengan larutan developer (24 gram serbuk Na2CO3, 1.2 mL formaldehida, dan 160 µL larutan Natrium-tiosulfat 10 mg/mL dilarutkan dengan aquadest hingga volume 1 L) sambil di-shaker hingga pita-pita DNA muncul. Proses pewarnaan dihentikan setelah semua pita DNA muncul dengan menambahkan larutan fiksasi dari tahap sebelumnya. Gel kemudian dicuci kembali dengan 1 L aquadest dan dikeringkan. Selanjutnya pita DNA difoto dengan menggunakan kamera digital. Analisis Marka Mikrosatelit (SSR)
Penilaian Hasil Amplifikasi (Scoring). Setiap pita yang muncul pada gel merepresentasikan alel. Nilai (+) diberikan apabila terdapat pita dan nilai (-) bila tidak terdapat pita. Penilaian muncul atau tidaknya pita genetik dilakukan secara manual. Setiap pita DNA yang terbentuk pada marka SSR menunjukkan posisi alel pada lokus. Satu marka SSR merupakan satu lokus (Mulsanti 2011).
6
i= 1,2,3,...n, dimana pi2 dan pj2 adalah frekuensi alel ke-i dan ke-j. Rumus Botstein et al. (1980) pada penelitian ini diimplementasikan dengan menggunakan software PowerMarker V3.25.
HASIL
Kuantitas DNA Hasil Isolasi
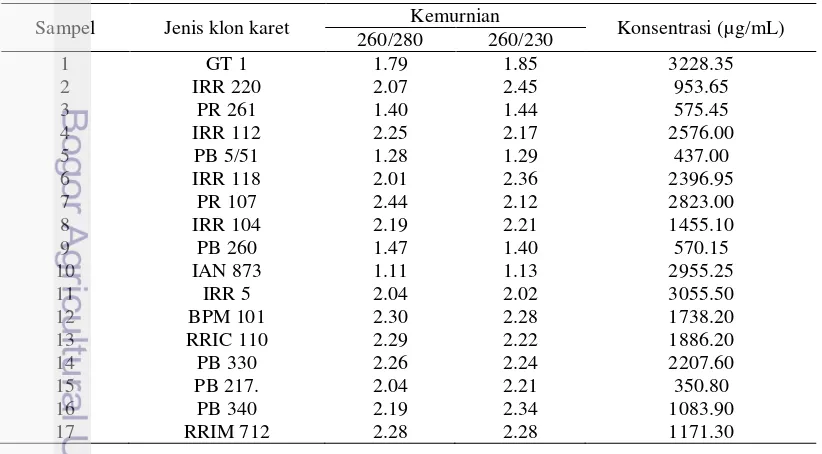
Sebanyak 17 sampel hasil isolasi DNA diukur konsentrasi dan kemurniannya. Kemurnian diperoleh dari perbandingan nilai absorbansi 260 dan 280 nm (perbandingan DNA dengan protein) dan nilai absorbansi 260 dan 230 nm (perbandingan DNA dengan polisakarida). Berdasarkan penelitian, konsentrasi DNA berkisar antara 350.80 – 3228.35 µg/mL, perbandingan A260/A280 antara 1.11 – 2.44 dan perbandingan A260/A230 antara 1.13 dan 2.45. Klon GT 1 merupakan klon dengan nilai konsentrasi tertinggi yaitu 3228.35 µg/mL dan klon PB 217 merupakan klon dengan konsentrasi terendah yaitu sebesar 350.80 µg/mL. Konsentrasi DNA yang diperoleh tidak seragam, oleh karena itu dilakukan pengenceran untuk menyeragamkan konsentrasi DNA hasil isolasi. Konsentrasi DNA diseragamkan menjadi 100 ng/µL. Sampel DNA murni akan menghasilkan nilai kemurnian yang berkisar antara 1.8-2.0 (Walker and Wilson 2000). Perbandingan nilai A260/A280 merupakan perbandingan nilai kemurnian DNA dengan protein, sedangkan perbandingan nilai A260/A230 merupakan perbandingan nilai kemurnian DNA dengan polisakarida. Kemurnian DNA hasil isolasi cukup baik karena banyak yang memenuhi nilai 1.8-2.0. Nilai kemurnian beberapa klon yang tidak sesuai dengan literatur disebabkan karena DNA tidak terlarut sempurna sehingga mempengaruhi pembacaan nilai absorbansi (Wulansari 2014).
Tabel 3 Kemurnian dan konsentrasi DNA tanaman karet hasil isolasi yang akan digunakan untuk SSR secara spektrofotometri
Sampel Jenis klon karet Kemurnian Konsentrasi (µg/mL)
7 Kualitas DNA Hasil Isolasi
Kualitas DNA hasil isolasi yang dielektroforesis menggunakan gel agarosa 0.8% ditunjukkan pada Gambar 1. Pita yang dihasilkan cukup jelas menunjukkan bahwa DNA berhasil diisolasi dan tidak rusak atau terfragmentasi sehingga dapat digunakan untuk analisis SSR. Smear seperti yang terlihat pada gambar dapat disebabkan karena terlalu banyak volume DNA yang dielektroforesis atau penggunaan tegangan yang terlalu besar (Ausubel 1990).
Gambar 1 Elektroforegram DNA karet hasil isolasi (Keterangan: M= Marker, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
Hasil Evaluasi Sembilan Primer SSR
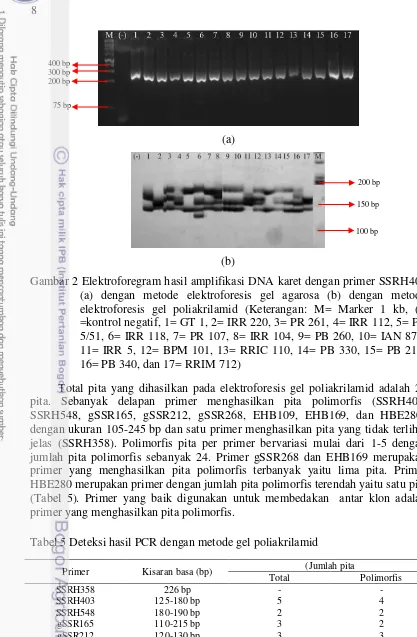
Elektroforesis menggunakan gel agarosa tidak dapat digunakan untuk mengidentifikasi klon-klon karet yang digunakan. Pita DNA pada elektroforesis gel agarosa tidak terpisahkan secara sempurna dibandingkan dengan hasil elektroforesis gel poliakrilamid (Gambar 2). Berdasarkan penelitian, hanya hasil elektroforesis gel poliakrilamid yang dapat digunakan untuk scoring dan analisis tingkat polimorfisme.
Total pita yang dihasilkan pada elektroforesis gel agarosa adalah 13 pita. Lima primer menghasilkan hanya satu pita (SSRH403, SSRH548, gSSR212, EHB 109 dan HBE280) dengan ukuran 150-350 bp dan empat primer menghasilkan dua pita (SSRH358, gSSR165, gSSR268, dan EHB169) dengan ukuran 180-280 bp (Tabel 4). Seluruh pita yang terbentuk merupakan pita monomorfis (Lampiran 2).
Tabel 4 Deteksi hasil PCR dengan metode gel agarosa
Primer Kisaran basa (bp) Jumlah pita
Total Polimorfis
SSRH358 220-270 2 0
SSRH403 230 1 0
SSRH548 250 1 0
gSSR165 230-280 2 0
gSSR212 180 1 0
gSSR268 150-190 2 0
EHB109 240 1 0
EHB169 200-230 2 0
HBE280 350 1 0
8
(a)
(b)
Gambar 2 Elektroforegram hasil amplifikasi DNA karet dengan primer SSRH403 (a) dengan metode elektroforesis gel agarosa (b) dengan metode elektroforesis gel poliakrilamid (Keterangan: M= Marker 1 kb, (-) =kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
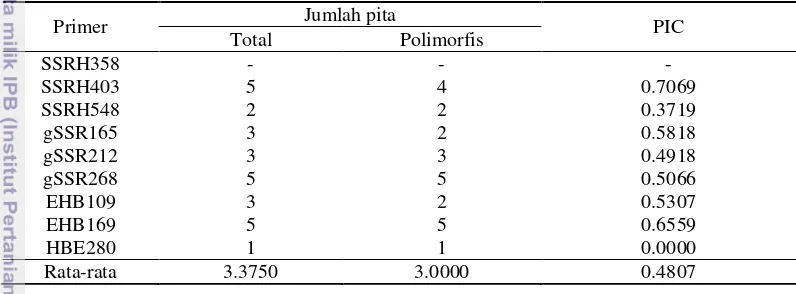
Total pita yang dihasilkan pada elektroforesis gel poliakrilamid adalah 27 pita. Sebanyak delapan primer menghasilkan pita polimorfis (SSRH403, SSRH548, gSSR165, gSSR212, gSSR268, EHB109, EHB169, dan HBE280) dengan ukuran 105-245 bp dan satu primer menghasilkan pita yang tidak terlihat jelas (SSRH358). Polimorfis pita per primer bervariasi mulai dari 1-5 dengan jumlah pita polimorfis sebanyak 24. Primer gSSR268 dan EHB169 merupakan primer yang menghasilkan pita polimorfis terbanyak yaitu lima pita. Primer HBE280 merupakan primer dengan jumlah pita polimorfis terendah yaitu satu pita (Tabel 5). Primer yang baik digunakan untuk membedakan antar klon adalah primer yang menghasilkan pita polimorfis.
Tabel 5 Deteksi hasil PCR dengan metode gel poliakrilamid
Primer Kisaran basa (bp) (Jumlah pita
Total Polimorfis
SSRH358 226 bp - -
SSRH403 125-180 bp 5 4
SSRH548 180-190 bp 2 2
gSSR165 110-215 bp 3 2
gSSR212 120-130 bp 3 3
gSSR268 105-140 bp 5 5
EHB109 140-160 bp 3 2
EHB169 145-160 bp 5 5
HBE280 245 bp 1 1
Jumlah 27 24
75 bp 200 bp 400 bp 300 bp
9 Elektroforegram hasil amplifikasi DNA dengan primer SSRH358 menunjukkan bahwa pita yang dihasilkan tidak jelas sehingga tidak dapat ditentukan ukuran pitanya (Gambar 3a), sedangkan hasil amplifikasi dengan primer SSRH403 menghasilkan lima pita dengan kisaran ukuran pita 125-180 bp (Gambar 3b). Elektroforegram hasil amplifikasi DNA dengan primer SSRH548 menghasilkan dua pita dengan ukuran berkisar 180-190 bp (Gambar 3c). Elektroforegram hasil amplifikasi DNA dengan primer gSSR165 menghasilkan tiga pita dengan ukuran yang lebih besar yaitu 110-215 bp (Gambar 3d).
(a)
(b)
(c)
(d)
Gambar 3 Elektroforegram hasil amplifikasi DNA karet dengan primer (a) SSRH358 (b) SSRH403 (c) SSRH548 (d) gSSR165 pada gel poliakrilamid (Keterangan: M= Marker 1 kb, (-)= kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
100 bp 150 bp 200 bp
100 bp 150 bp 200 bp
150 bp 200 bp
10
(a)
(b)
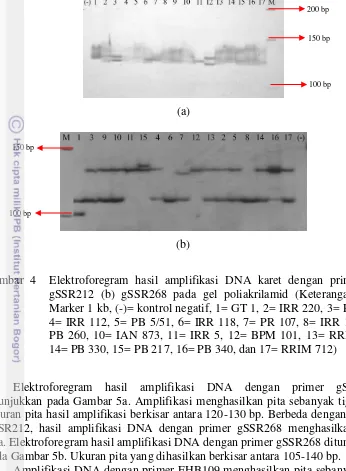
Gambar 4 Elektroforegram hasil amplifikasi DNA karet dengan primer (a) gSSR212 (b) gSSR268 pada gel poliakrilamid (Keterangan: M= Marker 1 kb, (-)= kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
Elektroforegram hasil amplifikasi DNA dengan primer gSSR212 ditunjukkan pada Gambar 5a. Amplifikasi menghasilkan pita sebanyak tiga pita. Ukuran pita hasil amplifikasi berkisar antara 120-130 bp. Berbeda dengan primer gSSR212, hasil amplifikasi DNA dengan primer gSSR268 menghasilkan lima pita. Elektroforegram hasil amplifikasi DNA dengan primer gSSR268 ditunjukkan pada Gambar 5b. Ukuran pita yang dihasilkan berkisar antara 105-140 bp.
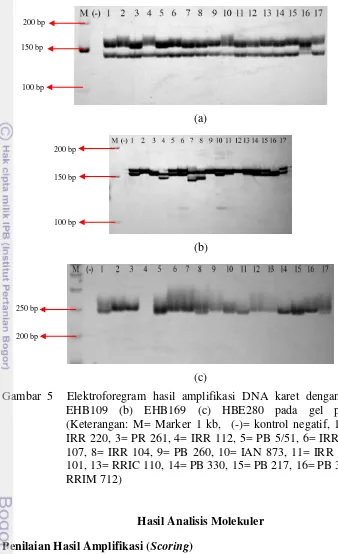
Amplifikasi DNA dengan primer EHB109 menghasilkan pita sebanyak tiga pita. Elektroforegram hasil amplifikasinya dapat dilihat pada Gambar 6a. Ukuran pita yang dihasilkan berkisar antara 140-160 bp. Primer selanjutnya yaitu EHB169 menghasilkan pita hasil amplifikasi sebanyak lima pita (Gambar 6b). Pita-pita tersebut memiliki ukuran 145-160 bp.
Elektroforegram hasil amplifikasi DNA dengan primer HBE280 ditunjukkan pada Gambar 6c. Jumlah pita yang dihasilkan pada amplifikasi ini hanya satu pita. Primer HBE280 menghasilkan pita dengan ukuran paling besar yaitu 245 bp. Pita yang dihasilkan antara satu klon dengan klon lainnya memiliki ukuran yang sama sehingga hasil amplifikasi dengan primer ini terlihat seperti monomorfis. Namun klon ke-4 yaitu IRR 112 pada elektroforegram tidak menghasilkan pita. Perbedaan pola pita ini menunjukkan bahwa pita hasil amplifikasi dengan primer HBE280 bukan bersifat monomorfis tetapi polimorfis. Pola pita dikatakan bersifat monomorfis jika semua klon menghasilkan pita dan berukuran sama.
100 bp 150 bp 200 bp
11
(a)
(b)
(c)
Gambar 5 Elektroforegram hasil amplifikasi DNA karet dengan primer (a) EHB109 (b) EHB169 (c) HBE280 pada gel poliakrilamid (Keterangan: M= Marker 1 kb, (-)= kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
Hasil Analisis Molekuler
Penilaian Hasil Amplifikasi (Scoring)
Sebanyak delapan dari sembilan primer yang digunakan menghasilkan pita yang polimorfis. Pita DNA yang dihasilkan memiliki ukuran yang berbeda untuk masing-masing primer. Ukuran pita yang dihasilkan pada penelitian ini (Tabel 6) berbeda dengan kisaran ukuran pita berdasarkan penelitian sebelumnya. Perbedaan ukuran pita yang diperoleh diduga karena penggunaan sampel yang berbeda.
Klon karet dapat diidentifikasi berdasarkan pola pita yang terbentuk (Tabel 6). Klon yang dapat diidentifikasi menunjukkan pola pita yang berbeda dengan
100 bp 150 bp 200 bp
100 bp 150 bp 200 bp
12
klon-klon karet lainnya dilihat dari hasil amplifikasi. Primer SSRH403 dapat mengidentifikasi klon GT 1, IRR 220, PR 261, IRR 118, dan IAN 873. Primer gSSR165 hanya dapat mengidentifikasi klon PR 107. Primer gSSR212 dapat mengidentifikasi klon GT 1 dan BPM 101. Primer gSSR268 dapat mengidentifikasi klon GT 1, PR 107, dan PB 217. Primer EHB109 hanya dapat mengidentifikasi klon PB 340. Primer EHB169 dapat mengidentifikasi klon PR 261, IRR 112, PR 107, IRR 104, BPM 101, dan PB 340. Primer HBE280 hanya dapat mengidentifikasi klon IRR 112. Primer SSRH548 hanya dapat membedakan 17 klon karet menjadi tiga kelompok yang memiliki pola pita berbeda. Klon-klon dalam satu kelompok tidak dapat dibedakan karena memiliki pola pita yang sama. Kelompok pertama terdiri dari klon GT 1, IRR 112, PB260, IAN 873 dan RRIC 110. Kelompok kedua terdiri dari klon IRR 220, PR 261, PB 5/51, IRR 118, PR 107, IRR 104, IRR 5, BPM 101, PB 340, dan RRIM 712. Kelompok ketiga terdiri dari klon PB 330 dan PB 217. Primer SSRH358 tidak dapat membedakan antar klon.
Tabel 6 Scoring hasil PCR dengan metode gel poliakrilamid
Primer No.
13
Polymorphic Information Content (PIC)
Tingkat polimorfisme (PIC) diperlukan untuk memilih marka yang dapat membedakan antar klon yang digunakan (Mulsanti 2011). De Vicente dan Fulton (2003) mengatakan bahwa nilai polimorfisme ditentukan oleh frekuensi kemunculan pitanya. Berdasarkan hasil analisis diperoleh rata-rata PIC sebesar 0.4807. Primer SSRH403 merupakan primer dengan nilai PIC paling tinggi yaitu 0.7069, sedangkan primer HBE280 merupakan primer dengan nilai PIC terendah yaitu 0. Primer SSRH358 tidak dapat dihitung nilai PIC-nya karena hasil amplifikasi yang tidak terlihat dengan jelas.
Tabel 7 Hasil analisis primer menggunakan software PowerMarker V3.25
Primer Jumlah pita PIC
Isolasi DNA tanaman karet dilakukan menggunakan metode Castillo et al. (1994) yang umum digunakan pada tanaman perkebunan (Ying dan Zaman 2006). Ain (2010) menyatakan bahwa metode ini termasuk praktis dan dapat menghasilkan DNA yang baik dari tanaman karet dibandingkan dengan metode yang lain. Bagian tanaman karet yang digunakan untuk isolasi adalah bagian daun yang masih muda. Daun yang masih muda banyak mengandung DNA karena bagian ini sedang aktif melakukan proses pembelahan dan pertumbuhan sel. Daun muda juga lebih memudahkan dalam penggerusan karena daun muda memiliki tekstur yang lebih lunak (Wulansari 2014).
14
Penyeragaman konsentrasi DNA dilakukan agar jumlah DNA yang digunakan diperkirakan sama untuk setiap tahapan PCR (Wulansari 2014). Konsentrasi DNA mempengaruhi kualitas fragmen hasil amplifikasi. Konsentrasi DNA yang rendah dapat menghasilkan fragmen sebagai pita yang tipis pada gel atau bahkan pita tidak terlihat secara visual, sedangkan konsentrasi DNA yang terlalu tinggi dapat menghasilkan fragmen sebagai pita yang terlalu tebal (Nurhaimi-Haris 2003).
Uji kualitatif DNA hasil isolasi dilakukan dengan menggunakan elektroforesis gel agarosa 0.8%. Kualitas DNA pada elektrofregram menunjukkan bahwa DNA hasil isolasi sudah cukup baik, namun terdapat smear pada beberapa klon yaitu klon PB 5/51, klon IRR 118, dan klon IAN 873. Smear terbentuk akibat degradasi DNA menjadi potongan-potongan pendek. Hal ini dapat terjadi karena perlakuan DNA selama isolasi. Smear juga dapat disebabkan oleh volume DNA yang digunakan terlalu besar saat elektroforesis, penggunaan tegangan yang terlalu besar (Ausubel 1990) atau sifat dari klon karet itu sendiri. Menurut Dualembang et al. (2011), metode SSR tidak mengharuskan menggunakan DNA yang murni sehingga DNA hasil isolasi sudah dapat digunakan untuk amplifikasi PCR-SSR. Kelebihan lain dari penggunaan metode SSR adalah konsentrasi DNA yang dibutuhkan relatif rendah, serta mampu menggunakan DNA dengan kualitas sedang (Dualembang et al. 2011).
Evaluasi Sembilan Primer SSR
Identifikasi genetik dengan pendekatan molekuler sangat dibutuhkan dalam kegiatan pemuliaan tanaman agar memperoleh hasil yang tepat. Marka molekuler merupakan solusi untuk pemecahan masalah dalam pemuliaan konvensional. Penemuan marka molekuler dapat membantu kelancaran pekerjaan seleksi. Marka molekuler DNA yang ideal harus memiliki tingkat polimorfisme yang sedang sampai tinggi, terdistribusi merata di seluruh genom, secara teknik sederhana, cepat dan murah, membutuhkan sedikit jaringan dan DNA sampel (Agarwal et al. 2008).
SSR merupakan marka DNA berbasis PCR yang dapat digunakan untuk menentukan genotipe individu dengan mendeteksi suatu segmen DNA yang mengandung pola perulangan sederhana dari basa nitrogen (Fadlilah 2014). Identifikasi 17 klon karet pada penelitian ini menggunakan sembilan primer yang mengacu pada penelitian sebelumnya. Primer-primer tersebut memiliki tingkat polimorfisme yang tinggi dan efektif membedakan klon Asia, klon dari Guetamala, dan klon Amerika (Garcia et al. 2011). Penggunaan mikrosatelit relatif mudah dengan menggunakan PCR karena dapat menjangkau seluruh kromosom (Prasetiyono dan Tasliah 2003).
15 yaitu 3%. Ukuran produk PCR-SSR tergolong kecil sehingga digunakan konsentrasi gel yang tinggi.
Elektroforegram hasil amplifikasi DNA pada penelitian ini digunakan untuk membedakan klon-klon karet. Chen (1998) mengemukakan bahwa kemampuan suatu primer dalam mengungkapkan keragaman genetik koleksi plasma nutfah ditunjukkan oleh jumlah pita DNA polimorfis yang dihasilkan. Primer polimorfis dibutuhkan untuk dapat menganalisis keragaman genetik satu populasi tanaman dengan memperlihatkan keragaman pola pita yang terbentuk (Chakravarthi dan Naravaneni 2006).
Hasil elektroforesis menggunakan gel poliakrilamid 6% mampu memisahkan DNA lebih sempurna dibandingkan dengan gel agarosa 3%. Hal ini disebabkan gel poliakrilamid memiliki pori-pori yang lebih kecil daripada gel agarosa (Diputra 2013). Gel poliakrilamid dapat memisahkan DNA meski perbedaan ukuran DNA-nya hanya 1 bp. Hal ini ditunjukkan dari jumlah dan pola pita yang terbentuk pada penelitian ini. Hasil elektroforesis dengan gel agarosa menghasilkan jumlah pita yang lebih sedikit (13 pita) dibandingkan dengan pita yang terbentuk pada elektroforesis gel poliakrilamid (27 pita).
Hasil elektroforesis gel agarosa menunjukkan bahwa pita DNA yang terbentuk bersifat monomorfis, sehingga tidak dapat digunakan untuk analisis selanjutnya. Pita-pita polimorfis pada penelitian ini ditunjukkan oleh hasil elektroforesis gel poliakrilamid. Sebanyak 24 dari 27 pita yang terbentuk bersifat polimorfis dengan variasi ukuran 105-245 bp. Pola pita polimorfis tersebut dihasilkan oleh delapan primer. Hanya primer SSRH358 yang menghasilkan pita yang tidak jelas (meragukan). Pita yang tidak jelas dapat terjadi karena kontaminasi urea pada permukaan sumur gel poliakrilamid (Millah et al. 2010), konsentrasi gel yang kurang tinggi atau tegangan yang digunakan terlalu besar sehingga pita DNA tidak terpisah secara sempurna. Optimasi konsentrasi gel dan besarnya tegangan diperlukan untuk menghasilkan pita yang jelas dan terpisah dengan sempurna.
Berdasarkan hasil penelitian, hampir seluruh klon karet dapat diamplifikasi oleh delapan primer kecuali klon IRR 112, klon PB 5/51, klon IRR 104, PB 260, IAN 873, RRIC 110, PB 330, PB 217, PB 340, dan RRIM 712 tidak dapat diamplifikasi oleh primer gSSR212. Hal ini dapat disebabkan primer yang tidak dapat menempel pada DNA target, DNA polimerase gagal memperpanjang daerah yang ditempeli primer untuk menyempurnakan daerah target (Lai et al. 2003), dan kemungkinan urutan basa nukleotida primer tersebut bukan merupakan komplemen dari basa nukleotida pada cetakan DNA target (Hartati et al. 2007). Keberhasilan amplifikasi mikrosatelit dipengaruhi oleh kualitas DNA genom, primer, konsentrasi larutan, dan kondisi suhu pada siklus PCR terutama pada suhu annealing (Brown 1991).
Profil Marka SSR
16
kemunculan alelnya (De Vicente and Fulton 2003). Marka yang menghasilkan pita lebih sedikit memiliki kemampuan yang lebih kecil untuk membedakan sampel yang diuji. Nilai PIC yang tinggi ditunjukkan pada marka yang menghasilkan banyak pita.
Nilai PIC merupakan standar yang baik untuk mengevaluasi marka genetik (Emrani et al. 2011). Rata-rata nilai PIC pada penelitian ini yaitu sebesar 0.4807. Nilai ini menunjukkan bahwa primer-primer yang digunakan mampu untuk membedakan klon-klon karet. Botstein et al. (1980) menggolongkan nilai PIC menjadi tiga kelas yaitu PIC>0.5 termasuk sangat informatif, 0.25>PIC>0.5 termasuk sedang dan PIC<0.25 termasuk memiliki nilai informatif yang rendah. Lima primer pada penelitian ini termasuk dalam kategori sangat informatif (SSRH403, gSSR165, gSSR268, EHB109, dan EHB169), dua primer termasuk dalam kategori sedang (SRH548 dan gSSR212), dan satu primer termasuk dalam kategori rendah (HBE280). Primer SSRH358 tidak dapat dihitung nilai PIC-nya karena hasil amplifikasi pada elektrofregram tidak menunjukkan pita DNA yang jelas. Primer SSRH403 yang memiliki nilai PIC tertinggi (PIC:0.7069) dapat mengidentifikasi lima klon karet, sedangkan primer HBE280 yang memiliki nilai PIC terendah (PIC:0) hanya dapat mengidentifkasi satu klon karet. Hal ini sesuai dengan penelitian yang dilakukan oleh Garcia et al. (2011) bahwa primer SSRH403 tergolong primer yang sangat informatif sehingga dapat mengidentifikasi klon karet dalam jumlah cukup banyak.
Tingkat polimorfisme yang tinggi mengindikasikan bahwa variasi diantara genotip yang dianalisis cukup besar (Dualembang et al. 2004). Struss dan Plieske (1998) mengemukakan bahwa tingkat polimorfisme yang tinggi juga dipengaruhi oleh tingkat polimorfisme marka SSR yang digunakan. Penggunaan primer yang lebih banyak dengan tingkat polimorfisme tinggi dapat memberikan pengelompokan yang lebih baik. Data molekuler sangat bergantung pada pemilihan primer yang digunakan. Szewe-McFadden et al. (1996) mengatakan bahwa penggunaan primer SSR sebanyak 10-20 memungkinkan untuk membedakan sejumlah genotip yang berkerabat dekat karena kemampuan menghasilkan polimorfisme tinggi.
17 gSSR268, dan EHB109 membuktikan bahwa klon IRR 112 merupakan keturunan klon RRIC 110 dan IAN 873 karena memiliki pola pita yang sama. Hasil amplifikasi dengan primer SSRH548 juga menunjukkan bahwa klon IRR 104 merupakan keturunan BPM 101. Pusat Penelitian Karet (2006) menyebutkan bahwa IRR 220 merupakan hasil persilangan antara PB 260 dan IAN 873. Hasil amplifikasi dengan primer EHB109 membuktikan bahwa klon IRR 220 memiliki pola pita yang sama dengan klon tetuanya yaitu klon PB 260 dan IAN 873. Selain itu, hasil amplifikasi dengan primer EHB169 juga menunjukkan bahwa klon IRR 220 dan PB 260 memiliki pola pita yang sama,
SIMPULAN DAN SARAN
Simpulan
Identifikasi beberapa klon karet dapat dilakukan dengan primer SSRH403, gSSR165, gSSR212, gSSR268, EHB109, EHB169, dan HBE280. Lima primer termasuk dalam kategori sangat informatif (SSRH403, gSSR165, gSSR268, EHB109, dan EHB169), dua primer termasuk dalam kategori sedang (SRH548 dan gSSR212), dan satu primer termasuk dalam kategori rendah (HBE280). Primer SSRH403 dengan nilai PIC tertinggi (PIC:0.7069) dapat mengidentifikasi lima klon karet, sedangkan primer HBE280 dengan nilai PIC terendah (PIC:0) hanya dapat mengidentifkasi satu klon karet.
Saran
Penelitian ini perlu dilanjutkan dengan menggunakan lebih banyak primer dan klon karet sehingga dapat diperoleh primer yang bukan hanya dapat membedakan antar klon tetapi juga sangat spesifik mengidentifikasi klon karet tententu dilihat dari pola pita yang dihasilkan. Sekuensing perlu dilakukan untuk mengkonfirmasi daerah yang teramplifikasi.
DAFTAR PUSTAKA
Agarwal M, Shrivastava N, Padh H. 2008. Advances in molecular marker techniques and their applications in Plan Sciences. Plant Cell Reporter 27: 617-631.
Ain YK. 2010. Isolasi DNA daun tanaman karet dengan menggunakan metode Khanuja, kit komersial, dan Castillo [laporan praktik lapang]. Bogor (ID): Institut Pertanian Bogor.
Ausubel FM. 1990. Current Protocols in Molecular Biology. Kanada (CA): John Willey & Sons.
18
Chakravarthi BK, Naravaneni R. 2006. SSR marker based DNA fingerprinting and diversity Study in Ice (Oryza sativa L.). African Journal of Biotechnology 5:684-688.
Chee KH, KM Zhong, TW Darmono. 1986. The occurance of eight races of Mycrocyclus ulei on hevea Rubber in Bahia, Brazil. Trans. Br. Mycol. Soc. 87:15-21.
Chen XM. 1998. Genome scanning for RGA in rice, barley, and wheat by high-resolution electrophoresis. TAG 97:345-355.
De Vicente MC, Fulton T. 2003. Using Molecular Marker Technique in Studies on Plant Genetic Diversity. [internet]. [diunduh 2 Juni 2014]. Tersedia pada: www.bioversityinternational.org/fileadmin/user_upload/online_library/publicat ions/pdfs/Molecular_Markers_Volume_1_en.pdf.
Diputra IM. 2013. Keragaman genetik Pinus merkusii Jungh. et Ed Vriese strain Tapanuli berdasarkan penanda mikrosatelit [skripsi]. Bogor (ID): Institut Pertanian Bogor.
[Disbun] Dinas Perkebunan Provinsi Kalimantan Timur. 2014. Tahun 2013, Terjadi Dua Kasus Benih Palsu [Internet]. [diacu 2014 2 Mei]. Tersedia pada: http://disbun.kaltimprov.go.id/berita-432-tahun-2013-terjadi-dua-kasus-benih-palsu.html.
[Ditjenbun] Direktorat Jenderal Perkebunan. 2006. Statistik Perkebunan Indonesia 2006-2011: Karet (Rubber). Jakarta (ID): Sekretariat Direktorat Jenderal Perkebunan Departemen Pertanian.
Dualembang E, Musa Y, Azrai M. 2004. Karakterisasi genetik koleksi plasma nutfah sorgum (Sorghum bicolor L. Moench) berbasis marka SSR (Simple Sequence Repeats). Journal of The Indonesia Nutrition Association: 1-15. Emrani H, Aminiria C, Arbabe MAR. 2011. Genetic variation and bottleneck in
japanese quail (Coturnix japonica) strain using twelve microsatelitte markers. African Jour. of Biotch. 10(20):4289-4295.
Fadlilah SH. 2014. Identifikasi gen Xa pada galur F1 Code x Angke dan Ciherang x IRBB21 serta uji patogenitas padi galur isogenik [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Feng S, Wu Y, Li W, Yu F, Wang J. 2012. Analysis of genetis diversity and SSR allelic variations in rubber tree (Hevea brasiliensis), The Molecular Basis of Plant Genetic Diversity. Prof. Mahmut Caliskan, editor. China (CN): InTech. Garcia IA, Gonzalez SM, Montoya D, Ariztizabal F. 2011. Identification in
19 Hartati D, Anto K, Taryono, Endang S, Widyatmoko A. 2007. Pendugaan keragaman genetik di dalam dan antar provenan Pulai (Alastonia scholars (L.) R. Br.) menggunakan penanda RAPD. Jurnal Pemuliaan Tanaman Hutan 1(2):1-8.
Hidayatun N, Chaerani, Utami DW. 2011. Sidik jari DNA plasma nutfah ubi jalar di Indonesia berdasarkan delapan penanda SSR. Jurnal Agro Biogen 7(2): 119-127.
Husniyati T. 2012. Analisis variasi genetik populasi tanaman karet (Hevea brasiliensis) sumber eksplan untuk perbanyakan in vitro berdasarkan RAPD [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Ilhami A. 2010. Analisis sidik jari DNA padi beras merah, padi aromatik, dan padi genjah [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Lai Y, Deepali S, Norman A, Fengzhu S. 2003. The mutation process of microsatellites during the polimerase Chain reaction. Jurnal of Computational Biology 10(2): 143-155.
Millah M, Habibah NA, Suwarni E. 2010. Analisis keanekaragaman genetik dan diferensiasi jati Jawa dan Madura berdasarkan marka mikrosatelit untuk mendukung fingerprinting jati. Biosaintifika 2(2):101-109.
Mulsanti IW, Surahman M, Wahyuni S, Utami DW. 2013. Identifikasi galur tetua padi hibrida dengan marka spesifik dan pemanfaatannya dalam uji kemurnian benih. Jurnal Penelitian Pertanian Tanaman Pangan 32(1): 1-8.
Mulsanti IW. 2011. Identifikasi dan evaluasi kemurnian genetik benih padi hibrida menggunakan marka mikrosatelit [tesis]. Bogor (ID): Institut Pertanian Bogor.
Novalina. 2009. Deteksi marka genetik yang terpaut dengan komponen produksi lateks pada tanaman karet [disertasi]. Bogor (ID): Institut Pertanian Bogor. Nurhaimi-Haris, Hajrial A, Nurita TM, Agus P. 2003. Kemiripan genetik klon
karet (Hevea brasiliensis Muell Arg.) berdasarkan metode amplified fragment lenght polymorphisms (AFLP). Menara Perkebunan 71(1): 1-15.
Penot E, CIRAD-CP, Rasidin A, IRRI Sembawa. 1994. Hevea Rubber Clones in Indonesia. Medan (ID): Badan Penelitian Sungei Putih.
Pootakham W, Juntima C, Jomchat N, Sangsrakru D, Yoocha T, Tragoonrung S, Tangphatsornruang S. 2012. Development of genomic-derived simple sequence repeat markers in Hevea brasiliensis from 454 genome shotgun sequences. Plant Breeding 131: 555-562. doi:10.1111/j.1439-0523.2012.01982.x.
Prasetiyono J, Tasliah. 2003. Strategi pendekatan bioteknologi untuk pemuliaan tanaman toleran keracunan aluminium. JIPI. 10(1):64-67.
Pusat Penelitian Karet. 2006. Pengenalan Klon Karet Penghasil Lateks dan Lateks Kayu. Medan (ID): Badan Penelitian Sungei Putih.
[Pusdatin] Pusat Data dan Sistem Informasi Pertanian. 2013. Ekspor impor komoditas pertanian menurut komoditas. Buletin Triwulanan Ekspor Impor Komoditas Pertanian 5(4): 4.
Septiningsih EM, Santoso TJ, Utami DW, Hidayatun N. 2004. Analisis sidik jari DNA varietas tanaman pangan. Kumpulan Makalah Seminar Hasil Penelitian BB-Biogen Tahun 2004: 140-151.
20
Szewe-McFadden AK, Kresovich S, Bliek SM, Mitchell SE, McFerson JR. 1996. Identification of polymorphic, conserved simple sequence repeats (SSRs) in cultivated Brassica spesies. Theor. Appl. Genet. 93:534-538.
Triwitayakorn K, Chatkulkawin P, Kanjanawattanawong S, Sraphet S, Yoocha T, Sangsrakru D, Chanprasert J, Ngamphiw C, Jomchai N, Therawattanasuk K et al. 2011. Transcriptome sequencing of Hevea brasiliensis for development of microsatellite markers and construction of a genetis linkage map. DNA Research 18:471-482.
Walker JM, Wilson K. 2000. Principles and Techniques of Practical Biochemistry. Cambridge (GB): Cambridge University Press.
Wulansari R. 2014. Studi kekerabatan dan morfologi padi lokal adan hasil mutasi sinar gama [skripsi]. Bogor (ID): Institut Pertanian Bogor.
Ying ST, Zaman FQ. 2006. DNA extraction from mature oil palm leaves. J. Of oil palm Research 18:219-224.
21 Lampiran 1 Diagram alir penelitian
Tanaman karet
Isolasi DNA (Castillo et al. 1994)
DNA
- Uji kualitas - Uji kuantitas
- SSR
- Elektroforesis
Pita DNA
22
Lampiran 2 Elektroforegram hasil amplifikasi DNA karet menggunakan gel agarosa 3%
(a)
(b)
(c)
(d)
Hasil amplifikasi DNA karet menggunakan primer (a) SSRH358 (b) SSRH403 (c) SSRH548 (d) gSSR165, (Keterangan: M= Marker 1 kb, (-)= kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
75 bp 200 bp 400 bp 300 bp
75 bp 200 bp 400 bp 300 bp
75 bp 200 bp 400 bp 300 bp
23
(a)
(b)
(c)
(d)
(e)
Hasil amplifikasi DNA karet menggunakan primer (a) gSSR212 (b) gSSR268 (c) EHB109 (d) EHB169 (e) HBE280 (Keterangan: M= Marker 1 kb, (-)= kontrol negatif, 1= GT 1, 2= IRR 220, 3= PR 261, 4= IRR 112, 5= PB 5/51, 6= IRR 118, 7= PR 107, 8= IRR 104, 9= PB 260, 10= IAN 873, 11= IRR 5, 12= BPM 101, 13= RRIC 110, 14= PB 330, 15= PB 217, 16= PB 340, dan 17= RRIM 712)
75 bp 200 bp 400 bp 300 bp
75 bp 200 bp 400 bp 300 bp
75 bp 200 bp 400 bp 300 bp
75 bp 200 bp 400 bp 300 bp
24
RIWAYAT HIDUP
Penulis dilahirkan di Tegal, Jawa Tengah pada tanggal 14 Februari 1993 dari ayah bernama Sopari dan ibu bernama Khuzaemah. Penulis merupakan anak pertama dari 2 bersaudara. Tahun 2010 penulis menyelesaikan pendidikan Sekolah Menengah Atas di SMA Negeri 1 Tegal dan pada tahun yang sama lolos seleksi masuk Institut Pertanian Bogor (IPB) melalui jalur Undangan Seleksi Masuk IPB (USMI) dan diterima di Departemen Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam.
Selama mengikuti perkuliahan penulis pernah menjadi asisten praktikum mata kuliah Metabolisme untuk mahasiswa Biokimia Tahun ajaran 2013/2014. Penulis juga aktif dalam kegiatan organisasi kampus, diantaranya Anggota Divisi CIC Community Research and Education of Biochemistry (CREB’s) periode 2011/2012, 2012/2013 dan anggota Bike to Campus Bogor 2011-2014. Penulis juga pernah aktif dalam beberapa kepanitiaan seperti panitia Masa Perkenalan Departemen Mahasiswa Biokimia tahun 2012, Biochemistry Fair 2012, Seminar dan Kajian Ilmiah Kehalalan 2012, Pesta Sains Nasional 2012, Bochemistry Expo 2013, dan Pesta Sains Nasional 2013.