STUDI MORFOLOGI DAN MOLEKULER
Boesenbergia
angustifolia
(Hallier f.) Schltr. DAN
Boesenbergia sp.
AINA MARDIYAH
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
AINA MARDIYAH. Studi Morfologi dan Molekuler
Boesenbergia angustifolia
(Hallier f.) Schltr. dan
Boesenbergia sp
. Dibimbing oleh DJAROT SASONGKO
HAMI SENO dan MARLINA ARDIYANI.
Zingiberaceae merupakan famili terbesar yang memiliki 53 genus dan lebih dari
1200 spesies yang tersebar di daerah tropis. Salah satu genus dari famili
Zingiberaceae adalah Boesenbergia. Identifikasi tumbuhan ini masih sangat sulit
dilakukan karena
Bosenbergia angustifolia
belum banyak dipelajari. Penelitian ini
melakukan
studi
filogenetik
terhadap
Boesenbergia
angustifolia
dan
Boesenbergia sp.
dari Sumatra dengan spesies Zingiberaceae lain khusunya
Boesenbergia berdasarkan karakter morfologi dan karakter molekuler yang
berdasarkan gen
Internal Transcribed Spacer
(ITS). Penelitian ini melakukan
isolasi DNA sampel dengan metode berbasis setiltrimetilamonium bromida
(CTAB) modifikasi Doyle & Doyle. Amplifikasi daerah ITS menghasilkan
amplikon ± 800 bp. Sedangkan panjang amplikon yang dihasilkan dari sekuensing
DNA adalah 788 basa untuk
Boesenbergia angustifolia
dan 789 basa untuk
sekuen
Boesenbergia sp.
Penyejajaran dari sampel-sampel yang diteliti
memperlihatkan tingkat evolusi yang tinggi. Rekonstruksi pohon filogenetik
dilakukan dengan menggunakan metode parsimoni. Berdasarkan pohon
filogenetik, genus Boesenbergia kemungkinan merupakan kelompok parafiletik.
Hasil pengamatan karakter
morfologi,
Boesenbergia angustifolia
dan
Boesenbergia sp
memiliki kemiripan karakter bunga dan perbungaan. Hal ini
didukung oleh data molekuler berdasarkan daerah ITS. Sekuen DNA dari kedua
jenis tumbuhan ini memiliki perbedaan 17 basa. Selain itu keduanya membentuk
percabangan yang dekat dengan nilai bootstrap 97%. Hal ini mendukung
ABSTRACT
AINA MARDIYAH. Study of Morphology dan Molecular of
Boesenbergia
angustifolia
(Hallier f.) Schltr. and
Boesenbergia sp
. Dibimbing oleh DJAROT
SASONGKO HAMI SENO and MARLINA ARDIYANI.
Zingiberaceae is the largest family of Zingiberales with 53 genera, over 1200
species and spread in the tropics. Boesenbergia is the one of Zingiberaceae
genera, that has not been explored, and therefore, in this research morphological
and phylogenetic studies were conducted using
Boesenbergia angustifolia
and
Boesenbergia sp.
of Sumatra. The method used in this study is the observation of
morphological characters and molecular characters in based of variation in
Internal Transcribed Spacer
(ITS) sequence are used for phylogenetic tree
construction. DNA sample was isolated using cetyltrimethylammonium bromide
based method of Doyle & Doyle modification. Amplification of ITS region
resulted in ± 800 bp amplicon. Sequencing DNA of ITS region resulted in ± 788
bp amplicon from
Boesenbergia angustifolia
and ± 789 bp amplicon from
AINA MARDIYAH
Skripsi
Sebagai salah satu syarat untuk memperoleh gelar
Sarjana Sains pada
Departemen Biokimia
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM
INSTITUT PERTANIAN BOGOR
Judul Skripsi : Studi Morfologi dan Molekuler
Boesenbergia angustifolia
(Hallier f.) Schltr. dan
Boesenbergia sp.
Nama
: Aina Mardiyah
NRP
: G84080063
Tanggal Lulus :
Disetujui
Komisi pembimbing
Dr. Djarot Sasongko Hami Seno, M.Sc
Ketua
Dr. Marlina Ardiyani, M.Sc.
Anggota
Diketahui
Untaian rasa syukur penulis panjatkan kepada Allah SWT atas segala
rahmat, berkah, nikmat dan karunia-
Nya penulisan skripsi yang berjudul “Studi
Morfologi dan Molekuler
Boesenbergia angustifolia
(Hallier f.) Schltr. dan
Boesenbergia sp.
” dapat diselesaikan.
Shalawat serta salam semoga selalu
tercurah kepada Nabi Muhammad SAW, keluarga, sahabat, dan para pengikutnya
sampai akhir zaman. Kegiatan penelitian ini dilaksanakan kurang lebih selama 4
bulan mulai bulan Januari sampai dengan Mei 2012, bertempat di Laboratorium
Sistematika Molekuler, Pusat Penelitian Biologi, Bidang Botani, Lembaga Ilmu
Pengetahuan Indonesia, Cibinong.
Penulis mengucapkan terima kasih kepada semua pihak yang telah
membantu dalam penyelesaian penelitian ini baik secara langsung maupun tidak
langsung. Ucapan terima kasih penulis sampaikan kepada Dr. Djarot Sasongko
Hami Seno, M.Sc sebagai ketua pembimbing dan Dr. Marlina Ardiyani, M.Sc
sebagai anggota pembimbing yang telah memberikan bimbingan, motivasi, saran,
dan kritiknya. Penulis juga mengucapkan terima kasih kepada kedua orang tua
tercinta, kakak, adik-adik yang tak henti-hentinya selalu memberikan dukungan,
perhatian, dan doa kepada penulis. Tidak lupa pula penulis ucapkan terima kasih
kepada bu Nita, bu Susi, dan segenap staf di Laboratorium Sistematika Molekuler,
Pusat Penelitian Biologi, Bidang Botani, Lembaga Ilmu Pengetahuan Indonesia
yang telah banyak membantu dalam menyelesaikan penelitian ini. Akhir kata,
penulis berharap tulisan ini dapat berguna bagi penulis sendiri maupun semua
pihak demi kemajuan ilmu pengetahuan.
Bogor, Juli 2012
RIWAYAT HIDUP
Penulis dilahirkan di Padalarang, Kabupaten Bandung Barat pada tanggal
24 Januari 1990 dari ayah bernama Iyon Suyana dan ibu bernama Nina Agustina.
Penulis merupakan anak kedua dari lima bersaudara.
Pendidikan penulis dimulai dari RA Al-Islamiyyah dan MI Al-Islamiyyah,
kemudian dilanjutkan ke jenjang Sekolah Menegah Pertama di SMP Negeri 1
Padalarang, kemudian pada tahun 2005 melanjutkan ke SMA Negeri 2 Cimahi,
Jawa Barat. Penulis lulus dari SMA Negeri 2 kota Cimahi pada tahun 2008. Pada
tahun yang sama, penulis lulus seleksi masuk IPB melalui jalur Undangan Seleksi
Masuk Institut Pertanian Bogor (USMI). Penulis memilih program studi mayor
Biokimia, Fakultas Matematika dan Ilmu Pengetahuan Alam. Penulis juga
mengambil program studi minor yaitu Pengolahan Pangan di bidang studi Ilmu
dan Teknologi Pangan, Fakultas Teknologi Pertanian.
Selama mengikuti perkuliahan, penulis pernah menjadi asisten praktikum
mata kuliah Kimia Dasar untuk mahasiswa Tingkat Persiapan Bersama tahun
ajaran 2010/2011. Penulis juga pernah menjadi staf pengajar mata Kuliah Dasar di
bimbingan belajar tahun ajaran 2010/2011 dan 2011/2012. Penulis juga aktif
dalam organisasi, penulis sempat menjadi staf divisi C-Core
Community Research
and Education of Biochemistry
(CREBs) pada tahun 2009/2010.
DAFTAR GAMBAR ... ix
DAFTAR LAMPIRAN ... x
PENDAHULUAN ... 1
TINJAUAN PUSTAKA... 1
Boesenbergia angustifolia
... 1
Filogenetik ... 2
Isolasi DNA ... 3
Polymerase Chain Reaction
(PCR) ... 3
Internal Transcribed Spacer
(ITS) ... 4
Elektroforesis DNA... 4
BAHAN DAN METODE ... 5
Alat dan Bahan ... 5
Metode Penelitian ... 5
HASIL DAN PEMBAHASAN ... 7
Morfologi
Boesenbergia angustifolia
dan
Boesenbergia sp.
... 7
Profil Pita Hasil Isolasi DNA ... 10
Profil Pita Hasil Amplifikasi DNA ... 11
Hasil Sekuensing DNA ... 11
Hasil Analisis Filogenetik ... 13
SIMPULAN DAN SARAN ... 16
DAFTAR PUSTAKA ... 16
DAFTAR GAMBAR
Halaman
1
Boesenbergia angustifolia
... 2
2
Siklus dalam proses PCR ... 4
3
Daerah
Internal Transcribed Spacer
... 4
4
Struktur agarosa ... 5
5 Morfologi bunga dalam
Boesenbergia sp
. dan
Boesenbergia angustifolia
.. 7
6
Bunga
Boesenbergia angustifolia
(A) dan
Boesenbergia sp.
(B) ... 8
7
Perbandingan tumbuhan dan perbungaan
Boesenbergia angustifolia
dan
Boesenbergia sp.
... 8
8
Hasil elektroforesis Isolasi DNA ... 10
9
Hasil Elektroforesis PCR ITS ... 11
10 Elektroferogram hasil sekuensing
DNA
Boesenbergia angustifolia
... 12
11 Penyatuan sekuen
forward
dan sekuen r
everse Boesenbergia angustifolia
menggunakan program ChromasPro ... 12
12 Hasil penyejajaran sekuen ITS sampel
Boesenbergia angustifolia
dan
sampel lain dari data GenBank. ... 13
1 Diagram alir penelitian ... 20
2 Proses isolasi DNA dengan metode Doyle dan Doyle (1987) ... 21
3 Urutan basa primer ITS yang digunakan... 22
4 Hasil Pengurutan Amplikon ITS sampel
Boesenbergia angustifolia.
... 23
5 Hasil Pengurutan Amplikon ITS sampel
Boesenbergia sp.
... 25
6 Sekuen DNA daerah ITS
Boesenbergia angustifolia
dan
Boesenbergia sp
27
7 Hasil penyejajaran (
alignment
) dari sekuen ITS ... 28
PENDAHULUAN
Zingiberaceae merupakan famili terbesar dari 8 famili yang berada dalam ordo Zingiberales. Zingiberaceae memiliki 53 genus dengan lebih dari 1200 spesies yang tersebar di daerah tropis. Zingiberaceae terdiri dari 4 subfamili yaitu Siphonochiloidae, Tamijioidae, Alpinioidae, dan Zingiberoideae (Kress et al. 2002). Salah satu genus dari famili Zingiberaceae adalah Boesenbergia.
Boesenbergia merupakan genus yang hidup di hutan tropis. Boesenbergia pandurata (Roxb.) Schltr atau yang lebih dikenal dengan temu kunci merupakan salah satu spesies dari genus Boesenbergia yang banyak dikenal. Boesenbergia pandurata
memiliki manfaat mulai dari obat peluruh dahak, batuk, penambah nafsu makan, sariawan hingga kanker (Kirana et al.2006, Sohn et al. 2005). Boesenbergia pandurata
juga telah terbukti memiliki aktivitas antiinflamasi, analgetik, antioksidan (Yun et al. 2006) dan antimutagenik (Trakoontivakorn
et al. 2001).
Spesies-spesies lain dalam genus Boesenbergia masih belum banyak dipelajari manfaatnya. Selain itu identitas beberapa spesies dalam genus ini juga belum jelas.
Boesenbergia angustifolia merupakan salah satu spesies dari genus Boesenbergia yang belum jelas identitasnya. Identifikasi tumbuhan ini masih sangat sulit dilakukan karena literatur mengenai tumbuhan ini masih sangat sedikit. Hal ini menyebabkan sulitnya dalam menentukan suatu tumbuhan apakah termasuk spesies Boesenbergia angustifolia
atau bukan.
Studi filogenetik genus Boesenbergia dapat dilakukan melalui studi morfologi dan molekuler. Studi filogenetik dengan data morfologi sangat mudah dipengaruhi oleh faktor lingkungan dan lebih subjektif. Sehingga karakter morfologi ini memiliki ketidakkonsistenen. Pada data molekuler menyediakan karakter dalam jumlah besar. Selain itu, data molekuler dapat terekspresi pada semua jaringan tumbuhan (Cintamulya 2011).
Penelitian ini melakukan studi filogenetik terhadap Boesenbergia angustifolia dan
Boesenbergia sp. yang berasal dari Sumatra. Penelitian ini merupakan penelitian pendahuluan untuk mengetahui identitas
Boesenbergia angustifolia. Hubungan kekerabatan tumbuhan ini dengan spesies Boesenbergia lainnya belum diketahui.
Boesenbergia sp. yang ditemukan di Sumatra
memiliki kemiripan karakter morfologi dengan Boesenbergia angustifolia. Dengan demikian perlu dilakukan studi kekerabatan antara kedua tumbuhan ini dengan spesies dalam genus Boesenbergia lainnya.
Metode yang digunakan dalam penelitian ini adalah dengan pengamatan karakter morfologi dan pendekatan molekuler guna mendapatkan hasil identifikasi dan status kekerabatan yang jelas. Studi molekuler dalam penelitian ini menggunakan marka molekuler Internal Transcribed Spacer (ITS). Variasi sekuen pada daerah ITS memungkinkan daerah ini untuk analisis filogenetik dari banyak organisme yang berbeda (Henry et al. 2000). Identitas suatu tumbuhan sangat penting karena untuk dapat mempelajari lebih lanjut, tumbuhan tersebut harus jelas identitasnya.
Penelitian ini bertujuan untuk mengetahui variasi urutan DNA daerah Internal Transcribed Spacer (ITS) menggunakan analisis bioinformatika dengan memanfaatkan data sekuen gen RNA ribosom daerah ITS. Penelitian ini ingin mengetahui hubungan kekerabatan Boesenbergia angustifolia
dengan spesies-spesies Boesenbergia serta spesies Zingiberaceae lainnya. Penelitian ini juga bertujuan untuk mendapatkan pohon filogenetik Boesenbergia khususnya untuk mengetahui kekerabatan Boesenbergia angustifolia dengan spesies Boesenbergia lainnya berdasarkan urutan DNA daerah ITS.
Penelitian ini diharapkan dapat mengidentifikasi Boesenbergia angustifolia
dan mengetahui hubungan kekerabatan antara
Boesenbergia angustifolia dengan spesies Boesenbergia lainnya berdasarkan marka molekuler ITS. Selain itu juga, penelitian ini diharapkan dapat menyumbangkan data sekuen DNA pada daerah ITS dari spesies-spesies Boesenbergia yang diteliti.
TINJAUAN PUSTAKA
Boesenbergia angustifolia
Zingiberaceae terdiri dari 53 genus dan lebih dari 1200 spesies. Zingiberaceae terdiri dari 4 subfamili dan 6 puak (tribe). Keenam puak ini diantaranya Siphonochiloideae (Siphonochileae), Tamijioideae (Tamijieae), Alpinioideae (Alpinieae, Riedelieae), dan Zingiberoideae (Zingibereae, Globbeae) (Kress et al. 2002).
Penelitian mengenai Zingiberaceae telah banyak dilakukan dan masih terus dilakukan hingga saat ini mengingat jumlah spesiesnya sangat banyak. Data molekuler saat ini telah menghasilkan informasi yang lebih akurat. Beberapa genus yang telah berhasil dipelajari pola hubungan evolusi dan kekerabatannya diantaranya yaitu Alpinia (Kress et al. 2005), Globba (Williams et al. 2004), Curcuma, Hedychium, dan Zingiber (Kress et al. 2002). Salah satu genus dari famili Zingiberaceae yang belum banyak dipelajari adalah Boesenbergia.
Boesenbergia memiliki rimpang bulat memanjang berukuran kecil. Boesenbergia sebagian besar hidup di daerah hutan tropis. Boesenbergia memiliki spesies yang banyak. Lebih dari 80 spesies yang termasuk dalam genus Boesenbergia telah teridentifikasi yang tersebar di Asia beriklim tropis (Saensouk & Larsen 2001). Namun banyak pula spesies dalam marga ini belum teridentifikasi.
Tumbuhan Boesenbergia angustifolia
(Gambar 1) merupakan salah satu spesies dari Boesenbergia. Boesenbergia angustifolia
pertama kali dipertelakan oleh Hans Hallier pada tahun 1898. Hans Hallier menamakan spesies ini sebagai Gastrochilus angustifolia
Hallier f (Boissier 1898). Kemudian pada tahun 1913 Rudolf Schlechter mengidentifikasi lebih lanjut tumbuhan ini. Schlechter mendapatkan bahwa tumbuhan ini merupakan genus Boesenbergia. Sejak saat itu nama resmi tumbuhan ini adalah
Boesenbergia angustifolia (Hallier f.) Schltr (Fedde 1913).
Gambar 1 Boesenbergia angustifolia
Filogenetik
Sistematika adalah ilmu tentang keanekaragaman hayati yang terdapat di bumi dan sejarah evolusi mereka. Tujuan utama ilmu sistematika yaitu menemukan dan mendeskripsikan perubahan yang terjadi selama evolusi dalam rangka menyusun sistem klasifikasi. Sistematika bertujuan untuk memahami dan mendeskripsikan keanekaragaman suatu organisme dan merekonstruksi hubungan kekerabatan dengan organisme lainnya (Hidayat & Pancoro 2008).
Filogenetik merupakan salah satu sistem klasifikasi yang didasarkan pada keeratan hubungan nenek moyang antara spesies atau kelompok spesies. Sistem klasifikasi ini sangat penting untuk digunakan dalam penelusuran kekerabatan diantara berbagai takson yang ada untuk memahami keanekaragaman makhluk hidup melalui rekonstruksi hubungan kekerabatan (Soltis et al. 1992). Dengan adanya kemajuan ilmu khususnya bidang biologi molekuler, data DNA saat ini telah digunakan dalam banyak penelitian filogenetik untuk menghasilkan informasi yang lebih akurat (Hidayat & Pancoro 2008).
Karakter morfologi yang telah lama digunakan dalam banyak penelitian filogenetik sangat mudah dipengaruhi oleh faktor lingkungan dan data lebih subjektif. Karakter DNA diketahui relatif lebih konsisten dibandingkan karakter morfologi. Sehingga penggunaan DNA menjadi pilihan dan telah diaplikasikan pada berbagai jenis tumbuhan.
Penggunaan sekuen DNA dalam penelitian filogenetik dengan menggunakan analisis bioinformatika telah meningkat secara pesat. Menurut Pevsner (2003) dan Yi-Ping (2005) bioinformatika merupakan bidang ilmu baru yang memanfaatkan perkembangan biologi molekuler dan ilmu komputer untuk mengolah dan menganalisis sejumlah besar data biologi.
Dasar penggunaan sekuen DNA dalam studi filogenetik adalah terjadi perubahan basa nukleotida menurut waktu sehingga akan dapat diperkirakan kecepatan evolusi. Berbagai teknik berdasarkan DNA telah banyak dikembangkan dan berpotensi menjadi penanda gen. Dengan pengetahuan ini, rekonstruksi pohon evolusi antara organisme dapat dilakukan (Karmana 2009).
3
lebih baik terhadap karakter-karakter yang ada (Baldwin et al. 1995). DNA mengandung informasi mengenai proses evolusi suatu kelompok organisme, sehingga mudah untuk dianalisis (Hillis et al. 1996). Sekuen DNA juga menyediakan banyak karakter karena perbedaan laju perubahan basa-basa nukleotida di dalam lokus yang berbeda adalah besar. Selain itu, sekuen DNA telah terbukti menghasilkan sebuah hubungan kekerabatan yang lebih alami (Chase et al.
1993).
Isolasi DNA
Deoxyribonucleic acid (DNA) adalah polimer linier yeng tersusun dari nukleotida. Setiap nukleotida terdiri dari gugus fosfat, deoksiribosa, basa purin atau basa pirimidin. Dua helai polimer DNA memutar satu sama lain untuk membentuk DNA heliks ganda. DNA merupakan informasi genetik yang mengendalikan seluruh aktivitas sel hidup (Lehninger et al. 2004).
Prinsip isolasi DNA ada tiga yaitu pemecahan sel, pemurnian, dan pemekatan DNA. DNA tumbuhan lebih sulit diisolasi daripada DNA hewan, karena memiliki struktur yang berbeda (Surzycki 2000). Sel tumbuhan memiliki dinding sel sehingga dalam tahap pemecahan sel biasanya digunakan cara mekanik dan kimia. Selain itu sel tumbuhan juga memiliki jumlah kandungan polisakarida yang tinggi, sehingga dalam tahap pemurnian diperlukan senyawa kimia yang mampu memisahkan polisakarida dengan DNA murni (Porebski et al. 1997, Surzycki 2000).
Dalam proses isolasi asam nukleat keberadaan polisakarida dan senyawa metabolit sekunder dalam sel tumbuhan sering menyulitkan. Struktur polisakarida yang mirip dengan asam nukleat akan menyebabkan polisakarida tersebut akan mengendap bersama dengan asam nukleat. Metabolit sekunder dan polisakarida juga dapat menghambat kerja enzim. Adanya polisakarida dalam tumbuhan ditandai dengan kekentalan pada hasil isolasi DNA yang menyebabkan kesulitan dalam reaksi PCR akibat penghambatan aktivitas enzim polimerase (Porebski et al. 1997). Oleh karena itu diperlukan suatu teknik isolasi DNA genom tumbuhan yang tepat sehingga diperoleh kualitas DNA yang baik bagi proses amplifikasi PCR. Cetyl Trimethyl Ammonium Bromide (CTAB) merupakan metode yang umum digunakan dalam ekstraksi DNA tumbuhan yang banyak mengandung
polisakarida dan senyawa polifenol (Jose & Usha, 2000). Penelitian ini akan dilakukan isolasi DNA kromosom atau DNA genom dengan metode CTAB yang dimodifikasi oleh Doyle dan Doyle (1987).
Polymerase Chain Reaction (PCR)
Polymerase Chain Reaction (PCR) merupakan teknik untuk memperbanyak DNA. Perbanyakan fragmen DNA dilakukan secara selektif oleh sepasang oligonukleotida yang dikenal sebagai primer. Primer merupakan sekuen DNA pendek dengan panjang 15-25 basa dan berutas tunggal. Jenis primer akan menentukan fragmen DNA mana yang akan terpotong dan teramplifikasi. Sehingga pemilihan primer dalam melakukan PCR sangat penting dilakukan untuk mendapatkan informasi yang diinginkan (Mikkelsen & Corton 2004).
Pada teknik PCR selain diperlukan primer juga diperlukan DNA cetakan dan enzim polimerase. DNA cetakan merupakan tempat sintesis utas DNA baru. Enzim polimerase diperlukan untuk mengkatalisis penempelan primer dengan fragmen yang ada pada rantai DNA. Taq polimerase berasal dari bakteri
Thermus aquaticus. Thermus aquaticus
pertama kali dikulturkan oleh Hudson Freeze.
Taq polimerase memiliki stabilitas termal tinggi (Reysenbach 2001).
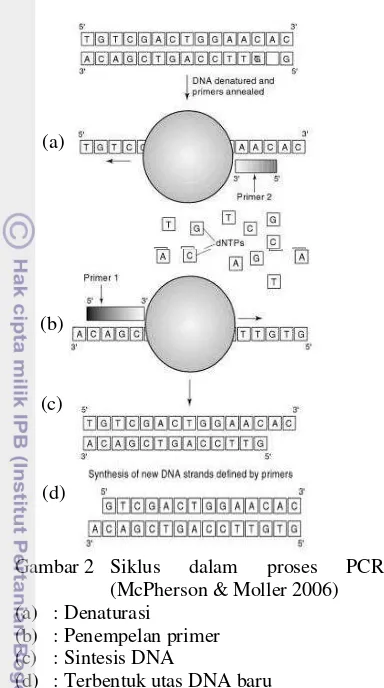
Tahap denaturasi merupakan tahap awal reaksi PCR yang berlangsung pada suhu tinggi yaitu antara 94-96°C sehingga ikatan hidrogen pada DNA terputus. Umumnya pada tahap awal ini dilakukan sampai 5 menit untuk memastikan semua utas DNA terpisah. DNA utas tunggal digunakan sebagai tempat menempelnya primer melalui proses
annealing pada suhu sekitar 42-65°C. Pada tahap annealing, primer menempel pada utas DNA yang urutan basanya komplementer dan bersifat spesifik. Semakin panjang oligonukleotidanya, maka semakin spesifik terhadap urutan DNA tertentu (McPherson & Moller 2006).
Gambar 2 Siklus dalam proses PCR (McPherson& Moller 2006) (a) : Denaturasi
(b) : Penempelan primer (c) : Sintesis DNA
(d) : Terbentuk utas DNA baru
Internal Transcribed Spacer (ITS)
Internal Transcribed Spacer (ITS) berasal dari DNA ribosom (rDNA). Sekuen DNA ribosom merupakan salah satu perangkat molekuler yang dapat dijadikan acuan dalam penelusuran filogenetik dari suatu organisme. Penggunaan ITS didasarkan pada rDNA yang secara alamiah terkonservasi. Semakin berbeda spesies secara filogenetik, semakin berbeda sekuen sebagian dari rDNA ini (McCullough et al. 1998). Gen 18S rDNA, termasuk dua daerah Internal Trancribed Spacer (ITS) dan gen 5.8S rDNA memiliki panjang total 2.600 bp, terpisah dari gen 26S rDNA (Gambar 3) yang memiliki panjang 3.300 bp (McCullough et al. 1998).
Variasi sekuen pada daerah ITS ini sering digunakan untuk analisis filogenetik. Daerah ITS lebih baik dibandingkan dengan molekul target lainnya karena sensitivitasnya yaitu berukuran kecil (kurang lebih 700 pasang basa) dan memiliki salinan yang banyak di dalam genom inti sekitar 100 kopi (Baldwin et al. 1995). Daerah ini mempunyai laju evolusi yang tinggi dan ada pada semua gen rRNA eukariot. Analisis perbandingan dari sekuen gen rDNA memberikan kemungkinan untuk analisis hubungan filogenetik dari berbagai level taksonomi yang berbeda (Henry et al.
2000).
Daerah ITS sering digunakan para ahli untuk analisis filogenetik molekuler pada tumbuhan dalam rangka memahami keanekaragaman dan menjawab beberapa masalah filogenetik. Pada penelitian ini digunakan primer ITS 5P dan ITS 8P yang merupakan modifikasi dari ITS4 dan ITS5. Primer ITS 5P dan ITS 8P memiliki panjang 22 basa dan 20 basa (lampiran 3). Melting time (Tm) primer ITS 5P dan ITS 8P masing-masing adalah 570C. Primer ini akan menghasilkan fragmen dengan ukuran kurang lebih 700-800 bp (Moller & Cronk 1997).
Elektroforesis DNA
Elektroforesis adalah suatu teknik pemisahan molekul selular berdasarkan ukuran dengan menggunakan medan listrik yang dialirkan pada suatu medium. Teknik ini dapat digunakan dengan memanfaatkan muatan listrik yang ada pada makromolekul. Laju perpindahan suatu molekul bergantung pada ukurannya, semakin kecil ukuran molekul maka molekul tersebut akan semakin cepat lajunya (Yuwono 2008).
Teknik elektroforesis dapat digunakan untuk analisis DNA, RNA maupun protein. Elektroforesis DNA dapat dilakukan untuk menganalisis fragmen-fragmen DNA hasil isolasi DNA ataupun hasil amplifikasi DNA. Arah pergerakan untuk DNA adalah menuju elektroda positif karena adanya muatan negatif pada rangka gula-fosfat yang dimiliki oleh DNA (Campbell et al. 2002).
Gambar 3 Daerah Internal Transcribed Spacer (ITS) (Tropp 2008). (a)
(b)
(c)
Gambar 4 Struktur agarosa (Horton 2011)
Gel yang digunakan untuk memisahkan DNA salah satunya adalah gel agarosa (Wilson & Walker 2000). Agarosa merupakan polimer linear dari D-galaktosa dan 3,6-anhidrogalaktosa (Gambar 4) yang diisolasi dari rumput laut. Gel agarosa mempunyai daya pemisahan lebih rendah dibandingkan dengan gel poliakrilamid, tetapi mempunyai rentang pemisahan lebih besar. DNA dari 200 basa sampei 50 kilobasa dapat dipisahkan dengan gel agarosa dengan berbagai konsentrasi agarosa (Sudjadi 2008).
Ukuran DNA dapat ditentukan dengan menyertakan marka atau penanda yang digunakan pada proses running. Setelah tahap
running selesai, dilakukan metode pewarnaan atau staining yang dilajutkan dengan penghilangan warna atau destaining. Pewarnaan DNA di dalam gel agarosa dilakukan dengan menggunakan larutan etidium bromida (EtBr) selama 15 menit agar molekul sampel berpendar dalam sinar UV (ultraviolet). Penghilangan warna atau
destaining dilakukan dengan perendaman dalam akuades selama 5-7 menit (Mikkelsen & Corton 2004).
BAHAN DAN METODE
Alat dan Bahan
Bahan-bahan yang digunakan dalam isolasi DNA adalah daun Boesenbergia angustifolia dan daun Boesenbergia sp. Daun yang digunakan adalah daun yang matang, pasir kuarsa, polivinilpirolidin (PVP), bufer
cethyl trimetyl ammonium bromide (CTAB), merkaptoetanol, kloroform-isoamilalkohol (CI) (24:1), isopropanol 100%, dan bufer TE.
Bahan-bahan yang digunakan dalam elektroforesis hasil isolasi yaitu agarosa, bufer tris-borat EDTA (TBE), loading dye, DNA
leader, etidium bromida, dan sampel hasil ekstraksi. Bahan-bahan yang digunakan dalam PCR yaitu dH2O, es, sampel hasil ekstraksi,
primer ITS dan MgCl2, buffer, dNTPs, dan
Taq polimerase. Elektroforesis hasil PCR diperlukan agarosa, bufer TBE, DNA leader, EtBr, dan sampel hasil PCR.
Alat-alat yang digunakan dalam ekstraksi DNA adalah mortar, tabung 1.5 mL, sudip,
waterbath dan shaker, sentrifus, pipet mikro, tips, lemari asam, dan tabung rak. Alat-alat yang digunakan dalam elektroforesis yaitu neraca analitik, labu Erlenmeyer, gelas ukur,
microwave, cetakan agar, sisir, bak elektroforesis, pipet mikro, tips, lemari asam, dan gel doc. Sedangkan alat-alat yang digunakan dalam PCR yaitu tabung 0.2 mL, bak es, pipet mikro, tips, sentrifuse mikro dan PCR.
Metode Penelitian Studi Morfologi
Determinasi tumbuhan merupakan metode yang digunakan dalam studi morfologi. Determinasi sampel dilakukan dengan mendeskripsikan morfologi setiap bagian tumbuhan segar secara langsung. Kemudian sampel tumbuhan segar dari Boesenbergia angustifolia dan Boesenbergia sp. dibuat spesimen kering untuk dapat dibandingkan secara langsung dengan spesimen kering koleksi Herbarium Kebun Raya Bogor dengan nama Gastrochilus angustifolia. Selain itu, dilakukan juga perbandingan sifat morfologi dengan protolog mengenai Boesenbergia angustifolia.
Pembuatan spesimen kering dilakukan pada sampel segar Boesenbergia angustifolia
dan Boesenbergia sp. yang masih utuh, kemudian dibersihkan dari kotoran. Setelah itu, kedua tumbuhan ini masing-masing ditumpuk diantara dua lembar kertas kemudian di lapisi dengan papan, diikat kuat. Sampel kemudian dikeringkan dalam oven 500C. Setelah sampel kering, sampel disimpan dalam freezer selama 5 hari.
Studi Molekuler
dan ditambahkan dengan CI sebanyak 1 kali volume sampel. Setelah itu, sampel dihomogenkan selama 15 menit. Lalu, disentrifus kembali dengan kecepatan 13000 rpm, selama 15 menit. Supernatan dipindahkan ke dalam tabung baru dan ditambahkan lagi dengan CI sebanyak 1 kali volume sampel. Lalu, disentrifus kembali dengan kecepatan 13000 rpm, selama 10 menit. Supernatan dipindahkan ke dalam tabung baru, ditambahkan isopropanol dingin sebanyak 2/3 kali volume, dan disimpan pada -200C selama semalam. Kemudian larutan disentrifugasi pada kecepatan 13000 rpm selama 10 menit. Supernatan yang terbentuk kemudian dibuang. Peletnya kemudian dikeringkan dalam oven pada 650C selama 1 jam. Pelet yang telah kering kemudian diencerkan dengan bufer TE pH 8.0.
Amplifikasi DNA. PCR ITS yaitu PCR dengan menggunakan primer ITS. Komposisi larutan PCR terdiri dari DNA sebanyak 0.5 µL, 2.5 µL green bufer, 0.5 µL MgCl, 0.75 µL primer ITS 5P sebagai forward (GGA AGG AGA AGT CGT AAC AAG G), 0.75 µL primer ITS 8P sebagai reverse (CAC GCT TCTC CCA GAC TAC A), 0.25 µL dNTP mix, dan 0.1 µL Taq polymerase, dan 7.15 µL dH2O. Kemudian larutan dihomogenisasi.
Setelah itu. diamplifikasi menggunakan PCR
thermal cycle dengan kondisi PCR yaitu 30 siklus dengan tahap pradenaturasi dan denaturasi pada suhu 94oC selama 3 menit dan 30 detik, tahap annealing pada suhu 55oC selama 1 menit, tahap sintesis pada suhu 72oC selama 90 detik, tahap pemanjangan pada suhu 72oC selama 5 menit, dan tahap penyimpanan pada suhu 4oC. Total waktu PCR ITS yaitu 2 jam 37 menit (Moller & Cronk 1997).
Elektroforesis Hasil Isolasi DNA dan Hasil PCR. Sebanyak 0.4 g agarose ditambahkan dengan 40 mL larutan 0.5 kali TBE. Kemudian dipanaskan dalam microwave
hingga agarose larut. Setelah larut dinginkan sebentar, kemudian gel agarosa dimasukkan ke dalam cetakan. Gel didiamkan hingga gel mengeras. Gel dimasukkan dalam bak elektroforesis dan ditambahkan dengan buffer TBE hingga sumur dalam gel terendam. Sebanyak 1 µL DNA leader dimasukkan ke dalam sumur pertama. Kemudian sebanyak 2 µL DNA, dimasukkan ke dalam sumur berikutnya. Setelah semua sampel dimasukkan ke dalam sumur, sampel dirunning selama 25 menit pada tegangan 100 volt. Setelah 25 menit, gel direndam dalam larutan EtBr selama 15 menit. Kemudian bilas gel dengan
air, gel pun dimasukkan ke dalam gel doc
yang di dalamnya terdapat sinar UV untuk dilihat hasilnya (Crandall & Barber 2007).
Purifikasi dan DNA Sequencing.
Penentuan urutan basa DNA terhadap produk amplifikasi dilakukan di 1st Base, Malaysia. Penentuan urutan basa dilakukan dua arah menggunakan pasangan primer ITS 5P dan ITS 8P. Proses DNA sequencing ini terdiri dari dua tahap yaitu siklus sekuensing (cycle sequencing) dengan menggunakan PCR dan
sequencing dengan menggunakan sequencer. Cycle sequencing ITS menggunakan 2 primer ITS yaitu ITS 5P dan ITS 8P. Komposisi reaksi PCR terdiri dari sampel DNA hasil PCR, bufer, MgCl, primer, dNTP, ddNTP,
Taq polimerase, dan dH2O. Kemudian larutan
dihomogenisasi hingga homogen. Setelah itu diamplifikasi menggunakan PCR thermal cycle. Sekuensing dilakukan dengan metode
automated sequencing ABI Prismy Big Dye Terminator Cycle Sequencing Ready Reaction Kit. Produk cycle sequencing dibersihkan dalam kolom Sephadex G-50 Centri-Sep spin. Kemudian dikeringkan, dan diurutkan basa-basanyadalam Automated Sequencer.
Penyejajaran Sekuen. Sekuen yang telah didapatkan kemudian dianalisis. Urutan basa DNA yang diperoleh dari daerah ITS diurutkan dengan menggunakan perangkat lunak Chromaspro. Setelah itu kemudian dilakukan penyejajaran dengan menggunakan perangkat lunak Molecular Evolutionary Genetics AnalysisVersion 4.0 (MEGA 4.0) (Tamura et al. 2007). Sekuen DNA
Boesenbergia angustifolia disejajarkan dengan sekuen DNA Boesenbergia sp.
Sekuen-sekuen DNA ini juga disejajarkan dengan sekuen DNA spesies Boesenbergia sp.
yang lain yang ada pada data bank DNA NCBI (http://www.ncbi.nlm.nih. gov). Tujuan penyejajaran sekuen ini adalah untuk menentukan homologi antara sekuen yang diteliti dengan sekuen yang lainnya yang ada pada genbank.
HASIL DAN PEMBAHASAN
Morfologi Boesenbergia angustifoliadan
Boesenbergia sp.
Determinasi tumbuhan dilakukan untuk mengungkapkan atau menetapkan identitas atau jati diri suatu tumbuhan. Langkah ini dilakukan untuk memastikan bahwa tumbuhan yang diteliti merupakan Boesenbergia angustifolia atau bukan. Bagian-bagian tumbuhan yang diamati meliputi daun, teruk berdaun, bunga, dan perbungaan.
Determinasi Boesenbergia angustifolia.
Hasil determinasi dapat diketahui bahwa
Boesenbergia angustifolia tumbuhan tidak berbatang (acaulis) dengan tinggi antara 40-50 cm. Daunnya berbentuk bulat memanjang (lanset), ramping, dan meruncing ke ujung daun. Ukuran daunnya sekitar 35 x 6 cm. Daun berbentuk bulat memanjang (lanset), ramping, dan meruncing ke ujung daun. Tangkai daun berbentuk kanal seperti perahu dan berwarna merah darah. (Gambar 7A).
Perbungaan tumbuhan ini tersembunyi di dalam daun. Tabung mahkotanya memiliki panjang 5.3 cm, tipis, dan berwarna putih. Braktea atau daun pelindung tumbuhan ini berukuran 2.3 cm dengan panjang brakteol 2 cm.
Bunga tumbuhan ini berwarna putih dan berukuran kecil. Labelum (bibir bunga) berwarna putih, berbentuk seperti telur, di dasar tandan berwarna ungu mozaik, tepinya bengkok, berwarna putih, kering, antar tepi dan tandan besar terdapat hiasan berwarna kekuningan. Labelum ini memiliki ukuran 2.5 x 1 cm. Staminodianya menghadap keluar, berbentuk lonjong seperti spatula, tumpul, menekuk terbuka, memilki 4 buah peruratan dan berukuran 1 x 0.3 cm (Gambar 5B). Ovarium berbentuk lonjong berukuran 0.4 cm dan calyx (kelopak bunga) berukuran 0.5 cm. Filamennya panjang berukuran 0.3 cm, berwana putih agak transparan. Stylus (tangkai putik) bersandar pada filamen dengan ukuran
6,8 cm. Anter (kepala sari) sari sejajar berukuran 0.6 cm. Serbuk sari berwarna kuning pucat (Gambar 6A).
Determinasi Boesenbergia sp. Tumbuhan merupakan tumbuhan acaulis dengan daun berbentuk bulat memanjang (laset), meruncing ke ujung daun, ujung daunnya membentuk ekor. Tulang daunnya menyirip. Permukaan daun bagian atas licin, mengkilap, beralur, dan berwarna hijau. Sedangkan permukaan daun bagian bawah berwarna hijau pucat dengan gradasi ungu, kasar, permukaannya licin, dan mengkilap. Tangkai daun membentuk kanal seperti perahu. Tangkai daunnya agak pendek, daunnya berpelepah, bagian bawah daun hingga tangkai daun berwrna ungu kecoklatan (Gambar 7B).
Perbungaan tumbuhan ini tersembunyi di dalam daun, bunganya jarang dan berwarna putih. Tabung mahkotanya agak panjang. Berwarna putih dan berukuran 4.1 cm. Braktea tumbuhan ini tumbuh selang-seling dikanan dan kiri perbungaan. Panjang braktea sekitar 2.4 cm dan brakteolnya 1.4 cm.
Bunga Boesenbergia sp. berwarna putih dan berukuran kecil. Labelum berbentuk lonjong dengan terdapat lekukan dan pada ujungnya sedikit mengkerut, ukuran labelum yaitu 2.2 x 1 cm. Warna labelum putih agak transparan pada bagian tengahnya berwana merah, jingga dengan bintik-bintik putih, dan pada bagian tengah bagian atas berwarna kuning pucat. Staminodianya yang menghadap keluar, berbentuk lonjong, tumpul, memiliki 4 buah peruratan, dan ukuran staminodia yaitu 1.2 x 0.7 cm (Gambar 5A). Ovarium berbentuk lonjong dengan ukuran 0.5 cm dan calyx berukuran 0.5 cm. Filamennya agak pendek yaitu berukuran 0.4 cm, berwana keputihan, agak transparan.
Stylus bersandar pada filamen dan dengan panjang 5.2 cm. Anter atau kantung serbuk sari sejajar berukuran 0.6 cm, dasarnya terbagi karena ada perbedaan longitudinal. Serbuk sari berwarna kuning pucat (Gambar 6B).
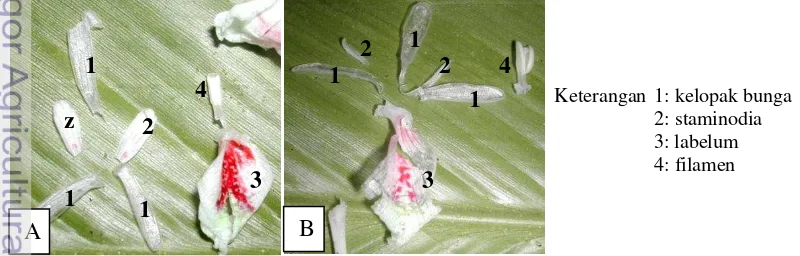
Gambar 5 Morfologi bunga dalam Boesenbergia sp. (A) dan Boesenbergia angustifolia (B)
1
1
1
1
1
1
2
2
2
z
3
3
4
4
A
B
Gambar 6 Bunga Boesenbergia angustifolia
(A) dan Boesenbergia sp.(B)
Hasil determinasi pada Boesenbergia angustifolia menunjukkan tingkat kesamaan morfologi yang tinggi dengan spesimen herbarium dan protolog. Kesamaan terlihat dari struktur tumbuhan dan perbungaan. Bentuk daun dan tata letak daun pada sampel
Boesenbergia angustifolia (Gambar 7A) dengan Boesenbergia angustifolia dari spesimen herbarium (Gambar 7C) terlihat
sangat mirip. Begitu juga dengan bentuk perbungaan sampel (Gambar 7D) dan perbungaan spesimen herbarium (Gambar 7F) yang mirip. Kemiripan terlihat dari bentuk tabung mahkota yang panjang. Sampel ini dapat dipastikan merupakan Boesenbergia angustifolia. Sedangkan antara Boesenbergia angustifolia dan Boesenbergia sp. memiliki beberapa perbedaan dan kemiripan.
Perbedaan ini terlihat pada bentuk daun, tata letak daun, serta warna tangkai bagian bawah (Gambar 7A dan 7B). Bentuk daun
Boesenbergia angustifolia lebih panjang dan ramping. Sedangkan pada Boesenbergia sp.
lebih lebar. Tata letak pada Boesenbergia angustifolia tebih rapi dan teratur. Pada
Boesenbergia angutifolia warna tangkai daun bagian bawah berwarna merah darah. Sedangkan pada Boesenbergia sp. berwarna merah kecoklatan dan warna ini tampak mulai dari permukaan bawah daun hingga bawah tangkai daun.
Gambar 7 Perbandingan tumbuhan dan perbungaan Boesenbergia angustifolia dan
Boesenbergia sp.
A : Tumbuhan Boesenbergia angutifolia
B : Tumbuhan Boesenbergia sp.
C : Tumbuhan spesimen tipe Boesenbergia angustifolia Herbarium Bogoriense D : Perbungaan Boesenbergia angutifolia
E : Perbungaan Boesenbergia sp.
F : Perbungaan spesimen tipe Boesenbergia angustifolia Herbarium Bogoriense.
A
D
B
E
C
F
9
Bunga dan perbungaan antara kedua tumbuhan ini memiliki tingkat kemiripan yang sangat tinggi (Gambar 6). Kemiripan terlihat dari bentuk labelum. Terdapat sedikit perbedaan, pada Boesenbergia angustifolia
labelum berbentuk bulat lonjong, terdapat banyak kerutan. Sedangkan labelum pada
Boesenbergia sp. terdapat lekukan pada bagian tengahnya. Ukuran bunga serta warna kedua tumbuhan ini hampir tidak dapat dibedakan. Bunga kedua tumbuhan ini sama-sama berukuran kecil dan berwarna putih. Corak warna pada labelumnya sangat mirip. Komponen-komponen bunga bagian dalam seperti filamen, anter, stylus, ovarium, braktea, brakteol memiliki ukuran yang
sangat mirip (Tabel 1). Namun ada beberapa perbedaan pada bunganya. Bentuk staminodia pada Boesenbergia angustifolia
lebih ramping (Gambar 5). Tabung mahkota (corolla tube) pada tumbuhan Boesenbergia angustifolia lebih panjang (Tabel 1). Selain itu, braktea dan brakteol pada Boesenbergia angustifolia jumlahnya lebih sedikit dari
Boesenbergia sp. Berdasarkan karakter morfologi bunga dan perbungaan, kemungkinan Boesenbergia angustifolia dan
Boesenbergia sp. untuk berkerabat dekat sangat besar. Hal ini perlu didukung oleh data molekuler.
Tabel 1 Karakter morfologi Boesenbergia angustifolia (Hallier f.) Schltr dan Boesenbergia sp.
No. Karakter B. angustifolia B. sp
1 Daun
Bentuk daun bulat memanjang, ramping, dan ujungnya runcing
bulat memanjang, agak lebar, dan ujungnya rucing berekor
Ukuran daun 35 x 6 cm 23 x 9 cm
Panjang tangkai daun 8-14 cm 5-11 cm
Warna pelepah daun hijau merah kecoklatan
Bentuk tangkai daun berbentuk kanal seperti perahu berbentuk kanal seperti perahu Warna tangkai daun merah darah pada bagian bawah
tangkai
merah kecoklatan
Tulang daun menyirip menyirip
2 Perbungaan
Ukuran bulir 1.2 x 0.3 cm 1.5 x 0.3cm
Panjang tabung mahkota
6.8 cm 5.2 cm
Braktea panjang 2.3 cm, dalam satu bunga jumlahnya sedikit
panjang 2.4 cm, dalam satu bunga jumlahnya banyak Brakteol panjang 2 cm, dalam satu bunga
jumlahnya sedikit
panjang 1.4 cm, dalam satu bunga jumlahnya banyak
Panjang ovarium 0.4 cm 0.5 cm
Panjang sepal (calyx) 0.5 cm 0.5 cm
3 Bunga
Panjang bunga 2.3 cm 2.4 cm
Warna bunga putih putih
Bentuk bibir bulat lonjong, terdapat banyak kerutan
bulat lonjong pada bagian tengahnya ada lekukan
Ukuran bibir 2.5 x 1 cm 2.2 x 1 cm
Bentuk staminodia lonjong seperti spatula lonjong dan lebih lebar
Ukuran staminodia 1 x 0.3 cm 1.2 x 0.7 cm
Panjang filamen 0.3 cm 0.4 cm
Panjang stylus 6.8 cm 5.2 cm
Profil Pita Hasil Isolasi DNA
Isolasi DNA merupakan tahap yang penting dalam analisis molekuler. DNA yang akan digunakan hendaknya berada dalam keadaan murni. Kualitas DNA yang baik dapat diperoleh dengan memperhatikan setiap tahapan pengerjaan isolasi DNA. Isolasi DNA yang digunakan adalah metode yang berbasis CTAB yang dimodifikasi oleh Doyle dan Doyle (1987). Tahapan isolasi DNA dalam metode ini sangat sederhana, mudah dilakukan, tidak membutuhkan waktu lama, dan hasil yang didapat memiliki kualitas yang cukup baik. Selain itu, metode ini dipilih karena telah digunakan dalam penelitian tumbuhan yang termasuk famili Zingiberaceae (Kress et al. 2002; Kress et al. 2005).
Sampel yang digunakan berasal dari daun yang masih muda dan tidak terlalu tua. Penggunaan daun sebagai sumber DNA karena daun merupakan bagian tumbuhan yang paling mudah diisolai dan DNA yang terdapat pada daun cukup banyak. Proses isolasi DNA diawali dengan pemecahan dinding sel. Pemecahan dinding sel dilakukan secara mekanik dengan menggunakan pasir kuarsa dan PVP. PVP berfungsi sebagai antioksidan dengan menghambat aktivitas polifenol oksidase. Polifenol aksidase merupakan enzim yang dapat menyebabkan DNA berwarna coklat (Bintang 2010). Pemecahan dinding sel juga dilakukan secara kimiawi dengan menggunakan CTAB.
CTAB mampu melisis sel serta mendenaturasi protein. Penambahan CTAB dengan kandungan garam tinggi dapat memisahkan polisakarida dari dinding sel (Santoso 2005). Penggunaan bufer CTAB untuk mengisolasi DNA dapat menghasilkan produk DNA yang berkualitas baik yang ditunjukkan oleh pita DNA genom pada beberapa tanaman. Penambahan CTAB menghasilkan cairan yang kental yang menunjukkan banyaknya polisakarida yang terdapat pada sampel daun. Setelah disentrifus, DNA berada pada fase supernatan. Penggunaan merkaptoetanol mencegah proses oksidasi senyawa fenolik sehingga menghambat aktivitas radikal bebas yang dihasilkan oleh oksidasi fenol terhadap asam nukleat (Wilkins & Smart 1996).
Polisakarida dan metabolit sekunder yang terdapat dalam sampel tumbuhan dapat dihilangkan dengan menggunakan kloroform-isoamilalkohol. Penambahan kloroform-isoamolalkohol dalam proses pemurnian ini membentuk tiga fase setelah disentrifus. Fase pertama (atas) adalah fase air tempat DNA
berada, fase kedua (tengah) merupakan protein, dan fase ketiga (bawah) adalah fase isoamilalkohol. Fungsi kloroform-isoamilalkohol yaitu mendenaturasi protein yang masih menempel pada kromosom. Larutan ini lebih efisien untuk mengisolasi asam nukleat (Ausubel et al. 1998). isopropanol dan penyimpanan selama selama semalam pada suhu -200C dilakukan untuk memekatkan DNA sehingga dapat diencerkan dengan bufer TE.
Hasil isolasi DNA dari sampel daun
Boesenbergia angustifolia dan daun
Boesenbergia sp. diuji kualitasnya dengan menggunakan elektroforesis agarosa 1%. Gel agarosa dengan konsentrasi 1% memiliki struktur serat yang baik, ukuran porinya besar, dan tahan terhadap gesekan. Kelebihan dari elektroforesis gel adalah pita DNA dapat dideteksi dengan kepekaan tinggi (Bintang 2010). Pengujian kualitas DNA Boesenbergia angustifolia dan DNA Boesenbergia sp.
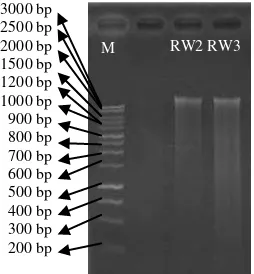
menunjukkan hasil yang baik. Hal ini ditunjukkan oleh DNA yang terlihat utuh, pita yang terbentuk memiliki intensitas yang jelas dan terang, dan pengotor atau smear yang terbentuk relatif sedikit. Gambar 8 menunjukkan DNA terbentuk pada pita diawal migrasi. Hal ini memperlihatkan bahwa DNA yang terisolasi merupakan DNA total dengan ukuran lebih dari 3000 bp.
Pada penelitian ini tidak dilakukan analisis kuantitatif DNA. Hal ini dikarenakan hasil analisis kualitatif DNA yang baik cukup untuk mengetahui bahwa DNA sampel dapat dilanjutkan untuk proses amplifikasi. Isolasi DNA dengan metode CTAB dapat menghasilkan 100-500 µg DNA per gram panjang jaringan tanaman yang segar dan muda (Ausubel et al 1998).
Gambar 8 Hasil elektroforesis Isolasi DNA Keterangan
M : Marker
RW2 : Boesenbergia angustifolia
RW3: Boesenbergia sp.
11
Profil Pita Hasil Amplifikasi DNA
Amplifikasi pada DNA Boesenbergia angustifolia dan DNA Boesenbergia sp. hasil isolasi dilakukan dengan menggunakan PCR. Primer yang digunakan adalah ITS 5P dan ITS 8P. Primer ini merupakan urutan basa penanda untuk daerah genom inti pengkode rDNA. Primer diperlukan untuk menyeleksi bagian sekuen DNA yang akan diamplifikasi. Daerah yang diamplifikasi adalah daerah Internal Transcribed Spacer (ITS). Karena itu urutan basa pada primer ini sangat menentukan keberhasilan dalam mendapatkan amplikon atau sekuen DNA target.
PCR merupakan salah satu tahapan yang penting. Untuk mendapatkan amplikon dengan kualitas yang baik, perlu diperhatikan komposisi setiap bahan PCR dan optimasi suhu dan waktu pada setiap tahapan dalam proses PCR. PCR diawali dengan tahap pradenaturasi, denturasi, penempelan primer, dan pemanjangan.
Pradenaturasi merupakan tahap awal dalam PCR. Pada penelitian ini, pradenaturasi dan denaturasi dilakukan pada suhu 94oC. Suhu tinggi menyebabkan putusnya ikatan hidogen sehingga DNA utas ganda menjadi DNA utas tunggal. Pada saat penempelan primer kondisi suhu sangat penting. Suhu penempelan tidak boleh melebihi titik leleh primer agar primer tidak rusak. Selain itu suhu penempelan juga tidak boleh terlalu rendah karena jika suhu rendah, maka akan menyebabkan terjadinya penempelan primer pada tempat yang tidak spesifik (mispriming) (Muladno 2002; Graham 1997). Primer ini akan membentuk ikatan hidrogen dengan DNA template pada daerah sekuens yang komplemen dengan sekuens primer. Suhu penempelan primer ITS 5P dan ITS 8P yaitu 550C.
Setelah penempelan primer dilanjutkan dengan pemanjangan pada suhu 72oC selama 1.5 menit. Suhu ini merupakan suhu optimum untuk kerja enzim DNA polimerase. Pada tahap ini Taq polimerase mengkatalisis mononukleotida pada primer yang sesuai dengan utas DNA komplemen yang berada di sebelahnya. Suhu pada setiap tahap diatur sedemikian rupa sehingga dihasilkan amplifikasi sekuen target DNA yang efisien. Proses polimerasi rantai DNA yang baru oleh
Taq polimerase berdasarkan sekuen yang ada pada DNA cetakan. Hal ini dilakukan dengan menambahkan dNTP yang komplemen
dengan DNA cetakan dari arah 5’P ke 3’OH.
Setelah siklus PCR berakhir, dilakukan penyempurnaan pemanjangan pada suhu 72oC
selama 5 menit untuk menjamin semua rantai DNA telah terbentuk. Dalam waktu kira-kira 5 menit, DNA target yang panjangnya bisa mencapai ratusan pasangan basa telah digandakan. Kemudian tahap pemanasan atau denaturasi dilakukan kembali, memulai siklus pemutusan utas ganda DNA, penempelan primer, dan sintesis DNA (Campbell et al.
2002). Siklus optimal PCR yaitu 25-45 siklus (Fairbanks & Andersen 1999). Siklus ini berjalan sebanyak 30 kali, hingga DNA target telah terduplikasi secara optimal.
DNA Boesenbergia angustifolia dan DNA
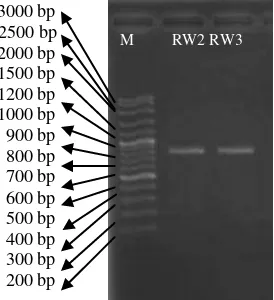
Boesenbergia sp. yang telah diamplifikasi pada daerah ITS diuji kualitasnya dengan metode elektroforesis gel agarosa 1%. Profil pita amplikon yang didapatkan memilki kualitas baik. Pita yang terbentuk jelas dan tebal untuk kedua sampel. Dari Gambar 9 diketahui bahwa amplikon hasil amplifikasi memiliki panjang sekitar 800 bp. Panjang amplikon ini sesuai harapan karena amplifikasi DNA dengan ITS akan memiliki panjang amplikon sekitar 700-800 bp. Amplikon DNA Boesenbergia angustifolia
dan DNA Boesenbergia sp bisa dilanjutkan proses sekuensing karena memiliki kualitas baik.
Gambar 9 Hasil Elektroforesis PCR ITS Keterangan
M: Marker
RW2 : Boesenbergia angustifolia
RW3: Boesenbergia sp.
Hasil Sekuensing DNA
Penentuan urutan basa dengan marka ITS dilakukan secara dua arah dengan menggunakan sepasang primer ITS 5P (forward) dan ITS 8P (reverse). Proses sekuensing DNA ini terdiri dari dua tahap yaitu siklus sekuensing (cycle sequencing) dengan menggunakan PCR dan sekuensing untuk mengetahui urutan basa DNA daerah ITS dengan menggunakan sequencer.
(a)
(b)
Gambar 10 Elektroferogram hasil sekuensing DNA Boesenbergia angustifolia; (a) forward; (b) reverse. (Merah) basa T, (hitam) basa G, (hijau) basa A, (biru) basa C.
Data hasil sekuensing DNA berupa elektroforegram yang berisi grafik hasil pembacaan basa nukleotida pada DNA.Setiap puncak yang terbentuk diterjemahkan menjadi basa andenin (A), guanine (G), sitosin (C), atau timim (T). Perbedaan basa ditunjukan dengan perbedaan warna setiap jenis basa. Secara keseluruhan elektroforegram hasil sekuensing DNA Boesenbergia angustifolia
(Gambar 10) dan Boesenbergia sp.
menunjukan kualitas puncak-puncak yang cukup baik dan dapat terbaca dengan jelas walaupun ada beberapa bagian puncak yang memiliki pemisahan yang kurang jelas dan bertumpuk. Pembentukkan puncak yang kurang jelas ini menimbulkan pembacaan basa nukleotida yang tidak akurat sehingga
dilakukan kembali pemeriksaan kembali untuk mengakuratkan hasil .
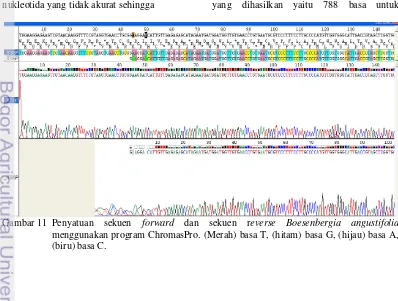
Sekuen DNA amplikon ITS 5P (forward) dan amplikon ITS 8P (reverse) kemudian diolah. Pengolahan dilakukan menggunakan program ChromasPro untuk mendapatkan sekuen lengkap dari daerah ITS. Langkah awal setiap sekuen dipotong pada kedua ujungnya. Pemotongan dilakukan pada sekuen DNA yang memiliki puncaknya tidak bagus (rusak). Karena puncakyang rusak tidak bisa dijadikan data karena tidak akurat. Setelah dipotong, sekuen DNA amplikon ITS 5P dan ITS 8P diurutkan atau digabungkan sehingga menghasilkan satu buah sekuen utuh dari masing-masing sampel. Panjang sekuen DNA yang dihasilkan yaitu 788 basa untuk
Gambar 11 Penyatuan sekuen forward dan sekuen reverse Boesenbergia angustifolia
13
Boesenbergia angustifolia dan 789 basa untuk sekuen Boesenbergia sp. Kedua sekuen ini memiliki perbedaan basa yang sedikit yaitu 17 basa. Lampiran 6 menunjukkan sekuen DNA
Boesenbergia angustifolia dan sekuen DNA
Boesenbergia sp. Huruf basa yang diberi tanda warna biru adalah basa-basa yang berbeda dari kedua sekuen DNA tersebut. Perbedaan ini terletak pada urutan basa ke-1, 3, 5, 7, 8, 175, 178, 202, 402, 427, 474, 510, 512, 523, 592, 596, dan 780.
Hasil Analisis Filogenetik
Analisis filogenetik diawali dengen melakukan penyejajaran sekuen DNA. Sekuen DNA hasil penggabungan dialignment atau disejajarkan dengan menggunakan program MEGA 5.0 (Molecular Evolutionary Genetics Analysis Version 5.0) Penyejajaran dilakukan dengan tujuan untuk menentukan tingkat homologi dari urutan basa DNA yang dianalisis. Untuk dapat menjelaskan hubungan kekerabatan tumbuhan Boesenbergia angustifolia, selain diperlukan sekuen DNA
Boesenbergia angustifolia dan Boesenbergia sp., diperlukan pula sekuen-sekuen dari spesies Boesenbergia, dan sekuen-sekuen dari spesies lain yang masih dalam famili Zingiberaceae yang berdasarkan marka ITS. Sekuen ini dapat diperoleh pada bank data DNA dari situs National Center for Biotechnology Information (NCBI) atau GenBank. Maka dilakukan pengunduhan data-data sekuen DNA yang diperlukan.
Pengambilan sekuen ini berdasarkan Kress et al. (2002) Penyejajaran DNA daerah ITS dilakukan pada 30 sekuen DNA. Sekuen-sekuen tersebut yaitu 2 Sekuen-sekuen Boesenbergia angustifolia dan Boesenbergia sp, 17 sekuen dari genus Boesenbergia, dan beberapa sekuen dari genus yang masih dalam keluarga Zingiberaceae sebanyak 11 sekuen.
Sekuen-sekuen tersebut disejajarkan dengan menggunakan align by muscle.
Setelah dilakukan penyejajaran, sekuen-sekuen DNA tersebut dipotong sama pada bagian awal dan akhirnya menjadi 740 bp. Pemotongan ini dilakukan berdasarkan daerah ITS yang terletak diantara daerah 18 S dan 26 S. Daerah ITS 1 terletak pada sekuen DNA urutan basa 1-228, urutan basa 229-449 merupakan daerah gen 5.8 S, dan posisi basa 450-734 adalah daerah ITS 2. Hasil penyejajaran menunjukkan tingkat homologi yang tinggi diantara sampel-sampel yang diteliti. Adanya homologi ditunjukkan dengan banyaknya daerah-daerah yang memiliki urutan yang sama (terkonservasi). Karena sifat ITS yang variatif, dalam penyejajaran ini muncul gap (ditandai oleh garis putus-putus). Adanya gap atau perbedaan sekuen ini menunjukkan terjadinya proses mutasi yang tinggi baik itu berupa substitusi insersi maupun delesi (Gambar 12). Terjadinya mutasi ini dapat disebabkan oleh peristiwa akibat kejadian alam, adaptasi, maupun aktivitas manusia.
Gambar 12 Hasil penyejajaran sekuen ITS sampel Boesenbergia angustifolia dan sampel lain dari data GenBank. (Merah) basa T, (ungu) basa G, (hijau) basa A, (biru) basa C.
Analisis filogenetik pada penelitian ini menggunakan program PAUP 4.b10 (Swofford 2002). Rekonstruksi pohon filogenetik menggunakan metode maksimum parsimoni (Felsensein 1985; Mort et al. 2000). Insersi dan delesi diperlakukan sebagai data yang hilang. Semua karakter diberi bobot yang sama. Evaluasi dari pohon filogenetik yang dihasilkan menggunakan analisis
bootstrap sebanyak 100 ulangan. Indeks konsistensi (CI) dan indeks retensi (RI) dihitung untuk pohon konsensus (Ubaidilah & Sutrisno 2009).
Hasil analisis filogenetik diperoleh 740 karakter, yang meliputi 429 karakter konstan, 129 karakter yang tidak informatif, dan 162 karakter informatif. Karakter konstan merupakan karakter yang seragam pada semua spesies yang diteliti. Karakter informatif adalah karakter yang tidak dihitung atau karakter yang dianggap hilang. Sedangkan karakter informatif merupakan karakter yang berperan dalam membentuk cabang dalam pohon filogenetik.
Analisis filogenetik dengan metode parsimoni diperoleh 36 pohon filogenetik, dengan nilai indeks konsistensi (CI) sebesar 0.637 dan indeks retensi (RI) sebesar 0.651. Nilai CI dan RI di atas menunjukkan konsistensi dan resolusi yang cukup tinggi dari pohon filogenetik yang dihasilkan (Swofford 1998). Indeks konsistensi menunjukkan tingkat kesamaan antarsekuen yang dianalisis. Sedangkan indeks retensi menunjukkan tingkat homoplasi atau kemiripan sekuen diantara spesies, tetapi berbeda secara keturunan (Estoup et al. 2002). Gambar 13 merupakan pohon filogenetik yang menggambarkan hubungan kedekatan atau kekerabatan beberapa spesies dalam genus Boesenbergia dan beberapa genus yang masih merupakan famili Zingiberaceae berdasarkan daerah ITS. Skala 10 menunjukkan jarak evolusi pada panjang cabang. Sedangkan angka pada cabang menunjukkan nilai bootstrap. Bootstrap
merupakan nilai yang menggambarkan tingkat kepercayaan dari sebuah titik cabang dalam sebuah topologi dengan menggunakan komputer. Jika nilai bootstrap antara 95-100% maka dapat disimpulkan bahwa percabangan tersebut mempunyai tingkat kepercayaan yang tinggi (Ubaidilah & Sutrisno 2009).
Untuk membangun sebuah pohon filogenetik, diperlukan penentuan akar untuk membedakan karakter primitif dari karakter yang berkembang lebih baru. Analisis filogenetik menggunakan suatu konsep yang
disebut pembandingan dengan luar kelompok (outgroup). Luar kelompok atau outgroup
adalah suatu spesies atau kelompok spesies yang relatif masih berkerabat dekat dengan kelompok spesies yang sedang dipelajari, tetapi tidak sedekat hubungannya dngan hubungan antar anggota spesies yang sedang dipelajari (Campbell et al. 2003).
Pada pohon filogenetik (Gambar 13),
Alpinia officinarum ditempatkan pada pohon sebagai outgroup. Menurut Kress et al. (2002) genus Alpinia merupakan kerabat terjauh dari genus Boesenbergia sehingga dapat diketahui kekerabatan sampai pada tingkat asal usul spesies tersebut. Penambahan outgroup ini sangat dibutuhkan untuk dapat menimbulkan polarisasi karakter apomorfik dan plesimorfik. Apomorfik adalah karakter yang berubah dan diturunkan dan terdapat pada ingroup, sedangkan plesimorfik merupakan karakter
primitive yang terdapat pada outgroup. (Hidayat & Pancoro 2008).
Pohon filogenetik (Gambar 13) membentuk 3 cabang utama. Cabang pertama merupakan outgroup yaitu spesies Alpinia officinarum. Spesies-spesies dalam genus Boesenbergia mengelompok pada cabang ketiga. Namun dalam cabang ketiga ini terdapat pula 5 spesies dari genus lain. Kelima genus lainnya yaitu genus Cornukaempferia, Kaemferia, Zingiber, Distichochlamys, Scaphoclamys, dan Curcumorpha.
Hal ini sama dengan pohon filogenetik berdasarkan Kress et al. (2002). Pada pohon filogenetik tersebut, spesies-spesies dari Boesenbergia membentuk satu cabang dengan kelima genus diatas (Lampiran 8). Karena dalam satu percabangan terdiri dari beberapa genus, sehingga genus Boesenbergia termasuk kelompok parafiletik. Parafiletik merupakan kelompok yang terdiri dari beberapa spesies yang berasal dari nenek moyang yang berbeda (Campbell et al. 2003).
15
Gambar 13 Pohon Filogenetik genus Boesenbergia berdasarkan daerah Internal Transcibed Spacer (ITS)
Alpinia offinarum AF478718
Stahlianthus involucratus AF478796
Curcuma petiolata AF202408
Hitchenia glauca AF478765
Hedychium flavescens AF478758
Boesenbergia cordata AJ388277
Boesenbergia armeniaca AY296723
Boesenbergia aurantiaca AY626271
Boesenbergia jangarunii AY626272
Boesenbergia parva AB097225
Boesenbergia belalongensis AJ388280
Boesenbergia orbiculata AJ388278
Boesenbergia rotunda AY626269
Boesenbergia flavorubra AY296726
Boesenbergia variegata AY626273
Cornukaemferia aurantiaca AF478736
Kaempferia rotunda AF478767
Zingiber ellipticum AF478799
Distichochlamys citrea AF478744
Scaphochlamys biloba AF478788
Boesenbergia plicata AJ388279
Boesenbergia albomaculata HM236118
Boesenbergia longiflora AY424745
Curcumorpha longiflora AF478742
Boesenbergia pulcherrima AF478725
Boesenbergia gelatinosa AY424744
Boesenbergia pulchella AY626270
Boesenbergia basispicata AY424743
Boesenbergia sp
Boesenbergia angustifolia
97.0 51.1
100.0 54.8
87.0 65.6
65.6
90.0 89.9
100.0
51.4 96.4 100.0
90.0
lebih dekat dengan Boesenbergia basispicata
dan Boesenbergia pulchella karena membentuk satu percabangan.
Fokus penelitian ini yaitu Boesenbergia angustifolia dan Boesenbergia sp. Kedua jenis ini berkerabat sangat dekat. Karena selain
Boesenbergia angustifolia dan Boesenbergia sp. terletak pada percabangan yang sama, percabangan jenis ini memiliki nilai bootstrap
yang cukup tinggi yaitu 97% maka tingkat kepercayaan kedua jenis ini untuk berkerabat dekat cukup tinggi. Kedekatan ini juga terlihat dari sedikitnya perbedaan basa antara
Boesenbergia angustifolia dan Boesenbergia sp yaitu hanya berbeda 17 basa (Lampiran 6).
Berdasarkan sifat morfologi Boesenbergia angustifolia dan Boesenbergia sp memiliki beberapa perbedaan. Namun, kedua tumbuhan ini memiliki tingkat kemiripan bunga dan perbungaan yang sangat tinggi. Bentuk dan corak warna labelum antara kedua jenis ini sangat mirip. Komponen-komponen bunga bagian dalam seperti filamen, anter, stylus,
ovarium, braktea, dan brakteol memiliki ukuran yang hampir sama. Berdasarkan karakter morfologi bunga dan perbungaan, kemungkinan kedua tumbuhan ini berkerabat dekat sangat tinggi. Kemungkinan ini didukung oleh hasil data molekuler yang didapat memperkuat kesimpulan data morfologi yang menyatakan bahwa
Boesenbergia sp. merupakan varietas dari
Boesenbergia angustifolia.
SIMPULAN DAN SARAN
Simpulan
Analisis filogenetik menggunakan gen ITS dapat memberikan informasi mengenai hubungan kekerabatan Boesenbergia angustifolia. Boesenbergia termasuk genus dari Zingiberaceae yang tergolong dalam kelompok parafiletik. Selain itu juga didapatkan Boesenbergia sp termasuk dalam varietas dari Boesenbergia angustifolia. Hal ini terlihat dari kemiripan karakter morfologi bunga dan perbungaan, sedikitnya (17 basa) perbedaan sekuen amplikon ITS dan posisi pohon filogenetik dengan nilai bootstrap 97%.
Saran
Penggunaan sekuen sampel yang lebih banyak disarankan dilakukan untuk mendapatkan hasil gambaran kekerabatan yang lebih jelas. Kemudian penggunaan marka molekuler perlu ditambah agar analisis filogenetik mengenai hubungan kekerabatan
Boesenbergia angustifolia dapat dipelajari
dengan lebih jelas. Selain itu diperlukan sampel-sampel yang memiliki sekuen DNA dengan panjang yang seragam sehingga dapat menunjang dalam analisis filogenetik.
DAFTAR PUSTAKA
Ausubel et al. 1998. Current protocols in molecular biology. New York. John Wiley and Sons. Inc.
Baldwin et al. 1995. The ITS Region of nuclear ribosomal DNA: a valuable sources of evidence on angiospermae phylogeny. Ann Missouri Bot Gard 82: 247-277.
Bintang M. 2010. Teknik penelitian Biokimia. Jakarta: Erlangga.
Boissier H. 1898. Bulletin de l'Herbier Boissier Volume 6. Genève Impr. Romet.
Campbell NA, Reece JB, Mitchell LG. 2002.
Biologi Jilid 1 Edisi 5. Jakarta: Erlangga.
Campbell NA, Reece JB, Mitchell LG. 2003.
Biologi Jilid 2 Edisi 5. Jakarta: Erlangga.
Chase MW, Soltis DE, Olmstead RG. 1993. Phylogenetics of seed plants: An analysis of nucleotide sequences from the plastid gene rbcL. Ann Missouri Bot Gard
80:528-580.
Cintamulya I. 2011. Aplikasi Penanda Molekuler Mikrosatelit/SSRs (Simple Sequence Repeats) Untuk Menunjang Program Pemuliaan Tanaman. Berk. Penel. Hayati Edisi Khusus 7A: 161–165.
Crandall E, Barber P. 2007. Laboratory Protocols: Agarose Electrophoresis. The Barber Lab. UCLA Department of Ecology and Evolutinary Biology.
Doyle JJ, Doyle JL. 1987. A rapid DNA isolation procedure from small quantities of fresh leaf tissue. Phytochem Bull 19: 11-15.
Estoup A, Jarne P, Cornuet JM. 2002. Homoplasy and mutationmodel at microsatellite loci and their consequences for population genetics analysis. Mol Ecol.
17
Fairbank DJ, Andersen WR. 1999. Genetics: The continuity of life. London International Thomsom Publishing Company: 277-282.
Fedde F. 1913. Repetorium Specierum Novarum Regni Vegetabilis Volume 12.
Berlin: Selbstverlag des Herausgebers.
Felsenstein J. 1985. Confidence intervals on phylogenies: An approach using the bootstrap. Evolution 39: 783-791.
Graham A, Newton CR. 1997. PCR (Polimerase Chain Reaction). Ed Ke-2. New York: Springer Verlag.
Henry T, Iwen PC, dan Hinrichs SH. 2000. Identification of Aspergillus species using internal transcribed spacer regions 1 and 2.
J Clin Microbiol 38: 1510–1515.
Hidayat T, Pancoro A. 2008. Ulasan kajian filogenetika molekuler dan peranannya dalam menyediakan informasi dasar untuk meningkatkan kualitas sumber genetik anggrek. Jurnal AgroBiogen 4:35-40.
Hillis DM, Moritz C, Mable BK. 1996. Molecular Systematic. 2nd Ed. Massachusetts :Sinauer Assocites.
Horton Derek. 2011. Advances in Carbohydrate Chemistry and Biochemistry Vol. 6. London: Academic Press.
Jose J, Usha R. 2000. Extraction of geminiviral DNA from a highly mucilaginous plant (Abelmoschus esculentus). Plant Mol Biol Rep 18: 349 - 355.
Karmana I Wayan. 2009. Kajian Evolusi Berbasis Urutan Nukleotida. GaneÇ Swara Edisi Khusus 3:75-81.
Kirana et al. 2006. Anticancer properties of Panduratin a isolated from Boesenbergia pandurata (Zingiberaceae).Journal of Natural Medicine 61:131-137.
Kress WJ, Prince LM, Williams KJ. 2002. The phylogeny and a new classification of the gingers (Zingiberaceae): evidence from molecular data. Am JBot89:1682-1696.
Kress et al. 2005. The molecular phylogeny of
Alpinia (Zingiberaceae): a complex and polyphyletic genus of Gingers. Am J Bot 9: 167-168.
Lehninger AL, Nelson DL, Cox MM. 2004. Lehninger Principles Of Biochemistry 4th Edition. New York: WH Freeman.
Tropp BE. 2008. Molecular Biology: Genes to Protein. London: Jones & Bartlett Publishers.
McCulloug MJ, Clemons KV, McCucker JH, Stevens DA. 1998. Intergenic transcribed spacer PCR ribotyping for differentiation of Saccharomyces species and interspecific hybrids. J Clin Microbiol
36:1035–1038.
McPherson M, Moller S. 2006. PCR 2nd Edition. New York: Taylor & Francis Group.
Mikkelsen SR, Corton E. 2004.
Bioanalytical Chemistry. New Jersey: John Wiley & Sons.
Moller M, Cronk QCB. 1997. Origin and relationships of Saintpaulia (gesneriaceae) based on ribosomal DNA internal transcribed spacer (ITS) sequences. Am J Bot 84: 956–965.
Mort ME, Soltis PS, Soltis DE, Maery ML. 2000. Comparison of three methods for estimating internal support for phylogenetic trees. Systematic Biology 49: 160-170.
Muladno. 2002. Teknologi Rekayasa Genetika. Bogor: Pustaka Wirausaha Muda.
Pevsner J. 2003. Bioinformatics andFunctional Genomics. New Jersey : Wiley-Liss, John Wiley & Sons Inc.
Porebski S, Bailey LG, Baum BR. 1997. Modification of CTAB DNA extractions protocol for plants cotaining high polysacharide and polyphenol components. Plant Molecul Biology reporter 15: 8-15.
Reysenbach AL, Voytek M, Mancinelli R. 2001. Thermophiles: Biodiversity, Ecology, and Evolution. New York: Plenum Publishers.
Santoso PJ. 2005. Modified CTAB-based DNA isolationprocedure for fruit crops.
Jurnal Stigma14:1-4.
Sohn et al. 2005. Protective effects of Panduratin egainst oxidative damage of tert-Butylhydroperoxide in human HepG2 cells. Biological and Pharmaceutical Bulletin 28:1083-1086.
Soltis PS, Soltis DE, Doyle JJ. 1992.
Molecular Systematics of Plant. New York: Chaman & Hall.
Sudjadi. 2008. Bioteknologi Kesehatan. Yogyakarta: Kanisius.
Surzycki,S. 2000. Basic Techniques in Molecular Biology. New York: Springer-Verlag Berlin.
Swofford DL. 1998. PAUP*4.0b10: Phylogenetic Analysis Using Parsimony (*and other Methods), Version 4. Sunderland: Sinauer Associates.
Swofford DL. 2002. PAUP*: phylogenetic analysis using parsimony (*and oth