MENGGUNAKAN TEKNIK
REAL TIME
RT-PCR
AMINAH
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Dengan ini saya menyatakan bahwa tesis Kajian Persistensi dan Penularan Virus Avian Influenza di Peternakan Itik menggunakan Teknik Real Time RT-PCR adalah karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apapun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan mauoun tidak dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Februari 2012
AMINAH. The Study of Persistence and Transmission of Avian Influenza Virus in Duck Farm using Real Time RT-PCR Technique. Under supervision of SURACHMI SETIYANINGSIH and IDWAN SUDIRMAN.
Highly pathogenic avian influenza (HPAI) H5N1 virus has been a major threat to poultry industry and human health in Indonesia over the past several years. The existence of backyard and free-range duck raising system has been hypothesized to play role in the disease circulation. This study investigated H5 virus infection and circulation occurring in three types of free-range duck farms in Indramayu District, West Java, Indonesia from September 2009 to March 2010. One hundred and eighty pairs of cloacal-oropharyngeal swab samples from sentinel ducks placed in six farms of the three farm types were collected every month for seven months period and screened in pools for influenza A virus. In addition to the sentinel ducks, 30 non-sentinel ducks were included at the first and the last sampling month. Of the total 648 pool samples collected for seven months, 91 pools (14%) were found influenza A positive at repeated events. The virus was more commonly found on type 1 farms followed by type 2 and 3 farms. The individual swab samples of influenza A positive pools were tested for H5 subtype. Of the total 91 pools of influenza A positive samples, 455 individual samples were tested for H5 subtype and 50 (11%) individual samples were found H5 positive. The H5 subtype was found at certain points of sampling time and was more commonly found in type 2 farms followed by type 1 and was not found on type 3 farms. The H5 virus was likely to be transmitted within duck farm but was unlikely to be maintained for a long time.
Avian influenza (AI) telah menjadi masalah global maupun nasional yang mengakibatkan kerugian besar bagi industri perunggasan. Virus highly pathogenic avian influenza (HPAI) H5N1 bersifat 100% mematikan bagi ayam dan unggas
menghilang seiring kematian itik yang terinfeksi atau pembersihan virus
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan
atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan,
penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau
tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan
yang wajar IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis
MENGGUNAKAN TEKNIK
REAL TIME
RT-PCR
AMINAH
Tesis
Sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Mikrobiologi Medik
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Nama : Aminah Nomor Pokok : B253090011
Program Studi : Mikrobiologi Medik
Disetujui: Komisi Pembimbing
drh. Surachmi Setiyaningsih, Ph.D Ketua
Dr. drh. Idwan Sudirman Anggota
Diketahui:
Ketua Program Studi Mikrobiologi Medik
A.n. Dekan Sekolah Pascasarjana IPB Sekretaris Program Magister
Prof. Dr. drh. Fachriyan H Pasaribu Prof. Dr. Ir. Nahrowi, M.Sc
Puji syukur kepada Allah SWT atas limpahan berkahNya sehingga penulis dapat menyelesaikan penelitian dan penyusunan tesis yang berjudul “Kajian Persistensi dan Penularan Virus Avian Influenza di Peternakan Itik mengunakan Teknik Real Time RT-PCR” ini. Karya ilmiah ini disusun sebagai salah satu syarat dalam menyelesaikan studi program Magister pada Sekolah Pascasarjana Institut Pertanian Bogor.
Terima kasih penulis sampaikan kepada drh. Surachmi Setiyaningsih, PhD atas kesempatan dan segala bimbingan yang diberikan sehingga karya ilmiah ini dapat terwujud, kepada Dr. drh. Idwan Sudirman atas bimbingan dan arahan dalam diskusi-diskusi selama penyusunan tesis, dan kepada Prof. Dr. drh. Retno D. Soejoedono, MS sebagai penguji luar komisi pada ujian tesis ini. Terimakasih kepada seluruh staf pengajar serta pegawai laboratorium program studi Mikrobiologi Medik atas curahan ilmu dan tenaga yang tiada henti selama penulis menyelesaikan studi.
Terima kasih kepada Colorado State University (CSU) atas dukungan dana penelitian yang penulis kerjakan, khususnya kepada Dr. Kristy Pabilonia dan Christina Weller dari CSU Veterinary Diagnostic Laboratory atas pelatihan yang diberikan. Demikian juga kepada rekan-rekan dokter hewan yang tergabung dalam Center for Indonesian Veterinary Analytical Studies (CIVAS) atas kerja keras selama pengambilan sampel di lapangan.
Terima kasih yang tak terhingga penulis sampaikan kepada Dr. Yety Rochwulaningsih, MS atas segala dukungan agar penulis melanjutkan studi serta kepada keluarga besar Bukittinggi dan Sragen atas kebersamaan dan kehangatan keluarga yang penulis terima. Tidak lupa kepada rekan-rekan drh. Emilia, Yuliana Radja Riwu, Wury Kadarsih, Wiwin Mukti, dan Zakiyah Widowati untuk semua persahabatan. Terimakasih juga kepada semua pihak yang mendukung penelitian serta penyusunan tesis ini.
Semoga tesis ini bermanfaat dan menginspirasi banyak pihak untuk giat melakukan penelitian dan memberikan yang terbaik bagi kemajuan ilmu pengetahuan dan kesejahteraan bangsa. Segala saran dan masukan yang menunjang demi kebaikan kedepan kami terima dengan tangan dan pikiran terbuka.
Bogor, Februari 2012
AI Avian influenza
Ct Cycle threshold
HA Hemaglutinin
HPAI Highly pathogenic avian influenza
LPAI Low pathogenic avian influenza
M1 Protein matriks 1
M2 Protein matriks 2 (ion channel) MA Matriks
NA Neuraminidase
NEP Nuclear export protein
NLS Nuclear localization signal
NP Nukleoprotein NS Non-struktural
ORF Open reading frame
PA Polymerase acidic
PB1 Polymerase basic 1
PB2 Polymerase basic 2
PCR Polymerase chain reaction
Rn Normalized reporter
RNA Ribonucleic acid
RNP Ribonukleoprotein
RRT-PCR Real time reverse transcriptase polymerase chain reaction
RT-PCR Reverse transcriptase polymerase chain reaction
SA Sialic acid
Sentinel Hewan yang sengaja ditempatkan untuk merekam satu atau rangkaian kejadian penyakit dalam program sureveilans prospektif
Halaman
DAFTAR TABEL ... xvi
DAFTAR GAMBAR ... xvii
DAFTAR LAMPIRAN ... xviii
PENDAHULUAN ... 1
Latar Belakang ... 1
Tujuan ... 2
Manfaat ... 3
Hipotesis ... 3
TINJAUAN PUSTAKA ... 4
Virus Influenza A ... 4
Siklus replikasi virus ... 8
Antigenic drift ... 11
Antigenic shift ... 12
Influenza A pada Unggas Liar ... 13
Influenza A pada Unggas Domestik ... 15
Ekologi Itik dan Perannya dalam Penyebaran Influenza A ... 15
Patobiologi Avian Influenza pada Itik ... 19
AI Patogenitas Rendah (low-pathogenic avian influenza, LPAI) ... 20
AI Patogenitas Tinggi (highly pathogenic avian influenza, HPAI) ... 21
Teknik Diagnostik Avian Influenza ... 23
RRT-PCR untuk Deteksi Avian Influenza ... 23
BAHAN DAN METODE ... 31
Waktu dan Tempat ... 31
Bahan dan Alat ... 31
Sampel ... 31
Metode ... 32
Pooling ... 32
Isolasi RNA ... 33
RT-PCR Konvensional ... 33
Real Time RT-PCR ... 34
Analisis Data ... 35
HASIL DAN PEMBAHASAN ... 36
RT-PCR Konvensional dan Real Time ... 36
Influenza A ... 38
Subtipe H5 ... 41
SIMPULAN DAN SARAN ... 45
DAFTAR PUSTAKA ... 46
Halaman
Tabel 1. Pasangan primer dan probe untuk deteksi gen tertentu. ... 29
Tabel 2. Virus AI di tiga tipe peternakan itik angon. ... 39
Tabel 3. Persentase sampel usap positif di 6 peternakan itik ... 40
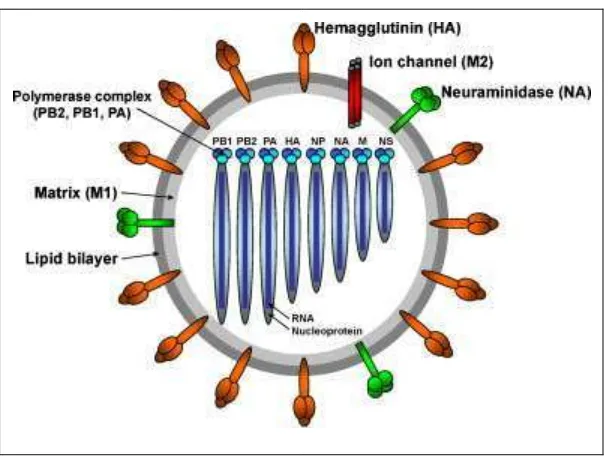
Gambar 1.Diagram skematis struktur virus influenza A ... 4
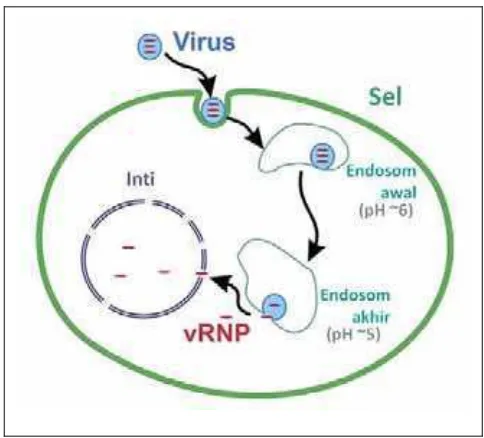
Gambar 2.Endositosis virus influenza ... 9
Gambar 3.Grafik tingkat kelangsungan hidup itik. ... 20
Gambar 5. Perbandingan hasil PCR konvensional dan real time ... 36
Gambar 6. Grafik amplifikasi real time RT-PCR. ... 37
Gambar 7. Distribusi temporal VAI di tiga tipe peternakan itik angon ... 41
Lampiran 1. Tabulasi nilai Ct MA dan H5 di peternakan 1 ... 59
Lampiran 2. Tabulasi nilai Ct MA dan H5 di peternakan 2 ... 60
Lampiran 3. Tabulasi nilai Ct MA dan H5 di peternakan 3 ... 61
Lampiran 4. Tabulasi nilai Ct MA dan H5 di peternakan 4 ... 62
Lampiran 5. Tabulasi nilai Ct MA dan H5 di peternakan 5 ... 63
Latar Belakang
Avian influenza (AI) telah menjadi masalah global maupun nasional yang mengakibatkan kerugian besar bagi industri perunggasan. Sejak terjadi wabah
highly pathogenic avian influenza (HPAI) pertama pada pada unggas di akhir tahun 2003, hingga Januari 2012 30 dari 33 Provinsi di Indonesia telah tertular (OIE 2012). Kasus AI pada manusia di Indonesia mulai terjadi pada bulan Juni 2005, dan hingga bulan Januari 2012 WHO telah mencatat 184 kasus dengan 152 (82,6%) diantaranya mengakibatkan kematian (WHO 2012). Hingga Oktober 2010 kasus AI pada manusia di Indonesia paling tinggi terjadi di Provinsi DKI Jakarta dan dari 46 kasus konfirmasi, 39 (84,8%) diantaranya meninggal dunia (Dinkes-Jabar 2010). Kasus AI pada manusia tertinggi kedua terjadi di Provinsi Jawa Barat yaitu dari 41 kasus konfirmasi, 36 (87,8%) diantaranya meninggal dunia dan tersebar di 14 Kabupaten/Kota (Dinkes-Jabar 2010).
Unggas air liar merupakan reservoir utama virus influenza A dan dapat menularkannya ke unggas domestik dan mamalia, termasuk manusia. Dari 16 subtipe HA virus influenza A, hanya subtipe H5 dan H7 yang dapat menyebabkan
VAI H5 pada populasi itik karena hasilnya tidak akan akurat, demikian juga dengan peran itik dalam wabah HPAI.
Di Indonesia beberapa jenis itik telah lama dikembangkan untuk dimanfaatkan sebagai sumber protein hewani berupa daging maupun telur. Jenis itik yang berkembang di beberapa daerah antara lain itik alabio di Kalimantan Selatan, itik tegal di Jawa Tengah, dan itik pitalah di Sumatera Barat (Suswono 2011a; Suswono 2011b; Suswono 2011c). Jawa Barat memiliki populasi itik paling tinggi di Indonesia yaitu mencapai 4,4 juta ekor pada tahun 2002 dan terus meningkat hingga 8,2 juta ekor pada tahun 2009, 2 juta ekor diantaranya berada di Kabupaten Indramayu (Ditjennak 2010) dengan mayoritas pemeliharaan menerapkan sistem angon sehingga dijadikan sebagai lokasi penelitian mengenai virus avian influenza (VAI) H5 di peternakan itik angon.
Keberadaan VAI dapat dideteksi menggunakan teknik reverse transcriptase
PCR (RT-PCR). Teknik ini memiliki beberapa keuntungan antara lain dapat digunakan untuk berbagai jenis sampel, lebih cepat dan lebih ramah lingkungan dibandingkan isolasi virus pada embrio ayam, dan karena virus tidak aktif sejak awal pemrosesan, keamanan dan keselamatan biologis juga lebih mudah untuk dipertahankan (Spackman dan Suarez 2008). Secara konvensional teknik ini memerlukan waktu beberapa jam dan pembacaan hasilnya melibatkan bahan kimia yang dapat merugikan pengguna maupun lingkungan. Perkembangan teknologi telah melahirkan real-time RT-PCR (RRT-PCR) yang mulai banyak digunakan sejak awal tahun 2000-an dalam rangka pengawasan rutin, selama wabah, dan untuk penelitian. Beberapa keuntungan RRT-PCR dibandingkan dengan RT-PCR konvensional antara lain dari segi sensitivitas dan spesifisitas yang lebih tinggi, memerlukan waktu yang lebih singkat, bersifat kuantitatif, ramah lingkungan, dan meskipun biaya yang diperlukan untuk investasi peralatan lebih tinggi namun untuk operasional dan pengamanan lingkungan teknik ini memerlukan biaya yang lebih sedikit.
Tujuan
Teknik RRT-PCR digunakan untuk mendeteksi persistensi dan penularan VAI H5 dalam peternakan itik angon serta menentukan rute pengeluaran (shedding) virus.
Manfaat
Memberi informasi tentang persistensi dan penularan VAI subtipe H5 di peternakan itik sebagai pertimbangan dalam pengendalian AI.
Hipotesis
Virus Influenza A
Virus influenza merupakan virus RNA untai negatif dengan genom tersegmentasi berisi tujuh sampai delapan segmen gen yang termasuk kedalam famili Orthomyxoviridae. Berdasarkan perbedaan sifat antigenik protein matriks dan nukleoprotein, virus influenza dikelompokkan kedalam tiga tipe yaitu A, B, dan C yang masing-masing memiliki kecenderungan inang dan patogenisitas berbeda. Virus influenza A dan B memiliki struktur yang tidak dapat dibedakan dibawah mikroskop elektron (Bouvier dan Palese 2008) berbeda dengan virus influenza C. Virus influenza A dan B memiliki delapan segmen gen RNA untai tunggal, sedangkan virus influenza C memiliki tujuh segmen dan masing-masing menyandi setidaknya satu protein (Murphy et al. 1999).
(RNP) virus. Protein M1 juga berikatan dengan protein pengeluaran dari inti (nuclear export protein NEP) yang memperantarai pengeluaran M1-RNP melalui nukleoporin ke dalam sitoplasma (Bouvier dan Palese 2008). Protein M2 yang berukuran kecil merupakan ion channeltransmembran dan hanya ditemukan pada virus influenza A. Protein M2 memiliki bagian luar yang berada di permukaan selubung virus bersama dengan HA dan NA. Protein M2 merupakan target obat anti influenza dari kelas amantadine yang memblokir aktivitas ion channel dan mencegah pelepasan selubung virus (Pinto et al. 1992; Wharton et al. 1994; Sheu
et al. 2011). Selain itu, M2 merupakan protein permukaan sehingga dijadikan sebagai komponen vaksin (Slepushkin et al. 1995; Neirynck et al. 1999).
Hemaglutinin merupakan protein membran integral tipe I terglikosilasi yang berfungsi sebagai protein pengikat reseptor dan protein fusi serta merupakan target utama netralisasi oleh antibodi inang (Cross et al. 2001; Hulse et al. 2004; Hoffmann et al. 2005; Gambaryan et al. 2006). Protein ini dapat mengenali asam sialat (N-acetyl neuraminic acid) yang terikat pada gula di ujung glikoprotein sel inang. Virus influenza A memiliki berbagai HA spesifik dengan isomerisasi ikatan glikosidik berbeda untuk disakarida yang terdiri atas sialic acid (SA) dan galaktosa atau N-asetilgalaktosamin (GalNAc). Reseptor HA pada unggas memiliki spesifisitas ikatan terhadap SA Į2,3 sel bersilia, sementara HA pada manusia memiliki spesifisitas ikatan yang lebih tinggi terhadap SA Į2,6 sel tidak bersilia (Matrosovich et al. 2004). Struktur kristal molekul HA berbentuk trimer dengan dua regio struktural berbeda yaitu bagian batang dan kepala (Wilson et al.
aktivasi (Chen et al. 1998). Pada manusia dan mamalia lain, enzim ini berupa triptase Clara yang diproduksi oleh sel epitel bronkiolus (Murakami et al. 2001). Aktivasi pembelahan HA dalam sel usus dan/atau pernafasan unggas kemungkinan juga memerlukan protease serupa. Situs pengenalan protease dapat berubah menjadi urutan menyerupai furin R-X-R/K-R pada subtipe H5 dan H7 bila mengalami mutasi insersional pada situs pembelahan HA. Perubahan situs pembelahan HA menjadi polibasa ini memperluas spesifisitas protease sehingga memungkinkan aktivasi pembelahan intraseluler dan replikasi virus secara sistemik pada unggas yang mengakibatkan influenza unggas sangat patogen (highly pathogenic avian influenza, HPAI) (Werner 2006). Akumulasi perubahan yang relatif kecil pada situs antigenik HA yang dikenali oleh antibodi disebut
antigenic drift yang menghasilkan strain virus yang tidak lagi dapat dinetralisir oleh antibodi sehingga inang menjadi rentan terhadap infeksi kembali oleh strain yang mengalami drift.
Neuraminidase (NA) merupakan tetramer berbentuk seperti jamur yang menancap pada selubung virus melalui domain transmembran (Colman et al.
1983; Varghese et al. 1983). Sebagai glikoprotein membran integral tipe II dengan aktivitas enzimatik sialidase (neuraminidase), NA diperlukan untuk pembelahan SA sel inang yang memungkinkan pelepasan virion baru dan melepaskan SA dari glikoprotein virus untuk mencegah agregasi partikel progeni virus (Palese et al.
1974). Hemaglutinin dan NA merupakan target antigenik utama respon imun humoral terhadap virus influenza A dengan NA menjadi target obat antivirus oseltamivir dan zanamivir (De Clercq 2006).
Sintesis RNA virus influenza A memerlukan polimerase yang terdiri atas tiga subunit PA, PB1, dan PB2. Kompleks heterotrimer polimerase terbentuk melalui interaksi PA dengan PB1 dan PB1 dengan PB2. Protein PA berperan penting dalam penempelan, katalisis, dan lokalisasi inti oleh polimerase (Guu et al. 2008). Protein PB1 berfungsi sebagai RNA polimerase sedangkan PB2 berperan dalam sintesis mRNA melalui pengikatan bagian kepala mRNA inang. Protein non struktural kecil lainnya yaitu PB1-F2 secara bervariasi disandi oleh gen PB1 melalui bingkai bacaan (reading frame) alternatif. Protein ini menjadikan membran dalam mitokondria sebagai target dan mungkin berperan dalam apoptosis selama infeksi virus influenza A selain memiliki aktivitas antagosnisme interferon (Dudek et al. 2011). Gen PB1 juga menyandi polipeptida ketiga yang diekspresikan melalui penggunaan kodon AUG diferensial yang disebut N40 (Wise et al. 2009).
Protein non-struktural 1 (NS1) memiliki beberapa domain fungsional antara lain: domain N-terminal pengikat RNA (residu 1-73) yang pada in vitro mengikat beberapa spesies RNA dengan afinitas rendah dan memiliki sinyal lokalisasi inti (nuclear localization signal, NLS) (Hatada dan Fukuda 1992; Qian et al. 1995; Chien et al. 2004), dan domain C-terminal 'efektor' (residu 74-230) yang memperantarai interaksi dengan protein sel inang dan secara fungsional menstabilkan domain pengikat RNA (Wang et al. 2002). Keseluruhan NS1 merupakan homodimer dengan domain pengikat RNA dan domain efektor berkontribusi terhadap multimerisasi (Nemeroff et al. 1995). NS1 memiliki fungsi pleiotropik, antara lain pengikatan dsRNA, peningkatan translasi mRNA virus, penghambatan proses mRNA inang dan antagonisme interferon tipe I (Palese dan Shaw 2007). Protein NS2 (disebut juga protein ekspor inti, NEP) ditemukan dalam virion dan memfasilitasi pengeluaran kompleks RNP virus dari dalam inti (O'Neill et al. 1998).
Berdasarkan karakterisasi antigen glikoprotein permukaan HA dan NA virus influenza A dikelompokkan kedalam 16 subtipe HA dan 9 NA (Fouchier et al.
2005). Secara teoritis kombinasi HA-NA dapat membentuk 144 subtipe, dan setidaknya 116 kombinasi subtipe ini telah diisolasi dari unggas (Krauss et al.
nomenklatur virus influenza adalah sebagai berikut: pertama, tipe virus (A, B, atau C), kemudian inang (jika bukan manusia), tempat isolasi, nomor isolasi dan tahun isolasi (dipisahkan dengan garis miring). Untuk virus influenza A, subtipe HA (H1-H16) dan NA (N1-9) ditulis dalam tanda kurung. Sebagai contoh, strain yang termasuk dalam vaksin trivalen influenza manusia untuk musim 2010-2011 di Amerika Serikat adalah: A/California/7/2009 (H1N1), A/Perth/16/2009 (H3N2) dan B/Brisbane/60/2008.
Virus influenza tipe B dan C menginfeksi dan hampir selalu diisolasi dari manusia meskipun virus influenza B pernah diisolasi dari anjing laut dan virus influenza C pernah diisolasi dari babi dan anjing (Wright et al. 2007). Sebaliknya, virus influenza A dapat menginfeksi berbagai hewan berdarah panas seperti unggas, babi, kuda dan manusia. Virus AI yang menjadi penyebab flu burung/AI termasuk kedalam virus influenza A dengan unggas air sebagai reservoir alami untuk semua subtipenya (Webster et al. 1992). Tiga sifat penting yang membuat virus influenza mudah beradaptasi, mampu menghindari respon kekebalan inang, dan mampu menginfeksi spesies inang baru (Webster et al. 1992; Bahl et al.
2009) yaitu: pertama, enzim polimerase yang mengkatalisis replikasi RNA dari cetakan RNA mudah melakukan kesalahan; kedua, kurangnya koreksi kesalahan selama replikasi; dan ketiga, struktur genom virus influenza memungkinkan untuk pertukaran segmen antar virus-virus yang menginfeksi sel di waktu bersamaan melalui proses yang disebut reassortment.
Siklus replikasi virus
juga terdapat pada epitel saluran pernafasan manusia meskipun jumlahnya lebih sedikit dibandingkan Į2,6 (Couceiro et al. 1993; Matrosovich et al. 2004) sehingga manusia dan primata lain juga dapat terinfeksi oleh VAI meskipun dengan efisiensi yang lebih rendah dibandingkan infeksi oleh strain manusia (Tian
et al. 1985; Beare dan Webster 1991). Perbedaan ekspresi SA pada saluran pernafasan mamalia membantu menjelaskan infektivitas rendah tetapi patogenisitas tinggi pada beberapa strain VAI. Pada manusia protein SA dengan ikatan Į2 ,3 dalam jumlah sedikit terdapat di saluran pernafasan bawah seperti bronkiolus dan alveoli. Akses partikel virus dari udara ke paru-paru tidak semudah virus mencapai saluran pernafasan bagian atas seperti nasofaring, sinus paranasal, trakea, dan bronkus, sehingga infeksi VAI relatif jarang terjadi pada manusia. Namun ketika strain VAI menginfeksi paru-paru manusia, pneumonia berat dan progresif dapat terjadi dengan angka kematian melebihi 60% (Gambotto
et al. 2008).
inang (Stegmann 2000; Sieczkarski dan Whittaker 2005). Ion hidrogen dari endosom dipompa ke dalam partikel virus melalui ion channel M2. Pengasaman internal virion influenza melalui channel M2 mengganggu interaksi protein-protein internal sehingga RNP dapat dilepaskan keluar dari matriks virus ke dalam sitoplasma sel (Martin dan Helenius 1991).
Setelah keluar dari virion, RNP masuk kedalam inti sel inang dengan memanfaatkan sinyal lokalisasi inti (NLS) oleh protein virus (NS1) yang memerintahkan protein sel untuk memasukkan RNP dan protein virus lainnya ke dalam inti sel inang (Cros dan Palese 2003). Inti merupakan tempat dimana semua sintesis RNA virus terjadi, tempat RNA poliadenilasi (mRNA) yang bertindak sebagai cetakan bagi sel inang untuk translasi, dan tempat segmen RNA virus yang membentuk genom progeni virus. Polimerase RNA yang merupakan komponen RNP juga masuk kedalam inti dan menggunakan RNA virus untai negatif sebagai cetakan untuk mensintesis dua RNA untai positif, yaitu cetakan mRNA untuk sintesis protein virus, dan RNA komplementer (cRNA) untuk membentuk lebih banyak RNA virus untai negatif penyusun genom (Bouvier dan Palese 2008).
Berbeda dengan mRNA sel inang yang terpoliadenilasi oleh poli (A) polimerase spesifik, ujung penutup poli (A) mRNA virus influenza disandi dalam bentuk RNA virus untai negatif dengan lima sampai tujuh residu urasil yang ditranskripsikan oleh polimerase virus menjadi untai positif dengan adenosin membentuk ekor poli (A) (Robertson et al. 1981; Li dan Palese 1994). Pembentukan ujung penutup RNA messenger juga terjadi dengan cara unik yang sama, di mana protein PB1 dan PB2 "mencuri" primer berujung penutup 5' dari transkrip pre-mRNA inang untuk memulai sintesis mRNA virus, proses ini disebut "cap snatching" (Krug 1981). Setelah terpoliadenilasi dan ujungnya ditutup, mRNA asal virus dapat keluar dan diterjemahkan seperti mRNA inang. Pengeluaran segmen RNA virus dari inti diperantarai oleh protein M1 dan NEP/NS2 virus (Cros dan Palese 2003).
kedalam aparatus Golgi untuk modifikasi pasca-translasi. Ketiga protein tersebut memiliki sinyal penyusun apikal yang kemudian mengarahkan mereka ke membran sel untuk perakitan virion. Meskipun relatif sedikit yang diketahui tentang translasi dan penyortiran protein yang bukan bagian dari selubung, M1 diperkirakan berperan dalam membawa kompleks RNP-NEP berkontak dengan protein selubung HA, NA, dan M2 untuk dikemas di membran sel inang (Palese dan Shaw 2007).
Virus influenza tidak sepenuhnya menular kecuali virion yang lengkap berisi genom delapan segmen, atau tujuh segmen untuk virus influenza C. Sebelumnya pengemasan RNA virus dianggap sebagai sebuah proses yang sepenuhnya acak, di mana segmen RNA virus secara tidak beraturan dimasukkan ke dalam tunas partikel virus dan hanya yang memiliki genom lengkap yang dapat menular. Namun bukti baru menunjukkan bahwa pengemasan merupakan proses selektif di mana sinyal pengemasan pada semua segmen RNA virus memastikan bahwa genom lengkap dimasukkan ke dalam setiap partikel virus (Bancroft dan Parslow 2002; Fujii et al. 2003).
Pertunasan (budding) virus influenza terjadi di membran sel yang dimulai dengan akumulasi protein matriks M1 di sisi sitoplasma dari lipid bilayer. Ketika
budding selesai, tonjolan HA tetap menempelkan virion pada SA di permukaan sel hingga partikel virus secara aktif dilepaskan oleh aktivitas sialidase protein NA (Colman et al. 1983; Varghese et al. 1983).
Antigenic drift
Virus influenza A terus berevolusi dengan tingkat mutasi tinggi yang berkisar antara 1×10-3 sampai 8×10-3 substitusi/situs/tahun (Chen dan Holmes 2006). Mutasi selektif pada domain antigenik yang terjadi secara bertahap dalam satu strain dan menghindarkan virus dari sistem kekebalan disebut antigenic drift
glikoprotein permukaan seperti HA dapat menguntungkan virus karena memungkinkan virus menghindar dari sistem kekebalan. Proses replikasi virus sangat rawan mutasi karena enzim polimerase yang mengkatalisis replikasi RNA dari cetakan RNA mudah melakukan kesalahan disertai kurangnya koreksi kesalahan selama replikasi. Hal ini menjadi penyebab terjadinya antigenic drift.
Antigenic drift merupakan salah satu strategi virus influenza untuk menghindar dari sistem kekebalan inang yang meningkat karena vaksinasi. Perubahan antigenik yang terjadi di daerah epitop merupakan hambatan untuk pengembangan vaksin karena vaksinasi yang efektif hanya dapat terjadi bila strain epidemik sesuai dengan strain vaksin (Stohr 2002). Gen HA sebagai target netralisasi antibodi menjadi contoh klasik protein antigen yang mengalami mutasi titik yang menumpuk pada epitop atau daerah yang dikenali antibodi (Webster et al. 1982; Wilson dan Cox 1990). Antigenic drift pada gen HA dapat dipercepat oleh vaksinasi (Lee et al. 2004) yang suboptimal karena tekanan oleh kekebalan hasil imunisasi terhadap virus yang sebelumnya bereplikasi dan beredar antar dan intra spesies (Abdelwhab dan Hafez 2011) memaksa virus untuk beradaptasi. Virus H5N1 dapat bermutasi secara intensif pada unggas yang divaksinasi sehingga berpotensi menimbulkan pandemi. Gen HA dari 4 strain H5N1 yang beredar di Mesir mengalami perubahan asam amino pada epitop HA sehingga berbeda dengan VAI H5N1 awal yang ditemukan sejak program vaksinasi dimulai pada tahun 2006 yang berdampak pada virulensi H5N1 pada mamalia (Abdel-Moneim et al. 2011). Contoh lain virus influenza A yang mengalami antigenic drift adalah virus pandemi H1N1 2009 (pH1N1 2009) yang berasal dari babi. Residu 227 HA pada H1N1 babi yang berupa asam amino alanin mengalami perubahan menjadi asam glutamat sehingga mampu menular dan menimbulkan pandemi pada manusia (van Doremalen et al. 2011).
Antigenic shift
penting dalam evolusi virus influenza A (Holmes et al. 2005; Dugan et al. 2008) dan adaptasi inang (Garten et al. 2009, Scholtissek et al. 1978). Secara teori dapat terjadi 256 (28) kombinasi 8 segmen gen hasil reassortment antara dua virus dalam satu inang. Rekombinasi homolog jarang terjadi pada virus RNA negatif seperti virus influenza A (Boni et al. 2008) tetapi rekombinasi dengan pertukaran segmen gen diketahui berperan dalam perubahan virulensi dan adaptasi inang (Wright et al. 2007).
Influenza A pada Unggas Liar
Virus influenza A memiliki keragaman genetik dan antigenik yang tinggi dan tersebar pada berbagai spesies unggas liar di seluruh dunia. Penularan virus influenza A pada unggas air liar terjadi melalui rute fekal-oral dan menginfeksi sel-sel epitel saluran pencernaan dengan sedikit atau tanpa gejala penyakit. Virus bertahan melalui infeksi asimtomatik (low pathogenic, LPAI) pada unggas air dari ordoAnseriformes seperti itik dan angsa, ordo Charadriiformes seperti camar dan burung laut, serta ordo Passeriformes dan setidaknya 105 spesies unggas liar telah teridentifikasi membawa virus influenza A (Munster et al. 2007). Distribusi subtipe HA dan NA virus pada isolat unggas liar tidak merata. Sebagian besar subtipe HA dapat ditemukan pada Anseriformes sedangkan subtipe H13 dan H16 ditemukan pada Charadriiformes (Munster et al. 2007).
keseimbangan (Dugan et al. 2008). Keragaman genetik yang lebih rendah dimiliki oleh lima segmen gen VAI lainnya (PB2, PB1, PA, NP dan M). Analisis filogenetik juga menunjukkan perbedaan urutan asam inti yang jelas antara VAI yang berasal dari unggas di belahan dunia timur dan barat, sesuai dengan evolusi cekaman alopatrik (Dugan et al. 2008; Munster dan Fouchier 2009).
Banyaknya kombinasi HA-NA yang ditemukan pada unggas liar menunjukkan bahwa infeksi campuran dan reassortment VAI sering terjadi pada unggas liar (Wang et al. 2008) dan bahwa subtipe HA-NA memiliki kombinasi spesifik yang rendah. Keragaman genetik yang tinggi pada HA, NA dan NS bertolak belakang dengan 5 segmen gen penyandi protein internal yang memiliki stabilitas tinggi di tingkat asam amino. Hal ini menandakan bahwa kelima segmen gen tersebut telah melalui alur seleksi pemurnian. Kecocokan kelima gen tersebut untuk saling terkait dalam genom ditentukan oleh viabilitas fungsional, dengan sedikit cekaman selektif untuk mempertahankan mutasi yang menguntungkan. Urutan asam amino yang sangat stabil menunjukkan bahwa reassortment terjadi antara segmen-segmen yang secara fungsional setara. Dugan et al. berhipotesis bahwa VAI pada unggas liar berperan sebagai kolam (pool) besar yang berisi segmen-segmen gen yang memiliki kesetaraan fungsional sehingga dapat saling tukar membentuk konstelasi genom sementara tanpa ada cekaman selektif yang kuat agar tetap bertahan sebagai genom (Dugan et al. 2008).
Influenza A pada Unggas Domestik
Unggas domestik dari ordo Galliformes seperti kalkun, ayam, dan burung puyuh bukan merupakan reservoir virus influenza A unggas namun rentan terhadap infeksi oleh virus influenza A dari unggas liar yang telah beradaptasi. Virus influenza A yang telah beradaptasi pada Galliformes jarang kembali dan beredar di unggas liar (Swayne 2007) kecuali virus HPAI H5N1 Eurasia yang baru-baru ini diisolasi dari populasi unggas liar di Eropa dan Asia. Virus panzootik HPAI H5N1 galur Asia memiliki keunikan (Webster et al. 2007) yang dapat mengakibatkan kematian jutaan unggas di 64 negara di tiga benua. Adaptasi virus influenza A pada inang Galliformes secara molekuler belum sepenuhnya dapat dijelaskan namun diketahui melibatkan seleksi positif mutasi HA, NA (Perez et al. 2003; Campitelli et al. 2004), dan protein RNP (Wasilenko et al.
2008).
Virus influenza A yang diisolasi dari unggas domestik umumnya mempertahankan spesifisitas pengikatan reseptor HA Į2,3-SA (Wright et al.
2007). Ciri lainnya yaitu penghapusan in-frame sekitar 20 asam amino di daerah batang NA yang mengurangi aktivitas enzimatik NA (Baigent dan McCauley 2001) sebagai kompensasi terhadap penurunan aktivitas pengikatan reseptor HA virus influenza A dari unggas liar yang beradaptasi untuk bereplikasi di saluran pernafasan unggas domestik (Matrosovich et al. 1999). Strain virus influenza A H5 atau H7 yang beradaptasi pada unggas domestik berkembang menjadi HPAI melalui akuisisi mutasi insersi yang mengakibatkan situs pembelahan asam amino polibasa pada HA (Wright et al. 2007).
Ekologi Itik dan Perannya dalam Penyebaran Influenza A
Itik adalah anggota subfamili Anatinae yang menaungi spesies unggas air
Anseriformes. Subfamili ini tersebar di seluruh dunia dan menempati hampir semua habitat perairan. Ekologi unggas ini memungkinkan pemeliharaan dan penyebaran VAI.
sering menginfeksi saluran pernafasan bagian atas. Virus LPAI dalam populasi itik ditularkan melalui rute fekal-oral (Webster et al. 1992) yang dicirikan oleh tingginya jumlah usap kloaka positif dibandingkan trakea dan titer virus yang tinggi pada kotoran serta didukung oleh stabilitas virion dalam air meskipun penularan melalui aerosol tidak dapat diabaikan. Itik yang diinfeksi secara eksperimental mengeluarkan virion H4N7, H7N3, dan H11N9 dalam waktu lebih lama dan titer lebih tinggi melalui feses dibandingkan melalui trakea (Webster et al. 1978). Virus AI memasuki lingkungan ketika inang defekasi atau mengeluarkan leleran kemudian menginfeksi inang yang rentan melaui proses makan dan minum. Ketika segerombolan itik berenang di kolam kecil, diperkirakan sebanyak 1010 EID50/g/hari virion ditularkan ke lingkungan melalui kotoran masing-masing itik yang terinfeksi (Webster et al. 1978) dan VAI relatif stabil dalam air (Stallknecht et al. 1990; Webster et al. 1992). Keadaan ini menjelaskan mengapa prevalensi infeksi pada itik yang makan di permukaan lebih tinggi dibandingkan itik yang mencari makan di air yang lebih dalam (Olsen et al.
2006).
Data surveilans menunjukkan bahwa penularan VAI dalam populasi itik terjadi sepanjang tahun. Prevalensi infeksi menunjukkan pola siklus tahunan pada populasi itik di Amerika Utara (Olsen et al. 2006) (Krauss et al. 2004) dan Eurasia (Munster et al. 2007) yang memuncak sebelum dan selama migrasi musim gugur sebagai akibat dari masuknya itik remaja yang secara imunologis naif kedalam populasi (Hinshaw et al. 1985; Webster et al. 1992; Olsen et al.
beredar dalam populasi itik atau penurunan transmisi karena penyebaran populasi (Hinshaw et al. 1985).
Secara umum prevalensi infeksi di tempat migrasi musim dingin dan di tempat bersarang musim semi lebih tinggi pada populasi itik Eropa dibandingkan populasi itik Amerika Utara. Penjelasan yang paling mungkin untuk perbedaan ini adalah variasi acak, karena penelitian surveilans pada populasi itik di beberapa daerah di Amerika Utara dan Eropa sering memperoleh nilai prevalensi yang sedikit berbeda. Banyak faktor dapat mempengaruhi prevalensi termasuk ukuran populasi itik, lokasi pengambilan sampel, waktu pengambilan sampel, dan lain-lain.
Prevalensi infeksi paling rendah terjadi selama migrasi musim semi namun kembali meningkat setelah musim kawin ketika itik pindah ke tempat bersarang musim panas (Hinshaw et al. 1985; Krauss et al. 2004; Wallensten et al. 2007). Tidak jelas bagaimana populasi itik memperoleh virus AI selama musim semi setiap tahun. Ada dua kemungkinan bahwa itik mungkin membawa virus terus menerus selama migrasi yang ditunjukkan oleh prevalensi pada itik yang terus ada sepanjang tahun, meskipun daya tahan virus di habitat beku juga dapat berperan dalam kelangsungan hidup virus (Olsen et al. 2006) karena virion infektif mungkin dapat bertahan di dalam air beku melewati musin dingin di tempat itik berkembang biak dan menginfeksi saat itik tersebut kembali pada musim semi (Webster et al. 1978; Webster et al. 1992).
(Wagner et al. 2002). Meskipun gen H6 berasal dari Eurasia dan secara luas tersebar pada itik di Amerika Utara, analisis genom virus menunjukkan bahwa pertukaran gen antar benua antara Eurasia dan Amerika sangat terbatas (Krauss et al. 2007). Oleh karena itu kemunculan genotipe virus baru harus melalui mutasi dan reassortment genom-genom yang bersirkulasi dalam wilayah geografis tertentu. Kesempatan untuk mutasi dan reassortment ini terbuka lebar di daerah tempat itik istirahat dan mencari makan karena populasi itik dari berbagai tempat dan koridor migrasi berbeda datang dengan membawa kombinasi subtipe masing-masing (Wallensten et al. 2007). Koinfeksi itik dengan dua atau lebih subtipe virus sering terjadi (Sharp et al. 1997) sama seperti reassortment memunculkan virus yang sangat virulen pada unggas Galliformes namun memiliki patogenisitas rendah pada inang itik (Sturm-Ramirez et al. 2005).
Peran itik dalam pemeliharaan dan penyebaran virus influenza, dan terutama dalam pemunculan genotipe baru tergantung pada perilaku migrasi. Itik yang bermigrasi setiap tahun cenderung menyebarkan virus influenza di sepanjang rute migrasi terutama pada populasi itik domestik dan peliharaan di berbagai lokasi persinggahan (Olsen et al. 2006; Wallensten et al. 2007). Selanjutnya itik domestik membawa virus berdekatan dengan spesies lain dan berperan dalam penyebaran LPAI dan HPAI pada unggas domestik dan unggas darat lainnya (Hulse-Post et al. 2005; Sturm-Ramirez et al. 2005; Gilbert et al. 2006).
seropositif terhadap H5 VAI masing-masing pada ayam 33,37% (n=1.038), itik 43,44% (n=63), entog 28,21% (n=68), dan angsa 42,3% (n=11) (BALITVET 2006).
Patobiologi Avian Influenza pada Itik
Wabah AI pertama kali dilaporkan tahun 1878 terjadi pada ayam dan burung di Italia yang saat itu disebut penyakit Lombardia kemudian pada tahun 1901 Centanini dan Savonucci dapat mengidentifikasi organisme berukuran mikro yang menyebabkan wabah tersebut namun baru pada tahun 1955 Schafer dapat menunjukkan ciri-ciri organisme itu sebagai virus influenza A (Werner 2006). Virus influenza A biasanya tidak patogenik terhadap reservoir alaminya yaitu itik dan unggas air lain. Namun virus HPAI telah berevolusi dari yang tidak mengakibatkan atau sedikit menimbulkan gejala infeksi pada saluran pernafasan itik menjadi virus yang menyebabkan penyakit sistemik parah dan kematian (Pantin-Jackwood dan Swayne 2007). Wabah virus highly pathogenic avian influenza (HPAI) H5N1 pertama kali dilaporkan di Cina Selatan pada tahun 1996-1997, kemudian menyebar dan menyebabkan kematian unggas di Vietnam, Thailand, Indonesia dan Negara Asia Timur sejak awal tahun 2004 (Smith et al.
2006).
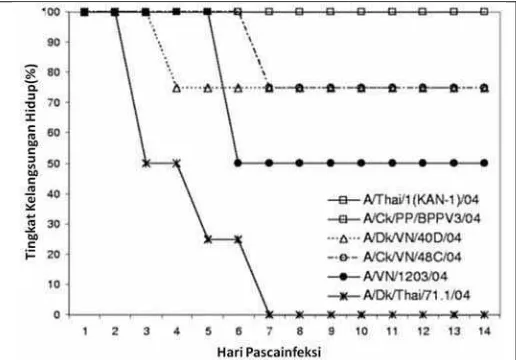
Gejala HPAI H5N1 sebelum tahun 2002 tidak terlihat pada itik. Penelitian eksperimental yang dilakukan oleh (Perkins dan Swayne 2002) yang menginfeksikan A/chicken/HK/220/97 pada itik tidak menemukan gejala klinis. Sedangkan infeksi virus HPAI H5N1 pada itik menggunakan isolat yang diperoleh pada tahun 2002 sampai 2004 menimbulkan gejala klinis seperti penurunan berat, lesu, diare, mata berkabut, dan ataksia kemudian mati meskipun beberapa strain yang diisolasi selama tahun 2002-2004 juga menunjukkan gejala yang ringan atau tidak ada sama sekali (Gambar 3) (Sturm-Ramirez et al. 2004). Itik yang diinfeksi virus A/duck/Thailand/71.1/2004 menunjukkan gejala panas tinggi, kesulitan bernafas, depresi, diare, gejala syaraf (ataksia, konvulsi, dan inkoordinasi), dan konjungtivitis dengan mortalitas 20-100% (Songserm et al.
paling menonjol ditemukan pada paru-paru seperti pneumonia, edema, kongesti, dan peradangan perivaskuler (Songserm et al. 2006). Pada itik yang menunjukkan gejala syaraf ditemukan kumpulan sel radang di sekitar pembuluh darah dan peradangan neuroglia. Pada itik ras pedaging ditemukan degenerasi otot jantung dengan perubahan patologis lain yang dapat terlihat antara lain hepatitis, tubulonefritis, pengecilan kelenjar limfoid dan enteritis (Songserm et al. 2006).
Gambar 3. Grafik tingkat kelangsungan hidup itik yang diinfeksi berbagai isolat VAI H5N1 diadaptasi dari Sturm-Ramirez et al. (2005). Kelompok virus LPAI: A/Thai/1(Kan-1)/04 dan A/Ck/PP/BPPV3/04, HPAI: A/Dk/VN/40D/04, A/Ck/VN/48C/04, A/Dk/Thai/71.1/04, dan A/VN/1203/04.
AI Patogenitas Rendah (low-pathogenic avian influenza, LPAI)
merupakan organ target virus LPAI pada itik sebagai tempat replikasi virus tanpa infeksi pada paru-paru. Lebih spesifik, replikasi virus LPAI diyakini terjadi di kriptus Lieberkühn usus besar (Kida et al. 1980).
Keragaman jenis itik juga berperan penting dalam patogenisitas virus influenza. Embrio itik Mallard yang diinokulasi dengan virus LPAI memiliki tingkat kematian lebih rendah daripada embrio entog. Antigen virus dapat ditemukan di organ-organ internal seperti sinus hidung, faring, trakea, bronkus, paru-paru, dan kantung hawa embrio itik Mallard tetapi tidak ditemukan pada embrio entog. Alasan mortalitas dan replikasi virus pada itik Mallard ini tidak jelas tetapi mendukung bukti bahwa itik Mallard berperan sebagai reservoir utama virus LPAI di alam (Mutinelli et al. 2003).
Pemahaman mengenai respon imun itik terhadap VAI masih terbatas meskipun beberapa penelitian mengenai respon antibodi serum itik yang terinfeksi secara alami maupun eksperimental telah dilakukan (Suarez dan Schultz-Cherry 2000). Itik Pekin putih yang diinokulasi virus LPAI H7N2 memberikan hasil titer antibodi HI yang sangat rendah tetapi virus tetap dikeluarkan hingga 7 hari pasca inokulasi. Inokulasi ulang setelah 46 hari dengan strain virus yang sama memberi respon antibodi yang lebih tinggi tetapi virus tidak ditemukan pada organ. Hasil ini disertai rendahnya respon imun sekunder setelah inokulasi menggunakan virus yang dilemahkan dalam formalin menunjukkan bahwa respon cepat imun pada itik yang diinfeksi ulang dapat membatasi infeksi influenza untuk rentang waktu tertentu (Kida et al. 1980). Infeksi yang pernah terjadi tidak dapat melindungi itik terhadap infeksi berikutnya oleh subtipe virus lain. Sebagai contoh, itik yang diinfeksi subtipe H4N6 terlindungi dari infeksi ulang dengan virus yang sama tetapi mengeluarkan virion selama 8 hari setelah ditantang dengan isolat H11N3 (Austin dan Hinshaw 1984).
AI Patogenitas Tinggi (highly pathogenic avian influenza, HPAI)
dan kepala gemetar meskipun tidak mati. Titer virus yang tinggi ditemukan pada organ pernafasan (paru-paru dan trakea), otak, hati, ginjal, dan usus besar disertai perubahan mikroskopik pada otak (ensefalitis), jantung (miokarditis dengan degenerasi dan nekrosis miosit), dan bursa (hiperplasia ringan pada folikel limfoid) (Kishida et al. 2005).
Neurotropisme dan pankreatotropisme virus terlihat pada penelitian lain yang menggunakan isolat virus HPAI. Itik yang ditantang dengan virus HPAI H5N1 pada dosis letal menunjukkan gejala neurologis berat, seperti tortikolis, inkoordinasi, tremor, dan kejang (Sturm-Ramirez et al. 2004; Vascellari et al.
2007). Imunohistokimia positif yang ditemukan pada otak dan batang otak serta hibridisasi in situ virus yang terlihat pada neuron dan sel glia materi abu-abu otak menunjukkan neurotropisme isolat setelah tahun 2002 (Sturm-Ramirez et al.
2004; Vascellari et al. 2007).
Meskipun rute masuknya virus ke dalam sistem saraf pusat belum dapat dipastikan, setidaknya dua hipotesis dapat menjelaskan. Hipotesis pertama yaitu transmisi virus dapat menjalar melalui serabut saraf vagus, olfaktorius, dan trigeminus, dan hipotesis kedua yaitu virus dapat melakukan penetrasi melewati
blood-brain barrier (Silvano et al. 1997; Park et al. 2002).
Ciri-ciri lain virus HPAI H5N1 pada itik adalah titer virus yang sering lebih tinggi pada usap orofaringeal dibandingkan usap kloaka (Sturm-Ramirez et al.
2004; Keawcharoen et al. 2008). Ekskresi virus HPAI H5N1 pada faring diduga berasal dari paru-paru dan/atau kantung hawa karena hanya kedua jaringan ini yang menunjukkan bukti replikasi virus secara imunohistokimia. Kecenderungan ekskresi pada faring ini menunjukkan bahwa usap faring juga harus diambil ketika melakukan surveilans VAI pada bebek liar selain usap kloaka yang selalu dilakukan (Keawcharoen et al. 2008). Jika tidak, prevalensi HPAI H5N1 dapat disalahperhitungkan.
kecenderungan risiko positif AI yang lebih tinggi (OR = 4,05) dibandingkan dengan yang tidak dicampur. Isolat virus HPAI H5N1 FKH/IPB/Duck/NG29 yang ditemukan pada bebek sehat dapat menginfeksi ayam yang berkontak sehingga pemeliharaan yang dicampur antara bebek dan ayam berpotensi meningkatkan
shedding virus dimana bebek berperan sebagai bank virus dan ayam sebagai media propagasi (FKH-IPB 2006).
Teknik Diagnostik Avian Influenza
Diagnosa AI dilakukan dengan isolasi virus atau melalui deteksi dan karakterisasi segmen genom virus karena gejala klinis yang ditimbulkan sangat beragam menurut spesies inang, strain virus, status kekebalan inang, keberadaan infeksi lain dan kondisi lingkungan (OIE 2009). Identifikasi VAI diawali dengan isolasi virus pada ruang alantois telur ayam berembrio (TAB) specific pathogen free (SPF). Selanjutnya cairan alantois diuji tapis dengan hemagglunation test
(HA) untuk mendeteksi keberadaan virus yang mampu mengaglutinasi sel darah merah, kemudian diuji dengan agar gel immunodiffusion test (AGID) atau enzyme linked immunosorbent assay (ELISA) yang masing-masing untuk mendeteksi tipe dan subtipe virus. Pengujian subtipe virus juga dapat dilakukan dengan
hemagglutination inhibition test (HI) dan neuraminidase inhibition test (NI). Alternatif lain untuk mendeteksi keberadaan VAI adalah dengan reverse-transcription polymerase chain reaction (RT-PCR) ataureal time RT-PCR (RRT-PCR) menggunakan primer spesifik matriks atau nukleoprotein. Selanjutnya subtipe virus ditentukan dengan menggunakan primer spesifik hemaglutinin dan neuraminidase. Uji serologis seperti AGID, HI, dan ELISA juga digunakan untuk mendeteksi antibodi dalam serum inang.
RRT-PCR untuk Deteksi Avian Influenza
Perkembangan teknologi yang pesat memberikan berbagai pilihan teknik dan produk yang dapat digunakan untuk mendukung pengujian diagnostik yang telah ada atau menjadi landasan untuk pengujian diagnostik yang baru. Teknik
real-time RT-PCR (RRT-PCR) dalam rangka pengawasan rutin, selama wabah, dan untuk penelitian karena lebih menguntungkan dari segi sensitivitas dan spesifisitas yang lebih tinggi, memerlukan waktu yang lebih singkat, bersifat kuantitatif, lebih ramah lingkungan, dan meskipun biaya yang diperlukan untuk investasi peralatan lebih tinggi namun untuk operasional dan pengamanan lingkungan teknik ini memerlukan biaya yang lebih sedikit.
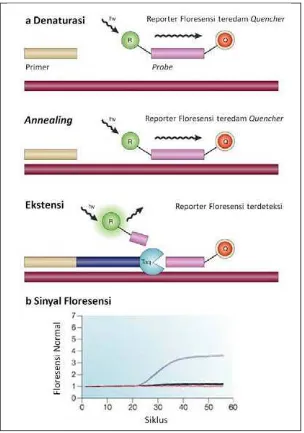
Pembacaan hasil RRT-PCR tidak memerlukan elektroforesis gel melainkan dapat dilihat secara langsung berupa grafik intensitas pendaran zat warna floresens yang meningkat secara eksponensial, linier, kemudian mendatar seiring siklus amplifikasi (Gambar 4). Hasil RRT-PCR berupa nilai Ct (cycle threshold) yang merupakan perpotongan antara kurva amplifikasi dengan garis threshold
yang menggambarkan konsentrasi relatif target PCR.Aktivitas nuklease ujung 5'- polimerase yang terdapat dalam polymerase chain reaction (PCR) memecah
probe hidrolisis saat ekstensi amplikon sehingga memisahkan reporter (R) florofor dari quencher (Q). Sinyal floresens yang dihasilkan ketika tereksitasi oleh cahaya dari luar di setiap siklus PCR sebanding dengan jumlah produk yang dihasilkan (Koch 2004).
Beberapa peningkatan yang penting demi perbaikan pengujian RRT-PCR telah tersedia untuk VAI, antara lain: pengembangan dan penggunaan kontrol internal untuk mengurangi negatif palsu reaksi dan pengembangan reagen kering beku (lyophilized) untuk meningkatkan kualitas kontrol (Das et al. 2006; Di Trani
et al. 2006); penggunaan robot untuk meningkatkan keluaran laboratorium agar mampu menangani peningkatan jumlah sampel selama wabah meskipun mungkin tidak memberikan sensitivitas yang lebih baik (Spackman et al. 2002; Spackman dan Suarez 2005); dan protokol baru untuk pengolahan sampel sulit seperti sampel kloaka atau jaringan (Das et al. 2006).
RT-PCR, dan urutan basa primer dan probe. Ketiga faktor tersebut harus diperhatikan agar pengujian menjadi sensitif dan spesifik.
Gambar 4. Prinsip probe hidrolisis TaqMan diadaptasi dari Koch (2004) (a) proses hidrolisis probe saat ekstensi memisahkan reporter floresensi dari quencher (b) sinyal floresensi meningkat secara eksponensial, linier, kemudian mendatar seiring siklus amplifikasi.
Ekstraksi RNA merupakan tahap yang penting dalam setiap uji diagnostik molekuler karena kualitas RNA akan mempengaruhi efisiensi amplifikasi. Berbagai teknologi ekstraksi RNA yang ada seperti ekstraksi organik, ekstraksi kolom silika, dan ekstraksi manik (beads) magnetik (Hale et al. 1996; Petrich et al.
Metode ekstraksi organik yang menggunakan fenol dan guanidinium memberikan efisiensi ekstraksi yang baik untuk sampel kompleks tetapi metode ini memungkinkan inhibitor PCR juga ikut terekstraksi sehingga dapat menyebabkan hasil negatif palsu (Das et al. 2006). Metode ekstraksi juga bervariasi dalam hal kemudahan penggunaan dan skalabilitas. Metode ekstraksi organik relatif memerlukan tenaga intensif dan sulit untuk pengerjaan sampel skala besar.
Beberapa metode ekstraksi berbasis kolom atau beads magnetik dapat digunakan untuk keluaran besar misalnya dengan pemrosesan pada plat 96 sumuran atau menggunakan robot. Banyak sistem robotik yang tersedia secara komersial dengan format reagen dan perlengkapan sendiri atau terbuka. Platform
kerja robotik juga sangat bervariasi dalam hal biaya tergantung pada kerumitan dan fitur mesin. Platform kerja robotik cukup menjanjikan untuk meningkatkan efisiensi laboratorium diagnostik, tetapi setiap robot dengan teknologi ekstraksi RNA masih memerlukan validasi sebelum dapat digunakan secara rutin pada sampel diagnostik. Kecil kemungkinan bagi sebuah mesin atau kit ekstraksi untuk dapat memiliki fleksibilitas dalam menangani berbagai jenis sampel yang dibawa ke laboratorium diagnostik dengan efisiensi ekstraksi RNA dan kemurnian yang diperlukan untuk memperoleh hasil yang konsisten (Aguero et al. 2007; Tewari et al. 2007). Diagnosis AI untuk ayam dan kalkun sebaiknya menggunakan sampel usap trakea atau orofaringeal karena tropisme virus pada kedua spesies tersebut adalah saluran pernafasan. Namun untuk spesies lain seperti itik direkomendasikan sampel usap kloaka karena pada spesies tersebut virus LPAI memiliki tropisme enterik. Sampel usap trakea/orofaringeal relatif mudah digunakan untuk ekstraksi RNA karena mengandung sedikit sekali sel. Sampel RNA lebih sulit diekstraksi dari usap kloaka dan jaringan karena keduanya mengandung bahan organik lebih tinggi, komposisi kimia yang kompleks, dan berpotensi mengandung inhibitor PCR (Cone et al. 1992; Buonagurio et al. 1999; Petrich et al. 2006).
dilengkapi dengan tahap reverse transkripsi dan amplifikasi PCR, biasanya dengan enzim yang berbeda untuk setiap tahapan. Kedua tahap tersebut sangat penting untuk pengujian diagnostik yang sensitif. Secara umum RT-PCR dapat dijalankan dengan prosedur dua tahap atau satu tahap (onestep). Pada prosedur dua tahap, reverse transkripsi RNA dan amplifikasi DNA dijalankan secara terpisah sehingga optimasi dilakukan di kedua reaksi. Sedangkan pada RT-PCR
onestep, semua reagen untuk tahapan reverse transkripsi RNA dan amplifikasi DNA dimasukkan kedalam tabung yang sama sehingga pengujian dapat selesai tanpa membuka tabung untuk memasukkan reagen tambahan. Prosedur dua tahap dianggap lebih sensitif daripada metode onestep karena kedua tahapan dilakukan pada kondisi yang optimal namun amplifikasi onestep menyederhanakan prosedur dan mengurangi kemungkinan kontaminasi silang sampel sehingga prosedur
onestepini lebih baik untuk berbagai situasi (OIE 2008b).
rendah, sensitivitas yang lebih tinggi, lebih mudah digunakan, meningkatkan keluaran, dan nilai tambah lain. APHIS telah menyetujui beberapa perubahan atau alternatif sejak protokol resmi disetujui pada tahun 2002 (Suarez et al. 2007).
Elemen kunci ketiga untuk pengujian diagnostik molekuler adalah desain primer dan probe yang menjadi landasan sensitivitas dan spesifisitas uji. Meskipun program-program untuk desain primer terus berkembang, sensitivitas pasangan primer tetap harus ditentukan secara empiris melalui optimasi konsentrasi primer dan probe, konsentrasi magnesium, dan kondisi siklus agar didapatkan sensitivitas tertinggi (OIE 2008b). Spesifisitas uji juga dapat dievaluasi secara in silico (menggunakan komputer) (Boutros dan Okey 2004) namun pengujian empiris tetap diperlukan untuk konfirmasi spesifisitas. Telah banyak pasangan primer AI untuk RT-PCR konvensional, namun baru beberapa yang telah dipublikasikan (Tabel 1). Variabilitas gen HA yang tinggi menyulitkan pengembangan primer dan probe yang dapat mendeteksi isolat beragam dalam satu subtipe HA, terutama untuk mengidentifikasi isolat dari garis keturunan Amerika dan Eurasia sehingga untuk wilayah geografis yang berbeda diperlukan pasangan primer dan probe berbeda (Spackman et al. 2002). Urutan basa primer dan probe menentukan spesifisitas dan sensitivitas uji diagnostik sehingga perubahan urutan basa memerlukan pengujian yang luas untuk validasi (OIE 2008b; OIE 2008a).
Pengujian RRT-PCR telah dikembangkan sebagai uji spesifik untuk influenza A dengan target regio lestari (conserved) pada matriks, nukleoprotein, atau gen nonstruktural lain yang informasi urutan basanya banyak tersedia. Beberapa pengujian spesifik untuk subtipe juga telah dipublikasikan dengan target gen HA untuk mendeteksi HPAI yang terbatas pada subtipe H5 atau H7 (Starick
et al. 2000; Munch et al. 2001; Collins et al. 2002; Spackman et al. 2002; Collins
(Spackman et al. 2002). Variabilitas HA menjadi salah satu alasan mengapa uji spesifik terhadap tipe A dengan target protein internal yang lebih lestari diperlukan sebagai uji tapis dalam pengujian molekuler AI dan pengujian subtipe HA berperan sebagai penyedia informasi tambahan dan konfirmasi sampel positif (Suarez et al. 2007).
Tabel 1. Pasangan primer dan probe untuk deteksi gen tertentu. Target Primer/ probe Urutan basa(5ƍ-3ƍ)
Amerika Utara dan Eurasia (Spackman et al. 2002)
Gen M1 M +25 AGATGAGTCTTCTAACCGAGGTCG
Mí124 TGCAAAAACATCTTCAAGTCTCTG
M +64 TCAGGCCCCCTCAAAGCCGA
Gen H5 (HA2) H5 +1456 ACGTATGACTAYCCRCARTAYTCA
H5í1685 AGACCAGCTACCATGATTGC H5 +1637 TCAACAGTGGCGAGTTCCCTAGCA Gen H7 (HA2) H7 +1244 ATTGGACACGAGACGCAATG
H7í1342 TTCTGAGTCCGCAAGATCTATTG H7 +1281 TAATGCTGAGCTGTTGGTGGCA Asia (Heine et al. 2005)
Gen M1 IVA-D161M AGATGAGYCTTCTAACCGAGGTCG IVA-D162M TGCAAANACATCYTCAAGTCTCTG
IVA-Ma TCAGGCCCCCTCAAAGCCGA
Gen H5 IVA-D148H5 AAACAGAGAGGAAATAAGTGGAGTAAAATT IVA-D149H5 AAAGATAGACCAGCTACCATGATTGC
IVA-H5a TCAACAGTGGCGAGTTCCCTAGCA Asia (Payungporn et al. 2006)
Gen M1 MF3 TGATCTTCTTGAAAATTTGCAG
MR1+ CCGTAGMAGGCCCTCTTTTCA
M-probe TTGTGGATTCTTGATCG
Gen H5 (HA2) H5F4 GACTCAAATGTCAAGAACCTTTA
H5R3 CCACTTATTTCCTCTCTGTTTAG
H5-probe ACGGAACGTATGACTAC
Gen N1 N1F2 GTTTGAGTCTGTTGCTTGGTC
N1R1 TGATAGTGTCTGTTATTATGCC
N1-probe TTGTATTTCAATACAGCCAC Qinghai (Hoffmann et al. 2007)
Gen H5 (situs pembelahan HA1 dan HA0)
FliH5-1028F GGGGAATGCCCCAAATATCT FliH5-1190R TCTACCATTCCCTGCCATCC
FliH5-CS-FAM AGAGAGAAGAAGAAAAAAGAGAGGACTA
FliH5-1148-HEX
TTGGAGCTATAGCAGGTTTTATAGAGG
Eurasia dan Afrika (Monne et al. 2008) AI virus
subtipe H5
H5-For TTATTCAACAGTGGCGAG
H5NE-Rev CCAG(T)AAAGATAGACCAGC H5probe CCCTAGCACTGGCAATCATG
1. M = A, C; R = A, G; Y = C, T
Pengujian gen matriks (M1) memiliki limit deteksi 10 fg atau sekitar 103 salinan gen dan dapat mendeteksi virus hingga 0,1 EID50(50% egg infective dose). Penelitian (Lee dan Suarez 2004) selanjutnya menunjukkan bahwa kuantitas RNA yang ditentukan dengan metode RRT-PCR berkorelasi erat dengan dan EID50 yang ditentukan dengan metode konvensional isolasi virus pada embrio ayam. Namun demikian, tingkat kesepakatan antara pengujian RRT-PCR matriks (MA RRT-PCR) dan isolasi virus (virus isolation, VI) pada embrio ayam tidak 100%. Positif RRT-PCR / negatif VI dan negatif RRT-PCR / positif VI pada sampel usap dapat terjadi (Spackman et al. 2002; Cattoli et al. 2004) sehingga hasil RRT-PCR sebaiknya diinterpretasikan pada tingkat kandang daripada tingkat individu. Berdasarkan analisis data wabah H7N2 LPAI di Virginia tahun 2002, sensitivitas diagnostik relatif RRT-PCR terhadap VI adalah 85,1% (probabilitas 95% interval: 71,9-95,7%), sedangkan spesifisitas diagnostik relatif terhadap VI mencapai 98,9% (probabilitas 95% interval: 98,0-99,5%) (Elvinger et al. 2007).
Pasangan primer dan probe spesifik untuk H5 dan H7 memiliki limit deteksi 100 fg target RNA atau sekitar 103-104 salinan gen dan dapat mendeteksi virus 10 EID50. Meskipun uji H5 yang telah ada terbukti mampu mendeteksi virus subtipe H5 Amerika Utara dan Eurasia, modifikasi untuk pengujian ini telah dilakukan untuk mengoptimalkan deteksi H5N1 Eurasia (Slomka et al. 2007b). Modifikasi primer forward dan reverse dilaporkan dapat meningkatkan sensitivitas analitik untuk virus H5N1 Eurasia hingga 1000 kali lipat (Heine et al. 2007).
Waktu dan Tempat
Penelitian dilakukan terhadap sampel yang dikoleksi selama tujuh bulan mulai September 2009 hingga Maret 2010 di Kabupaten Indramayu. Penelitian dilaksanakan di Laboratorium Virologi FKH IPB.
Bahan dan Alat
Media transport yang digunakan adalah brain heart infusion (BHI) dalam tabung eppendorf berukuran 2 ml. Virus (A/chicken/Indonesia/SmiWN18/2009); JF302895 digunakan sebagai kontrol positif. Sampel usap diambil menggunakan
cotton swab steril. Untuk isolasi genom virus digunakan MagMAX™ AI/ND Viral RNA Isolation kit dari Ambion®, plat ekstraksi 96 sumuran, dan magnetic stand. RT-PCR konvensional menggunakan SuperScript® III One Step RT-PCR with Platinum® Taq dari Invitrogen® dengan strip tabung PCR pada mesin BIO-RAD MJ Mini, sedangkan RRT-PCR dilakukan menggunakan Ag-Path ID™ One-Step RT-PCR kit dari Ambion® dengan plat optik 96 sumuran pada mesin Applied Biosystems 7500 Real Time PCR System. Peralatan dan bahan yang juga diperlukan selama penelitian antara lain mikropipet, tips mikropipet, titer plate shaker,vortex, alkohol, dan reservoir reagen.
Sampel
Sampel berasal dari sentinel dan non-sentinel yang ditempatkan di enam peternakan itik yang dibedakan berdasarkan cara itik memperoleh makanan, yaitu (tipe 1) peternakan itik angon; (tipe 2) peternakan itik dengan sistem angon dan pemberian pakan tambahan; (tipe 3) peternakan itik dengan pemberian pakan tanpa diangon dan hanya disediakan pekarangan untuk mencari makan tambahan. Pada masing-masing tipe, sentinel ditempatkan di dua peternakan dan bila terjadi kematian atau tanda terlepas maka sentinel diganti dengan itik yang ada di peternakan.
di enam peternakan (P1-P6). Sampel sentinel berasal dari bulan pengambilan 0-6 sedangkan sampel non-sentinel berasal dari bulan pengambilan 0 dan 6. Sehingga jumlah total sampel adalah 3240 sampel usap kloaka dan orofaring.
Sampel ditangani secara individual, dikemas, dan diberi kode berdasarkan jenis, asal, dan tanggal pengambilan sampel. Seluruh sampel ditransportasikan dalam rantai dingin (4-8°C) sebelum sampai ke laboratorium untuk dilakukan pengujian. Sampel usap disimpan pada kondisi deep freezer -80°C.
Metode
Pertama dilakukan pengujian untuk membandingkan PCR konvensional dan
real time dalam mendeteksi VAI H5. Kontrol positif H5 diencerkan secara serial dengan menambahkan sampel kedalam dH2O dengan perbandingan 1:1.
Sampel usap kloaka dan usap orofaringeal diuji keberadaan gen matriks dan VAI H5 dengan RRT-PCR. Sampel usap dipool di laboratorium sesuai jenis, peternakan asal, dan nomor individu dengan jumlah maksimal 5 sampel per pool
sebelum pengujian matriks (uji tapis influenza A). Sedangkan pengujian H5 dilakukan terhadap individu dalam pool yang positif influenza A. Semua proses penanganan sampel dilakukan dalam biological safety cabinet class II.
Pooling
Sampel usap dikelompokkan berdasarkan bulan pengambilan (0-6), asal peternakan, dan jenis (kloaka atau orofaring). Sampel usap dari tiap peternakan/bulan yang terdiri atas 30 individu (1-30) dikelompokkan menjadi 6
pool yang masing-masing terdiri atas 5 individu. Pool kloaka pertama terdiri atas sampel kloaka individu 1-5 yang masing-masing diambil 100 µl, pool kloaka kedua terdiri atas sampel kloaka individu 6-10 yang masing-masing diambil 100 µl, dan seterusnya. Setelah didapatkan pool, sampel individu kembali disimpan dalam deep freezer untuk pengujian H5 individu bila pool didapati positif influenza A. Uji tapis yang mendeteksi gen matriks dilakukan terhadap sampel
Isolasi RNA
Sebanyak 50 µl sampel dimasukkan kedalam 100 µl buffer lisis pada plat ekstraksi 96 sumuran kemudian ditambahkan larutan beadsmagnetik sebanyak 20 µl. Plat kemudian diagitasi selama 4 menit lalu didiamkan di atas magnetic stand
selama 2 menit untuk mengendapkan beads, setelah itu supernatan dibuang dengan cara disedot menggunakan mikropipet. Plat diturunkan dari magnetic stand kemudian ditambahkan 100 µl buffer pencuci I lalu diagitasi selama 30 detik dan didiamkan di atas magnetic stand selama 1 menit, setelah itu supernatan dibuang. Proses pencucian ini dilanjutkan menggunakan buffer pencuci II sebanyak dua kali setelah itu beads dikeringkan dengan cara agitasi selama 2 menit. Buffer elusi 50 µl ditambahkan untuk melarutkan RNA kemudian plat diagitasi selama 3 menit lalu didiamkan di atas magnetic stand selama 1 menit untuk mengendapkan beads, setelah itu supernatan (RNA) diambil dan dipindahkan kedalam plat 96 sumuran yang baru untuk nantinya digunakan sebagaitemplate PCR atau disimpan pada suhu -80°C hingga digunakan.
Untuk setiap proses isolasi RNA sampel disertakan satu kontrol positif (A/chicken/ Indonesia/SmiWN18/2009, accession number: JF302895) dengan nilai Ct 35 dan 1-2 kontrol negatif.
RT-PCR Konvensional
Campuran PCR disiapkan dalam di atas cold block dalam biosafety cabinet
(BSC). Secara berurutan reagen PCR dicampur kedalam tabung eppendorf kemudian disimpan pada -20oC hingga digunakan dengan urutan dan volume/reaksi sebagai berikut: RNAse-free dH2O 1,5 µl, buffer 2X 12,5 µl, primer forward (20ȝM) 1,0 µl, primer reverse (20ȝM) 1,0 µl, enzim mix 1,0 µl. Sekuens primer yang digunakan sebagai berikut: H5 forward (J3): 5’-GAT AAA TTC TAG CAT GCC ATT CC-3’ dan H5 reverse(B2a): 5’-TTT TGT CAA TGA TTG AGT TGA CCT TAT TGG-3’
1. Tahap 1 (1×): Reversetranskripsi 50oC 30 menit; denaturasi 94oC 4 menit. 2. Tahap 2 (35×): Denaturasi 94oC 45 detik; annealing 50oC 45 detik; ekstensi
72oC 2 menit.
Hasil PCR selanjutnya dielektroforesis menggunakan agarose 1% pada 120V selama 45 menit. Hasil elektroforesis dibaca di atas uv iluminator.
Real Time RT-PCR
Campuran PCR disiapkan dalam di atas cold block dalam biosafety cabinet
(BSC). Secara berurutan reagen PCR dicampur kedalam tabung eppendorf kemudian disimpan pada -20oC hingga digunakan dengan urutan dan volume/reaksi sebagai berikut: RNAse-free dH2O 1,08 µl, buffer 2X 12,50 µl, primer forward (20ȝM) 0,25 µl, primer reverse (20ȝM) 0,25 µl, enzim mix 25X 1,00 µl, probe (6ȝM) 0,25 µl, dan detection enhancer 1,67 µl.
Sekuens primer/probe yang digunakan sebagai berikut: matriks forward
(M+25): 5’-AGA TGA GTC TTC TAA CCG AGG TCG-3’; matriks reverse (M-124): 5’-TGC AAA AAC ATC TTC AAG TCT CTG-3’; probe AIV Matrix (M+64): 5’d FAM-TCA GGC CCC CTC AAA GCC GA-BHQ1-3’; H5 forward
(IVA-D148H5): 5’-AAA CAG AGA GGA AAT AAG TGG AGT AAA ATT-3’; H5reverse (IVA-D149H5): 5’-AAA GAT AGA CCA GCT ACC ATG ATT
GC-3’; probe IVA-H5a: 5’d FAM-TCA ACA GTG GCG AGT TCC CTA
GCA-BHQ1-3’.
Kedalam masing-masing sumuran plat optik 96 sumuran dimasukkan 17 µl campuran PCR kemudian ditambahkan 8 µl RNA template hasil isolasi. Plat ditutup dengan seal optik kemudian di tempatkan pada mesin Applied Biosystems 7500 Real Time PCR System dengan kondisi berbeda untuk PCR matriks dan H5. Untuk PCR matriks, mesin diatur agar siklus PCR berjalan sebagai berikut:
1. Tahap 1 (1×): Reverse transkripsi 45oC 10 menit; denaturasi 95oC 10 menit. 2. Tahap 2 (45×): Denaturasi 94oC 1 detik; annealing + ekstensi 60oC 30 detik. Sedangkan untuk PCR H5, mesin diatur agar siklus PCR berjalan sebagai berikut: 1. Tahap 1 (1×): Reversetranskripsi 45oC 10 menit; denaturasi 95oC 10 menit. 2. Tahap 2 (40×): Denaturasi 94oC 1 detik; annealing 57oC 30 detik; ekstensi
Untuk setiap run disertakan satu kontrol positif amplifikasi berupa RNA hasil isolasi dengan nilai Ct 25 dan satu kontrol negatif. Hasil PCR dianalisis menggunakan Applied Biosystems 7500 Real-Time PCR System software. Hasil dinyatakan layak bila diperoleh nilai Ct kontrol positif isolasi RNA 35, kontrol positif PCR 25, dan tidak satupun kontrol negatif memiliki nilai Ct (undetected). Sampel positif MA ditetapkan pada Ct 38 dan H5 36.
Analisis Data
RT-PCR Konvensional dan Real Time
Percobaan membandingkan RT-PCR konvensional dan real time dilakukan untuk mengetahui perbedaan sensitivitas kedua uji dalam mendeteksi VAI H5. Virus yang digunakan sebagai kontrol positif (A/chicken/Indonesia/SmiWN 18/2009; GenBank accession number: JF302895) diencerkan secara serial 1:1 kemudian diekstraksi dan dilakukan RT-PCR H5 secara konvensional maupun
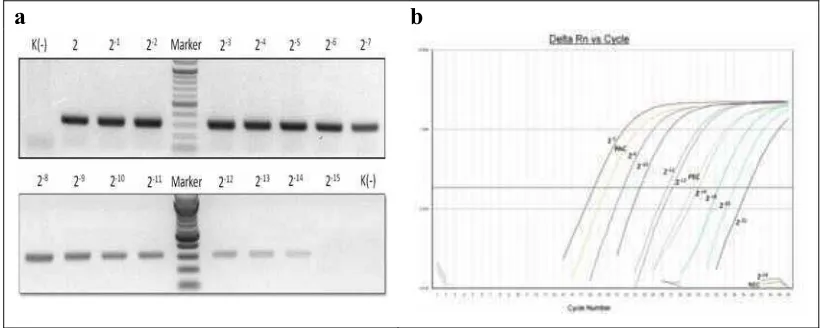
real time. Dengan RT-PCR konvensional, virus dapat terdeteksi hingga pengenceran 2-14 sedangkan menggunakan teknik real time, virus dapat terdeteksi hingga pengenceran 2-22 (Gambar 5). Perbedaan pengenceran hingga 28 ini menunjukkan bahwa RRT-PCR dapat mendeteksi sampel dengan konsentrasi hingga 250 kali lebih rendah dibandingkan RT-PCR konvensional. Lee dan Suarez (2004) menemukan bahwa RRT-PCR H5 memiliki limit deteksi 103-104 salinan gen atau 10 EID50 dan kuantitas RNA yang ditentukan dengan metode RRT-PCR berkorelasi erat dengan EID50 yang ditentukan dengan metode isolasi virus pada embrio ayam.
a b
Gambar 5. Perbandingan hasil PCR konvensional dan real time (a) elektroforesis gel RT-PCR konvensional, H5 terdeteksi hingga pengenceran 2-14 dan (b) grafik amplifikasi RRT-PCR hingga pengenceran 2-22.
volume satu atau dua kali run dilakukan untuk menghindari frezee-thaw. Untuk kontrol positif isolasi RNA, stok virus diencerkan 2-15 dan dibagi kedalam tabung-tabung berisi 100 µl sehingga dapat digunakan pada dua kali isolasi RNA.
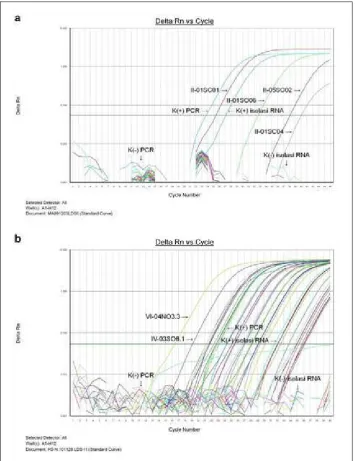
Gambar 6 menunjukkan hasil RRT-PCR MA sampel bulan kedua dan H5 berbagai bulan pengambilan sampel dengan kontrol positif dan negatif isolasi RNA dan PCR Teknik RT-PCR konvensional mendeteksi RNA setelah reaksi PCR selesai dan dilanjutkan dengan elektroforesis gel yang memerlukan waktu serta melibatkan bahan kimia berbahaya seperti etidium bromida dan sejenisnya. Sedangkan bahan kimia yang digunakan dalam RRT-PCR lebih aman dan memungkinkan deteksi dilakukan pada tahap awal reaksi sehingga dalam hal ini teknik real time lebih menguntungkan dibandingkan konvensional. Secara konvensional hasil yang diperoleh bersifat kualitatif sedangkan secara real time
hasil yang diperoleh merupakan konsentrasi relatif RNA target dalam bentuk nilai Ct dari perpotongan antara kurva amplifikasi dengan garis threshold.
Influenza A
Tingkat kesepakatan antara pengujian RRT-PCR matriks (MA RRT-PCR) untuk mendeteksi keberadaan virus influenza A dan isolasi virus pada embrio ayam tidak 100% sehingga PCR MA positif/isolasi virus negatif dan RRT-PCR MA negatif/isolasi virus positif pada sampel usap dapat terjadi (Spackman et al. 2002; Cattoli et al. 2004). Sensitivitas diagnostik relatif RRT-PCR terhadap isolasi virus adalah 85,1% dengan spesifisitas 98,9% (Elvinger et al. 2007) sehingga hasil RRT-PCR diinterpretasikan pada tingkat kandang/peternakan daripada tingkat individu.
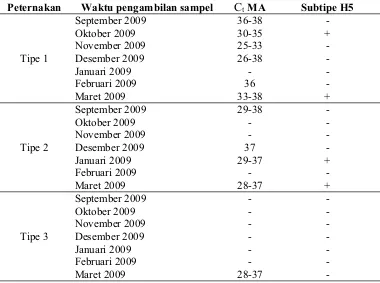
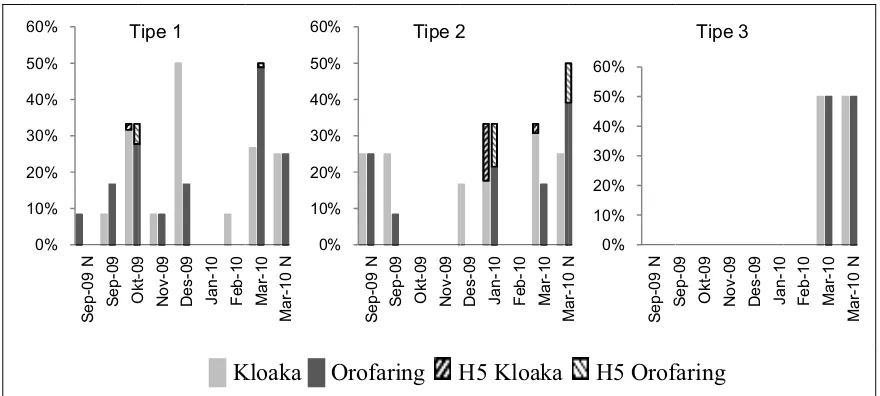
Dari 3.240 sampel yang diharapkan, berhasil dikoleksi 2.786 sampel karena faktor kematian atau itik yang tidak ditemukan saat pengambilan sampel. Virus influenza A dapat ditemukan di ketiga tipe peternakan, dan telah ada sejak awal pengambilan sampel di peternakan tipe 1 dan 2. Pada peternakan tipe 1, VAI terdeteksi hampir setiap bulan pengambilan sampel kecuali Januari (Tabel 2). Keberadaan VAI di peternakan tipe 2 terdeteksi saat screening di bulan September, Desember, Januari, dan Maret. Sedangkan pada peternakan tipe 3, VAI hanya ditemukan di akhir pengambilan sampel bulan Maret pada sampel usap kloaka dan orofaringeal itik sentinel maupun non sentinel.