SUPPORT
VECTOR MACHINE
(SVM)
MULYATI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul Identifikasi Ikan Tuna
dan Tenggiri Berdasarkan Sekuens DNA
Barcode
dengan Mengggunakan
Support
Vector Machine
(SVM). Pada penelitian ini
adalah benar karya saya dengan
arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada
perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya
yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam
teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut
Pertanian Bogor.
Bogor, September 2015
Mulyati
RINGKASAN
MULYATI. Identifikasi Ikan Tuna dan Tenggiri Berdasarkan Sekuens DNA
Barcode
dengan Menggunakan
Support Vector Machine
(SVM). Dibimbing oleh
WISNU ANANTA KUSUMA dan MALA NURILMALA.
Ikan tuna dan tenggiri merupakan salah satu produk perikanan yang banyak
diminati masyarakat dan memiliki nilai gizi yang baik bagi kesehatan. Banyak
dari produk olahan ikan tuna dan tenggiri ini telah mengalami pemalsuan,
yaitu
digantikannya kandungan produk yang memiliki nilai jual tinggi dengan
kandungan lain yang harganya lebih rendah. Untuk menjamin keamanan pangan,
pemalsuan ini harus dicegah, salah satunya dengan melakukan identifikasi
kandungan produk olahan tersebut
.
Identifikasi ini sangat penting untuk menjaga
standar kualitas pada industri makanan dan pasar.
Salah satu cara yang dilakukan untuk mengidentifikasi pemalsuan yaitu
dengan menggunakan metode berbasis DNA
barcode.
DNA
barcode
adalah
sekuen-sekuen pendek yang diambil dari bagian genom suatu makhluk hidup.
Identifikasi DNA
barcode
dilakukan dengan menggunakan pendekatan
komposisi, yaitu melakukan perhitungan jumlah frekuensi kemunculan pasangan
basa yang membentuk sekuens DNA
barcode
. Metode yang digunakan dalam
perhitungan frekuensi kemunculan pasangan basa ini adalah ekstraksi fitur
k-mers,
dengan
k
yang digunakan adalah 3-
mers
(
trinucleotide
) dan 4-
mers
(
tetranucletode
).
Hasil dari ekstraksi fitur menjadi vektor masukan untuk melakukan
identifikasi menggunakan
Support Vector Machine
(SVM) dengan kernel
Radial
Basis Function
(RBF)
.
Model yang terbentuk dari hasil identifikasi dengan SVM
dianalisis dengan menghitung nilai akurasi,
sensitivity
,
specificity
dan
Fmeasure.
Berdasarkan identifikasi dengan menggunakan SVM pada sekuens DNA
barcode
ikan tuna, tenggiri dan ikan lain hasil akurasi terbaik dengan
menggunakan
tetranucletide
pada tingkat genus yaitu sebesar 99,45% dan 88%
pada tingkat spesies.
SUMMARY
MULYATI. Identification of Tuna and Mackerel Fish Based on DNA Barcode
Sequences Using Support Vector Machine (SVM). Supervised by WISNU
ANANTA KUSUMA and MALA NURILMALA
Tuna and mackerel are some of the refined products that have great demand
in the community and contain good nutrients for health. Many of the refined
products have undergone fraudulent, by replacing the content of products that
have high sales value to other lower price one. For ensuring food safety,
fraudulent should be prevented by identifying the content of refined product. This
identification is very important to ensure the quality standards of the food industry
and markets.
One of the methods to identify fraudulent is using DNA barcode. DNA
barcode could be identified by using composition based approach, which calculate
the frequencies of substring or k-mers occurrences from the DNA barcode
sequences. The
k
values used in this research were k = 3 represented trinucleotide
(3-mers) and k = 4 represented tetranucleotide (4-mers).
The results of the feature extraction were the input vector for identification
by using Support Vector Machine (SVM) with kernel Radial Basis Function
(RBF). The model was evaluated by calculating accuracy, sensitivity, specificity,
and Fmeasure.
The evaluation results showed that the best accuracy was obtained by using
tetranucleotide with the value of 99.45% and 88% for the genus level and species
level, respectively.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan
atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan,
penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau
tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan
IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis ini
Tesis
Sebagai salah satu syarat untuk memperoleh gelar
Magister Komputer
pada
Program Studi Ilmu Komputer
IDENTIFIKASI IKAN TUNA DAN TENGGIRI BERDASARKAN
SEKUENS DNA
BARCODE
DENGAN MENGGUNAKAN
SUPPORT VECTOR MACHINE
(SVM)
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah SWT atas segala
karuniaNya sehingga karya ilmiah ini berhasil diselesaikan. Penelitian ini sudah
dikerjakan dari bulan Oktober 2013 dengan judul Identifikasi Ikan Tuna dan
Tenggiri Berdasarkan Sekuens DNA
Barcode
dengan Menggunakan
Support
Vector Machine
(SVM).
Terima kasih penulis ucapkan kepada Bapak DrEng Wisnu Ananta
Kusuma, ST MT dan Ibu Mala Nurilmala, SPi MSi selaku pembimbing yang telah
banyak memberi saran. Terima kasih juga kepada Ibu Dr Imas Sukaesih
Sitanggang, SSi MKom selaku penguji.
Ungkapan terima kasih juga disampaikan kepada ayah (Alm. Benlatief), ibu
(Siti Hawa), Kakak (Nurfadhli), Abang (Anwar Sadat, Syarkawi), dan adik
(Indriani, Rahmarani, Halimatussa’diah, Cut Husnul Fitri)
serta seluruh anggota
keluarga lainnya, atas doa dan kasih sayangnya. Selain itu ucapan terima kasih
juga kepada semua dosen dan staf Departemen Ilmu Komputer yang telah
membantu selama proses penelitian. Teman-teman sepembimbingan (Arini, Kana,
Yampi, Oci, Pak Saif), teman-teman kost (Grup FATIMA: Yuyun, Fuzy, Melly)
dan teman-teman seperjuangan angkatan 15 Ilmu Komputer IPB yang selalu
bersama penulis dua tahun ini, terima kasih atas dukungannya.
Semoga karya ilmiah ini bermanfaat.
Bogor, September 2015
DAFTAR TABEL
vi
DAFTAR GAMBAR
vi
1
PENDAHULUAN
1
Latar Belakang
1
Perumusan Masalah
3
Tujuan Penelitian
3
Manfaat Penelitian
4
Ruang Lingkup Penelitian
4
2
TINJAUAN PUSTAKA
4
DNA
Barcode
4
Support Vector Machine
(SVM)
5
3
METODE PENELITIAN
7
Data Penelitian
7
Ekstraksi Fitur
6
Normalisasi Data
8
Pelatihan SVM
8
Pengujian SVM
10
Evaluasi
10
4 HASIL DAN PEMBAHASAN
11
Ekstraksi Fitur
11
Normalisasi
11
Pelatihan dan Pengujian dengan SVM
12
Evaluasi
12
Pengujian dengan menggunakan BLAST
17
Kelebihan dan Kelemahan Sistem
17
5 SIMPULAN DAN SARAN
18
Simpulan
18
Saran
18
DAFTAR PUSTAKA 18
LAMPIRAN
21
DAFTAR TABEL
1
Data DNA barcode ikan tuna, tenggiri, dan ikan lain
7
2
Model klasifikasi biner dengan 3 kelas
10
3
Confusion Matrix
10
4
Nilai parameter terbaik untuk
gamma
dan
cost
12
5
Confusion matrix untuk genus dengan fekuensi trinucleotide
12
6
Confusion matrix untuk genus dengan fekuensi tetranucleotide
13
7
Confusion matrix untuk spesies dengan fekuensi trinucleotide
13
8
Confusion matrix untuk spesies dengan fekuensi tetranucleotide
13
9
Nilai
sensitivity
pada spesies ikan tuna, tenggiri dan ikan lain
14
10
Nilai
sensitivity
pada genus tuna, tenggiri dan ikan lain
15
11
Nilai
Specificity
pada spesies ikan tuna, tenggiri dan ikan lain
15
12
Nilai
specificity
pada genus ikan tuna, tenggiri dan ikan lain
15
13
Nilai
Precision
pada spesies tuna, tenggiri dan ikan lain
16
14
Nilai
Precision
pada genus tuna, tenggiri dan ikan lain
16
15
Nilai
Fmeasure
pada spesies ikan tuna, tenggiri dan ikan lain
16
16
Nilai
Fmeasure
pada genus tuna, tenggiri dan ikan lain
17
17
Tingkat kesamaan spesies tenggiri dengan spesies tuna
17
DAFTAR GAMBAR
1
Garis Pemisah terbaik yang memiliki margin terbesar
5
2
Flowchart kerangka pemikiran penelitian
7
3
Ekstraksi ciri
k-mers
dengan
k =
3
8
4
Contoh menggunakan 10
cross validation
9
5
Pembentukan matriks komposisi menggunakan trinucloetida
11
6
Hasil normalisasi dengan
trinucleotida
12
7
Perbandingan hasil akurasi pada tingkat genus dan spesies
14
DAFTAR LAMPIRAN
1
Daftar DNA barcode ikan tuna, tenggiri dan ikan lain
yang digunakan sebagai data latih
21
2
Daftar DNA barcode ikan tuna, tenggiri dan ikan lain
1.
PENDAHULUAN
Latar Belakang
Sektor perikanan merupakan salah satu sektor penting yang harus
dikembangkan guna meningkatkan daya saing dalam persaingan global. Agar
dapat bersaing secara kompetitif dan dapat memperluas pasar ekspor, diperlukan
kemampuan untuk mewujudkan produk perikanan yang memiliki nilai gizi dan
aman dikonsumsi. Adanya perdagangan bebas antar negara-negara ASEAN yang
akan diberlakukan pada Desember 2015 mendatang akan menyebabkan
banyaknya produk yang masuk dari berbagai negara, sehingga diperlukan
kemampuan untuk melacak (
traceability
) produk agar terjamin keamanannya.
Salah satu masalah dari jaminan keamanan adalah pada pemalsuan produk.
Pemalsuan yang dilakukan berupa substitusi daging ikan dengan spesies yang
mirip namun memiliki harga jual lebih rendah (Dudu
et al
. 2010; Abdullah
et al
.
2011). Efek negatif dari substitusi ini dapat merugikan konsumen dan juga
menyebabkan resiko gangguan kesehatan manusia. Salah satu produk perikanan
yang rawan pemalsuan dan sangat diminati masyarakat adalah produk olahan
berbahan dasar ikan khususnya ikan tuna dan tenggiri.
Ikan tuna memiliki nilai ekspor kedua tertinggi setelah udang. Menurut
Ditjen Perikanan dalam Badan Riset Kelautan dan Perikanan (BRKP) pada tahun
2013, sumbangan terbesar nilai ekspor hasil perikanan Indonesia yaitu dari
komoditi udang dan tuna, tongkol, cakalang (TTC) di mana masing-masing
menyumbang senilai US$997 juta dan US$515 juta. Adapun ikan tenggiri
merupakan ikan pelagis yang hidupnya menyebar hampir di seluruh perairan
Indonesia memiliki nilai ekonomis yang tinggi, di masa mendatang diperkirakan
permintaan komoditas ini baik dalam bentuk segar maupun olahan akan terus
mengalami peningkatan. Indikator yang menunjukkan hal tersebut adalah semakin
banyaknya diversifikasi produk olahan ikan. Hal ini diakibatkan semakin
meningkatnya permintaan terhadap sumberdaya tersebut (Sobari & Febriyanto
2010).
2
dalam waktu lama, komposisi kimianya dapat terdegradasi. Seiring
berkembangnya pengetahuan tentang identifikasi, sekarang ini banyak digunakan
identifikasi berbasis DNA
barcode
. Beberapa keunggulan identifikasi berbasis
DNA
barcode
menurut Virgilio
et al
. (2012) adalah memerlukan spesimen yang
sangat sedikit atau kecil, mampu mendokumentasikan keragamaan kelompok
taksonomi yang belum dikenal atau kelompok taksonomi yang berasal dari daerah
yang belum pernah teridentifikasi, dan juga mampu mengungkapkan variasi baru
atau keragaman baru pada spesies-spesies yang sebelumnya digolongkan pada
satu spesies saja.
Metode berbasis DNA
barcode
dapat juga mendeteksi keaslian suatu produk
bahan baku dalam bentuk segar yang akan dikemas sebagai produk olahan (Civera
2003) maupun produk yang sudah mengalami pemrosesan dan pembekuan
(Filonzi
et al
. 2010). Metode ini dapat juga diterapkan pada kandungan makanan
yang berbeda (Mafra
et al
. 2008). DNA
barcode
adalah s
ekuen-sekuen pendek
yang diambil dari bagian genom suatu organisme
(Hebert
et al
. 2003). Gen yang
digunakan sebagai penanda
barcode
ikan tuna dan tenggiri adalah dari gen
pengkode protein antara lain
Cytrochrome Oxidase
1 (CO1) dan
Cytochrome b
(
cyt
b) yang merupakan fragmen mitokondria.
Identifikasi berbasis DNA
barcode
dapat dilakukan melalui dua pendekatan
yaitu berdasarkan homologi dan berdasarkan komposisi (Pati
et al
. 2011).
Pendekatan homologi yaitu melakukan pencarian penjajaran sekuens yang
membandingkan fragmen DNA dengan sekuens referensi yang terdapat dalam
basis data yang digunakan, misalnya
National Center for Biotechnology
Information
(NCBI) dan
Barcode Of Life Database
(BOLD) dan hasilnya
disimpulkan pada tiap level taksonomi. Beberapa penelitian telah dilakukan
dengan pendekatan homologi antara lain Lin
et al.
(2005). Penelitian Lin
et al
ini
dilakukan terhadap 4 spesies tuna yaitu
Thunnus albacores, Thunnus thynnus
,
Thunnus alalunga,
dan
Thunnus obesus
dengan menggunakan
polymerase Chain
Reaction
(PCR). Hasilnya didapatkan bahwa 376 bp
cytochrome
b dari
Thunnus
obesus
terdapat perbedaan yang jelas yaitu 4.25% dibandingkan dengan 3 spesies
lainnya yaitu
Thunnus albacores, Thunnus thynnus
, dan
Thunnus alalunga
.
Lowenstein
et al
. (2009) juga melakukan penelitian pada identifikasi sushi tuna.
Hasil penelitiannya menunjukkan bahwa dengan berbasis karakter dan BLAST
metode yang diusulkan mampu mengidentifikasikan tuna sampai 100%.
Pendekatan lain yang dapat dilakukan adalah berdasarkan komposisi yaitu
melakukan perhitungan frekuensi ciri yang muncul dari pasangan basa yang
membentuk sekuens DNA. Pendekatan komposisi ini tidak perlu melakukan
penjajaran pada tiap sekuens DNA sehingga waktu yang diperlukan lebih cepat.
Pengekstraksian ciri dapat dilakukan dengan menggunakan metode perhitungan
frekuensi
k-mers
. Teknik ini
telah digunakan untuk mengekstraksi fitur DNA oleh
Karlin dan Burge pada tahun 1995. Sejak saat itu teknik tersebut digunakan secara
luas dengan panjang
k
yang berbeda-beda di mana semakin besar nilai
k
maka
hasil klasifikasi semakin akurat (McHardy 2007). Hasil dari ekstraksi fitur
selanjutnya akan menjadi vektor masukan untuk melakukan klasifikasi dan
identifikasi
.
Beberapa penelitian terkait yang telah dilakukan antara lain oleh
Weitschek
et al.
(2014) dengan menggunakan klasifikasi
supervised learning
(
Support Vector Machine
(SVM),
Naïve Bayes, RIPPER,
dan
C4.5). Hasil
yaitu sebesar 94.87% dibandingkan metode klasifikasi RIPPER dan C4.5. Seo
(2010) juga telah melakukan penelitian tentang klasifikasi sekuens
nucleotide
dengan menggunakan SVM, hasil yang diperoleh bahwa SVM berhasil
mengidentifikasikan lokasi pola spesifik pada spesies.
Berdasarkan latar belakang dan penelitian terkait maka dalam penelitian ini
identifikasi ikan tuna dan tenggiri berdasarkan sekuens DNA
barcode
dapat
dilakukan dengan mengunakan metode SVM sebagai
classifier
dan ekstraksi fitur
k-mers
sebagai pencirinya.
Perumusan Masalah
Ikan tuna dan tenggiri merupakan salah satu produk perikanan yang rawan
dilakukan pemalsuan baik dalam bentuk segar maupun olahan. Untuk
mengidentifikasi keaslian dari produk tersebut dapat dilakukan dengan
menggunakan beberapa cara yaitu berdasarkan ciri-ciri morfologi, komposisi
penyusun protein, dan berdasarkan DNA
barcode
. Namun identifikasi
berdasarkan ciri-ciri morfologi dan komponen penyusun protein hanya mampu
diidentifikasi pada tingkat taksonomi yang relatif tinggi dan tidak bisa
mengidentifikasi pada produk olahan maupun yang sudah mengalami pemanasan.
Oleh karena itu diperlukan suatu metode yang mampu mengidentifikasi sampai
tingkat relatif lebih rendah dan juga mampu mengidentifikasi pada produk yang
sudah diolah yaitu dengan menggunakan metode berbasis DNA
barcode
.
Identifikasi berbasis DNA
barcode
dilakukan dengan menggunakan dua
pendekatan yaitu pendekatan homologi dan pendekatan komposisi. Pendekatan
homologi memerlukan waktu yang lama karena melakukan perbandingan dan
pensejajaran pada setiap sekuens DNA. Adapun pendekatan komposisi yaitu
melakukan perhitungan frekuensi ciri yang muncul dari pasangan basa yang
membentuk sekuens DNA
barcode
dan tidak perlu melakukan pensejajaran pada
tiap sekuens DNA sehingga kompleksitas waktu yang diperlukan lebih cepat.
Perhitungan frekuensi ciri dapat dilakukan dengan menggunakan metode
perhitungan frekuensi
k-mers
, dengan
k
yang digunakan dalam penelitian ini
adalah 3
-mers
dan
4
-mers
Hasil dari pengekstraksi fitur dengan
k-mers
akan menjadi vektor masukan
untuk melakukan identifikasi. Menurut Weitschek
et al.
(2014) dan Seo (2010)
identifikasi dengan menggunakan
supervised learning
yaitu SVM memiliki
akurasi yang tinggi dan juga mampu mengidentifikasi pola spesifik pada spesies.
Oleh karena itu dalam penelitian ini identifikasi terhadap ikan tuna dan tenggiri
dilakukan dengan menggunakan metode klasifikasi
supervised learning
yaitu
Support Vector Machine
(SVM).
Adapun yang menjadi permasalahan dalam penelitian ini adalah bagaimana
melakukan identifikasi dengan menggunakan SVM sebagai
classifier
dan
menggunakan ekstraksi fitur
k
-
mers
sebagai pencirinya pada sekuens DNA
barcode
ikan tuna dan tenggiri.
Tujuan Penelitian
Tujuan dari penelitian ini adalah melakukan identifikasi ikan tuna dan
tenggiri berdasarkan sekuens DNA
barcode
dengan menggunakan SVM sebagai
4
Manfaat Penelitian
Manfaat dari penelitian ini adalah memudahkan pengidentifikasian terhadap
ikan tuna dan tenggiri dalam menghindari pemalsuan dengan ikan lainnya pada
produk segar maupun olahan.
Ruang Lingkup
Ruang lingkup dari penelitian ini adalah:
1.
Sekuens DNA
barcode
ikan yang digunakan adalah ikan tuna (
Thunnus
),
tenggiri (
Scomberomorus
) dan ikan lain seperti hiu (
Carcharhinus
), eskolar
(
Lepidocybium
), kakap (
Lutjanus
), ikan sapu (
Gadus
), dan ikan cod
(
Hypostomus
) yang diambil dari
Barcode Of Life Database
(BOLD).
2.
Panjang fragmen DNA
barcode
yang digunakan bervariasi (556-974 bp).
3.
Gen yang digunakan sebagai penanda
barcode
dari gen pengkode protein
yaitu
cytochrome oxidase
1 (CO1) dan
cytochrome
b (
cyt
b).
2.
TINJAUAN PUSTAKA
DNA
Barcode
DNA
barcode
sendiri
pertama kali menarik perhatian komunitas ilmuwan
pada tahun 2003 ketika kelompok penelitian Paul Hebert di Universitas Guelph
menerbitka
n sebuah makalah berjudul “i
dentifikasi biologis melalui DNA
barcode
”.
Di dalamnya, mereka mengusulkan sistem baru identifikasi spesies dan
penemuan menggunakan bagian pendek DNA dari daerah standar genom.
M
enurut Mitchel (2008) DNA
barcode
adalah urutan DNA pendek (± 500
pasangan basa) yang dapat digunakan untuk mengidentifikasi spesies. Tujuan
dari DNA
barcode
menurut Hollingsworth (2011) secara konseptual adalah
sederhana yaitu mencari satu atau beberapa daerah DNA yang akan membedakan
antara mayoritas spesies di dunia ini, dan melakukan pengurutan DNA dari
beragam sampel untuk menghasilkan referensi perpustakaan DNA skala besar di
dunia.
DNA
barcode
yang digunakan berasal dari DNA mitokondria. DNA
mitokondria mengandung sejumlah gen penting untuk respirasi dan fungsi
lainnya. Secara fisik mtDNA ini terpisah dari DNA lainnya, sehingga relatif lebih
mudah untuk mengisolasinya (berukuran relatif kecil yaitu hanya 16.000-20.000
pasang basa) dibandingkan jika harus mengisolasi milyaran nukleotida dari genom
inti (Wallace 1982).
Gen yang banyak digunakan sebagai penanda
barcode
adalah
Cytochrome c
oxidase
1 (CO1) dan
Cytochrome
b (
cyt
b) yang merupakan enzim mitokondria.
Support Vector Machine
(SVM)
SVM merupakan salah satu metode klasifikasi
supervised learning
dengan
konsep dasarnya adalah menemukan
hyperplane
(bidang pemisah) terbaik yang
dapat memisahkan data ke dalam 2 kelas dengan margin yang maksimal. Margin
adalah jarak antara garis
hyperplane
dengan anggota-anggota terdekat dari 2 kelas
(Gambar 1). SVM dapat melakukan klasifikasi data yang terpisah secara linier
(
linearly separable
) dan secara
non-
linier (
nonlinear separable
) (Burges 1998).
Menurut Osuna
et al.
(2007) suatu data yang dapat dipisahkan secara linear
disebut
linearly separable
. Misalkan himpunan
adalah dataset dan
yi
∈
{+1, -1} adalah label kelas dari data
xi
. Kondisi
linearly separable
terpenuhi
jika dapat dicari pasangan (
w, b
) sedemikian sehingga:
Gambar 1 Garis pemisah terbaik yang memiliki margin terbesar.
dengan
xi
adalah dataset
, w
adalah vektor bobot yang tegak lurus terhadap
hyperplane
(bidang normal) dan
b
adalah posisi fungsi pemisah relatif terhadap
pusat koordinat (titik asal). Bidang pemisah (
hyperplane
) terbaik adalah
hyperplane
yang terletak di tengah-tengah antara dua bidang pembatas kelas.
Untuk mendapatkan
hyperplane
terbaik dilakukan dengan memaksimalkan margin
atau jarak antara dua set objek dari kelas yang berbeda
.
Memaksimalkan nilai margin ekuivalen dengan meminimumkan nilai w. Margin
dapat dimaksimalkan dengan menggunakan fungsi optimisasi
lagrangian
seperti
berikut:
dengan
xi
adalah
support vector,
adalah jumlah
support vector
dan
xd
adalah
data yang akan diklasifikasikan.
Banyak kasus di lapangan yang penerapannya tidak dapat dipecahkan secara
linear, salah satu cara yang digunakan adalah dengan SVM
non linear.
Ide dasar
dari SVM
non linear
adalah memetakan data dari suatu bidang ke bidang yang
lebih tinggi dimensinya denga
n menggunakan fungsi kernel (Φ(x)). Data akan
dipetakan oleh fungsi Φ(x) ke ruang baru dengan dimensi lebih tinggi. Kemudian
SVM mencari
hyperplane
yang memisahkan kedua kelas secara linear di ruang
vektor yang baru tersebut. Pencarian
hyperplane
ini hanya bergantung pada
dot
(1)
(2)
6
product
dari data yang sudah dipetakan pada ruang baru yang berdimensi lebih
tinggi, Φ(
x
i).Φ(x
d). Perhitungan
dot product
diganti dengan fungsi
kernel.
sehingga fungsi keputusan adalah
Fungsi kernel yang umum digunakan adalah sebagai berikut:
1.
Gaussian radial basis function (RBF)
K (x
i,x
d) = exp (γ||x
i-x
d||
2)
dengan
γ
adalah parameter RBF
2.
Polinomial kernel,
K(x
i,y
d) =
pγ
merupakan parameter
slope
,
c
merupakan konstanta dan
p
merupakan
degree polynomial.
3.
Sigmoid Kernel
K(xi,xd) = tanh
γ
merupakan parameter
slope sigmoid
,
c
merupakan konstanta
.
Menurut Hsu
et al.
(2003) fungsi
kernel
yang direkomendasikan untuk diuji
pertama kali ialah fungsi kernel RBF karena memiliki performa yang sama
dengan SVM linear pada parameter tertentu dan memiliki perilaku seperti fungsi
kernel
sigmoid
dengan parameter tertentu dan rentang nilainya kecil [0,1].
Pada awalnya, SVM didesain untuk melakukan klasifikasi biner, yaitu
hanya dapat menangani data untuk dua kelas. Saat ini banyak riset yang
menggunakan lebih dari dua kelas, untuk mengembangkan SVM agar dapat
menangani kelas banyak, metode yang umum digunakan adalah
one-versus all
atau
metode one-againt-all
.
Metode ini, dibangun
k
buah model SVM biner (
k
adalah jumlah kelas). Setiap model klasifikasi ke-i dilatih dengan menggunakan
keseluruhan data, dengan label positif (kelas 1), sedangkan kelas lain sebagai label
negatif (kelas -1). Setelah dilakukan pelatihan, maka didapatkan
k
fungsi
keputusan dari
k
model tersebut. Misalkan ada data baru
x
yang diuji dengan
model ini maka
x
akan masuk ke dalam kelas yang memiliki nilai keputusan
terbesar.
Metode
multi class
lainnya yang digunakan adalah
one-versus one
atau
metode
one-againt-one
. Dengan menggunakan metode ini, dibangun
k(k-1)/2
buah model klasifikasi biner (
k
adalah jumlah kelas).
Setelah model klasifikasi
selesai dibangun, selanjutnya suatu data uji akan diklasifikasikan ke dalam kelas
yang paling banyak menang (Hsu & Lin 2002).
(5)
(6)
(7)
(4)
3.
METODE PENELITIAN
Metode pada penelitian ini terdiri atas beberapa tahapan proses, yaitu
pengumpulan data dari BOLD, melakukan ekstraksi fitur dengan perhitungan
frekuensi
k-mers
, normalisasi, pengklasifikasian dengan menggunakan SVM, dan
evaluasi. Proses penelitian digambarkan dalam bagan kerangka pemikiran yang
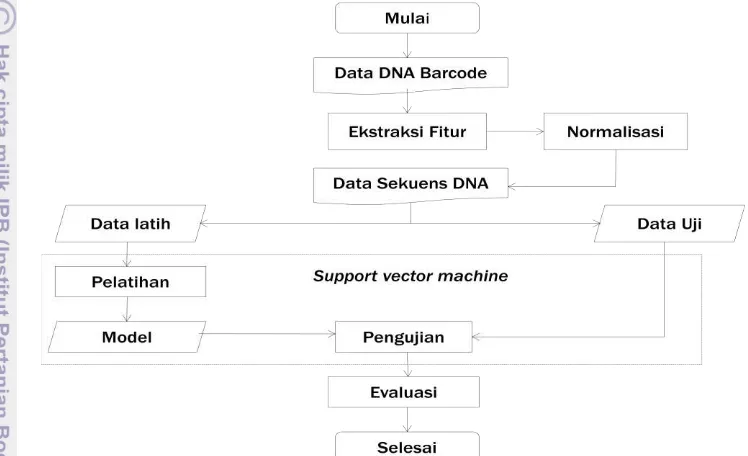
ditampilkan pada Gambar 2.
Gambar 2 Flowchart kerangka pemikiran penelitian
Data Penelitian
Data penelitian yang digunakan adalah DNA
barcode
ikan tuna, tenggiri
dan ikan lain (Tabel 1). DNA
barcode
ini diambil dari gen pengkode protein yaitu
Cytochrome Oxidase
1 (CO1) dan
Cytochrome
b (
cyt
b) yang merupakan fragmen
dari mitokondria. Data DNA
barcode
tersebut diperoleh dari BOLD
(
http:boldsystems.org
). BOLD adalah sebuah
workbench
informatika yang
membantu perolehan, penyimpanan, analisis dan publikasi record DNA
barcode
(Sujeevan 2007). Data DNA
barcode
ini direpresentasikan sebagai
string
dengan
formatnya berbentuk FASTA.
Tabel 1 Data Sekuens DNA
barcode
ikan tuna, tenggiri dan ikan lain
Kelas Genus Spesies
Banyaknya DNA barcode
Rata-rata panjang
DNA barcode (bp)
Keterangan
A Thunnus
T. albacares T. atlanticus T. thynnus T. alalunga T. tonggol T. orientalis
91 28 75 70 27 14
695 777 647 675 831 691
8
Kelas Genus Spesies
Banyaknya DNA barcode Rata-rata panjang DNA barcode (bp) Keterangan T. maccoyii T. obesus 16 88 752 679
B Scomberomorus
S. commerson S. niphonius S. regalis S. cavalla S. maculatus S. munroi S. brasiliensis S. semifasciatus 44 39 18 16 14 7 18 6 621 704 681 745 929 746 682 770 Tenggiri C
Carcharhinus C. limbatus C. obscures 45 38 673 669 Ikan Lain Lepidocybium L. flavobrunneum 24 699
Lutjanus L. analis L. campechanus
29 10
651 653 Gadus G. macrocephalus 45 706 Hypostomus H. plecostomus 3 658
Ekstraksi Fitur
Data latih dan data uji yang akan digunakan terlebih dahulu dilakukan
ekstraksi fitur. Metode ekstraksi fitur yang digunakan adalah perhitungan
frekuensi
k-mers
. Ekstraksi dengan frekuensi
k-mers
akan membentuk komposisi
sesuai dengan banyaknya data yang digunakan. Pola kemunculan
k
dalam sekuens
dihitung menggunakan empat basa utama (A,C,G, dan T) dipangkatkan dengan
rangkaian pasangan basa yang ingin digunakan (Pola kemunculan: 4^
k
, dengan
k
≥1)
(Kusuma 2012). Pada penelitian ini
k
yang digunakan adalah
trinucleotide
(3-mers
) dan
tetranucleotide
(4-
mers
). Ilustrasi perhitungan frekuensi pola
kemunculan dengan ekstraksi fitur
k-mers
dapat dilihat pada Gambar 3.
Gambar 3 Ekstraksi fitur
k-mers
dengan menggunakan 3
-mers
Normalisasi Data
Data yang digunakan memiliki panjang sekuens yang bervariasi sehingga
perlu dilakukan normalisasi. Normalisasi ini bertujuan untuk mendapatkan data
dengan nilai yang lebih kecil yang mewakili data asli tanpa kehilangan
karakteristik sendirinya (Han
et al
. 2012), di mana rentang nilai data yang
digunakan berkisar antara 0 dan 1. Rumus dari normalisasi yang digunakan yaitu:
A A A G A A C
dengan k = 3
3-
mers
Matrik Komposisi
Normalisasi= (nilai x) / (panjang sekuens)
dengan x adalah banyaknya frekuensi kemunculan
k-mers
.
Pelatihan SVM
Data latih yang sudah diekstraksi dan normalisasi selanjutnya dilakukan
pelatihan dengan SVM. SVM akan mencari model terbaik yang dapat
memisahkan kelas. SVM yang digunakan untuk bahasa pemrograman R tersedia
pada library e1071 (Meyer
et al.
2014). Pelatihan ini menggunakan fungsi
kernel
yaitu
Gaussian radial basis function
(RBF). Menurut Hsu
et al.
(2003) fungsi
kernel
yang direkomendasikan untuk diuji pertama kali ialah fungsi kernel RBF
karena memiliki performa yang sama dengan SVM linear pada parameter tertentu
dan memiliki perilaku seperti fungsi kernel
sigmoid
pada parameter tertentu
dengan rentang nilainya kecil yaitu [0,1].
Optimasi parameter dan pada kernel RBF menggunakan
grid search
dengan 10
fold
cross validation
(Gambar 4)
pada rentang 10
-6-10
-1untuk
parameter dan 10
-1-10
2untuk parameter .
Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train Train Train Train Train Train Train Train Train Train Test Train
Train Train Train Train Train Train Train Train Train Test
Gambar 4 Contoh menggunakan 10
fold cross validation
Pada Gambar 4 di atas adalah contoh menggunakan 10
fold cross
validation
, dataset dibagi sebanyak 10
fold
dengan melakukan iterasi sejumlah 10
kali untuk data latih dan data uji. Pada iterasi pertama, subset satu menjadi data
penguji sedangkan subset lainnya menjadi data pelatih. Pada iterasi kedua, subset
kedua digunakan sebagai data penguji dan subset lainnya menjadi data pelatih,
dan seterusnya hingga seluruh subset digunakan sebagai data penguji. Untuk
mendapatkan nilai akurasi ataupun ukuran penilaian lainnya dari hasil eksperimen
yang dilakukan, dapat diperoleh dari nilai rataan dari keseluruhan eksperimen
tersebut. Keuntungan
k-fold cross validation
adalah semua data digunakan baik
untuk data uji maupun data latih. Hal ini dilakukan untuk mendapatkan nilai
akurasi ataupun ukuran penilaian lainnya dari hasil eksperimen yang dilakukan
(Han
et al
. 2012).
Pada pelatihan ini menggunakan metode
multi class
SVM yaitu
one against
one
karena menggunakan 3 kelas yaitu kelas tuna, tenggiri, dan ikan lain. Dengan
menggunakan metode tersebut, terbentuk 3 buah model klasifikasi biner (Tabel 2)
Setiap model klasifikasi dilatih pada data dari dua kelas.
Fold 8
Fold 7 Fold 9 Fold 2 Fold 3 Fold 4
Fold 1
(9)
10
Tabel 2 Model klasifikasi biner dengan 3 kelas
y1=1 y2 = -1 Hipotesis
Kelas 1 Kelas 2 f 12 (x) = (w12)x + b12 Kelas 1 Kelas 3 f 13 (x) = (w13)x + b13 Kelas 2 Kelas 3 f 23 (x) = (w23)x + b23
Jika data x dimasukkan ke dalam fungsi hasil pelatihan
f
ij(x)=
(
w
ij)x
+
b
ij(i j
adalah indeks kelas) dan hasilnya
menyatakan x adalah kelas i, maka suara
untuk kelas i ditambah satu. Kelas dari
data x akan ditentukan dari jumlah suara
terbanyak. Jika terdapat dua buah kelas yang jumlah
suaranya sama, maka kelas
yang indeksnya lebih kecil dinyatakan sebagai kelas dari data tersebut (Shu & Lin
2002) .
Pengujian SVM
Model yang didapatkan dari hasil pelatihan sudah diuji dengan
menggunakan data uji yang diunduh dari BOLD dan juga dari Laboratorium
Fakultas Perikanan dan Ilmu Kelautan (FPIK) Institut Pertanian Bogor (IPB).
Pengujian dilakukan untuk mengidentifikasikan data uji ke dalam kelas ikan tuna,
tenggiri ataupun ikan lain.
Evaluasi
Berdasarkan hasil pelatihan dan pengujian SVM, didapatkan hasil yang
selanjutnya digunakan untuk mengevaluasi kinerja SVM. Pada tahap evaluasi
akan dihitung akurasi,
sensitivity, specificity
, dan
Fmeasure
berdasarkan tabel
confusion matrix
(Tabel 3). Tabel
confusion matrix
diperlukan untuk menentukan
kinerja suatu model klasifikasi (Tan
et al.
2005).
Tabel 3
Confusion matrix
Prediksi Kelas
Kelas Sebenarnya
Posittive Negative
Posittive A: True Positive B: False negative Negative C: False positive D: True negative keterangan:
True positive (TP) : jumlah data positif yang benar diklasifikasi oleh classifier. True negative (TN) : jumlah data negatif yang benar diklasifikasi oleh classifier. False positive (FP) : jumlah data negatif yang salah diklasifikasi sebagai data positif. False negative (FN): jumlah data positif yang salah diklasifikasi sebagai data negatif.
Berdasarkan tabel
confusion matrix
di atas maka akurasi,
sensitivity,
specificity
, dan
Fmeasure
dapat dihitung dengan persamaan berikut:
(10)
(13)
(11)
(14)
4.
HASIL DAN PEMBAHASAN
Penelitian ini menggunakan data sebanyak 765 sekuens DNA
barcode
yang
terbagi dalam 3 kelas, masing-masing kelas memiliki data yang tidak seimbang
yaitu 409 sekuens DNA tuna, 162 sekuens DNA tenggiri dan 194 sekuens DNA
ikan lain. Untuk data pengujian, data yang digunakan sebanyak 145 sekuens DNA
tuna yang terdiri dari 4 spesies yaitu
Thunnus alalunga
,
Thunnus albacores
,
Thunnus obesus
, dan
Thunnus thynnus.
4 sekuens DNA tenggiri dari spesies
Scomberomorus commerson
, dan 32 sekuens DNA ikan lain yang terdiri dari
spesies
Carcharhinus limbatus, Gadus macrocephalus,
dan
Hypostomus
plecostomus
.
Ekstraksi Fitur
Data sekuens DNA
barcode
ikan tuna, tenggiri dan ikan lainnya terlebih
dahulu dilakukan ekstraksi fitur dengan frekuensi
k-mers
untuk membentuk
matriks komposisi yang akan menjadi vektor masukan dalam proses identifikasi.
Frekuensi
k-mers
yang digunakan dalam penelitian ini adalah
trinucleotide
(3-mers
) dan
tetranucleotide
(4-
mers
). Banyaknya data yang diekstraksi adalah 765
sekuens DNA
barcode
untuk data latih dan 145 sekuens DNA
barcode
untuk data
uji. Matriks komposisi yang terbentuk dari data latih adalah 175 x 64 untuk
trinucleotide
dan 175 x 256 untuk
tetranucleotide
, Adapun untuk data uji matrik
komposisi yang terbentuk adalah 145 x 64 untuk
trinucletide
dan 145 x 256 untuk
tetranucleotide
. Gambar 5 adalah contoh hasil ekstraksi fitur dengan
menggunakan
trinucloetide
.
Gambar 5 Pembentukan matriks komposisi menggunakan trinucleotide
Normalisasi
Data sekuens DNA
barcode
ikan yang digunakan memiliki panjang yang
bervariasi sehingga perlu dinormalisasi. Normalisasi dilakukan dengan cara
membagi banyaknya frekuensi yang muncul dari hasil ekstraksi
k-mers
dengan
Data sekuens DNA
AAA AAC AAT … GGG
…
…
… … … … …
…
12
panjang data masing-masing sekuens. Contoh hasil dari normalisasi dengan
trinucleotide
ditunjukkan pada Gambar 6.
Gambar 6 Hasil normalisasi dengan
trinucleotide
Pelatihan dan Pengujian dengan SVM
Data latih (Lampiran 1) yang digunakan dalam penelitian ini adalah
sebanyak 765 sekuens DNA
barcode
ikan. Data latih ini selanjutnya dilakukan
pelatihan dengan SVM. SVM yang digunakan berupa
library
e1071 pada bahasa
Pemrograman R dengan fungsi kernel
radial basis function
(RBF) dan
C-classification
. Optimasi parameter dan pada kernel RBF menggunakan
grid
search
dengan 10
fold
cross validation
pada rentang 10
-6–
10
-1untuk parameter
dan 10
-1–
10
2untuk parameter menghasilkan parameter terbaik seperti yang
terlihat pada Tabel 4:
Tabel 4 Nilai parameter terbaik untuk
gamma
dan
cost
Parameter Trinucleotide (3-mers) Tetranucleotide (4-mers) Data 1 Data 2 Data 1 Data 2
Cost 100 100 10 100
Gamma 0.0001 0.01 0.001 0.001
Keterangan:
Data 1: Data sekuens DNA yang diambil dari BOLD (http:boldsystems.org) berdasarkan tingkat genus.
Data 2: Data sekuens DNA yang diambil dari BOLD (http:boldsystems.org) berdasarkan tingkat spesies.
Parameter terbaik yang diperoleh akan menjadi masukan pada pembentukan
model SVM. Selanjutnya model dilakukan pengujian dengan menggunakan data
uji (Lampiran 2) sebanyak 145 sekuens DNA
barcode
ikan.
Evaluasi
Penggunaan
confusion matrix
digunakan untuk mengevaluasi hasil
identifikasi pada data dengan menggunakan frekuensi
trinucleotide
dan
tetranucleotide
pada tingkat genus (Tabel 5 dan Tabel 6) maupun pada tingkat
spesies (Tabel 7 dan Tabel 8).
Tabel 5
Confusion matrix
untuk genus dengan frekuensi
trinucleotide
Kelas prediksi Kelas Sebenarnya
Tuna Tenggiri Ikan lain
Tuna 144 4 0
Tenggiri 1 0 10
ikan lain 0 0 22
Data hasil ekstraksi fitur
AAA AAC AAT … GGG
…
…
… … … … …
…
AAA AAC AAT … GGG
. . . … .
. . . … .
… … … … …
. . . … .
Tabel 6
Confusion matrix
untuk genus dengan frekuensi
tetranucleotide
Kelas prediksi Kelas Sebenarnya
Tuna Tenggiri Ikan lain
Tuna 145 1 0
Tenggiri 0 3 0
ikan lain 0 0 32
Tabel 7
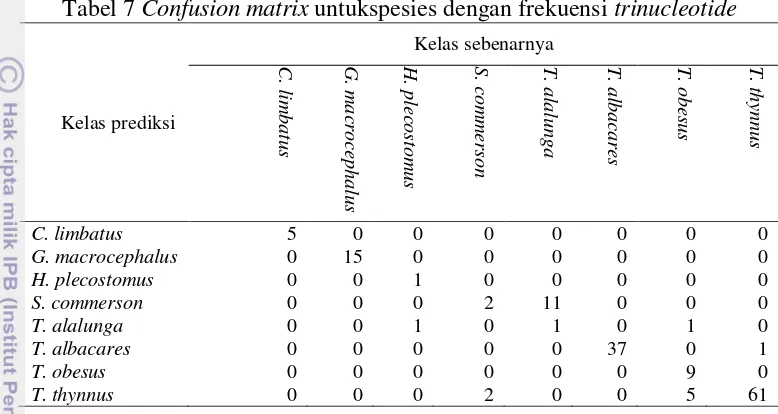
Confusion matrix
untukspesies dengan frekuensi
trinucleotide
Tabel 8
Confusion matrix
untuk spesies dengan frekuensi
tetranucleotide
Berdasarkan
confusion matrix
pada Tabel 5, 6, 7 dan 8 maka akurasi yang
diperoleh untuk data dengan menggunakan frekuensi
tetranucleotide
lebih tinggi
dibandingkan frekuensi
trinucleotide
(Gambar 7). Hal ini menunjukkan bahwa
pola kemunculan
k
pada ektraksi fitur mempengaruhi akurasi, karena semakin
besar nilai
k
maka semakin banyak fitur atau ciri yang terbentuk, informasi yang
diperoleh pun semakin banyak sehingga menyebabkan akurasi tinggi. Begitu juga
untuk data yang diidentifikasi berdasarkan tingkat genus juga lebih tinggi
akurasinya dibandingkan dengan data yang diidentifikasi berdasarkan tingkat
spesies.
Kelas prediksi Kelas sebenarnya C . limb a tu s G. ma cro ce p h a lu s H. p leco sto mu s S . co mme rs o n T. a la lu n g a T. a lb a ca res T. o b esu s T. th yn n u sC. limbatus 5 0 0 0 0 0 0 0
G. macrocephalus 0 15 0 0 0 0 0 0
H. plecostomus 0 0 1 0 0 0 0 0
S. commerson 0 0 0 2 11 0 0 0
T. alalunga 0 0 1 0 1 0 1 0
T. albacares 0 0 0 0 0 37 0 1
T. obesus 0 0 0 0 0 0 9 0
T. thynnus 0 0 0 2 0 0 5 61
Kelas prediksi Kelas sebenarnya C. li mb a tu s G. ma cro ce p h a lu s H. p lec o sto m u s S . c o mm ers o n T . a la lu n g a T . a lb a ca re s T . o b esu s T . th yn n u s
C. limbatus 5 0 0 0 0 0 0 0
G. macrocephalus 0 15 0 0 0 0 0 0
H. plecostomus 0 0 1 0 0 0 0 0
S. commerson 0 0 0 0 0 0 0 0
T. alalunga 0 0 1 1 11 0 1 0
T. albacares 0 0 0 0 0 47 0 0
T. obesus 0 0 0 0 0 0 9 0
14
Gambar 7 Perbandingan hasil akurasi pada tingkat genus dan spesies
Matriks yang digunakan untuk mengukur kemampuan SVM dalam
mengidentifikasi sekuens DNA
barcode
pada tiga kelas secara terpisah yaitu
sensitivity
dan
specificity
.
Sensitivity
adalah perbandingan ikan yang benar yang
berhasil diidentifikasi terhadap jumlah total ikan yang sebenarnya.
Specificity
menyatakan perbandingan ikan yang salah yang berhasil diidentifikasi terhadap
jumlah total ikan yang salah. Sementara nilai
precision
dan
recall
digunakan
untuk mengukur kemampuan SVM dalam mengidentifikasikan satu kelas saja,
dalam hal ini kelas ikan tuna, kelas tenggiri, atau kelas ikan lainnya saja.
Fmeasure
adalah matriks yang mengintegrasikan
precision
dan
recall
(Yen & Lee
2009). Tabel 9, 10, 11, 12, 13, 14, 15 dan 16 menunjukkan
performance
SVM
dalam mengidentifikasi data sekuens DNA untuk data ikan tuna, tenggiri dan ikan
lainnya.
Tabel 9 Nilai
sensitivity
pada spesies ikan tuna, tenggiri dan ikan lain
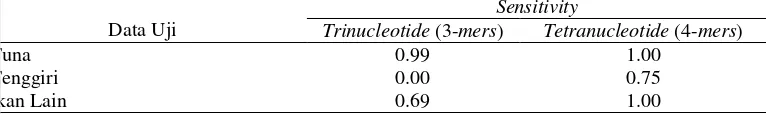
Pada Tabel 9, nilai
sensitivity
untuk tiap spesies dengan ekstraksi fitur
menggunakan
trinucleotide
dan
tetranucleotide
. Nilai rata-rata
sensitivity
yang
diperoleh untuk
trinucleotide
adalah 0.828, yang berarti bahwa setidaknya 82.8%
spesies ikan berhasil diidentifikasi ke kelas sebenarnya. Adapun dengan
menggunakan
tetranucleotide
rata-rata nilai
sensitivity
adalah 0.89, yaitu
sebanyak 89% spesies ikan dapat diidentifikasi ke kelas sebenarnya. Namun untuk
0 10 20 30 40 50 60 70 80 90 100
Genus Spesies
Pers
e
n
ta
se
Trinucleotide (3-mers)
Tetranucleotide (4-mers)
Data Uji
Sensitivity
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Carcharhinus limbatus 1.00 1.00
Gadus macrocephalus 1.00 1.00
Hypostomus plecostomus 0.50 0.50
Scomberomorus commerson 0.00 0.00
Thunnus alalunga 0.92 1.00
Thunnus albacares 0.80 1.00
Thunnus obesus 0.60 0.75
spesies
Scomberomorus commerson
memiliki nilai
sensitivity
yang paling rendah
yaitu 0, yang berarti tidak ada satupun spesies tersebut teridentifikasi ke dalam
kelas sebenarnya, hal ini disebabkan oleh ketidakseimbangan jumlah spesies
tersebut dengan spesies lainnya.
Tabel 10 Nilai
sensitivity
pada genus tuna, tenggiri dan ikan lain
Tabel 10 menunjukkan nilai
sensitivity
untuk genus tuna, tenggiri, dan
ikan lain. Nilai rata-rata
sensitivity
yang diperoleh pada ikan tuna dan ikan lain
untuk frekuensi
trinucleotide
adalah 0.84 yaitu sebanyak 84% ikan dapat
diidentifikasi ke kelas sebenarnya. Adapun pada pada tenggiri nilai
sensitivity
adalah 0, yang berarti ikan tenggiri tidak bisa diidentifikasi ke kelas sebenarnya.
Pada frekuensi
tetranucleotide
nilai rata-rata
sensitivity
yang diperoleh pada genus
ikan tuna, tenggiri dan ikan lain adalah 0.916, yang berarti 91.6% genus ikan
tersebut teridentifikasi ke dalam kelas sebenarnya.
Tabel 11 Nilai
Specificity
pada spesies ikan tuna, tenggiri dan ikan lain
Pada Tabel 11, nilai
specificity
untuk tiap spesies dengan ekstraksi fitur
menggunakan
trinucleotide
dan
tetranucleotide
. Nilai rata-rata
specificity
yang
diperoleh untuk
trinucleotide
adalah 0.977, yang berarti bahwa setidaknya 2.3%
spesies ikan teridentifikasi ke kelas yang bukan sebenarnya. Adapun dengan
menggunakan
tetranucleotide
rata-rata nilai
specificity
adalah 0.99, yaitu
sebanyak 1% spesies ikan yang teridentifikasi ke kelas yang bukan sebenarnya.
Tabel 12 Nilai
specificity
pada genus ikan tuna, tenggiri dan ikan lain
Tabel 12 menunjukkan nilai
specificity
untuk genus tuna, tenggiri, dan
ikan lain. Nilai rata-rata
specificity
yang diperoleh pada frekuensi
trinucleotide
adalah 0.93 yaitu sebanyak 7% ikan tidak mampu diidentifikasi ke kelas
sebenarnya. Adapun pada frekuensi
tetranucleotide
nilai rata-rata
specificity
yang
Data Uji
Sensitivity
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Tuna 0.99 1.00
Tenggiri 0.00 0.75
Ikan Lain 0.69 1.00
Data Uji Specificity
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Carcharhinus limbatus 1.00 1.00
Gadus macrocephalus 1.00 1.00
Hypostomus plecostomus 1.00 1.00
Scomberomorus commerson 1.00 1.00
Thunnus alalunga 0.96 0.98
Thunnus albacores 0.98 1.00
Thunnus obesus 0.96 1.00
Thunnus thynnus 0.92 0.96
Data Uji
Specificity
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Tuna 0.85 0.97
Tenggiri 0.94 1.00
16
diperoleh adalah 0.99, yang berarti hanya 1% ikan tersebut tidak mampu
teridentifikasi ke dalam kelas sebenarnya.
Tabel 13 Nilai
Precision
pada spesies tuna, tenggiri dan ikan lain
Pada Tabel 13, nilai
precision
untuk tiap spesies dengan ekstraksi fitur
menggunakan
trinucleotide
dan
tetranucleotide.
Nilai rata-rata
precision
yang
diperoleh untuk
trinucleotide
adalah 0.877, yang berarti bahwa setidaknya 87.7%
model dapat mengidentifikasi dengan tepat ke kelas sebenarnya. Adapun dengan
menggunakan
tetranucleotide
rata-rata nilai
precision
adalah 0.96, artinya model
dapat mengidentifikasi dengan tepat ke kelas sebenarnya sebesar 96%. Nilai
precision
terendah dimiliki oleh spesies
Scomberomorus commerson
yaitu bernilai
0.
Tabel 14 Nilai
Precision
pada genus tuna, tenggiri dan ikan lain
Tabel 14 menunjukkan nilai
precision
untuk genus tuna, tenggiri, dan ikan
lain. Nilai rata-rata
precision
yang diperoleh pada frekuensi
trinucleotide
adalah
0.985 yaitu sebanyak 98.5% model mampu mengidentifikasi ke kelas sebenarnya
pada genus ikan tuna dan ikan lain. Adapun pada tenggiri model tidak mampu
mengidentifikasi ke dalam kelas tenggiri. Pada frekuensi
tetranucleotide
nilai
rata-rata
precision
yang diperoleh adalah 0.916, yang berarti sebesar 91.6% model
mampu mengidentifikasi ke dalam kelas sebenarnya baik pada ikan tuna, tenggiri,
maupun ikan lain.
Tabel 15 Nilai
Fmeasure
pada spesies ikan tuna, tenggiri dan ikan lain
Data Uji Precision
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Carcharhinus limbatus 1.00 1.00
Gadus macrocephalus 1.00 1.00
Hypostomus plecostomus 1.00 1.00
Scomberomorus commerson 0.00 0.00
Thunnus alalunga 0.69 0.79
Thunnus albacores 0.95 1.00
Thunnus obesus 0.60 1.00
Thunnus thynnus 0.90 0.94
Data Uji
Precision
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Tuna 0.97 1.00
Tenggiri 0.00 0.75
Ikan Lain 1.00 1.00
Data Uji
Fmeasure
Trinucleotide (3-mers) Tetranucleotide (4-mers)
Carcharhinus limbatus 1.00 1.00
Gadus macrocephalus 1.00 1.00
Hypostomus plecostomus 0.67 0.67
Scomberomorus commerson 0.00 0.00
Thunnus alalunga 0.79 0.88
Thunnus albacores 0.87 1.00
Thunnus obesus 0.60 0.86
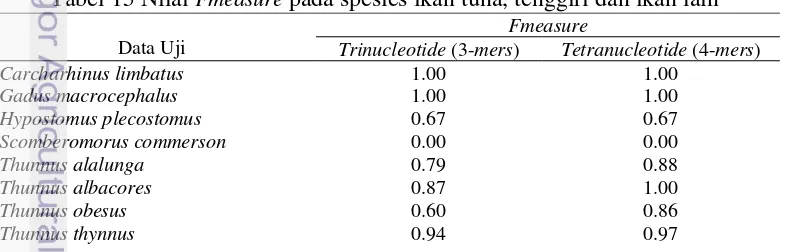
Pada Tabel 15, nilai
Fmeasure
untuk tiap spesies dengan ekstraksi fitur
menggunakan
trinucleotide
dan
tetranucleotide
. Nilai rata-rata
Fmeasure
yang
diperoleh untuk
trinucleotide
adalah 0.838, yang berarti bahwa setidaknya 83.8%
model dapat mengidentifikasi dengan tepat ke kelas sebenarnya. Adapun dengan
menggunakan
tetranucleotide
rata-rata nilai
Fmeasure
adalah 0.91, artinya model
dapat mengidentifikasi dengan tepat ke kelas sebenarnya sebesar 91%. Namun
nilai
Fmeasure
paling rendah juga dimiliki oleh spesies
Scomberomorus
commerson
yaitu sebesar 0.
Tabel 16 Nilai
Fmeasure
pada genus tuna, tenggiri dan ikan lain
Tabel 16 menunjukkan nilai
Fmeasure
untuk genus tuna, tenggiri, dan ikan
lain. Nilai rata-rata
specificity
yang diperoleh pada frekuensi
trinucleotide
adalah
0.89 yaitu sebanyak 89% model mampu mengidentifikasi dengan tepat ke kelas
sebenarnya pada genus ikan tuna dan ikan lain. Adapun pada tenggiri model tidak
mampu mengidentifikasi dengan tepat ke dalam kelas tenggiri. Pada frekuensi
tetranucleotide
nilai rata-rata
Fmeasure
yang diperoleh adalah 0.95, yang berarti
sebesar 95% model mampu mengidentifikasi dengan tepat ke dalam kelas
sebenarnya baik pada ikan tuna, tenggiri, maupun ikan lain.
Pengujian dengan Menggunakan BLAST
Spesies tenggiri yaitu
Scomberomorus commerson
memiliki nilai
sensitivity
dan
Fmeasure
yang rendah dan cenderung teridentifikasi ke dalam spesies tuna,
oleh karena itu pada penelitian ini juga menggunakan aplikasi
Basic Local
Alignment Search Tools
(BLAST) untuk melihat berapa persen tingkat
similarity
spesies tenggiri dengan spesies tuna.
Tabel 17 Tingkat kesamaan spesies tenggiri dengan spesies tuna
Data uji Spesies yang diduga SimilarityScomberomorus Commerson Thunnus Obesus 84% Thunnus Alalunga 84%
Hasil pengujian diperoleh berdasarkan Tabel 17 bahwa spesies dari
Scomberomorus commerson
memiliki kesamaan yang tinggi dengan spesies tuna
yaitu sebesar 84%. Hal ini yang menyebabkan spesies ikan tenggiri cenderung
teridentifikasi ke dalam spesies tuna yaitu
Thunnus obesus
dan
Thunnus alalunga
.
Kelebihan dan Kelemahan Sistem
Adapun kelebihan dari sistem identifikasian dengan SVM pada penelitian
ini adalah tidak membutuhkan memori yang banyak dalam melakukan identifikasi
karena dalam melakukan pengujian
hanya menggunakan
support vector
(data yang
berada di perbatasan antar kelas) yang mempengaruhi fungsi keputusan hasil
pengujian
. SVM juga memiliki kompleksitas yang linear sehingga waktu yang
Data Uji
Fmeasure
Trinucleotide (3-mers) Tetranucleotide (4-mers)
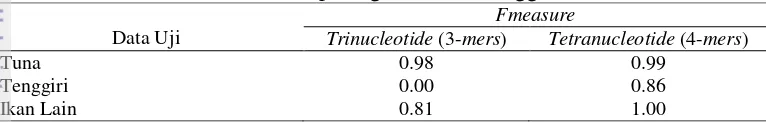
Tuna 0.98 0.99
Tenggiri 0.00 0.86
18
diperlukan lebih efisien. Adapun kekurangan dari sistem SVM ini adalah pada
data yang tidak seimbang tidak mampu diidentifikasi dengan baik sehingga
apabila ada data uji maka akan cenderung teridentifikasi ke dalam kelas yang
mayoritas.
5.
SIMPULAN DAN SARAN
Simpulan
Berdasarkan hasil yang diperoleh dari penelitian yang telah dilakukan,
metode
klasifikasi
dengan
menggunakan
model
SVM
berhasil
mengidentifikasikan sekuens DNA
barcode
untuk spesies ikan tuna dan spesies
ikan lain dengan baik, namun untuk spesies ikan tenggiri model tidak mampu
mengidentifikasi dengan baik. Nilai akurasi yang diperoleh untuk data dengan
menggunakan frekuensi
tetranucleotide
pada tingkat genus maupun spesies lebih
tinggi dibandingkan dengan menggunakan frekuensi
trinucleotide
yaitu sebesar
99.45% untuk genus dan 88% untuk spesies. Hal ini menunjukkan bahwa pola
kemunculan
k
pada ekstraksi ciri mempengaruhi akurasi, karena semakin besar
nilai
k
semakin banyak fitur yang terbentuk sehingga nilai akurasi juga semakin
tinggi.
Saran
Data yang digunakan pada penelitian ini tidak seimbang sehingga
mempengaruhi kinerja SVM, oleh karena itu untuk penelitian selanjutnya
diperlukan suatu metode untuk menyeimbangkan data misalnya dengan
menggunakan
undersampling
ataupun
oversampling
sehingga
dapat
meningkatkan akurasi dalam proses identifikasinya.
DAFTAR PUSTAKA
Abdullah A, Nurjanah, Kurnia N. 2011. Autentikasi tuna steak komersial dengan
metode PCR-Sequencing.
Jurnal Pengolahan Hasil Perikanan Indonesia
.
16(61) : 1-7.
[BRKP] Badan Riset Kelautan dan Perikanan. 2013.
Potret dan Strategi
Pengembangan Perikanan
Tuna, Udang dan Rumput Laut
. Jakarta: Badan
Riset Kelautan dan Perikanan.
Burges JC. 1998. A Toturial on Support Vector Machines for Pattern Recognition
.
Data Mining and Knowledge Discovery.
2: 955- 974.
Civera T. 2003. Species Identication and safety of fish products
. Vet Research
Communication.
27: 481.
Filonzi L, Stefania C, Marina V, Francesco NM. 2010. Molecular barcoding
reveals mislabelling of commercial fish products in Italy.
Food Research
International.
43: 1383-1388.
Han J, Kamber M, Pei J. 2012. Data Mining: Concepts and Techniques. 3
thed.
New York (US): Morgan Kaufman Elsevier Academic Pr.
Hebert PDN, Cywinska A, Ball SL, Dewaard JR. 2003.
Biological identification
through DNA barcodes.
Proc. R. Soc. Lond. B
. 270: 313-321.
Hollingsworth PM. 2011. Refining the DNA barcode for land plants.
PNAS
108(49): 19451-19452.
Hsu CW, Chang CC, Lin CJ. 2003. A practical guide to support vector
classification. Department of Computer Science and Information
Engineering (TW): National Taiwan University.
Hsu
CW,
Lin
CJ. 2002. A Comparison of methods for multi-class support vector
machines
. IEEE Transactions on Neural Networks
. 13:415-425.
Karlin S, Burge C. 1995. Dinucleotide relative abundance extremes: A genomic
signature.
Trends Genet.
11: 283
–
290.
Kusuma WA, 2012. Combined approaches for improving the performance of
denovo DNA sequence assembly and metagenomic classification of shorts
fragment from next generation sequencer [disertasi]. Tokyo(JP): Tokyo
Institut Of Technology.
Lin WF, Shiau CY, Hwang DF. 2005. Identification of four
thunnus
tuna speciesc
using mitochondrial cytochrome b gene sequence and PCR-RFLP Analysis.
Journal of food and drug analysis
. 13(4): 382-387.
Lowenstein JH, Amato G, Kolokotronis SO. 2009. The real maccoyii: identifying
tuna sushi with DNA barcodes-contrasting characteristic attributes and
genetic distances
. PloS ONE
4(11): e7866.
Mafra I, Ferreira IMPVO, Oliveira MBPPO. 2008. Food authentication by
PCR-based methods
. European Food Research and Technology
. 227: 649
–
665.
Matarese AC, Spies IB, Busby MS, dan Orr JW. 2011.
Early larvae of Zesticelus
Profundorum (Family Cottidae) Identified Using DNA Barcoding
. Ichthyol
Res 58:170
–
174.
McHardy AC, Martín HG, Tsirigos A, Hugenholtz P, Rigoutsos I. 2007. Accurate
phylogonetic classification of variabel-length DNA fragments
. Nature
Methods
. 4(1):63-72. doi: 10.1038/nmeth976.
Meyer D. 2014. e1071: Misc functions of the department of statistics, TU Wien. R
package
version
1.6-3.
Tersedia
pada
http://CRAN.R-project.org/package=e1071.
Mitchell A. 2008. DNA barcoding demystified.
Australia Journal Entomol
. 47:
169-173.
Myers MJ. 2011. Molecular identification of animal speciesin food: transition
from research laboratories to the regulatory laboratories.
Veterinary Journal
.
190: 7-8.
Osuna EE, Freund R, Girosi F. 1997. Support vector machines: training and
applications.
AI Memo.
1602.
20
Seo TK. 2010. Classification of nucleotide sequences using support vector
machines.
Journal of molecular evolution
. 71(4): 250-67.
Sobari MP, Febriyanto A. 2010. Kajian Bio-teknik pemanfaatan sumberdaya ikan
tenggiri dan distribusi pemasarannya di kabupaten Bangka.
Meritex
.
1(10):15-29.
Sujeevan R, Hebert PD. 2007. Bold: the barcode of life data system.
Mol Ecol
7(3): 355-364. doi: 10.1111/j.1471-8286.2007.01678.x.
Teresita MP, Joel FG, Shadi S, Donald JB, Brian G, Mehrdad H. 2013.
Rapid and
Accurate Taxonomic Classification of insect (class insect) Cytochrome c
Oxidase Sub Unit 1 (CO1) DNA Barcode Sequences Using a Naïve Bayes
Classifier.
Departement of Biology
, 1280, Main street Hamilton, on
Canada. Doi: 10.1111/1755-0998.12240.
Virgilio M, Jordaens K, Breman F, et al. 2012. Turning DNA barcodes into an
alternative tool for identification: African fruit flies as a model (Poster).
Consortium for the Barcode of Life
(CBOL).
Wallace. 1982. Structure and evolution of organelle genomes.
J Mikrobiologi
.
46(2):208-240 .
Weitschek E, Fiscon G, Felici G. 2014. Supervised DNA barcodes species
classification: analysis, comparison, and results.
BMC Bio Data Mining
. 7:
4.doi: 10.1186/1756-0381-7-4.
Lampiran 1 Daftar DNA barcode ikan tuna, tenggiri dan ikan lain yang digunakan
sebagai data latih
Genus Jenis DNA Barcode
Thunnus ANGBF7098-12|Thunnus albacares|COI-5P|HM007768 GBGC3261-07|Thunnus albacares|COI-5P|DQ835954 GBGC3263-07|Thunnus albacares|COI-5P|DQ835952 GBGC3265-07|Thunnus albacares|COI-5P|DQ835949 GBGC3267-07|Thunnus albacares|COI-5P|DQ835946 GBGC4264-08|Thunnus albacares|COI-5P|EU392206 GBGC7871-09|Thunnus albacares|COI-5P|DQ835956 GBGCA1334-13|Thunnus albacares|COI-5P|GU451793 GBGCA1346-13|Thunnus albacares|COI-5P|GU451781 GBGCA1348-13|Thunnus albacares|COI-5P|GU451779 GBGCA1366-13|Thunnus albacares|COI-5P|GU451761 GBGCA2127-13|Thunnus albacares|COI-5P|HM007771 GBGCA540-10|Thunnus albacares|COI-5P|GU324199 GBGCA541-10|Thunnus albacares|COI-5P|GU324198 GBGCA545-10|Thunnus albacares|COI-5P|GU324194 GBGCA682-10|Thunnus albacares|COI-5P|FJ605791 GBGCA685-10|Thunnus albacares|COI-5P|FJ605788 GBGCA688-10|Thunnus albacares|COI-5P|FJ605785 HIDNA034-14|Thunnus albacares|COI-5P
RFE248-05|Thunnus albacares|COI-5P|EU752224 SCFAC184-05|Thunnus albacares|COI-5P|KC015954 SCFAC280-06|Thunnus albacares|COI-5P|KC015956 SCFAC292-06|Thunnus albacares|COI-5P|KC015958 WLIND460-07|Thunnus albacares|COI-5P
ANGBF6823-12|Thunnus albacares|COI-5P|JN644293 ANGBF6833-12|Thunnus albacares|COI-5P|JN644300 ANGBF9217-12|Thunnus albacares|COI-5P|HM452166 ANGBF9228-12|Thunnus albacares|COI-5P|HM452165 CYTC1219-12|Thunnus albacares|COI-5P|JN086153 CYTC1219-12|Thunnus albacares|CYTB
FOA872-04|Thunnus albacares|COI-5P|DQ107651 FOA873-04|Thunnus albacares|COI-5P|DQ107652 GBGC3254-07|Thunnus albacares|COI-5P|DQ835947 GBGC3260-07|Thunnus albacares|COI-5P|DQ835955 GBGC7870-09|Thunnus albacares|COI-5P|DQ835957 GBGCA1345-13|Thunnus albacares|COI-5P|GU451782 GBGCA1361-13|Thunnus albacares|COI-5P|GU451766 GBGCA1367-13|Thunnus albacares|COI-5P|GU451760 GBGCA177-10|Thunnus albacares|COI-5P|NC_014061 GBGCA546-10|Thunnus albacares|COI-5P|GU324193 SAFC039-11|Thunnus albacares|COI-5P
SAFC040-11|Thunnus albacares|COI-5P
22
Genus Jenis DNA Barcode
Thunnus SAFC037-11|Thunnus albacares|COI-5P
SCFAC003-05|Thunnus albacares|COI-5P|KC015955 TUNA004-09|Thunnus albacares|COI-5P|GQ199630 WLIND459-07|Thunnus albacares|COI-5P|EF609628 BLOM114-14|Thunnus albacares|COI-5P
FOA869-04|Thunnus albacares|COI-5P|DQ107648 FOA870-04|Thunnus albacares|COI-5P|DQ107649 FOA871-04|Thunnus albacares|COI-5P|DQ107650 RFE250-05|Thunnus albacares|COI-5P|EU752225 TZMSB245-04|Thunnus albacares|COI-5P|DQ885060 TZMSB246-04|Thunnus albacares|COI-5P|DQ885058 TZMSB247-04|Thunnus albacares|COI-5P|DQ885059 TZMSC141-05|Thunnus albacares|COI-5P|DQ885062 TZMSC142-05|Thunnus albacares|COI-5P|DQ885061 BCIMS046-13|Thunnus albacares|COI-5P
BCIMS047-13|Thunnus albacares|COI-5P BCIMS048-13|Thunnus albacares|COI-5P
GBGC3253-07|Thunnus albacares|COI-5P|DQ835950 GBGC3266-07|Thunnus albacares|COI-5P|DQ835948 GBGC3268-07|Thunnus albacares|COI-5P|DQ835945 GBGCA1338-13|Thunnus albacares|COI-5P|GU451789 GBGCA1341-13|Thunnus albacares|COI-5P|GU451786 GBGCA1370-13|Thunnus albacares|COI-5P|GU451757 GBGCA1371-13|Thunnus albacares|COI-5P|GU451756 GBGCA695-10|Thunnus albacares|COI-5P|FJ605778 GBGCA705-10|Thunnus albacares|COI-5P|FJ605768 SAFC038-11|Thunnus albacares|COI-5P
SCFAC246-06|Thunnus albacares|COI-5P|KC015957 WLIND457-07|Thunnus albacares|COI-5P|EF609629 WLIND461-07|Thunnus albacares|COI-5P|EF609627 BCIMS045-13|Thunnus albacares|COI-5P
GBGCA1342-13|Thunnus albacares|COI-5P|GU451785 GBGCA1347-13|Thunnus albacares|COI-5P|GU451780 GBGCA1351-13|Thunnus albacares|COI-5P|GU451776 GBGCA1352-13|Thunnus albacares|COI-5P|GU451775 GBGCA1372-13|Thunnus albacares|COI-5P|GU451755 GBGCA2128-13|Thunnus albacares|COI-5P|HM007770 ANGBF1319-12|Thunnus alalunga|COI-5P|HQ167713 ANGBF7188-12|Thunnus alalunga|COI-5P|HM007773 CYTC1217-12|Thunnus alalunga|CYTB
Genus Jenis DNA Barcode Thunnus GBGC3386-07|Thunnus alalunga|COI-5P|DQ835824
GBGC3388-07|Thunnus alalunga|COI-5P|DQ835822 GBGC3390-07|Thunnus alalunga|COI-5P|DQ835820 GBGC3391-07|Thunnus alalunga|COI-5P|DQ835819 RFE233-05|Thunnus alalunga|COI-5P|EU752221 SAFC033-11|Thunnus alalunga|COI-5P|
ANGBF7873-12|Thunnus alalunga|COI-5P|JN007752 ANGBF7915-12|Thunnus alalunga|COI-5P|JN007761 ANGBF7916-12|Thunnus alalunga|COI-5P|JN007759 ANGBF7917-12|Thunnus alalunga|COI-5P|JN007757 ANGBF7918-12|Thunnus alalunga|COI-5P|JN007755 ANGBF7919-12|Thunnus alalunga|COI-5P|JN007753 BCIMS043-13|Thunnus alalunga|COI-5P|
BCIMS044-13|Thunnus alalunga|COI-5P| DNATR1704-13|Thunnus alalunga|COI-5P| FOA866-04|Thunnus alalunga|COI-5P|
FOA867-04|Thunnus alalunga|COI-5P|DQ107646 FOA868-04|Thunnus alalunga|COI-5P|DQ107647 GBGC0052-06|Thunnus alalunga|COI-5P|AB101291 GBGC1668-06|Thunnus alalunga|COI-5P|NC_005317 GBGC3387-07|Thunnus alalunga|COI-5P|DQ835823 GBGC3389-07|Thunnus alalunga|COI-5P|DQ835821 GBGC3392-07|Thunnus alalunga|COI-5P|DQ835818 GBGCA2125-13|Thunnus alalunga|COI-5