FILOGEOGRAFI KERANG HIJAU (
Perna viridis
) DI
INDONESIA DAN KAITANNYA DENGAN JALUR LINTAS
PELAYARAN
AYU DIAH PITALOKA
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa skripsi berjudul Filogeografi Kerang Hijau (Perna viridis) di Indonesia dan Kaitannya dengan Jalur Lintas Pelayaran adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Agustus 2015
Ayu Diah Pitaloka
ABSTRAK
AYU DIAH PITALOKA. Filogeografi Kerang Hijau (Perna viridis) di Indonesia dan Kaitannya dengan Jalur Lintas Pelayaran. Dibimbing oleh HAWIS MADDUPPA dan BEGINER SUBHAN.
Kerang hijau (Perna viridis) berpotensi untuk menyebar luas secara geografis melalui penyebaran larva atau air ballast kapal, yang berpotensi invasif di area geografis lainnya. Tujuan dari penelitian ini adalah untuk mengetahui konektivitas dan keragaman genetik spesies kerang hijau (Perna viridis) di Indonesia dan kaitannya dengan jalur lintas pelayaran, menggunakan marka mitokondria lokus Cytochrom oxidase 1 (CO1). Jarak genetik terjauh terdapat antara populasi Makassar dan Muara Kamal (D = 0.008), dan jarak genetik terdekat antara populasi Teluk Lada dan Ambon serta antara populasi Teluk Lada dan Palabuhan Ratu (D = 0.002). Differensiasi genetik terkecil terdapat antara Makassar dan Palabuhan Ratu (Fst = 0.05). Differensiasi genetik terbesar terbesar terdapat antara Ambon dan Palabuhan Ratu (Fst = 0.90) dan antara Ambon dan Teluk Lada (Fst = 0.90). Keragaman Genetik ditunjukkan oleh keragaman haplotipe yang tinggi. Keragaman haplotipe tertinggi terdapat pada populasi dari Muara Kamal (Hd = 0.27), diikuti populasi dari Palabuhan Ratu dan Teluk lada (Hd= 0.22 dan Hd = 0.18), dan haplotipe terendah terdapat pada populasi Ambon dan Makassar (Hd = 0.13). Hubungan genetik yang dekat antar lima populasi ini kemungkinan besar berkaitan dengan jalur lintas pelayaran Indonesia sebagai media persebaran spesies P. viridis.
Kata kunci: Perna viridis, DNA Barcoding, keragaman genetik, konektivitas
ABSTRACT
AYU DIAH PITALOKA. Phylogeography of Green Mussel (Perna viridis) in Indonesia and Its Relation to Cross Shipping Lanes. Supervised by HAWIS MADDUPPA and BEGINER SUBHAN.
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan
pada
Departemen Ilmu dan Teknologi Kelautan
FILOGEOGRAFI KERANG HIJAU (
Perna viridis
) DI
INDONESIA DAN KAITANNYA DENGAN JALUR LINTAS
PELAYARAN
AYU DIAH PITALOKA
DEPARTEMEN ILMU DAN TEKNOLOGI KELAUTAN FAKULTAS PERIKANAN DAN ILMU KELAUTAN
INSTITUT PERTANIAN BOGOR BOGOR
PRAKATA
Puji dan syukur penulis ucapkan kepada Allah subhanahu wa ta’ala atas segala rahmat dan karunia-Nya sehingga penulis dapat menyelesaikan skripsi yang berjudul “Filogeografi Kerang Hijau (Perna viridis) di Indonesia dan Kaitannya dengan Jalur Lintas Pelayaran”. Skripsi ini merupakan salah satu syarat untuk memperoleh gelar Sarjana Ilmu Kelautan pada Departemen Ilmu dan Teknologi Kelautan.
Pada kesempatan ini penulis mengucapkan terima kasih kepada semua pihak yang telah membantu dalam penulisan dan penyusunan skripsi ini terutama kepada:
1. Tuhan Yang Maha Esa atas segala rahmat yang diberikan kepada penulis hingga dapat menyelesaikan penulisan skripsi ini.
2. Bapak Dr. Hawis Madduppa, SPi, MSi dan Bapak Beginer Subhan, SPi MSi selaku pembimbing, yang telah banyak memberi segala saran, bimbingan, dan nasihat selama penelitian berlangsung hingga karya ilmiah ini selesai.
3. Mareike Huhn yang telah membantu dalam pengambilan sampel penelitian ini dan memberikan dukungan serta masukan.
4. Laboratorium Biosistematik dan Biodiversitas Kelautan ITK IPB, atas penyediaan fasilitas dalam proses pengolahaan data dalam dan penulisan skripsi ini.
5. Ayah Budi Purwanto, Bunda Wahyu Rohani dan seluruh keluarga besar atas kasih sayang yang diberikan.
6. Bang Edwin, Mbak Lita, Bang Pre, dan “Genetic Team” atas ketersediaannya menemani dan membantu dalam proses pengerjaan.
7. Keluarga besar Ilmu dan Teknologi Kelautan, khususnya ITK 48, atas dukungan dan semangat yang diberikan selama penyelesaian skripsi ini.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2015
DAFTAR ISI
DAFTAR TABEL ix
DAFTAR GAMBAR ix
DAFTAR LAMPIRAN ix
PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
METODE 2
Waktu dan Tempat 2
Koleksi Sampel 2
Alat dan Bahan 3
Prosedur 4
Ekstraksi 4
Amplifikasi DNA (PCR) 4
Elektroforesis Penentuan DNA 5
Siklus Pengurutan Nukleotida (cycle sequencing) 5
Analisis Data 5
HASIL DAN PEMBAHASAN 6
Struktur Genetik 6
Keragaman Genetik 7
SIMPULAN DAN SARAN 12
Simpulan 12
Saran 12
DAFTAR PUSTAKA 12
LAMPIRAN 14
DAFTAR TABEL
1 Lokasi, jumlah, dan kode akses sampel Perna viridis 3 2 Kategori nilai struktur populasi, jarak genetik, dan keragaman genetik 6 3 Jarak genetik (D) dalam dan antar populasi P. viridis 6 4 Analisis uji jarak berpasangan (Fst) pada lima populasi P. viridis 7 5 Deskripsi statistik keragaman genetik P. viridis 8
DAFTAR GAMBAR
1
Lokasi pengambilan sampel Perna viridis 22 Haplotype networkP. viridis di Ambon, Teluk Lada, Palabuhan Ratu,
Makassar, dan Muara Kamal 8
3 Haplotype network P. viridis dari populasi di Indonesia dan seluruh
dunia 9
4 Jalur lintas pelayaran Indonesia yang melewati lokasi pengambilan
sampel Perna viridis 10
5 Rekonstruksi pohon filogenetik Perna viridis dengan marka COI menggunakan metode Neigbour-Joining (NJ) dengan nilai bootstrap 1000 dengan total sekuens 158 dari populasi Indonesia dan seluruh
populasi dunia 11
DAFTAR LAMPIRAN
1
Komposisi Master Mix (MM) pada PCR 15PENDAHULUAN
Latar Belakang
Kerang hijau (Perna viridis, Linnaeus 1758) adalah hewan bercangkang yang termasuk jenis hewan lunak (moluska) yang hidup di laut terutama pada daerah intertidal, merupakan salah satu anggota dari kelas Bivalvia, dan memiliki sepasang cangkang berwarna hijau kebiruan (Cappenberg 2008). Umumnya hidup menempel dan bergerombol dengan menggunakan benang byssus pada dasar substrat yang keras, yaitu batu, karang, kayu, bambu, tali, atau lumpur keras pada perairan muara sungai, estuari, teluk dan daerah mangrove. Kerang ini tergolong dalam kelompok filter feeder, yaitu mendapatkan makanannya dengan cara menyaring air. Makanan kerang hijau berupa fitoplankton, detritus, diatom dan bahan organik lainnya yang terdapat di dalam air. Spesies ini merupakan sumber makanan yang berharga, telah dikonsumsi secara masal, dan dimanfaatkan dalam usaha budidaya.
Spesies dari Famili Mytilidae ini merupakan kerang spesifik Benua Asia. Kerang hijau tersebar luas dari Laut India, Teluk Persia hingga Filipina, Taiwan, Timur Laut Vietnam, dan China (Cappenberg 2008). Di Indonesia, kerang hijau tersebar dari Sumatera, Kalimantan, Jawa, Bali, hingga Sulawesi (Siddal 1980). Spesies ini mampu bertahan hidup dan berkembang biak pada lingkungan yang memiliki tekanan ekologis tinggi.
Jenis kerang ini berpotensi untuk menyebar luas secara geografis melalui penyebaran larvanya (Ahmed 2013). Larva kerang hijau terbentuk setelah 12 hingga 15 jam setelah telur dibuahi dan pada hari ke 8 otot kaki (byssus) mulai digunakan untuk merayap serta berenang bebas (Cappenberg 2008). Selama kurun waktu sekitar 8 hari, larva kerang hijau menyebar luas melalui arus maupun air ballast hingga akhirnya menempel pada suatu substrat.
2
Tujuan Penelitian
Penelitian ini bertujuan untuk mengetahui konektivitas dan keragaman genetik spesies kerang hijau (Perna viridis) di Indonesia dan kaitannya dengan jalur lintas pelayaran.
METODE
Waktu dan Tempat
Penelitian ini dilaksanakan pada bulan Oktober 2014 – Maret 2015 di Laboratorium Biodiversitas dan Biosistematika Kelautan Departemen Ilmu dan Teknologi Kelautan FPIK, IPB.
Gambar 1 Lokasi pengambilan sampel Perna viridis dari populasi di Indonesia dan seluruh populasi dunia
Koleksi Sampel
masing-3 masing sebanyak 5 sampel. Selain itu, penelitian ini juga menggunakan data sekunder berupa hasil sekuens yang diambil dari http://ncbi.nlm.nih.gov.
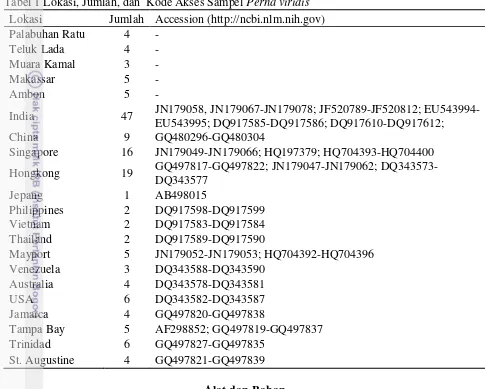
Tabel 1 Lokasi, Jumlah, dan Kode Akses Sampel Perna viridis
Lokasi Jumlah Accession (http://ncbi.nlm.nih.gov) Palabuhan Ratu 4 -
Teluk Lada 4 -
Muara Kamal 3 -
Makassar 5 -
Ambon 5 -
India 47 JN179058, JN179067-JN179078; JF520789-JF520812; EU543994-EU543995; DQ917585-DQ917586; DQ917610-DQ917612;
China 9 GQ480296-GQ480304
Singapore 16 JN179049-JN179066; HQ197379; HQ704393-HQ704400 Hongkong 19 GQ497817-GQ497822; JN179047-JN179062;
DQ343573-DQ343577
Jepang 1 AB498015
Philippines 2 DQ917598-DQ917599
Vietnam 2 DQ917583-DQ917584
Thailand 2 DQ917589-DQ917590
Mayport 5 JN179052-JN179053; HQ704392-HQ704396
Venezuela 3 DQ343588-DQ343590
Australia 4 DQ343578-DQ343581
USA 6 DQ343582-DQ343587
Jamaica 4 GQ497820-GQ497838
Tampa Bay 5 AF298852; GQ497819-GQ497837
Trinidad 6 GQ497827-GQ497835
St. Augustine 4 GQ497821-GQ497839
Alat dan Bahan
Alat yang digunakan dalam penelitian ini antara lain tube (1.5 ml, 0.6 ml, 0.3 ml, dan 0.2 ml), cutter, alat tulis, bunsen, cawan petri, tissue, gloves, tray,
forceps, vortex, microcentrifuge, heating block, pippettemen (10, 100, 1000 μL),
4
Prosedur
Ekstraksi
Ekstraksi DNA adalah prosedur umum memisahkan dan mengumpulkan DNA untuk analisis rekayasa genetika, forensik, bioinformatika, analisis asal usul dan antropologi. Ekstraksi DNA bertujuan untuk menghancurkan sel dan mengambil jaringan pada sampel, membuat ekstraksi secara murni serta melindungi DNA dari degradasi. Kegiatan ekstraksi pada penelitian ini menggunakan kit Invitek dan QiagenDNEasy.
Pada tahapan ekstraksi, hal yang pertama dilakukan adalah siapkan Elution Buffer D dan inkubasi pada suhu 52oC. Selanjutnya ambil bagian kecil dari sampel kerang hijau dan masukkan kedalam tube berukuran 1.5 ml. Tambahkan
Lysis Buffer G sebanyak 400 μl dan Proteinase K sebanyak 40 μl ke dalam tube.
Kemudian vortex selama 10 detik dan inkubasi selama 1 menit. Setelah itu senrtifuge selama 2 menit dengan kecepatan maksimum, kemudian transfer
supernatan dan tempatkan pada tube 1.5 ml yang baru. Tambahkan 200 μl
Binding Buffer T dan vortex selama 10 detik. Transfer suspensi ke dalam spin filter yang telah diletakkan pada tube, dan inkubasi selama 1 menit. Sentrifuge selama 2 menit pada 13,000 x g (12.000 rpm). Buang campurannya dan tempatkan
kembali spin filter pada tube tersebut. Tambakan 550 μl Wash Buffer, sentrifuge
selama 1 menit. Buang campurannya dan tempatkan kembali spin filter pada tube tersebut. Ulangi tahap penambahan Wash Buffer sekali lagi, dan senrifuge selama
2 menit. Tempatkan spin filter pada tube baru dan tambahkan 200 μl Elution
Buffer D. Inkubasi selama 3 menit pada suhu ruangan kemudian sentrifuge selama 2 menit pada 8,500 x g (9.500 rpm).
Amplifikasi DNA (PCR)
Amplifikasi dan visualisasi fragmen DNA dilakukan dengan PCR
(Polymerase Chain Reaction) menggunakan primer forward ACOIF
(50-CGDATTCARCTWTCTCAYCCHGG) dan reserve COIR4
5
Elektroforesis Penentuan DNA
Elektroforesis bertujuan untuk menguji kualitas DNA yang dihasilkan PCR. Tahap pertama yaitu pembuatan gel agarosa 1. % dengan 1x TAE sebanyak 0,5 gram dan buffer sebanyak 50 ml sebagai media elektroforesis. Agarose tersebut dipanaskan pada microwave, kemudian ditambahkan pewarna Etidium Bromide
(EtBr) sebanyak 4 μl dicetak dalam cetakanbersisir hingga membeku. Selanjutnya
4 μl produk Hasil PCR diambil dan dicampurkan dengan Loading dye (1 μl),
kemudian disisipkan di sumuran dalam cetakan agarose yang terendam dalam larutan buffer. Elektroforesis dilakukan pada tegangan 220 V dan arus 400 mA selama 25 menit. Pita hasil elektroforesis dapat dilihat dengan menggunakan sinar ultraviolet pada UV transluminator (Prehadi 2014).
Siklus Pengurutan Nukleotida (cycle sequencing)
Siklus pengurutan Nukleotida (Sequencing DNA) adalah metode untuk menentukan urutan nukleotida yang terdapat dalam DNA. Urutan DNA berhubungan dengan informasi genetik turunan dalam nukleus (inti), plasmid, mitokondria, dan kloroplas yang membentuk dasar pengembangan semua makhluk hidup (Randi dan Lucchini 1998). Produk PCR berupa DNA positif dikirim ke Cornel University, Amerika Serikat untuk penentuan urutan nukleotida dari sequens DNA dengan menggunakan mesin sequencher AB1377.
Analisis Data
Hasil sekuens yang didapatkan, merupakan hasil kerja mesin, sehingga kita perlu melakukan pengeditan jika terdapat kesalahan pembacaan kromatogram oleh mesin dan disejajarkan dengan urutan nukleotida anggota famili kerang hijau.
Analisis ini dilakukan dengan menggunakan software Mega 6 (Molecular Evolutionary Genetic Analysis) untuk pembacaan urutan nukleotida dan penjajaran (aligment) menggunakan ClustalW pada program tersebut untuk melihat adanya keragaman nukleotida (Tamura et al. 2007).
Data hasil penjajaran nukleotida yang diperoleh kemudian dicocokkan pada data yang tersedia pada GenBank di NCBI (National Centre for Biotechnology
Information) menggunakan BLAST (Basic Local Alignment Search Tool)
(http://blast.ncbi.nlm.nih.gov) (Prehadi et al. 2015).
Analisis filogeografi dilakukan dengan membuat konstruksi pohon filogenetik untuk semua sekuens dari semua sampel yang telah diedit dan disejajarkan dengan sekuen P. Viridis dari GenBank dan P. Perna dari GenBank
sebagai out group. Pembuatan pohon filogenetik menggunakan metode pengkelasan Neigbour-joining (NJ) (Ward et al. 2008) dengan nilai bootstrap sebesar 1000 karena metode ini efektif untuk melakukan perhitungan tingkat kesamaan dalam mengidentifikasi spesies melalui kekerabatan.
6
beberapa kategori berdasarkan nilai keragaman genetik, struktur populasi dan jarak genetik.
Tabel 2Kategori nilai struktur populasi, jarak genetik, dan keragaman genetik
Analisis Kategori Sumber
Rendah Sedang Tinggi
Struktur Populasi (Fst) 0.002 - 0.099 0.1 - 0.5 0.6 - 1.00 Excoffier et al. 1992 Jarak Genetik (D) 0.010 - 0.099 0.1 - 0.99 1.00 - 2.00 Nei 1972
Keragaman Haplotipe (Hd) 0.1 - 0.4 0.5 - 0.7 0.8 - 1.00 Nei 1987
HASIL DAN PEMBAHASAN
Struktur Genetik
Hasil analisis panjang fragmen DNA dari semua sampel menunjukan panjang fragmen 755-bp. Hasil seluruh sampel yang didapatkan sebanyak 21 fragmen genetik P. viridis yang terdiri dari 5 sampel Ambon, 4 sampe Teluk Lada, 4 sampel Palabuhanratu, 5 sampel Makassar, dan 3 sampel Muara Kamal. Verifikasi semua sekuens menunjukkan kemiripan sebesar 98-100% dengan P.
viridis pada Genebank.
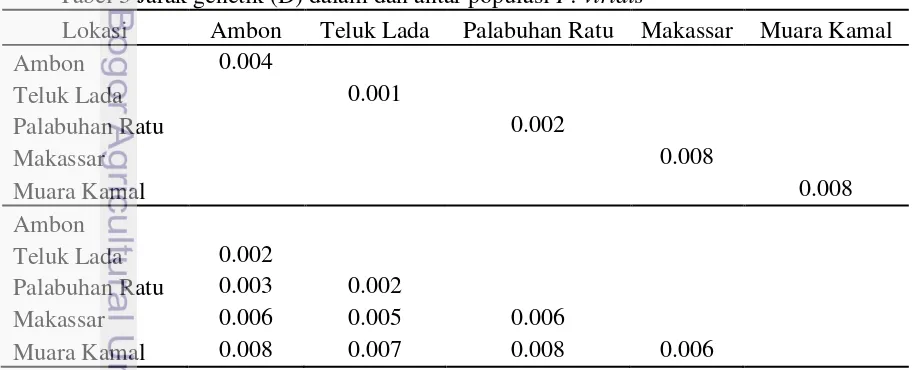
Analisis jarak genetik dalam populasi dapat dilihat pada Tabel 2. Jarak genetik P. viridis terbesar dimiliki oleh populasi dari Makassar dan Muara Kamal yaitu sebesar 0.008, dan jarak genetik terkecil pada populasi dari Teluk Lada sebesar 0.002. Jarak genetik menunjukan hubungan genetik yang terdapat antar individu dalam populasi. Jarak genetik di lima populasi menunjukan adanya hubungan genetik yang sangat dekat, sesuai dengan pernyataan Nei (1972). Presentasi nilai jarak genetik menjelaskan bahwa dari 755 pasangan basa (bp) yang diperoleh hanya terdapat 4 pasangan basa yang berbeda di dalam populasi dari Ambon, 1 pasangan basa yang berbeda dalam populasi dari Teluk Lada, 2 pasangan basa yang berbeda dalam populasi dari Palabuhan Ratu, dan 8 pasangan basa yang berbeda dalam populasi dari Makassar dan Muara Kamal.
Tabel 3Jarak genetik (D) dalam dan antar populasi P. viridis
Lokasi Ambon Teluk Lada Palabuhan Ratu Makassar Muara Kamal
Ambon 0.004
Teluk Lada 0.001
Palabuhan Ratu 0.002
Makassar 0.008
Muara Kamal 0.008
Ambon
Teluk Lada 0.002
Palabuhan Ratu 0.003 0.002
Makassar 0.006 0.005 0.006
7 Hasil analisis jarak genetik (D) antar populasi (Tabel 3) menunjukkan bahwa populasi berkisar antara 0.002 hingga 0.008. Semakin kecil nilai jarak genetik antar individu didalam maupun antar populasi, maka semakin dekat kedekatan genetik (Koh et al. 1999). Hal ini menunjukkkan adanya hubungan genetik yang sangat dekat di antara lima lokasi pengambilan sampel P. viridis.
Kedekatan hubungan kekerabatan antar populasi mungkin disebabkan karena antar populasi mempunyai asal-usul induk yang sama (Iskandar et al. 2010).
Analisis Uji jarak berpasangan (Fst) kerang hijau (P. viridis) dengan
significance level sebesar 0.05 menunjukan bahwa semakin kecil nilai Fst maka
semakin kecil diferensiasi genetik antar populasi. Berdasarkan hasil analisis, antara Makassar dan Palabuhan Ratu memiliki nilai Fst terkecil (Fst = 0.05), sedangkan antara Ambon dan Palabuhan Ratu serta Ambon dan Teluk Lada memiliki nilai Fst terbesar (Fst = 0.90) (Tabel 4). Selain itu, Makassar memiliki nilai Fst relatif kecil dengan keempat lokasi lainnya. Hal ini menunjukkan bahwa terjadi aliran gen yang tinggi antara Makassar dan keempat lokasi lainnya. Berbeda dengan Ambon yang memiliki nilai Fst yang cukup tinggi dengan Teluk Lada dan Palabuhan Ratu, hal ini menunjukkan bahwa Ambon dan Teluk Lada serta Ambon dan Palabuhan Ratu terjadi aliran gen yang rendah. Nilai Fst yang kecil menjelaskan bahwa terjadi aliran gen antar populasi yang sangat tinggi. Besarnya aliran gen kemungkinan disebabkan karena antar populasi saling memberikan pengaruh terhadap aliran genetik. Selain itu tingginya aliran gen yang masuk kedalam populasi per generasi turut mempengaruhi kedekatan genetik antar populasi. Mulyasari et al. (2010) menjelaskan bahwa populasi dengan tingkat differensiasi yang rendah, mungkin disebabkan oleh banyaknya kesamaan genetik antar populasi.
Tabel 4Analisis uji jarak berpasangan (Fst) pada lima populasi P. viridis
Lokasi Ambon Teluk
Lada
Palabuhan
Ratu Makassar
Muara Kamal Ambon
Teluk Lada 0.90
Palabuhan Ratu 0.90 0.22
Makassar 0.10 0.14 0.05
Muara Kamal 0.83 0.53 0.20 0.08
Keragaman Genetik
8
charu (Mytella charruana) yang merupakan spesies invasif juga, yaitu berkisar 0.9350 ± 0.0339 dan tergolong tinggi (Gillis et al. 2009).
Tabel 5Deskripsi statistik keragaman genetik P. viridis
No Lokasi Sampel Jumlah Sampel
Jumlah Haplotipe
Keragaman Haplotipe
(Hd)
Keragaman
Nukleotida (π (%))
1 Ambon 5 5 1.00 ± 0.13 0.15 ± 0.09
2 Teluk Lada 4 4 1.00 ± 0.18 0.15 ± 0.10
3 Palabuhan
Ratu 4 3 1.00 ± 0.22 0.03 ± 0.02
4 Makassar 5 5 1.00 ± 0.13 0.46 ± 0.28
5 Muara Kamal 3 3 1.00 ± 0.27 0.05 ± 0.04
Analisis haplotype network menjelaskan bahwa populasi dari Ambon, Teluk Lada, dan Palabuhan Ratu menghasilkan haplotipe yang berbeda – beda. Populasi dari Makassar dan Muara Kamal juga memiliki haplotipe yang berbeda – beda, tetapi ada satu sampel dari Makassar dan satu sampel dari Muara Kamal yang membentuk satu jenis haplotipe yang sama (Gambar 2).
Gambar 2 Haplotype network P. viridis di Ambon, Teluk Lada, Palabuhan Ratu, Makassar, dan Muara Kamal
9 berbagi haplotipe dengan Thailand. Populasi dari Indonesia, China, Jepang, Australia, USA, dan Venezuela menghasilkan haplotipe yang berbeda-beda. Tingkat keragaman haplotipe yang dihasilkan tergolong tinggi, yaitu sebesar 0.9780.
Gambar 3 Haplotype network P. viridis dari populasi di Indonesia dan seluruh populasi dunia (Lihat Tabel 1)
Keragaman genetik memberikan kemampuan untuk beradaptasi terhadap perubahan lingkungan dan iklim serta penyakit (Lande 1988). Nuryanto (2009) menyebutkan bahwa keragaman genetik yang tinggi mencerminkan besarnya ukuran populasi, sedangkan penurunan ukuran populasi akan mengurangi keragaman genetik. Keragaman genetik penting bagi kelangsungan hidup spesies karena spesies-spesies yang memiliki keragaman genetik kecil mungkin lebih rentan terhadap penyakit atau efek dari perubahan lingkungan. Peningkatan keragaman genetik menghasilkan keturunan dengan berbagai karakteristik yang dapat menjamin untuk menahan perubahan lingkungan dan mengurangi kemungkinan kerusakan gen merusak (seperti penyakit) muncul dalam populasi.
10
genetik dari populasi lain atau biasa disebut dengan migrasi genetik. Migrasi yang besar akan menyebabkan terjadinya perkawinan silang dan percampuran gen antar populasi yang berbeda, sehingga akan diperoleh variasi gen yang berbeda-beda (Kusuma 2014).
Gambar 4 Jalur lintas pelayaran Indonesia yang melewati lokasi pengambilan sampel Perna viridis
11
Gambar 5 Rekonstruksi pohon filogenetik Perna viridis dengan marka COI
menggunakan metode Neigbour-Joining (NJ) dengan nilai bootstrap 1000 dengan
total sekuens 158 dari populasi Indonesia dan seluruh populasi dunia (Lihat Tabel 1)
2 gi1181978 49
Singapore 9 gi341 657810
Australia 2 gi85718037 Jepang gi227202511 Venezuela 17 gi85718059 63
0 0
Australia 5 gi85718043 0
USA 7 gi85718045 0
USA 8 gi85718047 0
USA 10 gi85718051 0
China LSGB41203-2 gi29 0576422 0
China LSG
B41203-3 gi290576 424 0
China LSGB412 03-4 gi2
90576426 0
China LSGB41
203-6 gi290576 430 0
China LSGB
41203-7 gi29 0576432 0
China LSG B412
03-8 gi2905 76434 0
China HY YJ 09
03 gi30569 0304 India PVGOA
-13 gi33637 7113
0 India 30 gi341
657856 0 India 24 gi341657844 0 Hongkong HK5 gi85718
035
0 Hongkong 1 gi256557720
0 63-MKVP.02_PB__ACOIF 59-MAVP.02_PB__ACOIFIndia 26 gi341657848 Australia 4 gi85718041 0 P. viridis gi85718033
0 India K5 gi170791492
0 India PVO
RI-8 gi336377147
0 India PV
MAN-9 gi3363771 39
0 India PVKOL
-11 gi3363771 29
0 India P
VKAR
-3 gi3363771 25
0 India PVGO
12
SIMPULAN DAN SARAN
Simpulan
Hasil penelitian ini dapat disimpulkan bahwa struktur populasi genetik P.
viridis di lima lokasi menunjukan adanya hubungan genetik yang dekat antar
populasi. Konektivitas genetik terhubung diantara populasi dari Muara Kamal, Teluk Lada, Palabuhan Ratu, Makassar, dan Ambon. Keragaman haplotipe P.
viridis di lima lokasi tergolong tinggi sehingga kelima populasi tersebut memiliki
keragaman genetik yang tinggi. Hubungan genetik yang dekat antar populasi di lima lokasi ini kemungkinan besar berkaitan dengan jalur lintas pelayaran Indonesia sebagai media persebaran spesies P. viridis.
Saran
Berdasarkan hasil yang telah didapatkan, maka penulis menyarankan agar dilakukan penelitian lebih lanjut di beberapa negara yang diduga mengalami invasif kerang hijau, sehingga dapat dilakukan kerjasama internasional dengan negara yang bersangkutan untuk menanganinya.
DAFTAR PUSTAKA
Ahmed Y. 2013. Pengaruh simulasi transportasi kapal terhadap kerentanan kerang hijau Perna viridis pada stress suhu yang meningkat [tesis]. Bogor (ID): Institut Pertanian Bogor.
Cappenberg HAW. 2008. Beberapa Aspek Biologi Kerang Hijau (Perna viridis,
Linnaeus 1758). Oseana, volume 33(1): 33–40.
Excoffier L, Lischer H. 2009. Arlequin ver 3.5 user manual ; An integrated software package for population genetics data analysis. Swiss Institute of Bioinformatics.
Excoffier L, Smouse PE, Quattro JM. 1992. Analysis of moleculer variance inferred from metric distance among DNA haplotipes; application to human mitochondrial DNA restriction data. Genetics, volume 131: 479-491.
Gilg MR, Johnson EG, Gobin J, Bright BM, Ortolaza AL. 2012. Population genetics of introduced and native populations of the green mussel, Perna
viridis: determining patterns of introduction. Biol Invasions, DOI
10.1007/s10530-012-0301-2.
Gillis LK, Walters LJ, Fernandes FC, Hoffman EA. 2009. Higher genetic diversity in introduced than in native populations of the mussel Mytella
Charruana: evidence of population admixture at introduction sites.
Diversity and Distributions, (Diversity Distrib.)15: 784–795.
Kaluza P, Kölzsch A, Gastner MT, Blasius B. 2010. The complex network of global cargo ship movements. Interface journal of the royal society.
13 Kölzsch A, Blasius B. 2011. Indications of marine bioinvasion from network theory An analysis of the global cargo ship network. The European physical journal B. DOI: 10.1140/epjb/e2011-20228-5.
Kusuma, AB. 2014. Konektivitas dan keragaman genetik pada karang lunak
Sarcophyton trocheliophorum serta implikasinya terhadap kawasan
konservasi laut [Tesis]. Bogor (ID) Institut Pertanian Bogor.
Lande R. 1988. Genetics and demography in biological Conservation. Science, volume 241(4872): 1455-1460.
Lynch M, Crease TJ. 1990. The analysis of population survey data on DNA sequence variation. Moleculer Biology Evolution, volume 7: 337–394. Mead A, Carlton JT, Griffiths CL, Rius M. 2011. Revealing the scale of marine
bioinvasions in developing regions: a South African re-assessment. Biological invasion. doi:10.1007/s10530-011-0016-9
Mulyasari, SDT, Kristanto AH, Kusmini II. 2010. Karakteristik genetic enam populasi Nilem (Osteochilus hasselti) di Jawa Barat. Jurnal Riset Akuakultur, volume 5(2): 175-182.
Nei M. 1987. Moleculer evolutionary genetics. Columbia University. Press. New York. 512 hal.
Nei M. 1978. Estimation of average heterozygosity and genetic distance from a small number of individuals. Genetics, volume 89: 583-590.
Nei M. 1972. Genetic distance between population. American Nature, volume 106: 283-292.
Nuryanto A, Kochzius M. 2009. Highly restricted gene flow and deep evolutionary lineages in the giant clam Tridacna maxima. Coral Reefs, volume 28: 607–619.
Prehadi, Sembiring A, Kurniasih EM, Rahmad, Arafat D, Subhan B, Madduppa H. 2015. DNA barcoding and phylogenetic reconstruction of shark species landed in Muncar fisheries landing site in comparison with Southern Java fishing port. Biodiversitas, volume 16(1): 55–61.
Randi E, Lucchini V. 1998. Organization and evolution of the mitochondrial DNA control region in the avian genus Alectoris. Journal of Molecular Evolution, volume 47: 449-462.
Siddall SE. 1980. A Clarification of the Genus Perna (Mitylidae). Bullletin of Marine Science, volume 30(4): 858–870.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA: Molecular Evolutionary Genetic Analysis (MEGA) software version 4.0. Advance Access published May 7. Oxford University Press. Mol Bio 10. 1093/molbev/msm092.
14
15 Lampiran 1 Komposisi Master Mix (MM) pada PCR
Master mix ... tabung
STANDAR PROTOCOL ( 1μL DNA template)
MM 1 MM 2
ddH2O 5,5 9
10x Buffer PCR (PE-II) 1,5 1
dNTPs (8 mM) 2,5 ....
MgCl2 (25 mM) 2 ....
Primer 1 (10 mM) 1,25 ....
Primer 2 (10 mM) 1,25 ....
Amplitaq polymerase ( 5 unit/ µL) ... 0,125
16
Lampiran 2 Hasil pengurutan basa nukleotida (sequencing) pada Sticophus ocellatus
#AM.VP.01
CTTTCTCATCCCGGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCA TGCATTAGTAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACT TCCATTATGTATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGC ACCTAATGCTTTGTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAA CTATTTATCCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGT CTTTACATTTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAAT ATGCCTACAATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCG GTGTTCTTTTAATTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAA ATTTCAATACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTT GGTTTTTTGGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTA ATTATGCATTATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGT ATTGGTGTTATAGGTTGTGTGGTGTGGGCTCACCACAT---
#AM.VP.02
CGGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTA ATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGT ATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCT TTGTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCC ACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTT AATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAA TAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTT AATTATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATA CTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTG GTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCAT TATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATA---
#AM.VP.03
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCTTTCATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCATCA---
#AM.VP.04
AACAACTCATGCATTAGTAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAA TTGATTACTTCCATTATGTATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATT TTGGTTGGCACCTAATGCTTTGTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGA CAGGTTGAACTATTTATCCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTT TGATTACGTCTTTACATTTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTA ATAAGAATATGCCTACAATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTAC CGTAACTGGTGTTCTTTTAATTATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTT TGATCGAAATTTCAATACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGC ATGTATTTTGGTTTTTTGGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGT CAAAAGTAATTATGCATTATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCT ATAGTTGGTATTGGTGTTATAGGTTGTGTGGTGTGGGCTCATCATATATTTAC---
#AM.VP.05
17 TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCATCA---
#TL.VP.02
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCGTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCATCA---
#TL.VP.03
ATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAATAATTTTTTTTGCTGTAATG CCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTATTGGTGGTGTTGATTTAATT TTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTTGTACTTACTTATTTTGTCTT TTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCACCTTTATCTTCTGGGTTGTAC CATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAATTGGATTGAGTTCTTTATTA GGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATAAAAATAAAGGGTGAGAAAT CTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAATTATTTCTGTGCCAGTTCTGG CTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTAGGTTTTTTGATCCTATTGGAG GGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTCATCCTGAGGTGTACATTCTTA TTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTATTCTGGTAAAGATTCTGTTTTTG GTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATAGGTTGTGTGGTGTGGGCTCA---
#TL.VP.04
CCCGGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAG TAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTAT GTATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATG CTTTGTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTAT CCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACAT TTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTAC AATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTT TTAATTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAAT ACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTT GGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCA TTATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGT TATAGGTTGTGTGGTGTGGGCTCATCA---
#TL.VP.05
CCCGGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAG TAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTAT GTATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATG CTTTGTACTTACTTATTTTGTCTTTCATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTAT CCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACAT TTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTAC AATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTT TTAATTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAAT ACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTT GGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCA TTATTCTGGTAAAGATTCTGTTTTTGGTTCG---
18
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACTGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCATCA---
#PR.VP.02
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACTGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCA---
#PR.VP.04
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACTGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCA---
#PR.VP.05
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCGTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCATCAATATTTTAC---
#MA.VP.01
19 GTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCAT TATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATAT---
#MA.VP.02
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATACCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACTGTAACCGGTGTTCTTTTAA TCATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATCCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGGATTGGTGTTAT AGGTTGTGTAGTGTGGGCTCAC---
#MA.VP.03
TTTGGTTGGCACCTAATGCTTTGTACTTACTTATTTTGTCGTTTATAACGGAGAAAGGAGCTGGG ACAGGTTGAACTATTTATCCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATT TTGATTACGTCTTTACATTTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACT AATAAGAATATGCCTACAATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTA CCGTAACCGGTGTTCTTTTAATTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGT TTGATCGAAATTTCAATACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAG CATGTATTTTGGTTTTTTGGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATG TCAAAAGTAATTATGCATTATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGC TAT---
#MA.VP.04
GAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAATAATTTTTTTTGCTG TAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTATTGGTGGTGTTGATT TAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTTGTACTTACTTATTT TGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCACCTTTATCTTCTGGG TTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAATTGGATTGAGTTCT TTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATACCTACAATAAAAATAAAGGGTG AGAAATCTGAGTTGTATTTGTGGAGGATTACTGTAACCGGTGTTCTTTTAATCATTTCTGTGCCA GTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTAGGTTTTTTGATCCT ATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTCATCCTGAGGTGTA CATTCTTATCCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTATTCTGGTAAAGATT CTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGGATTGGTGTTATAGGTTGTGTAGTGT GGGCTCA---
#MA.VP.05
TGCATTAGTAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACT TCCATTATGTATTGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGC ACCTAATGCTTTGTACTTACTTATTTTGTCGTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAA CTATTTATCCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGT CTTTACATTTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAAT ATGCCTACAATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCG GTGTTCTTTTAATTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAA ATTTCAATACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTT GGTTTTTTGGTCATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAG---
#MK.VP.02
20
TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGGATTGGTGTTAT AGGTTGTGTAGTGTGGGCTCAC---
#MA.VP.04
GGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAGTAA TAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTATGTAT TGGTGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATGCTTT GTACTTACTTATTTTGTCGTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTATCCAC CTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACATTTAA TTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATGCCTACAATA AAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTTTTAA TTATTTCTGTGCCAGTTCTGGCTGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAATACTA GGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTTGGTC ATCCTGAGGTGTACATTCTTATTCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCATTAT TCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGTATTGGTGTTATA GGTTGTGTGGTGTGGGCTCAC---
#MA.VP.05
CCCGGGGGTAATTTTTTGAAAAATGAAAGGTTATATAATGTTGTAGTAACAACTCATGCATTAG TAATAATTTTTTTTGCTGTAATGCCTTTACTTATTGGTGCTTTTGGGAATTGATTACTTCCATTAT GTATTGGGGGTGTTGATTTAATTTTTCCTCGTTTAAATAATTTGAGATTTTGGTTGGCACCTAATG CTTTGTACTTACTTATTTTGTCTTTTATAACGGAGAAAGGAGCTGGGACAGGTTGAACTATTTAT CCACCTTTATCTTCTGGGTTGTACCATACTGGGCCTGCTGTTGATATTTTGATTACGTCTTTACAT TTAATTGGATTGAGTTCTTTATTAGGTTCGATTAATTTTGTGAGGACTAATAAGAATATACCTAC AATAAAAATAAAGGGTGAGAAATCTGAGTTGTATTTGTGGAGGATTACCGTAACCGGTGTTCTT TTAATCATTTCTGTGCCAGTTCTGGCCGGTGGGATTACTATATTGTTGTTTGATCGAAATTTCAAT ACTAGGTTTTTTGATCCTATTGGAGGGGGAGATCCTGTTTTATTTCAGCATGTATTTTGGTTTTTT GGTCATCCTGAGGTGTACATTCTTATCCTTCCGGCGTTTGGTGTGATGTCAAAAGTAATTATGCA TTATTCTGGTAAAGATTCTGTTTTTGGTTCGGTTGGGATATTATATGCTATAGTTGGGATTGGTGT TATAGGTTGTGTAGTGTGGGCTCACCACATATTTAC
21
RIWAYAT HIDUP
Penulis dilahirkan di Kediri pada tanggal 14 Oktober 1993 sebagai anak pertama dari tiga bersaudara dari orang tua bernama Budi Purwanto dan Wahyu Rohani. Penulis lulus dari Sekolah Menengah Pertama Negeri 1 Gampengrejo pada tahun 2008, dan Sekolah Menengah Atas Negeri 1 Kediri tahun 2011. Pada tahun 2011, penulis diterima sebagai mahasiswa Institut Pertanian Bogor, Fakultas Perikanan dan Ilmu Kelautan, Departemen Ilmu dan Teknologi Kelautan melalui jalur SNMPTN Undangan dan memperoleh beasiswa PPA tahun 2013-2014.
Selama kuliah di Institut Pertanian Bogor penulis pernah menjadi asisten mata kuliah Biologi Laut tahun 2014 dan 2015, asisten mata Kuliah Ekologi Laut Tropis tahun 2014. Penulis aktif dalam organisasi Himpunan Mahasiswa Ilmu dan Teknologi Kelautan (HIMITEKA-IPB) sebagai Bendahara II periode 2012-2013, dan Bendahara I periode 2013-2014.