DETEKSI KELAINAN GENETIK SAPI BALI
MENGGUNAKAN GEN FXI (
Factor XI
) DAN
SLC35A3 (
Solute Carrier Family 35 Member 3
)
SRI WAHYUNI SISWANTI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Deteksi Kelainan Genetik Sapi Bali Menggunakan Gen FXI (Factor XI) dan SLC35A3 (Solute Carrier Family 35 Member 3) adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
SRI WAHYUNI SISWANTI. Deteksi Kelainan Genetik Sapi Bali Menggunakan Gen FXI (Factor XI) dan SLC35A3 (Solute Carrier Family 35 Member 3). Dibimbing oleh JAKARIA dan CECE SUMANTRI.
Kelainan genetik terjadi karena adanya perubahan susunan basa nukleotida yang menyebabkan terjadinya kerusakan gen yang mengkodekan asam amino tertentu. Defisiensi factor XI pada suatu invidu menyebabkan terjadinya gangguan pada proses pembekuan darah. Kelainan genetik factor XI deficiency (FXID) disebabkan oleh adanya insersi poliadenin yang tidak sempurna yang menyebabkan terjadinya stop kodon prematur pada gen FXI. Substitusi guanin menjadi timin pada gen SLC35A3 menyebabkan kelainan genetik complex vertebral malformation (CVM). Keberadaan kelainan genetik pada suatu populasi menyebabkan kerugian ekonomi yang besar dalam program perbibitan. Beberapa penelitian mengenai deteksi kelainan genetik pada sapi perah telah banyak dilakukan namun demikian penelitian mengenai deteksi kelainan genetik pada sapi bali belum pernah dilakukan. Penelitian ini bertujuan untuk mendeteksi ada atau tidaknya kelainan genetik pada sapi bali yaitu CVM menggunakan gen SLC35A3 dan FXID menggunakan gen FXI.
Sebanyak 325 sampel DNA yang terdiri atas 303 sampel darah yang berasal dari pusat pembibitan sapi bali (BPTU-HMT Denpasar, BPT-HMT Serading Nusa Tenggara Barat dan VBC Kabupaten Barru Sulawesi Selatan) dan BBIB Singosari serta 22 sampel semen beku dari BIBD Baturiti digunakan untuk mendeteksi FXID dan CVM. Amplifikasi gen FXI menggunakan PCR sedangkan gen SLC35A3 menggunakan PCR-RFLP menggunakan enzim restriksi PstI. Sekuens primer gen FXI sesuai dengan Marron et al. 2004 dengan primer forward : 5’-CCC ACT GGC TAG GAA TCG TT-3’ dan primer reverse : 5’-CAA GGC AAT GTC ATA TCC AC-3’, sedangkan sekuens primer untuk gen SLC35A3 sesuai dengan Kanae et al. 2005 dengan primer forward : 5’-CAC AAT TTG TAG GTC TCA CTG CA-3’ dan primer reverse : 5’-CGA TGA AAA AGG AAC CAA AAG GG-3’. Produk PCR dianalisis dengan elektroforesis gel agarose 2%. Seluruh genotipe dikonfirmasi dengan sekuensing DNA untuk memastikan adanya alel mutan. Data dianalisis secara deskriptif terhadap ada atau tidaknya kelainan genetik FXID dan CVM pada sapi bali.
Hasil penelitian tidak ditemukan alel mutan (heterozigot) baik dengan gen FXI maupun SLC35A3. Hal ini menunjukkan bahwa tidak terdapat kejadian FXID dan CVM sapi bali di pusat pembibitan sapi bali dan balai inseminasi buatan.
SUMMARY
SRI WAHYUNI SISWANTI. Screening of Genetic Defect in Bali Cattle using Factor XI (Factor XI) and SLC35A3 (Solute Carrier Family 35 Member 3) Genes. Supervised by JAKARIA and CECE SUMANTRI.
Genetic defect occurs because the changing of the sequens nucleotide bases that leads defective gene coding amino acids. FXID is caused by imperfect of poly adenine which is resulted in introduction of premature stop codon in FXI gene. Substitution of guanine into thymine in SLC35A3 gene caused complex vertebral malformation (CVM). The research was aimed to detect the presence or absence of a genetic defect mainly CVM using SLC35A3 gene and FXID using FXI gene in Indonesian bali cattle. The presence of this genetic defect may have a significant economic impact in breeding program. The research of genetic defect almost in dairy cattles but there was no report for screening of genetic defect in bali cattles.
In this study, 303 fresh blood samples and 22 semen samples collected from Indonesian bali cattle breeding center (BPTU-HMT Denpasar, BPT HMT Serading West Nusa Tenggara and VBC district Barru South Sulawesi) and artificial insemination centre (BBIB Singosari and BIBD Baturiti) were used for screening of FXID and CVM. The amplicons of FXI gene were obtained using PCR and for SLC35A3 gene were obtained using PCR-RFLP method with PstI restriction enzymes. Primer used for FXI gene (Marron et al. 2004) with forward : 5’-CCC ACT GGC TAG GAA TCG TT-3’ and reverse : 5’-CAA GGC AAT GTC ATA TCC AC-3’, and primer used for SLC35A3 gene (Kanae et al. 2005) with forward : 5’-CAC AAT TTG TAG GTC TCA CTG CA-3’ and reverse : 5’ -CGA TGA AAA AGG AAC CAA AAG GG-3’. These PCR product were separated in 2% agarose gels stained with ethidium bromide and viewed under UV light. All genotypes were confirmed by DNA sequencing to determine an allele mutants. Data were analyzed descriptively to determine the presence or absence of genetis abnormalities of CVM and FXID in bali cattle.
The allele mutant was not found in all of the samples. The result of this study showed that CVM and FXID were not detected in bali cattles from Indonesian bali cattle breeding and artificial insemination centre.
© Hak Cipta Milik IPB, Tahun 2014
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
DETEKSI KELAINAN GENETIK SAPI BALI
MENGGUNAKAN GEN FXI (
Factor XI
) DAN
SLC35A3 (
Solute Carrier Family 35 Member 3
)
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2014
Judul Tesis : Deteksi Kelainan Genetik Sapi Bali Menggunakan Gen FXI (Factor XI) dan SLC35A3 (Solute Carrier Family 35 Member 3) Nama : Sri Wahyuni Siswanti
NIM : D151120181
Disetujui oleh Komisi Pembimbing
Dr Jakaria, SPt MSi Ketua
Prof Dr Ir Cece Sumantri, MAgr Sc Anggota
Diketahui oleh
Ketua Program Studi
Ilmu Produksi dan Teknologi Peternakan
Dr Ir Salundik, MSi
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Tema yang dipilih dalam penelitian yang dilaksanakan sejak bulan Oktober sampai dengan Desember 2013 ialah kelainan genetik pada sapi bali, dengan judul Deteksi Kelainan Genetik Sapi Bali Menggunakan Gen FXI (Factor XI) dan SLC35A3 (Solute Carrier Family 35 Member 3).
Terima kasih penulis ucapkan kepada Dr Jakaria, SPt MSi dan Prof Dr Ir Cece Sumantri, MAgrSc selaku komisi pembimbing, atas curahan waktu, arahan, bimbingan dan dorongan semangat sejak penyusunan proposal, penelitian hingga penulisan tesis. Penulis juga menghaturkan terima kasih kepada Dr Ir Dedy Duryadi Solihin, DEA selaku penguji luar komisi pada ujian sidang atas saran dan masukan yang diberikan. Kepada Sekretariat Sekolah Pasca Sarjana Fakultas Peternakan, penulis mengucapkan terima kasih atas pelayanan prima yang diberikan selama penulis menempuh studi di ITP IPB.
Penghargaan penulis sampaikan kepada Ir Tri Harsi, MP beserta staf Balai Embrio Ternak yang telah memberikan kesempatan kepada penulis untuk melanjutkan studi di Sekolah Pascasarjana IPB, drh Maidaswar, MSi beserta staf Balai Besar Inseminasi Buatan Singosari atas bantuan sampel yang diberikan, serta Kepala Laboratorium Genetika dan Molekuler Ternak Fakultas Peternakan Institut Pertanian Bogor atas koleksi sampel yang diberikan.
Kepada Eryck Andreas, Shelvi, Ferdy, Irene, Isyana, Furqon, Olin dan teman-teman ABGSci, penulis ucapkan terima kasih yang sebesar-besarnya atas kebersamaan selama di Laboratorium Genetika dan Molekuler Fakultas Peternakan IPB. Kepada teman-teman seperjuangan di Program Studi ITP angkatan 2012 khususnya grup Helix (Marlinda, Irma, Tia, Andre, Raden Jatu, Fuad, Pandu, Komang Alit dan Annisa) atas kebersamaan dalam diskusi selama ini, semoga kebersamaan dan kekeluargaan kita tetap terjalin hingga di masa yang akan datang. Kepada semua pihak yang telah membantu, yang tidak dapat disebutkan satu persatu penulis ucapkan terima kasih yang sebesar-besarnya.
Ungkapan terima kasih yang sebesar-besarnya penulis sampaikan kepada kedua orang tua, Bapak Siswanto dan Ibu Sri Dalyati, adik penulis Edi Nugraha, suami tercinta Gigih Ikhtiari Erfianto, putri-putriku tersayang Westra Bornie Kailani dan Alana Kikandriya Pramudhita serta seluruh keluarga, atas kesabaran, doa, kasih sayang dan motivasi yang diberikan selama ini.
Semoga karya ilmiah ini bermanfaat.
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 3
Kelainan Genetik 3
Kelainan Genetik FXID 3
Kelainan Genetik CVM 5
3 METODE 6
Lokasi dan Waktu Penelitian 6
Bahan 6
Prosedur Penelitian 8
Prosedur Analisis Data 10
4 HASIL DAN PEMBAHASAN 11
Amplifikasi Gen FXI dan gen SLC35A3 11
Deteksi Kelainan Genetik FXID dan CVM 12
Implementasi Deteksi Kelainan Genetik FXID dan CVM pada Sapi Bali di
Pusat Pembibitan Sapi Bali di Indonesia 20
5 SIMPULAN DAN SARAN 21
Simpulan 21
Saran 21
Ucapan Terimakasih 21
DAFTAR PUSTAKA 22
LAMPIRAN 27
DAFTAR TABEL
1 Asal dan jenis sampel sapi yang digunakan dalam penelitian 7 2 Tabulasi hasil deteksi kelainan genetik FXID pada sapi bali 13 3 Data penelitian dan deteksi kelainan genetik FXID pada beberapa
bangsa di beberapa negara 14
4 Tabulasi hasil deteksi kelainan genetik CVM pada sapi bali 17 5 Beberapa penelitian dan deteksi kelainan genetik CVM pada beberapa
negara pada berbagai spesies 18
DAFTAR GAMBAR
1 Rekonstruksi gen FXI berdasarkan sekuens gen FXI di GenBank
(Kunieda et al. 2005) 4
2 Rekonstruksi struktur gen SLC35A3 berdasarkan sekuens gen
SLC35A3 di GenBank (Thomsen et al. 2006) 6
3 Posisi penempelan primer terhadap gen FXI berdasarkan sekuens gen
FXI di GenBank (Marron et al. 2004) 7
4 Posisi penempelan primer gen SLC35A3 berdasarkan sekuens
GenBank (Kanae et al. 2005) 8
5 Hasil amplifikasi gen FXI pada gel agarose 1.5%, menghasilkan pita DNA berukuran 245 pb. M = marker DNA100 pb, 1-7 = sampel DNA
sapi bali 11
6 Hasil amplifikasi gen SLC35A3 pada gel agarose 1.5%, menghasilkan pita DNA berukuran 287 pb. M = marker DNA 100 pb, 1-7 = sampel
DNA sapi bali 12
7 Hasil deteksi kelainan gen FXI pada gel agarose 1.5%, menghasilkan pita DNA berukuran 245 pb. M = marker DNA100 pb, 1-7 = sampel
DNA sapi bali 13
8 Hasil pensejajaran sekuens gen FXI ekson 12 sapi bali dengan Bos
taurus 16
9 Hasil PCR-RFLP fragmen gen SLC35A3 ekson 4 dengan enzim restriksi PstI pada gel agarose 2%. M = marker DNA (100 pb), 1-6 =
sampel DNA sapi bali. 17
10 Hasil pensejajaran sekuens gen SLC35A3 ekson 4 pada sapi bali
dengan Bos taurus 19
DAFTAR LAMPIRAN
1 Bos taurus coagulation Factor XI gene, partial cds 26 2 Bos taurus coagulation Factor XI gene, complete cds 31 3 Bos taurus Solute Carrier Family 35 Member 3 (SLC35A3) gene,
complete cds 38
4 Bos taurus Solute Carrier Family 35 Member 3 (SLC35A3) gene, CVM
1
PENDAHULUAN
Latar Belakang
Sapi bali (Bos javanicus) merupakan salah satu sumberdaya genetik ternak asli Indonesia yang merupakan hasil domestikasi dari Banteng liar (Bibos banteng) (Martojo 2003) dengan populasi sebesar 4 789 521 ekor atau 32.31% dari seluruh sapi potong yang ada di Indonesia (BPS 2011). Sapi bali tersebar diseluruh Indonesia, dengan populasi terbesar terdapat di propinsi Sulawesi Selatan, Nusa Tenggara Timur, Nusa Tenggara Barat dan Bali dengan persentasi secara berurutan 19.94%, 14.28%, 14.04%, dan 13.31%.
Upaya pengembangan sapi bali dilakukan di beberapa wilayah Indonesia diantaranya adalah di BPTU-HPT Denpasar, BPT-HMT Serading Nusa Tenggara Barat dan Village Breeding Center (VBC) Kabupaten Barru Sulawesi Selatan. Balai inseminasi buatan berhasil menampung dan mendistribusikan semen dari sapi bali jantan unggul dalam upaya mendukung terciptanya sapi bali unggul, diantaranya adalah Balai Besar Inseminasi Buatan Singosari (BBIBS) dan Balai Inseminasi Buatan Baturiti (BIBD Baturiti).
Peran jantan dan betina sapi bali yang unggul dalam konteks perbibitan sangat dibutuhkan untuk meningkatkan mutu genetik mengingat sapi bali merupakan salah satu ternak asli Indonesia. Persyaratan bibit baik jantan maupun betina menjadi salah satu hal yang sangat penting dalam program perbibitan sapi bali, seperti tertuang dalam Permentan Nomor : 36/Permentan/OT.140/8/2006 tentang sistem perbibitan nasional yang menyebutkan bahwa salah satu syarat sapi bibit adalah bebas dari penyakit genetik. Kelainan genetik sangat tidak diharapkan keberadaannya dalam usaha perbibitan ternak, karena efek negatif yang ditimbulkannya, seperti penurunan kemampuan produksi dan reproduksi, anatomi tubuh yang abnormal dan beberapa kasus yang terjadi menyebabkan seekor ternak mati jika mutasi tersebut bersifat letal (Meydan et al. 2010).
Beberapa penelitian mengenai deteksi kelainan genetik pada ternak sapi terutama sapi perah telah banyak dilakukan diantaranya adalah Deficiency of Uridine Monophosphate Synthase (DUMPS) (Citek et al. 2006), Bovine Citrulinaemia (BC), Complex Vertebral Malformation (CVM), Factor XI Deficiency (FXID) dan Bovine Leucocyte Adhesion Deficiency (BLAD) (Meydan et al. 2010). Deteksi kelainan genetik CVM dan FXID pada sapi perah telah banyak dilakukan di beberapa negara karena CVM berpengaruh terhadap kemampuan reproduksi individu terutama dalam perkembangan fetus (Agerholm et al. 2004) dan FXID menyebabkan terjadinya berahi berulang (Ghanem et al. 2005) sehingga keberadaannya dalam usaha peternakan akan berakibat pada kerugian ekonomi dan perbaikan genetik ternak.
dilaporkan di Turki (Meydan et al. 2009), Amerika Serikat (Marron et al. 2004), India (Patel et al. 2007), Polandia (Gurgul et al. 2009), Jepang (Ghanem et al. 2005) dan Republik Czech (Citek et al. 2006). Selanjutnya diketahui kelainan genetik tersebut telah menyebar ke berbagai negara juga akibat penggunaan pejantan pembawa alel mutan FXID dalam program inseminasi buatan (Gentry et al. 1984). Informasi mengenai kelainan genetik pada sapi di Indonesia masih sangat terbatas. Deteksi kelainan genetik CVM dan FXID pada sapi potong terutama sapi bali sebagai salah satu sumber daya genetik ternak asli belum pernah dilakukan, sehingga penelitian mengenai deteksi kelainan genetik CVM dan FXID pada sapi bali perlu untuk dilakukan.
Perumusan Masalah
Upaya peningkatan mutu genetik sapi bali untuk menghasilkan bibit baik jantan maupun betina yang bebas dari kelainan genetik merupakan suatu keharusan untuk menjamin bahwa sapi bali yang digunakan sebagai bibit bebas dari kelainan genetik CVM dan FXID. CVM dan FXID adalah kelainan genetik yang bersifat resesif autosomal yang telah dideteksi pada sapi perah (Meydan et al. 2010). Deteksi kelainan genetik CVM dan FXID pada sapi bali belum pernah dilakukan di Indonesia sehingga penelitian ini perlu dilakukan karena sapi bali sebagai sumber daya genetik ternak asli Indonesia yang merupakan aset bangsa, bahkan dunia yang tak ternilai harganya.
Tujuan Penelitian
Penelitian ini bertujuan untuk melakukan deteksi terhadap ada tidaknya kelainan genetik FXID menggunakan gen FXI (Factor XI) dan CVM menggunakan gen SLC35A3 (Solute Carrier Family 35 Member 3) pada sapi bali serta sebagai informasi ilmiah tentang sapi bibit yang bebas dari kelainan genetik.
Manfaat Penelitian
Penelitian mengenai deteksi kelainan genetik pada sapi bali diharapkan mampu digunakan sebagai dasar kriteria seleksi sapi bibit khususnya sapi bali di pusat pembibitan sapi bali baik jantan maupun betina, selain itu juga dapat dijadikan sebagai dasar seleksi bibit sapi lokal, sapi impor maupun semen.
Ruang Lingkup Penelitian
dengan pendekatan Polymerase Chain-Reaction Fragment Length Polymorphism (PCR-RFLP) dengan menggunakan enzim restriksi PstI. Hasil genotipe yang diperoleh selanjutnya dilakukan analisis sekuensing.
2
TINJAUAN PUSTAKA
Kelainan Genetik
Beberapa istilah yang biasa digunakan untuk menyebut kelainan genetik diantaranya adalah genetic abnormality, genetic defect, genetic disorder, genetic anomaly dan syndrome. Kelainan genetik terjadi karena adanya mutasi secara acak pada genom. Kelainan genetik menyebar luas secara internasional seiring dengan perkembangan teknologi reproduksi transfer embrio dan inseminasi buatan. Kelainan genetik yang terjadi pada usaha peternakan menyebabkan kerugian secara ekonomi dan merusak usaha perbaikan genetik. (Whitlock et al.2008).
Kelainan genetik diwariskan dari tetua kepada keturunannya yang ditandai dengan kondisi yang menyimpang dari yang normal dengan efek negatif yang ditimbulkan baik secara fisik maupun fungsional yang berakibat pada kerugian yang besar pada usaha peternakan (Čítek et al. 2004). Alel karier dari gen mutan akan mensintesis protein non fungsional yang berakibat pada kerusakan perkembangan dan metabolisme tubuh (Uffo et al. 2009).
Perkawinan antara dua individu normal akan menghasilkan keturunan yang normal 100%, perkawinan antara dua individu heterozigot karier akan menghasilkan keturunan yang secara genetik homozigot normal sebesar 25%, heterozigot karier 50% dan homozigot resesif 25% sedangkan perkawinan antara individu karier dengan individu normal akan menghasilkan 50% homozigot normal dan 50% heterozigot karier (Buchanan 2010). Kelainan genetik yang terjadi pada sapi biasanya bersifat autosomal resesif, dimana kelainan genetik hanya akan muncul ketika terdapat dua gen resesif mutan secara genotipe pada individu (Meydan et al. 2010).
Kelainan Genetik FXID
Salah satu faktor penting dalam proses pembekuan darah adalah serin protease-factor XI atau disebut dengan Plasma Thromboplastin Antecedent (PTA), yang disintesis di dalam hati sebagai zymogen dan setelah konversi oleh enzim proteolitik berperan dalam proses intrinsik pembekuan darah (Gurgul et al. 2009). Kegagalan pembentukan serin protease-factor XI menyebabkan terjadinya kelainan genetik FXID yang dapat terjadi pada sapi jantan maupun betina (Gentry et al. 1975; Ghanem et al. 2008).
terjadinya insersi sebanyak 77 pasang basa pada ekson 12 gen FXI pada ternak heterozigot. Mutasi yang terjadi menyebabkan kodon stop prematur (TAA) (Marron et al. 1997; Ghanem et al. 2008) yang menyebabkan sebagian asam amino tidak disintesis (Mukhopadhyaya et al. 2006) sehingga protein yang terbentuk tidak lengkap (Gentry 1984). Pembentukan protein yang tidak sempurna menyebabkan protein tidak fungsional (Mukhopadyaya et al. 2006) sehingga ekspresi gen FXI terganggu dan serin protease tidak terbentuk (Ghanem et al. 2008). Kegagalan pembentukan serin protease mengakibatkan berbagai macam kelainan (Brush et al. 1987).
Kelainan genetik FXID pada seekor ternak dapat terjadi tanpa gejala yang terlihat maupun dengan gejala yang terlihat seperti pendarahan terus menerus, anemia, repeat breeding/berahi berulang (Gentry et al. 1980; Brush et al. 1987; Liptrap et al. 1995), lebih rentan terhadap pneumonia, mastitis dan metritis (Gentry et al. 1996), gangguan pembekuan darah, pertumbuhan lambat dan dysplasia (Ohba et al. 2008). Gejala kinis yang nampak pada sapi dengan defisiensi factor XI diantaranya adalah abortus, pedet mati setelah 48 jam, pada sapi homozigot terjadi pendarahan hebat pada paru-paru pasca partus, pada pedet homozigot terjadi pendarahan pada otak dan tulang belakang (Gentry et al. 1993). Kelainan genetik FXID pada individu homozigot ditandai dengan kadar aktivitas factor XI dalam plasma darah kurang dari 1% atau 2 ± 1%, heterozigot 38 ± 10% dan pada individu normal 94 ± 21% (Gentry et al. 1975; Gentry et al. 1993).
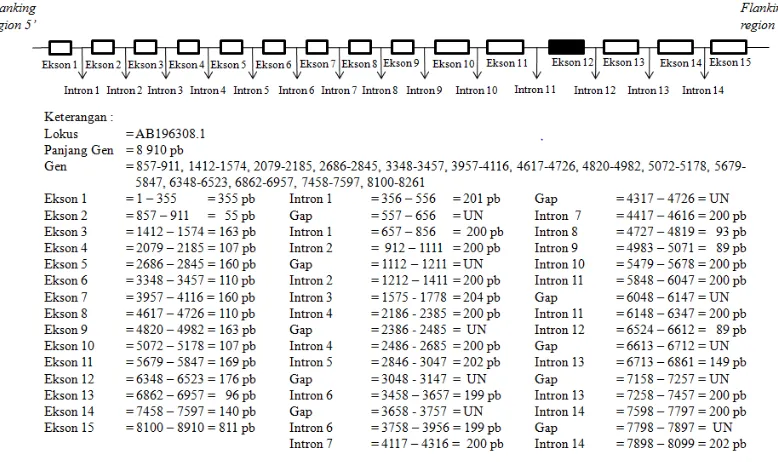
Gen FXI memiliki panjang 8 910 pasang basa, terletak pada kromosom 27 yang terdiri atas 15 ekson yang dipisahkan oleh 14 intron (Kunieda et al. 2005). Rekonstruksi gen FXI sesuai dengan kode akses GenBank AB196308.1 terlihat pada Gambar 1.
Defisiensi factor XI menyebabkan menurunnya kemampuan reproduksi ternak yang ditandai dengan ukuran garis tengah folikel yang kecil disertai dengan Gambar 1 Rekonstruksi gen FXI berdasarkan sekuens gen FXI di GenBank
konsentrasi estradiol yang lebih rendah dalam plasma saat ovulasi. Hal ini menyebabkan siklus berahi menjadi lebih lama dan proses luteolisis lambat. Sapi dengan FXID menunjukkan kemampuan beranak dan survival rate yang rendah (Liptrap et al. 1995). Mutasi pada factor XI menyebabkan berahi berulang (repeat breeding) yang berdampak pada kerugian ekonomi karena inefisiensi program inseminasi buatan (Ghanem et al. 2005).
Kelainan Genetik CVM
Kelainan genetik CVM pertama kali dikenali pada Danish Holstein Denmark pada tahun 1999 yang disebabkan oleh mutasi gen SLC35A3 yang mengkodekan uridine diphosphate-N-acetylglucosamine / UDP-GlcNAc transporter. Mutasi ini bersifat resesif autosomal dimana individu dalam kondisi homozigot resesif akan letal (Thomsen et al. 2006; Agerholm et al. 2004). CVM terjadi karena molekul protein UDP-N-acetylglucosamine atau golgi UDP-GlcNac transporter yang tidak sempurna di dalam aparatus golgi. Bovine SLC35A3 merupakan pengangkut Uridine diphosphate N-acetylglucosmaine atau UDP-GlcNAc dari sitosol/sitoplasma ke dalam lumen golgi sebelum proses glikosilasi protein, lemak dan proteoglican. UDP-GlcNAc berperan penting dalam struktur sitoskeleton (Patel 2012). Hal ini menunjukkan bahwa pembentukan tulang belakang dan tulang rusuk tergantung pada modifikasi karbohidrat di aparatus golgi (Thomsen et al. 2006).
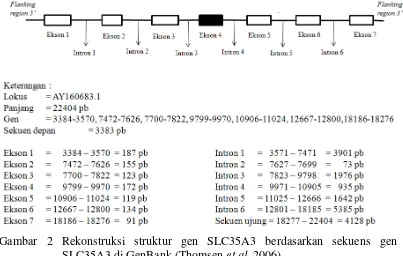
Gen SLC35A3 pada sapi memiliki ukuran 22 404 pasang basa (pb) yang terletak pada kromosom 3 terdiri atas 7 ekson yang dipisahkan oleh 6 intron, memiliki homologi 98% terhadap manusia dan anjing, 96% identik dengan SLC35A3 transporter tikus dan 83% identik dengan katak (Thomsen et al. 2006). Gen SLC35A3 merupakan anggota solute carrier family SLC35 yang mengangkut enzim nucleotide-sugars dari sitosol ke dalam lumen retikulum indoplasma dan atau aparatus golgi (Ishida et al. 2004). Dalam organel ini, nucleotide-sugars digunakan oleh glycosyltransferase untuk mensistesis rantai gula dari glikoprotein, glikolipid dan polimer karbohidrat. Rekonstruksi gen SLC35A3 sesuai dengan kode akses GenBank AY160683.1 dapat dilihat pada Gambar 2.
arthrogryposis antara tarsal dan metatarsophalangeal. Pemendekan leher biasa ditemukan pada pedet prematur, sapi hidup maupun pedet neonatal. Pada prinsipnya, identifikasi CVM diperoleh berdasarkan pada kelainan yang terjadi pada tulang belakang dan tulang rusuk. Kelainan genetik CVM pada suatu individu dapat diidentifikasi dengan kombinasi antara nekropsi dan genotiping (Agerholm et al. 2004).
Individu yang mengandung satu gen mutan akan menunjukkan penampilan yang normal namun individu tersebut merupakan individu yang karier yang akan mewariskannya kepada keturunannya. Individu yang teridentifikasi CVM akan ditandai dengan “CV” pada semua silsilahnya dan pada individu yang tidak karier akan ditandai dengan “TV”. Kode ini merupakan kode yang berlaku secara internasional (http://www.naab-css.org/education/CVMpressreleaseD.html, Grzy- bowski, 2003) (Čítek et al. 2004).
3
METODE
Lokasi dan Waktu Penelitian
Penelitian dilaksanakan di Laboratorium Genetika dan Molekuler Fakultas Peternakan IPB pada bulan Oktober hingga Desember 2013.
Bahan
Sampel Darah dan Semen
Sampel yang digunakan dalam penelitian ini berasal dari pusat pembibitan sapi bali (Tabel 1).
Tabel 1 Asal dan jenis sampel sapi yang digunakan dalam penelitian
Asal sampel Jenis sampel Jenis Kelamin Jumlah
Jantan Betina
BBIB Singosari Darah segar 28 0 28
BPT-HMT Serading NTB Darah segar 41 11 52
BPTU-HPT Denpasar Darah segar 98 59 157
BIBD Baturiti Semen beku 22 0 22
VBC Kab. Barru Sulawesi Selatan Darah segar 18 48 66
Total 207 118 325
Primer
Primer yang digunakan untuk mengamplifikasi gen FXI ekson 12 sesuai dengan Marron et al. 2004, dengan primer forward : 5’-CCC ACT GGC TAG GAA TCG TT-3’ dan primer reverse : 5’-CAA GGC AAT GTC ATA TCC AC-3’. Berdasarkan sekuens gen FXI dengan kode akses GenBank AH013749.2, primer menempel dari nukleotida ke 9 200 sampai dengan nukleotida ke 9 444, yang terletak pada sebagian intron K dan sebagian ekson 12. Individu yang mengalami mutasi akan terjadi insersi sebanyak 76 basa nukleotida yang dimulai dari nukleotida ke 9 402 (Marron et al. 2004). Posisi penempelan primer pada gen FXI dapat dilihat pada Gambar 3.
Alat
Primer yang digunakan untuk mengamplifikasi gen SLC35A3 ekson 4 sesuai dengan Kanae et al. 2005 dengan primer forward : 5’-CAC AAT TTG TAG GTC TCA CTG CA-3’ dan primer reverse : 5’-CGA TGA AAA AGG AAC CAA AAG GG-3’. Berdasarkan sekuens gen SLC35A3 dengan kode akses GenBank AY160683.1, primer gen SLC35A3 menempel pada runutan DNA yang dimulai dari nukleotida ke 9 848 hingga nukleotida ke 10 134, yang terletak di sebagian ekson 4 dan sebagian intron 4. Enzim restriksi PstI memotong pada situs CTGCA↓G. Posisi penempelan primer gen SLC35A3 dapat dilihat pada Gambar 4.
Keterangan :
huruf kecil : intron huruf besar : ekson
: awal dimulainya insersi pada gen FXI yang menyebabkan kelainan genetik FXID
Prosedur Penelitian
Ekstraksi DNA
Ekstraksi DNA dilakukan berdasarkan metode phenol-chloroform (Sambrook et al. 1989) yang dimodifikasi. Sampel darah diambil sebanyak 200 µl dan ditambahkan dengan 1 000 µl NaCl 0.2%, kemudian dihomogenkan menggunakan vortex dan didiamkan selama lima menit. Sampel disentrifugasi pada kecepatan 8 000 rpm selama lima menit kemudian bagian supernatan dibuang. Endapan yang ada ditambahkan dengan 10 µl Proteinase-K (5 mg/ml), 350 µl 1 x STE dan 40 µl SDS 10%, kemudian diinkubasi pada suhu 55 oC selama dua jam sambil dikocok perlahan. Larutan yang telah diinkubasi ditambahkan 40 µl NaCl 5 M, 400 µl phenol dan 400 µl CIAA kemudian dikocok perlahan pada suhu ruang selama satu jam. Kemudian larutan disentrifugasi dengan kecepatan 12 000 rpm selama lima menit sehingga terbentuk fase DNA yang ditandai dengan adanya lapisan bening yang nampak pada larutan paling atas. Fase DNA yang terbentuk diambil sebanyak 40 µl dan dipindahkan ke tabung 1.5 ml baru, ditambahkan dengan 40 µl NaCl 0.5 M dan 800 µl EtOH 96%, dihomogenkan, kemudian diistirahatkan overnight pada suhu -20 oC. Sampel DNA disentrifugasi pada kecepatan 12 000 rpm selama lima menit dan supernatan yang terbentuk dibuang, kemudian endapan ditiriskan sampai kering dan dilarutkan dalam 100 µl buffer TE 80%. DNA disimpan pada suhu -20 oC hingga sampel digunakan untuk amplifikasi PCR.
Pooling DNA
Pooling dilakukan dengan cara menggabungkan sampel DNA dengan konsentrasi tertentu dari beberapa individu untuk selanjutnya dilakukan analisis PCR. Satu pool terdiri dari lima sampel DNA. Sebanyak 1 µl sampel DNA masing-masing dari lima sampel DNA yang berbeda digabungkan ke dalam satu tabung eppendorf. Untuk memastikan bahwa semua sampel tercampur merata, maka pool DNA dihomogenkan dengan vortex kemudian menggunakan microsentrifuge. Sampel DNA yang sudah digabungkan kemudian dilanjutkan Keterangan :
huruf kecil : intron huruf besar : ekson
↓ : situs pemotongan enzim restriksi PstI
untuk amplifikasi PCR. Jika hasil amplifikasi PCR dalam satu pool DNA ditemukan adanya alel mutan, maka seluruh anggota pool diperiksa satu persatu.
Amplifikasi DNA
Amplifikasi in vitro dengan mesin thermal cycler dilakukan dengan kondisi suhu predenaturasi pada suhu 95 °C selama 5 menit, denaturasi pada suhu 95 °C selama 10 detik untuk 35 siklus, annealing pada suhu 60 °C selama 20 detik, elongasi pada suhu 72 °C selama 30 detik, dan elongasi akhir pada suhu 72 °C selama 5 menit.
Reaksi PCR dilakukan dengan volume 15 µl yang terdiri atas sampel DNA pool sebanyak 1 µl, primer forward-reverse 0.3 µl, Go Taq® Green Master mix 7.5 µl dan destilation water (DW) 6.2 µl. Produk PCR dielektroforesis menggunakan agarose 1.5%.
Elektroforesis Produk PCR Gen FXI dan Gen SLC35A3
Elektroforesis produk PCR gen FXI dilakukan dengan gel agarose 1.5% yang dibuat dengan cara melarutkan 0.45 g agarose dalam larutan 0.5 x TBE sebanyak 30 ml kemudian dipanaskan dalam microwave selama lima menit, selanjutnya dikocok dengan magnetic stirer dan ditambahkan EtBr sebanyak 2.5 µl. Larutan yang terbentuk didiamkan beberapa saat sampai uap airnya berkurang selanjutnya dituangkan ke dalam pencetak gel, sisir ditempatkan sesuai dengan tempatnya dan dibiarkan hingga mengeras. Selanjutnya sisir dicabut setelah agar mengeras dan terbentuk sumur-sumur gel yang dapat digunakan untuk proses elektroforesis selanjutnya.
Elektroforesis dilakukan dengan meletakkan gel yang sudah mengeras ke dalam gel tray elektroforesis yang berisi larutan buffer. Gel dipastikan terendam seluruhnya oleh larutan buffer. Produk PCR sebanyak 5 µl dimasukkan ke dalam sumur-sumur gel. Marker DNA 100 pb sebanyak 2 µl diletakkan ke dalam sumur paling pinggir sebagai penanda. Tahapan selanjutnya adalah dengan mengalirkan aliran listrik 100 V selama 30-45 menit. Proses elektroforesis dimulai ketika terlihat adanya gelembung-gelembung kecil pada sisi yang bermuatan negatif (katoda). Molekul DNA yang bermuatan negatif akan bergerak ke arah yang bermuatan positif (anoda) pada pH netral. Elektroforesis selesai sekitar 35-40 menit, selanjutnya gel diambil dan diletakkan ke dalam mesin UV transluminator untuk dilihat panjang pita DNA yang terbentuk.
Pita-pita DNA yang muncul dibandingkan dengan marker untuk diketahui panjang fragmennya, pita DNA dengan panjang pita yang sama menunjukkan genotipe yang sama. Produk PCR dari gen FXI akan menghasilkan pita DNA berukuran 245 pb dan gen SLC35A3 akan menghasilkan pita berukuran 287 pb. Mutasi pada gen FXI dapat diketahui jika hasil elektroforesis dari produk PCR diperoleh pita berukuran 320 pb. Deteksi mutasi pada gen SLC35A3 dilakukan dengan metode PCR-RFLP (Restriction Fragment Length Polymorphism).
Analisis PCR-RFLP
Kemudian produk PCR-RFLP disimpan dalam refrigerator hingga dilakukan elektroforesis.
Elektroforesis Produk PCR-RFLP Gen SLC35A3
Elektroforesis dilakukan terhadap produk PCR yang telah dipotong dengan enzim restriksi PstI menggunakan agarose 2% yang dibuat dengan cara melarutkan 0.6 g agarose dalam larutan 0.5 x TBE sebanyak 30 ml. Pembutan gel dilakukan dengan langkah yang sama yang dilakukan ketika genotiping pada gen FXI. Elektroforesis produk PCR yang telah dipotong dengan enzim PstI dilakukan dengan mengambil sampel produk PCR-RFLP sebanyak 7 µl kemudian dimasukkan ke dalam sumur-sumur gel. Marker DNA 100 pb diletakkan di sumur yang paling pinggir sebagai penanda. Pada sumur paling pinggir yang lainnya dimasukkan produk PCR yang tidak dipotong dengan enzim restriksi PstI sebagai pembanding untuk mengetahui bahwa proses pemotongan dengan enzim berhasil dilakukan. Tahapan elektroforesis yang sama dilakukan seperti ketika elektroforesis terhadap produk PCR dari gen FXI.
Pita-pita DNA yang terbentuk dibandingkan dengan marker dan produk PCR yang tidak dipotong dengan enzim restriksi PstI. Pita DNA dengan panjang pita yang sama menunjukkan genotipe yang sama. Sampel DNA yang berasal dari individu yang normal akan diperoleh ketika produk PCR berhasil dipotong oleh enzim restriksi PstI dan diperoleh pita DNA sepanjang 264 pb dan 23 pb (tidak nampak) sedangkan pada individu yang mengalami karier akan diperoleh pita DNA sepanjang 287 pb, 264 pb dan 23 pb (tidak nampak). Pada individu yang mutan akan diperoleh pita DNA dengan panjang pita yang sama dengan produk PCR yang tidak dipotong dengan enzim restriksi PstI dan akan diperoleh pita DNA sepanjang 287 pb (Mahfud 2009).
Sekuens Fragmen Gen FXI dan Gen SLC35A3
Sekuens dilakukan pada seluruh genotipe yang dihasilkan. Sekuensing dilakukan terhadap sampel baik yang beralel normal maupun yang karier dengan masing-masing berjumlah 2 sampel. Primer yang digunakan untuk sekuens adalah primer forward dan reverse. Sekuensing dilakukan dengan menggunakan mesin sekuenser oleh perusahaan sekuensing 1st Base.
Prosedur Analisis Data
Analisis Genotipe Gen FXI dan Gen SLC35A3
Data genotipe yang diperoleh dianalisis secara deskriptif terhadap ada tidaknya kelainan genetik CVM dan FXID pada sapi bali di kawasan pembibitan disertai dengan gambar dan tabel.
Analisis Hasil Sekuens
SLC35A3 ekson 4 dilakukan dengan menggunakan program Molecular Evolutionary Genetic Analysis (MEGA5).
4
HASIL DAN PEMBAHASAN
Amplifikasi Gen FXI dan gen SLC35A3
Amplifikasi Gen FXI
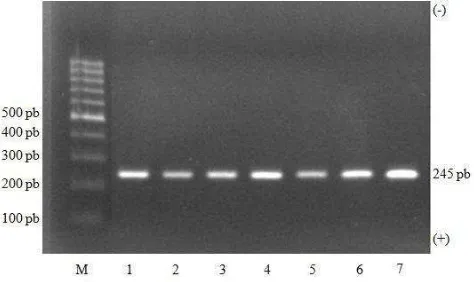
Seluruh sampel yang digunakan pada penelitian ini berhasil diamplifikasi dengan tingkat keberhasilan 100%. Berdasarkan referensi GenBank AH013749.2 primer yang digunakan terhadap gen FXI ekson 12 kromosom 27 (Marron et al. 2004) berhasil mengamplifikasi DNA sampel dan menghasilkan amplikon berukuran 245 pasang basa (pb) (Gambar 5).
Amplifikasi gen FXI pada sapi perah yang dilakukan oleh Meydan et al. (2010) berhasil dilakukan dengan suhu annealing/penempelan pada suhu 55 oC selama 60 detik. Bagheri et al. (2012) berhasil melakukan amplifikasi gen FXI pada suhu annealing 55 oC selama 1 menit. Sedangkan pada penelitian ini, amplifikasi fragmen gen FXI pada sapi bali berhasil dilakukan pada suhu annealing pada suhu 60 °C selama 20 detik. Dibandingkan dengan penelitian-penelitian sebelumnya, terdapat perbedaan kondisi PCR terutama suhu annealing yang digunakan untuk melakukan amplifikasi gen FXI. Tahapan-tahapan PCR yang sangat menentukan keberhasilan proses amplifikasi adalah suhu annealing/penempelan karena suhu annealing merupakan suhu optimum yang dibutuhkan yang memungkinkan primer untuk mengikat (anneal) dengan sekuens DNA komplemennya pada DNA tatakan (Nicolas 2004). Suhu annealing merupakan suhu yang sangat kritis yang sangat menentukan primer dapat menempel dengan baik dan diperoleh pita spesifik dari gen target. Perbedaan kondisi PCR disebabkan oleh mesin PCR yang digunakan, volume campuran PCR, konsentrasi primer dan gen target (Pelt-Varkuil et al. 2008) serta komposisi campuran penyusun reaksi PCR.
Amplifikasi Gen SLC35A3
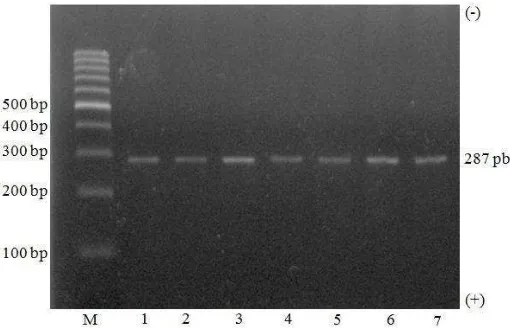
Amplifikasi DNA pada gen SLC35A3 ekson 4 dengan metode PCR-PIRA berhasil dilakukan pada keseluruhan sampel yang diperiksa dengan tingkat keberhasilan 100%. Primer yang digunakan berhasil mengamplifikasi gen SLC35A3 ekson 4 pada sapi bali dengan amplikon berukuran 287 pb (Gambar 6).
Amplifikasi terhadap gen SLC35A3 dilakukan dengan satu kali amplifikasi dengan menggunakann primer forward yang telah disisipkan dengan situs restriksi kedalamnya. Mahfud (2009) melakukan amplifikasi dua kali terhadap gen SLC35A3 pada sapi perah, amplifikasi pertama dilakukan dengan menggunakan primer forward sesuai dengan referensi GenBank nomor akses AY160683.1 dan amplifikasi kedua dilakukan dengan menggunakan primer forward yang telah disisipkan dengan situs restriksi kedalamnya. Amplifikasi yang dilakukan dua kali diperoleh hasil yang sama dengan yang hanya dengan satu kali amplifikasi. Amplifikasi gen SLC35A3 pada sapi bali berhasil dilakukan pada suhu annealing/penempelan pada suhu 60 °C selama 20 detik. Kondisi PCR yang berbeda juga terjadi ketika amplifikasi gen SLC35A3. Amplifikasi gen SLC35A3 pada sapi perah di Indonesia berhasil dilakukan pada suhu penempelan 56 oC selama 1 menit (Mahfud 2009). Kondisi PCR yang dilakukan oleh Meydan et al. (2010) berhasil mengamplifikasi gen SLC35A3 dengan suhu penempelan 52 oC selama 45 detik. Perbedaan yang terjadi selain karena mesin PCR yang digunakan dan komposisi reaksi PCR yang digunakan, mungkin disebabkan juga oleh susunan primer forward-reverse yang berbeda.
Deteksi Kelainan Genetik FXID dan CVM
Deteksi Kelainan Genetik FXID
pb sedangkan pada ternak mutan akan diperoleh pita berukuran 320 pb (Ghanem et al. 2008; Marron et al. 2004; Mahfud 2009). Patel et al. (2007) pada penelitiannya mengungkapkan diperoleh pita DNA berukuran 247 pada individu yang normal sedangkan pada individu yang karier diperoleh pita DNA berukuran 247 pb dan 324 pb karena adanya insersi 77 pasang basa pada gen pengontrol Factor XI.
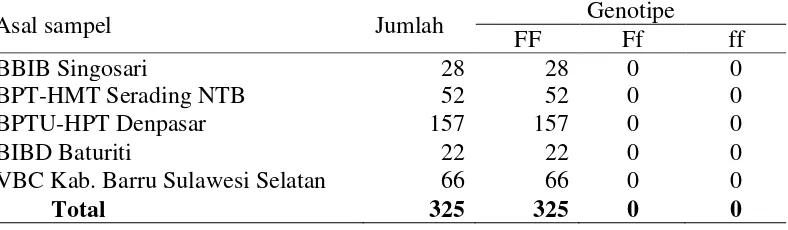
Mahfud (2009) dalam penelitiannya mengenai deteksi kelainan genetik FXID pada sapi perah di Indonesia menyatakan bahwa sapi dengan alel normal pada gen FXI merupakan sapi dengan genotipe FF (homozigot normal), sapi dengan alel karier merupakan sapi dengan genotipe Ff (heterozigot) sedangkan sapi dengan alel mutan merupakan sapi dengan genotipe ff (homozigot mutan). Seluruh sampel menunjukkan bahwa sapi bali yang diperiksa memiliki alel normal (genotipe homozigot normal : FF), tidak terdeteksi mengalami kelainan genetik FXID, tidak ditemukan genotipe Ff maupun genotipe ff. Sapi bali dengan genotipe ff (homozigot resesif) tidak akan ditemukan karena sapi bali yang digunakan sebagai sampel merupakan sapi yang masih hidup. Tabulasi hasil deteksi kelainan genetik FXID pada sapi bali di pusat pembibitan sapi bali dapat dilihat pada Tabel 2.
Tabel 2 Tabulasi hasil deteksi kelainan genetik FXID pada sapi bali
Asal sampel Jumlah Genotipe
FF Ff ff
BBIB Singosari 28 28 0 0
BPT-HMT Serading NTB 52 52 0 0
BPTU-HPT Denpasar 157 157 0 0
BIBD Baturiti 22 22 0 0
VBC Kab. Barru Sulawesi Selatan 66 66 0 0
Total 325 325 0 0
FXID pada sapi pertama kali ditemukan pada sapi Holstein di Ohio (Kociba et al. 1969) dan selanjutnya meluas ke beberapa negara yang lain karena program inseminasi buatan. Beberapa negara telah melakukan penelitian dan Gambar 7 Hasil deteksi kelainan gen FXI pada gel agarose 1.5%, menghasilkan pita DNA berukuran 245 pb. M = marker DNA100 pb, 1-7 = sampel DNA sapi bali
deteksi terhadap kejadian kelainan genetik FXID baik pada ternak perah maupun pada ternak lokal lainnya, seperti terlihat pada Tabel 3.
Penelitian mengenai kelainan genetik FXID di Indonesia pada sapi perah menunjukkan hasil bahwa dari 676 sampel yang diperiksa, ditemukan 5 individu mengalami karier FXID (frekuensi karier FXID/genotipe Ff = 0.74%; frekuensi alel mutan (f) = 0.004%). Tidak ditemukan individu mutan pada sapi perah di Indonesia karena sampel yang digunakan adalah sapi hidup (Mahfud 2009).
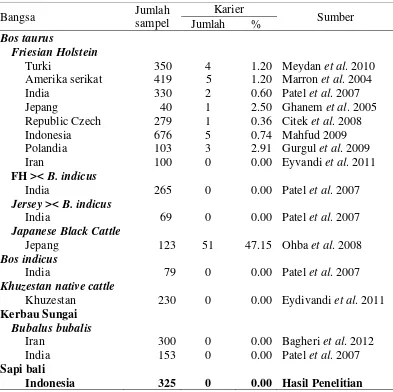
Beberapa penelitian dan deteksi kelainan genetik FXID yang dilakukan menunjukkan bahwa kelainan genetik FXID baru ditemukan pada sapi perah yang tersebar di beberapa negara dengan frekuensi kejadian yang sangat kecil, berkisar antara 0-2.91%. Kejadian FXID belum pernah dilaporkan terjadi pada kerbau sungai (Bagheri et al. 2012; Patel et al. 2007), Bos indicus (Patel et al. 2007), sapi lokal Khuzestan (Eyvandi et al. 2011) dan pada sapi persilangan antara Bos taurus dan Bos indicus (Patel et al. 2007). Kejadian FXID di Polandia terdeteksi pada pejantan unggul dengan nama Albit yang lahir pada tanggal 25 September 1998. Tabel 3 Data penelitian dan deteksi kelainan genetik FXID pada beberapa bangsa
di beberapa negara
Bangsa Jumlah
sampel
Karier
Sumber Jumlah %
Bos taurus
Friesian Holstein
Turki 350 4 1.20 Meydan et al. 2010 Amerika serikat 419 5 1.20 Marron et al. 2004
India 330 2 0.60 Patel et al. 2007
Jepang 40 1 2.50 Ghanem et al. 2005
Republic Czech 279 1 0.36 Citek et al. 2008
Indonesia 676 5 0.74 Mahfud 2009
Polandia 103 3 2.91 Gurgul et al. 2009
Iran 100 0 0.00 Eyvandi et al. 2011
FH >< B. indicus
India 265 0 0.00 Patel et al. 2007
Jersey >< B. indicus
India 69 0 0.00 Patel et al. 2007
Japanese Black Cattle
Jepang 123 51 47.15 Ohba et al. 2008
Bos indicus
India 79 0 0.00 Patel et al. 2007
Khuzestan native cattle
Khuzestan 230 0 0.00 Eydivandi et al. 2011
Kerbau Sungai
Bubalus bubalis
Iran 300 0 0.00 Bagheri et al. 2012
India 153 0 0.00 Patel et al. 2007
Sapi bali
Pejantan ini telah diproduksi semennya sebanyak 8 960 dosis dan telah diinseminasikan kepada 1 576 ekor betina, 78% betina yang dikawini tidak menunjukkan tanda-tanda kebuntingan setelah 56 hari, sehingga akhirnya semen yang dihasilkan dari pejantan tersebut tidak digunakan lagi untuk inseminasi buatan selanjutnya (Gurgul et al. 2009).
Kejadian FXID pada Japanese black cattle ditemukan individu-individu dengan alel mutan namun menunjukkan gejala klinis yang normal, tidak terjadi pendarahan yang berarti serta tidak terjadi penurunan performans pada keturunannya. Japanese black cattle yang mengalami kelainan FXID menunjukkan homeostatic deficiency, keterlambatan pertumbuhan dan hip displasia (Ohba et al. 2008). Kelainan genetik ini menyebar luas di Jepang karena penggunaan semen pada program inseminasi buatan. Kelainan FXID pada Japanese Black Cattle disebabkan oleh insersi 15 nukleotida pada ekson ke 9 dari gen FXI (Kunieda et al. 2005), sehingga kasus FXID antara sapi Holstein dan Japanese Black Cattle adalah kasus yang berbeda, yang mungkin disebabkan oleh perbedaan mutasi pada gen FXI, perbedaan lingkungan pemeliharaan dan pembibitan (Ohba et al. 2008).
Analisis Hasil Sekuensing Gen FXI
Sekuensing DNA dilakukan untuk mengkonfimasi keberadaan alel mutan sapi bali. Analisis BLAST dilakukan untuk melihat kemiripan yang terdapat pada dua atau lebih sekuens gen. Hasil analisis sekuens gen FXI menunjukkan bahwa gen FXI sapi bali memiliki kesamaan yang tinggi dengan sekuens gen FXI pada Bos taurus yang terdapat di GenBank (kode akses AH013749.2) dengan tingkat kesamaan (homologi) sebesar 100% dan diperoleh runutan basa nukleotida sepanjang 245 pb. Hal ini menunjukkan bahwan gen FXI pada sapi bali identik dengan gen FXI pada Bos taurus. Pada penelitian ini, tidak ditemukan individu karier maupun mutan, sehingga tidak ditemukan adanya insersi basa nukleotida pada runutan basa pada sampel yang disekuens seperti terlihat pada Gambar 8.
Hasil sekuensing pada sampel yang dianalisis secara individu maupun dalam pooling/gabungan menunjukkan hasil yang sama. Hal ini menunjukkan bahwa metode pool DNA yang digunakan dalam penelitian berhasil digunakan untuk melakukan deteksi mutasi gen FXI pada sapi bali. Jika dalam satu pool yang dianalisis ditemukan alel yang berbeda, maka seluruh anggota dalam pool tersebut dianalisis satu per satu.
Marron et al. (2004) menyatakan bahwa kelainan genetik FXID diketahui ketika sekuens dari individu yang normal dibandingkan dengan individu mutan, ditemukan adanya insersi sebanyak 76 pasang basa pada ekson 12 gen FXI. Mutasi insersi ini terdiri atas runutan poliadenin yang tidak sempurna yang diikuti oleh berulangnya 14 pasang basa pada sekuen normal di akhir insersi pada individu mutan (GAA ATA ATA ATT CA). Translasi dari sekuens yang mengalami mutasi ini menyebabkan terjadinya stop kodon (TAA) pada empat nukleotida pertama. Hal ini menyebabkan protein tidak terbentuk karena kehilangan serin protease yang dikodekan oleh ekson 13-15 (Asakai et al. 1987).
(BPTU-HMT Denpasar, BPT-HPT Serading Nusa Tenggara Barat, VBC Kabupaten Barru Sulawesi Selatan) dan balai inseminasi buatan (Balai Besar Inseminasi Buatan Singosari dan Balai Inseminasi Buatan Baturiti) merupakan sapi bali yang normal dan tidak terdeteksi kelainan genetik FXID.
Deteksi Kelainan Genetik CVM
Mutasi substitusi satu basa pada gen SLC35A3 menyebabkan terjadinya kelainan genetik CVM yang merupakan mutasi yang terjadi secara resesif Keterangan :
AB19608.1 : kode akses GenBank sekuens gen FXI Bos taurus complete cds
AH013749.2 : kode akses GenBank sekuens gen FXI Bos taurus partial cds
Marron et al : sekuens individu mutan (akses GenBank tidak diketahui)
: stop kodon prematur
autosomal dan bersifat lethal pada sapi. Hasil pemotongan gen SLC35A3 ekson 4 pada sapi bali dengan menggunakan enzim restriksi PstI ditunjukkan oleh fragmen terpotong pada posisi 264 pb dan 23 pb (tidak nampak) (Gambar 9). Produk PCR dari seluruh sampel yang diperiksa berhasil dipotong dengan menggunakan enzim restriksi PstI. Enzim restriksi PstI yang digunakan pada memotong runutan nukleotida pada situs 5’ CTGCA↓G 3’. Hal ini berarti bahwa jika tidak terjadi perubahan basa G (guanin) pada fragmen gen SLC35A3 maka produk PCR yang dihasilkan akan dipotong oleh enzim restriksi PstI. Demikian juga sebaliknya jika terjadi perubahan basa G (guanin) menjadi basa yang lain (T/timin, C/sitosin atau A/adenin) maka produk PCR tidak akan dipotong oleh enzim restriksi PstI.
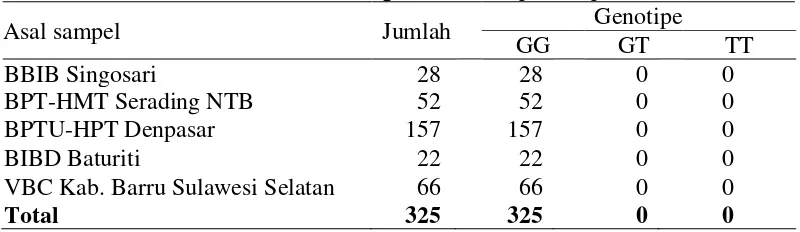
Kelainan CVM diketahui ketika produk PCR dari gen SLC35A3 tidak dapat dipotong dengan enzim restriksi PstI dan menghasilkan pita DNA berukuran 287 pb (alel mutan/genotipe TT). Jika pada hasil elektroforesis diperoleh 2 pita sepanjang 264 pb dan 23 pb maka individu tersebut merupakan individu yang normal (alel normal/genotipe GG). Jika pada hasil elektroforesis diperoleh 3 pita DNA yang berukuran 287 pb, 264 pb dan 23 pb, maka individu tersebut merupakan individu karier CVM (alel karier/genotipe GT) (Mahfud 2009; Gábor et al. 2012). Hasil penelitian menunjukkan bahwa hanya diperoleh satu macam genotipe yaitu genotipe GG, tidak ditemukan adanya individu karier CVM (genotipe GT) maupun individu mutan (genotipe TT) pada seluruh sampel yang diperiksa (Tabel 4).
Tabel 4 Tabulasi hasil deteksi kelainan genetik CVM pada sapi bali
Asal sampel Jumlah Genotipe
GG GT TT
BBIB Singosari 28 28 0 0
BPT-HMT Serading NTB 52 52 0 0
BPTU-HPT Denpasar 157 157 0 0
BIBD Baturiti 22 22 0 0
VBC Kab. Barru Sulawesi Selatan 66 66 0 0
Total 325 325 0 0
Kelainan genetik CVM pertama kali diidentifikasi pada Danish Holstein di Denmark yang terdeteksi pada elite bull Carlin-M Ivanhoe Bell yang dikenal luas mampu menghasilkan keturunan dengan produksi susu yang tinggi. Kelainan genetik ini dengan cepat menyebar ke beberapa negara karena penggunaan semen dari pejantan yang ternyata karier. Carlin-M Ivanhoe Bell juga karier BLAD. Gen yang menyebabkan kelainan genetik CVM dan BLAD berada pada kromosom yang berbeda sehingga pewarisan kelainan genetik berlangsung secara terpisah (Thomsen et al. 2006). Kelainan genetik CVM biasa ditemukan pada sapi holstein namun, tidak menutup kemungkinan terjadi juga pada sapi dari bangsa lain, baik jantan maupun betina, dewasa maupun pedet (EPS 2006). Beberapa penelitian dan deteksi kelainan genetik CVM pada beberapa negara pada beberapa spesies disajikan pada Tabel 5.
Tabel 5 Beberapa penelitian dan deteksi kelainan genetik CVM pada beberapa negara pada berbagai spesies
Bangsa Jumlah
sampel
Karier
Sumber Jumlah %
Bos taurus
Friesian Holstein
Republic Czech 517 21 4.00 Citek et al. 2006 Denmark no data no data 31.00 Thomsen et al. 2006 Polandia 605 150 24.79 Rusc & Kamiński 2007
Jepang 200 26 13.00 Ghanem et al. 2008
Slovakia 47 4 8.50 Gábor et al 2012
Swedia 228 58 23.00 Berglund et al. 2004
Turki 350 12 3.40 Meydan et al. 2010
China 587 56 9.54 Zhang et al. 2012
China 154 24 15.58 Wang et al. 2012
Indonesia 483 0 0.00 Mahfud 2009
Iran 144 0 0.00 Rezaee et al. 2008
Czech Pied
Republic Czech 146 0 0.00 Citek et al. 2006
Charolais
Republic Czech 8 0 0.00 Citek et al. 2006
Limousin
Republic Czech 6 0 0.00 Citek et al. 2006
Simmental
Republic Czech 2 0 0.00 Citek et al. 2006
Belgian blue
Republic Czech 9 0 0.00 Citek et al. 2006
Aberden Angus
Republic Czech 1 0 0.00 Citek et al. 2006
Sapi bali
Tabel 5 menunjukkan bahwa sapi bali di pusat pembibitan di Indonesia yang diperiksa tidak terdeteksi kelainan genetik CVM. Republik Chezh yang telah melakukan monitoring secara genetik terhadap populasi sapi pada negara tersebut juga tidak ditemukan kejadian CVM pada bangsa sapi selain Holstein (Čítek et al. 2006). Hal ini mungkin disebabkan karena jumlah sampel yang digunakan sangat sedikit. Walaupun kejadian CVM baru ditemukan pada sapi perah, namun hal ini harus terus diwaspadai dalam usaha pembibitan sapi.
Analisis Hasil Sekuensing Gen SLC35A3
Hasil analisis sekuens gen SLC35A3 menunjukkan bahwa gen SLC35A3 sapi bali memiliki kesamaan yang tinggi dengan sekuens gen SLC35A3 pada Bos taurus yang terdapat di Genbank (kode akses AY160683.1) dengan tingkat kesamaan (homologi) sebesar 100% dan diperoleh runutan basa nukleotida sepanjang 287 pb. Alignment/pensejajaran runutan nukleotida sapi bali dengan Bos taurus menunjukkan bahwa basa CT yang diinsersikan pada primer forward berhasil membuat situs restriksi baru sehingga dapat dikenali oleh enzim pemotong PstI. Tidak terjadi perubahan basa G (guanin) menjadi basa T (timin) pada runutan gen SLC35A3 ekson 4 pada sapi bali (Gambar 10).
Keterangan :
AY160683.1 : kode akses GenBank sekuens Bos taurus normal HM183013.1 : kode akses GenBank sekuens Bos taurus mutan
Kelainan genetik CVM terjadi karena adanya mutasi titik (point mutation)/missense mutation yang merupakan mutasi yang disebabkan karena terjadinya perubahan basa yang menyebabkan perubahan asam amino (Thomsen 2006). Hasil sekuens cDNA dari gen SLC35A3 menunjukkan terjadinya perubahan basa guanin (G) menjadi basa timin (T) pada nukleotida ke 559 pada sapi yang mengalami kelainan genetik CVM (Kanae et al. 2005). Mutasi ini menyebabkan terjadinya perubahan kodon yang menyebabkan perubahan asam amino valine (GTT/Val) pada posisi ke-180 menjadi phenylalanine (TTT/Phe) (Thomsen et al. 2006). Individu yang normal, pada runutan basa nukleotidanya menunjukkan bahwa pada basa nukleotida ke 9871 atau pada nukleotida ke 24 hasil sekuens dengan PCR-PIRA pada penelitian merupakan basa G (guanin). Sedangkan pada individu yang karier atau mutan, basa G (guanin) mengalami substitusi dengan basa T (timin). Hasil sekuens gen SLC35A3 pada sapi bali tidak ditemukan adanya mutasi basa guanin menjadi basa timin. Hal ini menunjukkan bahwa sampel sapi bali yang diambil dari pusat pembibitan sapi bali di Indonesia bebas dari kelainan genetik CVM.
Implementasi Deteksi Kelainan Genetik FXID dan CVM pada Sapi Bali di Pusat Pembibitan Sapi Bali di Indonesia
Deteksi kelainan genetik FXID dan CVM pada sapi bali diperoleh hasil bahwa kedua kelainan genetik tersebut tidak ditemukan pada sapi bali yang berada di pusat pembibitan sapi bali (BPTU-HPT Denpasar, BPT-HMT Serading NTB dan VBC Kabupaten Barru Sulawesi Selatan) serta di balai inseminasi buatan (BBIB Singosari dan BIBD Baturiti). Hal ini mungkin disebabkan karena sampel yang diperiksa berasal dari pusat-pusat pembibitan sapi bali dimana sapi bali yang yang berada pada lokasi-lokasi tersebut telah mengalami proses seleksi sebelumnya, sehingga kemungkinan untuk diperolehnya individu yang karier maupun individu mutan sangat kecil. Selain itu, individu yang karier memiliki kemampuan bertahan dan fitness yang rendah sehingga tidak mampu bertahan hingga sapi dewasa.
Meskipun tidak ditemukan kelainan genetik tersebut, deteksi kelainan genetik pada sapi bali khususnya dan sapi-sapi bibit pada umumnya baik sapi lokal maupun sapi impor di pusat pembibitan baik pemerintah maupun swasta sangat penting untuk dilakukan. Deteksi kelainan genetik juga perlu dilakukan pada semen dan embrio yang akan diimpor ke Indonesia untuk memastikan dan memberikan jaminan bahwa semen dan embrio yang akan digunakan merupakan semen dan embrio yang bebas dari kelainan genetik.
betina yang akan digunakan dalam program progeny test merupakan sasaran utama untuk dilakukan deteksi terhadap kelainan genetik. Dengan demikian, penyebaran kelainan genetik pada sapi-sapi bibit di Indonesia dapat dihindari.
Hal yang tidak kalah pentingnya adalah, rekording. Rekording yang dilakukan dalam program surveilan yang intensif bertujuan untuk mengetahui lebih awal kelainan-kelainan yang terjadi berdasarkan karakteristik morfologi. Program surveilan akan berhasil dengan baik jika ada laporan tentang kelainan-kelainan yang terjadi, koleksi dan penyimpanan sampel yang tepat serta informasi pedigree (Whitlock et al. 2008). Identifikasi terhadap sapi-sapi dengan kecenderungan menghasilkan keturunan yang mati baik abortus maupun mati sesaat setelah lahir sangat diperlukan untuk mendukung upaya pencegahan dan penyebaran kelainan genetik pada usaha peternakan (Whitlock et al. 2008).
5
SIMPULAN DAN SARAN
Simpulan
Penelitian ini menunjukkan bahwa tidak ditemukan adanya karier FXID maupun CVM pada (BPTU-HMT Denpasar, BPT-HPT Serading NTB, VBC Kabupaten Barru Sulawesi Selatan) dan balai inseminasi buatan (Balai Besar Inseminasi Buatan Singosari dan BIBD Baturiti). Seluruh sapi bali yang berada di pusat pembibitan dan balai inseminasi buatan di Indonesia merupakan individu dengan genotipe normal. Pendekatan molekuler dapat digunakan untuk melakukan deteksi terhadap kelainan genetik pada sapi dan dapat diterapkan pada pusat-pusat pembibitan di Indonesia baik pemerintah maupun swasta.
Saran
Upaya yang dilakukan untuk mencegah penyebaran kelainan genetik yang mungkin terjadi pada usaha peternakan maupun pembibitan adalah dengan melakukan identifikasi jika ditemukan ternak dengan morfologi yang menyimpang dari normal dengan melakukan pengecekan informasi pedigree, jika diperlukan dilakukan pengujian secara genetik terhadap organ tubuh dari induk, pejantan, pedet/sapi yang mengalami kelainan maupun semen yang digunakan. Ternak yang diketahui mengalami kelainan genetik sebaiknya dikeluarkan dari populasi.
Sebaiknya dilakukan juga deteksi kelainan genetik yang lain seperti BLAD, BC dan DUMPS (Meydan et al. 2010).
Ucapan Terimakasih
Laboratorium Genetika Molekuler Ternak Fakultas Peternakan Institut Pertanian Bogor.
DAFTAR PUSTAKA
Agerholm JS, Bendixen C, Arnbjerg J, Andersen O. 2001. Complex vertebral malformation in Holstein calves. J. Vet Diagn Invest 13:283-289.
Agerholm JS, Bendixen C, Arnbjerg J, Andersen O. 2004. Morphological variation of complex vertebral malformation in Holstein calves. J. Vet Diagn Invest 16:548-553.
Asakai R, Davie EW, Chung DW. 1987. Organization of the gene for human Factor XI. Biochemistry 26:7221-7228.
Bagheri S, Amirinia C, Chamani M, Aminafshar M, Sadeghi AA, Seyedabadi HR. 2012. Identification of Factor XI deficiency in Khuzestan buffalo population of Iran. Global Veterinaria 8(6):598-600.
Berglund B, Persson A, Stalhammar H. 2004. Effect of Complex vertebral malformation on fertility in Swedish Holstein cattle. Acta Vet Scand 45:161-165.
[BPS] Badan Pusat Statistik. 2011. Pendataan sapi potong, sapi perah dan kerbau 2011 (PSPK2011). Badan Pusat Statistik : 05303011.
Buchanan, DS. 2010. Genetic defects in cattle. Observational study. [Internet]. [Diunduh 2013 Oktober 17]; North Dakota (US). Tersedia pada http://www.ag.ndsu.edu/williamscountyextension/agriculture/livestock/gen tic-defects-in-cattle/files/genetic-defects-afflict-all-types-of-farm-livestock-2.
Brush PJ, Anderson PH, Gunning RF. 1987. Identification of Factor XI deficiency in Holstein-Friesian cattle in Britain. Vet Rec 121:14-17.
Chu Q, Sun D, Yu Y, Zhang Y, Zhang Y. 2008. Identification of complex vertebral malformation carriers in Chinese Holstein. J. Vet Diagn Invest 20:228-230.
Churchill GA, Giovannoni JJ, Tanksley SD. 1993. Pooled-sampling makes high-resolution mapping practical with DNA markers. Proc Natl Acad Sci 90:16-20.
Čítek J, Bláhová B. 2004. Recessive disorders-a serious health hazard?. J. Appl Biomed 2:187-194.
Čítek J, Rehout V, Hajkova, Pavkova J. 2006. Monitoring of genetic health of cattle in the Czech Republic. Veterinarni Medicina 51(6):333-339.
[EPS] European Patent Specification. 2006. Genetic test for the identification of carriers of complex vertebral malformations in cattle. WO 2002/040709 (23.05.2002 Gazette 2002/21).
Eyvandi C, Amirinia C, Kashan NE, Chamani M, Fayazi J. 2011. Study of Factor XI deficiency in Khuzestan cattle population of Iran. African J. Biotechnol 10(4):718-721.
Gentry PA, Crane S, Lotz F. 1975. Factor XI (plasma thromboplastin antecedent) deficiency in cattle. Can Vet J 16(6):160-163.
Gentry PA, Black BD. 1980. Prevalence and inheritance of Factor XI (plasma thromboplastin antecedent) deficiency in cattle. J Dairy Sci 63:616-620. Gentry PA. 1984. The relationship between Factor XI coagulant and Factor XI
antigenic activity in cattle. Can J Comp Med 48:58-62.
Gentry PA, Ross ML. 1993. Coagulation Factor XI deficiency in holstein cattle : expression and distribution of Factor XI activity. Can J Vet Res 57:242-247.
Gentry PA, Overton KM, Robinson JL. 1996. Bovine Factor XI deficiency. Illinois Dairy Report: 32-35.
Ghanem ME, Nishibori M, Nakao T, Nakatani K, Akita M. 2005. Factor XI mutation in a Holstein cow with repeat breeding in Japan. J. Vet. Med. Sci. 67(7):713-715.
Ghanem ME, Akita M, Suzuki T, Kasuga A, Nishibori M. 2008. Complex vertebral malformation in Holstein cow in Japan and its inheritance to crossbred F1 generation. Anim. Reprod. Sci 103:348-354.
Gurgul A, Rubiś, Slota E. 2009. Identification of carriers of the mutation causing coagulation Factor XI deficiency in Polish Holstein-Friesian cattle. J. Appl. Genet. 50(2):149-152.
Haton BM, Beever j. Robinson JL. 2000. Mutation that causes Factor XI deficiency in Holstein cattle. Illini DairyNet Papers:171-172.
Ishida N, Kawakita M. 2004. Molecular physiology and pathology of the nucleotide sugar transporter family (SLC35). Pfugers Arch. 447:768-775. Kanae Y, Endoh D, Nagahata H, Hayashi M. 2005. A method for detecting complex vertebral malformation in Holstein calves using polymerase chain reaction-primer introduced restriction analysis. J. Vet. Diagn. Invest. 17:258-262.
[Kementan] Kementerian Pertanian. 2006. Peraturan Menteri Pertanian Nomor : 36/Permentan/OT.140/8/2006 : Sistem perbibitan ternak nasional.
Kociba GD, Ratnoff OD, Loeb WF, Wall RL, Heider LE. 1969. Bovine thromboplastin antecedent (Factor XI) deficiency. J. Lab. Clin. Med. 74:37.
Kunieda M, Tsuji T, Abbasi AR, Khalaj M, Ikeda M, Miyadera K, Oqawa H, Kunieda T. 2005. An insertion mutation of the bovine FXI gene is responsible for Factor XI deficiency in Japanese black cattle. Mamm. Genome. 16(5):383-9. (Abstr.).
Liptrap RM, Gentry PA, Ross ML, Cummings E. 1995. Preliminary findings of altere follicular activity in Holstein cows with coagulation Factor XI deficiency. Vet. Res. Commun. 19:463-471.
Mahfud K. 2009. Deteksi dini kelainan genetik complex vertebral malformation dan Factor XI deficiency pada sapi perah Friesian-Holstein. [Thesis]. Bogor (ID). Institut Pertanian Bogor.
Martojo H. 2003. A simple selection program for smallholder Bali Cattle Farmers. In : Strategies to improve Bali Cattle in eastern indonesia. K. Entwistle and D.R. Lindsay (Eds). ACIAR Proc. No. 110. Canberra.
Meydan H., Yildiz MA, Agerholm JS. 2010. Screening for bovine leukocyte adhesion deficiency, deficiency of uridine monophosphate synthase, complex vertebral malformation, bovine citrullinemia, and Factor XI deficiency in Holstein cows reared in Turkey. Acta Veterinaria Scandinavia. 52:56.
Mukhopadhyaya PN, Jha M, Muraleedharan P, Rathod RN. 2006. Simulation of normal, carrier and affected controls for large scale genotiping of cattle for FXI deficiency. Genet Mol Res 5(2):323-332.
Nielsen US, Aamand GP, Andersen O. 2003. Effect of complex vertebral malformation on fertility traits in Holstein cattle. Livest Prod Sci 79:233-238.
Nicholas FX. 2004. Pengantar ke Genetika Veteriner. Muladno, penerjemah; Arianti L, editor. Bogor (ID) : Penerbit Pustaka Wirausaha Muda. Tejemahan dari : Introduction to Veterinary Genetics.
Ohba Y, Takasu M, Nishi N, Takeda E, Maeda S, Kunieda T, Kitagawa H. 2008. Pedigree analysis of Factor XI deficiency in Japanese black cattle. J. Vet. Med. Sci 70(3):297-299.
Patel RK, Soni KJ, Chauhan JB, Singh KM, Rao KRS. 2007. Factor XI deficiency in Indian Bos taurus, Bos indicus, Bos taurus x Bos indicus crossbreds and Bubalus bubalis. Genet. Molec. Biol., 30(3):580-583. Patel RK. 2012. Complex Vertebral Malformation (CVM) in Holstein.
Rezaee AR, Nassiry MR, Valizadeh R, Tahmoorespour M, Javadmanesh A, Zarei A, Janati H. 2008. Study of complex vertebral malformation disorder in Iranian Holstein bulls. World J. Zoo. 3(2):36-39.
Robinson JL, Beever JE, Leuw ND, Lewin HA, Gillings M. 1997. Ed. New York: Cold Spring Harbour Laboratory Press.
Sham P, Bader J, Craig I, O’Donovan M, Owen M. 2002. DNA pooling a tool for large-scale association studies. Nat. Rev. Genet 3(11):862-871.
Thomsen B, Horn P, Panitz F, Bendixen E, Anette HP. 2006. A missense mutation in the bovine SLC35A3 gene, encoding a UDP-N-acetylglucos-amine transporter, causes complex vertebral malformation. Genome Res. 16:97-105.
Uffo O, Acosta A. 2009. Molecular diagnosis and control strategies for relevant genetic disease of cattle. Biotech Apl. 26:204-208.
Voet D, Voet JG. 1995. Biochemistry. 2nd Ed. Canada : John Willey & Sons Inc.
complex vertebral malformation in Holstein cattle. J. Vet. Diagn. Invest. 24(3):568-571.
Whitlock BK, Kaiser L, Maxwell HS. 2008. Heritable bovine fetal abnormalities. Theriogenology 70:535-549.
Yang Y, Zhang J, Hoh J, Matsuda F, Xu P, Lathrop M, Jurg O. 2003. Eficiency of single-nucleotide polymorphism haplotype estimation from pooled DNA. Proc. Natl. Acad. Sci. 100(12):7225-7230.
Zhang Y, Fan X, Sun D, Wang Y, Yu Y, Xie Y, Zhang S, Zhang Y. 2012. A novel method for rapid and reliable detection of complex vertebral malformation and bovine leukocyte adhesion deficiency in Holstein cattle. J. Anim. Sci. & Biotech. 3(24):2-6.
Lampiran 1 Bos taurus coagulation Factor XI gene, partial cds
LOCUS SEG_AY570503S 10336 bp DNA linear MAM 29-JUL-2011 DEFINITION Bos taurus coagulation Factor XI gene, partial cds.
ACCESSION AH013749 AY570503 AY570504 VERSION AH013749.2 GI:342160192 KEYWORDS .
SOURCE Bos taurus (cattle) ORGANISM Bos taurus
Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Mammalia; Eutheria; Laurasiatheria; Cetartiodactyla; Ruminantia; Pecora; Bovidae; Bovinae; Bos.
REFERENCE 1 (bases 1 to 10336)
AUTHORS Marron,B.M., Robinson,J.L., Gentry,P.A. and Beever,J.E.
TITLE Identification of a mutation associated with Factor XI deficiency in Holstein cattle
JOURNAL Anim. Genet. 35 (6), 454-456 (2004) PUBMED 15566468
REFERENCE 2 (bases 1 to 10336)
AUTHORS Marron,B.M., Robinson,J.L., Gentry,P.A. and Beever,J.E. TITLE Direct Submission
JOURNAL Submitted (09-MAR-2004) Animal Sciences, University of Illinois at Urbana-Champaign, 1201 W. Gregory Drive, Urbana, IL 61801, USA COMMENT On or before Jul 29, 2011 this sequence version replaced
gi:45861246, gi:45861247, gi:45861245.
misc_feature join(8100..8242,9300..9475,10269..>10336)
misc_feature join(7515..7521,7615..7777,7867..7951)
misc_feature join(4242..4251,4960..5119,7412..7496)
/note="Subfamily of PAN/APPLE-like domains; present in
misc_feature join(1456..1462,2358..2517,4142..4226)
/note="Subfamily of PAN/APPLE-like domains; present in
misc_feature join(8097..8242,9300..9475,10269..>10336)
/note="Trypsin-like serine protease; Region: Tryp_SPc;
misc_feature order(7668..7670,7674..7676,7707..7709) /note="putative binding site; other site" /db_xref="CDD:238533"
misc_feature order(5010..5012,5016..5018,5049..5051) /note="putative binding site; other site" /db_xref="CDD:238533"
/number=D