Jurnal Biologi Indonesia 9(2): 2013
Jurnal Biologi Indonesia diterbitkan oleh Perhimpunan Biologi Indonesia. Jurnal ini memuat hasil penelitian ataupun kajian yang berkaitan dengan masalah biologi yang diterbitkan secara berkala dua kali setahun (Juni dan Desember).
Editor
Ketua
Prof. Dr. Ibnu Maryanto Anggota
Prof. Dr. I Made Sudiana Dr. Deby Arifiani Dr. Izu Andry Fijridiyanto
Dewan Editor Ilmiah
Dr. Abinawanto, F MIPA UI Dr. Achmad Farajalah, FMIPA IPB
Dr. Ambariyanto, F. Perikanan dan Kelautan UNDIP Dr. Aswin Usup F. Pertanian Universitas Palangkaraya Dr. Didik Widiyatmoko, PK Tumbuhan, Kebun Raya Cibodas-LIPI
Dr. Dwi Nugroho Wibowo, F. Biologi UNSOED Dr. Parikesit, F. MIPA UNPAD
Prof. Dr. Mohd.Tajuddin Abdullah, Universiti Malaysia Sarawak Malaysia Assoc. Prof. Monica Suleiman, Universiti Malaysia Sabah, Malaysia
Dr. Srihadi Agungpriyono, PAVet(K), F. Kedokteran Hewan IPB Y. Surjadi MSc, Pusat Penelitian ICABIOGRAD
Drs. Suharjono, Pusat Penelitian Biologi-LIPI Dr. Tri Widianto, Pusat Penelitian Limnologi-LIPI
Dr. Witjaksono Pusat Penelitian Biologi-LIPI
Sekretariat
Eko Sulistyadi MSi, Dewi Citra Murniati MSi, Hetty Irawati PU, S.Kom Alamat
d/a Pusat Penelitian Biologi - LIPI
Jl. Ir. H. Juanda No. 18, Bogor 16002 , Telp. (021) 8765056 Fax. (021) 8765068
Email : [email protected]; [email protected]; [email protected]; [email protected] Website : http://biologi.or.id
Jurnal Biologi Indonesia telah diakreditasi ulang berdasarkan SK Kepala LIPI 742/ E/2012 tanggal 7 Agustus 2012, Akreditasi:No. 460/AU2/P2MI-LIPI/08/2012.
Jurnal Biologi Indonesia 9(2): 2013
Perhimpunan Biologi Indonesia.
Bekerja sama dengan
Jurnal Biologi Indonesia 9 (2): 2013
Jurnal Biologi Indonesia 9(2): 2013
KATA PENGANTAR
Jurnal Biologi Indonesia yang diterbitkan oleh PERHIMPUNAN BIOLOGI INDONESIA bekerjasama dengan PUSLIT BIOLOGI-LIPI. Edisi Volume 9 No. 2 tahun 2013 memuat 15 artikel lengkap dan satu artikel tulisan pendek. Penulis pada edisi ini sangat beragam yaitu dari Balai Besar Penelitian Vete riner, Departemen Pertanian, Balai Penelitian dan Pengembangan Tanaman Obat dan Obat Tradisional. Badan Litbang Kesehatan, Kementerian Kesehatan, Balai Penelitian Pemulihan dan Konservasi Sumber Daya Ikan, Kementerian Perikanan dan Kelautan, Pusat Konservasi Tumbuhan Kebun Raya Bogor-LIPI, Puslit Bioteknologi-LIPI, Departemen Teknologi Pangan, Fakultas Teknologi Pangan, Institut Pertanian Bogor, Pusat Penelitian Biolo-gi LIPI; Fakultas Kehutanan, Institut Pertanian Bogor, Balai Konservasi Biota Laut-LIPI
Jurnal Biologi Indonesia 9(2): 2013
DAFTAR ISI
Halaman
Enzyme-Linked Immunosorbent Assay for Detection of Infectious Bronchitis
Antibody in Chickens using Local Isolate of PTS III
Risa Indriani & NLP Indi Dharmayanti
159
Karakterisasi Genetik Tempuyung (Sonchus arvensis L.) Berdasarkan Penanda Molekuler
Sequence-Related Amplified Polymorphism
Dyah Subositi & Rohmat Mujahid
167
Karakteristik Populasi Labi-labi Amyda cartilaginea (Boddaert, 1770) yang Tertangkap di Sumatera Selatan
Agus Arifin Sentosa, Danu Wijaya & Astri Suryandari
175
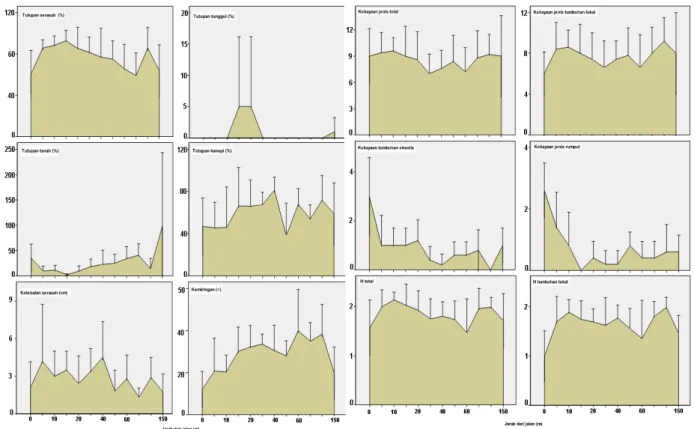
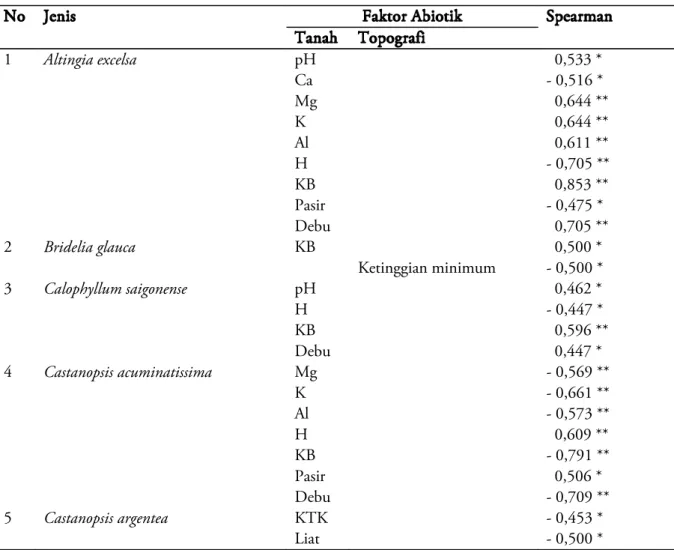
Pengaruh Jalan Terhadap Keragaman Jenis Tumbuhan Bawah dan Habitatnya di Koridor Taman Nasional Gunung Halimun Salak, Jawa Barat
Iyan Robiansyah & Danang W. Purnomo
183
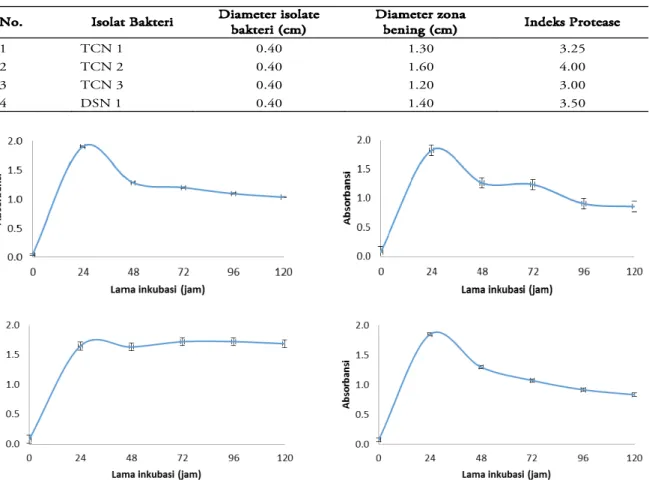
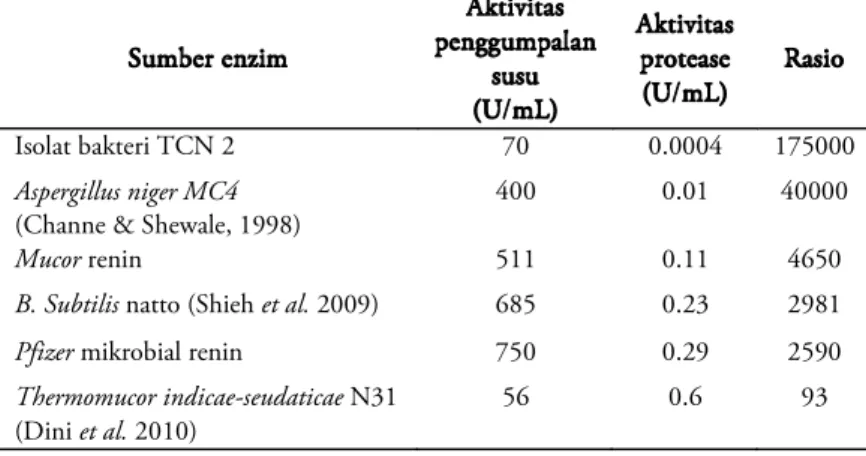
Isolat Bakteri Indigenous Penghasil Milk-Clotting Protease untuk Fermentasi Keju
Nanik Rahmani, Yana Nurita Sari, Nurheni Sri Palupi & Yopi
199
Preferensi Ekologis Jenis-Jenis Tumbuhan Dominan di Gunung Endut, Banten E.N. Sambas, C. Kusmana, L.B. Prasetyo & T. Partomihardjo
209
Pertumbuhan dan Produksi Jagung Pulut Lokal Sulawesi Selatan yang Ditanam di Polibag Pada Berbagai Kombinasi Perlakuan Pupuk Organik
Titi Juhaeti, N Hidayati& M Rahmansyah
219
Kajian Pemilihan Jenis Tumbuhan Untuk Restorasi Hutan Berdasarkan Beberapa Parameter Fotosintesis
Tinia Leyli Shofia Ahmad, Dede Setiadi, & Didik Widyatmoko
233
Kajian Pemberian Pakan Alternatif terhadap Konsumsi, Kecernaan, dan Efisiensi Penggunaan Pakan pada Jelarang Paha Putih (Ratufa Affinis Raffles, 1821)
Wartika Rosa Farida & Siti Nuramaliati Prijono
245
Keragaman genetik beberapa aksesi Jagung dari Nusa Tenggara Timur bagian berdasarkan profil Inter Short Sequence Repeat (ISSR)
Kusumadewi Sri Yulita & BP Naiola
255
Palem di Taman Nasional Bukit Baka-Bukit Raya, Kalimantan Barat
Himmah Rustiami
265
Biotransformasi 2E-6E-Farnesol oleh Jamur Endofit Botryosphaeria sp. CA2C-3 yang Diisolasi dari Temu Hitam (Curcuma aeruginosa ROXB.)
Andria Agusta
283
Penggunaan Ruang oleh Beruang Madu di Areal Konservasi IUPHHK-HTI PT. RAPP Estate Meranti
Nur Anita Gusnia, Agus Priyono Kartono, & Harnios Arief
289
Vocalizations of Microhyla achatina Tschudi, 1838 (Anura: Microhylidae) from the foot hills of Mount Salak, West Java
Hellen Kurniati
Jurnal Biologi Indonesia 9 (2): 2013
Halaman Sifat Fisik dan Kimia Daging Landak Jawa (Hystrix javanica F. Cuvier, 1823) yang Diberi
Tambahan Pakan konsentrat Wartika Rosa Farida
311
TULISAN PENDEK
Resolusi Kerancuan Perkiraan Luas dan Laju Deforestasi Hutan Mangrove di
Indonesia
Suyadi
Jurnal Biologi Indonesia 9(2): 2013
UCAPAN TERIMA KASIH
Jurnal Biologi Indonesia mengucapkan terima kasih dan penghargaan kepada para pakar yang telah turut sebagai penelaah dalam Volume 9, No 2, Juni2013:
Dr. Arjan Boonman , University of Tel Aviv, Israel Dr. Sri Sulandari Pusat Penelitian Biologi-LIPI Ir. Drs.Eko Harsono MSi, Puslit Limnologi-LIPI
Dr. Yuyu Suryasari Poerba, Pusat Penelitian Biologi-LIPI Dr. Tatik Kusniati Pusat Penelitian Biologi-LIPI
Ir.Mumpuni, Pusat Penelitian Biologi-LIPI Awal Riyanto, Pusat penelitian Biologi-LIPI
Drs Nunuk Widyastuti MSi, Pusat penelitian Biologi-LIPI Dr.Laode Alhamd, Pusat Penelitian Biologi-LIPI
Dr. Edi Mirmanto, Pusat Penelitian Biologi-LIPI Dr. Sarjiya Antonius, Pusat Penelitian Biologi-LIPI Dr. Dwi Astuti, Pusat Penelitian Biologi-LIPI Dr. Yopi. Pusat Penelitian Bioteknologi-LIPI Dr. Dedi Darnaedi, Pusat Penelitian Biologi-LIPI Dr. Atik Retnowati, Pusat Penelitian Biologi-LIPI Ir. Titi Juhaeti MSi. Pusat Penelitian Biologi-LIPI
Ir. SyamsulArifin Zein MSi, Pusat Penelitian Biologi-LIPI
Drh. Dewi Ratih PhD, APV. Fakultas Kedokteran Hewan IPBBIOLOGI
Jurnal Biologi Indonesia 9(2): 159-166 (2013)
Enzyme-Linked Immunosorbent Assay for Detection of Infectious Bronchitis Antibody in
Chickens using Local Isolate of PTS III
(Enzyme-linked immunosorbent assay untuk mendeteksi antibodi infectious bronchitis
pada ayam dengan isolat local)
Risa Indriani & NLP Indi Dharmayanti
Institute Centre Research of Veterinary, Jl. RE Martadinata no 30, Bogor 6114. E-mail: [email protected] Memasukan: September 2012, Diterima: April 2013
ABSTRACT
An indirect enzyme-linked immunosorbent assay (ELISA) was developed for screening of antibody to avian infec-tious bronchitis (IBV). Antigen was prepared from whole virus of infecinfec-tious bronchitis local isolate PTS-III serotype. Optimum dilution with minimum background for antigen concentration, rabbit anti-chicken conjugate and sera in developed ELISA were determined 0.4μg/well, 1:2000 and 1:100, respectively. Correlation optical densities (OD) were compared with a standard commercial ELISA (R2=0.933). The developed ELISA has a better sensitivity to he-magglutination inhibition (HI) test. The developed local isolate ELISA can be used to detect antibody against infec-tious bronchitis virus and it is suitable for sample screening at the diagnostic laboratories.
Keywords: ELISA, antibody, chicken, IB PTS-III local isolate
ABSTRAK
Pengembangan Enzyme-linked immunosorbent assay (ELISA) untuk mendeteksi antibodi virus infectious bronchitis (IB) pada ayam. Antigen ELISA IB disiapkan dari virus IB serotipe isolat lokal PTS-III. Pengenceran optimal pada konsentrasi antigen, konjugat rabbit anti-chicken, dan serum dengan background minimum dalam uji ELISA adalah 0.4μg, 1:2000 and 1:100 secara berurutan. Optical density (OD) dari serum IB positif pada uji ELISA yang dikem-bangkan dan ELISA standard mempunyai tingkat korelasi yang tinggi (R2=0.933). ELISA yang dikemdikem-bangkan mempunyai tingkat sensitifitas lebih baik terhadap uji hemagglutination inhibition (HI). Uji ELISA IB serotipe isolat lokal PTS-III dapat digunakan untuk screening sampel dalam laboratorium diagnostik.
Kata Kunci: ELISA, antibodi, ayam, IB isolat lokal PTS-III
INRODUCTION
Infectious Bronchitis virus (IBV) infects the respiratory tract, kidneys or oviduct of chick-en of all ages causing retarded growth, mortality, reduced egg production and inferior egg shell quality (King & Cavanagh 1991). The virus be-long to family of Coronaviridae. Corona viruses are enveloped, pleiomorphic, with a mean diame-ter of approximately 120 nm, club-shaped surface projections -the heavily glycosylate spike (S) gly-coprotein. IBVs contain four structural proteins. The S1 and S2 glycoproteins, from the spike or peplomer (S) located at surface of virion. In the
peplomer S, the S2 glycoprotein forms the stalk and is anchored in the membrane, whereas the S1 forms the outer bulbous part of the peplomer. The membrane (M) glycoprotein is embedded in the viral lipid bilayer and is only partially (10%) exposed on surface of virion. The nucleocapsid (N) protein is located inside the virion, associated with viral RNA (Cavanagh 1983a,b,c). The role of the S1 glycoprotein in induction of humoral anti-body responses, and it induces virus neutralizing (VN) and haemagglutination inhibiting (HI) an-tibodies (Cavanagh et al. 1984, Koch et al. 1990; Kant et al. 1992). The S1 glycoprotein has been considered to be the most likely inducer of
pro-Indriani & Dharmayanti
for 72 hours. A thousand ml allantoic fluid was harvested and clarified at 8000g for 30 minutes then EID50 was calculated. It was inactivated 0.05% β-PL and centrifuged at 90.000g for 90 minute in a sorvall AT- 629 (32ml) at 40C. The pooled result was pelleted in TEN buffer (150 mM NaCl, 10mM Tris-hydrochloride, and 1mM EDTA, pH 7.4) and analyzed for protein content by using Spectrophotometer/Nano Drop Tecnolo -gies ND-1000 base on Bradford’s method. The solution was finally divided into several aliquots and frozen at 700C.
Serum IBV was prepared from 20 of spe-cific pathogenic free chickens that reared under controlled conditions in the isolator (BSL-3) and vaccinated day 10 with live H120 (vaccine com-mercial), day 44 with inactivated oil-emulsion vaccine PTS-III (BBALITVET) and day 57 chal-lenge with 107EID
50 of the same virus. The chick-ens were bled at day 10, 24, 34, 44, 57, 70, 78 and 89 of age. A group of specific pathogenic free chicken were reared as negative control and bled at the same time.
Ortho-phenylene diamine (OPD) (Sigma P.23938) was used. Horeseradish peroxidase con-jugated rabbit anti-chicken IgG (Sigma A.9046) was used.
Antigen was diluted at concentrations 4 mg/ml, 2 mg/ml and 1 mg/ml in carbonate buff-er. Known positive IBV and negative sera was diluted at 1:100, 1:200 and 1:500 in dilution buffer. Conjugate was diluted at 1: 1000, 1:2000, 1:3000 and 1:4000. Six replicated reaction were done. The color of reaction was obtained from Ortho-phenylene diamine. The Optical density were measured at 450 nm using an automatic ELISA reader (Multiskan EX, Thermo Lybsys-tems) and the signal-to noise (S/N) ratio at the same dilution were evaluated.
The ELISA procedure was standardized on the method developed by Case et al (1982) with tection (Cavanagh et al. 1986, Ignjatovic & Galli
1995) and cross-ractive epitopes are the most like-ly to be involved in protection. The S2 glike-lycopro- glycopro-tein carries epitop which induce cross-reactive antibodies (Kusters et al. 1989; Lenstra et al. 1989; Koch et al. 1990). Immunity to IBV has most often been assessed using traditional serolog-ical assay; however, the enzyme-linked immuno-sorbent assay (ELISA) is used on a more frequent basis to measure IBV antibodies (Case et al.1982). The technique initially developed by Engvall & Perlmann (1971) and has been widely used. The use of ELISA offers a number of advantages com-pared with traditional serological assays, including increase sensitivity and simplicity of automation (Garcia & Bankowski 1981). The cross reactivity of the IB ELISA with several strains of the virus and detection antibodies against other serotypes (Zellen &Thorsen 1986 ; De Wit 2000) support the idea that ELISA is a promising tool for sero-logical studies, especially for use in evaluating the efficacy of vaccination regimens and monitoring the immune status of birds in a flock (Cavanagh & Naqi 2003; Wing et al. 2002). Indirect ELISAs were adapted and standardized for detecting anti-bodies against whole virus, and S1, S2 and N pro-tein of IBV (Ignjatovic & Galli 1995; Wing et al. 2002) and compared with other serological test (De Wit et al. 1997; Perrotta et al. 1988; Thater et
al. 1987). This paper describes study of indirect
ELISA for detection of antibody against IBV by using local isolate virus (PTS-III), which its according to the virus circulating in the field.
MATERIAL AND METHODS
The local isolate IBV of PTS III as de-scribed by Darminto (1992) was propagated in specific pathogenic free emberyonated chicken eggs at day 10 (Biofarma, Indonesia) by infecting the virus to allantoic fluid then incubated at 370C
Enzyme-Linked Immunosorbent Assay for Detection of Infectious Bronchitis Antibody
some modification. IBV local isolate PTS-III of antigen was assay at concentrations 0.4 μg/50 μl/ well in carbonate buffer (0.1M NaCO3, 0.02% NaN3, pH 9.6). Flat bottomed micro plates (Nunc) were coated antigen at same time. After incubate at 40C for one night, the wells were washed using washing buffer (0.15M NaCl, 2.5 mM KCL, 1.5 mM NaH2PO4 H2O , 9.0 mM Na2HPO4 and 0.05% Tween 20, pH 7.4) with soaking for 5 minutes at room temperature in each time and trapped out onto absorbent paper. Fifty μl blocking buffer (0.15M NaCl, 2.5 mM
KCL, 1.5 mM NaH2PO4 H2O, 9.0 mM
Na2HPO4, 1mM EDTA, 0.5% Casein, and 0.05 % Tween 20, pH7.4) was added to each well. The plate was washed after incubation at 370C for 120 minutes. Serum samples were prepared by diluting 1:100 in phosphate buffer saline (0.15M NaCl, 2.5 mM KCL, 1.5 mM NaH2PO4 H2O , 9.0 mM Na2HPO4, 1mM EDTA, 0.1% Casein, and 0.05 % Tween 20, pH7.4) then 50 μl was added to each well. The plate was washed after incubation at 370C for 60 minute. Horseradish peroxidase conjugated rabbit anti-chicken IgG was prepared by diluting 1:2000 μl of conjugate buffer (0.5M NaCl, 2.5 mM KCL, 1.5 mM NaH2PO4 H2O, 9.0mM Na2HPO4, 1mM EDTA, 0.1% Casein, and 0.05 % Tween 20, pH7.4) then 50 μl was added to each well. The plate was washed after incubation at 370C for 60 minute. A hundred μl of substrate (0.04% OPD, 0.04% H2O2, 0.2M NaHPO4, 0.1 M Citric acid, pH5) was added and incubated at room temperature for 45 minute then stopped by 1M H2SO4. Optical density of each serum was determined by using ELISA reader.
Cut-off value of the ELISA was determined by using optical density (OD) serum from unvac-cinated group (as negative control). The average value of OD serum was added three of standard
deviation (STDEV) as cut-off value of the ELISA. The specificity of the developed ELISA was calculated as the percentage of negative in unvac-cinated group and the sensitivity was calculated as percentage of positives in vaccinated group. The result of serum obtain by the Elisa (local isolate PTS-III) were compared with those obtained by a commercial kit (IDEXX).
Infectious Bronchitis antigen which tested by using ELISA was evaluated to determine anti-gen binding reaction and non specific antibodies against other respiratory viral diseases (Infectious Laryngotrachitis).
HI test which conducted by M41 antigen (kindly provided by BPMSOH), was used for the preparation of haemagglutinating antigen as de-scribed by King and Hopkins (1983) and Alexan-der et al (1983).
RESULTS
Antigen, serum and conjugate dilution
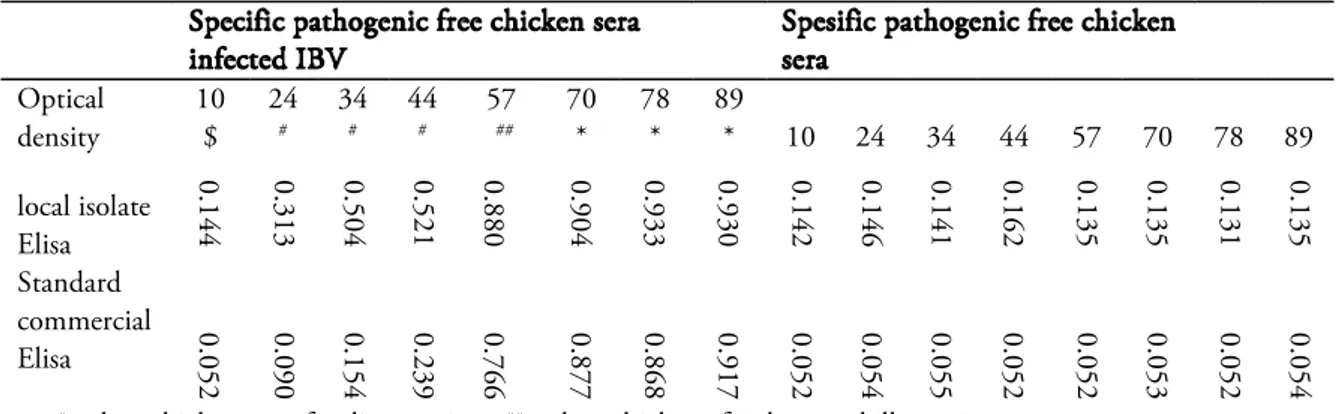
The protein content of prepared antigen of IBV local isolate PTS-III was 8.8 mg/ml base on Bradford’s method (Spectrophotometer/Nano Drop Tecnologies ND-1000). We obtained mini-mal nonspecific binding at 8 mg protein /well. According to titration result by 0,4 μg/well anti-gen (dilution 1:1000/well), 1:100 chicken serum dilution and 1:2000 conjugate HRPO in S/N ratio which showed expensiveness of conjugate, was chosen. Optical density of ELISA value for positive sera was 6 times greater to negative sera (Figure 1.).
Cross-reaction with other respiratory viral in-fection
Non specific antibody reaction other res-piratory disease (ILT) in local isolate ELISA IB test the average value OD was 0.144, with STDV
Indriani & Dharmayanti
The HI titer (log2) of the positive sera were 0.19, 3.45, 4.05, 3.95, 6.35, 8.3, 8.45, and 8.6 respectively. In non-vaccinated group, HI titer at first and the end of test period were 2.05 and 2.1 respectively. The peak titer in vaccinated group was recorded at day 44 or after 34 days post vac-cinated with H120 live vaccine and the titer in-creased at day 57 (after 2 weeks booster
vaccinat-Figure 1. Optimum dilution of antigen, sera and conjugate
IBV Antigen ELISA Control Antigen
? negative sera ? IB sera ? ILT sera Mean OD STDV Mean OD STDV
16 0.162 0.001 0.155 0.101
16 0.993 0.005 0.144 0.002
10 0.144 0.022 0.101 0.002
Tabel 1. Idetification reaction of non specific other respiratory virus (ILT)
Specific pathogenic free chicken sera
infected IBV
Spesific pathogenic free chicken
sera Optical density 10 $ 24 # 34 # 44 # 57 ## 70 * 78 * 89 * 10 24 34 44 57 70 78 89 local isolate Elisa 0.144 0.313 0.504 0.521 0.880 0.904 0.933 0.930 0.142 0.146 0.141 0.162 0.135 0.135 0.131 0.135 Standard commercial Elisa 0.052 0.090 0.154 0.239 0.766 0.877 0.868 0.917 0.052 0.054 0.055 0.052 0.052 0.053 0.052 0.054
Table 2. The result of 140 positive and 180 negative sera tested for IB antibody using local isolate PTS-III and commercial ELISAs
# = days chicken age after live vaccine, ## = days chicken after booster kill vaccine,
*= days chicken age after challenge , $= days before vaccinated Antigen content
Optical density ELISA
0.022, while the antigen control was 0.101 to 0.002 STDV (Tabel 1.)
Optical density of the local isolate ELISA Optical density obtained by local isolate PTS-III and commercial ELISAs were measured from 180 specific pathogenic free chickens sera and 140 specific pathogenic free chickens sera infected IBV (Table 2)
Enzyme-Linked Immunosorbent Assay for Detection of Infectious Bronchitis Antibody
ed). Also the titer was increased rapidly after chal-lenge in ELISAs. Correlation between the local isolate ELISA test and standard commercial ELI-SA was very high (R2=0.933) in vaccinated and challenged chicken sera, while at the unvaccinated chicken sera has no correlation (R2=0.010)
Cut off optical density of the ELISA
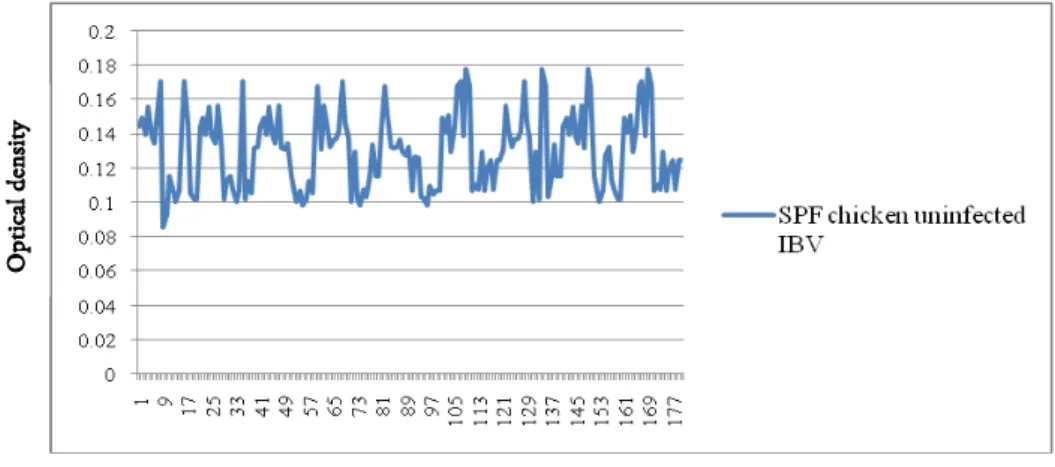
The cut off for the standardized ELISA test was determined as mean of negative sera plus thrice of the standard deviation 0.201 [0.141+ (3x0.020)] (Figure 2.)
Specificity and Sensitivity of the local isolate ELISA
Sensitivity and specificity were evaluated using sample positive (S/P) ratio (Table 2). Per-centage of sensitivity local isolate PTS-III ELISA
was slightly higher than hemaglutination inhibi-tion test, i.e. 96.42% and 90% respectively. While percentage of specificity between local iso-late PTS-III ELISA and hemaglutination inhibi-tion test were 100%.
DISCUSSION
Protective immunity to avian infectious bronchitis is not reflected by humoral antibodies, as shown by Davelaar & Kouwenhoven (1980). Nevertheless, monitoring antibodies after vaccina-tion is a valuable procedure for indicating that responses have occurred. The local isolate ELISA test for measuring antibody level against IBV was developed in this study. The test was standardized in term of reagent to obtain the significant S/N ratio. ELISA test has been developed to monitor
Figure 2. The cut off for standardized ELISA of local isolate PTS-III
E LISA reaction C hicken S era
SPF chicken infected IBV
SPF chicken
uninfected Precentation
Local isolate IB Elisa Positive 135 (a) 0 (b) Sensitivity 96,42%
Negative 5 ( c) 180 (d) Specificity 100%
Hemaglutination
Inhibition (HI) test Positive 126 (a) 0 (b) Sensitivity 90%
Negative 14 ( c) 180 (d) Specificity 100%
Table 4. Sensitivity and specificity the ELISA local isolate PTS-III dan HI
Sensitivity = (a) ; Specificity = (d) (BALDOCK 1988) (a)+(c) (d)+(b)
Number of sera
Indriani & Dharmayanti
antibody respond to vaccination against IB (Monreal et al. 1985, Mockett and Darbyshire 1981, Soula &Moreau 1981). Case et al. (1982) optimized the parameter of ELISA for detecting antibody against IBV. They obtained minimal nonspecific binding and very high sensitivity us-ing purified IB as antigen 50 ng protein /well and final NaCl concentration 1.0M in buffer. In this study it was obtained minimal nonspecific bind-ing at 0.4 μg protein/well. Accordbind-ing to titration result base on 0.4 μg protein/well at 1:2000 con-jugate showed similar significant difference in S/N ratio, and cheaper conjugate was chosen. To elim-inate non specific binding in the local isolate -III ELISA, we used blocking buffer (0,15M NaCl, 2,5 mM KCL, 1,5 mM KH2PO4, 9,0 mM NaHPO4, 0.5% Casein, and 0.05 % Tween 20, pH7.4) after coated antigen. The local isolate ELI-SA indicated low to moderate level after live vac-cination and moderate to relatively high level of antibody IB after injection of inactivated vaccine as well as commercial one. This data was agreed with HI results. In non-vaccinated group OD was lower then that the cut off (0.201) in the local isolate ELISA. Also, the titer of HI test which negative in this group parallel with the ELISAs results. The OD of the ELISAs and HI titer in-creased slowly following live H120 vaccination and more following oil vaccine injection.
The sensitivity of local isolate PTS-III ELI-SA was slightly higher than hemaglutination inhi-bition test, i.e. 96.42% and 91,42% respectively. The results showed specificity was equal 100% between local isolate PTS-III ELISA and hemag-lutination inhibition test. Some researcher demonstrated the high sensitivity of the ELISA and correlation of results obtained from the HI test in chicken sera infected intratarcheally with IB strain M41 (Mockett & Darbyshire 1981; Soula & Moreau 1981).
The result of antibodies titration showed
that the developed local isolate PTS-III ELISA could be reliable, repeatable and more sensitive for monitoring of vaccination schedules and for detection of early rising of antibodies against IB rapidly.
CONCLUSION
This study concluded that local isolate IB PTS III ELISA could be reliable, repeatable and sensitive for monitoring of vaccination schedules and detection of early rising of antibodies against IB rapidly.
AKNOWLEDGMENTS
This research was supported by funds from a researcher budget of BBALITVET DIPA in 2012. We thank Heri Hoerudin and Apipudin, and all collaborators helped for implementation of this study
REFERENCES
Alexander, DJ., WH. Allan, PM. Biggs, CD. Bracewell. JH. Darbyshire, PS. Dawson, AH. Harris. FTW. Jordan, I. Macpherson, JB. McFerran, CJ. Randall, O. Swarbrick, & GP. Wilding. 1983. A satandard tech-nique for haemagglutination inhibition test for antibodies to avian infectious bronchitis virus. Vet. Rec. 113:64
Baldock, FC. 1988 . Epidemiological evaluation of immunological test. Elisa technology in
diagnosis and research. JCU, Townsville,
Aus-tralia. 90-95.
Cavanagh, D. 1983a. Coronavirus IBV glycopoly-peptides: size of their polypeptide moieties and nature of their oligosaccharides. J. Gen.
Vir. 64: 1187-1191.
Enzyme-Linked Immunosorbent Assay for Detection of Infectious Bronchitis Antibody
evidence that the surface projections are asso-ciated with two glycopolypeptides. J. Gen.
Vir. 64: 1787-1791.
Cavanagh, D . (1983c) Coronavirus IBV: struc-tural characterization of the spike protein. J.
Gen. Vir. 64: 2577-2583.
Cavanagh, D., JH. Darbyshire, P. DAVIS, & RW. Peter. 1984. Induction of humoral neu-tralizing and haemagglutination-inhibiting antibody by the spike protein of avian infec-tious bronchitis virus. Avian Path. 13: 573-583.
Cavanagh, D., PJ. Davis, JH. Darbyshire, & RW. Peters. 1986. coronavirus IBV: virus retaining spike glycopolypeptide S2 but not S1 is unable to induce virus-neutralizing or haemagglutination-inhibiting antibody, or induce chicken tracheal protection. J. Gen.
Vir. 67: 1435-1442
Case, JT., AA. Ardans, DC. Bolton, & BJ. Reyn-olds. 1982. Optimization of parameter for detecting antibodies against infectious bron-chitis virus using an enzimed-linkes Im-munosorbent Assay: Temporal respon to vaccination and challenge with live virus.
Avian Diseases 27:196-210
Davelaar, PG. & Kouwenhoven, B. 1980. Vac-cination of DOC broiler against infectious bronchitis by eye drop application or coarse droplet spray and the effect of revaccination by spray. Avian Pathology 9:499-510
Darminto. 1992. Characterization of subtype In-fectious Bronchitis virus local isolate. J.
Pen-yakit Hewan 24: 78-81
De Wit, JJ., DR. Mekkes, B. Kouwenhoven, & JHM. Verheijden.1997. Sensitivity and spec-ificity of serological tests for infectious bron-chitis virus antibodies in broilers. Avian
Path. 26:105-118.
De Wit, JJ.(2000). Detection of infectious bron-chitis virus. Avian Pathology 29:71-93.
Engvall, E., & P. Parlmann. (1971). Enzyme linked immunosorbent assay (ELISA) quan-titative assay of immunoglobulin G. Immun. 8:871-874.
Garcia, Z., &. RA. Bankowski. 1981. Compara-tion of a tissue-culture virus-netraliz aCompara-tion test and the enzyme-linked immunosorbent assay for measurement of antibodies to infec-tious bronchitia. Avian Diseases 25:121-130. Ignjatovic, J., & L. Galli. 1995. Immune respon
to structural protein of avian infectious bron-chitis virus. Avian Path. 24:313-332
Kusters, JG., EJ. Jager, JA. Lenstra, G. Koch, WPA. Posthumus, RH. Meloen, & BAM. Van der Zeist. 1989. Analysis of an immu-nodominant region of infectious bronchitis virus. J. Immun. 143: 2692-2698.
Kant, A., G. Koch, DJ. van Roozelaar, JG. Kusters, FAJ. Poelwijk, & BMA. van der Zeijst, G. Koch, L. Hartog, A. Kant, & DJ. van Roozelaar. 1990. Antigenic domains on the peplomer protein of avian infectious bronchitis virus: correlation with biological functions. J. Gen. Vir.71: 1929-1935.
Kant, A., G. Koch, DJ. van Roozelaar, JG. Kusters, FAJ. Poelwijk, & BMA. van Der Zeijst 1992 Location of antigenic sites de-fined by neutralizing monoclonal antibodies on the S1 avian infectious bronchitis virus glycopolypeptide. J. Gen. Vir., 73, 591-596. King, DJ., & SR. Hopkins 1983. Evaluation of
the hemaggutination-inhibition test for measuring the response of chickens to avian infectious bronchitis virus vaccination, Avian
Diseases 27:100-112
King, DJ. & D. Cavanagh. 1991. Infectious bron-chitis, in: BW. Calnek, HJ. Barnes, CW. Beard,WM. Reid & HW. Yoder (Eds)
Dis-eases of Poultry, 9th edn, pp. 471-484
Koch,G., L. Hartog, A. Kant & DJ. van Roozelaar. 1990. Antigenic domains on the
Indriani & Dharmayanti
peplomer protein of avain influenza bronchi-tis virus: correlation with biological fungtion.
J. Gen. Vir. 71: 1929-1935.
Lenstra, JA., JG. Kusters, G. Koch. & BAM. van Der Zeijst. 1989 Antigenicity of the peplomer protein of infectious bronchitis virus. Mol. Immun. 26: 7-15.
Monreal, G., HJ. Baure & Wiegman. 1985. Comparasion of the enzyme-Linked Im-munosorbent Assay (ELISA). Hemagglutina-tion inhibiHemagglutina-tion test and agar gel precipitaHemagglutina-tion test for detection of antibodies to avian in-fectious bronchitis virus. Avian Path. 14:421 -434.
Mockett,APA., & JH. Darbyshire. 1981. Co-mapration studies with an enzyme-linked immunosorbent assay (ELISA) for antibodies to avian infectious bronchitis virus. Avian
Pathology 10:1-10.
Perrotta, C. C. Furtek, RA. Wilson, BS. Cowen & RJ. Eckroade. 1988. A standardization enzyme-linked immunosorbent assay for in-fectious bronchitis virus: Comparison with hemagglutination-inhibition and
virus-neutralization assays for measuring protective antibody levels in chickens. Avian Diseases 32:451-460.
Soula, A. & Y. Moreau. (1981). Antigen require-ments and specificity of a microplate enzyme -linked immunosorbent assay (ELISA) for detecting infectious bronchitis viral antibod-ies in chicken serum. Archives Vir. 67:283-295
Thater, SG., P. Villegas. & OJ. Fletcher. (1987). Comparison of two commercial enzyme-Linked Immunosorbent Assay and conven-tional methods for avian serology. Avian
Dis-eases 31:120-124.
Zellen, GK., &. J. Thorsen. (1986). Standardiza-tion and applicaStandardiza-tion of the Enzyme-Linked Immunosorbent Assay for infectious bron-chitis. Avian Diseases 30:695-698.
Wing, CH., CC. HONG, & JCH. SEAK. (2002). An ELISA for antibodies against infectious bronchitis virus using an S1 spike polypeptide. Vet. Micro. 85:333-342.
Jurnal Biologi Indonesia 9 (2): 167-174 (2013)
PENDAHULUAN
Tempuyung (Sonchus arvensis L.) merupa-kan tumbuhan anggota familia Asteraceae dan banyak ditemukan di Sumatera, Jawa, Bali,
Karakterisasi Genetik Tempuyung (Sonchus arvensis L.) Berdasarkan Penanda Molekuler
Sequence-Related Amplified Polymorphism
(Genetic Characterization of Tempuyung (Sonchus arvensis L.) Based on Sequence-Related
Amplified Polymorphism Molecular Markers)
Dyah Subositi* & Rohmat Mujahid
Balai Penelitian dan Pengembangan Tanaman Obat dan Obat Tradisional. Badan Litbang Kesehatan, Kementerian Kesehatan RI. Jl. Raya Lawu, Tawangmangu, Karanganyar, Jawa Tengah, Tel.: (0271) 697010,
Fax.: (0271) 697451, E-mail: [email protected] Memasukkan: Februari 2013, Diterima: April 2013
ABSTRACT
Tempuyung (Sonchus arvensis L.) is known as an important medicinal plant used as a diuretics and antihypertensives. This plant is widely distributed in Indonesia. Genetic diversity of tempuyung is important information as a database for further research especially in medicinal plant standardization. The objective of this study was to analyse genetic characterization of tempuyung based on SRAP (Sequence-related amplified polymorphism) molecular markers. Thirteen samples were collected from 8 different locations and amplified using 5 primer SRAP combinations. Similarity matrix was calculated using Dice coefficient. Unweighted Pair Group Method Using Arithmetic Mean (UPGMA) cluster analysis was performed to develop a dendrogram. The result indicates that there was a genetic variation among tempuyung accessions and divided into 4 clusters with similarity index of 0,7719. Citeureup and Turen3 accessions were the most closely similar with similarity index of 0,8936. In conclusion, SRAP markers may serve as an efficient and effective tools to analyze the genetic diversity among tempuyung accessions.
Keywords: genetic characterization, tempuyung (Sonchus arvensis L.), SRAP ABSTRAK
Tempuyung (Sonchus arvensis L.) banyak dimanfaatkan sebagai tumbuhan obat untuk menurunkan tekanan darah tinggi dan peluruh air seni. Tempuyung mudah dan banyak dijumpai di berbagai tempat di Indonesia. Keragaman genetik tempuyung merupakan informasi penting sebagai data base untuk penelitian lebih lanjut terutama mendukung standarisasi tumbuhan obat. Penelitian ini bertujuan untuk karakterisasi genetik tempuyung menggunakan penanda molekular SRAP (Sequence-related amplified polymorphism). Sebanyak 13 aksesi tempuyung yang dikoleksi dari 8 lokasi digunakan sebagai sampel dan diamplifikasi menggunakan 5 pasang kombinasi primer SRAP. Indeks similaritas dihitung menggunakan rumus indeks similaritas Dice kemudian disusun analisis klaster dan konstruksi dendogram dilakukan dengan menggunakan metode Unweighted Pair Grup Method Using Aritmetic
Method (UPGMA). Hasil penelitian menunjukkan adanya variasi genetik antar aksesi tempuyung dan terbagi
menjadi 4 klaster pada indeks similaritas 0,7719. Aksesi Citeurep dan aksesi Turen 3 mempunyai hubungan kemiripan yang terdekat pada indeks similaritas 0,8936.Penanda molekular SRAP merupakan teknik efektif dan efisien untuk karakterisasi genetik antar aksesi tempuyung.
Kata Kunci: Karakterisasi genetik, tempuyung (Sonchus arvensis L.), SRAP
Sulawesi dan Papua. Tumbuhan tersebut dapat tumbuh di tempat dengan ketinggian 50-2400 m dpl dan banyak dijumpai di sekitar persawahan, tepi jalan, dan tebing (Backer & van den Brink 1965). Tempuyung banyak digunakan untuk
Subositi & Mujahid.
kemudahan teknik RAPD dan hasil yang mempunyai keakuratan tinggi seperti AFLP serta mampu mendeteksi polimorfisme di coding
sequence yang umumnya terdapat di genom
kultivar dan mempunyai tingkat mutasi yang relatif rendah (Keyfi & Beiki 2012; Zeng et al. 2012). Penanda molekular telah banyak digunakan untuk berbagai tujuan anatara lain yaitu untuk identifikasi kultivar (Jianfeng et al. 2012), analisis keragaman genetik dan hubungan kemiripan plasma nutfah (Alghamdi et al. 2012), identifikasi dan karakterisasi genetik F1 hibrid (Zhang et al. 2012). Tujuan penelitian ini adalah karakterisasi genetik aksesi tempuyung berdasar-kan penanda molekular SRAP untuk mendukung standarisasi tumbuhan obat.
BAHAN DAN CARA KERJA
Koleksi sampel tempuyung dilakukan pada bulan Februari-Agustus 2012, wilayah pengum-pulan sampel meliputi provinsi Jawa Timur, Jawa Tengah, Daerah Istimewa Yogyakarta, Jawa Barat, Nusa Tenggara Barat dan Sumatera Barat. Sebanyak 52 aksesi tempuyung berhasil dikoleksi kemudian diamati pola spektrum FTIR (Fourier
transformed infrared spectrophotometer) dan
analisis hasil menggunakan PCA (Principal
Component Analysis). Sebanyak 13 aksesi yang
berbeda secara profil FTIR serta lokasi tumbuh dipilih sebagai sampel untuk analisis karakterisasi genetik (Tabel 1).
Sebanyak 0,1 gram daun muda tempuyung disimpan pada freezer -80°C kemudian diisolasi menggunakan protokol dan kit isolasi DNA (Sigma GenElute Plant Genomic DNA Miniprep
Kit). Hasil isolasi DNA genom tempuyung
diencerkan 1000X dan diukur absorbansi pada panjang gelombang 260 nm dan 280 nm (l 260/ 280) menggunakan spektrofotometer untuk menentukan kemurnian dan konsentrasi DNA. mengobati asma, batuk, beberapa keluhan sakit
pada dada, menenangkan saraf, anti radang, peluruh air seni dan menurunkan tekanan darah tinggi. Tempuyung juga dilaporkan berfungsi sebagai insektisida. Flavonol, glikosida flavonol, monoasil galaktosilgliserol, seskuiterpen lakton, dan asam kuinat berhasil diisolasi dari tanaman ini (Xiang & Yu 2010).
Keanekaragaman genetik pada suatu spesies sangat penting digunakan untuk menggambarkan daya adaptasi, seleksi genotip dan untuk merakit varietas baru, sidik jari genetik, serta manajemen plasma nutfah. Karakter morfologi merupakan karakter yang banyak digunakan untuk sebagai langkah awal untuk identifikasi dan analisis keanekaragaman genetik, karena karakter ini mudah diamati. Karakter morfologi mempunyai keterbatasan yaitu membutuhkan waktu penga-matan yang lama dan membutuhkan ketelitian dan kemampuan dalam mencandra saat di lapangan serta mudah terpengaruh oleh kondisi lingkungan (Fu et al. 2008; Meng et al. 2011).
Perkembangan teknik penanda molekular memungkinkan untuk menganalisis genom secara langsung dan akurat sehingga dapat meminima-lisasi kesalahan akibat faktor lingkungan. Berbagai macam teknik penanda molekular juga telah banyak digunakan untuk karakterisasi dan identifikasi varietas pada tumbuhan (Abdelmigid 2012). Penanda molekular tersebut antara lain
Restriction Fragment Length Polymorphism (RFLP), Random Amplified Polymorphic DNA (RAPD), Amplified Fragment Length Polmorphism (AFLP),
Mikrosatelit atau Simple Sequence Repeats (SSRs) dan Inter Simple Sequence Repeats (ISSRs) (Muthusamy et al. 2008).
Sequence-related amplified polymorphism
(SRAP) merupakan penanda molekular yang ditemukan lebih akhir dibandingkan penanda molekular lainnya seperti RFLP, AFLP, RAPD, SSR dan ISSR. SRAP mengkombinasikan
Karakterisasi Genetik Tempuyung (Sonchus arvensis L.)
No No
Koleksi Nama Aksesi Asal Keterangan
1 2 Turen-2 Turen, Malang Liar
2 3 Turen-3 Turen, Malang Liar
3 15 Purwokerto Purwokerto Liar
4 25 Kalisoro Tawangmangu, Karanganyar Liar
5 27 Patuk Gunung Kidul Liar
6 34 Materia Medika Materia Medika, Malang Budidaya
7 36 Mataram Mataram Liar
8 37 Citereup Citereup, Bandung Liar
9 47 B2P2TO-OT (1) Tawangmangu, Karanganyar Budidaya 10 48 B2P2TO-OT (2) Tawangmangu, Karanganyar Budidaya 11 49 B2P2TO-OT (3) Tawangmangu, Karanganyar Budidaya 12 50 B2P2TO-OT (4) Tawangmangu, Karanganyar Budidaya 13 52 B2P2TO-OT (campuran
untuk QC)
Tawangmangu, Karanganyar Budidaya
Tabel 1. Daftar aksesi tempuyung yang digunakan untuk karakterisasi genetik
Amplifikasi DNA aksesi tempuyung menggunakan 5 kombinasi primer SRAP yaitu ME1-EM2, ME1-EM3, ME2-EM1, ME3-EM2 dan ME4-EM3 (Tabel 2). Protokol SRAP berdasarkan Li & Quiros (2001) dengan modifi-kasi pada komposisi/campuran untuk reaksi PCR dan waktu elongasi terakhir. Sebanyak 3 μl
template (25 ng DNA genom), 0,6 μl primer reverse dan 0,6 μl primer forward, 13 μl PCR
Mix, kemudian ditambah distillated water sampai volume 25 μl. Campuran tersebut dimasukkan dalam Thermocycler (BioRad) dengan program sebagai berikut: pre-denaturasi 94°C selama 5 menit, kemudian siklus pertama sebanyak 5 siklus terdiri atas fase denaturasi 94°C selama 1 menit, fase penempelan pada suhu 35°C selama 1 menit dan fase elongasi/pemanjangan pada suhu 72°C selama 1 menit, selanjutnya diikuti oleh siklus kedua sebanyak 35 siklus terdiri atas fase denaturasi pada suhu 94°C selama 1 menit, fase penempelan pada suhu 50°C selama 1 menit dan fase elongasi pada suhu 72°C selama 1 menit. Reaksi PCR diakhiri fase elongasi pada suhu 72°C selama 8 menit dan 4°C sebagai holding
temperature.
Produk amplifikasi dicek menggunakan elektroforesis gel agarosa 2% yang telah diberi
SYBR safe green pada 80 volt selama 100 menit.
Visualisasi hasil elektroforesis menggunakan sinar UV pada alat Gel Documentation System (BioRad).
Data diperoleh dari hasil visualisasi frag-men DNA tiap aksesi tempuyung pada kombinasi primer SRAP yang berbeda. Bila terdapat fragmen diberikan skor 1 dan bila tidak terdapat fragmen diberi skor 0. Indeks similaritas dihitung menggu-nakan rumus indeks similaritas Dice kemudian disusun analisis kelompok (cluster analysis) dan konstruksi dendogram dilakukan dengan menggunakan metode Unweighted Pair Grup
Method Using Aritmetic Method (UPGMA). Data
tersebut diolah menggunakan program komputer (software) NTSYS ver 2.02.
HASIL
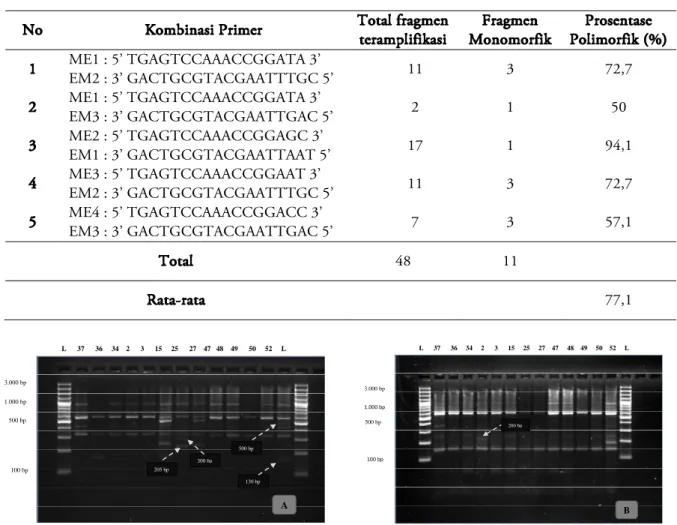
Sebanyak 48 fragmen DNA dihasilkan dari amplifikasi menggunakan 5 kombinasi primer SRAP dengan jumlah ≥ 2 fragmen DNA tiap primer (Tabel 3). Kombinasi primer ME2-EM1
Subositi & Mujahid.
menghasilkan jumlah fragmen paling banyak yaitu 17 fragmen, sedangkan primer ME1-EM3 menghasilkan fragmen paling sedikit yaitu 2 fragmen. Ukuran fragmen DNA yang dihasilkan dari kombinasi primer ME1-EM2 berkisar antara 130-1050 bp, primer ME1-EM3 menghasilkan fragmen 160-250 bp, primer ME2-EM1 mengha-silkan fragmen 40-1.620 bp, primer ME3-EM2 menghasilkan fragmen 20-1.000 bp, sedangkan kombinasi primer ME4-EM3 menghasilkan fragmen 50-450 bp.
Polimorfisme tertinggi dihasilkan amplifi-kasi menggunakan pasangan primer ME2-EM1 yaitu 94,1%, polimorfisme terendah 50% bila diamplifikasi menggunakan pasangan primer ME1-EM3, sedangkan polimorfisme rata-rata yaitu 77,1% (Tabel 3).
Lima kombinasi primer SRAP menghasil-kan fragmen DNA dengan polimorfisme rata-rata yang cukup tinggi (77,1%) dan 4 kombinasi primer menghasilkan beberapa fragmen spesifik tetapi kombinasi primer ME1-EM3 tidak dapat menghasilkan fragmen spesifik pada aksesi tertentu. Kombinasi primer ME4-EM3 mengha-silkan fragmen spesifik untuk aksesi B2P2TO-OT (4) pada ukuran sekitar 80 bp, sedangkan primer ME2-EM1 menghasilkan primer spesifik untuk aksesi Materia Medika Malang pada ukuran sekitar 1620 bp dan fragmen spesifik ukuran sekitar 1140 bp pada aksesi Mataram. Aksesi Turen-2 mempunyai fragmen spesifik ukuran sekitar 200 bp jika diamplifikasi menggunakan
kombinasi primer ME3-EM2, sedangkan kombinasi primer ME1 dan EM2 menghasilkan fragmen spesifik dengan ukuran sekitar 205 dan 300 bp pada aksesi Kalisoro dan fragmen spesifik berukuran 130 dan 500 bp pada aksesi B2P2TO-OT (Campuran) (Gambar 1).
Dendogram (Gambar 2) menunjukkan 13 aksesi tempuyung terbagi menjadi 4 klaster. Indeks kemiripan genetik 13 aksesi tempuyung berkisar 74,93%-89,36%. Klaster I terdiri dari aksesi Citeurep, aksei Turen 2 dan aksesi Mataram, Klaster II terdiri dari aksesi Materia Medika Malang, aksesi B2P2TO-OT campuran, aksesi B2P2TO-OT 1, aksesi B2P2TO-OT 2, aksesi B2P2TO-OT 4, dan aksesi B2P2TO-OT 3, klaster III hanya beranggotakan aksesi Purwokerto, sedangkan klaster IV terdiri dari aksesi Turen-3, aksesi Patuk dan aksesi Kalisoro. Aksesi Citeurep dan aksesi Turen 3 mempunyai hubungan kemiripan yang terdekat dengan nilai indeks kesamaan/kemiripan sebesar 89,36% atau 0,8936.
PEMBAHASAN
Lima kombinasi primer SRAP mengha-silkan fragmen dan tingkat polimorfisme yang cukup tinggi (77,1%), hal tersebut menunjukkan bahwa kombinasi primer SRAP terutama primer ME1-EM2, ME2-EM1 dan ME3-EM2 efektif digunakan untuk karakterisasi genetik tempuyung karena menghasilkan fragmen DNA banyak dan
No Nama Primer Sekuen
1 ME1 5’ TGAGTCCAAACCGGATA 3’ 2 ME2 5’ TGAGTCCAAACCGGAGC 3’ 3 ME3 5’ TGAGTCCAAACCGGAAT 3’ 4 ME4 5’ TGAGTCCAAACCGGACC 3’ 5 EM1 3’ GACTGCGTACGAATTAAT 5’ 6 EM2 3’ GACTGCGTACGAATTTGC 5’ 7 EM3 3’ GACTGCGTACGAATTGAC 5’
Karakterisasi Genetik Tempuyung (Sonchus arvensis L.)
polimorfisme tinggi. Variasi prosentase polimorfis -me menggambarkan perbedaan genetik antar dan di dalam populasi dan atau genotip yang diteliti serta kombinasi primer SRAP yang digunakan selain itu primer yang menghasilkan polimorfisme fragmen tinggi lebih efisien untuk studi keragaman genetik dan untuk membedakan antar genotip (Abdelmigid 2012; Alghamdi 2012).
Adanya fragmen spesifik yang terdapat pada beberapa aksesi jika diamplifikasi menggu-nakan kombinasi primer SRAP tertentu dapat digunakan sebagai penanda untuk karakterisasi ataupun identifikasi aksesi tersebut. Kusumadewi dkk. (2010) menyatakan bahwa pita-pita DNA yang unik dan dijumpai pada populasi tertentu
dapat dijadikan sebagai marka identifikasi, akan tetapi perlu dilakukan penelitian lebih lanjut terkait dengan jenis penanda molekular dan jumlah sampel yang digunakan. Primer SRAP terdiri dari primer forward yang mengamplifikasi daerah ekson dan primer reverse mengamplifikasi daerah intron dan daerah yang mempunyai promoter. Polimorfisme yang teramati berasal dari variasi panjang ekson, intron, promoter dan spacer baik antar individu maupun antar spesies (Li & Quiros 2001). Menurut Noormohammadi et al. (2011) adanya fragmen/lokus spesifik pada beberapa kultivar atau aksesi menunjukkan terjadinya insersi/ delesi di DNA pada genotip dimana hal tersebut berguna untuk perencanaan
No Kombinasi Primer Total fragmen
teramplifikasi Fragmen Monomorfik Prosentase Polimorfik (%) 1 ME1 : 5’ TGAGTCCAAACCGGATA 3’ EM2 : 3’ GACTGCGTACGAATTTGC 5’ 11 3 72,7 2 ME1 : 5’ TGAGTCCAAACCGGATA 3’ EM3 : 3’ GACTGCGTACGAATTGAC 5’ 2 1 50 3 ME2 : 5’ TGAGTCCAAACCGGAGC 3’ EM1 : 3’ GACTGCGTACGAATTAAT 5’ 17 1 94,1 4 ME3 : 5’ TGAGTCCAAACCGGAAT 3’ EM2 : 3’ GACTGCGTACGAATTTGC 5’ 11 3 72,7 5 ME4 : 5’ TGAGTCCAAACCGGACC 3’ EM3 : 3’ GACTGCGTACGAATTGAC 5’ 7 3 57,1 Total 48 11 Rata-rata 77,1
Tabel 3. Total fragmen DNA hasil amplifikasi dengan menggunakan 5 primer SRAP dan prosentase fragmen polimorfik pada 13 aksesi tempuyung.
L 37 36 34 2 3 15 25 27 47 48 49 50 52 L A 100 bp 500 bp 1.000 bp 3.000 bp 300 bp 130 bp 205 bp 500 bp L 37 36 34 2 3 15 25 27 47 48 49 50 52 L 100 bp 500 bp 1.000 bp 3.000 bp B 200 bp
Gambar 1. Fragmen DNA Tempuyung hasil amplifikasi dengan menggunakan primer SRAP (A: kombinasi primer ME1-EM2 dan B: kombinasi primer ME3-EM2)
Subositi & Mujahid.
hibridisasi. Primer SRAP mempunyai desain unik sehingga teknik tersebut lebih reprodusibel, stabil, lebih sederhana dibandingkan dengan teknik molekular lainnya.
Penanda molekular SRAP mampu menunjukkan adanya keragaman genetik diantara 13 aksesi tempuyung pada indeks kemiripan berkisar 74,93%-89,36%. Hal tersebut mengin-dikasikan keragaman genetik yang sempit antar aksesi tempuyung. Klaster II (Gambar 2) menun-jukkan anggota klaster terdiri dari sebagian besar aksesi berasal dari B2P2TO-OT Tawangmangu. Hal tersebut menunjukkan bahwa penanda molekular SRAP mampu mengelompokkan beberapa aksesi-aksesi menjadi satu klaster berda-sarkan lokasi asal sampel atau tempat tumbuh yang sama. Menurut Shafie et al. (2011) tingkat kemiripan yang tinggi berkorelasi dengan kondisi tempat tumbuh yang sama atau mirip. Lima aksesi B2P2TO-OT menjadi satu klaster kemungkinan disebabkan berasal dari tetua yang sama, dibudidayakan dalam jangka waktu lama dan tumbuh pada kondisi lingkungan yang sama. Al-Rawashdeh (2011) menyatakan bahwa
kemiripan atau kesamaan aksesi kemungkinan karena mempunyai tetua yang sama. Hasil yang sama terjadi pada karakterisasi genetik kultivar alfalfa menggunakan penanda molekular SRAP, terdapat klaster yang beranggotakan kultivar alfalfa berdasarkan kemiripan kondisi lingkungan asal (Yuan et al. 2011). Seluruh anggota klaster II merupakan aksesi yang telah dibudidayakan yaitu di B2P2TO-OT Tawangmangu dan di Materia Medika Malang, hal tersebut menunjukkan bahwa penanda molekular SRAP mampu menge-lompokkan aksesi berdasarkan karakter budidaya. Seehalak et al. (2006) menyatakan variasi genetik yang sempit antar aksesi yang telah dibudidayakan kemungkinan disebabkan karena dibudidayakan dalam jangka waktu yang lama sehingga aksesi telah beradaptasi dengan kondisi agroklimat.
Penanda molekular SRAP merupakan pe-nanda paling kuat untuk analisis keanekaragaman genetik pada kultivar rumput Bucholoe dactyloides dibandingkan dengan penanda molekular SSR, ISSR dan RAPD (Budak et al. 2004). Penanda molekular SRAP mampu menujukkan adanya variasi genetik yang sempit antar aksesi
Koefisien 0.70 0.75 0.80 0.85 0.90 Citeurep Turen-2 Mataram Materiamedika B2P2TO-OT(QC) B2P2TO-OT(1) B2P2TO-OT(2) B2P2TO-OT(4) B2P2TO-OT(3) Purwokerto Turen-3 Patuk Kalisoro
Karakterisasi Genetik Tempuyung (Sonchus arvensis L.)
tempuyung. Variasi genetik tersebut kemung-kinan disebabkan adanya perbedaan budidaya dan tetua aksesi tempuyung. Informasi tersebut merupakan salah satu data untuk mendukung standarisasi tempuyung sebagai bahan baku serta penelitian lebih lanjut pengembangan aksesi tempuyung. Penanda molekular SRAP dapat digunakan untuk identifikasi secara molekular pada tempuyung.
UCAPAN TERIMA KASIH
Penelitian ini ini adalah salah satu bagian penelitian yang dibiayai dari DIPA Tahun 2012 Balai Besar Penelitian dan Pengembangan Tum-buhan Obat dan Obat Tradisional (B2P2TO-OT), Badan Litbang Kesehatan.
DAFTAR PUSTAKA
Abdelmigid, HM. 2012. Efficiency of random amplified polymorphic DNA (RAPD) and inter-simple sequence repeats (ISSR) markers for genotype fingerprinting and genetic diversity studies in canola (Brassica napus).
Afri. J. Biotech. 11(24): 6409-6419.
Al-Rawashdeh, IM. 2011. Genetic Variability in a Medicinal Plant Artemisia judaica using Random Amplified Polymorphic DNA (RAPD) Markers. Inter. J. Agri. Ecol. 13:279 -282.
Alghamdi, SS., SA. Al-Faifi, HM. Migdadi, MA. Khan, EH. El-Harty, & MH. Ammar. 2012. Molecular Diversity Assessment Using Sequence Related Amplified Poly-morphism (SRAP) Markers in Vicia faba L. Int. J. Mol.
Sci. 13: 16457-16471.
Backer, CA. & RCB. van den Brink. 1968. Flora
of Java (spermatophytes only) Vol. 2. Walters
Nordoff, NY Groningen, The Netherlands.
Budak, H., RC. Shearman, I. Parmaksiz, RE. Gaussoin, TP. Riordan, & I. Dweikat. 2004. Molecular characterization of buf-falograss germplasm using sequence-related amplified polymorphism markers. Theor. Appl. Genet. 108: 328-334.
Fu, XP., GG. Ning, LP. Gao, & MZ. Bao. 2008. Genetic diversity of Dianthus accessions as assessed using two molecular marker systems (SRAPs and ISSRs) and morpho-logical traits. Sci. Horticult. 117: 263– 270.
Jianfeng, H., Q. Yonghua, M. Hingxia, Z. Chunyang, Y. Zixing, & H. Guibing. 2012. Molecular marker analysis of ‘Shatangju’ and ‘Wuzishatangju’ mandarin (Citrus reticulata Blanco). Afri. J. Biotech. 11(89):15501-15509.
Keyfi, F., & AH. Beiki. 2012. Exploitation of random amplified polymorphic DNA (RAPD) and sequence-related amplified polymorphism (SRAP) markers for genetic diversity of saffron collection. J. Med. Plants
Res. 6(14): 2761-2768.
Kusumadewi, Y., YS. Purba, & T. Partomihardjo. 2010. Keragaman Genetika Ramin [Gonystylus bancanus (Miq.) Kurz] dari Provinsi Riau Berdasarkan Profil Random Amplified Polymorphic DNA. J. Biol.
Indonesia 6(2): 173-183.
Li, G., & CF. Quiros. 2001. Sequence–related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: Its application to mapping and gene tagging in Brassica. Theor. Appl. Genet. 103: 455-461.
Meng, L., HX. Yang, PC. Mao, HW. Gao, & FD. Sun. 2011. Genetic diversity analysis of
Arrhenatherum elatius germplasm with
inter-simple sequence repeat (ISSR) markers. Afri.
Subositi & Mujahid.
Muthusamy, S., S. Kanagarajan, & S. Ponnusamy. 2008. Efficiency of RAPD and ISSR Markers System in Genetic Variation of Rice Bean (Vigna umbellata) Landraces.
Elec. J. Biotech. 11(3): 1-10.
Noormohammadi, Z., F. Shojaei-Jesvaghani, M. Sheidai, F. Farahani, & O. Alishah. 2011. Inter simple sequence repeats (ISSR) and random amplified polymorphic DNA (RAPD) analyses of genetic diversity in Mehr cotton cultivar and its crossing progenies.
Afri. J. Biotech. 10(56): 11839-11847.
Seehalak, W., N. Tomooka, A. Waranyuwat, P. Thipyapong, P. Laosuwan, A. Kaga, & DA. Vaughan. 2006. Genetic diversity of the
Vigna germplasm from Thailand and
neighbouring regions revealed by AFLP analysis. Gen. Res. Crop Evo. 53(5): 1043-1059.
Shafie, SB., SMZ. Hasan, AM. Zain, & RM. Shah. 2011. RAPD and ISSR markers for comparative analysis of genetic diversity in wormwood capillary (Artemisia capillaris) from Negeri Sembilan, Malaysia. J. Med.
Plants Rese. 5(18): 4426-4437.
Xiang, ZXIA., & LJ.Yu. 2010. Steroids and Phenols from Sonchus arvensis. Chin. J. Nat.
Med . 8(4): 267-269.
Yuan, Q., J. Gao, Z. Gui, Y. Wang, S. Wang, X. Zhao, B. Xia, & XL. Li. 2011. Genetic relationships among alfalfa gemplasms resistant to common leaf spot and selected Chinese cultivars assessed by sequence-related amplified polymorphism (SARP) markers. Afri. J. Biotech. 10(59): 12527-12534.
Zhang, F., YY. Ge, WY. Wang, XL. Shen, & XY. Yu. 2012. Assessing genetic divergence in interspecific hybrids of Aechmea gomosepala and A. recurvata var. recurvata using inflorescence characteristics and sequence-related amplified polymorphism markers.
Genet. Mol. Res. 11 (4): 4169-4178.
Zeng, B., GZ. Wang, FY. Zuo, ZH. Chen, & XQ. Zhang. 2012. Genetic diversity analysis of cocksfoot (Dactylis glomerata L.) accessions with sequence-related amplified polymorphism (SRAP) and inter-simple sequence repeat (ISSR) markers. Afri. J.
Jurnal Biologi Indonesia 9(2) 175-182 (2013)
Karakteristik Populasi Labi-labi Amyda cartilaginea (Boddaert, 1770) yang Tertangkap di
Sumatera Selatan
(Population Characteristics of the Asiatic Softshell Turtle Amyda cartilaginea (Boddaert,
1770) Harvested in South Sumatera)
Agus Arifin Sentosa, Danu Wijaya & Astri Suryandari
Balai Penelitian Pemulihan dan Konservasi Sumber Daya Ikan, Jl. Cilalawi No. 01 Jatiluhur, Purwakarta 41152; E-mail: [email protected]
Memasukan: Januari 2013, Diterima: April 2013
ABSTRACT
The Asiatic softshell turtle Amyda cartilaginea (Boddaert, 1770) is one of the reptile commodities included in CITES Appendix II with vulnerable status according to IUCN. The species has been harvesting, especially for export purpose in South Sumatera. The reseach was aimed to know the population characteristics of the Asiatic softshell turtle harvested in South Sumatera. The study was carried out based on enumerators approach from July to Desember 2012 in South Sumatera. The data enumeration also has been collected from the 1st collectors. Data analysis included
the size distribution of carapace curve length (CCL), carapace curve width (CCW), body weight, sex ratio, age structure, CCL-weight relationship and von Bertalanffy growth parameters. The results showed that there were recorded 306 individuals of A. cartilaginea (92% adult) with sex ratio male and female is 42:58. Its has carapace curve length range from 10 to 75.5 cm, carapace curve width 9 to 59.5 cm and body weight 0.02 to 40 kg. A.
cartilaginea growth pattern was negatively allometric (b = 2.727). The von Bertalanffy growth formula of A. cartilaginea in South Sumatera was PLK(t) = 78,75{1-exp[-0,18(t-(-0,72)]} cm.
Keywords: Amyda cartilaginea, population characteristics, South Sumatera ABSTRAK
Labi-labi Amyda cartilaginea (Boddaert, 1770) merupakan salah komoditi reptilia yang dikategorikan ada dalam CITES Appendiks II dengan status IUCN vulnerable. Jenis ini telah digunakan sebagai komoditi ekspor dari matera Selatan. Penelitian ini bertujuan untuk mengetahui karakteristik populasi labi-labi yang dipanen dari Su-matera Selatan. Kajian yang digunakan dalam penelitian ini menggunakan pendekatan yang didasarkan pada hasil penangkapan dari pengumpul selama bulan Juli-Desember 2012 di Sumatera Selatan. Data yang dikumpulkan ada-lah nisbah kelamin, struktur umur, bobot badan panjang lengkung karapas (PLK) dan lebar lengkung karapas (LLK), rasio panjang lengkung karpas dengan bobot badan, dan pola pertumbuhan von Bertalanffy. Hasil penelitian menunjukkan bahwa tercatat ada 306 individu yang digunakan dalam analisis ini yang terdiri dari A. cartilaginea (92% dewasa) dengan nisbah kelamin 42:58. Panjang lengkung karapas 10-75,5 cm, lebar karapas 9-59,5 cm dan bobot badan 0,02-40 kg dengan pola pertumbuhan negative (b=2,727). Formula pertumbuhan von Bertalanffy di Sumatera Selatan adalah PLK(t) = 78,75{1-exp[-0,18(t-(-0,72)]} cm
Kata Kunci: Amyda cartilaginea, karakteristik populasi, Sumatera Selatan
PENDAHULUAN
Labi-labi Amyda cartilaginea (Boddaert, 1770) merupakan jenis kura-kura air tawar dari famili Trionychidae, ordo Testudines yang menyebar luas di Asia Tenggara (Iskandar 2000; van Dijk 2000). Penyebaran A. cartilaginea di
Indonesia dijumpai di Kalimantan, Sumatera, Jawa, Bali, dan Lombok (Auliya 2007; Iverson 1992). Menurut Iskandar (2000), labi-labi ini umumnya dijumpai di daerah yang tenang dan berarus lambat.
Labi-labi, merupakan salah satu satwa air yang masuk ke dalam komoditas perikanan.
Labi-Sentosa, dkk.
labi-labi di provinsi tersebut. Penelitian ini bertujuan untuk mengetahui karakteristik populasi labi-labi (A. cartilaginea) yang tertangkap di Sumatera Selatan. Karakteristik populasi tersebut meliputi sebaran ukuran labi-labi hasil tangkapan, nisbah kelamin, struktur umur, pola pertumbuhan dan persamaan pertumbuhan von Bertalanffy. Hasil penelitian diharapkan dapat bermanfaat dalam rangka penyediaan data dan informasi ilmiah untuk mendukung pengelolaan dan penetapan status perlindungan labi-labi di Indonesia, khususnya di Sumatera Selatan.
BAHAN DAN CARA KERJA
Penelitian dilakukan di Kota Palembang, Kabupaten Ogan Komering Ilir dan Kabupaten Musi Banyuasin Provinsi Sumatera Selatan pada bulan Juli, Oktober dan Desember 2012 (Gambar 1). Pemantauan populasi labi-labi dilakukan dengan pendekatan kunjungan kepada para pengumpul di tingkat pertama (Riyanto & Mumpuni 2003) yang ditentukan secara snowball
sampling berdasarkan informasi dari pengumpul
besar di Palembang. Pengertian pengumpul tingkat pertama adalah pengumpul labi-labi yang menerima hasil tangkapan labi-labi dari alam secara langsung dari penangkap. Survei lapangan berupa peninjauan habitat labi-labi biasa tertangkap juga dilakukan untuk memperoleh gambaran kondisi habitat labi-labi secara umum.
Obyek penelitian adalah labi-labi dari spesies Amyda cartilaginea dengan identifikasi mengacu kepada Ernst & Barbour (1989) dan Iskandar (2000). Data ukuran A. cartilaginea diperoleh dari pengumpul pertama yang kemudian berperan sebagai enumerator pencatat data tangkapan labi-labi. Enumerator tersebut telah dilatih untuk mencatat data ukuran labi-labi yang tertangkap pada log book yang telah disediakan (Oktaviani & Samedi 2008). labi jenis tersebut di Indonesia telah dimanfaatkan
untuk kepentingan konsumsi dan sebagai peliharaan (Kusrini et al. 2009). Pemanfaatan labi -labi di Indonesia sudah berlangsung lama mengingat hewan tersebut termasuk satwa liar yang tidak dilindungi oleh peraturan di Indonesia. Walaupun demikian, secara interna-sional, spesies tersebut telah masuk ke dalam Appendix II CITES dan dikategorikan vulnerable (rentan) pada Red Data Book IUCN.
Salah satu daerah penyebaran labi-labi yang telah diketahui adalah di Sumatera Selatan (Kasmiruddin 1998; Oktaviani & Samedi 2008). Secara umum, wilayah Sumatera Selatan merupakan daerah potensi labi-labi mengingat 93,05% wilayahnya merupakan bagian dari daerah aliran sungai, termasuk di dalamnya daerah rawa. Menurut Oktaviani et al. (2008), topografi wilayah Sumatera Selatan dengan 25% daerah rawa yang mempunyai karakteristik berarus lambat dengan dasar lumpur atau gambut merupakan habitat bagi A. cartilaginea.
Pemanenan labi-labi di Sumatera Selatan yang cukup tinggi mendorong perlunya informasi mengenai aspek biologi populasi labi-labi di daerah tersebut. Penilaian Non Detrimental
Findings (NDF) terhadap pemanfaatan A. cartilaginea di Sumatera Selatan mengindikasikan
bahwa populasi spesies tersebut di alam dalam kondisi terancam kerusakan atau punah (Oktaviani & Samedi 2008).
Informasi ilmiah terkait keberadaan A.
cartilaginea di Sumatera Selatan masih relatif
terbatas, beberapa diantaranya telah dipublikasi-kan oleh Kasmiruddin (1998), Oktaviani & Samedi (2008), Oktaviani et al. (2008) dan Mumpuni & Riyanto (2010). Penelitian lanjutan mengenai beberapa aspek biologi populasi labi-labi di Sumatera Selatan masih diperlukan dalam rangka monitoring populasi dan melengkapi serta menyediakan informasi terkini terkait keberadaan
Karakteristik Populasi Labi-Labi Amyda cartilaginea (Boddaert, 1770)
Penapisan dan pensejajaran tingkat penumpul sebagai sumber data telah dilakukan dalam upaya untuk menghindari pengukuran/perhitungan ganda (Mumpuni & Riyanto 2010). Oleh karena itu, enumerator yang ditunjuk hanya pada tingkat pengumpul pertama yang saling independen.
Setiap individu labi-labi diukur oleh enu-merator segera pada saat kedatangan labi-labi dari penangkap/penjual ke pengumpul. Enumerator tersebut mencatat data hasil tangkapan labi-labi selama bulan Juli hingga Desember 2012. Data yang terkumpul oleh enumerator akan divalidasi terlebih dahulu sebelum digunakan dalam analisis untuk menghindari kesalahan pencatatan.
Parameter yang dicatat dalam format log
book enumerator adalah data morfologi, jenis
ke-lamin dan asal lokasi tangkap labi-labi. Penguku-ran morfologi dilakukan dengan metoda curveline menurut Nuitja (1992), meliputi pengukuran panjang lengkung karapas (PLK) dan lebar lengkung karapas (LLK) menggunakan pita ukuran dengan ketelitian 1 mm serta berat tubuh labi-labi menggunakan timbangan komersial.
PLK diukur mulai anterior hingga posterior pada bagian tengah karapas sedangkan LLK diukur dari sisi kiri ke kanan pada bagian tengah karapas (Oktaviani et al. 2008). Penguku-ran tersebut dilakukan terhadap keseluruhan hasil tangkapan labi-labi. Semua ukuran PLK dan LLK dicatat mengingat pengumpul tingkat pertama
menerima hasil tangkapan labi-labi untuk semua ukuran tanpa seleksi sebelumnya.
Jenis kelamin labi-labi ditentukan dengan membandingkan panjang dan bentuk ekor. Betina umumnya memiliki ekor yang pendek dan gempal, sementara jantan memiliki ekor yang lebih panjang dan ramping (Ernst & Barbour 1989).
Data morfologi yang diperoleh kemudian dianalisis dengan statistik deskriptif meliputi rerata (μ) dan simpangan baku (σ) dan distribusi frekuensi. Perbedaan dimorfisme antar jenis kelamin dilakukan uji t dimana sebelumnya sebaran data tersebut berdistribusi normal (Oktaviani et al. 2008). Analisis nisbah kelamin dilakukan untuk mengetahui proporsi jantan betinanya.
Analisis hubungan PLK-berat dilakukan untuk mengetahui pola pertumbuhan labi-labi (Oktaviani et al. 2008) menggunakan rumus:
W = a (PLK)b
dimana W adalah berat labi-labi (gram), PLK adalah panjang lengkung karapas (cm) serta a dan b adalah konstanta. Nilai b yang diperoleh diuji ketepatannya terhadap nilai b = 3 menggunakan uji-t dengan tingkat kepercayaan 95% (Effendie 2002)
Penentuan struktur umur didasarkan pada hasil pengukuran PLK labi-labi. Klasifikasi struk-tur umur disajikan pada Tabel 1. Data panjang lengkung karapas yang diperoleh dari pengukuran langsung. Data enumerator dikelompokkan da-lam suatu tabel distribusi frekuensi panjang untuk mengetahui parameter pertumbuhan labi-labi yang dianalisis menggunakan model pertumbu-han von Bertalanffy (Macale et al. 2009; Effendie 2002) dengan rumus:
PLK(t) = PLK∞{1-exp[-k(t-to)]}
Metode penentuan panjang lengkung karapas asimtot (PLK∞) dan koefisien pertum-buhan (k) diduga menggunakan metode
Sentosa, dkk.
pherd yang terdapat pada paket perangkat lunak FiSAT II untuk memaksimalkan nilai non parametriknya (Gayanilo et al. 2005). Umur teoritis (to) diduga menggunakan persamaan empiris Pauly (1983) dengan rumus:
log –(to)= -0,3922-0,2752 log PLK∞ - 1,038 log k
HASIL
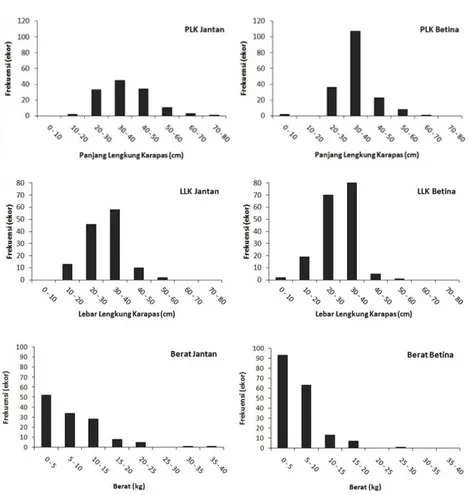
Morfologi dan Sebaran Ukuran
Data hasil pengukuran morfologi dan sebaran ukuran labi-labi disajikan pada Tabel 2 dan Gambar 2. Secara umum, nilai rerata PLK, LLK dan berat labi-labi jantan relatif lebih besar dibandingkan betina. Sebaran ukuran PLK labi-labi jantan cenderung lebih besar dibandingkan betina, namun sebaran LLK cenderung sama. Secara umum, labi-labi paling banyak tertangkap pada kisaran panjang 30 – 40 cm, baik PLK maupun LLK. Sebaran berat tubuh labi-labi jantan dan betina cenderung memiliki pola yang hampir sama dimana modus ukuran berat berada pada kisaran 0–5 kg.
Nisbah Kelamin dan Struktur Umur
Berdasarkan hasil pengamatan terhadap 306 ekor sampel labi-labi didapatkan nisbah kelamin jantan dan betina adalah 1:1,37 atau 42,16% jantan dan 57,84% betina. Walaupun individu betina lebih banyak tertangkap diban-dingkan jantan, namun kedua jenis kelamin labi-labi tersebut memiliki peluang tertangkap yang sama. Struktur umur sampel labi-labi yang berhasil diamati didominasi oleh labi-labi dewasa.
K elas U m u r P L K S tru ktu r U m u r
I = 5,9 cm Tukik (hatchling)
II 6 – 19,9 cm Rem aja III 20 – 24,9 cm D ewasa m uda
IV = 25 cm D ewasa
Pola Pertumbuhan
Hubungan panjang lengkung karapas dan berat labi-labi disajikan pada Tabel 3. Berdasarkan uji t terhadap nilai b = 3 diketahui bahwa nilai b jantan dan betina tidak sama dengan 3 (P<0,05) sehingga dikatakan bahwa pola pertumbuhan labi-labi tersebut bersifat allometrik. Perhitungan untuk labi-labi jantan dan gabungan keduanya menunjukkan pola allometrik negatif (b<3) dimana pertambahan panjang lengkung karapas lebih cepat dari pertambahan beratnya, sedangkan labi-labi betina menunjukkan pola allometrik positif (b>3) dimana pertambahan PLK lebih lambat dari pertambahan beratnya (Effendie 2002).
Pola pertumbuhan tersebut relatif sama dengan penelitian Oktaviani et al. (2008) yang menyebutkan bahwa pola pertumbuhan labi-labi di Sumatera Selatan bersifat allometrik negatif (b<3). Kondisi tersebut diduga terkait dengan kelimpahan makanan, genetik, dan kondisi lingkungan.
Pendugaan parameter pertumbuhan von Bertalanffy untuk A. cartilaginea dengan metode ELEFAN I pada paket program FiSAT menun-jukkan nilai panjang lengkung karapas asimtot (PLK∞) sebesar 78,75 cm, koefisien pertum-buhan (k) sebesar 0,18 tahun-1 dan t
0 sebesar -0,72 tahun dengan indek performansi pertumbu-han (growth performance index/Φ) berkisar antara 2,7 – 4,7 dengan persamaan sebagai berikut:
PLK(t) = 78,75{1-exp[-0,18(t-(-0,72)]} Profil pertumbuhan von Bertalanffy A.
cartilaginea di Sumatera Selatan disajikan pada
Gambar 4.
PEMBAHASAN
Secara umum, labi-labi yang tertangkap di Sumatera Selatan memiliki ukuran yang bervariasi, namun secara umum terdapat ukuran
Tabel 1. Struktur umur A. cartilaginea berdasarkan hasil pengukuran PLK (Kusrini et al. 2007)
Karakteristik Populasi Labi-Labi Amyda cartilaginea (Boddaert, 1770) JANTAN PLK (cm) LLK (cm) Berat (kg) Jumlah (ekor) 129 Min 13 12 0,4 Max 75,5 59,5 40 Rerata 38,34 30,31 8,17 Std 10,923 8,053 6,478 BETINA PLK (cm) LLK (cm) Berat (kg) Jumlah (ekor) 177 Min 10 9 0,02 Max 67 59 28 Rerata 34,97 29,25 5,87 Std 8,124 6,536 3,942
yang dominan tertangkap. Modus ukuran tersebut diduga merupakan labi-labi yang siap untuk ditangkap. Peluang labi-labi jantan dan betina untuk tertangkap adalah sama karena eksploitasi labi-labi oleh penangkap tidak mempertimbangkan jenis kelamin. Penangkapan labi-labi di Sumatera Selatan tidak ada seleksi hasil tangkapan berdasarkan ukurannya, sehingga peluang semua ukuran labi-labi untuk tertangkap adalah sama sehingga sebaran ukuran tangkapan tersebut dianggap dapat mencerminkan kondisi populasi di alam (Mumpuni & Riyanto 2010).
Secara statistik, uji t pada karakter morfologi labi-labi PLK dan berat menunjukkan ada perbedaan antara morfologi jantan dan betina (P<0,05), namun untuk karakter LLK relatif tidak berbeda nyata (P>0,05). Hal tersebut menunjuk-kan adanya dimorfisme seksual. Menurut Oktaviani et al. (2008), penentuan individu jantan dan betina juga dapat dilihat lebih jelas dari perbedaan bentuk dan ukuran ekornya pada individu dewasa yang mempunyai PLK ≥ 25 cm.
Berdasarkan hubungan PLK dengan berat diketahui bahwa labi-labi betina cenderung
memiliki pertumbuhan berat yang lebih cepat. Hal tersebut diduga terkait dengan metabolisme dan pola reproduksi dimana labi-labi betina mengandung telur setelah dibuahi oleh pejantan (Iskandar 2000).
Pendugaan parameter kurvatur PLK∞, k dan to bagi labi-labi dilakukan dengan asumsi bahwa pertumbuhan organisme akan mencapai suatu batas ukuran tertentu dan sampel yang diperoleh dari pengumpul dianggap sebagai sampel acak. Hal yang mendasari penggunaan model pertumbuhan von Bertalanffy bagi spesies
A. cartilaginea adalah penelitian Macale et al.
(2009) yang menduga pertumbuhan kura-kura Mesir (Testudo kleinmanni).
Nilai PLK∞ labi-labi di Sumatera Selatan sebesar 78,75 cm dan nilai tersebut lebih rendah dari yang dilaporkan oleh Lim & Das (1999) yang mencapai 83 cm. Iskandar (2000) menyebutkan bahwa diameter punggung labi-labi dapat mencapai 100 cm, meskipun umumnya hanya hingga 60 cm saja. Perbedaan nilai tersebut diduga terkait dengan perbedaan habitat. Laju eksploitasi yang semakin meningkat juga diduga akan menurunkan nilai panjang asimtot karena hewan tidak memiliki kesempatan untuk tumbuh (Effendie 2002).
A. cartilaginea betina banyak tertangkap di
Sumatera Selatan. Hasil penelitian tersebut sama dengan penelitian Lilly (2010) di Ketapang, Kalimantan Barat dimana labi-labi betina lebih banyak tertangkap dibandingkan jantan. Namun, yang perlu diperhatikan adalah ancaman terhadap pertumbuhan populasi labi mengingat labi-labi betina memiliki potensi reproduksi yang lebih tinggi (Iskandar 2000). Nisbah kelamin labi-labi betina yang lebih besar dibandingkan jantan di alam menunjukkan kondisi populasi yang baik karena labi-labi betina berperan dalam menghasilkan telur yang akan meregenerasi kelas
Tabel 2. Morfologi A. cartilaginea yang terukur di Sumatera Selatan