ANALISIS KEKERABATAN ANGGOTA FAMILIA SOLANACEAE BERDASARKAN SIKUEN DNA DAERAH ITS
(INTERNAL TRANSCRIBED SPACER)
SKRIPSI
Diajukan untuk memenuhi sebagian dari syarat untuk memperoleh gelar
Sarjana Sains Program Studi Biologi
Oleh:
Dina Karina Islami
1102832
PROGRAM STUDI BIOLOGI DEPARTEMEN PENDIDIKAN BIOLOGI
FAKULTAS PENDIDIKAN MATEMATIKA DAN ILMU PENGETAHUAN ALAM UNIVERSITAS PENDIDIKAN INDONESIA
ANALISIS KEKERABATAN ANGGOTA FAMILIA
SOLANACEAE BERDASARKAN SIKUEN DNA
DAERAH ITS (
INTERNAL TRANSCRIBED SPACER
)
Oleh
Dina Karina Islami
Sebuah skripsi yang diajukan untuk memenuhi salah satu syarat memperoleh gelar Sarjana pada Fakultas Pendidikan Matematika dan Ilmu Pengetahuan Alam
© Dina Karina Islami 2015 Universitas Pendidikan Indonesia
Agustus 2015
Hak Cipta dilindungi undang-undang.
LEMBAR PENGESAHAN
ANALISIS KEKERABATAN ANGGOTA FAMILIA SOLANACEAE BERDASARKAN SIKUEN DNA DAERAH ITS
(INTERNAL TRANSCRIBED SPACER) Oleh
Dina Karina Islami
1102832
DISETUJUI DAN DISAHKAN OLEH:
Pembimbing I
Dr. Topik Hidayat, M.Si. NIP. 197004101997021001
Pembimbing II
Dr. Didik Priyandoko, M.Si. NIP. 196912012001121001
Mengetahui,
Ketua Departemen Pendidikan Biologi
PERNYATAAN
Dengan ini saya menyatakan bahwa skripsi dengan judul "ANALISIS
KEKERABATAN ANGGOTA FAMILIA SOLANACEAE
BERDASARKAN SIKUEN DNA DAERAH ITS (INTERNAL TRANSCRIBED SPACER)” ini beserta seluruh isinya adalah benar-benar karya
saya sendiri dan saya tidak melakukan penjiplakan atau pengutipan dengan
cara-cara yang tidak sesuai dengan etika ilmu yang berlaku dalam masyarakat
keilmuan. Atas pernyataan ini, saya siap menanggung sanksi apabila kemudian
ditemukan adanya pelanggaran etika keilmuan atau ada klaim dari pihak lain
terhadap keaslian karya saya ini.
Bandung, Agustus 2015
Dina Karina Islami
KATA PENGANTAR
Puji dan syukur penulis panjatkan kehadirat Allah SWT yang telah
melimpahkan rahmat dan karunia-Nya, serta memberi kelancaran dan kemudahan
untuk melaksanakan penelitian dan penulisan skripsi yang berjudul “Analisis Kekerabatan Anggota Familia Solanaceae Berdasarkan Sikuen DNA Daerah ITS
(Internal Transcribed Spacer)”, sehingga penulis dapat menyelesaikan skripsi ini dengan baik.
Banyak pihak yang berperan dan membantu dalam melaksanakan penelitian
dan penulisan. Skripsi ini tidak akan terselesaikan tanpa bantuan dari pihak-pihak
tersebut. Penulis mengucapkan terima kasih dan penghargaan yang
sebesar-besarnya kepada:
1. Bapak Dr. Topik Hidayat, M. Si. selaku dosen pembimbing I yang dengan
sabar memberikan bimbingan, ilmu pengetahuan, dan motivasi selama
penelitian dan penulisan sehingga skripsi ini dapat diselesaikan dengan baik.
2. Bapak Dr. Didik Priyandoko, M. Si. selaku dosen pembimbing II dan Ketua
Program Studi Biologi FPMIPA UPI yang telah memberikan ilmu
pengetahuan, bimbingan, dan motivasi selama penelitian dan penulisan
skripsi.
3. Bapak Dr. Bambang Supriatno, M. Si. selaku Ketua Departemen Pendidikan
Biologi FPMIPA UPI.
4. Seluruh dosen dan staff Departemen Pendidikan Biologi FPMIPA UPI.
5. Bapak Rahadian Deden Juansah, S.Pd. selaku laboran Laboratorium
Mikrobiologi dan Bapak Sarna Suryana, M. Pd. Selaku laboran Laboratorium
Struktur Tumbuhan FPMIPA UPI yang telah sangat membantu memberikan
fasilitas, bimbingan, motivasi, serta berbagai kemudahan selama penelitian di
Laboratorium Mikrobiologi dan Struktur Tumbuhan.
6. Kedua orang tua, Mama Elly Tri Yuliani dan Papa Ayi Kustiaji, serta adik
Muhammad Amal Ramdhani dan Fadiah Idzni Maimuni atas segala doa,
motivasi, dukungan, pengorbanan, dan cinta yang tiada hentinya kepada
7. Putri Yunitha Wardiny, S. Si. selaku rekan satu penelitian sekaligus keluarga
di kampus yang dengan sabar untuk selalu bersama-sama berjuang sejak awal
perkuliahan, penelitian, penulisan skripsi, hingga akhirnya memperoleh gelar
sarjana ini.
8. Rekan-rekan seperjuangan Laboratorium Mikrobiologi Nilamsari K. I., S. Si.,
Pina Rosica, S. Si., Puji Nurhayat, S. Si., Amelia Qadaryanti, S. Si., atas
kebersamaan, diskusi, motivasi, dan segala suka duka yang dialami bersama.
9. Hestiarahma Purnamasari, S. Si., Angga Kly Sandy, S. Si., Sandy Ahmad
Herdiansyah, S. Si., Radita Maulasari, S. Si., dan Afri Irawan, S. Si. untuk
selalu ada dalam suka duka selama 4 tahun ini.
10. Keluarga Tocsic’11 yang selalu memberikan warna selama masa perkuliahan. 11. Semua kakak tingkat Biologi atas semua bantuan, saran, motivasi, dan
dukungan yang telah diberikan.
12. Semua pihak yang tidak dapat disebutkan satu persatu.
Semoga diberi keberkahan dan balasan yang lebih baik oleh Allah SWT atas
segala jasa yang diberikan kepada penulis.
Penulis menyadari masih banyak kekurangan dalam penulisan skripsi ini,
namun penulis berharap hasil penelitian ini dapat bermanfaat untuk penelitian
selanjutnya. Oleh karena itu, penulis mengharapkan saran dan kritik yang
membangun untuk perbaikan pada masa yang akan datang. Semoga skripsi ini
dapat memberikan sumbangan ilmu pengetahuan yang berperan dalam kemajuan
ilmu dan teknologi bagi pembaca.
Bandung, Agustus 2015
DAFTAR RIWAYAT HIDUP
Penulis bernama lengkap Dina Karina Islami, lahir di Bandung pada
tanggal 22 November 1993 merupakan anak pertama dari dua bersaudara dari
pasangan Ayi Kustiaji dan Elly Tri Yuliani. Penulis mulai mengenyam pendidikan
di Taman Kanak-Kanak Aisyiyah Bustanul Athfal XIX pada tahun 1998. Pada
tahun 1999 penulis melanjutkan studi ke SDN Ciporeat 3. Setelah lulus pada
tahun 2005, penulis melanjutkan pendidikan ke SMP Negeri 8 Bandung. Tiga
tahun setelahnya, penulis melanjutkan pendidikan di SMA Negeri 24 Bandung.
Tahun 2011 penulis melanjutkan jenjang pendidikannya di Universitas Pendidikan
Indonesia, Program Studi Biologi, Departemen Pendidikan Biologi. Selama
perkuliahan, penulis aktif sebagai asisten praktikum pada mata kuliah Botani
Cryptogamae selama tiga tahun ajaran berturut-turut, yaitu 2012-2013,
2013-2014, dan 2014-2015, serta pada mata kuliah Ekologi Umum pada tahun ajaran
ANALISIS KEKERABATAN ANGGOTA FAMILIA SOLANACEAE BERDASARKAN SIKUEN DNA DAERAH ITS
(INTERNAL TRANSCRIBED SPACER)
ABSTRAK
Solanaceae merupakan familia tumbuhan dengan tingkat keanekaragaman yang tinggi sehingga diperlukan penanda molekuler untuk memberikan karakter yang lebih akurat dalam membentuk pola kekerabatan. Internal Transcribed Spacer (ITS) merupakan penanda molekuler universal yang sering digunakan dalam analisis kekerabatan tumbuhan tinggi maupun rendah. Penelitian ini bertujuan untuk melihat pengelompokan anggota familia Solanaceae berdasarkan daerah ITS-1; 5,8S; dan ITS-2 secara terpisah, serta gabungan dari ketiganya, selain itu juga untuk mengetahui konsistensi pengelompokan tersebut dengan pengelompokan yang telah ada sebelumnya. Analisis filogenetik dilakukan terhadap dua puluh spesies anggota Solanaceae yang terdiri dari sebelas genus serta tiga outgroup yang berasal dari familia Convolvulaceae, Apocynaceae, dan Plantaginaceae. Pembentukan pohon filogeni menggunakan software Mega versi 4 dengan metode bootstrap test of phylogeny dan maximum parsimony dengan data yang berasal dari ITS-1; 5,8S; ITS-2, serta gabungan ketiganya. Hasil dari penelitian ini menunjukkan bahwa pohon dengan data yang berasal dari gabungan daerah ITS membentuk pola kekerabatan secara tepat dibandingkan jika hanya menggunakan salah satu daerah ITS saja. Terdapat tiga kelompok besar yang terbentuk yaitu kelompok I terdiri dari supergenus Datureae, Cestreae, dan Petuninae, kelompok II terdiri dari supergenus Withaninae dan Physalinae yang merupakan anggota dari tribe Physaleae, serta kelompok III terdiri dari supergenus Solaneae. Penggunaan ITS sebagai penanda molekuler mendukung pengelompokan yang telah ada sebelumnya.
PHYLOGENETIC ANALYSIS OF SOLANACEAE BASED ON ITS (INTERNAL TRANSCRIBED SPACER) SEQUENCE
ABSTRACT
Solanaceae is a family of plants with high diversity so that the molecular marker needed to provide more accurate character in determine of relationship among Solanaceae members. Internal Transcribed spacer (ITS) is a universal molecular marker that is often used for relationship analysis of plants. This study aims to find out relationship among the member of Solanaceae family based on ITS-1; 5,8S; ITS-2; and the combination of all, and also to determine the consistency of the relationship based on previous study. Phylogenetic analysis has been conducted on twenty species of eleven genera Solanaceae and three outgroup derived from family Convolvulaceae, Apocynaceae, and Plantaginaceae. Phylogeny tree analysis using software Mega version 4 with bootstrap test of phylogeny and maximum parsimony method with data derived from the ITS-1; 5,8S; ITS-2, and the combination of all. Results from this study indicate that phylogeny tree with the data derived from the combined sequences of ITS establish relationship patterns accurately than if use only one of ITS region. There are three major groups formed namely group I consists of supergenus Datureae, Cestreae, and Petuninae; group II consists of supergenus Withaninae and Physalinae that are member of tribe Physaleae, and group III consists of supergenus Solaneae. The use of ITS as a molecular markers support the Solanaceae relationship that has been studied before.
DAFTAR ISI
BAB II ANALISIS FILOGENETIK MOLEKULER FAMILIA SOLANACEAE
2.1 Sistem Klasifikasi……….
2.4 Daerah Internal Transcribed Spacer (ITS)………..
2.5 Polymerase Chain Reaction (PCR)………..
3.2 Sampel Penelitian……….
3.5.3 Pengukuran Konsentrasi dan Kemurnian DNA………...
3.5.4 Elektroforesis………...
3.5.5 Amplifikasi DNA daerah ITS………..
3.5.6 Sikuensing DNA………..
BAB IV HASIL PENELITIAN DAN PEMBAHASAN………......
4.1 Isolasi DNA………..
4.2 Amplifikasi DNA Daerah ITS……….
4.3 Verifikasi Data Hasil Sikuensing……….
4.4 Struktur daerah ITS………..
4.5 Hubungan Kekerabatan Anggota Solanaceae………...
4.6 Implikasi taksonomi……….
4.7 Aplikasi dalam Bidang Pertanian……….
36
BAB V KESIMPULAN DAN SARAN………
DAFTAR TABEL
Tabel Halaman
2.1 Konsentrasi gel agarosa untuk memisahkan DNA dengan berbagai
rentang berat molekul……….. 24
3.1 Daftar Tumbuhan yang Digunakan dalam Penelitian………. 26
3.2 Alat-alat yang Digunakan dalam Penelitian……… 28
3.3 Bahan-bahan yang Digunakan dalam Penelitian………. 28
4.1 Hasil Spektrofotometri DNA Solanaceae dan Outgroup……… 36
4.2 Hasil Spektrofotometri Amplikon DNA Daerah ITS Solanaceae dan Outgroup………... 40
4.3 Panjang daerah ITS pada Anggota Familia Solanaceae dan Outgroup 43 4.4 Sikuen DNA daerah ITS Anggota Solanaceae dan Outgroup……….. 45
DAFTAR GAMBAR
Gambar Halaman
2.1 Spesies-spesies Schizanthus………. 9
2.2 Heterostilus pada bunga………... 10
2.3 Peta persebaran Solanaceae………. 11
2.4 Pohon kekerabatan dan polarisasi karakter dalam analisis filogenetik……….... 14
2.5 Susunan nrDNA dan daerah ITS………. 16
2.6 Siklus PCR………... 21
2.7 Skema Elektroforesis………... 22
3.1 Skema Isolasi DNA………. 31
3.2 Data yang dihasilkan pada proses alignment setelah melalui proses editing………... 34
3.3 Alur Penelitian…………...……….. 35
4.1 Elektroforegram hasil isolasi DNA……….. 38
4.2 Elektroforegram hasil amplifikasi DNA daerah ITS………... 39
4.3 Contoh uji homologi Physalis angulata menggunakan program BLAST………... 41
4.4 Contoh hasil BLAST sikuen DNA sampel……….. 42
4.5 Pohon filogeni berdasarkan daerah ITS-1……… 50
4.6 Pohon filogeni berdasarkan daerah 5,8S……….. 51
4.7 Pohon filogeni berdasarkan daerah ITS-2……… 52
4.8 Pohon filogeni gabungan daerah ITS………... 54
4.9 Bunga anggota kelompok I……….. 55
4.10 Buah A. Brugmansia suaveolens, B. Brugmansia candida…………. 56
4.11 Bunga A. Brugmansia, B. Datura……… 56
4.12 Bunga A. Cestrum cultum, B. Cestrum nocturnunm………... 57
4.13 Buah yang diselimuti kaliks………. 58
lycopersicum, E. S. tuberosum, F. S. nigrum, G. S. pseudocapsicum,
H. Capsicum annuum………... 58
4.15 Plasentasi aksilaris pada buah A. Solanum melongena, B. S. viarum, C. S. lycopersicum, D. S. tuberosum, E. S. nigrum, F. S. pseudocapsicum, G. Capsicum annuum……….. 59
4.16 Bunga A. C. annuum, B. S. pseudocapsicum………... 61
4.17 Buah A. C. annuum, B. S. pseudocapsicum………. 61
DAFTAR LAMPIRAN
Lampiran Halaman
I Hasil Sikuensing Daerah ITS Solanaceae dan Outgroup……… 73
II Dokumentasi Morfologi Solanaceae dan Outgroup……… 113
III Daftar Alat dan Bahan………. 117
BAB I PENDAHULUAN
1.1Latar Belakang
Indonesia merupakan negara kepulauan terbesar di dunia yang tertelak
di garis khatulistiwa. Secara geografis, letak Indonesia berada pada garis 6°
LU-11° LS dan 95° BT-141° BT (Sukojo, 2003, hlm. 30). Iklim tropis serta
curah hujan yang tinggi sepanjang tahun merupakan akibat dari letak
geografis Indonesia. Selain itu, Indonesia pun memiliki topografi
berbeda-beda di setiap daerahnya sehingga menyebabkan Indonesia kaya akan sumber
daya hayati. Hal ini dikemukakan oleh Herbarium Bogoriense (2005, hlm.
28) yang menyatakan bahwa Indonesia sebagai salah satu negara mega
biodiversitas dengan tingkat keanekaragaman yang tinggi di dunia, salah
satunya keanekaragaman tumbuhan. Pernyataan tersebut diperkuat oleh hasil
penelitian yang dilakukan oleh Webb et al. (2010, hlm.7), bahwa hingga
tahun 2009, baru teridentifikasi sekitar 51.911 jenis tumbuhan yang terdapat
di Indonesia, 25.000 diantaranya merupakan tumbuhan berbiji.
Tumbuhan berbiji terbagi ke dalam dua kelas, yaitu Liliopsida
(monokotiledon), dan Magnoliopsida (dikotiledon). Familia-familia yang
tergolong ke dalam Liliopsida diantaranya Musaceae dan Zingiberaceae,
sedangkan familia-familia yang tergolong ke dalam Magnoliopsida
diantaranya Convolvulaceae, Plantaginaceae, Apocynaceae, dan Solanaceae.
Penyebaran tumbuhan berbiji di seluruh dunia tergolong tinggi.
Tumbuhan-tumbuhan tersebut dapat hidup di berbagai daerah di dunia dengan kondisi
yang sesuai bagi pertumbuhannya.
Salah satu familia tumbuhan yang memiliki tingkat penyebaran tinggi
di dunia adalah familia Solanaceae, meliputi 85 genus dengan jumlah spesies
mencapai 2800 dan paling banyak ditemukan di negara-negara beriklim tropis
(Cronquist, 1981, hlm. 895), salah satunya Indonesia. Hal ini disebabkan
karena iklim, curah hujan, dan faktor edafik di Indonesia sesuai bagi
2
Banyak anggota familia Solanaceae yang merupakan tumbuhan liar
(Cronquist, 1981, hlm. 895), tumbuh subur di beberapa daerah Indonesia.
Sebagai contoh, Solanum viarum (terong hutan berduri) dan Solanum
mauritianum (takokak hutan). Anggota Solanaceae selain tumbuh sebagai tanaman liar, banyak pula yang dikembangbiakkan di perkebunan dan
pertanian. Hal ini dikarenakan manfaat dari tanaman Solanaceae itu sendiri.
Berbagai manfaat tersebut antara lain sebagai sumber makanan, seperti pada
Solanum nigrum (leunca), Solanum lycopersicum (tomat), Solanum melongena (terong), dan Solanum tuberosum (kentang); sebagai tanaman hias seperti Petunia grandiflora (petunia), Solandra physalodes, dan Cestrum
(kembang dayang); sebagai bahan baku pembuatan rokok yaitu Nicotiana
tabacum (tembakau); dan juga sebagai tanaman obat, seperti Physalis angulata (ciplukan).
Beragam manfaat yang dimiliki tumbuhan-tumbuhan anggota
Solanaceae dapat dijadikan sebagai dasar pengelompokan tumbuhan.
Dasar-dasar pengelompokan seperti ini dikenal sebagai sistem klasifikasi alami
(Subagja, 2006, hlm. 102). Pengelompokan tumbuhan ini sangat diperlukan,
tujuannya yaitu untuk mempermudah dalam mempelajari tumbuhan tersebut
agar diketahui manfaatnya bagi kehidupan manusia. Selain sistem klasifikasi
alami, dikenal pula sistem klasifikasi buatan yang dipopulerkan oleh Linnaeus
pada zaman pertengahan. Sistem klasifikasi buatan ini mengelompokkan
organisme berdasarkan kesamaan ciri morfologi yang dimilikinya (Subagja,
2006, hlm. 102).
Selain untuk memudahkan dalam mempelajari dan mengetahui
manfaat dari suatu tumbuhan, sistem klasifikasi pun memiliki tujuan untuk
mengetahui kekerabatan antar satu jenis tumbuhan dengan jenis tumbuhan
lainnya. Pengelompokan yang biasa digunakan dalam menentukan
kekerabatan adalah pengelompokan secara filogenetik.
Sistem klasifikasi tumbuhan secara filogenetik merupakan
pengelompokan tumbuhan berdasarkan proses perjalanan evolusi. Kelompok
yang memiliki banyak persamaan ciri dianggap berkerabat dekat dan berasal
3
memerlukan outrgoup untuk menunjukkan polarisasi karakter (Hidayat &
Pancoro, 2006, hlm. 2). Karakter-karakter yang menunjukkan ciri khas dari
suatu tumbuhan perlu diketahui secara tepat agar pada akhirnya didapatkan
pohon filogenetik yang akurat.
Penggunaan karakteristik morfologi tumbuhan untuk menentukan
kekerabatan, meliputi karakteristik akar, batang, daun, buah, dan bunga saat
ini dirasakan kurang akurat. Banyak perdebatan mengenai pengelompokan
tersebut. Hal ini dikarenakan karakter morfologi tumbuhan sangat bervariasi
dan dapat berubah tergantung kondisi lingkungan. Berubahnya morfologi
tumbuhan akibat kondisi lingkungan seperti terjadinya plastisitas fenotip pada
tumbuhan. Plastisitas fenotip merupakan salah satu cara tumbuhan dalam
mengatasi bervariasinya faktor lingkungan (Gratani, 2014, hlm. 1). Selain itu,
perbedaan dari sudut pandang orang yang meneliti pun dapat memberikan
hasil yang tidak konsisten terhadap karakter morfologi suatu tumbuhan
(Hamdan et al, 2013, hlm. 30). Karena keterbatasan karakteristik morfologi
seperti yang telah disebutkan sebelumnya, mendorong perkembangan penanda
lain yang dapat langsung mengakses ke bagian material yang mengendalikan
karakter suatu individu yang dikenal dengan penanda molekuler (Zulfahmi,
2013, hlm. 41).
Penanda molekuler merupakan segmen DNA tertentu yang mewakili
perbedaan pada tingkat genom. Karena berkaitan dengan materi genetik,
tingkat akurasi penanda molekuler lebih sesuai dalam menghasilkan pola
kekerabatan. Penanda molekuler digolongkan ke dalam dua jenis, yaitu
penanda molekuler tanpa PCR dan penanda molekuler berdasarkan PCR.
Salah satu penanda molekuler berdasarkan PCR adalah Internal Transcribed
Spacer (ITS) (Zulfahmi, 2013, hlm. 46).
Penanda Internal Transcribed Spacer merupakan salah satu penanda
molekuler DNA yang sering digunakan oleh para ahli untuk menganalisis
kekerabatan pada tumbuhan, baik tumbuhan tingkat tinggi maupun tumbuhan
rendah (Schoch et al., 2012, hlm. 6244). Menurut Baldwin et al. (1995, hlm.
249), ITS memiliki beberapa keunggulan untuk analisis filogenetik
4
DNA genom, mudah mengalami mutasi sehingga dapat menunjukkan variasi
sikuen yang dapat membantu menentukan pola kekerabatan, dan berukuran
kecil yaitu kurang dari 700 pasang basa.
Penanda molekuler DNA daerah ITS ini dapat digunakan dalam
meneliti hubungan kekerabatan antar anggota familia Solanaceae. Hal ini
disebabkan karena belum banyak penelitian mengenai kekerabatan anggota
familia Solanaceae menggunakan penanda molekuler, khususnya di Indonesia,
padahal penyebaran tumbuhan anggota familia Solanaceae di Indonesia cukup
tinggi dengan berbagai manfaat yang dimilikinya. Penanda DNA daerah ITS
pun memiliki kemampuan-kemampuan yang membuatnya mudah untuk
ditangani. Berdasarkan hal-hal tersebut, maka dalam penelitian ini dilakukan
analisis kekerabatan anggota familia Solanaceae berdasarkan penanda DNA
daerah ITS.
1.2Rumusan Masalah
Berdasarkan latar belakang yang telah dikemukakan sebelumnya,
maka rumusan masalah pada penelitian ini adalah bagaimana kekerabatan
anggota familia Solanaceae berdasarkan Sikuen DNA daerah ITS?
1.3Pertanyaan Penelitian
Berdasarkan rumusan masalah yang telah diungkapkan, maka terdapat
beberapa pertanyaan penelitian sebagai berikut:
1.3.1 Bagaimana pola kekerabatan anggota familia Solanaceae jika
menggunakan sikuen DNA ITS-1; 5,8S; dan ITS-2 secara terpisah;
serta gabungan dari ketiga sikuen tersebut?
1.3.2 Apakah sikuen DNA daerah ITS mendukung pengelompokan anggota
familia Solanaceae yang telah ada sebelumnya?
1.4Batasan Masalah
Batasan masalah pada penelitian ini adalah:
1.4.1 Sampel tumbuhan yang digunakan dalam penelitian ini adalah dua
5
Physalis, Solanum, Brugmansia, Capsicum, Nicotiana, Withania, Cestrum, Petunia, Datura, Nicandra, dan Brunfelsia. Tiga outgroup digunakan dalam penelitian ini, yaitu familia Convolvulaceae diwakili
oleh Ipomoea, Plantaginaceae menggunakan genus Plantago, dan
Allamanda yang merupakan genus dari familia Apocynaceae.
1.4.2 Metode analisis kekerabatan yang digunakan adalah metode filogenetik.
1.4.3 Primer yang digunakan adalah primer ITS-4 (5’
-CCCGCCTGACCTGGGGTCGC-3’) sebagai reverse primer dan ITS-5 (5’-TAGAGGAAGGAGAAGTCGTAACAA-3’) sebagai forward primer.
1.5Tujuan Penelitian
Tujuan yang ingin dicapai dari penelitian ini adalah:
1.5.1 Mengetahui hubungan kekerabatan anggota familia Solanaceae
berdasarkan sikuen DNA daerah ITS-1; 5,8S; dan ITS-2 secara terpisah,
serta gabungan dari ketiganya.
1.5.2 Mengetahui konsistensi pengelompokan kekerabatan anggota familia
Solanaceae berdasarkan DNA daerah ITS dengan pengelompokan yang
telah ada sebelumnya.
1.6Asumsi
1.6.1 Analisis filogenetik berdasarkan sikuen DNA daerah ITS pada berbagai
spesies tumbuhan Solanaceae dalam satu genus menunjukkan adanya
kesamaan baik dalam analisis secara morfologi maupun menggunakan
penanda molekuler lainnya (Marshall et al., 2001, hlm. 1216).
1.6.2 Unit berulang DNA daerah ITS tidak berevolusi secara independen,
tetapi relatif homogen dalam suatu spesies (Fritz et al., 1994, hlm. 406).
1.7Manfaat Penelitian
Manfaat dari penelitian ini adalah:
1.7.1 Sebagai informasi awal mengenai sistem klasifikasi berbasis DNA bagi
6
1.7.2 Sebagai penelitian awal bagi penelitian lanjutan yang bertujuan untuk
mencari alternatif tumbuhan lain dengan kandungan senyawa kimia
yang sama berdasarkan hubungan kekerabatan yang paling dekat.
1.7.3 Mengetahui spesies-spesies yang berkerabat dekat dalam rangka
BAB III
METODE PENELITIAN
3.1 Jenis Penelitian
Penelitian ini merupakan jenis penelitian dasar dengan menggunakan
metode deskriptif, yaitu untuk menganalisis hubungan kekerabatan antar
anggota familia Solanaceae dengan cara merekonstruksi pohon filogenetik
berdasarkan siuken DNA daerah ITS.
3.2 Sampel Penelitian
Sampel pada penelitian ini adalah sebanyak dua puluh spesies tumbuhan
dari sebelas genus anggota familia Solanaceae. Familia Convolvulaceae,
Apocynaceae, dan Plantaginaceae digunakan sebagai outgroup. Pemilihan
familia Convolvulaceae sebagai outgroup didasarkan pada penelitian
Stefanovic et al., (2002, hlm. 1510) yang menyatakan bahwa Convolvulaceae
merupakan sister group dari familia Solanaceae. Sedangkan Plantaginaceae,
dan Apocynaceae dipilih sebagai outgroup karena memiliki kekerabatan yang
dekat dengan familia Solanaceae (Melotto-Passarin et al., 2008, hlm. 91;
Haston et al., 2009, hlm. 161). Sikuen DNA daerah ITS untuk genus
Capsicum dan Withania diperoleh dari genbank dengan alamat http://www.ncbi.nlm.nih.gov/. Daftar tumbuhan yang digunakan pada
penelitian ini tercantum dalam Tabel 3.1.
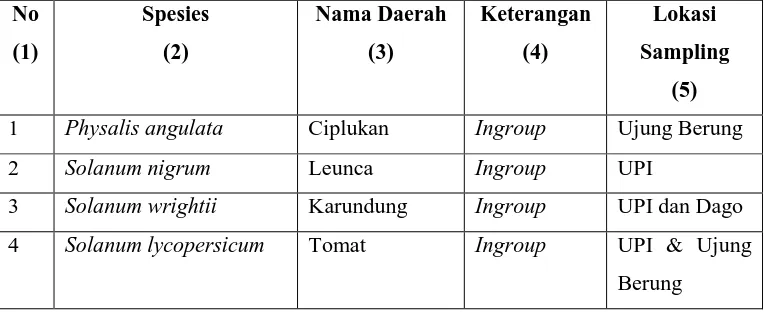
Tabel 3.1 Tumbuhan yang Digunakan dalam Penelitian
No
1 Physalis angulata Ciplukan Ingroup Ujung Berung
2 Solanum nigrum Leunca Ingroup UPI
3 Solanum wrightii Karundung Ingroup UPI dan Dago
4 Solanum lycopersicum Tomat Ingroup UPI & Ujung
27
5 Solanum viarum Terong hutan Ingroup UPI
(1) (2) (3) (4) (5)
6 Solanum melongena Terong ungu Ingroup Ujung Berung
7 Nicotiana tabacum Tembakau Ingroup Ujung Berung
8 Brugmansia suaveolens Kecubung orange Ingroup UPI
9 Brugmansia candida Kecubung putih Ingroup UPI
10 Brunfelsia uniflora Manacá Ingroup UPI
11 Cestrum nocturnum Kembang dayang Ingroup UPI
12 Solanum pseudocapsicum Leunca kuning Ingroup UPI
13 Capsicum annum Cabai merah Ingroup Genbank
14 Withania somnifera Ashwagandha Ingroup Genbank
15 Datura metel Kecubung ungu Ingroup Cihideung
16 Petunia grandiflora Petunia Ingroup Cihideung
17 Nicandra physalodes Ciplukan keras Ingroup UPI
18 Cestrum cultum Kembang dayang Ingroup Dago
19 Physalis peruviana Ciplukan Ingroup Dago
20 Solanum tuberosum Kentang Ingroup Cimahi
21 Plantago major Ki sendok Outgroup UPI & Dago
22 Allamanda cathartica Alamanda Outgroup UPI
23 Ipomoea cairica Bunga terompet Outgroup UPI
3.3 Lokasi Penelitian
Penelitian ini dilakukan di Laboratorium Mikrobiologi Departemen
Pendidikan Biologi Fakultas Pendidikan Matematika dan Ilmu Pengetahuan
Alam Universitas Pendidikan Indonesia, Bandung. Sikuensing hasil
amplifikasi DNA daerah ITS dari sampel-sampel tumbuhan dilakukan di
Macrogen Inc., Seoul-Korea.
3.4 Alat dan Bahan
Fungsi alat-alat yang digunakan dalam penelitian ini terdapat pada Tabel
28
Tabel 3.2 Alat-alat yang Digunakan dalam Penelitian
No Alat Fungsi
1 Autoclave Sterilisasi
2 Mikropipet Mengambil larutan
3 Tabung mikro Tempat isolasi DNA dan penyimpanan
DNA genom
4 Tabung PCR Tempat penyimpanan amplikon
5 Lumpang dan alu Menghaluskan sampel
6 Vorteks Menghomogenkan larutan
7 Timbangan digital Mengukur masa bahan
8 Spatula Mengambil bahan
9 Penangas air dan shaker Shocking saat isolasi DNA
10 Shaker Mencampurkan larutan
11 Mesin sentrifugasi Memisahkan fasa larutan
12 Freezer Menyimpan sampel
13 Spektrofotometer Mengukur konsentrasi DNA
14 Microwave Memanaskan bahan
15 Mesin elektroforesis Memisahkan sampel DNA
16 UV transluminator Melihat hasil elektroforesis
17 Mesin PCR Amplifikasi
18 Magnetic Stirrer with Hot Plate Menghomogenkan larutan
Bahan-bahan yang digunakan dalam penelitian beserta fungsinya terdapat
pada Tabel 3.3.
Tabel 3.3 Bahan-bahan yang Digunakan dalam Penelitian
No Bahan Fungsi
1 Kit isolasi DNA tumbuhan Isolasi DNA
2 Nitrogen cair Menghaluskan sampel
3 Buffer TAE Penyangga untuk elektroforesis DNA
4 ddH2O Melarutkan bahan
5 RNAse Mendegradasi molekul RNA
6 Agarose Media untuk mengelektroforesis DNA
7 DNA ledder Penanda ukuran pita DNA
8 Loading dye Pemberat dan pewarna molekul DNA
9 EtBr Pewarna DNA pada proses elektroforesis
10 Larutan TE Melarutkan bahan
11 PCR master mix Campuran bahan-bahan untuk amplifikasi
12 Primer ITS-5 dan ITS-4 Pembatas fragmen DNA target saat
amplifikasi
29
Daftar alat dan bahan yang digunakan dalam penelitian secara rinci
terdapat dalam lampiran III.
3.5 Prosedur Kerja 3.5.1. Sampling
Sampel tumbuhan didapatkan dari beberapa daerah di Bandung,
diantaranya Ujung Berung, Dago, Cihideung, dan Setiabudhi, serta Kota
Cimahi. Secara garis besar morfologi tumbuhan diamati dan
didokumentasikan. Sampel yang diambil dari tiap-tiap tumbuhan yaitu daun
muda, disimpan di dalam kantung plastik, diberi label, dan disimpan di
dalam box berisi es. Selanjutnya di laboratorium, sampel tumbuhan
disimpan di dalam freezer pada suhu -20°C. Penyimpanan sampel tumbuhan
pada suhu dibawah 0°C ini dimaksudkan untuk menjaga DNA sampel agar
tidak rusak.
3.5.2. Isolasi DNA Genom
DNA tumbuhan anggota suku Solanaceae diisolasi menggunakan kit
dari Thermo Scientific. Tahapan isolasi DNA menggunakan protokol dari
kit tersebut dengan sedikit modifikasi (Gambar 3.1). Sebanyak 100 mg daun
muda dihaluskan di dalam lumpang dan alu yang berisi nitrogen cair hingga
daun muda tersebut berbentuk serbuk. Selanjutnya, serbuk daun muda
dimasukkan ke dalam tabung mikro berisi 350 µl lysis buffer A, kemudian
sampel dihomogenkan menggunakan vorteks selama lima belas detik.
Sampel yang telah homogen ditambahkan lysis buffer B dan RNAse,
masing-masing sebanyak 50 µl dan 5 µl. Sampel diinkubasi di dalam
penangas air bersuhu 65°C selama sepuluh menit. Selama inkubasi, sampel
dihomogenkan menggunakan shaker dalam penangas.
Setelah diinkubasi, sampel diangkat, kemudian ditambahkan
precipitation solution sebanyak 130 µl. Larutan tersebut dihomogenkan dengan cara membolak-balikkan tabung mikro sebanyak 2-3 kali.
Selanjutnya, sampel dimasukkan ke dalam kotak berisi es selama lima
30
kecepatan 14.000 rpm. Supernatan yang diperoleh dari hasil sentrifugasi
dipindahkan kedalam tabung mikro baru kemudian diberi larutan plant
gDNA binding solution dan etanol 96%, masing-masing sebanyak 400 µl, selanjutnya dihomogenkan dengan cara membolak-balikkan tabung mikro.
Sebanyak setengah volume sampel (600-700 µl) dimasukkan ke dalam
spin column with collection tube dan disentrifugasi selama satu menit dengan kecepatan 8.000 rpm. Cairan yang terdapat pada bagain bawah dari
collection tube dibuang. Hal yang sama dilakukan untuk sisa volume sampel lainnya. Setelah semua sampel disaring pada spin column, ditambahkan
wash buffer I sebanyak 500 µl dan disentrifugasi selama satu menit dengan kecepatan 8.000 rpm. Setelah disentrifugasi, cairan yang berada dibawah
collection tube dibuang. Ditambahkan wash buffer II sebanyak 500 µl kedalam spin column, kemudian disentrifugasi selama tiga menit dengan
kecepatan 14.000 rpm. Setelah itu, tabung dikosongkan dan dispin kembali
selama satu menit dengan kecepatan 14.000 rpm.
Spin column dipindahkan ke dalam tabung mikro baru, ditambahkan 50 µl elution buffer ke tengah column membrane, kemudian diinkubasi
selama lima menit pada suhu ruangan. Setelah selesai inkubasi, spin column
dan tabung mikro tersebut disentrifugasi selama satu menit dengan
kecepatan 10.000 rpm. Ditambahkan kembali 50 µl elution buffer ke tengah
column membrane, dan disentrifugasi selama satu menit dengan kecepatan 10.000 rpm. Untuk penyimpanan jangka panjang, DNA hasil isolasi
31
(13) Supernatan dipindahkan ke
microtube baru (14) 400µl Plant gDNA Binding Solution
(15) 400µl Etanol 96%
(16) Dihomogenkan dengan cara membolak-balik tabung (17, 20) 600-700µl larutan dipindahkan
ke dalam spin column
(31) Inkubasi di suhu ruangan
sela a ’
(33) 50µl Elution Buffer
32
3.5.3 Pengukuran Konsentrasi dan Kemurnian DNA
Sebanyak 1 µl DNA hasil isolasi ditambahkan 499 µl ddH2O,
kemudian dihomogenkan. Konsentrasi dan kemurnian DNA genom diukur
dengan menggunakan spektrofotometer pada panjang gelombang 260 dan
280Å. Konsentrasi dan kemurnian DNA yang diperoleh dihitung
menggunakan rumus :
[DNA] = A x 50 x faktor pengenceran
Kemurnian DNA = A260 x A280
Keterangan :
A = Nilai absorbansi pada panjang gelombang tertentu
3.5.4 Elektroforesis
Gel agarose dibuat dengan konsentrasi 1% dalam buffer TAE,
dididihkan dalam microwave hingga campuran terlihat bening. Gel agarose
dituangkan ke dalam cetakan gel lengkap dengan sisir yang dipasang
dengan posisi tegak dan berjarak 0,5-1 mm dari dasar cetakan. Selanjutnya,
gel dibiarkan mengeras pada suhu ruang.
Sampel DNA disiapkan dan ditambahkan loading dye dengan
perbandingan 3:2, kemudian dimasukkan kedalam sumur gel yang berada di
dalam alat elektroforesis berisi buffer TAE 1x. Proses elektroforesis
dilakukan selama 120 menit dengan daya 100 volt. Gel agarose kemudian
direndam dalam EtBr selama 5 menit dan H2O selama 2 menit. Hasil
elektroforesis diamati dibawah lampu UV dan didokumentasikan (Hidayat,
2014, hlm. 5).
3.5.5 Amplifikasi DNA daerah ITS
33
Tabung Eppendorf dimasukkan kedalam mesin PCR dengan program
mengacu pada Hidayat et al. (2008, hlm. 17). Amplifikasi dimulai dengan
denaturasi awal pada suhu 95°C selama 2 menit (1 siklus), selanjutnya 35
siklus yang terdiri dari denaturasi pada suhu 95°C selama 30 detik,
annealing pada suhu 57°C selama 2 menit, dan ekstensi pada suhu 71°C selama 2 menit. Tahapan amplifikasi diakhiri oleh 1 siklus final extension
pada suhu 71°C selama 10 menit. Hasil amplifikasi dielektroforesis pada gel
agarosa 1% yang dilarutkan dalam buffer TAE 1x (Muchtar, 2008, hlm. 27).
3.5.6 Sikuensing DNA
Sebanyak 21 produk amplifikasi disikuensing di Macrogen Inc., Korea
Selatan menggunakan primer ITS-4 (5’
-CCCGCCTGACCTGGGGTCGC-3’) dan ITS-5 (5’-TAGAGGAAGGAGAAGTCGTAACAA-3’) dengan
menggunakan mesin ABI377A dan pewarnaan dengan kit ABI PRISMTM
Dye Terminator.
3.5.7 Analisis Data 3.5.7.1 Contig
Terdapat dua set data urutan basa nukleotida yang diperoleh dari hasil
sikuensing untuk setiap sampel hasil isolasi DNA daerah ITS. Satu set data
berasal dari sikuensing menggunakan primer ITS-5 dan satu set data lainnya
berasal dari hasil sikuensing menggunakan primer ITS-4. Dilakukan contig
menggunakan software CodonCode Aligner untuk kedua data tersebut. Pada
saat proses contig, terdapat daerah overlap dari masing-masing data. Hasil
yang diperoleh adalah urutan DNA daerah ITS secara lengkap. Penggunaan
kedua primer tersebut adalah untuk meminimalisir kesalahan baca yang
dilakukan mesin sequencer.
3.5.7.2 Verifikasi Data Hasil Sikuensing
Sikuen-sikuen DNA disejajarkan dengan sikuen DNA daerah ITS
yang terdapat di genbank dengan alamat http://ncbi.nlm.nih.gov/ untuk
memastikan bahwa sikuen DNA daerah ITS yang teramplifikasi merupakan
DNA tumbuhan. Untuk melihat homologinya, digunakan program BLAST
34
genus diambil satu sikuen DNA daerah ITS dari genbank untuk dijadikan
sebagai sikuen DNA pembanding.
3.5.7.3 Pencarian Daerah ITS
Pencarian daerah ITS yang terdiri dari ITS-1; 5,8S; dan ITS-2
dilakukan menggunakan program BLAST dengan alamat
http://blast.ncbi.nlm.nih.gov/Blast.cgi. Pencarian dilakukan dengan
penjajaran DNA sampel yang telah dicontig dengan DNA pembanding dari
genbank. Hasil yang didapatkan adalah letak urutan basa dimana daerah ITS-1; 5,8S; dan ITS-2 sampel berada. Selanjutnya, urutan tersebut dicatat
rangenya dan dihitung jumlahnya sehingga didapatkan urutan basa beserta jumlah dari daerah ITS sampel.
3.5.7.4 Editing Data
Editing data dilakukan sebelum pembentukan pohon filogeni dengan tujuan untuk membuat setiap data urutan basa nukleotida berada pada awal
dan akhir yang bersamaan sehingga diperoleh data yang informatif.
Dilakukan alignment terhadap semua sampel DNA menggunakan software
clustalX. Editing data dilakukan setelah proses alignment selesai dilakukan.
Data yang diedit adalah data dengan format .aln hasil dari alignment
35
3.5.7.5 Pembentukan Pohon Filogeni
Dilakukan pembentukan pohon filogeni terhadap data yang telah
melalui proses editing. Pembentukan pohon filogeni menggunakan software
Mega versi 4, dengan memilih menu bootstrap test of phylogeny dan
maximum parsimony untuk mengetahui nilai bootstrap dari pohon filogeni yang terbentuk.
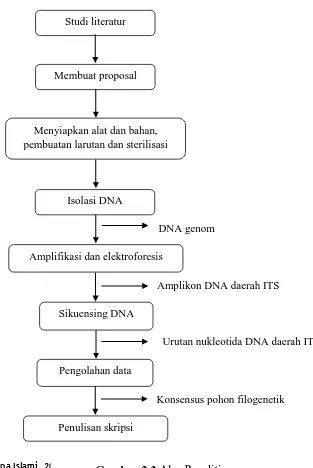
3.6 Alur Penelitian
Gambar 3.3 menunjukkan tahapan-tahapan serta hasil yang
didapatkan dari masing-masing langkah pada penelitian ini.
Studi literatur
Membuat proposal
Menyiapkan alat dan bahan, pembuatan larutan dan sterilisasi
Isolasi DNA
Amplifikasi dan elektroforesis
Sikuensing DNA
Pengolahan data
Penulisan skripsi
DNA genom
Amplikon DNA daerah ITS
Konsensus pohon filogenetik
BAB V
KESIMPULAN DAN SARAN
5.1 Kesimpulan
Kekerabatan anggota familia Solanaceae menggunakan sikuen DNA
daerah ITS secara terpisah menunjukkan pola yang tidak tepat. Sebaliknya,
kekerabatan yang sesuai ditunjukkan pada pohon filogeni yang menggunakan
keseluruhan sikuen DNA daerah ITS. Terdapat tiga kelompok besar yang
dihasilkan oleh pohon filogeni yang dibentuk oleh sikuen DNA daerah ITS.
Kelompok I terdiri dari supergenus Nicotianoideae, Datureae, Cestreae, serta
Petunieae; kelompok II terdiri dari supergenus Withaninae dan Physalinae
yang merupakan anggota tribe Physaleae; dan kelompok III terdiri dari
supergenus Solaneae. Posisi Capsicum annuum yang terletak satu node
dengan Solanum pseudocapsicum menunjukkan pola kekerabatan yang
berbeda dengan pengelompokan sebelumnya. Ditinjau dari segi morfologi,
kedua spesies ini memiliki banyak persamaan, sehingga berdasarkan
penelitian ini dapat diusulkan adanya penggantian nama Capsicum annuum,
misalnya menjadi Solanum capsicum. Namun secara keseluruhan,
penggunaan daerah ITS untuk menentukan kekerabatan anggota familia
Solanaceae mendukung pengelompokan sebelumnya.
5.2 Saran
Diperlukan lebih banyak lagi sampel anggota Solanaceae, baik pada
tingkat genus maupun spesies agar data yang didapatkan lebih valid. Karena
jika hanya diwakili oleh beberapa sampel saja, dapat dihasilkan
pengelompokan yang kurang tepat. Selain itu, penelitian mengenai Capsicum
annuum dan Solanum pseudocapsicum perlu dilakukan untuk memastikan kekerabatan diantara kedua spesies tersebut. Selain data molekuler, data
DAFTAR PUSTAKA
ABADS. (2006). Brugmansia suaveolens. [Online]. Tersedia: http://www.abads.org/abads/Gallery/species/brugmansia_suaveolens.htm [1
Juli 2015]
Alamy. (2011). Potato (Solanum tuberosum). [Online]. Tersedia: http://www.alamy.com/stock-photo-potato-solanum-tuberosum-stalk-with-small-green-fruit-and-laved-fruit-41743694.html [20 Juni 2015]
Ampliqon. (2013). MgCl2. Odense: Ampliqon PCR Enzymes & Reagents.
Andreasen, K. & Baldwin, B. G. (2003). Reexamination of Relationships, Habital Evolution, and Phylogeography of Checker Mallows (Sidalcea; Malvaceae) Based on Molecular Phylogenetic Data1. American Journal of Botany. 90, (3), hlm. 436–444.
Arsh. (2015). Jerusalem Cherry Flower. [Online]. Tersedia: http://www.flowersleaf.com/jerusalem-cherry-flower/ [12 Juni 2015]
Backer, C. A. & Brink, R. C. B. V. D. (1965). Flora of Java (Spermathophytes Only). Vol. II. Groningen: N. V. P. Noordhoff.
Bailey, L. H. (1963). How Plants Get Their Names. New York: Pover Publications, Inc.
Baldwin, B. G., Sanderson, M. J., Porter, J. M., Wojciechowski, M. F., Campbell, C. S., Donoghue, M. J. (1995). The ITS Region of Nuclear Ribosomal DNA: A Valuable Source of Evidence on Angiosperm Phylogeny. Annals of The Missouri Botanical Garden. 82 (2), hlm. 247-277.
Black Diamond. (2011). Solanaceae. [Online]. Tersedia: http://www.flickriver.com/photos/blackdiamondimages/sets/721576283503 87249/ [20 Juni 2015]
66
Boudreaux, B. (2011). Jerusalem Cherery (Solanum pseudocapsicum) Flower
0081. [Online]. Tersedia:
https://www.flickr.com/photos/84094713@N08/8086927471/ [20 Juni 2015]
Bryson, C. T. (2013). Solanum viarum. [Online]. Tersedia: http://www.texasinvasives.org/plant_database/detail.php?symbol=SOVI2 [20 Juni 2015]
Bumi Herbal. (2014). Pemanfaatna Tanaman Solanaceae pada Makanan Sehari-Hari. [Online]. Tersedia: http://bumiherbal.com/2014/06/pemanfaatan-tanaman-solanaceae-pada-makanan-sehari-hari/ [1 Juni 2015]
Cotterill, S. & Kearsey, S. (2002). Encyclopedia of Life Sciences. London: Macmillan Publishers Ltd.
Cronquist, A. (1981). An Integrated Systemof Classification of Flowering Plants. New York: Columbia University Press.
Dave. (2001). Shoofy Plant, Apple of Peru Nicandra physalodes. [Online]. Tersedia: http://davesgarden.com/guides/pf/go/654/ [20 Juni 2015]
Dennison, C. (2002). A Guide to Protein Isolation. Springer Netherlands.
Dharmayanti, N. L. P. I. (2011). Filogenetika Molekuler: Metode Taksonomi Organisme Berdasarkan Sejarah Evolusi. Wartoza. 21, (1), hlm. 1-10.
Encyclopaedia Britannica. (2015). Ilustrations Electrophoresis Gel DNA.
[Online]. Tersedia:
http://www.illustrationsource.com/stock/image/481364/gel-electrophoresis- uses-an-electric-field-and-positive-and-negative-electrodes-to-separate-dna-molecules-according-to-size/ [6 Juni 2015]
Faucon, P. (2005). Night Jessamine. [Online]. Tersedia: http://www.desert-tropicals.com/Plants/Solanaceae/Cestrum_nocturnum.html [20 Juni 2015]
Feiertag, S. (2014). Guam Boonies. [Online]. Tersedia: http://www.ethno- botanik.org/Capsicum/Guam-Boonies/Guam-Boonies-Capsicum-annuum-en.html [12 Juni 2015]
67
Anopheles nuneztovari (Diptera: Culicidae). Mol.Biol.Evol. 11, (3), hlm. 406-416.
Germer, J. (2005). Solanum melongena L. [Online]. Tersedia: http://www.virboga.de/Solanum_melongena.htm [20 Juni 2015]
Ghazanfar, S. (2011). Withania (Withania Somnifera). [Online]. Tersedia: http://www.arkive.org/withania/withania-somnifera/image-G118244.html [12 Juni 2015]
Gopalakrishnan, A. Gopinath, D., Vijayasaraswathy S. G., Kasa, J. (2014). Ethnomedicine in Cancer Therapy: A Review. World Journal of Pharmaceutical Research. 3, (6), hlm. 305-319.
Gratani, L. (2014). Plant Phenotypic Plasticity in Response to Environmental Factors. Advances in Botany, (208747), hlm. 1-17.
Haman, J. & Farmery, C. S. (2010). Ultraviolet Transilluminators. Health and Safety Department University of Edinburgh: Edinburgh.
Hamdan, N. Samad, A. A. Hidayat, T. Salleh, F. M. (2013). Phylogenetic Analysis of Eight Malaysian Pineapple Cultivars using a Chloroplastic Marker (rbcL gene). Jurnal Teknologi, 64 (2), hlm. 29-33.
Handoyo, D. & Rudiretna A. (2000). Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR). Unitas. 9, (1), hlm. 17-29.
Haston, E. Richardson, J. E. Stevens, P. F. Chase, M. F. Harris, D. J. (2009). The Linear Angiosperm Phylogeny Group (LAPG) III: A Linear Sequence of The Families in APG III. Botanical Journal of the Linnean Society, 161, hlm. 128-131.
Herbarium Bogoriense. (2005). Keanekaragaman Jenis dan Sumber Plasma Nutfah Durio (Durio spp.) di Indonesia. Buletin Plasma Nutfah, 11 (1), hlm. 28-33.
Hidayat, T. & Pancoro, A. (2006). Sistematika dan Filogenetika Molekuler. Makalah pada Kursus Singkat Perangkat Lunak PAUP dan MrBayes untuk Penelitian Filogenetika Molekuler SITH-ITB, Bandung.
68
Hidayat, T. Kusumawaty, D. Kusdianti, Yati, D. D. Muchtar, A. A. Mariana, D. (2008). Analisis Filogenetik Molekuler pada Phyllanthus niruri L. (Euphorbiaceae) Menggunakan Urutan Basa DNA Daerah Internal Transcribed Spacer (ITS). Jurnal Matematika dan Sains, 13 (1), hlm. 16-21.
Hidayat, T., & Pancoro, A. (2008). Kajian Filogenetika Molekuler dan Peranannya dalam Menyediakan Informasi Dasar untuk meningkatkan Kualitas Sumber Genetik Anggrek. AgroBiogen. 4, (1), hlm. 35-40.
Holmes, S. (2003). Bootstraping Phylogenetics Trees: Theory and Methods. Statistics Department, Stanford.
Hoof, B. (2014). Solanum lycopersicum. [Online]. Tersedia: http://www.ethno-botanik.org/Tomaten/Boars-Hoof/Boars-Hoof-Tomatensorte.html [20 Juni 2015]
Hunter, R. L. LaJeunesse, T. C. Santos, S. R. (2007). Structure and Evolution of The rDNA Internal Transcribed Spacer (ITS) Region 2 In The Symbiotic Dinoflagellates (Symbiodinium, Dinophyta). Phycol. 43, hlm. 120-128.
Janssens. (2009). Solanaceae. [Online]. Tersedia: http://www.mobot.org/mobot/research/apweb/orders/solanalesweb.htm [20 Juni 2015]
Jha, T. B. Samaddar, T. Nath, S. Das, A. (2014). Direct Organogenesis and Genetic Characterization of Solanum pseudocapsicum L. in vitro Regenerated Plants. Plant Tissue Cult & Biotech. 24, (1), hlm. 65-76.
Jobes, D. V. & Thien, L. B. (1997). A Conserved Motif in The 5,8S Ribosomal RNA (rRNA) Gene is a Useful Diagnostic Marker for Sequences. Plant Molecular Biology Reporter. 15, hlm. 326-334.
Khosravinia, H. Murthy, H. N. M. Parasad, D. T. Pirany, N. (2007). Optimizing Factor Influencing DNA Extraction from Fresh Whole Avian Blood. African Journal of Biotechnology. 6, (4), hlm. 481-486.
Marshall, J. A. Knapp, S. Davey, M. R. Power, J. B. Cocking, E. C. Bennett, M. D. Cox, A. V. (2001). Molecular systematics of Solanum section Lycopersicum (Lycopersicon) using the nuclear ITS rDNA region. Springer-Verlag, 103, hlm. 1216-1222.
69
Solanaceae and Related Species Based on cpDNA Sequence from Plastid trnEtrnT Region. Crop Breeding and Applied Biotechnology, 8, hlm. 85-95.
Moreira, P. A. & Oliviera, D. A. (2011). Leaf Age Affects The Quality of DNA Extracted from Dimorphandra mollis (Fabaceae), A Tropical Tree Species from The Cerrado Region of Brazil. Genetics and Molecular Research. 10, (1), hlm. 353-358.
Morris, S. C. Forbes-Smith, M. R. Scriven, F. M. (1989). Determination of Optimum Conditions for Suberization, wound Periderm Formation, Cellular Desiccation and Pathogen Resistance in wounded Solanum tuberosum Tubers. Physiological and Molecular Plant Pathology, 35 (2), hlm. 177-190.
Muchtar, A. A. (2008). Analisis Kekerabatan Marga-Marga pada Suku Euphorbiaceae berdasarkan Sikuen DNA daerah ITS. Skripsi Sarjana pada FPMIPA UPI Bandung: tidak diterbitkan.
Olmstead, R. G. Bohs, L. Migid, H. A. Valentin, E. S. Garcia, V. F. Collier S. M. (2008). A Molecular Phylogeny of The Solanaceae. Taxon, 57 (4), hlm. 1159-1181.
Pekarskyi, I. (2015). Red Chili Pepper with Slices and Seeds. [Online]. Tersedia: http://www.shutterstock.com/pic-110267027/stock-photo-red-chili-pepper-with-slices-and-seeds.html?src=0_qovdPatm7zhZ8jL-7RHQ-1-55 [20 Juni 2015]
70
Rahim, M. D. & Nasruddin, A. (2010). Deteksi Molekuler Rice Tungro Baciliform Virus di Sulawesi Selatan dengan Menggunakan PCR Genomik dan Optimalisasinya. Fitomedika. 7, (1), hlm. 55-61.
Rasbak. (2011). Solanum tuberosum flower. [Online]. Tersedia: https://commons.wikimedia.org/wiki/File:Solanum_tuberosum_flower,_%2 Bangladesh J. Agril, 35 (3), hlm. 525-534.
Sanders, R. (2003). D. metel var. fastuosa form fastuosa. [Online]. Tersedia: http://www.abads.org/abads/Rich_Sanders/datura_metel.htm [20 Juni 2015]
Schoc, C. L. Seifert, K. A. Huhndorf, S. Robert, V. Spouge, J. L. Levesque, C. A. Chen, W. Fungal Barcoding Consortium. (2012). Nuclear Ribosomal Internal Transcribed Spacer (ITS) Region as a Universal DNA Barcode Marker for Fungi. PNAS, 109 (16), hlm. 6241-6246.
Sibilio, G. (2015). Brunfelsia uniflora. [Online]. Tersedia: http://www.flickriver.com/photos/tags/brunfelsia/interesting/ [20 Juni 2015]
Simpson. (1998). Gel Electrophoresis and Photography An Application Note. UVP Inc.: Cambridge.
Stefanovic, S. Krueger, L. Olmstead, R. G. (2002). Monophyly of The Convolvulaceae and Circumscription of Their Major Lineages Based on DNA Sequences of Multiple Chloroplast Loci1. American Journal of Botany, 89 (9), hlm. 1510-1522.
71
Sukojo, B. M. (2003). Penggunaan Metode Analisa Ekologi Dan Penginderaan Jauh Untuk Pembangunan Sistem Informasi Geografis Ekosistem Pantai. Makara Sains, 7 (1), hlm. 30-37.
Teare, J. M. Islam, R. Flanagan, R. Gallagher, S. Davies, M. G. Grabau, C. (1997). Measurement of Nucleid Acid Concentrations Using The DyNA
Quant™ and The GeneQuant™. BioTechniques. 22, hlm. 1170-1174.
Tippery, N. P. & Les, D. H. (2008). Phylogenetic Analysis of The Internal Transcribed Spacer (ITS) Region in Menyanthaceae Using Predicted Secondary Structure. Molecular Phylogenetics and Evolution. 49, hlm. 526-537.
Top Tropicals. (2007). Solanum viarum. [Online]. Tersedia: http://toptropicals.com/catalog/uid/Solanum_viarum.htm [20 Juni 2015]
Torres, S. (2012). Jazmin de Noche. [Online]. Tersedia: https://commons.wikimedia.org/wiki/File:Jazm%C3%ADn_de_Noche.JPG [12 Juni 2015]
Vos, J. M. d. Hughes, C. E. Schneewiss, G. M. Moore, B. R. Conti, E. (2014). Heterostyly Accelerates Diversification via Reduced Extinction in Primeroses. Proceedings of The Royal Society. hlm. 1-9.
Webb, C. O. Slik, J. W. F. Triono, T. (2010). Biodiversity Inventory and Informatics in Southeast Asia. Biodivers Conserv. 10, hlm. 1-8.
Xeramtheum. (2007). Datura Brugmansia Whtat’s The Difference?. [Online]. Tersedia: https://davesgarden.com/community/forums/fp.php?pid=3333529 [12 Juni 2015]
Yati, D. D. (2008). Analisis Hubungan Kekerabatan pada Phyllanthus niruri L. Berdasarkan Karakter DNA. Skripsi Sarjana pada FPMIPA UPI Bandung: tidak diterbitkan.
Yilmaz, M. Ozic, C. Gok, I. (2012). Gel Electrophoresis. Shanghai: InTech China.
Yulianti, E. (2006). Pengembangan Teknik Isolasi DNA Tumbuhan Menggunakan Detergen Komersial. Makalah pada Seminar Nasional MIPA UNY, Yogyakarta.
72
Zell, H. (2009). Nicotiana tabacum. [Online]. Tersedia: https://commons.wikimedia.org/wiki/File:Nicotiana_tabacum_003.JPG [20 Juni 2015]