KERAGAMAN MIKROSATELIT DAN GEN
CYTOCHROME
OXIDASE SUBUNIT
I (COI) PADA
KERBAU INDONESIA
FERDY SAPUTRA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Keragaman Mikrosatelit dan Gen Cytochrome Oxidase Subunit I (COI) pada Kerbau Indonesia adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, November 2013
Ferdy Saputra
RINGKASAN
FERDY SAPUTRA. Keragaman Mikrosatelit dan Gen Cytochrome Oxidase
Subunit I(COI) pada Kerbau Indonesia. Dibimbing oleh CECE SUMANTRI dan
JAKARIA.
Penelitian ini bertujuan untuk mengidentifikasi keragaman genetik dan hubungan kekerabatan menggunakan mikrosatelit dan gen cytochrome oxidase
subunit I (COI). Sampel yang digunakan pada penelitian ini berjumlah 211 sampel kerbau terdiri atas masing-masing 30 sampel dari Aceh, Sumatra Utara, Banten, Jawa Tengah, Nusa Tenggara Barat, Sulawesi Selatan, sedangkan 19 sampel dari Riau dan 12 sampel kerbau sungai dari Sumatra Utara. Lokus mikrosatelit yang digunakan adalah CSSM66, ILSTS61 dan ILSTS17. Data mikrosatelit yang diamati adalah jumlah alel (na); alel efektif (ne); nilai heterozigositas pengamatan (Ho); nilai heterozigositas harapan (He) dan polymorphism information content (PIC) yang dianalisis menggunakan software GenAlEx 6.41 dan Cervus 3.0. Sekuen gen COI dianalisis menggunakan software MEGA 5.1 dengan metoda
Neighbor-joining dan model substitusi kimura 2 parameter. Lokus CSSM66
memiliki dua alel dan dua genotipe. Lokus ILSTS61 memiliki lima alel dan 14 genotipe. Lokus ILSTS17 memiliki dua alel dan tiga genotipe. Jarak genetik dengan menggunakan mikrosatelit mampu memisahkan antar populasi kerbau rawa di Indonesia. Pohon filogeni yang direkonstruksi berdasarkan sekuen gen COI hanya mampu membedakan perbedaan subspesies antara kerbau rawa dengan kerbau sungai. Sekuen gen COI memperlihatkan adanya empat haplotipe untuk kerbau rawa Indonesia yaitu H1, H2, H3 dan H4.
SUMMARY
FERDY SAPUTRA. Genetic Diversity of Microsatellite and Cytochrome Oxidase
Subunit I (COI) Gene in Indonesian Buffalo. Supervised by CECE SUMANTRI and JAKARIA.
This study aims to determine genetic diversity in Indonesian buffaloes using microsatellite markers and cytochrome oxidase subunit I gene. The sample used in this study were 211 samples of buffaloes (30 samples of swamp buffaloes from Aceh, North Sumatra, Banten, Central Java, West Nusa Tenggara, South Sulawesi, and 19 samples from Riau) and 12 samples of river buffaloes from North Sumatra. Microsatellite markers used were CSSM66, ILSTS61, and ILSTS17 loci. Microsatellite data in term of observed number of alleles (na); effective number of alleles (ne); Observed heterozygosity (Ho); Expected heterozygosity (He); and
polymorphism information content (PIC) were analyzed by GenAlEx 6.41 and
Cervus 3.1. COI gene sequences were analyzed using MEGA 5.1 software with the Neighbor-joining method and kimura 2 parameter substitution models. This study showed that CSSM66 loci had two alleles and two genotypes. ILSTS61 loci had five alleles and 14 genotypes. ILSTS17 loci had two alleles and three genotypes. Microsatellite genetic distance between populations could separate Indonesian swamp buffalo. The reconstructed phylogeny tree based on COI gene sequences were able to distinguish Subspecies of swamp buffalo and river buffalo. COI gene sequences showed four haplotypes for Indonesian swamp buffalo, namely H1, H2, H3 and H4.
© Hak Cipta Milik IPB, Tahun 2013
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Dilarang mengumumkan dan memperbanyak sebagian atau seluruh karya tulis ini
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Ilmu Produksi dan Teknologi Peternakan
KERAGAMAN MIKROSATELIT DAN GEN
CYTOCHROME
OXIDASE
SUBUNIT
I (COI) PADA
KERBAU INDONESIA
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2013
Judul Tesis : Keragaman Mikrosatelit dan Gen Cytochrome Oxidase Subunit I (COI) pada Kerbau Indonesia
Nama : Ferdy Saputra NIM : D151110141
Disetujui oleh Komisi Pembimbing
Prof Dr Ir Cece Sumantri, MAgrSc Ketua
Dr Jakaria, SPt MSi Anggota
Diketahui oleh
Ketua Program Studi
Ilmu Produksi dan Teknologi Peternakan
Prof Dr Ir Muladno, MSA
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah atas segala karunia-Nya sehingga karya ilmiah ini berhasil diselesaikan. Judul yang dipilih dalam penelitian yang dilaksanakan sejak bulan Agustus 2012 ini ialah keragaman mikrosatelit dan gen cytochrome oxidasesubunit I pada kerbau Indonesia. Terima kasih penulis ucapkan kepada Bapak Prof. Dr. Ir. Cece Sumantri, M.Agr.Sc dan Dr. Jakaria, S.Pt, M.Si selaku pembimbing, Dr. Ir. Achmad Farajallah, M.Si selaku penguji, Dr. Ir. Salundik, M.Si selaku perwakilan program studi ITP dan Prof. Dr. Ir Muladno, MSA selaku ketua program studi ITP yang telah banyak memberi saran dan dukungan. Di samping itu, penghargaan penulis sampaikan kepada Eryk Andreas, Irine, Wike Andre S, Galih Ari W, Isyana, Ahmad Furqon, Lusiana, Roaselin, Komang Alit, Yan Parta N, Dicky Tri U, Febriwendi F, C Willy Kia, Windi, Rika, Hikmawati, Hermanto H S dan Sekret PMK yang telah memberikan banyak dukungan. Ungkapan terima kasih juga disampaikan kepada ayah, ibu, serta seluruh keluarga, atas segala doa dan kasih sayangnya.
Semoga karya ilmiah ini bermanfaat.
Bogor, November 2013
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Perumusan Masalah 2
Tujuan Penelitian 2
Manfaat Penelitian 2
Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 3
Kerbau 3
Mikrosatelit 3
Gen Cytochrome Oxidase Subunit I (COI) 4
3 METODE 5
Waktu dan Tempat 5
Bahan 5
Alat 5
Prosedur 5
Prosedur Analisis Data 7
4 HASIL DAN PEMBAHASAN 8
Amplifikasi dan Genotyping TigaLokus Mikrosatelit 8
Frekuensi Alel Mikrosatelit 9
Keragaman Genetik Kerbau di Indonesia 10
Jarak Genetik Kerbau Indonesia 11
Keragaman Gen Cytochrome OxidaseSubunit I (COI) 12
5 SIMPULAN DAN SARAN 17
Simpulan 17
Saran 17
DAFTAR PUSTAKA 18
DAFTAR TABEL
1
Sekuens primer, letak di kromosom dan motif primer pada lokusCSSM66, ILSTS61, dan ILSTS17 5
2 Sekuens primer cytochrome oxidasesubunit I mengunakan
overlapping primer 6
3 Frekuensi alel lokus CSSM66, ILSTS61 dan ILSTS17 mikrosatelit
pada populasi kerbau di Indonesia 9
4 Keragaman genetik pada kerbau di Indonesia 10
5 Nilai Ho, He, PIC dan keseimbagan Hardy-Weinberg dari tiga lokus
mikrosatelit 11
6 Jarak genetik dari masing-masing populasi 11
7 Komposisi nukleotida sekuen gen cytochrome oxidasesubunit I
(COI) 13
8 Haplotipe dan posisi keragamannya berdasarkan sekuen gen COI 14
DAFTAR GAMBAR
1 Hasil elektroforesis lokus ILSTS17 dengan teknik PAGE 10% 8 2 Hasil elektroforesis lokus ILSTS61 dengan teknik PAGE 10% 8 3 Hasil elektroforesis lokus CSSM66 dengan teknik PAGE 10% 8 4 Dendogram populasi kerbau di Indonesia dengan metoda Neighbor
Joining 11
5 Hasil elektroforesis menggunakan primer FS1 dan FS2 dengan
agarose 1.5% 12
6 Hasil elektroforesis menggunakan primer FS3 dengan agrarose 1.5% 12 7 Sekuen gen cytochrome oxidase subunit I COI pada kerbau Indonesia 15 8 Pohon filogeni kerbau Indonesia berdasarkan sekuen gen COI dengan
metode neighbor-joining dan model substitusi kimura 2 parameter 15
1
1
PENDAHULUAN
Latar Belakang
Sumber daya genetik ternak merupakan salah satu bagian penting dalam menunjang keamanan pangan bagi kehidupan manusia. Sumber daya genetik ternak juga merupakan bahan baku esensial dalam pemanfaatannya secara berkelanjutan untuk produksi peternakan. Di sisi lain, konservasi dan pemanfaatan berkelanjutan dari sumber data genetik ternak juga merupakan bagian program dari program Food and Agriculture Organization (FAO) melalui program Global
Strategy for the Management of Farm Animal Genetic Resources untuk memandu
baik pada tingkat nasional, regional dan global untuk memperkuat kontribusi ternak dalam menjaga ketahanan pangan dan pembangunan, dan untuk mencegah terjadinya erosi sumber daya genetik ternak. Melalui program Molecular Genetic
Characterization of Animal Genetic Resources diharapkan membantu
negara-negara maju dan berkembang untuk merencanakan dan mengimplementasikan analisis yang efektif dalam penggunaan sumber daya genetik ternak untuk pangan dan pertanian (FAO 2011). Upaya untuk mendukung program tersebut adalah melalui karakterisasi genetik molekuler ternak terutama ternak kerbau, karena informasi genetik pada ternak kerbau di Indonesia masih terbatas.
Konservasi genetik penting dilakukan guna mempertahankan bangsa
(breed) atau rumpun yang murni atau dengan kata lain, mempertahankan
keragaman genetik (genetic diversity). Ada dua jenis utama dari ancaman kepunahan yaitu ancaman deterministic dan stochastic (Caughley 1994). Ancaman deterministic adalah perusakan habitat, polusi, eksploitasi berlebih, translokasi spesies, dan perubahan iklim global. Ancaman stochastic adalah perubahan genetik, demografi, atau faktor lingkungan. Genetic stochasticity
adalah perubahan genetik secara acak (genetic drift) dan peningkatan inbreeding
(Shaffer 1981). Genetic stochasticity menyebabkan hilangnya variasi genetik (termasuk alel yang menguntungkan) dan peningkatan alel yang berbahaya atau merugikan.
Mikrosatelit murupakan salah satu penciri (marker) yang sering digunakan dalam kajian genetika populasi karena memiliki keunikan, diantaranya adalah sifatnya kodominan, motif ulangan tinggi, dan pewarisannya sesuai hukum mendel. Studi mengenai mikrosatelit berkembang dengan pesat dan telah dilakukan secara luas pada berbagai jenis ternak di Indonesia seperti ternak sapi (Abdullah et al. 2008), kambing (Zein et al. 2012) dan domba (Sumantri et al. 2008; Jakaria et al. 2012). Kajian mikrosatelit pada kerbau di Indonesia telah dilaporkan namun masih terbatas di wilayah Bogor dan Sulawesi (Barker et al.
1997). Selain penciri mikrosatelit, juga penciri yang umumnya digunakan untuk studi keragaman adalah DNA Barcode yang merupakan penciri menggunakan gen
cytochrome oxidase subunit I (COI) (Hebert et al. 2004). Hal ini telah dilakukan
pada berbagai species seperti burung (Hebert et al. 2004), sapi (Syed-Shabthar et al. 2013), ayam (Gao et al. 2011), nyamuk (Cywinska et al. 2006), Cricula
trifenestrata (Surianaet al. 2012), lepidoptera (Wilson, 2010) dan kumbang (Funk
et al. 1995). Kajian keragaman genetik dengan menggunakan mikrosatelit dan gen
2
khususnya di wilayah Aceh, Sumatra Utara, Riau, Banten, Jawa Tengah, Nusa Tenggara Barat dan Sulawesi Selatan, sehingga penelitian ini penting dilakukan.
Perumusan Masalah
Indonesia sebagai salah satu negara yang memiliki sumber daya genetik ternak yang beraneka ragamng termasuk sumber daya genetik ternak kerbau yang tersebar di beberapa wilayah di Indonesia merupakan salah satu kekayaan dan aset bangsa yang telah dimanfaatkan untuk sumber pangan, tenaga dan sosial budaya. Namun di sisi lain, informasi mengenai sumber daya genetik ternak kerbau tersebut masih sangat terbatas dan bahkan belum ada data genetik terutama data penciri mikrosatelit dan gen COI.
Tujuan Penelitian
Penelitian ini bertujuan untuk mengidentifikasi keragaman genetik pada kerbau di Indonesia terutama di wilayah Aceh, Sumatra Utara, Riau, Banten, Jawa Tengah, Nusa Tenggara Barat dan Sulawesi Selatan berdasarkan penciri mikrosatelit lokus CSSM66, ILSTS61, ILSTS17 dan gen cytochrome oxidase subunit I(COI).
Manfaat Penelitian
Penelitian ini diharapkan dapat dijadikan sebagai informasi dasar dalam upaya mempertahankan keragaman genetik (biodiversity) dan program pemuliaan pada setiap populasi kerbau di Indonesia.
Ruang Lingkup Penelitian
3
2
TINJAUAN PUSTAKA
Kerbau
Kerbau diklasifikasikan ke dalam class Mamalia, subclass Ungulata, ordo
Artiodactyla, subordo Ruminantia, family Bovidae, subfamili Bovinae, genus
Bovini, yang meliputi mengikuti tiga kelompok: Bovina (sapi), Bubalina dan Syncerina. Syncerina hanya mencakup spesies Syncerus caffer (kerbau Afrika) (Borghese 2005). Bubalina (kerbau Asia) mencakup tiga spesies: Bubalus depressicornis atau Anoa yang hidup di Indonesia, Bubalus mindorensis yang kehidupan di Filipina dan Bubalus bubalis yang berasal dari domestikasi Bubalus arnee, kerbau liar (Borghese 2005). Domestikasi spesies ini (Bubalus bubalis) terjadi relatif baru-baru (5.000 tahun yang lalu) dibandingkan dengan domestikasi
Bos taurus dan Bos indicus (10.000 tahun lalu) (Ross 1975). Kerbau asia
mencakup dua subspesies yang dikenal sebagai kerbau sungai (river/water) dan kerbau rawa/lumpur (swamp). Kerbau sungai memiliki 50 kromosom dengan lima pasang yang submetasentrik, sementara 20 pasang adalah akrosentrik. Kerbau rawa memiliki 48 kromosom, dimana 19 pasangan adalah metasentrik. Perbedaan jumlah diploid yang hanya bersifat semu. Bahkan, kromosom 1 kerbau Rawa yang besar berasal dari tandem fusi translokasi antara kromosom 4 kerbau Sungai (telomere p-lengan) dan kromosom 9 (sentromer) (Iannuzzi 1998). Kerbau sungai tersebar di India, Pakistan, Sri Lanka dan beberapa negara di Eropa seperti Italia, Bulgaria, Yunani dan Yugoslavia. Kerbau rawa tersebar di Asia Timur termasuk China, Indonesia, Filipina dan Thailand (Ross 1975).
Mikrosatelit
4
akan tinggi bila menggunakan lokus dengan ulangan dua nuckleotida (dinucleotide), semakin banyak jumlah basa ulangan maka akan mengurangi peluang munculnya stutter band/peak. Mekanisme mutasi inilah yang sering dipelajari dalam Microsatellite Instability.
Gen Cytochrome Oxidase Subunit I (COI)
Salah satu mekanisme yang terjadi di dalam sel adalah produksi ATP melalui respon terhadap dan kebutuhan energi melalui perubahan dalam komposisi mitokondria (Duggan et al. 2011). Biogeneis mitokondria dipengaruhi banyak gen, contohnya biosintesis sitokrom c oksidase (COX) membutuhkan koordinasi gen COX dalam dua genom: 10 subunit yang dikodekan oleh DNA inti dan tiga subunit dikodekan oleh DNA mitokondria (Duggan et al. 2011). Gen
cytochrome oxidasesubunit I (COI) yang terdapat di DNA mitokondria (mtDNA)
5
3
METODE
Waktu dan Tempat
Penelitian dilakukan di Laboratorium Genetika Molekuler Ternak, Departemen Ilmu Produksi dan Teknologi Peternakan, Fakultas Peternakan IPB yang dimulai dari Agustus 2012 sampai April 2013.
Bahan
Sampel DNA kerbau yang digunakan berasal dari Laboratorium Genetika Molekuler Ternak. Sebanyak 199 sampel DNA kerbau rawa yang terdiri dari populasi Aceh, Sumatra utara, Banten, Jawa Tengah, Nusa Tenggara Barat, Sulawesi Selatan masing-masing 30 sampel, sedangkan populasi Riau 19 sampel. Sebanyak 12 sampel DNA kerbau sungai dari populasi Sumatra Utara digunakan sebagai pembanding. Bahan yang digunakan dalam penelitian ini adalah dH2O, TE 80%, primer, dNTPs, MgCl2, 10xBuffer, Taq Polymerase, Polyacrilamid, TEMED, 5xTBE, APS, AgNO3, Formaldehid, NaOH, Amonia, Agrose dan 0.5xTBE.
Alat
Alat yang digunakan dalam penelitian ini adalah Micropipette, Inkubator,
Vortex, Sentrifuse, Freezer, Mesin Thermal Cycler Geneamp 9700 (AB System) dan Peralatan elektroforesis (CBS Scientific dan Mupid).
Prosedur
Amplifikasi DNA Mikrosatelit dan Gen Cytochrome Oxidase Subunit I
Lokus DNA Mikrosatelit yang digunakan dalam penelitian ini adalah lokus CSSM066, ILSTS61 dan ILSTS17. Sekuens primer, letak di kromosom dan motif primer lokus yang disajikan pada Tabel 1.
Tabel 1 Sekuens primer, letak di kromosom dan motif primer pada lokus CSSM66, ILSTS61, dan ILSTS17
Lokus Sekuens Primer
(5’-3’) KromosomLetak di
Motif Ulangan CSSM66 Forward: ACA CAA ATC CTT TCT GCC AGC TGA
Reverse: AAT TTA ATG CAC TGA GGA GCT TGG
14 (AC)17
ILSTS61 Forward: AAA TTA TAG GGG CCA TAC GG Reverse: TGG CCT ACC CTA CCA TTT CC
15 (CA)14
ILSTS17 Forward: GTC CCT AAA ATC GAA ATG CC Reverse: GCA TCT CTA TAA CCT GTT CC
X (TG)17
Sumber: Kathiravan et al. (2009)
6
25 mM MgCl2; 1.2 µl 10xBuffer; 0.05 µl Taq Polymerase; dH2O sampai 12 µl dan selanjutnya dijalankan pada mesin Gene Amp PCR System 9700 (AB System) dengan kondisi sebagai berikut: denaturasi awal 95oC 5 menit, denaturasi akhir 95oC 15 detik, aneling 55oC 15 detik, ekstensi awal 72oC 15 detik, dan selanjutnya ekstensi akhir 72oC 5 menit.
Sekuens primer yang digunakan untuk mengamplifikasi gen cytochome
oxidase subunit I disajikan pada Tabel 2. Primer dibuat dengan menggunakan
Primer-Blast dengan acuan nomor akses genbank sebagai berikut: NC_020615, EF536351, JN632607, AY488491, AF547270, NC_006295 dan AY702618. Tabel 2 Sekuens primer cytochrome oxidasesubunit I mengunakan overlapping
primer
Keterangan: *berdasarkan genbank AF547270, F (Forward), R (Reverse)
Amplifikasi gen cytochrome oxidasesubunit I (COI) menggunakan bahan yaitu 1 µl sampel DNA; 0.5 µl primer (forward dan reverse); 0.5 µl 10 mM dNTPs; 0.5 µl 25 mM MgCl2; 3.5 µl 10xBuffer; 0.1 µl Taq Polymerase; dH2O sampai 35 µl dan selanjutnya dijalankan pada mesin Gene Amp PCR System 9700
(AB System) dengan kondisi sebagai berikut: denaturasi awal 95oC 5 menit, denaturasi akhir 95oC 45 detik, aneling 60oC 45 detik, ekstensi awal 72oC 1 menit, dan ekstensi akhir 72oC 5 menit. Amplicon COI dilelektroforesis dengan menggunakan agarose 1.5%.
Elektroforesis Lokus DNA Mikrosatelit
Hasil amplifikasi lokus DNA mikrosatelit (produk PCR) selanjutnya dielektroforesis menggunakan Polyacrilamid Gel Elektroforesis (PAGE) 10% dengan bufer 0.5xTBE. Hasil elektroforesis kemudian dilakukan pewarnaan perak atau silver staining mengacu pada Byun et al. (2009). Setelah itu, dilakukan
genotyping terhadap pita yang muncul dari hasil elektroforesis secara manual.
Genotyping dilakukan secara berurutan pada alel yang lebih awal atau tercepat (alel dengan panjang basa paling pendek) sampai alel paling akhir atau lambat (alel dengan panjang basa yang lebih panjang) diidentifikasi secara berurutan yaitu dari alel A sampai dengan alel Z (alfabetik)..
Sekuensing Daerah Cytochrome Oxidase Subunit I
7 dilakukan dengan teknik multiple sequence alignment analysis dengan menggunakan perangkat lunak (software) MEGA version 5.10 (Tamura et al. 2011).
Prosedur Analisis Data
Data setiap lokus mikrosatelit dianalisis menggunakan perangkat lunak
(software) GenAlEx 6.41 (Peakall et al. 2012) dan Cervus 3.0 (Kalinowski et al.
2007). Adapun peubah yang diamati adalah jumlah alel (na), alel efektif (ne), nilai heterozigositas pengamatan (Ho), nilai heterozigositas harapan (He) dan
polymorphism information content (PIC). Jarak genetik Nei dan dendogram
diperoleh dari frekuensi alel dengan menggunakan software POPTREE2 (Takezaki et al. 2010). Dendogram menggunakan metoda Neighbor-Joining
8
4
HASIL DAN PEMBAHASAN
Amplifikasi dan Genotyping Tiga Lokus Mikrosatelit
Amplifikasi lokus ILSTS17, ILSTS61 dan CSSM66 mikrosatelit diperoleh hasil optimal yaitu dengan suhu annealing masing-masing lokus adalah 55oC (Gambar 1, 2 dan 3). Suhu annealing yang diperoleh sama dengan Kathiravan et al. (2009) yaitu 55oC.
Keterangan:
M adalah marker 20 bp
Gambar 1 Hasil elektroforesis lokus ILSTS17 dengan teknik PAGE 10%
Keterangan:
M adalah marker 20 bp
Gambar 2 Hasil elektroforesis lokus ILSTS61 dengan teknik PAGE 10%
Keterangan:
M adalah marker 20 bp
9 CB (0.071), CC (0.118), DB (0.014), DC (0.104), DD (0.047), EA (0.009), EB (0.052), EC (0.100), ED (0.076), dan EE (0.014). Lokus ILSTS memiliki tiga genotipe dengan frekuensi masing-masing terdiri atas genotipe AA (0.497), BA (0.081) dan BB (0.422). Lokus CSSM66 dan ILSTS17 memiliki jumlah genotipe yang sangat terbatas, dibandingkan dengan lokus ILSTS61, padahal lokus CSSM66 dan ILSTS17 memiliki motif ulangan yang lebih banyak dibandingkan dengan lokus ILSTS61 (Kathiravan et al. 2009).
Frekuensi Alel Mikrosatelit
Hasil analisis lokus CSSM66, ILSTS61 dan ILSTS17 pada populasi kerbau di Indonesia diperoleh jumlah alel masing-masing dua alel, lima alel dan dua alel. Adapun frekuensi alel setiap lokus dan setiap populasi kerbau yang dianalisis disajikan pada Tabel 3.
Tabel 3 Frekuensi alel lokus CSSM66, ILSTS61 dan ILSTS17 pada populasi kerbau di Indonesia Sulawesi Selatan; KS: Kerbau Sungai
10
Keragaman Genetik Kerbau di Indonesia
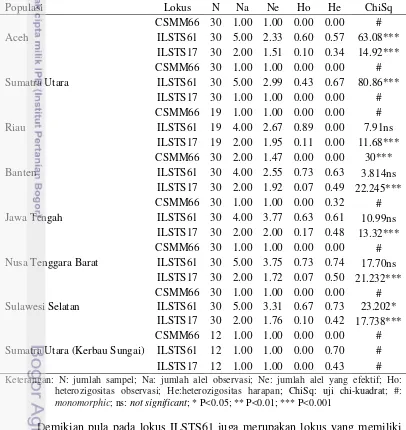
Hasil analisis mikrosatelit menunjukkan bahwa lokus ILSTS61 memiliki nilai heterozigositas yang tinggi pada semua populasi kerbau rawa kecuali pada populasi kerbau sungai di Sumatra Utara. Hasil uji keseimbangan Hardy-Weinberg menunjukkan bahwa lokus ILSTS61 pada populasi Riau, Banten, Jawa Tengah dan Nusa Tenggara Barat berada dalam keseimbangan Hardy-Weinberg yang berarti bahwa pada populasi tersebut tidak terjadi seleksi, mutasi, migrasi dan genetic drift.
Tabel 4 Keragaman genetik pada kerbau di Indonesia
Populasi Lokus N Na Ne Ho He ChiSq
Aceh
CSMM66 30 1.00 1.00 0.00 0.00 #
ILSTS61 30 5.00 2.33 0.60 0.57 63.08***
ILSTS17 30 2.00 1.51 0.10 0.34 14.92***
Sumatra Utara
CSMM66 30 1.00 1.00 0.00 0.00 #
ILSTS61 30 5.00 2.99 0.43 0.67 80.86***
ILSTS17 30 1.00 1.00 0.00 0.00 #
Riau
CSMM66 19 1.00 1.00 0.00 0.00 #
ILSTS61 19 4.00 2.67 0.89 0.00 7.91ns
ILSTS17 19 2.00 1.95 0.11 0.00 11.68***
Banten
CSMM66 30 2.00 1.47 0.00 0.00 30***
ILSTS61 30 4.00 2.55 0.73 0.63 3.814ns
ILSTS17 30 2.00 1.92 0.07 0.49 22.245***
Jawa Tengah
CSMM66 30 1.00 1.00 0.00 0.32 #
ILSTS61 30 4.00 3.77 0.63 0.61 10.99ns
ILSTS17 30 2.00 2.00 0.17 0.48 13.32***
Nusa Tenggara Barat
CSMM66 30 1.00 1.00 0.00 0.00 #
ILSTS61 30 5.00 3.75 0.73 0.74 17.70ns
ILSTS17 30 2.00 1.72 0.07 0.50 21.232***
Sulawesi Selatan
CSMM66 30 1.00 1.00 0.00 0.00 #
ILSTS61 30 5.00 3.31 0.67 0.73 23.202*
ILSTS17 30 2.00 1.76 0.10 0.42 17.738***
Sumatra Utara (Kerbau Sungai)
CSMM66 12 1.00 1.00 0.00 0.00 #
ILSTS61 12 1.00 1.00 0.00 0.70 #
ILSTS17 12 1.00 1.00 0.00 0.43 #
Keterangan: N: jumlah sampel; Na: jumlah alel observasi; Ne: jumlah alel yang efektif; Ho: heterozigositas observasi; He:heterozigositas harapan; ChiSq: uji chi-kuadrat; #:
monomorphic; ns: not significant; * P<0.05; ** P<0.01; *** P<0.001
11
Keterangan: Na: jumlah alel observasi; N: jumlah sampel; Hobs: heterozigositas observasi; Hexp: heterozigositas harapan; PIC: Polymorphism Information Content; HW: keseimbangan hardy-weinberg; nd: not done; ns: not significant, * P<0.05, ** P<0.01, *** P<0.001
Jarak Genetik Kerbau Indonesia
Hasil analisis jarak genetik Nei (Da) (Nei et al. 1983) menunjukkan bahwa hubungan kekerabatan terutama pada populasi kerbau yang terdapat di setiap wilayah yang diamati yaitu pada populasi kerbau di Aceh, Sumatra Utara, Sumatra Utara (kerbau sungai), Riau, Banten, Jawa Tengah, Nusa Tenggara Barat dan Sulawesi Selatan menunjukkan jarak genetik yang berbeda pada setiap populasi kerbau (Tabel 6). Berdasarkan data jarak genetik, dibuat rekonstruksi pohon genetik (filogenetik) pada setiap populasi kerbau di Indonesia sebagaimana disajikan pada Gambar 4.
Tabel 6 Jarak genetik dari masing-masing populasi
Populasi 2 3 4 5 6 7 8
Keterangan : 1) Aceh, 2) Sumatra Utara, 3) Riau, 4) Banten, 5) Jawa Tengah, 6) Nusa Tenggara Barat , 7) Sulawesi Selatan dan 8) Sumatra Utara (Kerbau Sungai)
12
Gambar 4 memperlihatkan bahwa setiap populasi kerbau yang dianalisis menggunakan tiga lokus mikrosatelit mampu memisahkan antar populasi kerbau di Indonesia yaitu populasi kerbau di wilayah Aceh, Sumatra Utara, Riau, Banten, Jawa Tengah, Nusa Tenggara Barat, Sulawesi Selatan dan Sumatra Utara (kerbau sungai). Berdasarkan pohon genetik jelas memperlihatkan bahwa populasi kerbau yang terdapat di Sumatra Utara (kerbau sungai) memiliki jarak genetik yang jauh dibandingkan dengan populasi kerbau lainnya. Hal ini disebabkan bahwa populasi kerbau yang terdapat di Sumatra Utara (kerbau sungai) merupakan kerbau tipe perah/sungai (river buffalo), berbeda dengan populasi kerbau yang terdapat di Aceh, Sumatra Utara, Riau, Banten, Jawa Tengah, Nusa Tenggara Barat dan Sulawesi Selatan yang merupakan tipe daging/lumpur (swamp buffalo).
Keragaman Gen Cytochrome Oxidase Subunit I (COI)
Hasil amplifikasi cytochrome oxidase subunit I menggunakan overlapping
primer dengan dua pasang primer untuk melengkapi sekuen cytochrome oxidase subunit I (Gambar 5). Kerbau yang berasal dari Aceh tidak dapat diamplifikasi dengan kedua primer (FS1 dan FS2), namun dapat diamplifikasi dengan menggunakan primer FS3. Hal ini dapat diperkirakan bahwa adanya perbedaan basa nukleotida primer dengan basa nukleotida pada sampel kerbau Aceh sehingga kedua primer tersebut tidak dapat diamplifikasi.
Keterangan:
M adalah marker 100 bp, Primer FS1 (884 bp) dan Primer FS2 (1047 bp)
Gambar 5 Hasil elektroforesis menggunakan primer FS1 dan FS2 dengan agarose 1.5 %
Keterangan:
M adalah marker 100 bp, Primer FS2 (781 bp)
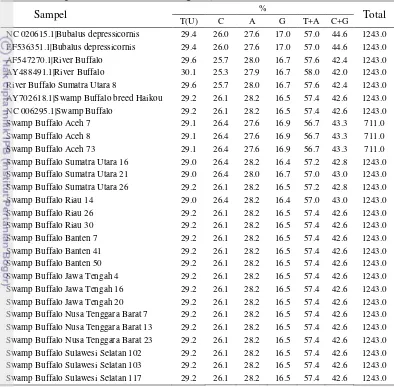
13 Komposisi nukleotida pada semua sampel yang digunakan pada penelitian ini hanya mempunyai sedikit perbedaan terutama pada sampel kerbau rawa di Aceh dan sampel kerbau rawa Sumatra Utara (Tabel 7).
Tabel 7 Komposisi nukleotida sekuen gen cytochrome oxidase subunit I (COI)
Sampel % Total
T(U) C A G T+A C+G
NC 020615.1|Bubalus depressicornis 29.4 26.0 27.6 17.0 57.0 44.6 1243.0 EF536351.1|Bubalus depressicornis 29.4 26.0 27.6 17.0 57.0 44.6 1243.0
AF547270.1|River Buffalo 29.6 25.7 28.0 16.7 57.6 42.4 1243.0
14
Tabel 8 Haplotipe dan posisi keragamannya berdasarkan sekuen gen COI
H
15
Anoa dan kerbau sungai memiliki memiliki nukleotida guanine (G) pada posisi 555 bp di gen cytochrome oxidase subunit I (COI), sedangkan kerbau rawa memiliki basa nukleotida adenine (A) kecuali kerbau rawa Aceh memiliki basa nukleotida thymine (T) sebagaimana disajikan pada Tabel 8 dan Gambar 7.
Gambar 7 Sekuen gen cytochrome oxidase subunit I COI pada kerbau Indonesia
Gambar 8 Pohon filogeni kerbau Indonesia berdasarkan sekuen gen COI dengan metode neighbor-joining dan model substitusi kimura 2 parameter
Swamp Buffalo Sumatra Utara 21 Swamp Buffalo Sulawesi Selatan 102
Swamp Buffalo Nusa Tenggara Barat 13 Swamp Buffalo Jawa Tengah 16 Swamp Buffalo Banten 41 Swamp Buffalo Riau 26 Swamp Buffalo Sumatra Utara 16 Swamp Buffalo Aceh 7
EF536351.1|Bubalus depressicornis AY702618.1|Swamp Buffalo breed Haikou Swamp Buffalo Aceh 8
Swamp Buffalo Sumatra Utara 26 Swamp Buffalo Riau 30 Swamp Buffalo Banten 50 Swamp Buffalo Jawa Tengah 20
AF547270.1|River Buffalo River Buffalo Sumatra Utara 8 NC 020615.1|Bubalus depressicornis NC 006295.1|Swamp Buffalo Swamp Buffalo Aceh 73 Swamp Buffalo Riau 14 Swamp Buffalo Banten 7 Swamp Buffalo Jawa Tengah 4 Swamp Buffalo Nusa Tenggara Barat 7 Swamp Buffalo Nusa Tenggara Barat 23 Swamp Buffalo Sulawesi Selatan 103 Swamp Buffalo Sulawesi Selatan 117
AY488491.1|River Buffalo
63
16
Hasil penelitian menunjukkan terdapat kedekatan secara genetic kerbau rawa Indonesia dengan kerbau rawa asal China (Kode akses genbank AY702618 dan NC 006295). Hal ini mengukuhkan penelitian yang menyatakan bahwa kerbau rawa merupakan hasil domestikasi kerbau liar (Bubalus arnee) yang dilakukan di China sekitar 7000 tahun yang lalu (Chen dan Li 1989 & Kumar et al. 2007). Pohon filogeni dalam penelitian ini menggambarkan perbedaan kerbau sungai (Bubalus bubalis) dengan kerbau rawa (Bubalus bubalis carabanesis) dalam tingkat subspecies. Keragaman genetik menggunakan D-loop dan
cytochrome b ditemukan delapan haplotipe untuk D-loop dan satu haplotipe untuk
cytochrome b pada kerbau rawa di Bogor dan Sulawesi (Lau et al. 1998). Lei et al.
(2007) melaporkan bahwa ditemukan 12 haplotipe untuk kerbau rawa Cina menggunakan D-loop mitokondria dan dua garis keturunan maternal. Hasil penelitian juga ditemukan sekuens “unik” pada haplotipe 3. Hal yang sama ditemukan pada penelitian Kierstein et al. (2004) yang menemukan “eccentric
group” dengan menggunkan D-loop mitokondria. Kierstein et al. (2004)
berasumsi bahwa “eccentric group” ini terjadi selama migrasi ke Indochina dan
17
5
SIMPULAN DAN SARAN
Simpulan
Keragaman mikrosatelit tinggi terdapat pada lokus ILSTS61 dan ILSTS17, sedangkan pada lokus CSMM66 memiliki keragaman rendah. Ketiga lokus yang digunakan dapat menentukan perbedaan genetik pada setiap populasi kerbau di Indonesia. Keragaman genetik dengan menggunakan pendekatan gen COI di mitokondria pada populasi kerbau di Indonesia rendah dan tidak dapat dibedakan secara jelas antar populasi kerbau, namun ditemukan empat macam haplotipe pada kerbau rawa Indonesia.
Saran
18
DAFTAR PUSTAKA
Abdullah MAN, Noor RR, Martojo H, Solihin DD. 2008. Genetics characterization of Aceh cattle utilizing microsatellite DNA analyses. JITAA
33: 165-175.
Barker JSF, Moore SS, Hetzel DJS, Evans D, Tan SG, Byrne K. 1997. Genetic diversity of Asian water buffalo (Bubalus bubalis): microsatellite variation and a comparisonwith protein-coding loci. Anim. Gene. 28: 103–115.
Borghese A. 2005. Buffalo production and research. FAO. Rome.
Byun SO, Fang Q, Zhou H, Hickford JGH. 2009. An effective method for silver-staining DNA in large numbers of polyacrylamide gels. Anal. Biochem. 385: 174–175
Caughley G. 1994. Directions in conservation biology. J. Anim. Eco. 63: 215– 244.Chen YC, Li XH. 1989. New evidence of the origin and domestication of the Chinese swamp buffalo (Bubalus bubalis). Buffalo Journal 1: 51–5.
Cywinska AC, Hunter FF, Hebert PDN. 2006. Identifying Canadian mosquito species through DNA barcodes. Med. Veter. Entomol. 20: 413-424.
Duggan AT, Kocha KM, Monk CT, Bremer K, Moyes CD. 2011. Coordination of cytochrome c oxidase gene expression in the remodelling of skeletal muscle. J.
Exper. Biol 214: 1880-1887
Ellegren H. 2004. Microsatellites: Simple sequences with complex evolution: Nature Reviews. Genetics 5: 435-445.
FAO. 2011. Molecular genetic characterization of animal genetic resources. FAO Animal Production and Health Guidelines. No. 9. Rome.
Funk DJ, Futuyma DJ, Orti G, Meyer A. 1995. Mitochondrial DNA sequence and multiple data sets: A phylogenetic study of phyotophagus beetles (Chrysomelidae: Ophraella). Mol. Biol. Evol. 12: 627-640.
Gao YS, Tu YJ, Lu JX, Zhang XY. 2011. Studies on the DNA barcoding of two newly discovered chicken breeds by mtDNA COI gene. J. Anim. Vet. Adv. 10: 1711-1713.
Gasser RB, Zhu X, McManus DP. 1999. NADH dehydrogenase subunit 1 and cytochrome c oxidase subunit I sequences compared for members of the genus
Taenia (Cestoda). Inter. J. Parasit 29: 1965-1970.
Hartl DL, Clark AG. 1997. Principles of population genetics 3rd ed. Sinauer Associates. Sunderland, MA.
Hebert PDN, Stoeckle MY, Zemlak TS, Francis CM. 2004. Identification of Birds through DNA Barcodes. PLoS Biol. 2: e312.
Iannuzzi L. 1998. A genetic physical map in river buffalo (Bubalus bubalis,
2n=50 ). Caryologia. 51: 311-318
Jakaria, M.S.A. Zein MSA, Sulandari S, Subandriyo, Muladno. 2012. The use of microsatellite markers to study genetic diversity in Indonesian sheep. JITAA
37: 1-9.
Jeffreys AJ, NJ Royle, Willson V, Wong Z. 1988. Spontaneous mutation rates to new length allels at tandem- repetitive hypervariable loci in human DNA.
Nature. 332: 278-281.
19 Kalinowski ST, Taper ML, Marshall TC. 2007. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Mol. Eco. 16: 1099-1006.
Kathiravan P, Mishra BP, Kataria RS, Sadana DK. 2009. Evaluation of genetic architecture and mutation drift equilibrium of marathwada buffalo population in central India. J. Livsci 121: 288-289
Kierstein G, Vallinoto M, Silva A, Schneider MP, Iannuzzi L, Breniga B. 2004. Analysis of mitochondrial D-loop region casts new light on domestic water buffalo (Bubalus bubalis) phylogeny. Mol. Phylogenet. Evol. 30: 308–32. Kimura M, Crow JF. 1964. The number of alleles that can be maintained in a
finite population. Genetics. 49: 725-738.
Kimura M, Ohta T. 1978. Stepwise mutation model and distribution of allelic frequencies in a finite population. Proceedings of the National Academy of Sciences of the United States of America 75: 2868-2872.
Kumar S, Nagarajan M, Sandhu JS, Kumar N, Behl V, Nishanth G. 2007. Mitochondrial DNA analyses of Indian water buffalo support a distinct genetic origin of river and swamp buffalo. Anim Genet. 8: 227-32.
Lau, C. H., R. D. Drinkwater, K.Yusoff, S. G. Tan, D. J. Hetzel, J. S. Barker. 1998. Genetic diversity of Asian water buffalo (Bubalus bubalis): mitochondrial DNA D-loop and cytochrome b sequence variation. Anim Genet. 29: 253-64.
Lehman T, Wiliam AH, Collins FH. 1996. An evolutionary constrains on microsatellite loci using null allels. J. Genet. 144: 1155-1167.
Levinson G, Gutman GA. 1987. Slipped-satrand mispairing: a major mechanism for DNA sequence evolution. Mol. Biol. Evo. 4: 203-221
Lei CZ, Zhang W, Chen H, Lu F, Ge QL, Liu RY, Dang RH, Yao YY, Yao LB, Lu ZF, Zhao ZL. 2007. Two Maternal Lineages Revealed by Mitochondrial DNA D-loop Sequences in Chinese Native Water Buffaloes (Bubalus bubalis). Asian-Aust. J. Anim. Sci. 20: 471-476
Li YC, Korol AB, Fahima T, Beiles A, Nevo E. 2002. Microsatellites: Genomic distribution, putative functions and mutational mechanisms: A review. Mol.
Eco. 11: 2453-2465.
Nei M, Tajima F, Tateno Y. 1983. Accuracy of estimated phylogenetic trees from molecular data. J. Mol. Evol. 19: 153-170.
Nei M. 1987. Molecular Evolutionary Genetics. Columbia University Press. New York, USA.
Palumbi SR. 1996. Nucleic acids II: the polymerase chain reaction. Dalam
Molecular systematics. Ed ke-2. Edited by Hillis DM, Moritz C, Mable BK. Massachusetts: Sinauer.
Peakall R, Smouse O. 2012. GenAlEx 6.5: Genetic analysis in Excel. Population genetic software for teaching and research – an update. Bioinformatics 28: 1-3. Ross C. 1975. Alternative Livestock: with particular response to the water buffalo
(Bubalus bubalis). In: Meat. 1st publication: 507-524.
Selkoe KA, Toonen RJ. 2006. Microsatellites for ecologists: a practical guide to using and evaluating microsatellite markers. Eco. Letters. 9: 615-629.
Shaffer ML. 1981. Minimum population sizes for species conservation.
20
Sumantri C, Farajallah A, Fauzi U, Salamena JF. 2008. Keragaman Genetik DNA Mikrosatelit dan Hubungannya dengan Performa Bobot Badan pada Domba Lokal. Med. Pet. 31: 1-13
Suriana D, Solihin D, Noor RR, Thohari AM. 2012. The Characteristics of Cytochrome C Oxidase Gene Subunit I in Wild Silkmoth Cricula trifenestrata
Helfer and Its Evaluation for Species Marker. Med. Pet. 35: 102-110
Syed-Shabthar SM, Rosli MK, Mohd-Zin NA, Romaino SM, Fazly-Ann ZA, Mahani MC, Abas-Mazni O, Zainuddin R, Yaakop S, Md-Zain BM. 2013. The molecular phyogenetic signature of Bali cattle revealed by maternal and paternal markers. Mol Biol Rep. [Epub ahead to print]
Takezaki N, Nei M, Tamura K. 2010. POPTREE2: Software for Constructing Population Trees from Allele Frequency Data and Computing Other Population Statistics with Windows Interface. Mol. Biol. Evol. 27: 747–752
Tamura K, Dudley J, Nei M, Kumar S. 2011. MEGA software (version 5): Molecular Evolutionary Genetics Analysis. Center of Evolutionary Functional Genomics Biodesign Institute. Arizona State University.
White HW, Kusukawa N. 1997. Agarose-based system for separation of short tandem repeat loci. BioTechniques. 22: 976-980.
Wilson JJ. 2010. Assessing the value of DNA barcodes and other priority gene regions for molecular phylogenetics of Lepidoptera. Plos One 5: e10525. Wu W, Schmidt TR, Goodman M, Grossman LI. 2000. Molecular evolution of
cytochrome c oxidase subunit 1 in primates: is there coevolution between mitochondrial and nuclear genomes. Mol. Phylogenet. Evol. 17: 294-304.
21