ANALISIS MOLEKULER
FUSARIUM
SPECIES COMPLEX
DARI
BEBERAPA DAERAH DI INDONESIA BERDASARKAN SEKUEN DNA
DAERAH ITS rDNA DAN GEN
EF-1
α
FENI TUNARSIH
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Analisis Molekuler
Fusarium Species Complex dari Beberapa Daerah di Indonesia Berdasarkan Sekuen DNA Daerah ITS rDNA dan Gen EF-1α adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
RINGKASAN
FENI TUNARSIH. Analisis Molekuler FusariumSpecies Complex dari Beberapa Daerah di Indonesia Berdasarkan Sekuen DNA Daerah ITS rDNA dan Gen
EF-1α. Dibimbing oleh GAYUH RAHAYU dan IMAN HIDAYAT.
Fusarium merupakan salah satu cendawan yang bersifat patogen atau endofit pada beragam inang atau hidup sebagai saprob. Fusarium dibangun oleh Link pada tahun 1809. Hingga akhir tahun 1980-an spesies Fusarium dikenali melalui ciri morfologinya. Ciri morfologi mencakup ciri koloni dan ciri mikroskopisnya pada media tumbuh tertentu. Secara mikroskopis, Fusarium
dicirikan oleh adanya makrokonidia, mikrokonidia, klamidospora, konidiofor, dan sel konidiogen. Seiring dengan perkembangan teknologi, spesies Fusarium
diidentifikasi dengan menggunakan pendekatan molekuler, salah satunya ialah teknik Polymerase Chain Reaction (PCR) pada marka molekuler tertentu.
Di Indonesia umumnya spesies Fusarium diidentifikasi dengan pendekatan morfologi. Identifikasi secara molekuler sudah dilakukan namun identitas ditetapkan sebatas pencarian BLAST yang tersedia pada GenBank. Identifikasi tidak dilengkapi dengan analisis filogenetik. Oleh sebab itu, penelitian ini bertujuan menganalisis keragaman dan hubungan kekerabatan beberapa Fusarium species complex asal Indonesia koleksi Institut Pertanian Bogor Culture Collection (IPBCC) dengan menggunakan gen ITS rDNA dan EF-1α.
Identifikasi secara molekuler dilakukan dengan analisis filogenetik berdasarkan data sekuen DNA dari daerah ITS rDNA dan gen EF-1α dengan menggunakan metode Maximum Likelihood (ML). Sekuen DNA Fusarium hasil amplifikasi daerah ITS rDNA dan gen EF-1α selanjutnya didepositkan pada
database GenBank.
Berdasarkan analisis filogenetik sekuen DNA daerah ITS rDNA, 41 galur
Fusarium pada penelitian ini diklasifikasikan ke dalam 5 species complex
monofiletik, yaitu F. decemcellulare species complex (FDSC), F. graminearum species complex (FGSC), F. oxysporum species complex (FOSC), F. solani species complex (FSSC), dan F. tricinctum species complex (FTSC), serta ke dalam F. fujikuroi species complex (FFSC) yang polifiletik. Berdasarkan hubungan kekerabatannya dengan sekuen spesies rujukan, satu galur diidentifikasi sebagai F. cerealis,5 galur F. decemcellulare, 16 galur F. oxysporum, 9 galur F. solani, dan 10 galur tidak dapat diidentifikasi ke tingkat spesies.
Diantara 41 galur yang dipelajari, gen EF-1α hanya dapat diamplifikasi dari 11 galur. Hasil analisis filogenetik gen EF-1α mendukung hasil analisis filogenetik daerah ITS. Galur-galur yang sebelumnya diidentifikasi sebagai F. cerealis, F. decemcellulare, F. oxysporum, F. solani, memiliki identitas yang sama pada analisis filogenetik gen EF-1-nya Beberapa galur yang sebelumnya tidak teridentifikasi sampai dengan spesies, dapat diberi nama spesies yaitu F. ambrosium, F. concentricum, dan F. verticillioides.
Analisis filogenetik juga menunjukkan tidak adanya spesifisitas inang dari spesies Fusarium yang dianalisis. Fusarium pada tingkat spesies dan Fusarium
pada tingkat species complex dapat memiliki berbagai strategi hidup, yaitu saprob, endofit, dan patogen.
SUMMARY
FENI TUNARSIH. Molecular Analysis of Fusarium Species Complex from Several Regions in Indonesia Based on DNA Sequence of ITS rDNA Region dan
EF-1α Gene. Supervised by GAYUH RAHAYU and IMAN HIDAYAT.
Fusarium is a fungal genus associated with various sources, and living as pathogens, endophytes or saprobes. Fusarium was first described by Link in 1809. Up to end of 1980’s, Fusarium were identified based on their morphological traits. The morphological approach involves microscopic characters examination, viz. macroconidia, microconidia, chlamydospore, and conidiogenous cell on a certain agar medium. With the availability of molecular technology, identification of species within Fusarium have shifted from morphological to molecular approach through Polymerase Chain Reaction (PCR) technique using specific molecular marker.
Morphological-based identification had mainly been used to recognize
Fusarium species in Indonesia. Several molecular-based Fusarium identification from Indonesia has been done, but they were heavily relied on BLAST search result and not involving phylogenetic analysis. In this study, the diversity and phylogenetic relationship of Fusarium species complex from Indonesia deposited at IPBCC (IPB Culture Collection) is elucidated using phylogenetic analysis based on nucleotide sequences generated from the ITS rDNA region and EF-1α gene.
Molecular identification of Fusarium was carried out by phylogenetic analysis based on DNA sequence generated from ITS rDNA region and EF-1α gene using Maximum Likelihood (ML) method. All DNA sequences generated from ITS rDNA region and EF-1α gene from this study have been submitted to GenBank.
Phylogenetic ITS tree showed that 41 Fusarium strains belong to five monophyletic species complexes, namely F. decemcellulare species complex (FDSC), F. graminearum species complex (FGSC), F. oxysporum species complex (FOSC), F. solani species complex (FSSC) and F. tricinctum species complex (FTSC), and one polyphyletic F. fujikuroi species complex (FFSC). Based on their relationship with reference strains, one strain was identified as F. cerealis, 5 strains were F. decemcellulare, 16 strains were F. oxysporum, 9 strains were F. solani and 10 strains could not be identified into species level.
EF-1α gene of 11 out of 41 Fusarium strains could be amplified. The phylogenetic analysis based on this gene supported the above phylogenetic analyses. Strains that were identified as F. cerealis, F. decemcellulare, F. oxysporum, F. solani has the same species name based on phylogenetic analysis of EF-1gene By using ITS rDNA some strains can be identified as F. ambrosium, F. concentricum and F. verticillioides.
The phylogenetic analysis indicated that Fusarium species shown no host specificity. One Fusarium species may asscociate with different sources. Further, one species complex may contains Fusarium of different lifestyles, viz. saprobe, endophyte, and pathogen.
© Hak Cipta Milik IPB, Tahun 2015
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Mikrobiologi
ANALISIS MOLEKULER
FUSARIUM
SPECIES COMPLEX
DARI BEBERAPA DAERAH DI INDONESIA BERDASARKAN
SEKUEN DNA DAERAH ITS rDNA DAN GEN
EF-1
α
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
BOGOR 2015
PRAKATA
Puji dan syukur penulis panjatkan kepada Allah subhanahu wa ta’ala atas segala karunia-Nya sehingga penulis dapat menyelesaikan penelitian yang berjudul Analisis Molekuler Fusarium Species Complex dari Beberapa Daerah di Indonesia Berdasarkan Sekuen DNA Daerah ITS rDNA dan Gen EF-1α. Penelitian ini didanai oleh BOPTN IPB atas nama Dr Gayuh Rahayu. Penulis mengucapkan terima kasih kepada Dr Gayuh Rahayu sebagai ketua komisi pembimbing dan Dr Iman Hidayat sebagai anggota komisi pembimbing atas segala bimbingan, arahan, waktu, tenaga, dan nasehat sehingga saya dapat menyelesaikan penulisan karya ilmiah ini. Ucapan terima kasih saya sampaikan kepada Dr Nilam Fadmaulidha Wulandari sebagai penguji luar komisi dan Prof Dr Anja Meryandini, MS sebagai ketua program studi Mikrobiologi yang telah memberikan saran demi kesempurnaan karya ilmiah ini.
Ucapan terima kasih penulis sampaikan kepada Departemen Pendidikan Tinggi Republik Indonesia (DIKTI) atas beasiswa Fresh Graduate tahun 2012 sehingga penulis dapat melanjutkan studi S2 di Institut Pertanian Bogor. Penulis mengucapkan terima kasih kepada staf Laboratorium IPBCC yang telah membantu penulis selama penelitian di laboratorium serta teman-teman SPS Mikrobiologi 2012-2013 yang senantiasa memberikan doa dan dukungan.
Semoga karya ilmiah ini bermanfaat.
Bogor, Agustus 2015
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
Latar Belakang 1
Tujuan Penelitian 2
Manfaat Penelitian 2
2 METODE 2
Waktu dan Tempat Penelitian 2
Pengumpulan Sampel 2
Ekstraksi DNA, Amplifikasi PCR, dan Sekuensing 5
Analisis Filogenetik 5
3 HASIL DAN PEMBAHASAN 6
Hasil 6
Pohon filogenetik berdasarkan sekuen DNA daerah ITS rDNA 6
Pohon filogenetik berdasarkan gen EF-1α 7
Pembahasan 10
4 SIMPULAN DAN SARAN 13
Simpulan 13
Saran 13
DAFTAR PUSTAKA 13
LAMPIRAN 17
DAFTAR TABEL
1 Daftar galur Fusarium yang digunakan dalam penelitian 3
DAFTAR GAMBAR
1 Pohon filogenetik galur Fusarium asal berbagai sumber/inang di Indonesia berdasarkan daerah ITS rDNA dengan metode maximum
likelihood 8
2 Pohon filogenetik galur Fusarium asal berbagai sumber/inang di Indonesia berdasarkan gen EF-1α dengan metode maximum likelihood
10
DAFTAR LAMPIRAN
1 Daftar sekuen rujukan ITS galur Fusarium yang digunakan pada
penelitian 17
2 Daftar sekuen rujukan EF-1α galur Fusarium yang digunakan pada
1
PENDAHULUAN
Latar Belakang
Fusarium merupakan cendawan yang dapat bersifat patogen, endofit, dan saprob. Fusaria patogen dan endofit telah dilaporkan menginfeksi berbagai tanaman inang, seperti Asparagus officinalis, Begonia × hiemalis, Brassica oleracea, Capsicumannuum, Cucumis melo, Cyclamen persicum, Daucus carota,
Dianthus caryophyllus, Hosta sp., Hyacinthus sp., Matthiola incana, Miscanthus sinensis, Musa acuminata, Phaseolus sp., Phyllostachys sp., Pisum sativum,
Saintpaulia ionantha (Schroers et al. 2004), Bryophyllum sp., Citrus sp., Fagus sylvatica, Gymnocalcium damsii, Hoodia gordonii, Solanum lycopersicum
(Schroers et al. 2009), dan M. sapientum (Summerbell & Schroers 2002). Fusaria patogen dilaporkan pula menginfeksi hewan, diantaranya adalah serangga (Acari, Coleoptera, Diptera, Hemiptera, Lepidoptera, Orthoptera, Thysanoptera) dan nematoda (Tylenchida) (O’Donnell et al. 2012). Selain itu, fusaria saprob terdistribusi secara luas pada tanah, humus, dan kayu lapuk (Schroers et al. 2009; Silvestro et al. 2013).
Genus Fusarium pertama kali dideskripsikan oleh Link pada tahun 1809 (Aoki et al. 2014). Fusarium (Nectriaceae, Hypocreales, Ascomycetes,
Ascomycota) dicirikan oleh adanya struktur mikroskopik, yaitu makrokonidia, mikrokonidia, klamidospora, dan fialid. Fusarium berasosiasi dengan beberapa fase teleomorf, yaitu Gibberella, Hematonectria, dan Albonectria (Moretti 2009). Hingga akhir tahun 1980-an, identifikasi spesies Fusarium masih dilakukan dengan menggunakan pendekatan morfologi. Taksonomi dan identifikasi
Fusarium mengalami perkembangan dari pendekatan morfologi ke molekuler dengan menggunakan teknik ekstraksi DNA dan Polymerase Chain Reaction
(PCR) sehingga identifikasi dapat dilakukan lebih cepat (Oechsler et al. 2009). Pada tahun 1989, Guadet et al. (1989) mempelajari hubungan kekerabatan
Fusarium dengan menggunakan sekuen subunit besar rRNA. Pada tahun 1991, Bruns et al. (1991) mempelajari sistematika molekuler cendawan Fusarium
berdasarkan pada karakter morfologi dan genetik. Seiring dengan perkembangan teknologi, identifikasi fusaria saat ini dilakukan dengan pendekatan molekuler yang didukung oleh karakter morfologi (Baayen et al. 2001; Geiser et al. 2004; O’Donnell et al. 2008; O’Donnell et al. 2012).
Teknik PCR dapat dilakukan dengan menggunakan marka molekuler. Marka molekuler yang umum digunakan untuk mengidentifikasi cendawan ialah gen ITS rDNA dan EF-1α (Schroers et al. 2004; Ceng et al. 2008; Zhao & Lu 2008; Schroers et al. 2009; Pinaria et al. 2010; Hove et al. 2011; Wang et al. 2011; O’Donnell et al. 2012). Daerah ITS rDNA memiliki tingkat keberhasilan tinggi untuk mengidentifikasi cendawan melalui pendekatan molekuler (Schoch et al. 2012). Evaluasi dari 18S rDNA, ITS1, 5.8S rDNA, 28S rDNA, β-tubulin, dan aminoadipate reductase (lys2) untuk mengidentifikasi spesies Fusarium
yang memuat sekuen molekular dari spesies fusaria tidak tersedia secara lengkap pada database GenBank (Geiser et al. 2004; Oechsler et al. 2009).
Identifikasi Fusarium di Indonesia umumnya dilakukan berdasarkan karakter morfologi. Jumjunidang et al. (2012) mengidentifikasi Fusarium dengan menggunakan analisis vegetative compatibility group. Saat ini, identifikasi
Fusarium di Indonesia telah dilakukan dengan menggunakan pendekatan molekuler (Pinaria et al. 2010, Nugroho et al. 2013, Nurbaya et al. 2014). Pada studi tersebut, identifikasi spesies dari Fusarium hanya dilakukan dengan menggunakan aplikasi pencarian BLAST serta tidak dilengkapi dengan analisis filogenetik. Pada studi ini, identifikasi secara molekuler dilakukan dengan menggunakan koleksi fusaria IPBCC dengan mikrokonidia yang berlimpah, sedikit ataupun tidak ada.
Tujuan Penelitian
Penelitian ini bertujuan menganalisis keragaman dan hubungan kekerabatan
Fusarium spesies complex di Indonesia melalui pendekatan molekuler dengan menggunakan data sekuen DNA dari daerah ITS rDNA dan gen EF-1α.
Manfaat Penelitian
Hasil penelitian ini diharapkan dapat memberikan informasi tentang keragaman cendawan pada umumnya, khususnya cendawan Fusarium species complex yang ada di Indonesia.
2
METODE
Waktu dan Tempat Penelitian
Penelitian dilaksanakan mulai dari bulan Maret 2013 sampai Oktober 2014 di laboratorium Institut Pertanian Bogor Culture Collection (IPBCC) dan laboratorium Mikologi, Departemen Biologi, FMIPA IPB, Dramaga, Bogor.
Pengumpulan Sampel
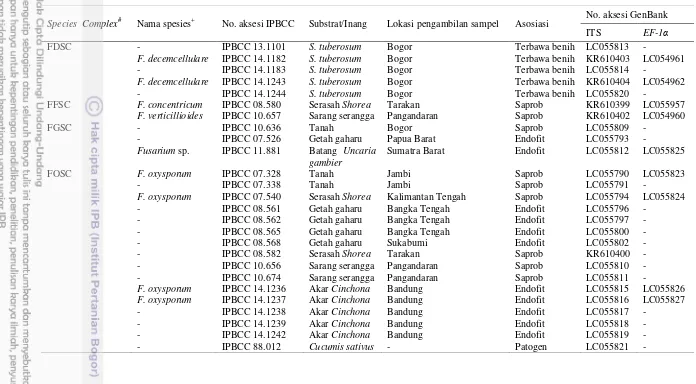
Sebanyak 41 galur cendawan Fusarium (Tabel 1) digunakan dalam penelitian ini yang terdiri atas 19 galur endofit asal tanaman obat-obatan, 14 galur saprob asal tanah dan serasah, 5 galur terbawa benih pada Solanum tuberosum, 2 galur patogen masing-masing asal Cucumis sativus dan S. tuberosum, dan 1 galur yang tidak diketahui sumber atau inangnya (IPBCC 2010).
Tabel 1 Daftar galur Fusarium yang digunakan dalam penelitian
Species Complex# Nama spesies+ No. aksesi IPBCC Substrat/Inang Lokasi pengambilan sampel Asosiasi No. aksesi GenBank ITS EF-1α
FDSC - IPBCC 13.1101 S. tuberosum Bogor Terbawa benih LC055813 -
F. decemcellulare IPBCC 14.1182 S. tuberosum Bogor Terbawa benih KR610403 LC054961
- IPBCC 14.1183 S. tuberosum Bogor Terbawa benih LC055814 -
F. decemcellulare IPBCC 14.1243 S. tuberosum Bogor Terbawa benih KR610404 LC054962
- IPBCC 14.1244 S. tuberosum Bogor Terbawa benih LC055820 -
FFSC F. concentricum IPBCC 08.580 Serasah Shorea Tarakan Saprob KR610399 LC055957
F. verticillioides IPBCC 10.657 Sarang serangga Pangandaran Saprob KR610402 LC054960
FGSC - IPBCC 10.636 Tanah Bogor Saprob LC055809 -
- IPBCC 07.526 Getah gaharu Papua Barat Endofit LC055793 -
Fusarium sp. IPBCC 11.881 Batang Uncaria
gambier
Sumatra Barat Endofit LC055812 LC055825
FOSC F. oxysporum IPBCC 07.328 Tanah Jambi Saprob LC055790 LC055823
- IPBCC 07.338 Tanah Jambi Saprob LC055791 -
F. oxysporum IPBCC 07.540 Serasah Shorea Kalimantan Tengah Saprob LC055794 LC055824
- IPBCC 08.561 Getah gaharu Bangka Tengah Endofit LC055796 - - IPBCC 08.562 Getah gaharu Bangka Tengah Endofit LC055797 - - IPBCC 08.565 Getah gaharu Bangka Tengah Endofit LC055800 - - IPBCC 08.568 Getah gaharu Sukabumi Endofit LC055802 - - IPBCC 08.582 Serasah Shorea Tarakan Saprob KR610400 - - IPBCC 10.656 Sarang serangga Pangandaran Saprob LC055810 - - IPBCC 10.674 Sarang serangga Pangandaran Saprob LC055811 -
F. oxysporum IPBCC 14.1236 Akar Cinchona Bandung Endofit LC055815 LC055826
F. oxysporum IPBCC 14.1237 Akar Cinchona Bandung Endofit LC055816 LC055827
- IPBCC 14.1238 Akar Cinchona Bandung Endofit LC055817 - - IPBCC 14.1239 Akar Cinchona Bandung Endofit LC055818 - - IPBCC 14.1242 Akar Cinchona Bandung Endofit LC055819 - - IPBCC 88.012 Cucumis sativus - Patogen LC055821 -
Tabel 1 Daftar galur Fusarium yang digunakan dalam penelitian (lanjutan...)
- : tidak diketahui sumber/inang, asosiasi, dan lokasi pengambilan sampel. + : nama spesies berdasarkan gen EF-1α.
* : galur Fusarium yang diperoleh dari kultur koleksi CBS (Centraalbureau voor Schimmelcultures), Utrecht, Belanda.
# : FDSC (F. decemcellulare species complex), FFSC (F. fujikuroi species complex), FGSC (F. graminearum species complex), FOSC (F. oxysporumspecies complex),FSSC (F. solanispecies complex), FTSC (F. tricinctumspecies complex).
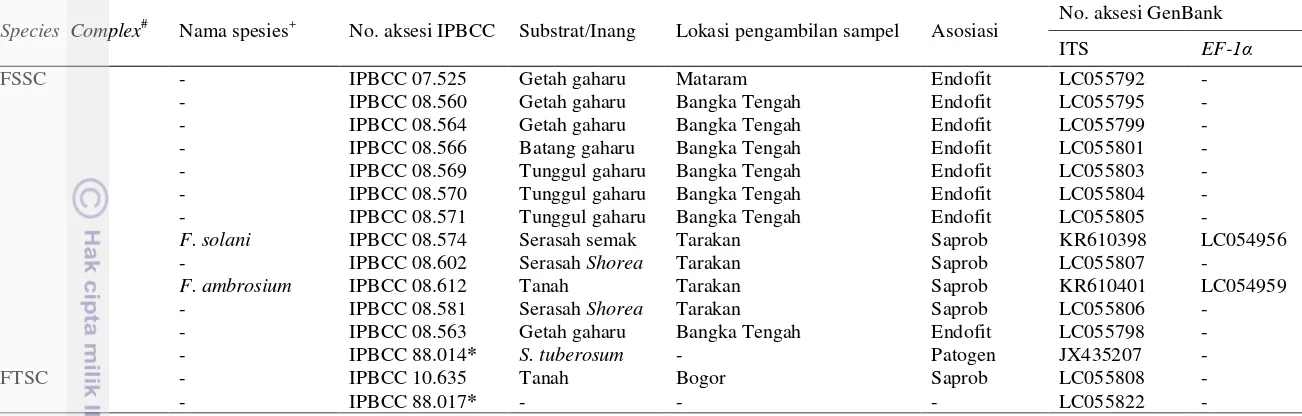
Species Complex# Nama spesies+ No. aksesi IPBCC Substrat/Inang Lokasi pengambilan sampel Asosiasi No. aksesi GenBank ITS EF-1α
FSSC - IPBCC 07.525 Getah gaharu Mataram Endofit LC055792 - - IPBCC 08.560 Getah gaharu Bangka Tengah Endofit LC055795 - - IPBCC 08.564 Getah gaharu Bangka Tengah Endofit LC055799 - - IPBCC 08.566 Batang gaharu Bangka Tengah Endofit LC055801 - - IPBCC 08.569 Tunggul gaharu Bangka Tengah Endofit LC055803 - - IPBCC 08.570 Tunggul gaharu Bangka Tengah Endofit LC055804 - - IPBCC 08.571 Tunggul gaharu Bangka Tengah Endofit LC055805 -
F. solani IPBCC 08.574 Serasah semak Tarakan Saprob KR610398 LC054956
- IPBCC 08.602 Serasah Shorea Tarakan Saprob LC055807 -
F. ambrosium IPBCC 08.612 Tanah Tarakan Saprob KR610401 LC054959
- IPBCC 08.581 Serasah Shorea Tarakan Saprob LC055806 - - IPBCC 08.563 Getah gaharu Bangka Tengah Endofit LC055798 - - IPBCC 88.014* S. tuberosum - Patogen JX435207 -
FTSC - IPBCC 10.635 Tanah Bogor Saprob LC055808 -
- IPBCC 88.017* - - - LC055822 -
Ekstraksi DNA, Amplifikasi PCR, dan Sekuensing
Ekstraksi DNA Fusarium dilakukan dengan cara menumbuhkan miselium pada media Potato Dextrose Broth (PDB) (Difco, USA) selama 5 hari pada suhu ruang di mesin penggoyang. Miselium cendawan selanjutnya dipanen, dikeringanginkan, dan dikeringdingingkan dengan menggunakan nitrogen cair. Proses ekstraksi DNA dilakukan dengan mengikuti prosedur Raeder & Broda (1985) dengan modifikasi.
Reaksi amplifikasi DNA dilakukan dengan menggunakan DNA konsentrasi 100 ng/µL. Amplifikasi daerah ITS dilakukan dengan membuat campuran PCR sebanyak 30 µL yang terdiri atas 1.2 µL DNA template, 3 µL Dream Taq Buffer
(mengandung MgCl2) (Thermo scientific, USA), 3 µL 2 mM dNTP (Thermo
scientific, USA), 0.6 µL 10 pmol primer ITS5 (forward) (5’ -GGAAGTAAAAGTCGTAACAAGG-3’) dan ITS4 (reverse) (5’ -TCCTCCGCTTATTGATAT-3’) (Thermo scientific, USA) (White et al. 1990), 0.75 µL DreamTaqpolymerase (Thermo scientific, USA), dan 20.85 µL ddH2O.
Reaksi PCR dilakukan dengan prosedur predenaturasi 94°C selama 5 menit, denaturasi 94°C selama 30 detik, penempelan 52°C selama 30 detik, pemanjangan 72°C selama 30 detik, dan pemanjangan akhir 72°C selama 7 menit. Amplifikasi gen EF dilakukan dengan membuat campuran PCR sebanyak 32 µL campuran PCR yang terdiri atas 0.65 µL DNA template, 3.25 µL Dream Taq Buffer
(mengandung MgCl2) (Thermo Scientific, USA), 2.6 µL 2 mM dNTP (Thermo
Scientific, USA), 1.63 µL masing-masing 10 pmol primer EF1 (forward) (5’ -GGARGTACCAGTSATCATG-3’) dan EF2 (reverse) (5’ -ATGGGTAAGGARGACAAGAC-3’) (Thermo Scientific, USA) (O’Donnell et al. 1998), 0.098 µL Dream Taq polymerase (Thermo Scientific, USA), dan 22.64 µL ddH2O. Reaksi PCR dilakukan dengan menggunakan prosedur predenaturasi
94°C selama 2 menit, denaturasi 94°C selama 35 detik, penempelan 53°C selama 50 detik, pemanjangan 72°C selama 2 menit, dan pemanjangan akhir 72°C selama 6 menit. Produk PCR selanjutnya dielektroforesis dengan menggunakan gel agarosa 1% pada 80 Volt selama 30 menit. Ladder 1 kb (Thermo Scientific, USA) digunakan sebagai marka ukuran amplikon. Gel agarosa selanjutnya direndam pada Ethidium Bromide (EtBr) selama 30 menit dan direndam pada akuades untuk membilas EtBr yang tersisa pada gel agarosa. Produk hasil PCR dikirim ke 1stBASE (Malaysia) untuk proses perunutan nukleotida.
Analisis Filogenetik
Sekuen nukleotida dari gen ITS dan EF-1α diedit dengan menggunakan program ChromasPro versi 1.7.7 (Technelysium, Australia). Sekuen disejajarkan dengan sekuen rujukan yang diunduh dari GenBank (http://www.ncbi.nlm.nih.gov). Analisis filogenetik dilakukan dengan menggunakan program MEGA6 (Molecular Evolutionary Genetics Analysis versi 6.0) (Tamura et al. 2013) dengan menggunakan parameter Maximum Likelihood
(ML). Kekuatan cabang internal pada pohon filogenetik diuji dengan analisis
atau lebih ditampilkan pada pohon filogenetik. Pohon filogenetik selanjutnya diolah dengan menggunakan program TreeGraph versi 2 (Stöver dan Müller 2010). Sekuen Hypocrea lixii CBS 226.95 dengan nomor aksesi GenBank AF057606 digunakan sebagai outgroup pohon filogenetik ITS. Sekuen F. tricinctum CBS 393.93 dengan nomor aksesi GenBank AB674295 digunakan sebagai outgroup pada pohon filogenetik EF-1α. Sekuen ITS dan EF-1α dari galur
Fusarium selanjutnya didepositkan pada GenBank (http://www.ncbi.nlm.nih.gov) dan DDBJ (http://www.ddbj.nig.ac.jp) (Tabel 2).
3
HASIL DAN PEMBAHASAN
Hasil
Pohon Filogenetik berdasarkan Sekuen DNA Daerah ITS rDNA
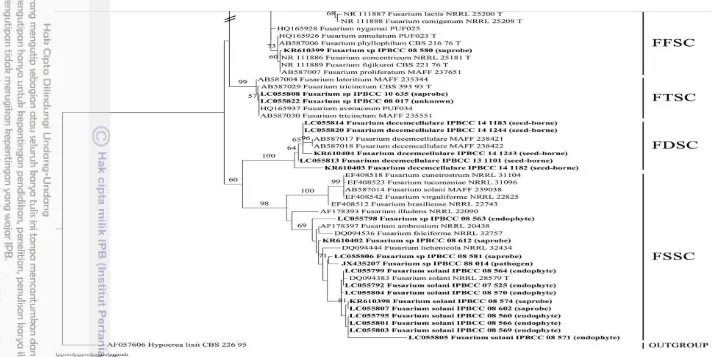
Pohon filogenetik dirancang dengan menggunakan sekuen tunggal ITS dengan Hypocrea lixii CBS 226.95 (AF057606) sebagai outgroup dan 88 sekuen ITS dari galur Fusarium. Hasil analisis menunjukkan bahwa keempat puluh satu galur Fusarium yang dipelajari diklasifikasikan ke dalam 5 species complex yang monofiletik, yaitu FGSC (BS=99%), FTSC (BS=99%), FDSC (BS=100%), FSSC (BS=98%), dan FOSC (BS=60%) (Oechsler et al. 2009, Schroers et al. 2009, O’Donnell et al. 2012, dan Aoki et al. 2014) dan clade FFSC yang polifiletik (Gambar 1). Hal ini menunjukkan bahwa ITS rDNA dapat digunakan untuk mengidentifikasi galur-galur Fusarium yang berada dalam clade FDSC, FGSC, FOSC, dan FSSC. Galur Fusarium pada clade tersebut diidentifikasi sebagai F. cerealis, F. decemcellulare, F. oxysporum dan F. solani.
Lima galur yaitu Fusarium IPBCC 13.1101, IPBCC 14.1182, IPBCC 14.1183, IPBCC 14.1243, dan IPBCC 14.1244 berada pada clade FDSC dan diidentifikasi sebagai F. decemcellulare (BS=100%). Satu galur, IPBCC 10.636 diidentifikasi sebagai F. cerealis (BS=67%) dan berada dalam clade FGSC. diidentifikasi sebagai F. solani (BS=81%).
Namun demikian terdapat beberapa galur yang sekuennya tidak membentuk
cluster yang kuat dengan sekuen rujukan. Galur-galur ini membentuk garis turunan tunggal atau membentuk cluster bersama dengan berbagai spesies. Galur-galur ini tidak dapat diidentifikasi hingga tingkat spesies. Galur Fusarium tersebut berada pada clade FFSC (IPBCC 10.657 dan IPBCC 08.580), FGSC (IPBCC 11.881 dan IPBCC 07.526), FTSC (IPBCC 88.017 dan IPBCC 10.635), dan FSSC (IPBCC 08.563, IPBCC 08.612, IPBCC 08.581, dan IPBCC 88.014) tidak dapat
diidentifikasi dengan menggunakan gen ITS rDNA. Kesepuluh galur tersebut diidentifikasi sebagai Fusarium sp.
Pada pohon filogenetik ini, satu Fusarium species complex terdiri dari galur-galur Fusarium yang berasal dari beragam substrat atau inang dan memiliki strategi hidup yang beragam, yaitu saprob, endofit, dan patogen. Pada clade
FOSC, galur Fusarium (n=16) bersifat endofit (n=9) pada tanaman gaharu dan kina, patogen (n=1) pada C. sativus, dan saprob (n=6) pada serasah, sarang serangga, dan tanah. Clade FSSC terdiri atas 13 galur Fusarium yang berasosiasi dengan beragam inang, yaitu endofit (n=8) pada gaharu, patogen (n=1), saprob (n=4) pada serasah dan tanah. Terdapat tiga galur Fusarium pada clade FGSC yang terdiri atas 2 galur Fusarium endofit (n= 2) pada gubal gaharu dan U. gambier, dan saprob (n=1) pada tanah. Clade FDSC terdiri atas 5 galur Fusarium
yang bersifat terbawa benih pada S. tuberosum. Clade FTSC terdiri atas 2 galur
Fusarium yang bersifat saprob dan berasal dari inang yang tidak diketahui (Gambar 1).
Pohon Filogenetik berdasarkan Gen EF-1α
Pohon filogenetik gen EF 1-disusun dengan menggunakan satu sekuen F. tricinctum CBS 393.93 (AB674295) sebagai outgroup dan 48 sekuen EF-1α dari galur Fusarium. Sebanyak 11 galur Fusarium yang diteliti termasuk ke dalam 4
clade yang monofiletik yaitu FOSC (BS=99%), FFSC (BS=99%), FDSC (BS=99%), dan FGSC (BS=88%) serta satu clade FFSC yang polifiletik (Gambar 1). Hasil analisis ini mendukung hasil analisis filogenetik berdasarkan ITS rDNA. Galur Fusarium IPBCC07.328, IPBCC 07.540, IPBCC 14.1236, dan IPBCC 14.1237 diidentifikasi sebagai F. oxysporum. Galur Fusarium IPBCC 08.580 diidentifikasi sebagai F. concentricum. Galur Fusarium IPBCC 10.657 sebagai F. verticillioides. Sebanyak dua galur Fusarium IPBCC 14.1243 dan IPBCC 14.1182 diidentifikasi sebagai F. decemcellulare. Galur Fusarium IPBCC 08.612 dan IPBCC 08.574 masing-masing diidentifikasi sebagai F. ambrosium dan sebagai F. solani (Tabel 1).
9
Gambar 1 Pohon filogenetik galur Fusarium asal berbagai sumber/inang di Indonesia berdasarkan daerah ITS rDNA dengan metode
maximum likelihood. Percabangan dengan nilai bootstrap (BS) ≥ 50% dari analisis yang ditampilkan. Huruf cetak tebal
Gambar 1 Pohon filogenetik galur Fusarium asal berbagai sumber/inang di Indonesia berdasarkan gen EF-1α dengan metode Maximum Likelihood. Percabangan dengan nilai bootstrap (BS) ≥ 50% dari analisis yang ditampilkan. Huruf cetak tebal menunjukkan spesimen yang diuji.
Pembahasan
Indonesia baru pertama kali dilakukan. Pada studi ini, kekerabatan dievaluasi dengan menggunakan sekuen DNA dari daerah ITS rDNA dan gen EF-1α. Pada penelitian ini, analisis filogenetik ITS rDNA menghasilkan lima cluster yang monofiletik, yaitu F. decemcellulare species complex (FDSC), F. graminearum species complex (FGSC), F. oxysporum species complex (FOSC), F. solani species complex (FSSC), dan F. tricinctum species complex (FTSC) serta F. fujikuroi species complex (FFSC) yang polifiletik.
Pembagian Fusarium spesies ke dalam sistem species complex berubah-ubah berdasarkan analisis marka yang digunakan dalam analisis kekerabatan. Menurut Watanabe et al. (2011) FDSC terdiri dari satu spesies yaitu F. decemcellulare, sehingga pada penelitian ini hanya sekuen rujukan F. decemcellulare yang digunakan untuk identifikasi spesies. FDSC terdiri dari dua spesies yaitu F. decemcellulare dan F. albosuccineum. Analisis filogenetik FDSC dilakukan dengan menggunakan gen RPB1 dan RPB2 (O’Donnell et al. 2013). Berdasarkan informasi ini, F. decemcellulare koleksi IPBCC perlu dievaluasi kembali identitasnya.
FGSC terdiri dari F. culmorum, F. graminearum, F. poae, F. sambucinum, dan F. sporotrichioides. Analisis filogenetik FGSC dilakukan berdasarkan LSU rDNA (Schroers et al. 2009). Namun, pada publikasi Aoki et al. (2014) FGSC terdiri dari F. graminearum dan F. asiaticum, sedangkan F. cerealis, F. culmorum, F. poae, F. pseudograminearum, F. sambucinum, dan F. sporotrichioides termasuk ke dalam F. sambucinum species complex. Pada penelitian ini sekuen rujukan yang digunakan terbatas pada F. acaciae-mearnsii,
F. acuminatum, F. asiaticum, F. boothii, F. cerealis, F. graminearum, F. kyuhuense, F. langsethiae, F. poae, dan F. sporotrichioides.
FOSC berdasarkan analisis filogenetik gen EF-1α, β-tubulin, mtSSU rDNA (Schroers et al. 2004), RPB1 dan RPB2 (O’Donnell et al. 2013) menunjukkan bahwa FOSC hanya terdiri dari F. oxysporum. Pada penelitian ini hanya sekuen rujukan F. oxysporum yang digunakan untuk mengidentifikasi spesies.
FSSC terdiri dari F. falciforme dan F. striatum. Analisis filogenetik pohon kombinasi FSSC dilakukan berdasarkan gen EF-1α dan RPB2 (O’Donnell et al.
2012). Berdasarkan analisis filogenetik kombinasi dari gen RPB1 dan RPB2, FSSC terdiri dari F. ambrosium, F. falciforme, F. illudens, F. neocosmosporiellum, F. phaseoli, F. plagianthi, F. solani, dan F. virguliforme
(O’Donnell et al. 2013). Fusarium brasiliense, F. cuneirostrum, F. lichenicola, dan Neocosmospora vasinfecta, termasuk ke dalam FSSC berdasarkan analisis filogenetik gen EF-1α, rRNA, dan RPB2 (O’Donnell et al. 2008). Pada penelitian ini digunakan sekuen rujukan dari F. ambrosium, F. brasiliense, F. cuneirostrum,
F. falciforme, F. illudens, F. lichenicola, F. solani, F. tucumaniae, dan F. virguliforme.
Menurut Aoki et al. (2014) FTSC terdiri dari F. acuminatum, F. avenaceum, dan F. tricinctum. Berdasarkan analisis filogenetik FTSC menggunakan gen RPB1
dan RPB2 menunjukan bahwa FTSC terdiri dari F. avenaceum, F. flocciferum, F. torulosum, dan F. tricinctum(O’Donnell et al. 2013). Pada analisis filogenetik ini digunakan sekuen rujukan F. tricinctum.
filogenetik kombinasi 28S rDNA, mtSSU rDNA, dan β-tubulin dari FFSC menunjukkan bahwa FFSC terdiri dari F. acutatum, F. annulatum, F. anthophilum, F. bactridioides, F. begoniae, F. bravicetanulatum, F. bulbicola, F. circinatum, F. concentricum, F. denticulatum, F. dlamini, F. fujikuroi, F. globosum, F. guttiforme, F. lactis, F. napiforme, F. neoceras, F. nygamai, F. phyllophilum, F. proliferatum, F. pseudoanthophilum, F. pseudocircinatum, F. pseudonygamai, F. ramigenum, F. sacchari, F. subglutinans, F. succisae, F. thapsinum, F. udum, dan F. verticillioides (O’Donnell et al. 1998). Pada penelitian ini digunakan sekuen rujukan dari F. annulatum, F. bactridioides, F. concentricum, F. fujikuroi, F. guttiforme, F. napiforme, F. nygamai, F. phyllophilum, F. pseudonygamai, F. proliferatum, dan F. verticillioides.
ITS rDNA dapat digunakan untuk mengidentifikasi Fusarium sampai dengan tingkat spesies, yaitu F. cerealis, F. decemcellulare, F. oxysporum, dan F. solani. Pengklasifikasian galur-galur Fusarium ke dalam species complex dengan menggunakan sekuen ITS rDNA memiliki beberapa kelebihan, yaitu pohon filogenetik yang lebih sederhana, efisien, biaya lebih murah, dan efektif (O’Donnell et al. 2008). Daerah ITS rDNA dapat digunakan untuk memisahkan spesies-spesies yang berkerabat dekat seperti F. verticillioides dan F. proliferatum
(Visentin et al. 2009). Namun, daerah ITS rDNA tidak mampu memisahkan anggota-anggota dari F. fujikuroi species complex (FFSC), seperti F. verticillioides dengan F. subglutinans (O’Donnell & Cigelnik 1999) serta F. fujikuroi dengan F. proliferatum (Waalwijk et al. 1996). Daerah ITS rDNA memiliki variasi nukleotida yang rendah dan kurang informatif untuk mendefinisikan beberapa spesies complex dalam Fusarium (Oechsler et al. 2009, Wang et al. 2011). Oleh karena itu, diperlukan kemampuan interpretasi yang baik terhadap analisis pohon filogenetik yang dirancang dengan menggunakan sekuen DNA dari daerah ITS (Cheng et al. 2008, O’Donnell et al. 2008).
Studi ini menunjukkan bahwa ITS rDNA hanya dapat digunakan untuk mengklasifikasikan galur-galur Fusarium ke dalam species complex. Studi ini juga menunjukkan bahwa galur-galur Fusarium yang diisolasi dari berbagai inang berada pada satu species complex yang sama. Hal ini menunjukkan tidak adanya
host-specificity anggota Fusarium pada tingkat species complex atau spesies. Analisis filogenetik juga menunjukkan bahwa satu Fusarium species complex
dimungkinkan memiliki anggota dengan strategi hidup yang berbeda-beda baik sebagai endofit, patogen, saprob atau terbawa benih. Pada studi ini tidak ditemukan galur Fusarium dengan strategi hidup sebagai patogen pada hewan dan manusia, padahal sebelumnya dilaporkan bahwa F. coccophilumspecies complex
yang ditemukan di Indonesia hidup berasosiasi dengan serangga (O’Donnell et al. 2012).
Galur yang berada dalam cluster yang sama (pada pohon ITSnya) dengan galur yang teridentifikasi ke dalam spesies berdasarkan sekuen EFnya dapat diberi nama yang sama. Namun demikian konfirmasi nama tersebut masih diperlukan dengan menggunakan gen EF-1α. Gen EF-1α memiliki tingkat similaritas tinggi dengan spesies Fusarium pada database Fusarium-ID dan analisis pohon filogenetik EF-1α mampu menguraikan beberapa anggota pada semua clade
informatif pada tingkat spesies dari Fusarium dan universal untuk mempelajari filogeni genus tersebut. O’Donnell et al. (2012) menyatakan bahwa gen EF-1α
dapat digunakan untuk mempelajari konsep spesies pada Fusarium.
Marka molekuler untuk identifikasi Fusarium sampai dengan tingkat spesies masih diperdebatkan. Pada penelitian ini gen EF-1α digunakan sebagai marka untuk identifikasi sampai dengan tingkat spesies. O’Donnell et al. (2010) merekomendasikan gen EF-1α, RPB1, dan RPB2 untuk mengidentifikasi spesies
Fusarium. Evaluasi beberapa marka molekuler oleh Wang et al. (2011) mengindikasikan bahwa ITS rDNA, mtSSU rDNA, dan LSU rRNA merupakan gen yang kurang informatif untuk analisis molekuler filogenetik. Wang et al. (2011) merekomendasikan IGS sebagai lokus yang paling informatif dan mampu memisahkan galur-galur Fusarium dengan baik dibandingkan gen RPB2.
4
SIMPULAN DAN SARAN
Simpulan
Dalam penelitian ini, daerah ITS rDNA dapat digunakan untuk mengelompokkan galur-galur Fusarium ke dalam lima species complex yang monofiletik, yaitu FDSC, FGSC, FOSC, FSSC, dan FTSC. FFSC adalah species complex yang polifiletik. Berdasarkan analisis filogenetik gen EF-1α dan asumsi kesamaan clade ITS dan EF-1α, 34 galur Fusarium dapat diidentifikasi, yaitu 1 galur F. ambrosium, 1 galur F. cerealis, 1 galur F. concentricum, 5 galur F. decemcellulare, 16 galur F. oxysporum, 9 galur F. solani, dan 1 galur F. verticillioides. Galur-galur yang berkerabat memiliki fleksibilitas strategi hidup.
Saran
Analisis molekuler galur-galur Fusarium perlu dilakukan dengan menggunakan marka molekuler lainnya yaitu EF-1α, IGS, RPB1, dan RPB2.
DAFTAR PUSTAKA
Aoki T, O’Donnell K, Geiser DM. 2014. Systematic of key phytopathogenic
Fusarium species: current status and future challenges. J Gen Plant Pathol
80:189-201. Doi: 10.1007/s10327-014-0509-3.
Baayen RP, O’Donnell K, Breeuwsma S, Geiser DM, Waalwijk C. 2001. Molecular relationships of fungi within the Fusarium redolens-F. hostae
clade. Phytopathology 91: 1031-1044.
Bruns TD, White TJ, Taylor JW. 1991. Fungal molecular systematic. Annu Rev Ecol Syst 22: 525-564.
Cheng Z, Tang W, Su Z, Cai Y, Sun S, Chen Q, Wang F, Lin Y, She Z, Vrijmoed LLP. 2008. Identification of mangrove endophytic fungus 1403 (Fusarium
proliferatum) based on morphological and molecular evidence. J For Res
19: 219-224. Doi: 10.1007/s11676-008-0030-7.
Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783-791.
Geiser DM, Jime´nez-Gasco MM, Kang S, Makalowska I, Veeraraghavan N, Ward TJ, Zhang N, Kuldau GA, O’Donnell K. 2004. FUSARIUM-ID v. 1.0: A DNA sequence database for identifying Fusarium. Eur J Plant Pathol 110: 473-479.
Guadet J, Julien J, Lafay JF, Brygoo Y. 1989. Phylogeny of some Fusarium
species, as determined by large-subunit rRNA sequence comparison. Mol Biol Evol 6: 227-242.
Hove FV, Waalwijk C, Logrieco A, Munaut F, Moretti A. 2011. Gibberella musae (Fusarium musae) sp. nov., a recently discovered species from banana is sister to F. verticillioides. Mycologia 103: 570-585. Doi: 10.3852/10-038.
[IPBCC] Institut Pertanian Bogor Culture Collection. 2010. Katalog Koleksi Biakan Mikroorganisme Institut Pertanian Bogor. Bogor (ID): IPBCC. Jumjunidang, Riska, Soemargono. 2012. Identification and distribution of
Fusarium oxysporum f. sp. cubense galures through analysis of vegetative compatibility group in Lampung Province, Indonesia. ARPN J Agric Biol Sci 7: 279-284.
Nugroho PA, Setyabudi FMCS, Saleh B, Rahayu ES. 2013. Fumonisin-Producing
Fusarium from Maize Grains in Tretep, Indonesia. J Food Sci and Engin 3: 534-540.
Nurbaya, Kuswinanti T, Rosmana A, Baharuddin, Millang S. 2014. Growth rate and identification of Fusarium spp. associated with Aquillaria spp. from Nunukan regency, North Kalimantan. Int J Curr Res Aca Rev 2: 33-40. O’Donnell K, Cigelnik E. 1999. Two divergent intragenomic rDNA ITS2 types
within a monophyletic lineage of the fungus Fusarium are nonorthologous.
Mol Phylo Evol 1:1-14.
O’Donnell et al. 2010. Internet-accessible DNA sequence database for identifying fusaria from human and animal infections. J Clin Microbiol 48: 3708-3718. O’Donnell K, Humber RA, Geiser DM, Kang S, Crous PW, Johnston PR, Aoki T,
Rooney AP, Rehner SA. 2012. Phylogenetic diversity of insecticolous fusaria inferred from multilocus DNA sequence data and their molecular identification via FUSARIUM-ID and Fusarium MLST. Mycologia 104: 427-445.
O’Donnell K, Kistler HC, Cigelnik E, Ploetz RC. 1998. Multiple evolutionary origins of the fungus causing Panama disease of banana: Concordant evidence from nuclear and mitochondrial gene genealogies. Proc Natl Acad Sci USA 95: 2044-2049.
O’Donnell K et al. 2013. Phylogenetic analyses of RPB1 and RPB2 support a middle cretaceous origin for a clade comprising all agriculturally and medically important fusaria. Fungal Genet Biol 52: 20- 31.
O’Donnell K, Sutton DA, Fothergill A, McCarthy D, Rinaldi MG, Brandt ME, Zhang N, Geiser DM. 2008. Molecular phylogenetic diversity, multilocus haplotype nomenclature, and in vitro antifungal resistance within the
Oechsler RA, Feilmeier MR, Ledee DR, Miller D, Diaz MR, Fini ME, Fell JW, Alfonso EC. 2009. Utility of molecular sequence analysis of the ITS rRNA region for identification of Fusarium spp. from ocular sources. IOVS 50: 2230-2236. Doi: 10.1167/iovs.08-2757.
Pinaria AG, Liew ECY, Burgess LW. 2010. Fusarium species associated with vanilla stem rot in Indonesia. Australasian Plant Pathol 39: 176-183. Raeder U, Broda P. 1985. Rapid preparation of DNA from filamentous fungi.
Letters in Applied Microbiology 1: 17-20.
Schroers HJ, Baayen RP, Meffert JP, Gruyter J, Hooftman M, O’Donnell K. 2004.
Fusarium foetens, a new species pathogenic to begonia elatior hybrids (Begonia × hiemalis) and the sister taxon of the Fusarium oxysporum
species complex. Mycologia 96: 393-406.
Schroers HJ, O’Donnell KO, Lamprecht SC, Kammeyer PL, Johnson S, Sutton DA, Rinaldi MG, Geiser DM, Summerbell RC. 2009. Taxonomy and phylogeny of the Fusarium dimerum species group. Mycologia 101: 44-70. Doi: 10.3852/08-002.
Schoch CL et al. 2012. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi. Proceedings of the National from different phylogenetic analyses. BMC Bioinformatics 11: 7. Doi: 10.1186/1471-2105-11-7.
Summerbell RC, Schroers HJ. 2002. Analysis of phylogenetic relationship of
Cylindrocarpon lichenicola and Acremonium falciforme to the Fusarium solani species complex and a review of similarities in the spectrum of
Visentin I, Tamietti G, Valentino D, Portis E, Karlovsky P, Moretti A, Cardinale F. 2009 – The ITS region as a taxonomic discriminator between Fusarium verticillioides and Fusarium proliferatum. Mycol Res 113, 1137–1145. Doi: 10.1016/j.mycres.2009.07.011.
Waalwijk C, De Koning JRA, Baayen RP, Bayen RP, Gams W. 1996. Discordant grouping of Fusarium spp. from sections Elegans, Liseola, Dlaminia based on ribosomal ITS1 and ITS2 sequences. Mycologia 88: 361-368.
Wang H, Xiao M, Kong F, Chen S, Dou HT, Sorrell T, Li RY, Xu YC. 2011. Accurate and practical identification of 20 Fusarium species by seven-locus sequence analysis and reverse line blot hybridization, and an in vitro antifungal susceptibility study. J Clin Microbiol 49: 1890-1898. Doi: 10.1128/JCM.02415-10.
Watanabe M, Yonezawa T, Lee KI, Kumagai S, Konishi YS, Goto K, Kudo YH. 2011. Molecular phylogeny of the higher and lower taxonomy of the
Fusarium genus and differences in the evolutionary histories of multiple genes. BMC Evol Biol 11: 1-16.
White TJ, Bruns T, Lee S, Taylor J. 1990. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. Di dalam: Innis MA, Gelfand DH, Sninsky JJ, White TJ, editor. PCR Protocols: A Guide to Methods and Applications. New York (US): Academic Pr. hlm 315–322. Zhao ZH, Lu G. 2008. Fusarium sinensis sp. nov., a new species from wheat in
Lampiran 1 Daftar sekuen rujukan ITS galur Fusarium yang digunakan pada
24 FGSC F. sporotrichioides MAFF 236639 33 FOSC F. oxysporum CBS 133023 KF255448 Type
Lampiran 1 Daftar sekuen rujukan ITS galur Fusarium yang digunakan pada penelitian (lanjutan...)
No. ID galur Fusarium yang digunakan pada analisis filogenetik: ATCC:
American Type Culture Collection, Manassas, Virginia; MAFF: Ministry of Agriculture and Forestry, NIAS, Tsukuba, Japan; NRRL: Northern Regional Research Laboratory, NCAUR Peoria, Illinois; CBS-KNAW: Centraalbureau voor Schimmelcultures—Fungal Biodiversity Center, Utrecht, Belanda; FRC:
Fusarium Research Center, Pennsylvania State University, Pennsylvania; PUF: No. ID galur Fusarium dari Beijing Tongren Hospital, Beijing, dan Shandong Eye Institute, Qingdao, provinsi Shandong, Cina.
No. Species
complex
Nama pada database
GenBank, NCBI
No. ID galur No. aksesi
GenBank Keterangan 34 FSSC F. ambrosium NRRL 20438 AF178397 -
35 FSSC F. falciforme NRRL 32757 DQ094536 - 36 FSSC F. lichenicola NRRL 32434 DQ094444 - 37 FSSC F. solani NRRL 28579 DQ094383 Type
38 FSSC F. solani MAFF 239038 AB587014 - 39 FSSC F. illudens NRRL 22090 AF178393 -
40 FSSC F. tucumaniae NRRL 31096 EF408523 -
41 FSSC F. brasiliense NRRL 22743 EF408512 -
42 FSSC F. cuneirostrum NRRL 31104 EF408518 -
43 FSSC F. virguliforme NRRL 22825 EF408542 -
44 FTSC F. lateritium MAFF 235344 AB587004 -
45 FTSC F. avenaceum PUF034 HQ165937 -
46 FTSC F. tricinctum CBS 393.93 AB587029 -
47 FTSC F. tricinctum MAFF 235551 AB587030 -
48 - H. lixii CBS 226.95 AF057606 Outgroup
Lampiran 2 Daftar sekuen rujukan EF-1α galur Fusarium yang digunakan pada penelitian (lanjutan...)
No. Species
complex
Nama pada
database GenBank, NCBI
No. ID galur
No. aksesi
GenBank Keterangan
37 FSSC F. illudens NRRL
22090
AF178326 -
38 FTSC F. tricinctum CBS 393.93 AB674263 Outgroup
No. ID galur Fusarium yang digunakan pada analisis filogenetik: ATCC:
American Type Culture Collection, Manassas, Virginia; MAFF: Ministry of Agriculture and Forestry, NIAS, Tsukuba, Japan; NRRL: Northern Regional Research Laboratory, NCAUR Peoria, Illinois; CBS-KNAW: Centraalbureau voor Schimmelcultures—Fungal Biodiversity Center, Utrecht, Belanda; PUF: No. ID galur Fusarium dari Beijing Tongren Hospital, Beijing, dan Shandong Eye Institute, Qingdao, provinsi Shandong, Cina.
RIWAYAT HIDUP
Penulis dilahirkan di kebumen pada tanggal 30 Agustus 1991. Penulis merupakan putri pertama dari Bapak Waluyo dan Ibu Rotingah. Penulis menyelesaikan pendidikan sekolah dasar pada tahun 2003 di SD Negeri 3 Panjer, sekolah menengah pertama di SMP Negeri 1 Kebumen pada tahun 2006, dan sekolah menengah atas di SMA Negeri 2 Kebumen pada tahun 2009.
Penulis menyelesaikan pendidikan program sarjana di Departemen Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Institut Pertanian Bogor pada tahun 2012. Pada tahun 2012, penulis melanjutkan pendidikan pascasarjana melalui beasiswa Fresh Graduate Direktorat Jenderal Pendidikan Tinggi (DIKTI). Penulis menyelesaikan pendidikan program pascasarjana di program studi Mikrobiologi, Institut Pertanian Bogor pada tahun 2015.
Hasil dari penelitian ini telah dipresentasikan pada seminar ―5th International Conference on Green Technology‖ pada tanggal 7-8 November 2014 di Malang. Hasil dari penelitian ini sedang dalam proses publikasi pada jurnal Plant Pathology & Quarantine dengan judul ―Molecular Phylogenetic Analysis of Indonesian Fusarium isolates from different lifestyle based on ITS rDNA sequence‖.