ISOLASI DAN PENGKLONAN FRAGMEN cDNA
GEN PENYANDI PROTEIN STEROID NUCLEAR RECEPTOR
DARI Melastoma malabathricum L.
AGUSTINA SENJAYANI
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2011
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI
Dengan ini saya menyatakan bahwa tesis Isolasi dan Pengklonan Fragmen cDNA Gen Penyandi Protein Steroid Nuclear Receptor dari Melastoma
malabathricum L. adalah karya saya dan pembimbing dan belum diajukan dalam
bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Bogor, Juli 2011
Agustina Senjayani
ABSTRACT
AGUSTINA SENJAYANI. Isolation and Cloning of cDNA Fragment of Gene Encoding for Steroid Nuclear Receptor Protein from Melastoma malabathricum L. Under the direction of SUHARSONO and MIFTAHUDIN.
We have isolated a cDNA fragment of gene encoding for Steroid Nuclear Receptor protein from Melastoma malabathricum L. (MmFSNR). Started by successfully total RNA isolation, we then generated total cDNA using total RNA as template by reverse transcription. The fragment of MmFSNR (700bp) had been isolated by PCR using total cDNA as template and specific primers snrF and
snrR. The MmFSNR fragment was succesfully inserted into pGEM®–T Easy
plasmid, then the recombinant plasmid was introduced into Eschericia coli DH5α. Sequencing of MmFSNR fragment using ABI Prism 310 Automated DNA sequencer resulted sequence of 700bp of MmFSNR fragment. The MmFSNR fragment does not contain EcoR1 restriction site founded in the multi cloning site of pGEM®–T Easy.
Alignment analysis of nucleotide using BLASTN showed that the
MmFSNR fragment is 65% identical to cDNA fragment encoding for
uncharacterized membran protein of Arabidopsis thaliana (At3g27390), identical to (GN : At3g27390; Uniprot :Y3739_ARATH). Alignment analysis of deduced amino acid showed that MmFSNR protein is 53% identical to At3g27390 protein and 47% identical to SNR protein of Medicago truncantula. At3g27390 is a protein induced by flagellin elicited by pathogen and SNR is protein related to xenobiotic sensor.
Keywords : gene cloning, cDNA, steroid nuclear receptor, Melastoma
RINGKASAN
AGUSTINA SENJAYANI. Isolasi dan Pengklonan Fragmen cDNA Gen Penyandi Protein Steroid Nuclear Receptor dari Melastoma malabathricum L. Dibimbing oleh SUHARSONO DAN MIFTAHUDIN.
Respon tanaman terhadap senyawa xenobiotik baik yang berasal dari biotik maupun abiotik melibatkan sejumlah mekanisme pertahanan yang spesifik dan terkoordinasi. Inisiasi respon selulernya dilakukan oleh reseptor xenobiotik yang berperan mendeteksi senyawa dan menginduksi beragam aktivitas enzimatik dan transporter untuk mendetoksifikasi dan menghilangkan senyawa asing.
Steroid nuclear receptor, adalah bagian dari kelas reseptor inti (Nuclear receptors/NRs). NR adalah faktor transkripsi yang diaktivasi oleh ligan yang
mengatur ekspresi gen melalui interaksi dengan sekuen DNA hulu dari gen-gen targetnya dan merupakan sensor xenobiotik. Protein ini juga mempunyai fungsi sebagai sensor pertahanan terhadap patogen dalam sistem imunitas tanaman.
Fragmen cDNA dari gen penyandi protein Steroid Nuclear Receptor dari
Melastoma malabathricum. L (MmFSNR) telah berhasil diisolasi dan diklon di
dalam pGEM®–T Easy. Isolasi dan pengklonan MmFSNR dimulai dengan RNA total. RNA total telah berhasil diisolasi. cDNA total telah berhasil disintesis dengan menggunakan RNA total sebagai cetakan melalui proses transkripsi balik. Fragmen cDNA MmFSNR berukuran 700 pb telah berhasil diisolasi melalui PCR dengan cDNA total sebagai cetakan dan primer spesifik snrF dan snrR. Fragmen
MmFSNR ini kemudian diinsersikan kedalam plasmid pGEM®–T Easy,
selanjutnya plasmid rekombinan ini diintroduksikan ke dalam Eschericia coli DH5α. Pengurutan DNA fragmen MmFSNR dengan menggunakan ABI Prism 310
Automated DNA Sequencer menghasilkan urutan fragmen cDNA MmFSNR
sebesar 700 pb. Fragmen ini mengandung beberapa situs enzim restriksi tetapi tidak mengandung situs pemotongan EcoR1 yang terdapat di dalam situs ganda pengklonan dari pGEM®–T Easy.
Analisis kesejajaran nukleotida menggunakan program BLASTN menunjukkan bahwa fragmen MmFSNR mempunyai kemiripan 65% dengan mRNA penyandi protein membran uncharacterized dari Arabidopsis thaliana L. (At3g27390). Analisis penyejajaran deduksi asam amino dari fragmen MmFSNR menggunakan program BLASTP menunjukkan bahwa protein deduksi dari cDNA MmFSNR mempunyai kemiripan 53% dengan protein At3g27390 dan 47% dengan SNR dari Medicago truncantula. At3g27390 adalah protein yang diinduksi oleh elisitor flagelin yang dihasilkan oleh patogen dan SNR adalah sensor xenobiotik, sehingga MmFSNR kemungkinan mempunyai peranan dalam detoksifikasi Al yang dapat menjadi senyawa xenobotik.
Kata kunci : pengklonan gen, cDNA, steroid nuclear receptor, Melastoma
©
Hak Cipta milik IPB, Tahun 2011
Hak Cipta dilindungi Undang-Undang
1. Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumber.
a. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah.
b. Pengutipan tidak merugikan kepentingan yang wajar bagi IPB. 2. Dilarang mengumumkan dan memperbanyak sebagian atau seluruh Karya
ISOLASI DAN PENGKLONAN FRAGMEN cDNA
GEN PENYANDI PROTEIN STEROID NUCLEAR RECEPTOR
DARI Melastoma malabathricum L.
AGUSTINA SENJAYANI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains pada
Program Studi Bioteknologi
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2011
PRAKATA
Bismillaahirrahmaanirrahiim.
Segala puji hanya milik Allah SWT yang telah melimpahkan rahmat dan karunia-Nya sehingga karya ilmiah yang berjudul Isolasi dan Pengklonan
Fragmen cDNA Gen Penyandi Protein Steroid Nuclear Receptor dari
Melastoma malabathricum L. akhirnya dapat diselesaikan. Penelitian ini dibiayai oleh Proyek Hibah Penelitian Tim Pascasarjana Angkatan III dengan judul ”Isolasi dan karakterisasi gen-gen yang berhubungan dengan toleransi tanaman terhadap pH rendah dan aluminium tinggi” atas nama Prof. Dr. Ir. Suharsono, DEA sebagai peneliti utama.
Selama proses penulisan karya ilmiah ini, penulis mendapat bantuan berbagai pihak baik secara langsung maupun tidak langsung. Untuk itu penulis bermaksud menyampaikan rasa terima kasih kepada Prof. Dr. Ir. Suharsono, DEA dan Dr. Ir. Miftahudin, M.Si selaku Komisi Pembimbing atas segala bimbingan dan arahannya serta Dr. Aris Tjahjoleksono, DEA selaku Penguji Luar Komisi atas berbagai saran dan masukannya. Terima kasih juga Penulis ucapkan kepada Dr. Ir. Utut Widyastuti dan Dr. Ir. M. Jusuf (alm) atas seluruh bimbingannya. Terimakasih disampaikan pada Pusat Penelitian Sumberdaya Hayati dan Bioteknologi IPB yang telah menyediakan sarana dan prasarana untuk penelitian.
Terimakasih untuk Ayahanda, Ibunda, adinda Aris dan Anis serta seluruh keluarga atas semua do’a, pengorbanan dan kasih sayangnya. Terimakasih juga untuk Kak Hanum, Firdaus, Zendi, Wiwid, Pak Mul, Mbak Pepy, Pak Muzuni, seluruh keluarga besar Laboratorium BIORIN, mami Nanik, adinda Yeni, Kak Rina, WA-1 dan Kutilang family, serta semua pihak yang telah banyak membantu dan tidak dapat disebutkan satu per satu.
Semoga karya ilmiah ini dapat memberikan manfaat.
Bogor, Juli 2011
RIWAYAT HIDUP
Penulis dilahirkan di Malang pada tanggal 3 Agustus 1978 dari ayah Mulyadi Tampi dan ibu Sismani. Penulis merupakan putri pertama dari tiga bersaudara.
Pendidikan sarjana ditempuh di Program Studi Ilmu dan Teknologi Benih, Jurusan Budi Daya Pertanian, Fakultas Pertanian IPB, lulus pada tahun 2001. Pada tahun 2003, penulis diterima di Program Studi Bioteknologi pada Program Pascasarjana IPB.
Penulis pernah bekerja di Laboratorium Genetika Molekular Tumbuhan
Cibinong Science Center Lembaga Ilmu Pengetahuan Indonesia (LIPI) Bogor
DAFTAR ISI
Halaman DAFTAR TABEL ... iv DAFTAR GAMBAR ... v DAFTAR LAMPIRAN ... vi PENDAHULUAN Latar Belakang ... 1 Tujuan Penelitian ... 2 TINJAUAN PUSTAKA Pertahanan terhadap cekaman xenobiotik ... 3Steroid Nuclear Receptor (SNR) ... 8
Toleransi Melastoma malabathricum L. terhadap xenobiotik ... 11
BAHAN DAN METODE Waktu dan Tempat Penelitian ... 13
Bahan ... 13
Metode Penelitian ... 13
Isolasi RNA total ... 14
Sintesis cDNA total ... 14
Isolasi Fragmen cDNA MmFSNR melalui PCR ... 15
Pengklonan cDNA ke dalam vektor pGEM®-T Easy ... 15
Transformasi genetik bakteri E. coli DH5α dengan vektor rekombinan ... 16
Analisis cDNA sisipan ... 17
Analisis urutan nukleotida MmFSNR ... 17
HASIL DAN PEMBAHASAN Isolasi RNA Total ... 18
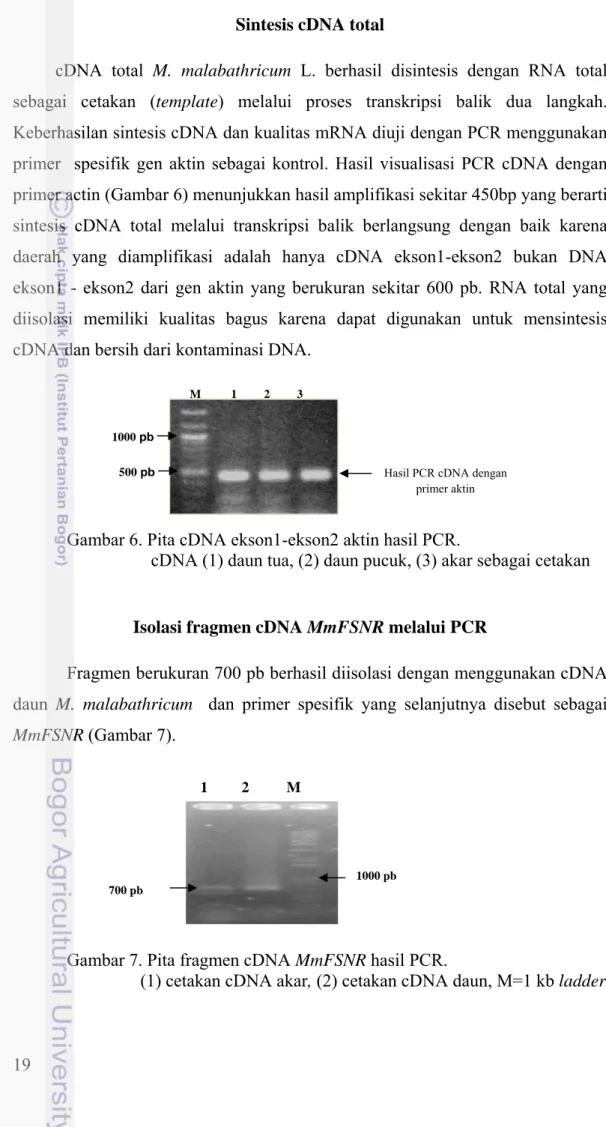
Sintesis cDNA Total ... 19
Isolasi Fragmen cDNA MmFSNR Melalui PCR ... 19
Pengklonan Fragmen MmFSNR ke dalam Plasmid pGEM®-T Easy ... 20
Analisis Fragmen MmFSNR ... 21
SIMPULAN DAN SARAN Kesimpulan ... 32
Saran ... 32
DAFTAR PUSTAKA ... 33 LAMPIRAN
DAFTAR TABEL
Halaman
1. Data hasil isolasi RNA total ... 18 2. Hasil analisis kemiripan sekuen nukleotida MmFSNR dengan program
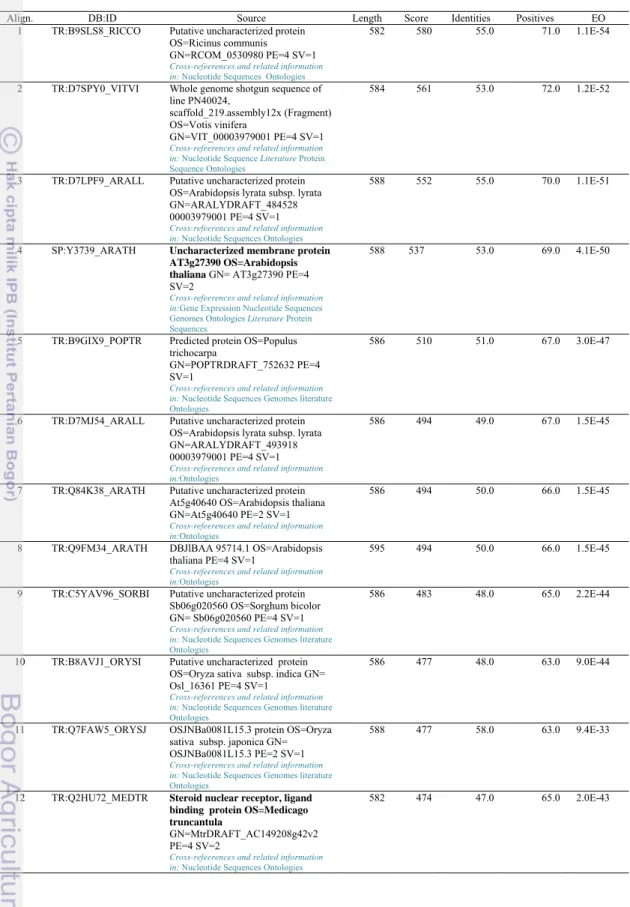
BLASTN (http://www.ebi.ac.uk/BLASTN) ... 22 3. Hasil analisis kemiripan sekuen nukleotida MmFSNRcut dengan
program BLASTN (http://www.ebi.ac.uk/BLASTN) ... 24 4. Hasil analisis kemiripan sekuen deduksi asam amino MmFSNR
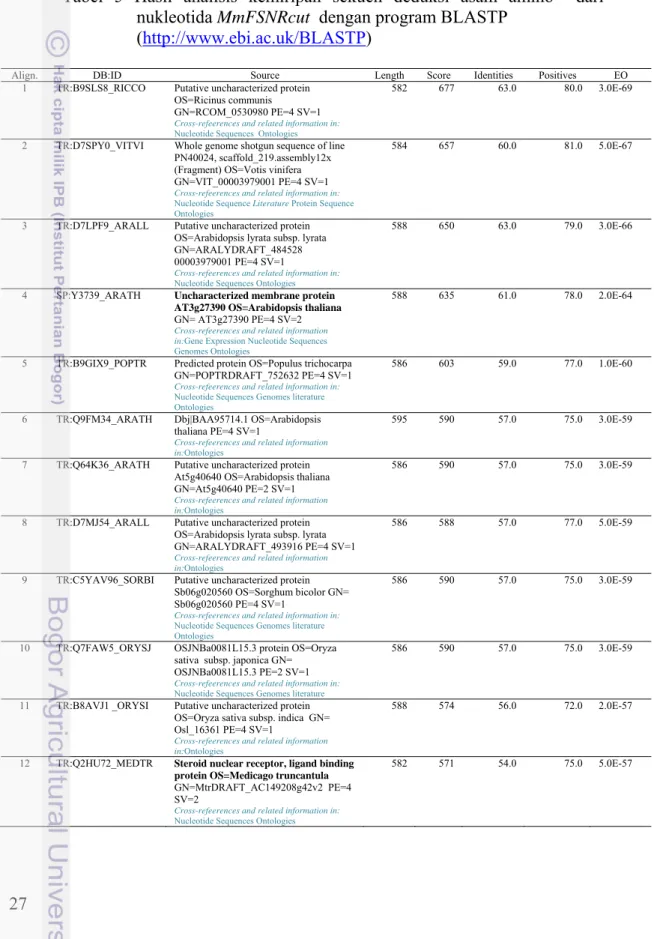
dengan program BLASTP (http://www.ebi.ac.uk/BLASTP) ... 26 5. Hasil analisis kemiripan sekuen deduksi asam amino nukleotida
MmFSNRcut dengan program BLASTP
DAFTAR GAMBAR
Halaman
1. Proses umum metabolisme senyawa xenobiotik ... 3
2. Mekanisme pertahanan tanaman terhadap patogen... 7
3. Melastoma malabathricum L. ... 11
4. Struktur kimia malabathrin yang merupakan metabolit sekunder yang dihasilkan oleh Melastoma malabathricum L ... 12
5. Hasil elektroforesis RNA total ... 18
6. Pita cDNA ekson1-ekson2 aktin hasil PCR ... 19
7. Pita Fragmen cDNA MmFSNR hasil PCR ... 19
8. Hasil transformasi E. coli DH5α dan PCR terhadap koloni putih ... 20
9. Hasil pengurutan DNA sisipan (MmFSNR) pada pGEM®–T Easy menggunakan primer T7 ... 21
10. Peta restriksi MmFSNR ... 22
11. Hasil analisis kesejajaran ganda nukleotida MmFSNR dengan nukleotida SNR pada padi dan M. truncantula ... 23
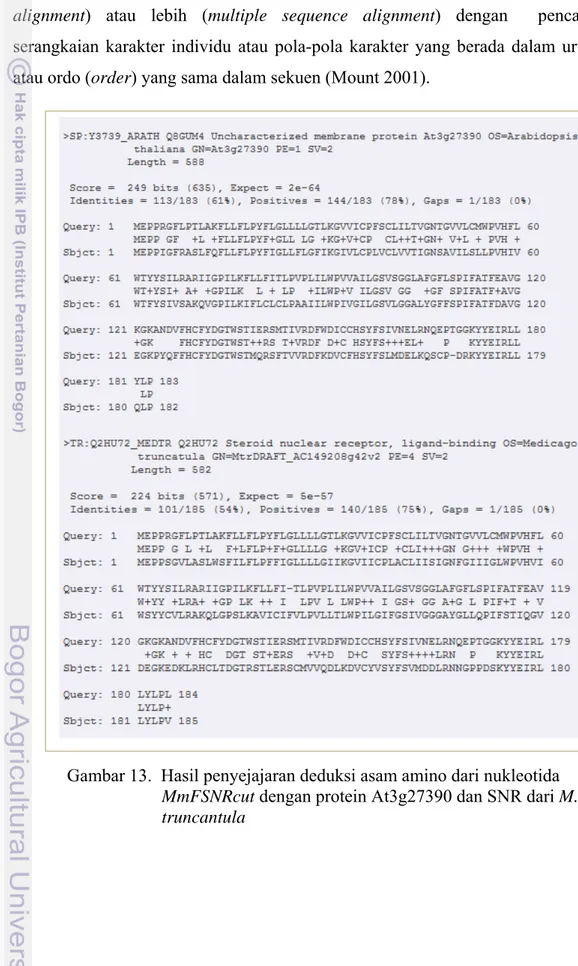
12. Deduksi asam amino dari urutan nukleotida fragmen cDNA MmFSNR 25 13. Hasil penyejajaran deduksi asam amino dari nukleotida MmFSNRcut dengan protein At3g27390 dan SNR dari M. truncantula ... 28
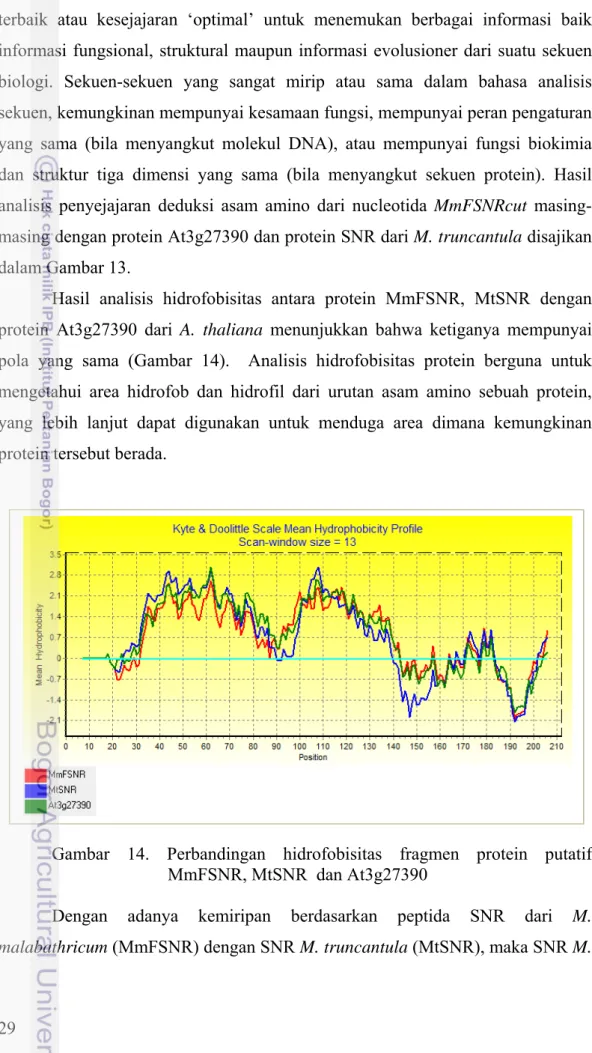
14. Perbandingan hidrofobisitas fragmen protein putatif MmFSNR, MtSNR dan At3g27390 ... 29
DAFTAR LAMPIRAN
Halaman
1. Data elektroferogram hasil pengurutan DNA sisipan MmFSNR dengan primer sp6 ... 38 2. Data elektroferogram hasil pengurutan DNA sisipan MmFSNR dengan
1
PENDAHULUAN
Latar Belakang
Sejumlah organisme mempunyai mekanisme pertahanan terhadap cekaman xenobiotik baik yang berasal dari biotik maupun abiotik. Mekanisme pertahanan terhadap cekaman xenobiotik ini melibatkan kerja berbagai gen-gen pertahanan. Melastoma malabathricum L. merupakan tumbuhan yang dikenal sebagai gulma invasif di lahan asam berkadar Aluminium (Al) tinggi. M.
malabathricum L. mampu mengakumulasi Al pada daun muda, daun dewasa,
daun tua dan akarnya masing-masing sebesar 8000 part per million (ppm), 9200 ppm, 14400 ppm dan 10400 ppm tanpa menunjukkan gejala-gejala keracunan (Watanabe et al. 1998). Pertumbuhan dan penyerapan hara pada M.
malabathricum L. bahkan semakin meningkat dengan keberadaan Al (Osaki et al.
1997), padahal Al sangat beracun bagi tanaman.
M. malabathricum L. juga menghasilkan metabolit berbasis stachyurin,
kompleks tannin malabathrin A, E dan F (Yoshida et al. 2010). Metabolit ini adalah bagian dari tanin komplek (flavono-elagitanin), yang merupakan senyawa metabolit sekunder yang bersifat antioksidan, anti tumor dan anti bakteri (Yoshida
et al. 2000). M. malabathricum L. juga menghasilkan sejumlah senyawa metabolit
yang bersifat alelopatik seperti senyawa terpenoid, flovonoid dan senyawa fenolik (Faravani et al. 2008). Menurut Zhao et al. (2005), akumulasi metabolit sekunder sering terjadi pada tanaman yang terpapar oleh cekaman termasuk oleh berbagai elisitor atau molekul sinyal. Kemampuan mengakumulasi Al pada daun dan akar, serta menghasilkan berbagai senyawa metabolit anti bakteri dan zat alelopatik menunjukkan bahwa M. malabathricum L. memiliki mekanisme toleransi terhadap berbagai cekaman xenobiotik melalui sebuah sistem metabolisme detoksifikasi, sehingga tanaman ini berpotensi sebagai sumber gen-gen pertahanan terhadap cekaman xenobiotik.
Sandermann (1992) mengemukakan konsep green liver untuk menjelaskan bagaimana tanaman dapat bertindak sebagai penampungan umum berbagai senyawa berbahaya melalui metabolisme xenobiotik yang mirip dengan yang terjadi pada hati (liver). Proses tersebut melibatkan berbagai enzim yang bekerja
2
melalui tahapan transformasi, konjugasi dan kompartementasi. Respon seluler terhadap masuknya senyawa xenobiotik diinisiasi oleh keberadaan reseptor xenobiotik yang melakukan deteksi (sensor). Reseptor ini selanjutnya berperan menginduksi berbagai enzim dan transporter untuk mendetoksifikasi dan menghilangkan senyawa tersebut.
Steroid nuclear reseptor (SNR) adalah protein subkelas Nuclear reseptor
(NR). Protein NR merupakan faktor transkripsi yaitu protein yang meningkatkan atau menurunkan transkripsi gen-gen. Aktivitasnya secara spesifik adalah mengatur pengikatan RNA polimerase pada DNA. Protein NR bekerjasama dengan protein lain mengendalikan proses metabolisme, perkembangan dan homeostasis organisme melalui pengaturan ekspresi gen-gen spesifik (Germain et
al 2003). Menurut Gronemeyer (2004), NR membentuk sebuah superfamily
protein yang mengendalikan fungsi-fungsi terkait penyakit utama seperti diabetes, osteoporosis dan kanker. Menurut Morel et al. (2000), ketika berikatan dengan sebuah ligan senyawa xenobiotik, protein NR berperan krusial dalam induksi enzim-enzim detoksifikasi. Pada metazoan, Steroid Nuclear Receptor dikenal sebagai sensor xenobiotik dan menjadi master pengendali beberapa gen pertahanan terutama untuk drug resistance (Blumberg et al 1998). Pada tanaman SNR diduga mempunyai fungsi paralel dalam sensor pertahanan tehadap patogen yang mendeteksi molekul mikrobial dalam sistem imunitas (Shen and Shulze-lefert 2007).
Berdasarkan luasnya peran Steroid Nuclear Receptor pada aktivasi berbagai gen-gen pertahanan tersebut dan masih terbatasnya kajian pada tumbuhan, maka peran SNR dalam resistensi terhadap senyawa xenobiotik pada
M. malabathricum L. perlu diteliti.
Tujuan
Penelitian ini bertujuan untuk melakukan isolasi dan pengklonan fragmen cDNA penyandi protein Steroid Nuclear Receptor dari M. malabathricum L.
TINJAUAN PUSTAKA
Pertahanan tanaman terhadap cekaman xenobiotik
Menurut Encyclopedia Britannica (2011), xenobiotik dipahami sebagai
senyawa yang tidak secara alami dihasilkan oleh spesies biologi dan karenanya bersifat asing (xeno : asing; bios : kehidupan). Senyawa xenobiotik juga mengacu pada zat-zat kimiawi yang membahayakan atau berdampak racun ketika diakumulasi oleh sistem hidup. Sandermann (1992) mengemukakan konsep green
liver untuk menjelaskan bagaimana tanaman dapat bertindak sebagai
penampungan umum berbagai senyawa berbahaya melalui metabolisme xenobiotik yang mirip dengan yang terjadi pada hati (liver). Proses detoksifikasi tersebut melibatkan berbagai enzim yang bekerja melalui tahapan transformasi, konjugasi dan kompartementasi (Gambar 1). Tahapan transformasi terjadi melalui peningkatan polaritas senyawa xenobiotik biasanya oleh enzim semacam sitokrom P450 (fase I), tahapan konjugasi adalah pengikatan senyawa xenobiotik polar
dengan biomolekul tanaman seperti glukosa dan asam amino (fase II) sedangkan tahapan kompartementasi merupakan tahapan penimbunan senyawa xenobiotik (fase III).
Gambar 1. Proses umum metabolisme senyawa xenobiotik
Respon seluler terhadap masuknya senyawa xenobiotik diinisiasi oleh keberadaan reseptor xenobiotik yang melakukan deteksi (sensor). Reseptor ini selanjutnya berperan menginduksi berbagai enzim dan transporter yang berperan
4
mendetoksifikasi dan menghilangkan senyawa tersebut. Sistem sensor dan detoksifikasi berjalan manakala reseptor dan enzim-enzim yang melakukan metabolisme memiliki kesamaan spesifitas dan terjadi koordinasi induksi berbagai enzim oleh senyawa xenobiotik (Morel et al. 2000)
Toleransi Tanaman terhadap Aluminium
Aluminium terlarut merupakan salah satu senyawa xenobiotik yang menyebabkan toksisitas pada lahan asam. Meningkatnya kelarutan Al di dalam tanah asam menyebabkan terjadinya defisiensi nutrien seperti Mg, Ca dan P serta unsur mikro seperti Zn, Pb, dan Mo (Foy & Flemming 1978). Menurut Mossor-Pietraszewska (2001), Al menghambat proses pembelahan sel, sebab Al mengubah fisik-kimia dinding sel dan DNA serta menurunkan produksi dan translokasi sitokinin. Kelainan akar karena cekaman aluminium akan mengganggu proses absorbsi sejumlah unsur esensial seperti fosfor, kalsium, hara mikro seperti molibdenum, seng dan tembaga oleh rambut akar (Marschner 1995). Cekaman aluminium juga menyebabkan terhambatnya penyerapan NH4 dan NO3 serta
terganggunya aktivitas enzim nitrat reduktase (Cumming and Taylor 1990). Al mempengaruhi gen-gen yang berhubungan dengan pembelahan sel seperti heat
shock protein (hsp), protein histon H3, H4 dan S-adenosyl-L-homosystein (SHH).
Salah satu fungsi hsp adalah untuk pelipatan protein (protein folding) dan diekspresikan di berbagai tipe sel dalam kondisi tidak mendapat cekaman panas. Tingkat transkripsi H3 dan H4 menurun dengan berlanjutnya perlakuan Al baik pada varietas toleran maupun varietas peka. Hal ini menunjukkan bahwa Al menghambat peran histon dalam pembelahan sel (Richards and Gardner 1994).
Menurut Taylor (1991), pada prinsipnya terdapat dua mekanisme toleransi tanaman terhadap Al yaitu mekanisme eksternal dan mekanisme internal. Mekanisme eksternal dilakukan dengan mencegah Al masuk ke dalam simplas dan mencapai daerah metabolik yang peka. Hal ini dilakukan melalui eksklusi Al3+ toksik dari ujung akar dengan cara melepaskan ligan pengkelat Al3+ seperti asam-asam organik dan fosfat atau dengan melepaskan OH untuk meningkatkan pH eksternal. Tanaman juga melakukan melakukan imobilisasi Al pada dinding sel, mengatur permeabilitas membran, mengatur pH permukaan rizosfer, eksudasi
5
ligan pengkelat, eksudasi fosfat dan efluk Al. Metabolisme internal dilakukan dengan imobilisasi, kompartementasi dan detoksifikasi saat Al masuk ke dalam simplas. Hal ini dilakukan melalui pengikatan Al oleh protein tertentu, kompartementasi dalam vakuola, induksi dan peningkatan aktivitas enzim-enzim tertentu (Taylor 1991).
Beberapa spesies tanaman diketahui memiliki toleransi terhadap Al melalui eksudasi asam-asam organik lewat akar. Efluks malat dikeluarkan oleh wheat/Triticum aestivum (Delhaize et al. 1993; Basu et al. 1994; Pellet et al. 1996). Sitrat dikeluarkan oleh jagung, snapbean dan Casia tora (Miyasaka et al. 1991; Pellet et al. 1996; Ma et al. 1997) sedangkan oksalat dikeluarkan oleh
buckwheat dan taro (Ma and Miyasaka 1998; Zheng et al. 1998). Tanaman teh
dan hydrangea sebaliknya justru dikenal sebagai tanaman akumulator Al. Daun teh yang tua mampu mengakumulasi Al diatas 30.000 mg/kg berat kering (Matsumoto et al. 1976) dan daun tanaman hydrangea yang bunganya berkelopak biru dapat mengakumulasi Al diatas 3000 mg/kg (Ma et al. 1997). Tanaman M.
malabathricum L. dan Vaccinium macrocarpon mengakumulasi Al dalam
konsentrasi tinggi baik di akar maupun daunnya (Osaki et al. 1997).
Pertahanan Tanaman terhadap Patogen
Tanaman mempunyai sejumlah mekanisme pertahanan untuk menghadapi berbagai serangan dari lingkungan baik serangan serangga, nematoda, tumbuhan parasit, fungi, virus maupun bakteri. Mekanisme pertahanan tersebut dapat berupa kemampuan yang telah terbentuk (pre-formed) berwujud hambatan mekanik dan kimiawi yang bersifat konstitutif maupun sistem pertahanan yang bersifat
inducible (inducible defense system) (Montesano et al. 2003).
Sistem pertahanan inducible berpusat pada ketepatan persepsi sel terhadap patogen. Tanaman dalam hal ini, mampu mengenali senyawa yang dihasilkan patogen (elisitor) dan menggunakannya untuk memicu pensinyalan pertahanan (defence signalling). Mekanisme pertahanan inducible melibatkan dua tipe pertahanan berbeda yaitu tipe gene for gene interaction dan pertahanan dasar (basal defense). Tipe pertahanan gene for gene interaction dicirikan : (1)diinduksi oleh patogen spesifik; (2) berdasarkan pengenalan (recognition) produk gen Avr
6
yang spesifik dan sesuai (match) dengan produk gen R; (3) keragaman individu yang sangat tinggi akibat perubahan susunan gen (gene shuffling) meiosis; (4) reaksi cepat termasuk respon hipersensitivitas. Adapun pertahanan dasar (basal
defence) dicirikan antara lain : (1) diinduksi melalui infeksi oleh hampir semua
mikroba; (2) berdasarkan pengenalan general elicitor; (3) aktivasi pertahanan cepat tetapi umumnya tanpa respon hipersensitif (Boller et al 2004). Persepsi kedua tipe pertahanan ini selanjutnya mengaktivasi tahapan (cascade) transduksi sinyal dan respon pertahanan awal untuk mencegah perluasan serangan patogen ke dalam jaringan sel inang.
Istilah elisitor awalnya hanya digunakan untuk menyebut molekul yang menginduksi produksi fitoaleksin, sedangkan saat ini istilah tersebut menunjuk secara luas pada senyawa yang mampu menstimulasi berbagai tipe pertahanan tanaman (Nürnberger 1999), baik senyawa tersebut berasal dari patogen (exogenous elicitors) maupun senyawa yang dikeluarkan oleh tanaman akibat tindakan patogen (endogenous elicitors) (Boller 1995). Berdasarkan spesifitasnya, elisitor dapat diklasifikasikan ke dalam dua kelompok, yaitu elisitor umum (general elisitor), yang mampu memicu pertahanan baik pada tanaman inang maupun bukan inang dan elisitor spesifik ras (race spesific elicitor) yang menginduksi respon pertahanan dan menghasilkan resistensi hanya pada kultivar inang spesifik. Elisitor tidak mempunyai struktur kimiawi yang umum, melainkan termasuk ke dalam berbagai kelas senyawa yang berbeda secara luas mulai dari oligosakarida, peptida, protein dan lipid. Hal ini menunjukkan bahwa tanaman mempunyai kemampuan untuk mengenali berbagai molekul yang secara struktural berbeda sebagai sinyal bagi pertahanan terhadap patogen (Boller 1995).
Salah satu jenis elisitor umum adalah flagelin. Flagelin adalah protein penyusun utama flagela bakteri. Protein yang terdapat dalam jumlah besar pada hampir seluruh bakteri berflagela ini, bermassa 30 kDa hingga 60 kDa dan membentuk lubang silinder yang menyusun filamen flagela bakteri. Struktur flagelin inilah yang membangun bentuk heliks filamen flagela yang sangat penting untuk menjalankan fungsi flagela, sedangkan terminal N dan C flagelin memberinya kemampuan untuk melakukan polimerisasi menjadi filamen. Pengenalan terhadap flagelin melibatkan reseptor semacam kinase (receptor like
7
kinase) FLS2 dan mengaktivasi respon hilir (downstream) antara lain produksi
ROS (reactive oxygen species), biosintesis etilen, aktivasi kaskade MAPK (mitogen-activated protein kinases) dan aktivasi ekspresi gen pertahanan (Gomez-Gomez et al. 1999, Nuhse et al. 2000, Asai et al. 2002). Pada Arabidopsis, persepsi terhadap flg22 meningkatkan resistensi untuk melawan bakteri patogen (Zipfel et al. 2004).
Menurut Boller et al. (1995), pengenalan patogen pada tumbuhan melibatkan interaksi reseptor tanaman dengan elisitor dan induksi cascade fosforilasi yang mengarah pada respon-respon pertahanan (Gambar 2). Beberapa kejadian pensinyalan awal (early signalling events) adalah antara lain fluks ion melalui membran plasma, alkalinisasi ekstraseluler, peningkatan sementara konsentrasi kalsium (Ca) sitosolik, fosforilasi dan defosforilasi melalui aktivasi MAPK dan CDPK (calcium-dependent protein kinases) dan produksi ROS ekstraseluler melalui NADPH oksidase yang terletak pada plasma membran dan peroksidase apoplastik serta biosintesis etilen dan asam jasmonat (Menke et al. 1999; Scheel 1998).
Gambar 2. Mekanisme pertahanan tanaman terhadap patogen
Protein-protein yang membentuk jalur sinyal transduksi, kemunculannya pada sel berdasarkan persepsi terhadap elisitor. Aktivasi berbagai protein tersebut terjadi melalui modifikasi pascatranslasi, perubahan konformasi dan/atau perubahan dalam formasi kompleks. Modifikasi pascatranslasi yang diketahui
8
paling luas terlibat dalam transduksi sinyal adalah fosforilasi protein. Inisiasi pensinyalan elisitor umum diprediksi terjadi melalui interaksi antara elisitor dengan reseptor yang terletak pada membran plasma. Pensinyalan dan respon awal tampaknya dimediasi melalui protein-protein berasosiasi membran, dimana hasil studi terbaru menunjukkan bahwa protein-protein tersebut mempunyai satu atau lebih situs fosforilasi (Nuhse et al. 2004).
Metabolit sekunder pada tanaman memainkan peran utama dalam menghadapi perubahan lingkungan dan mengatasi cekaman. Peran penting tersebut terbentuk dari kompleksitas tipe kimiawinya yang luas maupun interaksinya yang mendasari beragam fungsi seperti menstabilkan struktur; sebagai penentu polimerisasi dan kondensasi fenol dan quinon; interaksi elektrostatis antara poliamin dengan lokus bermuatan negatif dalam komponen sel; fotoprotektif yang terkait dengan absorbansi cahaya tampak dan radiasi ultraviolet akibat adanya ikatan ganda terkonjugasi; antioksidan dan anti radikal yang diatur oleh ketersediaan kelompok –OH, –NH2, dan –SH serta transduksi
sinyal (Edreva et al. 2008).
Menurut Zhao et al. (2005), akumulasi metabolit sekunder tanaman sering terjadi pada tanaman yang terpapar oleh cekaman termasuk oleh berbagai elisitor atau molekul sinyal. Sejumlah aspek transduksi sinyal elisitor yang menghantarkan pada produksi metabolit sekunder tanaman diantaranya adalah : persepsi elisitor oleh berbagai reseptor tanaman; determinan avirulen dan keterkaitannya dengan protein R; protein pengikat small GTP dan heterotrimerik; fluks ion; alkalinisasi media dan asidifikasi sitoplasma; pecahan oksidatif dan ROS; inositol trifosfat dan nukleotida siklik (cAMP dan cGMP); asam salisilat dan nitrit oksida; pensinyalan asam jasmonat, etilen dan asam absisik; sinyal oksilipin dan sebagainya. Seluruh komponen sinyal ini diatur baik secara langsung ataupun tidak langsung oleh elisitor untuk menginduksi akumulasi metabolit sekunder tanaman (Zhao et al. 2005).
Steroid Nuclear Receptor
Steroid Nuclear Receptor (SNR) adalah protein subkelas Nuclear reseptor
9
faktor transkripsi yaitu protein yang meningkatkan atau menurunkan transkripsi gen-gen. Aktivitasnya secara spesifik adalah mengatur pengikatan RNA polimerase pada DNA. NR bekerjasama dengan protein lain mengendalikan proses metabolisme, perkembangan dan homeostasis organisme melalui pengaturan ekspresi gen-gen spesifik (Germain et al. 2003). Reseptor ini adalah aktivator transkripsi yang teraktivasi ligan. Ketika terikat dengan ligan (hormon), maka kompleks tersebut akan mampu melintasi membran inti masuk ke dalam nukleus dan dapat berikatan dengan gen tertentu yang karenanya dapat menginduksi transkripsi dan selanjutnya menghasilkan protein.
Ligan-ligan khusus untuk reseptor inti adalah hormon-hormon lipofilik dengan hormon steroid dan turunan vitamin A dan D. Hormon-hormon ini memainkan peran penting dalam regulasi metabolisme, fungsi organ, proses perkembangan dan diferensiasi sel. Reseptor inti yang telah diaktivasi hormon-hormon mengikat DNA spesifik reseptor HRE (hormon-hormone response element)yaitu sekuen DNA yang terletak pada daerah promotor gen yang diaktivasi oleh komplek hormon-reseptor. Karena proses ini selanjutnya menyebabkan terjadinya transkripsi gen-gen terkait, maka hormon-hormon yang terlibat juga disebut sebagai penginduksi (inducer) ekspresi gen. Aktivasi transkripsi gen lebih lambat dibandingkan sinyal-sinyal yang secara langsung mempengaruhi protein yang ada. Sebagai konsekuensinya, efek hormon-hormon yang menggunakan reseptor inti biasanya bersifat jangka panjang. Secara primer, reseptor ini terletak di dalam sitosol. Dengan ketiadaan hormon steroid, reseptor-reseptor dapat melekat bersama-sama membentuk kompleks yang disebut sebagai kompleks aporeseptor, yang juga mengandung protein chaperone (juga dikenal sebagai heat shock
protein atau Hsps). Protein heatshock diperlukan untuk mengaktivasi reseptor
dengan membantu protein untuk melipat dengan suatu langkah yang dengannya sekuen sinyal dapat melalui jalan lintasan ke nukleus. Reseptor steroid juga memiliki efek represif terhadap ekspresi gen ketika domain transaktivasinya tersembunyi sehingga protein tersebut tidak dapat mengaktivasi transkripsi. Lebih jauh, aktivitas reseptor steroid dapat meningkatkan fosforilasi residu serin pada N terminalnya, sebagai hasil lintasan transduksi sinyal lain misalnya oleh
10
faktor pertumbuhan (growth factor). Tingkah laku demikian ini dikenal dengan istilah Crosstalk (Germain et al. 2003).
Menurut Shen dan Schulze-lefert (2007), pada tanaman, NR diduga berperan secara signifikan dalam sensor imunitas. Lintasan pensinyalan didalamnya adalah paralel dengan mekanisme kontrol regulator pada reseptor steroid hewan. Blumberg et al. (1998) berhasil mengisolasi Steroid Xenobiotic
Receptor (SXR), salah satu SNR yang mengaktivasi gen penyandi sitokrom P450
3A4 (CYP3A4). CYP3A4 dikenal sebagai enzim pertahanan pertama yang melakukan metabolisme terhadap obat dan berbagai senyawa toksik. Bock and Kohle (2004) menyatakan bahwa Xenobiotic Nuclear Receptor (Pregnane X
Receptor/PXR, Constitutive Androstane Receptor/CAR, dan Aryl hidrocarbon Receptor/Ah) secara terkoordinasi menginduksi gen-gen yang terlibat pada
seluruh fase metabolisme xenobiotik baik fase metabolisme oksidatif, fase konjugasi maupun fase transport.
Auoabdi et al. (2006) menyatakan bahwa PXR mampu mengatur
ekspresi 40 gen penyandi enzim-enzim metabolisme xenobiotik dan drug
transporter penting. PXR mengintegrasikan input dari berbagai faktor transkripsi
teraktivasi ligan ke dalam satu respon akhir sehingga dikenal sebagai master xenobiotik/metabolit. Selain mengaktivasi CYP3A4 (El sankary et al. 2000) dan CYP2B8 (Goodwin et al. 2001), PXR juga dilaporkan mengaktivasi GST A2 (Glutathione-S-Transferase) (Falkner et al. 2001). GST dikenal sebagai antioksidan (Ezaki et al. 1995) yang berperan mengkatalisis hasil konjugasi senyawa toksin dengan glutathione menjadi bentuk nontoksik (Xiang & Oliver 1998) dan pada Arabidopsis thaliana diinduksi oleh Al (Richard et al. 1998). Maglich et al. (2002) melaporkan bahwa PXR juga mengaktivasi OATP2 (Organic Anion Transporting Polypeptide) dan MDR1a (Multidrug resistance). MDR berperan dalam resistensi sel terhadap berbagai obat, yang pada ragi ekspresinya diinduksi oleh cekaman kadmium (Li et al. 1997), sedangkan pada barley diinduksi oleh cekaman Al (Sasaki et al. 2002). Gen penyandi MRP (multidrug resistance associated protein), jenis MDR lainnya berhasil diisolasi dari Arabidopsis yaitu AtMrp1 (Lu et al. 1997; 1998) dan ekspresinya diinduksi oleh berbagai senyawa seperti benoksakor, kloquintocet, klorodinitrobenzene
11
(CNDB), asam salisilat dan menadion (Gaedeke et al. 2001). Fragmen cDNA
mrp berukuran 633 pb yang menyandi daerah antara TM1 dan NBF1 juga telah
berhasil diisolasi dari Melastoma affine (Suharsono et al. 2008).
Toleransi Melastoma malabathricum L. terhadap xenobiotik
M. malabathricum L. (Gambar 3) merupakan salah satu jenis gulma
invasif yang ditemukan tumbuh baik di berbagai lahan asam di Indonesia, di Asia Tenggara. Jenis tanaman semak tropis anggota family Melastomaceae yang di Indonesia dikenal bernama Harendong ini mendominasi lahan-lahan asam di Kalimantan, Sumatera dan Irian Jaya. M. malabathricum L. dikenal sebagai hiperakumulator aluminium yang menjadi tanaman dominan pada tanah asam daerah tropis. Melastoma mampu mengakumulasi Al diatas 10000 mg kg-1 aluminium pada daun dewasa dan diatas 7000 pada daun mudanya (Watanabe et
al. 1997). Pertumbuhan dan penyerapan hara pada melastoma justru semakin
meningkat dengan keberadaan Al (Osaki et al. 1997). Mekanisme induksi pertumbuhan Melastoma oleh Al ini masih belum jelas. Cekaman Al pada tanaman toleran akan menginduksi sejumlah gen untuk menghindari pengaruh ion Al. Pada Melastoma, gen-gen ini diduga tidak hanya berperan dalam mendetoksifikasi Al, akan tetapi juga berperan dalam menginduksi hormon pertumbuhan.
Gambar 3. Melastoma malabathricum L.
M. malabathricum L. dan famili Melastomaceae dari ordo Myrtales
dilaporkan menghasilkan elagitanin yang potensial untuk pencegahan penyakit dan mempunyai aktivitas biologis baik bersifat efek anti tumor maupun anti
12
bakteri. Di Malaysia, M. malabathricum dikenal secara lokal sebagai senduduk. Akar, daun dan pucuknya telah digunakan oleh penduduk Malaysia untuk perlakuan pasca kelahiran (post natal), mencegah bekas cacar (smallpox), disentri, diare, radang pencernaan, dan obat kumur (Yoshida et al. 2000).
Menurut Yoshida et al. (2010), M. malabathricum menghasilkan metabolit berbasis stachyurin kompleks tannin malabathrin A, E dan F (Gambar 4). Metabolit ini adalah bagian dari tanin kompleks (flavono-elagitanin) yang ditentukan oleh struktur unik C-C terkondensasi dari tanin C-glikosidik (tipe
vescalagin atau tipe stachyurin) dengan flavan-3-ol (katekin atau epikatekin).
Tidak sebagaimana tanin C-glikosidik, tanin jenis ini ditemukan terbatas hanya pada sejumlah species tanaman yang termasuk ke dalam famili Combretaceae,
Myrtaceae, Fagaceae dan Theaceae.
Gambar 4. Struktur kimia malabathrin yang merupakan metabolit sekunder yang dihasilkan Melastoma malabathricum L. Kedua kompleks tannin berbasis vescalagin dan stachyurin yang diisolasi hingga saat ini, seluruhnya dicirikan dengan keberadaan sebuah ikatan C-C berorientasi β pada glukosa C-1 [1]. Terdapat banyak contoh hemisintesis kompleks tanin oleh reaksi diantara tanin C-glikosidik dan (+)- katekin atau (-)-epikatekin yang dikatalisis oleh asam sederhana (Yoshida et al. 2010). Kompleks polifenol, tanin yang dapat dihidrolisis dan termasuk dalam oligomer elagitanin ini menarik perhatian dalam dua dekade terakhir ini karena keragaman struktur maupun aktivitas biologisnya terutama karena efek antioksidatif, antiviral dan anti tumor yang dihasilkannya (Yoshida et al. 2000).
METODE PENELITIAN
Waktu dan Tempat Penelitian
Penelitian ini dilakukan mulai bulan Februari 2005 hingga bulan Maret 2008 di Laboratorium Biologi Molekuler dan Seluler Tanaman dan Laboratorium BIORIN (Biotechnology Research Indonesia – The Netherlands), Pusat Penelitian Sumberdaya Hayati dan Bioteknologi Institut Pertanian Bogor.
Bahan
Bahan tanaman yang digunakan adalah daun dan akar M. malabathricum
L. yang diperoleh dari lahan asam Jasinga, Bogor. Plasmid pGEM®-T-Easy
(Promega) digunakan sebagai vektor pengklonan dan E. coli galur DH5α digunakan sebagai inang vektor rekombinan. Primer aktin
ActF:TCACCAACTGGGACGACATG dan ActR: TCATGAGGTAGTCAGTCAGGT
digunakan sebagai alat evaluasi cDNA total. Primer spesifik dari gen SNR A.
thaliana L. yaitu snrF: ATGGATATTGATCACGGCAGA dan snrR: ATAACCACGTCAGG CAAAGG digunakan untuk mengisolasi cDNA MmFSNR.
Metode penelitian
Isolasi dan pengklonan fragmen MmFSNR dilakukan melalui beberapa tahapan yaitu isolasi RNA total, sintesis cDNA total, isolasi cDNA MmFSNR, pengklonan cDNA MmFSNR ke dalam vektor, transformasi genetik E. coli dan analisis MmFSNR.
Isolasi RNA total. RNA total diisolasi menggunakan metode Chang et al.
(1993) yang dimodifikasi. Daun dibuang tulangnya, ditimbang sebanyak 1 gram lalu ditambah 10 ml buffer 2XCTAB (2% CTAB, 2% PVP 25000, 0.1 M tris pH 9.5, 20 mM EDTA, 1.4 M NaCl, 1% ß-mercaptoetanol dan 0.1% DEPC) yang sebelumnya telah dihangatkan pada suhu 65ºC, digerus dengan bantuan pasir kuarsa hingga menjadi serbuk halus. Campuran serbuk daun dan buffer ekstraksi diinkubasi selama 10 menit pada suhu 65ºC dan digoyang beberapa kali, diekstraksi dengan 10 ml Kloroform:Isoamilalkohol (24:1) lalu disentrifugasi pada 10000 rpm pada suhu 4oC (Sorval Ultra Pro 80) selama 10 menit. Bagian atas
14
cairan (supernatan) dipindahkan ke tabung baru kemudian ditambah ¼ volume LiCl 10M. Campuran diinkubasi pada suhu -20ºC selama 2.5 jam kemudian disentrifugasi (Sorvall Ultra Pro 80) pada kecepatan 10000 rpm selama 10 menit pada suhu 4ºC. Fase cairan dibuang, RNA total yang mengendap disuspensi dengan 500μl TE 1x (10mM Tris HCl pH 7.4 dan 1 mM EDTA) dan dipindah ke tabung eppendorf. RNA diekstraksi dengan 1xvolume fenol pH 9, disentrifugasi pada kecepatan 14000 rpm (Jouan BR4i) selama 10 menit pada suhu 20 ºC. Suspensi RNA (di bagian atas) dan dipresipitasi dengan penambahan 1 x volume PCI (25:24:1) dan disentrifugasi pada kecepatan 14000rpm (Jouan BR4i) selama 10 menit pada suhu 20ºC. Supernatan diambil dan dipresipitasi dengan penambahan ¼ x volume LiCl 10M dan kemudian diinkubasi selama semalam pada suhu -20ºC. Untuk mengendapkan RNA total, campuran disentrifugasi pada kecepatan 14000rpm (Jouan BR4i) selama 10 menit pada suhu 4ºC. Cairan dibuang dan endapan RNA dibilas dengan penambahan 500μl ethanol 70% kemudian disentrifugasi pada kecepatan 14000 rpm (Jouan BR4i) selama 10 menit pada suhu 4ºC. Endapan dikeringkan dengan vacuum dryer selama 15 menit kemudian diresuspensi dengan H2O yang telah diperlakukan dengan DEPC.
Kuantitas RNA total hasil isolasi ditentukan dengan spektrofotometer UV-VIS (Cecil CE 2020) pada panjang gelombang 260 nm. Konsentrasi RNA ditentukan dengan menetapkan satu satuan absorban pada panjang gelombang 260 nm setara dengan 40 μg/ml RNA. Kemurnian RNA total ditentukan berdasarkan perbandingan nilai absorban pada panjang gelombang 260 nm dengan 280 nm (Saunders & Parker 1999). Keutuhan RNA ditentukan berdasarkan elektroforesis pada gel agarose 1% (FMC, USA) dengan larutan MOPS (4.2g/l MOPs, 0.41 g/l Na-asetat, 0.37 g/l EDTA (2Na)H2O). RNA sebanyak 1μl dicampur dengan 12 μl
larutan premiks [MOPS, 50% (v/v) formamide,17.5% (v/v) formaldehid dan 27.5% (v/v) air DEPC] dipanaskan 65 ºC 10 menit, didinginkan di es 5 menit dan ditambahkan 1/6 x volume loading dye (0.25% bromophenol blue, 0.25% xylene cyanol FF, 30% gliserol) kemudian dielektroforesis pada 100 volt selama 30 menit. Visualisasi RNA dilakukan pada transiluminator GelDoc (Labquip) setelah diwarnai dengan EtBr selama 30 menit dan dibilas dengan H2O.
15
Sintesis cDNA total. cDNA total disintesis melalui reaksi transkripsi balik
(ReverseTranscriptase/RT) menggunakan Superscript III Reverse Transcriptase (Invitrogen). Komposisi sintesis cDNA total adalah 5 μg RNA total, 1x RT buffer, 20 pmol oligodT, 4mM dNTP, 10mM DTT, 40 U Enzim SuperScript TM
III RTase
dan dH2O yang telah diperlakukan dengan DEPC hingga volume reaksi mencapai 20μl. Sintesis cDNA dilakukan pada suhu 52°C selama 50 menit. Keberhasilan terbentuknya cDNA dan kemurnian cDNA diuji dengan menggunakan PCR dengan primer spesifik aktin. Komposisi reaksi PCR adalah 0.75 μl cDNA total hasil RT, 1x Buffer taq, 30 mM MgCl2, 3 mM dNTP mix, 15 pmol primer ActF, 15 pmol primer ActR, 4% Me2SO, 0.75 U enzim taq DNA polymerase (Fermentas
Inc.) dan ddH2O dengan volume reaksi 15 μl. PCR dilakukan pada kondisi praPCR pada 95 °C 5 menit, denaturasi pada 94°C 30 detik, penempelan primer pada 57°C selama 30 detik dan pemanjangan pada 72° C selama1.5 menit. PCR dilakukan sebanyak 35 siklus dan pascaPCR dilakukan pada suhu 72°C selama 5 menit. Hasil PCR actin dielektroforesis dalam gel agarose 1.2% dengan larutan penyangga TAE 1X (4.84 g/l Tris Hydroximetilaminomethan, 0.1142 ml/l asetat glasial, 2 ml 0.5M EDTA pH 8). Elektroforesis dilakukan pada voltase konstan 50 volt selama 1 jam. Visualisasi hasil elektroforesis dilakukan diatas transiluminator UV (Hoever) dan difoto menggunakan kamera digital yang dihubungkan ke komputer dengan menggunakan program Digidoc. Bila hasil visualisasi menunjukkan pita ukuran 450bp maka RNA yang digunakan sebagai bahan reaksi RT murni dan cDNA total tidak terkontaminasi DNA genom sehingga cDNA total ini dapat digunakan untuk mengisolasi cDNA MmFSNR.
Isolasi cDNA MmFSNR. Reaksi PCR dengan primer spesifik dilakukan
menggunakan cDNA hasil RT dan sepasang primer spesifik untuk snrF dan snrR yang didisain dari A. thaliana. Campuran PCR terdiri dari 2 μl 10X PCR Buffer (200mM Tris HCl pH 8.4, 500mM KCl) (Fermentas), 2 μl 25mM MgCl2
(Fermentas), 1 μl 10mM dNTP Mix, 1 μl primer forward (10 pmol), 1 μl primer
reverse (10 pmol), 0.2 μl Taq DNA polymerase (5 Unit/μl),1 μl cDNA (hasil RT)
dan ddH2O hingga volume mencapai 20 μl. PCR dilakukan dengan kondisi
pra-PCR pada 94°C, 5 menit; denaturasi pada 94°C, 30 detik; penempelan primer pada 55°C , 30 detik; dan pemanjangan pada 72°C, 7 menit sebanyak 35 siklus
16
menggunakan alat PCR (MJ Research TM 100). Hasil PCR dengan primer spesifik kemudian diuji dengan elektroforesis.
Pengklonan cDNA MmFSNR kedalam vektor pGEM®-T-Easy. Hasil
sintesis cDNA spesifik dari mRNA Melastoma selanjutnya disisipkan ke dalam vektor pGEM®-T-Easy (Promega) mengikuti prosedur Promega. Ligasi dilakukan dengan mencampurkan 1- 3 μl cDNA spesifik dengan 5 μl buffer 2X Rapid
Ligation,1μl vektor pGEM®-T-Easy (50ng), 1 μl T4 DNA Ligase (3 Weiss
units/μl), dan ddH2O sampai volume akhir 10 μl. Campuran reaksi diinkubasi
pada 4 OC selama semalam.
Transformasi genetik E. coli. Transformasi E. coli dengan vektor
rekombinan dilakukan berdasarkan prosedur Suharsono (2002). Satu koloni bakteri E. coli galur DH5α dikulturkan dalam 2 ml LB cair (10g/l Bacto-tryptone, 5 g/l Yeast extract, 10 g/l NaCl) menggunakan shaking incubator berkecepatan 250 rpm pada suhu 37ºC selama semalam. Kemudian, 200 μl disubkultur dalam 20 ml LB cair dengan kondisi yang sama hingga OD600=0.4-0.5. Bakteri
dipindahkan ke tabung eppendorf 1.5 ml dan diinkubasi dalam es selama 10 menit lalu disentrifugasi pada kecepatan 3000rpm (Jouan BR4i) pada suhu 4ºC selama 5 menit. Endapan bakteri disuspensi dalam 495 μl buffer transformasi TB (10mM PIPES, 15mM CaCl2.2H2O, 250 mM KCl, 55 mM MnCl2.4H2O, pH 6.7) dan
diinkubasi dalam es selama 10 menit kemudian disentrifugasi dengan kecepatan 3000rpm pada suhu 4ºC selama 5 menit. Endapan bakteri ditambah 82.5 μl TB dan disuspensi dengan hati-hati kemudian ditambah 3.3 μl DMSO dan diinkubasi di dalam es selama 10 menit sehingga diperoleh bakteri kompeten. Sebanyak 50µl dari bakteri kompeten tersebut dicampur dengan 10µl (50-100ng) DNA plasmid hasil ligasi dan diinkubasi dalam es selama 25 menit. Campuran dipanaskan pada suhu 45ºC selama 45 detik dan diinkubasi kembali ke dalam es selama 5 menit. Campuran ini ditambah 100µl media 2xYT (16g/l Bacto-tryptone, 10g/l Yeast extract, 5 g/l NaCl pH 7.0) dan diinkubasi pada suhu 37ºC selama 20 menit dalam shaking incubator berkecepatan 250 rpm. Bakteri disebar merata diatas media LB yang mengandung ampicilin menggunakan batang kaca yang dibengkokkan lalu diinkubasi pada suhu 37ºC selama semalam. Seleksi transforman rekombinan dilakukan dengan memilih koloni putih yang tumbuh.
17
Analisis cDNA sisipan. Analisis cDNA sisipan dimulai dengan isolasi
plasmid rekombinan. Plasmid diisolasi menggunakan prosedur Suharsono (2002). Kultur E. coli sebanyak 150 µl ditumbuhkan dalam 15 ml media LB dan diinkubasi di inkubator bergoyang (250 rpm) pada suhu 37OC semalam. Kultur bakteri disentrifugasi dengan kecepatan 12000 rpm (Jouan BR4i) pada suhu 4OC selama 30 menit. Pelet disuspensikan dengan 300μl buffer suspensi (50 mM Tris-HCl, pH 7,5 dan 10 mM EDTA). Suspensi bakteri ditambah 300μl buffer lisis (0,2 M NaOH dan 1% SDS) dan dibolak balik perlahan beberapa kali. Campuran ditambah 300μl buffer netralisasi (5 M Na-Asetat, Asam asetat glasial, dan H2O
PH 4,8) dan disentrifugasi 13000 rpm pada suhu 4OC selama 20 menit. Supernatan diambil dan diekstrak dengan 1xvolume PCI (25:24:1), divorteks dan disentrifugasi dengan kecepatan 13000 rpm pada suhu 4OC selama 2 menit. Supernatan diambil dan diperlakukan dengan RNAse pada suhu 37OC semalam. Larutan diekstrak kembali dengan PCI dan disentrifugasi seperti ekstraksi sebelumnya. Supernatan diambil dengan hati-hati dan ditambahkan 0,1x volume 3 M NaOAc pH 5.2 dan 2x volume EtOH absolut. Larutan diinkubasi selama 2 jam pada suhu -20 OC dan disentrifugasi dengan kecepatan 13000 rpm pada suhu 4OC selama 20 menit. Pellet dibilas dengan 1 x volume EtOH 70% dan dikeringkan dengan alat vakum. DNA kering disuspensikan di 10-20 μl ddH2O.
Analisis urutan nukleotida MmFSNR dan protein MmFSNR.
Pengurutan DNA dilakukan menggunakan DNA Sequencer ABI Prism Model 310 versi 3.7. Identifikasi urutan nukleotida dilakukan menggunakan beberapa analisis. Analisis kesejajaran lokal fragmen MmFSNR berdasarkan urutan nukleotida dan asam amino dengan data yang ada di GenBank dilakukan dengan menggunakan program BLAST (Basic Local Alignment Search Tools) yang tersedia dalam NCBI (National Center for Biotechnology Information) melalui akses situs http://www.ebi.ac.uk/BLAST (Mount, 2001). Analisis situs restriksi dilakukan dengan menggunakan program Bioedit versi 7.0.0 dan NEBCutter
(http://www.firstmarket.com/cutter/cut2.htm). Program Bioedit versi 7.0.0 juga
digunakan untuk melakukan analisis deduksi asam amino dari urutan DNA dan analisis hidrofobisitas protein MmFSNR yang dibandingkan dengan protein SNR pada padi dan pada Medicago truncantula.
HASIL DAN PEMBAHASAN
Isolasi RNA Total
RNA total M. malabathricum telah berhasil diisolasi melalui modifikasi
metode Chang et al. (1993). Modifikasi dilakukan pada larutan penyangga dengan peningkatan konsentrasi β-mercaptoetanol sampai 200μl dan penambahan 1% PVP (Polivinil polivirolidon) dan pada fase ekstraksi yaitu menggunakan fenol pH 9. Pengujian hasil isolasi RNA total menunjukkan rendemen antara 171-247 μg/g bahan tumbuhan. Analisis kemurnian RNA melalui metode spektrofotometri menunjukkan nilai rasio absorban pada panjang gelombang 260nm dan 280nm sebesar 1.87 hingga 1.9 (Tabel 1). Menurut Farrel (1993) jika nilai rasio absorban pada panjang gelombang 260nm dan 280nm berkisar 1.8-2.0 maka RNA total yang diisolasi adalah murni yang bebas dari kontaminan protein.
Tabel 1 Data hasil isolasi RNA total
No. Bahan Tumbuhan Nilai Absorbansi Absorbansi Rasio (λ 260 /λ 280) Rendemen RNA (μg/g bahan tumbuhan) λ 260 λ 280 1 Akar 0.229 0.112 1.87 192 2 Daun 0.203 0.109 1.86 171 3 Daun pucuk 0.294 0.155 1.90 247
Pengujian keutuhan RNA dengan elektroforesis menunjukkan adanya dua pita dominan pada RNA total yang diisolasi dari akar, daun tua dan daun pucuk. Dua pita dominan ini kemungkinan adalah RNA ribosomal (rRNA) 28S dan 18S (Gambar 5). Munculnya pita rRNA dalam bentuk utuh mengindikasikan bahwa RNA total yang telah berhasil diisolasi adalah utuh.
Gambar 5. Hasil elektroforesis RNA total.
RNA total dari (1) akar, (2) daun, (3) daun pucuk
1 2 3 28S
19
Sintesis cDNA total
cDNA total M. malabathricum L. berhasil disintesis dengan RNA total
sebagai cetakan (template) melalui proses transkripsi balik dua langkah. Keberhasilan sintesis cDNA dan kualitas mRNA diuji dengan PCR menggunakan primer spesifik gen aktin sebagai kontrol. Hasil visualisasi PCR cDNA dengan primer actin (Gambar 6) menunjukkan hasil amplifikasi sekitar 450bp yang berarti sintesis cDNA total melalui transkripsi balik berlangsung dengan baik karena daerah yang diamplifikasi adalah hanya cDNA ekson1-ekson2 bukan DNA ekson1 - ekson2 dari gen aktin yang berukuran sekitar 600 pb. RNA total yang diisolasi memiliki kualitas bagus karena dapat digunakan untuk mensintesis cDNA dan bersih dari kontaminasi DNA.
Gambar 6. Pita cDNA ekson1-ekson2 aktin hasil PCR.
cDNA (1) daun tua, (2) daun pucuk, (3) akar sebagai cetakan
Isolasi fragmen cDNA MmFSNR melalui PCR
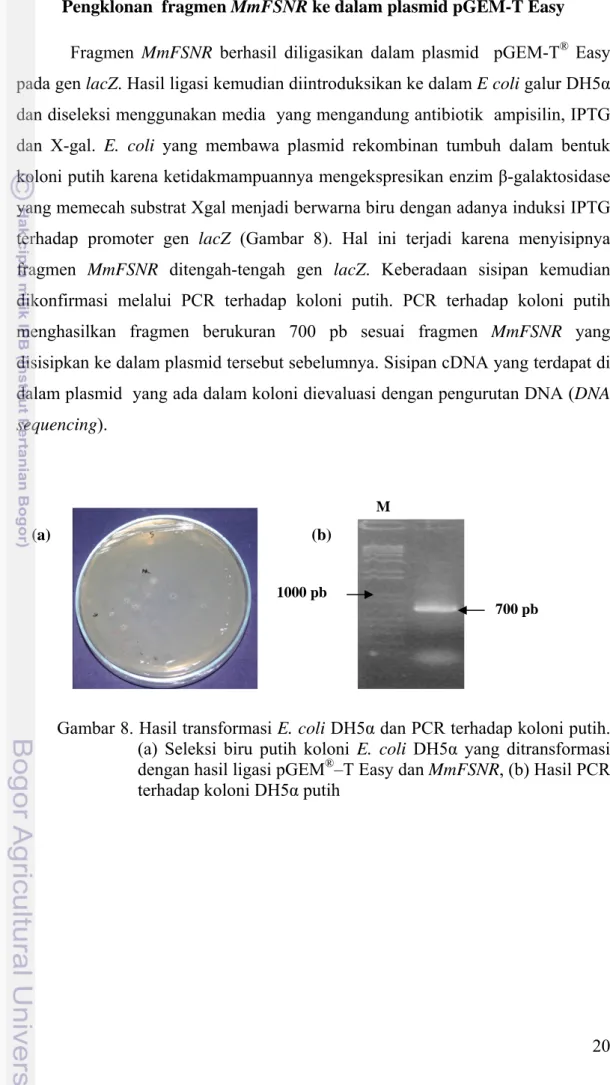
Fragmen berukuran 700 pb berhasil diisolasi dengan menggunakan cDNA daun M. malabathricum dan primer spesifik yang selanjutnya disebut sebagai
MmFSNR (Gambar 7).
Gambar 7. Pita fragmen cDNA MmFSNR hasil PCR.
(1) cetakan cDNA akar, (2) cetakan cDNA daun, M=1 kb ladder 700 pb
1 2 M
1000 pb
Hasil PCR cDNA dengan primer aktin
M 1 2 3
1000 pb
20
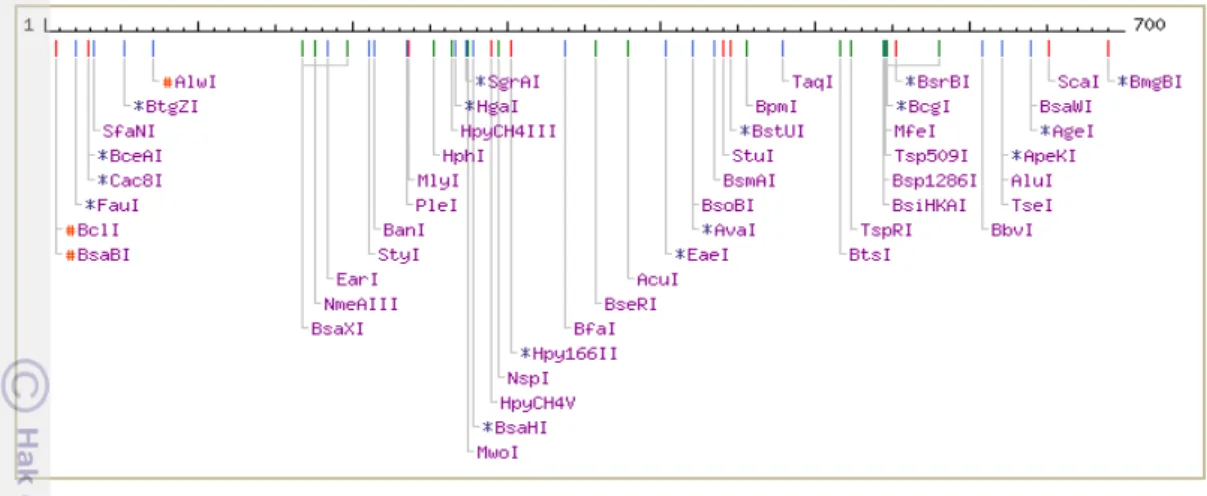
Pengklonan fragmen MmFSNR ke dalam plasmid pGEM-T Easy
Fragmen MmFSNR berhasil diligasikan dalam plasmid pGEM-T® Easy pada gen lacZ. Hasil ligasi kemudian diintroduksikan ke dalam E coli galur DH5α dan diseleksi menggunakan media yang mengandung antibiotik ampisilin, IPTG dan X-gal. E. coli yang membawa plasmid rekombinan tumbuh dalam bentuk koloni putih karena ketidakmampuannya mengekspresikan enzim β-galaktosidase yang memecah substrat Xgal menjadi berwarna biru dengan adanya induksi IPTG terhadap promoter gen lacZ (Gambar 8). Hal ini terjadi karena menyisipnya fragmen MmFSNR ditengah-tengah gen lacZ. Keberadaan sisipan kemudian dikonfirmasi melalui PCR terhadap koloni putih. PCR terhadap koloni putih menghasilkan fragmen berukuran 700 pb sesuai fragmen MmFSNR yang disisipkan ke dalam plasmid tersebut sebelumnya. Sisipan cDNA yang terdapat di dalam plasmid yang ada dalam koloni dievaluasi dengan pengurutan DNA (DNA
sequencing).
Gambar 8. Hasil transformasi E. coli DH5α dan PCR terhadap koloni putih. (a) Seleksi biru putih koloni E. coli DH5α yang ditransformasi dengan hasil ligasi pGEM®–T Easy dan MmFSNR, (b) Hasil PCR terhadap koloni DH5α putih
(a) (b)
M
700 pb 1000 pb
21
Analisis fragmen MmFSNR
Hasil pengurutan nukleotida fragmen cDNA sisipan menunjukkan bahwa DNA sisipan MmFSNR berukuran 700 pb (Lampiran 1 dan 2). Urutan DNA
MmFSNR termasuk posisi primer dan situs pengklonan pada pGEM®-T Easy,
disajikan dalam Gambar 9.
Gambar 9. Hasil pengurutan DNA sisipan (MmFSNR) pada pGEM®-T Easy menggunakan primer T7
Analisis situs pemotongan enzim restriksi terhadap fragmen MmFSNR menunjukkan bahwa fragmen ini tidak mengandung situs EcoR1 sehingga situs
EcoR1 dapat digunakan untuk mengeluarkan sisipan fragmen MmFSNR dari
vektor pengklonan plasmid pGEM-T® Easy. Analisis peta restriksi pada fragmen
MmFSNR berguna sebagai dasar informasi dalam konstruksi gen SNR dan secara
umum penting diketahui untuk melakukan rekayasa genetika. Peta restriksi fragmen MmFSNR disajikan pada Gambar 10.
T7
GCGGAGTCCATGCTGCTGCTGCTAGGTGGCGCTTGAATTCGATTATGGATATTGATCACGGCA GATAAGCGGCGGGCGAGGGCGATGATGCTTTATGGGGTTTCTTGAGATTTTGCTTTTGATCCC GAAGAAGAAGAAGAAGAAGAAGAAGAAGGAGAAGAAGAAGGGGGGAGGGGGATGGAGCC TCCGAGGGGATTCCTGCCTACCCTCGCCAAGTTCTTACTCTTCCTCCCCTATTTCCTCGGCTTATT GCTCCTTGGCACCCTCAAGGGAGTCGTCATCTGCCCTTTCTCTTGCCTTATCCTCACCGTCGGCA ACACCGGCGTCGTCCTGTGCATGTGGCCCGTCCACTTCCTCTGGACGTATTACTCCATCTTGAGG GCTAGGATTATCGGCCCCATCCTGAAGTTTCTCCTCTTTATTACCCTGCCTGTTCCGTTGATCTTG TGGCCGGTGGTTGCTATCCTCGGGAGTGTCTCTGGAGGCCTCGCGTTCGGCTTTCTTTCTCCTAT CTTCGCTACTTTCGAGGCAGTAGGGAAGGGGAAGGCCAACGATGTGTTCCACTGCTTTTATGA TGGAACGTGGAGCACAATTGAGCGGAGCATGACCATCGTGAGGGACTTTTGGGACATTTGTTG CCATTCATATTTCTCAATCGTGAATGAGCTGCGGAATCAAGAGCCTACCGGTGGGAAGTACTAC GAGATCAGATTGTTGTACCTTCCTTTGCCTGACGTGGTTATAATCACTAGTGAATTCGC
Keterangan
Hitam : DNA sisipan Merah : Primer spesifik A1‐F dan A1‐R Hijau : Situs restriksi Eco RI Kuning : Situs restriksi Spe I
22
Gambar 10. Peta restriksi MmFSNR
Analisis kesejajaran lokal berdasarkan nukleotida menggunakan program BLASTN (http://www.ebi.ac.uk/BLASTN) menunjukkan bahwa fragmen
MmFSNR mempunyai kemiripan sebesar 65% dengan mRNA dari unknown protein At3g27390 Arabidopsis thaliana (Tabel 2).
Tabel 2. Hasil analisis kemiripan sekuen nukleotida MmFSNR dengan program BLASTN (http://www.ebi.ac.uk/BLASTN)
Align. DB:ID Source Length Score Identities Positives EO 1 EM_EST:CN916233 030203ABPB004005HT (ABPB) M9
root tips Malus x domestica cDNA clone ABPB004005, mRNA sequence.
Cross-refeerences and related information in:Ontologies
710 1032 65.0 65.0 1.8E-38
2 EM_EST:CN916808 030108ABPB001620HT (ABPB) M9 root tips Malus x domestica cDNA clone ABPB001620, mRNA sequence.
Cross-refeerences and related information in:Ontologies
650 1032 65.0 65.0 1,9E-38
3 EM_EST:CX542902 UCRPT01_5_003_D08_T3 Poncirus trifoliate CTV-chalanged cDNA library - UCRPT01-UCR2 citrus trifoliate cDNA clone UCRPT01_003_T3D08, mRNA sequence.
Cross-refeerences and related information in:Ontologies
807 961 64.0 64.0 2.5E-35
4 EM_EST:FS338946 Lotus japonicus cDNA, clone: LjFL3-063-CB12, 5’-end sequence.
Cross-refeerences and related information in:Ontologies
786 954 65.0 65.0 5.3E-35
5 EM_PL:BT002384 Arabidopsis thaliana Unknown
protein (At3g27390) mRNA, complete cds.
Cross-refeerences and related information in:Nucleotide Sequence Genomes Ontologies Protein Sequence
2011 957 65.0 65.0 6.1E-35
6 EM_EST: FS323108 Lotus japonicus cDNA, clone: LjFL3-015-AG09, 5’-end sequence.
Cross-refeerences and related information in:Ontologies
795 944 65.0 65.0 1.5E-34
7 EM_EST: FS322413 Lotus japonicus cDNA, clone: LjFL3-013-AG06, 5’-end sequence.
Cross-refeerences and related information in:Ontologies
23
Berdasarkan analisis kesejajaran ganda (multiple sequence alignment) menggunakan ClustalW pada program Bioedit versi 7.0.0, DNA sisipan
MmFSNR mempunyai kesamaan dengan sekuen nukleotida SNR dari padi
(Os02g0597800) dan Medicago truncantula yang dimulai dari nukleotida ke 133 dari primer yang digunakan untuk mengisolasi fragmen MmFSNR (Gambar 11).
Gambar 11. Hasil analisis kesejajaran ganda nukleotida MmFSNR dengan nukleotida SNR padi dan M. truncantula
Fragmen 132 nukleotida di ujung 5’ dari MmFSNR tidak mempunyai kemiripan baik dengan SNR dari padi (OsSNR) maupun SNR dari M. truncantula (MtSNR). Karena tidak mempunyai kesamaan dengan OsSNR dan MtSNR, bagian nukleotida MmFSNR yang berbeda tersebut yakni 132 pasang basa dari primer, dihilangkan dan hasilnya disebut sebagai MmFSNRcut. Fragmen nukleotida
MmFSNRcut selanjutnya dianalisis kembali menggunakan program BLASTN
untuk melihat apakah pemotongan 132 basa berpengaruh terhadap hasil analisis kemiripan DNA sisipan dengan database sekuen di GenBank.
Analisis kesejajaran lokal berdasarkan nukleotida menggunakan program BLASTN (http://www.ebi.ac.uk/BLASTN) menunjukkan bahwa fragmen
MmFSNRcut mempunyai kemiripan sebesar 66% atau hanya meningkatkan 1%
24
dari nilai kemiripan sebelumnya dengan mRNA dari unknown protein At3g27390
A. thaliana (Tabel 3). Pemotongan 132 pasang basa pada sekuen MmFSNR
ternyata tidak menghasilkan pengaruh nyata dalam pencarian hasil kemiripan DNA sisipan dengan database sekuen dalam GenBank.
Tabel 3 Hasil analisis kemiripan sekuen nukleotida MmFSNRcut dengan program BLASTN (http://www.ebi.ac.uk/BLASTN)
Align. DB:ID Source Length Score Identities Positives EO
1 EM_EST:CN916233 Brassica oleraceae var. achephala mRNA, clone: KALE-009K18, 5’ end sequence.
Cross-refeerences and related information in: Ontologies
679 531 67.0 67.0 5.8E-32
2 EM_EST:CN916808 Arabidopsis thaliana Unknown
protein (At3g27390) mRNA, complete cds.
Cross-refeerences and related information in:Ontologies
2011 555 66.0 66.0 6.5E-32
3 EM_EST:CX542902 vsu-ars_011439_LI10Contig_15980 VSU-ARS-L10 Phaseolus acutifolius cDNA, mRNA sequence.
Cross-refeerences and related information in:Ontologies
712 575 67.0 67.0 8.8E-32
4 EM_EST:FS338946 Brassica oleraceae var. achephala mRNA, clone: KALE-084E22, 5’ end sequence.
Cross-refeerences and related information in:Ontologies
818 531 67.0 67.0 1.7E-31
5 EM_PL:BT002384 Pv026B_M13F_E05. ab1 Phaseolus vulgaris cv Early gallatin Phaseolus vulgaris cDNA, mRNA sequence.
Cross-refeerences and related information in:Nucleotide Sequence Genomes Ontologies Protein Sequence
826 548 66.0 66.0 4.8E-31
6 EM_EST:CN916233 Brassica oleraceae var. achephala mRNA, clone: KALE-016H10, 5’ end sequence.
Cross-refeerences and related information in:Ontologies
846 503 66.0 66.0 7.1E-31
7 EM_EST:FS323108 Brassica oleraceae var. achephala mRNA, clone: KALE-154D20, 5’ end sequence.
Cross-refeerences and related information in:Ontologies
780 503 66.0 66.0 7.7E-31
8 EM_EST:GW468405 CA00-XXFR-022-C01-CB.F Coffea arabica FR2 Coffea arabica cDNA cloneCA00-XX-FR2-022-C01-CB mRNA sequence
Cross-refeerences and related information in:Ontologies
686 927 64.0 64.0 1.0E-33
9 EM_EST:HO786406
vsu-ars_011439_LI110contig_15980_VSU _ARS-L10 Phaseolus acutifolius cDNA, mRNA sequence
Cross-refeerences and related information in:Ontologies
712 906 64.0 64.0 8.7E-33
10 EM_EST :DK459251 Brassica oleracea var.acephala mRNA, clone KALE-009K18, 5’end sequence
Cross-refeerences and related information in:Ontologies
879 897 64.0 64.0 1.8E-32
11 EM_EST:DK461847 Brassica oleracea var.acephala mRNA, clone KALE-0016H10, 5’end sequence
Cross-refeerences and related information in:Ontologies
846 890 65.0 65.0 3.9E-32
12 EM_EST:DK514523 Brassica oleracea var.acephala mRNA, clone KALE-154D20, 5’end sequence
Cross-refeerences and related information in:Ontologies
25
Hasil deduksi asam amino menunjukkan bahwa MmFSNR menyandi 232 asam amino (Gambar 12).
Gambar 12. Deduksi asam amino dari urutan nukleotida fragmen cDNA
MmFSNR
Hasil kesejajaran berdasarkan urutan asam amino dengan program BLASTP (http://www.ebi.ac.uk/BLASTP) menunjukkan bahwa MmFSNR mempunyai kesamaan 53% dengan protein Uncharacterized membrane protein GN : At3g27390 OS : Arabidopsis thaliana (Uniprot : Y3739_ARATH) dan 47% dengan protein Steroid Nuclear Reseptor dari M. truncantula (Tabel 4).
ATGG ATA TTG ATC ACG GCA GAT AAG CGG CGG GCG AGG GCG ATG ATG 46 1 Trp Ile Leu Ile Thr Ala Asp Lys Arg Arg Ala Arg Ala Met Met 15 47 CTT TAT GGG GTT TCT TGA GAT TTT GCT TTT GAT CCC GAA GAA GAA 91 16 Leu Tyr Gly Val Ser End Asp Phe Ala Phe Asp Pro Glu Glu Glu 30 92 GAA GAA GAA GAA GAA GAA GGA GAA GAA GAA GGG GGG AGG GGG ATG 136 31 Glu Glu Glu Glu Glu Glu Gly Glu Glu Glu Gly Gly Arg Gly Met 45 137 GAG CCT CCG AGG GGA TTC CTG CCT ACC CTC GCC AAG TTC TTA CTC 181 46 Glu Pro Pro Arg Gly Phe Leu Pro Thr Leu Ala Lys Phe Leu Leu 60 182 TTC CTC CCC TAT TTC CTC GGC TTA TTG CTC CTT GGC ACC CTC AAG 226 61 Phe Leu Pro Tyr Phe Leu Gly Leu Leu Leu Leu Gly Thr Leu Lys 75 227 GGA GTC GTC ATC TGC CCT TTC TCT TGC CTT ATC CTC ACC GTC GGC 271 76 Gly Val Val Ile Cys Pro Phe Ser Cys Leu Ile Leu Thr Val Gly 90 272 AAC ACC GGC GTC GTC CTG TGC ATG TGG CCC GTC CAC TTC CTC TGG 316 91 Asn Thr Gly Val Val Leu Cys Met Trp Pro Val His Phe Leu Trp 105 317 ACG TAT TAC TCC ATC TTG AGG GCT AGG ATT ATC GGC CCC ATC CTG 361 106 Thr Tyr Tyr Ser Ile Leu Arg Ala Arg Ile Ile Gly Pro Ile Leu 120 362 AAG TTT CTC CTC TTT ATT ACC CTG CCT GTT CCG TTG ATC TTG TGG 406 121 Lys Phe Leu Leu Phe Ile Thr Leu Pro Val Pro Leu Ile Leu Trp 135 407 CCG GTG GTT GCT ATC CTC GGG AGT GTC TCT GGA GGC CTC GCG TTC 451 136 Pro Val Val Ala Ile Leu Gly Ser Val Ser Gly Gly Leu Ala Phe 150 452 GGC TTT CTT TCT CCT ATC TTC GCT ACT TTC GAG GCA GTA GGG AAG 496 151 Gly Phe Leu Ser Pro Ile Phe Ala Thr Phe Glu Ala Val Gly Lys 165 497 GGG AAG GCC AAC GAT GTG TTC CAC TGC TTT TAT GAT GGA ACG TGG 541 166 Gly Lys Ala Asn Asp Val Phe His Cys Phe Tyr Asp Gly Thr Trp 180 542 AGC ACA ATT GAG CGG AGC ATG ACC ATC GTG AGG GAC TTT TGG GAC 586 181 Ser Thr Ile Glu Arg Ser Met Thr Ile Val Arg Asp Phe Trp Asp 195 587 ATT TGT TGC CAT TCA TAT TTC TCA ATC GTG AAT GAG CTG CGG AAT 631 196 Ile Cys Cys His Ser Tyr Phe Ser Ile Val Asn Glu Leu Arg Asn 210 632 CAA GAG CCT ACC GGT GGG AAG TAC TAC GAG ATC AGA TTG TTG TAC 676 211 Gln Glu Pro Thr Gly Gly Lys Tyr Tyr Glu Ile Arg Leu Leu Tyr 225 677 CTT CCT TTG CCT GAC GTG GTT ATA 700
26
Tabel 4 Hasil analisis kemiripan sekuen deduksi asam amino MmFSNR dengan program BLASTP (http://www.ebi.ac.uk/BLASTP)
Align. DB:ID Source Length Score Identities Positives EO
1 TR:B9SLS8_RICCO Putative uncharacterized protein OS=Ricinus communis GN=RCOM_0530980 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Ontologies
582 580 55.0 71.0 1.1E-54
2 TR:D7SPY0_VITVI Whole genome shotgun sequence of line PN40024,
scaffold_219.assembly12x (Fragment) OS=Votis vinifera
GN=VIT_00003979001 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequence Literature Protein Sequence Ontologies
584 561 53.0 72.0 1.2E-52
3 TR:D7LPF9_ARALL Putative uncharacterized protein OS=Arabidopsis lyrata subsp. lyrata GN=ARALYDRAFT_484528 00003979001 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Ontologies
588 552 55.0 70.0 1.1E-51
4 SP:Y3739_ARATH Uncharacterized membrane protein
AT3g27390 OS=Arabidopsis thaliana GN= AT3g27390 PE=4
SV=2
Cross-refeerences and related information in:Gene Expression Nucleotide Sequences Genomes Ontologies Literature Protein Sequences
588 537 53.0 69.0 4.1E-50
5 TR:B9GIX9_POPTR Predicted protein OS=Populus trichocarpa
GN=POPTRDRAFT_752632 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Genomes literature Ontologies
586 510 51.0 67.0 3.0E-47
6 TR:D7MJ54_ARALL Putative uncharacterized protein OS=Arabidopsis lyrata subsp. lyrata GN=ARALYDRAFT_493918 00003979001 PE=4 SV=1
Cross-refeerences and related information in:Ontologies
586 494 49.0 67.0 1.5E-45
7 TR:Q84K38_ARATH Putative uncharacterized protein At5g40640 OS=Arabidopsis thaliana GN=At5g40640 PE=2 SV=1
Cross-refeerences and related information in:Ontologies
586 494 50.0 66.0 1.5E-45
8 TR:Q9FM34_ARATH DBJlBAA 95714.1 OS=Arabidopsis thaliana PE=4 SV=1
Cross-refeerences and related information in:Ontologies
595 494 50.0 66.0 1.5E-45
9 TR:C5YAV96_SORBI Putative uncharacterized protein Sb06g020560 OS=Sorghum bicolor GN= Sb06g020560 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Genomes literature Ontologies
586 483 48.0 65.0 2.2E-44
10 TR:B8AVJ1_ORYSI Putative uncharacterized protein OS=Oryza sativa subsp. indica GN= Osl_16361 PE=4 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Genomes literature Ontologies
586 477 48.0 63.0 9.0E-44
11 TR:Q7FAW5_ORYSJ OSJNBa0081L15.3 protein OS=Oryza sativa subsp. japonica GN= OSJNBa0081L15.3 PE=2 SV=1
Cross-refeerences and related information in: Nucleotide Sequences Genomes literature Ontologies
588 477 58.0 63.0 9.4E-33
12 TR:Q2HU72_MEDTR Steroid nuclear receptor, ligand
binding protein OS=Medicago truncantula
GN=MtrDRAFT_AC149208g42v2 PE=4 SV=2
Cross-refeerences and related information in: Nucleotide Sequences Ontologies
582 474 47.0 65.0 2.0E-43
Adapun hasil kesejajaran terhadap deduksi asam amino dari fragmen sisipan yang sudah dihilangkan 132 pasang basa dari primer (MmFSNRcut)