Estimasi Jarak Genetik Galur Jagung Pulut Berbasis Marka
Mikrosatelit dan Korelasinya dengan Karakter Morfologi
Andi Takdir M.1, Hajrial Aswidinnoor2, Trikoesoemaningtyas2, dan Jajah Koswara2 1
Mahasiswa Sekolah Pascasarjana Institut Pertanian Bogor (IPB) 2Dosen Sekolah Pascasarjana Institut Pertanian Bogor (IPB)
Dramaga, Bogor, Jawa Barat
ABSTRACT. Genetic Distance Estimation of Waxy Corn Lines Based on Microsatellite Marker and Its Correlation to Morphology Characters. Maize breeding program for high yielding hybrid varieties requires the availability of pair populations having different heterotic pattern. The present study was aimed to estimate genetic distance among 39 waxy corn lines based on SSRs (Single Sequence Repeats) markers or micro satellites and its correlation with the morphology characters. The research was carried out at ICERI biology molecular laboratory, Maros, South Sulawesi and Cikeumeuh Experimental Farm, Bogor, West Java from January to April 2008. The data were analyzed by PIC (polymorphism information content) and Jaccard’s coefficient. Results showed
that total of 64 alleles were detected with an average allele number of 3.20 alleles per locus, range form 2-5 alleles/locus. The genetic distance values ranged from 0.18 to 0.99 indicating the broad genetic variability among lines. Construction of dendrogram differentiates among lines, and divides the lines in three clusters with coefficient cofenitif value (r) 0.87 indicating a good fit based on genetic similarities and consistent with the pedigree data. Estimate of simple correlation value (r) between genetic distance value and morphology characters of the line performance was positive, suggesting there was relation between both characters. While correlation between similarity matrices SSR-based and similarity matrices morphological-based using goodness of fit criteria is very weak.
Keywords: Waxy corn, genetic distance, micro satellite, correlation
ABSTRAK. Untuk mendapatkan jagung hibrida berpotensi hasil tinggi diperlukan pasangan populasi yang memiliki kelompok heterotik berbeda. Penelitian bertujuan untuk mengetahui jarak genetik 39 galur jagung pulut (waxy corn) berbasis marka SSRs (Single Sequence Repeats) atau mikrosatelit dan korelasinya dengan karakter fenotipik. Penelitian dilaksanakan di laboratorium molekuler Balitsereal, Maros, dan KP Cikeumeuh BB-Biogen, Bogor, pada Januari-April 2008. Tingkat polimorfisme primer dihitung meng-gunakan formula PIC (polymorphism information content) dan tingkat kemiripan genetik dengan koefisien Jaccard. Hasil penelitian menunjukkan bahwa 20 primer yang digunakan memiliki tingkat polimorfisme rata-rata 0,62, sebanyak 64 alel terdeteksi berkisar dari 2-5 alel/lokus dengan rata-rata 3,20 alel/lokus, nilai jarak genetik rata-rata 0,61 dengan kisaran 0,18-0,99. Pengelompokan galur berdasarkan marka SSR sebanyak tiga klaster dengan nilai koefisien kofenitif (r) sebesar 0,87 tergolong baik (good fit) terhadap matrik kemiripan genetik, dengan kode pedigree yang sama untuk masing-masing klaster. Berdasarkan estimasi nilai korelasi sederhana (r) antara nilai jarak genetik dengan karakter morfologi penampilan galur bernilai positif atau terdapat hubungan di antara kedua karakter tersebut. Korelasi antara matrik kemiripan berdasarkan SSRs dengan matrik kemiripan berdasarkan morfologi, berdasarkan kriteria goodness of fit maka nilai korelasi tersebut tergolong sangat lemah. Kata kunci: Jagung pulut, jarak genetik, mikrosatelit, korelasi
K
arakterisasi keragaman genetik koleksi plasmanutfah jagung pulut (waxy corn) yang didukung oleh data molekuler belum pernah dilakukan di Indonesia. Informasi pedigree dan karakter morfologi sangat diperlukan untuk menunjang perakitan varietas hibrida. Dalam program pembentukan jagung hibrida diperlukan pasangan galur murni yang memiliki latar belakang genetik yang jauh agar hibrida yang dihasilkan memiliki tingkat heterosis yang tinggi. Robinson (2000) mengemukakan bahwa metode seleksi yang umum digunakan dalam pembentukan tetua hibrida adalah metode silsilah (pedigree). Ketidaktepatan karakter morfologi menjelaskan hubungan genetik antarindividu adalah akibat adanya interaksi dengan lingkungan. Penggunaan marka molekuler sebagai alat bantu yang langsung melihat perbedaan genetik di antara galur nampaknya merupakan alternatif yang tepat.Marka SSRs (Simple Sequence Repeats) atau mikro-satelit telah digunakan secara ekstensif sebagai marka genetik pada studi genetik jagung, seperti pada konstruksi pemetaan keterpautan gen dan pemetaan QTL (Romero-Severson 1998; Frova et al. 1999), atau analisis keragaman genetik dan evolusi (Senior et al. 1998; Pejic et al. 1998; Lu and Bernardo 2001; Matsuoka 2002). Teknik ini sering pula digunakan dalam mem-bedakan genotipe, evaluasi kemurnian benih, pemetaan gen, sebagai alat bantu seleksi, studi genetik populasi, dan analisis diversitas genetik. Akhir-akhir ini, mikrosatelit lebih banyak digunakan untuk karakterisasi dan pemetaan genetik tanaman, di antaranya jagung, padi, anggur, kedelai, jewawut, gandum, kelapa, dan tomat (Perera et al. 2000; Rivera et al. 1999; dan Tuelat et al. 2000). SSRs memiliki polimorfisme dan resolusi lebih tinggi dibandingkan dengan penanda DNA lainnya, (Tuelat et al. 2000). Primer spesifik hanya mengamplifikasi sekuen-sekuen tertentu, sehingga dapat meningkatkan keakuratan deteksi sekuen DNA yang mirip atau berbeda antarindividu. Karena tingkat keakuratannya tinggi (reproducibility), maka metode SSRs dapat digunakan oleh peneliti berbeda dengan hasil yang relatif sama (Karp et al. 1997). Beberapa pertimbangan lain sehingga marka mikrosatelit banyak digunakan dalam studi
genetik di antaranya adalah: terdistribusi secara melimpah dan merata dalam genom, variabilitasnya sangat tinggi (banyak alel dalam lokus), dan sifatnya yang kodominan dengan lokasi genom yang telah diketahui. Program perakitan varietas hibrida yang berpotensi hasil tinggi membutuhkan pasangan populasi yang memiliki kelompok heterotik yang berbeda. Oleh karena itu, dalam pembentukan varietas hibrida, pemilihan tetua memerlukan perhatian khusus karena untuk mendapat-kan peluang munculnya heterosis pada generasi turunannya (F1) diperlukan tetua penyusun varietas hibrida yang memiliki jarak genetik yang jauh.
Penelitian ini bertujuan untuk mengetahui hubungan kekerabatan atau jarak genetik galur jagung pulut berdasarkan pola pita DNA memanfaatkan marka SSRs (Simple Sequence Repeats), dan korelasi antara matriks rata-rata jarak taksonomi berdasarkan penampilan morfologi dan koefisien kemiripan berdasarkan pola pita DNA berbasis marka SSRs.
BAHAN DAN METODE
Penelitian dilaksanakan di KP Cikeumeuh, Balai Besar Penelitian Pengembangan Bioteknologi dan Sumber Daya Genetik (BB-Biogen) Bogor dan di Laboratorium Biologi Molekuler, Balai Penelitian Tanaman Serealia (Balitsereal) Maros dari Januari hingga April 2008. Materi genetik yang digunakan sebanyak 39 galur jagung pulut koleksi Balitsereal (Tabel 1) digunakan untuk me-ngetahui jarak genetik berdasarkan marka SSRs dan 10 galur hasil seleksi memiliki jarak genetik jauh digunakan untuk pengamatan morfologi.
Primer mikrosatelit yang digunakan sebanyak 20 pasang basa (forward dan reverse) diperoleh dari Research Genetik Inc, dan dari Invitrogen. Primer-primer tersebut diseleksi berdasarkan tingkat polimorfisme (referensi dari CIMMYT) (Tabel 2).
Kegiatan di Laboratorium Isolasi DNA
Biji ditanam sebanyak 10 individu untuk masing-masing galur pada baki menggunakan media tanah. Materi yang digunakan untuk ekstraksi DNA adalah tanaman yang berumur 10-15 HST. Bagian tanaman yang diambil adalah daun muda yang telah membuka sempurna, dari 5-10 individu tanaman dipotong-potong kecil, dicampur, dan kemudian ditimbang 0,4 g/sampel. Kegiatan isolasi, pemurnian dan penentuan konsentrasi DNA mengikuti prosedur George et al. (2004) dengan beberapa modifikasi. Kuantitas dan kualitas DNA hasil ekstraksi
diukur berdasarkan standar -DNA melalui proses elektroforesis horizontal menggunakan gel agarose 0,9%.
Amplifikasi dan Visualisasi Pola Pita DNA Proses amplifikasi, pemisahan DNA pada PAGE (Polyacrylamide Gel Electrophoresis), juga mengikuti prosedur George et al. (2004) dengan beberapa modifikasi. Modifikasi dilakukan sesuai dengan kondisi laboratorium karena penggunaan enzim dari sumber yang berbeda dan juga untuk efisiensi penggunaan bahan tanpa mengurangi kualitas visualisasi DNA. Inbrida yang menghasilkan tingkat heterosigositas >20% dan missing data >10% dikeluarkan. Denaturasi poliakrilamid gel elektroforesis dengan 4,5% akrilamid Tabel 1. Materi genetik galur-galur jagung pulut (waxy corn) yang
digunakan.
No. Pedigree Populasi asal Warna/tipe biji 1. PTBC4-7-5-B Tuxpeno/Pulut Takalar Putih/Semi flint 2. PTBC4-7-11-B Tuxpeno/Pulut Takalar Putih/Semi flint 3. PTBC4-17-1-B Tuxpeno/Pulut Takalar Putih/Semi flint 4. PTBC4-9-3-B Tuxpeno/Pulut Takalar Putih/Semi flint 5. PTBC4-9-5-B Tuxpeno/Pulut Takalar Putih/Semi flint 6. PTBC4-10-1-B Tuxpeno/Pulut Takalar Putih/Semi flint 7. PTBC4-10-7-B Tuxpeno/Pulut Takalar Putih/Semi flint 8. PTBC4-11-6-B Tuxpeno/Pulut Takalar Putih/Semi flint 9. PTBC4-11-8-B Tuxpeno/Pulut Takalar Putih/Semi flint 10. PTBC4-12-3-B Tuxpeno/Pulut Takalar Putih/Semi flint 11. PTBC4-12-12-B Tuxpeno/Pulut Takalar Putih/Semi flint 12. PTBC4-13-6-B Tuxpeno/Pulut Takalar Putih/Semi flint 13. PTBC4-14-7-B Tuxpeno/Pulut Takalar Putih/Semi flint 14. PTBC4-14-12-B Tuxpeno/Pulut Takalar Putih/Semi flint 15. PTBC4-15-1-B Tuxpeno/Pulut Takalar Putih/Semi flint 16. PTBC4-15-4-B Tuxpeno/Pulut Takalar Putih/Semi flint 17. PTBC4-16-2-B Tuxpeno/Pulut Takalar Putih/Semi flint 18. PTBC4-16-8-B Tuxpeno/Pulut Takalar Putih/Semi flint 19. PTBC4-18-43-B Tuxpeno/Pulut Takalar Putih/Semi flint 20. PTBC4-20-1-B Tuxpeno/Pulut Takalar Putih/Semi flint 21. PTBC4-21-2-B Tuxpeno/Pulut Takalar Putih/Semi flint 22. PTBC4-21-4-B Tuxpeno/Pulut Takalar Putih/Semi flint 23. PTBC4-22-2-B Tuxpeno/Pulut Takalar Putih/Semi flint 24. MSP2(10)-82-1-B Populasi Pulut Sul-Sel Putih/flint 25. MSP2(10)-90-1-B Populasi Pulut Sul-Sel Putih/flint 26. MSP2(10)-95-1-B Populasi Pulut Sul-Sel Putih/flint 27. MSP2(10)-96-1-B Populasi Pulut Sul-Sel Putih/flint 28. MSP2(10)-103-1-B Populasi Pulut Sul-Sel Putih/flint 29. MSP2(10)-113-1-B Populasi Pulut Sul-Sel Putih/flint 30. MSP2(10)-120-1-B Populasi Pulut Sul-Sel Putih/flint 31. MSP2(10)-122-1-B Populasi Pulut Sul-Sel Putih/flint 32. MSP2(10)-125-2-B Populasi Pulut Sul-Sel Putih/flint 33. MSP2(10)-145-1-B Populasi Pulut Sul-Sel Putih/flint 34. MSP2(10)-149-1-B Populasi Pulut Sul-Sel Putih/flint 35. MSP2(10)-163-1-B Populasi Pulut Sul-Sel Putih/flint 36. MSP2(10)-169-1-B Populasi Pulut Sul-Sel Putih/flint 37. MSP2(10)-182-1-B Populasi Pulut Sul-Sel Putih/flint 38. MSP2(10)-187-1-B Populasi Pulut Sul-Sel Putih/flint 39. MSP2(10)-192-1-B Populasi Pulut Sul-Sel Putih/flint
protokol Bio-Rad Laboratories Inc. Hercules, CA, USA. Pita-pita DNA dideteksi melalui proses silver staining menggunakan protokol sistem sekuensing DNA Promega Silver Sequence.
Skoring Data
Alel-alel diberi label berdasarkan posisi relatif dari pita-pita terhadap fragmen öX174/Hin f I. Data diskor dalam bentuk data biner, jika ada pita ditulis satu (1), tidak ada pita ditulis nol (0). Pita-pita yang kabur dan terlalu sukar untuk diskoring diberi tanda missing data dengan angka sembilan (9). Jika ada galur yang menghasilkan banyak pita maka pita yang paling jelas diskor ‘1’ sedangkan yang lainnya diskor ‘9’.
Analisis Data Genotipik
Analisis data molekuler dilakukan berdasarkan hasil skoring pita DNA yang muncul pada plate, hasil skoring dalam bentuk data biner. Tingkat polimorfisme (PIC = Polimorphism Information Content) dari primer yang digunakan dihitung untuk masing-masing marka SSRs (Smith et al. 1997), dengan formula:
n if
PIC
1 21
i = 1, 2, 3,………n ,di mana
f
i2 adalah frekuensi alel ke-i.Estimasi Kekerabatan Genetik dan Analisis Klaster Tingkat kemiripan genetik (GS=genetic similarity) diestimasi dengan menggunakan koefisien Jaccard (Rohlf 2000) dengan formula:
n
u
m
S
di mana:m = jumlah pita (alel) DNA yang sama posisinya, n = total pita (alel) DNA,
u = jumlah pita (alel) DNA yang tidak sama posisinya. Kemiripan genetik dianalisis dengan menggunakan software NTSYS-pc versi 2.1 (Rohlf 2000). Analisis matriks jarak genetik diperoleh dari hasil analisis kemiripan genetik (Lee 1998), dengan formula:
S = 1 – GS di mana:
S = jarak genetik,
GS = Kemiripan genetik (genetic similarity).
Analisis Boot-Strapping dilakukan untuk mengetahui tingkat kepercayaan pengelompokan dengan menggunakan program WinBoot. Koefisien korelasi kofenetik (r) juga dihitung dan dilanjutkan dengan uji Mantel (Mantel 1967) untuk melihat goodness of fit dari hasil analisis klaster. Principal Coordinate Analysis (PCA) Tabel 2. Sekuen dari 20 marka mikrosatelit yang digunakan dalam penelitian.
No. Primer Bin No. Repeat type Primer sequence
1 phi109275 1.03 AGCT CGGTTCATGCTAGCTCTGC // GTTGTGGCTGTGGTGGTG 2 phi96100 2.01 ACCT AGGAGGACCCCAACTCCTG // TTGCACGAGCCATCGTAT 3 phi374118 3.02 ACC TACCCGGACATGGTTGAGC // TGAAGGGTGTCCTTCCGAT
4 phi079 4.05 AGATG TGGTGCTCGTTGCCAAATCTACGA // GCAGTGGTGGTTTCGAACAGACAA 5 phi109188 5.03 AAAG AAGCTCAGAAGCCGGAGC // GGTCATCAAGCTCTCTGATCG
6 phi299852 6.07 AGC GATGTGGGTGCTACGAGCC // AGATCTCGGAGCTCGGCTA 7 phi328175 7.04 AGG GGGAAGTGCTCCTTGCAG // CGGTAGGTGAACGCGGTA 8 phi233376 8.09 CCG CCGGCAGTCGATTACTCC // CGAGACCAAGAGAACCCTCA
9 phi065 9.03 CACTT AGGGACAAATACGTGGAGACACAG // CGATCTGCACAAAGTGGAGTAGTC 10 umc1196 10.07 CACACG CGTGCTACTACTGCTACAAAGCGA // AGTCGTTCGTGTCTTCCGAAACT 11 phi109642 2.03-2.04 ACGG CTCTCTTTCCTTCCGACTTTCC // GAGCGAGCGAGAGAGATCG 12 phi101049 2.10 AGAT CCGGGAACTTGTTCATCG // CCACGTCCATGATCACACC 13 phi102228 3.06 AAGC ATTCCGACGCAATCAACA // TTCATCTCCTCCAGGAGCCTT
14 phi093 4.08 AGCT AGTGCGTCAGCTTCATCGCCTACAAG // AGGCCATGCATGCTTGCAACAATGGATACA 15 umc1153 5.09 (TCA)4 CAGCATCTATAGCTTGCTTGCATT // TGGGTTTTGTTTGTTTGTTTGTTG
16 phi423796 6.01 AGATG CACTACTCGATCTGAACCACCA // CGCTCTGTGAATTTGCTAGCTC 17 phi114 7.03 GCCT CCGAGACCGTCAAGACCATCAA // AGCTCCAAACGATTCTGAACTCGC 18 phi420701 8.00 CCG GATGTTTCAAAACCACCCAGA // ATGGCACGAATAGCAACAGG 19 phi448880 9.06-9.07 AAG CGATCCGGAGGAGTTCCTTA // CCATGAACATGCCAATGC 20 phi96342 10.02 ATCC GTAATCCCACGTCCTATCAGCC // TCCAACTTGAACGAACTCCTC * Sumber: Applied Biotechnology Center at CIMMYT, Mexico
(Dillon and Goldstein 1984) dilakukan untuk mengetahui posisi relatif dari inbrida yang dianalisis pada ruang dua dimensi.
Kegiatan di Lapangan
Materi genetik yang terseleksi memiliki nilai jarak genetik jauh berdasarkan hasil analisis keragaman genetik di laboratorium sebanyak 10 galur di tanam di lapangan masing-masing galur pada barisan terpisah meng-gunakan jarak tanam 0,75 m x 0,20 m panjang 5 m dan diulang sebanyak tiga kali, benih sebelum ditanam diberikan perlakuan dengan metalaksil untuk mencegah penyakit bulai (Perenosclerospora maydis), saat tanam lubang tanam diberi karbofuran untuk mencegah serangan ulat bibit, penanaman dilakukan dengan menggunakan sistem tugal, pemupukan pertama diberikan saat tanaman berumur tujuh hari setelah tanam dengan takaran pemupukan 150 kg/ha urea, 200 kg/ha SP36, dan 100 kg/ha KCl, dengan menugal di samping tanaman, pemupukan kedua diberikan saat tanaman berumur 30 hari setelah tanam dengan takaran pemupukan 150 kg/ha urea.
Pengamatan Data Molekuler
Analisis data molekuler dilakukan berdasarkan hasil skoring pita DNA yang muncul pada plate. Pita DNA diberi skor berdasarkan penampilan pita DNA ditransformasi ke dalam kode data biner dengan cara: jika ada pita diberi skor satu (1) dan jika tidak ada pita diberi skoring nol (0). Pita yang tidak sempurna dan tidak jelas diberi skor 9 (missing data). Jika ada galur yang menghasilkan banyak pita maka pita yang paling jelas diberi skor ‘1’ sedangkan yang lainnya diberi skor ‘9’. Data pada kolom
menunjukkan inbrida sedangkan baris menunjukkan lokus SSR.
Pengamatan Data Morfologi
Pengamatan utama morfologi dilakukan meliputi 10 variabel pengamatan meliputi: tinggi tanaman (cm), tinggi letak tongkol (cm), diameter batang (cm), lingkar batang (cm), indeks luas daun (cm), panjang daun (cm), lebar daun (cm), kandungan klorofil daun di bawah tongkol (unit), kandungan klorofil daun di tongkol (unit), kandungan klorofil daun bendera (unit) dengan pengukuran kandungan klorofil menggunakan alat SPAD 502 model MINOLTA.
HASIL DAN PEMBAHASAN
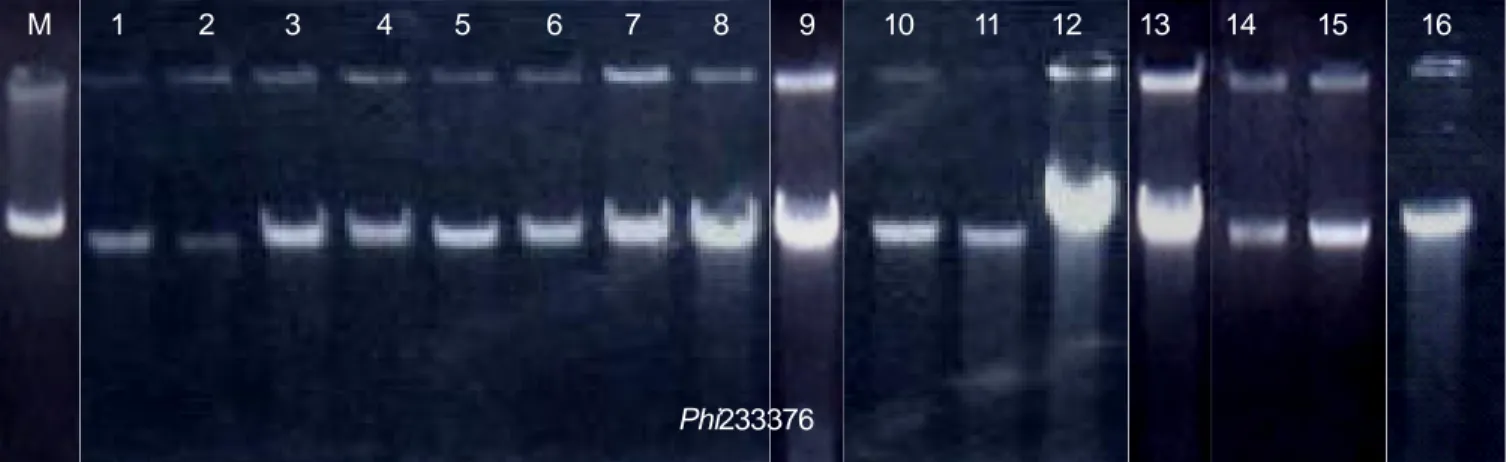
Profil Marka MikrosatelitKegiatan ini merupakan studi untuk memprediksi galur jagung pulut yang kemungkinan menghasilkan heterosis tinggi. Dari total 20 primer yang digunakan dalam penelitian semuanya dapat teramplifikasi. Jumlah primer yang disekuensing adalah 20 primer. Untuk meng-hasilkan data dengan validasi tinggi maka primer yang menghasilkan missing data >10% dikeluarkan. Hal yang sama juga dilakukan terhadap galur-galur yang meng-hasilkan tingkat heterosigositas >20%. Pada kegiatan ini tidak terdapat primer yang menghasilkan missing data >10%, tetapi terdapat satu galur dalam kondisi hetero-sigositas >20%, yaitu MSP2(10)-105-1-B sehingga tidak diikutkan dalam analisis lebih lanjut. Gambar 1 adalah salah satu lokus SSRs yang memperlihatkan variasi genetik dari inbrida yang dikarakterisasi.
Gambar 1. Visualisasi pola pita DNA menggunakan marka SSR phi233376 melalui elektroforesis vertikal 4,5% PAGE (Polyacrylamide Gel Electrophoresis).
5
M 1 2 3 4 6 7 8 9 10 11 12 13 14 15 16
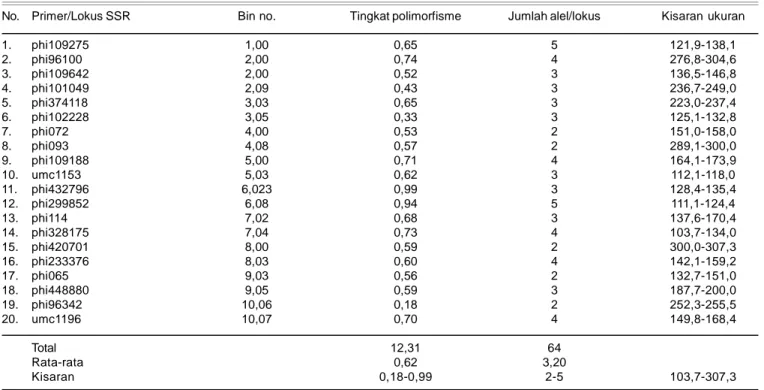
Profil data 20 marka mikrosatelit untuk galur jagung pulut menunjukkan tingkat polimorfisme 0,18-0,99 dengan rata-rata 0,62. Total alel yang diperoleh sebanyak 64 berkisar dari 2-5 dengan nilai rata-rata 3,20 alel/lokus (Tabel 3). Semakin tinggi keragaman genetik yang diperoleh dari sejumlah koleksi galur maka akan lebih leluasa melakukan eksploitasi heterosis. Koleksi galur yang dikarakterisasi ini menunjukkan variasi genetik yang cukup beragam berdasarkan data pedigree. Hal ini menegaskan bahwa materi genetik yang dikarakterisasi mempunyai variasi yang tinggi. Tingginya tingkat poli-morfisme juga dipengaruhi oleh tingkat polipoli-morfisme marka SSRs yang digunakan. Oleh karena itu, pemilihan marka polimorfisme yang tinggi mempunyai kontribusi terhadap nilai PIC (Polimorphism Information Content). Penggunaan primer yang lebih banyak dengan tingkat polimorfisme tinggi diharapkan dapat memberikan pengelompokan yang lebih komprehensif. Chen et al. (2000), menyatakan bahwa data molekuler sangat bergantung pada pemilihan primer yang digunakan.
Dendrogram Galur Jagung Pulut
Analisis keragaman genetik berguna untuk mengetahui pola pengelompokan populasi genotipe yang dimiliki dan untuk mengetahui karakter penciri setiap kelompok genotipe yang terbentuk, sehingga dapat digunakan dalam kegiatan seleksi tetua untuk perakitan varietas
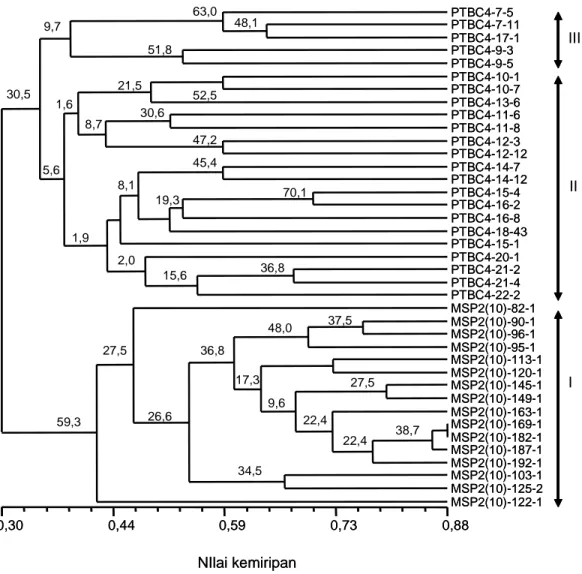
unggul baru. Koefisien korelasi kofenetik (r) sebesar 0,88 tergolong good fit untuk kelompok galur jagung pulut yang ditangani. Hal ini menggambarkan bahwa tingkat akurasi pengelompokan secara genetik dengan jumlah primer yang digunakan sangat akurat atau semua primer polimorfis. Pejic et al. (1998) mengemukakan, nilai r menggambarkan akurasi pengelompokan secara genotipik, yang dapat dihasilkan berdasarkan estimasi kemiripan genetik di antara galur yang dikarakterisasi dengan jumlah primer yang digunakan. Semakin banyak primer polimorfis yang digunakan maka nilai r akan semakin besar. Hasil dendrogram menunjukkan semua galur dapat dibedakan antara yang satu dengan lainnya (Gambar 3).
Dalam program hibridisasi, besarnya keragaman genetik antarpopulasi calon tetua perlu diperhatikan. Semakin luas tingkat keragaman antarcalon tetua semakin besar peluang terbentuknya segregan potensial. Walaupun demikian, pertimbangan tidak hanya didasarkan atas besarnya diversitas genetik, tetapi juga potensi hasil, dan ketahanan terhadap hama penyakit. Dengan terjadinya tingkat diversitas genetik yang luas, dan adanya pencirian suatu kluster oleh karakteristik tanaman tertentu, maka jumlah kombinasi persilangan yang mungkin dilakukan semakin besar, sehingga peluang untuk memperoleh segregan yang mempunyai tingkat heterosis tinggi menjadi semakin besar.
Tabel 3. Profil data marka mikrosatelit hasil karakterisasi pada galur jagung pulut.
No. Primer/Lokus SSR Bin no. Tingkat polimorfisme Jumlah alel/lokus Kisaran ukuran
1. phi109275 1,00 0,65 5 121,9-138,1 2. phi96100 2,00 0,74 4 276,8-304,6 3. phi109642 2,00 0,52 3 136,5-146,8 4. phi101049 2,09 0,43 3 236,7-249,0 5. phi374118 3,03 0,65 3 223,0-237,4 6. phi102228 3,05 0,33 3 125,1-132,8 7. phi072 4,00 0,53 2 151,0-158,0 8. phi093 4,08 0,57 2 289,1-300,0 9. phi109188 5,00 0,71 4 164,1-173,9 10. umc1153 5,03 0,62 3 112,1-118,0 11. phi432796 6,023 0,99 3 128,4-135,4 12. phi299852 6,08 0,94 5 111,1-124,4 13. phi114 7,02 0,68 3 137,6-170,4 14. phi328175 7,04 0,73 4 103,7-134,0 15. phi420701 8,00 0,59 2 300,0-307,3 16. phi233376 8,03 0,60 4 142,1-159,2 17. phi065 9,03 0,56 2 132,7-151,0 18. phi448880 9,05 0,59 3 187,7-200,0 19. phi96342 10,06 0,18 2 252,3-255,5 20. umc1196 10,07 0,70 4 149,8-168,4 Total 12,31 64 Rata-rata 0,62 3,20 Kisaran 0,18-0,99 2-5 103,7-307,3
Angka yang berada di atas garis (Gambar 3) me-nunjukkan tingkat kepercayaan pengelompokan. Semakin tinggi tingkat kepercayaan pengelompokan semakin kuat kemiripan genetik galur dalam kelompok tersebut. Dengan demikian, peluang inbreeding akan semakin tinggi jika dilakukan persilangan satu dengan yang lain dalam kelompok pada tingkat kepercayaan pengelompokan yang tinggi. Oleh karena itu, persilangan antargenotipe dalam kelompok yang sama tersebut disarankan untuk dihindari. Menurut Warburton et al. (2005), galur-galur tidak terklaster berdasarkan fenotipe, adaptasi lingkungan, tipe atau warna biji, umur panen, atau respon heterotik, tapi galur-galur yang berkerabat secara pedigree biasanya berada pada klaster yang sama. Pada galur jagung pulut, pasangan-pasangan persilangan yang harus dihindari adalah di antara galur yang terdapat dalam satu kelompok.
Dari 39 galur jagung pulut yang menggunakan 20 marka SSRs, terdapat tiga klaster dan dapat dibedakan, klaster yang sama dicirikan oleh galur dengan kode pedigree hampir sama (Gambar 2). Hal tersebut mem-perlihatkan bahwa galur tersebut dibentuk dari populasi yang sama, sehingga tingkat kekerabatannya lebih dekat. Klaster pertama merupakan kelompok kedua terbesar, terdiri atas16 galur dan didominasi oleh galur dengan inisial pedigree MSP2. Klaster II merupakan kelompok terbesar, terdiri atas 18 galur dan didominasi oleh galur dengan inisial pedigree PTBC4. Klaster III merupakan kelompok terkecil, terdiri atas lima galur dan didominasi oleh galur dengan inisial pedigree PTBC4. Nilai matriks jarak genetik cukup tinggi. Hal ini mengindikasikan materi genetik berasal dari materi populasi dasar dengan variasi genetik cukup luas dan memberikan peluang besar untuk mendapatkan pasangan tetua hibrida potensial. Gambar 2. Dendrogram 39 galur jagung pulut menggunakan 20 marka SSRs dan dikonstruksi berdasarkan koefisien kemiripan Jaccard.
NIlai kemiripan 0,30 0,44 0,59 0,73 0,88 PTBC4-7-5 PTBC4-7-11 PTBC4-17-1 PTBC4-9-3 PTBC4-9-5 PTBC4-10-1 PTBC4-10-7 PTBC4-13-6 PTBC4-11-6 PTBC4-11-8 PTBC4-12-3 PTBC4-12-12 PTBC4-14-7 PTBC4-14-12 PTBC4-15-4 PTBC4-16-2 PTBC4-16-8 PTBC4-18-43 PTBC4-15-1 PTBC4-20-1 PTBC4-21-2 PTBC4-21-4 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-90-1 MSP2(10)-96-1 MSP2(10)-95-1 MSP2(10)-113-1 MSP2(10)-120-1 MSP2(10)-145-1 MSP2(10)-149-1 MSP2(10)-163-1 MSP2(10)-169-1 MSP2(10)-182-1 MSP2(10)-187-1 MSP2(10)-192-1 MSP2(10)-103-1 MSP2(10)-125-2 MSP2(10)-122-1 45,4 30,5 9,7 63,0 48,1 51,8 52,5 21,5 5,6 1,9 30,6 8,7 47,2 59,3 27,5 15,6 2,0 36,8 70,1 37,5 27,5 38,7 34,5 26,6 22,4 22,4 9,6 17,3 48,0 36,8 19,3 8,1 1,6 III II I NIlai kemiripan 0,30 0,44 0,59 0,73 0,88 PTBC4-7-5 PTBC4-7-11 PTBC4-17-1 PTBC4-9-3 PTBC4-9-5 PTBC4-10-1 PTBC4-10-7 PTBC4-13-6 PTBC4-11-6 PTBC4-11-8 PTBC4-12-3 PTBC4-12-12 PTBC4-14-7 PTBC4-14-12 PTBC4-15-4 PTBC4-16-2 PTBC4-16-8 PTBC4-18-43 PTBC4-15-1 PTBC4-20-1 PTBC4-21-2 PTBC4-21-4 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-90-1 MSP2(10)-96-1 MSP2(10)-95-1 MSP2(10)-113-1 MSP2(10)-120-1 MSP2(10)-145-1 MSP2(10)-149-1 MSP2(10)-163-1 MSP2(10)-169-1 MSP2(10)-182-1 MSP2(10)-187-1 MSP2(10)-192-1 MSP2(10)-103-1 MSP2(10)-125-2 MSP2(10)-122-1 45,4 30,5 9,7 63,0 48,1 51,8 52,5 21,5 5,6 1,9 30,6 8,7 47,2 59,3 27,5 15,6 2,0 36,8 70,1 37,5 27,5 38,7 34,5 26,6 22,4 22,4 9,6 17,3 48,0 36,8 19,3 8,1 1,6 III II I
Posisi Relatif Galur Jagung Pulut
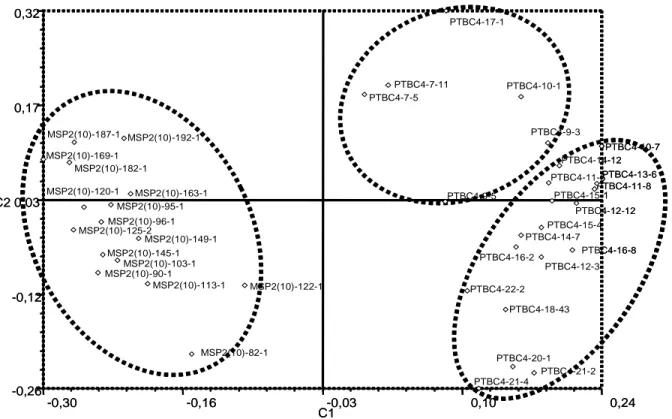
Posisi relatif dari masing-masing galur yang dianalisis pada ruang dua dimensi (Gambar 3) yang memberikan peluang untuk memasangkan galur satu dengan lainnya lebih diperketat lagi, di mana galur yang menggerombol bersama dihindari untuk dipasangkan.
Secara umum, berdasarkan analisis PCA (Principal Coordinate Analysis) terbentuk tiga kelompok galur yang sesuai dengan pengelompokkan pada dendogram. Galur PTBC4-7-5, PTBC4-7-11 dan PTBC4-17-1 dapat direkomendasikan untuk dipasangkan dengan 26 galur jagung pulut lain di luar kelompoknya. Kelompok-kelompok yang terbentuk disebut Kelompok-kelompok heterotik. Artinya, galur yang berada dalam kelompok yang sama jika disilangkan dengan galur di luar kelompoknya akan memberi peluang besar untuk memunculkan heterosis. Oleh karena itu, dalam proses mencari pasangan tetua hibrida potensial, dengan bantuan informasi marka molekuler, maka pasangan-pasangan tidak potensial dapat dihindari lebih awal untuk efisiensi waktu, tenaga, dan dana. Pengujian pasangan-pasangan heterotik di lapangan lebih difokuskan kepada potensi genetik dari inbrida-inbrida yang terpilih.
Analisis gerombol juga digunakan untuk mengetahui hubungan kekerabatan genotipe yang diuji (Sumertajaya et al. 2003). Analisis biplot digunakan untuk mengetahui peubah penciri setiap gerombol genotipe. Sumertajaya et al. (2003). juga menyebutkan bahwa analisis biplot dapat digunakan untuk mengetahui kemiripan atau kedekatan relatif antarobyek pengujian (gerombol genotipe) dan keragaman setiap peubah.
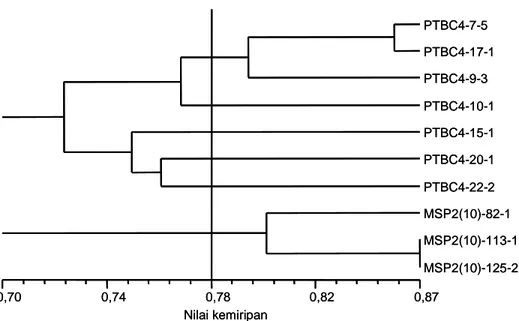
Korelasi antara Karakter Genotifik dan Morfologi Untuk melihat keselarasan antara morfologi dengan pola pita SSRs, dilakukan pengujian goodness of fit dengan menggunakan analisis korelasi antara matriks jarak morfologi dengan matriks tingkat kemiripan genetik. Analisis dilakukan pada 10 genotipe, dendrogram 10 jagung pulut berdasarkan pola pita SSRs disajikan pada Gambar 4. Dengan membandingkan antara dendrogram berdasarkan morfologi (Gambar 5) dan pola pita SSRs (Gambar 4) terhadap 10 genotipe yang dianalisis diperoleh hasil pengelompokan yang tidak konsisten. Genotipe dengan kode pedigree PTBC4 dan MSP2 berdasarkan morfologi tidak berada pada satu kelompok, sedang berdasarkan pola pita SSRs me-ngelompok masing-masing menjadi satu kelompok. Gambar 3. Posisi relatif 39 galur jagung pulut menggunakan 20 marka SSRs berdasarkan hasil analisis PCoA dua dimensi.
C1 -0,30 -0,16 -0,03 0,10 0,24 C2 -0,26 -0,12 0,03 0,17 0,32 PTBC4-17-1 PTBC4-10-7 PTBC4-11-8 PTBC4-12-3 PTBC4-13-6 PTBC4-16-2 PTBC4-16-8 PTBC4-18-43 PTBC4-20-1 PTBC4-21-2 PTBC4-21-4 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-90-1 MSP2(10)-95-1 MSP2(10)-96-1 MSP2(10)-103-1 MSP2(10)-113-1 MSP2(10)-120-1 MSP2(10)-122-1 MSP2(10)-125-2 MSP2(10)-145-1 MSP2(10)-149-1 MSP2(10)-163-1 MSP2(10)-169-1 MSP2(10)-182-1 MSP2(10)-187-1 MSP2(10)-192-1 PTBC4-7-5 PTBC4-7-11 PTBC4-9-3 PTBC4-9-5 PTBC4-10-1 PTBC4-11-6 PTBC4-12-12 PTBC4-14-7 PTBC4-14-12 PTBC4-15-1 PTBC4-15-4 C1 -0,30 -0,16 -0,03 0,10 0,24 C2 -0,26 -0,12 0,03 0,17 0,32 PTBC4-17-1 PTBC4-10-7 PTBC4-11-8 PTBC4-12-3 PTBC4-13-6 PTBC4-16-2 PTBC4-16-8 PTBC4-18-43 PTBC4-20-1 PTBC4-21-2 PTBC4-21-4 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-90-1 MSP2(10)-95-1 MSP2(10)-96-1 MSP2(10)-103-1 MSP2(10)-113-1 MSP2(10)-120-1 MSP2(10)-122-1 MSP2(10)-125-2 MSP2(10)-145-1 MSP2(10)-149-1 MSP2(10)-163-1 MSP2(10)-169-1 MSP2(10)-182-1 MSP2(10)-187-1 MSP2(10)-192-1 PTBC4-7-5 PTBC4-7-11 PTBC4-9-3 PTBC4-9-5 PTBC4-10-1 PTBC4-11-6 PTBC4-12-12 PTBC4-14-7 PTBC4-14-12 PTBC4-15-1 PTBC4-15-4
Fenomena perbedaan kelompok beberapa genotipe antara dendrogram berdasarkan morfologi dan pola pita DNA tersebut kemungkinan terjadi karena persentase genom yang terwakili masih sedikit atau masing-masing penanda mengungkap daerah genom yang berbeda. Apabila kedua matriks jarak taksonomi dikorelasikan dengan matriks tingkat kemiripan berdasarkan pola pita DNA (Tabel 4) dan diuji dengan statistik Z Mantel dihasilkan korelasi (r) yang digunakan
untuk melihat kesesuaian atau keselarasan dua dendrogram. Korelasi antara matriks kemiripan berdasarkan pola pita DNA dengan matriks kemiripan berdasarkan morfologi data kualitatif biner adalah 0, 111. Berdasarkan kriteria goodness of fit maka nilai korelasi tersebut tergolong sangat lemah. Dari nilai korelasi tersebut dapat dikatakan bahwa penggunaan data kuantitatif sangat kecil peranannya dalam pengelompok-kan genotipe, sehingga data kuantitatif dapat diabaipengelompok-kan.
Gambar 4. Dendrogram 10 galur jagung pulut (waxy corn) hasil analisis klaster berdasarkan pola pita DNA dengan metode UPGMA menggunakan 20 praimer SSRs. Nilai kemiripan 0,70 0,74 0,78 0,82 0,87 PTBC4-7-5 PTBC4-17-1 PTBC4-9-3 PTBC4-10-1 PTBC4-15-1 PTBC4-20-1 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-113-1 MSP2(10)-125-2 Nilai kemiripan 0,70 0,74 0,78 0,82 0,87 PTBC4-7-5 PTBC4-17-1 PTBC4-9-3 PTBC4-10-1 PTBC4-15-1 PTBC4-20-1 PTBC4-22-2 MSP2(10)-82-1 MSP2(10)-113-1 MSP2(10)-125-2 Nilai kemiripan 0,81 0,98 1,16 1,34 1,52 PTBC4-7-5 PTBC4-17-1 PTBC4-10-1 PTBC4-20-1 PTBC4-22-2 MSP2(10)-113-1 PTBC4-9-3 PTBC4-15-1 MSP2(10)-82-1 MSP2(10)-125-2 Nilai kemiripan 0,81 0,98 1,16 1,34 1,52 PTBC4-7-5 PTBC4-17-1 PTBC4-10-1 PTBC4-20-1 PTBC4-22-2 MSP2(10)-113-1 PTBC4-9-3 PTBC4-15-1 MSP2(10)-82-1 MSP2(10)-125-2
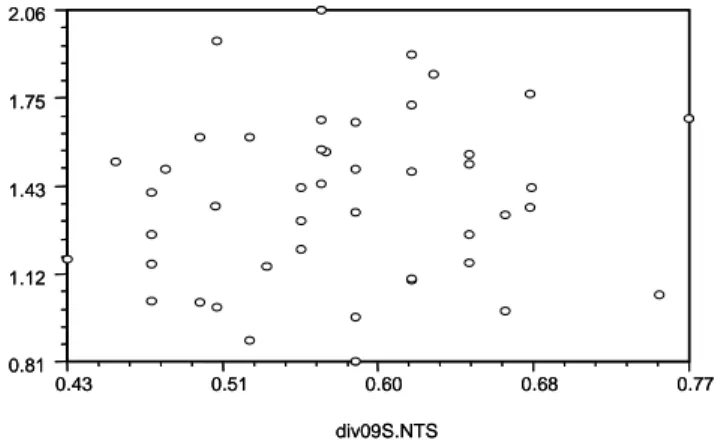
Nilai korelasi sangat lemah diperoleh karena membandingkan antara tingkat kemiripan dengan jarak. Kemiripan genetik menurut Nei (1987) adalah merupakan kebalikan dari jarak genetik yang secara luas menunjukkan kemiripan sifat dari dua aksesi tanaman. Ukuran jarak genetik dan kemiripan genetik dari dua aksesi merupakan kovarian dan frekuensi alel seluruh sifat yang diamati (Smith 1984). Diagram hubungan antara matriks jarak taksonomi dengan matriks tingkat kemiripan disajikan pada Gambar 6.
KESIMPULAN
1. Rata-rata tingkat polimorfisme 20 marka SSRs yang digunakan adalah 0,62 dan total alel 64 dengan rata-rata 3,20 alel/lokus dan nilai koefisien korelasi kofenitik (r) 0,87 tergolong good fit pada kelompok galur jagung pulut yang ditangani.
2. Pengelompokan galur berdasarkan marka SSRs diperoleh tiga klaster atau kelompok heterotik, dengan satu kelompok memiliki satu kelompok pedigree mengelompok tersendiri, namun galur lain dengan pedigree yang mirip terbagi menjadi dua kelompok.
3. Pengelompokan galur berdasarkan marka SSRs dan pengelompokan berdasarkan kemiripan morfologi memiliki nilai korelasi sangat lemah.
4. Terdapat empat galur yang dapat direkomendasi-kan untuk dipasangdirekomendasi-kan dengan galur lainnya adalah: PTBC4-7-5-BB, PTBC4-7-11-BB, PTBC4 -17-1-BB, dan PTBC4-9-3-BB.
DAFTAR PUSTAKA
Allard, R. W. 1960. Principle of plant breeding. John Wiley and Sons, Inc. New York. 485 p.
Birchler, J.A., D.L. Auger, and N.C. Riddle. 2003. In search of molecular basis of heterosis. Plant Cell 15(10):2236-2240. Chahal, G. S. dan S. S. Gosal. 2002. Principles and procedures of
plant breeding biotechnological and convencional approaches. Narosa Publ. House. New Delhi, India. 604 p. Chen, Z., W. Song, and A. Warren. 2000. Studies on six Euplotes
spp. (Ciliophora: Hypotrichida) using RAPD fingerprinting, including a comparison with morphometric analyses. Acta. Protozool. 39:209-216.
Dillon, W.R. and M. Goldstein. 1984. Multivariate analysis methods and applications. John Willey and Sons. p. 581.
Frova, C., P. Krajewski, N. Di Fonzo, M. Villa, and M. Sari-Gorla. 1999. Genetic analysis of drought tolerance in maize by molecular markers. I. Yield components. Theor. Appl. Genet. 99:280-288. div09S.NTS 0.43 0.51 0.60 0.68 0.77 0.81 1.12 1.43 1.75 2.06 div09S.NTS 0.43 0.51 0.60 0.68 0.77 0.81 1.12 1.43 1.75 2.06
Gambar 6. Diagram pencar korelasi antara matrik jarak taksonomi berdasarkan morfologi dengan matrik kemiripan genetik berdasarkan pola pita DNA dari 10 genotip jagung pulut (waxy corn).
Tabel 4. Matrik rata-rata jarak taksonomi di antara genotipe jagung pulut berdasarkan penampilan morfologi (di bawah diagonal) dan matrik tingkat kemiripan genetik berdasarkan pola pita DNA (di atas diagonal).
Genotipe 1 2 3 4 5 6 7 8 9 10 PTBC4-7-5BB 0,571 0,857 0,380 0,428 0,428 0,095 0,750 0,228 0,619 PTBC4-17-1BB 1,045 0,095 0,857 0,857 0,428 0,380 0,250 0,746 0,904 PTBC4-9-3BB 1,509 1,355 0,857 0,380 0,142 0,619 0,750 0,228 0,666 PTBC4-10-1BB 1,492 1,157 1,259 0,619 0,380 0,904 0,001 0,987 0,142 PTBC4-15-1BB 1,406 1,542 9,633 1,719 0,380 0,619 0,500 0,228 0,428 PTBC4-20-1BB 1,155 1,020 1,206 1,055 1,659 0,619 0,250 0,228 0,666 PTBC4-22-2BB 1,761 1,336 1,097 1,819 1,484 1,102 0,001 0,710 0,142 MSP2(10)-82-1BB 1,946 1,518 1,001 1,171 1,490 1,555 1,329 0,101 0,001 MSP2(10)-113-1BB 1,562 1,361 1,668 1,146 2,058 1,439 1,828 1,425 0,674 MSP2(10)125-2BB 1,900 1,606 1,016 1,425 1,260 1,606 1,309 2,875 1,673 (1) PTBC4-7-5BB, (2) PTBC4-17-1BB, (3) PTBC4-9-3BB, (4) PTBC4-10-1BB, (5) PTBC4-15-1BB, (6) PTBC4-20-1BB, (7) PTBC4-22-2BB, (8) MSP2(10)-82-1BB, (9) MSP2(10)-113-1BB, dan (10) MSP2(10)125-2BB
George, M.L.C., E. Regalado, M. Warburton, S. Vasal, and D. Hoisington. 2004. Genetic diversity of maize inbred lines in relation to downy mildew. Euphytica 135:145-155.
Karp A., S. Kresnovich, K.V. Bhat, W.G. Ayad, and T. Hodgkin. 1997. Moleculer tools in plant genetic resources conservation: a guide to the technologies. IPGR. Rome. 46 p.
Lee, M. 1998. DNA markers for detecting genetic relationship among germplasm revealed for establishing heterotic groups. Presented at the Maize Training Course, CIMMYT, Texcoco, Mexico, August 25, 1998.
Lu, H., and R. Bernardo. 2001. Molecular marker diversity among current and historical maize inbreds. Theor. Appl. Genet. 103:613-617.
Mantel, N. 1967. The ditection of disease clustering and a generalized regression approach. Cancer Res. 27:209-220. Matsuoka, Y., S.E. Mitchell, S. Kresovich, M. Goodman, and J.
Doebley. 2002. Microsatellite in Zea-variability, patterns of mutation, and use for evolutionary studies. Theor. Appl. Genet. 104:436-450.
Nei, M. 1987. Estimation of average heterozygosity and genetic distance from a small number of individuals: Genetic 89:583-590.
Pejic, I., P. Ajmone-Marshan, M. Morgante, V. Kozumplick, P. Castiglioni, G. Taramino, and M. Motto. 1998. Comparative analysis of genetic similarity among maize inbred lines detected by RFLPs, RAPDs, SSRs and AFLPs. Theor. Appl. Genet. 97:1248-1255.
Perera, L., J.R. Russell, Provan, and W. Powell. 2000. Use micrsatellite DNA marker to investigate the level of genetic diversity and population genetic structure of coconut (Cocos nucifera L.) Genom. 43:15-21.
Rivera, R. 1999. Isolation and characterization of polymorphic microsatellites in Cococs nucifera L. Genom. 42:668-675. Robinson, R.W. 2000. Rational and method for producing hybrid
cucurbit seed. In: A.S. Basra (ed.). Hybrid seed production in vegetable: rational and method in selected crops. The Haworth Press, Inc. New York. 135 p.
Rohlf, F.J. 2000. NTSYS-pc. numerical taxonomy, and multivariate analysis system, Version 2.1. Exeter Publications, NY. Romero-Severson, J. 1998. Maize microsatellite-RFLP consensus
map. Maize DB (http://www.agron.missouri.edu).
Sumertajaya, I.M., B. Sartono, F.M. Affendi, U.D. Syafitri, dan Y. Angraeni. 2003. Modul teori analisis peubah ganda. Jurusan Statistik FMIPA IPB. Bogor.
Senior, M.L., J.P. Murphy, M.M. Goodman, and C.W. Stuber. 1998. Utitity of SSRs for determining genetic similarities and relationship in maize using agarose gel system. Crop Sci. 38:1088-1098.
Smith, J.S.C., E.C.L. Liu, H. Shu, O.S. Smith, S.J. Wall, M.L. Senior, S.E. Mitchell, S. Kresovich, and J. Zeigle. 1997. An evaluation of the utility of SSR loci as molecular markers in maize (Zea mays L.): Comparison with data from RFLPs and pedigree. Theor. Appl. Genet. 95:163-173.
Tuelat. 2000. An analysis of genetic diversity in coconut (Cocos nucifera) population from across the geographic range using sequence-tagged microsatellites (SSRs) and AFLPs. Theory Application Genetics 100:764-771.
Warburton, M.L., J.M. Ribaut, J. Franco, J. Crossa, P. Dubreuil & F.J. Betran. 2005. Genetic characterization of 218 elite CIMMYT maize inbred lines using RFLP markers. Euphytica 142:97-106.