129
Keragaman dan Hubungan Genetik Antara Kelapa Tetua Genjah Kuning Nias
(GKN) dan Dalam Tenga (DTA) serta Hibrida KHINA-1
Berdasarkan Marka Mikrosatelit
Genetic Diversity and Association among Nias Yellow Dwarf (NYD), Tenga Tall
(TAT) and KHINA-1 Hybrid Coconuts Based on Microsatellite Markers
ANNEKE PESIK
1,2, DARDA EFENDI
1, HENGKY NOVARIANTO
3, DINY DINARTY
1,
ISMAIL MASKROMO
3, ELSJE T. TENDA
3, dan SUDARSONO
11
PMB Laboratorium Departemen Agronomi dan Hortikultura, Fakultas Pertanian,
Institut Pertanian Bogor, Jln. Meranti-Kampus Darmaga, Bogor 16680, Indonesia
2Jurusan Biologi, Fakultas Matematika dan Ilmu Pengetahuan Alam, Universitas Pattimura
Jalan Ir. M. Putuhena Kampus Poka, Ambon 97233, Indonesia
3Balai Penelitian Tanaman Palma
Jalan Raya Mapanget, Kotak Pos 1004, Manado 95001
E-mail: [email protected]
Diterima 28 September 2015 / Direvisi 30 Oktober 2015 / Disetujui 9 Nopember 2015
ABSTRAK
Informasi keragaman dan hubungan genetik antar tetua dan progeni berperan penting dalam strategi pemuliaan tanaman kelapa. Identitas genetik antar progeni dari persilangan terkontrol perlu diidentifikasi untuk memastikan bahwa progeni berasal dari tetuanya, dan bukan akibat kontaminasi serbuk sari. Penggunaan marka mikrosatelit mampu mendeteksi keragaman genetik aksesi dan menduga kebenaran tetuanya. Tujuan penelitian ini adalah untuk mengevaluasi keragaman genetik populasi tetua GKN dan DTA serta hibrida KHINA-1, dan menganalisis hubungan genetik antara tetua GKN dan DTA dengan hibrida KHINA-1 menggunakan 19 marka mikrosatelit. Hasil penelitian menunjukkan nilai polimorfisme 19 marka mikrosatelit berkisar antara 0.18—0.72 dengan rataan jumlah alel 3.68 per lokus. Berdasarkan hasil analisis filogenetik dengan metode Neighbour Joining menggunakan program DARwin5, dapat
diketahui bahwa populasi GKN tergolong dalam satu group; populasi DTA merupakan populasi yang heterogenus dengan sebagian besar individu tergabung ke dalam satu kelompok dan sisanya ke dalam minimal dua kelompok yang berbeda; dan populasi hibrida KHINA-1 terbagi ke dalam minimal lima kelompok yang berbeda. Estimasi struktur populasi dengan perangkat lunak STRUCTURE menunjukkan adanya tingkat segregasi dan atau rekombinasi yang tinggi pada populasi KHINA-1, yang mengindikasikan telah terjadi percampuran materi genetik antara tetua GKN dan DTA pada populasi hibrida KHINA-1. Seleksi yang lebih akurat menggunakan marka molekuler dapat membantu perakitan kelapa hibrida yang lebih produktif.
Kata kunci: Keragaman genetik, kelapa hibrida, marka mikrosatelit.
ABSTRACT
Information on genetic diversity and association among parents and progenies plays an important role in plant breeding. Genetic identity among progenies derived from controlled population need to be determined to make sure that they are from hybridization of parents and they are not because of pollen contamination. Microsatellite markers can be used to evaluate genetic diversity and relationship among parents and their hybrid populations. The objectives of this study are to evaluate the genetic diversity of NYD, TAT and KHINA-1 hybrid populations; and to analyze the genetic relationship among the NYD and TAT as parents, with the KHINA-1 hybrid populations using 19 microsatellite markers. The results showed that the polymorphic information content (PIC) of 19 microsatellite markers loci was ranged from 0.18—0.72 and the average allele per locus was 3.68. Based on phylogenetic analysis using Neighbour-Joining for Tree Construction conducted by using DARwin5 software, individuals of the NYD population belong to a single group; the TAT population are heterogeneous population with the majority of the individuals are belonged into a single group and the rest are belonged to at least two separate groups; and those of KHINA-1 hybrid population are divided into at least five groups. Estimation of the population structure using STRUCTURE software shows that the presence of high segregation and or recombination event among the KHINA-1 progeny, indicating there has been a mixture of genetic materials from NYD and TAT parents in the KHINA-1 hybrids. More accurate selection using molecular marker should aid the development of more productive coconut hybrids.
130
PENDAHULUAN
Pemuliaan tanaman merupakan suatu metode pemanfaatan keragaman genetik plasma nutfah secara sistematis untuk menghasilkan varietas baru yang lebih baik dari sebelumnya. Upaya menghasilkan varietas unggul kelapa dapat mempercepat program peremajaan kelapa, dan untuk itu diperlukan bahan tanaman yang me-miliki karakteristik hasil kopra tinggi dan cepat berbuah. Metode pemuliaan yang dapat ditempuh adalah seleksi dan hibridisasi untuk merakit jenis kelapa hibrida, terutama kelapa hibrida hasil persilangan antara kelapa Genjah x kelapa Dalam (Pandin, 2009).
Kelapa hibrida intervarietas diperoleh dari hasil persilangan dua tetua berbeda yang mem-punyai keunggulan tertentu. Berdasarkan hasil evaluasi dan seleksi plasma nutfah kelapa, pemulia kelapa Balai Penelitian Palma (Balit Palma) menetapkan kelapa Genjah Kuning Nias (GKN) asal Pulau Nias, Sumatera Utara sebagai tetua betina (Novarianto, 2008). Kelapa Genjah Kuning Nias berbuah pada umur tiga tahun dengan produksi buah 100—120 butir/pohon/ tahun. Untuk tetua jantan dipilih varietas kelapa Dalam yaitu Dalam Tenga (DTA) asal Sulawesi Utara (Novarianto, 2008), dengan karakteristik produksi tinggi, jumlah buah banyak dengan ukuran buah sedang sampai besar. Varietas yang dihasilkan dari persilangan antara GKN dan DTA adalah Kelapa Hibrida Indonesia-1 (KHINA-1) yang mulai berbuah pada umur 3—4 tahun, hasil kopra rata-rata 4 ton/ha/tahun dengan hasil tertinggi 5 ton/ha/tahun, dan kadar minyak kopra 64%. KHINA-1 telah dilepas sebagai Kelapa Unggul Nasional (Novarianto, 2008).
Tetua GKN, DTA dan hibrida KHINA-1 koleksi Balai Penelitian Tanaman Palma perlu dianalisis hubungan kekerabatan dan struktur populasinya untuk mengetahui variasi genetik antar individu tetua GKN atau DTA dan antar hibrida KHINA-1. Selain itu, analisis hubungan antara KHINA-1 dengan tetua GKN dan DTA juga dapat digunakan untuk menduga kebenaran individu hibrida KHINA-1 sebagai hasil per-silangan antara kedua tetuanya (GKN dan DTA). Evaluasi hubungan kekerabatan antara individu tetua dengan progeninya dapat memberikan hasil yang kurang akurat, jika hanya berdasarkan fenotipe progeni. Populasi hibrida dapat berupa progeni illegitimate (outcrossing - hasil hibridisasi dengan serbuk sari dari tetua yang berbeda) atau berupa hasil penyerbukan sendiri (selfing – hasil hibridisasi dengan serbuk sari sendiri). Illegitimate hybrid merupakan hal yang perlu mendapat
perhatian dari pemulia karena dapat memberikan pengaruh negatif terhadap keragaan hibrida. Illegitimate hybrid dapat terjadi pada berbagai tahapan persilangan terkontrol mulai dari tahap awal seleksi, pelabelan tetua dan pelabelan benih di lapangan (Hama-Ali et al., 2014). Pada kelapa, persilangan outcrossing dengan bantuan serangga pada kelapa Dalam atau Genjah (Larekeng et al., 2015a,b), juga berpotensi menyebabkan terjadinya illegitimate hybrid baik. Penelitian untuk mendapat-kan informasi kebenaran tetua dalam persilangan yang menghasilkan hibrida KHINA-1 dapat dievaluasi menggunakan marka molekuler, belum pernah dilakukan.
Analisis berbasis marka molekuler diguna-kan untuk mengetahui hubungan genetik antar individu dalam suatu populasi secara akurat. Penggunaan marka molekuler khususnya mikro-satelit sangat direkomendasikan karena bersifat ko-dominan, secara alami multi-alel dan memiliki kapasitas untuk mendeteksi polimorfisme yang tinggi (Rajesh et al., 2008). Marka ini sangat sering digunakan dalam penelitian tanaman kelapa untuk analisis keragaman genetik dan penyebaran serbuk sari (Kumaunang dan Maskromo, 2007; Rajesh et al., 2008; Devakumar et al., 2010; Xiao et al., 2013; Rajesh et al., 2014; Maskromo et al., 2015; Larekeng et al., 2015a,b). Selain pada kelapa, marka mikrosatelit telah digunakan untuk analisis parental pada kelapa sawit dan kakao (Thawaro dan Te-chato 2009; Thongthawee et al., 2010; Ajambang et al., 2012; Hama-Ali et al., 2014; Tinche et al., 2014; Ajijah et al., 2016). Penelitian ini ber-tujuan untuk mengevaluasi keragaman genetik populasi tetua GKN dan DTA serta hibrida KHINA-1; dan untuk menganalisis hubungan genetik antara tetua GKN dan DTA dengan hibrida KHINA-1 menggunakan 19 marka mikro-satelit.
BAHAN DAN METODE
Analisis molekuler dilakukan di Laborato-rium Biologi Molekuler Tanaman (PMB Lab.), Departemen Agronomi dan Hortikultura, Fakultas Pertanian, Institut Pertanian Bogor, dari bulan Januari sampai dengan bulan Agustus tahun 2015. Bahan tanaman yang digunakan diperoleh dari Balai Penelitian Tanaman Palma, Manado, terdiri dari 10 individu kelapa GKN sebagai representasi tetua betina, 15 individu kelapa DTA sebagai representasi tetua jantan, dan 72 individu KHINA-1 sebagai hibrida hasil persilangan antara GKN dengan campuran serbuk sari dari sejumlah individu DTA.
131
Ekstraksi DNA
Isolasi DNA dilakukan dengan mengguna-kan metode CTAB (Rohde et al., 1996) dengan sedikit modifikasi yang telah dibuktikan efektif untuk mengisolasi DNA dari jaringan daun kelapa, kelapa sawit, kakao, pisang dan padi (Pandin, 2009; Sutanto et al., 2013; 2014; Rachmat et al., 2014; Ajijah et al., 2015; Maskromo et al., 2015). Sebanyak 0.3 g daun kelapa dimasukkan ke dalam mortar yang berisi 10 ml larutan penyangga lisis (100 mM Tris–HCl pH 8, CTAB 2% (b/v), 1.4 M NaCl, 20 mM EDTA, dan 0.2% β–mercapto-etanol), selanjutnya digerus sampai halus. Serbuk daun dipindahkan ke dalam satu tabung mikro 1.5 ml dan diinkubasi dalam waterbath dengan suhu 60 °C selama 1 jam. Selanjutnya campuran gerusan daun dan larutan penyangga disentrifugasi pada kecepatan 14000 rpm selama 3 menit, dan dilanjutkan dengan menambahkan 1 volume kloroform:isoamil alkohol (24:1). Larutan disentri-fugasi pada kecepatan 14000 rpm, kemudian supernatan dipisahkan ke dalam tabung baru. Supernatan DNA dipresipitasi dengan menambah-kan sodium asetat 3 M pH 5.2 sebanyak 0.1 volume dari supernatan dan isopropanol dingin sebanyak 0.8 volume dari supernatan. Endapan DNA yang dihasilkan melalui sentrifugasi pada 14000 rpm selama 15 menit, dicuci dengan etanol 70% dan dikering-anginkan. Endapan DNA di-suspensi dalam 500 μl larutan TE 1x sebagai stok utama DNA dan disimpan dalam freezer bersuhu 20°C. Kualitas DNA yang dihasilkan diuji dengan elektroforesis untuk menentukan kualitas dan kuantitasnya (Sambrook et al., 1989) menggunakan gel agarosa (Vivantis Inc., USA) 0.8% (b/v) dan larutan penyangga TBE 1x. DNA yang dielek-troforesis diwarnai menggunakan pewarnaan GelRedTM 33% (v/v) (Biotium Inc., USA) di bawah
sinar UV (Vilber Lourmat Super Bright TFX-20 MX, Sigma-Aldrich) dan difoto dengan kamera digital untuk dokumentasi. Sebelum digunakan sebagai templat untuk PCR, stok utama DNA diencerkan dalam ddH2O menyesuaikan dengan
kuantitas DNA awalnya dan disimpan dalam freezer bersuhu -20 °C hingga siap digunakan. Amplifikasi DNA Dengan Marka Mikrosatelit
Sebanyak 19 pasangan primer mikrosatelit spesifik kelapa digunakan dalam penelitian ini. Dua primer yang dikembangkan oleh (Rivera, 1999), yaitu primer CNZ 21 dan CNZ 51; dan tujuh belas primer lainnya dikembangkan oleh CIRAD (Centre for International Cooperation in Agri-cultural Research for Development, 2002) dengan kode CnCir (Tabel 1). Amplifikasi PCR dilakukan
dengan menggunakan KAPA2GTM PCR kit (Kapa Biosystems Inc., USA). Komposisi reaksi amplifikasi PCR terdiri dari 5.0 µl 5x buffer PCR, 0.5 µl MgCl2
25 mM, 0.5 µl dNTPs 10 mM, masing-masing 0.75 µl untuk primer forward dan primer reverse dengan konsentrasi 10 µM, 0.1 µl Taq DNA Polymerase (5 U µl-1) dan ditambah dengan ultra purewater (H2O) steril hingga mencapai volume
akhir total 25 µl. Amplifikasi PCR dilakukan dengan menggunakan mesin PCR BioRad T100TM Thermal Cycler. Tahapan amplifikasi terdiri atas: satu siklus pre–denaturasi pada suhu 95°C selama 3 menit; diikuti dengan 35 siklus yang terdiri atas tahapan denaturasi pada suhu 95°C selama 15 detik, penempelan primer pada suhu 48.8—55.4°C selama 15 detik (sesuai dengan suhu Tm masing-masing primer), pemanjangan primer pada suhu 72°C selama 1 detik; dan diakhiri dengan satu siklus pemanjangan akhir pada suhu 72°C selama 10 menit. Keragaman alel hasil amplifikasi PCR dipisahkan menggunakan gel akrilamid 6% dengan buffer SB 1X (Brody dan Kern, 2004) dan dielektroforesis menggunakan Cole-Parmer® Dedicated Height Sequencer. Hasil elektroforesis diwarnai dengan menggunakan pewarna perak nitrat (Creste et al., 2001) untuk menampilkan alel mikrosatelit. Hasil pewarnaan didokumentasi menggunakan scanner.
Skoring Alel dan Analisis Data
Pita DNA hasil amplifikasi PCR diskor secara manual sehingga menghasilkan data pasangan alel untuk masing-masing individu yang dianalisis. Selanjutnya data hasil skoring diubah menjadi data biner dengan kode 1 jika ada pita dan kode 0 jika tidak ada pita, dan dikompilasi untuk menentukan matriks jarak genetik dan parameter keragaman genetik (rata–rata jumlah alel yang diamati, rata–rata jumlah alel efektif yang diamati, heterozigositas, dan persentase lokus yang poli-morfik) menggunakan perangkat lunak GenAlEx 6.5 (Peakall dan Smouse 2012). Analisis filogenetik dilakukan menggunakan perangkat lunak DARwin5 (Perrier et al., 2003), sedangkan perhi-tungan PIC dilakukan dengan menggunakan perangkat lunak PowerMarker V3.25 (Liu dan Muse 2005). Perangkat lunak STRUCTURE V2.3.4 (Pritchard et al., 2000) digunakan untuk menduga struktur populasi dari populasi GKN, DTA, dan KHINA-1, yang menunjukkan kemiripan genotipe masing-masing individu dalam sub-grup yang didapatkan. Selanjutnya nilai rata-rata dan ragam diplot dalam ―likelihood per K‖ dihitung dengan menggunakan STRUCTURE Harvester v.0.6.94 (Earl dan vonHoldt 2012) dan metode Evanno (Evanno et al., 2005).
132
Tabel 1. Pasangan primer mikrosatelit untuk sembilan belas lokus yang digunakan dalam penelitian. Table 1. Microsatellite primer pairs of nineteen loci used in this study.
Nama Primer Primer name Sekuens oligonukleotida (5’– 3’) Oligonucleotide sequences (5’– 3’) Ukuran Primer Primer size (pb/bp) TM (°C) Ukuran Alel Allele size (pb/bp) CNZ 21 F:ATAACATCCTCCAACCTG R:GACTGAATCCAACCCTT 18 17 55 170-185 CNZ 51 F:AAAGTGAAGTGGATAATGTG R:AGAGAGGATCTAGGGTTGT 20 19 55 170-190 CnCir 56 F:AGTCCTAAAAGTGTTGGC R:GTAATCCTATGGCTGCTT 18 18 55 230-250 CnCir A9 F:GGACACTGGGTTCTGTT R:CTCTGTAATCTGCGGG 17 16 55 213-230 CnCir 87 F:AACCAGAACTTAAATGTCG R:TTTGAACTCTTCTATTGGG 19 19 55 210-230 CnCir 123 F:TCATTCAGAGGACAAAAGTT R:TAAAAATTCATAAAGGTAAAA 20 21 46 190-210 CnCir 121 F:TTGGTCTATTGCATGTTC R:TGGCATTGAGAGGGT 18 15 55 150-170 CnCir C5 F:CTGAAGATATGTGTTTATGC R:TGTTCCAGATTGAGGTT 20 17 52 270-290 CnCir C9 F:ATGTTTTAGCTTCACCATGAA R:TCAAGTTCAAGAAGACCTTTG 21 21 55 260-280 CnCir E4 F:CTTTAGGGAAAAAGGACTGAG R:ATCCATGAGCTGAGCTTGAAC 21 21 55 170-190 CnCir A3 F:AATCTAAATCTACGAAAGCA R:AATAATGTGAAAAAGCAAAG 20 20 55 250-260 CnCir H11 F:AATGTTTGTGTCTTTGTGCGTGTGT R:TCCTAATTTTTCTTCCCCTTCCTCA 25 25 60 80-110 CnCir 73 F:TCGCTGATGAATGCTTGCT R:GGGGCTGAGGGATAAACC 19 18 55 140-165 CnCir 1 F:CAGAAAGGAGAAAGGAAAT R:CTACGATAGAGGAATGAGC 19 19 55 280-310 CnCir 226 F:ACCAACAAAGCCAGAGC R:GCAGCCACTACCTAAAAA 17 18 55 120-140
CnCir E11 F:GCATGGTATTCGGATTTG R:ATGGTTCAGATTTGGACAGT 18 20 52 180-190 CnCir 119 F:CAGAAACAGCCAATCAAGCAATA R:ATCCATAATAGCCACTCAACAAAAA 23 25 58 240-270 CnCir E2 F:TAGAAGGAAAGGGAGATG R:GTCCATCACTAAGATAGATCA 18 21 55 240-255 CnCir 2 F:AGAACCCTTGCTCCAC R:TCCAGCCATTCCATC 16 15 55 200-260
Keterangan/Note: F = Primer/Forward; R = Primer/Reverse; pb/bp = pasang basa/base pair.
HASIL DAN PEMBAHASAN
Analisis Keragaman Genetik Tetua GKN dan DTA
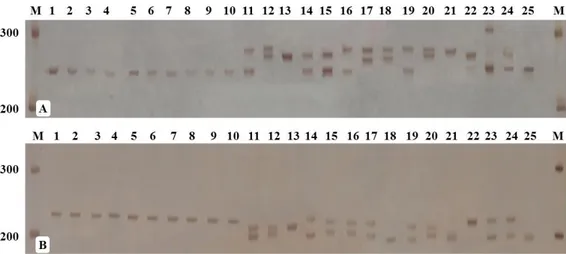
Keseluruhan primer mikrosatelit yang diuji menghasilkan pita polimorfik dengan jumlah alel total sebanyak 69. Dari total 69 alel yang terdeteksi, jumlah alel per lokus terbanyak adalah 4 alel, yaitu masing-masing untuk lokus CNZ 21, CNZ 51, CnCir C5, CnCir H11, CnCir 73, CnCir 1, CnCir E2 dan CnCir 2. Contoh alel yang teridentifikasi di antara sejumlah individu tetua GKN dan DTA yang diamplifikasi dengan primer CNZ 51 dan CnCir 56 disajikan pada Gambar 1. Pada gambar ini dapat dilihat contoh keseragaman alel pada semua individu GKN dan keberagaman alel antar 15 individu DTA yang dievaluasi untuk lokus CNZ 51 dan CnCir 56.
Gambar 2 dan Tabel 2 disajikan untuk menunjukkan parameter keragaman yang didapat pada dua populasi tetua GKN dan DTA dalam analisis keragaman genetik populasi. Jumlah alel per lokus untuk masing-masing lokus bervariasi antara 2—6 dengan rataan 3.63 per lokus. Jumlah alel efektif (Ne) untuk setiap lokus berkisar antara 1.34—2.99 dengan rataan 1.99 per lokus. Nilai heterozigositas yang diamati (Ho) menunjukkan jumlah penyebaran gen atau alel yang diamati pada suatu populasi; untuk populasi tetua yang dianalisis berkisar antara 0.12—0.88 dengan nilai rataan 0.40. Nilai heterozigositas harapan (He) untuk populasi tetua berkisar antara 0.25—0.67 dengan nilai rataan 0.48. Nilai Polymorphic Information Content (PIC), yang merupakan pengukuran tingkat polimorfisme setiap lokus, bervariasi dari 0.24 (CnCir E2) sampai 0.61 (CnCir C5) dengan rataan 0.43 (Tabel 2).
133
Gambar 1. Profil alel untuk marka mikrosatelit yang dihasilkan dengan menggunakan primer (A) CNZ 51 dan (B) CnCir 56 - untuk kelapa Genjah Kuning Nias (1-10) dan Dalam Tenga (11-25). M: DNA marker (100 bp ladder).
Figure 1. Allele profiles of microsatellite markers generated using both (A) CNZ 51 and (B) CnCir 56 – primer for
Nias Yellow Dwarf (1-10) and Tenga Tall (11-25) coconuts. M: marker DNA (100 bp ladder).
Tabel 2. Data jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai Polymorphic Information Content (PIC) pada populasi tetua (GKN dan DTA) berdasarkan hasil analisis dengan 19 lokus marka mikrosatelit.
Table 2. Number of alleles (Na), number of effective alleles (Ne), level of heterozigosity (Ho and He), and Polymorphic Information Content (PIC) of GKN and DTA parental populations based on 19 microsatellite markers loci.
Nama Primer
Primer name Na Ne Ho He PIC
CNZ 21 4 2.34 0.48 0.57 0.52 CNZ 51 4 1.98 0.48 0.50 0.46 CnCir 56 3 1.85 0.44 0.46 0.41 CnCir A9 3 2.04 0.36 0.51 0.41 CnCir 87 2 1.81 0.44 0.45 0.35 CnCir 123 5 2.07 0.40 0.52 0.47 CnCir 121 3 1.71 0.20 0.42 0.38 CnCir C5 4 2.99 0.32 0.67 0.61 CnCir C9 5 1.91 0.56 0.48 0.44 CnCir E4 3 1.39 0.28 0.28 0.26 CnCir A3 2 1.72 0.28 0.42 0.33 CnCir H11 4 2.01 0.32 0.50 0.47 CnCir 73 4 2.62 0.12 0.62 0.54 CnCir 1 4 1.91 0.52 0.48 0.44 CnCir 226 2 1.97 0.88 0.49 0.37 CnCir E11 6 1.93 0.36 0.48 0.46 CnCir 119 3 1.93 0.56 0.48 0.43 CnCir E2 4 1.34 0.20 0.25 0.24 CnCir 2 4 2.35 0.52 0.57 0.51 Total 69 37.87 9.14 7.72 8.11 Rata-rata 3.63 1.99 0.40 0.48 0.43
Keterangan/Note: Ho = heterozigositas yang diamati/observed heterozygosity, He = heterozigositas yang diharapkan/expected heterozigosity.
134
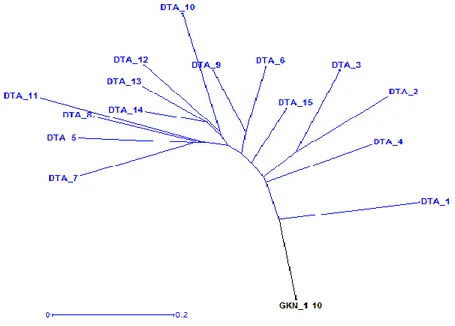
Gambar 2. Hasil analisis filogenetik dengan metode Neighbour Joining menggunakan perangkat lunak DARwin5 untuk 10 individu genjah kuning Nias (GKN) dan 15 individu Dalam Tenga (DTA) berdasarkan keragaman alel pada 19 lokus marka mikrosatelit.
Figure 2. Results of the phylogenetic analysis using Neighbour Joining method with DARwin5 software for 10 individuals of Nias Yellow Dwarf (GKN) and Tenga Tall (DTA) based on allele diversities of 19 microsatellite markers loci.
Gambar 2 menunjukkan adanya kesera-gaman antar 10 individu GKN yang dievaluasi dan keragaman yang tinggi antar 15 individu DTA. Dari Gambar 2 juga ditunjukkan bahwa 10 individu GKN yang dievaluasi memiliki kom-posisi genetik yang sangat berbeda dengan 15 individu DTA. Hal ini dapat terjadi, diduga karena adanya perbedaan tipe penyerbukan antara kelapa Dalam dan kelapa Genjah. Kelapa Dalam umum-nya menyerbuk silang sehingga variasi alel pada setiap individu lebih tinggi dibandingkan dengan kelapa Genjah yang menyerbuk sendiri (Novarianto, 2010). Hasil pengamatan di lapang menunjukkan adanya perbedaan ciri morfologi yang sangat signifikan antar kedua populasi tetua karena induk GKN merupakan kelapa Genjah dan tetua jantan DTA merupakan kelapa Dalam (Manaroinsong dan Kumaunang, 2005; Novarianto, 2011). Tetua GKN memiliki warna buah kuning; warna tandan buah kuning; warna petiole kuning kehijauan; panjang pada 11 bekas daun 55.03 cm; umur mulai berbuah 40 bulan (3.3 tahun), umur mulai panen 48 bulan (4 tahun). Tetua DTA memiliki warna buah hijau, hijau kekuningan atau merah kekuningan; warna tandan buah hijau atau hijau kekuningan; warna petiole hijau; panjang pada 11 bekas daun 104 cm; umur mulai berbuah 5 tahun, umur mulai panen 6 tahun (Kementerian Pertanian, 2015). Hubungan kekerabatan antar populasi kelapa GKN dan DTA
sangat jauh dan tingkat heterogenitas tinggi antar individu kelapa DTA. Hal ini memberikan harapan bahwa populasi hibrida hasil persilangan antar keduanya dapat menghasilkan hibrida yang lebih baik, dibandingkan kedua tetuanya sebagai akibat terjadinya heterosis.
Analisis Keragaman Genetik Populasi GKN,
DTA dan KHINA-1
Gambar 3 dan Tabel 3 disajikan untuk menunjukkan ada tidaknya perubahan parameter keragaman populasi setelah ditambahkan individu hibrida KHINA-1 ke dalam analisis keragaman genetik.Dengan menambahkan populasi KHINA-1, analisis dengan 19 lokus mikrosatelit menghasil-kan 70 pita polimorfik pada total populasi GKN, DTA dan KHINA-1. Dari total 70 alel yang ter-deteksi, jumlah alel terdeteksi per lokus (Na) bervariasi antara dengan 2 alel (CnCir 87, CnCir A3 dan CnCir 226) hingga 6 alel (CnCir E11) dan dengan rataan 3.68 alel per lokus. Jumlah alel efektif (Ne) untuk setiap lokus berkisar antara 1.24—4.16 dengan rataan 2.32 alel efektif per lokus. Pada populasi total, nilai heterozigositas teramati (Ho) berkisar antara 0.14—0.90 dengan nilai rataan 0.41, heterozigositas harapan (He) berkisar antara 0.18—0.49 dengan nilai rataan 0.38. Nilai Poly-morphic information content (PIC), bervariasi antara 0.18 (E2) sampai dengan 0.72 (CNZ 51) dengan rataan 0.47 (Tabel 3). Kimura dan Crow (1964)
135
Tabel 3. Data jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai Polymorphic Information Content (PIC) pada populasi GKN, DTA dan KHINA-1 berdasarkan hasil analisis dengan 19 lokus marka mikrosatelit.
Table 3. Number of alleles (Na), number of effective alleles (Ne), level of heterozigosity (Ho and He), and Polymorphic Information Content (PIC) of GKN, DTA and KHINA-1 populations based on 19 microsatellite markers loci.
Nama Primer Na Ne Ho He PIC
CNZ 21 4 2.73 0.43 0.45 0.58 CNZ 51 5 4.16 0.49 0.48 0.72 CnCir 56 3 2.55 0.45 0.41 0.54 CnCir A9 3 1.97 0.41 0.33 0.41 CnCir 87 2 1.98 0.50 0.33 0.37 CnCir 123 5 2.31 0.33 0.35 0.51 CnCir 121 3 2.30 0.23 0.39 0.50 CnCir C5 4 2.31 0.39 0.40 0.48 CnCir C9 5 3.00 0.44 0.45 0.61 CnCir E4 3 1.68 0.20 0.28 0.35 CnCir A3 2 1.80 0.33 0.31 0.34 CnCir H11 4 2.08 0.35 0.40 0.48 CnCir 73 4 3.13 0.34 0.38 0.63 CnCir 1 4 2.13 0.53 0.40 0.47 CnCir 226 2 1.99 0.90 0.49 0.34 CnCir E11 6 1.94 0.34 0.38 0.45 CnCir 119 3 2.29 0.44 0.36 0.49 CnCir E2 4 1.24 0.14 0.18 0.18 CnCir 2 4 2.41 0.59 0.41 0.50 Total 70 44.0 10.28 9.81 9.05 Rata-rata 3.68 2.32 0.41 0.38 0.47
Keterangan/Note: Ho = Heterozigositas yang diamati/Observed heterozygosity, He = Heterozigositas yang diharapkan/ Expected heterozigosity.
menyatakan bahwa jumlah alel efektif merupakan gambaran dari jumlah alel yang terdapat pada suatu populasi sampai tahap individu siap me-lakukan persilangan, alel-alel tersebut tetap ter-pelihara dalam populasi.
Jika dibandingkan antara hasil analisis ke-ragaman populasi tetua (GKN dan DTA, Tabel 2 dan Gambar 2) dengan populasi total (GKN, DTA dan KHINA-1, Tabel 3 dan Gambar 3) yang dievaluasi, terdapat peningkatan jumlah alel total dari 69 (populasi tetua) menjadi 70 (populasi total) dan rataan jumlah alel per lokus 3.63 menjadi 3.68 (Tabel 2 dan 3); jumlah alel efektif berkisar 1.34— 2.99 (populasi tetua) menjadi 1.24—4.16 (populasi total) dan rataan alel efektif per lokus 1.99 menjadi 2.32 (Tabel 2 dan 3); Ho berkisar 0.12—0.88 (populasi tetua) menjadi 0.14—0.90 (populasi total) dan rataan 0.40 menjadi 0.41 (Tabel 2 dan 3); He berkisar 0.25—0.67 (populasi tetua) menjadi dengan 0.18—0.49 (populasi total) dan rataan 0.48 menjadi 0.38 (Tabel 2 dan 3) serta PIC berkisar 0.24—0.61 (populasi tetua) menjadi 0.18—0.72 (populasi total) dan rataan 0.43 menjadi 0.47 (Tabel 2 dan 3). Berbagai perubahan nilai parameter populasi antara populasi tetua dan populasi total tersebut mengindikasikan ada informasi genetik
baru (bukan berasal dari dua tetuanya) yang masuk kedalam populasi KHINA-1. Hal ini mengindikasikan bahwa sebagian dari populasi KHINA-1 yang dianalisis diduga merupakan illegitimate hybrid.
Nilai PIC menunjukkan tingkat informatif lokus-lokus yang digunakan sebagai ciri tingkat keragaman alel. Nilai PIC diklasifikasikan menjadi tiga kelas, yaitu PIC>0.5 sebagai sangat informatif, 0.25>PIC>0.5 sebagai moderat informatif, dan PIC<0.25 sebagai kurang informatif (Botstein et al., 1980). Dalam penelitian ini dapat ditunjukkan bahwa marka CNZ 51 adalah lokus yang paling informatif dan marka CnCir E2 adalah lokus yang kurang informatif. Nilai PIC dan jumlah alel per lokus sangat tergantung dari keragaman aksesi plasma nutfah yang diuji dan lokus marka mikro-satelit yang digunakan (Tasma, 2014). Dalam penelitian ini, rataan nilai PIC 0.47 mengindi-kasikan bahwa sebagian besar lokus yang diguna-kan mampu menerangdiguna-kan keragaman genetik pada populasi yang dievaluasi.
136
I
V
IV
III
II
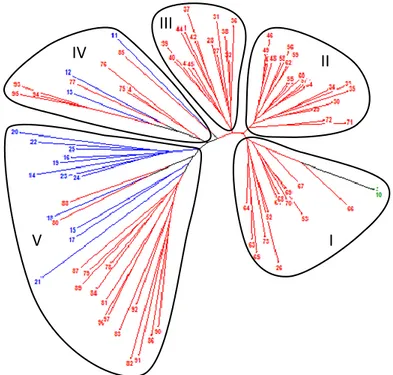
Gambar 3. Pohon filogenetik berdasarkan metode Tree Construction-Neighbour Joining dari individu GKN, DTA dan KHINA-1 menggunakan 19 marker mikrosatelit. Warna hitam adalah tetua GKN, warna biru adalah tetua DTA dan warna merah adalah KHINA-1. Individu nomor 1-10 adalah tetua GKN, 11-25 adalah tetua DTA, 26-97 adalah progeni KHINA-1.
Figure 3. Phylogenetic tree based on Tree Construction-Neighbour Joining method of individual GKN and DTA using 19 microsatellite markers. GKN in black, DTA in blue, KHINA-1 in red. Number 1-10 are GKN parent, 11-25 are DTA parent, 26-97 are KHINA-1 progeny.
Analisis filogenetik populasi yang diuji berdasarkan 19 marka mikrosatelit menunjukkan lima kelompok yang berbeda (Gambar 3). Kelom-pok I, sepuluh individu tetua GKN menge-lomKelom-pok menjadi satu dengan 13 individu KHINA-1, yang menunjukkan bahwa individu KHINA-1 tersebut mempunyai latar belakang genetik yang sama berdasarkan pola pita dari 19 marka mikrosatelit, yang mayoritasnya berasal dari tetua GKN. Jika dibandingkan dengan populasi tetua jantan DTA (kelompok V), 13 individu KHINA-1 tersebut mempunyai jarak genetik yang jauh. Data tersebut mengindikasikan bahwa tetua jantan dari 13 individu KHINA-1 kemungkinan besar bukan turunan dari populasi DTA yang diuji. Individu KHINA-1 yang masuk ke dalam kelompok II dan III, berada di antara tetua GKN dan DTA. Hal ini mengindikasikan bahwa semua individu KHINA-1 di kelompok II dan III merupakan hasil per-silangan GKN x DTA. Dalam kelompok IV, ter-dapat tiga individu DTA bercampur dengan delapan individu KHINA-1, dan dalam kelompok V, terdapat enam belas individu KHINA-1 ber-campur dengan dua belas tetua DTA. Semua individu KHINA-1 dalam kelompok IV dan V mempunyai jarak genetik yang jauh dengan tetua
GKN. Hal tersebut mengindikasikan bahwa individu KHINA-1 yang ada dalam kelompok IV dan V kemungkinan besar merupakan illegitimate hybrid, yang tetuanya bukan hasil persilangan dari GKN x DTA.
Analisis Struktur Populasi GKN, DTA dan KHINA-1
Struktur genetik populasi kelapa GKN, DTA dan KHINA-1 dianalisis menggunakan perangkat lunak STRUCTURE V2.3.4 dengan pendekatan Bayesian untuk menentukan nilai K, yaitu jumlah sub populasi dalam suatu koleksi dan meng-estimasi proporsi genom setiap aksesi yang ber-asal dari setiap sub populasi. Penggunaan STRUCTURE dapat melakukan pengelompokan individu-individu di dalam populasi dengan akurat. Perangkat lunak ini digunakan untuk mengidentifikasi struktur populasi dan mengatasi masalah terdapatnya individu yang meragukan di dalam subpopulasi (Pritchard et al., 2000). Per-hitungan ad hoc maksimum ΔK diperoleh pada K=2, yang menyatakan bahwa populasi uji dibagi atas dua subgrup (Gambar 4).
137
Gambar 4. Perubahan delta K (ΔK) sesuai perbedaan K untuk populasi GKN, DTA dan KHINA-1 yang dihasilkan oleh perangkat lunak STRUCTURE.
Figure 4. Delta K changed according to different K of population GKN, DTA and KHINA-1 by STRUCTURE software.
Gambar 5. Estimasi struktur populasi berdasarkan data genotyping 19 marka mikrosatelit pada populasi GKN, DTA dan KHINA-1 menggunakan perangkat lunak STRUCTURE. Kelompok 1 adalah tetua GKN, kelompok 2 adalah tetua DTA, kelompok 3 adalah KHINA-1.
Figure 5. Estimation of population structure based on genotyping of 19 microsatellite markers on population GKN, DTA and 1 using STRUCTURE software. GKN in group 1, DTA in group 2, KHINA-1 in group 3.
Alel-alel spesifik yang berasal dari tetua GKN direpresentasikan oleh warna hijau dan tetua DTA dengan warna merah (Gambar 5). Hasil analisis STRUCTURE menunjukkan meskipun komposisi genetik antara tetua GKN dan DTA berbeda jauh, tetapi pada populasi tetua DTA terdapat sejumlah alel yang sama dengan alel yang dimiliki oleh tetua GKN. Individu KHINA-1 sebagai hasil persilangan tetua GKN dan DTA menunjukkan variasi campuran warna merah dan hijau yang mengindikasikan adanya keragaman genetik luas antar individu dalam populasi
KHINA-1. Gambar 5 menunjukkan bahwa individu nomor 76—97 mempunyai konstitusi genetik mendekati tetua jantan DTA (mayoritas warna merah). Individu tersebut merupakan KHINA-1 yang masuk dalam kelompok IV dan V pada Gambar 3. Sebaliknya, individu KHINA-1 nomor 48 dan 51 dengan latar belakang genetik mendekati GKN, merupakan individu KHINA-1 yang masuk dalam kelompok I pada Gambar 3. Sebagian individu KHINA-1 yang mempunyai konstitusi genetik campuran proporsional antara tetua GKN dan DTA (campuran proporsional
138
warna merah dan hijau) merupakan representasi legitimate hybrid. Individu tersebut adalah KHINA-1 yang masuk dalam kelompok II dan III pada Gambar 3.
Berbagai data hasil analisis yang dilakukan dalam penelitian ini mengilustrasikan contoh penerapan marka molekuler dalam membantu pemuliaan tanaman. Hasil analisis marka mole-kuler mikrosatelit telah dilakukan untuk meng-evaluasi keragaman genetik diantara populasi tetua GKN, DTA dan hibrida KHINA-1. Hasil penelitian ini dapat ditindaklanjuti dengan peng-amatan daya hasil dan komponen hasil atau karakteristik kualitatif dan kuantitatif penting lainnya, yang diinginkan dari setiap individu KHINA-1 yang telah dievaluasi secara molekuler. Selanjutnya, hasil analisis marka molekuler yang telah memilah individu KHINA-1 ke dalam kelompok I—V diasosiasikan dengan berbagai karakter kualitatif dan kuantitatif yang dikum-pulkan. Lebih lanjut, hasil analisis asosiasi penge-lompokan berdasarkan marka molekuler dapat digunakan untuk melakukan seleksi yang lebih akurat dalam perakitan kelapa hibrida yang lebih produktif, misalnya dalam pengembangan varietas hibrida kelapa kopyor Indonesia (Novarianto et al. 2014).
KESIMPULAN
Hasil analisis molekuler menggunakan 19 lokus marka mikrosatelit terhadap populasi tetua DTA dan GKN mendukung pemilihan keduanya sebagai tetua dalam pembentukan KHINA-1 karena kedua tetua terbukti mempunyai jarak genetik yang tinggi. Hasil analisis molekuler menggunakan marka mikrosatelit yang sama terhadap populasi KHINA-1 mengelompokkan individu KHINA-1 ke dalam lima kelompok (Kelompok I—V). Individu KHINA-1 yang tergolong ke dalam Kelompok I, IV, dan V berpotensi merupakan illegitimate hybrid karena memiliki kemiripan genetik yang tinggi dengan tetua GKN (kelompok I) atau DTA (kelompok IV dan V). Individu KHINA-1 yang tergolong ke dalam kelompok II dan III berpotensi sebagai legitimate hybrid antar GKN dan DTA. Hasil analisis filogenetik diperkuat oleh hasil analisis STRUCTURE yang mengidentifikasi adanya illegitimate hybrids di antara populasi KHINA-1. Implikasi hasil penelitian ini dapat digunakan untuk mendukung perakitan kelapa hibrida baru melalui proses seleksi yang lebih akurat meng-gunakan marka molekuler yang informatif.
UCAPAN TERIMA KASIH
Penulis menyampaikan terima kasih kepada Kementerian Riset, Teknologi, dan Pendidikan Tinggi RI atas pembiayaan pendidikan pasca-sarjana tahun 2011—2014. Disampaikan terima kasih pula kepada Pimpinan Badan Litbang Pertanian, Kementerian Pertanian RI atas pem-biayaan Program Penelitian KKP3N - Kelapa Kopyor (tahun 2013—2014) dan Program Peneli-tian KKP3S - Kelapa (tahun 2015), a.n. Prof. Dr. Sudarsono, MSc.
DAFTAR PUSTAKA
Ajambang, W., Sudarsono, D. Asmono, N. Toruan. 2012. Microsatellite markers reveal Cameroon's wild oil palm population as a possible solution to broaden the genetic base in the Indonesia-Malaysia oil palm breeding programs. African Journal of Biotechnology 11(69): 13244-13249.
Ajijah, N., R.S. Hartati, R. Rubiyo, D. Sukma, S. Sudarsono. 2016. Effective cacao somatic embryo regeneration on kinetin supple-mented DKW medium and somaclonal variation assessment using SSR markers. Agrivita 38(1): 80-92.
[CIRAD] Centre for International Cooperation in Agricultural Research for Development 2002. A laboratory manual. Coconut micro-satellite kit. France: Montpellier.
Botstein, D., R. White, M. Skolnick, R. Davis. 1980. Construction of a genetic linkage map ini man using restriction fragment lenght polymorphism. Am J Hum Genet 32(3): 314-331.
Brody, J., S. Kern. 2004. Sodium boric acid: a Tris free, cooler conductive medium for DNA electrophoresis. BioTechniques 36: 214-216. Creste, S., A. Neto, A. Figueira. 2001. Detection of
single sequence repeats polymorphisms in denaturing polyacrilamide sequencing gels by silver staining. Plant Molecular Biology Reporter 19: 299-306.
Devakumar, K., V. Niral, B. Jerard, C. Jayabose, R. Chandramohanan, P. Jacob. 2010. Micro-satellite analysis of distinct coconut accessions from Agati and Kavaratti islands, Lakshadweep, India. Sci Horti 125: 309-315. Earl, D., B.M. vonHoldt. 2012. STRUCTURE
HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources 4(2): 359-361.
139
Evanno, G., J. Regnaut, Goudet. 2005. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study. Molecular Ecology 14: 2611-2620.
Hama-Ali, E., S. Tan, S. Alwee, J. Panandam, P. Namasivayam, H. Peng, H. Ling. 2014. Illegitimacy and sibship assignments in oil palm (Elaeis guineensis Jacq.) half-sib families using single locus DNA microsatellite markers. Mol Biol Rep. doi: 10.1007/s11033-014-3829-7.
Kementerian Pertanian. 2015. Sistem informasi database varietas tanaman komoditas kelapa. http://ppvt.setjen.pertanian.go.id/ varietas/tamu/utama.asp.
Kimura, M., J. Crow. 1964. The number of alleles that can be maintained in a finite population. Genetics 49(4): 725-738.
Kumaunang, J., I. Maskromo. 2007. Keragaman genetik plasma nutfah kelapa Dalam (Cocos nucifera L.) di kebun percobaan Mapanget berdasarkan penanda DNA SSRs. Buletin Palma 33: 18-27.
Larekeng, S., I. Maskromo, A. Purwito, N. Matjik, S. Sudarsono. 2015a. Pollen dispersal and pollination patterns studies in Pati kopyor coconut using molecular markers. International Journal on Coconut R & D 31(1): 46-60.
Larekeng, S.H., I. Maskromo, A. Purwito, N.A. Matjik, dan Sudarsono. 2015b. Penyebaran polen berdasarkan analisis SSR membuk-tikan penyerbukan kelapa Dalam Kalianda Normal ke Kopyor. Buletin Palma 16(1): 77-92.
Liu, K., S. Muse. 2005. PowerMarker: An integrated analysis environment for genetic marker analysis. Bioinformatics 21: 2128-2137.
Manaroinsong, E., J. Kumaunang. 2005. Karakter fisiologi beberapa kultivar kelapa Dalam. Buletin Palma 29: 14-22.
Maskromo, I., T. Tenda, M. Tulalo, H. Novarianto, D. Sukma, Sukendah, Sudarsono. 2015. Keragaman fenotipe dan genetik tiga varietas kelapa genjah kopyor asal Pati Jawa Tengah. Jurnal Littri 2(1): 1-8.
Novarianto, H. 2008. Perakitan kelapa unggul melalui teknik molekuler dan implikasinya terhadap peremajaan kelapa di Indonesia. Pengembangan Inovasi Pertanian 1(4): 259-273.
Novarianto, H. 2010. Karakteristik bunga dan buah hasil persilangan kelapa hibrida genjah x genjah. Buletin Palma 39: 100-110.
Novarianto, H. 2011. Penampilan bibit kelapa hibrida genjah x genjah. Buletin Palma 12(1): 18-26.
Novarianto, H., I. Maskromo, D. Dinarti, and Sudarsono. 2014. Production technology for Kopyor coconut seednuts and seedlings in Indonesia. International Journal on Coconut R & D 30(2): 31-40.
Pandin, D. 2009. Keragaman genetik kultivar kelapa Dalam Mapanget (DMT) dan Dalam Tenga (DTA) berdasarkan penanda Random Amplified Polymorphic DNA (RAPD). Buletin Palma 36: 17-29.
Peakall, R., P. Smouse. 2012. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics 28: 2537-2539. doi: 10.1093/bioinformatics/bts460.
Perrier, X., A. Flori, F. Bonnot. 2003. Data analysis methods. Montpellier: Enfield Science Publishers.
Pritchard, J., M. Stephens, P. Donnelly. 2000. Inference of population structure using multilocus genotype data. Genetics 155: 945-959.
Rachmat, A., S. Nugroho, D. Sukma, H Aswidinnoor, Sudarsono. 2014. Overexpression of OsNAC6 transcription factor from Indonesia rice cultivar enhances drought and salt tolerance. Emir. J. Food Agric 26(6): 519-527. Rajesh, M., V. Arunachalam, P. Nagarajan, P.
Lebrun, K. Samsudeen, C. Thamban. 2008. Genetic survey of 10 Indian coconut landraces by Simple Sequence Repeats (SSRs). Scientia Horticulturae 118: 282-287. Rajesh, M., K. Samsudeen, B. Jerard, P. Rejusha, A.
Karun. 2014. Genetic and phylogenetic relationships of coconut populations from Amini and Kadmat Islands, Lakshadweep (India). Emir. J. Food Agric 26(10): 898-906. Rivera, R. 1999. Isolation and characterization of
polymorphic microsatellites in Cocos nucifera L. Genom 42: 668-675.
Rohde, W., A. Kullaya, M.J. Rodriguez, E. Ritter. 1996. Genome analysis of Cocos nucifera L. by PCR amplification of spacer sequences separating a subset of Copia-like EcoR1 repetitive elements. J Genet Breed 49: 170-186.
Sambrook, J., E.F. Fritch, T. Maniatis. 1989. Molecular cloning: A laboratory manual: Cold Spring Harbor Laboratory Press. Sutanto, A., D. Sukma, C. Hermanto, Sudarsono.
2014. Isolation and characterization of Resistance Gene Analogue (RGA) from
140
Fusarium resistant banana cultivars. Emir. J. Food Agric 26(6): 508-518.
Sutanto, A., C. Hermanto, D. Sukma, Sudarsono. 2013. Development of SNAP marker based on resistance gene analogue genomic sequences in banana (Musa spp.) [Pengem-bangan marka SNAP berbasis resistance gene analogue pada tanaman pisang (Musa spp.). Jurnal Hortikultura 23(4): 300-309. Tasma, I. 2014. Skrining marka SSR untuk analisis
diversitas genetik aksesi kelapa sawit. Buletin Palma 15(1): 1-13.
Thawaro, S., S. Te-chato. 2009. Application of molecular markers in the hybrid verification and assessment of somaclonal variation from oil palm propagated in vitro. Sci Asia 35: 142-149.
Thongthawee, S., P. Tittinutchanon, H. Volkaert. 2010. Microsatellites for parentage analysis in an oil palm breeding population. Thai Journal of Genetics 3(2): 172-181.
Tinche, D. Asmono, D. Dinarty, Sudarsono. 2014. Keragaman genetik kelapa sawit (Elaeis guineensis Jacq.) populasi nigeria berdasar-kan analisis marka SSR (Simple Sequence Repeats). Buletin Palma 15(1): 14-23.
Xiao, Y., L. Yi, Y. Yaodong, F. Haikuo, X. Wei, M. Annaliese, Z. Songlin, S. Ross, Q. Fei. 2013. Development of microsatellite markers in Cocos nucifera and their application in evaluating the level of genetic diversity of Cocos nucifera. Plant Omics J 6: 193-200.