EVALUASI STRUKTUR KRISTAL 4HMA SEBAGAI TARGET VIRTUAL ENZIM MATRIX METALLOPROTEINASE 9 PADA PENAPISAN VIRTUAL
BERBASIS STRUKTUR
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S. Farm)
Program Studi Farmasi
Oleh:
FX Nyoman Indra Putra Nugraha
NIM: 138114119
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA YOGYAKARTA
i
HALAMAN JUDUL
EVALUASI STRUKTUR KRISTAL 4HMA SEBAGAI TARGET VIRTUAL ENZIM MATRIX METALLOPROTEINASE 9 PADA PENAPISAN
VIRTUAL BERBASIS STRUKTUR
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh:
FX Nyoman Indra Putra Nugraha
NIM: 138114119
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA YOGYAKARTA
iv
HALAMAN PERSEMBAHAN
“It is OK not to know all the answers.
It is better to admit our ignorance than to believe the answers that might be wrong.
Pretending to know everything, closes the door to finding out what is really there”
(from “Cosmos: A Space Time Odyssey”)
Karya ini kupersembahkan untuk Tuhan Yesus Kristus yang selalu menyertaiku Papi, Mami dan Mama yang selalu mendoakanku
Teman-teman seperjuanganku
vii PRAKATA
Puji dan syukur penulis panjatkan ke hadirat Tuhan Yang Maha Esa karena atas kasih dan rahmat-Nya penulis dapat menyelesaikan skripsi yang berjudul “Evaluasi Struktur Kristal 4HMA Sebagai Target Virtual Enzim Matrix
Metalloproteinase 9 Pada Penapisan Virtual Berbasis Struktur” sebagai syarat
untuk memperoleh gelar sarjana farmasi (S.Farm.) di Universitas Sanata Dharma Yogyakarta. Keberhasilan dalam penulisan skripsi ini tidak lepas dari bantuan dan dukungan berbagai pihak. Oleh karena itu dengan kerendahan hati penulis ingin mengucapkan terima kasih kepada:
1. Bapak Enade Perdana Istyastono, Ph.D., Apt. selaku pembimbing yang telah sabar dan banyak memberikan pengarahan dan pendampingan selama proses penyusunan skripsi.
2. Bapak Maywan Hariono, Ph.D., Apt. dan Bapak Florentinus Dika Octa Riswanto, M.Sc. selaku penguji yang telah mendukung terlaksananya penelitian dan penyusunan skripsi ini dan telah memberikan saran berharga bagi penulis.
3. Ibu Dita Maria Virginia, M. Sc., Apt. selaku Dosen Pembimbing Akademik yang telah membimbing dari awal hingga akhir tahun perkuliahan.
4. Seluruh dosen dan staf Fakultas Farmasi Universitas Sanata Dharma yang telah membantu kelancaran proses pembelajaran selama penulis menempuh kuliah dari awal hingga akhir dan yang telah membantu mempermudah dalam urusan berkas-berkas.
5. Keluarga penulis tercinta yang selalu memberikan semangat dan dukungan selama proses penyusunan skripsi.
6. Teman-teman kelompok skripsi: Donny Suparto, Yolanda Tyas Prameswari, dan Catharina Rency yang telah banyak memberikan pembelajaran selama proses penelitian berlangsung.
7. Vonny Dewi Lestari Putra yang selalu memberikan semangat dan dukungan doa dari awal hingga akhir.
viii
8. Marianno William Jefferson Sili yang selalu menemani penulis dalam proses penyusunan skripsi.
9. Fenny Marisza Sihaloho dan Kenny Kowira yang telah banyak membantu dan memberi dukungan kepada penulis.
10. Teman-teman FSM C 2013, FST A 2013 dan seluruh angkatan 2013 yang telah berbagi pengalaman, canda tawa, dan suka duka selama berada di Fakultas Farmasi Universitas Sanata Dharma.
11. Seluruh pihak yang tidak dapat penulis ucapkan namanya satu per satu yang telah mendukung penulis selama proses penyusunan skripsi.
Akhir kata, penulis menyadari bahwa skripsi ini masih sangat jauh dari kesempurnaan. Oleh karena itu penulis sangat mengharapkan kritik dan saran yang membangun dalam perbaikan skripsi ini. Semoga skripsi ini dapat memberikan manfaat bagi setiap pembacanya. Terima kasih.
ix DAFTAR ISI
HALAMAN JUDUL ...i
HALAMAN PERSETUJUAN ...ii
HALAMAN PENGESAHAN ...iii
HALAMAN PERSEMBAHAN ...iv
PERNYATAAN KEASLIAN KARYA ...v
LEMBAR PERNYATAAN PERSETUJUAN PUBLIKASI...vi
PRAKATA ...vii
DAFTAR ISI ...ix
DAFTAR TABEL ...xi
DAFTAR GAMBAR ...xii
DAFTAR LAMPIRAN ...xiii
ABSTRAK ...xiv ABSTRACT ...xv PENDAHULUAN ...1 METODE PENELITIAN ...2 Bahan ...2 Instrumentasi ...2 Metode ...2 Preparasi ligan LC20 ...2
Penambatan molekul ligan LC20...2
Konversi .mol2 menjadi .pdb...2
Penyesuaian koordinat atom ligan ...3
Perhitungan RMSD dan visualisasi ...3
Analisis statistik ...3
HASIL DAN PEMBAHASAN ...3
x
DAFTAR PUSTAKA ...8 LAMPIRAN ...10 BIOGRAFI PENULIS ...18
xi DAFTAR TABEL
xii
DAFTAR GAMBAR
Gambar 1. Struktur 2D dan 3D REF.pdb dengan koordinat yang telah disesuaikan dengan ligand_0ZD306_0.pdb ... 4 Gambar 2. Histogram seribu pose yang dikelompokkan menjadi delapan
klaster ... 5 Gambar 3. Pose ikatan REF (a), K4 (b), K5 (c), K6 (d), K7 (e), dan K8
xiii
DAFTAR LAMPIRAN
Lampiran 1. Script untuk melakukan preparasi ligan LC20 ... 11
Lampiran 2. Konfigurasi PLANTS (plants.config) ... 12
Lampiran 3. Script untuk melakukan penambatan ulang ligan LC20 ... 13
Lampiran 4. Script untuk menentukan 1000 pose terbaik ... 14
Lampiran 5. Script untuk mengubah 1000 pose terbaik format .mol2 menjadi format .pdb ... 15
Lampiran 6. Script untuk menghitung nilai RMSD 1000 pose terbaik ... 16
xiv ABSTRAK
Diabetic foor ulcer (DFU) merupakan luka tebal di bawah pergelangan
kaki hingga menembus melalui dermis. Jika dibandingkan dengan neuropati, komplikasi mikrovaskuler, dan komplikasi lain, DFU menjadi komplikasi utama pada penderita diabetes. Pada penderita DFU, matriks metalloproteinase 9 (MMP-9) diekspresikan secara berlebih. Kadar matriks metalloproteinase yang tinggi pada DFU dapat menghambat proses penyembuhan luka. Ligan LC20 dalam struktur kristal 4HMA merupakan senyawa karboksilat bifungsional yang dapat bertindak sebagai inhibitor MMP-9. Penelitian ini bertujuan untuk mengevaluasi struktur kristal 4HMA sebagai target virtual dalam penapisan virtual berbasis struktur. Ligan LC20 dipreparasi menggunakan SPORES 1.3, kemudian ditambatkan pada kantung ikatan MMP-9 menggunakan PLANTS 1.2 dengan konfigurasi yang dikembangkan oleh Anita et al. (2012). Penambatan dilakukan sebanyak 1000 replikasi dengan lima iterasi. Keberhasilan penambatan ulang diketahui melalui nilai root mean square distance (RMSD) yang tidak lebih besar daripada 2,0 Å. Data RMSD untuk 1000 pose diuji statistik dengan metode one
sample t-test untuk mengetahui nilai RMSD yang seharusnya tidak lebih besar
daripada 2,0 Å. Hasil penelitian ini menunjukkan bahwa struktur kristal 4HMA tidak sesuai sebagai target virtual dalam penapisan virtual berbasis struktur menggunakan metode PLANTS sebagai docking software dan konfigurasi yang dikembangkan oleh Anita et al. (2012) karena nilai RMSD 1000 pose lebih besar daripada 2,0 Å.
xv ABSTRACT
Diabetic foot ulcer (DFU) is defined as a full-thickness lesion below the ankle and a wound penetrating through the dermis. Compared to neuropathy, microvascular complication, and other complications, DFU becomes the main complication in diabetic patient. In DFU’s patients, matrix metalloproteinase 9 is highly expressed, therefore its high level can delay the wound healing process. LC20 ligand in the 4HMA crystal structure is a bi-functional carboxylate that is known able to act as MMP-9 inhibitor. The aim of this study was to evaluate the 4HMA crystal structure as a virtual target in structure based virtual screening. LC20 ligand were prepared and docked using SPORES 1.3 and PLANTS 1.2, respectively utilizing configuration developed by Anita et al. (2012). The docking was run in 1000 replications with five iterations. The successful control docking is indicated by the root mean square distance (RMSD) value that should not be greater than 2,0 Å. The RMSD data was statistically tested using one sample t-test to determine the RMSD should not be greater than 2,0 Å. The result showed that the 4HMA crystal structure is unsuitable used as the virtual target in structure based virtual screening employing PLANTS as a docking software and configuration developed by Anita et al. (2012) because the RMSD value is greater than 2,0 Å.
1
Diabetes merupakan sekelompok penyakit metabolik yang ditandai dengan hiperglikemia akibat kecacatan pada sekresi insulin, kinerja insulin, atau keduanya (American Diabetes Association, 2013). Sebanyak 60% populasi dunia yang menderita diabetes berasal dari Benua Asia (Ramachandran et al., 2012), dan di Indonesia sendiri diperkirakan penderita diabetes menjadi 14,1 juta jiwa pada tahun 2035 dari 8,5 juta jiwa pada tahun 2013 (Guariguata et al., 2014). Diabetic foor ulcer (DFU) merupakan luka tebal di bawah pergelangan kaki hingga menembus melalui dermis (Schaper, 2004). Jika dibandingkan dengan neuropati, komplikasi mikrovaskuler, dan komplikasi lain, DFU menjadi komplikasi utama pada penderita diabetes (Yusuf et al., 2016). DFU juga menjadi sumber utama morbiditas dan menjadi faktor risiko amputasi yang signifikan bagi penderita diabetes (Zimny et al., 2004).
Gangguan ekspresi dan aktivasi matriks metalloproteinase (MMP) menjadi salah satu penemuan penting pada diabetes. MMP merupakan sekelompok enzim yang berperan dalam degradasi matriks ekstraseluler (McLennan et al., 2008). DFU menjadi sulit disembuhkan karena tingginya kadar sitokin pro-inflamasi pada luka, yang mengakibatkan kadar MMP meningkat (Lobmann et al., 2006). Kadar MMP yang tinggi pada penderita DFU dapat menghambat proses penyembuhan luka tersebut (Muller et al., 2008), dengan mendegradasi material matriks ekstraseluler secara tidak terkendali (Sam and Krishnan, 2015).
Struktur kristal 4HMA mengandung senyawa karboksilat bifungsional N,N'-bis(2- [(biphenyl-4ylsulfonyl)[(2R)-1-hydroxyl-3-methyl-1-oxobutan-2-yl]-amino]ethyl)benzene-1,3-dicarboxamide (ligand_0ZD306_0 atau ligan LC20) yang dikristalkan dalam MMP-9 (Antoni et al., 2013). Senyawa karboksilat diketahui dapat bertindak sebagai inhibitor MMP-9 karena memiliki zinc binding group (ZBG) (Kontogiorgis et al., 2005). Penelitian ini bertujuan untuk mensimulasikan penambatan ligan LC20 pada kantung ikatan MMP-9 untuk mengetahui nilai root mean square distance (RMSD) sehingga dapat ditentukan validitas struktur kristal 4HMA sebagai target virtual dalam penapisan virtual berbasis struktur (PVBS).
2 METODE PENELITIAN
Bahan
Konfigurasi PLANTS yang dikembangkan oleh Anita et al. (2012). Struktur kristal MMP-9 dengan kode 4HMA (diambil dari RCSB PDB). Perangkat lunak SPORES 1.3 (ten Brink and Exner, 2009), PLANTS 1.2 (Korb et al., 2006), PyMOL 1.8.4, Open Babel 2.4.0, dan R statistical software 3.3.0.
Instrumentasi
Server Fakultas Farmasi Universitas Sanata Dharma (alamat IP 103.247.10.66 (pharcomp.web.id)) dan HP ProBook 4420s dengan spesifikasi: Intel Core i3 M350 @2,27 GHz, RAM 2,00 GB, 32-bit Operating System, Linux Ubuntu 16.04 LTS.
Metode
Preparasi ligan LC20
Berdasarkan inspeksi visual struktur kristal 4HMA menggunakan PyMOL 1.8.4, diambil rantai A dari dua rantai yang ada dengan perintah “grep” pada Linux Ubuntu 16.04 LTS. Ligan dan protein dipreparasi menggunakan SPORES 1.3 dengan luaran ligand.mol2 dan protein.mol2. Kemudian ligand.mol2 diubah menjadi ligand_input.smi dan dilakukan gen3d menggunakan Open Babel 2.4.0. Hasil luaran tersebut di-reprot menggunakan SPORES 1.3 menghasilkan ligan yang siap ditambatkan.
Penambatan molekul ligan LC20
Ligan ditambatkan pada kantung ikatan MMP-9 menggunakan PLANTS 1.2 melalui server dengan mengacu pada konfigurasi yang dikembangkan oleh Anita et al. (2012). Setiap satu kali replikasi (1 run) dilakukan lima kali iterasi penambatan. Luaran yang dihasilkan adalah lima pose berupa skor ChemPLP, satu pose dengan skor ChemPLP terkecil diambil. Seribu kali run dilakukan dengan lima kali iterasi sehingga didapatkan data seribu pose terbaik dari setiap run.
Konversi .mol2 menjadi .pdb
Hasil seribu pose terbaik dalam bentuk file.mol2 dari setiap run diubah menjadi file.pdb (atom hidrogen dihilangkan) dengan menggunakan Open Babel 2.4.0.
3 Penyesuaian koordinat atom ligan
Ligan yang digunakan untuk penambatan dan ligand.mol2 hasil luaran SPORES 1.3 diubah terlebih dahulu menjadi ligand_1.pdb dan ligand.pdb (atom hidrogen dihilangkan) dengan menggunakan Open Babel 2.4.0. Kemudian koordinat masing-masing atom pada ligand_1.pdb disesuaikan dengan koordinat setiap atom pada ligand.pdb. Nama ligand_1.pdb diubah menjadi ref.pdb.
Perhitungan nilai RMSD dan visualisasi
Nilai RMSD 1000 pose terbaik dihitung menggunakan PyMOL 1.8.4. Perhitungan nilai RMSD dilakukan antara ref.pdb dengan seribu pose terbaik .pdb. Hasil nilai RMSD dikelompokkan menjadi beberapa klaster dan kemudian ditentukan median setiap klasternya untuk dilakukan visualisasi pose ikatan antara ligan dan kantung ikatan MMP-9 menggunakan PyMOL 1.8.4.
Analisis statistik
Data nilai RMSD untuk 1000 pose terbaik diuji secara statistik dengan metode one sample t-test untuk mengetahui hasil nilai RMSD yang seharusnya tidak lebih besar daripada 2,0 Å menggunakan R statistical software 3.3.0.
HASIL DAN PEMBAHASAN
Penelitian ini bertujuan untuk menentukan validitas struktur kristal 4HMA sebagai target virtual enzim MMP-9 pada penapisan virtual berbasis struktur. Berdasarkan inspeksi visual struktur kristal 4HMA dengan menggunakan PyMOL 1.8.4, terdapat dua rantai enzim matriks metalloproteinase, yaitu rantai A dan rantai B. Rantai yang diambil adalah rantai A karena pada rantai ini terdapat ligan LC20. Rantai A dipisahkan dengan rantai B menggunakan perintah grep pada Linux Ubuntu 16.04 LTS dan selanjutnya dipreparasi menggunakan SPORES 1.3 dengan hasil luaran ligand_0ZD306_0.mol2 (LC20) dan protein.mol2. Kemudian ligand_0ZD306_0.mol2 diubah menjadi
ligand_input.smi dan dilakukan gen3d dengan menggunakan Open Babel 2.4.0 sehingga
didapatkan ligand_input_babel.mol2. Mode reprot dilakukan terhadap luaran tersebut dengan menggunakan SPORES 1.3 untuk menghasilkan ligand_input.mol2 yang siap ditambatkan pada kantung ikatan MMP-9.
4
sebagai acuan untuk melakukan penambatan antara ligand_input.mol2 dengan kantung ikatan MMP-9 (protein.mol2) menggunakan PLANTS 1.2. Penambatan dilakukan sebanyak 1000 replikasi dengan lima kali iterasi. Setiap iterasi dalam satu kali replikasi menghasilkan skor ChemPLP terkecil. ChemPLP merupakan fungsi penilaian dari PLANTS 1.2. Skor ChemPLP terkecil di antara lima iterasi dalam satu kali replikasi diambil sebagai pose penambatan terbaik sehingga akan didapatkan sebanyak 1000 pose terbaik. Seribu pose dalam bentuk .mol2 tersebut diubah ke dalam bentuk .pdb (atom hidrogen dihilangkan) dengan menggunakan Open Babel 2.4.0 untuk kemudian digunakan dalam perhitungan nilai RMSD.
Koordinat atom pada ligand_input.mol2 perlu disesuaikan dahulu dengan koordinat atom pada ligand_0ZD306_0.mol2. Sebelum koordinat atom disesuaikan,
ligand_input.mol2 dan ligand_0ZD306_0.mol2 diubah ke dalam bentuk .pdb (atom
hidrogen dihilangkan) dengan menggunakan Open Babel 2.4.0. Penyesuaian koordinat atom dilakukan supaya ligand_input.pdb memiliki koordinat atom yang sama dengan
ligand_0ZD306_0.pdb sehingga dapat dijadikan sebagai pembanding terhadap 1000 pose
penambatan terbaik untuk menentukan nilai RMSD masing-masing pose penambatan. Ligan yang telah disesuaikan koordinat atomnya diberi nama REF.pdb (Gambar 1).
Gambar 1. Struktur 2D dan 3D REF.pdb dengan koordinat yang telah
5
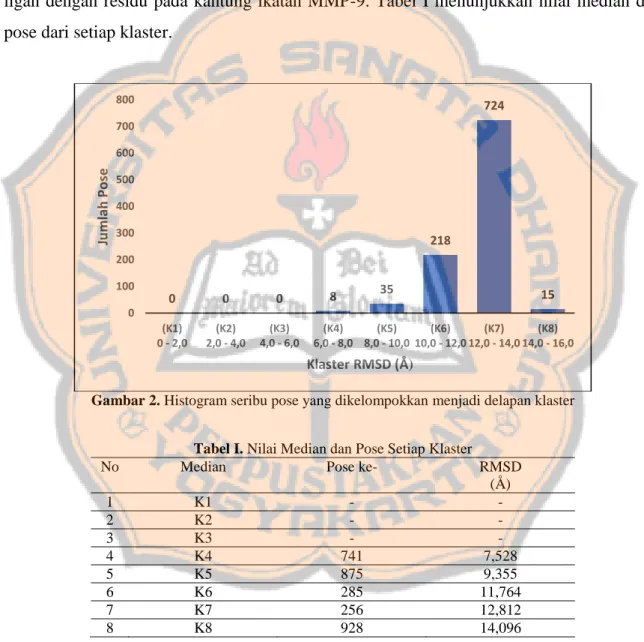
1000 pose terbaik .pdb dengan menggunakan PyMOL 1.8.4. Nilai RMSD yang dihasilkan adalah pada rentang 6,679 Å (paling rendah) sampai dengan 14,633 Å (paling tinggi). Persebaran nilai RMSD dibagi menjadi delapan klaster dan dapat dilihat dalam bentuk histogram pada Gambar 2. Klaster-klaster tersebut dibagi berdasarkan cluster_rmsd pada konfigurasi PLANTS, yaitu 2,0. Nilai median setiap klaster ditentukan untuk memperoleh data representatif dari setiap klaster yang akan digunakan untuk visualisasi pose antara ligan dengan residu pada kantung ikatan MMP-9. Tabel I menunjukkan nilai median dan pose dari setiap klaster.
Gambar 2. Histogram seribu pose yang dikelompokkan menjadi delapan klaster
Tabel I. Nilai Median dan Pose Setiap Klaster
No Median Pose ke- RMSD
(Å) 1 K1 - - 2 K2 - - 3 K3 - - 4 K4 741 7,528 5 K5 875 9,355 6 K6 285 11,764 7 K7 256 12,812 8 K8 928 14,096
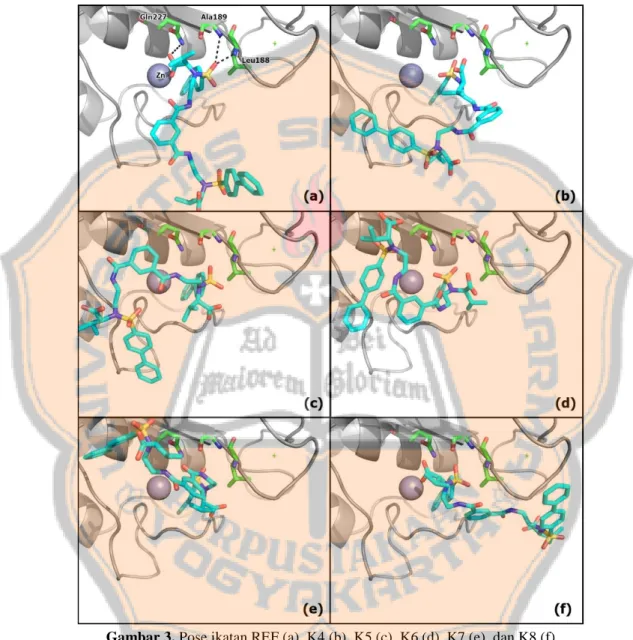
Visualisasi setiap pose ligan dilakukan untuk menggambarkan interaksi ikatan antara ligan LC20 dengan residu yang terdapat pada kantung ikatan MMP-9. Terdapat enam pose penambatan yang divisualisasikan, yaitu pose ligan REF (Gambar 3.a), K4 (Gambar 3.b), K5 (Gambar 3.c), K6 (Gambar 3.d), K7 (Gambar 3.e), dan K8 (Gambar 3.f).
0 0 0 8 35 218 724 15 0 100 200 300 400 500 600 700 800 (K1) 0 - 2,0 (K2) 2,0 - 4,0 (K3) 4,0 - 6,0 (K4) 6,0 - 8,0 (K5) 8,0 - 10,0 (K6) 10,0 - 12,0 (K7) 12,0 - 14,0 (K8) 14,0 - 16,0 Ju m lah Po se Klaster RMSD (Å)
6
yang berikatan dengan ligan LC20 ditunjukkan oleh struktur mode stick yang berwarna hijau, interaksi antara ligan dan residu ditunjukkan oleh garis putus-putus berwarna hitam. K1, K2, dan K3 tidak divisualisasikan karena tidak terdapat pose pada rentang nilai RMSD dalam klaster tersebut.
Gambar 3. Pose ikatan REF (a), K4 (b), K5 (c), K6 (d), K7 (e), dan K8 (f)
dengan residu pada kantung ikatan MMP-9
Berdasarkan inspeksi visual antara REF dengan residu pada kantung ikatan MMP-9, terdapat tiga ikatan hidrogen dan satu ikatan ionik Zn2+–O yang diduga menjadi ikatan penting. Ikatan hidrogen pertama dan kedua terjadi antara atom hidrogen pada gugus sulfonamida dengan Leu188 dan Ala189. Ikatan hidrogen ketiga terjadi antara atom hidrogen pada gugus karboksilat dengan Gln227. Ikatan ionik Zn2+–O terjadi antara atom oksigen pada gugus karboksilat dengan Zn2+ pada kantung ikatan MMP-9. Ikatan hidrogen
7
sedangkan ikatan ionik Zn –O dapat terjadi jika jarak yang dihasilkan lebih kurang 2,005 Å (Evarestov, 2015). K4, K5, K6, K7, dan K8 tidak memiliki ikatan hidrogen karena jarak yang dihasilkan antara donor-akseptor melebihi 3,5 Å, namun pada K8 terjadi ikatan ionik Zn2+–O.
Suatu ligan dapat diterima dalam protokol PVBS apabila ligan tersebut mampu menghasilkan mode ikatan yang reliabel dengan protein (Verdonk et al., 2004) dan memiliki nilai RMSD tidak lebih dari 2,0 Å (Marcou and Rognan, 2007). Nilai RMSD untuk 1000 pose terbaik diuji secara statistik dengan metode one sample t-test menggunakan R statistical software 3.3.0 untuk mengetahui nilai RMSD yang seharusnya tidak lebih besar daripada 2,0 Å. Uji statistik one sample t-test memberikan hasil nilai
p-value sama dengan 2,2 x 10-16 (p-value < 0,05). Berdasarkan hasil uji statistik one sample
t-test, struktur kristal 4HMA tidak sesuai sebagai target virtual dalam PVBS dengan
menggunakan PLANTS sebagai docking software dan konfigurasi yang dikembangkan oleh Anita et al. (2012) karena nilai RMSD 1000 pose terbaik lebih besar daripada 2,0 Å.
KESIMPULAN
Evaluasi struktur kristal 4HMA sebagai target virtual MMP-9 menggunakan konfigurasi PLANTS yang dikembangkan oleh Anita et al. (2012) menghasilkan nilai RMSD 1000 pose terbaik lebih besar daripada 2,0 Å dan bahwa lebih dari 50% pose menunjukkan ligan berada di luar target molekul sehingga menjadi fleksibel serta perhitungan RMSD dengan ko-kristal ligan menjadi tidak relevan. Oleh karena itu berdasarkan metode PLANTS sebagai docking software dan konfigurasi yang dikembangkan oleh Anita et al. (2012), struktur kristal 4HMA tidak sesuai sebagai target virtual dalam penapisan virtual berbasis struktur. Saran dari penelitian ini adalah perlu dilakukan pengembangan konfigurasi PLANTS yang sebelumnya telah dikembangkan oleh Anita et al. (2012).
8
American Diabetes Association, 2013. Diagnosis and classification of diabetes mellitus.
Diabetes Care, 36 (Suppl.1), 67 – 74.
Anita, Y., Radifar, M., Kardono, L.B., Hanafi, M., and Istyastono, E.P., 2012. Hypothesis Structure-based design of eugenol analogs as potential estrogen receptor antagonists. Bioinformation, 8 (19), 901 – 906.
Antoni, C., Vera, L., Devel, L., Catalani, M.P., Czarny, B., Cassar-Lajeunesse, E., Nuti, E., Rossello, A., Dive, V., and Stura, E.A., 2013. Crystallization of bi-functional ligand protein complexes. Journal of Structural Biology, 182 (3), 246 – 254. Evarestov, R.A., 2015. Theoretical Modeling of Inorganic Nanostructure – Symmetry and
ab-initio Calculations of Nanolayers, Nanotubes and Nanowires. London:
Springer.
Guariguata, L., Whiting, D.R., Hambleton, I., Beagley, J., Linnenkamp, U., and Shaw, J.E., 2014. Global estimates of diabetes prevalence for 2013 and projections for 2035.
Diabetes Research and Clinical Practice, 103 (2), 137 – 149.
Korb, O., Stützle, T., Exner, T.E., 2006. PLANTS: Application of Ant Colony Optimization to Structure-Based Drug Design. Journal of Chemical Information
and Modeling, 4150 (22), 247 – 258.
Lobmann, R., Zemlin, C., Motzkau, M., Reschke, K., and Lehnert, H., 2006. Expression of matrix metalloproteinases and growth factors in diabetic foot wounds treated with a protease absorbent dressing. Journal of Diabetes and Its Complications, 20 (5), 329 – 335.
Marcou, G. and Rognan, D., 2007. Optimizing fragment and scaffold docking by use of molecular interaction fingerprints. Journal of Chemical Information and
Modeling, 47 (1), 195 – 207.
McLennan, S. V., Min, D., and Yue, D.K., 2008. Matrix metalloproteinases and their roles in poor wound healing in diabetes. Wound Practice & Research, 16 (3), 116–121. Muller, M., Trocme, C., Lardy, B., Morel, F., Halimi, S., and Benhamou, P.Y., 2008.
Matrix metalloproteinases and diabetic foot ulcers: the ratio of MMP-1 to TIMP-1 is a predictor of wound healing. Diabetic Medicine, 25 (4), 419 – 426.
Ramachandran, A., Snehalatha, C., Shetty, A.S., and Nanditha, A., 2012. Trends in prevalence of diabetes in Asian countries. World Journal of Diabetes, 3 (6), 110 – 117.
Sam, J.S. and Krishnan, B., 2015. Study on matrix metalloproteinases in diabetic foot ulcer disease. Journal of Biomedical and Pharmaceutical Research, 4 (3), 56 – 65. Schaper, N.C., 2004. Diabetic foot ulcer classification system for research purposes: A
progress report on criteria for including patients in research studies.
Diabetes/Metabolism Research and Reviews, 20 (Suppl. 1), 90–95.
Shahbazi, Z., 2015.Mechanical Model of Hydrogen Bonds in Protein Molecules. American
Journal of Mechanical Engineering, 3 (2), 47 – 54.
ten Brink, T., and Exner, T.E., 2009. Influence of Protonation, Tautomeric, and Stereoisomeric States on Protein–Ligand Docking Results. Journal of Chemical
Information and Modeling, 49, 1535 – 1546.
Verdonk, M.L., Berdini, V., Hartshorn, M.J., Mooij, W.T.M., Murray, C.W., Taylor, R.D., Watson, P., 2004. Virtual Screening Using Protein–Ligand Docking: Avoiding Artificial Enrichment. Journal of Chemical Information and Modeling, 44 (3), 793-806.
9
Nakatani, T., and Sugama, J., 2016. Prevalence and Risk Factor of Diabetic Foot Ulcers in a Regional Hospital, Eastern Indonesia. Open Journal of Nursing, 6 (1), 1–10.
Zimny, S., Schatz, H., and Pfohl, M., 2004. The effects of ulcer size on the wound radius reductions and healing times in neuropathic diabetic foot ulcers. Experimental and
10
11
Lampiran 1. Script untuk melakukan preparasi ligan LC20
> wget https://files.rcsb.org/download/4HMA.pdb.gz > gunzip 4HMA.pdb.gz
> grep " A " 4HMA.pdb > 4HMAA.pdb
> /home/enade/program/PLANTS/SPORES1.3 --mode splitpdb 4HMAA.pdb
> /home/enade/program/PLANTS/PLANTS1.2 --mode bind ligand_0ZD306_0.mol2 5 protein.mol2
> cp
/home/enade/program/master_ER/Bioinformation_Anita201 2_ER-ANT/validated/WAT/VS/plants.config .
> grep -Ev bindingsite_ plants.config | grep -Ev water > plants.config2
> cat plants.config2 bindingsite.def > plants.config > mv ligand.mol2 ligand_input.mol2
> babel -imol2 ligand_input.mol2 -osmi ligand_input.smi > babel --gen3d -ismi ligand_input.smi -omol2
ligand_input_babel.mol2
> /home/enade/program/PLANTS/SPORES1.3 --mode reprot ligand_input_babel.mol2 ligand_input.mol2
12
Lampiran 2. Konfigurasi PLANTS (plants.config) # scoring function and search settings scoring_function chemplp search_speed speed2 # input protein_file protein.mol2 ligand_file ligand_input.mol2 # output output_dir results
# write single mol2 files (e.g., for RMSD calculation) write_multi_mol2 0
# cluster algorithm cluster_structures 50 cluster_rmsd 2.0
# binding site definition
bindingsite_center 11.7363 24.5261 -7.36469 bindingsite_radius 16.0101
13
Lampiran 3. Script untuk melakukan penambatan ulang ligan LC20 #!/bin/sh mkdir /home/nyomanindra/cobacoba/newdock100 cd /home/nyomanindra/cobacoba/newdock100/ for i in $(seq 1 100); do mkdir /home/nyomanindra/cobacoba/newdock100/rep_$i cd /home/nyomanindra/cobacoba/newdock100/rep_$i/ for j in $(seq 1 5); do mkdir dock_$j cd dock_$j/ cp /home/nyomanindra/cobacoba/plants.config . cp /home/nyomanindra/cobacoba/protein.mol2 . cp /home/nyomanindra/cobacoba/ligand_input.mol2 . /home/enade/program/PLANTS/PLANTS1.2 --mode screen plants.config rm plants_mod.config protein.mol2 ligand_input.mol2 cd ../ done cd ../../ done
tar -czvf seratus.tar.gz newdock100 rm -rf newdock100
14
Lampiran 4. Script untuk menentukan 1000 pose terbaik > for j in $(seq 1 1000); do for i in $(seq 1 5); do echo "`cat /home/nyomanindra/cobacoba/best/SERIBU/rep_$j/ dock_$i/ results/bestranking.csv`,$i";
done | grep -Ev TOTAL_SCORE | sort -t, -k2n | head -1 | awk -F, '{print $12}';
15
Lampiran 5. Script untuk mengubah 1000 pose terbaik format .mol2 menjadi format .pdb > best_1000=($(cat best_1000.lst)) > n=${#best_1000[@]} > for ((j=0; j<=$n; j++)); do echo "cp/home/nyomanindra/cobacoba/best/SERIBU/rep_$[j+ 1]/ dock_${best_1000[$j]}/results/4HMAA_ligand_0_entry _00001_ conf_01.mol2 rep_$ [j+1].${best_1000[$j]}.mol2"; done > copy1000.sh
> chmod u+x copy1000.sh > ./copy1000.sh
16
Lampiran 6. Script untuk menghitung nilai RMSD 1000 pose terbaik > mkdir rmsd.pilot
> cd rmsd.pilot
> ls ../pdb | grep rep | sort -t_ -k2,2n > hasil.pdb.all
> cp ../best/bestpdb/ligand_ref.pdb ref.pdb > gedit all.rmsd.sh &
#!/bin/sh
for i in $(cat hasil.pdb.all); do
cp ../pdb/$i res.pdb
pymol -c rmsd.pml | grep RMS | awk '{print $4}' > $i.rmsd done rm res.pdb > gedit rmsd.pml & load ref.pdb load res.pdb rms_cur ref,res quit
> chmod u+x all.rmsd.sh > ./all.rmsd.sh
17
Lampiran 7. Lampiran data statistik one sample t-test hasil 1000 RMSD > data=read.csv(“hasil.csv”, header=FALSE)
> t.test(data, mu=2, alt="greater") One Sample t-test
data: data
t = 296.38, df = 999, p-value < 2.2e-16
alternative hypothesis: true mean is greater than 2 95 percent confidence interval:
12.39662 Inf sample estimates: mean of x
18
BIOGRAFI PENULIS
FX Nyoman Indra Putra Nugraha lahir di Denpasar pada tanggal 3 Desember 1995. Riwayat pendidikan formal yang telah ditempuh oleh penulis adalah TK Katolik Santo Yoseph, Denpasar (2000-2001), SD Katolik Santo Yoseph II, Denpasar (2001-2007), Seminari Menengah Roh Kudus, Tuka – Bali (2007-2010), SMA Katolik Santo Yoseph, Denpasar (2010-2013). Setamat dari pendidikan SMA, penulis melanjutkan pendidikan ke jenjang yang lebih tinggi di Fakultas Farmasi Sanata Dharma Yogyakarta. Selama menjalani perkuliahan, penulis pernah aktif dalam beberapa kegiatan kepanitiaan dan sebagai asisten praktikum Kimia Dasar (2016).