HASIL DAN PEMBAHASAN Amplifikasi Gen Pit1
Gen Pit1 ekson 6 pada sapi Friesian Holstein (FH) dari lokasi BIB Lembang, BBIB singosari dan BET Cipelang; sapi pedaging (Simmental, Limousin, Angus, dan Brahman) yang berasal dari BET Cipelang telah berhasil di amplifikasi dengan menggunakan teknik Polymerase Chain Reaction (PCR). Persentase keberhasilan amplifikasi gen Pit1 dalam penelitian ini adalah 100%. Hasil amplifikasi gen Pit1 pada gel agarose 1,5% disajikan pada Gambar 4.
Keterangan : M = Marker
1 – 8 = sampel sapi pedaging 9 – 10 = sampel sapi FH
Gambar 4. Visualisasi Hasil Amplifikasi Gen Pit1 Sapi FH dan Sapi Pedaging pada Gel Agarose 1,5%
Amplifikasi gen Pit1 menggunakan mesin Termal Cycler pada penelitian ini menghasilkan fragmen dengan panjang 611 pasang basa (pb). Perkiraan panjang fragmen yang diamplifikasi dapat diketahui dengan menyesuaikan sekuens gen Pit1 yang diperoleh dari GeneBank no.akses Y15995 dan AM490263 dengan pasangan primer berdasarkan literatur Javanmard et al. (2005) disajikan pada Gambar 5.
Menurut Viljoen et al. (2005), yang harus diperhatikan dalam optimasi PCR diantaranya adalah suhu anneling, konsentrasi Mg2+, konsentrasi primer, konsentrasi DNA target; selain itu Muladno (2002) menambahkan komponen lainnya yang dibutuhkan adalah ensim Taq DNA polymerase, deoxynucleoside triphosphat (dNTP), dan larutan penyangga (buffer). Pada gen Pit1 untuk penelitian ini kondisi suhu anneling yang digunakan adalah 60 °C selama 45 detik sebagaimana yang
digunakan oleh Javanmard et al. (2005). Suhu anneling adalah suhu yang memungkinkan terjadinya penempelan primer pada DNA cetakan dan perpanjangan DNA dimulai dari primer selama proses PCR. Setiap gen memiliki suhu anneling yang berbeda untuk dapat menghasilkan panjang fragmen yang diinginkan untuk diamati.
Pendeteksian Keragaman Gen Pit1 dengan Teknik PCR-RFLP
Metode Restriction Fragment Length Polymorphism (RFLP) digunakan untuk mengindentifikasi keragaman gen Pit1 pada sapi FH dan sapi pedaging dari produk PCR dengan memanfaatkan runutan nukleotida yang bisa dikenali oleh enzim rerstriksi yang disebut sebagai situs restriksi. Sumantri et al. (2007), analisis RFLP sering digunakan untuk mendeteksi adanya keragman gen yang terkait dengan sejumlah sifat nilai ekonomis, seperti produksi dan kualitas susu; selain itu analisis RFLP digunakan untuk mendeteksi lokasi genetik dalam kromosom yang menyandikan penyakit yang diturunkan(Orita et al., 1989).
Ada tidaknya situs restriksi kemudian dapat digunakan untuk mengetahui ada tidaknya mutasi. Proses pemotongan gen Pit1 dilakukan dengan menggunakan enzim restriksi Hinf1 yang mengenali situs pemotongan pada posisi G|AnTC.
781 tgcatacaga attattttct tctcagtaag tcagtgccct cttgtggcag aaagtggata
841 aacaatgtcg gggttccctc cttaatttct tcctgtgact ctggtaaaag gagcctacat
901 gagacaagca tctaaatgtt caaaaaaact tcacatttat tattgttgaa aagctttgaa
961 ggtgttttca gcgtctttag gtttcctttt tacgttaatg ttagtactaa tatttaggaa
1021 atgtaaccta acttgatttt gatgggccta aaccatcatc tcccttcttt cctgccaact
1081 ccccacctcc cagtattgct gctaaagacg ccctggagag acactttgga gaacagaata
1141 agccttcctc tcaggagatc ctgcggatgg ctgaagaact aaacctggag aaagaagtgg
1201 tgagggtttg gttttgtaac cgaaggcaga gagaaaaacg ggtgaagaca agcctgaatc
1261 agagtttatt tactatttct aaggagcatc tcgaatgcag ataggctctc ctattgtgta
1321 atagcgagtg tttctacttt tcattccttt ctcttctcca gccaaaatag aaattagtta
1381 tttggttagc ttcaaaaaat cacatcagta atttttgcag aagtgtttct tttctacttt
1441 aaaaataaat acaatttaaa ttatgttgat gaattattct cagaaggcac attgtacatt
1501 t Keterangan : = Primer = Situs Pemotongan Alel A : 5’ --- GACAAGCCTAAATCAGAGTTTAT ---3’ Alel B : 5’ --- GACAAGCCTGAATCAGAGTTTAT ---3’
Gambar 5. Posisi Penempelan Primer pada sekuen Gen Pit1 (nomor akses Y115995 dan AM490263). Terjadi Mutasi pada Situs Pemotongan Hinf1 (G|AnTC) posisi 1256 (Javanmard et al., 2005)
Keragaman gen Pit1 yang terjadi pada penelitian ini disebabkan adanya mutasi titik yang terjadi pada basa ke 1256. Mutasi pada gen Pit1 ialah mutasi subsitusi basa (transisi) dari Guanin (G) menjadi Adenin (A) (Gambar 5). Menurut Toland (2008), keragaman DNA dapat terjadi karena akibat dari mutasi. Mutasi adalah suatu perubahan struktur kimia gen yang menyebabkan berubahnya fungsi gen. Mutasi titik atau Point Mutation adalah mutasi yang terjadi hanya pada satu titik nukleotida. Menurut Paolella (1998), mutasi titik dapat dibedakan berdasarkan tipe perubahan runutan nukleotida yaitu penghilangan (delesi), penyisipan (insersi), subtitusi (transisi dan transversi), kesalahan pembacaan (profreading errors), dan
perubahan struktur kimia pada basa.
Keterangan: M = Marker
1-10 = sampel AA, AB, BB = Genotipe
Gambar 6. Pola Pita Gen Pit1|Hinf1 pada Gel Agarose 2%
Pendeteksian keragaman gen Pit1 pada sampel dengan panjang fragmen 611 pb. Posisi pemotongan dengan menggunakan Hinf1, menghasilkan panjang fragmen 367 pb dan 244 pb. Produk PCR fragmen gen Pit1 yang telah dipotong dengan enzim restriksi Hinf1 menghasilkan tiga macam fragmen, yaitu fragmen yang tidak terpotong menghasilkan satu pita yang dikenal dengan genotipe AA dengan panjang fragmen 611 pb, menghasilkan dua pita 367 pb dan 244 pb yang dikenal dengan genotipe BB, dan fragmen gabungan yang menghasilkan tiga pita dengan panjang fragmen 611 pb, 367 pb, dan 244 pb yang dikenal dengan genotipe AB (Gambar 6).
Penelitian ini sesuai dengan Jawasreh et al. (2009) pendeteksian keragaman gen Pit1 dengan teknik PCR-RFLP dengan enzim Hinf1yang dilakukan pada sapi
Friesian di Jordania menghasilkan tiga macam fragmen, yaitu fragmen yang tidak terpotong menghasilkan satu pita yang dikenal dengan genotipe AA (611 pb), menghasilkan dua pita yang dikenal dengan genotipe BB (367 pb dan 244 pb), dan fragmen gabungan yang menghasilkan tiga pita dengan panjang fragmen yang dikenal dengan genotipe AB (611 pb, 367 pb, dan 244 pb).
Frekuensi Genotipe dan Alel Gen Pit1
Hasil analisis frekuensi genotipe dan alel sapi FH dari lokasi BIB lembang, BBIB Singosari dan BET Cipelang yang berjumlah 89 ekor; serta sapi pedaging dari BET Cipelang yang berjumlah 36 ekor disajkan pada Tabel.2.
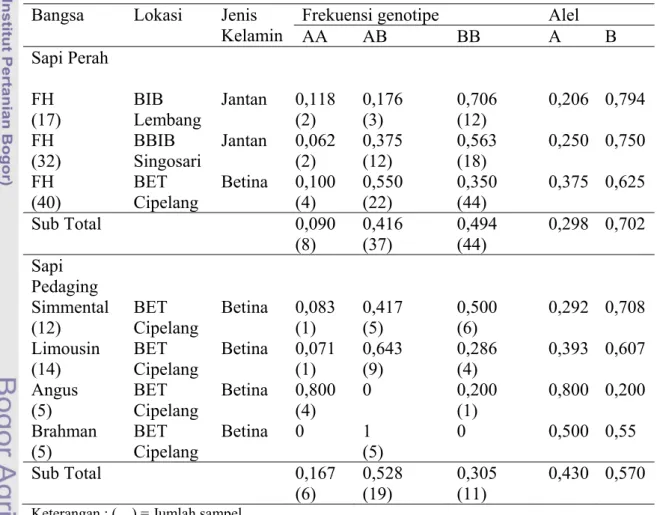
Tabel 2. Frekuensi Genotipe dan Alel Gen Pit1|Hinf1 pada Sapi Friesian Holstein
dan Sapi Pedaging
Bangsa Lokasi Jenis
Kelamin
Frekuensi genotipe Alel
AA AB BB A B Sapi Perah FH BIB Lembang Jantan 0,118 0,176 0,706 0,206 0,794 (17) (2) (3) (12) FH BBIB Singosari Jantan 0,062 0,375 0,563 0,250 0,750 (32) (2) (12) (18) FH BET Cipelang Betina 0,100 0,550 0,350 0,375 0,625 (40) (4) (22) (44) Sub Total 0,090 0,416 0,494 0,298 0,702 (8) (37) (44) Sapi Pedaging Simmental
(12) BET Cipelang Betina 0,083 0,417 (1) (5) 0,500 (6) 0,292 0,708 Limousin (14) BET Cipelang Betina 0,071 0,643 0,286 0,393 0,607 (1) (9) (4) Angus BET Cipelang Betina 0,800 0 0,200 0,800 0,200 (5) (4) (1) Brahman (5) BET Cipelang Betina 0 1 0 0,500 0,55 (5) Sub Total 0,167 0,528 0,305 0,430 0,570 (6) (19) (11)
Keterangan : (…) = Jumlah sampel
Gen Pituitary-Specific Positive Transcription Factor 1 (Pit1) merupakan faktor transkripsi spesifik pituitari yang berperan untuk perkembangan pituitari dan ekspresi hormon pada mamalia. Gen Pit1 merupakan anggota dominan POU yang
mengandung protein, yaitu kelompok regulator transkripsi yang mempunyai peran kunci dalam diferensiasi dan pembelahan sel (Mangalam et al., 1989).
Frekuensi Genotipe sapi FH di lokasi BIB Lembang frekuensi genotipe yang tertinggi adalah genotipe BB (0,706), begitu pula dengan yang terdapat di lokasi BBIB Singosari yang tertinggi adalah genotipe BB (0,563); sedangkan di lokasi BET Cipelang frekuensi genotipe yang tertinggi adalah genotipe AB (0,550) (Tabel 2). Frekuensi alel adalah frekuensi relatif dari suatu alel dalam populasi atau jumlah
suatu alel terhadap jumlah total yang terdapat dalam suatu populasi (Nei dan Kumar, 2000).
Frekuensi alel sapi FH dari ketiga lokasi tersebut memperoleh alel B (0,702) lebih tinggi dari alel A (0,298). Sapi pedaging yang berasal dari BET Cipelang terdiri bangsa Simmental, Limousin, Angus, dan Brahman. Frekuensi genotipe yang terdapat pada sapi Simmental yang tertinggi genotipe BB (0,500), sapi Limousin yang tertinggi genotipe AB, sapi Angus yang tertinggi genotipe AA (0,800); serta sapi Brahman yang tertinggi genotipe AB (1), hal ini dikarenakan dari seluruh sampel sapi Brahman yang digunakan bergenotipe AB.
Frekuensi alel yang terdapat pada sapi pedaging berbeda disetiap bangsa. Sapi Simmental mempunyai frekuensi alel B (0,708) yang lebih tinggi dari alel A (0,292), begitu pula yang terjadi pada sapi Limousin yang mempunyai frekuensi alel B (0,607) yang lebih tinggi dari alel A (0,393), sapi Angus mempunyai frekuensi alel A (0,800) yang lebih tinggi dari alel B (0,200), sedangkan pada sapi Brahman antara alel A dan alel B mempunyai frekuensi yang sama (0,500). Menurut Nei (1987) menyatakan bahwa suatu alel dapat dikatan polimorfik jika memiliki frekuensi alel sama dengan atau kurang dari 0,99. Oleh karena itu, gen Pit1 sapi FH dari ketiga lokasi yang diamati dan sapi pedaging yang berasal dari BET Cipelang bersifat polimorfik (beragam).
Keragaman genotipe gen Pit1 sapi FH dari ketiga lokasi tersebut dapat dibandingkan dengan beberapa penelitian sebelumnya. Penelitian ini sesuai dengan Edriss et al. (2008) bahwa sapi Holstein di Isafahan memiliki tiga variasi genotipe yaitu AA (0,018-0,050), AB (0,350-0,450), dan BB (0,500-0,600) dan yang tertinggi adalah genoptipe BB (0,500-0,600). Frekuensi alel yang didapatkan yaitu alel B (0,700-0,775) lebih tinggi daripada alel A (0,225-0,300). Jawasreh et al. (2009) juga
melaporkan hasil penelitiannya terhadap sapi Friesian di Jordania yaitu mempunyai frekuensi alel B (0,8255) lebih tinggi dari alel A (0,1744).
Gen Pit1 merupakan salah satu gen kandidat yang perlu dicari hubungan atau keterkaitannya dengan performa pertumbuhan, kualitas karkas dan juga performa laktasi pada beberapa bangsa sapi seperti yang telah dilaporkan oleh beberapa penelitian sebelumnya (Woollard et al. 1994; Moody et al. 1995; Zwierzhowski et al. 2001; Dybus et al. 2003; Oprzadek et al. 2003; Zhao et al. 2004; Viorica et al. 2007). Hasil penelitian yang dilakukan oleh Renaville et al. (1997) dapat diasosiasikan bahwa alel A pada lokus Pit1|HinfI dengan produksi susu dan protein superior, dan inferior untuk persentase lemak pada sapi perah. Sehubungan dengan itu, Zwierzchowski et al. (2002) menunjukkan bahwa alel A pada lokus Pit-1 berdampak positif pada sifat produksi susu. Selain itu menurut Zhao et al. (2000) melaporkan bahwa polimorfisme Pit1|HinfI menampakkan efek pada sifat pertumbuhan sapi Angus dan menjadi kandidat gen untuk digunakan dalam Marker-Assisted Selection
(MAS). Polimorfisme gen Pit-1 yang terjadi pada ekson 6, selain bisa berasosiasi dengan sifat karkas dan produksi susu, juga bisa dengan sifat seleksi yang lain, seperti berat badan umur 1 tahun (Zhao et al., 2004). Berdasarkan laporan Mattos et al. (2004), keragaman genetik gen Pit1 yang berinteraksi dengan bGH/MspI berpengaruh teradap variasi persentase lemak pada ternak perah.
Keseimbangan Hardy-Weinberg
Keseimbangan Hardy-Weinberg merupakan suatu keadaan dimana dalam suatu populasi yang terdapat faktor penghambat (mutasi, seleksi, migrasi, dan genetic
drift), maka frekuensi gen dominan dan resesif dalam suatu populasi tidak akan berubah dari satu generasi ke generasi lainnya. Keseimbangan variasi genotipe penelitian ini dengan menggunakan uji Chi-square (χ2). Hasil uji Chi-square tersebut
dapat disajikan pada Tabel 3.
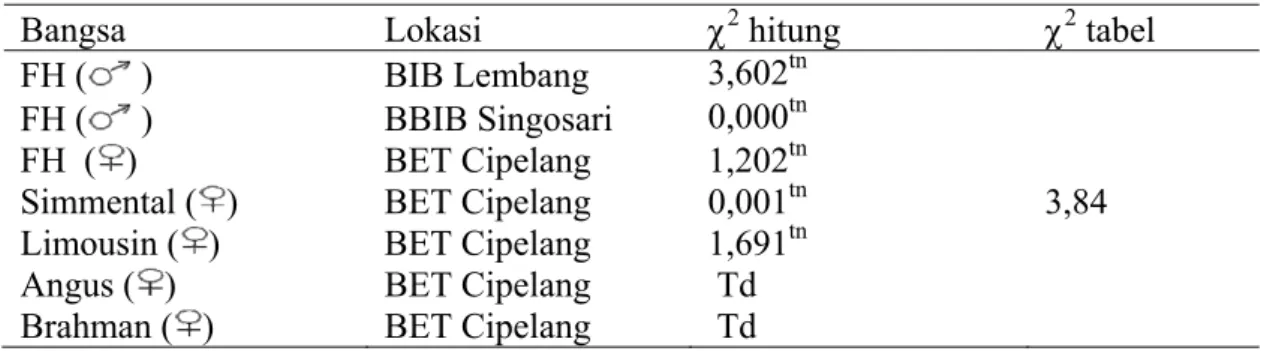
Berdasarkan hasil analisis Chi-square diperoleh χ2 hitung < χ2tabelterdapat
padasapi FH dari lokasi BIB Lembang, BBIB Singosari dan BET Cipelang berada dalam keseimbangan Hardy-Weinberg, begitu pula yang terjadi pada sapi Simmental, Limousin yang berasalah dari BET Cipelang. Menurut Noor (2000), keseimbangan
gen dalam populasi yang cukup besar terjadi jika tidak ada seleksi, mutasi, migrasi dan genetic drift.
Tabel 3. Nilai Chi-Square Gen Pit1|Hinf1
Bangsa Lokasi χ2 hitung χ2 tabel
3,602tn BIB Lembang FH ( ) 0,000tn BBIB Singosari FH ( ) FH ( ) BET Cipelang 1,202tn
Simmental ( ) BET Cipelang 0,001tn 3,84
Limousin ( ) BET Cipelang 1,691tn
Angus ( ) BET Cipelang Td
Brahman ( ) BET Cipelang Td
Keterangan : tn = tidak berbeda nyata Td = tidak dapat dianalisis
Keseimbangan yang terjadi pada sapi FH dari ketiga lokasi yang diamati, sapi Simmental dan sapi Limousin terjadi bukan dikarenakan tidak adanya seleksi, karena seluruh sapi yang berada pada lokasi tersebut merupakan hasil seleksi. Hal tersebut dapat terjadi diduga karena sifat yang diseleksi tidak terkait langsung dengan gen
Pit1, sehingga dalam keadaan keseimbangan. Pada lokasi BET Cipelang untuk sapi pedaging bangsa Angus tidak dapat dianalisis karena memiliki derajat bebas χ2
adalah 0, disebabkan pada bangsa tersebut hanya terdapat 2 macam genotipe dan 2 macam alel; sedangkan untuk bangsa Brahman tidak dapat dianalisis karena memiliki derajat bebas χ2 adalah -1, disebabkan pada bangsa tersebut hanya terdapat
1 macam genotipe dan 2 macam alel. Derajat bebas χ2 merupakan hasil pengurangan
antara jumlah genotipe dengan jumlah alel (Allendorf dan Luikart, 2007). Heterozigositas
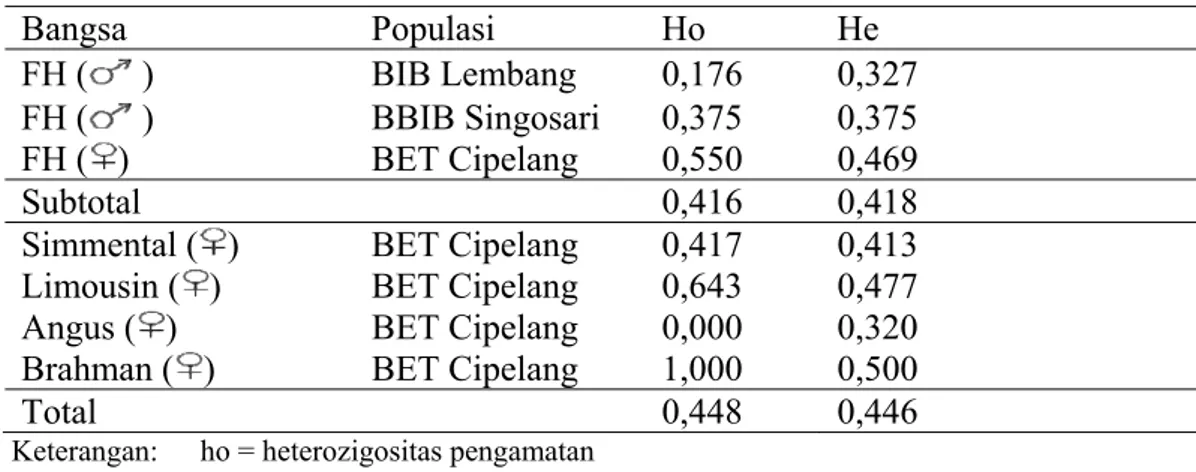
Heterozigositas disebut juga sebagai rataan keragaman genetik. Pendugaan nilai heterozigositas memiliki arti penting untuk diketahui, yaitu untuk mendapatkan gambaran variabilitas genetik (Marson et al., 2005). Berdasarkan hasil analisis nilai heterozigositas sapi FH dari tiga lokasi, nilai heterozigositas yang tertinggi adalah sapi FH dari BET Cipelang 0,550; sedangkan pada sapi pedaging nilai heterozigositas yang tetinggi adalah bangsa Brahman yaitu 1. Menurut Javanmard et al. (2005) nilai heterozigositas kurang dari 0,5 mengindikasi rendahnya variasi gen dalam suatu populasi. Avise (1994) menyatakan bahwa semakin tinggi derajat
heterozigositas suatu populasi maka daya tahan hidup populasi tersebut akan semakin tinggi. Hasil analisis heterozigositas pada tiga populasi sapi FH dan pada populasi BET Cipelang untuk sapi Simmental, Limousin, Angus dan Brahman tersaji pada Tabel 4.
Tabel 4. Heterozigositas Gen Pit1|Hinf1
Bangsa Populasi Ho He FH ( ) BIB Lembang 0,176 0,327 BBIB Singosari 0,375 0,375 FH ( ) FH ( ) BET Cipelang 0,550 0,469 Subtotal 0,416 0,418
Simmental ( ) BET Cipelang 0,417 0,413
Limousin ( ) BET Cipelang 0,643 0,477
Angus ( ) BET Cipelang 0,000 0,320
Brahman ( ) BET Cipelang 1,000 0,500
Total 0,448 0,446
Keterangan: ho = heterozigositas pengamatan he = heterozigositas harapan
Sapi FH dari BIB Lembang mempunyai nilai heterozigositas pengamatan (Ho) lebih rendah dari nilai heterozigositas harapan (He), Sapi FH dari BBIB Singosari mempunyai nilai heterozigositas pengamatan (Ho) sama dengan nilai heterozigositas harapan (He); sedangkan Sapi FH dari BET Cipelang mempunyai nilai heterozigositas pengamatan (Ho) lebih tinggi dari nilai heterozigositas harapan (He). Sapi pedaging bangsa Simmental, Limousin, dan Brahman mempunyai nilai heterozigositas pengamatan (Ho) lebih tinggi dari nilai heterozigositas harapan (He); sedangkan sapi pedaging bangsa Angus nilai heterozigositas pengamatan (Ho) lebih rendah dari nilai heterozigositas harapan (He). Tingginya nilai heterozigositas (Ho) menunjukkan adanya keragaman alel dalam populasi tersebut.