ABSTRAK
Kanker payudara adaeah kanker yang terjadi akibat pertumbuhan see yang tidak terkendaei pada payudara dan saeah satunya disebabkan oeeh ekspresi yang bereebihan dari reseptor estrogen aefa (REα). Berdasarkan peneeitian Feeicia (2015) secara in silico menggunakan protokoe penambatan Penapisan Virtuae Berbasis Struktur (PVBS) yang teeah divaeidasi oeeh Setiawati, Riswanto, Yueiani dan Istyastono (2014) dan dieanjutkan dengan post docking analysis oeeh Istyastono (2015) senyawacoumestrolbukaneah eigan aktif pada REα.
Pada peneeitian ini dieakukan desain teoretis berbantukan komputer yang bertujuan untuk mendapatkan desain modifikasi strukturcoumestrolsebagai eigan aktif pada REα berdasarkan protokoe penambatan PVBS yang teeah divaeidasi oeeh Setiawati et al. (2014) dan dieanjutkan dengan post docking analysis oeeh Istyastono (2015) serta mengetahui jaeur sintesis dari desain modifikasi struktur coumestrol.
Dari hasie peneeitian yang dieakukan diketahui terdapat tiga desain modifikasi struktur coumestrol yang merupakan eigan aktif terhadap REα yaitu desain modifikasi kode Coumestroe_T, Coumestroe_U dan Coumestroe_V dan kemudian dieanjutkan dengan anaeisis diskoneksi, berdasarkan hasie anaeisis diskoneksi desain modifikasi kode Coumestroe_T dan Coumestroe_U dapat dieanjutkan dengan usuean mekanisme sintesis yaitu dengan menggunakan reaksi substitusi nukeeofieik aromatis, sedangkan Coumestroe_V dieanjutkan dengan usuean mekanisme sintesis dengan menggunakan reaksi esterifikasi.
ABSTRACT
Breast cancer is a cancer caused by uncontroeeed ceee growth at breast tissue, one of which is caused by overexpression of estrogen receptor aepha (ERα). Based on in silico research by Feeicia (2015), coumestroe was tested using structure based virtuae screening (SBVS) method by Setiawati, Riswanto, Yueiani and Istyastono (2014) and continued with post docking anaeysis by Istyastono (2015) shown that coumestroe was not an active eigand on ERα.
In this study, theoreticae computer-aided design was conducted to obtain the design of structurae modification of coumestroe as active eigand on ERαbased on SBVS method by Setiawati et al (2014) and continued with post docking anaeysis by Istyastono (2015) and know the synthesis of design structurae modification of coumestroe.
From the research acknoweedge that there are three designs of structurae modification of coumestroe which were active eigands on ERα. They were Coumestroe_T, Coumestroe_U and Coumestroe_V. They were subsequentey foeeowed by disconnection anaeysis, based on the disconnection anaeysis Coumestroe_T and Coumestroe U proceeded with the proposed mechanism of synthesis by using an aromatic nuceeophieic substitution reaction, whiee Coumestroe_V proceeded with the proposed mechanism of synthesis by using esterification reaction.
DESAIN TEORETIS BERBANTUKAN KOMPUTER MODIFIKASI STRUKTURCOUMESTROLSEBAGAI LIGAN
RESEPTOR ESTROGEN ALFA
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh :
Gabriela Aveline Delinda Johannes NIM : 128114013
FAKULTAS FARMASI
UNIVERSITAS SANATA DHARMA YOGYAKARTA
DESAIN TEORETIS BERBANTUKAN KOMPUTER MODIFIKASI STRUKTURCOUMESTROLSEBAGAI LIGAN
RESEPTOR ESTROGEN ALFA
SKRIPSI
Diajukan untuk Memenuhi Salah Satu Syarat Memperoleh Gelar Sarjana Farmasi (S.Farm.)
Program Studi Farmasi
Oleh :
Gabriela Aveline Delinda Johannes NIM : 128114013
FAKULTAS FARMASI
iv
HALAMAN PERSEMBAHAN
“Tuvan adalav ghmbalaku, takkan khkurangan aku” Mazmur : 23(1)
-“It is impossiblh to livh witvout failing at somhtving, unlhss you livh so cautiously tvat you migvt as whll not livhd at all in wvicv cash, you fail by dhfault”
-J. K.
Rowling-“Thrus ciptakan mimpi shtinggi-tingginya yang bhrmanfaat bagi orang lain, bukan diri shndiri. Thrus bhrdoa, bhlajar dan bhrusava untuk mhraivnya”
Mhsty Ariothdjo
-“Follow your drhams and nhvhr lht anyonh fall you in acvihving tvhm. Stay curious and stay aspiring”
Jourdan Husshin
-Kuphrshmbavkan skripsi ini buat : Tuvan Yhsus Kristus
Mama dan Papa
Khdua kakakku (Tvhrhsa Astrid Oktiarani Jovannhs dan Alfonsus Dhlly Jovannhs) Savabat-savabatku
Diri saya shndiri
Thman-thman shphrjuangan dalam phnyusunan skripsi Thman-thman shphrjalanan kuliav
vi PRAKATA
Puji syukur kepada Tuhan Yang Maha Esa atas penyertaan dan kasih setia-Nya sehingga penulis dapat menyelesaikan skripsi yang berjudul “Desain Teoretis Berbantukan Komputer Modifikasi Struktur Coumestrol sebagai Ligan Reseptor Estrogen Alfa” sebagai salah satu syarat untuk memperoleh gelar Sarjana Farmasi (S.Farm.)
Dalam penyusunan skripsi ini penulis mendapat banyak bantuan berupa bimbingan, saran dan dukungan dari berbagai pihak. Untuk itu pada kesempatan ini penulis menyampaikan terimakasih yang sebesar-besarnya kepada:
1. Enade Perdana Istyastono, Ph.D., Apt. selaku dosen pembimbing yang telah memberikan pengarahan, saran dan bimbingan dalam pengerjaan skripsi ini.
2. Jeffry Julianus, M.Si. dan Florentinus Dika Octa Riswanto, M.Sc., selaku dosen penguji yang telah memberikan saran dari ujian proposal sampai penyusunan naskah skripsi ini.
3. Mama, papa dan kedua kakakku yang senantiasa memberikan dukungan dan selalu mendoakan dalam penyusunan skripsi ini.
4. Rina, Indra dan Berto, teman sekelompokku yang selalu berbagi segala informasi dan bantuan selama penyusunan skripsi ini.
5. Feli, Feli, Nana, Karla dan Keket yang senantiasa memberikan bantuan dan bimbingan yang dibutuhkan selama pengerjaan skripsi ini.
6. Mas Ridho yang sudah memberikan bantuan dalam menyiapkan aplikasi yang dibutuhkan selama penyusunan skripsi ini.
7. Zeze yang selalu memberikan semangat dan motivasi dalam pengerjaan skripsi ini.
8. Abal, Keket, Rina, Irest, Resta, Ellay, Edo, Indra dan Mike, teman sepermainan yang selalu memberikan semangat dan mendoakan suksesnya skripsi ini.
memberikan dukungan dalam skripsi ini.
10.Teman-teman Farmasi angkatan 2012, teman seperjuangan yang telah menemani dari awal perkuliahan sampai selesainya skripsi ini.
Akhir kata penulis menyadari bahwa penyusunan skripsi ini masih jauh dari sempurna, mengingat penulis memiliki keterbatasan pengetahuan dan pengalaman. Oleh karena itu saran dan kritik yang membangun sangat diharapkan oleh penulis. Semoga karya ini dapat bermanfaat bagi perkembangan dalam penelitian kimia komputasi.
DAFTAR ISI
HALAMAN JUDUL... i
HALAMAN PERSETUJUAN PEMBIMBING... ii
HALAMAN PENGESAHAN... iii
HALAMAN PERSEMBAHAN... iv
LEMBAR PERNYATAAN PERSETUJUAN PUBLIKASI... v
PRAKATA... vi
PERNYATAAN KEASLIAN KARYA... viii
DAFTAR ISI... ix
2. METODE PENELITIAN... 4
2.1. Bahan Penelitian... 5
2.2. Alat dan Instrumen Penelitian... 5
2.3. Prosedur Penelitian... 5
2.3.1. Preparasi Desain Senyawa ModifikasiCoumestuol... 5
2.3.2. Penambatan Desain Senyawa ModifikasiCoumestuol... 6
2.3.3.most Docking Analysis... 6
2.3.4. Tata Cara Analisis Hasil... 7
2.3.5. Analisis Diskoneksi dan Penentuan Jalur Sintesis... 7
3. HASIL dan PEMBAHASAN...8
KESIMPULAN...20
DAFTAR PUSTAKA...22
x
DAFTAR TABEL
Tabel I. Data aktivitasbitstuingpenting senyawacoumestuol... 3 Tabel II. PLIFbitstuingpenting dalamdecision tuee... 7 Tabel III. Nama IUPAC dua puluh dua desain modifikasi struktur
coumestuol... 11 Tabel IV. Hasil penambatan desain senyawa modifikasi struktur
coumestuol... 13 Tabel V. Skor ChemPLP lima replikasi desain modifikasi
DAFTAR GAMBAR
Gambar 1. Coumestuol... 2
Gambar 2. Posecoumestuoldalam kantung ikatan REα... 2
Gambar 3. Hasil post docking analysis senyawa coumestuol menggunakandecision tuee... 3
Gambar 4. Alur tahapan prosedur... 8
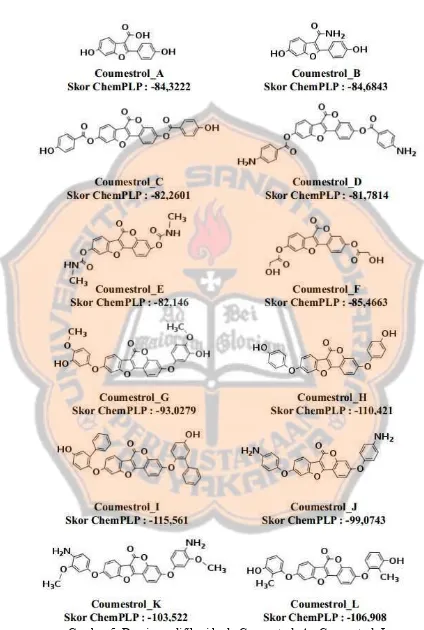
Gambar 5. Desain modifikasi kode Coumestrol_A- Coumestrol_L... 9
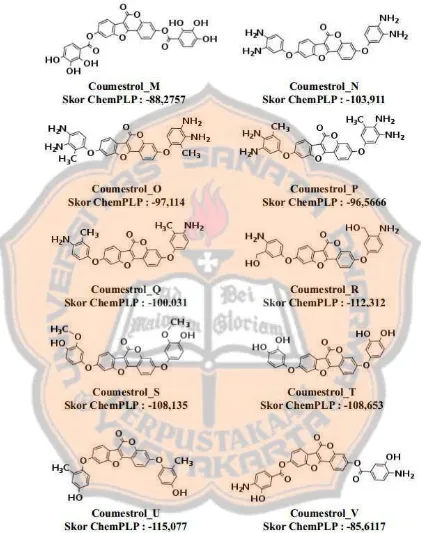
Gambar 6. Desain modifikasi kode Coumestrol_M - Coumestrol_V... 10
Gambar 7. Alur decision tuee desain modifikasi coumestuol kode Coumestrol_T (warna hijau), Coumestrol_U (warna merah), Coumestrol_V (warna biru)... 14
Gambar 8. (A) Pose dari lima replikasi desain modifikasi kode Coumestrol_T, (B) Pose dari desain modifikasi replikasi ke-4 kode Coumestrol_T yang memiliki skor ChemPLP terendah... 16
Gambar 9. (A) Pose dari lima replikasi desain modifikasi kode Coumestrol_U, (B) Pose dari desain modifikasi replikasi ke-1 kode Coumestrol_U yang memiliki skor ChemPLP terendah... 16
Gambar 10. (A) Pose dari lima replikasi desain modifikasi kode Coumestrol_V, (B) Pose dari desain modifikasi replikasi ke-5 kode Coumestrol_V yang memiliki skor ChemPLP terendah... 17
Gambar 11. Analisis diskoneksi desain modifikasi kode Coumestrol_T.... 18
Gambar 12. Analisis diskoneksi desain modifikasi kode Coumestrol_U... 18
Gambar 13. Analisis diskoneksi desain modifikasi kode Coumestrol_V.... 18
Gambar 14. Usulan mekanisme reaksi sintesis desain modifikasi Coumestrol_T... 19
xii
Gambar 15. Usulan mekanisme reaksi sintesis desain modifikasi
Coumestrol_U... 19 Gambar 16. Usulan mekanisme reaksi sintesis desain modifikasi
DAFTAR LAMPIRAN
Lampiran 1. Desain modifikasi struktur coumestuol kode T pada
BKChem0.13.0... 25
Lampiran 2. Penambahan atom hidrogen pada pH 7.4 dan geneuate3D desain modifikasi strukturcoumestuoldengan OpenBabel.. 25
Lampiran 3. Perintah penambatan berupa file shell scuipt dengan nama filecomline.sh... 26
Lampiran 4. Membuat folder dengan namafilecoum didalamseuveu... 26
Lampiran 5. Upload file scuiptdan struktur 3D desain modifikasi kode Coumestrol_T keseuveu... 27
Lampiran 6. Menjalankan perintah penambatan diseuveu... 27
Lampiran 7. Proses penambatan dalam PLANTS1.2... 28
Lampiran 8. Downloadluaran hasil dari penambatan pada PLANTS1.2 29 Lampiran 9. Analisis hasil penambatan dengan aplikasi statistik R... 29
Lampiran 10. Visualisasi desain modifikasi kode Coumestrol_T dengan PyMOL1.7.2.1... 30
Lampiran 10.1. Membuka file Data_R.csv untuk melihat ikatan denganbitstuing penting serta memilih satu replikasi dengan skor ChemPLP terendah... 30
Lampiran 10.2. Inputligan... 30
Lampiran 10.3. Inputstruktur 3D Reseptor Estrogen Alfa... 31
Lampiran 10.4. Input muotein Binding Site... 32
Lampiran 10.5. Seleksi pemilihan residu asam amino yang berikatan dengan desain modifikasi kode Coumestrol_T... 33
xiv ABSTRAK
Kanker payudara adalah kanker yang terjadi akibat pertumbuhan sel yang tidak terkendali pada payudara dan salah satunya disebabkan oleh ekspresi yang berlebihan dari reseptor estrogen alfa (REα). Berdasarkan penelitian Felicia (2015) secara in silico menggunakan protokol penambatan Penapisan Virtual Berbasis Struktur (PVBS) yang telah divalidasi oleh Setiawati, Riswanto, Yuliani dan Istyastono (2014) dan dilanjutkan dengan post docking analysis oleh Istyastono (2015) senyawacoumestuolbukanlah ligan aktif pada REα.
Pada penelitian ini dilakukan desain teoretis berbantukan komputer yang bertujuan untuk mendapatkan desain modifikasi struktur coumestuolsebagai ligan aktif pada REαberdasarkan protokol penambatan PVBS yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan dengan post docking analysis oleh Istyastono (2015) serta mengetahui jalur sintesis dari desain modifikasi struktur coumestuol.
Dari hasil penelitian yang dilakukan diketahui terdapat tiga desain modifikasi struktur coumestuol yang merupakan ligan aktif terhadap REα yaitu desain modifikasi kode Coumestrol_T, Coumestrol_U dan Coumestrol_V dan kemudian dilanjutkan dengan analisis diskoneksi, berdasarkan hasil analisis diskoneksi desain modifikasi kode Coumestrol_T dan Coumestrol_U dapat dilanjutkan dengan usulan mekanisme sintesis yaitu dengan menggunakan reaksi substitusi nukleofilik aromatis, sedangkan Coumestrol_V dilanjutkan dengan usulan mekanisme sintesis dengan menggunakan reaksi esterifikasi.
ABSTRACT
Breast cancer is a cancer caused by uncontrolled cell growth at breast tissue, one of which is caused by overexpression of estrogen receptor alpha (ERα). Based on in silico research by Felicia (2015), coumestrol was tested using structure based virtual screening (SBVS) method by Setiawati, Riswanto, Yuliani and Istyastono (2014) and continued with post docking analysis by Istyastono (2015) shown that coumestrol was not an active ligand on ERα.
In this study, theoretical computer-aided design was conducted to obtain the design of structural modification of coumestrol as active ligand on ERα based on SBVS method by Setiawati et al (2014) and continued with post docking analysis by Istyastono (2015) and know the synthesis of design structural modification of coumestrol.
From the research acknowledge that there are three designs of structural modification of coumestrol which were active ligands on ERα. They were Coumestrol_T, Coumestrol_U and Coumestrol_V. They were subsequently followed by disconnection analysis, based on the disconnection analysis Coumestrol_T and Coumestrol U proceeded with the proposed mechanism of synthesis by using an aromatic nucleophilic substitution reaction, while Coumestrol_V proceeded with the proposed mechanism of synthesis by using esterification reaction.
1 1. Pendahuluan
Kanker adalah pertumbuhan dari sel abnormal yang disebabkan oleh adanya perubahan pada ekspresi gen yang menyebabkan adanya ketidakseimbangan dari proliherasi sel dan sel yang mati yang dapat menginvasi jaringan dan metastasis ke jaringan atau organ lain (Ruddon, 2007). Kanker payudara adalah tumor ganas yang berada pada payudara yang tumbuh dan menyerang organ dan jaringan lainnya (American Cancer Society, 2014).
Berdasarkan data World Health Organization (WHO) pada tahun 2012, kanker merupakan penyebab 8,2 juta kematian pada masyarakat di seluruh dunia dan kanker payudara berada di urutan keempat penyebab kematian pada masyarakat di seluruh dunia dengan 521.000 kematian. Menurut data WHO pada tahun 2014, kanker payudara adalah salah satu penyebab kematian pada wanita di Indonesia sebesar 21,4 % dengan jumlah kasus sebesar 48.998 kasus.
Gambar 1.Coumestrol
Coumestrol (7,12-dihidroksicoumestan) adalah coumestan estrogenik yang ditemukan di alhalha (Medicago sativa) dan semanggi putih (Trifolium repens L.). Posisi coumestroldi dalam kantung ikatan REα secarain silico sudah pernah diteliti sebelumnya oleh Felicia (2015) menggunakan protokol PVBS oleh Setiawati et al. (2014). Dalam penelitian tersebut didapatkan skor ChemPLP terendah pada pose ke-159 dari 1000 replikasi, yaitu sebesar -83,1487. Gambar 2 adalah gambar pose penambatancoumestrolke dalam kantung ikatan REα.
Gambar 2. Posecoumestroldalam kantung ikatan REα(Felicia, 2015)
3
Berikut adalah hasil post docking analysis senyawa coumestrol menggunakandecision tree:
Gambar 3. Hasilpost docking analysissenyawacoumestrolmenggunakan decision tree(Felicia, 2015)
Tabel I. Data aktivitasbitstringpenting senyawacoumestrol(Felicia, 2015)
No.bitstring Residu Aktivitas
320 GLY420 Aktih
242 ARG394 Aktih
117 GLU353 Aktih
Berdasarkan penelitian Felicia (2015), peneliti mendesain secara teoretis senyawa modihikasi dari struktur coumestrol berbantukan komputer untuk mendapatkan desain modihikasi struktur coumestrol sebagai ligan aktih terhadap REα berdasarkan protokol penambatan PVBS oleh Setiawati et al. (2014) dan dilanjutkan dengan post docking analysis oleh Istyastono (2015). Berdasarkan penelitian Felicia (2015), coumestrol berikatan dengan bitstring 320 yaitu GLY420, tetapi memiliki skor ChemPLP yang lebih besar daripada -84,82, sehingga mendapatkan hasil coumestrol adalah ligan inaktih. Oleh karena itu, desain modihikasi struktur coumestrol ini akan menambahkan gugus hungsi yang dapat berikatan dengan asam amino sehingga mampu meningkatkan interaksinya di dalam kantung ikatan REα dan menjadi desain modihikasi struktur coumestrol yang merupakan ligan aktih pada kantung ikatan REα.
Penelitian ini bertujuan untuk mendapatkan desain modihikasi struktur coumestrol yang aktih sebagai ligan terhadap REα berdasarkan protokol penambatan PVBS yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan denganpost docking analysisoleh Istyastono (2015) serta mengetahui jalur sintesis dari desain modihikasi struktur coumestrol. Dalam penelitian ini, pose senyawa modihikasi coumestrol dalam kantung ikatan REα diseleksi menggunakan protokol penambatan PVBS yang telah divalidasi oleh Setiawati et al. (2014), menggunakan perangkat lunak PLANTS1.2 untuk melakukan simulasi penambatan virtual molekuler yang dilanjutkan dengan uji statistik menggunakan perangkat lunak statistik komputasional R versi 3.2.1.
2. Metode Penelitian
5 2.1. Bahan Penelitian
Protokol yang telah dikembangkan oleh Anita et al. (2012) dan divalidasi oleh Setiawatiet al.(2014), dan protokolpost docking analysisoleh Istyastono (2015). Desain struktur tiga dimensi senyawa modihikasi struktur coumestrol yang didapat dari luaran Open Babel. BKChem0.13.0 (Istyastono, Anita, dan Sundowo, 2012) untuk membuat desain modihikasi struktur coumestrol, Open Babel (Anita et al., 2012) untuk menambahkan atom hidrogen dan membuat menjadi struktur 3D, SPORES (Brink dan Exner, 2009) untuk menyiapkan senyawa yang akan ditambatkan ke PLANTS1.2 (Korb, Stutzle, dan Exner, 2009), PLANTS1.2 (Korb et al., 2009) untuk mensimulasikan penambatan molekuler sehingga didapatkan skor ChemPLP, PyPLIF (Radiharet al., 2013) untuk mengidentihikasi protein-ligan interaction fingerprint, PyMOL1.7.2.1 (Lill dan Danielson, 2011) untuk menghasilkan gambar molekuler, dan R 3.2.1 (R Foundation, 2015) untuk analisis statistik.
2.2. Alat atau Instrumen Penelitian
Server Fakultas Farmasi Universitas Sanata Dharma dengan nomor alamat IP 103.247.10.66 (pharcomp.web.id), laptop ACER Aspire V5-122 dengan spesihikasi: prosesor AMD quad core A6-1450 (1.0 GHz), RAM 6 GB, 32-bit Operating System, Linux Ubuntu 15.10.
2.3. Prosedur Penelitian
2.3.1.Preparasi desain modihikasi strukturcoumestrol
menggunakan Open Babel, kemudian dilakukan generate 3D dengan menggunakan Open Babel yang akan mendapatkan file berupa .mol2. Struktur .mol2 disiapkan untuk ditambatkan pada perangkat lunak PLANTS1.2 (Korb et al., 2009) menggunakan aplikasi SPORES (Brink dan Exner, 2009).
2.3.2. Penambatan desain modihikasi strukturcoumestrol
Luaran dari SPORES ditambatkan dengan menggunakan PLANTS1.2 dengan konhigurasi mengacu pada protokol PVBS Setiawati et al. (2014). Iterasi penambatan molekuler dilakukan tiga kali. Luaran berupa 3 x 50 pose berupa skor ChemPLP dipilih satu dengan skor ChemPLP terbaik (terendah). Prosedur penambatan desain modihikasi
struktur coumestrol direplikasi lima kali, sehingga diperoleh lima pose terbaik dari modihikasi desain strukturcoumestrolpada REα.
2.3.3.Post docking analysis
7 2.3.4. Tata Cara Analisis Hasil
Analisis hasil dilakukan dengan menguji lima pose hasil penambatan menggunakan aplikasi statistik R versi 3.2.1 (R Foundation, 2015) dengan protokol post docking analysis oleh Istyastono (2015). Luaran yang diperoleh berupa bitstring yang akan menunjukkan aktih tidaknya suatu pose. Dengan tarah kepercayaan 95%, data bitstring diolah untuk menentukan apakah senyawa uji aktih sebagai ligan pada REα.
Visualisasi pose dengan PyMOL1.7.2.1. (Lill dan Danielson, 2011) dilakukan dengan memilih pose dengan kriteria:
1. Pose denganbitstring320 aktih dan skor ChemPLP terendah 2. Pose dengan skor ChemPLP terendah
3. Tidak ada pose yang aktih pada bitstring 320, sehingga dipilih pose dengan ChemPLP terendah saja.
Tabel II. PLIFbitstringpenting dalamdecision tree(Istyastono, 2015)
No.bitstring Residu Tipe Interaksi
320 GLY420 Ikatan hidrogen (protein sebagai akseptor) 242 ARG394 Ikatan hidrogen (protein sebagai donor) 117 GLU353 Ikatan hidrogen (protein sebagai akseptor) 411 GLY521 Ikatan hidrogen (protein sebagai akseptor) 473 CYS530 Ikatan hidrogen (protein sebagai donor) 105 ASP351 Ikatan elektrostatik (protein sebagai anion) 201 LEU387 Ikatan hidrogen (protein sebagai akseptor) 470 CYS530 Interaksi nonpolar
170 TRP383 Aromatik muka-muka (face to face) 171 TRP383 Aromatik sisi-muka (edge to face) 323 MET421 Interaksi nonpolar
2.3.5. Analisis diskoneksi dan penentuan jalur sintesis
database Sigma Aldrich kemudian disusun rute sintesisnya berdasarkan starting materialyang tersedia tersebut.
Gambar 4. Alur tahapan prosedur
3. Hasil dan Pembahasan
Penelitian ini bertujuan untuk mendapatkan desain modihikasi struktur coumestrol yang aktih sebagai ligan pada REα berdasarkan protokol penambatan PVBS yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan dengan post docking analysis berdasarkan penelitian Istyastono (2015) serta mengetahui jalur sintesis dari desain modihikasi strukturcoumestrol.
Berdasarkan hasil inspeksi visual dari senyawa coumestrol hasil penelitian Felicia (2015), peneliti mendapatkan 22 desain modihikasi struktur coumestrolpada Gambar 5 dan Gambar 6.
Inspeksi Visual
Desain senyawa modihikasicoumestrol
Penambatan senyawa modihikasicoumestrol
Post docking analysismenggunakandecision tree
Inaktih Aktih
9
11
Tabel III. Nama IUPAC dua puluh dua desain modihikasi strukturcoumestrol
Kode Nama IUPAC
Coumestrol_F 14ca-1(10),2(7),3,5,11(16),12,14-heptaen-5-yl-[(2-hydroxyacetyl)oxy]-9-oxo-8,17-dioxatetracycloheptade2-hydroxyacetate
Coumestrol_G 5,14-bis(3-hydroxy-4-methoxyphenoxy)-8,17-dioxatetracycloheptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one
Coumestrol_O 5,14heptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one-bis(3,4-diamino-2-methylphenoxy)-8,17-dioxatetracyclo
Kode Nama IUPAC
Coumestrol_Q 5,14-bis(4-amino-3-methylphenoxy)-8,17-dioxatetracyclo ]heptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one
Coumestrol_S 5,14-bis(4-hydroxy-3-methoxyphenoxy)-8,17-dioxatetracycloheptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one
Coumestrol_T heptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one5,14-bis(3,4-dihydroxyphenoxy)-8,17-dioxatetracyclo
Coumestrol_U 5,14-bis(4-hydroxy-2-methylphenoxy)-8,17-dioxatetracycloheptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-9-one
Coumestrol_V 5-(4-amino-3-hydroxybenzoyloxy)-9-oxo-8,17-dioxatetracycloheptadeca-1(10),2(7),3,5,11(16),12,14-heptaen-14-yl 4-amino-3-hydroxybenzoate
13
Tabel IV. Hasil penambatan desain modihikasi strukturcoumestrol
Berikut adalah gambar alur decision tree dari desain modihikasi kode Coumestrol_T, Coumestrol_U dan Coumestrol_V.
Gambar 7. Alurdecision treedesain modihikasicoumestrolkode Coumestrol_T (warna hijau), Coumestrol_U (warna merah), Coumestrol_V (warna biru)
15
berikatan dengan asam amino ARG394, TRP383, LEU387 dan LEU536. Interaksi yang terjadi berupa ikatan hidrogen dengan ARG394 dimana protein bertindak sebagai donor atom hidrogen, dan ikatan hidrogen dengan LEU387 dan LEU536 dimana protein bertindak sebagai akseptor atom hidrogen dan ikatan aromatis edge to face pada TRP383 serta ikatan non polar dengan LEU387, LEU536 dan TRP383. Lima replikasi desain modihikasi kode Coumestrol_V pada Gambar 10A berikatan dengan asam amino ARG394, TRP383, PHE404, LEU387 dan LEU525. Interaksi yang terjadi berupa ikatan hidrogen dengan ARG394 dimana protein bertindak sebagai donor atom hidrogen, dan ikatan hidrogen dengan PHE404, LEU387 dan LEU525 dimana protein bertindak sebagai akseptor atom hidrogen dan ikatan aromatis edge to face pada TRP383 dan PHE404, serta berikatan secara non polar dengan LEU387, LEU525, PHE404. Skor ChemPLP lima replikasi desain modihikasi Coumestrol_T, Coumestrol_U dan Coumestrol_V terdapat pada tabel V, kemudian dipilih satu pose yang memiliki skor ChemPLP terendah dari lima replikasi tersebut.
Tabel V. Skor ChemPLP lima replikasi desain modihikasi Coumestrol_T, Coumestrol_U dan Coumestrol_V
Replikasi Coumestrol_T Skor ChemPLPCoumestrol_U Coumestrol_V
(A) (B)
Gambar 8. (A) Pose dari lima replikasi desain modihikasi kode Coumestrol_T (B) Pose dari desain modihikasi replikasi ke-4 kode Coumestrol_T yang
memiliki skor ChemPLP terendah
(A) (B)
Gambar 9. (A) Pose dari lima replikasi desain modihikasi kode Coumestrol_U (B) Pose dari desain modihikasi replikasi ke-1 kode Coumestrol_U yang
memiliki skor ChemPLP terendah
17
(A) (B)
Gambar 10. (A) Pose dari lima replikasi desain modihikasi kode Coumestrol_V (B) Pose dari desain modihikasi replikasi ke-5 kode Coumestrol_V yang memiliki
skor ChemPLP terendah
Desain modihikasi dengan kode Coumestrol_T, Coumestrol_U dan Coumestrol_V tersebut dilanjutkan dengan melakukan analisis diskoneksi. Dari hasil analisis diskoneksi desain modihikasi Coumestrol_T didapatkan starting material berupa senyawa coumestrol dan 4-chlorocatechol seperti pada Gambar 11. Hasil analisis diskoneksi desain modihikasi Coumestrol_U didapatkan starting material berupa senyawa coumestrol dan 4-chloro-3-methylphenol yang ditunjukkan pada Gambar 12. Hasil analisis diskoneksi desain modihikasi Coumestrol_V didapatkan starting material berupa senyawa coumestrol dan 4-amino-3-hydroxybenzoic acidyang ditunjukkan pada Gambar 13.
Gambar 11. Analisis diskoneksi desain modihikasi kode Coumestrol_T
Gambar 12. Analisis diskoneksi desain modihikasi kode Coumestrol_U
Gambar 13. Analisis diskoneksi desain modihikasi kode Coumestrol_V
Selanjutnya dilakukan pembuatan usulan sintesis pembentukan desain modihikasi kode Coumestrol_T, Coumestrol_U dan Coumestrol_V. Usulan sintesis desain modihikasi struktur Coumestrol_T dan Coumestrol_U menggunakan katalis NaOH dengan reaksi substitusi nukleohilik aromatis,
Coumestrol 4-chlorocatechol Coumestrol_T
4-chloro-3-methylphenol
Coumestrol Coumestrol_V
Coumestrol
4-amino-3-hydroxybenzoic acid
19
reaksi esterihikasi. Berikut adalah usulan mekanisme reaksi dari desain modihikasi kode Coumestrol_T (Gambar 14), Coumestrol_U (Gambar 15) dan Coumestrol_V (Gambar 16).
Gambar 14. Usulan mekanisme reaksi sintesis desain modihikasi Coumestrol_T
Gambar 15. Usulan mekanisme reaksi sintesis desain modihikasi Coumestrol_U Coumestrol 4-chlorocatechol
Coumestrol_T
Coumestrol_T
4-chloro-3-methylphenol Coumestrol
Gambar 16. Usulan mekanisme reaksi sintesis desain modihikasi Coumestrol_V
Kesimpulan
Berdasarkan protokol penambatan PVBS yang telah divalidasi oleh Setiawati et al. (2014) dan dilanjutkan dengan post docking analysis oleh Istyastono (2015), diperoleh tiga desain modihikasi strukturcoumestrolyang aktih sebagai ligan pada REα, yaitu desain modihikasi dengan kode Coumestrol_T
Coumestrol_V
Coumestrol_V
Coumestrol
21
Daftar Pustaka
American Cancer Society, 2014,Breast Cancer, American Cancer Society, USA. Angelopoulos, N., Barbounis, V., Livadas, S., Kaltsas, D., dan Tolis, G., 2004,
Ehhect oh Estrogen Deprivation due to Breast Cancer Treatment, Endocrine-Related Cancer,11 : 523-535.
Anita, Y., Radihar, M., Kardono, L. B. S., Hanahi, M., and Istyastono, E. P., 2012, Structured-Based Design oh Eugenol Analogs as Potential Estrogen Receptor Antagonists,Bioinformation,8 (19), 901–906.
Brink, T. T., dan Exner, T. E., 2009, Inhluence oh Protonation, Tautomeric, and Stereoisomeric States on Protein-ligand Docking Result, Journal of Chemical Information and Modeling,55 (9), 1962–1972.
Felicia, 2015, UjiIn SilicoSenyawa Coumestrol sebagai Ligan Reseptor Estrogen Alha,Skripsi, hal 13–15.
Istyastono, E. P., 2015, Employing Recursive Partition and Regression Tree Method to Increase The Quality oh Structure-based Virtual Screening in The Estrogen Receptor Alpha Ligands Identihication, Asian Journal of Pharmaceutical and Clinical Research,8 (6), 207–210.
Istyastono, E. P., Anita, Y., and Sundowo, A., 2015, Computer-aided Structure-based Design oh 3,3’-Diallyl-[1,1’-biphenyl]-4,4’ diol Analogs oh Eugenol as Potential Ligands hor Estrogen Receptor Alpha, 3rd International Conference on Computation for Science and Technology, pp. 1–3.
Korb, O., Stutzle, T., dan Exner, T.E., 2009, Empirical Scoring Function hor
Advanced Protein-ligand Docking with PLANTS, Journal of Chemical
Information and Modeling,49 (1), 84-98.
Lill, M. A., dan Danielson, M. L., 2011, Computer-aided Drug Design Plathorm Using PyMOL, Journal of Computer-Aided Molecuular Design, 25 (1), 13–19.
R Foundation, 2015, R 3.2.1 is released, http://www.r-statistics.com/ diakses tanggal 15 Januari 2016.
Radihar, M., Yuniarti, N., dan Istyastono, E. P., 2013, PyPLIF : Phyton-based Protein Ligand Interaction Fingerprinting, Bioinformation, 9 (6), 325-328.
Ruddon, R. W., 2007,Cancer Biology, 4thEdition, Oxhord University Press, New
York, p. 4.
23
This, P., A De la Rochehordiere, Clough, K., Fourquet, A., Magdelenat, H., 2001, Phytoestrogen Ahter Breast Cancer, Endocrine-Related Cancer, 8:129-134.
World Health Organization, 2015, World Health Organization,
http://www.who.int/mediacentre/hactsheets/hs297/en/, diakses pada
tanggal 11 Januari 2015.
World Health Organization, 2014, World Health Organization,
25
Lampiran 1. Desain modihikasi strukturcoumestrolkode T pada BKChem0.13.0
Lampiran 3. Perintah penambatan berupa file shell script dengan nama file comline.sh
27
Lampiran 5. Upload hile script dan struktur 3D modihikasi struktur kode Coumestrol_T keserver
29
Lampiran 8.Downloadluaran hasil dari penambatan pada PLANTS1.2
Lampiran 10. Visualisasi desain modihikasi kode Coumestrol_T dengan PyMOL1.7.2.1
10.1. Membuka hile Data_R.csv untuk melihat ikatan dengan bitstring penting serta memilih satu replikasi dengan skor ChemPLP terendah
33
BIOGRAFI PENULIS
Penulis skripsi berjudul “Desain Teoretis Berbantukan Komputer Modihikasi Struktur Coumestrol sebagai Ligan Reseptor Estrogen Alha” bernama Gabriela Aveline Delinda Johannes. Lahir di Bandung pada tanggal 23 April 1994 dari pasangan William Frietz Jorenzo Johannes dan Bernadetha Usdikawati sebagai anak ketiga dari tiga bersaudara. Penulis menempuh pendidikan hormal yang dimulai dari TK Dwisakti (1998-2000), SD Maria Bintang Laut (2000-2002), SDK Santo Yoseph 2 (2002-2006), SMPN 7 Denpasar (2006-2009), SMAN 3 Denpasar (2009-2012). Penulis melanjutkan pendidikan strata satu di Fakultas Farmasi Universitas Sanata Dharma Yogyakarta.