Fakultas Ilmu Komputer
3810
Clustering
Pasien Kanker Berdasarkan Struktur Protein Dalam Tubuh
Menggunakan Metode
K-Medoids
Laily Putri Rizby1, Marji2, Lailil Muflikhah3
Program Studi Teknik Informatika, Fakultas Ilmu Komputer, Universitas Brawijaya Email: 1[email protected], 2[email protected], 3[email protected]
Abstrak
Kanker merupakan penyakit yang kerap menjadi momok bagi sebagian besar orang memang telah memakan banyak korban. Semakin berkembangnya zaman semakin banyak virus yang tersebar di masyarakat. Kanker adalah istilah yang digunakan untuk menggambarkan ratusan penyakit berbeda dengan fitur tertentu yang sama. Kanker dimulai dengan perubahan dalam struktur dan fungsi sel yang menyebabkan sel membelah dan menggandakan diri tanpa terkontrol. Umumnya kanker dinamai sesuai organ dan jenisnya tempat pertama kali ia berkembang. Mutasi gen yang paling sering ditemukan pada kanker manusia adalah Gen P53. Gen P53 merupakan gen penekan tumor yang mengkode atau mengekspresikan protein 53. Dari berbagai banyak data yang ada perlu dilakukan proses klusterisasi yaitu pengelompokkan jenis kanker berdasarkan kelasnya. Salah satu metode klustering yang mulai banyak digunakan adalah metode K-Medoids. K-medoids atau dikenal pula dengan PAM (Partitioning Around Medoids) menggunakan metode partisi clustering untuk mengelompokkan sekumpulan n objek menjadi sejumlah k cluster. Algoritma ini menggunakan objek pada kumpulan objek untuk mewakili sebuah cluster. Objek yang terpilih untuk mewakili sebuah cluster disebut medoid. Pada penelitian clustering pasien kanker menggunakan metode K-Medoids ini menunjukkan nilai persentase kualitas cluster sebesar 77% pada percobaan pada nilai k 14 dan menggunakan 116 data.
Kata kunci: clustering, k-medoids, kanker, protein tubuh, silhouette coefficient
Abstract
Cancer is a disease that often becomes a scourge for most people has indeed taken many victims. In this era, more viruses are scattered in the community. Cancer is a term used to describe hundreds of different diseases with the same particular features. Cancer begins with changes in the structure and function of cells that cause cells to divide and multiply uncontrollably. Cancer is generally named after the organ and its type where it first developed. The most common mutation of genes found in human cancers is the P53 Genes. The P53 gene is a tumor suppressor gene that encodes or expresses protein 53. From a wide range of data there is a clustering process that classifies types of cancer by its class. One of the most widely used methods of clustering is the K-Medoids method. K-medoids or also known as PAM (Partitioning Around Medoids) using the clustering partition method to group a set of n objects into a number of cluster k. This algorithm uses objects on a collection of objects to represent a cluster. The object chosen to represent a cluster is called the medoid. In clustering research, cancer patients using K-Medoids method showed cluster quality percentage of 77% in experiments at k 14 and using 116 data.
Keywords: clustering, k-medoids, cancer, body protein, silhouette coefficient
1. PENDAHULUAN
Kanker adalah istilah yang digunakan untuk menggambarkan ratusan penyakit berbeda dengan fitur tertentu yang sama. Kanker dimulai dengan perubahan dalam struktur dan fungsi sel yang menyebabkan sel membelah dan menggandakan diri tanpa terkontrol. Sel
kemudian dapat menyerang dan merusak jaringan sekitar, dan sel dapat memisahkan diri dan menyebar ke area lain dalam tubuh. Umumnya kanker dinamai sesuai organ dan jenisnya tempat pertama kali ia berkembang (Kelvin, Joanne Frankel., dkk., 2011).
bentuk pengobatan dengan target gen masih terus dikembangkan, temuan ini membawa harapan baru untuk melawan penyakit mematikan tersebut. Peta genetik yang dibuat para ilmuwan Amerika Serikat menunjukkan setidaknya terdapat sekitar 200 gen termutasi yang berperan mengatur pembentukan, pertumbuhan, dan penyebaran tumor. Sebagian besar gen yang termutasi ini belum diketahui sebelumnya (Diananda, Rama, 2011).
Mutasi gen yang paling sering ditemukan pada kanker manusia adalah Gen P53. Gen P53 merupakan gen penekan tumor yang mengkode atau mengekspresikan protein 53. Protein p53 merupakan faktor transkripsi terhadap gen-gen yang terlibat dalam regulasi siklus sel, induksi apoptosis, repair DNA, dan stabilitas genome.
Dengan semakin berkembang pesatnya kecanggihan teknologi juga diharapkan adanya sebuah sistem atau aplikasi yang mampu mengidentifikasi penyakit kanker. Pengidentifikasian dilakukan dengan cara mengclusterkan data pasien ke dalam kelas “non-cancer”, “breast cancer”, “colorectal cancer” dan kelas “lung cancer”. Pengklusteran merupakan pengelompokan record, pengamatan, atau memperhatikan dan membentuk kelas pada objek-objek yang memiliki kemiripan.
Dalam penelitian ini metode clustering K-Medoids dipilih sebagai metode yang mampu melakukan klustering data protein dengan baik. K-Medoids adalah sebuah algoritma yang menggunakan metode partisi clustering untuk mengelompokkan sejumlah n objek menjadi k cluster.
Metode ini telah digunakan pada penelitian sebelumnya untuk klasterisasi penyakit kanker berdasarkan kandungan logam dalam darah. Dan tingkat akurasinya mencapai 57.14% (Nastiti, Shofi., dkk., 2014). Sedangkan dengan data kanker yang sama yaitu berdasarkan struktur protein dalam tubuh tetapi menggunakan algoritma Modified K-Nearest Neighbor (MKNN) tingkat akurasi maksimum yang dicapai adalah 43.53% (Retwitasari, Arintha, 2016).
Dari pendahuluan yang telah dipaparkan dan berdasarkan pada penelitian sebelumnya maka dibuatlah penelitian tentang clustering pasien kanker berdasarkan struktur protein dalam tubuh menggunakan metode K-Medoids ini.
2. PROTEIN TUBUH
Molekul protein tersusun dari satuan-satuan dasar kimia yaitu asam amino. Satu molekul protein dapat terdiri dari 12 sampai 20 macam asam amino dan dapat mencapai jumlah ratusan asam amino (Wibisono, Yudi, 2011).
Semenjak tahun 1960 semakin nyata bahwa ada paling sedikit tiga residu nukleotida DNA diperlukan untuk mengkode untuk masing-masing asam amino. Empat huruf kode DNA yaitu A, T, G dan C tersusun membentuk tiga huruf yang disebut dengan kodon.
Pada prosesnya di dalam sel, terjadi proses transkripsi yaitu sintesis RNA dengan DNA sebagai cetakannya. RNA yang membawa sandi yang sama dengan resep pada DNA ini bertindak sebagai cetakan untuk sintesis protein. Setiap kodon mengkodekan 1 asam amino. Sementara itu jumlah asam amino penyusun protein diketahui hanya 20 saja.
3. GEN P53
Kode genetik yang mengatur pembentukan sel-sel kanker berhasil dipecahkan. Peta genetik yang dibuat para ilmuwan Amerika Serikat menunjukkan setidaknya terdapat sekitar 200 gen termutasi yang berperan mengatur pembentukan, pertumbuhan, dan penyebaran tumor. Ditemukan sejenis protein yang dikenal dengan nama “P53” berhubungan kuat dengan kanker.
Proses mutasi dalam gen P53 yang mampu mengontrol produksi protein, dipercaya sebagai penyebab dari 50% kasus keganasan. Gen P53 merupakan gen penekan tumor yang mengkode atau mengekspresikan protein 53, nama ini diambil dari berat molekulnya yang sebesar 53 kilodalton. Protein 53 merupakan faktor tranksripsi terhadap gen-gen yang terlibat dalam regulasi siklus sel, induksi apoptosis, repair DNA, dan stabilitas genome.
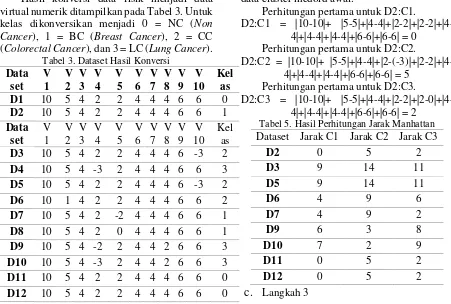
4. POINT ACCEPTED MUTATION
Gambar 1. Tabel Matrik PAM250
Ria, 2013). Pada tabel PAM titik yang termutasi pada protein adalah perubahan pada salah satu asam amino, yang terpilih secara alami.
Jumlah dari matriks PAM (PAM1, PAM250) menunjukkan sebuah evolusi jarak. Semakin besar jumlahnya maka semakin besar pula jaraknya (Dor, Shifra Ben, 2007). Untuk memperoleh nilai pada PAM maka dilakukan perkalian, contoh PAM2 diperoleh dari perkalian antara PAM1 dan PAM1, begitu pula dengan PAM3 diperoleh dari perkalian antara PAM1dan PAM2, begitu seterusnnya. Matrik PAM1 merupakan dasar untuk menghitung matrik yang lain dengan anggapan mutasi yang berulang akan mengikuti aturan yang sama dengan matrik PAM1, dengan logika tersebut dapat diperoleh matrik PAM250.
Tabel di atas diketahui terdapat 20 macam asam amino. ). Macam-macam residu asam amino yang diurutkan dari atas ke bawah yaitu Sistein Alanin (Ala) = A, Arginin (Arg) = R,
Algoritma K-medoids atau dikenal pula dengan PAM (Partitioning Around Medoids) menggunakan metode partisi clustering untuk mengelompokkan sekumpulan n objek menjadi sejumlah k cluster. Algoritma ini menggunakan objek pada kumpulan objek untuk mewakili sebuah cluster. Objek yang terpilih untuk mewakili sebuah cluster disebut medoid. Cluster dibangun dengan menghitung kedekatan yang dimiliki antara medoid dengan objek non-medoid (Han, J., M. Kamber, 2006).
Menurut Han dan Kamber (2006)
algoritma K-medoids adalah sebagai berikut. 1. Secara acak pilih k objek pada sekumpulan
n objek sebagai medoid. 2. Ulangi:
3. Tempatkan objek non-medoid ke dalam cluster yang paling dekat dengan medoid. 4. Secara acak pilih oacak: sebuah objek
non-medoid.
5. Hitung total biaya, S, dari pertukaran medoid oj dengan orandom.
6. Jika S < 0 maka tukar oj dengan oacak untuk membentuk sekumpulan k objek barusebagai medoid.
6. PERANCANGAN DAN IMPLEMENTASI
Dataset protein yang tersedia adalah data bertipe String sehingga langkah pertama yang harus dilakukan dalam sistem di penelitian ini adalah melakukan konversi data menjadi data numerik sehingga data menjadi bertipe integer dan dapat dilakukan proses clustering. Langkah selanjutnya adalah melakukan perhitungan clustering menggunakan metode K-Medoids. Dan langkah terakhir adalah melakukan uji kualitas kluster menggunakan silhouette coefficient.
Pengkonversian data dengan cara mencocokkan dataset dengan data wild yang telah ada melalui tabel PAM250.
Tabel 1. Data Wild bentuk Fisik
Tabel 2. Dataset bentuk Fisik
Hasil konversi data fisik menjadi data virtual numerik ditampilkan pada Tabel 3. Untuk kelas dikonversikan menjadi 0 = NC (Non Cancer), 1 = BC (Breast Cancer), 2 = CC (Colorectal Cancer), dan 3 = LC (Lung Cancer).
Tabel 3. Dataset Hasil Konversi
Data
6.1. Perhitungan K-Medoids
Pada perhitungan manual nilai k yang digunakan adalah 3. Sehingga data yang diperoses adalah mengelompokkan dataset sebanyak 3 cluster.
a.
Langkah 1Menentukan secara acak medoid awal yang berbeda sebanyak 3 medoid dari dataset protein yang telah dikonversikan. Dalam perhitungan ini medoid awal yang digunakan adalah D1, D4, dan D8.
Tabel 4. Dataset Medoid Awal
Clus
Menghitung jarak setiap data dengan medoid awal C1, C2 dan C3 menggunakan rumus jarak manhattan (Manhattan Distance). Rumus jarak manhattan :
𝑑(𝑖, 𝑗) = ∑ |𝑥(𝑖) − 𝑥(𝑗)| + |𝑦(𝑖) − 𝑦(𝑗)|𝑛
𝑖=1 (1)
Sebagai contoh ditampilkan perhitungan secara detail pada data D2 yang diambil dari dataset protein. Data D2 akan dilakukan
perhitungan dengan data C1, C2 dan C3 pada data cluster medoid awal.
Perhitungan pertama untuk D2:C1.
D2:C1 = |10-10|+ |5-5|+|4-4|+|2-2|+|2-2|+|4-4|+|4-4|+|4-4|+|6-6|+|6-6| = 0
Perhitungan pertama untuk D2:C2.
D2:C2 = |10-10|+ |5-5|+|4-4|+|2-(-3)|+|2-2|+|4-4|+|4-4|+|4-4|+|6-6|+|6-6| = 5
Perhitungan pertama untuk D2:C3.
D2:C3 = |10-10|+ |5-5|+|4-4|+|2-2|+|2-0|+|4-4|+|4-4|+|4-4|+|6-6|+|6-6| = 2
Tabel 5. Hasil Perhitungan Jarak Manhattan
Dataset Jarak C1 Jarak C2 Jarak C3
Langkah selanjutnya adalah menghitung nilai cost dari tiga cluster pada setiap data yang telah dihitung jaraknya dengan medoid awal, dengan rumus :
𝑐𝑜𝑠𝑡(𝑖) = min {𝑑𝐶1(𝑖), 𝑑𝐶2(𝑖), … , 𝑑𝐶𝑛(𝑖) (2) Contoh perhitungan pada D2 adalah sebagai berikut.
Cost (D2) = min {0,5,2} = 0
Hasil perhitungan untuk semua dataset protein dapat dilihat pada tabel 6.
Tabel 6. Nilai Cost Cluster pada Medoid Awal
d.
Langkah 4Pada langkah ini iterasi pertama mulai dilakukan. Perhitungan dilakukan dengan mengganti salah satu medoid. Medoid baru yang digunakan adalah salah satu data non medoid yang berada pada cluster yang sama pada medoid awal. Dalam perhitungan ini medoid yang diganti adalah cluster 2, yaitu D4 diganti dengan D9.
Tabel 7. Medoid Iterasi 1
Clu
Langkah selanjutnya adalah mengulangi langkah 2-4 sampai semua data pernah menjadi medoid.
Tabel 8. Hasil Jarak Manhattan dan Nilai Cost Iterasi 1
Hasil perhitungan jarak manhattan dan nilai cost pada setiap data di iterasi 1 dapat dilihat pada Tabel 8. Maka didapatkan anggota Cluster 1 : D1, D2, D3, D5, D6, D11 dan D12, anggota Cluster 2 : D4, D9 dan D10, anggota Cluster 3 : D7 dan D8. Lalu total cost yang didapatkan adalah 28. Karena total cost lebih kecil dari total cost medoid sebelumnya maka medoid dan anggota cluster berubah.
Perhitungan diulang terus sampai semua data pernah menjadi medoid. Setelah itu dilanjutkan dengan menghitung kualitas cluster.
6.2. Perhitungan Silhouette Coefficient
Hasil dari silhouette coefficient ini berada antara nilai -1 sampai 1. Jika s(i) = 1 maka data telah berada pada cluster yang tepat, jika s(i) = 0
maka data berada pada posisi di tengah, maksudnya data terdapat kemungkinan berada pada cluster yang tepat tetapi bisa juga seharusnya berada pada cluster yang lain, jika s(i) = -1 maka data berada pada cluster yang salah sehingga seharusnya data berada pada cluster yang lain.
a. Langkah 1
Menghitung jarak D1 terhadap semua data yang berada pada cluster yang sama, yaitu cluster 3.
Tabel 9. Jarak D1 dengan Anggota Cluster 3
Dataset Dataset Jarak D1 dengan semua anggota C3
D1
Menghitung jarak D1 terhadap semua data yang berada pada cluster 1 dan cluster 2, kemudian cari nilai rata-rata jarak yang paling kecil.
Tabel 10. Jarak D1 dengan Anggota Cluster 1 dan Cluster 2 silhouette coefficient bisa dilakukan menggunakan dengan rumus :
𝑆(𝑖) = 𝑏(𝑖) − 𝑎(𝑖)
Gambar 2. Implementasi Antarmuka Sistem Clustering Pasien Kanker
Diketahui 𝑎(1) = 1,7 dan 𝑏(1) = 6, sehingga 𝑎(1) < 𝑏(1) nilai silhouette coefficient-nya adalah :
𝑆 = 1 − 𝑎(1)𝑏(1)
= 1 − 1,76 = 1 − 0,28 = 0,72
Perhitungan Silhouette Coefficient dilakukan pada semua data dan dihitung rata-rata akhir untuk mengetahui nilai SC metode K-Medoids.
6.3. Implementasi Antarmuka
Antarmuka dibuat untuk memudahkan pengguna dalam menggunakan sistem yang dapat dilihat pada gambar 2.
Yang pertama adalah bagian input data yang digunakan oleh pengguna untuk memasukkan data-data yang diperlukan dalam proses clustering.
Bagian kedua adalah tabel dataset protein, bagian ketiga adalah menunjukkan jumlah dataset dan cluster serta menampilkan nilai akhir dari Silhouette Coefficient, dan bagian keempat adalah sebuah textarea yang berisi hasil clustering yang dilakukan sistem.
7. HASIL DAN ANALISIS
7.1. Pengujian Pengaruh Jumlah Cluster
Prosedur pengujian pengaruh jumlah
cluster ini adalah dengan memasukkan nilai
k
yang berarti adalah jumlah cluster dengan
nilai yang berbeda-beda dan akan dilakukan
percobaan beberapa kali. Nilai
k
atau jumlah
cluster yang digunakan adalah 2 sampai 20.
Dan nilai tertinggi berada pada jumlah
Gambar 3. Grafik Hasil Pengujian Pengaruh Jumlah Cluster terhadap Silhouette Coefficient
Gambar 4. Grafik Hasil Pengujian Pengaruh Jumlah Dataset terhadap Silhouette Coefficient
cluster 14 dan 17 yaitu 0,726.
7.1. Pengujian Pengaruh Jumlah Dataset
Jumlah data yang digunakan bervariasi
yaitu dari 100% data atau 588 dataset,
kemudian diambil secara acak menjadi 20%,
30%, 40%, 50%, dan 60% data dari data
total. Grafik hasil percobaan dapat dilihat
pada gambar 4.
Dari percobaan yang telah dilakukan
terlihat hasil yang bervariasi dan hasil
terbaik didapatkan ketika data yang
digunakan 20% yaitu 116 dataset protein,
rata-rata
nilai
silhouette
coefficient
mencapai 0,778.
KESIMPULAN
Berdasarkan hasil perancangan, implementasi, dan hasil pengujian sistem yang telah dilakukan dapat didapatkan kesimpulan :
1.
Metode K-Medoids dapatdiimplementasikan pada sistem clustering pasien kanker berdasarkan struktur protein dalam tubuh. Data total yang digunakan sebanyak 588 dataset. Proses awal yang dilakukan adalah mengkonversi data menggunakan matriks PAM, kemudian dipilih pusat medoid secara acak sebanyak nilai k diinginkan, selanjutnya menghitung
0,52 0,612
0,66 0,716 0,726 0,726 0,686
0 0,2 0,4 0,6 0,8
2 5 8 11 14 17 20
S
il
ho
uette
C
o
ef
fi
ci
ent
Jumlah Cluster
Hasil Pengujian Pengaruh Jumlah Cluster Terhadap Silhouette Coefficient
0,778
0,724 0,732 0,736 0,74 0,698
0,73
0,65 0,7 0,75 0,8
20% 30% 40% 50% 60% 100% Total
S
il
ho
uette
C
o
ef
fi
ci
ent
Jumlah Dataset
jarak masing-masing data terhadap pusat medoid menggunakan perhitungan jarak manhattan, lalu dicari nilai cost pada setiap data, pilih cost minimal, dan total semua cost minimal yang didapatkan, proses akan berulang seperti ini sampai semua data pernah menjadi medoid. Jika nilai total cost lebih kecil maka medoid diganti, tetapi jika nilai total cost lebih besar maka medoid tetap. Kemudian sistem akan menampilkan hasil clustering sesuai dengan jumlah cluster yang diinputkan. Dan menampilkan hasil silhouette coefficient yang merupakan kualitas cluster yang dihasilkan.
2.
Dari dua pengujian yang telah dilakukan didapatkan hasil terbaik yaitu dengan jumlah cluster = 14 yang memiliki nilai silhouette coefficient 0,726 dan jumlah dataset 116 data atau data 20% yang memiliki nilai silhouette coefficient 0,778.DAFTAR PUSTAKA
Candra, Sefia, Antonius R. C., Lucia Dwi K., 2012. Clustering Tag Status Facebook Dengan Menggunakan Algoritma K-Medoids. Fakultas Teknologi Informasi. Universitas Kristen Duta Wacana. Yogyakarta.
Diananda, Rama, 2011. Mengenal Seluk-Beluk Kanker. Ar-Ruzz Media Group. Jogjakarta.
Dizon, Don S., Michael L. Krychman, Paul A. DiSilvestro, 2011. 100 Questions and Answers about Cervical Cancer. Jones and Bartlett Publishers, LLC. Sudbury, MA.
Dor, Shifra Ben, 2007, Scoring Matrices, Weizmann Institute Of Science, Rehovot.
Han, J., M. Kamber, 2006. Data Mining Concepts and Techniques Second Edition. Morgan Kauffman Publisher. San Fransisco.
Kelvin, Joanne Frankel., Leslie B. Tyson, 2011. 100 Questions & Answers about Cancer Symptoms and Cancer Treatment Side Effects, second Edition. Jones and Bartlett Publishers, LLC. Sudbury, MA. Kurnianti, Ria, 2013, Penggunaan Metode Pengelompokkan K-Means Pada Klasifikasi KNN Untuk Penentuan Jenis Kanker Berdasarkan Susunan Protein, Universitas Brawijaya, Malang.
Nastiti, Shofi., Faisal P., Rizky Ramadhan, 2014. Clustering Pasien Kanker Berdasarkan Konsentrasi Logam Dalam Darah Menggunakan Metode K. Program Teknologi Informasi dan Ilmu Komputer, Universitas Brawijaya. Malang.
Retwitasari, Arintha, 2016. Penentuan Jenis Kanker Berdasarkan Struktur Protein Menggunakan Algoritma Modified K-Nearest Neighbor (MKNN). Fakultas Ilmu Komputer, Universitas Brawijaya Malang.
Shibab, A., 2000. Fuzzy Clustering Algorithm and Their Application to Medical Image Analysis. Disertation, University of London. London.
Uyha, 2010, http://uyha06.wordpress.com/ilmu- keperawatan/laporan-praktikum-biokimia/protein/.