commit to user
i
DAN NON PONDOH MELALUI ANALISIS RAPD-PCR
Skripsi
Untuk memenuhi sebagian persyaratan guna memperoleh derajat Sarjana Pertanian
di Fakultas Pertanian Universitas Sebelas Maret
Jurusan/Program Studi Agronomi
Oleh :
TRIPURNAMI CANDRADEWI H0106109
FAKULTAS PERTANIAN UNIVERSITAS SEBELAS MARET
commit to user
ii
DETEKSI KERAGAMAN SALAK (Salacca zalacca ) VARIETAS PONDOH DAN NON PONDOH MELALUI ANALISIS RAPD-PCR
yang dipersiapkan dan disusun oleh TRIPURNAMI CANDRADEWI
H 0106109
telah dipertahankan di depan Dewan Penguji Pada tanggal, Oktober 2012
Dan dinyatakan telah memenuhi syarat
Susunan Tim Penguji
Ketua Anggota I Anggota II
Prof. Dr. Ir. Nandariyah, MS NIP. 19540805 198103 2 002
Prof. Dr. Ir. Djoko Purnomo, MP NIP. 19480426 197609 1 001
Dr. Ir. Pardono, MS NIP. 19550806 198303 1 003
Surakarta, Oktober 2012 Universitas Sebelas Maret Surakarta
FakultasPertanian Dekan
commit to user
iii
Berilmu, Ilmu adalah hiasan, Keutamaan dan bertanda segala pujian
Dalam setiap hari berusahalah selalu bertambah dalam penguasaan ilmu
Dan menyelamlah ke dalam lautan ilmu
(HR. Muhammad Ibnul Hasan bin Abdullah)
Karya kecil ini Kupersembahkan untuk : Ayah dan Ibu Serta Adikku Tercinta dan seluruh Keluarga
commit to user
iv
Penulis dilahirkan di Klaten pada tanggal 31 Maret 1988 sebagai anak Pertama dari Tiga bersaudara dari Bapak Prawoto, Spd dan Ibu Sri Indriyastuti, BA. Penulis adalah seorang muslim dan lahir dari orang tua dengan latar pendidikan Pegawai Negeri. Penulis tinggal bersama kedua orang tua di Jabungan (21/09), Gondang, Kebonarum, Klaten dari tahun 1993 sampai sekarang.
Pendidikan Dasar sampai perguruan tinggi, diselesaikan penulis di kota Klaten dan Surakarta, Jawa Tengah. Tahun 1993 penulis pernah sekolah di TK Pertiwi 1 Boyolali pada tingkat A dan melanjutkan ke tingkat B di TK Pertiwi 1 Sumberejo, Klaten selatan. Pada tahun 1993-1994 penulis telah menyelesaikan pendidikan TK. Tahun 1995-1996 penulis pernah mengikuti Taman Pendidikan Al‟ Quran “ AL- FIRDAUS ”. Tahun 2000 penulis lulus dari SD Negeri 1 Sumberejo, Klaten selatan. Kemudian tahun 2003 penulis menyelesaikan pendidikan Lanjutan Pertama di SMP Negeri 3 Klaten dan Pendidikan Lanjutan Atas tahun 2006 di SMA Negeri 2 Klaten. Pada tahun yang sama, penulis diterima di Jurusan Agronomi Fakultas Pertanian Universitas Sebelas Maret Surakarta pada Agustus 2006. Selama mengeyam pendidikan di Fakultas Pertanian Penulis aktif dalam kegiatan rohani Mahasiswa sebagai pengurus FUSI tahun 2007/2008 dan penulis menjadi anggota tetap Himpunan Mahasiwa Agronomi (HIMAGRON). Penulis juga aktif dalam mengikuti kegiatan kepanitian OSMARU tahun 2007. Pada tahun 2008/2009 penulis pernah mengikuti kegiatan MAGANG mahasiswa di CV. NURSERY, Prambanan.
commit to user
v
Puji syukur kehadirat Allah SWT atas berkat rahmat, hidayah dan ridho-Nya penulis dapat menyelesaikan skripsi dengan judul “ Deteksi Keragaman Salak (Salacca Zalacca ) Varietas Pondoh dan Non Pondoh Melalui Analisis RAPD-PCR”. Skripsi ini disusun dan diajukan sebagai salah satu syarat untuk memperoleh gelar Sarjana Pertanian di Fakultas Pertanian Universitas Sebelas Maret Surakarta. Skripsi ini adalah sebagian dari penelitian Prof. Dr. Ir. Nandariyah, MS.
Penulis menyadari bahwa dalam penyelesaian skripsi ini tidak lepas dari dukungan berbagai pihak, oleh karena itu penulis menyampaikan terima kasih kepada:
1. Prof. Dr. Ir. Bambang Pujiasmanto, MS selaku Dekan Fakultas Pertanian Universitas Sebelas Maret Surakarta.
2. Prof. Dr. Ir. Nandariyah, MS selaku Dosen Pembimbing Utama Skripsi yang telah memfasilitasi penelitian ini, atas segala bimbingan, bantuan, evaluasi, saran dan ilmu yang telah diberikan serta pengarahan demi lebih baiknya skripsi ini.
3. Prof. Dr. Ir. Djoko Purnomo, MP selaku Dosen Pembimbing Pendamping Skripsi, yang telah memberikan bimbingan, masukan maupun pengarahan. 4. Dr. Ir. Pardono, MS selaku Dosen Pembahas dan Dosen Pembimbing
Akademik yang telah memberikan bimbingan, saran, masukan dan pengarahan.
5. Bapak Ibu dosen serta karyawan-karyawan Fakultas Pertanian Universitas Sebelas Maret Surakarta.
6. Pak Ahmad himawan pemilik CV Agrobiotech yang telah memfasilitasi segala kegiatan yang berkaitan dengan penelitian.
commit to user
vi
teman seperjuangan (Nanik dan Latifah), dan mas Agus yang selalu memberikan bantuan, masukan, semangat, doa dan dukungan selama penelitian.
9. Rekan-rekan angkatan 2006 Fakultas Pertanian UNS (IMAGO „06) dan segenap pihak yang telah membantu demi kelancaran penelitian ini.
Penulis menyadari bahwa penulisan skripsi ini masih jauh dari sempurna. Penulis mengharapkan kritik dan saran yang membangun. Demikian, semoga skripsi ini bermanfaat bagi penulis khususnya dan pembaca pada umumnya.
Surakarta, Oktober 2012
commit to user
vii
Halaman
HALAMAN JUDUL ... i
HALAMAN PENGESAHAN ... ii
MOTTO DAN PERSEMBAHAN ... . iii
DAFTAR RIWAYAT HIDUP ... iv
3. Sistem klasifikasi tanaman salak ... 7
4. Keragaman jenis salak . ... 8
B. Analisis RAPD-PCR ... 12
1. PCR (Polymerase Chain Reaction) ...13
2. RAPD (Random Amplified Polymorphic DNA) ... . 17
3. Aplikasi analisis RAPD ... . 18
4. Isolasi DNA ... . 19
5. Elektroforesis ... 20
commit to user
viii
A. Waktu dan Tempat Penelitian ... . 23
B. Bahan dan Alat Penelitian ... . 23
C. Cara kerja Penelitian... . 24
1. Pengambilan sampel daun ... . 24
2. Isolasi DNA ... . 24
3. Uji kualitas DNA ... . 26
4. Seleksi primer ... . 26
5. RAPD-PCR/Amplifikasi DNA ... 27
6. Elektroforesis ... 27
7. Visualisasi hasil RAPD ... 28
D. Analisis Data ... 28
1. Analisis similaritas ... 29
2. Analisis gerombol ... 30
IV.HASIL DAN PEMBAHASAN... . 31
A. Sistem klasifikasi kultivar tanaman salak pondoh dan non pondoh... .. 31
1. Hasil isolasi dan uji kualitas DNA ... .. 31
2. Hasil seleksi primer ... .. 36
3. Hasil amplifikasi DNA dengan penanda RAPD ... .. 37
V. KESIMPULAN DAN SARAN ... 49
A. Kesimpulan ... 49
B. Saran ... 50
DAFTAR PUSTAKA ... 51
commit to user
ix
Nomor Judul Halaman
commit to user
x
Nomor Judul Halaman
1 Prinsip amplifikasi fragmen DNA dengan mesin PCR ... ... 16 2 Profil hasil uji kualitas tujuh sampel daun salak hasil metode CTAB
(Pondoh Super, Pondoh Lawu, Pondoh Hijau, Gading, Kembang arum, Manggala, dan Lumut) dengan penanda marker DNA 1 kb ... 33 3 Profil hasil uji kualitas isolat DNA empat sampel daun salak dengan
metode CTAB (salak Kecandran, Banjar, pondoh Madu, dan pondoh Hitam dengan marker 1 kb ... 34 4 Hasil uji kualitas DNA salak (Saratan, Bejalen, Nglumut, Pondoh
lawu, pondoh Madu, dan Kecandran) dengan metode Plant Kit menggunakan penanda marker 1 kb (250-10000 pb)... 35 5 Hasil uji kualitas tujuh sampel DNA salak (pondoh Hitam, pondoh
Lawu, Nglumut, Bejalen, pondoh Super, Kecandran, dan Gading) dengan metode Plant Kit ... 36 6 Hasil uji seleksi primer OPA-11, OPA-17, OPA-16, 17,
OPX-15, dan OPA-18 ... 37 7 Hasil amplifikasi PCR varietas salak (Kembang arum, Manggala, dan
Kecandran) dengan primer OPA-11 menghasilkan tiga pita monomorfik ... 38 8 Hasil amplifikasi PCR isolat DNA salak (pondoh Madu, pondoh
Hitam, dan Kembang arum) dengan primer OPA-16 yang tidak berhasil amplifikasi ... 40 9 Hasil amplifikasi DNA dua puluh dua kultivar S. zalacca
menggunakan primer OPA-11... .... 41 10 Hasil amplifikasi dua puluh dua kultivar salak dengan pasangan basa
terendah 100 pb... 42 11 Hasil amplifikasi lima belas kultivar salak dari hasil seleksi dua puluh
commit to user
xi
pola pita dengan jumlah pita dua pita pada kultivar Kelapa bali dan Manggala... ... 44 13 Pola pita no. 3 dan no. 4 mempunyai kesamaan pola pita yang terletak
antara 100 pb - 400 pb pada kultivar Kediri dan pondoh Hitam... 44 14 Pola pita no. 5, no. 8, no. 10, dan no.12 pada kultivar pondoh Madu,
Thailand, Bejalen, dan pondoh Lawu yang terletak 300 pb - 600 pb. ... 45 15 Pola pita no. 9 dan no. 11 pada kultivar Banjar dan Kecandran terletak
300 pb - 600 pb mempunyai kesamaan pola pita... 46 16 Pola pita yang berkerabat jauh dijumpai pada kultivar Gula pasir,
commit to user
xii
Nomor Judul Halaman
Lampiran 1. Foto Sampel daun salak bahan ekstraksi... 54 Lampiran 2. Foto Hasil ektraksi DNA ... 55 Lampiran 3. Alat ektraksi DNA dengan metode CTAB ... 57 Lampiran 4. Alat dan bahan ekstraksi dan elektroforesis DNA dengan
metode Plant Kit ... 59 Lampiran 5. Identifikasi terjemahan pola dan jumlah pita lima belas
commit to user
xiii
DAN NON PONDOH MELALUI ANALISIS RAPD-PCR
Tripurnami Candradewi H 0106109
RINGKASAN
Salak (Salacca zalacca) merupakan tanaman asli Indonesia yang mempunyai keragaman jenis dan berpotensi tinggi untuk dikembangkan sebagai salah satu produk ekspor. Informasi tentang keragaman genetik salak masih sangat kurang, sehingga perlu adanya pengujian dan penelitian dengan metode RAPD - PCR. Penanda RAPD merupakan salah satu penanda molekuler yang dapat digunakan untuk mempelajari keragaman genetik tanaman tahunan. Penelitian ini bertujuan untuk mengetahui keragaman tanaman salak pondoh dan salak non pondoh. Tujuan lain yaitu mengetahui apakah terdapat hubungan kekerabatan kultivar tanaman salak pondoh dan salak non pondoh berdasarkan penanda RAPD.
Penelitian ini sebagai bagian dari penelitian Prof. Dr. Ir. Nandariyah, MS pada tahun 2012. Penelitian dilaksanakan pada bulan April sampai Mei 2012 bertempat di CV. Agribiotech Jl. Jambon No. 605 Gang Batan Jatimulyo, Kricak, Tegalrejo, Yogyakarta, dan Laboratorium Fisiologi dan Bioteknologi Fakultas Pertanian Universitas Sebelas Maret Surakarta. Penelitian dilaksanakan dengan dua metode isolasi DNA yaitu metode CTAB dan Dinamid Plant Kit, kemudian DNA hasil isolasi diuji secara kualitatif dengan elektroforesis gel agarose. Hasil isolat berkualitas baik diamplifikasi dengan penanda RAPD-PCR, hasilnya kemudian di running dengan elektroforesis gel agarose. Selanjutnya hasil elektroforesis diamati dibawah sinar ultraviolet agar dapat didokumentasikan melalui kamera makro. Dari dua puluh dua kultivar salak baik non pondoh dan pondoh hanya lima belas kultivar yang berhasil amplifikasi dengan primer OPA-11 dan susunan pola pita polimorfik antara 100 pb – 600 pb.
commit to user
xiv
VARIABILITY THROUGH RAPD-PCR ANALYSIS
Tripurnami Candradewi H 0106109
SUMMARY
Salak (Salacca zalacca) is Indonesia native fruit crop, which has several
kinds of variety and has high potential to be developed as one of the export
commodity. For developing the crop, genetic information is necessary (still
limited untill now) through RAPD - PCR testing and research methods. RAPD
marker is one of the molecular markers that can be used to study the genetic
diversity of annual plants. The research aims is to determine the genetic
variability of salak pondoh and non pondoh. The other aim of the research is to
determine the trait relationship of two salak kinds as mention above by RAPD
markers.
The study as apart of result of nandariyah research in 2012 years. The
study was conducted from April to May 2012 held at CV. Agribiotech Jl. No.
Jambon. 605 Gang Jatimulyo Batan, Kricak, Tegalrejo, Yogyakarta, and at Plant
Physiology and Biotechnology Laboratory, Faculty of Agriculture, University of
Sebelas Maret, Surakarta. The research was conducted by two kinds method of
DNA isolation there are CATB and Dinamid Plant Kid method. The quality of
isolation product then to be test by gel agarose electrophoresis. High quality
isolate then amplified by RAPD-PCR marker, the result then running by gel
agarose electrophoresis and observed under ultraviolet ray for documentating
through micro camera. Among twenty two salak cultivars (pondoh and non) just
fifteen cultivars succeeded amplifical by OPA-11 primer with polymorphic
banding pattern between 100 bp – 600 bp.
The result of research showed based on RAPD marker the relationship
commit to user
DETECTION OF SALAK (Salacca zalacca) PONDOH AND NON PONDOH VARIABILITY THROUGH RAPD-PCR ANALYSIS
Nandariyah 1) Tripurnami Candradewi 2)
H 0106109
ABSTRACT
Salak (Salacca zalacca) is Indonesia native fruit crop, which has several kinds of variety and has high potential to be developed as one of the export commodity. For developing the crop, genetic information is necessary (still limited untill now) through RAPD - PCR testing and research methods. RAPD Jambon. 605 Gang Jatimulyo Batan, Kricak, Tegalrejo, Yogyakarta, and at Plant Physiology and Biotechnology Laboratory, Faculty of Agriculture, University of Sebelas Maret, Surakarta. The research was conducted by two kinds method of DNA isolation there are CATB and Dinamid Plant Kid method. The quality of isolation product then to be test by gel agarose electrophoresis. High quality isolate then amplified by RAPD-PCR marker, the result then running by gel agarose electrophoresis and observed under ultraviolet ray for documentating through micro camera. Among twenty two salak cultivars (pondoh and non) just fifteen cultivars succeeded amplifical by OPA-11 primer with polymorphic banding pattern between 100 bp – 600 bp.
The result of research showed based on RAPD marker the relationship between salak pondoh and non pondoh cultivars.
Keywords: genetic diversity, Pondoh and non pondoh salak, RAPD-PCR marker
1) Study as part of a research project leading Prof. Dr. Ir. Nandariyah, MS
commit to user
1 BAB. I PENDAHULUAN
A. Latar Belakang
Salak (Salacca zalacca) merupakan tanaman asli Indonesia yang berpotensi tinggi untuk dikembangkan sebagai salah satu produk ekspor ke pasar internasional. Buah salak merupakan salah satu buah tropis yang banyak diminati oleh masyarakat Jepang, Amerika, dan Eropa. Akan tetapi untuk memenuhi standar kualitas produksi buah, kultivar salak di Indonesia seharusnya diperbaiki melalui program pemuliaan yang menyeluruh. Banyak aspek tanaman salak yang harus diteliti lagi, antara lain penelitian fisiologi dan bioteknologi untuk mendukung program pemuliaan tanaman.
Salak pondoh merupakan buah yang sangat populer di Yogyakarta, bahkan menjadi ciri khas daerah tersebut. Salak pondoh memiliki bermacam-macam varietas seperti salak pondoh super, salak pondoh hijau, salak pondoh hitam, dan salak pondoh madu. Salak pondoh merupakan salah satu jenis salak yang mengalami peningkatan produksi. Kelebihan salak pondoh antara lain rasa buah yang manis meskipun belum matang, memiliki kandungan air cukup, berbuah sepanjang tahun, masa simpan buah lebih dari 20 hari, bila dimakan dalam jumlah banyak tidak menimbulkan rasa tidak enak di perut, dan harga jual relatif tinggi (Purnomo, 2001). Selain itu di Indonesia khususnya di Jawa dan di Bali terdapat juga varietas lokal non pondoh seperti Salak Lawu (Matesih, Karanganyar), Salak Saratan (Magelang), Salak Nglumut (Magelang), Salak Kecandran (Salatiga), Salak Bejalen (Ambarawa), Salak Manggala (Sleman), Salak Gading (Sleman), Salak Kediri, Salak Banjarnegara, Salak Gula pasir, Salak Kelapa bali (Bali) dan Salak Kembang Arum (Sleman) yang kesemuanya merupakan sumber keragaman genetik tanaman salak.
commit to user
unggul dan produksi tinggi, evaluasi keragaman terhadap kultivar-kultivar sejauh ini masih terbatas pada sifat-sifat morfologis yang sangat dipengaruhi oleh keadaan lingkungan, dan belum ada eksplorasi dan penggalian informasi genetik yang menyeluruh dari seluruh potensi plasma nutfah tanaman salak.
Hingga saat ini informasi genetik salak masih sangat terbatas, hal ini menjadi salah satu kendala dalam usaha pemuliaan dan pengembangan salak unggul. Pemberian nama dan pengelompokan tanaman salak sampai saat ini umumnya masih didasarkan pada sifat yang masih tradisional. Pengetahuan tentang keragaman genetik sebenarnya merupakan modal dasar bagi para ahli pemuliaan dan genetika populasi dalam pengembangan dan perbaikan tanaman, terutama sebagai langkah awal seleksi tanaman (Thorman and Osborn, 1992). Langkah ini penting terutama untuk membedakan individu dalam spesies serta identifikasi genotip secara tepat dan identifikasi gen-gen yang berpotensi sebagai pembawa karakter unggul.
B. Perumusan Masalah
Salak merupakan tanaman asli Indonesia, buah banyak digemari masyarakat karena rasa manis, renyah dan kandungan gizi yang tinggi. Jawa Tengah dan Yogyakarta sebagai salah satu pusat keragaman kultivar salak mempunyai potensi yang cukup besar untuk menghasilkan varietas-varietas unggul yang lebih bernilai ekonomis dan kompetitif. Salak Pondoh merupakan komoditas unggulan dan perlu untuk ditingkatkan kualitas maupun kuantitas produksinya. Meskipun salak lokal non Pondoh kurang diminati konsumen bukan berarti keberadaannya tidak penting untuk pemuliaan tanaman.
commit to user
mendeteksi keragaman genetik pada tingkat DNA, salah satunya yaitu dengan teknik Random Amplified Polymorphic DNA (RAPD). Teknik RAPD mampu menghasilkan potongan DNA hasil pelipat gandaan dalam jumlah yang tak terbatas dan setiap potongan dapat diperlakukan sebagai karakter untuk keperluan analisis. Penanda RAPD merupakan salah satu penanda molekuler yang dapat digunakan untuk mempelajari keragaman genetik tanaman tahunan. Salah satu contohnya mendeteksi kesalahan pengelompokan varietas dan kultivar (Novey et.al, 1994). Berdasarkan uraian di atas, diteliti bermaksud menjawab pertanyaan sebagai berikut:
1. Bagaimanakah keragaman tanaman Salak varietas salak pondoh dan non pondoh melaui analisis RAPD-PCR?
2. Bagaimana hubungan kekerabatan tanaman Salak varietas salak pondoh dan non pondoh melalui analisis RAPD?
C. Tujuan Penelitian
Penelitian ini bertujuan untuk :
1. Mengetahui keragaman jenis tanaman Salak pondoh dan non pondoh pada kultivar masing-masing.
2. Mengetahui apakah terdapat hubungan kekerabatan kultivar tanaman salak pondoh dan non pondoh berdasarkan penanda RAPD.
D. Manfaat Penelitian
Penelitian ini mempunyai manfaat yaitu:
1. Memastikan/menentukan tingkat keragaman dan kekerabatan kultivar salak pondoh maupun non pondoh.
2. Bagi peneliti dapat mengenal lebih dalam keragaman kultivar salak melalui teknik penanda molekuler RAPD-PCR
commit to user
4 BAB. II
TINJAUAN PUSTAKA
A. Salak (Salacca zalacca) 1. Taksonomi
Kerajaan : Plantae
Divisi : Spermatophyta Kelas : Monocotyledoneae Ordo : Arecales
Famili : Arecaceae Genus : Salacca
Spesies : Salacca zalacca
2. Morfologi
Bentuk morfologi tanaman salak sangat dipengaruhi oleh faktor lingkungan seperti iklim, tanah, dan topografi yang saling terkait, sehingga mempengaruhi fungsi fisiologis dan morfologis. Dalam beradaptasi dengan lingkungan yang tidak sesuai, maka tanaman salak akan melakukan beberapa perubahan baik fisiologi maupun morfologi. Salak adalah sejenis palma dengan buah yang biasa dimakan. Dalam bahasa Inggris disebut snake fruit, karena kulit buah mirip dengan sisik ular. Salak termasuk palma berbentuk perdu atau hampir tidak berbatang, berduri banyak, melata dan beranak banyak, tumbuh menjadi rumpun yang rapat dan kuat.
commit to user
Batang menjalar di bawah atau di atas tanah, membentuk rimpang, sering bercabang, dan diameter 10-15 cm.
Daun salak berbentuk pinnate atau berupa sisir terdiri atas pelepah, tangkai, dan helaian anak daun yang tersusun menyirip, tangkai daun tertutup oleh duri tajam (Ashari, 1995). Daun majemuk menyirip, panjang 3-7 m, tangkai daun, pelepah dan anak daun berduri panjang, tipis dan banyak, warna duri kelabu sampai kehitaman. Anak daun berbentuk lanset dengan ujung meruncing, berukuran sampai 8 x 85 cm, sisi bawah keputihan oleh lapisan lilin. Bagian bawah dan tepi tangkai berduri tajam, ukuran dan warna daun tergantung varietas (Anonim, 1992).
Salak umumnya berumah dua (dioesis), karangan bunga terletak dalam tongkol majemuk yang muncul di ketiak daun, bertangkai, mula-mula tertutup oleh seludang yang belakangan mengering dan mengurai menjadi serupa serabut. Bunga kecil muncul di ketiak pelepah, mekar selama 1-3 hari. Ketika masih muda diselubungi seludang yang berbentuk perahu simetri radial. Mempunyai tiga daun kelopak dan tiga daun mahkota, kadang-kadang struktur kelopak dan mahkota tidak dapat dibedakan. Kuntum bunga dibedakan menjadi kuntum besar dan kecil. Keduanya bersatu dalam satu dasar bunga yang memiliki putik dengan satu bakal biji. Bunga jantan, terdiri atas stamen, banyak, rapat, panjang, tersusun seperti genteng, simetri radial.
commit to user
bunga jantan dan betina terdapat pula bunga hermaprodit (Steenis, 1975). Panjang tongkol bunga jantan 50-100 cm, terdiri atas 4-12 bulir silindris masing-masing antara 7-15 cm, dan banyak bunga kemerahan terletak di ketiak sisik-sisik yang tersusun rapat. Tongkol bunga betina 20-30 cm, bertangkai panjang, terdiri atas 1-3 bulir dengan panjang mencapai 10 cm.
Tanaman salak mempunyai sistem perakaran dangkal, batang jarang terlihat karena tertutup oleh pelepah daun yang tersusun roset dan sangat rapat sekali. Akar serabut, menjalar datar di bawah tanah. Daerah perakaran tidak luas, dangkal dan mudah rusak jika kekeringan atau kelebihan air. Perkembangan akar terutama dipengaruhi oleh cara pengolahan tanah, pemupukan, tekstur tanah, sifat fisik tanah, sifat kimia tanah, air tanah. Untuk menjaga akar tetap tumbuh, maka perlu diadakan penimbunan dan setelah muncul akar-akar muda, akar yang tua dipotong (Tjahjadi, 1995; Santoso, 1990).
Buah salak tipe buah batu berbentuk segitiga agak bulat atau bulat telur terbalik, runcing di pangkal dan membulat di ujung, panjang 2,5-10 cm, terbungkus oleh sisik-sisik berwarna kuning coklat sampai coklat merah mengkilap yang tersusun seperti genting, dengan banyak duri kecil yang mudah putus di ujung tiap-tiap sisik. Dinding buah tengah (sarkotesta) tebal berdaging, kuning krem sampai keputihan, berasa manis, masam, atau sepat. Biji 1-3 butir, berwarna coklat hingga kehitaman, keras, panjang 2-3 cm. Salak terutama ditanam untuk dimanfaatkan buahnya, yang populer sebagai buah meja. Selain dimakan segar, buah ini juga biasa dibuat manisan, asinan, dikalengkan, atau dikemas sebagai keripik. Buah yang muda digunakan untuk bahan rujak.
commit to user
pun kerap digunakan untuk mempersenjatai pagar, atau untuk melindungi pohon yang tengah berbuah dari pencuri.
3. Sistem klasifikasi tanaman salak
Ada beragam cara untuk mengelompokkan tanaman antara lain pengelompokan berdasarkan kegunaan untuk manusia, menurut tempat tumbuh, kebiasaan tumbuh, menurut adaptasi iklim, menurut wujud, bentuk dan struktur (morfologi) serta klasifikasi menurut struktur reproduksi dan klasifikasi yang lebih modern dengan menggunakan teknik pembedaan kromosom serta analisis DNA yang lebih menekankan pada analisis genetika (Harjadi, 1982 dalam Nandariyah, 2007). Penggolongan berdasarkan morfologi merupakan penggolongan yang masih umum di gunakan pada saat ini.
Klasifikasi keragaman tanaman salak yang ada saat ini umumnya adalah penggolongan berdasarkan ciri-ciri morfologi vegetatif dan generatif. Identifikasi dan klasifikasi pada tanaman salak telah dicoba oleh beberapa peneliti. Suskendriyati et al., (2000) membedakan tanaman salak pondoh di dataran tinggi Sleman berdasarkan perbedaan morfologi batang, daun, bunga, buah dan duri. Sukaya (2003) meneliti keragaman dan mengelompokkan kultivar-kultivar salak di Sleman berdasarkan karakter morfologi dan mendapatkan kesimpulan bahwa peubah morfologi buah mempunyai keragaman terbesar. Harsono (1994) mengelompokkan kultivar salak di Madura berdasarkan ciri morfologi dan menggabungkan dengan pola pita isozim namun belum diperoleh hasil yang maksimal.
commit to user
dan molekuler DNA (Melchinger, 1992). Beberapa macam penanda yang dapat digunakan untuk membedakan varietas antara lain: morfologi tanaman, pola pita isozim, dan pola pita DNA. Penanda molekuler memberikan suatu kemungkinan untuk mendapatkan hubungan genetik yang lebih akurat dibandingkan dengan penanda-penanda yang lain. Penanda molekuler memberikan kemungkinan yang lebih luas dan akurat dalam mendapatkan hubungan genetik karena, secara potensial memiliki jumlah penanda yang tidak terbatas, tidak dipengaruhi oleh kondisi lingkungan, dapat diarahkan untuk analisis keterpautan, dapat mengidentifikasi bahan persilangan dalam jumlah banyak, dan dapat mengidentifikasi tanaman pada stadia awal (Nienhuis et al., 1994).
Karakter morfologi seringkali dipengaruhi oleh perubahan lingkungan. Pengamatan morfologi juga harus memperhatikan umur tanaman, karena perubahan umur mempengaruhi perubahan morfologi. Sifat genetik cenderung stabil terhadap perubahan lingkungan, dan tidak dipengaruhi oleh umur, sehingga penanda genetik dapat memberikan informasi yang relatif lebih akurat (Pratamaningtyas, 1997; Sukartini, 2001).
4. Keragaman Jenis Salak
Berbagai aksesi tanaman salak tumbuh di pusat-pusat budidaya tanaman salak khususnya di Jawa. Di daerah pusat budidaya salak, seringkali dijumpai keragaman aksesi yang dinamai sesuai dengan ciri khas warna kulit buah, daging buah, rasa, aroma dan daerah asal. Ragam kultivar salak terjadi karena pada umumnya diperbanyak dengan menggunakan biji tanaman salak dan menyerbuk silang (Ashari, (1995) dalam Nandariyah, 2007).
commit to user
Queensland dan juga Fiji. Sebagian ahli menganggap salak yang tumbuh di Sumatra bagian utara berasal dari jenis yang berbeda, yang dibedakan atas dua varietas botani, yakni var. zalacca dari Jawa dan var. amboinensis (Becc.) Mogea dari Bali dan Ambon. Jenis spesies salak yang pernah ditemukan di dunia kurang lebih 20 spesies, namun baru sekitar 13 spesies yang diketahui dengan pasti identitasnya. Dari 13 spesies itu ternyata ragam salak paling banyak dan dibudidayakan di Indonesia. Dewasa ini ada beberapa varietas salak yang telah dilepas oleh pemerintah antara lain salak Pondoh, Nglumut, Enrekang, Bali, dan Suwaru (Nandariyah, 2007).
Tabel 1. Varietas salak yang dilepas dari beberapa wilayah di Indonesia
Varietas Salak Wilayah
Gondok, Nangka, Nenas, Gula Pasir, Bule Kuning/Golla-golla Sumber : Puslibang Hortikultura 1995 dalam Sri Kaidah, 1999.
Salak Lawu berada di desa Matesih, Kecamatan Tawangmangu Kabupaten Karanganyar. Berada pada ketinggian 600 mdpl di lereng Gunung Lawu. Salak Lawu dibudidayakan oleh petani bersama dengan Salak Pondoh dan diperbanyak secara generatif (Nandariyah, 2007).
commit to user
generatif. Salak Saratan terus mengalami penurunan jumlah populasi dikarenakan terdesak oleh pemukiman penduduk (Nandariyah, 2007).
Salak Kecandran berasal dari desa Kecandran, Salatiga ± 5 km dari Desa Bejalen Ambarawa. Desa Kecandran merupakan sentra produksi salak karena sebagian besar penduduk memiliki tanaman salak. Namun sebanyak 60% keseluruhan jumlah tanaman salak telah beralih menjadi Salak Pondoh (Nandariyah, 2007).
Salak Bejalen berasal dari Desa Bejalen Kecamatan Ambarawa Kabupaten Semarang berada pada ketinggian 450 mdpl di tepi danau Rawa Pening. Salak Bejalen dibudidayakan oleh petani setempat diperoleh dari perbanyakan secara generatif. Salak bejalen mempunyai ciri rasa manis agak sepet sampai manis dengan tinggi rata-rata 429 cm. Panjang pelepah rata-rata 367 cm. Populasi tanaman salak terdiri atas sekelompok tanaman betina dan tanaman jantan dengan jumlah lebih sedikit dibandingkan tanaman betina (Nandariyah, 2007).
commit to user
Salak merupakan tanaman asli Indonesia, yang sampai saat ini belum diketahui secara pasti sejak kapan tanaman tersebut dibudidayakan pertama kali. Hanya diduga tanaman salak ini sudah dibudidayakan sejak ratusan tahun silam. Tanaman salak memiliki nama ilmiah Salacca edulis Reinw dan termasuk famili Palmae serumpun dengan kelapa, kelapa sawit,
aren (enau), palem, pakis yang bercabang rendah dan tegak.
Sejarah Salak pondoh bermula ketika Orangtua Muhadiwinarto, yang bernama Partodimejo menerima souvenir dari seorang berkebangsaan Belanda yang akan kembali ke negara asalnya. Salah satu dari souvenir tersebut adalah empat buah pohon salak.Kemudian pohon-pohon salak tersebut ditanam oleh Muhadiwinarto, disebuah desa Sukobinangun, Merdikorejo, Tempel sejak tahun 1948. Saat ini Salak pondoh telah menyebar keseluruh pelosok Sleman dan bahkan seluruh Indonesia. (afendry.com, 2012).
Salak pondoh merupakan jenis salak berdaging buah empuk seperti embut, rasanya renyah, tidak sepat dan tidak masam. Salak Pondoh Hitam berasal dari daerah Sleman, Yogyakarta, merupakan varietas salak unggul yang sudah sangat populer. Salah satu keunggulan yang menonjol adalah meskipun buah masih muda, tetapi rasa sudah manis. Buah berbentuk segitiga atau bulat telur terbalik. Daging buah terdiri atas tiga septa dalam setiap buah dan berwarna putih kapur. Ketebalan antara 0,8-1,5 cm dan tekstur keras. Dalam setiap buah terdapat 1-3 biji yang keras dan berwarna cokelat kehitaman. Ukuran buah antara 2,5-7,5 cm dan berat 30-100 g/buah. Jumlah buah pertandan antara 10-27. Salak ini mempunyai kulit buah yang paling gelap bila dibandingkan dengan salak pondoh lain dan berbentuk paling bulat.
commit to user
bersisik tersusun seperti genting pendek dan berwarna cokelat kekuningan. Dinding kulit bagian dalam berserat dan berdaging putih kekuningan. Buah muda berasa manis keasaman dan setelah tua berasa manis. Jumlah biji dalam setiap buah antara 2-3, biji berwarna kecokelatan, keras, dan terdapat sisi cembung serta datar. Ukuran buah cukup besar, panjang antara 2,5-8 cm dan berat sekitar 70 g/buah. Jumlah buah per tandan antara 10-50 buah (Nandariyah, 2007).
Salak Pondoh Super berasal dari Kabupaten Sleman memiliki kulit buah berwarna coklat kekuningan dengan daging buah berwarna coklat, tebal, rasa manis, renyah dan masir. Buah berbentuk memanjang dan berukuran besar, tiap kilogram berisi 9-11 butir buah, memiliki kulit bersisik yang tersusun rapi seperti genteng dan berduri halus serta biji berwarna coklat kehitaman (Nandariyah, 2007).
Salak Pondoh Manggala juga berasal dari Kabupaten Sleman memiliki kulit buah berwarna coklat kekuningan dan sisik pada bagian pangkal kulit buah tersusun membentuk lorek (ada warna putih diantara sisik) serat daging buah berwarna putih susu.
commit to user
1. Salak Madu Balerante, yang sudah dilepas sebagai varietas unggulan. 2. Salak Madu Sukomartani yang juga dikenal sebagai salak madu
probo.
Salak madu memang lebih enak dari salak pondoh super, apabila daging buah dipencet dengan jari akan keluar cairan seperti madu, cairan ini tidak dijumpai pada salak pondoh dan salak gading (Nandariyah, 2007).
B. Analisis RAPD-PCR
Metode RAPD-PCR merupakan kombinasi teknik PCR (Polymerase Chain reaction) menggunakan primer-primer dengan sekuen acak untuk
keperluan amplifikasi lokus acak dari genom (Rafalski et al., 1991). Metode ini mempunyai keunggulan pada kesederhanaan teknik dan pengerjaan cepat (Hu dan Quiros, 1991), sehingga RAPD layak digunakan dalam suatu analisis yang menggunakan jumlah sampel cukup besar dan dimanfaatkan dalam upaya pemuliaan tanaman, genetika populasi, dan studi biodiversitas (William et al.,1990; Yang dan Quiros, 1993). Penggunaan penanda RAPD akan menguntungkan industri benih karena dapat meningkatkan efisiensi identifikasi kultivar dan menurunkan biaya (Horejsi dan Staub, 1998). Untuk tingkat DNA teknik RAPD yang didasarkan pada reaksi berantai oleh polimerase merupakan analisis yang banyak dipakai karena disamping mudah, cepat, dan memerlukan DNA dalam jumlah sedikit.
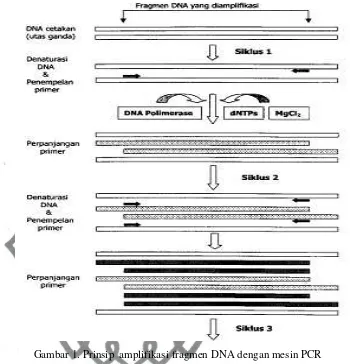
commit to user 1. PCR (polymerase chain reaction)
Penggunaan PCR diawali dengan ditemukannya DNA polimerase dari Eschercia coli. Enzim yang dihasilkan dari E. coli ini bersifat sensitif terhadap panas dan akan mudah rusak atau tidak aktif pada suhu yang diperlukan untuk memisahkan DNA pita ganda. Oleh karena itu dibutuhkan suatu enzim tertentu yang tahan panas pada suhu 940C agar kedua rantai DNA cetakan terpisah.
Kajian keragaman genetik tanaman sangat erat berhubungan dengan kajian tentang gen, DNA, dan kromosom. Seiring dengan perkembangan teknologi molekuler modern maka pengetahuan tentang DNA telah banyak dimanfaatkan dalam bidang biologi, kedokteran dan pemuliaan tanaman pertanian. Suatu teknik amplifikasi potongan DNA yang dikembangkan oleh Karry Mulis pada tahun 1985 yang telah banyak digunakan dalam analisis genetik tingkat molekuler adalah metode Polymerase Chain Raection. PCR adalah sebuah teknik biologi molekuler
untuk mereplikasikan DNA dengan menggunakan enzim Taq polimerase. Sedangkan menurut (Newton dan Graham, 1997) PCR adalah suatu metode amplifikasi DNA secara enzimatik terdiri dari serangkaian siklus denaturasi DNA, penempelan dan pemanjangan primer pada DNA cetakan berulang-ulang pada kondisi suhu yang disesuaikan. PCR digunakan untuk amplifikasi bagian DNA yang pendek (sampai 10 kb). Sejak ditemukan oleh Kary Mullis pada tahun 1985, teknik ini telah melahirkan teknik PCR-based marker teknik lainnya yang sangat bervariasi. Protokol dasar PCR adalah:
DNA utas ganda denaturasi pada suhu 95°C sehingga membentuk DNA utas tunggal yang berfungsi sebagai cetakan.
commit to user
Suhu ditingkatkan menjadi 72°C sehingga enzim DNA polymerase dapat melakukan sintesis DNA membentuk utas ganda DNA baru.
Utas ganda DNA yang baru disintesis, didenaturasi pada suhu tinggi dan siklus berulang.
Produk PCR diamati dengan gel elektroforesis dengan menggunakan gel agarose ataupun gel poliakrilamida dan diamati dengan uv-transiluminator.
Pada waktu melakukan amplifikasi, selama dua sampai lima siklus amplifikasi terdiri dari beberapa tahapan, yaitu:
a. Tahap Denaturasi
Pada tahap pertama ini utas ganda molekul DNA akan terpisah sempurna dan menghasilkan pita tunggal yang merupakan cetakan bagi primer. Penyebab kegagalan PCR yang paling umum adalah denaturasi yang tidak sempurna dari cetakan DNA (Innis dan Gelfand,
1980 dalam Sri kaidah, 1999). Suhu denaturasi biasanya 940C selama 30 detik atau 970C selama 15 detik. Denaturasi yang kurang sempurna akan memacu benang DNA snabback dan mengurangi hasil produk amplifikasi. Apabila denaturasi terlalu tinggi atau terlalu lama akan menyebabkan kehilangan aktivitas enzim yang seharusnya tidak perlu terjadi (Innis dan Gelfand, 1980 dalam Sri kaidah, 1999).
b. Tahap Annealling
Pada proses annealing suhu dan lama waktu tergantung pada komposisi, panjang, dan konsentrasi primer. Pauls et al. (1993) dalam Kaidah (1999) menyatakan bahwa suhu annealing dipengaruhi oleh panjang, banyak G dan C dalam primer dan konsentrasi garam larutan buffer. Adapun faktor- faktor tersebut mengikuti rumus berikut:
Tm = 81,5 + 16,6 (log M) + 0,41 (%GC) – (500/n)
commit to user
aktif dalam selang temperatur yang cukup lebar (Taylor, 1992 dalam Kaidah, 1999).
Temperatur annealing mempunyai selang antara 550 C-720 C. Untuk kisaran temperatur antara 200 C dan 850C untuk primer 20-mer sedangkan 340C - 400C untuk primer 10-mer (Uphoff dan Wricke, 1992 dalm Kaidah, 1999). Suhu annealing lebih rendah dari suhu denaturasi yang bertujuan untuk memberikan kesempatan bagi primer
untuk menempel pada urutan komplemen yang ada dalam molekul DNA target. Suhu annealing yang terlalu tinggi akan mempertinggi misextention dari nukleotida. Sedangkan suhu annealing yang terlalu
rendah akan menimbulkan misincroporation nukleotida. Suhu annealing merupakan variabel kunci yang menentukan kekhususan
amplifikasi suatu DNA. c. Tahap Ekstensian
commit to user
Gambar 1. Prinsip amplifikasi fragmen DNA dengan mesin PCR
Metode RAPD mampu mendeteksi sekuen nukleotida dengan hanya menggunakan satu primer. Primer tersebut akan berikatan dengan utas DNA pasangannya dengan arah orientasi yang berlawanan. Selama penempelan primer masih berada dalam jarak yang masih dapat diamplifikasi, maka akan diperoleh produk DNA amplifikasi (Tingey et al., 1992; Weising et al., 1995; Hayati, 1999). Pelaksanaan amplifikasi DNA dengan menggunakan reaksi PCR diketahui bahwa hasil akhir sangat ditentukan oleh banyak faktor seperti konsentrasi DNA cetakan, ion magnesium, Dntp, Taq polymerase, jenis dan konsentrasi primer, serta kondisi yang
commit to user
2. RAPD (Random Amplified Polymorphic DNA)
Salah satu teknik molecular marker yang menggunakan PCR adalah Random Amplified Polymorphism DNA (RAPD). Metode RAPD merupakan pengembangan teknik PCR untuk mendeteksi keragaman genetik atau mengidentifikasi jenis dengan mengamplifikasi potongan DNA spesifik yang berkomplementer dengan cetakan DNA. RAPD bertujuan untuk menghasilkan banyak copy dari DNA cetakan. Potongan-potongan acak yang umumnya berukuran antara 250-2000 pasangan basa diamplifikasi menggunakan thermocycle dengan primer tunggal, yang berukuran 10 pasangan basa.
Metode standar RAPD menggunakan oligonukleotida tunggal pendek (10-12 basa) dengan urutan acak sebagai primer untuk mengamplifikasi genomik DNA dalam jumlah nanogram dengan temperatur annealing yang rendah. Produk amplifikasi PCR dipisahkan dengan agarose gel diwarnai dengan ethidium bromide. Primer decamer secara komersial tersedia di berbagai sumber (misalnya Operon Technologies Inc., Alameda, California atau University of British Columbia, Canada). Analisis RAPD berbeda dengan kondisi PCR standar dimana hanya menggunakan satu primer dan tidak memerlukan informasi sekuen DNA awal (Nandariyah, 2007).
Pada temperatur annealing yang tepat selama siklus thermal, oligonukleotida primer dengan urutan sekuen acak berikatan pada beberapa priming site pada sekuen komplementer pada template DNA genomik dan menghasilkan produk jika priming site berada dalam wilayah/jarak yang dapat diamplifikasi. Profil amplifikasi DNA tergantung pada homologi sekuen nukleotida antara template/cetakan DNA dengan oligonucleotide primer. Variasi nukleotida antar template DNA menghasilkan ada tidaknya band karena perubahan priming site.
commit to user
tomat, kentang, dan jagung (Newton and Graham,1997). Setiyo Eko (2001) menggunakan RAPD untuk pemetaan dan keragaman genetik kelapa sawit. Hayati (1999) telah meneliti penggunaan penanda RAPD untuk mendeteksi keragaman genetik kelapa genjah di Jombang. Dwiatmini (2002) menggunakan penanda RAPD untuk analisis pengelompokan dan hubungan kekerabatan spesies anggrek Phalaenopsis. Septimayani (2002) meneliti keragaman genetik blewah (Cucumis melo L). Sedangkan Robi’ah (2004) menggunakan penanda RAPD untuk menganalisis keragaman genetik pisang introduksi (Musa spp). Apriyani (2005) memakai teknik RAPD untuk analisis keragaman
buah nanas. Galingging (2005) memanfaatkan penanda RAPD untuk menganilis keragaman buah pepaya. Sedangkan Pandin (2000) menggunakan RAPD untuk mengetahui kemiripan genetik populasi kelapa. Identifikasi keragaman kultivar tanaman jeruk berdasarkan penanda RAPD dilakukan oleh Karsinah et al. (2002). Mansyah et al., (2003) memanfaatkan teknik RAPD untuk analisis keragaman genetik manggis (Garcinia mangostana L). Sedangkan Nurhaimi dan Darusamin meneliti penggunaan penanda RAPD untuk mendetekasi dan mengelompokkan klon kelapa sawit yang berbuah normal dan abnormal (Nurhaimi dan Darusamin, 1997).
3. Aplikasi analisis RAPD
Teknik RAPD yang sederhana memerlukan biaya lebih murah sehingga aplikasi yang sangat luas dari RAPD pada berbagai area biologi. Beberapa area tersebut antara lain: Kemampuan RAPD mendeteksi variasi intra-specifik dapat digunakan untuk melakukan screnning untuk tingkat inbreeding pada tanaman komersial untuk mencegah peningkatan frekuensi alel resesif yang merugikan dalam populasi.
Marker species-specific digunakan dalam inter-specific gene flow
commit to user
bereproduksi secara seksual. Karena bereproduksi secara aseksual, maka fragmen polimorfik antar individual dapat digunakan untuk menentukan identitas klonal.
Walaupun metode RAPD relatif cepat, murah dan gampang dilaksanakan dibandingkan metode marker DNA lain, berita tentang konsistensi/reproducibility menjadi perhatian sejak teknik ini diperkenalkan. RAPD sangat sensitif terhadap perubahan kondisi reaksi PCR. Problem reproducibility/konsistensi biasanya terjadi pada band/pita dengan intensitas yang rendah. Hal ini mungkin terjadi karena primer tidak cocok secara sempurna pada sekuen priming site, amplifikasi pada beberapa siklus mungkin tidak terjadi sehingga band tetap samar.
4. Isolasi DNA
Isolasi merupakan pemisahan materi dari materi lain untuk di identifikasi. Pada dasarnya isolasi DNA dapat dilakukan dari berbagai sumber, antara lain organ manusia, darah, daun, daging buah, serangga, kalus, akar batang, daging. Bahan isolasi DNA tanaman yang diisolasi dari tanaman seringkali terkontaminasi oleh polisakarida dan metabolit sekunder seperti tanin, pigmen, alkaloid dan flavonoid. Sedangkan DNA dari hewan lebih banyak mengandung protein. Salah satu kesulitan isolasi DNA dari tanaman tinggi adalah proses destruksi dinding sel untuk melepaskan isi sel. Hal ini disebabkan karena tanaman memiliki dinding sel yang kuat dan seringkali pada beberapa jenis tanaman, kontaminasi tersebut sulit dipisahkan dari ekstrak asam nukleat. Kehadiran kontaminasi di atas dapat menghambat aktivitas enzim, misalnya DNA tidak sensitif oleh enzim restriksi dan menggangu proses amplifikasi DNA dengan PCR. Demikian pula pada hewan yang memiliki kandungan kitin (seperti serangga), memerlukan teknik dan metode khusus untuk menghancurkan sel hingga isi dapat terpisah atau keluar dari sel (Nandariyah, 2007).
commit to user
Elektroforesis adalah teknik pemisahan komponen atau molekul bermuatan berdasarkan perbedaan tingkat migrasinya dalam sebuah medan listrik. Medan listrik dialirkan pada suatu medium yang mengandung sampel yang akan dipisahkan. Teknik ini dapat digunakan dengan memanfaatkan muatan listrik yang ada pada makromolekul, misalnya DNA yang bermuatan negatif. Jika molekul yang bermuatan negatif dilewatkan melalui suatu medium, kemudian dialiri arus listrik dari suatu kutub ke kutub yang berlawanan muatannya maka molekul tersebut akan bergerak dari kutub negatif ke kutub positif. Kecepatan gerak molekul tersebut tergantung pada nisbah muatan terhadap massanya serta tergantung pula pada bentuk molekulnya. Pergerakan ini dapat dijelaskan dengan gaya Lorentz, yang terkait dengan sifat-sifat dasar elektris bahan yang diamati dan kondisi elektris lingkungan:
F adalah gaya Lorentz, q adalah muatan yang dibawa oleh objek, E adalah medan listrik.
Secara umum, elektroforesis digunakan untuk memisahkan, mengidentifikasi, dan memurnikan fragmen DNA. Elektroforesis gel merupakan suatu teknik analisis penting dan sangat sering dipakai dalam bidang biokimia dan biologi molekular. Secara prinsip, teknik ini mirip dengan kromatografi, memisahkan campuran bahan-bahan berdasarkan perbedaan sifat. Dalam elektroforesis gel, pemisahan dilakukan terhadap campuran bahan dengan muatan listrik yang berbeda-beda. Dalam elektroforesis gel terdapat dua material dasar yang disebut fase diam dan fase bergerak (eluen). Fase diam berfungsi "menyaring" objek yang akan
commit to user
terjadi aliran elektron dan zat objek akan bergerak dari elektrode negatif ke arah sisi elektrode positif. Kecepatan pergerakan ini berbeda-beda, tergantung dari muatan dan berat molekul DNA. Kisi-kisi gel berfungsi sebagai pemisah. Objek yang berberat molekul lebih besar akan lebih lambat berpindah.
Elektroforesis Gel Agarosa didasarkan pada pergerakan mulekul bermuatan dalam media penyangga matriks stabil di bawah pengaruh medan listrik. Media yang umum digunakan adalah gel agarosa atau poliakrilamid. Elektroforesis gel agarosa digunakan untuk memisahkan fragmen DNA yang berukuran lebih besar dari 100 pb dan dijalankan secara horizontal, sedangkan elektroforesis poliakrilamid dapat memisahkan 1 pb dan dijalankan secara vertikal. Elektroforesis poliakrilamid biasanya digunakan untuk menentukan urutan DNA (sekuensing).
commit to user C. Hipotesis
Hipotesis penelitian ini dapat disusun sebagai berikut:
1. Keragaman kultivar salak pondoh dan non pondoh dapat diklasifikasikan dengan penanda RAPD-PCR.
commit to user
23 BAB. III
METODE PENELITIAN
A. Waktu dan Tempat Penelitian
Penelitian ini dilaksanakan pada bulan April – Mei 2012 di CV. Agribiotech dan Laboraturium Fisiologi dan Bioteknologi Fakultas Pertanian Universitas Sebelas Maret Surakarta.
B. Bahan dan Alat Penelitian
Bahan yang digunakan dalam penelitian ini adalah daun tanaman salak varietas pondoh dan non pondoh. Sampel yang diteliti sebanyak dua puluh dua kultivar yang dibedakan:
1. Kultivar salak non pondoh: Salak Saratan (magelang), salak Bejalen (Ambarawa), salak Kecandran (Salatiga), salak Gading (Sleman), salak Nglumut (Magelang), salak Kelapa bali (Bali), salak Gula pasir, salak Kediri, salak Madura, salak Manggala, salak Engrekang, salak Lumajang, salak Thailand, salak Banjar, salak Merah, salak Suaru, dan salak Tasik.
2. Kultivar salak pondoh: Salak Pondoh Super, Pondoh Hitam, Salak Pondoh Madu (Sleman D.I. Yogyakarta), Pondoh Lawu (Jawa Tengah), Salak Kembang arum (Sleman), dan pondoh Hijau.
Bahan-bahan yang dibutuhkan dalam analisis DNA total dengan RAPD-PCR ada dua metode yaitu CTAB dengan buffer dan Dynamid Plant Kit. Bahan- bahan yang digunakan isolat DNA total adalah: Dynamid plant kit yang terdiri atas 3 jenis larutan yang terdiri atas larutan LA (lisis Acid),
PA (Protein acid) dan CA (Capture Acid), akuades steril, etanol absolut, alkohol 70%, agarosa 1%, megamix blue, good view II sebagai pewarna, loading dyne sebagai pemberat dan primer. Primer yang digunakan sebanyak
commit to user
total dengan metode CTAB adalah larutan buffer reaksi, TAE 1X, Chloroform, 2,5 M sodium asetat, Isopopanol, Aquades steril dan etanol 70%. Alat-alat yang digunakan dalam penelitian adalah mortal dan pistil, gunting, ice box, kertas label, tabung eppendorf 1,5 ml, pipet mikro Eppendorf
(10-100 µl, (10-100-(10-1000 µl, dan 2-10 µl), pipet tetes, Erlenmeyer, spatula, tabung eppendurf PCR, Centrifugasi, water bath, tips Eppendorf (yellow tips dan blue tips), frezer, Gelas ukur 500 ml, Elektroforesis neraca analitik (4 desimal), micro wave, cetakan agarosa dan sisiran, sumuran, transluminator T 220V, mesin PCR (Thermocycler), Lampu UV, dan kamera digital makro. C. Cara kerja Penelitian
1. Pengambilan sampel
Populasi tanaman salak yang digunakan untuk penelitian laboratorium dengan mengambil 10 sampel daun sebagai sumber isolat DNA. Sampel daun yang diambil adalah daun yang masih muda dari bagian pucuk dan baru berkembang penuh. Daun diambil sebanyak 5-10 daun dari lokasi yang berbeda-beda. Setelah daun dipotong dari tangkai, kemudian dimasukan dalam kantong plastik diberi label, agar tetap terjaga kesegaran daun dimasukkan dalam coolbox/kotak pendingin yang berisi es dan dapat disimpan pada alat pendingin -700C untuk kemudian dapat di isolasi/ekstraksi.
2. Isolasi/ekstraksi DNA
commit to user
sebentar ditambahkan larutan TE pH 8 lalu sentrifugasi pada 5000 rpm selama 5 menit. Sampel pellet DNA disimpan dalam suhu -200C.
commit to user
telah terbentuk, kemudian menambahkan dengan etanol 70% sebanyak 70 µl, kemudian pellet DNA dapat disimpan pada suhu -20 0C.
3. Uji kualitas DNA
Kuantitas DNA diukur dengan menggunakan alat Spektrofotometer, dengan cara memasukkan 2µl/100µl DNA dan sebagai pembanding 100 µl aquadest ke dalam kufet pada alat spektrofotometer yang sudah diatur dengan panjang gelombang 260 nm dan 280 nm. Kuantitas DNA diketahui dari hasil pengukuran optical density (OD) 260/280. Kualitas DNA diuji dengan melakukan elektroforesis gel agarosa dan divisualisai pada alat foto polaroid merk MP4 Land Camera. Gel dibuat dengan cara melarutkan 0,20% (b/v) agarosa dalam larutan buffer TAE 1X, lalu dipanaskan dalam micro wave selama 5 menit hingga larut semua. Setelah itu didinginkan
selama 10 menit, kemudian ditambah 2 µl goodView II dan dikocok perlahan merata. Larutan tersebut lalu dituang kedalam cetakan gel yang telah disiapkan. Sisir pembuat sumur diletakkan dengan jarak 0,5-0,1 mm dari dasar cetakan. Gel dibiarkan memadat selama kurang lebih satu jam.
Gel yang telah memadat dimasukkan ke dalam bak elektroforesis yang berisi larutan buffer TAE 1X yang sudah diberi Good View II sampai seluruh gel terendam. Contoh DNA yang telah ditambah loading buffer dimasukkan ke dalam lubang sumur yang ada pada gel. Elektroforesis dilakukan pada voltage 100 volt selama kurang lebih 30 menit. Ekstraksi DNA diulangi apabila DNA yang dihasilkan belum menghasilkan pita yang jelas. Hasil eletroforesis dapat dilihat dengan UV transluminator. 4. Seleksi primer
commit to user
Tabel 1. Jenis primer dan urutan nukleotida penyusunnya
No Primer Sekuens nukleotida (5’-3’)
1. temperatur 950C dilanjutkan tahap II adalah proses amplifikasi berlangsung sebanyak 40 siklus, dimulai dengan pemisahan utas DNA genom (denaturasi) pada temperatur 940C selama 1 menit, penempelan primer (annealing) pada temperatur 370C selama 45 detik, pemanjangan utas DNA (extension) pada temperatur 720C selama 1,5 menit, diakhiri dengan tahap perpanjangan akhir (tahap III), pada temperatur 720C selama 5 menit (Nandariyah, 2008).
Primer yang digunakan untuk amplifikasi DNA (RAPD) terdiri dari tiga macam primer: OPA-11, OPA-16, dan OPA-17 yang diseleksi dari 12 macam primer yaitu X-17, D-18, D-16, X-15, D-17, D-15, D-13, 12, A-18, OPA-11, OPA-16 dan OPA-17. Kuantitas DNA diketahui dari hasil pengukuran optical density (OD) 260/280 dan kualitas DNA diketahui dari hasil elektroforesis gel dan divisualisasi pada alat foto polaroid merek MP4 Land Camera. Visualisasi pita DNA hasil amplifikasi dilakukan dengan mengamati pita-pita DNA setelah dielektroforesis pada alat UV-transilluminator (Hoefer) dan dilanjutkan pemotretan menggunakan tustel
Polaroid MP4 Land Cameradiafragma 8, jarak lensa 14,8 ketinggian 52,3 cm pada film polaroid 667.
6. Elektroforesis
commit to user
kertas parafin ditambah dengan 10μl marker DNA leader 1 kb. Running dalam cetakan agarose selama 15 menit 100 volt.
7. Visualisasi hasil RAPD
Visualisasi pita DNA hasil amplifikasi dilakukan dengan mengamati pita-pita DNA setelah dielektroforesis pada alat UV-transilluminator (Hoefer) dan dilanjutkan pemotretan tustel Polaroid MP4 Land Camera.
D. Analisis Data
Data yang diperoleh dari pemotretan gel hasil RAPD yang diharapkan adalah pita-pita diksrit (tajam dan jelas) dengan ukuran tertentu dari masing-masing nomor genotipe salak. Jarak pita diukur dari batas bawah sampai batas atas yang masih nampak. Nomor pita diurutkan dari jarak pita terdekat dengan batas bawah sumur. Setiap pita dianggap sebagai satu karakter dan
dinilai „1‟ bila terdapat pita dan „0‟ bila tidak ada pita. Pita DNA yang dinilai
adalah pita yang terlihat baik bersolusi tajam atau lemah. Berdasarkan ada tidaknya suatu pita, disusun matriks data biner yang diturunkan menjadi matriks persamaan (matriks jarak genetic). Untuk menentukan kesamaan pasangan genotipe yang terdapat pada individu yang berbeda digunakan rumus Nei dan Li sebagai berikut:
F = 2 n ab/ ( na + nb)
F adalah koefisien persamaan, nab adalah jumlah pita sama posisinya, baik individu a dan b, na dan nb adalah jumlah pita masing-masing individu a dan b
Pada program NTSYS, data matriks dihitung melalui koefisien “Dice”
yang pada prinsipnya sama dengan rumus Nei dan Li, yakni: Sb = 2a / n1 +n2
Untuk menghitung koefisien jarak data harus dikonversikan dengan ; d = 1- S
d adalah jarak, dan S adalah nilai matrik persamaan yang dihitung dengan rumus Nei dan Li.
commit to user
dilakukan analisis cluster (pengelompokkan) secara manual dengan cara dipilih nilai jarak paling kecil dalam jarak matriks, dua individu dibuat cabang, dan cabangnya adalah setengah jarak tersebut, mengelompokkan dua individu lainnya berdekatan dan membuat cabang dari setengah jarak tersebut, dan antar cabang juga dibuat, sampai akhir terbentuk diagram pohon. Dalam penelitian ini seluruh pengolahan data menggunakan program-program dalam computer. Prosedur pengolahan data tersebut adalah sebagai berikut:
1. Penyusunan data dalam bentuk data biner pada program Excel dengan keluaran data berbentuk Formatted Text/ Space delimited (.prn)
2. Data dalam bentuk .prn dimasukkan dalam program Notepad, diubah menjadi .dat.
3. Data berbentuk .dat dimasukkan dalam program Windisk dan diolah menjadi data matrik (.mat)
4. Pemrosesan data, matriks menggunakan program NTSYS-pc, dengan tahap: Data .mat dimasukan dalam analisis UPGMA dalam program Shan clustering menghasilkan pengelompokkan data berdasarkan jarak genetik. Hasil pengelompokkan dimasukkan dalam program Tree Display untuk menghasilkan dendogram.
5. Untuk mendapatkan hasil cetakan yang baik, hasil analisis TreeDisplay dipindahkan ke dalam program MS Word.
6. Pengujian validitas pengelompokkan dilakukan oleh NTSys, digunakan analisis bootstrap didalam program WinBoot.
7. Presentase pengelompokkan yang dilakukan WinBoot dituliskan dalam dendogram pada program MSWord.
8. Untuk mengetahui korelasi antar primer digunakan analisis Comparison dalam program NTSys. Data yang dipakai berasal dari data .mat keluaran data berupa nilai r (korelasi).
a. Analisis Similaritas
commit to user
sama berdasarkan penanda RAPD, dan data diolah dengan NTSYS pc versi 2.02 dengan proses Similarity for Qualitative Data ( SIMQUAL) dan dihitung berdasarkan metode Simple Matching Coefficient (SM) dari Sokal dan Sneath (Rohlf, F.J. 1998) dengan rumus sebagai berikut
SM = m/n
SM adalah koefisien similaritas, m adalah banyaknya data dengan pola yang sama, dan n adalah jumlah data. Analisis keragaman genotipik berdasarkan keragaman dianalisis dengan langkah: membuat matrik karakter vs genotipe, data hasil skoring dalam sofware untuk analisis keragaman, membuat dendogram hasil analisis gerombol. Data fenotipik tersebut dianalisis dengan NTSYSpc versi 2.02.
b. Analisis gerombol (Cluster analysis)
Analisis gerombol (Cluster analysis) menggunakan metode Sequential, Agglomerative, Hierarchical and Nested Clusthering (SAHN).
Setiap karakter diubah dalam bentuk data biner. Nilai 1 diberikan pada subkarakter yang terdapat pada genotip dan yang tidak tampak diberi 0. Pola pita DNA hasil amplifikasi pada tanaman akan menunjukkan jumlah pita dan jarak migrasi. Penilaian dilakukan terhadap pita yang jelas dan terang secara konsisten yakni skor nol (0) jika tidak ada pita dan satu (1) jika ada pita pada posisi yang sama individu yang dibandingkan. Analisis pengelompokan adalah berdasarkan kesamaan genetik yang disajikan dalam bentuk dendogram sehingga dendogram yang dihasilkan adalah analisis gerombol berdasarkan penanda genotipik.
commit to user
31 BAB. IV
HASIL PENELITIAN DAN PEMBAHASAN
A.Klasifikasi keragaman tanaman salak pondoh dan non pondoh berdasarkan penanda molekuler RAPD
Pengklasifikasian pengelompokan kultivar salak pondoh dan non pondoh yang dibedakan atas dasar kesamaan sifat-sifat atau tingkat kekerabatan kelompok masing-masing. Penelitian dimulai dengan ekstraksi DNA dan uji kualitas DNA hasil ekstraksi, menyeleksi primer untuk penanda RAPD, amplifikasi DNA dengan teknologi PCR, elektroforesis hasil amplifikasi DNA, analisis cluster data dan penggolongan kekerabatan berdasarkan RAPD dan analisis diskripstif.
Keragaman genetik 22 genotipe ditentukan berdasarkan hubungan kesamaan genetik antar genotipe tanaman salak yang berbeda dengan cara membandingkan pita DNA hasil amplifikasi PCR dengan primer tertentu pada setiap genotip tanaman. Hasil analisis kesamaan genetik dengan menggunakan koefisien kesamaan genotipe dengan metode Nei dan Lei (Kaidah, 1999).
1. Hasil isolasi dan uji kualitas DNA 1a). Isolasi DNA
commit to user
berbeda. Teknik CTAB menggunakan buffer ekstraksi phenol, dapat ditambah dengan chloroform dan isopropanol. Pada lapisan phenol akan melarutkan lipid, polisakarida dan protein. Chloroform merupakan pelarut yang menstabilkan ikatan antara lapisan organik dan aquaeus (sampel DNA yng tertinggal). Metode Dynamid Plant Kit menggunakan tiga bahan praktiks yang sudah ada dan dibuat dari
pabrikan, terdiri dari empat jenis bahan yaitu LA (Lisis Acid) merupakan larutan resuspensi (buffer dan pengkelat), larutan PA (Protein Acid) terdiri dari SDS dan NaOH (pelisis) dapat mendenaturasi protein sedangkan CA berfungsi merenaturasi kembali dan RNase untuk menghilangkan sisa-sisa RNA atau protein yang masih tertinggal pada DNA. Sebagai dasar untuk analisis lanjutan seperti PCR, RFLP, kloning atau sekuensing, maka DNA yang digunakan harus bersih dari kontaminan (kemurnian tinggi) dan memiliki berat molekul yang tinggi, selama proses ekstraksi beberapa hal yang dapat terjadi:
a. DNA patah-patah selama proses isolasi b. DNA terdegradasi oleh enzim nuklease c. Terjadi kontaminasi oleh polisakadarida
d. Metabolit sekunder ikut terisolalsi (Fatchiyah et al., 2011)
commit to user
diberikan pada setiap sampel yang dimasukkan dalam sumur gel agarosa sebelum gel dicetak dalam cetakan gel.
1b). Uji Kualitas DNA
Metode standar yang digunakan untuk identifikasi, pemisahan, dan purifikasi fragmen DNA adalah menggunakan elektroforesis gel agarosa. Selama proses pengeringan pellet DNA, disiapkan agarosa 1% (0,20 gram agarosa dalam 20 ml TBE 1x). Untuk proses eletroforesis ditambahkan TE 70 µl pada pellet DNA lalu sentrifugasi, diambil 5 µl DNA ditambhkan 2 µl blue Juice (BJ) 10x dan running/elektroforesis pada tegangan 100 volt selama 30 menit. Hasil elektroforesis kemudian dilihat pada UV transiluminator.
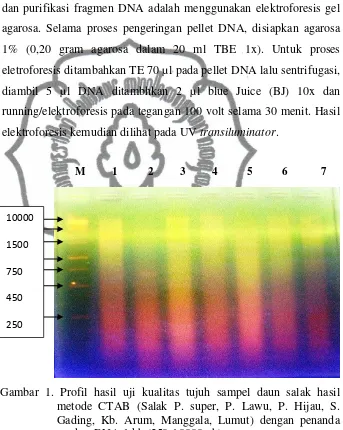
Gambar 1. Profil hasil uji kualitas tujuh sampel daun salak hasil metode CTAB (Salak P. super, P. Lawu, P. Hijau, S. Gading, Kb. Arum, Manggala, Lumut) dengan penanda marker DNA 1 kb (250-10000 pb)
Hasil uji kualitas tujuh sampel DNA dengan elektroforesis agarosa dari isolat DNA hasil isolasi dengan metode CTAB menunjukkan bahwa masih terdapat protein atau RNA sehingga hasil kualitas masih belum baik, belum menghasilkan DNA murni. Adanya
M 1 2 3 4 5 6 7
10000
1500
750
450
commit to user
RNA yang masih tercampur berupa garis berwarna merah kebawah. Hal ini dikarenakan beberapa faktor yaitu ikut terbawa debris (sampah) saat pengambilan supernatan dengan mikropipet, sehingga debris masih dapat tercampur kedalam supernatan yang akan menjadi isolat DNA, berpengaruh pada hasil DNA. Pada tahap akhir isolasi tidak ditambahkan RNase untuk melisiskan RNA supaya hasil isolasi berupa DNA murni, tidak tercampur oleh RNA sehingga isolat DNA yang dihasilkan belum murni.
M 1 2 3 4
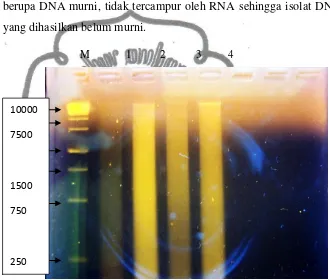
Gambar 2. Profil hasil uji kualitas DNA 4 sampel daun salak dengan metode CTAB (S. Kecandran, Banjar, P.madu, P. Hitam) dengan marker 1 kb (250-10000 pb)
Hasil elekroforesis menunjukkan dengan jelas posisi pita DNA dari empat sampel DNA. Hasil eletroforesis pada percobaan menghasilkan pita DNA pada bagian atas gel sedangkan pita RNA pada bagian bawah gel (apabila pada waktu dilakukan running tidak diberikan RNase). DNA mempunyai berat molekul lebih besar dibandingkan dengan berat molekul RNase. Menurut Sambrook et al. (1989) dalam Sri Kaidah (1999) kecepatan migrasi DNA
tergantung pada ukuran molekulnya. Sampel DNA hasil running 10000
7500
1500
750
commit to user
yang paling jelas terlihat terdapat pada sampel dua dan empat yaitu salak banjar dan pondoh hitam. Namun dari ke empat sampel DNA masih terdapat RNA. Hasil elektroforesis tidak dipotong dengan enzim restriksi akan memberikan pita DNA yang utuh. Hal ini menunjukkan bahwa semua isolat DNA dari daun salak mempunyai kualitas yang baik sehingga layak digunakan dalam penelitian analis DNA antara lain sebagai DNA cetakan untuk proses PCR.
M 1 2 3 4 5 6 7
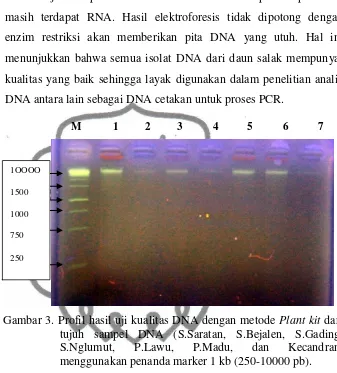
Gambar 3. Profil hasil uji kualitas DNA dengan metode Plant kit dari tujuh sampel DNA (S.Saratan, S.Bejalen, S.Gading, S.Nglumut, P.Lawu, P.Madu, dan Kecandran, menggunakan penanda marker 1 kb (250-10000 pb).
Hasil uji kualitas DNA dengan metode Dynamid Plant Kit menghasilkan isolat DNA murni tanpa campuran RNA, berupa garis tipis pada setiap sampel. Metode Dynamid plant kit dapat membersihkan sisa debris atau sampah. Selain itu garis RNA sudah tidak tampak seperti pada hasil isolasi dengan metode CTAB, dikarenakan pada tahap akhir isolasi dilakukan penambahan RNase untuk menghilangkan RNA yang masih menempel pada DNA. Untuk hasil isolat DNA murni yang berkualitas baik.
1OOOO
1500
1000
750
commit to user
M 1 2 3 4 5 6 7
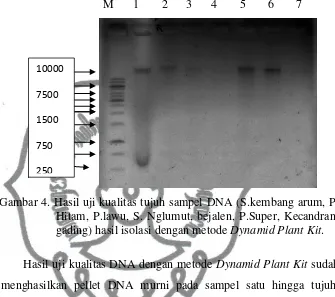
Gambar 4. Hasil uji kualitas tujuh sampel DNA (S.kembang arum, P. Hitam, P.lawu, S. Nglumut, bejalen, P.Super, Kecandran, gading) hasil isolasi dengan metode Dynamid Plant Kit.
Hasil uji kualitas DNA dengan metode Dynamid Plant Kit sudah menghasilkan pellet DNA murni pada sampel satu hingga tujuh, namun pada sampel satu varietas kembang arum masih terdapat RNA. Sedangkan pada sampel tiga dan empat yaitu varietas p.lawu dan S. nglumut sudah terdapat pellet DNA namun pita yang dihasilkan sangat tipis (konsentrasi sangat sedikit) sehingga pada waktu PCR konsentrasi harus ditingkatkan agar dapat amplifikasi dengan sempurna.
2. Hasil seleksi primer
Seleksi primer dilakukan untuk mendapatkan primer yang dapat mengamplikasi DNA. Dalam penelitian ini ada tujuh primer yang dipilih secara random. Primer diseleksi dari dua belas macam primer yaitu: X-17, X-15, A-12, A-18, OPA-11, OPA-16 dan OPA-17. Dari hasil penelitian seleksi primer menunjukkan bahwa tidak seluruh primer yang digunakan (tujuh primer) mampu mengamplifikasi setiap contoh genotipe salak. Hal
10000
7500
1500
750
commit to user
ini diduga tingkat kemurnian DNA template dari hasil bulking atau pooling masih rendah atau masih terdapat senyawa kontaminan seperti polifenol yang dapat menghambat terjadi reaksi PCR. Metode bulking atau pooling untuk seleksi primer belum dapat menjamin hasil amplifikasi yang diharapkan memperoleh pita-pita beragam atau polimorfis.
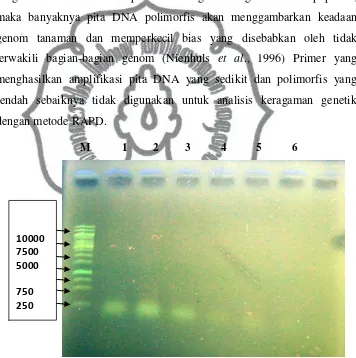
Jumlah pita DNA polimorfisme dalam analisis keragaman genetik sangat menentukan dalam penentuan tingkat keragaman suatu populasi, maka banyaknya pita DNA polimorfis akan menggambarkan keadaan genom tanaman dan memperkecil bias yang disebabkan oleh tidak terwakili bagian-bagian genom (Nienhuls et al., 1996) Primer yang menghasilkan amplifikasi pita DNA yang sedikit dan polimorfis yang rendah sebaiknya tidak digunakan untuk analisis keragaman genetik dengan metode RAPD.
M 1 2 3 4 5 6
Gambar 5. Hasil uji seleksi Primer OPA11, OPA17, OPA 16, OPX17, OPX -15, dan OPA- 18.
Dalam pemilihan primer sebaiknya primer- primer yang menghasilkan amplifikasi pita DNA yang sedikit dan polimorfis yang rendah tidak digunakan untuk analisis keragaman genetik dengan metode RAPD.
10000 7500 5000
commit to user
3. Hasil amplifikasi DNA dengan penanda RAPD
Penanda RAPD adalah penanda yang bersifat dominan yang berarti sifat tersebut tidak dapat dibedakan antara genotip heterosigot dengan homosigot tetapi dapat dibedakan terhadap sifat resesif dengan cara mendeteksi ada tidak pita DNA (Rafalski et al. 1994 dalam Nandariyah, 2007).
Secara umum analisis RAPD menunjukkan pola pita fragmen DNA yang baik berdasarkan tinggi konsistensi profil pita DNA antara tahap skrening primer dan tahap penggenotipan populasi pemetaan. Menurut Waugh (1997) banyak faktor yang mempengaruhi reproducibility profil RAPD, antara lain kondisi dan konsentrasi DNA, ko-ekstraksi pengkontaminasi DNA dan interferensi amplikasi.
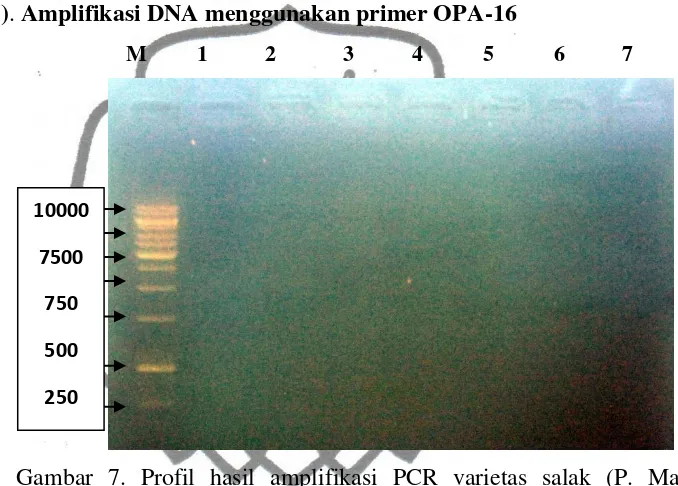
Persentase pita RAPD polimorfik pada beberapa penelitian keragaman genetik menunjukkan rata-rata tiap populasi sebesar 69% pada kelapa (Hayati et al., 2000) dan 56 % pada kelapa sawit (Rajanaidu, Maizura dan Cheah, 2000). Faktor yang menyebabkan perbedaan ini adalah jumlah dan jenis primer dan populasi yang digunakan. Berdasarkan tipe populasi yang digunakan, Rajanaidu et al. (2000) menunjukkan selang persentase polimorfik 8-94%, yang lebih besar dari Hayati et al. (2000) 62-76%. a). Amplifikasi DNA menggunakan primer OPA-11