ANALISIS KEBENARAN TETUA DAN KERAGAMAN
GENETIK POPULASI KELAPA SAWIT (
Elaeis guineensis
Jacq.) ASAL KAMERUN MENGGUNAKAN MARKA SSR
(SIMPLE SEQUENCE REPEAT)
LALU FIRMAN BUDIMAN
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa tesis berjudul Analisis Kebenaran Tetua dan Keragaman Genetik Populasi Kelapa Sawit (Elaeis guineensis Jacq.) Asal Kamerun Menggunakan Marka SSR (Simple Sequence Repeat) adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2016
Lalu Firman Budiman
RINGKASAN
LALU FIRMAN BUDIMAN. Analisis Kebenaran Tetua dan Keragaman Genetik Populasi Kelapa Sawit (Elaeis Guineensis Jacq.) Asal Kamerun Menggunakan Marka SSR (Simple Sequence Repeat). Dibimbing oleh SUDARSONO dan ADI PANCORO.
Penelitian mengenai analisis kebenaran tetua terhadap 8 populasi persilangan kelapa sawit asal Kamerun telah dilakukan di Kumai, Kalimantan Tengah, Indonesia, sejak April 2014 - April 2015. Penelitian ini bertujuanuntuk : 1) Mendeteksi terjadinya illegitimasi persilangan material introduksi Kamerun, 2) Memperoleh informasi keragaman genetik yang dimiliki material genetik origin (asal) Kamerun, 3) Mengetahui hubungan kekerabatan antar populasi.
Sebanyak 260 individu dari 8 populasi genotipe yang berbeda dianalisis menggunakan marka SSR dan dianalisis lebih lanjut menggunakan Software
GenAlex, Cervus, Colony, Principal Coordinate Analysis, dan Structure.Dari 16 marka SSR yang diuji, 14 marka SSR memiliki tingkat polimorfisme yang tinggi (sangat informatif) kecuali pada lokus mEgCIR0173 (LG 3) dan mEgCIR0353 (LG 16) yang memiliki nilai PIC yang rendah. Pengelompokan terjadi pada populasi dura (A125, A127, A140) dan populasi B01 (T x T). Sedangkan populasi B02 dan B57 memiliki jarak genetik yang jauh terhadap populasi dura. Pada populasi hasil persilangan D x P yaitu C22 dan C72, terlihat bila populasi C22 memiliki jarak genetik yang jauh terhadap populasi dura, namun sebaliknya, populasi C72 memiliki jarak genetik yang relatif dekat dengan populasi dura. Tingkat kemurnian dari seluruh populasi yang diuji sangat tinggi. Hanya ditemukan satu individu yang diduga ilegitim yaitu individu B01 (21-4).
SUMMARY
LALU FIRMAN BUDIMAN. Analysis of Genetic Diversity and Parent Legitimation in Oil Palm (Elaeis guineensis Jacq) Origin Cameroon Using SSR Marker. Supervised by SUDARSONO and ADI PANCORO.
A research to analyze genetic diversity from 8 (Eight) populations of palm crossing Cameroon origin have been conducted in Kumai, Central Kalimantan, Indonesia since April 2014 until April 2015. This study aims to 1) Detect the illegitimation of genetic material of Cameroon origin, 2) Obtain genetic diversity information from genetic material of Cameroon origin, 3) Know the kinship among populations.
A total of 260 individuals from eight populations of different genotypes were analyzed using Simple Sequence Repeat (SSR). Data were analyzed using Software GenAlex, Cervus, and Structure. From sixteen SSR markers were tested, fourteen SSR markers have a high degree of polymorphism (very informative) except at the locus mEgCIR0173 (LG 3) and mEgCIR0353 (LG 16) having a low value of the PIC. Grouping occurs in a population dura (A125, A127, A140) and a population of B01 (T x T). While the population of B02 and B57 had a genetic distance away on the population of the dura. In a population of cross D x P namely C22 and C72, C22 population has a genetic distance away to the dura population, but on the contrary, the population C72 has relatively close to dura population. The degree of purity of the population tested was quite high. Using Colony software, we only found one individual suspected ilegitim B01 (21-4).
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Pemuliaan dan Bioteknologi Tanaman
ANALISIS KEBENARAN TETUA DAN KERAGAMAN
GENETIK POPULASI KELAPA SAWIT
(
Elaeis guineensis
JACQ.)
ASAL KAMERUN MENGGUNAKAN MARKA
Simple Sequence Repeat
(SSR)
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
BOGOR
2016
Judul Tesis : Analisis Kebenaran Tetua dan Keragaman Genetik Populasi Kelapa Sawit (Elaeis guineensis Jacq.) Asal Kamerun Menggunakan Marka SSR(Simple Sequence Repeat)
Nama : Lalu Fiman Budiman
NIM : A253120261
Disetujui oleh Komisi Pembimbing
Prof. Dr.Ir. Sudarsono,MSc Ketua
Dr Adi Pancoro Anggota
Diketahui oleh
Ketua Program Studi Komunikasi Pembangunan Pertanian dan Pedesaan
Dr Yudiwanti Wahyu. E.K, MS
Dekan Sekolah Pascasarjana
Dr Ir Dahrul Syah, MScAgr
Tanggal Ujian: (12 Agustus 2016)
PRAKATA
Puji syukur penulis panjatkan kehadirat Allah SWT atas segala karunia-Nya sehingga penulis dapat menyelesaikan penelitian ini dengan judul ” Analisis Kebenaran Tetua Dan Keragaman Genetik Populasi Kelapa Sawit (Elaeis Guineensis Jacq.) Asal Kamerun Menggunakan Marka SSR(Simple Sequence Repeat)”sesuai dengan waktu yang telah ditetapkan. Pendidikan dan penelitian ini merupakan program pengembangan riset berkelanjutan dari PT Astra Agro Lestari Tbk. dan seluruh pembiayaannya didanai sepenuhnya oleh PT Astra Agro Lestari Tbk. Penulis mengucapkan terima kasih yang sebesar-besarnya kepada Bapak Prof. Dr. Ir. Sudarsono, MSc dan Bapak Dr. Adi Pancoro selaku pembimbing yang telah banyak memberikan masukan dan arahan hingga terselesaikannya penelitian ini dengan baik. Ucapan terima kasih yang tidak berhingga secara khusus penulis sampaikan kepada Bapak Widya Wiryawan Presiden Direktur dan Bapak Bambang Palgoenadi selaku Direktur PT. Astra Agro Lestari Tbk. yang peduli terhadap peningkatan sumber daya manusia Indonesia, penulis bersyukur menjadi salah seorang yang diberi kesempatan untuk meningkatkan potensi diri melalui jenjang akademik. Terima kasih secara khusus juga penulis haturkan kepada Bapak Satiyoso yang telah memberikan kesempatan dan motivasi kepada penulis sehingga kesempatan ini dapat dijalani dengan baik. Kepada segenap rekan-rekan peneliti di Research Center PT. Astra Agro Lestari Tbk, khususnya para peneliti di Laboratorium Molekular Kumai, Ardha, Hani, dan Reza yang banyak membantu didalam penyelesaian penelitian ini sehingga dapat kami selesaikan dengan baik. Kami juga mengucapkan terima kasih kepada rekan-rekan di Laboratorium Biologi Molekuler Tanaman, Departemen Agronomi dan Hortikultura, Fakultas Pertanian IPB yang banyak memberikan masukan dan dukungannya dalam terselesaikannya penelitian ini.
Kepada motivator sejati, istri tercinta Almh.Rustie Marlina Kartikasari yang menemani dan memotivasi walaupun dalam kondisi sakit hingga akhir hayatnyadan anak-anakku Lalu Muhammad Fauzan, Lalu Farhan Hamizan, dan Baiq Farah Syafwa Kamiliyya, penulis ucapkan terima kasih karena selalu memberi dorongan semangat dan doa. Khusus kepada Ibunda tercinta Almh. Sutati Wiyani dan Ayahanda tercinta Lalu Muchdin penulis haturkan doa dan terima kasih karena cita-cita beliau menjadi semangat kepada penulis untuk terus maju walaupun jenjang yang dicapai saat ini tidak pernah terbayangkan beliau. Penulis menyadari sepenuhnya bahwa tesis ini masih jauh dari sempurna, namun demikian penulis berharap semoga tesis ini bermanfaat kepada pembaca dan semua pihak yang membutuhkannya.
Bogor, September 2016
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
1 PENDAHULUAN 1
1.1 Latar Belakang 1
1.2 Tujuan Penelitian 2
1.3 Hipotesis Penelitian 2
1.4 Manfaat Penelitian 2
1.5 Ruang Lingkup Penelitian 2
2 TINJAUAN PUSTAKA 4
2.1 Asal Tanaman Kelapa Sawit 4
2.2 Botani dan Morfologi Kelapa Sawit 5
2.3 Syarat Tumbuh 7
2.4 Metoda Seleksi Pemuliaan Kelapa Sawit 7
2.5 Penggunaan Marka Molekular dalam Pemuliaan Kelapa Sawit 10
2.6 Metoda Parentage analysis 11
2.7 Penggunaan Penanda Molekuler Simple Sequence Repeat
(SSR/Microsatelit) dengan metoda Mutiplex PCR 13
3 GENETIC DIVERSITY ANALYSIS BASED ON SSRMARKERS OF TXT CROSS AND DURA SELF OIL PALM (Elaeis guineensis Jacq.) PARENTAL
POPULATIONS ORIGINATEDFROM CAMEROON 15
Abstract 15
3.1 Introduction 15
3.2 Materials And Methods 17
3.3Results Ans Discussion 20
3.4 Discussions 32
3.5 Conclusion 34
4 ANALISIS KERAGAMAN GENETIK DAN KEBENARAN TETUA POPULASI DURA(D x D) A125 DAN A140, POPULASI TENERA (TX T) B57 DAN PERSILANGAN DURADELI X (LA ME X SP 540T) 36
Abstrak 36
4.1 Pendahuluan 36
4.2 Bahan dan Metode 38
4.3 Hasil Dan Pembahasan 40
4.4 Simpulan 50
5 ANALISIS KERAGAMAN GENETIK DAN KEBENARAN TETUA POPULASI DURA A125 DAN A140 (D X D)POPULASI TENERA B01,
Abstrak 51
5.1 Pendahuluan 51
5.2Bahan dan Metode 53
5.3 Hasil Dan Pembahasan 55
5.4 Simpulan 63
6 PEMBAHASAN UMUM 65
7 SIMPULAN DAN SARAN 66
7.1 Simpulan 66
7.2 Saran 66
DAFTAR PUSTAKA 67
DAFTAR TABEL
1. List of the type of oil palm populations evaluated in this research consisting of individuals derived from Tenera/Pisifera Crosses with La Me and La Me x SP540 genetic background and Dura self 18 2. Sixteen SSR marker loci, developed by CIRAD and available through
TropGENE Database (http://tropgenedb.cirad.fr/) 18 3. The observed number of alleles for each SSR marker locus among loci in
the evaluated oil palm populations 21
4. Expected allele frequencies based on Mendel genetics model crosses of
two parents 21
5. Loci having number of alleles per locus and ratio of allele frequencies that are fit or unfit to the expected based on the model crosses for each
studied oil palm population 22
6. The polymorphic information content (PIC) for each SSR marker locus
among evaluated oil palm populations 24
7. The mean values of allele per locus, effective allele (Ne), private allele, locally common alleles presence in < 50% of populations, and polymorphic information content (PIC) over loci for each oil palm
population 25
8. Allele classification according to allelic frequencies based on the approach of Buchert et al. (1997) (high, intermediate, low and rare) and
Marshall and Brown (1975) (common and rare) 26
9. The mean values of genetic diversity parameters over loci for each
studied oil palm population 27
10. Summary results of analysis of molecular variance (AMOVA) using input as allelic distance matrix for F-statistics analysis. 28 11. Daftar populasi kelapa sawit yang dievaluasi dalam penelitian 38 12. The observed number of allele for each SSR marker locus among the
evaluated oil palm populations 41
13. Kesesuaian jumlah lokus alel-1 dan rasio frekuensi alel 41 14. Nilai PIC (Polymorphic Information Content) pada setiap lokus marka
SSR 43
15. Klasifikasi alel berdasarkan frekuensi alel menggunakan pendekatan Buchert et al. (1997) (Tinggi, intermediet, rendah dan langka) dan
Marshall dan Brown (1975) (umum dan langka) 44
16. Tingkat heterozigositas dan nilai Polymorphic Information Content (PIC) pada setiap Lokus yang melibatkan induk Dura menunjukkan primer yang digunakan memiliki tingkat PIC yang cukup tinggi (rerata
0.616) 46
17. Tabel AMOVA (Analisis Ragam Molekular) 46
18. Daftar populasi kelapa sawit yang dievaluasi dalam penelitian 53 19. Jumlah alel pada setiap marka SSR pada populasi kelapa sawit yang
diuji 56
20. Kesesuaian jumlah alel lokus-1 dan rasio alel frekuensi fit atau tidak fit sesuai yang diharapkan berdasarkan model persilangan untuk setiap
21. Nilai PIC (Polymorphic Information Content) pada setiap lokus marka
SSR 58
22. Klasifikasi alel berdasarkan frekuensi alel menggunakan pendekatan Buchert et al. (1997) (Tinggi, intermediet, rendah dan langka) dan
Marshall dan Brown (1975) (umum dan langka) 59
23. Parameter keragamatan genetik pada populasi kelapa sawit berdasarkan nilai heterozigositas pengamatan (Hobs),aheterozigositas harapan
(Hexp) dan nilai F Null 60
24. Tabel Analisis Ragam molekular 60
DAFTAR GAMBAR
1. Tipe buah Dura yang memiliki cangkang tebal 6
2. Tipe buah pisifera yang tidak memiliki cangkang 6
3. Tipe buah Tenera yang memiliki cangkang tipis 7
4. Diagram program seleksi Reciprocal Recurrent Selection . (Digambar ulang dari Baudouin et al., 1997 dalam Corley dan Tinker. 2003 ) 8 5. Bagan penyebab terjadinya Illegitimasi pada persilangan pemuliaan
kelapa sawit (Hama-Ali et al., 2015) 13
6. The most likely pedigree construction from COLONY. Identified parent IDs on the top bar and offspring IDs at the bottom. Red lines emanate downwards from male parents and yellow lines from females 23 7. Six oil palm populations inferred from the STRUCTURE analysis. The
vertical coordinate of each subgroup indicates the membership coefficients for each individual, and the digits on the horizontal
coordinate represent the group IDs in Table 1 29
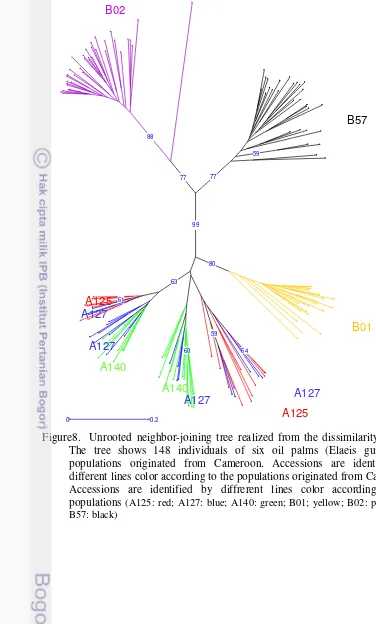
8. Unrooted neighbor-joining tree realized from the dissimilarity matrix. The tree shows 148 individuals of six oil palms (Elaeis guineensis) populations originated from Cameroon. Accessions are identified by different lines color according to the populations originated from Cameroon. Accessions are identified by diffrerent lines color according to the populations (A125: red; A127: blue; A140: green; B01; yellow;
B02: purple and B57: black) 31
9. PCoA of 148 individuals in the six oil palm (E. guineensis) populations for the first two dimensions. It shows the conformation of the six populations into different groups (B01, B02, B57 and A122, A127, and A150). Accessions are identified by different lines color according to the populations (A125: red; A127: blue; A140: green; B01; yellow;
B02:purple and B57: black). 32
10. Kontruksi pedigree dari COLONY. Identitas ID tetua pad bagian atas dan ID keturunannya pada bagian bwah. Garis merah menunjukkan tetua jantan dan garis kuning menunjukkan tetua betina 42 11. Analisis Structure memperlihatkan terbentuknya dua grup populasi yaitu
(1) B57 dan C22 berwarna merah, dan (2) A140 dan A125 berwarna hijau 47
12. Berdasarkan pendekatan metode Evanno dari 4 populasi yang diamati
13. Pohon filogenetik empat populasi berdasarkan metoda Tree Construction-Neighnor Joining menunjukkan terbentuknya 3 grup populasi. Tiap populasi dibedakan berdasarkan warna (populasi A125, hitam; A140, coklat; C22, biru muda; dan B57, ungu) 49 14. Sebaran sampel berdasarkan Principal Coordinat Analisis (PCoA)
menunjukkan bahwa 4 populasi yang dianalisis mengelompok menjadi 4 grup. Grup 1 terdiri dari populasi Dura (A140 dan A125) , sedangkan populasi B57 (TxT/P) dan C22 (DxT) membentuk grup yang berbeda. Populasi C22 membentuk dua grup dengan jarak genetik yang berbeda 50 15. Kontruksi pedigree dari COLONY. Identitas ID tetua pada bagian atas
dan ID keturunannya pada bagian bawah. Garis merah menunjukkan tetua jantan dan garis kuning menunjukkan tetua betina 57 16. Analisis Structure memperlihatkan terbentuknya dua grup populasi yaitu
(1) B01, C72, A140, A125 berwarna hijau dan B02 berwarna merah 61 17. Pohon filogenetik empat populasi berdasarkan metoda Tree
Construction-Neighnor Joining menunjukkan terbentuknya 4 grup populasi. Grup 1 yaitu populasi B02 (merah); grup 2, populasi B01 (biru) ; grup 3, populasi dura A125 dan A140 ( hitam dan coklat); grup
4, populasi C72 (hijau) 62
18. Sebaran sampel berdasarkan Principal Coordinat Analisis (PCoA) menunjukkan bahwa 5 populasi yang dianalisis mengelompok menjadi 3 grup. Grup 1 terdiri dari populasi Dura (A140 dan A125) dan populasi B01 (T x T), grup 2 adalah populasi C72 (D x P) dan grup 3 adalah
1
PENDAHULUAN
1.1 Latar Belakang
Kelapa sawit merupakan komoditas penghasil minyak nabati paling paling produktif di dunia. Per hektar luas tanam, kelapa sawit mampu menghasilkan minyak 3 s.d 8 kali lebih tinggi dibandingkan dengan tanamam penghasil minyak nabati lainnya (Corley, 2009). Sebagai gambaran pada tahun 2012, 56.2 juta ton minyak kelapa sawit dihasilkan dari 17.24 juta ha. Tapi bandingkan dengan ekstrak minyak rapeseed yang hanya mampu menghasilkan 23.6 juta ton minyak dari 36.4 juta ha(Barcelos et al., 2015).Bahkan menurut Corley (2009), kelapa sawit mampu memenuhi seluruh kebutuhan minyak nabati dunia sebesar 240 juta ton pada tahun 2050.
Berdasarkan data Ditjenbun (2014) luas lahan kelapa sawit mengalami peningkatan pesat dari tahun ke tahun. Pada tahun 2004 luas perkebunan kelapa sawit mencapai 5,7755,28 ha, namun pada tahun 2014 terjadi peningkatan luas lahan hampir dua kali lipat menjadi 10.956.231 ha. Namun saat ini dengan adanya keterbatasan kesesuaian lahan bagi tanaman kelapa sawit dan adanya moratorium alih fungsi lahan dari hutan ke perkebunan, menyebabkan luasan perkebunan kelapa sawit sudah sulit lagi untuk ditingkatkan. Peningkatan produktivitas kelapa sawit harus lebih mengandalkan efisiensi dan intensifikasi lahan dengan penggunaan bahan tanaman unggul berkualitas. Untuk menghasilkan bahan tanaman unggul dibutuhkan ketersediaan plasma nutfah yang memadai. Keberhasilan program pemuliaan sangat ditentukan oleh ketersediaan plasma nutfah yang memadai dan bagaimana penanganan proses seleksi hingga diperoleh tanaman kelapa sawit yang memiliki kualitas unggul (Arias et al. 2012). Menyadari hal tersebut, dalam upaya menghasilkan varietas unggul kelapa sawit maka PT. Astra Agro Lestari Tbk, bekerjasama dengan IRAD (The Institute of Agricultural Research for Development) Kamerun, telah melakukan persilangan terkontrol dan mengintroduksi plasma nutfah advanced genetic material Kamerun ke Indonesia yang dilakukan dalam kurun waktu 3 tahun mulai 2008 sampai 2011.
Namun demikian, upaya perbaikan genetik seringkali terkendala oleh adanya kontaminasi persilangan yang disebabkan oleh beberapa faktor. Faktor-faktor yang memungkinkan terjadinya kontaminiasi penyerbukan pada tanaman kelapa sawit antara lain yaitu faktor biologi tanaman seperti terjadinya bunga hermaprodit, akibat kesalahan manusia saat koleksi polen dan juga faktor eksternal lainnya seperti kerusakan kantong isolasi akibat gigitan serangga maupun kesalahan dalam pelaksanaan isolasi bunga (Thongthawee, et al. 2010; Hama-Ali et al. 2015).
2
(2005) yang menggunakan marka SSR di dalam melakukan fingerprinting pada tanaman kelapa sawit. Selain itu penggunaan marka SSR sangat bermanfaat dalam melakukan evaluasi keragaman genetik. Evaluasi keragaman genetik berdasarkan morfologi tanaman tidak lagi cukup karena rendahnya tingkat polimorfisme, lamanya fase, dan adanya pengaruh lingkungan yang besar (Okoye et al.2016). Selain itu juga penggunaan marka molukuler mampu mengurangi waktu program pemuliaan dan hasil yang lebih presisi.
1.2 Tujuan Penelitian
Tujuan Penelitian ini adalah :
1. Mendeteksi terjadinya illegitimasi persilangan material introduksi Kamerun 2. Memperoleh informasi keragaman genetik yang dimiliki material genetik origin
(asal) Kamerun
3. Mengetahui hubungan kekerabatan antar populasi 1.3 Hipotesis Penelitian
Hipotesis :
1. Marka molekular SSR mampu mendeteksi kebenaran tetua populasi genetik asal Kamerun
2. Terdapat keragaman genetik antar genotipe yang berasal dari Kamerun 3. Dapat diketahui hubungan kekerabatan antar populasi
1.4 Manfaat Penelitian
Penelitian ini memberikan informasi yang sangat bermanfaat didalam mendeteksi kemurnian dari benih hasil persilangan yang telah dilakukan. Hal tersebut memperkecil terjadinya kesalahan didalam program pemuliaan kelapa sawit kedepan. Dengan diperolehnya informasi mengenai keragaman genetik antar populasi dan hubungan kekerabatan antar populasi, memudahkan pemulia didalam menentukan rencana persilangan program pemuliaan berikutnya.
1.5 Ruang Lingkup Penelitian
3 Bagan Alir Penelitian
Analisis Kebenaran Tetua dan Keragaman Genetik Kelapa Sawit asal Kamerun Menggunakan Metode Parentage Analysis
Isolasi DNA Extractor; Centrifus; inkubator;
Pengambilan Sampel Daun (Tetua & Offspringnya)
PCR
SSR Fragment analysis
Analisis data(GenAlex, Cervus, Colony,Structure, Darwin)
Analisis Keragaman Genetik Identifikasi kebenaran Tetua
4
2
TINJAUAN PUSTAKA
2.1 Asal Tanaman Kelapa Sawit
Tanaman kelapa sawit ada dalam bentuk liar, semi liar, maupun dibudidayakan di daerah equator tropis seperti di Afrika, Asia Tenggara, maupun di Amerika Tengah dan Selatan. Umumnya tanaman kelapa sawit telah di domestifikasi oleh manusia. Sejarah kelapa sawit ditemukan pertama kali di sebuah makam di Abydos, Mesir sekitar 3000 SM yaitu dengan ditemukannya lemak yang diperkirakan berasal dari kelapa sawit. Fosil polen mirip dengan polen kelapa sawit yang dipelihara saat ini ditemukan di Afrika Barat dari jaman Miocene dan dari lapisan yang lebih muda di delta Niger, serta bukti lingustik yang menyebutkan ditemukannya spesies pohon mirip kelapa sawit. Sedangkan di Amerika Selatan ditemukan kelapa sawit dengan jenis Elaeis oleifera.
Menurut Zeven (1967) origin (daerah asal) kelapa sawit terletak di Guinea Afrika Tengah yang menyebar ke timur dan selatan melalui Sierra Leone , Ghana dan negara-negara Afrika Tengah lainnya. Hasil ini diperkuat oleh hasil analisis molekular yang dilakukan Zulkifli et.al.(2012) yang menunjukkan kedekatan genetik dari plasma nutfah dari negara-negara tersebut.
Sedangkan di Benua Amerika, tanaman kelapa sawit utamanya ditemukan di Brasil. Di Brasil, kelapa sawit dapat ditemukan tumbuh secara liar atau setengah liar di sepanjang tepi sungai, khususnya di sungai Amazon. Tanaman kelapa sawit yang termasuk dalam subfamili Cocoideae merupakan tanaman asli Amerika Selatan, termasuk spesies E.oleifera dan E. odora. Walaupun demikian, salah satu subfamili Cocoideae adalah tanaman asli Afrika. Zeven (1965) memastikan asal E.quineensis berdasarkan hasil deskripsi para ahli botani sebelumnya dan para penjajah di benua Afrika. Nama-nama kelapa sawit dalam bahasa deerah di kedua sisi lautan Atlantik juga mengacu pada nama Afrika.
Secara linguistik, nama kelapa sawit di Suriname merupakan perubahan dari kata dari Afrika seperti Yoruba, Fanti Twi dan Kokongo. Sementara di Brasil, Dnede kemungkinan merupakan perubahan dari kata Ndende di Kimbudu, Angola. Selain itu juga diperkuat dengan adanya penemuan fosil tepung sari dari kala Miosen di delta Nigeria yang bentuknya sangat mirip dengan tepung sari kelapa sawit. Spesies-spesies liar yang ada di Amerika diasumsikan keluar dari Afrika mengikuti perjalanan manusia pada masa prasejarah.
Perkembangan industri kelapa sawit menurut Hartley (1988), dimulai pada abad ke-19 yaitu dimulainya ekspor minyak dan inti sawit dari Afrika. Pada masa itu, sumber minyak kelapa sawit berasal dari tanaman kelapa sawit yang tumbuh liar dengan cara yang masih sangat sederhana. Setelah kelapa sawit memiliki nilai ekonomi yang baik bagi masyarakat, barulah kemudian tanaman kelapa sawit dibudidayakan dan berkembang menjadi perkebunan rakyat.
5 menyebutkan bahwa terdapat empat bibit kelapa sawit yang ditanam di Buitenzorg Botanical Garden (Kebun Raya Bogor) pada tahun 1848. Dari empat bibit tersebut, dua bibit diintroduksi dari Bourbon atau Mauritius pada Februari 1848 oleh D.T Pryce.
2.2 Botani dan Morfologi Kelapa Sawit
Kelapa sawit yang memiliki nama latin Elaeis gueneensis Jacq.berasal kata Elaion yang berarti minyak dalam bahasa Yunani, guineensis yaitu nama asal kelapa sawit ditemukan di Guinea (pantai barat Afrika), dan Jacq yang merupakan nama Botanis Amerika Jacquin. Saat ini terdapat tiga spesies kelapa sawit yaitu E.quineensis yang berasal dari Afrika dan E. Oleifera yang berasal dari Amerika latin. Spesies ketiga dikenal dengan Barcella odora yang sebelumnya bernama E.odora oleh Wessels-Boeer (1965) dalam Corley (2005). Mengapa dibedakan dari spesies Elaeis lainnya karena sifatnya yang berbunga secara bisexual. Namun berdasarkan hasil analisis marka molekular, B. odora
masih termasuk genus Elaeis (Barcelos et.al., 2002). Tanaman kelapa sawit klaifikasikan sebagai berikut :
Divisi : Embryophyta Siphonagama
Kelas : Angiospermae
Ordo : Monocotyledonae
Famili : Arecaceae
Sub Famili : Cocoideae
Egenus : Elaeis
Spesies :E. guineensis Jacq., E. oleifera (H.B.K.) Cortes, E. odora
Tanaman kelapa sawit tergolong tanaman monokotil. Batang tegak tidak bercabang, berdiameter 40 sampai 75 cm dengan tinggi batang sekitar 15 sampai 18 m. Daun majemuk dengan pelepah daun tersusun melingkari batang berbentuk spiral. Panjang pelepah daun mencapai 9 m dan panjang helaian daun mencapai 1.2 m dengan jumlah 100 sampai 160 pasang. Untuk perkebunan kelapa sawit, jumlah pelepah daun yang dipertahankan sekitar 30 sampai 50 pelepah (Hartley, 1988). Akarnya terdiri atas akar primer, sekunder, tersier dan kuarter yang merupakan akar serabut yang sebagian besar berada dekat permukaan tanaman dengan kedalaman 15 sampai 30 cm.
Tipe pembungaan kelapa sawit monoecious, yaitu bunga jantan dan bunga betina berada pada satu tanaman, tetapi pada tandan yang berbeda. Namun terkadang dijumpai juga bunga hermaprodit pada tanaman kelapa sawit muda yang berumur sekitar 2 sampai 4 tahun, atau pada tanaman induk yang mengalami beberapa kali persilangan sendiri (selfing). Bunga ini umumnya akan berkurang sejalan bertambahnya umur tanaman. Bunga kelapa sawit akan muncul pada setiap ketiak pelepah kecuali bila bunga telah aborsi pada fase pembungaan yang sangat muda. Akibat waktu berbunga yang berbeda, sehingga memungkinkan terjadinya penyerbukan silang antar tanaman kelapa sawit sangat tinggi. Bahkan hampir bisa dikatakan tidak ada persilangan sendiri yang terjadi secara alami.
6
pada perikarp (kulit buah) dan mesokarp (daging buah), sedangkan pada inti mengandung 4 sampai 6% minyak (Hartley, 1988).
Berdasarkan ketebalan cangkangnya, kelapa sawit dibedakan menjadi tipe dura, tenera dan pisifera dengan kriteria sebagai berikut:
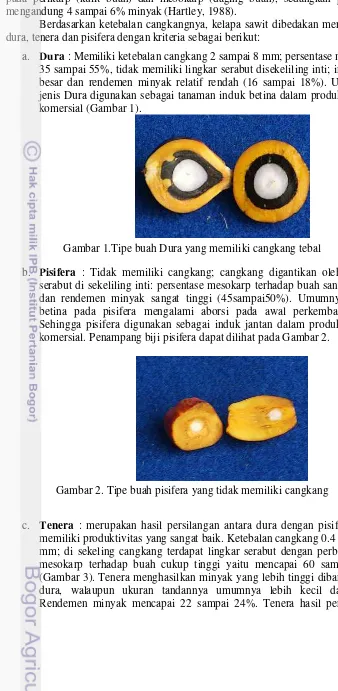
a. Dura : Memiliki ketebalan cangkang 2 sampai 8 mm; persentase mesokarp 35 sampai 55%, tidak memiliki lingkar serabut disekeliling inti; inti relatif besar dan rendemen minyak relatif rendah (16 sampai 18%). Umumnya jenis Dura digunakan sebagai tanaman induk betina dalam produksi benih komersial (Gambar 1).
Gambar 1.Tipe buah Dura yang memiliki cangkang tebal
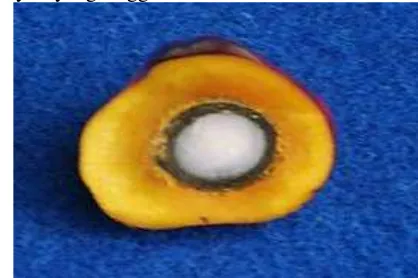
b. Pisifera : Tidak memiliki cangkang; cangkang digantikan oleh lingkar serabut di sekeliling inti: persentase mesokarp terhadap buah sangat besar dan rendemen minyak sangat tinggi (45sampai50%). Umumnya bunga betina pada pisifera mengalami aborsi pada awal perkembangannya. Sehingga pisifera digunakan sebagai induk jantan dalam produksi benih komersial. Penampang biji pisifera dapat dilihat pada Gambar 2.
Gambar 2. Tipe buah pisifera yang tidak memiliki cangkang
7 dura dan pisifera terseleksi dibudidayakan secara komersial karena menghasilkan minyak yang tinggi.
Gambar 3. Tipe buah Tenera yang memiliki cangkang tipis
Sifat ketebalan cangkang pada masing masing tipe kelapa sawit dikendalikan oleh satu lokus gen tetua (monogenik) dengan dua alel (Sh+ Sh-) yang berekpresi kodominan. Secara teoritis bila pohon dura (Sh+ Sh+) disilangkan dengan pohon tenera (Sh+ Sh-) maka dalam proses reproduksinya, pohon dura akan menyumbangkan satu jenis gamet (Sh+) sedangkan pohon tenera menyumbangkan dua jenis gamet (Sh+ dan Sh-). Dalam proses penyerbukan dan pembuahan, gamet dari masing-masing tetua akan berpadu bebas sehingga pada turunannya akan terbentuk 50% tipe dura dan 50% tipe tenera. Tetapi variasi ketebalan cangkang yang terlihat pada masing-masing tipe disebabkan oleh perbedaan perkembangan lignifikasi cangkang yang diwariskan secara kuantitatif dan dikendalikan oleh banyak gen (Corley et al. 1978). Tenera lebih disukai untuk digunakan sebagai bahan tanaman komersial karena mempunyai proporsi kandungan minyak di dalam mesokarpnya 30% lebih besar dibandingkan jenis dura. Untuk mendapatkan 100% tenera (DxP), maka tetua betina tipe dura (DxD) disilangkan dengan tetua jantan tipe pisifera hasil persilangan Tenera x Pisifera (TxP).
2.3 Syarat Tumbuh
Kelapa sawit dapat tumbuh dengan baik pada daerah tropika basah di katulistiwa, dengan ketinggian (elevasi) 0 sampai 500 m dari permukaan laut (dpl) pada suhu optimal 24 sampai 28oC. Namun tanaman kelapa sawit masih dapat tumbuh pada suhu minimal 8 oC hingga maksimal 38oC. Jumlah curah hujan yang optimum untuk tanaman kelapa sawit adalah 2000 sampai 2500 mm/tahun, tidak memiliki defisit air, dengan curah hujan yang merata sepanjang tahun. Defisit air tidak boleh mencapai 250 mm. Jumlah curah hujan yang melebihi 2500 mm/tahun juga kurang baik bagi kelapa sawit. Namun jumlah curah hujan diatas 2500 mm/tahun akan tidak menyebabkan gangguan bagi kelapa sawit bila jumlah hari hujan tidak lebih dari 180 hari.
2.4 Metoda Seleksi Pemuliaan Kelapa Sawit
8
toleransi terhadap stres biotik maupun abiotik, dan memiliki karakter morfologi yang bermanfaat seperti tangkai buah yang panjang yang memudahkan didalam pemanenan (NHCU, 2015). Skema seleksi yang umum dilakukan oleh lembaga penelian kelapa sawit yaitu menggunakan metoda Reciproal Recurrent Selection(RRS), metoda Family and Individuals Palm Selection (FIPS), metode silang balik (Back Cross), biak sel dan jaringan, serta MAS (marker assisted selection)(Zulhermana, 2009).
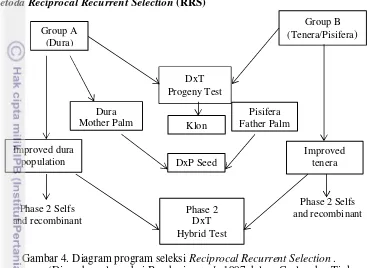
Metoda Reciprocal Recurrent Selection (RRS)
Metoda RRS merupakan metoda yang dikembangkan oleh Comstock dan Robinson (1948) pada pemuliaan tanaman jagung. Tujuan dari metoda RRS yaitu untuk meningkatkan alel-alel bermanfaat, mempertahankan keragaman genetik dan mengeksploitasi heterosis. Sejalan dengan temuan dua gugus heterotik pada kelapa sawit, dura dan pisifera, Gascon et al.(1989) menyarankan penggunaan metoda RRS untuk memperbaiki produktivitas minyak tanaman kelapa sawit. Efektivitas program RRS untuk mengeksploitasi heterosis pada kelapa sawit telah dibuktikan oleh Gascon et al. (1989) pada percobaan pengujian di Pantai Gading yang menunjukkan bahwa hasil siklus ke-1 RRS yang dilakukan mampu meningkatkan produktivitas minyak sebesar 18%. Pada akhir siklus ke-2 RRS, produktivitas minyak meningkat hingga 36%, relatif terhadap rataan produktivitas minyak pada populasi dasar DxP pra-RRS. Penerapan strategi RRS menurut Asmono et al(2005) melibatkan dua heterotik group yaitu, Group A yang mencakup materi tetua betina dura dan Group B tetua jantan, pisifera dari famili tenera/pisifera. Dari populasi dasar yang telah diseleksi dilakukan suatu tahapan evaluasi untuk menganalisis dan menentukan individu tanaman terbaik yang
Group A (Dura)
Group B (Tenera/Pisifera)
DxT Progeny Test
Klon Dura
Mother Palm
Pisifera Father Palm
Improved dura population
Improved tenera population DxP Seed
Phase 2 Selfs and recombinant
Phase 2 Selfs and recombinant Phase 2
DxT Hybrid Test
9 dilihat berdasarkan hasil uji keragaan keturunannya. Berdasarkan hasil pengujian tersebut, maka akan diketahui kemampuan dari daya gabung umum (GCA) maupun daya gabung khusus (SCA) dari masing-masing persilangan yang diuji. Uji keturunan akan memperlihatkan kemungkinan potensi dan daya gabung dari tetua-tetua yang digunakan. Berdasarkan hasil pengujian tersebut akan diperoleh kandidat-kandidat tetua potensial yang digunakan untuk siklus seleksi selanjutnya dan digunakan sebagai tanaman indukan untuk menghasilkan benih kebutuhan komersial.
Metoda Family and Individual Palm Selection (FIPS)
Metoda FIPS menerapkan strategi seleksi terhadap famili dan individu. Menurut Asmono et al(2005) seleksi dan rekombinasi dilakukan pada famili dura, sedangkan untuk pengujian dura-dura tersebut disilangkan dengan tester berupa pisifera unggul. Tujuan utama dari penggunaan FIPS adalah untuk memperbaiki produksi CPO. Prosedur seleksi ini juga dilakukan untuk memperbaiki sifat sekunder, seperti pertumbuhan meninggi yang lambat. Keberadaan varietas yang mengandung CPO tinggi dan mempunyai pertumbuhan meninggi yang lambat diharapkan dapat meningkatkan nilai ekonomi dari tanaman.
Metoda Backcross (Silang Balik)
Metoda silang balikbertujuan untuk memperbaiki suatu sifat pada suatu kultivar yang mengalami kekurangan. Pengertian backcross yaitu pengulangan persilangan dari progeni suatu hibrida ke salah satu tetuanya (Fehr, 1987). Tetua yang berkontribusi terhadap gen-gen yang mengontrol satu sifat yang diharapkan disebut tetua donor (Nonrecurrent). Tetua donor digunakan hanya sekali persilangan dan tidak diulang kembali. Sedangkan tetua dimana gen-gen tersebut ditransfer disebut recurrent parent. Recurrent parent mengidentifikasikan bahwa tetua tersebut digunakan berulang-ulang di dalam prosedur silang balik.
Pada tanaman kelapa sawit, species yang umum digunakan dalam budidaya adalah E. guineensis. Hal tersebut disebabkan karena produktivitas tanamannya yang tinggi seperti kandungan CPO dan PKO yang tinggi. Namun spesies ini memiliki kelemahan antara lain kandungan asam lemak tak jenuh (ALTJ) yang rendah (40-60%) dan pertumbuhan vegetatif yang cepat meninggi. Sedangkan E. oleifera beberapa keunggulan antara lain (1) laju pertumbuhan yang lambat sehingga memudahkan dalam pemanenan (Corley and Tinker, 2003); (2) Kandungan Asam lemak tak jenuh (ALTJ) yang tinggi yang mencapai70-80% (Montoyaetal,2014); (3) aktivitas lipase yang rendah pada mesokarp buah, memperpanjang waktu antara panen dan proses buah (Sambanthamurthi et al, 1995; Cadena et al., 2013); (4) kandungan vitamin A dan E yang tinggi, yang membuktikan nilai kandungan gizi yang tinggi (Rajanaidu et al, 2000) dan (5) kemampuan adaptasi yang luas terhadap lingkungan (Barcelos,2002). (6) relatif toleran terhadap beberapa penyakit (Corley and Tinker, 2003), termasuk bud-rot
10
E.guineensismelalui metoda pemuliaan silang balik. Tujuan utamanya adalah untuk menghasilkan kelapa sawit unggul yang memiliki produktivitas yang tinggi dengan kandungan CPO dan ALTJ yang tinggi. Menurut Corley dan Tinker (2003), hibrida antara E. oleifera. × E. guineensis (O x G) memiliki keunggulan karena pertumbuhannya lambat dan memiliki kandungan minyak desaturasi yang tinggi. Selain itu, hibrida O x G memiliki keunggulan dalam hal ketahanan terhadap fatal yellowing disease yang terdapat di Amerika Latin.
Perbanyakan Kultur Jaringan
Salah satu metoda peningkatan produktivitas kelapa sawit yaitu melalui perbanyakan kultur jaringan. Teknologi kultur jaringan merupakan teknologi yang mampu menghasilkan regenerasi jaringan tanaman secara cepat dalam jumlah yang banyak serta menghasilkan keturunan yang mampu berproduksi tinggi serta serupa dengan indukan asal jaringan dan seragam (Sleper dan Phoelman. 2006). Hasil pengamatan di lapang pada percobaan PPKS menunjukkan bahwa tanaman klon asal kultur jaringan mampu menghasilkan tandan buah segar (TBS) 30-40% lebih tinggi dari produksi TBS tanaman asal benih (Latief et al. 2003; Corley dan Tinker. 2003). Peningkatan produksi terjadi karena keseragaman tanaman klonal dan karena penggunaan pohon induk terpilih dari 5% terbaik populasi DxP hasil seleksi RRS. Permasalahan yang dihadapi dalam perbanyakan kultur jaringan adalah munculnya abnormalitas pembungaan dan pembuahan. Pada abnormalitas pembungaan, dihasilkan tanaman dengan bunga jantan 100% sehingga tidak dihasilkannya pembuahan. Sedangkan pada abnormalitas pembuahan, yaitu dengan dihasilkannya buah mantel.
Upaya perbanyakan kultur jaringan pada kelapa sawit dimulai pada tahun 1960-an dan pertengahan tahun 1970-an (Corley dan Tinker. 2003). Klonal pertama kali ditanam di Malaysia pada tahun 1977 (Plate VIIIB) dan pengulangan percobaan di tahun 1978 (Corley et al,1979). Mengikuti keberhasilan tersebut, secara cepat terjadi ekspansi dan dipertengahan tahun 1980-an setidaknya 10 laboratorium kultur jaringan di Malaysia telah dibuat dan beberapa dinegara lain.
11 Namun dengan ditemukannya marka molekular yang mampu mendeteksi genetik suatu organisme tertentu, mampu mempersingkat waktu dan sumber daya yang dibutuhkan. Penggunaan marka molekular dalam program pemuliaan tanaman dapat membantu memberikan informasi yang tepat di dalam mengetahui struktur genetik suatu populasi (Guesquière1985), menentukan identitas dan keragaman genetik, serta mengetahui gen yang terkait karakter tertentu sejak awal seleksi (Jack dan Mayes 1993; Shah et al. 1994). Selain itu penggunaan marka molekuler dapat juga digunakan untuk mendeteksi tetua dari suatu individu organisme maupun tanaman melalui metoda fingerprinting (Jeffrey, 1985) yang juga dikenal sebagai metoda parentage analysis.
2.6 Metoda Parentage analysis
Parentage analysis (Analisis Tetua) adalah pondasi dasar penelitian ekologi molekular. Pola tetua memainkan peranan penting di dalam studi mengenai perbedaan ekologi dan evolusi seperti seleksi tetua (Yezerinac et al.
1995; Jones & Avise 1997), pola sebaran dan rekruitment (Dow & Ashley 1996; Haredesty et al . 2006), estimasi dari parameter genetik kuantitatif (Kruuk et al.
2000: Garant & Kruuk 2005) dan konservasi biologi (Haig 1998; Planes et al .
2009). Namun demikian parentage analysis termasuk disiplin ilmu yang masih baru. Revolusi ekologi molekular dimulai pada akhir 1960 dengan penemuan allozyme elekctrophoresis (Hubby & Lewontin 1966). Pada awal molekular ekologi, analisis tetua hampir tidak mungkin dilaksanakan karena beberapa permasalahan (Ellstrand 1984; Gowaty & Karlin 1984), karena rendahnya level karakter polymorphisme berdasarkan marka protein. Studi mengenai tetua diawali dengan ditemukannya probe DNA yang dapat digunakan pada manusia dan beberapa organisme untuk mengetahui variasi pada lokus minisatelit. Teknik ini dikenal saat ini dengan “DNA finger printing” (Jefreys et al. 1985). Beberapa tahun setelah pengembangan DNA fingerprinting, ditemukan metoda marka microsatelite (Tautz 1989), yang lebih dikenal dengan simple sequence repeats, yang menjadi awal masa keemasan analisis tetua (parentage analysis).
Marka mikrosatelit adalah metoda yang paling tepat di dalam melakukan
parentage analysis karena mampu dengan mudah menguji lokus tunggal, codominant, dan berbagai macam marka (Avise 2004; Pemberton 2009). Meskipun multi lokus DNA fingerprinting metoda yang relatif sederhana, pola pitanya sangat sulit untuk ditangani secara statistik, sehingga parentage analisis harus dihitung dengan jumlah pita yang diharapkan dari pembagian antar kelas yang berbeda. Mikrosatelit dapat mengikuti pola penurunan Mendel dan bagian kunci dari teori statistik yang berhubungan dengan parentage analysis telah dikembangkan (Thompson 1975; Meagher 1986; Devlin et al . 1988).
Jones et al.(2010) mengelompokan teknik analisis parental kedalam empat kategori yaitu exclusion, categorical allocation, fractional allocation dan
12
terhadap progeninya, maka sampel parental tidak termasuk kedalam pertimbangan sebagai true parent (parental yang sebenarnya). Namun teknik ini memiliki beberapa kekurangan diantaranya:
1. Beberapa karakteristik penanda molekul dalam penurunan Mendelian bersifat ketat sehingga dapat memunculkan pengecualian yang salah terhadap true parents. Mikrosatelit sangat rentan mengalami permasalahan ini. Sehingga akan memunculkan kesalahan dalam scoring.
2. Pengecualian secara utuh akan sulit dilakukan terhadap populasi progeni dan parental dalam jumlah yang sangat besar.
Teknik categorical allocation dikembangkan untuk melengkapi teknik
exclusion yang sulit untuk melakukan pengecualian secara utuh (Meagher & Thomson, 1986). Salah satu kelebihan categorical allocation ialah adanya metode untuk memilih parental tunggal yang paling mirip (most likely) dari grup yang diduga sebagai parental. Hal ini berdasarkan dari observasi yang menunjukan bahwa parental dengan genotip yang berbeda kemungkinan menghasilkan probabilitas genotip progeni yang berbeda (Meagher & Thompson, 1986). Pada umumnya teknik ini menggunakan pendekatan likelihood atau pendekatan Bayesian (Marshall et al., 1998).
Dasar pemikiran dari pendekatan likelihood ini dikembangkan oleh Meagher & Thompson (1986) dan Marshall et al (1998). Categorical allocation
dapat diaplikasikan dengan satu parental yang diketahui atau kedua parentalnya diketahui. Keduanya menggunakan persamaan likelihood yang berbeda (Marshall et al., 1998). Nilai LOD dengan pendekatan likelihood merupakan nilai logaritma dari rasio multilokus likelihood (Meagher & Thomson, 1986).
Perangkat lunak Cervus(Marshall et al., 1998) merupakan salah satu perangkat lunak yang dapat digunakan untuk analisis parental. Pendekatan sederhana yang digunakan ialah persamaan likelihood (kemiripan) menggunakan metode exclusion dan categorical allocation. Kandidat genotipe indukan akan dibandingkan dengan genotipe progeni dan akan dilakukan pengecualian jika terdapat genotipe indukan yang tidak sesuai pada satu atau lebih lokus. Penggunakan beberapa kandidat indukan dan lokus yang memiliki polimorfisme tinggi akanmenghasilkan hanya satu kandidat indukan tanpa pengecualian. Sistem
likelihood menggunakan informasi dari setiap lokus untuk memastikan frekuensi alel pada progeni berasal dari kandidat indukan dan mengidentifikasi kandidat indukan bersifat heterozigot atau homozigot (Marshall et al. 1998).
13
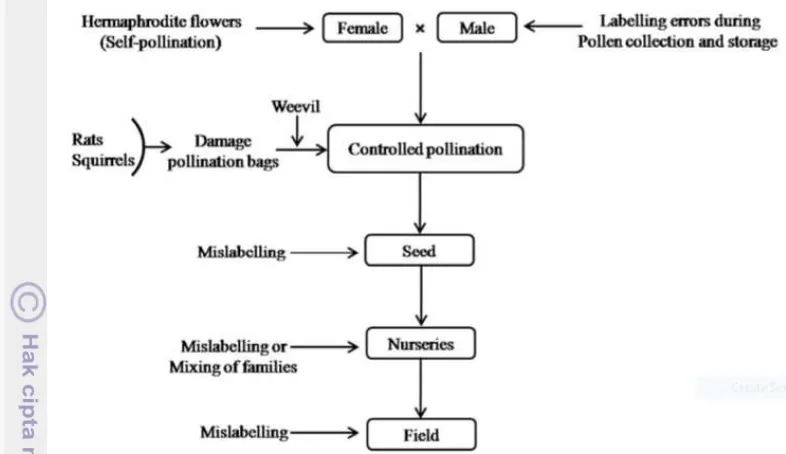
Gambar 5. Bagan penyebab terjadinya Illegitimasi pada persilangan pemuliaan kelapa sawit (Hama-Ali et al. 2015)
Aplikasi penggunaan parentage analisis dalam kegiatan pemuliaan kelapa sawit salah satunya adalah untuk mengetahui kebenaran tetua persilangan. Illegitimasi (kontaminasi persilangan) menjadi faktor yang tidak terlihat yang memiliki pengaruh negatif pada kegiataan persilangan. Menurut Hama-Ali (2015) Illegitimasi dapat terjadi pada berbagai fase persilangan, dari mulai fase inisiasi pembungaan, pelabelan tetua, sampai pelaksanaan penanaman bibit dilapangan (Gambar 5). Banyak faktor yang dapat menyebabkan terjadinya kontaminasi persilangan baik secara biologi tanamannya (seperti munculnya pembungaan hermaprodit), akibat kesalahan manusia saat pengambilan pollen dan faktor-faktor eksternal seperti rusaknya kantong polinasi (sungkup) yang disebabkan oleh serangga maupun tikus, serta terjadinya penyerbukan bebas oleh serangga penyerbuk Elaeidobius kamerunicus. Sebelum adanya teknologi berbasis molekular dan marka codominant seperti mikrosatelit, sangat sulit menentukan terjadinya kontaminasi persilangan. Para pemulia harus menunggu beberapa tahun sampai tanaman mengalami fase generatif, untuk mengecek terjadinya kontaminasi dengan cara yang tradisional dengan memeriksa ketebalan cangkang dan membandingkan pola segregasinya.
2.7 Penggunaan Penanda Molekuler Simple Sequence Repeat (SSR/Microsatelit) dengan metoda Mutiplex PCR
[image:30.612.118.511.88.315.2]14
Beberapa penulis mendefinisikan mikrosatelit sebagai pengulangan dari 2 sampai 8 bp, atau pengulangan dari1sampai 6 bp atau bahkan 1 sampai 5 bp. Mikrosatelit dihasilkan/berasal dari daerah yang merupakan ragam sederhana dari motif berulang sekuen DNA yang telah terwakili. Mekanisme mutasi dominan pada mikrosatelit merupakan fenomena 'tergelincirnya proses perpasangan untai DNA'. Proses terjadi ketika sintesa DNA dalam array mikrosatelit mengalami
slipped strand mispairing, yang berakibat pada bertambah atau berkurang satu, atau lebih sekuens unit berulang, bergantung pada apakah yang keluar adalah dari rantai DNA loop yang baru disintesis atau rantai template-nya.
Reaksi PCR untuk SSR dijalankan dengan primer forward dan reverse
yang menempel pada ujung 5 `dan 3` dari masing-masing template DNA. Fragmen PCR biasanya dipisahkan pada gel poliakrilamida yang dikombinasikan dengan pewarnaan AgNO3, autoradiografi atau sistem deteksi fluoresens. Agarose gel (biasanya 3%) dengan EtBr juga dapat digunakan ketika perbedaan ukuran alel antara sampel lebih besar dari 10 bp. Namun penggunaan SSR dibatasi oleh lambatnya data yang dihasilkan dan terbataskan pasangan marka serta dibutuhkannya biaya yang tinggi untuk melakukan pengembangan marka. Namun demikian, perbaikan dibidang teknologi molekular terus dikembangkan seperti adanya metoda multiplex PCR untuk amplifikasi DNA.
Multipleks PCR yaitu metoda PCR yang menggunakan beberapa marka dalam satu reaksi. Metoda ini bertujuan meningkatkan jumlah informasi dari yang diperoleh per reaksi, dan untuk mengurangi pemakaian bahan dan biaya tenaga kerja. Kombinasi penggunaan bahan yang berbasis fluorescen (berpendar) mampu secara otomasis mendeteksi DNA dan ukuran fragment sehingga mampu mempercepat proses, hasil yang lebih akurat dan perolehan data dengan biaya yang lebih murah (Mitchell et al.1997). Namun pengembangan multiplex PCR pada tanaman seringkali mengalami kesulitan karena besarnya ukuran genom dan
polyploidy. Namun saat ini telah dikembangkan metoda baru yang dikenal dengan metoda “Multiplex ready PCR” yang dikembangkan oleh Hayden et al. (2008) yang menggabungkan teknologi berbasis fluorescens dengan penggunaan primer M-13 . Metoda ini menggunakan deteksi SSR yang berpendar dan analiser fragmen DNA otomatis untuk mengukur ukuran alel yang merupakan metoda analisis genotipe SSR yang paling cepat dan akurat (Ziegle et al. 1992). Prosedur ini berdasarkan pemisahan SSR yang berlabel oleh gel elektroforesis. Keuntungkan dari SSR yang berpendar adalah SSR dapat dipisahkan dalam gel (kapiler) berdasarkan ukuran fragmennya tanpa tumpang tindih satu sama lain. Pada kasus saat SSR memiliki ukuran alel yang sama akan terjadi tumpang tindih, maka pemisahan dapat dilakukan dengan melabel produk SSR dengan dye
15
3
GENETIC DIVERSITY ANALYSIS BASED ON
SSRMARKERS OF TXT CROSS AND DURA SELF OIL
PALM (
Elaeis guineensis
Jacq.) PARENTAL POPULATIONS
ORIGINATEDFROM CAMEROON
Abstract
The success of oil palm breeding activities depends on the availability of diverse germplasm parental populations, especially between the Dura and the Pisifera types. Therefore, evaluation of the potential Dura and Pisifera genetic diversity is neccessary. The objectives of this research are to analyze the T x T crosses and Dura self oil palm (Elaeis guineensis Jacq.) parental populations genetic diversity and evaluate their potential value for creating hybrid progenies in oil palm breeding program. A total of 148 individuals from three T x T crosses and three Dura self populations were evaluated. Genotyping was conducted using 16 SSR marker loci. DNA isolation and PCR amplifications for all of the SSR loci were conducted at the Biotechnology Laboratory, PT. Astra Agro Lestari Tbk. The genotype data were analyzed using software for population genetic and genetic diversity analysis. Results of the analysis indicated the T x T male parent populations were more diverse than the Dura self. The 16 evaluated microsatellite markers are either highly or moderately polymorphic based on their PIC values. Hence, they could be used for further analysis for larger number of Astra Agro Lestari’s T x T and Dura Self population samples. The results of clustering and PCA analysis showed the all populations are grouped into three groups consisting of (1) B02, (2) B57, and (3) the rest of populations (B01 and the three Dura Self populations). In the meantime, the third group is further divided into five subgroups, consisting of sub-group 1: the B01, and sub-group two to five comprised of a mix individuals from members of at least two different Dura self populations. All the studied T x T populations could potentially be used as improved male parents for producing future oil palm hybrid varieties. The T x T populations has a wider genetic distance than that of the D Self populations. Moreover, member of Dura self oil palm population should not be grouped based on the family but it should be based on results of clustering analysis. The reported data should be beneficial for aiding future oil palm breeding in Indonesia.
Keywords: Afican oil palm, Dura, Pisifera type oil palm, Population structure, Simple Sequence Repeat
3.1 Introduction
16
The 2014 data from Directorate General of Plantations, Republic of Indonesian (Ditjenbun, 2014) indicated there were rapid increases in oil palm plantation areas in Indonesia. The total areas of oil palm plantations in 2004 were only 5,284,723 ha while in 2012 were 10,956,231 ha, respectively. However, the Indonesian government through Presidential Instruction, Republic of Indonesia No 10/2011, has implemented a moratorium on forest to plantation land conversion since 2011 (Ditjetbun, 2013). Moreover, availability of arable land suitable for growing oil palm has also become a limiting factor for the opening of new oil palm plantation (Danielsen et al., 2009). Therefore, meeting the increasing future demand for palm oil has to come from more productive planting materials while those better yielding planting materials should come from effective oil palm breeding programs (Barcelos et al., 2015).
One of the critical success factors in generating more productive and superior quality oil palm planting material through breeding program is the availability of diverse oil palm breeding materials. Diverse oil palm breeding materials provide oil palm breeders raw materialsfor developing new and improved cultivars with desirable characteristics (Govindaraj et al., 2015).Therefore, understanding genetic diversity and population structure of the breeding materials is an important aspect of oil palm breeding(Thongthawee et al., 2010; Barcelos et al., 2015). Breeding for superior oil palm variety is generally conducted through reciprocal recurrent selection (RRS) method (Purba et al., 2000). In one generation of RRS, potential female (Dura type) and male (Pisifera type) parental lines are cross hybridized to generate D x P progenies. The D x P progenies are evaluated for their superior characters in the field. To generate improved parental lines, the Dura types are self-pollinated and the Pisifera types are generated through crossing of two Tenera types (T x T crosses)(NCHU et al.,2015). The cycle is continually repeated to eventually identify improved parental lines capable of producing the new superior D x P cultivars.
In RRS method, the existence of illegitimate progeny can happen in any stage of the controlled crosses in the oil palm breeding activities, from the initial stage of selecting and labeling parents, to the final stage of field evaluation trials. Many factors affect the percentage of illegitimacy or contamination in oil palm breeding such as the biology of the plant flower (i.e. the existence of a few hermaphrodite flowers), human errors during pollen collection and damage of the pollination bag for covering the flowers because of animals and the environment factors allowing insect pollinators to cross pollinate the female flowers (Hama-Ali et al., 2015). Improved parental lines in RSS oil palm breeding methods should not contains illegitimate progenies; therefore, identification of illegitimacy in the improved parental line populations is important. Moreover, new superior variety should only be obtained from hybridization of genetically diverse parental populations. Therefore, generating basic information about genetic diversity of parental populations is also necessary to identify the parental line combinations having the largest potential of hybrid vigor (Arias et al., 2012).
17 markers (Purba et al., 2000; Billotte et al., 2001, Lim and Rao, 2005; Zulhermana et al., 2010; Ajambang et al., 2012). Although they are informative for studying genetic diversity of oil palm, RAPD, ISSR and AFLP are dominant markers. Therefore, there are some limitations on the use of RAPD, ISSR and AFLP markers in genetic diversity because those markers are not able to differentiate the genotype of heterozygous and homozygous individuals (Okoye et al., 2016).
Simple-sequence repeats (SSRs), also known as microsatellites, are tandem repeats of two to four nucleotide motifs that are widely distributed in the eukaryotic genomes (Delseny et al., 1983; Tautz and Renz, 1984; Tautz, 1989; Billotte et al., 2001). The SSR markers derived from PCR amplification of SSR sequences have a high degree of allelic variability; therefore, it is a powerful tool for genetic analysis, germplasm identification and genetic mapping of intra- or interspecific of many plants including the genus Elaeis. The reasons SSR marker are preferable in plant genetic analysis include: (1) Its existence is abundant and uniformly distributed in the genome, (2) it is highly polymorphic and a co-dominant marker, (3) it is relatively easy and fast to generate by using PCR technique and relatively simple and easy to interpret the allele configurations, and (4) its relatively easily accessed by other laboratories if the SSR sequences are available (Saghai-Maroof et al., 1994). The SSR marker is a co-dominant marker; therefore, it can differentiate genotype of heterozygous and homozygous individuals (Akkaya et al., 1992; Jones et al., 2010). The SSR marker is also advantageous because of its high discriminative character which makes it capable of detecting polymorphism even among closely related members of a population (Ferreira and Grattapaglia, 1998; Mohammadi and Prasanna, 2003). It is also highly reproducible, relatively low cost and require low training level for its implementation. More importantly, SSR marker can be used to analyze large number of samplessince it is possible to automate genotyping using SSR markers (Hayden et al., 2008). The SSR marker has been used to evaluate oil palm genetic diversity and population structure of breeding materials and parental lines (Thongthawee et al., 2010; Okoye et al., 2016) and natural populations of African (E. guineensis) and American (E. oleifera) origin of oil palms (Ting et al., 2010).
To develop parental lines for generating new superior oil palm varieties, PT. Astra Agro Lestari Tbk. has set up collaborative activities withthe Institute of Agricultural Research for Development (IRAD) of Cameroon by introducing selected Dura self, T x T, and D x P crosses in the year 2008 – 2011 for evaluation under Indonesian environment conditions. Although they are generated through a comprehensive breeding program, the genetic background of those populations has never been evaluated using molecular tools. The objectives of this study are to analyze genetic diversity and determine the population structure of Dura self (D x D) and Tenera by Tenera/Pisifera crosses(T x T/P) of oil palm (E. guineensis
Jacq.) parental populations originated from Cameroon and evaluate their potential value for creating hybrid progenies in oil palm breeding program in Indonesia.
3.2 Materials And Methods
18
Pangkalan Bun, Central Kalimantan. Samples were collected from nursery and experimental area of PT. Astra Agro Lestari in Pangkalan Bun, Central of Kalimantan. A total of 148 individuals from six different populations were evaluated. The populations consisted of three populations of D x D and three populations of T x T/P (Table 1). The number of evaluated individuals ranged from 20 to 73 individuals per population. Leaf tissues were sampled from the spear leaf of three years old plants in the trial field station.
Table1. List of the type of oil palm populations evaluated in this research consisting of individuals derived from Tenera/Pisifera Crosses with La Me and La Me x SP540 genetic background and Dura self
Family Genetic background No. of
individuals Type
Female Male
B01 LM2T LM2T 20 TxP
B02 LM2T LM2T 36 TxP
B57 (LM5TxLM10T) X RS3T AF
(LM5TxLM10T)
X RS3T AF 29 TxT
A140 DA115D x DA3D DA115D x DA3D 23 D self
A125 DA10D x DA115D
DA10D x
DA115D 20 D self
A127 DA10D x DA3D DA10D x DA3D 20 D self
Note: D – Dura, P – Pisifera, and T – Tenera type of oil palm, D self – selfing of individual Dura palm, T x P – Tenera by Pisifera cross, and T x T – Tenera by Tenera cross.
3.2.1 DNA Extraction and SSR Marker Amplification
Total DNA was extracted from a total of 100 mg of leaf samples following standard protocols using commercial plant DNA extraction kit (Geneaid). RNA was removed from total nucleic acid by standard RNAse treatment (Sambrook et al., 1989). Quality and quantity of the extracted DNA were evaluated using Nanodrop ND-1000 spectrophotometer (Nano-Drop Technologies Inc.) and by electrophoresis of 2 l of sampled DNA in 1 % agarose gel. Sixteen SSR marker loci, developed by CIRAD and available through TropGENE Database (http://tropgenedb.cirad.fr/) were used to genotype all of the evaluated individuals. The list of primers used to generate 16 SSR marker loci are presented in Table 2.
19 sub-contracted to a third party genotyping provider (First Base Laboratories, Malaysia) and were done using the 96-capillary ABI Prism 3730 XL DNA Analyzer (Applied Biosystems Inc, USA) using GENESCAN-500 (Liz) size standard.
Table2Sixteen SSR marker loci, developed by CIRAD and available through TropGENE Database (http://tropgenedb.cirad.fr/)
3.2.2 Data Analysis
Allele scoring data provided by third party genotyping provider (First Base Laboratories, Malaysia) were organized for analysis using Microsoft Office Excel
Primer Chromo
some Primer sequence (5’- 3’) Motif
Allele size (bp)
Ann. temp
mEgCIR0802 1 F CTCCTTTCGGCGTATCCTTTA (GA)12 217 52ºC
R TACGTGCAGTGGGTTCTTTC
mEgCIR3282 2 F GTAACAGCATCCACACTAAC (GA)20 245 52ºC
R GCAGGACAGGAGTAATGAGT
mEgCIR0173 3 F TGAACAAGAAGGCGGAAAGAGA (GA)18 132 52ºC
R TGCGGGCGAGGAAAGGT
mEgCIR3533 4 F TCTATGGCTCTGTCGTGTAT (GA)18 139 52ºC
R CGAGCCGGTAGAAACTAT
mEgCIR2813 5 F GCTTTGTTGCAGTTTGACTA (GT)7
(GA)11 52ºC R GTTTAGGATGTTGCGTGAT
mEgCIR3543 6 F GTTCCCTGACCATCTTTGAG (GA)17 232 52ºC
R GTCGGCGATTGATTAGATTC
mEgCIR0894 7 F TGCTTCTTGTCCTTGATACA (GA)8ta
(GA)9 186 52ºC R CCACGTCTACGAAATGATAA
mEgCIR0886 8
F GATCTGCCGGTGCTCCTA
(GA)9 157 52ºC R CTCAGTTTAGTCGATCCTTCCATT
G
mEgCIR3886 9 F
TTCTAGGGTCTATCAAAGTCATA
AG (GA)5GT(GA)2
0
187 52ºC R AGCCACCACCACCATCTACT
mEgCIR3785 10 F AAGCAATATAGGTTCAGTTC (GA)21
D 284 52ºC
R TCATTTTCTAATTCCAAACAAG
mEgCIR3362 11 F CCCATCATCTGCTCAGGATAGAC (GA)19 151 52ºC R ACCCTCTCCTCTTGGGAAGA
mEgCIR2414 12 F CAATCATTGGCGAGAGA (GA)10 195 52ºC
R CGTCACCTTTCAGGATATG
mCnCIR0038 13 F CAAGTATATGTGTGTGTATGC (GA)8 113 52ºC
R ATCTGCTGTTGATTATGG
mEgCIR3546 14 F GCCTATCCCCTGAACTATCT (GA)15 286 52ºC
R TGCACATACCAGCAACAGAG
mEgCIR3292 15 F AGCCATGAGTGAATCATATC (GA)20 173 52ºC
R ACCACGATGTCAATCTCTAT
mEgCIR0353 16 F ATTTCGTAAGGTGGGTGT (GT)11(
20
and the appropriate data files were prepare for subsequent analysis using various software for genetic diversity, population parameter, and population structure analysis. Population genetic parameters were analyzed using CERVUS version 3.0 (Kalinowski et al., 2007) and GENALEX version 6.3 (Peakall and Smouse, 2012). Analysis of molecular variance (AMOVA) (Excoffier et al., 1992) using GENALEX version 6.3 (Peakall and Smouse, 2012) was done to calculate partition of total genetic variation between and within populations. Principal coordinat and Neighbor-Joining analysis were done using DARWIN Software version 6.0.013 (Perier and Jacquemoud-Collet, 2006) while the population structures were determined using the STRUCTURE software version 2.3.4 (Falush et al., 2003) and STRUCTURE HARVESTER version Web 0.6.94 (Earl et al., 2012). The approach by Evanno et al. (2005) was used to select the appropriate K clusters for the studied populations. To validate the presence of illegitimate individuals among the studied populations, the genotype data were subjected to analysis using COLONY software (Jones and Wang, 2010).
3.3Results Ans Discussion
3.3.1 Allele numbers in the evaluated oil palm population
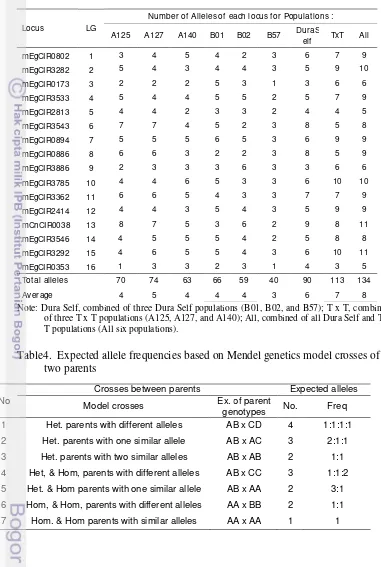
In the combined six populations (All population), the evaluated 16 loci of simple sequence repeat (SSR) markers generated a total of 134 alleles and the average of 8 alleles per locus (Table 2). In the combined all populations, both the mEgCIR0038 (LG 13) and mEgCIR3292 (LG 15) SSR primer pairs generated the highest number of alleles/locus (11 alleles) (Table 2). On the other hand, both the mEgCIR2813 (LG 5) and mEgCIR0353 (LG 16) SSR primer pairs generated the lowest number of alleles/locus (5 alleles) (Table 3). For the evaluated T x T population, the total alleles were 113 and the average was 7 alleles per locus while for Dura self - the total alleles were 90 and the average was 6 alleles per locus (Table 3). Moreover, the total number of alleles ranged from 40 – 74 alleles while the average ranged from 3 – 5 alleles per locus for each of the oil palm populations (Table 3).
21
Table3. The observed number of alleles for each SSR marker locus among loci in the evaluated oil palm populations
Locus LG
Num ber of Alleles of each locus f or Populat ions :
A125 A127 A140 B01 B02 B57 DuraS
elf TxT All
mEgCIR0802 1 3 4 5 4 2 3 6 7 9
mEgCIR3282 2 5 4 3 4 4 3 5 9 10
mEgCIR0173 3 2 2 2 5 3 1 3 6 6
mEgCIR3533 4 5 4 4 5 5 2 5 7 9
mEgCIR2813 5 4 4 2 3 3 2 4 4 5
mEgCIR3543 6 7 7 4 5 2 3 8 5 8
mEgCIR0894 7 5 5 5 6 5 3 6 9 9
mEgCIR0886 8 6 6 3 2 2 3 8 5 9
mEgCIR3886 9 2 3 3 3 6 3 3 6 6
mEgCIR3785 10 4 4 6 5 3 3 6 10 10
mEgCIR3362 11 6 6 5 4 3 3 7 7 9
mEgCIR2414 12 4 4 3 5 4 3 5 9 9
mCnCIR0038 13 8 7 5 3 6 2 9 8 11
mEgCIR3546 14 4 5 5 5 4 2 5 8 8
mEgCIR3292 15 4 6 5 5 4 3 6 10 11
mEgCIR0353 16 1 3 3 2 3 1 4 3 5
Tot al alleles 70 74 63 66 59 40 90 113 134
Average 4 5 4 4 4 3 6 7 8
Note: Dura Self, combined of three Dura Self populations (B01, B02, and B57); T x T, combined of three T x T populations (A125, A127, and A140); All, combined of all Dura Self and T x T populations (All six populations).
Table4. Expected allele frequencies based on Mendel genetics model crosses of two parents
No
Crosses between parents Expected alleles Model crosses Ex. of parent
22
The evaluated populations are derived from individual palm to palm crosses (Table 1); therefore, the expected number of allele presence for each locus within each population depended on the genotype of the parents. There would be seven possible models of segregation based on Mendel genetics, as presented in Table 4. The expected number of alleles per population and the expected allele frequencies in each model cross are also presented (Table 4).
The number of alleles per locus for some loci in the evaluated oil palm populations is larger than the expected values of the model crosses. Based on the hybridization model (Table 4), the expected number of alleles/locus ranges from 1 – 4 while in the evaluated populations, they range from 1 – 7 alleles/locus. Moreover, some loci showing the expected number of alleles per locus do not show the expected allele segregation ratios for each population (Table 4). This finding was similar to those of Okoye et al. (2016a,b) who evaluated genetic diversity of NIFOR oil palm main breeding parent genotypes using SSR markers. Okoye et al. (2016a,b) also found alleles of the studied loci in the evaluated oil palm populations were also more than the expected and the allele frequencies did not fit to the expected segregation ratios.
Table5. Loci having number of alleles per locus and ratio of allele frequencies that are fit or unfit to the expected based on the model crosses for each studied oil palm population
Population Total loci
Loci having number of allele/locus Loci having ratio of allele frequency* Fit to the expected Larger than
expected Fit to expected Unfit to expected
B01 16 8 8 2 6
B02 16 12 4 6 6
B57 16 16 0 8 8
A140 16 9 7 5 4
A125 16 9 7 4 5
A127 16 9 7 3 6
Note: *Ratios of allele frequency were evaluated only for loci having number of alleles fit to the expected.
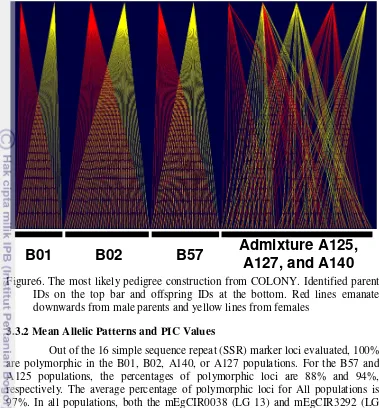
23
Figure6. The most likely pedigree construction from COLONY. Identified parent IDs on the top bar and offspring IDs at the bottom. Red lines emanate downwards from male parents and yellow lines from females
3.3.2 Mean Allelic Patterns and PIC Values
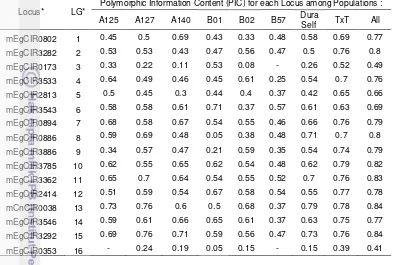
Out of the 16 simple sequence repeat (SSR) marker loci evaluated, 100% are polymorphic in the B01, B02, A140, or A127 populations. For the B57 and A125 populations, the percentages of polymorphic loci are 88% and 94%, respectively. The average percentage of polymorphic loci for All populations is 97%. In all populations, both the mEgCIR0038 (LG 13) and mEgCIR3292 (LG 15) SSR marker loci showed the highest average polymorphic information content (PIC=0.84). Meanwhile, the mEgCIR3785 (LG 10) showed the highest PIC in T x T (PIC=0.79) while and the mEgCIR0038 (LG 13) in D x D population (PIC=0.79), respectively (Table 5). The mEgCIR0353 (LG 16) SSR marker locus (Table 5) showed the lowest PIC in the all populations (PIC=0.41), in T x T populations (PIC=0.39), and in D self population (PIC=0.15). For the All populations, the average PIC over 16loci of SSR markers is 0.74 while that for the evaluated T x T population is 0.70 and for Dura self is 0.56 (Table 6). Meanwhile, the average PIC over loci for each of the evaluated oil palm populations ranged from 0.39 – 0.55 (Table 6).
According to Okoye et al. (2016a,b), the marker loci having PIC value > 0.7 is informative while PIC value between 0.4 – 0.7 is moderately informative and less than 0.4 is not informative. Highvalue of PIC for each locus indicates the locus is