ISOLASI DAN IDENTIFIKASI BAKTERI PENGHASIL GEN
AZOREDUCTASE DALAM PRODUK SUSU BERWARNA
RED 40
RATIH RACHMAWATI
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI SKRIPSI DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA*
Dengan ini saya menyatakan bahwa skripsi berjudul Isolasi dan Identifikasi Bakteri Penghasil Gen Azoreductase dalam Produk Susu Berwarna Red 40 adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir skripsi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Juni 2013
Ratih Rachmawati
ABSTRAK
RATIH RACHMAWATI. Isolasi dan Identifikasi Bakteri Penghasil Gen
Azoreductase dalam Produk Susu Berwarna Red 40. Dibimbing oleh I MADE ARTIKA dan NOVIK NURHIDAYAT.
Azoreductase merupakan enzim yang memiliki kemampuan mendekolorisasi warna red 40 yang tergolong senyawa azo menjadi senyawa amina aromatik sehingga dapat merugikan industri pengolahan susu berwarna. Tujuan dari penelitian adalah menganalisis keberadaan bakteri penghasil gen azoreductase yang berperan pada dekolorisasi dalam produk susu. Uji kualitatif dari 17 isolat bakteri menunjukkan semua sampel susu berwarna merah menjadi putih setelah diinkubasi selama 48 jam. Reduksi zat warna terbaik sampel 25.6 dan 25.1 dengan absorbansi terkecil 0.171 dan 0.173 dilanjutkan analisis statistika perlakuan sampel susu dengan isolat 25.6 – F berpengaruh nyata dengan kontrol. Uji aktivitas degradasi
azoreductase semi kuantitatif dengan kode isolat 91MO73, A, E dan F menunjukkan diameter zona degradasi terbesar kode isolat F sebesar 3.2 cm dan zona degradasi terkecil kode isolat E yaitu sebesar 1.7 cm. Identifikasi dengan
RATIH RACHMAWATI. Isolation and Identification for Bacteria which Produce Gene Azoreductase in Colour Milk Products. Supervised by I MADE ARTIKA and NOVIK NURHIDAYAT.
Azoreductase is an enzyme that has an ability to docolourize red colour 40 were clacified as an azo compound become an aromatic so that it will make an unprofitable for colour milk production industry. The aim of this research was analyzed existence of bacteria which produce an azoreductase gene that has a function to decolourize in milk product. The qualitative analyze from 17 bacteria isolates showed that all of the red colour milk samples become white colour after 48 hours incubated.The best reduction of colour substance are sample 25.6 and 25.1 with smallest absorbance are 0.171 and 0.173 then continue into statistic analyze that milk sample with isolate 25.6 – F had a real influence against the a control. Activities analyze of azoreductase degradation with semi quantitative in isolate code 91MO73, A, E and F showed that the biggest degradation zone diameter of isolate code F was 3.2 cm and the smallest degradation zone diameter in isolate code E was 1.7 cm. Identification with amplification azoreductase gene product by qPCR had a Ct 24.57, 30.87, 33.48 and 35.32 showed that those bacteria isolate samples contain an azoreductase gene
Skripsi
sebagai salah satu syarat untuk memperoleh gelar Sarjana Sains
pada
Departemen Biokimia
ISOLASI DAN IDENTIFIKASI BAKTERI PENGHASIL
GEN AZOREDUCTASE DALAM PRODUK SUSU BERWARNA
RED 40
RATIH RACHMAWATI
DEPARTEMEN BIOKIMIA
FAKULTAS MATEMATIKA DAN ILMU PENGETAHUAN ALAM INSTITUT PERTANIAN BOGOR
DAFTAR ISI
DAFTAR TABEL vi
DAFTAR GAMBAR vi
DAFTAR LAMPIRAN vi
PENDAHULUAN 1
METODE 2
Waktu dan Tempat Penelitian 2
Bahan 2
Alat 2
Prosedur Analisis Data 2
HASIL DAN PEMBAHASAN 5
Hasil 5
Pembahasan 9
SIMPULAN DAN SARAN 14
Simpulan 14
Saran 15
DAFTAR PUSTAKA 15
LAMPIRAN 17
DAFTAR GAMBAR
1 Hasil uji kualitatif inkubasi selama 24 jam 5 2 Hasil uji kualitatif inkubasi selama 48 jam 6 3 Hasil analisis reduksi zat warna red 40 secara kuantitaif kontrol dan
sampel isolat bakteri dengan nilai absorbansi rerata 511 6
4 Hasil zona degradasi azoreductase 7
5 Diamter zona degradasi azoreductase 8
6. Kurva amplifikasi Real Time PCR gen azoreductase pada sampel isolat
bakteri 9
DAFTAR LAMPIRAN
1 Diagram Alir Penelitian 17
2 Hasil daerah terkonservasi gen azoreductase pada sekuen mikroba
dengan homologi tertinggi 18
3 Hasil analisis BLASTN gen azoreductase 19
4 Hasil daerah terkonservasi gen azoreductase dengan menggunakan
program Mega 5 20
1
PENDAHULUAN
Susu merupakan bahan pangan yang diperlukan bagi tubuh manusia yang mempunyai nilai gizi yang lengkap dan seimbang. Kandungan nutrisi dalam susu seperti laktosa, lemak, protein, vitamin, mineral, enzim dan beberapa mikroba. Susu dapat diperoleh dari hasil pemerahan hewan seperti sapi, kuda, kerbau dan kambing. Susu dapat berfungsi untuk menguatkan otot, tulang dan gigi pertumbuhan dan pemeliharaan, membantu fungsi normal otak dan sistem syaraf serta pembentukan sel darah merah (vitamin B12). Di negara maju dan berkembang susu dijadikan sebagai sumber utama kalsium bagi tubuh dan merupakan bahan pangan yang memiliki kandungan gizi tinggi (Kalkwarf et al
2003).
Tingkat konsumsi susu penduduk di Indonesia sekitar 11.09 liter per kapita per tahun. Nilai tersebut jauh lebih rendah bila dibandingkan dengan tingkat konsumsi penduduk di beberapa negara di kawasan Asia Tenggara misalnya negara Malaysia yang mencapai 40 liter per kapita per tahun. Hal tersebut menjadikan pihak produsen susu di Indonesia mengembangkan suatu metode untuk menarik dan meningkatkan selera konsumen dengan menambahkan bahan tambah pangan (Badan Pusat Statistik 2012).
Bahan tambah pangan adalah bahan yang secara alamiah bukan merupakan bagian dari bahan makanan, tetapi terdapat dalam bahan makanan tersebut karena perlakuan pada saat proses pengolahan, penyimpanan atau pengemasan (Cahyadi 2006). Penggunaan bahan tambah pangan seperti produk susu yang telah diberikan pewarna akan dapat memperbaiki dan memberikan daya tarik tersendiri pada produk yang dihasilkan sehingga minuman akan berpenampilan lebih menarik dan menimbulkan selera dengan warna yang indah.
Penambahan pewarna yang digunakan dalam produk susu menggunakan pewarna sintetis yang berjenis pewarna azo. Senyawa azo merupakan zat warna sintetis yang mempunyai sistem kromofor dari gugus azo (-N=N-) yang berikatan dengan gugus aromatik. Struktur umum R-N=N-R’ dengan R dan R’ adalah rantai organik yang sama atau berbeda (Padmavathy et al 2003). Adanya penambahan zat warna azo dalam pengolahan produk susu sapi segar atau fresh milk tidak bertahan lama atau cepat pudar karena memberikan efek penghilangan warna atau dekolorisasi pada susu. Isolat bakteri yang terdapat dalam susu berwarna red 40 mampu mengurangi pewarna jenis senyawa azo dengan bantuan reaksi enzim
azoreductase (Padmavathy et al 2003). Penghilangan warna dalam produk susu berwarna sebagai akibat tereduksinya senyawa azo oleh aktivitas azoreductase
yang berperan dalam sebagian besar bakteri yang belum teridentifikasi.
Penelitian ini bertujuan menganalisis keberadaan bakteri penghasil gen
2
METODE
Waktu dan Tempat Penelitian
Penelitian dilakukan di Laboratorium Genetika Mikroba, Pusat Penelitian Biologi LIPI Cibinong. Penelitian dilakukan mulai bulan Oktober 2012 sampai Mei 2013.
Bahan
Bahan-bahan yang digunakan adalah isolat bakteri tambahan dari LIPI dengan kode strain 84, 83, 92 NO 72, 91 MO73, 27, 3.1, 25.1, 25.3, 25.4, 25.5, 25.6 dan 3.2, susu yang telah berubah warna atau rusak, susu berwarna steril, media Luria Bertani Agar (Bacto agar, trypton, peptone, NaCl dan K2HPO4), akuades, zat warna azo.
Alat
Peralatan yang digunakan dalam penelitian ini adalah alat timbang, gelas ukur, Erlenmeyer, magnetic stirrer, vorteks, cawan petri, sudip, laminar air flow, tip, mikropipet, autoklaf, tabung reaksi, sumbat, spetrofotometer UV-Vis, tabung Effendorf, waterbath, stopwatch, freezer, balok es dan mesin RT-qPCR.
Prosedur Analisis Data
Pembuatan Media Luria Bertani/ LB (Bertani 1951)
Media yang digunakan untuk peremajaan bakteri adalah Luria Bertani (LB) sebanyak 3.75 g bacto agar, 3.75 g pepton, 0.75 g tripton, 1.25g NaCl dan 0.62 g K2HPO4 dilarutkan ke dalam 250 mL akuades dalam labu Erlenmeyer. Larutan diaduk menggunakan magnetic stirrer hingga homogen. Erlenmeyer ditutup rapat dengan alumunium foil dan disterilisasi dalam autoklaf selama 15 menit pada suhu 1210 C dan tekanan 1 atm. Media yang telah steril dibiarkan agak dingin dan dituang ke dalam cawan petri steril secara aseptik dalam laminar air flow dan dibiarkan hingga beku.
Isolasi Bakteri (Modifikasi Cowan 1984)
Sebanyak 100 µ L dan 300 µL susu yang telah rusak dan mengalami perubahan warna, diisolasi dengan media pertumbuhan LB agar dilanjutkan inkubasi selama 24 jam dengan suhu 370 C dalam inkubator. Isolat bakteri dari LIPI dengan kode strain 84, 83, 92 NO 72, 91 MO 73, 27, 3.1, 25.1, 25.3, 25.4, 25.5, 25.6 dan 3.2 diremajakan dengan metode yang sama..
Uji Kualitatif (Modifikasi Ladero et al 2010)
3 kali ulangan. Selanjutnya diinkubasi selama 24 jam pertama dan 48 jam kedua dalam inkubator suhu 370C. Sebagai kontrol yang pertama tabung reaksi steril berisi 9 mL susu berwarna segar tanpa penambahan isolat diletakkan dalam inkubator suhu 370C untuk inkubasi selama 48 jam. Sebagai kontrol kedua tabung reaksi berisi 9 mL susu berwarna tanpa penambahan isolat dan diletakkan di bawah sinar UV inkubasi selama 48 jam untuk melihat pengaruh sinar UV terhadap ketahanan warna red 40.
Analisis Reduksi Red 40 (Modifikasi Cappuccino dan Sherman 1999)
Seleksi hasil isolasi bakteri dari susu yang telah rusak dengan kode A, E, F, D dan B serta isolat dari LIPI 84, 83, 92NO72, 91MO73, 27, 3.1, 25.1, 25.3, 25.4, 25.5, 25.6, 3.2, dimasukkan 1 hingga 2 ose kedalam 17 tabung reaksi steril yang berisi LB cair yang telah disterilisasi. Masing-masing tabung diisi 5 mL untuk pengukuran OD 600 nm dan absorbansi 0.5 untuk dijadikan stok bakteri. Selanjutnya sebanyak 20 mL zat warna red 40 dengan konsentrasi 0.01% yang supernatan untuk analisis menggunakan spektrofotometri UV-VIS dengan maks 511 nm.
Analisis Statistika (Matjik & Sumertajaya 2002)
Analisis statistika yang digunakan adalah rancangan faktorial dalam Rancangan Acak Lengkap (RAL). Penelitian ini dilakukan dengan dua kali ulangan dan uji lanjut menggunakan uji Duncan. Hasil disajikan sebagai rata-rata nilai absorbansi ± SD (standar deviasi) atau rata-rata absorbansi ± SEM (standar error mean). Model linier yang digunakan adalah :
Yij = + Γi+ εij
Keterangan :
i = perlakuan pada sumbu x (control, 84, 83…….B) j = 1,2
Yij = Pengamatan pada perlakuan ke-I dan ulangan ke-j
= Rataan umum
Γi = Pengaruh perlakuan ke-i
εij = Pengaruh acak pada perlakuan ke-I dan ulangan ke-j
Data yang diperoleh dianalisis dengan Analysis of Variance (ANOVA) dengan tingkat kepercayaan 95% dan taraf 0.05. Analisis data dilakukan dengan program SPSS 18.0. Jika hasil uji berbeda nyata maka dilanjutkan dengan uji Duncan.
Uji Aktivitas Degradasi Azoreductase Semi Kuantitatif (Modifikasi Montira
L & Sukallaya B 2012).
4
telah diberikan penambahan red 40. Sebelumnya dilakukan pembuatan media LB agar sebanyak 100 mL dan penambahan 0.01 % red 40 dicampurkan dan diaduk secara homogen dilanjutkan proses sterilisasi dan kemudian diletakkan ke dalam cawan petri. Setelah media padat, isolat sampel diambil 1 jarum ose dan ditumbuhkan 1 titik spot pada media LB agar yang telah berwarna merah untuk dilihat zona bening dan diinkubasi selama 48 jam dan diukur zona beningnya. Adanya 1 spot isolat bakteri yang dikelilingi oleh zona tidak berwarna menunjukkan aktivitas degradasi oleh azoreductase. Rumus perhitungan ukuran zona bening sebagai berikut :
Ukuran zona bening ═
Studi Bioinformatika
Pencarian Daerah Terkonservasi Gen Azoreductase
Identifikasisekuen-sekuen yang homolog dari gen Azoreductase dapat dilakukan dengan menggunakan program dari National Center for Biotechnology Information (NCBI) atau (http ://www.ncbi.nlm.nih.gov/BLAST/) dan memilih analisis BLASTN untuk membandingkan sekuen nukleotida dengan sekuen nukletida dalam database. Hasil dari analisis BLAST, dapat dipilih beberapa sekuen yang menunjukkan homologi cukup tinggi dengan query (nilai bit score
tinggi). Selanjutnya menggunakan program Multiple sequence alignment Clustal W (http://www.ebi.ac.uk/clustalw//) untuk melihat hasil alignment yang menunjukkan daerah homolgi sekuen yang tinggi diantara seluruh sekuen atau daerah terkonservasi.
Perancangan Primer (Sulistyaningsih 2007)
Primer dirancang melalui situs http://blast.ncbi.nlm.nih.gov/ dan memilih tampilan menu Primer-Blast kemudian menyalin sekuen target dari daerah terkonservasi yang mengandung gen azoreductase pada box input sequence.
Perancangan primer dapat dipilih dengan kriteria yaitu, panjang primer berkisar antara 15 – 25 nukleotida, mempunyai 40-60 % GC content, kedua primer sebaiknya tidak mengalami self annealing sehingga sepasang primer tidak saling komplemen, suhu annealing tidak melebihi 700C dan perbedaan temperatur
annealing (melting point) antara kedua primer kurang lebih sama.
Ekstraksi DNA / Heat Shock (Modifikasi Ogier & Serror2002)
Isolat bakteri yang tumbuh dari zona bening diremajakan dalam LB agar miring. Setelah tumbuh, diambil 1 jarum ose dan dimasukkan ke dalam tabung Effendorf ukuran 1 mL yang berisi 500 µL akuades steril. Selanjutnya divorteks hingga homogen dan dipanaskan kedalam waterbath pada suhu 950C selama 15 menit dan dimasukkan ke dalam freezer pada suhu -200 C selama 15 menit. Tahapan selanjutnya dilakukan sentrifugasi kecepatan 5000 rpm selama 5 menit dan diambil supernatan untuk dijadikan sumber DNA template dalam pengujian qPCR.
Amplifikasi Gen Azoreductase (Alarcon et al 2006)
DNA dari sampel yang akan diujikan digunakan sebagai sumber DNA
5
Reverse, DNA template 1 µL, Syber Green Premix 10 µL dan deoxynucleoside triphosphate sebanyak 7 µL.
Reaksi amplifikasi dengan primer 5’ATTTTGACCTGGTTGCACGC3’
(AzoR F) dan 5’TAAAGCCGAGGAACGTGGAC3’ (AzoR R) yang akan
digunakan dalam program qPCR dengan tahapan pradenaturasi 950 C selama 3 menit, denaturasi akhir 950 C selama 10 detik dan annealing pada suhu 560 C selama 30 detik. Amplifikasi gen azoreductase dilakukan sebanyak 40 siklus.
HASIL DAN PEMBAHASAN
Hasil
Analisis Uji Kualitatif
Hasil analisis secara kualitatif (Gambar 1) sampel susu berwarna ditambahkan dengan isolat bakteri yang telah diseleksi dari susu yang telah rusak atau berubah warna dengan kode A, E, F, D dan B serta isolat tambahan dari LIPI dengan kode 84, 83, 92MO72, 91MO73, 27, 3.1, 25.1, 25.3, 25.4, 25.5, 25.6, 3.2 dicampur akuades steril sebanyak 1 mL menunjukkan bahwa, pada inkubasi selama 24 jam pertama baik ulangan 1 dan ulangan 2 terjadi perubahan warna dari warna merah muda menjadi agak keputihan. Sampel kontrol yang memiliki perlakuan sama namun tidak ditambahkan dengan isolat bakteri tidak mengalami perubahan warna. Sampel yang diletakkan di bawah sinar ultraviolet juga tidak mengalami perubahan warna dengan warna tetap merah muda.
Sampel pada inkubasi selama 48 jam, dapat dilihat pada Gambar 2, yang menunjukkan bahwa hampir semua sampel susu dengan penambahan 17 kode isolat bakteri telah mengalami perubahan warna menjadi putih dan mengalami gumpalan padat atau koagulasi baik pada ulangan 1 maupun ulangan 2. Sampel kontrol dan sampel yang diletakkan di bawah sinar ultraviolet tidak mengalami perubahan warna dan berbentuk cair
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 Ulangan 1
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 Ulangan 2
6
Keterangan :
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 Ulangan 1
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 Ulangan 2
Gambar 2 Hasil uji kualitatif inkubasi 48 jam ulangan 1 dan ulangan 2
Keterangan :
Analisis Reduksi Zat Warna Red 40
Hasil analisis uji reduksi red 40 pada 511 nm oleh 17 isolat bakteri dan 1 kontrol dengan dua kali ulangan menunjukkan bahwa sampel isolat dengan kode 25.6 dan 25.1 memiliki kemampuan mereduksi zat warna red 40 paling banyak dengan hasil absorbansi rerata paling rendah dengan nilai 0.171 dan 0.173.
Gambar 3 Hasil analisis secara kuantitaif kontrol dan sampel isolat bakteri dengan nilai absorbansi rerata 511
1 = Kontrol + 5 = 92NO72 9 = F 13 = 25.3 17 = 3.2
2 = Sinar UV 6 = 91MO73 10 = 27 14 =25.4 18 = D
3 = 84 7 = A 11 = 3.1 15 =25.5 19 = B
4 = 83 8 = E 12 = 25.1 16 =25.6
1 = Kontrol + 5 = 92NO72 9 = F 13 = 25.3 17 = 3.2
2 = Sinar UV 6 = 91MO73 10 = 27 14 =25.4 18 = D
3 = 84 7 = A 11 = 3.1 15 =25.5 19 = B
7 Berdasarkan analisis uji secara statistika, dapat dilihat pada tabel analisis ANOVA (Lampiran 7) bahwa memiliki nilai signifikan sebesar 0.017 yang menandakan bahwa pengamatan pada uji reduksi zat warna red 40 memiliki nilai berpengaruh nyata dengan perlakuan lainnya sehingga diperlukan uji lanjut dengan uji Duncan.
Hasil uji Duncan pada reduksi zat warna red 40 dalam penelitian ini berkisar antara 0.171- 0.679. Nilai ini menunjukkan bahwa reduksi zat warna red
40 oleh beberapa sampel bakteri berpengaruh nyata antara kontrol dan perlakuan sampel dengan isolat bakteri 25.6 – F, namun sampel 84 tidak berpengaruh nyata dengan kontrol. Sampel 25.6 hingga sampel F menunjukkan bahwa nilai reduksi zat warna red 40 kelompok kontrol nyata P > 0.05 lebih tinggi daripada kelompok perlakuan lainnya. Kelompok 25.6 dan 25.1 memiliki nilai absorbansi lebih rendah yang berarti isolat 25.6 dan 25.1 memiliki kemampuan mereduksi zat warna red 40 paling kuat.
Aktivitas Degradasi Azoreductase
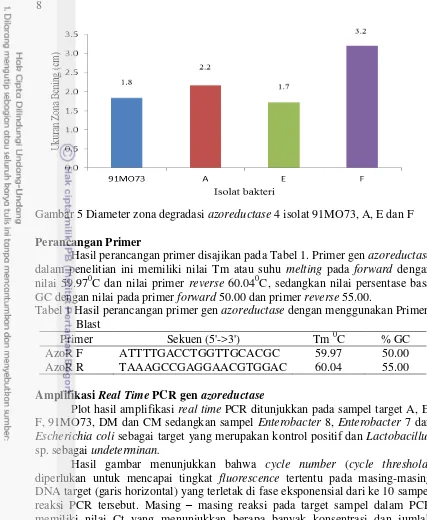
Isolasi bakteri yang telah diseleksi pada 17 isolat (84, 83, 92MO72, 91MO73, A, E, F, 27, 3.1, 25.1, 25.3, 25.4, 25.5, 25.6, 3.2, D dan B) menunjukkan bahwa hanya 4 isolat dengan kode 91MO73, A, E dan F memiliki aktivitas degradasi AzoR yang kuat. Aktivitas degradasi ini ditunjukkan dengan terbentuknya zona bening yang tidak berwarna merah di sekitar koloni (Gambar 3).
1 4
2 3
Gambar 4 Hasil zona degradasi azoR 4 sampel A, E, F dan 91MO73 pada media pertumbuhan LB agar dan zat warna
Keterangan :
1 = A 3 = F
2 = E 4 = 91MO73
8
Gambar 5 Diameter zona degradasi azoreductase 4 isolat 91MO73, A, E dan F
Perancangan Primer
Hasil perancangan primer disajikan pada Tabel 1. Primer gen azoreductase
dalam penelitian ini memiliki nilai Tm atau suhu melting pada forward dengan nilai 59.970C dan nilai primer reverse 60.040C, sedangkan nilai persentase basa GC dengan nilai pada primer forward 50.00 dan primer reverse 55.00.
Tabel 1 Hasil perancangan primer gen azoreductase dengan menggunakan Primer Blast
Primer Sekuen (5'->3') Tm 0C % GC
AzoR F ATTTTGACCTGGTTGCACGC 59.97 50.00
AzoR R TAAAGCCGAGGAACGTGGAC 60.04 55.00
Amplifikasi Real Time PCR gen azoreductase
Plot hasil amplifikasi real time PCR ditunjukkan pada sampel target A, E, F, 91MO73, DM dan CM sedangkan sampel Enterobacter 8, Enterobacter 7 dan
Escherichia coli sebagai target yang merupakan kontrol positif dan Lactobacillus
sp. sebagai undeterminan.
Hasil gambar menunjukkan bahwa cycle number (cycle threshold) diperlukan untuk mencapai tingkat fluorescence tertentu pada masing-masing DNA target (garis horizontal) yang terletak di fase eksponensial dari ke 10 sampel reaksi PCR tersebut. Masing – masing reaksi pada target sampel dalam PCR memiliki nilai Ct yang menunjukkan berapa banyak konsentrasi dan jumlah molekul DNA yang telah diamplifikasi pada siklus yang dihasilkan.
Sampel E.coli sebagai kontrol positif, dapat menghasilkan amplifikasi atau
copy DNA terbanyak pada siklus ke 20.71 bila dibandingkan dengan sampel kontrol positif lainnya. Sampel Enterobacter 8 dapat mengamplifikasi DNA pada siklus 21.79 dan Enterobacter 7 pada siklus 23.02. Sebagai target sampel DNA dari isolat bakteri yang berbeda dengan kode 91MO73, F, Colour milk, E, DM,
9
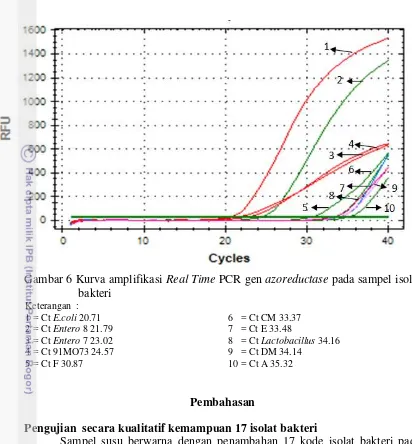
Gambar 6 Kurva amplifikasi Real Time PCR gen azoreductase pada sampel isolat bakteri
Keterangan :
1 = Ct E.coli 20.71 6 = Ct CM 33.37
2 = Ct Entero 8 21.79 7 = Ct E 33.48
3 = Ct Entero 7 23.02 8 = Ct Lactobacillus 34.16
4 = Ct 91MO73 24.57 9 = Ct DM 34.14
5 = Ct F 30.87 10 = Ct A 35.32
Pembahasan
Pengujian secara kualitatif kemampuan 17 isolat bakteri
Sampel susu berwarna dengan penambahan 17 kode isolat bakteri pada masa inkubasi selama 24 jam pertama dan 48 jam kedua dengan suhu 370 C menunjukkan bahwa, terjadi perubahan warna dari merah muda menjadi putih dan sebagian besar mengalami koagulasi atau membentuk gumpalan. Sampel kontrol tidak mengalami perubahan warna karena tidak ada penambahan isolat bakteri. Sampel susu berwarna yang diletakkan di bawah paparan sinar UV juga tidak mengalami perubahan warna selama inkubasi 24 jam pertama dan 48 jam kedua.
Sampel susu yang telah mengalami perubahan warna berbeda dengan sampel kontrol yang warnanya tetap bertahan sebagai akibat adanya aktivitas biologis dalam susu yang menyebabkan terjadinya reaksi enzimatik. Reaksi enzimatik yang berperan adalah azoreductase. Adanya kontrol susu berwarna tanpa penambahan isolat bakteri diletakkan di bawah sinar UV mengindikasikan bahwa sinar UV dapat berpotensi dalam meminimalisir pertumbuhan bakteri dalam susu sehingga mencegah terjadinya ekspresi gen AzoR.
Menurut Purwakakusumah 2007, adanya penyinaran oleh sinar ultraviolet menyebabkan bakteri yang terkandung dalam susu tersebut atau yang berada di lapisan permukaan tabung reaksi yang digunakan akan mati dan proses metabolisme bakteri tersebut tidak ada. Selain itu, menurut Stolz 2001 dan Pandey
10
tahan terhadap sinar cahaya matahari maupun sinar UV sehingga pada sampel kontrol yang diletakkan di bawah paparan sinar UV warnanya akan tetap bertahan.
Reduksi zat warna Red 40
Penghilangan zat warna red 40 menunjukkan adanya aktivitas reduksi gen penyandi azoreductase oleh beberapa sampel bakteri. Hal ini dapat ditunjukkan dengan penambahan red 40 dengan konsentrasi 0.01 % dicampur dengan media LB cair. Sampel perlakuan dengan jumlah 17 isolat bakteri dan 1 kontrol dengan dua kali ulangan diukur dengan 511 nm menunjukkan bahwa isolat dengan kode 25.6 dan 25.1 memiliki ukuran absorbansi rerata paling kecil yaitu sebesar 0.171 dan 0.173.
Nilai tersebut dibuktikan dengan uji statistika yang menunjukkan bahwa, sampel isolat bakteri dengan kode isolat 25.6 – F berpengaruh nyata dengan kontrol, namun sampel 84 tidak berpengaruh nyata dengan kontrol. Kelompok isolat dengan kode 25.6 dan 25.1 memiliki nilai absorbansi terendah yang menunjukkan bahwa isolat bakteri tersebut memiliki kemampuan mereduksi zat warna azo red 40 paling kuat bila dibandingkan dengan perlakuan lainnya.
Semakin rendah nilai absorbansi suatu sampel maka menandakan bahwa isolat tersebut mampu mereduksi zat warna red 40 yang menyebabkan penghilangan warna. Menurut Liu et al 2008 dan Liu et al 2009 bahwa adanya keterlibatan aktivitas oleh azoreductase dalam proses percepatan penghilangan zat warna azo atau dekolorisasi. Selain itu, adanya peran azoreductase yang mengkatalisis NADPH menjadi NADP dalam mereduksi senyawa azo dengan memecah ikatan gugus -N═N- yang menyebabkan terjadinya degradasi pewarna azo (Chen 2006).
Sampel dengan kode isolat bakteri 84 tidak berpengaruh nyata dengan sampel kontrol. Hal tersebut karena pada pewarna red 40 bukan merupakan substrat yang baik untuk aktivitas azoreductase sehingga pewarna red 40 ini akan lebih sulit untuk diangkut kedalam sel bakteri kode 84 dan karena adanya gugus fungsi yang terdapat dalam pewarna red 40 tersebut. Studi terbaru menunjukkan bahwa adanya gugus fungsi dalam struktur pewarna azo misalnya gugus orto, meta dan para dapat mempengaruhi kerentanan terhadap dekolorisasi atau proses penghilangan warna (Hsueh dan Chen 2007, Hsueh dan Chen 2008, Hsueh et al
2009).
Molekul zat warna merupakan suatu gabungan dari zat organik tidak jenuh dengan kromofor sebagai pewarna dan auksokrom sebagai pengikat warna dengan serat. Sekitar 60-70 % zat warna dalam daftar Color Index merupakan jenis zat warna azo yang digunakan dalam makanan dan minuman. Zat warna azo mempunyai sistem kromofor dari gugus azo (-N=N-) yang berikatan dengan gugus aromatik. Struktur umum R-N=N-R’ dengan R dan R’ adalah rantai organik yang sama atau berbeda (Padmavathy et al 2003).
11 tahap, dua elektron ditransfer ke zat warna azo yang bertindak sebagai akseptor elektron. Hal ini berkaitan dengan mediator redoks seperti NADH, NADPH, FMNH2 dan FADH2 bertindak sebagai koenzim untuk mempercepat pemecahan ikatan azo (Guo et al 2010).
Aktivitas degradasi warna red 40 semi kuantitatif 17 isolat bakteri
Aktivitas degradasi azoR dilakukan untuk menunjukkan bakteri yang dapat berperan dalam degradasi zat warna azo Red 40.Degradasi azoR dilakukan dengan menambahkan 1 spot isolat bakteri pada media LB agar yang telah mengandung senyawa zat warna azo Red 40 dengan konsentrasi 0.01 %. Adanya 1 spot bakteri pada media LB warna yang dikelilingi oleh zona tidak berwarna merah atau zona bening menunjukkan adanya aktivitas degradasi azoR yang dihasilkan oleh bakteri tersebut.
Aktivitas degradasi azoR terhadap 17 sampel isolat menunjukkan bahwa hanya 4 isolat dengan kode 91MO73, A, E dan F memiliki kemampuan dalam mendegradasi zat warna azo red 40 dalam media LB berwarna. Diameter zona degradasi terbesar ditunjukkan dengan kode isolat F yaitu sebesar 3.2 cm dan zona degradasi terkecil ditunjukkan dengan kode isolat E yaitu sebesar 1.7 cm. Hal ini menunjukkan perbedaan aktivitas degradasi oleh gen AzoR yang dihasilkan.
Semakin lebar ukuran zona bening menandakan semakin besar potensi isolat bakteri tersebut dalam mengekspresikan gen AzoR sehingga pewarna azo
red 40 yang digunakan dapat terdegradasi. Zona tidak berwarna ini atau zona bening disekeliling koloni menunjukkan adanya gen AzoR yang berperan dalam mereduksi pewarna azo red 40 pada media LB (Montira L & Sukallaya B 2012) .
Alignment Daerah Terkonservasi
Pencarian daerah terkonservasi pada bakteri yang mengandung gen
azoreductase dapat dilakukan melalui situs National Center for Biotechnology Information (NCBI). Identifikasi gen azoreductase ini, dirancang berdasarkan sekuen Escherichia coli strain K -12 yang diperoleh dari situs http://www. ncbi.nlm.nih.gov/. Selanjutnya dicari tingkat homologi dengan menggunakan program Basic Local Alignment Search Tool (BLAST) melalui situs
(http://www.ncbi.nlm.nih.gov/BLAST/) dan memilih analisis BLASTN untuk membandingkan sekuen nukleotida dengan sekuen nukleotida dalam database.
Tingkat homologi suatu sekuen DNA dari gen azoreductase dapat ditentukan berdasarkan parameter hasil BLASTN yaitu bit score, expect value (E) dan query. Bit score merupakan nilai perhitungan statistik hasil perbandingan antara data query sequence dan subject sequence. Semakin tinggi nilai bit score, maka semakin tinggi tingkat hubungan kekerabatan bakteri satu dengan yang lainnya (Thompson et al 2002).
Adanya nilai E merupakan jumlah sekuen pada subject sequence yang tidak terkait dengan query sequence. Semakin kecil nila E value maka semakin tinggi tingkat kepercayaan terhadap similaritas sekuen tersebut (Thompson et al
2002). Selanjutnya daerah terkonservasi yang telah dipilih dengan sekuen tertinggi dari gen azoreductase dapat ditentukan dengan menggunakan program
12
Perancangan Primer
Hasil alignment daerah terkonservasi sekuen gen azoreductase dari program Bioedit kemudian dapat dilakukan analisis perancangan Primer dengan
PRIMER-BLAST dari situs (http://www.ncbi.nlm.nih.gov/BLAST/). Pasangan primer AzoR reverse dan AzoR forward untuk amplifikasi gen azoreductase dari daerah terkonservasi memiliki ukuran panjang ( 323 – 490 bp ).
Berdasarkan Tabel 1, primer AzoR forward memiliki sekuen 5’
ATTTTGACCTGGTTGCACGC3’. Primer ini memiliki nilai melting temperature
(Tm) sebesar 59.970 C dan % GC sebesar 50 % sedangkan primer AzoR reverse dengan sekuen 5’ TAAAGCCGAGGAACGTGGAC3’ memiliki nilai Tm 60.040C dan % GC 55 %. Nilai Tm diatas menunjukkan bahwa kedua nilai primer memiliki hasil terbaik berkisar pada suhu 550 – 600C dan kedua primer memiliki nilai yang saling berdekatan sehingga spesifitas dan terjadinya amplifikasi PCR dapat dicapai (Maier et al 2009)). Hasil % GC forward primer dengan nilai 50.00 dan reverse primer pada 55.00 menunjukkan bahwa presentase jumlah G dan C terhadap jumlah basa total pada primer berkisar antara 40-60%.
Hasil primer forward dan reverse memiliki panjang 20 bp. Nilai tersebut sesuai dengan literatur bahwa panjang primer optimal adalah 18-22 bp. Panjang ini cukup panjang untuk mencapai spesifisitas yang cukup dan cukup pendek bagi primer untuk terikat dengan mudah pada DNA template pada suhu annealing-nya (Sulistyaningsih 2007). Berdasarkan analisis dengan program Gene Runner,
primer AzoR forward dan reverse terdapat kemungkinan dimer sekitar 4 bp dan primer
Amplifikasi gen azoreductase
Isolat bakteri dari hasil uji aktivitas degradasi azoreductase dengan kode 91MO73, A, E dan F serta isolat induk dari E.coli, Lactobacillus, Enterobacter 7 dan 8 diekstraksi dengan metode heat shock untuk diambil bagian DNA. Fungsi ekstraksi DNA dalam bakteri dengan metode kejut panas atau heat shock agar dapat melisis bagian membrane sel sehingga DNA yang diharapkan dapat dijadikan sebagai sumber DNA template(Sambrook & Russel 2003).
Analisis amplifikasi gen azoreductase (AzoR) dilakukan melalui analisis DNA menggunakan real time PCR (qPCR). Analisis qPCR dilakukan terhadap 10 target sampel isolat bakteri yang berhasil diisolasi dan dijadikan sebagai sumber DNA template. Sampel isolat tersebut dipilih karena pada uji secara semi kuantitatif menunjukkan hasil pengukuran zona degradasi warna koloni yang jelas pada isolat bakteri dengan kode A, E, F dan 91MO73.
Kontrol positif diambil dari isolat bakteri E.coli dan Enterobacter dengan kode 8 dan 7 yang merupakan hasil sekuen dari analisis BLASTN dalam database pada NCBI dan mengandung gen azoreductase dengan daerah homologi tertinggi (Thompson et al 2002). Isolat dengan kode DM dan CM didapatkan dari susu yang telah mengalami perubahan warna dan susu yang masih dalam keadaan steril. Begitu juga dengan sampel Lactobacillus, yang belum diketahui ada atau tidaknya gen azoreductase.
13 dengan dsDNA dan memancarkan sinyal. SYBR Green ketika terikat dengan dsDNA tidak dapat berpendar. Setelah mengalami satu siklus pada tahap denaturasi atau pemanasan suhu 950 C, dsDNA akan menjadi ssDNA akibat pemanasan (Smith 2008). Selanjutnya, ketika template menjadi ssDNA, tahap
annealing atau penempelan primer yang spesifik terjadi dan pewarna SYBR Green
disini akan berpendar mengeluarkan deteksi sinyal fluorescence yang menandakan bahwa gen azoreductase tersebut dapat teridentifikasi secara kualitatif (Manit et al
2005).
Sampel Escherichia coli yang bertindak sebagai kontrol positif pada Gambar 6 menunjukkan bahwa, bakteri tersebut dapat menghasilkan copy DNA atau amplikon DNA yang mengandung gen azoreductase terbanyak dengan nilai Ct 20.71. Hasil amplifikasi DNA pada sampel target tersebut jauh lebih tinggi bila dibandingkan dengan sampel target bakteri dengan kode A yang dalam menghasilkan produk amplifikasi gen azoreductase baru didapatkan pada Ct 35.32.
Hal tersebut dapat dilihat selama proses amplifikasi DNA terjadi pada saat fase eksponensial yang dapat menghasilkan 2 kali lipat amplikon DNA setiap satu siklus. Hal ini terkait dengan adanya nilai Ct (Cycle threshold) yang berbanding terbalik dengan jumlah DNA awal. Semakin cepat atau semakin rendah nilai Ct (cycle threshold) yang didapatkan maka semakin banyak dan semakin tinggi konsentrasi DNA awal yang dihasilkan sehingga banyaknya jumlah molekul gen
azoreductase yang didapatkan juga semakin banyak. Sebaliknya, semakin lama atau semakin tinggi nilai Ct (cycle threshold) yang didapatkan maka semakin sedikit dan semakin rendah jumlah DNA awal sehingga dihasilkan jumlah molekul amplikon gen azoreductase semakin sedikit (Manit et al 2005).
Target sampel dengan kode 91MO73, F, E dan A dapat menghasilkan produk amplifikasi DNA pada Ct berturut-turut 24.57, 30.87, 33.48 dan 35.32. Nilai Ct tersebut menunjukkan bahwa bakteri tersebut dapat mengamplifikasi gen
azoreductase tersebut dengan jumlah molekul atau copy number DNA yang berbeda-beda tergantung pada hasil pancaran sinar dari SYBR Green yang didapatkan pada fase eksponensial. Sampel bakteri Lactobacillus yang diduga tidak mengandung gen azoreductase, namun pada Gambar 6, ditunjukkan bahwa pada siklus 34.16, bakteri ini dapat menghasilkan produk amplifikasi DNA yang mengandung gen azoreductase yang dapat ditangkap oleh pewarna SYBR Green
pada fase eksponensial (Manit et al 2005).
Hasil Ct di atas merupakan hasil visualisasi secara kualitatif ada tidaknya gen azoreductase dalam masing-masing sampel bakteri, namun belum diketahui berapa banyak konsentrasi dan jumlah molekul DNA secara kuantitatif yang dihasilkan pada nilai Ct yang didapatkan. Hal ini karena perlu dilakukan perhitungan lebih lanjut dimana diperlukan adanya kurva standar DNA yang selanjutnya konsentrasi DNA pada masing-masing sampel target dapat ditentukan dengan membandingkan nilai Ct terhadap nilai kurva standar DNA (Victor et al
2012)
14
memiliki nilai Ct yang sedikit jauh lebih tinggi bila dibandingkan pada sampel DM yakni sebesar 33.37.
Hal tersebut mengindikasikan bahwa pada sampel CM yang berupa susu steril dapat menghasilkan jumlah molekul DNA lebih banyak bila dibandingkan dengan sampel DM. Hasil produk amplifikasi sampel CM sedikit lebih tinggi dibandingkan dengan DM, karena adanya bakteri penghasil gen azoreductase
yang terdapat pada susu steril yang dapat mereduksi substrat pewarna red 40. Akibatnya dalam jangka waktu tertentu, susu steril tersebut dapat menghasilkan lebih banyak lagi jumlah molekul dan konsentrasi DNA seiring dengan terjadinya perubahan warna dalam sampel susu tersebut (Montira L & Sukallaya B 2012).
Hasil sampel DM yang telah mengalami perubahan warna dapat menghasilkan jumlah molekul DNA lebih sedikit bila dibandingkan dengan sampel CM yang merupakan susu steril. Hal ini karena adanya perbedaan aktivitas
azoreductase dalam mereduksi substrat dari pewarna red 40 dimana pada sampel DM, komponen substrat dari pewarna red 40 telah direduksi sebelumnya sehingga pada saat dilakukan qPCR sampel DM telah mengalami pengurangan aktivitas atau masa aktif sehingga menghasilkan hasil produk amplifikasi lebih sedikit (Montira L & Sukallaya B 2012).
Pengamatan secara kuantitatif dan kualitatif dengan menggunakan Real Time PCR dapat dilihat pada reaksi yang berlangsung cepat hanya pada fase eksponensial yang dapat menghasilkan produk amplifikasi DNA dengan nilai dua kali lipat setiap siklusnya secara tepat dan akurat (Manit et al 2005). Grafik yang menunjukkan fase linier merupakan reaksi amplifikasi yang terjadi mulai melambat dan produk PCR tidak lagi menjadi dua kali lipat pada setiap siklusnya. Fase plateau menunjukkan bahwa reaksi telah berhenti dan tidak ada lagi produk amplifikasi yang sedang dibuat (Vanguilder et al 2008).
Keuntungan dari penggunaan real time PCR bila dibandingkan dengan PCR standar biasa adalah bila dilihat dari segi kuantitatif real time PCR dapat dilihat jumlah produk PCR pada fase eksponensial namun pada PCR standar biasa hanya dapat membandingkan intensitas dari band hasil elektroforesis yang membutuhkan pewarna EtBr secara semi kuantitatif (Smith 2008).
SIMPULAN
Simpulan
15
Saran
Karakterisasi lebih lanjut perlu dilakukan dengan identifikasi jenis bakteri menggunakan DNA sequencing. Bagi pihak industri minuman susu, agar dalam pengolahan produk susu berwarna dalam hal proses sterilisasi disesuaikan dengan kondisi optimum bakteri yang telah diidentifikasi. Selain itu, penambahan pewarna sintetis jenis azo sesuai dengan batas optimum pewarna yang telah ditentukan.
DAFTAR PUSTAKA
[BPS] Badan Pusat Statistik. 2012. Statistik Peternakan 2012. Konsumsi Susu Per Kapita Per Tahun 2009 – 2012. Jakarta : Badan Pusat Statistik.
Alarcon B, B Vicedo, and R. Aznar. 2006. PCR based procedures for detection and quantification of Staphylococcus aureus and their application in food. J Appl Microbiol 100:352−364.
Cahyadi W. 2006. Analisis dan Aspek kesehatan Bahan Tambahan Pangan.Edisi Kedua. Jakarta : Penerbit Bunga Aksara.
Cappuccino GJ, Sherman N. 1999. Fourth Edition. Microbiology A. Laboratory Manual. J Addison Wesley. 37(4):129-183.
Chen H. 2006. Recent advances in azo dye degrading enzyme research. Current Protein and Peptide Science 7:101-111.
Cowan ST. 1984. Manual for the Identification of Medical Bacteria. Second Edition. Cambridge : Cambridge University Press.
Guo J.Li K, Wang X, Yang J. 2010. The Handbook of Environmental Chemistry.
New York : Springer Heidelberg Inc.
Hsueh CC, Chen BY, Yen CY. 2009. Understanding effects of chemical structure on azo dye decolorization characteristics by Aeromonas hydrophila. J Hazard Materials.167:995-1001.
Hsueh CC, Chen BY. 2007. Comparative study on reaction selectivity of azo dye decolorization by Pseudomonas luteola. J Hazard Materials 141:842-849. Hsueh CC, Chen BY. 2008. Exploring effects of chemical structure on azo dye
decolorization characteristics by Pseudomonas luteola. J Hazard Materials 154:703-710.
Kalkwarf et al. 2003. Milk Intake During Childhood and Adolescence, Adult Bone Density and Osteoporosis Fractures in US Women. American Journal Clinical Nutrition 77:257–265.
Ladero V, Martinez N, Martin MC, Fernandez M, Alvarez MA. 2010. qPCR for quantitative detection of tyramine producing bacteria in dairy products. J Food Microbiology 43:289-295.
Liu G, Zhou J, Jin R, Zhou M, Wang J, Lu H, Qu Y. 2008. Enhancing survival of
Escherichia coli by expression of azoreductase AZR possessing quinone reductase activity. Applied Microbiology Biotechnology. 80: 409-416. Liu G, Zhou J, Wang J, Zhou M , Lu H, Jin R. 2009. Acceleration of azo dye
16
Maier RM, Pepper IL, Gerba CP. 2009. Environmental Microbiology. London : Elsevier Inc.
Manit A, Iqbal S, Magali W, Lyndon G, Neehar A, Hitendra. 2005. Basic principles of real-time quantitative PCR. FD Expert Rev Mol Diagn 5(2). Mattjik AA, Sumertajaya M. 2002. Perancangan Percobaan dengan Aplikasi
SAS. Bogor : IPB Press.
Montira L, B Sukallaya. 2012.Characterization of the decolorizing activity of azo dyes by Bacillus subtilis azoreductase AzoR1. Songklanakkarin Journal of Science and Technology. 34(5):509–516
Ogier JC, Serror P. 2008. Safety Assessment of Dairy Microorganism the
Enterococcus genus. J Food Microbiol 126:291–301.
Padmavathy SS, Sandhya K, Swaminathan YV, Subrahmanyam T. Chakrabarti, and SN Kaul. 2003. Aerobic Decolorization of Reactive Azo Dyes in Presence of Various Cosubstrates. Chem Biochem Eng.17(2):147–151. Padmavathy SS, Sandhya K, Swaminathan YV, Subrahmanyam T.Chakrabarti,
SN Kaul. 2003. Aerobic Decolorization of Reactive Azo Dyes in Presence of Various Cosubstrates. Chem Biochem Eng. 17(2):147–151.
Pandey A, Singh P, Iyengar L. 2007. Bacterial decolorization and degradation of azo dyes. International Biodeterioration & Biodegradation. 59:73-84. Purwakusumah W. 2007. Filter Ultra Violet Sinar UV [Internet] ; [diunduh 2013
Juni 01]. Tersedia pada : http://www.o-fish.com/Filter/filter uv.php. Sambrook J, Russel DW. 2003. Molecular Clonning : A laboratory manual
Edition 3. New York : Cold Spring Harbor Laboratory Press.
Smith C, Mark O. 2008. Advantages and limitations of quantitative PCR based approaches in microbial ecology [editorial]. FEMS Department of Animal and Plant Sciences : University of Sheffield, Westren Bank.
Stolz A. 2001. Basic and applied aspects in the microbial degradation of azo dyes.
Applied Microbiology Biotechnology. 56:69-80.
Sulistyaningsih E. 2007. Polymerase Chain Reacton (PCR) : Era Baru Diagnosis dan Manajemen Penyakit Infeksi. Jurnal Biomedis Vol 1.
Thompson JD, Gibson TJ, Higgins DG. 2002. Multiple sequence alignment using ClustalW and ClustalX. Curr Protoc Bioinformatics. Chapter 2:3.
VanGuilder HD, Vrana KE, Freeman WM (2008).Twenty-five years of quantitative PCR for gene expression analysis. J Biotech 44: 619–626.
17
LAMPIRAN
Lampiran 1 Diagram Alir Penelitian
Analisis Uji Kualitatif
Analisis reduksi warna red 40 secara Kuantitatif
Pembuatan Media dan Isolasi Bakteri
Studi Bioinformatika Analisis Statistika
Perancangan Primer
Amplifikasi DNA dengan qPCR Uji Aktivitas Degradasi Azoreductase Semi
18
Lampiran 2 Hasil Pensejajaran (alignment) daerah terkonservasi gen azoreductase
pada sekuen bakteri dengan homologi tertinggi
ATGAGCAAGGTATTAGTTCTTAAATCCAGCATCCTGGCAGGGTACTCT CAGTCTAATCAGTTGTCCGATTATTTTGTTGAACAATGGCGCGAAAAG CACTCCGCTGATGAAATCACCGTTCGCGACCTGGCTGCAAATCCGATT CCGGTACTGGATGGCGAACTGGTTGGCGCTCTGCGTCCGAGCGATGCG CCGCTGACTCCGCGTCAGCAGGAAGCTCTGGCGCTTTCCGATGAGCTG ATTGCCGAGCTGAAAGCCCACGACGTTATCGTTATTGCGGCACCGATG TATAACTTCAACATCTCAACTCAGTTGAAAAACTATTTTGACCTGGTTG CACGCGCAGGCGTTACTTTCCGCTATACCGAGAACGGTCCGGAAGGTC TGGTAACGGGTAAAAAAGCCATCGTTATTACCAGCCGCGGTGGGATCC ACAAAGATGGACCAACGGACCTGGTGACGCCGTATCTGTCCACGTTCC TCGGCTTTATCGGCATTACCGATGTGAAATTTGTCTTCGCCGAAGGGAT CGCATACGGTCCGGAAATGGCAGCGAAAGCGCAGTCTGACGCGAAAG CAGCCATCGACAGCATTGTTGCTGCATAA
20
Lampiran 4 Hasil daerah terkonservasi gen azoreductase dengan menggunakan program Mega 5
21 Lampiran 6 Hasil analisis Primer dimer AzoR reverse program Genne Runner
Lampiran 7 Hasil pengukuran zona degradasi azoR 4 isolat terpilih No Kode Isolat Diamater Zona
Bening (cm)
Diameter Koloni (cm)
Rasio Zona Bening
1 91MO73 1.1 0.6 1.8
2 A 1.3 0.6 2.2
3 E 1.2 0.7 1.7
4 F 1.6 0.5 3.2
Lampiran 8 Hasil pengujian secara kuantitatif reduksi zat warna 511 nm
Sampel A rerata Standar Deviasi Nilai Perbedaan Rata-rata
Kontrol 0.679 0.001 0.679a ± 0.001
84 0.481 0.266 0.481a ± 0.266
83 0.309 0.171 0.309bc ± 0.171
92No72 0.226 0.072 0.226c ± 0.072
91Mo73 0.303 0.177 0.303bc ± 0.177
A 0.240 0.078 0.24bc ± 0.078
E 0.247 0.049 0.247bc ± 0.049
22
Sampel A rerata Standar Deviasi Nilai Perbedaan Rata-rata
27 0.215 0.013 0.215c ± 0.013
Lampiran 9 Analisis statistika Reduksi zat warna red 40 ANOVA
Lampiran 10 Uji lanjut Duncan terhadap reduksi zat warna red 40
23 Lampiran 11 Hasil amplifikasi produk DNA dengan Real Time PCR
Well Fluor Target Content Sample Cq Color SQ
A01 SYBR Azr Unkn A 35,32 Green N/A
A02 SYBR Azr Unkn E 33,48 Green N/A
A03 SYBR Azr Unkn F 30,87 Green N/A
A04 SYBR Azr Unkn 91M073 24,57 Green N/A A05 SYBR Azr UnDeterm Lactobacillus 34,16 Blue N/A A06 SYBR Azr Pos Ctrl Entero 8 21,79 Red N/A A07 SYBR Azr Pos Ctrl Entero7 23,02 Red N/A A08 SYBR Azr Pos Ctrl E.coli 20,71 Red N/A
A09 SYBR Azr Unkn DM 34,14 PalePink N/A
24
RIWAYAT HIDUP
Penulis dilahirkan di Kudus pada tanggal 29 Desember 1990 dari ayahanda Mr Munarto dan ibunda GCH.Susita Hermiyati. Penulis merupakan anak ketiga dari tiga bersaudara. Pada tahun 2009 penulis lulus dari SMA Negeri 1 Kudus dan melanjutkan sekolah di Institut Pertanian Bogor Program Studi Biokimia Fakultas Matematika dan Ilmu Pengetahuan Alam melalui jalur Undangan Seleksi Masuk IPB (USMI).
Selama masa perkuliahan, penulis aktif menjadi anggota Kesatuan Mahasiswa Katolik IPB (KEMAKI). Penulis juga aktif menjadi anggota beberapa kepanitiaan yang diadakan oleh IPB seperti menjadi tim Humas pada acara Hari Olahraga Kemaki tahun 2010, anggota tim divisi bidang keilmuan Bioanalisis di Crebs tahun 2011, anggota tim medis dalam Masa Perkenalan Fakultas (MPF) tahun 2011.
Pada tahun 2012, penulis melaksanakan praktik lapang di Pusat Penelitian