KERAGAMAN GENETIK PLASMA NUTFAH KELAPA
INDONESIA DAN PENENTUAN IDENTITAS KELAPA
HIBRIDA BERDASARKAN MARKA MOLEKULER
ANNEKE PESIK
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Dengan ini saya menyatakan bahwa disertasi berjudul “Keragaman Genetik Plasma Nutfah Kelapa Indonesia dan Penentuan Identitas Kelapa Hibrida Berdasarkan Marka Molekuler” adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi mana pun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir disertasi ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, September 2016
ANNEKE PESIK. Keragaman Genetik Plasma Nutfah Kelapa Indonesia dan Penentuan Identitas Kelapa Hibrida Berdasarkan Marka Molekuler. Dibimbing oleh DARDA EFENDI, SUDARSONO, HENGKY NOVARIANTO dan DINY DINARTI.
Keragaman genetik plasma nutfah kelapa yang ada di Indonesia sangat tinggi, sebagian besar populasi tersebut telah dikoleksi oleh Balai Penelitian Palma, Manado. Namun, kajian genetik untuk semua aksesi kelapa kurang lengkap. Informasi keragaman dan hubungan genetik tanaman kelapa menjadi penting dalam strategi pemuliaan tanaman kelapa. Kelapa hasil persilangan terkontrol perlu diidentifikasi secara genetik untuk memastikan kebenaran tetuanya, karena kontaminasi serbuk sari dapat berpotensi menjadi masalah. Penggunaan marka molekuler mampu mendeteksi keragaman genetik aksesi dan menduga kebenaran tetuanya untuk efisiensi waktu yang diperlukan dalam proses seleksi. Rangkaian penelitian dilakukan bertujuan untuk 1) mengembangkan marka SNAP berbasis gen WRKY dengan reaksi multiplex PCR; 2) mengevaluasi keragaman genetik dan struktur populasi kelapa Indonesia dengan marka SNAP berbasis gen WRKY, SUS, SACPD dan ABI; 3) mengevaluasi keragaman genetik dan struktur populasi kelapa Indonesia dengan marka SSR; 4) menganalisis hubungan genetik antara tetua GKN dan DTA dengan hibrida KHINA-1 menggunakan marka SSR; 5) mengidentifikasi marka SSR yang berasosiasi dengan karakter jumlah buah dan komponen buah; dan 6) mengidentifikasi hibrida kelapa kopyor di Pati dan Lampung berdasarkan marka SNAP dan SSR.
Penelitian diawali dengan pengembangan marka SNAP berbasis gen WRKY yang divalidasi menggunakan kelapa kopyor. Disain primer SNAP menggunakan tiga puluh lima sekuen bank data NCBI dari gen WRKY2, WRKY6, WRKY7, WRKY19 dan WRKY21. Delapan pasang primer SNAP berhasil didisain. Amplifikasi DNA menggunakan primer SNAP dilakukan dengan metode multiplex PCR. Selanjutnya delapan pasang primer SNAP gen WRKY digunakan untuk analisis keragaman genetik kelapa.
Analisis keragaman genetik kelapa Indonesia menggunakan marka bi-alel SNAP berbasis gen WRKY, SUS, SACPD dan ABI3 dilakukan untuk 28 aksesi kelapa Dalam dan 13 aksesi kelapa Genjah. Hasil analisis berdasarkan metode
Neighbour Joining menunjukkan ada tiga kelompok utama yang terbagi ke dalam subkelompok berbeda. Umumnya aksesi kelapa Dalam menyebar dengan aksesi yang lain, hanya 2 aksesi yang mengelompok yaitu DBI dan DTE. Hal yang sama juga terjadi pada kelapa Genjah hanya 2 aksesi yang mengelompok (GKN dan GSK). Struktur populasi berdasarkan 16 marka SNAP menunjukkan bahwa populasi kelapa terbagi atas dua subgrup dengan pencampuran alel yang beragam.
Keragaman genetik kelapa juga dilakukan dengan marka molekuler multialel yaitu SSR untuk populasi yang sama. Hasil analisis keragaman genetik kelapa Indonesia menggunakan 20 marka SSR menunjukkan bahwa terdapat keragaman genetik yang sangat tinggi pada populasi uji. Analisis filogenetik berdasarkan metode
DTA memberikan harapan bahwa populasi hibrida hasil persilangan antar keduanya dapat menghasilkan hibrida yang sangat heterogen sehingga diharapkan lebih baik dibandingkan kedua tetuanya sebagai akibat terjadinya heterosis. Pengelompokkan populasi tetua dan KHINA-1 dengan metode Neighbour Joining menghasilkan lima kelompok. Individu KHINA-1 di kelompok I, IV, dan V merupakan illegitimate
hybrid karena mempunyai kemiripan genetik yang tinggi dengan tetua GKN
(Kelompok I) atau DTA (Kelompok IV dan V) sedangkan individu KHINA-1 pada kelompok II dan III yang memiliki alel kedua tetuanya merupakan individu
legitimate hybrid.
Analisis marka tunggal digunakan untuk mengidentifikasi lokus marka SSR yang berasosiasi dengan karakter jumlah buah dan komponen buah KHINA-1. Sebanyak 35 individu legitimate hybrid KHINA-1 dilakukan pengamatan jumlah buah dan komponen buah. Parameter komponen buah terdiri dari: berat buah utuh, berat buah tanpa sabut, berat buah tanpa air, berat daging buah dan tebal daging buah. Data pengamatan digabungkan dengan data genotyping marka SSR pada penelitian hubungan genetik tetua dengan hibrida KHINA-1, kemudian dianalisis menggunakan piranti lunak SAS versi 9.1. Hasil asosiasi marka tunggal diperoleh dua lokus (CNZ 21 dan CNZ 51) berkorelasi positif dengan karakter jumlah buah per tandan. Lokus CnCir 87 berasosiasi dengan karakter berat buah utuh dan berat buah tanpa sabut, sedangkan karakter tebal daging buah berasosiasi dengan lokus CnCir 56. Dua karakter lain (berat buah tanpa air dan berat daging buah) tidak menunjukkan adanya asosiasi dengan lokus marka SSR yang diuji.
Identifikasi hibrida kopyor menggunakan marka SNAP berbasis gen WRKY, SUS, SACPD, ABI3 dan marka SSR telah dilakukan untuk persilangan kelapa kopyor di Pati dan Lampung. Seleksi 16 primer SNAP dan 20 primer SSR dilakukan untuk membedakan tetua betina dan jantan. Hasil seleksi primer SNAP untuk membedakan tetua betina dan tetua jantan, ada 4 primer gen WRKY (WRKY6#1, WRKY6#3, WRKY19#1 dan WRKY21#3) untuk persilangan di Pati, dan 2 primer gen WRKY (WRKY19#1 dan WRKY21#3) untuk persilangan di Lampung. Primer-primer tersebut digunakan untuk mengamplifikasi DNA hibrida kopyor di Pati dan Lampung. Marka SNAP berbasis gen SUS, SACPD, ABI3 tidak digunakan untuk identifikasi hibrida kopyor karena teruji tidak dapat membedakan tetua betina dan tetua jantan. Untuk primer SSR diperoleh 4 primer (CNZ 21, CNZ 51, CnCir 56 dan CnCir A9) yang dapat digunakan untuk identifikasi hibrida kopyor. Identifikasi hibrida koypor dengan marka SNAP dan SSR menunjukkan bahwa persilangan di Pati hanya sebagian progeni yang legitimate hybrid sedangkan persilangan di Lampung semua progeni merupakan individu legitimate hybrid.
Identifikasi hibrida kelapa Kopyor di Lampung memiliki tingkat keberhasilan persilangan lebih tinggi dibandingkan di Pati. Rata-rata persentasi persilangan di Lampung mencapai 84.4% (total 36 individu) legitimate hybrid dan 15.6% (total 7 individu) illegitimate hybrid, sedangkan di Pati hanya mencapai 63.5% (total 127 individu) legitimate hybrid dan 36.5% (total 73 individu) illegitimate hybrid.
ANNEKE PESIK. The Genetic Diversity of Indonesian Coconut Germplasm and Determining the Identity of Coconut Hybrid Based on Molecular Markers. Supervised by DARDA EFENDI, SUDARSONO, HENGKY NOVARIANTO and DINY DINARTI.
The genetic diversity of coconut germplasm in Indonesia is very high which majority of the population has been collected by the Indonesian Palm Research Institute, Manado (North Sulawesi). However, genetic studies for all coconut accessions are considered less complete. The information of plant genetic diversity and relationships become important in the coconut palm plant breeding programme. Similarly, progeny of the pollination controlled need to be identified to ensure the correctness of parents, because pollen contamination can potentially be a problem. The use of molecular markers capable of detecting the genetic diversity of accessions and guessed the truth parents for time efficiency is needed in the selection process. The series of studies conducted aiming to 1) develop SNAP markers based on WRKY gene with multiplex PCR technique; 2) evaluate the genetic diversity and population structure of Indonesian coconut with SNAP markers based on WRKY, SUS, SACPD and ABI gene; 3) evaluate the genetic diversity and population structure of Indonesian coconut with SSR markers; 4) analyze the genetic relationship between the parents, i.e. DTA and GKN with the hybrid KHINA-1 using SSR markers; 5) identify SSR markers associated with the character of the amount of fruit and the character of fruit components; and 6) identify hybrid coconut kopyor in Pati and Lampung using SNAP and SSR markers.
The study began with the development of markers SNAP based on WRKY were validated using kopyor coconut. SNAP primer were designed using thirty five sequences from the NCBI genebank for WRKY2, WRKY6, WRKY7, WRKY19 and WRKY21. Eight primer pairs WRKY SNAP were successfully designed. DNA amplification using the SNAP primers was performed by multiplex PCR technique. Furthermore, the eight primers SNAP of WRKY genes were used for analysis of the coconut genetic diversity.
Analysis of genetic diversity of Indonesian coconut using bi-allelic SNAP markers of WRKY, SUS, SACPD and ABI3 gene were conducted for Dwarf and Tall coconut of 28 and 13 accessions, respectively. The phylogenetic result using Neighbour Joining method shows there is three main groups which also divided into different subgroups. On the general, coconut accession spread within other accession; however, two accessions (DBI and DTE) clustered together. The same thing occurs in only 2 Dwarf coconut accessions clustered (GKN and GSK). The structure of the population by 16 SNAP markers showed that coconut population is divided into two subgroups with diverse mixing allele.
Population structure based on the SSR markers dividing population into two subgroups of coconut.
The research of analysis of genetic relationships between parents and KHINA-1 hybrid using 19 SSR markers. Kinship between GKN and DTA populations were long distance and a high degree of heterogeneity among individuals DTA coconut gives good expectations that the population hybrids between them can generate highly heterogeneous hybrid that could be better than both parents as a result of heterosis. Grouping the parental population and KHINA-1 with the Neighbour Joining method produces five groups. Individual KHINA-1 in group I, IV, and V is illegitimate hybrid because it has genetic similarity with parents GKN (Group I) or DTA (Group IV and V), while individual KHINA-1 in group II and III which have alleles from both parents as a legitimate hybrid.
An analysis of single markers were used to identify whether the loci SSR markers associated with some characters of amount of fruit and fruit components of KHINA-1. A total of 35 individuals KHINA-1 legitimate hybrid were observed onto fruit number and fruit components. Parameter fruit components consist of: whole fruit weight, the weight of the fruit without the fiber, fruit weight without water, heavy flesh and thick flesh. Observation data combined with genotyping data of SSR markers in genetic association studies with parents and KHINA-1 hybrid, and then analyzed using SAS software version 9.1. The results of a single marker association acquired two loci (CNZ 21 and CNZ 51) is positively correlated with the number of characters per bunch. Locus of CnCir 87 associated with the character of the whole fruit weight and the weight of the fruit without the fiber, while the character of the fruit flesh associated with locus CnCir 56. Two other characters (heavy fruit without water and heavy flesh) did not show any relation with loci of SSR markers.
Kopyor hybrid identification using the SNAP markers based on WRKY, SUS, SACPD, ABI3 gene and the SSR markers were done to coconut kopyor progeny in Pati (Central Java) and Lampung (South Sumatra). Selection of 16 oligonucleotide SNAP primers and 20 SSR primers were carried out to distinguish male and female parents. The selection result of SNAP primer for distinguish the male and female parents, there are 4 primer of WRKY gene (WRKY6#1, WRKY6#3, WRKY19#1 and WRKY21#3) suitable for progeny in Pati, while 2 primer of WRKY gene (WRKY-19#1 and WRKY21#3) fitted for progeny in Lampung. The primers were tested to amplify DNA kopyor hybrid both in Pati and Lampung. SNAP markers based on SUS, SACPD, ABI3 gene were failed for identification of kopyor hybrid as proven did not distinguish the female and male parents. For SSR marker, there were obtained 4 primers (CNZ 21, CNZ 51, CnCir 56, and CnCir A9) that can be used for identify-cation of kopyor hybrid. Identifiidentify-cation of kopyor hybrid with SNAP and SSR markers showed that the molecular approach allows early identification of the controlled crossing of coconut, prior the plant produce the fruits with kopyor phenotypically.
Identification of controlled crossing of coconut Kopyor in Lampung has a success rate higher than the breeding in Pati. The average percentage of breeding in Lampung reached 84.4% (36 individuals for total) legitimate hybrid and 15.6% (7 individuals) illegitimate hybrid, whereas in Pati only reached 63.5% (127 individuals for total) legitimate hybrid and 36.5% (73 individuals) illegitimate hybrid.
Hak Cipta
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
KERAGAMAN GENETIK PLASMA NUTFAH KELAPA
INDONESIA DAN PENENTUAN IDENTITAS KELAPA
HIBRIDA BERDASARKAN MARKA MOLEKULER
ANNEKE PESIK
Disertasi
sebagai salah satu syarat untuk memperoleh gelar Doktor pada
Program Studi Pemuliaan dan Bioteknologi Tanaman
SEKOLAH PASCASARJANA
INSTITUT PERTANIAN BOGOR
Penguji Luar
Penguji pada Ujian Tertutup:
Dr Ir Asep Setiawan, MS
(Staf pengajar pada Departemen Agronomi, Fakultas Pertanian, Institut Pertanian Bogor)
Dr Ir Rubiyo, MSi
(BPTP Bangka Belitung, Kementerian Pertanian RI)
Penguji pada Sidang Promosi Doktor:
Dr Ir Asep Setiawan, MS
(Staf pengajar pada Departemen Agronomi, Fakultas Pertanian, Institut Pertanian Bogor)
Dr Ir RR Sri Hartati MP
Puji dan syukur penulis panjatkan kepada Tuhan Yang Maha Esa atas segala karunia-Nya sehingga penulis berhasil menyelesaikan Disertasi yang berjudul Keragaman Genetik Plasma Nutfah Kelapa Indonesia dan Penentuan Identitas Kelapa Hibrida Berdasarkan Marka Molekuler.
Terima kasih penulis ucapkan kepada yang dihormati: Dr Ir Darda Efendi MSi, Prof Dr Ir Sudarsono MSc, Prof (R) Dr Ir Hengky Novarianto MS, dan Dr Ir Diny Dinarti MSi selaku pembimbing yang banyak memberi arahan, saran, dan wawasan kepada penulis dalam menyelesaikan penelitian dan penulisan disertasi. Ucapan terima kasih disampaikan juga kepada para Penguji Luar Komisi: Dr Ir Asep Setiawan, MS, Dr Ir Rubiyo, MSi, dan Dr Ir RR Sri Hartati MP; dan kepada Ketua Program Studi Pemuliaan dan Bioteknologi Tanaman, Ketua Departemen Agronomi dan Hortikultura, Dekan Fakultas Pertanian IPB, Dekan Sekolah Pascasarjana IPB, Rektor IPB; serta Ketua Jurusan Biologi FMIPA Unpatti, Dekan FMIPA Unpatti, dan Rektor Universitas Pattimura di Ambon.
Ucapan terima kasih juga penulis sampaikan kepada Kementerian Riset, Teknologi dan Pendidikan Tinggi yang telah memberikan beasiswa pendidikan, Program Kerja sama Kemitraan Penelitian dan Pengembangan Pertanian Nasional (KKP3N) Kementerian Pertanian yang telah memberikan dana penelitian, Kepala Balai Penelitian Palma (Balit Palma) Manado yang telah mengijinkan penulis untuk pengambilan sampel, teknisi Kebun Percobaan Mapanget Balit Palma Manado (Pak Uga, Pak Merdi, Pak Niko) yang banyak membantu pelaksanaan penelitian di lapang.
Terima kasih dan penghargaan penulis sampaikan kepada teman-teman seangkatan 2011 program studi Pemuliaan dan Bioteknologi Tanaman, Fitri Rachmawati, Heni Safitri, Marlina Mustafa, Angelita Puji Lestari, Rini Hermanasari, Agus Zainudin atas dukungan semangat dan persahabatan selama studi. Terima kasih juga penulis sampaikan kepada teman-teman dan staf di Laboratorium Biologi Molekuler Tanaman, Departemen Agronomi dan Hortikultura IPB terlebih khusus untuk Zikril Ilahi SP MSi, Fitria Lukita SP MSi dan Rahmawati Nur Fitriana SSi yang telah banyak membantu selama penelitian di laboratorium. Terimakasih juga kepada semua anggota Persatuan Mahasiswa Maluku dan Pembina PERMAMA, Prof Dr Ir GA Wattimena atas semua dukungan moril, serta kepada pihak lain yang tidak disebutkan.
Ungkapan terima kasih juga disampaikan kepada ayah Herling Pesik, ibu Hetty Waworga (Almah), suami tercinta Pieter Agusthinus Riupassa dan ketiga anak tersayang, Ascendiazorg Riupassa, Ancela Riupassa, Alynne Riupassa serta seluruh keluarga besar, terutama Kel. Mas Sulis Usdoko, atas segala doa, motivasi, bantuan, pengorbanan dan kesabarannya dalam mendampingi penulis selama menempuh pendidikan.
Penulis menyadari masih banyak kekurangan dalam disertasi ini. Semoga disertasi ini bermanfaat bagi kemajuan ilmu pemuliaan dan biologi molekuler tanaman, khususnya tanaman kelapa di Indonesia.
DAFTAR GAMBAR xix
1 PENDAHULUAN 1
1.1 Latar Belakang 1
1.2 Perumusan Masalah 5
1.3 Tujuan Penelitian 5
1.4 Manfaat dan Kebaruan Penelitian 6
1.5 Ruang Lingkup Penelitian 6
1.6 Kerangka Berpikir dan Garis Besar Disertasi 7
2 TINJAUAN PUSTAKA 10
2.1 Keanekaragaman Tanaman Kelapa 10
2.2 Pemuliaan Tanaman Kelapa Indonesia 12
2.3 Marka Molekuler 14
Daftar Pustaka 17
3 PENGEMBANGAN MARKA SNAP BERBASIS GEN WRKY DAN
VALIDASI MENGGUNAKAN MULTIPLEX PCR 23
Abstrak 23
Abstract 24
3.1 Pendahuluan 25
3.2 Bahan dan Metode 27
3.3 Hasil dan Pembahasan 29
3.4 Simpulan 37
Daftar Pustaka 38
4 KERAGAMAN GENETIK KELAPA (Cocos nucifera L.)
INDONESIA BERDASARKAN MARKA SNAP BERBASIS GEN
WRKY, SUS, SACPD DAN ABI3 40
Abstrak 40
Abstract 41
4.1 Pendahuluan 42
4.2 Bahan dan Metode 43
4.3 Hasil dan Pembahasan 47
4.4 Simpulan 59
Daftar Pustaka 60
5 KERAGAMAN GENETIK KELAPA (Cocos nucifera L.)
INDONESIA BERDASARKAN MARKA SSR 63
Abstrak 63
Abstract 64
5.1 Pendahuluan 65
5.2 Bahan dan Metode 66
5.3 Hasil dan Pembahasan 68
5.4 Simpulan 83
Abstrak 87
Abstract 88
6.1 Pendahuluan 89
6.2 Bahan dan Metode 90
6.3 Hasil dan Pembahasan 91
6.4 Simpulan 101
Daftar Pustaka 103
7 IDENTIFIKASI MARKA SSR YANG BERASOSIASI DENGAN
KARAKTER JUMLAH BUAH DAN KOMPONEN BUAH 106
Abstrak 106
Abstract 107
7.1 Pendahuluan 108
7.2 Bahan dan Metode 109
7.3 Hasil dan Pembahasan 109
7.4 Simpulan 115
Daftar Pustaka 116
8 IDENTIFIKASI KELAPA HIBRIDA KOPYOR BERDASARKAN
MARKA SNAP DAN SSR 117
Abstrak 117
Abstract 118
8.1 Pendahuluan 119
8.2 Bahan dan Metode 120
8.3 Hasil dan Pembahasan 122
8.4 Simpulan 131
Daftar Pustaka 132
9 PEMBAHASAN UMUM 133
10 SIMPULAN UMUM 136
DAFTAR PUSTAKA 138
terdapat di database GenBank dan jumlah single nucleotide polymorphism (SNP) berdasarkan hasil multiple sequence analysis
(MSA) 29
3.2 Daftar titik SNP berdasarkan multiple sequence alignment gen WRKY
dari tanaman kelapa 30
3.3 Daftar primer SNAP yang didisain berdasarkan gen WRKY 31 4.1 Daftar primer SNAP gen WRKY, SUS, SACPD, ABI3 yang
digunakan pada penelitian 45
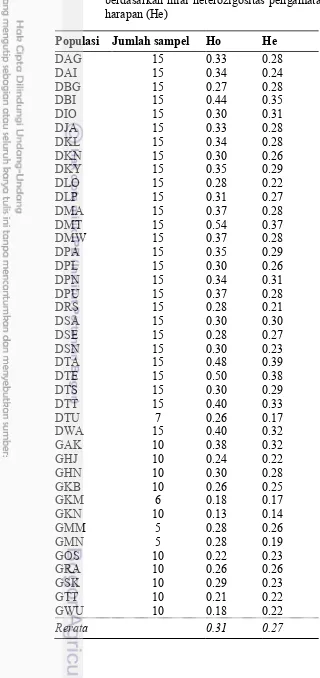
4.2 Jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai PIC pada populasi kelapa Dalam dan Genjah
berdasarkan 16 lokus marka SNAP 48
4.3 Jumlah alel yang diamati pada setiap lokus marka SNAP untuk
populasi kelapa Dalam dan Genjah 49
4.4 Klasifikasi alel berdasarkan frekuensi alel menurut Buchert et al. (1997) dan Marshall dan Brown (1975) pada populasi plasma nutfah
kelapa Dalam dan Genjah 50
4.5 Parameter keragaman genetik pada populasi kelapa Dalam dan Genjah berdasarkan nilai heterozigositas pengamatan (Ho) dan heterozigositas
harapan (He) 53
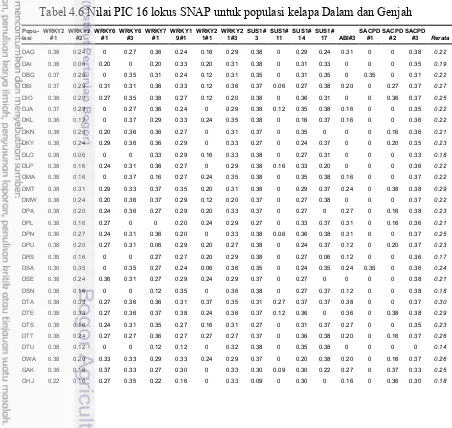
4.6 Nilai PIC 16 lokus SNAP untuk populasi kelapa Dalam dan Genjah 54 5.1 Daftar 20 pasang primer SSR dan suhu penempelannya dalam reaksi
amplifikasi DNA 67
5.2 Jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai PIC pada populasi kelapa Dalam dan Genjah
berdasarkan 20 marka SSR 69
5.3 Jumlah alel yang diamati pada setiap lokus marka SSR untuk populasi
kelapa Dalam dan Genjah 71
5.4 Klasifikasi alel berdasarkan frekuensi alel menurut Buchert et al. (1997) dan Marshall dan Brown (1975) pada populasi plasma nutfah
kelapa 72
5.5 Parameter keragaman genetik pada populasi kelapa Dalam dan Genjah berdasarkan nilai heterozigositas pengamatan (Ho) dan heterozigositas
harapan (He) 75
5.6 Nilai PIC (Polymorphic Information Content) 20 lokus SSR untuk
populasi kelapa Dalam dan Genjah 76
6.1 Data jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai Polymorphic Information Content (PIC) pada populasi tetua (GKN dan DTA) berdasarkan hasil analisis dengan 19
lokus marka SSR 92
6.2 Data jumlah alel (Na), jumlah alel efektif (Ne), tingkat heterozigositas (Ho dan He) dan nilai Polymorphic Information Content (PIC) pada populasi GKN, DTA dan KHINA-1 berdasarkan hasil analisis dengan
19 lokus marka SSR 95
6.3 Jumlah alel yang diamati pada setiap lokus marka SSR untuk populasi
6.5 Parameter keragaman genetik pada populasi GKN, DTA dan KHINA-1 berdasarkan nilai heterozigositas pengamatan (Ho) dan
heterozigositas harapan (He) 98
6.6 Nilai PIC (Polymorphic Information Content) 19 lokus SSR untuk
populasi GKN, DTA dan KHINA-1 99
7.1 Deskripsi data dan uji normalitas karakter jumlah buah dan komponen
buah KHINA-1 110
7.2 Hasil analisis asosiasi marka tunggal dengan 19 primer SSR untuk karakter jumlah buah per tandan dan komponen buah 112 8.1 Primer SNAP gen WRKY yang terpilih untuk identifikasi hibrida
kelapa kopyor 123
3.1 Multiple sequence alignment dari gen WRKY6 pada kelapa menunjukkan adanya posisi Single Nucleotide Polymorphisms (SNP) pada posisi ke-3 di ukuran basa 795 dari sekuens. Variasi nukleotida sebesar berturut-turut untuk T (57.1%) atau C (42.9%) 30 3.2 Reaksi singleplex PCR untuk validasi efektivitas primer SNAP pada
kelapa Kopyor Genjah Pati. Reaksi PCR menggunakan pasangan primer: (1) WRKY6#1 (2) WRKY19#1 (3) WRKY21#3 (4) WRKY2#2 (5) WRKY21#1 (6) WRKY2#1 (7) WRKY6#3 dan (8)
WRKY21#1 dan WRKY19#1 32
3.3 Pengaruh suhu pada multiplex PCR untuk amplifikasi DNA kelapa Kopyor Dalam Kalianda menggunakan marka SNAP. Setiap reaksi PCR, dua pasangan primer masing-masing (WRKY6#1 dan WRKY2#2) dan (WRKY6#1 dan WRKY2#1) digunakan untuk amplifikasi dua lokus SNAP yang berbeda. Amplifikasi DNA menggunakan WRKY6#1 menghasilkan fragmen berukuran 107 pb, WRKY2#1 berukuran 319 pb, sedangkan WRKY2#2 berukuran 260
pb 33
3.4 Optimasi volume primer untuk multiplex PCR pada kelapa Kopyor Dalam Kalianda menggunakan primer WRKY2#2 (260 pb) dan WRKY6#1 (107 pb); volume primer 0.75 µL (A) and 0.375 µL (B);
suhu annealing adalah 55.4oC 34
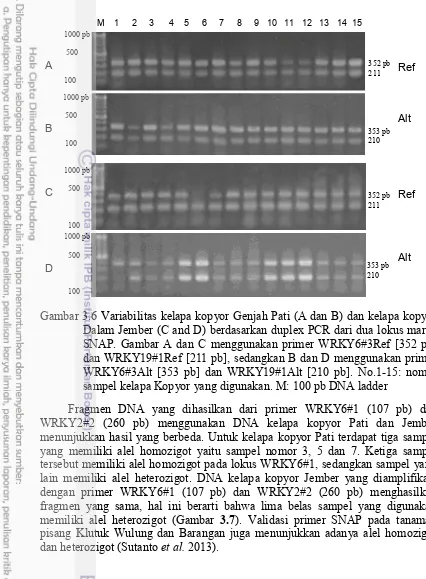
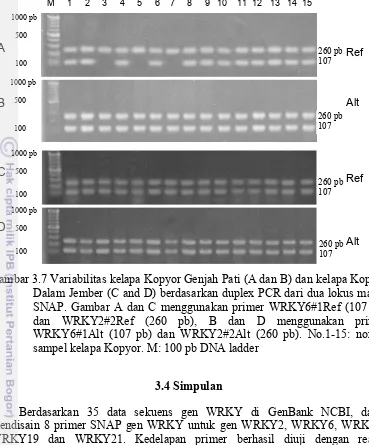
3.5 Amplifikasi primer SNAP untuk multiplex pada kelapa Kopyor Genjah Pati. Reaksi duplex PCR, M2-1: WRKY6#1 Alt (107 pb) dan WRKY2#2 Alt (260 pb); M2-2: WRKY6#1 Alt (107 pb) dan WRKY2#2 Alt (260 pb); M2-3: WRKY6#1 Ref (107 pb) dan WRKY2#1 Ref (319 pb); M2-4: WRKY6#1 Alt (107 pb) dan WRKY2#1 Alt (319 pb). Reaksi triplex PCR menggunakan WRKY6#1 Ref (107 pb), WRKY2#2 Ref (260 pb) dan WRKY2#1 Ref (319 pb), M3-2: WRKY6#1 Alt (107 pb), WRKY2#2 Alt (260 pb) dan WRKY2#1 Alt (319 pb); M3-3: WRKY6#1 Ref (107 pb), WRKY21#1 Ref (272 pb) dan WRKY7#1 (354 pb) Ref; M3-4: WRKY6#1 Alt (107 pb), WRKY21#1 Alt (272 pb) dan WRKY7#1 Alt (354 pb). Fragmen berukuran 354 pb tidak diperoleh 35 3.6 Variabilitas kelapa kopyor Genjah Pati (A dan B) dan kelapa kopyor
Dalam Jember (C and D) berdasarkan duplex PCR dari dua lokus marka SNAP. Gambar A dan C menggunakan primer WRKY6#3Ref [352 pb] dan WRKY19#1Ref [211 pb], sedangkan B dan D menggunakan primer WRKY6#3Alt [353 pb] dan WRKY19#1Alt [210 pb]. No.1-15: nomor sampel kelapa Kopyor yang digunakan. M:
100 pb DNA ladder 36
dan (B) skema foto elektroforesis dan skoring. Pola ini digunakan untuk validasi marka putatif SNAP pada fragmen gen WRKY, SUS,
SACPD dan ABI3 dari tanaman kelapa 46
4.2 Profil alel marka SNAP yang dihasilkan dengan menggunakan pasangan primer WRKY6#3 (352 pb) dan WRKY19#1 (211 pb) masing-masing untuk kelapa Genjah Orange Sagerat (A) dan Dalam
Bali (B). M: DNA marker (100 pb ladder) 47
4.3 Pohon filogenetik kelapa Dalam dan Genjah berdasarkan metode Neighbour Joining menggunakan piranti lunak DARwin berdasarkan
16 lokus marka SNAP 56
4.4 Berdasarkan marka SNAP dan pendekatan metode Evanno populasi kelapa Dalam dan Genjah dibagi menjadi 2 subgrup populasi sesuai
delta K maksimum pada K=2 57
4.5 Estimasi struktur populasi kelapa Dalam (label hitam) dan Genjah (label merah) berdasarkan 16 primer SNAP menggunakan piranti lunak STRUCTURE. Label individu berupa 3 huruf nama aksesi yang
diikuti dengan nomor individu aksesi 58
5.1 Profil alel marka SSR yang dihasilkan menggunakan pasangan primer (A) CNZ 51 dan (B) CnCir 56 masing-masing untuk kelapa Genjah Kuning Nias (1–10) dan Dalam Tenga (11–25). M: DNA marker (100 pb ladder). Nomor dalam foto gel adalah skoring alel dari pita di
sebelah kiri nomor dimaksud 68
5.2 Pohon filogenetik kelapa Dalam dan Genjah berdasarkan metode Neighbour Joining menggunakan piranti lunak DARwin berdasarkan
20 lokus marka SSR 79
5.3 Berdasarkan marka SSR dan pendekatan metode Evanno populasi kelapa Dalam dan Genjah dibagi menjadi 2 subgrup populasi sesuai
dengan delta K maksimum pada K=2 81
5.4 Estimasi struktur populasi kelapa Dalam (label nama populasi berwarna hitam) dan Genjah (label nama populasi berwarna merah) berdasarkan data genotyping 20 lokus marka SSR menggunakan
piranti lunak STRUCTURE 82
6.1 Profil alel untuk marka SSR yang dihasilkan dengan menggunakan pasangan primer (A) CNZ 51 dan (B) CnCir 56 untuk kelapa Genjah Kuning Nias (1–10) dan Dalam Tenga (11–25). M: DNA marker (100 pb ladder). Nomor dalam foto gel adalah skoring alel dari pita di
sebelah kiri nomor dimaksud 91
6.2 Hasil analisis filogenetik dengan metode Neighbour Joining menggunakan piranti lunak DARwin untuk 10 individu Genjah Kuning Nias (GKN) dan 15 individu Dalam Tenga (DTA) berdasarkan keragaman alel pada 19 lokus marka SSR 93 6.3 Profil alel untuk marka SSR menggunakan pasangan primer (A) CNZ
betina 97 6.5 Pohon filogenetik berdasarkan metode Neighbour Joining dari
individu GKN, DTA dan KHINA-1 menggunakan 19 lokus marka SSR. Individu nomor 1–10 adalah tetua GKN, 11–25 adalah tetua
DTA, 26–97 adalah KHINA-1 99
6.6 Perubahan delta K (ΔK) sesuai perbedaan K untuk populasi GKN, DTA dan KHINA-1 yang dihasilkan oleh piranti lunak STRUCTURE 100 6.7 Estimasi struktur populasi berdasarkan data genotyping 19 marka SSR
pada populasi GKN, DTA dan KHINA-1 menggunakan piranti lunak STRUCTURE. Tulisan warna merah adalah tetua GKN, warna hitam adalah tetua DTA, dan warna biru adalah KHINA-1 101 7.1 Grafik frekuensi dari jumlah buah per tandan dan komponen buah
KHINA-1. Sebaran jumlah buah per tandan memenuhi sebaran normal, sedangkan karakter lainnya tidak memenuhi sebaran normal 111 7.2 Variasi alel dan rataan jumlah buah per tandan pada dua lokus SSR
(CNZ 21 dan CNZ 51). Simbol ● menunjukkan nilai rata-rata karakter
dari masing-masing alel 113
7.3 Variasi alel dan rataan berat buah utuh, berat buah tanpa sabut, dan tebal daging pada dua lokus SSR (CnCir 87 dan CnCir 56). Simbol ● menunjukkan nilai rata-rata karakter dari masing-masing alel 114 8.1 Profil alel primer SNAP gen WRKY6#1, WRKY6#3, WRKY19#1,
WRKY21#3 pada persilangan di Pati (A) dan primer WRKY19#1, WRKY21#3 pada persilangan di Lampung (B). R: primer reference; A: primer alternate. M: DNA marker (100 pb ladder) 123 8.2 Profil alel primer CnCir 56 yang membedakan tetua betina dan tetua
jantan pada persilangan di Pati. H1-H4 & K1-K4 adalah tetua betina. DTE, DMT, GSK DBI adalah tetua jantan. M: DNA marker (100 pb
ladder) 124
8.3 Profil alel tetua dan progeni persilangan di Pati. (A) primer SNAP gen WRKY19#1 (210 pb) dan WRKY6#3 (352 pb); K3 adalah tetua betina; GSK adalah tetua jantan; (B) primer SSR CnCir 56; K1, K2, K4, H4 adalah tetua betina; DTE, DMT, DBI adalah tetua jantan 124 8.4 Konstruksi pedigree menggunakan piranti lunak COLONY. Identitas
tetua pada bagian atas dan keturunannya pada bagian bawah. Garis merah (*) menunjukkan tetua jantan dan garis kuning (#)
menunjukkan tetua betina 125
8.5 Pohon filogenetik berdasarkan metode Neighbour Joining dari individu H4, DBI dan progeni. Individu H4xDBI14 sampai dengan H4xDBI25 adalah progeni legitimate yaitu individu yang terletak di antara kedua tetua (H4 dan DBI), sedangkan individu H4xDBI1 sampai dengan H4xDBI13 adalah yang illegitimate 126 8.6 Grafik tingkat keberhasilan persilangan di Pati berdasar jumlah
individu (A) dan persentasi (B) menggunakan tetua jantan DTE, DMT,
betina; DBI adalah tetua jantan. M: DNA marker (100 pb ladder). Nomor dalam foto gel adalah skoring alel dari pita di sebelah kiri
nomor dimaksud 128
8.8 Konstruksi pedigree menggunakan piranti lunak COLONY. Identitas tetua pada bagian atas dan keturunannya pada bagian bawah. Garis merah (*) menunjukkan tetua jantan dan garis kuning (#)
menunjukkan tetua betina 129
8.9 Pohon filogenetik berdasarkan metode Neighbour Joining dari individu 46H, DBI dan progeni. Individu 46HxDBI1 dan 46HxDBI3 adalah progeni legitimate, sedangkan individu lainnya adalah
illegitimate 129
8.10 Grafik tingkat keberhasilan persilangan di Lampung berdasar jumlah individu (A) dan persentasi (B) menggunakan tetua jantan DTE, DMT,
1
PENDAHULUAN
1.1 Latar Belakang
Kelapa (Cocos nucifera L.) adalah tanaman tahunan yang menjadi bagian penting dalam kehidupan masyarakat. Semua bagian tanaman kelapa dapat dimanfaatkan untuk memenuhi kebutuhan ekonomi, sosial, dan budaya sehingga disebut juga tree of life, tree of abundance dan tree of heaven (Rajesh et al. 2013). Kelapa sebagai penghasil minyak sayur dunia (2.4%) berada pada urutan kedelapan setelah kelapa sawit (33.4%), minyak kedelai (26.4%), biji minyak lobak (15.8%), biji bunga matahari (9.6%), dan komoditi lainnya. Indonesia merupakan negara produsen utama kelapa di Asia Pasifik setelah Filipina dan Sri Lanka dengan luas area 3 808 ha, produksi buah kelapa 15.25 juta ton/tahun dan produksi kopra 3 049 juta ton/tahun (Idroes 2012). Berdasarkan data Asian Pacific Coconut Community 2012 permintaan kelapa tidak hanya untuk industri bahan pangan (71%) tetapi juga untuk industri kimia (25%) dan konsumsi rumah tangga (4%). Untuk memenuhi permintaan tersebut perlu dilakukan pengelolaan perkebunan secara baik dan penggunaan bibit yang berkarakter unggul.
Arti penting kelapa bagi masyarakat Indonesia tercermin dari luas areal perkebunan rakyat yang mencapai 98% dari total luas areal kelapa nasional yaitu sekitar 3.86 juta ha. Luas perkebunan kelapa terdiri dari perkebunan rakyat seluas 3.7 juta ha; perkebunan milik pemerintah seluas 4.7 ribu ha dan swasta seluas 66.2 ribu ha. Daerah persebaran tanaman kelapa di Indonesia yang luas berada di Sumatera (34.5%), Jawa (23.2%), Sulawesi (19.6%), Bali, NTB, NTT (8%), Kalimantan (7.5%), Maluku dan Papua (7.5%). Produksi kelapa nasional mencapai 3.29 juta ton/tahun. Pengusahaan kelapa juga membuka kesempatan kerja dari kegiatan pengolahan produk turunan dan hasil samping yang sangat beragam (Direktorat Jenderal Perkebunan 2015). Beberapa industri sangat bergantung pada bahan baku kelapa, seperti industri minyak, santan, tepung kelapa, oleo kimia, nata de coco, cuka, minyak murni, karbon aktif, asap cair, serat sabut, dan industri kreatif pengolahan batang kelapa (Pandin 2009).
Pengusahaan kelapa pada tingkat petani mengalami masalah yaitu rendahnya produktivitas dengan rata-rata 1 ton kopra/ha/tahun, padahal potensi produksi kelapa dapat mencapai 3–5 ton kopra/ha/tahun. Hal ini disebabkan petani belum menggunakan benih kelapa unggul (Novarianto 2008). Varietas kelapa di Indonesia umumnya kelapa Dalam dengan karakter pohon tinggi dan sulitnya mencari tenaga pemanjat saat panen sehingga biaya panen semakin mahal. Sekitar 30% perkebunan kelapa rakyat sudah tua dan kurang produktif. Akibatnya produktivitas kelapa dan pendapatan petani rendah. Beberapa alternatif untuk memenuhi permintaan buah kelapa adalah memaksimalkan pengelolaan perkebunan yang ada melalui peremajaan kelapa, penggunaan varietas kelapa hibrida yang memiliki karakter pohon pendek, cepat berbuah, produksi buah banyak dan kadar minyak tinggi (Novarianto 2010).
digunakan untuk analisis keragaman genetik yang terdiri dari 28 aksesi kelapa Dalam dan 13 aksesi kelapa Genjah.
Pemberian nama aksesi kelapa umumnya disesuaikan dengan daerah asal misalnya kelapa Dalam Mapanget berasal dari desa Mapanget, kabupaten Minahasa, Sulawesi Utara; atau misalnya nama kelapa Genjah Kuning Nias berasal dari pulau Nias, Sumatera Utara. Beberapa nama aksesi lainnya tidak menggunakan nama daerah asalnya karena sudah diberi nama oleh masyarakat setempat seperti kelapa Dalam Takome dan Genjah Raja yang berasal dari Kepulauan Maluku dan kelapa Genjah Salak berasal dari Kalimantan Selatan (Pandin 2009).
Populasi kelapa koleksi Balit Palma telah dikarakterisasi untuk sifat kecepatan berbunga pertama, jumlah buah, kadar kopra dan kadar minyak tinggi. Kelapa yang cepat berbunga adalah kelapa Genjah Salak (GSK), Genjah Kuning Nias (GKN) dan Dalam Sawarna (DSA) berturut-turut adalah 17 bulan, 28 bulan dan 36 bulan. Kelapa berbuah banyak adalah kelapa Dalam Takome (DTE) berkisar 200–230 butir/pohon/tahun dan kelapa GKN sekitar 80–120 butir/pohon/tahun. Kelapa yang memiliki kadar kopra tinggi adalah DMT, DTA, DBI, DPU, DSA dan DRU berkisar 270–300 gram/butir. Kelapa yang mempunyai kadar minyak tinggi adalah DMT berkisar 67–71% (Rompas 1993).
Migrasi suatu individu atau populasi tanaman dari suatu tempat ke tempat lain yang diikuti oleh isolasi geografi dan hibridisasi dapat menyebabkan terjadinya aliran gen. Konsekuensi dari adanya aliran gen antar populasi tanaman akan menyebabkan meningkatnya keanekaragaman genetik, menimbulkan kom-binasi gen baru dan memindahkan kemampuan adaptasi dari satu populasi ke populasi lainnya. Keanekaragaman genetik suatu populasi semakin meningkat dengan adanya mutasi (Nagy 1997). Keanekaragaman genetik yang tinggi dari suatu populasi tanaman sangat bermanfaat sebagai sumber keanekaragaman gen yang memberikan prospek seleksi untuk program pemuliaan tanaman dalam rangka usaha perbaikan produksi tanaman dan memelihara kesinambungan gen. Untuk menghindari terjadinya erosi genetik atau kehilangan karakter-karakter genetik kelapa yang potensial maka perlu dilakukan penyelamatan plasma nutfah kelapa yang diawali dengan pengamatan dan analisis keanekaragaman genetik (Mawikere 2005).
Dalam Bali asal Bali, dan Dalam Palu asal Sulawesi Tengah. Tiga varietas kelapa hibrida yang dihasilkan adalah KHINA-1 (Genjah Kuning Nias x Dalam Tenga), KHINA-2 (Genjah Kuning Nias x Dalam Bali), dan KHINA-3 (Genjah Kuning Nias x Dalam Palu). Dengan pemeliharaan yang intensif, ketiga hibrida mampu berproduksi 4–5 ton kopra/ha/tahun dan telah dilepas oleh Menteri Pertanian pada tahun 1984 (Novarianto 2008).
Pengembangan kelapa selain kelapa hibrida Indonesia, juga terdapat kelapa eksotik yaitu kelapa kopyor yang berpeluang untuk dikembangkan dalam peningkatan pendapatan petani kelapa. Kelapa kopyor bernilai ekonomi tinggi namun produksi pohon kelapa kopyor masih relatif rendah, hanya berkisar 25% buah yang terbentuk per tandan. Pada tahun 2010 sudah dilepas tiga varietas kelapa unggul lokal genjah kopyor asal Pati Jawa Tengah. Sebagai tindak lanjutnya tahun 2013 mulai dirakit varietas kelapa hibrida kopyor baru, melalui kerjasama IPB dan Balit Palma dengan memanfaatkan varietas unggul hasil penelitian Balit Palma yaitu kelapa Dalam Mapanget (DMT), Dalam Takome (DTE), Dalam Bali (DBI) dan Genjah Salak (GSK). Keempat varietas kelapa tersebut memiliki karakter produksi yang unggul meliputi daging buah tebal, jumlah buah banyak, ukuran buah besar dan umur tanaman berbuah cepat.
Perakitan kelapa hibrida memanfaatkan plasma nutfah koleksi Balit Palma telah banyak dilakukan, namun informasi tentang keragaman genetik semua aksesi kurang tersedia lengkap. Penelitian sebelumnya telah dilakukan oleh Matondang (2000) untuk 9 populasi kelapa Dalam asal Maluku; Hayati et al. (2000) untuk keragaman genetik kelapa Genjah Jombang; Ifadatin (2002) dan Roslim et al. (2003) masing-masing menggunakan enam dan tiga populasi kelapa Dalam; dan Hannum et al. (2003) dengan 4 populasi kelapa Genjah, hanya menggunakan jumlah populasi terbatas dan marka molekuler RAPD yang bersifat dominan.
Untuk mempelajari kontrol pewarisan suatu karakter pada tanaman, maka diperlukan silsilah yang lengkap dan jelas asal usul persilangan pada setiap generasi. Pola pewarisan suatu karakter dapat ditelusuri melalui silsilah hasil persilangan dari generasi yang jelas tetuanya. Persilangan antar kelapa Genjah dengan kelapa Dalam menghasilkan kelapa unggul KHINA-1, KHINA-2 dan KHINA-3. Namun, polen yang digunakan merupakan campuran polen dari beberapa pohon kelapa terseleksi dan benih yang diperoleh pada setiap generasi tidak dilabel dengan baik sehingga identitas individu tetua pohon sebagai jantan dari setiap generasi zuriat tidak diketahui. Evaluasi awal secara molekuler untuk kelapa hibrida Indonesia dan bibit hasil persilangan kelapa kopyor dengan beberapa kelapa unggul lainnya perlu dilakukan untuk mengetahui silsilah hasil persilangan dan keberhasilan persilangan. DNA total dari tetua betina, tetua jantan, dan hibrida dijadikan sebagai referensi untuk menganalisis kemurnian bibit (anakan) kelapa hasil persilangan terkontrol.
dibuat berdasarkan jumlah sekuen DNA sederhana yang berulang-ulang. Single Nucleotide Polymorphism (SNP) adalah variasi sekuens tunggal DNA dalam genom, yang biasanya terdapat dua alel di alam sehingga sangat mudah dianalisis. SNP menunjukkan tempat sekuens DNA yang dibedakan oleh satu basa dan berperan penting dalam mekanisme molekuler terkait suatu karakter (Kumar et al. 2012). Berbagai marka molekuler berbasis PCR telah digunakan untuk keragaman genetik kelapa namun informasi genetik berdasarkan marka SSR dan SNAP masih kurang, khususnya untuk populasi kelapa Indonesia. Pada penelitian ini marka tersebut dipilih untuk analisis keragaman genetik dan penentuan identitas kelapa hibrida karena jumlahnya yang melimpah dan terdistribusi merata dalam genom tanaman, dihasilkan cepat melalui teknik PCR, mudah diskor dan informasi sekuen primernya mudah diakses melalui publikasi (Govindaraj et al. 2015).
Marka SNAP berbasis gen WRKY, SUS, SACPD dan ABI3 belum pernah digunakan dalam riset pada populasi kelapa Indonesia. Gen WRKY merupakan faktor transkripsi, secara langsung berperan dalam respon cekaman biotik dan abiotik tanaman seperti senesen, pertumbuhan tanaman, dan pembentukan embrio (Eulgem et al. 2000; Zhang dan Wang 2005). Gen SUS (Sucrose Synthase), SACPD (Stearoyl Acyl Carrier Protein Desaturase) dan ABI (Abscisic Acid Insensitive) berperan pada saat perkecambahan embrio. Selama perkecambahan embrio, energi dan nutrisi diperoleh dari air kelapa (sukrosa) dan endosperma (karbodirat/galaktomanan dan lipid). Gen SUS berperan dalam pembentukan sukrosa sedangkan gen SACPD untuk pembentukan asam lemak, dengan mengubah asam stearat menjadi asam oleat (Kachroo et al. 2008). Gen ABI berperan dalam pergerakan galaktomanan yang terakumulasi di dinding sel endosperma (Potomati dan Buckeridge 2002).
Kemampuan primer SNAP dievaluasi untuk menghasilkan marka SNAP. Evaluasi efektivitas primer SNAP menggunakan teknik multiplex PCR untuk meningkatkan kecepatan dan efisiensi genotyping SNP. Multiplex PCR terdiri dari dua atau lebih pasang primer dalam satu campuran reaksi PCR untuk menghasilkan beberapa ukuran amplikon yang spesifik (Liu dan Wu 2012). Keunggulan teknik multiplex PCR adalah mengurangi biaya, waktu dan tenaga. Namun ada kendala teknik multiplex PCR pada tanaman yaitu ukuran genom tanaman yang besar dan poliploidi, interaksi primer yang tidak diinginkan, dan amplifikasi yang tidak spesifik (Hayden et al. 2008). Selain itu menurut Sheng et al. (2003) masalah lainnya yaitu terbatasnya lokus target yang dapat diamplifikasi dan kesulitan mendapatkan kembali hasil yang sama.
marka DNA. Marka SSR dan SNAP sangat sesuai untuk pembuatan peta pautan genetik karena bersifat ko-dominan, lebih mudah ditransfer dan lebih bermakna dibanding marka yang dominan (Yan et al. 2013).
Integrasi marka molekuler ke dalam program marker assisted selection (MAS) diketahui mampu meningkatkan efektivitas seleksi. Lokus DNA yang berasosiasi dengan komponen produksi dan mempunyai pengaruh genetik yang besar akan bermanfaat untuk meningkatkan efektifitas seleksi (Lebrun et al. 2001). Salah satu cara untuk mengidentifikasi keberadaan QTL yang terkait dengan suatu karakter fenotipik adalah analisis marka tunggal (Champoux et al. 1995; Collard et al. 2005).
Teknik yang merupakan kombinasi seleksi berbasis marka molekuler dengan teknik pemuliaan konvensional merupakan alat bantu strategis yang dapat mempersingkat waktu seleksi, sehingga dapat mempercepat pencapaian tujuan pemuliaan tanaman, untuk menyediakan sumberdaya genetik yang memiliki karakter unggul dalam waktu yang singkat.
1.2 Perumusan Masalah
Plasma nutfah kelapa merupakan sumber sifat keturunan untuk merakit dan mengembangkan kultivar kelapa unggul. Evaluasi keragaman genetik koleksi plasma nutfah kelapa Indonesia perlu dilakukan untuk memperoleh informasi genetik dalam menunjang program pemuliaan dan pelestarian plasma nutfah kelapa Indonesia. Pengembangan marka molekuler untuk analisis keragaman genetik inter dan intra populasi kelapa Indonesia koleksi Balit Palma Manado telah banyak dilakukan namun masih terbatas untuk aksesi tertentu. Untuk memperkaya informasi genetik plasma nutfah kelapa di Balit Palma Manado maka dilakukan analisis keragaman genetik dengan marka SNAP (berbasis gen WRKY, SUS, SACPD, ABI3) dan marka SSR untuk 28 aksesi kelapa Dalam dan 13 aksesi kelapa Genjah.
Kelapa hasil persilangan terkontrol perlu diidentifikasi secara genetik untuk memastikan kebenaran tetuanya, karena kontaminasi serbuk sari dapat berpotensi menjadi masalah. Penggunaan marka molekuler mampu mendeteksi keragaman genetik aksesi dan menduga kebenaran tetuanya. Oleh karena itu dilakukan analisis dengan marka SSR untuk populasi KHINA-1, marka SNAP (berbasis gen WRKY, SUS, SACPD, ABI3) dan marka SSR untuk persilangan kelapa kopyor yang dilakukan di Pati dan Lampung. Individu KHINA-1 yang teridentifikasi sebagai legitimate hybrid dilanjutkan pengamatan di lapang. Parameter yang diamati adalah jumlah buah per tandan dan komponen buah untuk analisis asosiasi antar marka SSR terhadap karakter jumlah buah dan komponen buah.
1.3 Tujuan Penelitian
Tujuan umum penelitian ini adalah mendapatkan kandidat marka molekuler untuk analisis keragaman genetik kelapa Dalam dan Genjah, analisis hubungan genetik kelapa hibrida dan analisis marka yang berasosiasi dengan karakter jumlah buah dan komponen buah. Adapun tujuan khusus penelitian adalah:
penentuan identitas hibrida hasil persilangan beberapa varietas kelapa Indonesia;
2. Menganalisis keragaman genetik intra dan inter populasi plasma nutfah kelapa Indonesia menggunakan marka SNAP (berbasis gen WRKY, SUS, SACPD, ABI3) dan marka SSR;
3. Mendapatkan marka SNAP (berbasis gen WRKY, SUS, SACPD, ABI3) dan marka SSR untuk penentuan identitas hibrida KHINA-1 dan kelapa kopyor; dan 4. Mendapatkan marka SSR yang berasosiasi dengan karakter jumlah buah dan
komponen buah kelapa.
1.4 Manfaat dan Kebaruan Penelitian
Manfaat dan kebaruan penelitian ini adalah:
1. Diperolehnya marka SNAP (Single Nucleotide Amplified Polymorphism) berbasis gen WRKY dengan reaksi multiplex PCR, yang diaplikasikan untuk analisis keragaman genetik plasma nutfah kelapa;
2. Didapatkan metode reaksi multiplex PCR dengan marka SNAP gen WRKY, yang pertama kali digunakan untuk tanaman kelapa;
3. Informasi keragaman genetik 28 aksesi kelapa Dalam dan 13 aksesi kelapa Genjah berdasarkan marka SNAP berbasis gen WRKY, SUS, SACPD dan ABI3, informasi ini bermanfaat untuk menyusun program pemuliaan tanaman kelapa;
4. Informasi keragaman genetik 28 aksesi kelapa Dalam dan 13 aksesi kelapa Genjah berdasarkan marka SSR (Simple Sequence Repeat), informasi ini bermanfaat untuk menyusun program pemuliaan tanaman kelapa;
5. Informasi keragaman tetua yang digunakan untuk persilangan KHINA-1 (Kelapa Hibrida Indonesia-1) dan hubungan genetik antara KHINA-1 dengan tetua berdasarkan marka SSR, informasi ini bermanfaat untuk mendapatkan individu KHINA-1 yang illegitimate dan legitimate hybrid;
6. Diperolehnya marka SSR yang berasosiasi dengan karakter jumlah buah dan komponen buah KHINA-1 sehingga dapat dijadikan sebagai alat untuk seleksi berbasis marka (Marker Assisted Selection atau MAS), yang dapat mempersingkat waktu pemuliaan kelapa;
7. Informasi identitas hibrida kelapa kopyor berdasarkan marka SNAP (berbasis gen WRKY, SUS, SACPD dan ABI3) dan marka SSR, untuk mengetahui keberhasilan persilangan terkontrol yang dilakukan di Pati dan Lampung.
1.5 Ruang Lingkup Penelitian
buah dan komponen buah dari legitimate hybrid KHINA-1. Kelima, mengetahui identitas hibrida dari persilangan kelapa kopyor dengan kelapa unggul nasional (DTA, DTE, DMT, GSK) berdasarkan marka SNAP (berbasis gen WRKY, SUS, SACPD, ABI3) dan marka SSR. Untuk mendefinisikan batasan penelitian ini secara skematis disajikan dalam kerangka penelitian seperti Gambar 1.1.
Gambar 1.1 Diagram alir penelitian
1.6 Kerangka Berpikir dan Garis Besar Disertasi
Keragaman genetik plasma nutfah kelapa yang ada di Indonesia sangat tinggi, sebagian besar populasi tersebut telah dikoleksi oleh Balit Palma Manado. Akan tetapi informasi genetik untuk semua aksesi kelapa belum lengkap. Beberapa penelitian telah dilakukan oleh peneliti Balit Palma Manado meliputi analisis keragaman morfologi dan genetik secara terbatas pada beberapa aksesi kelapa tertentu menggunakan jumlah sampel dan aksesi yang terbatas, misalnya hanya 2–5 individu setiap aksesi. Informasi keragaman genetik tanaman kelapa koleksi Balit Palma menjadi penting dalam strategi pemuliaan tanaman kelapa.
II. Keragaman Genetik
Berdasarkan Marka SNAP & SSR
~ [Bab 4 & 5]
III. Identifikasi Tetua Persilangan KHINA-1 Berdasarkan Marka SSR dan Persilangan Kelapa Kopyor Berdasarkan Marka SNAP & SSR ~ [Bab 6 & 8]
IV. Identifikasi Marka SSR yang Berasosiasi dengan Karakter Jumlah Buah dan Komponen Buah ~ [Bab 7]
I. Pengembangan Marka SNAP Berbasis Gen WRKY ~ [Bab 3]
PLASMA NUTFAH KELAPA INDONESIA
Marka Polimorfik untuk Keragaman Genetik, Penentuan Identitas Hibrida, dan Marka yang Berasosiasi dengan Karakter Jumlah Buah
Selain itu informasi hubungan genetik kelapa hasil persilangan terkontrol perlu diidentifikasi untuk memastikan kebenaran tetuanya, karena kontaminasi serbuk sari dapat berpotensi menjadi masalah. KHINA-1 yang merupakan hasil persilangan antar kelapa Genjah Kuning Nias (GKN) dan Dalam Tenga (DTA) belum pernah dianalisis hubungan genetik dengan tetuanya. Individu KHINA-1 yang tidak memiliki hubungan genetik dengan tetuanya tidak dapat digunakan untuk tahapan seleksi. Setelah diperoleh informasi individu yang diduga merupakan progeninya maka dilanjutkan dengan pengamatan jumlah buah dan komponen buah untuk menentukan marka-marka yang berasosiasi dengan karakter tersebut. Untuk hasil persilangan kelapa koypor di Pati dan Lampung dilakukan analisis pola segregasi antara buah kelapa fenotipe normal dengan buah kelapa kopyor. Jumlah progeni yang digunakan sangat sedikit untuk persilangan yang dilakukan di Lampung karena saat penelitian dilakukan masih sedikit buah yang berkecambah. Dengan demikian untuk melengkapi informasi hubungan genetik plasma nutfah kelapa maka dilakukan serangkaian penelitian terkait aspek pemuliaan dan molekuler untuk menjawab permasalahan yang ada dan disajikan dalam disertasi ini (Gambar 1.1). Bab 1 diuraikan tentang latar belakang, masalah, tujuan, manfaat, kebaruan penelitian, ruang lingkup penelitian, kerangka berpikir dan garis besar disertasi. Bab 2 berisi kajian literatur tentang keanekaragaman dan pemuliaan tanaman kelapa serta marka molekuler. Selain itu juga diuraikan hasil penelitian yang terkait dengan beberapa aspek yang diteliti pada disertasi ini.
Perkembangan teknologi molekuler DNA telah banyak dimanfaatkan untuk mendukung program pemuliaan pada banyak spesies tanaman. Single Nucleotide Amplified Polymorphisms (SNAP) adalah salah satu teknologi marka molekuler untuk mendeteksi putatif SNP menggunakan primer alel spesifik. Pengembangan marka molekuler SNAP berbasis gen WRKY membutuhkan informasi struktur dan keragaman nukleotida dari gen pengendali sifat yang diinginkan. Informasi tersebut didapatkan melalui data di GenBank untuk mendisain primer. Tahapan disain primer SNAP gen WRKY dan validasinya menggunakan reaksi multiplex PCR disajikan di Bab 3. Reaksi multiplex dioptimasi terlebih dahulu untuk mendapatkan hasil yang optimum. Primer-primer yang diperoleh selanjutnya diaplikasikan pada penelitian keragaman genetik kelapa dan identifikasi tetua KHINA-1 dan kelapa kopyor.
Primer SNAP gen WRKY kemudian digunakan untuk analisis keragaman genetik dan struktur populasi tanaman kelapa Dalam dan Genjah dengan reaksi multiplex PCR. Selain berbasis gen WRKY juga digunakan gen SUS, SACPD dan ABI3 untuk melengkapi informasi keragaman genetik kelapa. Total jumlah individu yang digunakan pada penelitian ini yaitu 507 individu sehingga untuk efisiensi penelitian (waktu dan biaya) maka metode reaksi multiplex PCR ini dilakukan. Namun, metode ini dilakukan bila perbedaan ukuran produk PCR lebih dari 30 pasangan basa nukleotida (pb), sehingga untuk gen SUS, SACPD dan ABI3 metode ini tidak dapat digunakan karena ukuran produk PCR masing-masing gen sangat dekat atau kurang dari 30 pb. Topik yang membahas tentang penelitian ini disajikan pada Bab 4. Untuk penggunaan marka SSR, metode reaksi multiplex PCR tidak dilakukan karena tidak dioptimasi.
digunakan merupakan marka yang polimorfik sehingga tidak dilakukan seleksi primer. Secara umum aksesi kelapa Dalam dan Genjah yang diuji menunjukkan perbedaan genetik yang signifikan dengan membentuk sembilan kelompok berdasarkan metode Neighbour Joining. Informasi lengkapnya terdapat di Bab 5.
Selain untuk analisis keragaman genetik kelapa, marka SSR tersebut juga digunakan dalam analisis keragaman genetik tetua yang digunakan untuk persilangan KHINA-1 dan untuk mengevaluasi hubungan genetik tetua dengan hibrida KHINA-1. Sebanyak 19 marka SSR yang digunakan dapat memisahkan tetua betina (Genjah Kuning Nias) dan tetua jantan (Dalam Tenga) dari hibrida KHINA-1 ke dalam lima kelompok yang terpisah. Identitas hibrida KHINA-1 yang illegitimate dan legitimate hybrid dapat diketahui dengan 19 marka SSR. Individu yang legitimate hybrid sebanyak 35 individu dan illegitimate hybrid sebanyak 37 individu dari total 72 individu KHINA-1. Hasil penelitian ini dapat ditinjau pada Bab 6.
Selanjutnya pada Bab 7 diungkapkan penggunaan marka untuk identifikasi adanya asosiasi dengan karakter morfologi. Sebanyak 35 individu legitimate hybrid dilakukan perhitungan jumlah buah dan pengukuran komponen buah (berat buah utuh, berat buah tanpa sabut, berat buah tanpa air, berat daging buah dan tebal daging buah). Data yang diperoleh digabungkan dengan data skoring identitas hibrida KHINA-1 (Bab 6) untuk mengidentifikasi marka SSR yang berasosiasi dengan karakter jumlah buah dan komponen buah. Penggunaan marka molekuler sebagai alat bantu seleksi dalam program pemuliaan tanaman dapat mengidentifikasi lokus-lokus yang berasosiasi dengan karakter yang diinginkan dan dapat dilakukan pada stadia awal pertumbuhan tanaman.
2
TINJAUAN PUSTAKA
2.1 Keanekaragaman Tanaman Kelapa
Tanaman kelapa (Cocos nucifera L.) adalah anggota famili Arecaceae (Palmaceae). Satu spesies kelapa terdiri dari banyak kultivar, akibat adanya penyebaran melalui aliran air, manusia, hibridisasi, mutasi, isolasi geografi dan seleksi sehingga keanekaragaman kelapa menjadi sangat tinggi (Foale 2005).
Waktu permulaan budidaya kelapa masih menjadi perdebatan hingga kini. Chan dan Craig (2006) menyatakan bahwa tanaman kelapa terdistribusi secara luas di daerah tropik dan sub tropik 23° Lintang Utara dan Selatan dari ekuator. Ada tanaman kelapa yang ditemukan di luar lintang ini, tetapi hanya sampai berbunga, buahnya tidak akan berkembang sempurna. Migrasi bangsa Polynesia ke Pasifik 4500 tahun yang lalu menyebabkan seleksi alami tanaman kelapa. Suku Indo-Malaya yang menempati pulau Mikronesia, pedagang dari Arab dan Melayu menyebarkan kelapa ke India, Sri Lanka dan Afrika Timur sekitar 3000 tahun yang lalu. Kelapa diintroduksi ke Afrika Barat dan Karibia (termasuk pantai Atlantik Karibia) selama abad ke-16 oleh penjelajah Eropa. Fakta yang lain mendukung bahwa kelapa berasal dari Asia Tenggara (Burma, Filipina, Malaysia, Indonesia, Cina Selatan, Thailand dan Vietnam). Pada negara-negara ini tanaman kelapa lebih banyak dikembangkan dan mempunyai produktivitas serta keanekaragaman yang tinggi.
Perpindahan suatu individu atau populasi tanaman dari satu tempat ke tempat yang lain diikuti oleh isolasi geografi dan hibridisasi dapat menyebabkan terjadinya aliran gen. Aliran gen antar populasi tanaman dapat meningkatkan keanekaragaman karakter genetik, menimbulkan kombinasi gen baru, dan memindahkan kemampuan beradaptasi di suatu tempat dari satu populasi ke populasi yang lain (Nagy 1997).
Untuk menghindari terjadinya erosi genetik atau kehilangan karakter-karakter genetik kelapa yang potensial maka perlu dilakukan penyelamatan plasma nutfah yang masih tersisa, diawali dengan pengamatan keanekaragaman karakter genetik. Keanekaragaman karakter genetik akibat migrasi, isolasi geografi, seleksi, hibridisasi dan mutasi dapat terjadi dalam populasi kelapa. Spesies Cocos nucifera L. memiliki berbagai jenis kultivar yang diberi nama sesuai tempat tumbuhnya, warna buah dan umur tanaman kelapa. Keanekaragaman komposisi genetik yang tinggi dari suatu populasi tanaman sangat bermanfaat sebagai sumber keragaman gen untuk program pemuliaan tanaman dan pemeliharaan kesinambungan sumber gen.
bunga jantan dan bunga betina. Kedua, autogami tak langsung dengan ciri waktu kemasakan bunga betina pendek, tidak terjadi kesamaan waktu kemasakan bunga betina dan bunga jantan pada seludang yang sama tetapi terjadi kesamaan waktu kemasakan bunga betina dan bunga jantan pada seludang berikutnya. Ketiga, autogami langsung dengan ciri kemasakan bunga betina lama, terdapat kesamaan waktu kemasakan bunga betina dan bunga jantan pada manggar yang sama. Keempat, autogami semi langsung dengan ciri waktu kemasakan bunga betina pendek, terdapat kesamaan waktu kemasakan bunga betina dan bunga jantan pada manggar yang berbeda (Sangare et al. 1978).
Secara umum tanaman kelapa yang tumbuh secara lokal diklasifikasikan ke dalam tiga tipe yaitu, tipe kelapa Dalam (Typica), tipe kelapa Genjah (Nana) dan tipe intermediate (Aurantica) antar Typica dan Nana. Tipe Aurantica sangat jarang dikembangkan. Pengelompokan tersebut berdasarkan pola penyerbukan, karakter morfologi seperti tinggi pohon, warna buah dan perbedaan kuantitatif dan kualitatif dalam komponen buah (Liyanage dan Corputy 1976). Pada tahun 1978 diusulkan pengelompokan kelapa Dalam menjadi Niu kafa yang berkembang secara alami dan disebarkan oleh arus laut dan Niu vai yang berkembang sebagai hasil seleksi dari Niu kafa melalui penanaman dan disebarkan oleh manusia (Harries 1978).
Berdasarkan kecepatan berbunga pertama, tanaman kelapa digolongkan atas 2 tipe yaitu tipe Dalam dan Genjah. Kelapa tipe Dalam umumnya memiliki batang yang tinggi, bagian pangkal membengkak (disebut bole); daun dengan mahkota terbuka penuh 30–40 daun, panjang daun 5–7 meter; berbunga pertama lambat yaitu 7–10 tahun setelah tanam; buah masak sekitar 12 bulan setelah penyerbukan; umur tanaman dapat mencapai 80–90 tahun; lebih toleran terhadap macam-macam jenis tanah dan kondisi iklim, kualitas kopra dan minyak, serta sabut umumnya baik, menyerbuk silang (Santos et al. 1996).
Kelapa tipe Genjah pada umumnya memiliki batang pendek berkisar 12 meter dan agak kecil, tidak memiliki bole, panjang daun berkisar 3–4 meter, berbunga pertama berkisar 3–4 tahun setelah tanam, buah masak berkisar 11–12 bulan sesudah penyerbukan, umur tanaman dapat mencapai 35–40 tahun, kualitas kopra dan minyak serta sabut kurang baik, menyerbuk sendiri (Foale 1992).
Tanaman kelapa di Indonesia terdiri atas beberapa jenis kelapa yang dianggap unik yaitu 1) kelapa Hibrida, jenis kelapa hasil persilangan antara tipe kelapa Genjah dan kelapa Dalam dengan sifat lebih unggul dari kedua tetuanya; 2) kelapa Kopyor (Jawa dan Sumatera), daging buahnya sangat lunak dan mudah lepas dari tempurungnya; 3) kelapa Kenari (Maluku) berada secara individu dalam suatu populasi memiliki struktur daging buah berbeda dari kelapa normal, rasanya sangat renyah sehingga sangat cocok untuk dikonsumsi sebagai kelapa muda; 4) kelapa Sabut Merah (Gorontalo dan NTT), terdapat warna merah muda pada pangkal buah; 5) kelapa Bercabang (Sulut dan Sulteng), anakan muncul dari pangkal batang pohon pertama; 6) kelapa Dalam Mamuaya (Sulut), Dalam Palapi (Sulteng), Dalam Dobo yang berbuah besar, sabut tipis dengan potensi hasil 4–5 ton kopra/ha, Dalam Santongbolang (Sulut) berbuah banyak yaitu 60–100 buah/tandan (Novarianto dan Miftahorachman 2000).
Rp. 20 000–Rp. 30 000/butir atau 10 kali lebih mahal dibanding buah kelapa normal. Kelapa kopyor adalah kelapa mutan asli Indonesia dengan endosperma bergumpal-gumpal yang lepas dari tempurungnya. Endosperma yang tidak normal tersebut sangat disukai oleh konsumen tetapi buah kelapa kopyor tidak selalu tersedia di pasar (Maskromo et al. 2015).
Kelapa Makapuno asal Filipina juga mempunyai endosperma yang abnormal sehingga bentuknya seperti jeli, jika buahnya terlalu tua sebagian dari endosperma akan terlarut dalam air kelapa. Karakteristik mutan pada kelapa makapuno tersebut dilaporkan dapat diturunkan secara genetik dari tetua ke progeninya (Santos 1999).
Asal usul kelapa kopyor memiliki informasi yang kurang, sampai saat ini. Secara umum tanaman kelapa berbuah kopyor diketahui ada dan tersebar di pulau Jawa dan Sumatera. Di pulau Jawa, kelapa kopyor banyak ditemukan di banyak kabupaten di provinsi Jawa Tengah, di daerah Jember dan Sumenep, provinsi Jawa Timur dan di Tangerang provinsi Banten. Di pulau Sumatera, kelapa kopyor berada di Kalianda, provinsi Lampung (Novarianto dan Miftahorachman 2000).
Keanekaragaman tanaman kelapa yang tersedia merupakan materi dasar untuk program pemuliaan sehingga kelestariannya menjadi hal yang utama agar tidak terjadi erosi genetik atau kehilangan sumber gen yang potensial. Penyebab erosi genetik pada pertanaman kelapa yaitu peningkatan jumlah penduduk sehingga terjadi pengalihan fungsi lahan kebun kelapa menjadi perumahan, penggantian tanaman kelapa dengan komoditas lain yang dianggap lebih bernilai ekonomis, organisasi di bidang konservasi genetik kelapa belum memadai (Pandin 2009).
2.2 Pemuliaan Tanaman Kelapa Indonesia
Koleksi plasma nutfah merupakan usaha konservasi sumber daya alam dan keperluan utama dalam kegiatan pemuliaan tanaman, sebagai sumber bahan dasar untuk perakitan menghasilkan varietas unggul dengan nilai ekonomi yang tinggi. Manfaat plasma nutfah adalah menyediakan materi genetik tanaman tertentu yang kemungkinan akan hilang di dalam populasi tanaman budidaya sehingga karakter genetik tetap tersedia.
Pemuliaan tanaman merupakan suatu metode pemanfaatan keragaman genetik plasma nutfah secara sistematis untuk menghasilkan varietas baru yang lebih baik dari sebelumnya. Kegiatan pemuliaan kelapa membutuhkan waktu pembungaan pertama yang sangat lama yaitu sekitar 4–10 tahun. Pengujian satu keturunan tunggal membutuhkan minimal 70 pohon atau lahan setara 0.5 ha untuk masa penelitian minimal selama 10 tahun setelah tanam. Selain itu jumlah zuriat yang bisa diperoleh dari satu tetua betina dalam setahun sangat rendah. Pada kondisi ini diperlukan metode yang dapat meningkatkan efisiensi untuk pemuliaan tanaman kelapa (Pandin 2000).
Karakterisasi adalah proses deskripsi suatu karakter atau kualitas suatu individu. Karakterisasi sumber genetik merujuk pada proses identifikasi atau diferensiasi aksesi. Terminologi yang disepakati oleh GenBank dan germplasm management, karakterisasi adalah deskripsi karakter yang pewarisannya tinggi, mudah dilihat secara kasat mata, dan terekspresi stabil pada berbagai lingkungan ([IPGRI] The International Board of Plant Genetic Resources Institute 2003).
Pemuliaan tanaman kelapa di Indonesia telah dilakukan sejak 1911/1912 saat pemerintah Belanda mengoleksi 240 buah kelapa yang berasal dari kelapa Dalam terpilih di pulau Jawa. Pada tahun 1927 pemerintah Belanda mendirikan
Klapper Proef Station di Manado dengan kegiatan eksplorasi, seleksi dan koleksi plasma nutfah kelapa yang berada di Desa Mapanget oleh seorang ahli agronomi yaitu Dr. P.L.M. Tammes. Seleksi dilakukan berdasarkan jumlah produksi per pohon dan terseleksi 100 pohon, dari setiap pohon diambil buahnya untuk ditanam di Kebun Percobaan Mapanget. Setiap nomor tetua ditanam dalam bentuk barisan sebanyak 10 pohon sehingga seluruhnya berjumlah 1 000 pohon (Reyne 1948). Populasi ini dikenal sebagai koleksi Tammes. Pada tahun 1955, seorang pemulia tanaman berkebangsaan Jerman yaitu Dipl. Ing. A.F. Ihne melakukan seleksi negatif berdasarkan produksi di setiap pohon dari koleksi Tammes dan didapatkan 42 nomor pohon terpilih. Hasil seleksi negatif tersebut selanjutnya diseleksi lagi dan didapatkan 29 nomor pohon yang digunakan sebagai tetua dalam persilangan terbuka dan buatan untuk uji keturunan. Program pemuliaan kelapa dilanjutkan pada tahun 1973/1974 oleh pemulia tanaman Balit Palma dan Dr. Liyanage pemulia dari UNDP/FAO. Mereka melakukan survei pada beberapa provinsi di Indonesia untuk mengidentifikasi karakter tanaman kelapa yang cocok untuk ditanam di Kebun Percobaan Mapanget (Novarianto et al. 1998).
Kelapa kopyor Indonesia dengan endosperma abnormal, menurut Sudarsono
et al. (2012) diduga dikendalikan oleh gen mutan resesif “k” sedangkan endosperma normal dikendalikan oleh gen dominan “K”. Secara genetik, segregasi genotipe tanaman kelapa kopyor dan fenotipe endosperma memiliki tiga dugaan. Pertama, buah kelapa kopyor (kk) mempunyai embrio zigotik dengan genotipe homozigot “kk” dan endosperma genotipe homozigot “kkk”. Buah kelapa kopyor tidak akan berkecambah secara alami karena abnormalitas endosperma sehingga buahnya tidak dapat dijadikan sebagai bibit. Untuk mendapatkan bibit dari buah kelapa kopyor, embrio zigotik harus dikulturkan secara in vitro. Setelah berkecambah, embrio tersebut akan berkembang menjadi bibit homosigot “kk”. Keunggulan bibit ini adalah kemampuan menghasilkan buah kopyor 100% per tandannya, dengan asumsi tidak ada pohon kelapa berbuah normal (genotipe KK atau Kk) di sekitarnya yang memungkinkan terjadi persilangan. Kelemahan bibit ini, di pasaran bibit kelapa kopyor hasil kultur embrio zigotik masih belum terjangkau oleh petani karena harganya sangat mahal (Rp. 350 000/bibit dan pesanan minimal 100 bibit).
produksi yang mahal. Harga jual lebih murah Rp. 35 000, akan tetapi hetero-zigositas gen pengendali sifat kopyor, dapat menghasilkan buah kopyor dari bibit kelapa kopyor heterozigot bervariasi antara 20–50% per tandan tergantung tipe kelapa (Dalam atau Genjah) dan adanya tegakan kelapa normal dapat menyumbangkan serbuk sari normal (K) di sekitar kelapa kopyor heterozigot. Adanya kelapa berbuah normal akan menurunkan persentase pembentukan buah kopyor.
Ketiga, buah kelapa normal (KK) mempunyai embrio zigotik dengan genotipe homozigot “KK” dan endosperma normal dengan genotipe homozigot “KKK”. Buah kelapa ini dapat berkecambah secara alami karena endosperma normal sehingga buah tidak dapat dijadikan bibit. Bibit tersebut akan menghasilkan kelapa normal homozigot, yang tidak pernah menghasilkan buah kopyor.
Bibit kelapa kopyor heterozigot “Kk” dapat dihasilkan melalui persilangan terkontrol antara induk betina heterozigot “Kk” dengan induk jantan homozigot “kk” (hasil kultur in vitro). Hasilnya 50% buah kelapa kopyor (embrio zigotik “kk” dan endosperma “kkk”) dan 50% buah kelapa normal heterozigot (embrio zigotik “Kk” dan endosperma “Kkk atau “KKk”).
Program pemuliaan tanaman kelapa di Indonesia dibagi atas: 1) program jangka pendek terutama ditujukan untuk menghasilkan benih yang berkualitas baik, yang diharapkan produksinya untuk memenuhi kebutuhan bahan baku industri kelapa; dan 2) program jangka panjang bertujuan untuk memperbaiki populasi tanaman kelapa. Populasi ini diharapkan mampu memproduksi kopra/ha/thn tinggi, lebih cepat berbuah, kadar minyak tinggi, resisten terhadap penyakit busuk pucuk dan gugur buah kelapa, pemeliharaan sederhana, toleran terhadap lahan gambut atau pasang surut, tahan kekeringan serta kadar asam laurat dan protein tinggi (Novarianto 2008).
Penyediaan benih dan kebun kelapa merupakan bagian penting dalam upaya perakitan kelapa. Program penyediaan benih jangka pendek dapat dilakukan melalui pemanfaatan kelapa Dalam unggul lokal, sedangkan program pembangunan kebun induk kelapa untuk jangka panjang dilaksanakan dengan membangun Kebun Induk Kelapa Dalam Komposit (KIKD Komposit). Untuk mempercepat seleksi varietas kelapa unggul di setiap daerah dan sebagai tetua dalam perakitan kelapa Dalam komposit, dapat dilakukan dengan memanfaatkan teknik molekuler (Novarianto 2008).
2.3 Marka Molekuler
Marka molekuler dapat menunjukkan perbedaan genetik antara individu organisme atau spesies. Umumnya, marka tidak mewakili gen target pembeda komposisi genetik, tetapi bertindak sebagai tanda (Collard et al. 2005). Variasi alel dalam genom dari spesies yang sama diklasifikasikan dalam tiga kelompok utama berdasarkan perbedaan jumlah tandem repeats pada sebagian lokus yaitu
Marka SSR berbasis PCR digunakan untuk DNA fingerprinting, pemetaan gen, analisis parental, studi keragaman genetik dan populasi genetik. Marka molekuler ini memiliki polimorfisme yang tinggi, kodominan dan dapat membedakan berbagai alel pada spesies tanaman karena variasi jumlah unit/motif berulang, yang terdiri atas 1–6 pb sekuens DNA, seperti pengulangan dinukleotida [contohnya (AT)n atau (CT)n] dan trinukleotida [contohnya (ATT)n], yang tersebar
terutama pada daerah antara gen-gen dan daerah nonkoding dalam genom (Meksem dan Kahl 2005).
Marka SSR sangat sering digunakan dalam penelitian tanaman kelapa, yang pertama kali dikembangkan oleh Rivera (1999) menguji 41 lokus SSR pada 20 aksesi kelapa. Teulat et al. (2000) menggunakan 37 lokus SSR yang dikembangkan (Rivera 1999) untuk menentukan keragaman genetik 31 individu tanaman dari 14 populasi yang berbeda secara geografi. Selanjutnya penggunaan marka SSR lebih banyak diaplikasikan untuk analisis keragaman genetik tanaman kelapa di antaranya yang dilakukan