PENELUSURAN KEASLIAN IKAN TUNA (
Thunnus
sp.)
DAN PRODUK OLAHANNYA MELALUI TEKNIK
MOLEKULER DNA
BARCODING
NURING WULANSARI
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PERNYATAAN MENGENAI TESIS DAN
SUMBER INFORMASI SERTA PELIMPAHAN HAK CIPTA
Dengan ini saya menyatakan bahwa tesis berjudul “Penelusuran Keaslian
Ikan Tuna (Thunnus sp.) dan Produk Olahannya melalui Teknik Molekuler DNA Barcoding” adalah benar karya saya dengan arahan dari komisi pembimbing dan belum diajukan dalam bentuk apa pun kepada perguruan tinggi manapun. Sumber informasi yang berasal atau dikutip dari karya yang diterbitkan maupun tidak diterbitkan dari penulis lain telah disebutkan dalam teks dan dicantumkan dalam Daftar Pustaka di bagian akhir tesis ini.
Dengan ini saya melimpahkan hak cipta dari karya tulis saya kepada Institut Pertanian Bogor.
Bogor, Mei 2016
RINGKASAN
NURING WULANSARI. Penelusuran Keaslian Ikan Tuna (Thunnus sp.) dan Produk Olahannya melalui Teknik Molekuler DNA Barcoding. Dibimbing oleh NURJANAH dan MALA NURILMALA.
Kasus mislabeling atau ketidaksesuaian dengan label pada produk perikanan telah banyak dilaporkan di beberapa negara, namun di Indonesia belum banyak dilaporkan. Pengujian keaslian ikan atau autentikasi diperlukan untuk meyakinkan konsumen tentang keakuratan pelabelan. DNA barcoding merupakan urutan pendek DNA dari wilayah standar pada genom yang digunakan untuk identifikasi spesies. Penelitian ini bertujuan mengidentifikasi keaslian ikan tuna dan olahannya melalui teknik molekuler DNA barcoding menggunakan DNA mitokondria cyt b dan COI (cytochrome c oxidase subunit I) sehingga dapat digunakan untuk mencegah adanya pemalsuan/ketidaksesuaian label dan economic fraud.
Tahapan penelitian terdiri atas identifikasi morfologi, pengukuran proporsi tuna segar utuh, pengukuran komposisi proksimat, identifikasi berbasis protein, dan identifikasi molekuler. Sampel terdiri atas 5 sampel tuna segar utuh dan 22 sampel olahan tuna berupa tuna steak, sushi tuna, bakso tuna, abon tuna dan tuna kaleng.
Identifikasi secara morfologi menunjukkan ke lima sampel tuna segar utuh teridentifikasi sebagai tuna sirip kuning (Thunnus albacares). Komposisi rendemen daging merah ikan tuna 15,88%, daging putih 32,90%, sedangkan rendemen jeroan, kepala, tulang dan kulit masing-masing berkisar 8,19%; 22,87%; 16,03%; dan 4,14%. Komposisi protein tuna segar utuh 25,57%, tuna steak 22,37% dan tuna kaleng dalam minyak 14,91%. Identifikasi berbasis protein menunjukkan profil pola protein tuna segar utuh dan tuna steak terlihat lebih tebal dan jelas, untuk sushi tuna walaupun tidak tebal, namun pola proteinnya sama dengan tuna segar utuh dan tuna steak, sedangkan pola protein olahan bakso tuna, abon tuna dan tuna kaleng terlihat namun tipis.
Hasil desain primer dengan gen cyt b menghasilkan urutan basa nukleotida untuk foward 5‟GGAATAGGGAGAAGTAGAGGACG3‟ dan reverse
5‟CTYCTATCCGCAGTCCCATATGTYGG3‟ dengan panjang fragmen 620 bp, sedangkan menggunakan gen COI dihasilkan primer dengan urutan basa nukleotida untuk foward dan reverse masing-masing
5‟GTGCATGAGCTGGAATAGTTG3‟ dan 5‟CAGGGTCGAAGAAGGTTG3‟
dengan panjang fragmen 583 bp.
ditandai dengan tidak adanya pita target, hal ini diduga bahwa sampel tuna kaleng dalam minyak (Ka3) tidak berisi ikan tuna.
Identifikasi spesies berdasarkan analisis BLAST menunjukkan bahwa tuna segar (Ts1) baik menggunakan primer cyt b dan COI teridentifikasi sebagai tuna mata besar (Thunnus obesus) dengan tingkat homologi sebesar 99%, namun secara morfologi teridentifikasi sebagai T. albacares, hal ini disebabkan oleh ukuran yang masih kecil, yaitu panjang cagak (fork length) antara Ts1 dan Ts2 kurang dari 40 cm, sehingga sering terjadi kesalahan identifikasi. Semua olahan
tuna baik menggunakan gen cyt b maupun COI teridentifikasi sebagai T. albacares dengan homologi berkisar 98-100%, kecuali untuk tuna steak (St5)
SUMMARY
NURING WULANSARI. Authentication on Tuna Fish (Thunnus sp.) and Its
Processed Product‟s through Molecular DNA Barcoding Technique. Supervised
by NURJANAH and MALA NURILMALA.
Mislabeling of fishery products has been reported many times in several countries, but yet in Indonesia. Fishery product investigation or authentication is needed for ensuring consumer trust on labelling accuracy. DNA barcoding is a short DNA sequence from a standard part of the genome that is used to identify
species. This study aimed to identify tuna fish and its processed product‟s
authenticity through molecular DNA barcoding technique using mitochondrial DNA cyt b and COI (cytochrome c oxidase subunit I) that can be used to prevent mislabeling and economic fraud.
Research methods consisted of morphological identification, tuna‟s proportion measurement from fresh whole, proximate composition measurement, identification based on protein, and identification based on DNA. Samples were consisted of 5 fresh whole tuna fish and 22 processed tuna samples such as steak, sushi, meatballs, shredded tuna, and canned tuna.
Morphological identification showed that all five fresh whole tuna samples were identified as yellowfin tuna (Thunnus albacares). Yield composition of the red meat of tuna fish was 15.88% and the white meat was 32.90%, while yield composition of viscera, head, bone, and skin were 8.19%; 22.87%; 16.03%; and 4.14%, respectively. Protein composition of fresh whole tuna was 25.57%, 22.37% for tuna steak, and 14.91% for canned tuna in oil. Protein-based identification indicates that the protein pattern profile of fresh whole tuna and tuna
steak‟s protein appeared thicker and clearer. Although the protein pattern of tuna sushi was not thick, its protein pattern was similar to fresh whole tuna and tuna steak. The protein patterns of processed tuna such as meatballs, shredded tuna, and canned tuna were appear although a little.
Primer design result with cyt b gene generated nucleotide base sequence
for foward as 5‟GGAATAGGGAGAAGTAGAGGACG3‟ and reverse as 5‟CTYCTATCCGCAGTCCCATATGTYGG3‟ with amplicon length 620 bp, while COI gene generated 5‟GTGCATGAGCTGGAATAGTTG3‟ and 5‟CAGGGTCGAAGAAGGTTG3‟ for each foward and reverse nucleotide base sequence with amplicon length 583 bp.
Species identification based on BLAST analysis indicates that fresh whole tuna (Ts1) both using cyt b and COI primers were identified as bigeye tuna (T. obesus) with homology levels of 99%, but they were morphologically identified as T. albacares. This was because of the small size of tuna which fork length between Ts1 and Ts2 was less than 40 cm, therefore misidentification could be frequently occured. All processed tuna products, either using cyt b gene or COI gene were identified as T. albacares with homological range between 98-100%, except for tuna steak (St5) that was identified as T. obesus with 99% homological rate. Phylogenetic tree construction with cyt b primer showed that fresh whole tuna (Ts2, Ts3, and Ts5) samples, as well as processed tuna formed a clade with T. albacares, while fresh whole tuna (Ts1) and tuna steak (St5) formed a clade with T. obesus, except for Ts4 fresh whole tuna that formed its own category. Phylogenetic tree construction with primary COI showed that fresh whole tuna (Ts2, Ts3, Ts4, and Ts5) samples, as well as processed tuna formed a clade with T. albacares, while fresh whole tuna (Ts1) and tuna steak (St5) formed a clade with T. obesus. These results show that BLAST analysis was compatible with branch characteristics formed by phylogenetic tree.
© Hak Cipta Milik IPB, Tahun 2016
Hak Cipta Dilindungi Undang-Undang
Dilarang mengutip sebagian atau seluruh karya tulis ini tanpa mencantumkan atau menyebutkan sumbernya. Pengutipan hanya untuk kepentingan pendidikan, penelitian, penulisan karya ilmiah, penyusunan laporan, penulisan kritik, atau tinjauan suatu masalah; dan pengutipan tersebut tidak merugikan kepentingan IPB
PENELUSURAN KEASLIAN IKAN TUNA (
Thunnus
sp.)
DAN PRODUK OLAHANNYA MELALUI TEKNIK
MOLEKULER DNA
BARCODING
NURING WULANSARI
Tesis
sebagai salah satu syarat untuk memperoleh gelar Magister Sains
pada
Program Studi Teknologi Hasil Perairan
SEKOLAH PASCASARJANA INSTITUT PERTANIAN BOGOR
PRAKATA
Puji dan syukur penulis panjatkan kehadirat Allah SWT atas rahmat dan karuniaNya, sehingga penyusunan tesis ini dapat terselesaikan. Judul tesis ini
adalah “Penelusuran Keaslian Ikan Tuna (Thunnus sp.) dan Produk Olahannya melalui Teknik Molekuler DNA Barcoding”. Penelitian ini didanai Ditjen Pendidikan Tinggi melalui penelitian Hibah Kompetensi tahun 2015 atas nama
Dr Mala Nurilmala, SPi, MSi dengan judul “Identifikasi Hasil Perikanan melalui
Teknik Molekuler DNA (DNA Barcoding) dalam Rangka Mencegah Pemalsuan
dan Meningkatkan Keamanan Pangan”
Penulis mengucapkan terimakasih yang sebesar-besarnya kepada:
1. Prof Dr Ir Nurjanah, MS dan Dr Mala Nurilmala, SPi, MSi selaku dosen pembimbing yang telah sabar memberikan bimbingan, masukan, dukungan dan semangat kepada penulis sehingga penulis dapat menyelesaikan tesis ini. 2. Dr Tati Nurhayati, SPi, MSi selaku dosen penguji luar komisi yang telah
memberikan, masukan, dan arahan sehingga penulis mampu menyelesaikan tesis ini.
3. Dr Asadatun Abdullah, SPi, MSM, MSi selaku perwakilan tim gugus kendali mutu atas saran serta masukannya kepada penulis.
4. Prof Dr Ir Joko Santoso, MSi selaku Ketua Departemen Teknologi Hasil Perairan, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor. 5. Dr Ir Wini Trilaksani, MSc selaku Ketua Program Studi Teknologi Hasil
Perairan, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor. 6. Suami (Dhafing Agung Nugroho, S.Pi), anak-anak tercinta (Kayana Indira
Nareswari dan Naya Rania Paramesti) serta kedua orang tua (H. Djumono dan Hj. Sri Sugiyanti) atas doa dan dukungan baik materil maupun spiritual kepada penulis.
7. Teman-teman satu tim penelitian molekuler (Nur Fajrin Nisa, Deden Maulid Yusman, Yustin Widyastuti, Lita Ayu, Hilda Lulu) serta Paqih atas bantuan yang tulus.
8. Teman-teman Pascasarjana THP 2013 atas dukungan, semangat dan doa yang diberikan kepada penulis.
9. Badan Pengembangan Sumberdaya Manusia Kelautan dan Perikanan KKP yang telah membiayai penelitian ini melalui beasiswa Tugas Belajar Tahun 2013.
10. Semua pihak yang telah membantu penyelesaian tesis ini.
Penulis menyadari bahwa masih terdapat kekurangan dalam penyusunan tesis ini, oleh karena itu jika terdapat kesalahan penulis memohon maaf yang sebesar-besarnya. Penulis sangat mengharapkan kritik dan saran yang membangun sehingga bermanfaat untuk penyelesaian tesis ini. Semoga tesis ini bermanfaat.
DAFTAR ISI
Identifikasi Berbasis Protein (Profil Protein) 9
Identifikasi Berbasis DNA 9
Desain Primer cyt b dan COI 9
Panjang, Berat dan Ciri Fisik Ikan Tuna Segar Utuh 13
Proporsi Tuna Segar Utuh 13
Komposisi Kimia Tuna Segar, Tuna Steak dan Tuna Kaleng 14
Profil Protein (SDS-PAGE) 15
Hasil Isolasi, Pengukuran Konsentrasi dan Kemurnian DNA 17 Hasil Amplifikasi DNA dan Elektroforesis Hasil PCR 19
Hasil Analisis Bioinformatika 22
Pohon Filogenetik 23
4 SIMPULAN DAN SARAN 26
Simpulan 26
Saran 26
DAFTAR PUSTAKA 27
LAMPIRAN 31
DAFTAR TABEL
1. Koleksi sampel tuna segar utuh dan olahan tuna 6
2. Tahapan PCR amplifikasi 11
3. Proporsi ikan tuna (%) 14
4. Pengukuran proksimat ikan tuna dan olahan tuna 15
5. Hasil oligoevaluator desain primer ikan tuna 17
6. Konsentrasi dan kemurnian DNA 18
7. Kasus mislabeling ikan tuna menggunakan gen target COI 22
8. Identifikasi spesies dengan analisis BLAST 22
DAFTAR GAMBAR
1. Diagram alir penelitian 5
2. Skema morfometrik ikan tuna segar utuh 7
3. Ikan tuna segar utuh 13
4. Pola protein tuna segar utuh dan olahan tuna 16 5. Elektroforegram hasil PCR ikan tuna dan olahan tuna dengan primer
cyt b untuk tuna segar utuh (a); tuna steak (b); sushi tuna (c); bakso tuna (d); abon tuna (e); dan tuna kaleng (f)
20
6. Elektroforegram hasil PCR ikan tuna dan olahan tuna dengan primer COI untuk tuna segar utuh (a); tuna steak (b); sushi tuna (c); bakso tuna (d); abon tuna (e); dan tuna kaleng (f)
21
7. Pohon filogenetik tuna segar utuh dan olahan tuna dengan primer cyt b 24 8. Pohon filogenetik tuna segar utuh dan olahan tuna dengan primer COI 25
DAFTAR LAMPIRAN
1. Bahan yang digunakan 32
2. Alat yang digunakan 32
3. Koleksi sampel 34
1
1
PENDAHULUAN
Latar Belakang
Tuna termasuk dalam famili scombridae dan diklasifikasikan dalam 14 genus antara lain Acanthocybium, Allothunnus, Auxis, Cybiosarda, Euthynnus, Gasterochisma, Grammatorcynus, Gymnosarda, Katsuwonus, Rastrelliger, Sarda, Scomber, Scomberomorus, dan Thunnus (www.fishbase.org). Tuna dari genus
Thunnus yang merupakan komoditas utama antara lain tuna albakora (T. alalunga), tuna mata besar/bigeye tuna (T. obesus), tuna sirip kuning/yellowfin
tuna (T. albacares), tuna sirip biru Atlantik/Atlantic bluefin tuna (T. thynnus), tuna sirip biru Pasifik/Pacific bluefin tuna (T. orientalis), dan tuna sirip biru selatan/Southern bluefin tuna (T. maccoyii) (Majkowski 2007).
Di Indonesia tuna/tongkol/cakalang (TTC) merupakan komoditi perikanan terbesar kedua setelah udang. Data statistik KKP (2014) menunjukkan bahwa volume ekspor untuk TTC tahun 2009 sebesar 150.989 ton, tahun 2010 sebesar 122.450, tahun 2011-2013 masing-masing 141.747 ton, 201.159 ton dan 209.072 ton, sedangkan data volume ekspor hingga bulan september 2014 sebesar 155.130 ton. Kenaikan rata-rata volume ekspor TTC antara tahun 2009 sampai 2013 sebesar 13,67%, sedangkan tahun 2013 mengalami kenaikan rata-rata sebesar 3,9% dibandingkan dengan tahun 2012. Permintaan yang tinggi akan ikan tuna dan semakin terbatasnya stok ikan tuna terutama tuna sirip biru memberikan peluang untuk terjadinya pemalsuan.
Beberapa penelitian mengenai ikan tuna Indonesia antara lain perubahan komposisi nutrisi ikan cakalang akibat proses pemasakan (Nurjanah et al. 2015), autentikasi ikan dan produk olahan tuna dari Indonesia menggunakan DNA barcoding dan DNA nukleus (Abdullah dan Rehbein 2014) dan penilaian kualitas daging ikan tuna Thunnus albacares komersial berdasarkan kandungan mioglobin (Nurilmala et al. 2013).
Kasus mislabeling atau ketidaksesuaian dengan label, substitusi spesies dengan spesies yang lebih murah telah banyak dilaporkan beberapa negara. Di Brazil dilaporkan bahwa 24% dari sampel yang diperoleh merupakan mislabeling, yaitu spesies dengan harga tinggi (flounder fish, pink cusk-eel dan cod) disubtitusi
dengan spesies yang bernilai rendah (Basa dan Alaska pollock) (Carvalho et al. 2015). Substitusi juga ditemukan di Mesir, yaitu ikan berlabel
2
primer cyt b dengan gen target 380 bp, olahan kerupuk tenggiri teridentifikasi tidak mengandung ikan tenggiri (Maulid dan Nurilmala 2015)
Penipuan konsumen dengan mislabeling tidak hanya berdampak pada pengelolaan sumberdaya kelautan yang berkelanjutan (Jacquet dan Pauly 2007), namun juga berdampak pada keamanan pangan dan berpotensi menimbulkan masalah kesehatan (Burger et al. 2004). Identifikasi dari 68 sampel tuna sushi di 31 restauran di New York, Denver dan Colardo menunjukkan bahwa 9 restauran
di antaranya menjual sushi tuna sebagai “white tuna” atau albacore tuna (T. alalunga) namun teridentifikasi sebagai escolar (Lepidocybium flavorunneum), spesies tersebut di Jepang dan Italia telah dilarang dijual karena dapat menimbulkan masalah kesehatan (diare) (Lowenstein et al. 2009)
Pemberlakuan ASEAN Free Trade Area (AFTA) atau dikenal dengan Masyarakat Ekonomi Asean (MEA) akan meningkatkan persaingan perdagangan antar negara ASEAN, tarif bea masuk produk perikanan diturunkan 0 sampai 5%, hal ini tidak hanya akan membawa peluang dan pemanfaatan namun juga permasalahan, hambatan dan tantangan bagi Indonesia di sektor kelautan dan perikanan. Peningkatan kualitas dan mutu perikanan salah satunya keaslian akan produk perikanan dapat meningkatan daya saing Indonesia menghadapi MEA.
Autentikasi diperlukan untuk meyakinkan konsumen tentang keakuratan pelabelan serta menjaga kualitas dan keamanan pangan (Klossa-Kilia et al. 2002). Autentikasi dilakukan karena banyaknya spesies hasil perairan yang dapat digunakan sebagai bahan baku dalam pembuatan produk perikanan (Martinez et al. 2005).
Metode berbasis protein yang digunakan untuk identifikasi spesies antara lain Sodium Dedocyl Sulfate Polyacrylamide Gel Electrophoresis (SDS-PAGE), urea Isoelectric Focusing (IEF) (Moretti et al. 2003) dan menggunakan metode Enzyme-Linked Immunosorbent Assay (ELISA) (Asensio et al. 2003).
Penggunaan metode berbasis DNA memiliki beberapa kelebihan dibanding dengan protein diantaranya DNA bersifat termostabil, DNA dapat digunakan untuk produk yang telah mengalami pengolahan, DNA terdapat di sebagian besar sel, DNA berpotensi memberikan informasi lebih dibanding protein karena degenerasi dari kode genetik dan adanya daerah non coding (Lockley dan Bradsley 2000).
DNA barcoding merupakan urutan pendek DNA dari wilayah standar pada genom yang digunakan untuk identifikasi spesies. Teknik DNA barcoding merupakan teknik yang banyak dikembangkan, karena relatif mudah dilakukan apabila dibandingkan dengan teknik lainnya (Wong dan Hanner 2008). DNA barcoding telah berhasil digunakan untuk mengidentifikasi kelompok tertentu dari jenis ikan seperti ikan tuna (Terol et al. 2002), flatfish (Espineira et al. 2008), anchovy (Jerome et al. 2008) dan sharks (Barbuto et al. 2010).
Penelitian ini menggunakan gen cytochrome c oksidase subunit I (COI) dan cytochrome b (cyt b) dari DNA mitokondria sebagai gen target. DNA barcoding menggunakan COI telah banyak digunakan untuk identifikasi spesies (Bucklin et al. 2011). Kelebihan gen COI di antaranya (1) memiliki sifat
3
karena laju evolusi pada COI lebih tinggi dibandingkan rRNA dan disebabkan oleh tingginya laju substitusi pada posisi ketiga di kodon (Bucklin et al. 2011).
Menurut Tobe et al. (2011) gen cyt b menunjukkan variasi yang besar pada sekuen yang pendek, variasi interspesies cyt b lebih besar dibandingkan COI namun variasi intraspesiesnya sama dengan COI. Randi (1996) menyatakan bahwa adanya variasi urutan pada gen cyt b, sehingga sering digunakan sebagai pembanding analisis filogenetik untuk tingkat spesies, genus atau famili yang sama.
Teknik molekuler DNA barcoding diperlukan untuk mendeteksi adanya pemalsuan dalam rangka rangka perlindungan terhadap konsumen dan mencegah economic fraud.
Perumusan Masalah
1. Mulai diberlakukannya perdagangan bebas di kawasan ASEAN atau Masyarakat Ekonomi ASEAN (MEA) pada awal Januari 2016, seluruh produk perikanan dapat masuk ke Indonesia, sehingga teknik molekuler dapat dijadikan metode deteksi terhadap produk perikanan yang akan masuk ke Indonesia
2. Metoda yang cepat dan akurat untuk mendeteksi keaslian suatu produk perikanan diperlukan untuk melindungi konsumen.
Tujuan Penelitian
Penelitian ini bertujuan mengidentifikasi keaslian ikan tuna dan olahannya melalui teknik molekuler DNA barcoding menggunakan gen DNA mitokondria cyt b dan COI sehingga dapat digunakan untuk mencegah adanya pemalsuan/ketidaksesuaian label dan economic fraud.
Manfaat Penelitian
`
4
2
METODE
Waktu dan Tempat
Penelitian dilaksanakan pada bulan November 2014 sampai Agustus 2015 bertempat di Laboratorium Karakteristik Bahan Baku, Departemen Teknologi Hasil Perairan, Laboratorium Biodiversitas dan Biosistematika Kelautan, Departemen Ilmu dan Teknologi Kelautan, Laboratorium Terpadu, Fakultas Perikanan dan Ilmu Kelautan, Institut Pertanian Bogor.
Bahan
Bahan yang digunakan yaitu ikan tuna segar utuh dan olahan tuna (tuna steak, sushi tuna, bakso tuna, abon tuna, dan tuna kaleng), DNeasy blood and tissue kit (Qiagen, Venlo, Netherlands), DNeasy mericon food kit (Qiagen, Venlo, Netherlands), etanol 96%, UltraPureTM 10x TBE buffer (Invitrogen, Massachusetts, US), kloroform, PCR kit commercial KAPA Taq EXtra HotStart ReadyMix Kit (Kapabiosystems, Massachusetts, US), primer foward dan reverse, agarose (Invitrogen, Massachusetts, US), ethidium bromide, dan DNA ladder 1 kb (Invitrogen, Massachusetts, US).
Alat
Alat yang digunakan antara lain tabung mikro (Qiagen, Venlo, Netherlands), peqTwist vortex mixer (Peqlab Biotechnologie GmbH, Erlangen, Jerman), mikro pipet Finnpipette F1 (Thermo Scientific, Vantaa, Finland), pipet tips (Axygen Scientific, California, USA), sentrifuse PerfectSpin 24 plus (Peqlab Biotechnologie GmbH, Erlangen, Jerman), mesin PCR Termocycler Biometra T1 (Biometra GmbH, Gottingen, Jerman), elektroforesis horizontal Mupid-Exu Submarine Electrophoresis System (Advance, Tokyo, Jepang), elektroforesis vertikal TU100YK (Scie-Plas Ltd, Cambridge, England), timbangan digital Adam PW254 (Adam Equipment Co, Milton Keynes, United Kingdom), spindown PerfectSpin mini (Peqlab Biotechnologie GmbH, Erlangen, Jerman), microwave Panasonic NN-SM320M, freezer, alat sinar Ultraviolet Viewer (UV-1) (ExtraGene Inc, Taichung City, Taiwan), dan Nanophotometer P360 (Implen GmbH, Schatzbogen, Germany).
Prosedur Penelitian
5
kemurnian DNA, amplifikasi DNA, sekuensing, analisis bioinformatika dan konstruksi pohon filogenetik (Gambar 1).
Gambar 1 Diagram alir penelitian
Sampel
Tuna segar utuh Olahan tuna
Morfologi
Collette dan Nauen (1983) Proporsi tuna segar utuh
Analisis proksimat (AOAC 2005)
SDS-PAGE (Laemmli 1970)
Isolasi DNA
(DNeasy® blood and tissue protocol, 2011) dan (DNeasy mericon food handbook, 2014)
Pengukuran konsentrasi dan kemurnian DNA (Muladno, 2010)
Amplifikasi DNA
(Modifikasi Kappa HotStart Protokol)
Elektroforesis
Sekuensing (Sanger et al. 1977)
Analisis bioinformatika MEGA 6 (Tamura et al. 2013)
Pohon filogenetik Desain primer
(Borah, 2011; Sambrook dan Rusell, 2001)
Profil protein
6
Koleksi Sampel
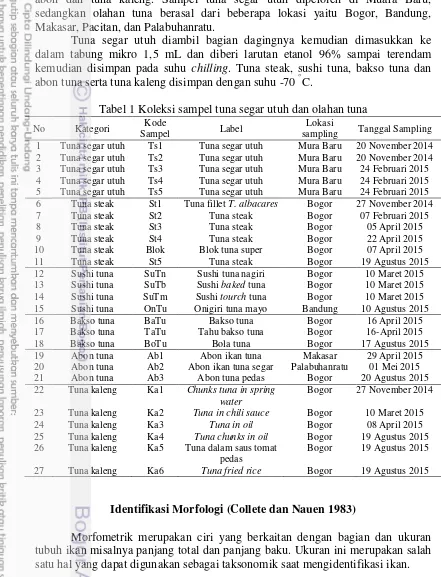
Sampel terdiri atas tuna segar utuh sebanyak 5 sampel dan produk olahan tuna sebanyak 22 sampel (Tabel 1). Olahan tuna terdiri atas steak, sushi, bakso, abon dan tuna kaleng. Sampel tuna segar utuh diperoleh di Muara Baru, sedangkan olahan tuna berasal dari beberapa lokasi yaitu Bogor, Bandung, Makasar, Pacitan, dan Palabuhanratu.
Tuna segar utuh diambil bagian dagingnya kemudian dimasukkan ke dalam tabung mikro 1,5 mL dan diberi larutan etanol 96% sampai terendam kemudian disimpan pada suhu chilling. Tuna steak, sushi tuna, bakso tuna dan abon tuna serta tuna kaleng disimpan dengan suhu -70 °C.
Tabel 1 Koleksi sampel tuna segar utuh dan olahan tuna No Kategori Kode
Identifikasi Morfologi (Collete dan Nauen 1983)
7
Pengukuran Proporsi Tuna Segar Utuh
Proporsi tuna segar merupakan persentase masing-masing bobot bagian tubuh (daging merah, daging putih, kulit, tulang, dan jeroan) ikan tuna dari bobot awal. Proporsi dihitung sebagai berikut:
Proporsi (%) = x 100%
Analisis Proksimat (AOAC 2005)
Analisis proksimat dilakukan untuk menentukan komposisi kimia bahan baku. Analisis proksimat meliputi kadar air, abu, protein, lemak, dan karbohidrat (by difference).
Analisis Kadar Air
Prinsip analisis kadar air yaitu untuk mengetahui kandungan air yang terdapat pada suatu bahan. Tahap pertama adalah mengeringkan cawan porselen dalam oven pada suhu 102-105 °C selama 10-15 jam, kemudian cawan diletakkan ke dalam desikator (kurang lebih 30 menit) dan dibiarkan sampai dingin lalu ditimbang. Cawan tersebut ditimbang kembali hingga beratnya konstan, kemudian cawan dan 5 g sampel yang telah dipotong-potong ditimbang, selanjutnya cawan dimasukkan ke dalam oven dengan suhu 102-105 °C selama 3-5 jam. Cawan dimasukkan ke dalam desikator dan dibiarkan sampai dingin kemudian ditimbang. Perhitungan kadar air yaitu:
Kadar air (%) = x 100% Keterangan:
A = Berat cawan kosong (gram)
B = Berat cawan dengan sampel (gram)
C = Berat cawan dengan sampel setelah dikeringkan (gram)
Analisis Kadar Abu
Prinsip penetapan kadar abu yaitu abu dalam bahan pangan ditetapkan dengan menimbang sisa mineral hasil pembakaran bahan organik pada suhu
PB
Skema ikan menunjukkan ciri morfometrik: P=Sirip Pectoral; A=Sirip Anal; C=Sirip Caudal; D=Sirip Dorsal; PB=Panjang Baku; PC=Panjang Cagak; PT=Panjang Total
Gambar 2 Skema morfometrik ikan tuna segar utuh
Bobot bagian tubuh ikan (g) Bobot utuh ikan (g)
8
sekitar 500-600 °C. Cawan proselin dikeringkan dalam oven pada suhu 102-105 °C selama 30 menit. Sebanyak 1-2 g sampel ditimbang dalam cawan porselin yang telah diketahui beratnya. Contoh dikeringkan dalam oven dan diarangkan, selanjutnya diabukan dalam tanur pada suhu 600 °C selama 6-8 jam sampai pengabuan sempurna (abu bewarna putih). Sampel didinginkan dalam desikator kemudian ditimbang. Kadar abu hitung menggunakan rumus sebagai berikut:
Kadar abu (%) = x 100%
Analisis Protein
Sampel ditimbang sebanyak 0,25 g dimasukkan ke dalam labu kjeldahl 100 mL, kemudian ditambahkan selenium 0,25 g dan 3 mL H2SO4 pekat, selanjutnya didekstruksi (pemanasan dalam keadaan mendidih) selama 1 jam atau sampai larutan menjadi jernih. Larutan dibiarkan dingin, kemudian 50 mL aquades dan 20 mL NaOH 40% ditambahkan lalu didestilasi. Hasil destilasi ditampung ke dalam labu erlenmeyer yang berisi campuran 10 mL H3BO3 2% dan 2 tetes indikator Brom Cresol Green-Methyl Red, setelah volume hasil tampungan (destilat) menjadi 10 mL dan berwarna hijau kebiruan maka destilasi dihentikan. Destilasi dititrasi dengan HCl 0,1 N sampai bewarna merah muda. Perlakuan yang sama dilakukan juga terhadap blanko. Kadar protein dihitung dengan rumus: Kadar protein (%) =
Keterangan:
VA : mL HCl untuk titrasi sampel VB : mL HCl untuk titrasi blanko
N : normalitas HCl standar yang digunakan 14,007 : berat atom Nitrogen
6,25 : faktor konversi protein untuk ikan W : berat sampel dalam gram
Analisis Lemak
Analisis kadar lemak dilakukan dengan metode sokhlet. Labu lemak yang akan digunakan di oven selama 30 menit pada suhu 100-105 °C, kemudian didinginkan dalam desikator untuk menghilangkan uap air dan ditimbang (A). Sampel ditimbang sebanyak 2 g (B) lalu dibungkus dengan kertas saring, ditutup dengan kapas bebas lemak dan dimasukkan ke dalam alat ekstraksi sokhlet yang telah dihubungkan dengan labu lemak yang telah di oven dan diketahui bobotnya. Pelarut heksan atau pelarut lemak lain dituangkan sampai sampel terendam dan dilakukan refluks atau ektraksi lemak selama 5-6 jam atau sampai palarut lemak yang turun ke labu lemak berwarna jernih. Pelarut lemak yang telah digunakan, disuling dan ditampung setelah itu ekstrak lemak yang ada dalam labu lemak dikeringkan dalam oven bersuhu 100-105 °C selama 1 jam, lalu labu lemak didinginkan dalam desikator dan ditimbang (C). Tahap pengeringan labu lemak
Berat abu Berat contoh
9
diulangi sampai diperoleh bobot yang konstan. Kadar lemak dihitung dengan rumus:
Kadar lemak (%) = Keterangan:
A : berat labu kosong (gram) B : berat sampel (gram)
C : berat labu dan lemak hasil ekstraksi (gram)
Analisis Karbohidrat (by difference)
Kadar karbohidrat dihitung dengan menghitung sisa (by difference) yaitu dengan rumus sebagai berikut:
Kadar karbohidrat (%) = 100% - (%air + %abu + %protein + %lemak)
Identifikasi Berbasis Protein (Profil Protein)
SDS-PAGE merupakan teknik untuk memisahkan protein berdasarkan kemampuan untuk bergerak dalam arus listrik. Metode SDS-PAGE pada penelitian ini menggunakan separating gel 12,5% dan stacking gel 3%. Sampel diekstrak proteinnya dengan cara 1 g sampel ditambahkan 3 mL buffer fosfat di homogenisasi kemudian di sentrifugasi dengan kecepatan 5.000 rpm dan suhu 4 °C selama 30 menit kemudian diambil supernatannya. Hasil ekstraksi kemudian dipanaskan pada suhu 95 °C selama 3 menit.
Sampel dan marker dimasukkan ke dalam sumur gel SDS-PAGE sebanyak 5 µL dengan mikropipet. Chamber elektroforesis dihubungkan dengan arus listrik pada arus 13 mA dan voltase 150 V selama 3 jam. Running buffer dipisahkan dan gel diambil dari plate pembentuk gel. Proses staining (pewarnaan) menggunakan 0,125% comassie briliant blue selama 2 jam hingga gel berwarna biru, kemudian dilanjutkan proses destaning (puncucian) menggunakan 25% methanol dan 10% asam asetat selama satu malam hingga gel berwarna bening dan pola protein sudah terlihat.
Identifikasi Berbasis DNA
Desain Primer cyt b dan COI
Primer merupakan oligonukleotida atau nukleotida pendek yang berfungsi sebagai inisiasi pada proses pemanjangan DNA. Desain primer diperlukan untuk mendapatkan primer yang spesifik pada proses amplifikasi DNA melalui metode PCR.
Parameter yang digunakan dalam mendesain primer antara lain jumlah basa berkisar 18-30 basa (Borah 2011). Suhu melting (Tm) yang optimum berkisar 50-70 °C dan presentase G atau C berkisar 40-65% (Sambrook dan Russell 2001).
10
Data sekuen ikan tuna untuk gen cyt b dan COI diambil melalui http://www.ncbi.nlm.nih.gov/nucleotide/ situs web National Center for Biotechnology Information (NCBI), kemudian sekuen disejajarkan (alignment) untuk mendapatkan daerah lestari (conserve) baik foward dan reverse. Hasil primer yang telah didesain kemudian di uji melalui situs oligoevaluator http://www.oligoevaluator.com/OligoCalcServlet#.
Isolasi DNA
Isolasi DNA menggunakan standar protokol DNeasy blood & tissue kit (Qiagen) untuk tuna segar utuh, sedangkan olahan tuna menggunakan standar DNeasy mericon food kit (Qiagen).
Tahapan isolasi DNA tuna segar utuh yaitu sampel sebanyak 10-25 mg
ditambahkan 180 μL buffer ATL dan 20 μL proteinase K kemudian di vortex dan di inkubasi pada suhu 56 °C selama satu jam atau sampai lisis dan di vortex setiap
15 menit. Sebanyak 200 μL buffer AL ditambahkan kemudian disentrifugasi selama 10 menit pada kecepatan 8.000 rpm (rotation per minute). Sampel yang telah disentrifugasi diberi 500 μL etanol (96-100%) dan di vortex. Hasil campuran dipipet ke mini spin coloumn dan ditempatkan ke dalam tabung 2 mL kemudian disentrifugasi selama 1 menit pada 8.000 rpm dan cairannya dibuang. Spin coloumn ditempatkan ke dalam tabung 2 mL baru dan ditambahkan 500 μL buffer AW1, kemudian disentrifugasi selama 1 menit dan dibuang cairannya. Spin coloumn ditempatkan ke dalam tabung 2 mL baru dan ditambahkan 500 μL buffer AW2, kemudian disentrifugasi selama 3 menit pada 14.000 rpm dan dibuang cairannya. Spin coloumn ditempatkan pada tabung 2 mL baru, kemudian DNA di
elusi dengan menambahkan 200 μL buffer AE tepat pada bagian tengah membran
spin coloumn dan diinkubasi pada suhu ruang (15-25 °C) selama 1 menit, kemudian disentrifugasi selama 1 menit pada 8.000 rpm.
Tahapan isolasi untuk olahan tuna yaitu sebanyak 200 mg olahan tuna ditambahkan 1 mL food lysis dan 2,5 µL proteinase K, kemudian di inkubasi pada suhu 60 °C semalaman atau sampai lisis dan disentrifugasi pada kecepatan 2.500 g selama 5 menit. Supernatan diambil sebanyak 700 µL dan ditambahkan kloroform 500 µL kemudian di vortex dan disentrifugasi pada kecepatan 14.000 g selama 15 menit, selanjutnya supernatan diambil sebanyak 350 µL dan ditambahkan 350 µL buffer PB kemudian di vortex dan dipindahkan ke spin coloumn. Sentrifugasi pada kecepatan 17.900 g selama 1 menit dan cairannya dibuang, selanjutnya ditambahkan 500 µL buffer AW2. Sentrifugasi pada kecepatan 17.900 g selama 1 menit dan cairannya dibuang, kemudian pindahkan ke spin coloumn ke tabung baru dan ditambahkan 150 µL buffer EB, selanjutnya sentrifugasi pada kecepatan 17.900 g.
Pengukuran Konsentrasi dan Kemurnian DNA
11
Konsentrasi DNA (µg/mL)
Kemurnian DNA ditentukan dengan cara menghitung rasio antara nilai OD (optical density) 260 dan 280 pada sampel DNA yang diukur melalui spektrofotometer. Jika nilai rasio OD260/OD280 berkisar antara 1,8 sampai 2,0 maka DNA dinyatakan murni (Muladno, 2010). Rasio OD260/OD280 kurang dari 1,8 menunjukkan adanya kontaminasi berupa fenol atau protein pada hasil ekstraksi (Devereux dan Wilkinson 2004), sedangkan jika rasio OD260/OD280 lebih dari 2,0 menjelaskan bahwa DNA terkontaminasi RNA (Khosravinia et al. 2007).
Amplifikasi DNA
Hasil isolasi DNA kemudian diamplifikasi secara in vitro melaui teknik PCR. Bahan utama pada reaksi ini terdiri atas air, buffer, dNTPs, primer dan Taq DNA polymerase yang telah disiapkan dalam suatu tabung, kemudian dibuat menjadi volume yang lebih kecil dalam tabung mikro, lalu ditambahkan DNA template dan MgCl2 (Basit 2009).
Bahan PCR mix yang digunakan mengikuti protokol KAPA Taq Extra HotStart ReadyMix yang dimodifikasi terdiri atas ddH2O sebanyak 21,5 μL, primer foward dan reverse masing-masing 1,25 μL, DNA template sebanyak
1 μL, dan KAPA Taq Extra HotStart ReadyMix PCR Kit sebanyak 25 μL, sehingga diperoleh bahan PCR mix sebanyak 50 μL dengan 40 siklus. Bahan PCR
mix kemudian dimasukkan ke mesin thermocycler yang telah diatur suhu dan siklus yang akan digunakan. Tahap PCR amplifikasi DNA mengikuti protokol KAPA Taq Extra HotStart ReadyMix PCR Kit yang telah dimodifikasi (Tabel 2).
Tabel 2 Tahapan PCR amplifikasi
Tahapan Keterangan
Pre denaturation Persiapan denaturasi, suhu diatur 95 °C selama 3 menit
Denaturation Pemutusan untaian ganda menjadi untaian tunggal (T = 95 °C
selama 30 detik)
Annealing Penempelan primer (Tm - 5 °C) selama 30 detik
Extention Persiapan polimerasi (T = 72 °C selama 1 menit)
Post extention Polimerasi (T = 72 °C selama 1 menit), terdapat jutaan salinan
DNA
Preservation Penyimpanan (T = 4-10 °C selama 5 menit)
Elektroforesis
Elektroforesis agarose digunakan untuk memastikan adanya DNA setelah proses isolasi dan untuk mengidentifikasi keberhasilan proses amplifikasi setelah PCR. Gel agorose 1% dibuat dengan cara mensuspensikan agarose kering 5 g dalam larutan buffer TBE 1X 50 mL, kemudian dimasukkan ke microvawe selama 3-5 menit hingga larutan berwarna jernih. Sebanyak 4 µL ethidium bromide ditambahkan dan dituangkan ke dalam casting tray yang telah diletakkan comb (sisir) hingga gel mengeras. Gel kemudian direndam dengan larutan buffer TBE serta dihubungkan dengan elektroda positif dan negatif selama 25 menit.
12
berjalan melewati pori-pori pada gel dengan adanya arus listrik. DNA akan bergerak menuju kutub positif dan menjauhi kutub negatif karena adanya arus listrik, hasil elektroforesis selanjutnya divisualisasikan di bawah gelombang pendek sinar UV (Basit 2009).
Sekuensing
Produk PCR yang berhasil teramplifikasi disiapkan untuk proses penentuan urutan nukleotidanya menggunakan DNA sequencer dengan metode Sanger (Sanger et al. 1977). Penentuan urutan nukleotida dilakukan dengan cara mengirim produk PCR ke DNA Sequencing Services 1st Base Laboratories Sdn Bhd, Taman Serdang Perdana, Selangor-Malaysia melalui jasa PT. Genetika Science.
Analisis Bioinformatika
Data hasil sekuensing diolah menggunakan program MEGA 6 (Molecular Evolutionary Genetic Analysis) (Tamura et al. 2013). Data tersebut disejajarkan menggunakan CustalW (1,6) pada program MEGA 6 untuk melihat keragaman basa nukleotidanya. Urutan basa nukleotida yang diperoleh kemudian dicocokkan pada data yang tersedia pada GenBank di NCBI menggunakan BLAST (http://blast.ncbi.nlm.nih.-gov), hasil pencocokan berupa presentase kecocokkan, semakin tingi nilai presentasenya maka semakin mendekati atau menyamai spesies.
Pohon Filogenetik
13
3
HASIL DAN PEMBAHASAN
Pengukuran Panjang, Berat dan Ciri Fisik Ikan Tuna Segar Utuh
Pengamatan morfologi 5 ekor sampel tuna segar utuh meliputi pengukuran panjang total, panjang baku, panjang cagak, pengukuran berat ikan tuna dan ciri fisik ikan tuna segar utuh.
Menurut Robinson dan Simonds (2006) tuna sirip kuning dan tuna mata besar diklasifikasikan berdasarkan ukurannya, yaitu panjang <20 cm dikategorikan sebagai larva; panjang antara 20 cm sampai 99 cm sebagai juvenil atau subdewasa, sedangkan panjang >100 cm digolongkan sebagai ukuran dewasa. Panjang total ikan tuna segar utuh berkisar 34,5-63,8 cm, sehingga tuna segar utuh pada penelitian ini digolongkan sebagai juvenil atau subdewasa .
Gambar 3 Ikan tuna segar utuh
Panjang baku tuna segar utuh berkisar antara 29 cm sampai 50,5 cm, panjang cagak berkisar 31,3-53,8 cm, dan panjang total berkisar 34,5-63,8 cm dengan berat berkisar antara 0,46 kg sampai 2,27 kg. Pengukuran secara morfologi menurut Collette dan Nauen (1983) bahwa tuna segar utuh 1 (Ts1), Ts2, Ts3, Ts4, dan Ts5 teridentifikasi sebagai yellowfin tuna (T. albacares) dengan ciri-ciri sebagian besar spesies dari memiliki sirip kuning cerah, tubuh ditutupi sisik yang sangat kecil, tidak ada bintik hitam pada tubuh permukaan (Gambar 3) ventral hati tanpa garis-garis, cuping (lobus) hati bagian kanan lebih panjang dan tipis dibandingkan dengan bagian tengah dan kiri.
Identifikasi secara morfologi hanya bisa dilakukan untuk ikan utuh, sedangkan ikan yang karakteristik fisiknya telah hilang (fillet, steak, ikan yang dikalengkan) identifikasi morfologi akan sulit dilakukan sehingga dilanjutkan dengan identifikasi berdasarkan protein.
Proporsi Tuna Segar Utuh
14
Tabel 3 Proporsi ikan tuna (%) Spesies Daging
merah
Daging
putih Jeroan Kepala Tulang Kulit Tuna segar
utuh 15,88±1,41 32,90±2,52 8,19±1,10 22,87±3,06 16,03±4,14 4,14±1,05
Euthynnus
affinis1) 12,82 37,18 41,3 5,13
Thunnus
alalunga2) 46,90±1,60 6,10±1,00 23,90±1,80 19,00±1,20 4,2±0,6
Skipjack
Daging merah ikan tuna merupakan lapisan daging yang mempunyai pigmen kemerahan di bawah kulit tubuh. Komposisi daging merah 15,88%, daging putih 32,90%, sedangkan rendemen jeroan, kepala, tulang dan kulit masing-masing berkisar 8,19%; 22,87%; 16,03%; dan 4,14%. Hasil ini tidak jauh berbeda dengan komposisi rendemen ikan tuna lainnya. Secara umum bagian ikan yang dapat dimakan berkisar antara 45 sampai 50% (Suzuki 1981). Pemanfaatan hasil perikanan tidak hanya bagian yang dapat dimakan, namun juga perlu memanfaatkan bagian lainnya seperti jeroan, kepala, tulang dan kulit, sehingga tidak ada bagian ikan yang terbuang. Trilaksani et al. (2006) memanfaatkan limbah tulang ikan tuna (Thunnus sp.), sedangkan Agustin dan Sompie (2015) memanfaatkan limbah kulit ikan tuna (Thunnus albacares) sebagai sumber gelatin.
Komposisi Kimia Ikan Tuna Segar Utuh, Tuna Steak dan Tuna Kaleng
Analisis proksimat meliputi pengukuran kadar air, abu, lemak, protein dan karbohidrat dengan perhitungan by difference. Analisis proksimat pada penelitian dilakukan sebagai dugaan awal adanya kandungan protein pada olahan tuna serta untuk mengetahui komposisi kimia yang terkandung pada tuna segar utuh dan olahan tuna. Pengukuran proksimat diwakili oleh tuna segar utuh, tuna steak dan tuna kaleng (Tabel 4).
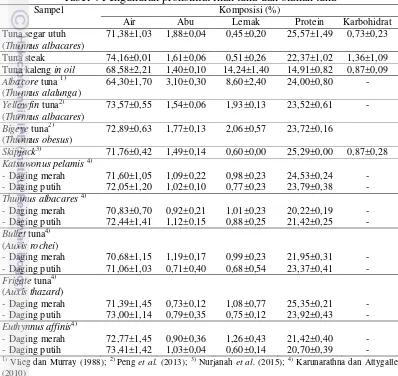
Komposisi protein tuna segar utuh (Thunnus albacares) 25,57%, tuna steak 22,37% dan tuna kaleng in oil 14,91%. Komposisi protein ini tidak jauh berbeda dengan ikan tuna lainnya yaitu 24,00% untuk T. alalunga, 23,52% untuk T. albacares dan 23,72% untuk T. obesus, juga untuk famili scombridae lainnya (Euthynnus affinis, Auxis rocei, Auxis thazard, dan Katsuwonus pelamis) komposisi protein berkisar 20-25%.
Komposisi lemak tuna segar utuh, tuna steak dan tuna kaleng dalam minyak masing-masing 0,45%; 0,51% dan 14,24%. Kadar lemak yang tinggi pada tuna kaleng dalam minyak disebabkan adanya bahan tambahan berupa minyak, kadar lemak akan meningkat karena adanya penyerapan minyak selama penyimpanan.
15
daging putih (20-23%), sedangkan komposisi lemak lebih banyak ditemukan pada daging merah (0,98-1,26%) dibandingkan pada daging putih (0,60-0,88%). Menurut Sanchez-Zapata et al. (2011) daging merah terletak dibawah kulit dan pada ikan tuna daging merah juga terletak dekat tulang punggung. Ikan berlemak mengandung proporsi daging merah lebih tinggi hingga mencapai 48% (Sanchez-Zapata dan Perez Alvarez 2007), hal ini dikarenakan ikan berlemak merupakan spesies migrasi, sehingga membutuhkan lemak, glikogen dan mioglobin untuk melakukan perjalanan panjang (Sanchez-Zapata et al. 2011).
Tabel 4 Pengukuran proksimat ikan tuna dan olahan tuna
Sampel Komposisi (%)
Air Abu Lemak Protein Karbohidrat Tuna segar utuh
(Thunnus albacares)
71,38±1,03 1,88±0,04 0,45±0,20 25,57±1,49 0,73±0,23
Tuna steak 74,16±0,01 1,61±0,06 0,51±0,26 22,37±1,02 1,36±1,09 Tuna kaleng in oil 68,58±2,21 1,40±0,10 14,24±1,40 14,91±0,82 0,87±0,09
Albacore tuna1)
72,89±0,63 1,77±0,13 2,06±0,57 23,72±0,16
Skipjack3) 71,76±0,42 1,49±0,14 0,60±0,00 25,29±0,00 0,87±0,28
Katsuwonus pelamis 4)
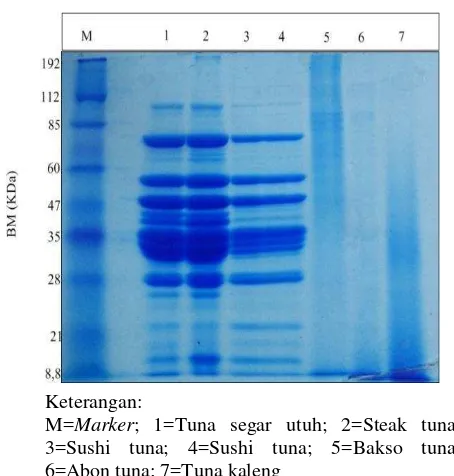
Pola protein yang terbentuk pada sampel tuna segar utuh, tuna steak, sushi, bakso, abon dan tuna kaleng dapat dilihat pada Gambar 4. Pola protein pada tuna segar utuh dan tuna steak terlihat lebih tebal dan jelas, sedangkan sushi walaupun tidak tebal pola proteinnya sama dengan tuna segar utuh dan tuna steak. Pola protein olahan bakso tuna, abon tuna dan tuna kaleng terlihat, namun pola proteinnya cukup tipis.
16
Gambar 4 Pola protein tuna segar utuh dan olahan tuna
Identifikasi spesies berbasis protein hanya bisa dilakukan untuk ikan utuh (tuna segar) maupun produk yang mengalami pengolahan (tuna steak dan sushi tuna), namun untuk olahan yang telah mengalami proses pemanasan (bakso tuna, abon tuna dan tuna kaleng) metode berbasis protein kurang efektif karena protein telah terdegradasi, sehingga perlu dilakukan identifikasi lanjut secara molekuler, hal ini sesuai dengan Rasmussen dan Morrissey (2008) bahwa metode berbasis protein umumnya dapat digunakan untuk ikan segar, namun kurang efektif untuk produk yang mengalami pemanasan karena protein telah terdegradasi.
Desain Primer cyt b dan COI
Data sekuen ikan tuna diperoleh dari NCBI melalui situs web http://www.ncbi.nlm.nih.gov/nucleotide/ berdasarkan parsial DNA mitokondria COI dan cyt b.
Sekuen tuna untuk cyt b terdiri atas T. obesus, T. orientalis, T. albacares, T. alalunga, T. thynnus, T. maccoyii, T. tonggol. Daerah conserve untuk foward gen cyt b diperoleh pada rentang 445 bp dan 1064 bp untuk reverse, sehingga diperoleh panjang fragmen 620 bp. Primer cyt b diberi label F dan Tuna1-R. Sekuen tuna untuk COI disusun dari T. alalunga, T. orientalis, T. obesus, T. atlanticus, dan T. macoyii. Daerah conserve untuk COI diperoleh pada rentang daerah 20 bp untuk foward dan 602 bp untuk reverse, sehingga diperoleh panjang fragmen 583 bp. Primer yang dirancang untuk COI diberi label Tuna-F2 dan Tuna-R2.
Panjang primer cyt b untuk foward diperoleh 26 bp (base pairs) dan reverse diperolah 23 bp, sedangkan untuk COI diperolah 21 bp untuk foward dan 18 bp untuk reverse. Menurut (Borah 2011) primer dengan kisaran panjang 18-30 bp cukup untuk primer mengikat template pada suhu annealing. Urutan
Keterangan:
M=Marker; 1=Tuna segar utuh; 2=Steak tuna;
17
basa nukleotida foward dan reverse yang diperoleh kemudian diuji dengan situs web oligoevaluator http://www.oligoevaluator.com/OligoCalcServlet#. Hasil uji tersebut dapat dilihat pada Tabel 5.
Tabel 5 Hasil oligoevaluator desain primer ikan tuna Primer Label Sekuens Panjang
(bp)
Tuna1-F 5‟CTYCTATCCGCAGTCCCATATGTYGG3‟ 26 62,9 50,0 Tidak Tidak
Tuna1-R 5‟GGAATAGGGAGAAGTAGAGGACG3‟ 23 63,0 52,2 Tidak Tidak
COI
Tuna2-F 5‟GTGCATGAGCTGGAATAGTTG3‟ 21 62,3 47,6 Tidak Tidak
Tuna2-R 5‟CAGGGTCGAAGAAGGTTG3‟ 18 60,9 55,6 Tidak Tidak
Suhu melting (Tm) untuk foward dan reverse cyt b diperoleh masing-masing 62,9 °C dan 63 °C, sedangkan untuk COI diperoleh 62,3°C untuk foward dan 60,9 °C untuk reverse, hal ini sesuai dengan Sambrook dan Russell (2001) bahwa suhu melting (Tm) yang optimum berkisar antara 50 sampai 70 °C. Suhu melting digunakan untuk estimasi suhu annealing (yang berperan untuk menempelnya primer pada DNA template/cetakan), bila Tm terlalu tinggi menghasilkan produk PCR yang dihasilkan rendah, sedangkan Tm yang terlalu rendah dapat menyebabkan produk non spesifik disebabkan oleh tingginya jumlah pasangan basa yang tidak sesuai (Borah 2010).
Presentase GC (GC%) diperoleh untuk foward dan reverse primer cyt b masing-masing 50,0% dan 52,2%, sedangkan untuk primer COI diperoleh 47,6% untuk foward dan 55,6% untuk reverse. Presentase GC menunjukkan banyaknya kandungan guanin dan citosin dalam primer. Presentase GC merupakan ciri penting dari DNA dan memberikan informasi mengenai kekuatan suhu annealing. Presentase GC sebaiknya pada rentang 45-60% (Dieffenbach et al. 1993), sehingga primer yang diperoleh masih dalam kisaran GC% yang disarankan. Primer dengan GC% yang rendah dapat menurunkan efisiensi proses PCR, karena primer tidak mampu untuk menempel secara efektif pada cetakan (Handoyo dan Rudiretna 2001).
Hasil uji oligoevaluator menunjukkan bahwa primer yang dirancang tidak membentuk primer dimer dan struktur sekunder (hairpins). Dimer pada primer akan menyebabkan primer dapat berikatan dengan primer lainnya yang sejenis (self dimer) atau primer dapat berikatan dengan primer pasangannya (cross dimer). Struktur sekunder (hairpins) pada primer dapat mengganggu proses penempelan primer pada DNA template (Borah 2011).
Primer yang didesain baik untuk cyt b dan COI telah memenuhi beberapa kriteria, sehingga dapat digunakan pada proses amplifikasi dengan PCR. Menurut Abd-Essalam (2003) primer yang kurang optimal akan menghasilkan sedikit atau tidak ada produk yang teramplifikasi, sedangkan primer yang optimal sangat penting untuk spesifisitas dan efisiensi pada proses PCR.
Hasil Isolasi, Pengukuran Konsentrasi dan Kemurnian DNA
18
perusakan dinding sel, menghilangkan protein dan RNA. Hasil isolasi DNA yang diperoleh kemudian dilakukan pengukuran konsentrasi DNA dan menghitung kemurnian DNA.
Konsentrasi DNA berkisar antara 2 ng/µ L sampai 444 ng/µ L, kecuali untuk olahan tuna kaleng (Ka1, Ka2 dan Ka4), bola tuna (BoTu) dan sushi tourch tuna (SuTm) konsentrasi DNA yang diperoleh rendah, sehingga kemurnian DNA nya tidak terbaca (Tabel 6). Konsentrasi DNA hasil isolasi yang diperoleh bervariasi, hal ini disebabkan oleh sampel yang diisolasi berasal dari sumber yang berbeda (tuna segar utuh dan produk olahan tuna). Kandungan bahan campuran yang terdapat dalam produk juga dapat mempengaruhi konsentrasi DNA yang diperoleh. Penambahan bumbu-bumbu, tepung, lemak dan bahan lain dalam produk olahan menyebabkan DNA yang diisolasi masih tercampur dengan senyawa kontaminan seperti oligopeptida, polisakarida, protein dan bahan-bahan organik lainnya (Nuraini 2004).
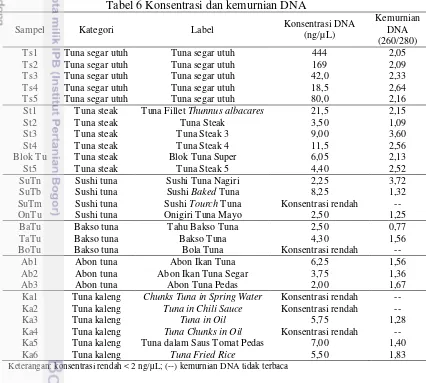
Tabel 6 Konsentrasi dan kemurnian DNA
Sampel Kategori Label Konsentrasi DNA
(ng/µL) St1 Tuna steak Tuna Fillet Thunnus albacares 21,5 2,15
St2 Tuna steak Tuna Steak 3,50 1,09
Ka1 Tuna kaleng Chunks Tuna in Spring Water Konsentrasi rendah -- Ka2 Tuna kaleng Tuna in Chili Sauce Konsentrasi rendah --
Ka3 Tuna kaleng Tuna in Oil 5,75 1,28
Ka4 Tuna kaleng Tuna Chunks in Oil Konsentrasi rendah -- Ka5 Tuna kaleng Tuna dalam Saus Tomat Pedas 7,00 1,40
Ka6 Tuna kaleng Tuna Fried Rice 5,50 1,83
Keterangan: konsentrasi rendah < 2 ng/µL; (--) kemurnian DNA tidak terbaca
19
hal ini diduga pada tahap presipitasi dengan kloroform kurang maksimal. Kloroform sendiri berfungsi untuk membersihkan protein serta molekul lainya (polisakarida), sehingga DNA yang diperoleh bebas kontaminasi. Masih terdapatnya kontaminasi RNA pada sampel tuna segar utuh (Ts1, Ts2, Ts3, Ts4, dan Ts5), tuna steak (St1, St3, St4, Blok Tu, dan St5), dan sushi tuna (SuTn) disebabkan oleh pada tahap isolasi tidak ditambahkannya enzim RNAse, sehingga RNA tidak terdegradasi.
Hasil Amplifikasi DNA dan Elektroforesis Hasil PCR
Isolat DNA tuna segar utuh dan olahan tuna kemudian diamplifikasi menggunakan primer cyt b dan COI. Tahapan PCR amplifikasi yaitu predenaturasi 94 °C selama 5 menit, denaturasi 94 °C selama 30 detik, annealing 58 °C selama 1 menit untuk cyt b dan untuk COI suhu annealing 57,3 °C selama 1 menit, ektensi 72 °C selama 1 menit dengan 40 siklus, paska ektensi 72 °C selama 7 menit dan penyimpanan 8 °C selama 5 menit.
(c)
M=Marker; 1=Sushi tuna nagiri (SuTn); 2=Sushi baked tuna (SuTb); 3=Sushi tourch tuna (SuTm); 4=Onigiri tuna mayo (OnTu); (-)=Kontrol negatif
(d)
M=Marker; 1=Bakso tuna (BaTu); 2=Tahu bakso tuna (TaTu); 3=Bola tuna (BoTu); (-)=Kontrol negatif (a)
M=Marker; (-)=Kontrol negatif; 1=Tuna segar utuh 1 (Ts1); 2=Tuna segar utuh 2 (Ts2); 3=Tuna segar utuh 3 (Ts3); 4=Tuna segar utuh 4 (Ts4); dan 5=Tuna segar utuh 5 (Ts5); (-)=Kontrol negatif
(b)
20
Gambar 5 Elektroforegram hasil PCR ikan tuna dan olahan tuna dengan primer cyt b untuk tuna segar utuh (a); tuna steak (b); sushi tuna (c); bakso tuna (d); abon tuna (e); dan tuna kaleng (f)
Elektroforegram PCR tuna dan olahannya dengan primer cyt b menunjukkan bahwa tuna segar utuh, tuna steak, tuna sushi, bakso tuna, abon tuna
dan tuna kaleng (Gambar 5) berhasil teramplifikasi pada rentang antara
500 sampai 750 bp, hal ini sesuai dengan DNA target cyt b yaitu 620 bp, namun untuk tuna kaleng in oil (Ka3) sampel tidak teramplifikasi ditandai dengan tidak munculnya pita target. 2=Tuna segar utuh 2 (Ts2); 3=Tuna segar utuh 3 (Ts3); 4=Tuna segar utuh 4 (Ts4); dan 5=Tuna segar utuh 5 (Ts5); (-)=Kontrol negatif
(b)
M=Marker; 1=Tuna fillet Thunnus albacares (St1); 2=Tuna steak2 (St2); 3=Tuna steak3 (St3); 4=Tuna steak4 (St4); 5=Blok tuna super (Blok Tu); dan 6=Tuna steak5 (St5); (-)=Kontrol negatif
(c)
M=Marker; 1=Sushi tuna nagiri (SuTn); 2=Sushi baked tuna (SuTb); 3=Sushi tourch tuna (SuTm); 4=Onigiri tuna mayo (OnTu); (-)=Kontrol negatif
(f)
M=Marker; 1= Chunks tuna in spring water (Ka1); 2=Tuna in chili sauce (Ka2); 3=Tuna in oil (Ka3); 4= Tuna chunks in oil (Ka4); 5= Tuna dalam saus tomat pedas (Ka5); 6= Tuna fried rice (Ka6); (-)=Kontrol negatif
21
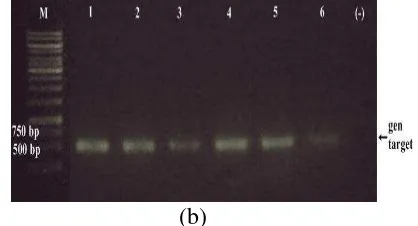
Gambar 6 Elektroforegram hasil PCR ikan tuna dan olahan tuna dengan primer COI untuk tuna segar utuh (a); tuna steak (b); sushi tuna (c); bakso tuna (d); abon tuna (e); dan tuna kaleng (f)
Elektroforegram PCR tuna dan olahannya dengan primer COI menunjukkan bahwa tuna segar utuh, tuna steak, tuna sushi, bakso tuna, abon tuna dan tuna kaleng (Gambar 6) menunjukkan bahwa sampel juga berhasil teramplifikasi pada rentang 500-750 bp, hal ini sesuai dengan DNA target COI yaitu 583 bp, namun untuk sampel tuna kaleng in oil (Ka3) tidak teramplifikasi ditandai dengan tidak adanya pita target.
Menurut Cawthorn et al. (2012) sembilan sampel kaleng berlabel sebagai
“tuna chunks”, “pink salmon” dan “mackarel” tidak teramplifikasi dengan COI, disebabkan karena rendahnya kualitas DNA pada sembilan sampel tersebut. Sampel tuna kaleng in oil (Ka3) pada penelitian ini tidak teramplifikasi dan konsentrasi DNA yang diperoleh 5,7 ng/µ L, sehingga sampel Ka3 diduga tidak berisi ikan tuna, namun menurut Pardo dan Perez-Villareal (2004) perlakuan panas pada proses pengalengan dapat menyebabkan degradasi DNA menjadi fragmen yang lebih kecil dari 200 bp. Penggunaan primer universal dengan gen
target yang lebih pendek (“mini barcode”) telah digunakan untuk mengidentifikasi spesimen DNA yang terdegradasi (Hajibabaei et al. 2006; Meusnier et al. 2008). Chapela et al. (2006) mendesain primer menggunakan cyt b dengan panjang fragmen 100 bp sampai 300 bp untuk mengamplifikasi 4 variasi tuna kaleng (Thunnus albacares) yaitu light tuna in brine, oil, vinegar dan tuna dengan saus tomat.
Kasus mislabeling terkait ikan tuna yang dilaporkan dari beberapa negara menggunakan gen taget COI dapat dilihat pada Tabel 7.
(f)
M=Marker; 1= Chunks tuna in spring water (Ka1); 2=Tuna in chili sauce (Ka2); 3=Tuna in oil (Ka3); 4= Tuna chunks in oil (Ka4); 5= Tuna dalam saus tomat pedas (Ka5); dan 6= Tuna fried rice (Ka6); (-)=Kontrol negatif
(e)
22
Tabel 7 Kasus mislabeling ikan tuna menggunakan gen target COI Dipasarkan sebagai:
Coryphaena hippurus 96 Nagalakshmi et al.
2016
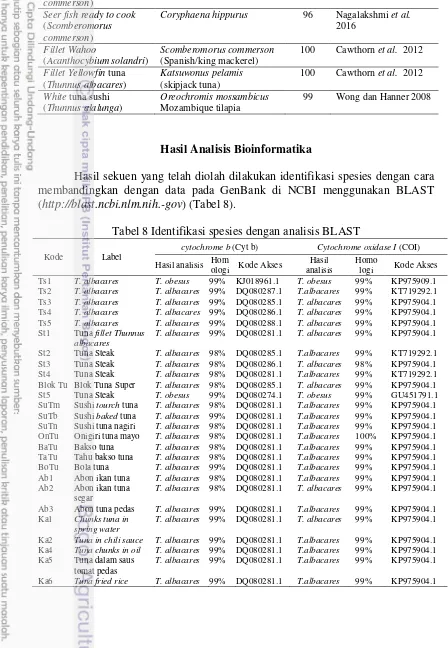
Hasil sekuen yang telah diolah dilakukan identifikasi spesies dengan cara membandingkan dengan data pada GenBank di NCBI menggunakan BLAST (http://blast.ncbi.nlm.nih.-gov) (Tabel 8).
Tabel 8 Identifikasi spesies dengan analisis BLAST Kode Label
cytochrome b (Cyt b) Cytochrome oxidase I (COI)
Hasil analisis Hom Ts2 T. albacares T. albacares 99% DQ080287.1 T.albacares 99% KT719292.1 Ts3 T. albacares T. albacares 99% DQ080285.1 T. albacares 99% KP975904.1 Ts4 T. albacares T. albacares 99% DQ080286.1 T. albacares 99% KP975904.1 Ts5 T. albacares T. albacares 99% DQ080288.1 T. albacares 99% KP975904.1 St1 Tuna fillet Thunnus
albacares
T. albacares 99% DQ080281.1 T. albacares 99% KP975904.1
St2 Tuna Steak T. albacares 98% DQ080285.1 T.albacares 99% KT719292.1 St3 Tuna Steak T. albacares 98% DQ080286.1 T. albacares 98% KP975904.1 St4 Tuna Steak T. albacares 98% DQ080281.1 T.albacares 99% KT719292.1 Blok Tu Blok Tuna Super T. albacares 98% DQ080285.1 T. albacares 99% KP975904.1 St5 Tuna Steak T. obesus 99% DQ080274.1 T. obesus 99% GU451791.1 SuTm Sushi tourch tuna T. albacares 98% DQ080281.1 T.albacares 99% KP975904.1 SuTb Sushi baked tuna T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1 SuTn Sushi tuna nagiri T. albacares 98% DQ080281.1 T.albacares 99% KP975904.1 OnTu Onigiri tuna mayo T. albacares 98% DQ080281.1 T.albacares 100% KP975904.1 BaTu Bakso tuna T. albacares 98% DQ080281.1 T.albacares 99% KP975904.1 TaTu Tahu bakso tuna T. albacares 98% DQ080281.1 T.albacares 99% KP975904.1 BoTu Bola tuna T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1 Ab1 Abon ikan tuna T. albacares 98% DQ080281.1 T.albacares 99% KP975904.1 Ab2 Abon ikan tuna
segar
T. albacares 98% DQ080281.1 T. albacares 99% KP975904.1
Ab3 Abon tuna pedas T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1 Ka1 Chunks tuna in
spring water
T. albacares 99% DQ080281.1 T. albacares 99% KP975904.1
Ka2 Tuna in chili sauce T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1 Ka4 Tuna chunks in oil T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1 Ka5 Tuna dalam saus
tomat pedas
T. albacares 99% DQ080281.1 T.albacares 99% KP975904.1
23
Identifikasi spesies dengan analisis BLAST dapat dilihat pada Tabel 8. Tuna segar utuh 1 (Ts1) baik menggunakan primer cyt b dan COI teridentifikasi sebagai T. obesus dengan tingkat homologi 99%, namun secara morfologi sampel teridentifikasi sebagai T. albacares, hal ini disebabkan oleh ukuran yang masih kecil, yaitu panjang cagak (fork length) antara Ts1 dan Ts2 kurang dari 40 cm yaitu 37,5 cm dan 31,3 cm. Menurut Gerasmio (2012) yellowfin tuna dan bigeye tuna pada ukuran juvenil terutama pada ukuran kurang dari 40 cm sering menimbulkan kesalahan identifikasi, sebanyak 48 sampel ikan tuna dengan fork length antara 13 sampai 31 cm, berdasarkan analisis morpho-meristik menunjukkan sebanyak 12 sampel (25%) terindentifikasi bigeye tuna, sedangkan 36 sampel (75%) merupakan yellowfin tuna, namun dengan fenotipe hati dan genetik menunjukkan bahwa hanya 5 sampel (10%) yang teridentifikasi big eye tuna, sedangkan 43 sampel (90%) lainnya merupakan yellowfin tuna. Semua olahan tuna steak, sushi tuna, bakso tuna, abon tuna dan tuna kaleng baik menggunakan gen cyt b maupun COI teridentifikasi sebagai T. albacares dengan homologi berkisar 98-100%, kecuali untuk tuna steak (St5) teridentifikasi sebagai T. obesus dengan homologi 99%. Adapun proses submit ke GenBank data sedang dilakukan baik untuk marka molekuler COI maupun cyt b.
Pohon Filogenetik
Konstruksi pohon filogenetik tuna segar utuh dan olahan tuna, baik primer cyt b dan COI menggunakan metode neighbor joining tree dengan nilai boostrap 1000 dan model p-distance. Spesies Balaenoptera omurai dijadikan outgroup untuk cyt b dan Chelonia mydas untuk COI. Spesies tersebut dijadikan outgroup karena mempunyai perbedaan basa nukleotida yang signifikan dengan sampel ikan tuna.
Konstruksi pohon filogenetik dengan primer cyt b menunjukkan bahwa sampel tuna segar utuh (Ts2, Ts3, dan Ts5), serta olahan tuna membentuk kelompok dengan T. albacares, sedangkan tuna segar utuh (Ts1) dan olahan tuna steak (St5) membentuk kelompok dengan T. obesus (Gambar 7), namun pada sampel ikan tuna segar utuh (Ts4) yang membentuk kelompok sendiri. Konstruksi pohon filogenetik dengan primer COI menunjukkan bahwa sampel tuna segar utuh (Ts2, Ts3, Ts4, dan Ts5), serta olahan tuna membentuk kelompok dengan T. albacares, kecuali tuna segar utuh (Ts1) dan olahan tuna steak (St5) membentuk kelompok dengan T. obesus (Gambar 8), hasil tersebut memperlihatkan bahwa hasil analisis BLAST sesuai dengan karakteristik cabang yang dibentuk oleh pohon filogenetik.
24
25
26
4
SIMPULAN DAN SARAN
Simpulan
Identifikasi secara morfologi hanya bisa dilakukan untuk ikan utuh yang ciri fisiknya terlihat, sedangkan identifikasi berbasis protein dapat digunakan baik untuk tuna segar utuh maupun tuna steak dan sushi tuna, namun untuk produk perikanan yang telah mengalami proses pemasakan perlu dilakukan pengujian berbasis DNA.
Elektroforegram PCR menunjukkan bahwa sampel tuna dan olahannya berhasil teramplifikasi sesuai dengan gen target baik dengan primer cyt b dan COI, kecuali tuna kaleng Ka3 tidak teramplifikasi. Identifikasi spesies secara molekuler baik menggunakan primer cyt b dan COI menunjukkan tuna segar utuh 1 (Ts1) dan tuna steak 5 (St5) teridentifikasi sebagai bigeye tuna (T.obesus) dengan tingkat homologi 99%, sedangkan tuna segar utuh (Ts2, Ts3, Ts4 dan Ts5) serta olahan tuna lainnya teridentifikasi T. albacares dengan tingkat homologi 98% sampai 100%.
Saran
1. Penelitian selanjutnya perlu dilakukan desain primer dengan ukuran gen target yang lebih pendek sekitar 100-300 bp („mini barcode’) terutama untuk produk-produk yang mendapat perlakuan panas (tuna kaleng).
27
DAFTAR PUSTAKA
[AOAC] Association of Official Analitycal Chemist. 2005. Official Method of the Association of Official Analitycal of Chemist. Arlington, Virginia, USA: Published by The Association of Analitycal Chemist, Inc.
[KKP] Kementerian Kelautan dan Perikanan. 2014. Kelautan dan Perikanan dalam Angka Tahun 2014. Jakarta. 330 hlm.
Abd-Elsalam KA. 2003. Bioinformatic tools and guideline for PCR primer design. African Journal of Biotechnology. 2(5):91-95.
Abdullah A, Rehbein H. 2014. Authentication of raw processed tuna from Indonesian market using DNA barcoding, nuclear gene and character-based approach. European Food Research and Technology. doi: 10.1007/s00217-014-2266-0.
Agustin AT, Sompie M. 2015. Kajian gelatin kulit ikan tuna (Thunnus albacares) yang diproses menggunakan asam asetat. Pros Sem Nas Masy Biodiv Indon. 1(5):1186-1189.
Asensio L. Gonzales I, Rodriguez MA, Mayoral B. Lopez-Calleja I, Hemandez PE, Garcia T, Martin R. 2003. Identification of grouper (Epinephelus guaza), wreck fish (Polyprion americanus), and Nile perch (Lates niloticus) fillets by polyclonal antibody-based enzymelinked immunosorbent assay. Journal of Agricultural and Food Chemistry. 51(5):1169-72.
Barbuto M, Galimberti A, Ferri E, Labra M, Malandra R, Galli P. 2010. DNA barcoding reveals fraudulent substitutions in shark seafood products: The
Italian case of “palombo” (Mustelus spp.). Food Research International. 43:3761-381.
Basit SA. 2009. Comparison of DNA Extraction Methods for Muscle Tissues of Tuna and Tuna-Like Species: Its Implications for Identification Using Polymerase Chain Reaction. Artikel: p 1-8.
Borah P. 2011. Primer designing for PCR. Science Vision. 11(3):134-136.
Bucklin A, Steinke D, Blanko-Bercial L. 2011. DNA barcoding of marine metazoa. Annual Review of Marine Science. 3:471-508.
Burger J, Stern AH, Dixon C, Jeitner C, Shukla S, Burke S, Gochfeld M. 2004. Fish availability in supermarkets and fish markets in New Jersey. Science of the Total Environment. 333:89-97.
Carvalho DC, Palhares RM, Drummond MG, Frigo TB. 2015. DNA barcoding identification of commercialized seafood in South Brazil: A governmental regulatory forensic program. Food Control. 50:784-788.
Cawthorn DM, Steinman HA, Witthuhn RC. DNA barcoding reveals a high incidence of spesies misrepresentation and substitution on the South African market. Food Research International. 46:30-40.
Changizi R, Farahmand H, Soltani M, Asareh R, Ghiasvand Z. 2013. Species identification reveals mislabeling of important fish products in Iran by DNA barcoding. Iranian Journal Fisheries Sciences. 12(4):783-791. Chapela MJ. Sotelo CG, Perez-Martin RI, Pardo MA, Perez-Villareal B, Giliardi
28
Cline E. 2012. Marketplace substitution of Atlantic salmon for Pacific salmon in Washington State detected by DNA barcoding. Food Research International. 45:388-393.
Collette BB, Nauen CE. 1983. FAO species catalogue. Vol. 2. Scombrids of the world. An annotated and illustrated catalogue of tunas, mackerels, bonitos and related species known to date. Rome: FAO Fisheries Department. Devereux R, Wilkinson SS. 2004. Amplification of Ribosomal RNA Sequences.
Kluwer Academic Publisher. Netherlands.
Dieffenbach CW, Lowe TMJ, Dveksler GS. 1993. General Concepts for PCR Primer Design. Cold Spring Harbor Laboratory Press. New York. p 30-37. Espineira M, Gonzalez-Lavin N, Vieites JM, Santaclara FJ. 2008. Development of
a method for the genetic identification of flatfish species on the basis of mitochondrial DNA sequences. Journal of Agricultural and Food Chemistry. 56(19):8954-8961.
Galal-Khallaf A, Ardura A, Mohammed-Geba K, Borell YJ, Garcia-Vazquez E. 2014. DNA barcoding reveals a high level of mislabeling in Egyptian fish fillets. Food Control. 46:441-445.
Gerasmio IRP, Babaran RP, Santos MD. 2012. Discrimination of juvenile yellowfin (Thunnus albacares) and bigeye (T. obesus) tunas using mitochondrial DNA control region and liver morphology. PLoS ONE 7(4). Hafiludin. 2011. Karakteristik proksimat dan kandungan senyawa kimia daging putih dan daging merah ikan tongkol (Euthynnus affinis). Jurnal Kelautan. 4(1): 10 hlm.
Hajibabaei M, Smith MA, Janzen DH, Rodriguez JJ, Whitfield JB, Hebert PDN. 2006. A minimalist barcode can identify a specimen whose DNA is degraded. Molecular Ecology Notes. 6:959–964.
Handoyo D, Rudiretna A. 2000. Prinsip Umum dan Pelaksanaan Polymerase Chain Reaction (PCR) [General Principles and Implementation of Polymerase Chain Reaction]. Unitas. 9(1):17-29.
Jacquet JL, Pauly D. 2007. Trade secrets: renaming and mislabelling of seafood. Marine Policy. 32:309-318.
Jerome M, Martinsohn JT, Ortega D, Carraeau P, Verrez-Bagnis V, Mouchel O. 2008. Toward fish and seafood traceability: Anchovy species determination in fish products by molecular markers and support through a public domain database. Journal of Agricultural and Food Chemistry. 56(10):3460-3469.
Karunarathna KAAU, Attygalle MVE. Nutritional evaluation in five species of tuna. Vidyodaya Journal of Science. 15:7-16.
Khosravinia H, Murthy HNN, Parasad DT, Pirany N. 2007. Optimizing Factors Influencing DNA Extraction from Fresh Whole Avian Blood. African Journal of Biotechnology. 6(4): 481-486.
Klossa-Kilia E, Papasotiropoulos V, Kilias G, Alahiotis S. 2002. Authentication of Messolongi (Greece) fish roe using PCR-RFLP analysis of 16s rRNA mtDNA segment. Food Control. 13:169-172.
Laemmli UK. 1970. Cleavage of structural protein during the assembly of the heat of bacteriophag T4. Nature. 227(10):680-685.