I. PENDAHULUAN
Latar Belakang
Kelapa (Cocos nucifera L.) merupakan salah satu tanaman penghasil minyak yang paling penting di daerah tropis. Peranan kelapa sebagai komoditi perkebunan bagi masyarakat Indonesia dan negara sangat besar. Produksi kelapa selama bertahun-tahun menunjukkan penurunan bertahap karena berbagai alasan, tetapi masih bernilai ekonomi penting dengan adanya permintaan industri yang tinggi untuk minyak laurat. Agar produksi kelapa tidak menurun, maka pelaksanaan peremajaan dan rehabilitasi harus dilakukan pada sekitar 20-30% pertanaman kelapa (Prastowo 2007). Perbaikannya dilakukan melalui pemuliaan konvensional dan bioteknologi. Jenis kelapa di Indonesia sangat variatif diantaranya adalah kelapa kopyor yang merupakan kelapa mutan asli Indonesia dan berbeda fenotipenya dengan kelapa Makapuno yang berasal dari Filipina.
Keberadaan kelapa kopyor yang unik dan asli Indonesia perlu terus dilestarikan dan dikembangkan lebih lanjut agar sumberdaya genetik asli Indonesia tersebut dapat dimanfaatkan sepenuhnya untuk kesejahteraan rakyat Indonesia. Berbeda dengan kelapa kopyor Indonesia, abnormalitas endosperma pada kelapa Makapuno menyebabkan jaringan ini menjadi lunak seperti jeli dan jika terlalu tua sebagian dari endospermanya akan terlarut dalam air kelapa, sehingga air kelapanya menjadi kental seperti minyak pelumas (Gambar 1.1).
Perbedaan utama antara abnormalitas endosperma kelapa kopyor dan kelapa makapuno adalah pada kelapa kopyor endospermanya tetap mempunyai penampakan seperti endosperma kelapa tetapi terlepas dari cangkangnya (Gambar 1.1), rasa air kelapa dan endospermanya lebih manis dari kelapa normal dan tekstur endospermanya lembut seperti tekstur gabus (Maskromo 2005). Semakin tua buah kelapanya umumnya air kelapanya semakin berkurang dan volume endosperma yang terlepas serta mengumpul dalam rongga dalam cangkang biji kelapanya semakin banyak (Maskromo et al. 2007).
Gambar 1.1 Variasi endosperma pada kelapa. Perbandingan endosperma abnormal pada (a) kelapa kopyor asal Indonesia, (b) kelapa Makapuno asal Filipina dan (c) endosperma normal pada kelapa normal (Sudarsono et al. 2014a)
Makapuno tidak memiliki aktivitas galaktosidase yang memungkinkan mengubah struktur dinding sel dan adhesi sehingga menghasilkan endosperm yang sangat kental (Luengwilai 2014). Besar kemungkinan bahwa abnormalitas fenotipe endosperma kelapa kopyor juga diduga juga sebagai akibat dari defisiensi enzim penting tertentu selama dalam proses perkembangan endospermanya. Namun demikian, identitas enzim yang defisien dari endosperma kelapa kopyor sampai saat ini masih belum diketahui. Karakteristik mutan pada kelapa kopyor juga dapat diturunkan secara genetik dari tetua ke progeninya (Sukendah 2009).
Salah satu permasalahan yang dihadapi oleh petani kopyor adalah rendahnya kuantitas hasil buah kopyor yang dipanen. Akibatnya produksi buah kelapa kopyor masih belum dapat memenuhi kebutuhan konsumen. Adanya kelapa normal di antara pertanaman kelapa kopyor diduga mempengaruhi produktivitas buah kopyor yang didapat (Sudarsono et al. 2012). Keberadaan pohon dewasa kelapa Dalam berbuah normal cenderung menyerbuk silang (Pandin 2009a) diduga berpengaruh negatif terhadap produksi buah kopyor. Pohon tersebut dapat menyebarkan serbuk sari pembawa sifat normal pada bunga betina pohon kelapa kopyor, akibatnya buah yang terbentuk dari penyerbukan akan menjadi buah normal (Sudarsono et al. 2012). Keberhasilan penyerbukan pada tanaman juga memerlukan bantuan polinator untuk persebaran serbuk sari tanaman kelapa maupun pada tanaman lainnya.
Studi evaluasi persebaran serbuk sari pada pertanaman kelapa kopyor dianggap penting untuk dilakukan untuk membuktikan dan memberikan informasi kepada petani mengenai jarak dan arah persebaran serbuk sari serta faktor yang berpengaruh pada penyerbukan kelapa kopyor. Studi persebaran serbuk sari dapat dipelajari dengan metode pewarnaan serbuk sari (Blair dan Williamson 2010) atau menggunakan marka molekular (Austerlitz et al. 2004). Marka yang biasa digunakan dalam analisis persebaran serbuk sari adalah marka RAPD pada Ilex paraguariensis (Cansian et al. 2010) dan marka SSR pada tanaman Hymenaea courbaril (Carneiro et al. 2011), tanaman pinus (Feng et al. 2010). Marka yang sering digunakan adalah marka SSR (Single Sequence Repeat) yang mempunyai keunggulan yaitu bersifat kodominan, polimorfismenya tinggi, lokus tersebar di dalam genom dalam jumlah banyak (Lowe et al. 2004) dan sampel DNA yang dibutuhkan sedikit karena dalam melakukan deteksi menggunakan PCR (Polymerase chain reaction) yang dapat menggandakan DNA (Semagn et al. 2006). Penanda DNA berbasis SNAP adalah satu-satunya penanda DNA yang memiliki sifat bi–alel dan kodominan, sehingga penanda SNAP mampu membedakan alel homozigot dari heterozigot yang efisien (Morin et al. 2004). Beberapa marker SSR dan marker SNAP sedang dikembangkan oleh PMB Laboratorium dari kegiatan penelitian awal namun demikian penelitian lebih lanjut masih sangat diperlukan.
memberikan informasi dasar genomik dan molekuler yang akan sangat berguna dalam mendukung program pemuliaan kelapa kopyor di masa yang akan datang.
Tujuan Penelitian
1. Mengetahui besarnya persentase penyerbukan silang dan penyerbukan sendiri yang terjadi pada populasi pertanaman kelapa kopyor tipe Genjah
2. Mengetahui besarnya persentase penyerbukan silang dan penyerbukan sendiri yang terjadi pada populasi pertanaman kelapa kopyor tipe Dalam
3. Mengetahui peranan pendonor serbuk sari kelapa normal dalam pembentukan progeni kelapa kopyor (efek xenia pada tanaman)
4. Mengetahui kemampuan dan efektifitas polinator lebah madu sebagai agen penyerbuk dominan
Manfaat Penelitian
Dengan diperolehnya informasi mengenai pola persebaran dan agen pernyerbuk yang dominan diharapkan akan memberikan informasi kepada petani mengenai efek keberadaan pohon kelapa berbuah normal di dalam pertanaman kelapa berbuah kopyor, efek polinator terhadap produksi buah kopyor sehingga diharapkan produktivitas buah kelapa kopyor dapat ditingkatkan, selain itu juga memberikan informasi dasar genomik dan molekuler yang akan sangat berguna dalam mendukung program pemuliaan kelapa kopyor di masa yang akan datang.
Kerangka Penelitian
Pemanfaatan marka molekuler dalam mendeteksi dan membantu analisis persebaran serbuk sari dalam populasi kelapa kopyor diharapkan mampu memberi efek positif dalam peningkatan produktifitas hasil bagi petani. Dengan pertimbangan tersebut maka dilakukan penelitian untuk melihat berbagai pengaruh persebaran serbuk sari terhadap berbagai studi penelitian.
Bagan Alir Penelitian
Gambar 1.2. Bagan alir penelitian disertasi Analisis Persebaran Serbuk Sari Kelapa Kopyor (Cocos nucifera L.) Asal Pati dan Kalianda Menggunakan Marka SSR dan SNAP Sebagai Penunjang Program Pemuliaan Tanaman
Plasma nutfah kelapa kopyor Indonesia ( Pati dan Lampung)
Karakteristik produktifitas kelapa
kopyor
Analisis persebaran serbuk sari kelapa
kopyor
Percobaan 1
Persebaran serbuk sari pada kelapa kopyor Genjah Pati mengindikasikan pentingnya
peranan polinator serangga dalam penyerbukan
Percobaan 2
Persebaran serbuk sari pada kelapa Dalam Kalianda membuktikan adanya penyerbukan silang pada kelapa dalam
Percobaan 4
Lebah sebagai polinator merubah pola persebaran serbuk sari pada kelapa
kopyor Pati
Data skoring tetua dan progeni setiap populasi
Analisis parental
menggunakan CERVUS
Percobaan 3
Persebaran serbuk sari membuktikan potensi pengaruh negatif kelapa normal
terhadap hasil buah kopyor
1. Mengetahui besarnya persentase penyerbukan silang dan penyerbukan sendiri yang terjadi pada populasi pertanaman kelapa kopyor tipe Genjah
2. Mengetahui besarnya persentase penyerbukan sendiri dan penyerbukan silang yang terjadi pada populasi pertanaman kelapa kopyor tipe Dalam 3. Mengetahui peranan pendonor serbuk sari
kelapa normal dalam pembentukan progeni kelapa kopyor (efek xenia pada tanaman)
II. TINJAUAN PUSTAKA
Tanaman Kelapa
Tanaman kelapa (Cocos nucifera L.) adalah satu satunya spesies dari cocos yang merupakan anggota dari subfamili Cocoideae dan famili Aracaceae (Palmaceae). Kelapa merupakan tanaman diploid yang memiliki jumlah kromosom 32 (2n=2×=32). Tanaman ini merupakan tanaman tahunan (perenial) yang bersifat monoecious yaitu memiliki bunga jantan dan betina pada tandan atau infloresensia yang sama (Chan dan Elevitch 2006).
Ciri-ciri pohon kelapa menurut Chan dan Elevitch (2006) adalah memiliki batang tunggal dan beruas dengan tinggi mencapai 30 m dan diameter kanopi 8-9 m. Akar berbentuk serabut, tebal, berkayu dan adaptif pada lahan berpasir pantai. Daun tersusun secara majemuk dan menyirip sejajar tunggal, pelepah terletak pada ibu tangkai daun, duduk pada batang (roset batang). Warna pada tangkai daun (petiole) mengindikasikan warna buah pada kelapa. Bunga kelapa merupakan bunga majemuk yang dilindungi oleh spatha. Bunga jantan dan betina terdapat pada satu tangkai utama yang disebut spadix, setiap spadix terdiri atas 40-60 cabang (spikelet) dengan ribuan bunga jantan. Letak bunga bunga betina terletak di pangkal, sedangkan bunga jantan di bagian atas bunga betina hingga ujung spikelet. Buah kelapa memiliki tiga lapisan yaitu eksokarp (kulit tipis terluar yang memiliki lapisan lilin) berwarna kuning, hijau, jingga atau coklat, mesokarp berupa lapisan serat yang lebih tebal atau sering disebut sabut dan endokarp yang keras disebut batok yang melindungi biji. Endokarp dan biji hanya dipisahkan oleh membran yang melekat pada sisi dalam dari endokarp. Biji kelapa memiliki tiga mikrofil (micropyle) dan hanya satu yang mengindikasikan keberadaan embrio. Embrio kelapa berukuran kecil dan akan membesar ketika buah siap untuk berkecambah. Endosperma biji kelapa terdiri atas endosperma cair yang mengandung banyak enzim dan endosperma padat yang mengendap pada dinding endokarp ketika buah menua (kernel).
Secara umum tanaman kelapa dibedakan atas dua tipe yaitu tipe Dalam (typica) dan tipe Genjah (nana) (Kumar et al. 2011). Penggolongan kedua tipe ini terutama didasarkan atas sifat munculnya pembungaan pertama, tinggi tanaman, komponen buah dan tipe penyerbukan. Kelapa Dalam mempunyai tinggi sekitar 15-18 m, batang kekar dengan dasar membengkak atau disebut bole. Bunga pertama muncul pada umur 6-10 tahun setelah tanam tetapi umur produktif dapat mencapai 90 tahun. Mahkota pohon memiliki 25-40 daun yang terbuka penuh, dengan panjang daun sekitar 5-7 m. Umumnya kelapa Dalam menyerbuk silang dan dari penyerbukan sampai buah masak memerlukan waktu sekitar 12 bulan dengan jumlah buah pertandan 6-12 butir. Ukuran buah besar sehingga produksi kopra, minyak dan sabut umumnya berkualitas baik.
Pohon kelapa tipe Genjah berpenampilan pendek sekitar 8-10 m saat berumur 20 tahun dengan batang agak kecil dan tanpa bole. Daunnya terbuka
kualitas buah dan kopranya kurang baik. Produksi akan mulai menurun setelah tanaman berumur 25 tahun (Chan dan Elevitch 2006).
Penyerbukan atau polinasi adalah jatuhnya serbuk sari dari kotak sari (antera) ke kepala putik (stigma) dalam satu bunga atau bunga yang berbeda. Penyerbukan tumbuhan dapat terjadi secara biotik dan abiotik. Penyerbukan biotik terjadi dengan bantuan hewan, sedangkan penyerbukan abiotik terjadi dengan bantuan angin, air dan gravitasi (Liferdi 2008). Jarak persebaran serbuk sari pada tanaman yang menyerbuk sendiri (autogamy) lebih rendah dibandingkan dengan tanaman menyerbuk silang (Boer 2007).
Kelapa Dalam pada umumnya merupakan tanaman menyerbuk silang sehingga tampilannya sangat beragam (Pandin 2009b). Kelapa Dalam memiliki bunga jantan yang matang lebih dulu dibanding bunga betina. Bunga betina siap diserbuki ketika bunga jantan umumnya sudah rontok sehinga terjadi penyerbukan silang. Kelapa Genjah pada umumnya memiliki pola penyerbukan sendiri meskipun memungkinkan terjadinya penyerbukan silang sehingga menyebabkan tingginya tingkat kemiripan genetik pada kelapa Genjah. Bunga betina dan bunga jantan pada kelapa Genjah masak secara bersamaan sehingga peluang untuk menyerbuk sendiri sangat besar (Hannum et al. 2003).
Penelitian Ramirez et al. (2004) menyatakan sebanyak 59% penyerbukan kelapa dibantu oleh serangga lebah madu. Lebah membantu proses penyerbukan silang, sehingga meningkatkan produktivitas tanaman budidaya. Potensi ini dimanfaatkan dengan cara meletakkan koloni lebah pada areal tanaman budidaya yang daya serbuknya rendah. Perpindahan lebah dari satu bunga ke bunga yang lain mempercepat proses polinasi karena serbuk sari banyak menempel pada kaki dan perut dari lebah (Liferdi 2008).
Kelapa Kopyor
Kelapa berbuah kopyor dari segi morfologi sama dengan tanaman kelapa lainnya. Maskromo et al. (2007) mengatakan buah kelapa kopyor hanya bisa dipastikan setelah buah dipanen dengan cara mengguncang buah kelapanya. Pada saat diguncang kelapa kopyor akan menghasilkan bunyi yang kurang nyaring dibanding kelapa normal, karena sebagian atau seluruh endosperma fase padatnya sudah lepas dari tempurungnya. Buah kopyor juga dapat diidentifikasi dengan ketukan, tetapi memerlukan keterampilan khusus untuk dapat melakukannya. Tukang ketuk kelapa yang sudah ahli dalam identifikasi buah kopyor disebut
“tukang totok”. Tingkat akurasi penentuan buah kopyornya dapat mencapai 99%
(Sudarsono et al. 2014a). Buah dengan sifat kopyor dihasilkan dari pohon kelapa tertentu yang sebagian besar buahnya mempunyai endosperma normal dan sebagian kecil abnormal (kopyor). Pohon kelapa kopyor hanya mempunyai buah kelapa kopyor dengan frekuensi antara 3-4 buah kopyor per tandan.
Gambar 2.1 Perbedaan fenotipe kelapa normal (kiri) dan kelapa kopyor (kanan)
Melalui serangkaian penelitian yang dilakukan sejak tahun 2005, pada tahun 2010 Balai Penelitian Tanaman Kelapa dan Palma Lain Manado telah berhasil melepas tiga varietas unggul lokal kelapa Genjah kopyor asal Pati, Jawa Tengah dengan potensi persentase rata-rata buah kopyor sekitar 40% pertandannya. Potensi produksi buah kopyor tersebut masih berpeluang untuk ditingkatkan dengan pemuliaan tanaman melalui penyerbukan silang buatan dengan tetua yang memiliki persentase buah kopyor tinggi yaitu dengan serbuk sari dari tanaman hasil kultur embryo yang menghasilkan buah kopyor mencapai 100% (Novarianto dan Miftahorrachman 2000).
Kelapa kopyor ditemukan baik pada kelapa tipe Dalam maupun tipe Genjah, dengan perbanyakan melalui kultur embrio dan menggunakan bibit alami. Pada pengembangan menggunakan bibit alami, kelapa kopyor tipe Dalam hanya menghasilkan buah kopyor antara 10 – 20 %, sedangkan tipe Genjah berpotensi mencapai di atas 50 % pertandannya, dengan tingkat produksi yang beragam. Ini terkait dengan pola pembungaan masing-masing tipe kelapa tersebut. Kelapa tipe Dalam memiliki pola penyerbukan silang, sedangkan tipe Genjah menyerbuk sendiri. Hal tersebut menyebabkan masih rendahnya jumlah buah kopyor yang diperoleh petani, dan berdampak pada rendahnya produksi buah kopyor.
Penanda Genetik
Konservasi dan penggunaan sumber genetik tanaman sangat penting dalam rangka produksi tanaman pertanian dan perkebunan serta pemeliharaan tanaman secara berkesinambungan. Oleh karena itu plasma nutfah merupakan sumber genetik tanaman yang perlu mendapat perhatian, tidak hanya pada tahap pengumpulan dan pemeliharaan tetapi juga bagaimana mengkarakterisasi keanekaragaman genetiknya, mengevaluasi sifat sifat yang dikehendaki dan memanfaatkan untuk pemuliaan tanaman.
tersebut mempunyai kelemahan karena karakter yang diamati kemungkinan dipengaruhi oleh lingkungan. Suatu metode karakterisasi yang dikenal dengan nama penanda molekuler telah dikembangkan untuk pengulangan keterbatasan penanda morfologi.
Pemecahan kendala dalam pemuliaan konvensional mulai mendapat titik terang dengan ditemukannya marka molekuler. Marka molekuler yang pertama kali dikenal adalah marka protein yang secara genetik dikenal sebagai isozim (Amar et al. 2011). Meskipun marka telah banyak digunakan dalam analisis genetik tanaman namun dalam perkembangannya marka isozim masih sangat terbatas jumlahnya. Beberapa sistem enzim tertentu dipengaruhi oleh regulasi perkembangan jaringan, yaitu hanya mengekspresikan suatu sifat pada jaringan tertentu dan pada stadia pertumbuhan tanaman. Kedua faktor tersebut merupakan kendala utama pengunaan marka isozim dalam mengeksploitasi potensi genetik tanaman (Mondini et al. 2009).
Semakin berkembangnya ilmu pengetahuan, maka pada awal tahun 1980-an ditemukan teknologi molekuler yang berbasis pada DNA. Marka molekuler tersebut dapat menutpi kekurangan dari marka isozim, karena jumlah yang tidak terbatas dan dapat melingkupi seluruh genom tanaman, tidak dipengaruhi oleh regulasi perkembangan jaringan, sehingga dapat dideteksi pada seluruh jaringan, dan memiliki kemampuan yang sangat tinggi dalam menganalisis keragaman karakter antar individu. Seiring dengan semakin berkembangnya teknologi yang berbasis marka DNA, maka saat ini telah ditemukan tiga tipe marka DNA dengan segala kelebihan dan kekurangan masing-masing. Semagn et al (2006) menyatakan bahwa penanda molekuler secara garis besar dibagi menjadi tiga kelompok berdasarkan metode deteksinya, yaitu (i) marka berbasis hibridisasi seperti RFLP, (ii) marka berbasis PCR seperti RAPD, AFLP, ISSR, SSR, dan (iii) marka berbasis sekuens DNA seperti SNP.
Teknologi marka DNA berdasarkan teknik PCR dapat bersifat spesifik atau acak sesuai dengan tipe primer yang digunakan (Gupta et al. 2002). Marka DNA hasil amplifikasi primer spesifik adalah marka yang bersifat kodominan. Pengembangan marka-marka kodominan membutuhkan informasi sekuen dari DNA target yang digunakan untuk merancang primer spesifik, umumnya memiliki ukuran panjang 18-24 basa. Marka DNA hasil amplifikasi primer acak (random primer) adalah marka DNA yang bersifat dominan, yaitu tidak dapat membedakan antara genotipe tanaman homozigot dan heterozigot. RAPD (Random Amplified Polymorphic DNA) adalah generasi pertama dari teknologi marka DNA yang bersifat dominan. Generasi kedua dari marka-marka berdasarkan teknik PCR adalah Amplified Fragment Length Polymorphisms (AFLP) yang bersifat dominan dan Simple Sequence Repeats (SSR) yang bersifat kodominan (Panaud et al. 1996).
Primer spesifik dirancang pada runutan basa yang terkonservasi dan selanjutnya digunakan untuk mengamplifikasi dan mengidentifikasi lokus yang polimorfik dengan menggunakan metode standar elektroforesis pada gel poliakrilamida (Jannati et al. 2009). Keunggulan analisis SSR adalah : 1) mengidentifikasi polimorfisme secara akurat, 2) bersifat kodominan, dan 3) sangat reproducible. Kemampuan teknik SSR membedakan individu-individu berdasarkan kombinasi alel, menjadikan teknik ini sering digunakan untuk mengidentifikasi dan menganalisis tetua (Zane et al. 2002) pada berbagai populasi spesies tanaman (Holton et al. 2002). Kelemahan teknologi SSR adalah memerlukan biaya dan curahan waktu yang tinggi untuk mengembangkannya, sehingga penggunaan marka SSR terbatas pada tanaman-tanaman yang memiliki nilai ekonomi tinggi (Ruan 2010).
Teknologi marka SSR telah mendominasi analisis genotyping tanaman sebelum teknologi SNPs dikembangkan. Sejak sepuluh tahun terakhir, teknologi marka SNPs mulai menggantikan teknologi marka SSR pada penelitian-penelitian genetika. Marka SNPs adalah marka berdasarkan variasi perubahan satu basa (A, T, G, atau C) pada situs-situs tertentu dari runutan basa DNA dalam genom organisme (Ganal et al. 2009). Polimorfisme SNP tersedia paling melimpah dan terdistribusi secara merata pada genom organism hidup (Aitken et al. 2009) sehingga metode analisis marka DNA berdasarkan SNP mampu mengidentifikasi variasi keragaman yang lebih tinggi dari metode analisis marka DNA berdasarkan SSR (Li et al. 2009).
SNP (Single nucleotide polymorphism) adalah kelas mutasi yang disebabkan oleh subsitusi atau insersi-delesi (indel) yang umum terjadi di genom organisme hidup dan merupakan unit terkecil dari variasi genetik yang ditransmisikan dari generasi ke generasi. Marka berdasarkan SNPs banyak digunakan pada studi-studi tentang proses evolusi dari suatu genom atau gen, karena umumnya evolusi pada sifat-sifat penting di tanaman adalah atribut dari keberadaan SNPs dan variasinya (Shamay et al. 2004). SNP adalah perubahan posisi spesifik satu atau dua basa nukleotida yang sifatnya melimpah dalam genom eukariot. Perbedaan basa nukleotida diduga berpengaruh terhadap sifat fenotipik pada tiap-tiap individu (McCouch et al. 2010). Jumlah SNP yang melimpah membuat marka SNAP lebih menarik dibanding marka lainnya, termasuk dalam mengembangkan penanda bagi gen target tertentu (Lestari dan Koh 2013). Deteksi marka SNAP yang bersifat ko-dominan, berdasarkan pada amplifikasi PCR dengan primer yang berbasis pada informasi sekuen untuk gen spesifik Marka SNAP saat ini telah digunakan sebagai penanda genetik untuk berbagai fungsi pemuliaan tanaman, misalnya analisis keragaman genetik, pembuatan linkage map dan Marker Assisted Selection (Chen et al. 2011). Kelemahan dari teknik SNAP adalah memerlukan informasi keragaman sekuen untuk suatu gen yang menjadi target analisis (Mammadov et al. 2012).
Persebaran Serbuk Sari
Proses aliran gen dapat terjadi pada tanaman yang memiliki keserasian secara seksual antara tanaman domestik maupun kerabat liarnya (Pandin 2009a). Analisis aliran gen melalui serbuk sari dalam suatu populasi dapat digunakan untuk menduga apakah terjadi perkawinan antara tanaman yang berbeda (outcrossing) atau dengan tanaman yang sama (selfing) (Boer 2007). Hamrick dan Trapnell (2011) mengatakan bahwa pola persebaran biji dapat dianalisis menggunakan dua metode, yaitu :
a. Metode tak langsung meliputi analisis struktur genetik populasi menggunakan marka genetik yang diwariskan secara maternal misalnya menggunakan cpDNA (DNA kloroplas) dan mtDNA (DNA mitokondria) dalam satu populasi.
b. Metode langsung menggambarkan pola persebaran biji menggunakan marka molekuler untuk mengidentifikasi induk dari biji atau analisis parental. Analisis metode langsung dibagi menjadi dua yaitu analisis induk jantan dan betina dari biji dan analisis kecocokan antara induk jantan dengan induk betina terhadap keturunannya.
Sistem perkawinan pada tanaman dapat diketahui melalui analisis pola persebaran serbuk sari. Penelitian Carneiro et al. (2011) menyatakan bahwa tanaman Hymenaea coubaril melakukan penyerbukan sendiri. Hal tersebut bertentangan dengan penelitian sebelumnya oleh Dunphy et al. (2004) yang menyatakan bahwa H. coubaril memiliki ketidaksesuaian secara seksual (self incompability). Penebangan pohon H. coubaril secara bebas dalam areal perhutanan dapat mengakibatkan berkurangnya pohon yang reproduktif. Kondisi tersebut mengakibatkan tanaman terisolasi, sehingga persentase penyerbukan sendiri dapat meningkat (Carneiro et al. 2011).
III. PERSEBARAN SERBUK SARI PADA KELAPA KOPYOR GENJAH PATI MENGINDIKASIKAN PERANAN POLINATOR
SERANGGA DALAM PENYERBUKAN
Abstrak
Analisis parental dapat digunakan untuk mengevaluasi persebaran serbuk sari pada kelapa kopyor. Tujuan penelitian untuk mengevaluasi (i) persebaran serbuk sari, (ii) kisaran penyerbukan sendiri dan penyerbukan silang yang terjadi, dan (iii) jarak serbuk sari ke tetua betina dalam populasi kelapa kopyor Pati. Hal ini sangat penting dilakukan untuk petani dalam peningkatan panen buah kopyor dan juga untuk mendukung pemuliaan kelapa mutan. Sebanyak 84 progeni dipanen dari 15 tetua betina. Kandidat tetua sebanyak 95 pohon dianalisis untuk melihat tetua jantan yang berptoensi untuk seluruh progeni. Provenan kelapa dewasa dipetakan sesuai posisi GPS. Seluruh sampel di identifikasi menggunakan 6 lokus SSR dan 4 lokus SNAP. Analisis parental dilakukan menggunakan CERVUS version 2.0 software. Hasil analisis mengindikasikan marka yang digunakan efektif untuk mengidentifikasi kandidat tetua di seluruh progeni. Tidak ada arah tertentu dari serbuk sari yang didonorkan tetua jantan ke tetua betina. Serbuk sari yang didonorkan berasal tetua jantan dari arah relatif terhadap posisi induk betina. Penyerbukan silang terjadi sebesar 82.1 % dari seluruh progeni yang dianalisis. Penyerbukan silang antara kelapa dalam dengan kelapa dalam (D x D), kelapa genjah dengan kelapa genjah (G x G), kelapa hibrida dengan kelapa hibrida (H x H), D x G, G x D, D x H, G x H dan H x G seluruhnya diobservasi. Penyerbukan sendiri (D x D dan G x G) terjadi sebesar 17,9% dari keseluruhan progeni. Kelapa genjah tidak selamanya mengalami penyerbukan sendiri. Keberadaan penyerbukan silang antara kelapa genjah dengan kelapa genjah, kelapa dalam dengan kelapa genjah dan kelapa hibrida dengan kelapa genjah juga diobservasi. Serbuk sari yang didonasikan dapat berasal dari serbuk sari donor yang berada dalam kisaran jarak tempuh 0-58 meter dari tetua betina resipien yang dievaluasi. Oleh karena itu, selain dengan adanya keberadaan angin, polinator serangga juga dapat berperan penting dalam polinasi kelapa Kopyor.
III. Pollen Dispersal of Pati Kopyor Coconut Indicating Importance Roles Of Insect Pollinator In Its Pollination *)
Abstract
Parentage analysis has been used to evaluate pollen dispersal in Kopyor coconut (Cocos nucifera L.). The objectives of this research were to evaluate (i) the dispersal of pollen, (ii) the rate of self and out-crossing pollination, and (iii) the distance of pollen travel in Pati kopyor coconut population. The finding of this activities should be beneficial to kopyor coconut farmers to increase their kopyor fruit harvest and to support breeding of this unique coconut mutant. As many as 84 progeny arrays were harvested from 15 female parents. As many as 95 adults coconut provenances surrounding the female parents were analyses as the potential male parents for the progenies. The adult coconut provenances were mapped according to their GPS position. All samples were genotyped using six SSR and four SNAP marker loci. Parentage analysis was done using CERVUS version 2.0 software. Results of the analysis indicated the evaluated markers were effective for assigning candidate male parents to all evaluated seedlings. There is no specific direction of donated pollen movement from assigned donor parents to the female ones. The donated pollens could come from assigned male parents in any directions relative to the female parent positions. Cross pollination occured in as many as 82.1% of the progenies analyzed. Outcrossing among tall by tall (TxT), dwarf by dwarf (DxD), hybrid by hybrid (HxH), TxD, DxT, TxH, DxH, and HxD were observed. Self-pollination (TxT and DxD) occurred in as many as 17.9% of the progenies. The dwarf coconut is not always self pollinated. The presence of DxD, TxD, and HxD outcrossing were also observed. The donated pollens could come from pollen donor in a range of at least 0-58 m apart from the evaluated female recipients. Therefore, in addition to the wind, insect pollinators may play an important role in Kopyor coconut pollination.
KEY WORDS : Coconut mutant, abnormal endosperm, parentage analysis, pollen movement, outcrossing rate, self pollination, SSR marker, SNAP marker
---
Introduction
Kopyor coconuts are natural coconut mutants having abnormal endosperm and only exist in Indonesia. The endosperm is soft, crumbly and detached from the shell, forming flakes filling up the shell (Maskromo et al. 2007; Novarianto et al. 2014). The Makapuno coconut grown in the Philipines and other Asian countries is another example of coconut mutant exhibiting endosperm abnormality (Samonthe et al. 1989; Wattanayothin 2010). This mutant has been used as parent for hybridizations in coconut breeding (Wattanayothin 2005). The Macapuno coconut exhibits a soft and jelly-like endosperm (Santos 1999) that is phenotypically different to Indonesian Kopyor coconut.
The kopyor coconut mutant phenotype is genetically inherited from parents to their progenies (Sukendah 2009) and most probably is controlled by a single locus (K locus) regulating the endosperm development of coconut (Sudarsono et al. 2014a). However, the identity of the regulatory locus has not yet been resolved.
The abnormal endosperm phenotype in kopyor coconut is controlled by the recessive k allele; therefore, the genotype of kopyor fruit of coconut would be homozygous kk for the zygotic embryos and homozygous kkk for the endosperm. On the other hand, the genotype of the normal fruit of coconut would either be a homozygous KK or a heterozygous Kk for the zygotic embryo and either a homozygous KKK, heterozygous KKk, or heterozygous Kkk, respectively.
The origin of Kopyor coconut mutant is not well documented; however, currently the kopyor provenances are found in a number of areas in Java and southern part of Sumatera (Novarianto and Miftahorrachman 2000). The district of Pati, Central Java Province is recognized as one of the Kopyor coconut production centers. Kopyor coconuts have existed in this region for generations, especially the dwarf type of Kopyor coconuts. Although only in a limited numbers, Kopyor Tall and Kopyor Hybrid coconut types also exist along side of the dwarf one.
The tall and dwarf coconut have different morphological characters and pollination strategy. Tall coconuts are generally outcrossing since male flower mature earlier than the female counterpart in the same inflorescence. Dwarf coconut tends to self-pollinate because of an overlapping maturation period between male and female flowers (Deb Mandal and Shyamapada 2011).
Pollination in coconut most probably is assisted by insect pollinators or by the wind (Ramirez et. al. 2004). The family of Diptera, Coleoptera and Hymenoptera are reported as effective pollinators of coconut (Ramirez et al. 2004). Distances of pollen transfer between male and female parents may be used to predict the type of pollinator assisting pollination in coconut. Such question may be answerred by studying pollen dispersal.
self-pollination and outcrossing rates in a certain population (Milleron et al. 2012).
To our understanding, pollen dispersal analysis has not been evaluated in coconuts. With the development of kopyor coconut in Indonesia, availability of information associated with pollen dispersal should be beneficial considering the recessive nature of the kopyor character. Such coconut pollen dispersal evaluation requires availability of some coconut progeny arrays and polymorphic loci for molecular markers of coconut genome.
Co-dominant markers, such as SSR and SNAP markers for coconut have been developed and routinely evaluated at PMB Lab, Department of Agronomy and Horticulture, Faculty of Agriculture, Bogor Agricultural University (IPB), Bogor, Indonesia for a number of plant species. These include coconut (Sudarsono et al. 2014), cacao (Kurniasih 2012), and nut meg – Myristica sp. (Soenarsih 2012). Moreover, the gene specific SNAP markers have also been developed and used successfully in coconut (Sudarsono et al. 2014).
The SSR markers have successfully been used in gene flow analysis of pines (Lian et al. 2001; Burczyk and Koralewski. 2005). SNAP marker have also been reported as an effective co-dominant marker for plant analysis (Morin et al. 2004, Sutanto et al. 2013) and proven to generate better data quality for the majority of samples on plant genetic studies (Brumfield et al. 2003) and population structure analysis (Herrera et al. 2007).
The objectives of this research were is to evaluate (i) the dispersal of pollen, (ii) the rate of self and out-crossing pollination, and (iii) the distance of pollen travel in Pati kopyor coconut population. The finding of these activities should be beneficial to kopyor coconut farmers to increase their kopyor fruit yield and to support breeding and cultivar development of this unique mutant.
Materials and Methods
Time and Location of Research
This research was conducted during the period of July 2012 up to January 2014. The field activities were at the Kopyor coconut plantation belonging to local
farmer‟s at Sambiroto, Pati District, Central Java, Indonesia. The research site was at the following GPS location: S 6 32.182 E 11 03.354. The soil in the evaluated Kopyor coconut plantation is sandy soil. The laboratory activities were done at Plant Molecular Biology Laboratory (PMB Lab), Department of Agronomy and Horticulture, Faculty of Agriculture, Bogor Agricultural University, Bogor, Indonesia.
Selection of Parents and Progeny Arrays
Six dwarf, seven tall, and two hybrid coconuts among the kopyor heterozygous Kk trees were selected as female parents. They were selected using purposive random sampling to represent different sites in the sampled population. A single fruit bunch from each female parent containing 2-10 fruits/bunch was harvested 10-11 months after pollination. The total harvested fruits were collected and identified as either kopyor or normal fruits. The identified normal fruits were germinated and DNA was isolated from leaf tissue of the germinated seedlings (63 seedlings of normal fruits). The kopyor fruits are not able to naturally germinate since this character is lethal. Zygotic embryos were isolated from the identified kopyor fruits and DNA was isolated directly from the whole zygotic embryo tissues (21 zygotic embryos). Among the 84 DNA samples, 26 samples were from tall, 45 from dwarf, and 13 from hybrid female parents.
Genotyping of Parents and Progenies
DNA isolation was conducted using the CTAB method (Rohde et al. 1995). Either young coconut leaf or zygotic embryo (0.3-0.4 g) was homogenized in 2 ml of lysis buffer, containing 0.007 g PVP and 10 μl2-mercaptoetanol. The homogenized tissues were then incubated in 65°C waterbath for 60 minutes and the mixtures were centrifuged at 11000 rpm for 10 minutes using using the Eppendorf 5416 centrifuge. Supernatant was then transferred to an Eppendorf tube and an equal volume of chloroform:isoamyl-alcohol (24:1) was added. The mixtures were mixed well; centrifuged at 11000 rpm for 10 minutes and the supernatant was transferred into new microtube.
Cold isopropanol (0.8 volume of supernatant) and sodium acetate (0.1 volume of supernatant) were added into the supernatant. After overnight incubation, the mixture was centrifuged at 11000 rpm for 10 minutes and DNA pellet was retained. The DNA pellet was washed using 500 μl of cold 70% ethanol, centrifuged and air dried before it was diluted into100 μl aquabidest. RNA contaminants were remove using RNase treatment following standard procedures (Sambrook and Russel 2001).
SSR marker at 37 loci (Lebrun et al. 2001) were evaluated for their polymorphism 6 polimorphic loci were selected. In addition, four SNAP marker loci developed based on nucleotide sequence variabilities of both SUS and WRKY genes were also used to genotype all of the parents and progeny arrays. To generate markers, PCR amplifications were conducted using the following reaction mixtures: 2µl of DNA; 0.625 µl of primers, 6.25 µl PCR mix (KAPA Biosystem), and 3 µl ddH20. Amplifications were conducted using the following steps: one cycle of pre-amplification at 95°C for 3 minutes, 35 cycles of amplification steps at 95 °C for 15 seconds (template denaturation), annealing temperature for 15 seconds (primer annealing), and 72 °C for 5 seconds (primer extension), and one cycle of final extention at 72 °C for 10 minutes as suggested by KAPA Biosystem kit.
The generated SNAP markers were separated using 1% agarose gel electrophoresis using TBE 1x buffer and stained using standard DNA staining procedures (Sambrook and Russel 2001). The electrophoregrams were visualized over the UV transluminescence table and recorded using digital camera. The recorded pictures were used to determine the genotype of the evaluated samples.
Identification of the Candidate Male Parents
Each sample of the progeny arrays has a known female parent but unknown pollen donor (the male parent). The candidate male parents could be any one of the sampled adult population including the female parents. This steps were conducted to determine the assigned male parent donating pollen to generate any fruit in the progeny arrays.
Identification of the assigned male parent was done by analyzing genotype of progeny and the respective female parent versus the genotype of all adult trees in the selected samples. The ID of the potential male parent for any progeny was determine based on the results of parentage analysis. Simulation was conducted to determine the threshold level for confidence interval of 80% (relax) and 95% (strict) levels before the final parentage analysis steps. Parentage analysis using the genotype of progenies, female parents, and potential male parents was done using CERVUS version 2.0 software (Marshall et al. 1998). Most likely approach (potential male parent with the highest LOD score) based on the matching genotype of progeny, female parent and potential male parent were used as the basis for assigning certain adult individual as the potential male parent or pollen donor of a progeny. The progeny and female parent genotype were compared with those of other adult trees and the assigned male parent was selected based on the output of CERVUS version 2.0 analysis results (Marshall et al. 1998).
Pattern of Pollen Dispersal
The location of the female and the asignmed male parents were plotted in the map of adult individuals generated by Garmin MapSource GPS mapping software version 76C5x. The distance between the known female parent and the assigned male parent was calculated using the same software. The distances and positions of both female and male parents in the generated map was then used to ilustrate pattern of pollen dispersal in the location. Self pollination was defined if the assigned male parent was the same as the female parent. Otherwise, they were assigned as outcrossing. The outcrossing were further grouped as outcrossing between either dwarf (dwarf parent pollinated other dwarf), tall (tall parent pollinated other tall), or hybrid (hybrid parent pollinated other hybrid); outcrossing between dwarf and either tall or hybrid (either tall or hybrid parent pollinated by dwarf); outcrossing between tall by hybrid coconuts (tall parent pollinated hybrid) or vice versa. The numbers of both self pollination and the respective cross pollination were calculated.
Results and Discussions
The Parents and Progeny Arrays
normal homozygous KK and kopyor heterozygous Kk individuals and a mixture of dwarf, tall and hybrid coconuts. All of these adult trees were used as potential male parents capable of donating pollens to and pollinating the selected female parents and generating the evaluated progeny arrays. The position of the selected female parents (6 dwarf, 7 tall, and 2 hybrid kopyor heterozygous Kk coconuts) are indicated in Figure 3.1. The harvested progenies from selected female parents ranged from 2-10 progenies per female parent. Out of 84 selected progenies, 21 were kopyor nuts and 63 were normal ones. They were harvested from tall (26 progenies), dwarf (45 progenies), and hybrid (13 progenies) female parents, respectively.
Genotyping of Parents and Progeny Arrays
The selected SSR and SNAP marker loci generated polymorphic markers in the evaluated coconut population. An example of the polymorphic marker generated by either the selected SSR (CnCir_56 locus) and SNAP (SUS 1_3 locus) primer pairs producing polimorphic markers is presented in Figure 2. and 3. In Figure 2, the evaluated individuals are either homozygous cc (sample # 1), bb (sample # 7-10), heterozygous bc (sample # 2-6, and 11), or heterozygous ab (sample # 12) for the CnCir_56 SSR locus. On the other hand, the evaluated individuals (sample # 1, 3, 4, 6) are heterozygous for reference and alternate SNAP alleles and the other two (sample # 2 and 5) are homozygous for the reference allele (Figure 3.3). All individuals were genotyped using the same approaches. The summary of genotping results for a total of 179 individuals using six SSR and four SNAP marker loci are presented in Table 3.1. The marker loci generated a range of 2-4 alleles per locus (Table 3.1).
Mean number of alleles per locus is 3.4 and mean PIC for all marker loci was 0.47. The polymorphic information content (PIC) for SSR marker loci ranges from 0.31-0.68 while that of SNAP markers ranges from 0.28-0.37 (Table 3.1). The PIC values represents measures of polimorphism between genotypes in a locus using information of the allele numbers (Sajib et al. 2012). Total exclusionary power using the ten marker loci is either 0.85 (first parent) or 0.97 (second parent), indicating the SSR and SNAP markers should be informative enough for analyzing the evaluated coconut population.
Identification of the Candidate Male Parents
parents. According to Marshall et al. (1998), the higher the LOD value the the higher the possibility the assigned male parent is the actual parent (Marshal et al. 1998).
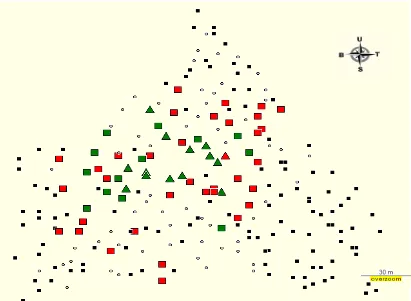
Figure 3.1.Map of study site with the existed coconut provenances at Pati, Central Java, Indonesia. The marks in this map indicated the positions of the coconut provenances. The sampled provenances in an approximately one hectare area are in the square box. The flags indicate the positions of the selected female parents.
Note: The marks indicate the position of ( ) normal tall,( ) kopyor tall, ( ) normal dwarf, ( ) kopyor dwarf, ( ) normal hybrid, and ( ) kopyor hybrid coconuts, respectively.
Figure 3.2. Polymorphism of SSR markers generated by PCR of the genomic DNA sample # 1-12 with a pair of CnCir_56 SSR primers. M: 100 bp DNA ladder markers. The a, b, and c are the three specific alleles of the CnCir_56 locus.
Figure3.3. Polymorphism of SNAP markers generated by PCR of the genomic DNA samples 1-6 with two pairs (the R: reference and the A: alternate primer pairs) of the SUS 1_3 SNAP locus. M: 100 bp DNA ladder markers. The R – PCR product of the R primer pairs and the A – PCR product of the A primer pairs. The occurrences of both R and A PCR products indicating the evaluated individuals are heterozygous, while if either only A or B indicating they were homozygous.
Table 3.1. Numbers of alleles and individuals, numbers of heterozygous and homozygous, observed (O) and expected (E) heterozygosity, and polymorphic information content (PIC) at 10 molecular marker loci of Kopyor coconut.
Locus Name No. of alleles
No. of individual
No. Of Heterozygosity
PIC
Hetero-zygous
Homozy-gous O E
CnCir_87 2 179 27 152 0.15 0.39 0.31
CnCir_86 4 179 100 79 0.56 0.72 0.67
CnZ-18 4 179 74 105 0.41 0.61 0.57
CnZ_51 5 179 72 107 0.40 0.58 0.54
CnCir_B12 6 176 64 112 0.37 0.72 0.68
CnCir_56 5 179 78 101 0.44 0.64 0.57
CnSus1#14 2 179 143 36 0.80 0.49 0.37
CnSus1#3 2 174 147 27 0.85 0.50 0.37
WRKY19#1 2 176 111 65 0.63 0.47 0.36
WRKY 6#3 2 179 76 103 0.43 0.34 0.28
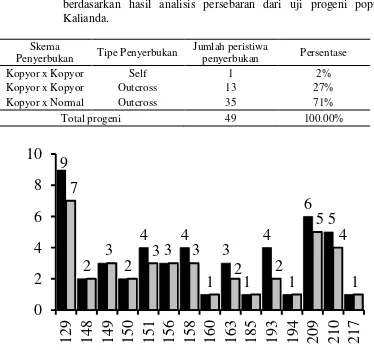
Cross pollination is pollination of female flower by male pollen from different parents. Cross pollination produces half-sib progenies. The tall, dwarf and hybrid coconuts could reciprocally donate their pollens. Based on the assigned male parent of the 84 progeny arrays, cross pollination occured in as many as 69 events (82.1 %). Among those identified as outcrossing, 4 events are cross pollination between tall x tall (TxT), 16 tall by dwarf (TxD), and 4 tall by hybrid (TxH) parents. Moreover, outcrossing among DxD (15 events), DxT (6
1
2
3
4
5
6
M
events), DxH (11 events), HxH (2 event) and HxD coconuts (11 events) were also observed. Complete scheme and pollination types identified based on results of pollen dispersal analysis are presented in Table 3.2.
The general understanding stated that because of the open flower morphology and the differences in flower maturation, tall coconut is probably always cross pollinated (Ramirez et al. 2004; Maskromo et al. 2011). However, our data indicated there are at least 2.38% of self pollination among the tall coconut (Table 3.2).
Self pollination is characterized by the pollination of female flower by male pollen of the same parent. Self pollination produces full-sib progenies. Total numbers of self pollination are observed in as many as 15 events (17.9 %) in the evaluated progeny arrays (Table 3.2). They consist of two self pollination events in the tall kopyor coconut (2.38 %) and 13 self pollination events in the dwarf kopyor one (15.48%). Based on 13 progeny arrays harvested from the hybrid parents, no self pollination in the hybrid coconut is recorded (Table 3.2).
The general understanding stated that because of the overlapping period between male and female flower maturation, dwarf coconut is always self pollinated (Maskromo et al. 2011). However, our data indicated the dwarf coconut is not always self pollinated. Contrary to the basic understanding, our data indicated the presence of more dwarf to dwarf (15 events, 17.86%), dwarf to tall (6 events, 7.14%) and dwarf to hybrid (11 events, 13.1%) outcrossing (Table 3.2). Finding by Rajesh et al. (2008) has previously indicated cross pollination did occur in dwarf coconuts. Availability of new tools, such as molecular markers, for analyzing outcrossing rate may change the previous understanding. Such changes have been shown in Hymenaea coubaril which was previously reported as more cross pollinated because of self incompatibility (Dunphy et al. 2004).
Table 3.2. Crossing schemes and pollination types identified based on results of pollen dispersal analysis of the progeny arrays
Crossing
Scheme Pollination types Event numbers Percentage
TxT Self 2 2.4%
DxD Self 13 15.7%
HxH Self 0 0
Sub-total Self 15 17.9
TxT Outcross 4 4.8%
TxD Outcross 16 19.3%
TxH Outcross 4 4.8%
DxD Outcross 15 18.1%
DxT Outcross 6 7.2%
DxH Outcross 11 13.3%
HxH Outcross 2 2.4%
HxT Outcross 0 0
HxD Outcross 11 13.1%
Sub-total Outcross 69 82.1%
Total progenies 84 100.0%
However, more recent pollen dispersal studies indicated that H. coubaril is more self pollinated (Carneiro et al. 2011).
Other alternative explanation for this findings is it is just a special case in the evaluated site. In the study site, coconut provenances were planted in high density planting. Moreover, population of honey bees exist in the coconut plantation. Honey bees are known to roam around the male and female flowers and function as effective pollinators for coconuts. The high density planting and the availability of pollinators may have caused the unexpected outcrossing rate. However, those are subjects of further investigations.
One assigned male parent may donate one or more pollens to the evaluated female coconut parent, with a range of 1-5 pollens per assigned male one. Number of assigned male parents donating certain numbers of pollen to the evaluated female parents is presented in Figure 3.4. The data indicate that most of the assigned male parents contribute only one pollen to the evaluated female parents. Only three assigned male parents (two dwarf, and one hybrid coconuts) donated 4 or 5 pollens to the surrounding female parents.
The same female parents may receive donated pollens from different numbers of assigned male parents, with a ranged of 1-7 assigned male parents donated pollen to the same female one. The numbers female parents receiving donated pollens from different number of assigned male parent iss presented in Figure 3.5. The data indicated a single female parent most frequently received pollens from 2, 4 or 5 different assigned male parents. Only three female parents evaluated in this experiment (two dwarf and one hybrid coconuts) are found receiving pollens from at least 6 assigned male parents (Figure 3.5).
Figure 3.4. Numbers of assigned male parent donating different numbers of
pollen to evaluated female parents.
N
um
ber
of
m
ale
pa
rent
Number of donated pollen
27
11
7
2
1
0
10
20
30
40
Figure 3.5. Numbers of female parent receiving donated pollens from different
number of assigned male parents.
Pattern of Pollen Dispersal
The distances between female to the assigned male parents have been determined based on their GPS positions. The distance of pollen travel between assigned male to female parents as measured in this evaluation ranged from 0 - 58 m. Numbers of pollination events of each distance class from the assigned male to the female coconut parents are presented in Figure 3.6. The assigned male parents are distributed almost evenly in the different class distances from the female parents. The 0 m distance between parents indicates self pollination events.
Figure 3.6. Numbers of pollination events for each distance class from the assigned male to the female coconut parents.
Number of assigned male parents
N
um
ber
of
f
em
ale
parent
1
3
1
4
3
1
2
0
2
4
6
1
2
3
4
5
6
7
N
um
ber
of
e
v
ent
s
Class distance between male to female (m)
15
6
13
18
12
12
7
0
10
20
To evaluate pattern of pollen dispersal among the assigned male parent to the female, the positions of assigned male parents as pollen donors to one female parent are plotted to a map using their GPS positions. Representative samples of the assigned male parent positions to a single female recipient parent are presented in Figures 3.7-3.11.
As the female parent, Hybrid kopyor # 059 (Figure 3.7) received 6 donated pollens from six different assigned pollen donors. The pollen contributors to the progeny array harvested from Hybrid kopyor # 059 female parent were all kopyor heterozygous Kk coconuts. However, the seven progenies harvested from this female parent were all phenotypically normal, i.e. genetically either a normal heterozygous Kk or homozygous KK. The positions of the assigned male parents relative to the female parent # 059 in the study site were presented in Figure 3.7.
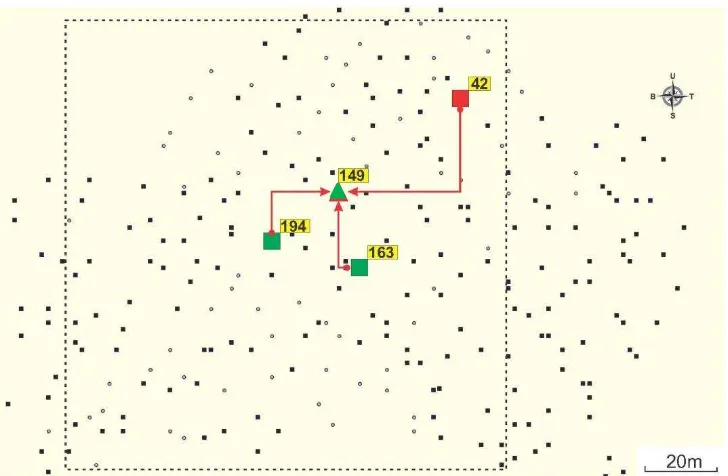
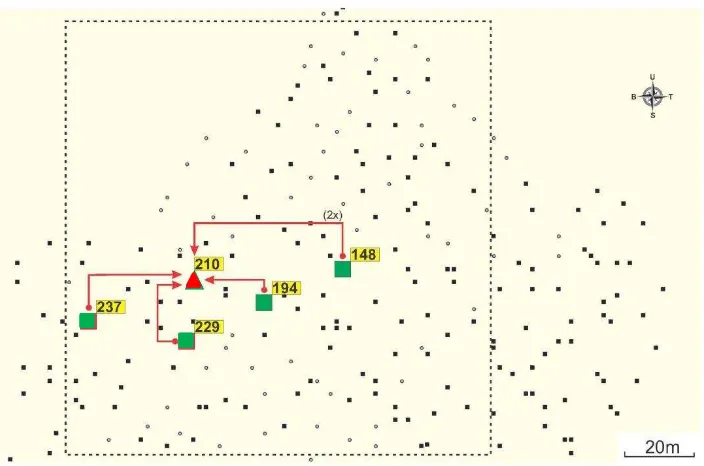
The Dwarf kopyor # 067 (Figure 3.8) received 10 donated pollens from eight different assigned male parents. The assigned pollen contributors to the Dwarf kopyor # 067 female parent were all kopyor heterozygous Kk coconuts. Only one out of the 10 progenies harvested from this female parent was phenotypically kopyor. The assigned male parent for the harvested kopyor fruit was the tall kopyor # 089. The positions of the assigned male parents relative to the female parent # 067 were presented in Figure 3.8.
Figure 3.8. Pattern of pollen movement to female parent # 067 inferred from parentage analysis. The marks indicate position of ( ) Dwaf kopyor, ( ) Hybrid kopyor, ( ) Tall kopyor as the assigned male (pollen donor), and ( ) Dwarf kopyor # 067 as the donor pollens and female recipient, respectively.
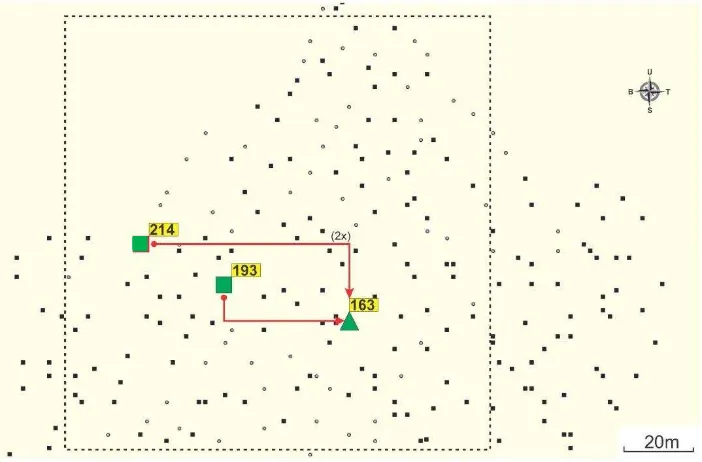
Dwarf kopyor # 068 (Figure 3.9) received 9 donated pollens from four assigned male parents. The four progenies were the result of outcross with either hybrid (# 59) or dwarf (#87 or # 90) and from self pollination. The assigned pollen contributors to the Dwarf kopyor # 068 were all kopyor heterozygous Kk coconuts. Three out of the 9 progenies harvested from Dwarf kopyor # 069 were phenotypically kopyor. These kopyor fruits received one donated pollen from either the tall kopyor # 059, dwarf kopyor # 68 or # 87. The positions of the assigned male parents relative to the female parent # 68 were presented in Figure 3.9.
Figure 3.9. Pattern of pollen movement to female parent # 068 inferred from parentage analysis. The marks indicate position of ( ) Dwaf kopyor and ( ) Hybrid kopyor as the assigned male parents (pollen donors), and ( ) Dwarf kopyor # 068 as the donor pollens and female recipient, respectively.
Dwarf kopyor # 089 (Figure 3.11) received 7 donated pollens from surrounding pollen donors. The pollen contributors to the Dwarf kopyor # 089 female parent were all kopyor coconuts. None of the 7 progenies harvested from Dwarf kopyor # 089 was phenotypically kopyor. The positions of the assigned male parents relative to the female parent # 089 were presented in Figure 3.11.
Figures 3.7-3.11 indicated there is no specific direction of donated pollen movement from assigned male parents to the female parents. The donated pollen could come from assigned male parents in any directions relative to the female parent positions. The other positions of the assigned male parents were presented in Figure attachment 5-14.
In the reseach location, wind blows from left to right during the night and from right to left during the day. If the wind is the major pollinators, there should be a specific pattern of pollen movement. Moreover, the distance of pollen dispersals should be close to the pollen donors. Our data did not support the wind as the only major pollinator in Kopyor coconut since pollens disperse in random directions and the assigned male parents are as far as 58 m apart from the evaluated female recipients. Our data also indicated that insect pollinators may play an important role in Kopyor coconut pollination. Numbers of insects are associated with inflorescence of kopyor coconuts. Such insects may aid pollination and promote cross pollination in kopyor coconuts, as it happens to other plant species (Bown 1988). These findings, however, do not rule out the role of wind in the Kopyor coconut pollination, especially from closely spacing male pollen donors.
This might have been the first report of using molecular marker to study pollen dispersal in coconut. Results of this study point to new finding about pollen dispersal and pollination, selfing and out-crossing rates among dwarf, hybrid, and tall coconuts, respectively. However, to generalize the finding will require more further research and evaluation since these findings may be only specific for the current study site.
Conclusion
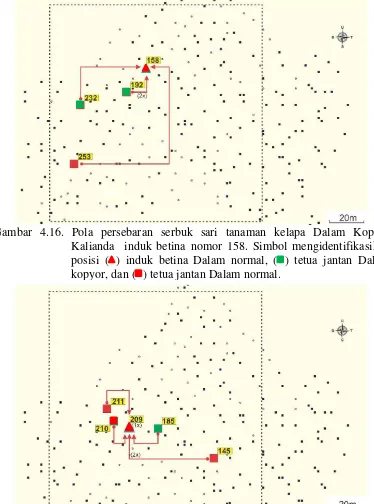
IV. PERSEBARAN SERBUK SARI PADA KELAPA DALAM KALIANDA MEMBUKTIKAN ADANYA PENYERBUKAN SILANG PADA KELAPA
DALAM
Abstrak
Analisis paternitas digunakan untuk mengetahui pola persebaran serbuk sari pada kelapa kopyor (Cocos nucifera L.) Dalam Kalianda. Tujuan spesifik penelitian ini untuk mengevaluasi (i) persebaran serbuk sari serta (ii) kisaran besarnya penyerbukan sendiri dan penyerbukan silang pada Kelapa Dalam Kopyor Kalianda, asal Kalianda, Lampung. Populasi yang digunakan dalam penelitian ini terdiri atas 60 pohon kelapa dewasa yang semuanya tipe kelapa Dalam. Dari 60 pohon kelapa dewasa tersebut dipilih 14 pohon dewasa yang dijadikan sebagai induk betina. Sebanyak 49 progeni dipanen dari 14 induk betina terpilih dan dikecambahkan untuk sumber DNA dalam analisis paternitas. Calon tetua jantan terdiri atas 47 pohon kelapa berbuah normal (homozigot KK) dan 13 pohon kelapa berbuah kopyor (heterozigot Kk) sebanyak 13 pohon. Enam lokus marker SSR polimorfik yaitu CnCir_B12, CnCir_86, CnCir_87, CnCir_56, CnZ_51, CnZ_18 dan empat lokus marker SNAP polimorfik yaitu CNSUS1#14, CNSUS1#3, WRKY6#3 dan WRKY19#1 digunakan untuk menentukan genotipe seluruh progeni, seluruh kandidat tetua jantan, dan semua tetua betina yang digunakan. Hasil percobaan menunjukkan serbuk sari kelapa kopyor Dalam Kalianda menyebar dengan jarak sejauh 63 m. Jarak penyebaran serbuk sari terbanyak yang teramati adalah pada jarak 40-50 m, dengan frekuensi sebesar 13 kejadian polinasi. Diantara 47 progeni yang dievaluasi, hanya satu (2%) progeni yang berasal dari penyerbukan sendiri (self pollination) dan 48 (98%) berasal dari penyerbukan silang. Hasil penelitian juga menunjukkan 13 progeni (27%) berasal dari penyerbukan silang antar tetua yang sama-sama kopyor heterosigot Kk dan 35 (71%) progeni berasal dari penyerbukan silang antara tetua kopyor heterosigot Kk dengan donor serbuk sari yang berasal dari kelapa normal homosigot KK.
IV. POLLEN DISPERSAL IN KALIANDA TALL COCONUT PROVED OUTCROSSING NATURE OF TALL COCONUT POLLINATION
Abstract
Paternity analysis is used to determine the pattern of pollen spread among kopyor coconut (Cocos nucifera L.) in Kalianda, Lampung. The specific objective of this study are to evaluate (i) the spread of pollen (ii) the magnitude of the range of self-pollination and cross-pollination in Kalianda Tall Kopyor coconut at Kalianda, Lampung. The population used in this study consisted of 60 adult palm trees, mixtures of heterozygous kopyor (Kk) and homozygous normal (KK) coconut trees. Progeny arrays (47 nuts) were harvested from 14 kopyor heterozygous Kk female parents and the progeny were germinated. As many as 60 adult trees surrounding the identified female parents were selected as potential male parents, consisted of 47 normal homozygous KK palm and 13 kopyor heterozygous Kk parents. Six polymorphic SSR marker loci used were CnCir_B12, CnCir_86, CnCir_87, CnCir_56, CnZ_51, CnZ_18 and the four polymorphic SNAP markers used were CNSUS1 # 14, CNSUS1 # 3, WRKY6#1 and WRKY19 # 3. The markers were used to genotype all the progeny, the potential male and the female parents. Results of the experiment indicated pollen of Kalianda Tall Kopyor coconut disperse as far as 63 m. The most frequent pollen dispersal distance are between 40-50 m with the frequency of 13 pollination events. Among the evaluated progenies, only one (2%) comes from self pollination event and 48 (98%) comes from cross pollination ones. Results of the progeny evaluation also indicated that 13 progenies (27%) are the results of outcrossing among kopyor heterozygous Kk parents and 35 events (71%) are from outcrossing among kopyor heterozygous Kk parents and the normal homozygous KK assigned male parents.
Pendahuluan
Kelapa kopyor merupakan komoditas andalan yang bernilai ekonomi tinggi dan dicirikan oleh daging buah yang bertekstur gembur dan sebagian besar tidak melekat di tempurungnya serta rasa yang gurih pada buah yang muda. Di Filipina, jenis kelapa ini disebut makapuno,di Sri Lanka dan Thailand disebut dikiri. Buah kopyor ini diduga berasal dari tanaman kelapa yang mengalami mutasi genetik secara alamiah. Kelapa berbuah kopyor adalah mutan kelapa yang ditemukan di antara populasi kelapa normal. Buah kelapa kopyor dapat dipasarkan dalam bentuk segar dan siap saji maupun melalui pengolahan lebih dahulu. Di Indonesia, pemanfaatan kelapa kopyor lebih ditujukan untuk kebutuhan konsumsi bahan pangan berupa es kopyor, es krim kopyor, koktil, selei kopyor dan bahan campuran kue (Sudarsono et al. 2012) .
Hasil survei yang dilaksanakan Balitka pada tahun 2006 menunjukkan bahwa kelapa kopyor terdiri atas dua tipe, yaitu tipe Dalam dan tipe Genjah. Tipe Dalam terdapat di Kalianda (Lampung Selatan), Ciomas (Bogor), Sumenep dan Jombang (Jawa Timur) dan Pati (Jawa Tengah). Pertanaman kelapa kopyor yang ditanam secara populasi dijumpai di Dukuhseti, Kabupaten Pati (Jawa Tengah), Sumenep (Jawa Timur), Ciomas (Bogor, Jawa Barat), Kalianda (Lampung Selatan), Riau dan Kalimantan Timur. Kelapa kopyor yang ditanam secara individu terdapat di Kabupaten Pati, Jombang dan Sumenep (Sudarsono et al. 2014a). Secara alami, tanaman kelapa kopyor tipe Dalam hanya menghasilkan buah kopyor 1-2 butir per tandan. Hal ini disebabkan kelapa tipe Dalam termasuk tanaman menyerbuk silang sehingga peluang bertemunya gen resisif pada bunga betina dan serbuk sari relatif kecil. Kelapa kopyor tipe Genjah menghasilkan buah kopyor per tandan lebih banyak dari tipe Dalam, kadang-kadang dapat mencapai 50%.
Kabupaten Lampung merupakan salah satu sentra kelapa di provinsi Lampung, dengan total areal pertanaman pada tahun 2010 seluas 34.730 ha dan produksi 30 435.60 ton. Luas areal pertanaman kelapa tersebut terus menurun seiring dengan alih fungsi lahan untuk pengembangan komoditi lainnya dan untuk perluasan areal pemukiman. Pada pertanaman kelapa untuk produksi kopra maupun pemanfaatan lainnya ternyata terdapat kelapa Dalam kopyor dengan luasan dan jumlah tegakan yang bervariasi. Kondisi pertanamannya dalam bentuk populasi dan individu tanaman yang menyebar di area kebun kelapa beberapa petani setempat. Hingga saat ini asal usul tanamannya belum diketahui. Umumnya tanaman kelapa Dalam kopyor di Lampung berasal dari warisan orang tua atau pengembangan dari tanaman kelapa Dalam kopyor yang diperoleh dari relasi petani setempat (Sudarsono et al. 2014b).
individu pohon yang sama. Walaupun bunga jantan dan betina ada pada individu pohon yang sama, tetapi bunga jantan dan betina tersebut biasanya mekar pada waktu yang berbeda.
Persebaran serbuk sari pada berbagai spesies tanaman kini telah banyak diteliti contohnya pada tanaman pinus (Schuster dan Mitoon 2000) dan Quercus garryana famili Fagaceae (Marsico et al. 2009). Dengan mengidentifikasi semua sumber serbuk sari yang potensial dalam suatu wilayah studi, penanda dari tetua secara langsung dapat digunakan untuk mengidentifikasi tetua jantan dari progeni yang dianalisis, dan dengan demikian memberikan wawasan tentang bagaimana sifat-sifat individu yang diberikan oleh tetua jantan dan menduga kisaran peluang tanaman menyerbuk sendiri atau menyerbuk silang dalam suatu populasi (Milleron et al. 2012). Untuk tanaman kelapa belum pernah dilaporkan mengenai pola persebaran serbuk sarinya secara spesifik.
Untuk mengembangkan pertanaman kelapa Dalam kopyor di Kabupaten Lampung Selatan, maka dianggap perlu untuk melakukan analisis persebaran serbuk sari pada kelapa Dalam Kalianda sebagai sumber informasi berbagai tipe penyerbukan tanaman kelapa Dalam. Adapun tujuan spesifik penelitian ini untuk mengevaluasi (i) persebaran serbuk sari (ii) kisaran besarnya penyerbukan sendiri dan penyerbukan silang pada Kelapa Dalam Kalianda Lampung.
BAHAN DAN METODE
Waktu dan Lokasi Penelitian
Penelitian dilaksanakan mulai bulan Juli 2013 hingga Agustus 2014. Kegiatan lapang dilakukan di perkebunan kelapa kopyor di Agom Jaya, Kalianda Kabupaten Lampung Selatan, Lampung dengan lokasi GPS S5 39.462 E105 34.962. Tipe tanah yang dievaluasi pada perkebunan kelapa kopyor merupakan tipe tanah berpasir. Kegiatan laboratorium dilakukan di Laboratorium Biologi Molekuler Tanaman (PMB Lab) Departemen Agronomi dan Hortikultura Fakultas Pertanian Institut Pertanian Bogor.
Pemilihan Tetua dan Pemanenan Progeni
Populasi terdiri atas 282 pohon. Populasi yang dipilih berdasarkan jumlah tanaman yang lebih dari 10 pohon kopyor. Populasi tersebut merupakan campuran dari pohon kelapa kopyor heterozigot (Kk) dengan pohon kelapa normal (KK) atau kelapa yang tidak pernah menghasilkan buah kopyor. Berdasarkan tipe kelapa tersebut, populasi yang digunakan dalam penelitian ini terdiri dari 60 pohon kelapa dewasa yang semuanya tipe kelapa Dalam, dari 60 pohon tersebut dipilih 14 pohon yang dijadikan sebagai kandidat induk betina. Lebih lanjut berdasarkan fenotipenya, pohon kelapa berbuah normal (homozigot KK) sebanyak 47 pohon dan pohon kelapa berbuah kopyor (heterozigot Kk) sebanyak 13 pohon. Keberadaan setiap pohon kelapa di perkebunan ditunjukkan dengan menggunakan pemetaan GPS.
Genotyping tetua dan progeni
Isolasi DNA dilaksanakan menggunakan metode CTAB (Rohde et al. 1995) dengan modifikasi. Daun kelapa muda maupun embrio kelapa (0.3-0.4 g) digerus dengan buffer lisis 2 ml, yang mengandung PVP 0.007 g dan 2-mercaptoetanol
sebanyak 10 μl. Hasil gerusan daun atau embrio diinkubasi dalam waterbath pada suhu 65 °C selama 60 menit dan dihomogenkan menggunakan sentrifugasi Eppendorf Centrifuge 5416 dengan kecepatan 11000 rpm selama 10 menit. Supernatan dipindahkan ke tube eppendorf kemudian ditambahkan kloroform:isoamil-alkohol (24:1) sebanyak volume supernatant lalu disentrifugasi dengan kecepatan 11000 rpm selama 10 menit dan dipindahkan ke mikrotube baru.
Supernatan dipindah ke tube eppendorf baru kemudian ditambahkan isopropanol dingin (0.8 volume dari supernatan) dan sodium asetat (0.1 volume supernatant). Setelah diinkubasi dalam freezer semalaman,suspensi kemudian disentrifugasi dengan kecepatan 11000 rpm selama 10 menit hingga diperoleh
pellet DNA. Pellet DNA dicuci dengan menggunakan ethanol 70% dingin 500 μl.
lalu disentrifugasi dan dikeringkan. Pellet DNA diencerkan menggunakan aquabidest sebagai suspensi DNA. Kontaminan RNA dihilangkan dengan menggunakan RNase sesuai standar prosedur (Sambrook dan Russel 2001).
Primer SSR yang diseleksi dari 36 primer (Lebrun et al. 2001) (Lampiran 1) diperoleh 6 primer yang polimorfik. Sebagai tambahan, empat lokus marka SNAP berdasarkan keragaman sekuens nukleotida (gen SUS dan WRKY) (Lampiran 2) juga digunakan untuk menganalisis seluruh tetua dan progeni. Amplifikasi PCR dilakukan dengan menggunakan 2µl DNA working solution, 0.625 µl primer, 6.25 pcr mix (KAPA Biosystem), dan 3 µl ddH2O untuk setiap reaksi. Amplifikasi dilakukan sebanyak 35 siklus, yang dimulai pada suhu 95 °C selama 3 menit sebagai denaturasi awal, diikuti 95 °C selama 15 detik sebagai denaturasi siklus pertama, penempelan primer spesifik - suhu disesuaikan dengan masing-masing pasangan primer - selama 15 detik, pemanjangan primer pada suhu 72 °C selama 5 detik. Final extention 72 °C selama 10 menit sesuai rekomendasi kit KAPA Biosystem.
Produk amplifikasi PCR untuk primer SSR dipisahkan dengan elektroforesis gel poliakrilamid 6% menggunakan Buffer SB 1x (Brody dan Kern 2004) dan pewarnaan gel dengan pewarna silver. Pewarnaan silver mengikuti metode Creste et al. (2001) dengan modifikasi. Elektroforegram di visualisasi diatas meja UV transluminesen di foto menggunakan kamera digital. Pemberian skor setiap genotipe dilakukan juga di atas light table.
Produk amplifikasi PCR untuk primer SNAP dipisahkan menggunakan gel agarose 1% menggunakan buffer TBE 1x dan pewarnaan menggunakan gel untuk melihat hasil skoring yang dihasilkan (Sambrook and Russel 2001). Elektroforegram di visualisasi diatas meja UV transluminesen di foto menggunakan kamera digital. Pemberian skor setiap genotipe dilakukan untuk setiap sampel.
Identifikasi Kandidat Tetua Jantan
ini dilakukan untuk memprediksi calon pendonor serbuk sari diantara calon tetua jantan yang dievaluasi di setiap progeni.
Identifikasi donor serbuk sari dilakukan dengan menganalisis genotipe progeni versus seluruh induk jantan yang dievaluasi. Semua induk jantan atau induk betinanya dievaluasi dan berpotensi sebagai donor serbuk sari. Analisis molekuler menggunakan software CERVUS analysis parentage program komputer Cervus 2.0 (Marshall 1998). Hasil Cervus diperoleh data alel frekuensi, PIC, heterozigositas, homozigositas. Dilanjutkan dengan data simulasi dan data analisis tetua. Data analisis tetua akan diperoleh kandidat tetua dengan lambang (*): artinya tingkat kepercayaan 95%. (+): tingkat kepercayaan 80%. dan (-): tingkat kepercayaan < 80% .
Analisis Pola Persebaran Serbuk Sari
Lokasi induk betina dan tetua jantan terpilih hasil analisis Cervus diplotkan menggunakan perangkat lunak pemetaan Garmin MapSource GPS versi 76C5x. Jarak antara induk betina dan tetua jantan dihitung menggunakan software yang sama. Jarak dan posisi dari kedua induk betina dan tetua jantan yang dihasilkan kemudian digunakan untuk mengilustrasikan pola persebaran serbuk sari di lokasi penelitian. Penyerbukan sendiri didefinisikan jika induk jantan yang teridentifikasi sama dengan tetua betinanya. Selain itu semuanya didefinisikan sebagai penyerbukan silang. Penyerbukan silang bisa dikelompokkan dengan kategori penyerbukan silang genjah (induk betina genjah diserbuki oleh tetua jantan genjah lainnya), penyerbukan silang dalam (induk betina dalam diserbuki oleh tetua jantan dalam lainnya), penyerbukan silang hibrida ((induk betina hibrida diserbuki o